- 研究
- 开放获取
- 发表:
通过对胶乳转录组的比较分析,揭示了胶乳分子量变化的潜在机制橡胶树取代巴西橡胶树克隆RRIM600和Reyan7-33-97
BMC植物生物学体积21文章编号:244(2021)
摘要
背景
天然橡胶的加工性能和力学性能很大程度上取决于其分子量和分子量分布。然而,橡胶生物合成过程中分子量调控的机制尚不清楚。
结果
在本研究中,我们确定了来自1岁的圣母树和30岁的定期敲击了乳胶的MW和粒度橡胶树克隆Reyan7-33–97和RRIM600。结果表明,两个无性系的乳胶分子量和乳胶粒径均不同,并随树龄的增加而增加。来自RRIM600树的乳胶的平均粒径比来自相同年龄的Reyan7-33–97树的乳胶的平均粒径小。在1年生树木中,Reyan7-33–97乳胶的分子量略高于RRIM600,而在30年生树木中,RRIM600乳胶的分子量显著高于Reyan7-33–97乳胶。转录组图谱的比较分析表明,平均橡胶粒径与橡胶粒相关蛋白的表达水平呈负相关,胶乳的高分子量特性与异戊烯基焦磷酸(IPP)单体生成途径基因和下游烯丙基二磷酸(APP)引发剂消耗非橡胶途径的表达增强密切相关。通过生物信息学分析,我们进一步确定了一组可能调控IPP生物合成的转录因子。
结论
综上所述,我们的研究结果揭示了IPP产生途径和非橡胶类异戊二烯途径中基因表达的变化可能的调控机制,这些变化影响IPP和APP启动子的比例和含量,从而导致同龄树木橡胶MW显著变化橡胶树克隆Reyan7-33-97和RRIM600。我们的研究结果为进一步了解橡胶的生物合成提供了依据,并为橡胶品质的遗传改良奠定了基础h .取代巴西橡胶树.
背景
天然橡胶(NR)是制造数千种工业产品不可缺少的原材料。巴西橡胶树(橡胶树取代巴西橡胶树)是目前唯一确定的商业天然橡胶来源,因其生产的橡胶具有优异的物理性能[1]。在橡胶树以焦磷酸异戊酯(IPP)为单体亚基生物合成NR分子,IPP主要由acetyl-CoA [2,3.]。在胞质中,IPP最初被IPP异构酶转化为其异构体二甲基烯丙基二磷酸(DMAPP),随后与IPP在胞质中缩合反式-结构形成香叶基焦磷酸盐(GPP)、法尼基焦磷酸盐(FPP)和香叶基香叶基焦磷酸盐(GGPP) [4]。与这些反式-短链戊烯基焦磷酸盐作为烯丙基引物底物,橡胶分子通过IPP的顺序缩合而生物合成独联体-通过碳阳离子反应的构型[3.,5]。酶橡胶转移酶(RT-ASE)催化在含型乳溶胶细胞的细胞溶胶中的单层膜包围的专用细胞石状结构的表面的这种反应,其称为橡胶颗粒(RP),以及所得的疏水性橡胶链在这些小球的内部分区化[6,7,8]。值得注意的是,在橡胶树在树木中,相邻的乳管细胞相互吻合,在树皮组织内形成广泛的乳管网络,橡胶颗粒相互排斥,以避免在称为乳胶的水悬浮体中融合[9,10]。结果,可以从中收获原料聚合物橡胶树通过在树皮中进行切口并收集乳胶自由流出船只来进行树木[5]。通过这种方式,以高效、可持续和环境友好的方式生产大量NR,供大规模商业使用。
天然橡胶的分子量和分子量分布(MWD)对橡胶制品的加工性能和力学性能有很大的影响[1]。分子量较高的NR具有较好的力学性能,如较高的抗拉强度、撕裂强度、回弹性和耐磨性[11,12]。研究发现,与MWD较窄的NR相比,MWD较宽的NR具有更快的炭黑掺入时间,即更好的加工性能[13,14]。在体外橡胶合成测定中,橡胶分子引发和聚合的速率和最终聚合物分子量大大依赖于底物浓度,IPP和初始烯丙基分子的比例,以及橡胶转移酶的内在性质[7,15]。一些金属离子也被报道为将底物与活性位点结合的必要辅助因子,从而调节体外产生的橡胶的起始速率、生物合成速率和分子量[16]。因此,Cherian等人[3.提示乳汁管环境可能在分子质量调控中起重要作用。此外,胶粒粒径是影响胶乳分子量的一个重要参数橡胶树树。小橡胶颗粒由高分子量橡胶组成,本质上比大橡胶颗粒具有更高的橡胶转移酶活性[17,18,19,20.]。而在这两种橡胶颗粒之间分布明显的橡胶颗粒蛋白,如小橡胶颗粒蛋白(SRPP)和橡胶伸长因子(REF),可能参与了聚合物链伸长和成熟橡胶分子质量的调控[21,22]。同样值得注意的是,橡胶的MW和MWD是密切相关的橡胶树克隆和年龄。老橡胶树的橡胶当量增加[23],而在同一橡胶试验中生长的多个无性系的新鲜乳胶在分子量和MWD上有很大的差异[24,25]。这些实验结果表明,橡胶MW由多种因素控制,包括含型乳草环境,橡胶粒子类型,遗传和发育阶段。
随着当前新一代测序技术的发展,一些基因组、转录组和蛋白质组的研究得到了广泛的应用h .取代巴西橡胶树克隆和其他橡胶生产品种已被报道[26,27,28,29,30.,31]。然而,这些研究主要集中在橡胶生物合成、乳胶流动、耐受力、乳汁管细胞发育和攻丝板干燥(TPD)等过程中涉及的关键基因的鉴定。对橡胶分子量调控机制的研究较少。基于上述的无性系和年龄相关差异橡胶树橡胶的分子量和相对分子质量大小以及不同来源乳胶的转录组图谱进行比较分析是值得的橡胶树并将分子量和MWD的变化与基因表达联系起来,为了解橡胶生物合成过程中分子量的调控机制提供了可能。
RRIM600是一个橡胶树源自TJIR1之间杂交的克隆 × PB86位于马来西亚,是世界上种植最广泛的橡胶克隆之一[32]. RRIM600是一个平均产乳量为 ~ 每年每公顷1350公斤乳胶,而它的杂交后代Reyan7-33–97是一个高产克隆(~ 1959公斤/公顷/年),具有较强的抗风害和低温能力,是中国最受欢迎的橡胶树无性系[2,33,34]。在我们目前的研究中,我们研究了1岁的圣母树和30岁的克隆雷森7-33-97和RRIM600的30岁的乳胶乳胶乳胶粒度和分布。通过对乳胶转录瘤的比较分析,我们研究了橡胶生物合成相关途径,橡胶颗粒相关蛋白和转录因子的表达,这些因子可能涉及胶乳中克隆差异的形成。我们的结果丰富了公开橡胶树转录组资源和在天然橡胶生物合成中的分子量调节机制的见解,也可能加速橡胶树的遗传改善以生产高质量的橡胶。
结果
胶乳分子量与橡胶粒径的比较分析
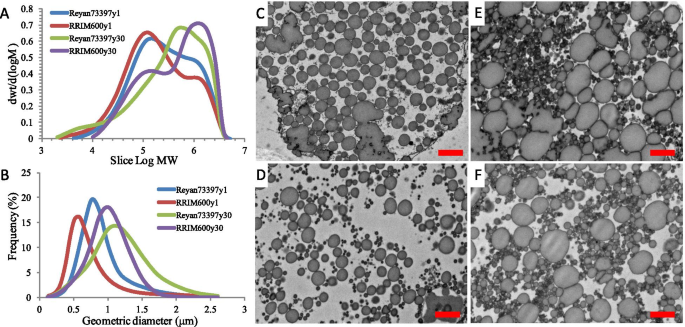
克隆热yan7-33 - 97和RRIM600的1年生未成熟树和30年定期抽丝树的样本分别命名为热yan73397y1、RRIM600y1、热yan73397y30和RRIM600y30。用GPC测定胶乳样品的数均分子量(MN)、分子量(MW)和MWD (MW/MN),如表所示1和无花果。1.作为年龄橡胶树树木从1到30年增加,乳胶乳胶的MW从4.77×10增加5到6.51 × 105, MWD也从5.53增加到7.19,而RRIM600树胶乳的MW和MN从4.26 × 10显著增加5至8.28 × 105和7.18 × 104至17.73 × 104分别略微减少MWD(从5.94到4.67)。在同一年龄的树木中,REYAN73397Y1乳胶显示出比RRIM600Y1略高的MW,而RRIM600Y30 LATEX在同一试验中的REYAN73397Y30的MW显着更高(表1).如图所示。1a,这些乳胶样品的MW范围从2.0 × 103.5.0×106并包含高分子量级分(> 106)和低分子量馏分(< 105).热延7-33 - 97 1年和30年树龄的乳胶树的乳液呈偏态单峰分布,在高分子量区域有一个肩,而RRIM600树的乳液呈典型的双峰分布(图2)。1一个)。随着树龄的增加,REYAN7-33-97和RRIM600树的乳胶表现出高分子量级分的显着增加。在1岁的树上,来自REYAN73397Y1的乳胶有比RRIM600Y1更高的MW橡胶。在30岁的树上,Reyan73397Y30的乳胶包含更多低MW橡胶(<2.0×104),高MW峰的位置比RRIM600y30乳液的位置要小(图6)。1一个)。
用激光光散射(LLS)对新鲜乳胶样品进行分析,以确定粒径,这是影响NR分子量的一个潜在因素1和无花果。1b).结果表明,随着年龄和无性系的不同,RPs的几何平均直径也不同。1岁树的乳胶主要粒径小于1 μm, 30岁树的乳胶主要粒径在0.5 ~ 2 μm之间。平均而言,RRIM600树的乳胶中RP的大小比同年龄的Reyan7-33-97树的要小。超薄截面的透射电子显微镜(TEM)图像显示,树皮乳汁管内橡胶颗粒以球形为主。如图所示。1C-f, the size of the RPs correlates well with the LLS measurements: the latex from 30-year-old trees has larger RPs than that from 1-year-old trees, and the RPs in RRIM600 latex tend to be smaller than those in Reyan7-33–97 latex. In both laticifers of Reyan7-33–97 at ages 1 and 30, we observed that some elongated particles appeared to result from aggregation, while in the analyzed sections of RRIM600 at ages 1 and 30, the rubber particles are uniformly dispersed.
转录组测序和差异表达基因的分析(DEGS)
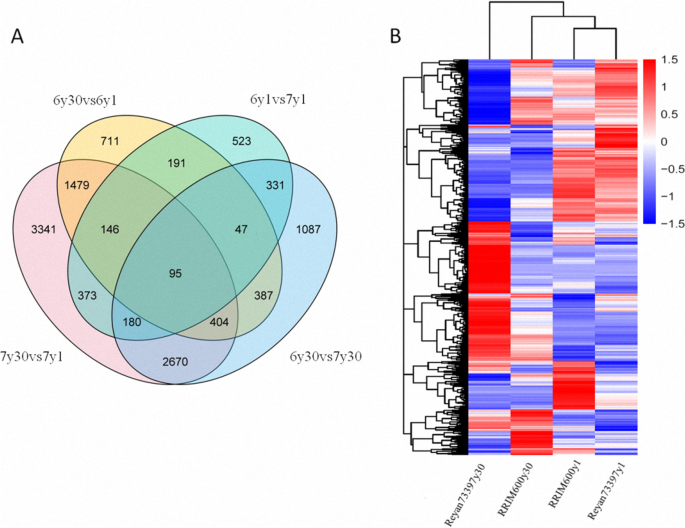
要了解橡胶MW克隆差异的转录变化,我们测序并分析了这些乳胶样品的转录组文件。使用Illumina成对端测序方法分离并测序总RNA。滤除了适配器,低质量读取和污染的读数,为REYAN73397Y1,RRIM600Y1,REYAN7397Y30和RRIM600Y30分别生成了平均数为6.45 GB,6.60 GB,6.35 GB和6.39 GB清洁基座。然后将这些清洁读数映射到橡胶树参考基因组,作图率为95.53% ~ 96.51%S1).将每个胶乳样品的基因表达模式计算为每百万片段的每千碱基每千碱基映射(FPKM)的片段。从4个胶乳样品中获得总共34,907个具有可检测表达水平的基因。为了分析测序结果,进行RRIM600Y1与RRIM633397Y1(6Y1VS7Y1)和RRIM600Y30与REYAN73397Y30(6Y30VS7Y30)的比较,以研究克隆MW差异的转录调节,而RRIM600Y30(6Y30)和REYAN73397Y30(7Y30)分别比较使用RRIM600Y1(6Y1)和REYAN73397Y1(7Y1)研究年龄相关的乳胶变异。这四个比较的DEG的数量如表所示2.与热研73397y30相比,RRIM600y30乳胶中有2648个基因表达上调,2553个基因表达下调。有趣的是,对比7y30vs7y1显示出最多的DEGs,在8688,表明热延7-33 - 97乳胶随着树龄的增加有明显的转录调控。构造了维恩图来表示不同比较之间的差异值关系。如图2a所示,6y30vs6y1和7y30vs7y1比较中有2124个DEGs重叠,说明它们在乳胶样品中年龄相关差异中的作用。比较6y1vs7y1和6y30vs7y30之间的653个重叠DEGs应该具有不同的克隆表达模式。基因表达谱的层次聚类分析表明,转录组测序分析得到的DEGs可分为4个簇,同一亚簇中的基因在乳胶样品中的表达模式相似(图)。2b和无花果。S1).Reyan73397y30中DEGs的表达模式与RRIM600y30、RRIM600y1和Reyan73397y1的表达模式不一致(图)。2b).为了验证转录组分析的结果,我们采用qRT-PCR进一步研究了17个关键橡胶生物合成基因和调控因子的表达水平。如图所示。S2, qRT-PCR结果与RNA-seq (R2= 0.950),说明转录组测序得到的表达数据是可靠的。
橡胶生物合成相关基因的表达水平
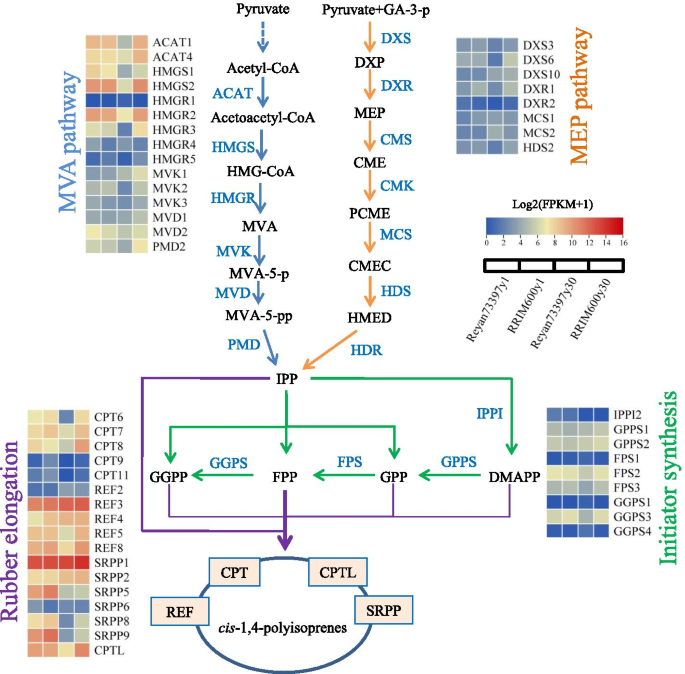
此前共鉴定出85个基因为橡胶生物合成基因,其中18个参与MVA途径,22个参与MEP, 15个参与APP引发生物合成,30个参与橡胶颗粒相关橡胶伸长[3.,6,26]。在四种比较中将这些橡胶生物合成基因中的四十九个基因鉴定为DEG。绘制橡胶生物合成途径的简单模式图,并且在四个胶乳样品中橡胶生物合成基因的FPKM值以热图的形式示出(图。3.)并在表格中列出S2.Reyan73397y1与RRIM600y1相比,2个DEGs (HMGR4和SRPP6)表达上调,3个DEGs (REF4、CPT9和DXR1)表达下调。除HMGR4外,其余MVA通路基因在Reyan73397y1中的表达水平略高于RRIM600y1,但未达到DEG阈值。与Reyan73397y30相比,RRIM600y30共上调27个deg,下调3个deg (HMGR1、MCS2和GGPS4),其中MVA通路13个基因上调,MEP通路9个基因上调(表)S2和无花果。3.).为了引发橡胶生物合成,首先将IPP转化为其高精电泳的异构体DMApp,其被异戊基二磷酸异构酶(IPPI)基因催化。我们的研究结果表明,IPPI1和IPPI2都存在于乳胶中相对较低的水平。IPPI1在四种乳胶样品中相当表达,而IPPI2在1岁的树上显示出乳胶的表达水平,而不是来自30岁的树木(表S2和无花果。3.).一群反式-戊二烯转移酶,包括GGPS、FPPS和GGPPS,催化生物合成app,可作为橡胶分子引发剂,也可作为非橡胶类异戊二烯前体。它们的表达水平反式Reyan73397y30与RRIM600y30相比,-戊酰基转移酶基因表达下调,而RRIM600y1与Reyan73397y1之间无显著差异。此外,RRIM600y30胶乳中CPT和CPTL转录本的积累量显著高于Reyan73397y30胶乳。
差异表达的橡胶生物合成基因FPKM值的热图(Log2(FPKM + 1))从左到右各列分别代表重复基因,各列分别代表乳胶样品Reyan73397y1、RRIM600y1、Reyan73397y30和RRIM600y30。缩略语:ACAT、acetyl-CoA acetyltransferase;HMGS:羟甲基戊二酰辅酶A合成酶;HMG-CoA,羟甲基戊二酰辅酶A;HMGR:羟甲基戊二酰辅酶A还原酶;MVA,甲羟戊酸;MVK,甲羟戊酸激酶;MVA-5-p mevalonate-5-phosphate;MVD phosphomevalonate激酶; MVA-5-pp, mevalonate-5-diphosphate; PMD, diphosphomevalonate decarboxylase; GA-3-p, D-Glyceraldehyde 3-phosphate; DXS, 1-deoxy-D-xylulose 5-phosphate synthase; DXP, 1-deoxy-D-xylulose-5-phosphate; DXR, 1-deoxy-D-xylulose 5-phosphate reductoisomerase; MEP, 2-C-methyl-D-erythritol 4-phosphate; CMS, 2-C-methyl-D-erythritol-4-phosphate cytidylyltransferase; CME, 2-C-methyl-D-erythritol-4-phosphate; CMK, 4-(cytidine 5’-diphospho)-2-C-methyl-D-erythritol kinase; PCME, 4-(cytidine 5’-diphospho)-2-C-methyl-D-erythritol; MCS, 2-C-methyl-D-erythritol 2,4-cyclodiphosphate synthase; CMEC, 2-C-methyl-D-erythritol 2,4-cyclodiphosphate; HDS, 4-hydroxy-3-methylbut-2-enyl diphosphate synthase; HMED, 4-hydroxy-3-methyl-but-2-en-1-yl diphosphate; HDR, 4-hydroxy-3-methylbut-2-enyl diphosphate reductase; IPPI, isopentenyl diphosphate isomerase; GPPS, geranyl pyrophosphate synthase; FPS, farnesyl pyrophosphate synthase; GGPS, geranylgeranyl pyrophosphate synthase; CPT, cis-prenyltransferase; REF, rubber elongation factor; SRPP, small rubber particle protein; CPTL, CPT-like
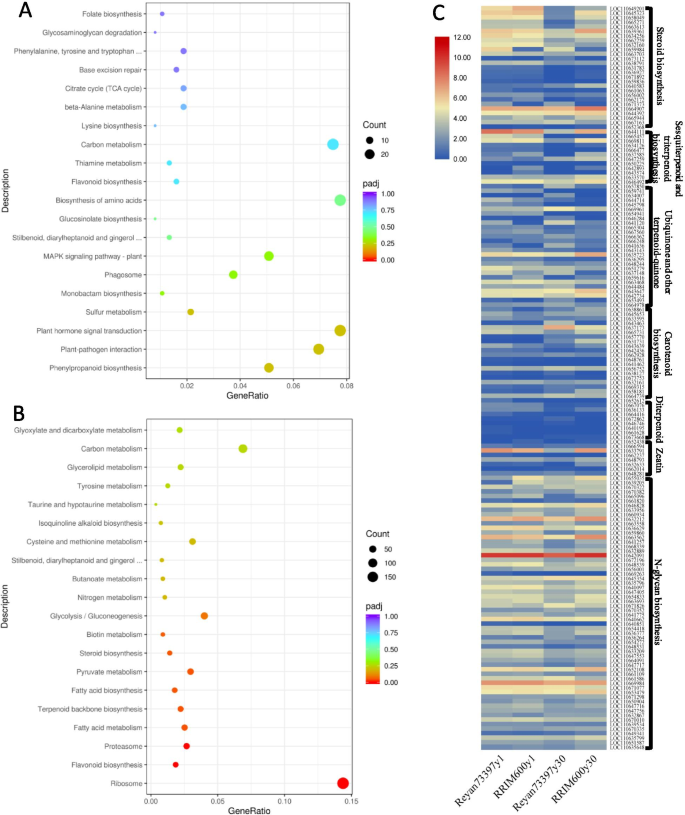
通过KEGG途径富集分析和基因集富集分析(GSEA)进一步研究橡胶生物合成相关途径基因的表达水平。图中分别显示了6y1vs7y1和6y30vs7y30的KEGG通路的富集。4表中列出了GSEA中与类异戊二烯途径相关的富集基因集S3.与Reyan73397y30相比,RRIM600y30富集了丙酮酸代谢(pop00620)和糖酵解/糖异生(pop00010)途径,这两种途径是产生丙酮酸和乙酰辅酶a前体的IPP生物合成途径。萜类主干生物合成途径(pop00900)在RRIM600y30中显著富集,这有助于提高橡胶生物合成酶的水平。与RRIM600y30相比,Reyan73397y30的核糖体、蛋白酶体和类黄酮通路显著上调,而脂肪酸生物合成和代谢基因下调(图3)。4b).与RRIM600y1比较,Reyan73397y1乳胶中富集了非橡胶类异戊二烯途径,包括类胡萝卜素生物合成(pop00906)、玉米素生物合成(pop00908)、泛素酮等萜醌生物合成(pop00130)和二萜生物合成(pop00904)。n -聚糖生物合成(pop00510)途径在RRIM600y1中富集(表)S3).在乳胶中,来自30岁的树木,非橡胶异戊二烯分支,包括癫痫萜类化合物和三萜类生物合成(POP00909),类固醇生物合成(POP00100),Zeepin生物合成(POP00908)和N-聚糖生物合成(POP00510),表达了更高的表达水平在RRIM600乳胶,而不是在Reyan7-33-97乳胶(表S3).为清晰显示这些富集的非橡胶类异戊二烯途径中DEGs的表达模式,其FPKM值见表S4和无花果。4c。
橡胶颗粒相关蛋白基因的表达水平
REF和SRPP是与橡胶颗粒相关的主要蛋白质。本研究的比较转录组分析表明,这些乳胶样品中REF和SRPP基因存在差异表达。如图所示。3.和表S2,大多数ref基因在30岁的树上显示出乳胶的表达水平高于1岁的树木,除了REF5和REF8,在REYAN73397Y30中显示出较低的表达水平。此外,来自30岁的树木的乳胶呈现出4个SRPP同种型的较高的转录物(SRPP1,SRPP2,SRPP3和SRPP4),同时表达了比1岁树龄的较低水平的其余SRPP基因(无花果。3.和表S2).
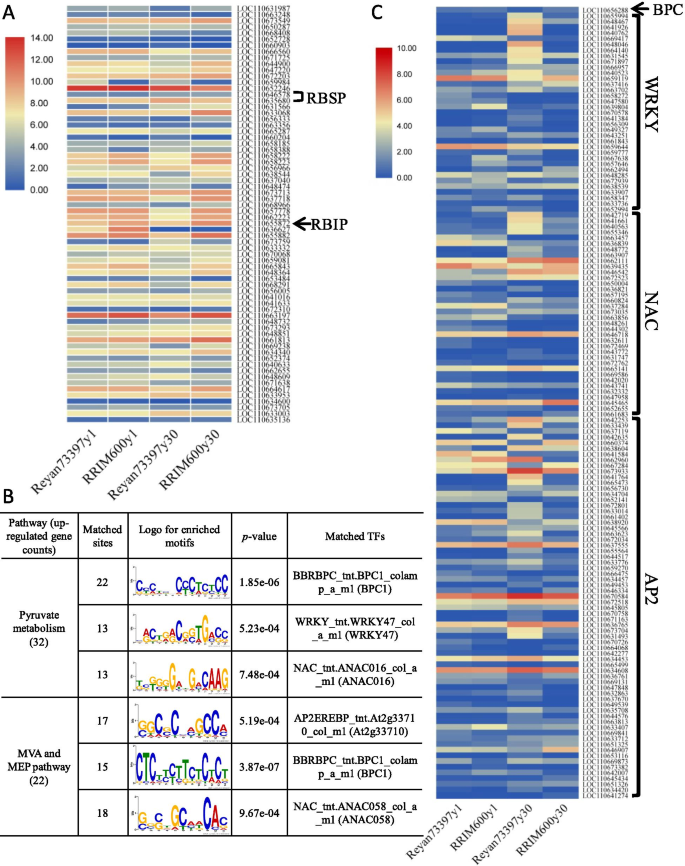
除了REF/SRPP基因外,基于之前发表的橡胶颗粒蛋白质组数据,我们鉴定了136个编码橡胶颗粒相关蛋白的转录本[27]。在这些基因中,在四个乳胶样品中,68差异表达,表达水平列于表中S5和无花果。5a.展示了几个以前被鉴定为橡胶生物合成调节因子的基因,包括橡胶生物合成刺激蛋白(RBSP)和橡胶生物合成抑制蛋白(RBIP)。在同龄期的树木中,RRIM600的乳胶比热闫7-33 - 97树的乳胶RP蛋白表达量高,而RP蛋白表达量小。
分析可能与IPP生物合成相关的差异表达转录因子
较高的IPP前驱体供给对提高橡胶的产率和分子量至关重要。然而,目前关于ipp生成途径的转录调控的知识橡胶树是非常有限的。在我们目前的研究中,总共有3077个基因在橡胶树乳胶被鉴定为转录调控因子。其中,466个基因在四种乳胶样品中有差异表达,其FPKM值列于表中S6.IPP以蔗糖为原料进行生物合成,蔗糖首先代谢成丙酮酸,然后代谢成乙酰辅酶a [2,35]。因此,我们研究了可能参与丙酮酸代谢和IPP生物合成途径调控的转录因子。由此可见,RRIM600y30中丙酮酸代谢的32个基因以及MVA和MEP途径的22个基因的表达量显著高于热研73397y30。因此,独联体-这些上调基因启动子上的调控元件(从转录起始位点的−1到−1000 bp)使用MEME套件寻找富集的基序[36]。统计上最重要的三个主题(取决于E-value)及其对应的TF族如图所示。5b.结果显示,在RRIM600y30中丙酮酸代谢、MVA和MEP通路中上调的DEGs的启动子中,BASIC PENTACYSTEINE (BPC)、WRKY、NAC和AP2四个TF家族的识别位点显著富集。这些tf被认为参与了IPP生物合成的调控。如图所示。5c,在差异表达的转录因子中,我们只鉴定到一个表达水平相对较低的BPC基因(LOC110656288)橡胶树与IPP生物合成途径表达水平呈正相关。有趣的是,大量WRKY、NAC和AP2基因在Reyan73397y30中的表达水平显著高于RRIM600y30,表明它们在IPP产生的调控中具有潜在的负调控作用。
讨论
育种橡胶树取代巴西橡胶树具有更高橡胶质量的克隆体是我们的长期目标。因此,需要全面了解橡胶生物合成过程中分子量的调控,以指导有效的分子修饰以预期地提高橡胶的质量橡胶树.根据先前的研究结果[23,25],橡胶的分子量存在无性系差异,且与龄期密切相关橡胶树为橡胶分子量调控机理的研究提供了方向。因此,我们测定了1年原始树和30年常规抽穗树乳胶的数平均分子量(MN)、重量平均分子量(MW)和分子量分布(MWD)橡胶树克隆Reyan7-33-97和RRIM600。GPC结果表明,平均MW橡胶树Reyan7-33-97无性系和RRIM600无性系的乳胶液含量不同,且随树龄增加而增加1). 在同一试验中,Reyan73397y1乳胶的分子量略高于RRIM600y1乳胶,而RRIM600y30乳胶的分子量明显高于Reyan73397y30乳胶(表1)1).这些结果揭示了热研7-33 - 97和RRIM600树种中胶乳分子量的明显调控规律,为后续研究胶乳分子量调控机理提供了基础。
橡胶生物合成是在橡胶颗粒表面进行的,橡胶颗粒是内质网衍生的细胞器,由疏水聚异戊二烯核和由脂质和蛋白质构成的包围单层膜组成[18]. MW和RP尺寸之间的相关性已被广泛研究。塔拉奇温等人[17[报道,RP的大小与乳胶分子量呈负相关。较小的RPS往往具有较高的平均分子量的橡胶橡胶树树。在我们的研究中,LLS和TEM结果表明,RRIM600树的平均RP尺寸小于同一年龄的热闫7-33 - 97树。结果表明,四种样品的平均粒径与胶乳分子量没有直接的相关性。最近的一项研究表明,rp的粒径并不是NR分子粒径的决定因素[19],需要进一步研究以确定RPs的大小变化是否在乳胶分子量的克隆和年龄相关差异的形成中起作用。
胶乳和RP大小的差异可能与橡胶颗粒膜蛋白有关。与较大的RPs相比,较小的RPs包含更多的蛋白质,并表现出更高的橡胶转移酶(RT-ase)酶活性[19,20.]。REFs和SRPPs是橡胶颗粒上含量最多的蛋白质,是具有不同性质的同源蛋白。SRPPs对膜的亲和性较低,覆盖脂质头基而不干扰膜的完整性,而REFs具有较高的膜亲和性,可插入膜并扰动膜的完整性[37]。Yamashita等人[19]显示SRPP亚型主要出现在较小的RP组分中,而REF亚型主要出现在较大的RP组分中。转录组数据显示,与1年生树的乳胶相比,30年生树的乳胶显示了更多的REF和4个SRPP亚型(SRPP1、SRPP2、SRPP3和SRPP4)的转录本,而其余的SRPP基因表达水平较低。在同年龄的树木中,与热闫7-33 - 97乳胶中相比,RRIM600乳胶中REF和SRPP基因均高表达(图7-33 - 97)。3.和表S2).这些发现表明,四种乳胶样品中RPs的大小和形态的显著差异可能与这些差异表达的REF和SRPP蛋白有关。此前已有研究表明,REF基因表达模式与乳胶产量呈正相关[38]。我们目前的研究显示了不一致的结果:无论是1岁还是30岁橡胶树树木,RRIM600胶乳的丰度在高产克隆REYAN7-33-97中的RRIM600胶乳中的丰度较高。类似地,RRIM600的RRIM同种型的表达水平高于高产克隆PB350和RRIM901 [33]。因此,REF/SRPP在橡胶生物合成中的作用有待进一步研究。
除了Ref / SRPP蛋白外,我们的结果表明,RP相关蛋白的整体转录性丰度似乎与平均RP尺寸负相关(图。1b,无花果。5和表S5).此外,通过透射电镜对橡胶颗粒的微观分析表明,热炎7-33 - 97乔木中的橡胶颗粒存在聚集现象,而RRIM600乔木中的橡胶颗粒则保持单生(图2)。1).根据Dai等人[39, RP蛋白使带负电荷的粒子均匀地分散在其中橡胶树乳胶。我们认为,在相同年龄的树木中,RRIM600胶乳中橡胶颗粒尺寸较小的原因可能是橡胶颗粒蛋白质的丰度较高,这提供了增强的静电斥力,防止橡胶颗粒聚集成更大的颗粒。
在这些rp定位蛋白中,一种patatin样蛋白(LOC110655872)此前被鉴定为橡胶生物合成抑制蛋白(RBIP) [24]。结果表明,LOC110655872在RRIM600胶乳中的表达量显著高于热岩7-33 - 97高产树胶乳中的表达量(见表2)S5和无花果。5a).真核起始因子5A (Eukaryotic initiation factor 5A, eIF-5A)是蛋白质合成过程中参与第一个肽形成的因子,此前被鉴定为橡胶生物合成刺激蛋白(rubber biosynthesis stimulator protein, RBSP) [40]。在两个差异表达的eIF基因中,eIF- 5a (LOC110635680)在高产无性系热炎7-33 - 97的乳胶中表达量是RRIM600的2倍(表2)S5和无花果。5a).我们的研究结果表明,这些差异表达的橡胶生物合成调节剂可能与RRIM600和Reyan7-33-97不同产量性状有关。
研究了橡胶生物合成相关基因的表达水平,结果表明,在1岁的树上,REYAN7-33-97乳胶表达水平略高于RRIM600乳胶,表明IPP生物合成水平较高REYAN73397Y1乳胶比RRIM600Y1乳胶。在30岁的树木的乳胶中,糖酵解/葡糖生成(POP00010),丙酮酸代谢(POP00620),TRIM600Y30的萜类骨架生物合成(POP00900),MVA和MEP途径高于REYAN73397Y30(表S2,无花果。4b和表S3).总之,我们目前的研究显示了有趣的结果,在30年的定期抽穗的树木中,来自平均产量的RRIM600无性系的乳胶似乎比来自高产的热研7-33 - 97无性系的乳胶具有更高水平的乙酰辅酶a、IPP前体和橡胶生物合成活性。这种相对转录活性的趋势似乎反映了此前RRIM600与高产无性系PB350和RRIM901之间的比较研究[33]。
一些早期的研究表明了基因调节各种App生物合成对橡胶生产植物MW和产率的影响。Euippi的过度表达,其催化了所有APP引发剂的生物合成中的第一步,导致转基因的产量和MW的产量和MW大肠ulmoides[41]. 在转基因植物中合成了更多的橡胶分子p . argentatum植物overexpressing帧/秒和GGPPS.但这些分子的分子量较低[42]。同样,我们正在进行的另一项研究表明橡胶树过表现出外源的树木帧/秒基因表现出两者的表达增加IPPI它们的产量性状需要进一步研究(未发表的数据)。正如之前在体外实验中所观察到的,在有限的IPP浓度下,增加引发剂会以较低的MW产生较高的乳液产率[7,16]。这些发现揭示了生产和消耗IPP和APPs的过程在调节胶乳分子量和产量方面的重要作用。
转录组图谱的进一步比较分析为RRIM600和Reyan7-33-97常规抽穗株系不同的乳胶MW和产量性状的机制提供了依据。我们的结果表明,从乳胶橡胶树在相同年龄的树中,IPPI基因被比较地表达(图。3.和表S2),表明RRIM600和Reyan7-33-97胶乳间IPP转化为DMAPP的效率相当。值得注意的是,这些下游非橡胶类异戊二烯途径的基因集在来自相同年龄的RRIM600和Reyan7-33-97树的乳胶中存在差异表达(表1)S3),消耗APP引发剂生产功能和结构多样化的非橡胶类异戊二烯产品。在Reyan73397y1和RRIM600y1的比较中,类胡萝卜素生物合成(pop00906)、玉米素生物合成(pop00908)、泛素酮和其他萜类奎宁生物合成(pop00130)和二萜类生物合成(pop00904)途径在Reyan73397y1乳胶中显著富集。在RRIM600y1基因中富集了n -聚糖生物合成(pop00510)途径(见表2)S3).在乳胶中,来自30岁的树木,非橡胶异戊二烯分支,包括癫痫萜类化合物和三萜类生物合成(POP00909),类固醇生物合成(POP00100),Zeepin生物合成(POP00908)和N-聚糖生物合成(POP00510),表达了更高的表达水平在RRIM600乳胶,而不是在Reyan7-33-97乳胶(表S3).因此,在DMAPP供应有限的情况下,RRIM600y30胶乳在橡胶生物合成分支中APP引发剂通量较少,再加上IPP供应较多,导致橡胶分子较少,链长增加。此外,在1年生的树木中,与RRIM600y1乳胶相比,mw较高的Reyan73397乳胶具有较高的IPP水平和较低的APPs水平,这分别是由于表达的MVA途径略高和非橡胶类异戊二烯途径丰富(图)。4c和表S3).
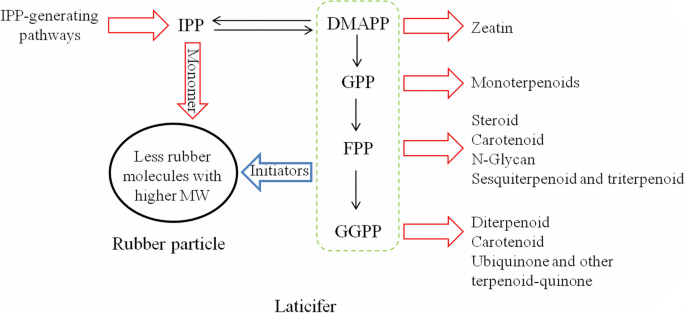
基于以上研究结果,我们认为相同树龄的RRIM600和Reyan7-33-97树胶乳MW的变化可能是由于不同的ipp生成途径和利用app生成橡胶和非橡胶类异戊二烯的途径的表达模式造成的(图7)。6). 当应用程序受到限制时,橡胶分子量随着IPP浓度的增加而增加。在DMAPP和APP供应有限的情况下,上调的非橡胶类异戊二烯途径会降低橡胶生物合成分支中的APP通量,导致橡胶分子量增加和乳胶产量降低,而下调非橡胶类途径则会产生相反的效果。需要进一步的生化研究来量化途径中间体IPP、DMAPP和APP引发剂以及下游橡胶和非橡胶产品。此外,还需要比较RT酶和非橡胶APP酶对APP的亲和力,以了解橡胶和非橡胶分支如何竞争APP引发剂,或者这是如何调节的。
然而,将1年的原始树的乳胶和30年的定期采穗树的乳胶进行比较,表明还有其他因素影响橡胶分子链的长度。尽管MVA和MEP途径以及下游非橡胶类异戊二烯途径的表达较高,但来自1年未采伐树的乳胶分子量明显低于30年常规采伐树的乳胶(见表2)1,无花果。3.和表S3).这可能是由于在快速生长的幼树中,IPP被用来生产大量植物生长过程所必需的化合物,只有当IPP超过它们的需求时,才会生产橡胶。如Cherian等人之前所述[3.],迄今所调查的所有rt -酶均具有高KmIPP防止橡胶在与必需的IPP酶竞争中聚合IPP。
基于乳胶中的乙酰辅酶和IPP前体的较高供应对于增加橡胶的产量和MW至关重要,我们研究了可能参与乙酰-CoA和IPP生物合成途径的调节的TFS.我们确定了属于四个家庭的139个差异表达的TFS(BPC,WRKY,NAC和AP2);他们的识别位点在RRIM600Y30中的丙酮酸代谢,MVA和MEP途径的上调次数的启动子中富集。shanks等人。[43提示原料药参与了对细胞分裂素的应答,并在植物的生长发育中起着不同的作用。目前,还没有关于原料药如何调节橡胶生物合成的信息橡胶树树。差异表达的BPC基因(LOM110656288)虽然以非常低的水平存在,但似乎与IPP生物合成途径的表达水平正相关,揭示其促进橡胶生物合成中的潜在作用橡胶树.一组NAC转录因子之前被鉴定为冷应激反应的调节因子[44]。Cao等[45)报道,HbNAC1(LOC110641661)参与脱水诱导乳汁管分化和乳胶生物合成。我们的结果表明HbNAC1随着树龄的增加,Reyan73397y30显著高于RRIM600y30,这与ipp产生途径的表达不一致,但与这两者的乳胶产量性状相一致橡胶树克隆。之前已经证明HbWRKY1(LOC110661399)和HbWRKY14(LOC110662072)抑制SRPP的表达,作为NR生物合成的负调控因子[3.,46,47]。虽然HbWRKY1和HbWRKY14结果表明,与RRIM600y30相比,Reyan73397y30中大量其他wrky的表达水平显著升高,这可能是Reyan73397y30中SRPP转录本丰度较低的原因。AP2转录因子主要参与乙烯信号通路,调控橡胶的生物合成橡胶树[2]。Makita等人[33]在CPT基因的启动子中证实了AP2的潜在结合元素橡胶树树。4个样品中AP2基因有67个差异表达,与RRIM600y30相比,Reyan73397y30中AP2基因表达上调的有20个,下调的有8个(表)S6),说明这些AP2基因可能在Reyan73397y30和RRIM600y30乳胶中受不同的转录调控。需要进一步的研究来阐明这些差异表达的转录因子在调控橡胶生物合成和分子量方面的作用橡胶树树。
结论
在我们目前的研究中,我们比较分析了1年生的原始树和30年定期抽穗树的乳胶的分子量、橡胶颗粒大小和转录组谱橡胶树克隆Reyan7-33-97和RRIM600。结果表明,热研7-33 - 97和RRIM600无性系的胶乳平均分子量和RP大小不同,且随树龄增加而增加。转录组分析表明,RRIM600乳胶中较小的平均粒径可能归因于较高的rp相关蛋白表达水平,Reyan7-33-97和RRIM600乳胶间的MW差异似乎是由于橡胶和非橡胶类异戊二烯提供和利用IPP和APP引发剂的途径的表达模式不同所致(图2)。6). 此外,还发现了一组可能在IPP生物合成中起作用的转录因子。综上所述,我们的结果表明,通过微调非橡胶类异戊二烯类的IPP生成途径和APP消耗途径的表达,精确调节橡胶生物合成中IPP和APP的水平和比率,可能是提高乳胶分子量和产量的可行工程策略。
方法
植物材料
的橡胶树本研究采用热研7-33 - 97和RRIM600接穗嫁接到种子-幼苗砧木上,在国家热带植物种质资源中心和海南省实验园种植。新鲜乳胶和树皮样本分别取自克隆热yan7-33 - 97和RRIM600的1年生未成熟树和30年定期抽丝树,分别命名为热yan73397y1、RRIM600y1、热yan73397y30和RRIM600y30。每个无性系在每个年龄取样三棵独立树。采集这些样本的必要许可由CATAS(中国热带农业科学院,中国海南儋州)提供。树皮样品用于研究乳汁管内橡胶颗粒的形态,乳胶样品用于分析MW、MWD、转录组和乳胶颗粒分布。
测定乳胶的分子量和粒径
用凝胶渗透色谱法(GPC)测定了胶乳样品的分子量分布。用于GPC的测量,干燥的乳胶样品溶解在四氢呋喃(THF)中,浓度为5 mg/ml。Waters GPC体系由两柱组成,排除限为2.0 × 1075.0 × 104,并对样品应用微分折射率探测器。以四氢呋喃为流动相,流速为0.5 ml/min。用市场上获得的已知分子量的聚异戊二烯标准品来校准色谱柱。测定温度为35℃,进样量为10 μl。利用Agilent (Santa Clara, USA) GPC/SEC软件(version 1.2)对每个乳胶样品的MW信息进行收集和分析。计算了3个生物重复的乳胶分子量的平均值和标准偏差。
每个胶乳样品中RPS的尺寸和分布由LA-960(Horiba Scientific,Japan)光散射粒度分布分析仪在30°C中测定,具有90°C的确定角度。
树皮解剖与透射电镜
如前所述,对树皮样品进行了超薄切片[48]。使用HT7700 TEM系统(日本,日本)在80 kV下,在800 kV下进行4000倍。
乳胶RNA分离和转录组测序
乳胶样品立即浸入液氮中冷冻,并研磨成细粉。根据制造商的协议,使用TRIzol试剂(Invitrogen公司)提取总RNA,并使用额外的DNA酶I去除任何基因组DNA的痕迹。使用NanoDrop 2000分光光度计(Thermo Fisher Scientific, USA)对纯化的RNA进行定量。全转录组cDNA文库构建和Illumina测序由Novogene公司(天津,中国)进行。每个乳胶样品,三个生物重复序列和分析。
序列数据组装和功能注释
FASTQ格式的原始数据经过严格的过滤条件,去除适配器序列、低质量读和碱基模糊读。乳胶样本的所有清洁读数均使用Trinity 2.0组装[49]并映射到之前发表的参考基因组橡胶树取代巴西橡胶树(NCBI生物工程:PRJNA394253) [26]。通过在以下公共数据库中搜索序列,对已组装的unigenes进行功能性注释:NCBI非冗余蛋白序列(Nr)、NCBI非冗余核苷酸序列(Nt)、SwissProt蛋白序列数据库、同源组蛋白簇(COG)、京都基因与基因组百科全书数据库(KEGG)、blastx基因本体(GO)。通过每千碱基/百万片段法计算组装的unigenes的表达水平[50]。评估乳胶样品之间的含量P.-value < 0.005 and |log2FoldChange|> 1 threshold [51]。使用TBtools构建热图和维恩图,分析不同比较之间的deg表达式和关系[52]。使用基于Wallenius非中央超几何分布的Goseq R包进行DEG的富集分析[53]。分别使用KOBAS和GSEA软件对DEGs进行KEGG途径富集和基因集富集分析(GSEA) [54,55]。
QRT-PCR分析
为了验证转录组分析的数据,进行了qRT-PCR。利用该方法测定了4个胶乳样品中编码关键橡胶生物合成酶和调节剂的17个基因的表达模式HbRH8(LOC110669478)基因作为内部对照。使用带有gDNA去除剂的Easyscript cDNA合成试剂盒(Transgen,中国)将总RNA反转录成cDNA。用特异性引物扩增稀释的cDNA(见表1)S7)与TransStart Tip Green qPCR SuperMix (Transgen, China)在CFX96 Real-Time PCR系统(Bio-Rad)上反应,94℃5 s、56℃15 s和72℃10 s循环45次。按公式2计算各基因的表达量−△△CT..对于每个胶乳样品,进行三种生物重复。使用这些17基因的相对表达水平计算转录组分析与QRT-PCR数据之间的相关性。
胶乳中转录调节因子的鉴定与分类
将乳胶样品中表达的转录本提交到在线iTAK分析工具(http://itak.feilab.net/cgi-bin/itak/index.cgi)以识别及自动分类转录因子[56]。
启动子主题分析
识别潜力独联体我们研究了30年树龄的RRIM600乳胶树样品中ipp生成途径中上调基因的启动子。提取这些基因转录起始位点上游1000 bp的序列,以便发现基序。MEME套件以经典模式使用,以识别丰富的主题[36],具有以下参数:最大图案数= 12,最小图案宽度= 6,最大图案宽度= 14和背景模型设置为1阶。使用带有默认参数的MEME套件中的TomTom工具,前三名统计学意义(低E-value)基序对每个基因集进行搜索拟南芥通过DAP motif数据库鉴定潜在的转录因子(TFs),这些转录因子能够识别这些富集的motif。进一步分析这些TF基因在四种乳胶样品中的表达水平。
数据和材料的可用性
本研究产生的所有原始序列数据已在美国国家生物技术信息中心(NCBI)的Short Read Archive数据库中保存,生物项目登录号为:PRJNA688380。
缩写
- 兆瓦:
-
衡量分子量
- 米歇尔。内格罗蓬特:
-
相对分子量
- 随钻测量:
-
分子量分布
- RP:
-
橡胶颗粒
- 透射电镜:
-
传输电子显微镜
- 接拍
-
激光光散射
- FPKM:
-
碎片每千碱基每百万碎片
- MVA:
-
甲羟戊酸
- MEP:
-
2-C-Methyl-D-erythritol 4-phosphate
- ACAT:
-
乙酰辅酶a乙酰转移酶
- HMGS:
-
3-Hydroxy-3-methylglutaryl-CoA合酶
- β-:
-
Hydroxymethylglutaryl辅酶A
- HMGR:
-
3-Hydroxyl-3-methylglutaryl-CoA还原酶
- MVK:
-
甲羟戊酸激酶
- MVA-5-p:
-
5-磷酸甲戊酯
- MVD:
-
Phosphomevalonate激酶
- MVA-5-pp:
-
甲羟戊酸-5-二磷酸盐
- PMD:
-
二磷酸戊戊酸脱羧酶
- GA-3-p:
-
d -甘油醛3 -磷酸
- dx:
-
1-脱氧-D-木酮糖5-磷酸合酶
- DXP:
-
1-脱氧-D-木糖糖-5-磷酸盐
- DXR:
-
1-Deoxy-D-xylulose 5-phosphate reductoisomerase
- CMS:
-
2-C-甲基-D-赤藓糖醇4-磷酸胞苷基转移酶
- CME:
-
2-C-甲基-D-赤藓糖醇-4-磷酸盐
- CMK:
-
4-(胞苷5 ' -二磷酸)-2- c -甲基-d -赤藓糖激酶
- PCME:
-
(4) -胞嘧啶核苷5 ' -diphospho 2-c-methyl-d-erythritol
- MCS:
-
2-C-methyl-D-erythritol 4-cyclodiphosphate合成酶
- CMEC:
-
2-C-methyl-D-erythritol 2, 4-cyclodiphosphate
- HDS:
-
4-Hydroxy-3-methylbut-2-enyl二磷酸合酶
- 嗯:
-
4-Hydroxy-3——methyl-but-2-en-1-yl二磷酸
- HDR:
-
4-Hydroxy-3-methylbut-2-enyl二磷酸还原酶
- IPPI:
-
Isopentenyl二磷酸异构酶
- gpp:
-
香叶焦磷酸合酶
- 帧:
-
通过焦磷酸合酶
- GGPS:
-
Geranylgeranyl焦磷酸合酶
- CPT:
-
独联体-Prenyltransferase
- CPTL:
-
CPT-like
- 裁判:
-
橡胶延长因子
- SRPP:
-
小橡胶颗粒蛋白
参考
- 1.
康沃尔K.橡胶生产。在:Thomas B, Murray BG, Murphy DJ,编辑。应用植物科学百科全书(第二版)。牛津大学:学术出版社;2017.410 - 9页。
- 2.
黄浩:热岩7-33-97橡胶树基因组的结构、组成及应用。在:橡胶树基因组。松井M,周k - s编辑。Cham:施普林格国际出版;2020: 13-40。
- 3.
Cherian S,Ryu Sb,康沃尔K.天然橡胶生物合成植物,橡胶转移酶复合物,以及代谢工程进展和前景。植物Biotechnol J. 2019; 17(11):2041-61。
- 4.
天然聚异戊二烯的结构和生物合成机制。Prog Polym SCI。1989; 14(3):339-71。
- 5.
普斯卡什·杰,戈特里奥·E,德菲厄·A,肯尼迪·JP。天然橡胶生物合成-活性碳正离子聚合?科学通报。2006;31(6):533-48。
- 6.
Yamashita S,Yamaguchi H,Waki T,Aoki Y,Mizuno M,Yanbe F,Ishii T,Funaki A,Tozawa Y,Miyagi-Inoue Y等。HEVEA Brasiliensis橡胶生物合成机械橡胶生物合成机械的识别与重构。Elife。2016; 5:E19022。
- 7.
植物橡胶生物化学的异同。植物化学。2001;57(7):1123 - 34。
- 8.
在三种进化上不同的植物中橡胶颗粒结构和功能的基本相似性。J Nat Rubber Res. 1993; 8:275-85。
- 9.
Nawamawat K, Sakdapipanich JT, Ho CC, Ma YJ, Song J, Vancso JG。巴西橡胶树天然胶乳颗粒的表面纳米结构。材料科学与工程。2011;39(1-3):157-66。
- 10.
Whalen M,McMahan C,Shintani D.制作生产工业上有用的天然橡胶的作物。在:植物和微生物中的异戊二烯合成。纽约纽约:Springer;2013. p。329-45。
- 11.
Gee G, Treloar LRG。橡胶组分分子量与物理性能的关系。橡胶化学技术。1941;16(3):580-9。
- 12.
Kok CM:分子量对uv降解天然橡胶物理性能的影响。欧元体系J1985年,21(1):37-40。
- 13.
Dick JS,Harmon C,VARE A.使用橡胶工艺分析仪的天然橡胶质量保证。聚合物测试。1999年; 18(5):327-62。
- 14.
Swanson Cl,Buchanan Ra,Otey FH。来自选定的温带区植物的天然橡胶的分子量。J Appl Polym SCI。1979; 23(3):743-8。
- 15
刘志刚,刘志刚,刘志刚。顺-1,4-聚异戊二烯的合成及其分子结构的研究进展[J]。植物化学。1999;(1):51 43-51。
- 16
金属辅助因子对橡胶树橡胶生物合成速率和分子量的调控。Biomacromol。2005;6(1):279 - 89。
- 17.
Tarachiwin L,Sakdapipanich JT,Tanaka Y. Hevea Brasiliensis粒度与分子量之间的关系。橡胶化学技术。2005; 78(4):694-704。
- 18.
王志强,王志强,王志强,等。天然橡胶α -末端基团的结构表征。《生物高分子。2005;6(4):1858 - 63。
- 19.
Yamashita S,Mizuno M,Hayashi H,Yamaguchi H,Miyagi-Inoue Y,Fushihara K,Koyama T,Nakayama T,Takahashi S. Hevea Brasiliensis的小型和大橡胶颗粒的纯化和表征。Biosci Biotechnol Biochem。2018; 82(6):1011-20。
- 20。
王东,谢强,孙勇,仝志,常磊,于磊,张旭,袁波,何鹏,金旭,等。蛋白质组学揭示了小橡胶颗粒是天然橡胶生产中刺激乙烯的关键橡胶生物合成机器。中华医学杂志。2019;20(20):5082。
- 21。
Berthelot K,Lecomte S,Estevez Y,Peruch F.HEVEA Brasiliensis Ref(HEV B 1)和SRPP(HEV B 3):概述橡胶粒子蛋白质。生物chimie。2014; 106:1-9。
- 22.
Collins-Silva J, Nural AT, Skaggs A, Scott D, Hathwaik U, Woolsey R, Schegg K, McMahan C, Whalen M, Cornish K, et al.;俄罗斯蒲公英(Taraxacum koka -saghyz)橡胶小颗粒蛋白TkSRPP3水平的改变导致橡胶代谢的定性和定量变化。植物化学。2012;79:46-56。
- 23.
关键词:橡胶树,胶乳,粒径,分子质量橡胶学报1999;2(3):150-9。
- 24.
Chow K-S,Bahari A,Taylor MA,Marshall DF:Hevea Brasiliensis橡胶生物合成的基因组学。在:橡胶树基因组。松井M,周k - s编辑。Cham:施普林格国际出版;2020:93-115。
- 25.
Kovuttikulrangsie S,Sakdapipanich J.来自不同年龄和克隆HEVEA树的NR的分子量(MW)和分子量分布(MWD)。Songklanakarin J SCI Technol。2005; 27(2):337-42。
- 26.
唐C,杨米,方y,罗y,gao s,xiao x,z,周b,张b,tan x等。橡胶树基因组揭示了橡胶生产和物种适应的新见解。Nat植物。2016; 2(6):16073。
- 27
戴林,康刚,李勇,聂振华,段晨,曾蓉。橡胶树胶粒蛋白质组学分析。acta botanica sinica(云南植物学报). 2013;42(2):457 - 462。
- 28
Luo Z, Iaffaldano BJ, Zhuang X, Fresnedo-Ramirez J, Cornish K. Analysis of first Taraxacum koko -saghyz transcriptome reveals potential rubber yield related SNPs。Sci众议员2017;7(1):9939。
- 29.
周克勤,万克良,杨海英。橡胶树胶乳的转录组分析对橡胶生物合成的影响。中国生物医学工程学报。2007;58(10):2429-40。
- 30.
赵军,陈勇,吴胜,田文敏。通过对橡胶树克隆CATAS8-79和PR107胶乳的转录组比较分析,揭示了调控胶乳再生和胶乳流动时间的新线索。植物学报。2015;15(1):104。
- 31。
邓杨H (X, Y, Z,刘H,陈J,戴L,夏Z, G,李·d·转录组分析揭示分子机制潜在增长杂种优势和弱点的橡胶树苗。BMC Plant biology . 2018;18(1):10。
- 32。
Lau N-S,Makita Y,Othman As,Matsui M:RRIM 600橡胶树基因组:首屈一指的谱系克隆的测序和分析策略。在:橡胶树基因组。编辑:松井M,周K-S。湛:斯普林格国际出版公司;2020: 41-54.
- 33。
Makita Y, Ng KK, Veera Singham G, Kawashima M, Hirakawa H, Sato S, Othman AS, Matsui M.橡胶树全长cDNA的大规模收集和转录组分析。DNA研究》2017;24(2):159 - 67。
- 34.
黄H:中国橡胶育种50年。中国农业出版社;2005.
- 35.
Long X,Li H,Yang J,Xin L,Fang Y,He B,Huang D,Tang C.来自Hevea Brasiliensis的真空蔗糖转运蛋白的表征:通过调节树皮中的细胞内蔗糖输送来涉及乳胶产生和含衣草。BMC植物BIOL。2019; 19(1):591。
- 36.
Bailey TL, Boden M, Buske FA, Frith M, Grant CE, Clementi L, Ren J, Li WW, Noble WS: MEME SUITE: motif发现和搜索工具。核酸Res2009, 37(Web服务器版本):W202-208。
- 37.
Berthelot K, Lecomte S, Estevez Y, Zhendre V, Henry S, Thevenot J, Dufourc EJ, Alves ID, Peruch f。BBA-Biomembranes。2014, 1838(1): 287 - 99。
- 38.
Priya P, Venkatachalam P, Thulaseedharan A:橡胶树(Hevea brasiliensis Muell)高产和低产无性系橡胶延伸因子(REF) mRNA转录的差异表达模式。参数)。植物细胞代表2007,26(10): 1833 - 1838。
- 39
戴林,聂志强,康刚,李勇,曾锐。橡胶树橡胶颗粒中橡胶伸长因子亚型的鉴定和亚细胞定位分析。植物生理与生物化学。2017;111:97-106。
- 40.
富含eIF-5A蛋白的细菌裂解物对巴西橡胶树橡胶生物合成的刺激作用。J橡胶Res2006年:251-259。
- 41.
杜仲二磷酸异戊二烯异构酶基因的表达及其对杜仲反式聚异戊二烯合成的影响生物科技BMC》。2012;12(1):78。
- 42.
2年转基因银胶菊的生长、橡胶和树脂评价。作物学报,2005;22(1):65-74。
- 43。
Shanks CM, Hecker A, Cheng CY, Brand L, Collani S, Schmid M, Schaller GE, Wanke D, Harter K, Kieber JJ。BASIC PENTACYSTEINE转录因子在细胞分裂素信号反应中的作用。植物j . 2018; 95(3): 458 - 73。
- 44。
龚XX,严斌,胡杰,杨长平,李永杰,刘建平,廖文斌。橡胶树(巴西橡胶树)的转录组分析发现了冷胁迫反应的候选调控因子。基因组的基因。2018;(11):1181 - 97。
- 45。
关键词:橡胶树,脱水诱导乳汁管分化,胶乳生物合成足底。2017;245(1):31-44。
- 46.
王勇,郭东,李海林,彭树清。巴西橡胶树WRKY转录因子HbWRKY1的特性分析植物生理与生物化学。2013;71:283-9。
- 47.
李海林,曲磊,郭东,王勇,朱建华,彭树清。组蛋白去乙酰化酶与WRKY转录因子相互作用,调控巴西橡胶树小橡胶颗粒蛋白基因的表达。Ind crop Prod. 2020;145:111989。
- 48.
Post J, van Deenen N, Fricke J, Kowalski N, Wurbs D, Schaller H, Eisenreich W, Huber C, Twyman RM, Prufer D,等。乳汁特异性顺式戊烯基转移酶沉默影响短角蒲蒿橡胶、三萜和菊粉含量。植物杂志。2012;158(3):1406 - 17所示。
- 49.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q, et al.;没有参考基因组的RNA-Seq数据的全长度转录组组装。生物科技Nat》。2011;29(7):644 - 52。
- 50.
基于RNA-Seq的哺乳动物转录组的定位和定量研究。Nat方法。2008;5(7):621 - 8。
- 51.
王L,Feng Z,王X,王X,张X.Degseq:用于从RNA-SEQ数据鉴定差异表达基因的R包。生物信息学。2010; 26(1):136-8。
- 52.
关键词:TBtools,大生物数据,交互分析,集成工具,大生物数据摩尔。2020;13(8):1194 - 202。
- 53.
年轻MD,Wakefield MJ,Smyth GK,Oshlack A. RNA-SEQ的基因本体分析:选择偏差。基因组Biol。2010; 11(2):R14。
- 54.
基于KEGG Orthology (KO)的自动化基因组注释和路径识别。生物信息学。2005;21(19):3787 - 93。
- 55。
Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES等。基因集富集分析:一种解释全基因组表达谱的基于知识的方法。中国科学院院刊2005;102(43):15545-50。
- 56。
郑义,娇c,孙h,rosli hg,pombo ma,zhang p,banf m,戴x,martin gb,giovannoni jj,等。ITAK:用于植物转录因子,转录调节剂和蛋白激酶的基因组预测和分类的程序。莫尔植物。2016; 9(12):1667-70。
致谢
不适用。
资金
本研究由国家自然科学基金项目(No. 31872708)、中央公益性科研机构基础研究基金项目(No. 1630022019019)和国家重点研发计划项目(No. 2019YFD1001102和No. 2019YFD1001105)资助。资助方支付了本研究的实验费用和发表费用,但不参与研究的设计、数据的收集、分析和解释以及手稿的撰写。
作者信息
隶属关系
贡献
Y.H.和T.H.构思了这些实验;s . x进行GPC、TEM实验,分析转录组数据,撰写主要手稿文本;J.L.对乳胶和树皮取样;X.D.和x.y分析了数据;联合大学修改了语言;H.H.和T.H.监督了这个项目。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:
表S1.乳胶转录组测序统计摘要。
额外的文件2:
表S2.四种巴西橡胶树胶乳中橡胶生物合成基因的表达水平(FPKM)
附加文件3:
表S3.与乳胶样品中的异戊二烯途径有关的差异富集的基因。
额外的文件4:
表S4.非橡胶类异戊二烯通路基因在四种巴西橡胶树胶乳中的表达水平(FPKM)
额外的文件5:
表S5.四种橡胶树胶乳样品中橡胶颗粒相关蛋白的差异表达。
额外的文件6:
表S6.乳胶样品中差异转录因子的分类及FPKM值
额外的文件7:
表S7.利用特异性引物对17个关键基因进行实时定量PCR。
附加文件8:
图S1. 层次聚类分析表明,通过转录组测序分析获得的DEGs表达模式可分为4个亚簇。
额外的文件9:
图S2.RNA-SEQ(FPKM)和QRT-PCR结果的相关分析。表达水平折叠在四个胶乳样品中的17个选定基因的转录组数据(y轴)的转录组数据(y轴)的转录物数据(y轴)变化的Log2的值的值折叠的Log2的值。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
李建军,李建军,李永华。et al。通过对胶乳转录组的比较分析,揭示了胶乳分子量变化的潜在机制橡胶树取代巴西橡胶树克隆RRIM600和Reyan7-33-97。植物生物学21,244(2021)。https://doi.org/10.1186/s12870-021-03022-5
已收到:
接受:
发表:
关键字
- 天然橡胶
- 橡胶树取代巴西橡胶树
- 橡胶分子量
- 乳胶转录组
- 橡胶颗粒大小