抽象的
背景
草原的草(叶片catharticus)是一种典型的凉爽季节饲料作物,冬季和春季生物量高的生物量产量和快速增长率。然而,由于可用的基因组资源有限,其遗传研究和育种仍然停滞不前。本研究的目的是使用高通量转录组测序产生大规模的基因组数据,并执行EST-SSR标记的初步验证b . catharticus.
结果
从大草原BCS1103的新的高产株收集包括种子,叶子和茎的11种组织样品。获得了总共257,773个unigenes,其中193,082(74.90%)注释。组织之间的比较分析鉴定为1803,3030和1570个基因,分别在种子,叶和茎中特别高度表达。从Unigene序列中鉴定了总共37,288个EST-SSR,并且设计了超过80,000个引物对。我们合成420个底漆对,并选择52种,具有高多态性,以估计24个遗传多样性和人口结构b . catharticus登记入册。虽然平均遗传距离为0.364,多样性较低,但来自南美、亚洲和野生的遗传多样性较高。此外,南美洲的遗传类型表现为纯血统,而亚洲的遗传类型表现为混合的内部关系,这表明基因流动的概率不同。系统发育分析将研究的植物分为4个支系,与表型聚类结果一致。最后,Mantel分析表明,表型变异主要由遗传组成。茎粗、株高、叶宽、生物量产量与遗传资料(r> 0.6,P< 0.001),可用于今后的选育。
结论
产生了一种可以利用遗传和分类学研究的基因组资源,以及分子育种b . catharticus及其未来的亲戚。
背景
蜡烛L.是一个广泛分布于温带世界的属,包含约150种C3草,其中许多尚未分类鉴定[1那2].叶片catharticus是主要的农用草种之一,属于Ceratochloa.部分(六倍体,2n = 6x = 42),在许多地区已归化,具有兼性闭花受精生殖行为和一年生或短寿多年生生长形式[1].由于生物量高产量,冬季和弹簧期间快速增长,适应性强,以及种子成熟后保持绿色的能力,b . catharticus在中国西南部的山区和丘陵地区的凉爽季节牧草草中变得越来越受欢迎[3.那4.].然而,作为栽培品种的b . catharticus其基因组资源相当有限,导致分子育种和分类研究进展缓慢。
下一代测序技术提供了高通量序列生成的尖端方法,允许快速和全面的基因组和转录本分析。广泛的转录组学研究已经应用于各种植物生物学环境中。例如,使用不同组织和阶段的比较转录组分析来揭示组织发育的调控模块[5.那6.那7.]或特定生化组件的代谢[8.那9.那10.,而组织特异性表达谱在揭示生物通路和调控网络方面已被证明是有效的。尽管基因组方法的普遍应用,到目前为止,这些还没有应用于研究b . catharticus.因此,仍然需要提供原始参考转录组型材b . catharticus及其亲属。
转录组测序也已用于鉴定分子标记,特别是简单的序列重复(SSR)标记,在许多饲料草种中,例如紫花苜蓿[11.],Elymus sibiricus.[12.那13.),而多花黑麦草[14.]由于其成本效益。分子标记在现代植物育种过程中具有很大的潜力,并且已被广泛应用于确定遗传多样性,基因型鉴定以及标记辅助选择(MAS)[13.那15.].但是,分子系统发育和遗传结构有限公司缺乏标志信息(蜡烛物种的EST序列的收集、保护和利用,目前尚不清楚蜡烛L.在GenBank数据库(https://www.ncbi.nlm.nih.gov/).以前,扩增的片段长度多态性(AFLP)估计在32名南美和一个北美蜡烛属登记入册(2].RAPD和AFLP分析b . catharticus从法国和新加坡展示了品种的狭窄基础,为使用分子标记技术提供了选择父母基因型以拓宽这些品种的遗传基础[16.].利用SSRs和aflp比较了分子标记与显著遗传变异之间的相关性叶片tectorum, SSRs被认为是强近交系物种群体遗传研究中表型性状的良好替代品[17.].由于缺乏来自自身种或属遗传数据的有效分子标记,小麦(Triticum Aestivum.)受雇于叶片评价标记的可转移性,显示出共显性、稳定性和高重现性等优点[18.].EST-SSRs已被证实在同一属的不同种之间,甚至在不同属之间具有良好的可转移性。此外,转录组测序(RNA-seq)可以用于大量SSR标记的鉴定和开发,与传统的SSR开发过程相比,速度更快,成本更低[13.].
本研究报告了一个全面的转录组测序b . catharticus基因型。主要目的是(a)通过转录组测序从不同发育阶段的不同组织中生成estb . catharticus,以及注释a德诺维转录组组装;(b)比较不同组织间的转录本表达,识别组织特异性基因;(c)进行大规模的在网上从转录组数据识别SSRS;(d)在与表型数据相结合的一组种质中,进行遗传多样性和群体结构分析。产生基因组资源,其显示在分子辅助选择中施用的初步潜力,这可以为进一步调查遗传和分类提供依据蜡烛及其亲属。
结果
Illumina公司测序,德诺维装配和功能注释
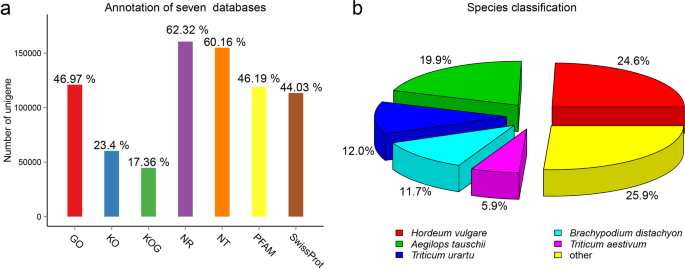
Illumina对11个种子、叶子和茎组织库进行测序,平均产生48945,309个原始reads和46,614,613个干净reads,后续分析使用高质量的干净reads(见表)1).数量为450,361的转录物组装,N50长度为1346bp,N90长度为281bp,从201到14,257bp的平均值为766bp。然后将转录物聚集成1129bp,1629bp和531bp分别为平均长度,n50和n90的总共257,773个undigenes(表2和图1).11个样本中FPKM密度分布的不同表达模式和正态分布如图S所示2.共有193,082(74.90%)的unigenes在公共蛋白数据库中被注释,其中160 662(62.32%)、155,087(60.16%)、119,080(46.19%)、113,501(44.03%)、60,321(23.4%)、44,775(17.36%)和121,098(46.97%)的unigenes分别来自NCBI Nr、Nt、PFAM、瑞士- prot、KEGG、KOG和GO数据库(图1)。1a).在所有7个数据库中,共有25739个(9.98%)Unigenes被注释,64,691个(25.1%)Unigenes没有任何匹配,这些Unigenes可能是在进化过程中产生的新基因b . catharticus基因组。基于NR数据库注释,与700多种物种相比,unigenesb . catharticus以顶部匹配显示大麦芽(24.6%),紧随其后的是Aegilops Tauschii.(19.9%),小麦属植物urartu(12.0%),Brachypodium distachyon(11.7%)和Triticum Aestivum.(5.9%;图。1b)。未经成熟的富集分析表明,在分子函数类别中高度富集,特别是“结合”(66,849)和“催化活性”(53,923)和“细胞”(38,314)(38,314)的“结合”(66,849)和“催化活性”(53,923)细胞组分中的“细胞部分”(38,293)(图。3.a).在五类KEGG通路中,代谢通路最大,有11个亚群和28653个基因,其中最显著的转录亚群有7828个基因(图S)3.b)。
组织中DEGs的分析b . catharticus
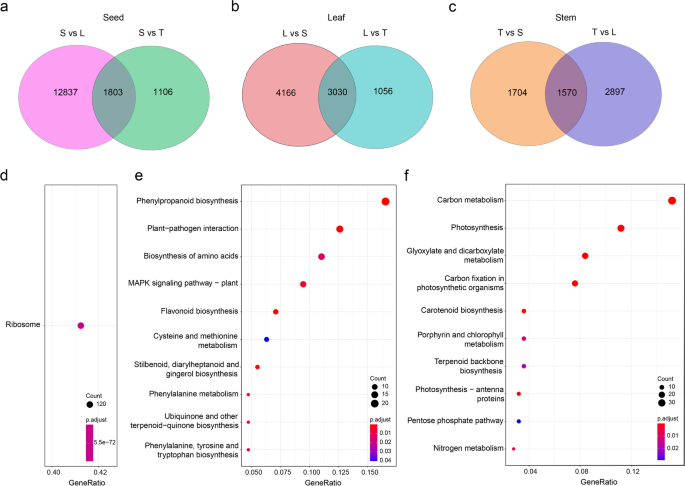
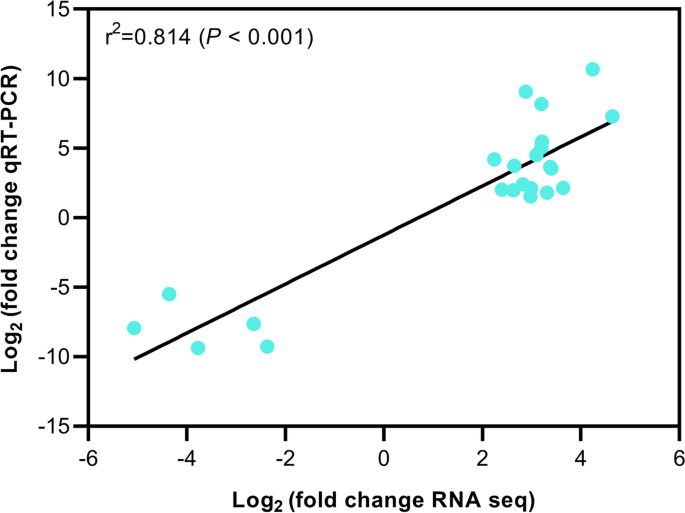
为了鉴定组织特异性基因,我们将10个转录组样本分为3组,种子S1、S2、F1、F2为S组,叶片L1、L2、L3为L组,茎T1、T2、T3为T组。我们对DEGs的S、L、T进行了比较分析(P<0.05), and determined tissue-specific highly expressed genes in one tissue, but not in the other tissues. We plotted Venn diagrams for the upregulated DEGs in the three pair-wise comparisons (i.e., S vs L, S vs T, and L vs T). A total of 1803 genes were found to be specifically and highly expressed in developing and germinating seeds (overlapping DEGs in “S vs L” and “S vs T,” Fig.2a),但不是在其他两种组织的叶子和茎中。在叶子中特别且高度表达3,030和1,570个基因(在“L VS S”和“L VS T中的重叠次数”图。2b)和茎(“T vs S”和“T vs L”的重叠DEGs,图。2c)分别。我们发现在“核糖体”的Kegg途径中显着富集在显影和发芽种子中具有高度表达的基因(图。2d).叶片特异性高表达基因在“碳代谢”、“光合作用”、“乙醛酸和二羧酸代谢”和“光合生物碳固定”等基因中显著富集(图4)。2e)。在“苯丙烷生物合成”,“植物 - 病原体相互作用”,“氨基酸生物合成”和“MAPK信号通路”中,茎特异性高表达基因显着富集。(植物 - 病原体相互作用“和”MAPK信号通路“(图。2f).为了验证RNA-seq中获得的deg,我们对3个组织样本(F2、L2和T2)中11个选择的基因进行了qRT-PCR分析,结果与RNA-seq结果吻合良好(r2 = 0.814,P< 0.001,无花果。3.).
识别EST-SSR标记和遗传多样性
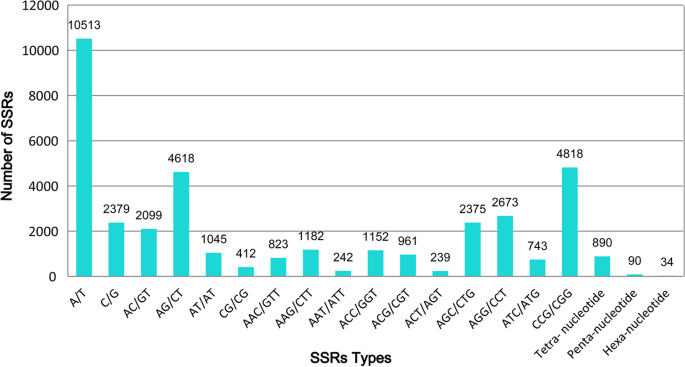
在32,370(14.47%)个unigenes中共鉴定出37288个SSR,其中4238个转录本中存在1个以上SSR。三核苷酸SSR重复最多(15,208个),其次是单核苷酸(12,892个)和二核苷酸(8,174个)。在单核苷酸重复中,A或T重复(10513次)比C或G重复(2379次)多4或5倍。CCG/CGG(4,818)、AGG/CCT(2,673)和AGC/CTG(2,375)组成了SSRs中所有三核苷酸重复基元的64.87%(图)。4.,表S.1).aag /CTTT、ATCCG/ATCGG和ACCTCC/AGGTGG重复序列分别占四、五、六核苷酸重复序列的主导地位,但这些重复序列很少出现。成功利用27540个SSRs设计了> 80000引物;随机选取420对引物进行多态性和性能分析,最终350对引物成功扩增24个DNA片段b . catharticus加入。为了进一步验证SSR基因座的序列,测序来自九个SSR标记的10个PCR产物。在所有情况下,所选载体的测序等位基因对原始轨迹的原始轨迹同源,有效地保证了可接受的下游分析(图S4.).
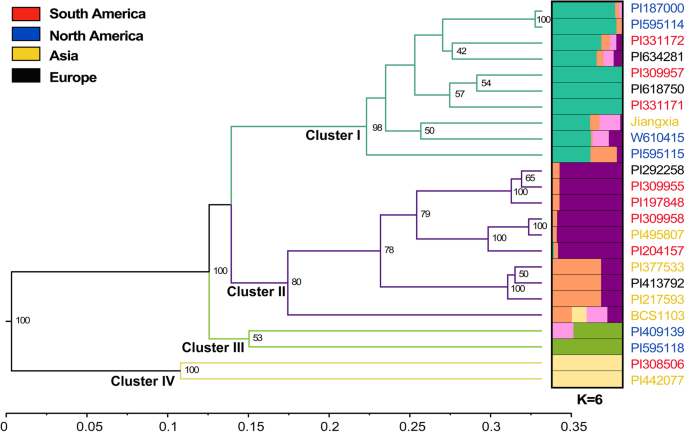
我们根据5-10个重复的二、三、四核苷酸SSRs设计了52对有效引物(表S)2).平均pp,pic,rp和mi值分别为91.60%,0.254,1.14和0.67个。在三种重复类型中,Tetra-核苷酸SSR显示相对较高的PIC(0.290),RP(1.29)和MI(0.73)值,表明高辨别力。来自五重复SSR的引物显示出更高的PIC值(0.285),而七个重复SSR显示出较高的RP(1.29)和MI(0.86)值(表3.).利用52对引物扩增出的152条条带对24份材料的Nei遗传距离进行估算。遗传距离为0.008 ~ 0.790,平均距离为0.364。最大值为0.790,最小值为0.008,最小值为PI187000和PI595114。目的:研究黄牛的遗传关系和种群结构b . catharticus进行UPGMA聚类分析和结构分析(图。5.).根据最大似然和δ K值(K = 6),将本研究的入选材料分为5个STRUCTURE亚组(以蓝绿、紫紫、橙、绿、淡黄5种颜色表示)。4个UPGMA聚类(聚类I ~ IV)与STRUCTURE分析结果一致。簇II包含两个STRUCTURE子群(紫色和橙色)。集群I和II均包含10个资源,而集群III和IV分别只有2个资源。此外,14个品种(> 50%)的隶属度系数较高(q值> 80%),其中南美、亚洲、欧洲和北美分别有7个、2个、2个和3个品种(图2)。5.).亚贫民中的遗传多样性大多符合地理位置。四大洲的遗传多样性指数(他)在0.173(欧洲)到0.238(亚洲)之间的不同,平均为0.209(表4.).此外,从野外收集的遗传多样性(0.259),其次是栽培品种(0.251),然后是不确定的育种状态(0.179)的材料。
表型变异和曼特尔分析
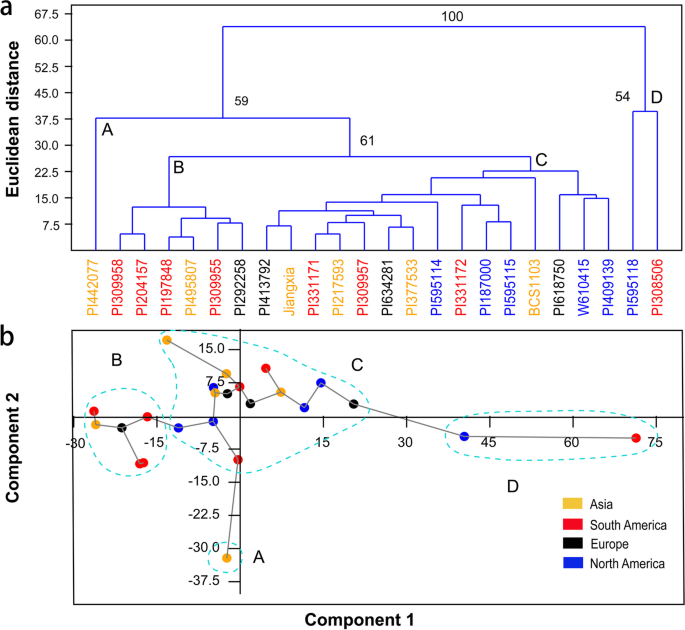
评估植物种质资源的基础是表型。在这项研究中,24中的11种定量特征b . cartharticus研究材料,估计育种潜力和表型变异。描述性统计显示,变异系数为2.21% ~ 24.87%,平均为11.61%,表型变异水平较高(表S3.).UPGMA集群和PCA分析表明,所有24种载体都分为四组(A,B,C和D),其显示与地理分布薄弱(图。6.).A组只有一个来自亚洲的物种(PI 442077),生物产量低,PH、SD、TN值低,旗叶宽。B组4份来自南美,2份来自欧洲和亚洲,PH、SD、叶宽较高,TN和LFI较低。C组几乎囊括了来自北美(5份)和欧洲(3份),以及来自亚洲(4份)和南美(3份)的其他材料,表现出高TN和LFI,低SD和叶宽。在D组中有2个品系,来自南美的PI308506和来自北美的PI595118,均具有特别高的TN和低SD,且具有矮秆表型(图2)。6.一个,表s3.).PI308506叶片窄,抽穗晚,PI595118叶片宽,抽穗早。PCA分析还表明,前两个分量占方差的91.97%(分别为76.45%和15.53%),共分为4个组(图2)。6.b)。
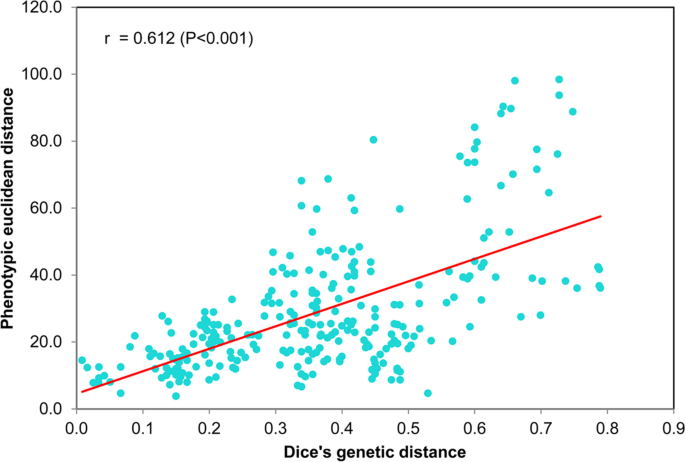
通过Mantel分析了解遗传距离与11个性状表型距离的关系,表型距离与遗传距离呈高度相关(r = 0.612,P< 0.01)(图7.).同时采用Mantel分析方法分析了24份材料11个表型性状的欧氏距离与遗传距离的相关性(表1)5.).结果表明,4个性状(PH、FLW、SD、FMY)的相关系数在0.340 ~ 0.666之间,呈极显著相关(p < 0.05)。r> 0.6,P<0.001).此外,表型基团与系统发育簇一致。遗传数据主要可以解释表型差异。例如,A组中PI442077的低生物收率,pH,SD,TN和宽旗叶可能是由于簇IV中的特定遗传信息(图。5.那6.和表S.3.).在C组中发现集群中的所有十种载体,B组中的所有六个载体都在Cluster II中。D组PI308506和PI595118均显示矮化表型,具有更多分蘖和较薄的茎(图。6.,表S.3.)但是在簇IV中的PI308506中PI308506之间的标题和叶宽差异存在显着差异,并且可以通过遗传聚类中的分裂解释。
讨论
转录组是关键遗传资源蜡烛物种
努力分子系统和分子育种蜡烛由于基因组资源有限,L.的产量停滞不前。在本研究中,我们的完整转录组分析b . catharticus覆盖11个组织样品,并且可能与几乎所有表达基因组装,共有257,773个unigenes,其中193,082(74.9%)被注释。虽然我们的数据代表了大量表达基因,可用于未来的遗传研究和改进计划b . catharticus甚至其他蜡烛例如,这些数量比之前报道的其他一些饲料物种的数量要高Dactylis glomerata[19.],Elymus sibiricus.[12.),狼尾草purpureum[20.].众所周知,PacBio和Nanopore的第三代测序可以产生高达1 Mb的reads,但全长转录本总是倾向于做各种组织的池化,以降低测序成本;此外,其序列误差也高于Illumina测序平台[21.那22.].我们的数据可作为后续转录组分析的基因参考b . catharticus在未来。
特定组织的基因表达
草种子是天然草地恢复和人工草地建立的基础,而茎和叶是饲料产量和营养价值的重要因素[23.那24.].很少有针对牧草组织特异性转录组的研究,以鉴定参与组织发育和饲料质量改进的候选基因。象草研究(狼尾草purpureum)茎木质纤维素鉴定了3,852只参与木质纤维素生物合成的43种候选基因,这可能促进高质量的大象草品种的发展[7.].我们发现了1570个茎特异基因,其中显著富集于苯丙素生物合成途径的基因可能影响饲料价值b . catharticus作为苯基丙醇代谢是木质纤维素生物合成的主要途径[7.那25.].同时鉴定了3030个主要与光合途径相关的叶片特异性基因,这些基因可能有助于提高叶片的光合效率和生物量参数。通过对紫花苜蓿叶片转录组的分析,鉴定出5133个DEGs和衰老相关途径,包括核糖体和苯丙素生物合成途径,淀粉和蔗糖代谢途径,并对叶片衰老的分子机制提出了新的解释[26.].此外,我们的比较分析表明,核糖体途径是1,803种种子特异性基因中最具显着富集的途径。众所周知,需要核糖体来合成储存和其他重要蛋白质,例如晚期胚胎发生的蛋白质(Lea)蛋白,具有种子成熟和干燥中的关键作用[27.],它们也需要种子萌发,其中新的核糖体在吸收几小时的吸收后产生,以合成与DNA修复,RNA合成,细胞器组件和能量供应相关的蛋白质[28.那29.].简单地说,这项工作提供了对组织特异性表达谱的初步理解b . catharticus这可能是有价值的基因组数据,以提高产量和质量b . catharticus.
丰富的EST-SSRsb . catharticus
EST-SSRs是一种强大的遗传标记,由于其具有共显性鉴定、多态性丰富、可移植性高、检测方法快速方便等优点,已广泛应用于遗传多样性、群体结构、品种鉴定、遗传图谱和标记辅助选择育种等方面[13.那30.].迄今为止,est - ssr的应用较少b . catharticus,多样性估计大多基于表型特征。在我们不同发育和生长阶段的转录组中,共有37,288个SSRs, EST-SSRs的出现频率为每7.8 kb 1个。该频率高于紫花苜蓿(1/12.06 kb) [11.),羊草(1/10.78 kb) (31.,但低于大肠sibiricus(前两份报告中的1 / 6.95 KB和1 / 6.2 KB [12.那13.])。此外,SSRs的总数也远远大于大肠sibiricus(两份报告共有8,769及8,871份[12.那13.]),多花黑麦草(11254) (14.]和苜蓿(1,649)[11.].EST-SSR标记多态性数量高的比例低于紫花苜蓿(27/100)[11.] 要么大肠sibiricus(112/500) (12.,这可能与底漆的设计和选择有关。SSR数量和频率的差异也很大程度上取决于物种、数据库大小、SSR搜索标准和使用的挖掘工具。本研究中各类SSRs中,三核苷酸重复序列最多,其次是单核苷酸重复序列和二核苷酸重复序列,三核苷酸重复序列最多的是CCG/CGG重复序列,与本研究中发现的相同E. sibiricus和L. multiflorum,但不同于GAA/CTT重复最丰富的在芝麻纪录和卢比斯花丁.SSR的丰富似乎是特定的物种[12.那13.那32.那33.].简而言之,本研究确定了许多有利于破坏遗传研究瓶颈问题的分子标记b . catharticus.
EST-SSRs是植物种质鉴定和分子育种的重要工具b . catharticus
了解遗传多样性和群体结构对种质资源的有效管理和利用具有重要意义[34.].评估饲料资源的遗传变异可以为选择资源提供基础,从而加快高质量的饲料育种的进展。评价b . catharticus种质比较相对滞后,分子标记的分化估计显着较高的量和分辨率明显高于形态学和细胞标志物特别罕见的[35.].以前关于遗传多样性的研究b . catharticus表明,商业品种的遗传基础非常窄,平均遗传相似系数为0.96 [16.].本研究共收集了来自四大洲的24份材料,包括野生型和不确定材料,其遗传相似度平均为0.636,甚至低于商品品种。来自亚洲的大部分种质被归为一个聚类,66.67%的种质为混合亲缘关系,而来自南美的87.50%的种质为纯亲缘关系(q值> 80%),说明两个区域间的基因流动概率不同。从育种情况来看,野生材料的多样性高于品种和栽培材料,这可能是长期人工驯化的贡献[36.].经过测试的野生资源显示出更高的遗传多样性,这也表明未来繁殖野生资源的综合品种可能益处。
在遗传和环境影响下,在遗传和环境影响下形成了许多种质,导致了丰富的形态多样性。对所有梳理的系统发育分析与表型群体一致。相关性相对较高(r = 0.612,P<0.001)的遗传变异与表型变异之间的关系,表型聚类与地理分布的关系较弱,说明表型多样性b . catharticus主要是由于遗传因素的贡献。这可以由之前的工作支持Ceratochloa.,显示高度相关(r= 0.70,p= 0.001)利用AFLP标记分析形态学变异与DNA多态性之间的关系[2]和低相关性(r=−0.243)蜡烛登记入册(3.].阿根廷居群表型多样性分析表明,阿根廷居群的总表型变异b . catharticus主要是由于环境因素[37.].本研究与其他研究不一致的原因可能是供试种质来源不同,表型性状采集不同。在本研究中,我们还认为FLW、PH、SD和FMY可能是一个选择育种项目中更可靠和有效的性状;其中,SD作为主要生物量性状的指标,变异系数较高,与分子数据的相关性较高,在标记-性状关联或QTL定位中具有较高的有效性和稳定性。所鉴定的EST-SSR标记可用于进一步的遗传选择和标记辅助育种。
结论
在这项研究中b . catharticus通过RNA-seq分析,分别鉴定了1803、3030和1570个在种子、叶片和茎中特异和高表达的基因。基于11个RNA-seq数据集,我们生成了257,773个unigenes,用于开发37,288个EST-SSRs。选取52个多态性EST-SSR标记对24个群体进行遗传多样性分析b . catharticus加入。在所有的过程中都发现了低遗传多样性,尽管南美和亚洲和亚洲和野生加入较高。系统发育分析将所有含义聚集成四种簇,这与表型聚类结果一致。此外,Mantel分析表明,总表型变异主要是遗传组分的贡献,以及四个表型参数,包括茎直径,植物高度,叶宽和生物质产量,与遗传变异显着相关,表明遗传选择的巨大潜力配种。一般而言,产生了一种可以使遗传和分类学研究的综合基因组资源,以及分子育种b . catharticus及其未来的亲戚。
方法
RNA分离和转录组测序
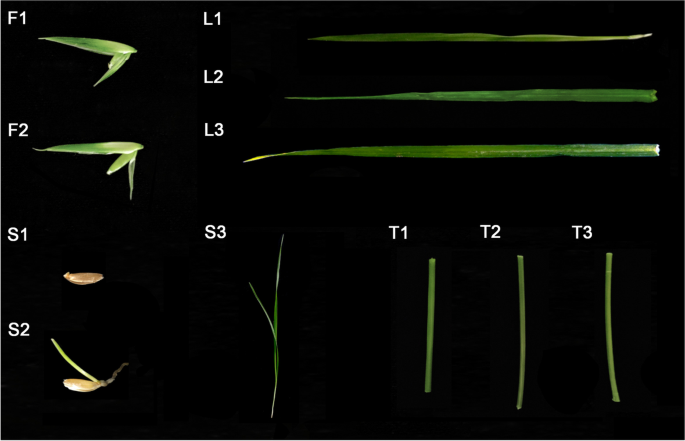
BCS1103,一个新的行b . catharticus具有优良的产量,最初是通过中国西南部四川省收集的野生种质选择的。这株植物在四川雅安的四川农业大学按照推荐的栽培方法在花盆中成熟。本研究共采集了10个健康个体的11个组织样本,包括胚根长1 - 2mm (S1)、茎长1cm (S2)的种子、三叶期地上苗(S3)、分蘖期叶片(L1)、旗叶(L2)、第二叶(L3)在受精后8天(DAF);分蘖期茎(T1) 8 DAF (T2)和20 DAF (T3);8 DAF (F1)和20 DAF (F2)的种子(图2)。8.).所有组织均直接放入液氮中,置于−80℃保存。采用Ghawana等人描述的方法分离总RNA [38.].每个样品的总量为1.5μgRNA用于文库结构。使用Nebnext®Ultra™RNA库预备套件为Illumina®(Neb,USA)生成测序库,在制造商的建议之后,并将索引代码添加到每个样本的属性序列。在Illumina Hiseq 2500平台上测序了库。
德诺维组装和基因注释
In this study, clean reads were obtained by removing those reads that contained adapters or poly-N (i.e., ambiguous sequences), and low-quality reads that had more than 50% of bases with Q-value ≤ 20. The unigenes were binned by Corset [39.]由使用Trinity的集合的contigs [40].所有unigenes分别在NCBI非冗余蛋白数据库(Nr)、NCBI核苷酸数据库(Nt)、Pfam、Swiss-Prot、Cluster of Orthologous Group、Gene Ontology (GO)和Kyoto Encyclopedia of Genes and Genomes (KEGG)等7个数据库中进行注释。如果这些数据库的结果不一致,NCBI Nt和Nr比对被认为是较好的注释。
基因表达和DEGS鉴定
RSEM软件用于将干净的读值映射到unigenes上[41].基于FPKM估计基因表达水平(每百万次映射片段的外显子模型的每千碱基片段)值。基于日志绘制由FPKM值密度分布反射的每个样品的表达式图案10.(FPKM)尺度为折叠变化。此外,通过使用DEGSEQ与调整的阈值集合重叠在两种比较中的相比之下来鉴定组织特异性基因p-Value <0.05和|日志2折叠变化|> 1 [42].
定量实时PCR(QRT-PCR)分析
为了通过实验验证我们的结果,选择了11个样品上的QRT-PCR分析的11个基因(用于种子的F2,茎的叶片,L2,用于茎的T2),具有三种生物学重复。表S中显示了11个基因及其引物对的细节4..根据还原辅助效率逆转录酶试剂盒(Thermo Scientific™)合成第一链cDNA。该实验是在CFX96实时系统(BIO-RAD,USA)中进行的GAPDH(甘油醛-3-磷酸脱氢酶)基因作为内部对照。每个PCR反应含有10μL的Sybr-Green主混合物和4pmol的每个引物,并在以下条件下设定扩增:初始步骤为95℃,3分钟,三步循环为95°C对于10 s,57℃,10 s和72℃,15秒,重复40次。使用ΔΔCT法测定基因定量。使用日志标准化相对表达式值2(fold change)计算RNA-seq与qRT-PCR结果的相关系数。
EST-SSR检测和底漆设计
利用MISA软件检测unigenes中潜在的SSR标记[43].默认参数设置为单核苷酸重复次数超过10X,二核苷酸重复次数超过6X,三、四、五、六核苷酸重复次数超过5X。引物3以默认参数为每个SSR设计PCR引物[44].PCR引物在清音生物科技公司(中国北京)合成。
SSRs验证和遗传变异
共收集了来自四大洲(北美、南美、欧洲和亚洲)十个国家的24份材料,包括野生材料、品种和育种状态不确定的材料(见表)6.).BCS1103和江夏种子由四川农业大学提供,其他种子来自美国国家植物种质系统。2016年和2017年,在四川农业大学(中国四川雅安)采用随机完全区组设计,将种子种植在盆内直至三叶期。每个地块行距为0.5 m,行距为0.5 m,每行可种植4株,共12株。将体积较大的样品与20个个体的新鲜叶片组织混合,并按照制造商的说明使用植物DNA提取试剂盒(中国北京天根)进行DNA提取。PCR扩增反应体积为15 μl,包含1.5 ng模板DNA, 1u Taq聚合酶,6 μM正反引物,7.5 μM Mix溶液。PCR反应循环曲线为95℃10 min, 95℃50 s,退火温度50 s, 72℃1 min, 35个循环,72℃10 min。将EST-SSR PCR产物装入6%尿素聚丙烯酰胺凝胶中,置于1X TBE缓冲液中,根据扩增的DNA片段大小,在200 V下电泳2 h。然后,凝胶经过2-3个初始洗涤步骤,然后用0.1%硝酸银染色15分钟。凝胶立即洗涤并固定在显影液中,以显示核苷酸带。
计算多态信息内容(PIC),标记指数(MI)和分辨率(RP)以评估SSRS的放大效率。根据公式计算每个底漆对计算PIC:PIC我 = 2f我(1−f我),其中PIC为标记i的多态信息内容,f我是存在的片段的频率,存在,“1 - f我“是缺点的片段的频率。PIC平均在每个引物对的碎片上。从Powell等人的公式计算MI。[45]:MI = PIC×EMR,其中EMR(有效多比率,EMR = PP×NPF)定义为多晶型基因座(PP)的产物和多态性片段(NPF)的数量。每个引物对的RP根据前静电和威尔金酮的公式计算[46]: Rp =∑Ib,其中Ib为片段信息量,计算为:Ib = 1 - [2 ×|0.5−p|],其中p为包含片段的组分比例。遗传多样性由不同等位基因数(Na)、有效等位基因数(Ne)、Shannon信息指数(I)和期望杂合度(He)测定,采用GENALEX 6.5版本[47].
计算成对(Dice)遗传距离,并利用矩阵在自由树软件中使用UPGMA (Unweighted Pair Group Method with Arithmetic, ungma)构建1000次bootstrap重复的聚类[48].此外,使用基于贝叶斯模型的群集方法使用结构软件(2.3.4版)将群组中的群组群集成群体[49[k-means聚类用于通过计算ΔK来识别最可能数量的遗传基团(k)[50].采用混合模型进行结构模拟,并基于首字母试验,在K = 2到K = 10的遗传簇范围内检验每个基因型的隶属度(每个独立运行10次)。对于每个K值,将老化周期设置为50,000次迭代,并在每次运行中对100,000个马尔可夫链蒙特卡罗重复进行估计。CLUMPP [51],使用DISTRUCT生成种群结构的图形显示(barplot) [52].
表型和Mantel分析
除了生物量产量和标题之日外,在2017年4月的全开花阶段收集了表型数据。从三个复制图中随机选择九种单个植物,用于观察11个形态特征:植物高度(pH,cm),茎直径(SD,mm),旗叶长度(FLL,mm),旗叶宽度(FLW,MM),倒数叶长度(PLL,CM),倒数第二叶片宽度(PLW,MM),第一节间(LFI,CM),分蘖号(TN),播种的天数(DSH,天),干物质产率(DMY,G /植物)和新鲜物质产量(FMY,G /植物)[37.)(计量方法见表S5.).使用NTSYS (version 2.2)和PAST (version 3.02)软件进行数据分析[53那54,包括描述性统计、PCA和UPGMA聚类。最后,利用NTSYS计算形态性状的欧氏距离矩阵,利用GENALEX 6.5版本的遗传距离和表型距离矩阵进行Mantel分析[47].
可用性数据和材料
目前研究中使用的原始测序数据可在国家生物技术信息中心(NCBI)序列阅读档案数据库获得,该数据库位于生物工程PRJNA670326 (https://www.ncbi.nlm.nih.gov).支持本文结论的其他数据集包含在文章及其附加文件中。相应作者可以获得任何合理的请求。
缩写
- AFLP:
-
扩增片段长度多态性
- COG:
-
同源群的群集
- 度:
-
差异表达基因
- EST-SSR:
-
表达序列标签-简单序列重复
- FPKM:
-
碎片每千碱基每百万
- 走:
-
基因本体论
- KEGG:
-
Kyoto基因和基因组的百科全书
- 密度:
-
马尔可夫链蒙特卡洛
- 门店:
-
新一代测序
- 主成分分析:
-
主成分分析
- RAPD:
-
随机扩增多态性
- UPGMA:
-
带算术的非加权对群方法
参考文献
- 1。
Williams WM, Stewart AV, Williamson ML. Bromus。出自:Kole C,编辑。野生作物亲缘:基因组和育种资源:谷子和草。海德堡:施普林格;2011.15 - 30页。
- 2。
马萨,Larson SR,Jensen KB,Hole DJ。AFLP变异蜡烛部分Ceratochloa.巴塔哥尼亚的种质。作物科学。2001;41(5):1609 - 16。
- 3.
Massa An,Jensen KB,Larson SR,Hole DJ。形态学变异蜡烛教派。Ceratochloa.巴塔哥尼亚的种质。可以j bot。2011; 82(1):136-44。
- 4.
Rugambwa VK, Holmes CW, Chu ACP。研究了季节和牧草质量对牧草营养价值的影响。马图亚草原和多年生黑麦草。中国科学(d辑:生命科学)1991;51:167-72。
- 5.
郑亚平,张海涛,张海涛,张海涛。番茄组织特异性转录组研究进展[j]。植物杂志。2015;168(4):1684 - 701。
- 6.
陈志强,陈志强,陈志强,等。萌发大麦籽粒中赤霉素表达及含量的转录和生化分析。[J] .中国生物医学工程学报。2020;71(6):1870-944。
- 7。
张W,张S,Lu X,Li C,Liu X,Dong G等。组织特异性转录组分析显示大象草中的木质纤维素合成调节(狼尾草purpurem.Schum)。植物生态学报。2020;20(1):528。
- 8。
赵飞,孙敏,张伟,姜超,滕建,盛伟,等。根、茎、叶的比较转录组分析Isodonamethystoides.揭示了参与王早津生物合成的候选基因。acta botanica yunnanica(云南植物研究). 2018;18(1):272。
- 9。
廖伟,梅志,苗林,刘鹏,高锐。水稻根、茎和叶组织的转录组比较分析Entadaphaseoloides揭示潜在的基因参与三萜类皂苷生物合成。BMC基因组学。2020; 21(1):639。
- 10.
胡H,Gutierrez-Gonzalez JJ,Liu X,Yeats Th,Garvin DF,Hoekenga OA等。遗传颞型基因表达模式与开发六倍体燕麦种子中的代谢组种子含量相关。植物Biotechnol J. 2019; 18(5):1211-22。
- 11.
刘铮,陈涛,马磊,赵智,赵px,南泽,等。利用Illumina平台对同源四倍体紫花苜蓿进行全球转录组测序及EST-SSR标记的开发PLoS ONE。2013; 8 (12): e83549。
- 12.
周强,罗东,马磊,谢伟,王颖,王颖,等。西伯利亚野生植物EST-SSR标记的开发及跨种可移植性研究(大sibiricus.L.)使用Illumina测序。Sci众议员2016;6:20549。
- 13。
张赵谢张Z, W, Y, J,王N, Ntakirutimana F, et al。基于rna测序的EST-SSR标记开发E..sibiricus.及其在17种植物系统发育关系分析中的应用大物种。植物学报。2019;19(1):235。
- 14。
Pan L,Huang T,Yang Z,Tang L,Cheng Y,Wang J等。基于RNA排序的EST-SSR标记表征洛利姆multiflorum与相关物种交叉可转移性。mol品种。2018; 38(6):80。
- 15.
刘k,徐h,刘g,关p,周x,peng h等。Qtl映射旗帜叶与小麦的特征(小子aestivum.L.)。Al Appl Genet。2018; 131(4):839-49。
- 16.
Puecher di,Robredo Cg,Rimieri P.遗传变异措施蜡烛catharticusVahl。群体和品种的RAPD和AFLP标记。Euphytica。2001;121(3):229 - 36。
- 17。
Ramakrishnan AP,Meyer Se,Waters J,Stevens Mr,Coleman Ce,Fairbanks DJ。分子标记与自适应显着的遗传变异之间的相关性蜡烛tectorum(Poaceae),近亲繁殖的年度草。我是J机器人。2004; 91(6):797-803。
- 18。
程X,王Y,Pang Y,Chen X,Wu J,Zhao J.Triticum Aestivum对SSR和EST-SSR标记的引物转移性分析蜡烛in.植物学报2014;32(1):27-33。
- 19。
冯国栋,黄磊,李军,王军,徐磊,潘磊,等。综合转录组分析揭示了果园草春化和花芽发育过程中不同的调控程序(dactylis.glomerataL.)。BMC植物BIOL。2017; 17(1):216。
- 20.
周胜,王超,陈鹏,陈志,等。第一个Illumina-based德诺维龙柏草转录组分析与分子标记发育(狼尾草purpurem.).摩尔品种。2018;38(7):1 - 14。
- 21.
Zuo C,Blow M,Sreedasyam A,Kuo RC,Ramamoorthy GK,Torres-Jerez I等。揭示了PACBIO长读测序对切换的转录组复杂性。Biotechnol生物燃料。2018; 11(1):170。
- 22.
王M,王p,梁f,ye z,li j,shen c,等。Allopolyploid棉中替代拼接的全球调查:景观,复杂性和监管。新植物。2018; 217(1):163-78。
- 23.
Collins M, Fritz JO。饲料质量。在:Barnes RF, Nelson C, Collins M, Moore KJ,编辑。牧草:草地农业概论。爱荷华州:爱荷华州出版社;2003.p . 363 - 90。
- 24。
Pontes Ls,Maire V,Schellberg J,Lou.F.草策略和草原社区对环境司机的回应:审查。Agron Center Dev。2015; 35(4):1297-318。
- 25。
王志强,王志强,王志强。木质素的生物合成。植物学报。2003;54(1):519-46。
- 26。
元J,Sun X,Guo T,Chao Y,Han L.全局转录族分析苜蓿揭示了六个关键的衰老生物过程。同行J. 2020; 8:E8426。
- 27.
Hundertmark M, Hincha DK。LEA (Late Embryogenesis abundance)蛋白及其编码基因拟南芥芥.BMC基因组学。2008;9:118。
- 28.
布雷JD。种子萌发和休眠。植物细胞。1997;9(7):1055 - 66。
- 29。
Nonogaki H, Bassel GW, Bewley JD。Germination-Still是个谜。植物科学。2010;179(6):574 - 81。
- 30.
王东,杨涛,刘锐,李宁,王旭东,Sarker A,等。扁豆SSR和KASP标记的RNA-Seq分析与开发镜头culinarisMedikus无性系种群。culinaris).作物J. 2020; 8(6):953-65。
- 31.
陈氏,黄X,yan x,梁y,王y,li x等。在羊草中的转录组分析(Leymus对):欧亚干草原的主要常年草。Plos一个。2013; 8(7):E67974。
- 32.
魏伟,齐旭,王磊,张永强,华伟,李东,等。芝麻的特性(胡麻属indigum.L.)使用Illumina成对序列测序和EST-SSR标记的发育的全局转录组。BMC基因组学。2011; 12(1):451。
- 33.
Parra-González LB, Aravena-Abarzúa GA, Navarro-Navarro CS, Udall J, Maughan J, Peterson LM,等。黄色卢平(羽扇豆山l转录组测序:分子标记的发展和比较研究。BMC基因组学。2012;13(1):425。
- 34.
Ribaut JM,Hoiseton D.标记辅助选择:新工具和策略。趋势植物SCI。1998; 3(6):236-9。
- 35.
Gupta p,varshney r,sharma p,ramesh bl。分子标记及其在小麦育种中的应用。植物品种。1999; 118(5):369-90。
- 36。
多种群遗传分析。在分子生态学。纽约:威利;2011.p . 129 - 77。
- 37。
Aulicino MB, Arturi MJ。阿根廷居群的表型多样性蜡烛catharticus(禾本科)。数量性状的遗传和环境组成。植物学报。2002;40(2):223-34。
- 38。
一种富含次生代谢物的植物组织的RNA分离系统。BMC Res Notes. 2011;4(1):85。
- 39.
戴维森N,Oshlack A. Corset:实现差异基因表达分析德诺维组装转录om。基因组Biol。2014; 15:410。
- 40.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I,等。从RNA-Seq数据中重建一个没有基因组的全长转录组。生物科技Nat》。2011;29(7):644。
- 41.
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12(1):323。
- 42.
Anders S,Huber W.序列计数数据的差异表达分析。基因组Biol。2010; 11(10):R106。
- 43.
Beier S,Thiel T,MünchT,Scholz U,Mascher M. Misa-Web:用于微卫星预测的Web服务器。生物信息学。2017; 33(16):2583-5。
- 44.
刘志强,王志强,王志强,等。初级3 -新功能和界面。核酸学报2012;40(15):e115。
- 45.
Powell W,Morgante M,Andre C,Hanafey M,Vogel J,Tingey S等人。种质分析RFLP,RAPD,AFLP和SSR(微卫星)标志物的比较。mol品种。1996年; 2(3):225-38。
- 46.
普及A,Wilkinson M.一种比较PCR引物应用于土豆品种的ISSR指纹识别的新系统。Al Appl Genet。1999; 98(1):107-12。
- 47.
Speall R,Smouse Pe。Genalex 6:Excel中的遗传分析。人口基因软件教学与研究。Mol Ecol Notes。2006; 6(1):288-95。
- 48。
HAMPL V,PavlícekA,Flegr J.基于DNA指纹识别的系统发育树的施工和自卷草引导分析与Freeware Program FreeTreee:应用于Trichomonad寄生虫。INT J SYST EVOL Microbiol。2001; 51(3):731-5。
- 49。
Hubisz MJ, Falush D, Stephens M, Pritchard JK。借助样本群信息推断弱总体结构。生态学报。2009;9(5):1322-32。
- 50。
基于多位点基因型数据的群体结构推断。遗传学。2000;155(2):945 - 59。
- 51.
Jakobsson M,Rosenberg Na。CLUMPP:用于处理群体结构分析中的标签交换和多模的集群匹配和排列程序。生物信息学。2007; 23(14):1801-6。
- 52.
罗森伯格na。Distruct:人口结构图形显示的程序。Mol Ecol Notes。2004; 4(1):137-8。
- 53.
哈默Ø,哈珀地方检察官,瑞安警局。过去:用于教育和数据分析的古生物统计软件包。Palaeontol电子乐。2001;4(1):9。
- 54.
Rohlf FJ。NTSYS-pc:数字分类学和多元分析系统-版本2.2。纽约州:埃克塞特软件公司;2005.
确认
作者感谢美国农业部国家植物种质系统(NPGS)提供的部分植物材料,即带有PI编号的材料。
资金
这项工作得到了中国国家自然科学基金(3177131276),是青藏高原高级饲料种质的重点实验室(2020年ZJ-Y03)和中国大学科学基金(2019TC257和2020TC189)。
作者信息
从属关系
贡献
x.m.、M.S.、S.J.构思设计作品;m.s.、w.w.、Z.D.和J.Y.进行了实验;m.s.、C.Z.和S.J.对数据进行了分析;M.S.写了手稿;许敏、尚杰、江孜一、江孜二、杨鑫对手稿进行了复核。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
作者遵守了所有相关的机构、国家和国际准则。
同意出版
不适用。
利益争夺
作者声明他们没有利益冲突。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
单基因和转录本的长度分布。
附加文件2:图S2。
基因表达模式。(a)基于log10的FPKM密度分布(FPKM)显示了基因在组织和发育阶段的表达水平。(b)在所有水平上检测不同组织下FPKM间隔分布。
附加文件3:图S3。
带注释的unigenesb . catharticus.(a)去分类。X轴表示每个GO类别内的子类别,Y轴表示特定类别的基因数;(b)Kegg分类。X轴表示分配给特定途径的基因的百分比,Y轴表示KEGG途径,包括蜂窝过程(A),环境信息处理(B),遗传信息处理(C),代谢(D)和有机体系统(E)。
附加文件4:图S4。
ESP-178和ESP-372引物对验证EST-SSRS扩增。裁剪两个凝胶图像以复合组合图,该图可以清楚地显示EST-SSR验证结果,并且在附加文件中提供原始和全长凝胶图像5..
附加文件5:图S5。
剖面型辅助施加零件多态性引物对的原始全长凝胶图像。附加号码在表中显示6.被标记在每个凝胶图像上,通过测序扩增的产品来验证由ESP-178和ESP-372的红色帧包围的带。标记在每个凝胶图像的两侧。
附加文件6:表S1。
单、二、三、四、五、六核苷酸的重复分布。
附加文件7:表S2。
52个相关EST-SSR标记详细信息。
附加文件8:表S3。
24的11个特征的描述性统计b . catharticus加入。
附加文件9:表S4。
Eleven基因及其引物的细节,用于QRT-PCR分析。
附加文件10:表S5。
形态特征名单b . catharticus:名称,缩写和测量方法。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
孙敏,董正东,杨军。等等。草原草转录组资源(叶片catharticus):表达的转录本,组织特异性基因,EST-SSR标记的鉴定和验证。BMC植物杂志21,264(2021)。https://doi.org/10.1186/s12870-021-03037-y
收到了:
公认:
发表:
关键词
- 叶片catharticus
- 转录组测序
- 差异表达基因
- 标记的发展
- 多样性分析