抽象的
背景
米 (奥雅萨苜蓿)细菌叶枯萎(BLB),由血疱引起的稻黄单胞菌PV。oryzae.(XOO),是影响全世界水稻生产的最毁灭性的疾病之一。抗性水稻品种或基因的开发和使用是目前控制BLB最有效的策略。
结果
在这里,我们使用了259种稻米净化,其基因分型用2 888 332高置信单核苷酸多态性(SNP)。结合259米线的电阻变化数据XOO在2年内观察到的种族,我们进行了一种基因组 - 宽的协会研究(GWA),以鉴定赋予植物阻力的定量性状基因座(QTL)对抗BLB。在病原体接种后的四个时间点,还通过转录组分析在抗干和敏感的稻米之间鉴定在GWAS结果中的基因的表达水平。从该109候选抗性基因揭开显示抗性和易感水稻系之间的显着差异表达的抗性基因。此外,单倍型嵌段结构分析预测基于CHR的短式耐抗体的候选基因。7_707158最小P.-Value(-Log 10P. = 9.72). Among them, two NLR protein-encoding genes, LOC_Os07g02560 and LOC_Os07g02570, exhibited significantly high expression in the resistant line, but had low expression in the susceptible line of rice.
结论
研究结果为水稻BLB抗性育种提供了重要的遗传基础。
背景
米 (奥雅萨苜蓿)为世界人民提供大约20%的饮食能源供应[1]. 然而,水稻白叶枯病(BLB)严重威胁着世界水稻生产稻黄单胞菌PV。oryzae.(XOO) [2].XOO现在在大米的稻米地区普遍存在,但由于其宿主的能力,这种细菌也威胁到南美洲和亚洲的小麦产量[3.那4.].在印度和菲律宾,这种疾病的危害已导致水稻生产损失20%-30%,达到80%-90%的破坏性水平[5.那6.].一般来说,这种伤害在分蘖期开始,随着疾病发病率随着开花阶段的宿主植物生长达到峰值而变得更广泛[7.].预防速度的最简单方法是应用化学杀虫剂;然而,大规模使用各种杀虫剂威胁着米饭食品的安全性。此外,由于BLB迅速传播,因此在季风气候中施加的这种化学对照最终是不可思议的,因为一旦大规模发生了肺泡侵袭,即如果不是不可能的,它的有效控制很难困难[8.].因此,开发和施加耐水稻品种是控制这种疾病的最有效的方法,并确保粮食安全。
迄今为止,在水稻植物中检测到超过42例抗抗性基因和数百个抗性基因座(QTL)。在这些基因中,一些来自野生物种的一些[9.那10那11].通过突变培养的水稻线发现了其他抗性基因或等位基因[12那13];例如Xa1型那xa3 / xa26,xa21那XA23那XA27那xa5型那XA13那xa25., 和XA41[14那15那16那17那18那19那20那21那22]. 此外,据报道,BLB R基因的全谱由16个基因组成:xa5型那xa8型那xa9型那XA13那XA15那XA19那XA20那XA24那xa25.那xa26b型那XA28那XA31那XA32那XA33那XA34, 和xa42[19那22].重要的是,BLB具有高竞争特异性,并且含有单抗性基因的水稻线中的短植物抗性的快速损失仍然是育种者的压迫问题。组合多种电阻基因或QTL可以有助于广泛的频谱和耐用性XOO这是有效的[23]. 因此,在许多水稻品系中检测新的抗病基因和qtl,是成功培育抗BLB水稻的关键。
大多数BLB抗性的研究都集中在单亲或双亲抗性遗传图谱群体上。基于高密度单核苷酸多态性(SNPs)和下一代测序的全基因组关联研究(GWAS)已被广泛应用于植物的许多农艺性状和抗病基因座的遗传定位[24那25].例如,李等人。(2015)通过GWAS方法检测到与抗耐抗条纹锈病相关的97个底座;和赋予耐受性的基因大丽花黄萎病在棉花中被GWAS鉴定[26].对于水稻,研究人员使用GWAS检测了27个与稻瘟病抗性相关的位点[27]. 然而,由于标记密度不足和连锁不平衡,GWAS不能在给定的位点上提供准确的目标基因。转录组分析可以通过检测和区分不同基因型候选基因的表达来克服这一限制。最近,Wen等人[28通过组合转录组和GWA方法,鉴定了一组与大豆豆中的白色耐塑性相关的候选基因。
在这项研究中,259条米线接种了两条XOO分别测定P3和P6两个小种对BLB的抗性。我们使用288332个高置信度SNP(缺失数据)进行BLB抗性的GWAS < 20%; 次要等位基因频率 > 1%). 在此基础上,我们通过分析最抗病水稻品系NSIC RC154和最感病水稻品系CT 9737–6-1-3P-M在感染后5个时间点(0、12、24、48和72h)的转录组,探索候选抗病基因。我们的结果使我们能够在这些水稻品系中检测到与BLB抗性相关的候选基因。这为水稻抗病育种提供了重要的基因资源。
结果
基因组变异和人口结构
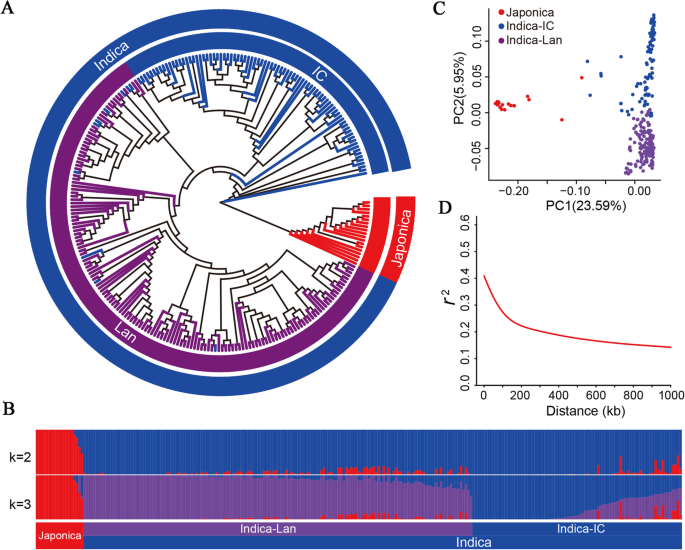
为了寻找Blb抗性基因,使用Illumina Hi-SEQ平台测序259种水稻净化(图。1A.表S1).这产生了1.33 TB的原始读取(表S2)和2 888 332高频体SNP(缺少数据<20%;映射到Nipponbare Rice Genome(表)后获得次要的次数频率[MAF]> 1%)(表S3).在2 888 332高置信SNP中,分别有146 191,555 884,363 883,464 911和318 546,SNP分别位于非基因区域,外显子,上游区域,内含子和下游区域(表S3).在编码序列(CDS)中,找到16 416终止增益,312 857非同义词,2 850拼接和1 085终止SNP(表S3).12米染色体中的SNP的数量范围为334 353(CHR.1)至192 058(CHR.9),并且在CHR上发现了最高的SNPS频率。8(8.50 snps / kb)(表S4).该SNP数据集259系列提供了一种充足的资源,用于水稻耐血液的分子改善。
人口结构和系统发育和系统发育树分析表明,这259种水稻品种含有三个亚组(K = 3):Landrace indica品种(94),改善的籼稻品种(146)和粳稻(19)(图。1b)。根据这259条稻米的主要成分分析(PCA),在前两种PC上解释了29.54%的遗传变异(图。1C)。这些结果表明,本研究中使用的水稻品种含有丰富的水稻核心种质遗传变异。259种水稻品种的LD衰变估计为194 kB(图。1d),建议稻米表现出中度LD [29].
水稻品种中的表型变异
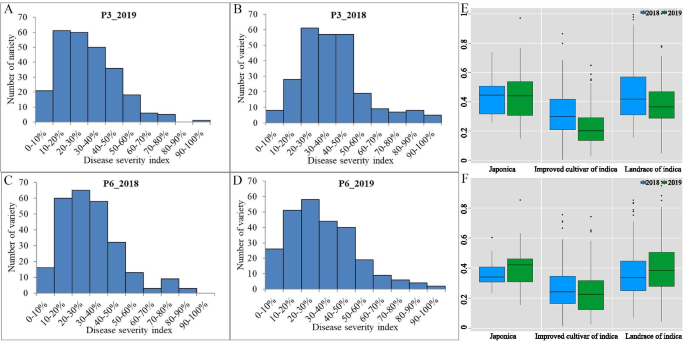
所有259条水稻线都接种了两个XOO在引导阶段进行比赛(即p3和p6),并且在2年内调查了每种抗性表型。对于p3.XOO2018年,所有品系的BLB疾病严重指数(DSI)在0.26%至99.46%之间(平均值) = 从2.82%到2019年的100%(平均 = 30.34%). 对于P6,相应的BLB DSI值为1.46–85.32%(2018年;平均的 = 30.92%和2.49–97.84%(2019年;平均的 = 31.92%(图。2广告;桌子S1). 在不同的水稻品系中观察到的广泛的BLB-DSI值(两个品系的差异分别为16倍和12倍)XOO比赛)显示了大量基因型可变性与稻米抗性有关。另外,两种种族的BLB发生率正常(图。2广告)。两种种族的BLB DSI的差异(ANOVA)分析揭示了基因型中的显着差异,暗示了赋予短置底基因座的存在。至关重要的是,在改善的品种和地板中的最低水位中,BLB抗性水平最高(图。2e,f)。后一种结果表明,人工选择在水稻育种应用中取得了成功。
Gwas用于细菌叶枯燥
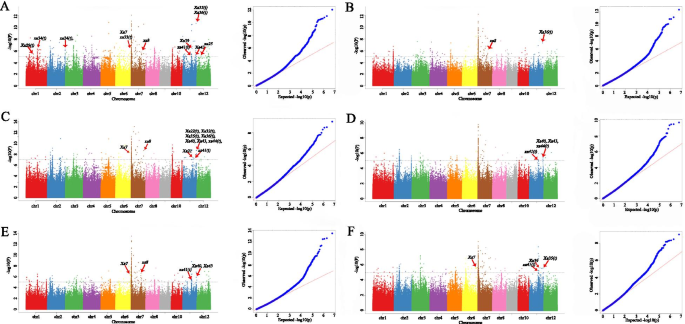
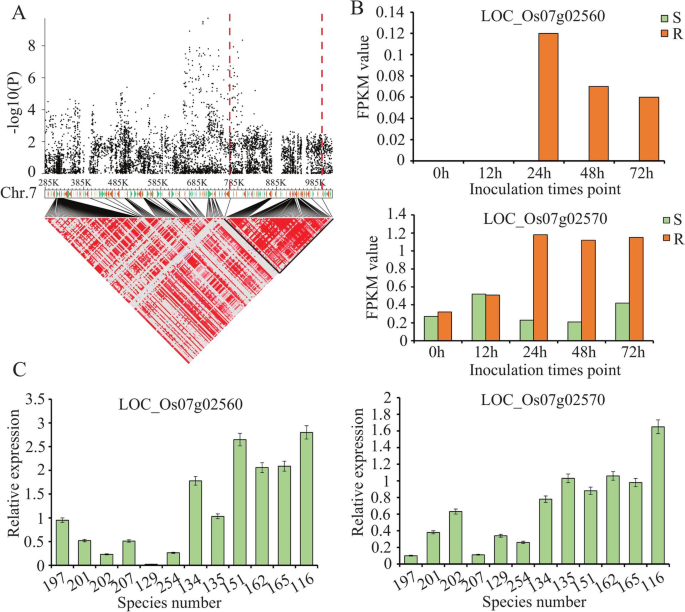
基于2 888 332高频率的SNP,两个抗性特征的GWA(每个XOO采用比赛选择不同植物特征)通过混合线性模型(MLM)进行。对于GWAS分析,使用这两个特征的表型数据(2年)和每年每个性质的最佳线性无偏的预测(BLUP)值。共有196和164个SNP基因座分别与P3和P6的抵抗显着相关( - log10P ≥ 5) (图。3.;桌子S5那S6那S7那S8那S9.和S10).有趣的是,它们的63个SNP基因座对所有特征都是共同的。接下来,将GWA应用于所选的240种籼稻。同样,使用了两个特征的表型数据及其年度增额值。在重要的关联SNP中,与为所有259种稻米品种实施的GWA的结果相比,CHR.7也识别出最强的信号(图S1).然而,一些已知的基因座未出现在240个籼稻品种的GWAS结果中(图。3.和图S1).因此,19种粳稻品种可以有助于BLB GWA,因此进一步研究了所有259种水稻品种的GWAS结果。根据水稻基因组的LD衰减,在显着相关SNP的上游和下游内检测候选基因在200kb的上游和下游(图。1d)。对于P3,检测到总共2081个基因(2年和2岁和增孔值),同样地检测到P6的1 297基因。其中,954个基因均为抗性特征(表格)S5那S6那S7那S8那S9.和S10).这些基因的数量在CHR中较高。7和chr。11,与其他水稻染色体相比(表S5那S6那S7那S8那S9.和S10).
我们总结了41先前报告了使用oryzaSAse数据库(以下)与BLB电阻相关的细映射QTLS或基因(http://www.shigen.nig.ac.jp/rice/oryzabase/gene/list.).为了进一步确认与BLB相关的明显基因或SNP,将结果与先前报告的细映射QTL或基因的结果进行比较。在P3菌株中检测到的基因座,鉴定了六个先前QTLS细映射QTL或基因(图。3.A-C)。在P6菌株中检测到的基因座中,有7个SNP与先前报道的QTL或基因相邻(图。3.D-F)。这证实了我们的结果是可靠的,具有更强探索水稻的新抗性基因的潜力。
抗性NSIC RC154的转录组和易感CT 9737-6-1-3P-M系列的比较
由于人口结构强,大米的较大程度的LD,Gwas鉴定的基因座通常落入基因沙漠中或具有许多同样合理的致病基因的地区。这使得难以稳健地识别功能基因。将GWA的结果与RNA-SEQ数据相结合,用于检测功能基因。
此外,先前的报道还表明,在不同抗性系中存在不同的表达水平的基因最有可能与抗病抵抗关系[28].
在2年的田间试验259个品系中,NSIC RC154 (DSI = 6.01%)和CT 9737-6-1-3P-M (DSI = 100%)分别是抗性最强和感病最强的品系。为了进一步鉴定与BLB抗性相关的基因,我们对接种后12、24、48和72 h的两个品系叶片进行了转录组分析XOO。总体而言,我们分别获得2,080,282,498清洁读数(表S11),分别为NSIC RC154和CT 9737-6-1-3P-M的映射读数值为91.89-94.29%和91.81%-94.37%(表S11). 抗性系NSIC RC154和感病系CT 9737–6-1-3P-M表现出明显不同的反应XOO接种;NSIC RC154中的差异表达基因(DEGS)的数量(11674; 3 167上调和8 507调节)(表S12)超过CT 9737–6-1-3P-M(11 436;2929上调,8254下调)(表S13). 在这些DEGs中,虽然两个水稻品种在不同时间点共有8851 DEGs,但只有在抗性系NSIC RC154中发现了2817 DEGs(表1)S14). 因此,后一种基因可能在BLB抗性中占有重要地位。在接种初期(12h和24h),抗病株NSIC RC154中检测到9023个DEGs(2477个上调,6546个下调),而感病株CT 9737–6-1-3P-M中检测到8040个DEGs(2201个上调,5839个下调)。当受到病原体的攻击时,NSIC RC154在早期阶段比CT 9737-6-1-3P-M含有更多的DEG。这表明NSIC RC154比CT 9737–6-1-3P-M对病原体的攻击和入侵反应更强烈。我们推测nsicrc154可能有许多与耐药性相关的基因,这些基因对病原体感染有快速反应。
gwas鉴定基因的表达分析
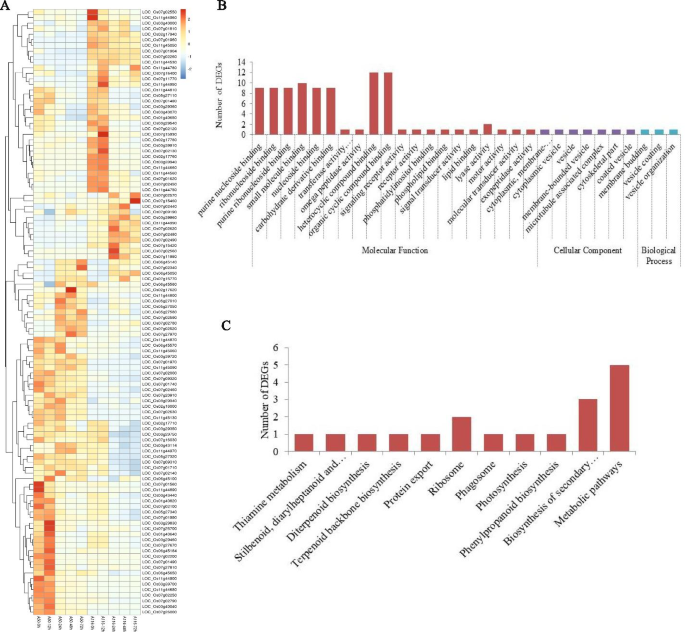
954个基因中发现与两者抗抵抗显着相关XOO抗性水稻nsicrc154中有161个小种(36个上调,125个下调)为DEGs,感病水稻ct9737-6-1-3P-M中有197个小种(64个上调,140个下调)。此外,还检测到一组109个DEGs在抗性和感病水稻系之间进行了显著差异表达(图。4.一种)。基因本体(GO)分析揭示了几种功能类别的DEG的富集富集,即信号传感器活性,嘌呤核苷结合,受体活性,电动机活性和分子传感器活性(图。4.B) 是的。KEGG(京都基因和基因组百科全书)注释表明,富含这些DEG的途径与苯丙酸生物合成、次级代谢产物生物合成、光合作用、二萜生物合成以及二苯乙烯类、二芳基庚类和姜辣素的生物合成密切相关(图。4.C) 是的。根据这些结果很难阐明BLB抗性的具体机制;然而,这些DEGs是非常重要的,因为它们很可能是增强BLB抗性的候选基因。
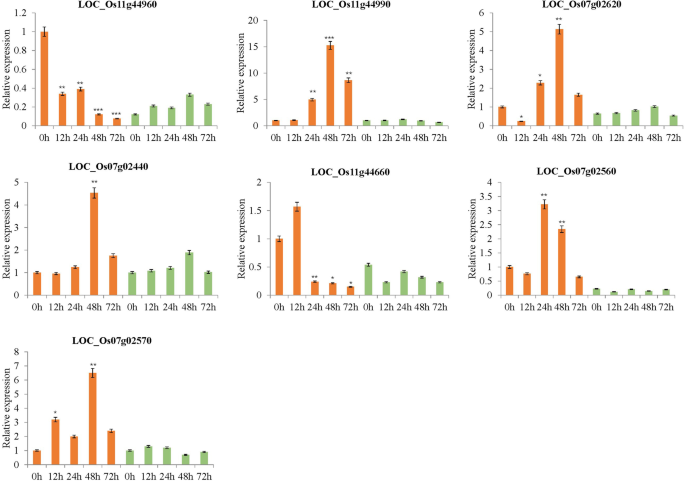
此外,在抗性和敏感的稻米之间具有显着差异表达水平的109°中,近半(45)的抗性比敏感线更大(图。4.一种)。这45个基因含有大量的防御相关蛋白质编码基因,例如编码基因的NBS-LRR抗病蛋白位置Os11g44960那位置:Os11g45050那位置:Os07g02570那loc_os11g44990.那loc_os07g02620, 和LOC_Os07g02560。众所周知,NBS(核苷酸结合状态)-LRR(富含亮氨酸的重复)抗性蛋白质批判性地参与植物 - 病原体相互作用,其NBS在该蛋白的调节活动中发挥关键作用[30].也许更重要的是,六种NBS-LRR疾病抗性蛋白质编码基因,位置:Os07g02570那loc_os11g44990.那loc_os07g02620, 和LOC_Os07g02560显示在24 HPI的抗性线上诱导上调,而在易感线之后,在易感线中没有显着变化。XOO接种。通过触发植物基础防御反应,反应性氧物种(ROS)在植物中是重要的[31].过氧化物酶前体编码基因,loc_os07g02440.,在24hpi的抗性线上上调,但在测试的所有时间点在易感线中诱导(图。4.一种)。该基因的表达意味着ROS可能是关键涉及对抗的XOO病原体早期在米饭感染。
此外,抗性相关的巨噬细胞蛋白(NRAMP)的抗体基因loc_os07g15460.,糖基水解酶系列3蛋白编码基因loc_os11g44950和细胞色素p450编码基因LOC_Os02g17760与易感线相比,所有接受耐药线中的表达明显更高。根据其他研究,铁是大多数生物体的关键因素,病原体可能与他们的主持人竞争[32]. 植物病原菌Dickeya Dadantii.依赖于其在几个宿主植物上的系统性疾病进展的纵向介导的铁摄取系统,包括拟南芥蒂利亚纳[33].在水稻植物中,几种金属离子如Zn2+,锰2+、铁2+和CD.2+通过NRAMP转运蛋白转运[34].在我们的研究中,NRAMP编码基因loc_os07g15460.在72 HPI的抗性线上发现了上调,但它在易感线上没有显着的表达变化。因此,该基因可以通过铁转移导致耐血压。此外,三种蛋白激酶编码基因(loc_os11g44660.那loc_os11g44560, 和loc_os07g02450.)被抗性线下调XOO接种,但在检查的任何时间点都没有在敏感线的转录组中诱导它们的表达水平。进一步的研究应尝试在功能上验证这些基因的效果,这是揭示水稻中复合短叶抗性特性的分子机制所需的效果。
QRT-PCR的表达验证候选基因
选择七种候选基因用于验证RNA-SEQ数据。这些由六种编码的NBS-LRR疾病抗性蛋白组成(位置Os11g44960那LOC_Os07g02560那位置:Os07g02570那loc_os11g44990., 和loc_os07g02440.)和一种蛋白激酶基因(loc_os11g44660.) (图。5.).每个基因的阈值周期(Ct)值相对于UBQ基因(内对照)归一化。采用qRT-PCR方法检测这些基因的相对表达量,并与转录组结果进行比较。结果表明,这些基因在抗性品系中表达差异较大XOOP6应变接种,以与RNA-SEQ数据一致的方式(图。5.).
通过单倍型和表达分析鉴定新的短曲抗基因
在这项研究中,在chr7上识别的最强信号是新的(图。3.),并且位于Chr.7上的SNP区域含有许多编码NBS-LRR抗性蛋白的基因。使用成对LD相关估计候选区域。我们专注于轨迹从0.747映射到0.981 MB,具有58个候选基因(图。6.A) 是的。在这些基因中,转录组数据发现了两个NLR蛋白编码基因,LOC_Os07g02560和位置:Os07g02570,其在抗性线中显着高表达,但在敏感的水稻中表达了低表达(图。6.B) 是的。确认LOC_Os07g02560和位置:Os07g02570在抗平台抗性中,选择六种抗性和六种敏感的品种以检查表达水平LOC_Os07g02560和位置:Os07g02570接种后XOO第6页。在24hpi时,这两个基因在抗病品种中的表达高于感病品种(图1)。6.C) 是的。我们还分析了这两个候选基因在抗病品种(NSIC RC154)和感病品种(CT 9737–6-1-3P-M)中的序列差异。我们发现了4个SNPs(Chr.7922336,T-C;第7章,T-C;第7章,A-G;7号染色体(922694,G-A)和6个单核苷酸多态性(7号染色体(920121),C-A;Chr.7920184,T-C公司;第7章,G-A;第7章,G-A;第7章,C-T;编码区含有Chr.7(920861,T-G)。对于LOC\ u Os07g02570,两个SNP(Chr.7\ u 927311,G-A;和两个单核苷酸多态性(Chr.7928738,A-G;检测编码区Chr.7(926479,A-G)。这些结果表明,这两个基因与BLB的抗性密切相关,有望成为该病的候选抗性基因。
讨论
对于259种米饭,我们将它们的抵抗水平评估为2年。结果表明,并非所有水稻群均同样耐药。此外,在259条稻米的抗性水平中,两者之间存在显着差异XOO经病原体种族测试,表明具有特异性抗性的R基因仅被某些水稻品种携带。这一发现与张等人的较小研究的发现一致。[35],其中六个抗米能水平在六个中有显着差异XOO后者根据病变的大小(长度值)分为三组。从各株系的发病率可以推断出各株系的抗性水平:< 10%区分抗性株系。在259份水稻材料中,有6个株系(IR 10M126、NSIC RC154、CT 16658 - 5-2- 3sr -2- 1- mmp、CT 15765-12-1 - 1-4-2 -1 - m、CT 15765-13-3-8 - 3-3-3 - m、IR61009-37-2-1-2 -2和HHZ 12-DT 10-SAL 1- dt 1)对这两种水稻均具有较高且稳定的抗性XOO比赛。这些结果表明了选择适当的材料,用于加速基础抗性育种和水稻的遗传学研究。
GWA是检测复杂性状的功能基因的重要方法。它已被用于检测与抗病抗性相关的新基因和植物中重要的农艺性状。例如,在玉米中,李等人。[36]使用gwas照亮的作用ZMFBL41编码F箱蛋白质的,在那种玉米植物对鞘枯萎的抗性。在该研究中,我们确定了63例BLB抗性基因座,含有954个显着相关的基因,尽管GWA为2,888,332个高频体SNP。发现了五种重要的SNP,即CHR。12_17579641,chr。11_28745675,Chr。11_21379864,chr。08_27163888和Chr。11_17915331分别位于克隆的R基因附近xa25.[37],XA26.[14],XA21[38],XA23[39],XA33.[40],XA13[16],和OS-11N3.[41] (图。3.).这些结果表明,使用相对较大的人群时可以实现相对高的GWA分辨率,从而大大加强了遗传多样性的调查和稻米的高密度SNP地图的产生。通过成功将GWA与转录组数据结合以发现水稻中的Blb抗性候选基因,在染色体7(Chr.7_707158)上鉴定了与P3和P6两者的耐水稻抗性有关的新型基因座。我们的数据为BLB电阻的未来基因功能研究提供了重要信息。预计这些发现可以作为函数基因发现在水稻和其他植物物种中的复杂性状作用的稳健参考。
与敏感线CT 9737-6-1-3P-M相比,在病原体接种后,抗性线NSIC RC154在早期点处具有更高调节的基因。此外,在组合GWA和转录组数据中,获得了109个与BLB抗性DEG的显着关联。将这些基因分配给Kegg功能注释后,发现仲成代谢物,光合作用,二萜类生物合成和斯蒂屈剂律,二芳肽和姜酚生物合成的苯基丙醇生物合成,生物合成,生物合成的途径全部富集。苯丙烷化合物在植物防御中起关键作用,从组成型或诱导物理和化学屏障免受致病感染的影响,作为参与局部和全身信号传导的信号分子,用于诱导一个或多个防御基因[42]. 丰富的苯丙酸生物合成表明,次生防御代谢产物在水稻对BLB的早期抗性中占有重要地位。然而,其他途径,如二萜生物合成和光合作用,也可能参与植物抗病性。例如,OSCY71Z2通过调节二萜植物植物的生物合成来治理Blb抗性[43].我们的研究结果表明,耐水稻抗性是一种相当复杂的特征,因为它取决于多种防御途径的良好协调和激活的网络。
许多BLB抗性基因已被寻找并应用于水稻育种[44].虽然有些人XOO现在已知抗性基因,大多数是特异性抗性基因;例如。,xa25.那XA26., 和Xa1型[30那45那46].因此,鉴于通过演变引起的植物抗性减少XOO因此,寻找新的抗性基因,并将其与已知的抗性基因结合,培育出持久、可持续的水稻抗性品系势在必行。在本研究中,发现多个NLR基因定位于Chr.7707158位点,其中几个NBS-LRR基因也聚集在一起。通过对NBS-LRR基因的单倍型和表达分析,得到了NBS-LRR基因簇(LOC_Os07g02560和位置:Os07g02570)这赋予了水稻对这两种作物的广谱抗性XOO赛跑被揭露了。但是所有这些突变实际上是否都推动了植物抵抗力的损失XOO需要进一步仔细调查。此外,植物基因组通常会编码数百个参与防御反应的NLR蛋白,其中一些蛋白在基因复制和扩增事件后发生在特定位点的集群中。以前的报告表明,基因连接的NLR基因可能共同作用,以识别病原体的无毒效应,如rps4./RPS1.那RGA4/RGA5, 和PIKP-1/PIKP-2。在这些对中,一种基因用作感知病原体效应器的“传感器”,而另一个基因是激活免疫信号所需的“辅助者”[47那48那49].同样的,LOC_Os07g02560和位置:Os07g02570在LD块中共同定位。然而,r-cluster中的这两个NBS-LRR基因是否受到相同的监管机制rps4./RPS1.那RGA4/RGA5或者PIKP-1/PIKP-2是未知的,并且可以进一步研究。
结论
我们整合了本研究的GWAS和转录组结果,以提供一些新的、有用的水稻白叶枯病基因资源。两个候选基因LOC_Os07g02560和位置:Os07g02570,由此获得。这些发现应提供用于评估候选基因的可靠目标,用于用于育种的育种。然而,仍有待完成更多的工作,以验证哪些额外的基因在水稻中抵抗抗性。
方法
植物材料和表型评价
本研究以259个水稻品系为材料。这些品种来自不同的国家,包括塞内加尔、中国、马来西亚、巴西、哥伦比亚和墨西哥。其中,国际水稻研究所(International Rice RealInstitute,IRI)提供了146条生产线,其余113条由中国四川农业大学水稻研究所保存。259个水稻品系的信息见表S1. 为了评价水稻对BLB的抗性,将259个水稻品种的种子在温室内播种。30天后移栽到四川农业大学的试验田,每行10株。
我们用了两个代表XOO菌株p3和p6以人为地接种植物。在30℃下单独培养菌株在蛋白右旋糖氧糖(PDA)培养基上2天;每个XOO种族暂停使用无菌水以108.细胞ml.-1作为接种物。在米饭分蘖阶段,每种米品种的最上层叶子中的15个被感染了两种XOO使用叶片剪切方法的比赛[50]. 在接种后14天,当病灶很容易看到时,测量病灶长度。每行记录BLB疾病评分,即病斑长度除以叶长。每个品种的平均病分是根据15片叶子计算的。数据采用microsoftexcel2010进行处理,在spssv16.0(IBM公司,Armonk,USA)中采用方差分析和Dunnett多重比较检验对不同水稻品种或亚群体的BLB值进行统计分析。
DNA提取和测序
对每个水稻品种21日龄幼苗的幼叶进行DNA提取。采用十六烷基三甲基溴化铵(CTAB)法提取基因组DNA[51].通过纳米光度计分光光度计(Implen,Ca,USA)和Qubit DNA测定试剂盒分别测定DNA的纯度和浓度,其具有Qubit 2.0 ulourometer(Life Technologies,USA)。所有259种水稻品种的DNA样品首先通过超声处理到350bp片段来破碎。这些DNA片段在终抛光,尾的末端和连接到全长适配器后进行测序。接下来,获得具有150bp读取长度的原始序列。在原始数据中,删除了包含适配器序列的读取的读取,或者具有低质量分数。高质量的配对末端读数被映射到Nipponbare Rie Genome(ftp://ftp.ensemblgenomes.org/pub/plants/release_36/fasta/oryza_indica/dna/)使用Burrows Wheeler软件工具和命令“mem-t 4-k 32–M”[52]. 在它们对齐后,通过基因组分析工具包(GATK)软件中的单倍型调用程序模块和GVCF模型来确定每个加入的GVCF格式的基因组变体[53].然后将所有GVCF文件合并在一起。然后通过这些参数过滤原始基因型文件:每个单独的深度≥4;次要等位基因频率(MAF)≥0.01,每个单独的基因型质量≥4;和;错过率≤0.2。以这种方式,总共获得2 888 332个SNP,并使用Annovar软件进一步注释(V2013-05-20)[42].这些SNP根据其注释分为五组:CDS SNP,上游SNP(位于转录开始点的1 KB内),基因间SNP,下游SNP(位于转录停止位点的1 KB内),以及内部SNP。
PCA,人口结构和LD分析
邻居加入(NJ)树通过了P.- 使用树最佳软件(V1.9.2)和1000个引导复制中的2,888,332个SNPS [54].通过程序混合物确定259条稻米的人口结构(V1.23)[55], k值范围为2 ~ 3。然后使用GCTA软件进行主成分分析(PCA) [56]. 为此,首先通过参数“–make grm”计算遗传关系矩阵;前三个主成分由参数“pca3”得到。用皮尔逊相关系数(r2)通过“POP-LD-Decay”软件工具计算成对SNP之间[57],其程序参数设置为“-Maxdist 1000 kB-Maf 0.05 -miss 0.1”。平均值R.2在1-kB窗口中的成对标记计算值,并且这些值在整个水稻基因组上平均值。
育种价值的估计
使用R计算平台中的“LME4”封装(v.3.2.2)中的“LME4”包来计算繁殖值(最佳线性无偏见的预测器)[58], 如下:
在哪里,呢y那μ.那线, 和座垫分别是表型、截距、品种效应和环境效应。的rep是重复次数,ε表示随机效应;的线 × 座垫术语指的是多样性和环境之间的相互作用,而rep × 座垫是复制与环境之间的互动。
GWAS分析
在GWA中仅使用具有测序深度≥4,缺失率<0.2和MAF≥0.01的SNP,后者使用EMMAX(Beta版本)软件包进行分析[59].使用EMMax计算的成对遗传距离的基质形成了随机效应的方差 - 协方差矩阵。
转录组分析
为了进一步鉴定定位在GWAS基因座周围的候选抗性基因,培育了抗性水稻品种NSIC RC154和感病品种CT 9737–6-1-3P-M(均已确认),并接种了毒性更强的菌株XOO叶片剪切方法中的温室中的比赛P6 [50].从每种水稻品种,分别在12,24,48和72 HPI获得叶样样品,每次治疗都有三种重复。还收集了12小时的非接种,新鲜叶片的对照样品。将所有叶片样品放入液氮中并储存在-80℃以进行RNA分离。根据制造商的说明,用植物总RNA隔离套件(上海桑龙生物科技)的植物全部RNA(Sangon Biotech)分离出来。我们使用NebNext Ultra™RNA库预备套件,用于illumina(Neb,USA),用于RNA-SEQ库建设。使用Illumina Hi-SEQ平台测序,并产生125-BP配对读数。在原始数据中,除去具有低质量分数的读取和包含适配器序列和延伸的-NS的读数。使用Bowtie V2.2.3建造了Nipponbare Rice参考基因组的指数,上述配对末端读取使用TOPHAT v2.0.12对齐[60.那61.那62.]. 为了计算每个基因的读取次数,使用了htseqv0.6.1软件[63.].使用Cuffdiff软件(V2.2.1)计算的基于FPKM(每百万百万百万百万千碱基的片段)存在每个基因的表达值。
使用“DESeq”软件包(v1.18.0)在R中进行两种处理(每个处理包含三个生物复制)的差异表达分析[64.].基于负二氯化分布测定了两种治疗中基因的差异表达水平。Benjamini和Hochberg的方法被用来调整P.- 用于控制虚假发现率(FDR)的值。基因与|原始转化|> 1和调整P.<0.05的值被指定为差异表达[65.].
单倍型分析
通过使用置信区间方法来区分单倍型块[66.,以及Haploview软件[67.].为此,Hardy-WeinbergP.-Value截止设定为0.001,MAF为0.05。
QRT-PCR.
利用qRT-PCR技术研究了7个候选基因在水稻植株中的相对表达水平。总RNA提取和反转录如前所述进行。PCR反应采用20μL体积,其中cDNA模板3μL,正向和反向基因特异性引物0.8μL。每个PCR集重复4次。泛素(UBQ)基因被用作数据标准化的内部对照。2号-ΔΔct使用方法计算基因表达水平。本实验中使用的引物在表中提供S15。
可用性数据和材料
在Bioproject SRR11747259-11,747,279(针对RNA-SEQ)和BiopoMe重新排序的BiopoMeject Prjna598020(用于基因组重新测序)下,沉积在国家生物技术中心(NCBI)序列读取档案(SRA)中沉积在国家生物技术中心(NCBI)序列读取档案(SRA)。
缩写
- BLB:
-
细菌叶会枯萎
- xoo:
-
Xanthomonas oryzae pv。oryzae.
- SNPS:
-
单核苷酸多态性
- GWAS:
-
基因组协会研究
- QTL:
-
数量性状基因座
- LD:
-
连锁不平衡
- 光盘:
-
编码序列
- 主成分分析:
-
主要成分分析
- DSI编号:
-
疾病严重程度指数
- 方差分析:
-
方差分析
- MLM:
-
混合线性模型
- 结影:
-
最好的线性无偏见预测
- 可见:
-
差异表达基因
- HPI:
-
H后接种后
- 去:
-
基因本体论
- Kegg:
-
京都基因和基因组百科全书
- ROS:
-
活性氧
- NJ:
-
邻接
- FDR:
-
假发现率
- FPKM:
-
每百万个转录物序列的每千碱基片段数
参考
- 1.
陈无,高啊,谢W,龚朗,陆K,王Ws等。基因组 - 范围协会分析为水稻代谢的自然变化提供遗传和生化洞察。NAT Genet。2014; 46(7):714-21。
- 2.
Nino Liu DO,Ronald PC,Bogdanove AJ。稻黄单胞菌致病变种:模式作物的模式病原。植物病理学。2010;7(5):303–24.
- 3.
Khush GS,Mackill DJ,Sidhu GS。育种水稻抗细菌枯萎病。在:Banta SJ,编辑。细菌枯萎水稻。LOS Banos:国际大米研究所;1989. p。207-17。
- 4.
张Q.遗传评价与利用耐水稻细菌枯萎病。科学科学罪。1991; 24(2):26-36。
- 5.
Saha S,Garg R,Biswas A,Rai A.细菌疾病稻米:概述。j纯苹果微生物。2015; 9:725-36。
- 6.
Srinivasan B,Gnanamanickam SS。鉴定野生稻中抗性的新源,oryza rufipogon对印度xanthomonas oryzae pv引起的稻米细菌枯萎病。oryzae。Curr SCI。2005; 88:1229-31。
- 7。
小川T。白叶枯病(xanthomonascampetrispv.)小种分布监测及抗性基因鉴定的方法和策略。(米饭)。日本农业研究所。1993;27:71–80.
- 8。
Lee Ks,Rasabandith S,Angeles Er,Khush GS。21种稻米细菌枯萎病的遗传。植物病理学。2003; 93:147-52。
- 9。
Busungu C,Taura S,Sakagami J-I,Ichitani K.来自XM14的新型稻米细菌抗性基因的鉴定和连杆分析来自IR24的突变线。品种sci。2016; 66:636-45。
- 10。
陈氏,黄泽,曾升,杨姬,刘Q,朱x高分辨率映射与Xanthomonas oryzae pv的基因预测。Oryzae抗性基因Xa7。mol品种。2008; 22:433-41。
- 11.
江痛,夏泽,周捷,万俊,李诗,陈卢斯,等。用遗传互补作用遗传互补稻米细菌枯萎基因XA5及其同源物TFIIA1的XA5(XA5)。Mol Genet基因组学。2006; 275:354-66。
- 12
歌曲WY,Wang GL,Chen Ll,Kim HS,Pi Ly,Holsten T,等。由水稻抗病基因XA21编码的受体激酶样蛋白。科学(80-)。1995年; 270:1804-6。
- 13
田DS,王JX,Zeng X,Gu Ky,秋CX,杨XB等。水稻抑制剂依赖性抗性蛋白XA10触发细胞死亡和内质网中的钙耗尽。植物细胞。2016; 26:497-515。
- 14
孙晓丽,曹永利,杨志峰,徐春春,李晓华,王绍平,等。Xa26是一种抗水稻白叶枯病菌的基因。编码一种LRR受体激酶样蛋白。植物j . 2004; 37(4): 517 - 27所示。
- 15
顾凯,杨斌,田德,吴力,王德,Sreekala C,等.aⅢ型效应子诱导R基因表达引发水稻抗病性。自然。2005;435:1122–5.
- 16
朱哲,傅比,杨华,徐聪,李泽科,桑切斯,等。水稻抗白叶枯病隐性基因xa13的定位研究。理论应用基因。2006;112(3):455–61.
- 17。
张F,卓德,张F,黄丽,王Ws,Xu Jl等。XA39,一种赋予Xanthomonas oryzae PV的广谱耐谱的新型显性基因。玉米酸盐。植物别墅。2014; 64:568-75。
- 18。
Kim Sm,Suh JP,Qin Y,Noh Th,Reinke Rf,Jena KK。新抗性基因,XA40,赋予水稻(Oryza Sativa L.)赋予抗细菌枯萎种群的鉴定和细映射。Al Appl Genet。2015; 128:1933-43。Springer Berlin Heidelberg。
- 19。
梁LQ,王海,曾吕克,王WJ,风JQ,Chen B等。水稻品种白征广州哈勃XA42(T)确定对Xanthomonas oryzae PV的抗性。oryzae。植物品种。2017; 136:603-9。
- 20
刘杰,王旭,米切尔T,胡勇,刘旭,戴力,等。水稻与稻瘟病菌相互作用的分子机制研究进展。植物病理学。2010;11:419–27.
- 21
Bandillo N,Raghavan C,Muyco Pa,塞维利亚Mal,Lobina It,Dilla-Ermita CJ,等。大米中的多家长高级生成交叉交叉(魔法)种群:遗传研究与育种的进展与潜力。米。2013; 6:11。
- 22
陈氏,刘X,曾升,欧阳D,杨杰,朱X。一种新型隐性基因XA34(T)对Xanthomonas抗性抗性的新型隐性基因Xa34(T)的分子映射。oryzae。Al Appl Genet。2011; 122:1331-8。
- 23
vikal y,Bhatia d.稻米细菌抗性的遗传学和基因组学。:国际大米研究进展。金泉李,Intechopen;2017. p。175-213。
- 24
Burghardt Lt,Young Nd,Tiffin P.植物基因组关联映射指南:植物中的基因组关联映射。Curr Protoc植物Biol。2017; 2:22-38。
- 25。
Lipka Ae,Tian F,Wang Q,Peiffer J,Li M,Bradbury PJ等。Gapit:基因组关联和预测综合工具。生物信息学。2012; 28:2397-9。
- 26。
李TG,MA XF,Li NY,周L,Liu Z,Han Hy等。基因组 - 宽协会研究发现了旱地棉花抗原抗性的候选基因(Gossymium hirsutum L.)。植物Biotechnol J. 2017; 15:1520-32。
- 27。
刘明华,康慧霞,徐玉成,彭勇,王丹,高丽娟,等。全基因组关联研究鉴定了一种对水稻Magnaporthe oryzae部分抗性的NLR基因。植物营养与肥料学报;2010;
- 28。
温Z,Tan R,张某,柯林斯PJ,Yuan J,Du W等人。整合GWAS和基因表达数据,以进行大豆耐白色模具的功能表征。植物Biotechnol J. 2018; 16:1825-35。
- 29。
黄Xh,魏Xh,桑t,赵q,冯q,zhao y等。基因组 - 水稻地质14种农艺性状的协会研究。NAT Genet。2010; 42:961-7。
- 30.
刘Qs,元M,周Y,李Xh,肖杰,王sp。MTN3 /唾液家族的一只副病毒性患者积聚地赋予水稻Xanthomonas oryzae的种族特异性抗性。植物细胞环境。2011; 34(11):1958-69。
- 31。
rohini g,Shalu J,Akhilesh Kt,Mukesh J. Genome-Sive测量和表达分析表明谷氨酸毒素基因家族成员在稻米的各种刺激期间的不同作用。DNA Res。2010; 17:353-67。
- 32。
Ogo Y,Kobayashi T,Nakanishi IR,Nakanishi H,Kakei Y,Takahashi M,等。识别铁缺乏响应元件2的新型NAC转录因子IDEF2调节植物中涉及铁袜的基因。J Biol Chem。2008; 283(19):13407-17。
- 33。
Kieu NP、Aznar A、Segond D、Rigault M、Simond-Côte E、Kunz C等。缺铁会影响植物的防御反应,并赋予对Dickeya dadanti和灰霉病菌的抗性。植物病理学。2012;13(8):816–27.
- 34。
Mani A,Sankaranarayanan K.在水稻植物中,几个金属离子如Zn2+,锰2+、铁2+,光盘2+等通过NRAMP TRANSICER蛋白运输。蛋白质J. 2018; 37:237-47。
- 35。
张F,吴ZC,王MM,张F,Dingkuhn M,XU JL等。基因组 - 范围的关联分析鉴定了在各种籼稻种质中集合中细菌枯萎病的抗性基因座。Plos一个。2017; 12(3):E0174598。
- 36
Li N,Lin B,Wang H,Li Xm,Yang Ff,Ding Xh等。ZMFBL41的自然变化赋予玉米中的带状叶和鞘枯燥的抗性。NAT Genet。2019; 51:1540-8。
- 37
陈海林,王绍平,张秋芳。水稻抗白叶枯病新基因定位于12号染色体上。植物病理学。2002;92(7):750 - 4。
- 38
Park CJ, Ronald PC。水稻XA21免疫受体的裂解和核定位。Nat Commun。2012;3(6):920。
- 39
周友,乌佐威vne,张ch,cheng lr,wang l,chen k等。野生稻(Oryza Rufipogon)XA23基因改善杂交水稻细菌枯萎病。作物科。2011; 30(6):637-44。
- 40.
Korinsak S,Sriprakhon S,Sirithanya P,Jairin J,Korinsak S,Vanavichit A等人。鉴定微卫星标志物(SSR)与水稻品种“BA7中的新细菌枯萎基因XA33(T)连接。”Maejo Int J SCI Technol。2009; 3(2):235-47。
- 41.
Antony G,周JH,Huang S,Li T,Liu B,White F,等。稻米XA13对细菌枯萎的隐性抗性被疾病易感性基因OS-11N3诱导击败。植物细胞。2010; 22(11):3864-76。
- 42.
Shadle GL,Wesley SV,Korth K,Chen F,Lamb C,Dixon R.转基因烟草中的苯基丙醇化合物和抗病性,具有改变的L-苯丙氨酸氨酶的表达。植物化学。2003; 64(1):153-61。
- 43.
李文清,邵明,杨杰,钟武,冈田K,亚马内H,等.水稻抗白叶枯病基因Oscyp71Z2涉及二萜类植物抗毒素的生物合成。植物科学。2013;207(完整):98–107。
- 44
李HJ,李Xh,肖耶,翼ra,王sp。XA3 / XA26轨迹的矫形器等位基因赋予水稻Xanthomonas Oryzae的储存种族特异性抗性。莫尔植物。2012; 5(1):281-90。
- 45
Cao Yl,Duan L,Li HJ,Sun XL,Zhao Y,Xu Cg等。XA3 / XA26系列耐水稻抗性Xanthomonas oryzae PV的功能分析。oryzae。Al Appl Genet。2007; 115(7):887-95。
- 46
王志祥,杨建平,王志祥,等。水稻抗白叶枯病基因Xa1的表达是通过细菌接种诱导的。自然科学学报,1998;95(4):1663-8
- 47
Baggs E,Dagdas G,Krasileva KV。NLR多样性,助手和集成域:了解NLR身份。CurrOp植物BIOL。2017; 38:59-67。
- 48
Cesari S,Bernoux M,Moncuquet P,Kroj T,Dodds PN。植物NLR蛋白对的新型保守机制:“综合诱饵”假设。前植物SCI。2014; 5:606。
- 49。
Wu CH、Krasileva KV、Banfield MJ、Terauchi R、Kamoun S。植物NLR蛋白的“传感域”:不仅仅是诱饵。前植物科学。2015;6:134.
- 50。
Kauffman He,Reddy APK,Hsieh Spy,Merca SD。水稻品种抗性对Xanthomonas oryzea的改进技术。植物DIS批准。1973; 57:537-41。
- 51。
uzunova m,ecke w,weissleder k,röbbeleng.映射油菜籽的基因组(芸苔Napus l.)。I.构建RFLP联系地图和QTL的定位,用于种子硫代葡萄糖苷含量。Al Appl Genet。1995年; 90:194-204。
- 52。
李H,Durbin R.快速准确的长读与挖洞轮转变换。生物信息学。2010; 26:589-95。
- 53。
McKenna A、Hanna M、Banks E、Sivachenko A、Cibulskis K、Kernytsky A等,《基因组分析工具包:分析下一代DNA测序数据的MapReduce框架》。基因组研究,2010年;20:1297–303.
- 54。
Vilella Aj,Severin J,Ureta-Vidal A,Heng L,Durbin R,Birney E.Seanemblcompara Geneteres:完整,复制意识到脊椎动物的系统发育树。Genome Res。2009; 19:327-35。
- 55。
亚历山大DH,Novembre J,Lange K.基于快速模型的无关个人祖先估算。Genome Res。2009; 19:1655-64。
- 56。
杨杰,乐施,我的天,维舍尔总理。GCTA:全基因组复杂性状分析工具。我是吉英。2011;88:76–82.
- 57。
张c,东ss,徐jy,他wm,杨tl。Poplddecay:基于变体呼叫格式文件的联动不平衡衰减分析的快速有效工具。生物信息学。2019; 35:1786-8。
- 58。
波兰贾,布拉德伯里PJ,克兰特州,纳尔逊rj。玉米北部叶片枯叶的全基因组嵌套术。Proc Natl Acad Sci USA。2011; 108:6893-8。
- 59。
Kang HM, Sul JH, Service SK, Zaitlen NA, Kong S, Freimer NB,等。全基因组关联研究中样本结构的方差成分模型。Nat麝猫。2010;42:348-54。
- 60
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg SL。TOPHAT 2:在存在插入,缺失和基因融合的情况下,转录om的精确对准。基因组Biol。2013; 14(4):R36。
- 61
Langmead B,Trapnell C,Pop M,Salzberg SL.短DNA序列与人类基因组的超快和记忆有效比对。基因组生物学。2009;10(3):R25。
- 62
Trapnell C,Williams Ba,Pertea G,Mortazavi A,Kwan G,Van Baren MJ,等。通过RNA-SEQ的转录程序组件和定量揭示了细胞分化期间未经发布的转录物和同种型切换。NAT BIOTECHNOL。2010; 28:511-5。
- 63.
Anders S,Huber W.序列计数数据的差异表达分析。基因组Biol。2010; 11:R106。
- 64.
安德斯,休伯W。RNA-Seq数据在基因水平的差异表达。海德堡:EMBL;2012
- 65.
Benjamini Y,Hochberg Y。控制错误发现率:一种实用而有效的多重测试方法。J R Stat Soc系列B方法。1995;57(1):289–300.
- 66.
Gabriel SB,Schaffner SF,Nguyen H,Moore JM,Roy J,Blumenstiel B等。人基因组中单倍型块的结构。科学。2002; 296:2225-9。
- 67.
Barrett Jc,Fry B,Maller J,Daly MJ。HaploView:LD和单倍型图的分析和可视化。生物信息学。2005; 21:263-5。
致谢
作者感谢所有贡献者的工作,并要感谢审稿人的宝贵意见和建议。
资金
这项工作得到了中国国家自然科学基金(第31400130和32001490号),四川科技计划(2019年No.2020yfh0117)和中国国家973项目(No.2014CB160304)。资金代理只提供了财政支持,并没有涉及设计的实验,收集,解释和数据的设计以及稿件的起草。
作者信息
从属关系
贡献
A.Z, A.W和X.S.设计了这个项目。A.W、X.S、Y.J、X.X和x.y进行了实验。X.S.进行了生物信息学分析。A.W.和X.S.写了手稿。研究,S.L Q.D。H.L。白雪,J.Z, Y.L, T.Z, L.W, P.L.提供有用的建议。所有作者阅读并批准最终稿件。
通讯作者
道德宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明没有竞争利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充资料
附加文件1:图S1。
来自基因组关联研究(GWAS)的分位数(Q-Q)和曼哈顿图谱结果对细菌叶枯萎(BLB)抗性使用240个籼稻品种。(A,B,C)GWA在(a)p3_2018,(b)p3_2019和(c)两年间P3的最佳线性无偏的预测(Blup)值。(D,E,F)GWA在(d)p6_2018,(e)p6_2019和(f)两年间P6的最佳线性无偏的预测(flup)值。X轴沿着每种染色体显示单个核苷酸多态性(SNP);Y轴是关联的-LOG10P。
附加文件2:表S1。
259种米饭净化的名称,原产地和人口结构。
附加文件3:表S2。
关于259种水稻种族基因组的序列信息。
附加文件4:表S3。
经过过滤的SNP注释的结果。
附加文件5:表S4。
使用测序数据映射到Nipponbare借口基因组的259米存取中的SNP数。MAF:次要等位基因频率。
附加文件6:表S5。
P3(BOLP)的GWAS与259条使用映射生成的SNP设置读取到Nipponbare的SNP集合。
附加文件7:表S6。
P3(2018年)GWA(2018年)育实,使用映射生成的SNP集合读取到Nipponbare。
附加文件8:表S7。
P3(2019年)的GWAS使用映射生成的SNP集合的259条米线,映射到Nipponbare。
附加文件9:表S8。
P6(BOLP)的GWAS使用映射生成的SNP集合与NipponBare产生的SNP集合的级别。
附加文件10:表S9。
P6(2018年)GWAS级育地,使用映射生成的SNP集读数读对Nipponbare。
附加文件11:表S10。
P6(2019年)的GWA(2019年)级别使用由映射生成的SNP集读对于Nipponbare而生成的SNP集合。
附加文件12:表S11。
Illumina转录组测序数据的统计数据。
附加文件13:表S12。
在不同接种时间的抗性线NSIC RC154中的DEGS列表。
附加文件14:表S13。
感病品系CT 9737-6-1-3P-M在不同接种时间的DEGs表。
附加文件15:表S14。
2817度是在电阻线NSIC RC154中唯一检测到的。
附加文件16:表S15。
QRT-PCR的候选基因序列的特异性引物。
权利和权限
开放存取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/. 知识共享公共领域放弃(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
舒,X.,王,A.,江,B。等。基因组 - 范围协会研究和转录组分析发现稻米细菌叶片抗性的新基因(奥雅萨苜蓿L.)。BMC植物生物学21,255(2021)。https://doi.org/10.1186/s12870-021-03041-2
已收到:
公认:
发表:
关键词
- 米
- 稻黄单胞菌PV。oryzae.
- 格干
- RNA-Seq
- 抗性基因