- 复习gydF4y2Ba
- 开放访问gydF4y2Ba
- 出版:gydF4y2Ba
全基因组关联研究及其在非模式作物中的应用gydF4y2Ba芝麻纪录gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba21.gydF4y2Ba,商品编号:gydF4y2Ba283gydF4y2Ba(gydF4y2Ba2021.gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
芝麻是一种罕见的非模式和小型作物,其许多遗传位点和候选基因的潜在特征已在相对高分辨率下被揭示。这些进展都得益于全基因组关联研究(GWAS)方法的应用。GWAS得益于芝麻高质量基因组的可用性、数千种基因型的重测序数据、广泛的转录组测序、单倍型图的开发和基于网络的功能数据库。gydF4y2Ba
结果gydF4y2Ba
本文综述了芝麻重要性状遗传发现的GWAS方法、基本统计模型及其应用。一种新型的在线数据库SiGeDiD(gydF4y2Bahttp://sigedid.ucad.sn/gydF4y2Ba)通过芝麻的GWAS提供所有遗传和基因组发现的途径。我们还首次测试了各种新的GWAS多位点模型在芝麻中的应用。gydF4y2Ba
结论gydF4y2Ba
总的来说,这项工作描述了在非模型作物sesame中高效实现GWAS的步骤并提供了指导。gydF4y2Ba
背景gydF4y2Ba
芝麻(gydF4y2Ba芝麻纪录gydF4y2Bal,2n = 2x = 26)属于gydF4y2Ba山茶科gydF4y2Ba家庭是驯养野生祖先驯化的最古老的油籽作物之一gydF4y2Ba年代gydF4y2Ba.gydF4y2Ba马拉巴里gydF4y2Ba在5000年前在近东,亚洲和非洲[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba].芝麻是其气候弹性,高油含量和独特的抗氧化特性[gydF4y2Ba3.gydF4y2Ba]. 是优质食用油和蛋白质食品的重要来源。芝麻籽的含油量在50-60%之间,含有大量的天然抗氧化剂,如芝麻素、芝麻素和芝麻醇,使其具有较长的保质期和稳定性[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba]. Ashakumary等人[gydF4y2Ba6gydF4y2Ba]据报道,芝麻含有19-25%的蛋白质,是铁、镁、铜、钙、维生素B1、E和植物甾醇的良好来源,有助于降低血液胆固醇水平。此外,芝麻籽中还含有各种必需氨基酸和脂肪酸[gydF4y2Ba7gydF4y2Ba]. 芝麻产业是一个价值数十亿美元的产业,支撑着全世界数百万农民的生计[gydF4y2Ba8gydF4y2Ba].在过去的十年中,总产量大幅增加,2017年达到600万吨(粮食和农业组织统计数据库[gydF4y2Ba9gydF4y2Ba].然而,芝麻生产和生产力面临不同的限制,包括有限的改进品种,成熟时胶囊破碎,非同步成熟度,差的建立,大量分支,低收获指数,干旱压力,涝渍和疾病[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba]. 为了加速芝麻改良,基因组学辅助育种已成为在短时间内培育优良品种的有效途径[gydF4y2Ba13gydF4y2Ba].于是,芝麻的参考基因组序列以及众多重要的基因组资源被送到了科学界[gydF4y2Ba14gydF4y2Ba].芝麻基因组的单倍型图是由全球705个不同品种的重新测序项目构建的,并进一步进行两种代表性基因组gydF4y2Ba德诺维gydF4y2Ba组装(gydF4y2Ba15gydF4y2Ba]. 这些资源对于芝麻研究的快速发展至关重要,因为它们加速了利用全基因组关联研究(GWAS)方法检测控制重要农艺性状的基因座。目前,已经发现了数百种与油质、抗逆性、种子产量等重要性状相关的遗传变异。这些发现有助于利用标记辅助选择和基因组选择来提高芝麻的遗传改良和综合生产力。这使得芝麻成为一种罕见的非模式和小作物,基因组研究,特别是GWAS,已经非常成功。gydF4y2Ba
在这篇综述论文中,我们首先提出GWAS方法和潜在的统计模型。然后详细介绍了GWAS在芝麻基因发现中的应用进展。为GWAS在芝麻中的更好应用提供了重要指导。gydF4y2Ba
正文gydF4y2Ba
GWAS方法,基础统计模型和在植物中的应用gydF4y2Ba
Gwas方法gydF4y2Ba
全基因组关联研究(GWAS)又称关联作图或连锁不平衡(LD)作图,充分利用了物种内高表型变异和自然种群中高数量的历史重组事件。与传统的数量性状位点(QTL)作图相比,以相对高的分辨率识别性状的遗传位点已成为一种替代方法[gydF4y2Ba15gydF4y2Ba]. GWAS一般适用于研究单核苷酸多态性(SNPs)与目标表型性状之间的关系。如今,利用先进的高通量基因分型技术进行SNP鉴定变得越来越容易。GWAS是根据LD通过对自然群体中的不同个体进行基因分型和表型分析来定量评价的。与传统的利用双亲分离群体的QTL定位方法不同,GWAS中感兴趣性状的致病基因的鉴定是在自然群体中进行的。GWAS的一个主要优点是,相同的基因型数据和相同的群体可以反复用于不同的性状。gydF4y2Ba
GWAS已成功应用于人类、动物和植物的高分辨率关联识别、候选基因检测和数量性状分析[gydF4y2Ba16.gydF4y2Ba,gydF4y2Ba17.gydF4y2Ba].在各种经济上有价值的作物中的Gwas被用来深入了解重要特征的遗传架构,包括前往花穗架,耐水稻黄色斑块病毒,生育恢复和稻米农艺性状的日子[gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21.gydF4y2Ba]; 遗传变化和进化的模式[gydF4y2Ba22.gydF4y2Ba,gydF4y2Ba23.gydF4y2Ba,组成和粘贴性质[gydF4y2Ba24.gydF4y2Ba,茎生物量[gydF4y2Ba25.gydF4y2Ba]叶片表皮电导[gydF4y2Ba26.gydF4y2Ba]在玉米;植物高度部件和花序结构[gydF4y2Ba27.gydF4y2Ba,粒度[gydF4y2Ba28.gydF4y2Ba]粮食品质[gydF4y2Ba29.gydF4y2Ba]高粱;玉米收获指数[gydF4y2Ba30.gydF4y2Ba],油菜开花时间[gydF4y2Ba31.gydF4y2Ba]应力耐受性,油含量和种子质量[gydF4y2Ba32.gydF4y2Ba芸苔属植物);油品产量及品质[gydF4y2Ba15gydF4y2Ba],产量相关的特征[gydF4y2Ba33.gydF4y2Ba,gydF4y2Ba34.gydF4y2Ba]、耐旱性[gydF4y2Ba35.gydF4y2Ba],维生素E [gydF4y2Ba36.gydF4y2Ba]芝麻。gydF4y2Ba
GWAS方法统计模型gydF4y2Ba
单轨模型gydF4y2Ba
使用GWAS的标记-性状关联已被广泛应用于群体的一维基因组扫描[gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba37.gydF4y2Ba,gydF4y2Ba38.gydF4y2Ba,gydF4y2Ba39.gydF4y2Ba].在该方法中,一次评估一个SNP。在使用描述为的一般线性模型(GLM)之后gydF4y2BaYgydF4y2Ba=gydF4y2BaβgydF4y2Ba0gydF4y2Ba+gydF4y2BaβgydF4y2Ba1gydF4y2BaXgydF4y2Ba[gydF4y2Ba40gydF4y2Ba](其中Y =因变量/预测变量/解释变量/响应变量,gydF4y2BaβgydF4y2Ba0gydF4y2Ba=gydF4y2Ba截获gydF4y2Ba;βgydF4y2Ba1gydF4y2Ba=gydF4y2Ba重量或坡度(系数);gydF4y2BaX =gydF4y2Ba变量),一种流行的模型,称为混合线性模型(MLM)(Q + K方法),其被描述为gydF4y2BaYgydF4y2Ba=gydF4y2BaXβ.gydF4y2Ba+gydF4y2Ba祖茂堂+ egydF4y2Ba[gydF4y2Ba41.gydF4y2Ba], (在哪里gydF4y2Bay =gydF4y2Ba观察表型矢量;gydF4y2Baβ =gydF4y2Ba含有固定效应的未知载体,包括遗传标记,人口结构(Q)和截距;gydF4y2Bau型=gydF4y2Ba个体/系多重背景QTL随机加性遗传效应的未知载体gydF4y2BaXgydF4y2Ba和gydF4y2BaZgydF4y2Ba=已知设计矩阵;和gydF4y2Ba电子=gydF4y2Ba为控制GWAS中群体分层的多重检验效应和偏倚,开发了未观察到的残差向量。然后,有报道称关联映射的精度有所提高[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba42.gydF4y2Ba,gydF4y2Ba43.gydF4y2Ba]. 随后,许多先进的基于传销的统计方法也被提出,以解决某些限制,如假阳性率,大的计算结果,和不准确的预测[gydF4y2Ba44.gydF4y2Ba].高效的混合模型协会(EMMA)[gydF4y2Ba45.gydF4y2Ba]压缩混合线性模型(CMLM)和先前确定的总体参数(P3D)[gydF4y2Ba46.gydF4y2Ba]和随机SNP效应混合线性模型(MRMLM)[gydF4y2Ba47.gydF4y2Ba]是迄今为止提出的基于传销的单位点基因组扫描的一些最新改进方法。这种先进的统计模型功能强大、灵活且计算效率高。EMMA是通过将QTN效应作为固定效应来最小化MLM概率函数中显示的计算负荷[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba44.gydF4y2Ba,gydF4y2Ba45.gydF4y2Ba];而CMLM则是通过将个体分组来控制庞大的基因型数据的大小,从而从聚类的个体中得到群体亲缘矩阵[gydF4y2Ba46.gydF4y2Ba]. 一般来说,尽管在复杂性状的标记效应的有效估计方面存在局限性,但单位点模型方法具有很好的处理多个标记的能力[gydF4y2Ba47.gydF4y2Ba,这是其值得报道的特点之一。gydF4y2Ba
虽然单轨模型分析是GWAS中每个SNP和表型之间关联分析的常见方法,但一些早期的报告表明,使用单轨模型分析具有局限性,以解决多次测试引起的潜在效果,历史基因型效应和脂肪熵作用[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba48.gydF4y2Ba]. 他们报告说,当一次只对单核苷酸多态性进行测试时,整个基因组中可用的基因变体之间的相互作用还没有得到深入的探讨。类似地,在这种方法中,用于控制由于多重检测引起的假阳性错误(FDR)的Bonferroni校正也是非常严格的,因此大量的重要基因座可能不会被单基因座模型识别,特别是对于由于表型数据和多基因座效应引起的较大错误[gydF4y2Ba49.gydF4y2Ba,gydF4y2Ba50.gydF4y2Ba].因此,这些单位点基因组扫描方法不便于检测分别受少数和/或多基因调控的效应大和效应小的数量性状[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba49.gydF4y2Ba].此外,单位点方法无法探究近缘基因内的上位性遗传效应[gydF4y2Ba51.gydF4y2Ba].gydF4y2Ba
Haplotype-based模型gydF4y2Ba
为了解决单基因座模型分析的一些局限性,基于单倍型的模型,基于随机SNP效应混合线性模型(MRMLM),描述如下:gydF4y2BaY=Xβ+ZgydF4y2BaK.gydF4y2BaygydF4y2BaK.gydF4y2Ba+ U + egydF4y2Ba(其中gydF4y2BaYgydF4y2Ba=所有品系的估计基因型值向量是作为种群结构的固定效应的关联矩阵,gydF4y2BaβgydF4y2Ba是固定效果的矢量,gydF4y2BaZgydF4y2BaK.gydF4y2Ba=gydF4y2Ba基因型指示剂的载体gydF4y2BaTH.gydF4y2Ba苏格兰民族党,gydF4y2BaYgydF4y2BaK.gydF4y2Ba=标记k对~N (0, k σgydF4y2Ba2gydF4y2BaK.gydF4y2Ba),gydF4y2Ba你gydF4y2Ba=N(0,σ)的亲缘矩阵(K)描述的多基因效应向量gydF4y2Ba2gydF4y2Ba一个gydF4y2Ba)以及gydF4y2BaegydF4y2Ba= ~N (0, IσgydF4y2Ba2gydF4y2BaegydF4y2Ba)),为小麦、水稻和大豆等主要作物开发和实施[gydF4y2Ba52.gydF4y2Ba,gydF4y2Ba53.gydF4y2Ba].高LD中的几个相邻标记在这种多变量方法中聚集成单个多基因座单倍型,因此单倍型在多个GLM系统中而不是单个SNP进行评估,并且已经观察到单倍型和选择的特性之间的关联[gydF4y2Ba48.gydF4y2Ba,gydF4y2Ba52.gydF4y2Ba,gydF4y2Ba54.gydF4y2Ba]. 在GWAS中,基于单倍型的模型比传统的单基因座模型更加有效和可靠,因为它有助于准确捕获等位基因多样性,优化高密度标记数据的使用,增强上位性互作的发现能力,并最小化多重测试[gydF4y2Ba51.gydF4y2Ba,gydF4y2Ba52.gydF4y2Ba].gydF4y2Ba
多位点模型gydF4y2Ba
多轨迹模型是GWAS中涉及两阶段算法的新发展的替代方法[gydF4y2Ba55.gydF4y2Ba,gydF4y2Ba56.gydF4y2Ba,gydF4y2Ba57.gydF4y2Ba]包括对整个基因组进行单位点扫描以检测所有可能的相关单核苷酸多态性(qtn),然后使用多位点GWAS模型检测所有相关单核苷酸多态性以检测真正的qtn。这些新发展的多基因座GWAS模型是检测受多基因/位点调控、受群体结构影响较小的复杂数量性状的理想模型。与单基因座模型相比,多基因座模型的一些优点是,能够高效地检测控制某一性状的多个基因,假阳性率低,并且不需要对已知排除重要基因座的多个检测进行Bonferroni校正[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba47.gydF4y2Ba,gydF4y2Ba58.gydF4y2Ba,gydF4y2Ba59.gydF4y2Ba].多位点模型也导致了GWAS中关联结果的质量和深度的实质性改进[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba42.gydF4y2Ba,gydF4y2Ba53.gydF4y2Ba,gydF4y2Ba57.gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba]. 目前在GWAS中主要实现的模型包括多位点混合模型(MLMM)[gydF4y2Ba57.gydF4y2Ba],多位点随机SNP效应混合线性模型(mrMLM)[gydF4y2Ba47.gydF4y2Ba]综合确定独立筛选期望最大化贝叶斯最小绝对收缩选择算子模型(ISIS EM-BLASSO)[gydF4y2Ba50.gydF4y2Ba]快速多基因座随机SNP效应高效混合模型关联(FastMremma)[gydF4y2Ba17.gydF4y2Ba]、基于多基因背景控制的最小角度回归+经验贝叶斯(pLARmEB) [gydF4y2Ba62gydF4y2Ba],kruskal-wallis测试与多基因背景控制下的经验贝叶斯(PKWMEB)[gydF4y2Ba58.gydF4y2Ba]和快速多位点随机SNP效应混合线性模型(FASTmrMLM)[gydF4y2Ba59.gydF4y2Ba,gydF4y2Ba63gydF4y2Ba].在迄今为止的众多多基因座模型中,Segura等人。[gydF4y2Ba57.gydF4y2Ba]提出了一种MLMM方法,该方法比其它已有的多位点方法(包括惩罚逻辑回归)具有优势[gydF4y2Ba64gydF4y2Ba],逐步回归[gydF4y2Ba65gydF4y2Ba]贝叶斯启发的惩罚极大似然法、计算效率、错误发现率检测以及解决GWAS中的种群结构问题。同样,Korte等人[gydF4y2Ba66gydF4y2Ba还提出了一种混合模型方法,称为多重性混合模型(MTMM),其检测用于精确相关的多种表型特征的因果区基因座,并同时处理具有分析内部和特性间方差分量。同样,Klasen等人。[gydF4y2Ba61gydF4y2Ba]提出了一种不采用群体校正技术的多位点关联的数量性状聚类分析方法,该模型在限制假阳性/假阴性关联方面有较好的效果。GWAS的多性状分析(MTAG)也是Turley等人开发的另一种具体方法[gydF4y2Ba67gydF4y2Ba]在GWAS中分析汇总统计(META分析)。Zhan等人。[gydF4y2Ba68gydF4y2Ba还提出了另一种方法,其命名为双核关联测试(DKAT),其包括两个单独的核矩阵来解释表型和基因型相似性。DKAT对现有方法的优点包括能够测试多个特征和多个SNP之间的关系而不进行参数假设,纠正I型错误率,统计上高效且计算可扩展[gydF4y2Ba60gydF4y2Ba,gydF4y2Ba68gydF4y2Ba].gydF4y2Ba
最近,人们进行了不同的比较研究来评估这些不同GWAS模型在不同植物物种中检测标记-性状关联的能力。在全球范围内,已经发现多基因座模型比单基因座模型更有效和强大,能够检测出兴趣性状的高度显著关联结果(表1)gydF4y2Ba1gydF4y2Ba).然而,将单基因座模型和多基因座模型结合在一起已经被证明可以提高GWAS中复杂性状关联分析的有效性和威力,因为单基因座模型可以检测出一些多基因座模型无法识别的基因座[gydF4y2Ba54.gydF4y2Ba,gydF4y2Ba70gydF4y2Ba].gydF4y2Ba
使用Pan-Genome与GWA的单一参考基因组gydF4y2Ba
研究给定群体的遗传变异的常见方法依赖于从现有参考基因组的序列中注释的基因和变体的解释依赖于基因和变体[gydF4y2Ba74gydF4y2Ba].目前,许多作物的参考基因组序列,包括稻米[gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba,gydF4y2Ba77gydF4y2Ba]、高粱[gydF4y2Ba78gydF4y2Ba],玉米[gydF4y2Ba79gydF4y2Ba],gydF4y2Ba芸苔属植物拉伯gydF4y2Ba[gydF4y2Ba80gydF4y2Ba], 仅仅 [gydF4y2Ba81gydF4y2Ba,gydF4y2Ba82gydF4y2Ba],小米[gydF4y2Ba83gydF4y2Ba], 土豆 [gydF4y2Ba84gydF4y2Ba],番茄[gydF4y2Ba85gydF4y2Ba],还有芝麻[gydF4y2Ba14gydF4y2Ba]已经有报道了。随着高质量参考基因组序列的产生,一些GWAS已经被用来发现不同群体之间的自然变异。然而,基于参考基因组的GWAS方法可能不足以区分群体之间或群体内部的任何差异,其中某些相关基因可能在参考基因组中不活跃,但可能在研究群体中表达[gydF4y2Ba86gydF4y2Ba].gydF4y2Ba
自发现泛基因组gydF4y2Ba链球菌阿拉糖gydF4y2Ba[gydF4y2Ba87gydF4y2Ba]通过比较不同来源的多个基因组,构建了不同的泛基因组gydF4y2Ba德诺维gydF4y2Ba同一物种包括水稻在内的不同个体的序列组装[gydF4y2Ba88gydF4y2Ba,gydF4y2Ba89gydF4y2Ba],玉米[gydF4y2Ba90gydF4y2Ba]),大豆[gydF4y2Ba91gydF4y2Ba],gydF4y2BaB那不勒斯gydF4y2Ba[gydF4y2Ba92gydF4y2Ba)、小麦(gydF4y2Ba93gydF4y2Ba]最近在芝麻[gydF4y2Ba94gydF4y2Ba](表gydF4y2Ba2gydF4y2Ba). 与基于参考基因组测序的GWAS方法不同,基于参考基因组测序的GWAS方法依赖于整个研究小组中的SNPs,泛基因组方法更具包容性,可以检测大量的变异,包括结构变异(SV)、拷贝数变异(CNV)、存在/不存在变异、倒位和翻译变异[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba86gydF4y2Ba].在这方面,Song等人。[gydF4y2Ba96gydF4y2Ba]报道了一种直接检测目标性状(西力克长度、种子重量和开花时间)结构变异的方法gydF4y2Ba甘蓝型油菜gydF4y2Ba基于PAV的全基因组关联研究(PAV-GWAS),使用从8个高质量基因组组装的泛基因组。他们还报告说,涉及单一参考基因组的SNP-GWAS方法表明没有检测到同一人群的因果结构变异。他们的研究结果表明,基于泛基因组的关联研究是一种强有力的方法,可以补充单一参考基因组方法检测新的SNP性状关联。同样,甘蔗花叶病毒抗性基因的物理位置(gydF4y2BaZMTRXH.gydF4y2Ba)在玉米中发现了由三种不同基因型组成的泛基因组,但没有使用单一的参考基因组[gydF4y2Ba90gydF4y2Ba].基于基于泛基因组的GWA已经在稻米和鸽豌豆等重要作物中进行了[gydF4y2Ba89gydF4y2Ba,gydF4y2Ba97gydF4y2Ba].gydF4y2Ba
芝麻GWAS种群的多样性与发育gydF4y2Ba
形态和遗传多样性gydF4y2Ba
芝麻属二倍体种,属于芝麻科gydF4y2Ba初生术gydF4y2Ba,细分gydF4y2Ba被子植物科gydF4y2Ba,类gydF4y2Ba双子叶植物gydF4y2Ba,订单gydF4y2BaTubifloraegydF4y2Ba,家庭gydF4y2Ba山茶科gydF4y2Ba,和属gydF4y2Basesamum.gydF4y2Ba.gydF4y2Ba山茶科gydF4y2Ba一个小科有16属60种,隶属于哪37种gydF4y2Basesamum.gydF4y2Ba属,只有gydF4y2Ba芝麻纪录gydF4y2BaL.是最常见的栽培品种[gydF4y2Ba10gydF4y2Ba,gydF4y2Ba39.gydF4y2Ba,gydF4y2Ba98gydF4y2Ba,gydF4y2Ba99gydF4y2Ba,gydF4y2Ba100gydF4y2Ba]. 世界上报道了大量的品种和生态型,对各种生态条件具有高度的适应性。细胞内有三个细胞遗传学组gydF4y2Basesamum.gydF4y2Ba其中2n=26由栽培的gydF4y2BaS. Indum.gydF4y2Ba随着gydF4y2BaS南阿拉坦。卡彭塞,S。申基群岛。马拉巴里gydF4y2Ba;2n = 32包括gydF4y2BaSprostratum,S。laciniatum,S。安哥拉,S。狭叶;gydF4y2Ba而gydF4y2BaS辐射,S。西方和南美洲。先秦gydF4y2Ba属于2n = 64 [gydF4y2Ba101gydF4y2Ba,gydF4y2Ba102gydF4y2Ba,gydF4y2Ba103gydF4y2Ba].到目前为止,广泛的形态变化,包括植物高度,高度到第一粒,高度到第一分支,分支数,开花时期,花色,每腋的鲜花数,胶囊的数量,每个腋数,胶囊边缘数日成熟,每粒种子的种子数,栽培芝麻的种子涂料,种子大小,种子油含量,种子产量和分支习惯的数量[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba104gydF4y2Ba,gydF4y2Ba105gydF4y2Ba,gydF4y2Ba106gydF4y2Ba,gydF4y2Ba107gydF4y2Ba].在世界各地收集的许多地方品种和品种中,除了芝麻种质存在巨大的表型变异外,也记录了各种基于分子标记的高水平遗传多样性(表1)gydF4y2Ba3.gydF4y2Ba) [gydF4y2Ba1gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba104gydF4y2Ba,gydF4y2Ba106gydF4y2Ba,gydF4y2Ba109gydF4y2Ba,gydF4y2Ba110gydF4y2Ba,gydF4y2Ba115gydF4y2Ba,gydF4y2Ba116gydF4y2Ba,gydF4y2Ba117gydF4y2Ba,gydF4y2Ba118gydF4y2Ba,gydF4y2Ba119gydF4y2Ba,gydF4y2Ba120.gydF4y2Ba,gydF4y2Ba121.gydF4y2Ba,gydF4y2Ba122.gydF4y2Ba,gydF4y2Ba123.gydF4y2Ba,gydF4y2Ba124.gydF4y2Ba,gydF4y2Ba125.gydF4y2Ba,gydF4y2Ba126.gydF4y2Ba,gydF4y2Ba127.gydF4y2Ba,gydF4y2Ba128.gydF4y2Ba,gydF4y2Ba129.gydF4y2Ba,gydF4y2Ba130.gydF4y2Ba,gydF4y2Ba131.gydF4y2Ba,gydF4y2Ba132.gydF4y2Ba,gydF4y2Ba133.gydF4y2Ba,gydF4y2Ba134.gydF4y2Ba]. 最近,新一代测序技术的进步促进了基于SNP的芝麻遗传多样性分析。在全球范围内,来自亚洲、欧洲、美洲和非洲的不同芝麻种质的遗传多样性水平较高(表1)gydF4y2Ba4gydF4y2Ba) [gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba36.gydF4y2Ba,gydF4y2Ba135.gydF4y2Ba,gydF4y2Ba136.gydF4y2Ba].gydF4y2Ba
GWAS人口的发展gydF4y2Ba
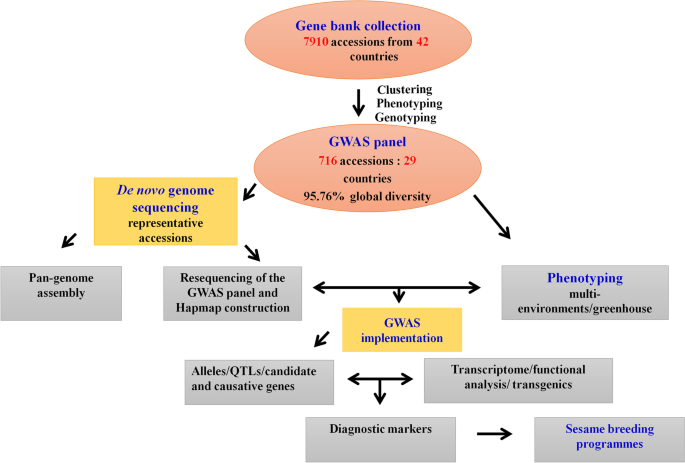
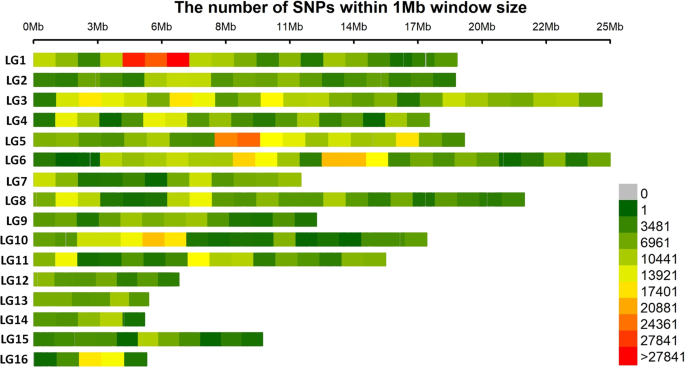
在中国,有8000多份芝麻种质存放在中国农业科学院油料作物研究所的中国中期基因库中[gydF4y2Ba14gydF4y2Ba]. 同样,在北京国家长期基因库中保存的约4500份芝麻种质[gydF4y2Ba107gydF4y2Ba](图。gydF4y2Ba1gydF4y2Ba). 基于这些大的收集,建立芝麻核心收集的策略早在2000年就开始使用形态学描述符和后来的分子工具[gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba106gydF4y2Ba,gydF4y2Ba107gydF4y2Ba,gydF4y2Ba137.gydF4y2Ba].最终,芝麻核心收集包括705个不同的住宅区,包括405个地位,来自中国的95个品种,以及28个其他国家的205种,在OCRI建立了[gydF4y2Ba15gydF4y2Ba].整个小组在Illumina HiSeq 2000上重新测序(http:/gydF4y2Bawww.ncgr.ac.cn/gydF4y2BaSesamehapmap),其中总共5,407,981个SNP在基因组中检测,平均每50 bp平均2 snP(图。gydF4y2Ba2gydF4y2Ba).该品种具有表型变异高、群体结构低、群体间遗传分化低、LD适度下降(~88 kb)等理想性状[gydF4y2Ba15gydF4y2Ba].然而,该小组包括的大多数成员(70.1%)只代表一个国家,而其他28个国家仅代表29.9%的成员。此外,虽然非洲是芝麻地方品种多样性的主要来源,但非洲芝麻入选的数量有限(~3%)[gydF4y2Ba108gydF4y2Ba].因此,为了开发重要农艺性状的遗传基础和潜在致病基因的检测,需要更新GWAS群体面板,纳入更多代表世界各地不同农业生态来源的材料。河南省农业科学院芝麻研究中心芝麻研究小组开发了另一种关联图谱面板人口[gydF4y2Ba122.gydF4y2Ba,gydF4y2Ba136.gydF4y2Ba]由366种种质豁免组成,占中国约89.9%,其余10.1%来自11个国家。该人群还表现出高表型和遗传多样性,相对良好的SNP密度(每2.6 kB,总共4.6kb,总共42,781个SNP)和中等衰减(〜99 KB)[gydF4y2Ba122.gydF4y2Ba]. 然而,该小组的地域代表性也有限。最近,从韩国核心收藏品中构建了更多的GWAS面板。然而,群体规模和SNP密度很低:96份材料和5962个SNP[gydF4y2Ba36.gydF4y2Ba]; 87份材料和8883个单核苷酸多态性[gydF4y2Ba135.gydF4y2Ba]. 总的来说,为了探索经济上重要的农艺性状的遗传基础并确定可能的致病基因,这些已开发的GWAS面板需要通过提供更多反映全球不同农业生态背景的材料来更新。gydF4y2Ba
在sesame中实现GWAS的优点和局限性gydF4y2Ba
优势gydF4y2Ba
基于高质量基因组序列的GWA的实现通常在更准确的预测和潜在致病基因的预测和开采中。沿整个染色体的基因组中SNP的高分辨率定位可以解开靶性特征的遗传架构;因此,GWA可以检测具有高功率和效率的更重要的关联,候选基因和基因组位置。自2014年以来,发展芝麻基因型的高质量草案草案“中志13”[gydF4y2Ba14gydF4y2Ba开启了芝麻基因组研究的大门。芝麻有一个小的二倍体基因组,估计为350 Mb,其中拼接了274 Mb草案基因组,预测了27148个蛋白编码基因。在同一时期,现代品种‘玉之1’的另一个基因组序列也发表了[gydF4y2Ba138.gydF4y2Ba].与减少测序成本相关的基因组测序技术的进展已经为芝麻中额外的基因组测序项目产生了机会。参考基因组被更新为具有更高分辨率[gydF4y2Ba39.gydF4y2Ba]以及“白芝麻”和“密苏里芝麻”等不同芝麻地方品种的基因组序列[gydF4y2Ba15gydF4y2Ba]和现代品种的“swetha”[gydF4y2Ba139.gydF4y2Ba]也发表了。此外,对5个不同基因组的芝麻盘基因组进行组装,鉴定出15890个可分离的基因,为利用GWAS进行全面的基因发现和优势等位基因挖掘提供了丰富的资源[gydF4y2Ba94gydF4y2Ba].同样,来自不同芝麻组织,各种生长条件和野生的巨大转录组数据的可用性gydF4y2Basesamum.gydF4y2Ba物种,如gydF4y2BaS放射体gydF4y2Ba和gydF4y2BaS木兰gydF4y2Ba(表gydF4y2Ba5gydF4y2Ba) (gydF4y2Bahttps://www.ncbi.nlm.nih.gov/bioproject/?Texm=((sesame)%20and2020%22sesamum%20indicum%22[orgn:__txid4182] )%20and%20bioproject_sra[filter]%20not%20bioproject_gap.[筛选gydF4y2Ba])促进GWAS尤其用于确定候选基因及其功能分析。几个映射种群的可用性[gydF4y2Ba11gydF4y2Ba]对于验证或完善GWAS发现也非常有用。此外,功能基因组数据库如Sinbase的可用性(gydF4y2Bahttp://ocri-genomics.org/Sinbase/index.htmlgydF4y2Ba),sesamefg(gydF4y2Bahttp://sesame-bioinfo.org/SesameFG/gydF4y2Ba)和已经部署以促进基因组挖掘,比较基因组学,基因表达分析的芝麻Hapmap对Gwas调查非常有用[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba105gydF4y2Ba,gydF4y2Ba140.gydF4y2Ba].gydF4y2Ba
为了进一步促进对GWAS结果的开发以及芝麻中可用的所有遗传发现,我们开发了一个名为的新型数据库gydF4y2Ba芝麻纪录gydF4y2Ba基因发现资料库(SiGeDiD) (gydF4y2Bahttp://sigedid.ucad.sn/gydF4y2Ba). SiGeDiD是一个灵活的所有遗传和基因组发现的在线目录,包括芝麻中的候选基因、QTL和功能分子标记(图。gydF4y2Ba3.gydF4y2Ba).它是芝麻GWAS项目的比较分析的重要平台,促进基因发现,特别是鉴定了从不同GWA和其他遗传/基因组研究中鉴定的抗性基因组区域/基因。该网站是用户友好的,我们集成了一个模块,允许研究人员在SigeDid中直接上传他们的发现。目前,BLAST功能不可用,但将更新SIGEDID以使其更加互动和功能。gydF4y2Ba
SiGeDiD:芝麻功能基因组发现的在线目录(gydF4y2Bahttp://sigedid.ucad.sn/gydF4y2Ba)gydF4y2Ba
统称,巨大的基因组资源的可用性,芝麻的小基因组大小,综合的GWAS面板,多样化的映射群体,高遗传多样性,低人口结构,相对较低的LD是在芝麻中的GWAS实施是有利的。gydF4y2Ba
局限性gydF4y2Ba
虽然GWAS为研究一系列与重要农艺性状相关的新基因提供了一个机会,但这种方法并不一定能确定致病性变体和基因[gydF4y2Ba141.gydF4y2Ba]. 当GWAS完成后,通常有必要采取额外的步骤来研究功能性和因果性变体以及它们的目标基因,在这些基因中转基因实验可能最终得以实施。然而,芝麻是一种难以进行遗传转化的植物,因此利用转基因方法对GWAS鉴定的SNPs进行验证是有限的。此外,虽然芝麻的LD衰减率相对低于水稻等其他自花授粉作物(~100-350) kb)[gydF4y2Ba142.gydF4y2Ba,gydF4y2Ba143.gydF4y2Ba],大豆(~574 kb)[gydF4y2Ba144.gydF4y2Ba,gydF4y2Ba145.gydF4y2Ba]和芸薹属植物(~405kb)[gydF4y2Ba146.gydF4y2Ba]与玉米等其它异花授粉种相比,其LD衰减率较高(约5.39-15.53kb)[gydF4y2Ba147.gydF4y2Ba].因此,芝麻中报道的LD衰减率(88kb)的适度水平表明,除非使用高标记密度,否则GWAS分辨率可能不容易地解析到致病基因。因此,GWA可以在没有高标记密度的情况下对基于特征的QTL区域或致病基因检测具有有限的效率。在芝麻中的另一个限制是许多芝麻的品种是高度光敏的,因此难以在世界各个地区中的现场表型和收集可靠的数据。gydF4y2Ba
GWAS在芝麻中的应用gydF4y2Ba
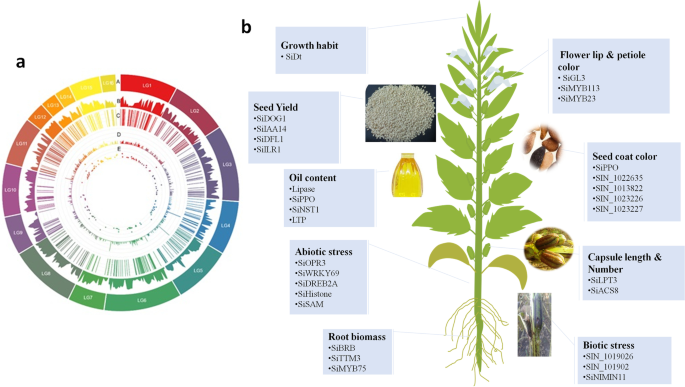
从2015年起,在芝麻上成功实施了多个GWAS项目,揭示了芝麻含油量、含油营养成分、种子产量、产量相关成分、种皮颜色、形态特征、抗病耐盐性、耐涝性、抗旱性等关键农艺性状的遗传基础,根系性状与营养价值[gydF4y2Ba15gydF4y2Ba,gydF4y2Ba33.gydF4y2Ba,gydF4y2Ba34.gydF4y2Ba,gydF4y2Ba35.gydF4y2Ba,gydF4y2Ba36.gydF4y2Ba,gydF4y2Ba135.gydF4y2Ba,gydF4y2Ba136.gydF4y2Ba,gydF4y2Ba148.gydF4y2Ba]. 据我们所知,到目前为止,在sesame进行的所有GWAS项目都是基于单轨迹方法(EMMA),大多数是在OCRI-CAAS开发的GWAS小组上实施的。在这项工作中,我们总结了不同的芝麻研究小组报告的所有GWAS结果(表1)gydF4y2Ba6gydF4y2Ba还有无花果。gydF4y2Ba4gydF4y2Ba).通过研究基于169组表型数据,包括油含量,营养成分,产量组分,形态学特征,生长循环,着色和抗病性,通过研究705芝麻涂层的自然变化进行大规模的GWA进行。总共使用1,805,413个SNP。这导致鉴定446显着相关的SNP,具有表型变异。在深入分析主要基因座之后,共有46个致病基因,包括与花唇颜色有关的基因(gydF4y2Ba西格尔3gydF4y2Ba),叶柄颜色(gydF4y2BaSimyb113.gydF4y2Ba和gydF4y2BaSIMYB23.gydF4y2Ba),含油量(gydF4y2BaSippogydF4y2Ba)脂肪酸生物合成(gydF4y2BaCXE17型gydF4y2Ba和gydF4y2BaGDSL公司gydF4y2Ba-如脂肪酶)和产量(gydF4y2BaSiACS公司gydF4y2Ba)已确定[gydF4y2Ba15gydF4y2Ba].同样,还进行了39个屈服相关性状的GWA [gydF4y2Ba34.gydF4y2Ba]使用与先前研究相同的人群[gydF4y2Ba15gydF4y2Ba]. 共鉴定出646个与感兴趣性状相关的基因座和48个与功能基因座显著相关的潜在基因。他们报道了几个与种子形成有关的候选同源基因和一些新的候选基因(gydF4y2Ba西尔普特3gydF4y2Ba和gydF4y2BaSiACS8型gydF4y2Ba)可以控制胶囊长度和胶囊号[gydF4y2Ba34.gydF4y2Ba].同样地,在490个不同的芝麻加入中研究了PEG诱导的干旱胁迫和盐胁迫耐受性的变化(代表亚洲,非洲,美国和欧洲的33个国家)基于GWA [gydF4y2Ba33.gydF4y2Ba]. 共有132个显著的SNPs解析为9个qtl,共有151个基因gydF4y2BaSiEMF1gydF4y2Ba,gydF4y2BaSiGRV2gydF4y2Ba,gydF4y2Basicyp76c7.gydF4y2Ba,gydF4y2BaSiGRF5型gydF4y2Ba,gydF4y2Ba硅镉8gydF4y2Ba,gydF4y2Ba信号3gydF4y2Ba,gydF4y2BaSIGDH2.gydF4y2Ba,gydF4y2Ba西拉巴1dgydF4y2Ba被检测为调节干旱胁迫的潜在基因,同时耐盐耐受,共有120例显着的SNP分解为15 QTLS和其中241个基因gydF4y2BaSilhcb6.gydF4y2Ba,gydF4y2BaSiMLP31型gydF4y2Ba,gydF4y2Ba西波德gydF4y2Ba,gydF4y2BaSiHSFA1型gydF4y2Ba,gydF4y2BaSiDUF538gydF4y2Ba,gydF4y2BaSiCC国家统计局gydF4y2Ba-LRR公司,gydF4y2Ba西乌德gydF4y2Ba,gydF4y2Ba信号3gydF4y2Ba,gydF4y2BaSINAC43.gydF4y2Ba,gydF4y2BaSIGDH2.gydF4y2Ba,gydF4y2BaSiCP24gydF4y2Ba,gydF4y2Ba西沃基14gydF4y2Ba,gydF4y2Ba六X五gydF4y2Ba,gydF4y2Ba六十六gydF4y2Ba,和gydF4y2BaSiG6PD1型gydF4y2Ba被检测为潜在基因[gydF4y2Ba33.gydF4y2Ba]. 随后,GWAS研究了400份芝麻种质的抗旱性遗传变异[gydF4y2Ba35.gydF4y2Ba]. 共鉴定出140个可靠稳定的qtl,解析为10个qtl。同样,120个基因,其中gydF4y2Ba锡亚比4gydF4y2Ba,gydF4y2BaSITTM3,SIGOLS1.gydF4y2Ba,gydF4y2Ba中华人民共和国1gydF4y2Ba,和gydF4y2BaSiSAMgydF4y2Ba对芝麻的抗旱性具有很高的调控潜力[gydF4y2Ba35.gydF4y2Ba]. 他们的研究首次用转基因方法验证了GWAS候选基因的功能。他们证明,尽管在干旱条件下有助于高产的等位基因远未被固定,但起源于易旱农业生态区的芝麻种质已固定了几个耐旱等位基因。因此,芝麻由于长期适应干旱多发区的农业生态环境,被认为是一种具有抗逆性的作物。最近还报告了其他新的GWAS结果[gydF4y2Ba36.gydF4y2Ba,gydF4y2Ba135.gydF4y2Ba,gydF4y2Ba136.gydF4y2Ba](表gydF4y2Ba6gydF4y2Ba). 基于测序法基因分型[gydF4y2Ba36.gydF4y2Ba]对维生素E进行GWAS,鉴定了8个强连锁SNPs和12个具有多种调控功能的基因,包括转录调节因子HTH、锌离子结合蛋白、糖基磷脂酰肌醇(GPI)锚定生物合成和核糖体蛋白。他们还鉴定了两个基因座LG\u03\u13104062,包含7个基因(gydF4y2Basin_1022039.gydF4y2Ba- - - - - -gydF4y2BaSIN_1022045.gydF4y2Ba)LG_08_6621957包含5个基因(gydF4y2BaSIN_1001936.gydF4y2Ba- - - - - -gydF4y2Ba辛努1001940gydF4y2Ba),利用两种不同的模型(GLM和MLM)分别在lg3和lg8上同时检测。因此,作者认为这两个同时检测到的位点具有很高的控制芝麻中维生素E的潜力。然而,由于在该GWAS中使用的SNPs数量有限(5,962个)和较小的面板大小,这一重要性状的潜在位点可能被遗漏[gydF4y2Ba136.gydF4y2Ba]. 利用42781个单核苷酸多态性的基因型数据和366份芝麻种质组成的关联图谱组的种皮颜色性状,鉴定了224个显著相关的单核苷酸多态性。基于与芝麻种皮颜色显著相关的四个最稳定的峰/单核苷酸多态性,他们保留了92个候选基因。在这些基因中,gydF4y2Ba辛1016759gydF4y2Ba(编码预测的PPO)也在以前的GWAS中报告[gydF4y2Ba15gydF4y2Ba]QTL定位研究[gydF4y2Ba39.gydF4y2Ba]. 利用87份芝麻种质和8883个SNPs的作图关联,对芝麻疫病抗性进行了GWAS分析[gydF4y2Ba135.gydF4y2Ba]. 这项研究的结果表明gydF4y2Ba辛努1019016gydF4y2Ba是与芝麻抗疫病性密切相关的候选基因之一。从GBS方法得到的有限SNP数目和本研究中使用的芝麻材料的相对较小的大小可能会影响与所研究性状相关的GWAS产量。最近,Dossa等人[gydF4y2Ba148.gydF4y2Ba]揭示了7个根系相关性状的遗传基础。他们共报告了409个重要信号,19个qtl,包含32个与芝麻根系性状相关的候选基因。更重要的是,他们发现了一种名为“gydF4y2Ba大根生物量gydF4y2Ba’ (gydF4y2BaSIN_1025576.gydF4y2Ba)通过生长素途径调节芝麻根生物量[gydF4y2Ba148.gydF4y2Ba]. 除了已发表的GWAS研究结果外,OCRI-CAAS芝麻研究小组还就各种农艺性状(包括淹水、叶绿素、苗期盐胁迫)发表了一些未发表的GWAS成果,有趣的是,基于代谢物的GWAS已经完成。这些结果将阐明芝麻中芝麻素/芝麻素等重要代谢物变异的遗传基础。所有候选基因、qtl和snp都将定期加载到SiGeDiD(http://SiGeDiD.ucad.sn/)中,以便在芝麻育种项目中进一步使用。gydF4y2Ba
新统计模型在提高芝麻GWAS准确性和有效性方面的潜力gydF4y2Ba
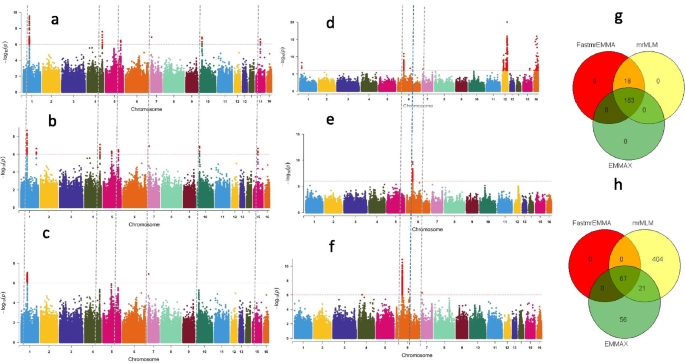
据我们所知,多基因座模型尚未在芝麻GWAS研究中使用,并且之前没有在芝麻中比较不同的GWAS模型(单个轨迹和多基因座型号)。在此,我们基于定量(根长)和定性(种子涂层颜色)特征在芝麻中测试了新的GWAS模型的应用。从Su等人开发的方法后,从场实验中收集了350芝麻饲料的根长的自然变化。[gydF4y2Ba149.gydF4y2Ba]基因型数据来自1000000个常见SNPs。对于种皮颜色GWAS,使用了600份芝麻材料和1000000个普通SNPs[gydF4y2Ba15gydF4y2Ba].为了探讨种子涂层的表型自然变化,收集来自5个胶囊的成熟的种子,并用高分辨率数码相机和种子 - 涂层颜色数据收集并拍摄,基于红色,绿色和蓝色(RGB)值按照张等人采用的方法学方法记录。[gydF4y2Ba150.gydF4y2Ba]. 随后,选择了三个独立的GWAS模型,包括两个多位点模型(mrMLM FASTmrEMMA和mrMLM)和一个单位点模型(EMMAX)(主要是因为它们不需要广泛的表型和基因型数据格式),并使用表型和基因型数据来实现。我们进一步比较了这三个模型的结果,以评估它们在揭示更多标记-性状关联和发现更多候选基因方面的潜力。gydF4y2Ba
我们对这两个性状的GWAS结果表明,FASTmrEMMA、mrMLM和EMMAX分别检测到190、181和162个与根长相关的显著snp(-log10(p)>6)。同样,fastmrema、mrMLM和EMMAX分别检测到67个、492个和143个与种皮颜色相关的显著单核苷酸多态性。gydF4y2Ba5gydF4y2Baf;表格gydF4y2Ba7gydF4y2Ba;表格gydF4y2BaS1gydF4y2Ba).在与根长相关的显著SNPs中,有163个SNPs被三种模型同时识别出来;EMMAX检测到的所有snp也被两种多位点模型同时检测到,而FASTmrEMMA和mrMLM同时检测到18个snp(图1)。gydF4y2Ba5gydF4y2Bag).在种皮颜色相关的SNPs中,3种模型和2种模型(mrMLM和EMMAX)分别检测到67个和27个SNPs(图5)。gydF4y2Ba5gydF4y2Bah).将所有与峰值SNPs在200 kb窗口内共聚为qtl [gydF4y2Ba35.gydF4y2Ba]三个模型共检测到根长和种皮颜色的qtl分别为19个和34个(表1)gydF4y2BaS1gydF4y2Ba).在这些QTL中,我们分别检索26和47个基因,分别用于根长度和种子涂层。基于由用于根长度识别的不同模型,九个潜在候选基因共同检测到的强大QTLS,包括gydF4y2BaSIN_1017810.gydF4y2Ba,gydF4y2BaSIN_101781,SIN_1017812gydF4y2Ba,gydF4y2BaSIN_1017815gydF4y2Ba,gydF4y2Ba辛1017843gydF4y2Ba,gydF4y2BaSIN_1007064gydF4y2Ba,gydF4y2BaSIN_1007065, SIN_1020072gydF4y2Ba和gydF4y2BaSIN_1017818gydF4y2Ba在种皮颜色方面,我们发现的潜在候选基因包括gydF4y2Ba辛欧1007188gydF4y2Ba,gydF4y2BaSIN_1007221.gydF4y2Ba,gydF4y2BaSIN\ U 1023226、SIN\ U 1023227gydF4y2Ba和gydF4y2Ba新宿1023228gydF4y2Ba.有趣的是,Mei等人先报道了本研究中检测到的三种基因。[gydF4y2Ba136.gydF4y2Ba].gydF4y2Ba
新统计多位点模型在芝麻中的应用。gydF4y2Ba一个gydF4y2Ba和gydF4y2BabgydF4y2Ba负对数10gydF4y2BaPgydF4y2Ba分别使用多位点模型mrMLM和FASTmrEMMA绘制根长度(y轴)与SNP位置(x轴)的关联值;gydF4y2BacgydF4y2Ba负对数10gydF4y2BaPgydF4y2Ba用于根长(Y轴)关联的值用单个轨迹型号绘制了根长(y轴)的关联(x轴)emmax;gydF4y2BadgydF4y2Ba和gydF4y2BaegydF4y2Ba负对数10gydF4y2BaPgydF4y2Ba-利用多位点模型mrMLM和FASTmrEMMA分别绘制了种皮颜色(Y轴)与SNP位置(X轴)的关联值;gydF4y2BafgydF4y2Ba负对数10gydF4y2BaPgydF4y2Ba-种皮颜色的关联值(Y轴)用单基因座模型EMMAX绘制在SNP位置(X轴)上。对于这两个特征,水平虚线-点线表示显著性差异gydF4y2BaPgydF4y2Ba值阈值(10gydF4y2Ba-6gydF4y2Ba)显著性单核苷酸多态性以红色突出显示,垂直线表示两个模型中至少有重叠的最显著峰;gydF4y2BaggydF4y2Ba文氏图分别显示了每个模型在根长GWAS中共享的和唯一检测到的显著snp;gydF4y2BaHgydF4y2Ba,文氏图描述了每种模型在种皮颜色GWAS中共享的和唯一检测到的重要snp。该分析的表型和基因型数据来自350份芝麻种质和1000000个根长的普通snp,以及705份芝麻种质和1805413个种皮颜色的普通snpgydF4y2Ba
总体而言,对不同GWAS模型的分析表明,使用综合方法(单点和多点模型)来提高sesame中GWAS的容量和能力是有潜力的。这将有助于发现更多新的标记性状关联和候选基因,特别是在研究数量性状时gydF4y2Ba.gydF4y2Ba值得注意的是,与仅采用单一模型检测的区域相比,在GWAS中被更多模型同时检测到的显著关联区域更有可能与所研究的性状高度相关。因此,为共同检测的相关区域开发诊断标记可加快芝麻分子育种进程。gydF4y2Ba
结论gydF4y2Ba
在过去的五年中,GWAS已经成功地应用于芝麻,并揭示了许多重要农艺性状的遗传基础。尽管已经确定了一系列的qtl(~300)和候选基因(~250)用于定性和定量性状,但更多的性状,包括叶绿素产量、代谢产物GWAS、耐涝性、耐热性等正在研究中。我们设想,所有这些结果将导致发展等位基因特异性诊断标记,作为芝麻育种计划的日常分子工具。尽管已开发出高质量的芝麻参考基因组序列,但更多情况下,在GWAS的峰值SNPs附近找到任何候选基因都存在局限性。为了克服这些限制,我们需要使用最近开发的芝麻泛基因组[gydF4y2Ba94gydF4y2Ba]用于将来的GWAS实现。应通过整合更多主要来自非洲的来自不同农业生态起源的种质和野生物种来改善最近可用的芝麻GWAS面板的多样性。为此,芝麻研究人员之间的国际合作是非常必要的。此外,研究人员之间的合作,利用精确的表型平台和对比环境,生成综合的种质特征数据,将使芝麻复杂性状的遗传结构得到更准确的解剖。努力在研究机构之间共享遗传材料对于加速基因发现至关重要。例如,OCRI产生的705个完全测序的GWAS小组的重新测序数据是公开的,如果种质资源(至少部分)可以与合作伙伴共享,就可以在芝麻上实施更多的GWAS项目,特别是在受环境高度影响的性状上。同样,开发一种SNP芯片可以作为一种快速、低成本、简单的新型芝麻种质基因分型的替代方法,用于未来的GWAS项目。gydF4y2Ba
新的多轨迹GWAS模型的应用以及单轨迹和多轨迹模型的集成将为sesame未来GWAS的实现提供更高的效率和能力。到目前为止,很少有研究证实了芝麻中的大量GWAS发现。因此,需要进行后续研究,以进一步验证在独立群体中从GWAS鉴定出的有利等位基因,并使用其他方法(经典双亲QTL定位、QTLseq等)。利用转基因方法验证GWAS的发现也有助于一些植物物种。在芝麻,遗传转化协议使用组织培养技术已被报道[gydF4y2Ba151.gydF4y2Ba]. 为了开发一种更有效的芝麻遗传转化方法,需要对这一课题进行更多的研究,例如采用浸花技术[gydF4y2Ba152.gydF4y2Ba]. 毛状根遗传转化技术也是一种灵活、快速的研究基因功能的技术,广泛应用于多种顽抗植物中[gydF4y2Ba153.gydF4y2Ba]. 我们建议开发芝麻毛状根遗传转化方案,并结合新的基因组编辑技术来确认一些重要的GWAS发现。最后,应鼓励开展基于GWAS峰SNPs及其有利等位基因的诊断分子标记的研究。这将大大加快芝麻分子育种。gydF4y2Ba
可用性数据和材料gydF4y2Ba
这篇评论文章中使用的数据可以在补充文件和手稿中找到。gydF4y2Ba
缩写gydF4y2Ba
- GWAS公司:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- 身份证号码:gydF4y2Ba
-
连锁不平衡gydF4y2Ba
- QTL:gydF4y2Ba
-
数量性状位点gydF4y2Ba
- 数量:gydF4y2Ba
-
定量性状核苷酸gydF4y2Ba
- sigedid:gydF4y2Ba
-
芝麻纪录gydF4y2Ba发现基因数据库gydF4y2Ba
- 苏格兰国家党:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
工具书类gydF4y2Ba
- 1.gydF4y2Ba
床上亚洲西南部的历史和芝麻的历史和洛略。econ bot。2004; 58(3):329-53。gydF4y2Ba
- 2。gydF4y2Ba
贝迪吉安D。芝麻科芝麻属植物的系统学和进化,第一部分:芝麻及其近缘植物起源的证据。韦比娅。2015;70(1):1–42.gydF4y2Ba
- 3.gydF4y2Ba
Ashri A.芝麻育种。植物品种Rev. 1989; 16:179-228。gydF4y2Ba
- 4.gydF4y2Ba
贝迪吉安D。芝麻:芝麻属。博卡拉顿:华润出版社;2010gydF4y2Ba
- 5。gydF4y2Ba
关键词:芝麻油,芝麻素,芝麻素,热氧化,亚油酸甲酯食品科学技术。2008;41(10):1871-5。gydF4y2Ba
- 6.gydF4y2Ba
Ashakumary L,Rouyer I,Takahashi Y,IDE T,Fukuda N,Aoyama T,等。Sesamin是一种芝麻Lignan,是大鼠肝脂肪酸氧化的有效诱导剂。代谢。1999; 48(10):1303-13。gydF4y2Ba
- 7.gydF4y2Ba
巴拉苏布拉曼尼扬P,帕拉尼亚潘S。大田作物:概述。农学原理与实践。农业生物学,印度,47;2001gydF4y2Ba
- 8.gydF4y2Ba
Alegbejo M,IWO G,Abo M,Idowu A. Sesame:尼日利亚的潜在工业和出口油籽作物。j维持农业。2003; 23(1):59-76。gydF4y2Ba
- 9gydF4y2Ba
FAOSTAT,F.统计数据库,渔业数据(2001)。罗马:联合国的粮食和农业组织;2018.可从互联网获取:gydF4y2Bahttp://www.fao.orggydF4y2BaurlgydF4y2Bahttp://www.fao.orggydF4y2Ba
- 10gydF4y2Ba
Ashri A.芝麻育种。植物品种Rev. 1998; 16:179-228。gydF4y2Ba
- 11gydF4y2Ba
多萨K,迪乌夫D,王L,魏X,张Y,娘M,等。新兴油料作物芝麻进入“组学”时代。前植物科学。2017;8:1154.gydF4y2Ba
- 12.gydF4y2Ba
Weiss E. Castor,Sesame和Safflower;1971年。gydF4y2Ba
- 13。gydF4y2Ba
沃尔斯特·瑞克,Ribaut J-M,Buckler Es,Tuberosa R,Rafalski Ja,Langridge P. Can Genomics促进孤儿作物的生产力吗?NAT BIOTECHNOL。2012; 30(12):1172-6。gydF4y2Ba
- 14。gydF4y2Ba
王L, Yu年代,通C,赵Y, Y,刘歌C, et al。高油料作物芝麻的基因组测序为油的生物合成提供了见解。基因组医学杂志。2014;15(2):1-13。gydF4y2Ba
- 15.gydF4y2Ba
刘魏X, K,张Y,冯Q,王L,赵Y, et al。芝麻油脂生产与品质的遗传发现。Nat Commun。2015;6:8609。gydF4y2Ba
- 16。gydF4y2Ba
黄晓,韩斌。作物全基因组关联研究进展。植物生物学杂志。2014;65:531 - 531。gydF4y2Ba
- 17gydF4y2Ba
温英杰,张宏,倪英利,黄波,张杰,冯英英,等。多基因座全基因组关联研究中混合线性模型的方法学实现。简要说明。2018;19(4):700–12.gydF4y2Ba
- 18gydF4y2Ba
Cubry P、Pidon H、Ta KN、Tranchant Dubreuil C、Thuillet A-C、Holzinger M等。全基因组关联研究精确定位了非洲稻的关键农艺QTL。生物十四。2020gydF4y2Ba
- 19gydF4y2Ba
黄×,桑T,赵Q,冯Q,赵Y,李C,等。水稻地方品种14个农艺性状的全基因组关联研究。纳特·吉内特。2010;42(11):961.gydF4y2Ba
- 20gydF4y2Ba
李鹏,周H,杨H,夏D,刘r,sun p等。基因组关联研究揭示了CMS-WA和CMS-HL在Xian / Indica和Aus ancedo rice(Oryza sativa L.)中生育恢复的遗传基础。米。2020; 13(1):11。gydF4y2Ba
- 21gydF4y2Ba
Yano K,Yamamoto E,Aya K,Takeuchi H,Lo P-C,Hu L等。使用全基因组测序的基因组关联研究迅速识别影响水稻农艺性状的新基因。NAT Genet。2016; 48(8):927。gydF4y2Ba
- 22。gydF4y2Ba
Hufford MB,Xu X,Van Heerwarden J,PyhäJärvi T,Chia J-M,Cartwright RA等。玉米驯化和改良的比较群体基因组学。纳特·吉内特。2012;44(7):808–11.gydF4y2Ba
- 23。gydF4y2Ba
acta photonica sinica, 2011, 41(5): 761 - 768 .焦勇,赵红,任玲,宋伟,曾斌,郭静,等。现代玉米育种中全基因组基因的变化。Nat麝猫。2012;44(7):812 - 5。gydF4y2Ba
- 24。gydF4y2Ba
Alves ML、Carbas B、Gaspar D、Paulo M、Brites C、Mendes Moreira P等。全麦玉米面粉中籽粒组成和面粉糊化行为的全基因组关联研究。BMC植物生物学。2019;19(1):123.gydF4y2Ba
- 25。gydF4y2Ba
Mazaheri M,Heckwolf M,Vaillancourt B,Gage JL,Burdo B,Heckwolf S,等。玉米秸秆生物量的基因组关联分析及玉米解剖学性状。BMC植物BIOL。2019; 19(1):1-17。gydF4y2Ba
- 26。gydF4y2Ba
林M,Matschi S,Vasquez M,Chamness J,Kaczmar N,Basgggio M,等。玉米叶片切割传导的基因组关联研究识别参与角质层发育调节的候选基因。G3基因基因组遗传学。2020; 10(5):1671-83。gydF4y2Ba
- 27。gydF4y2Ba
Morris GP,Ramu P,Deshpande Sp,Hash Ct,Shah T,Upadhyaya HD等。高粱抗组织抗组织特征的群体基因组和基因组基因组。PROC NATL ACAD SCI。2013; 110(2):453-8。gydF4y2Ba
- 28gydF4y2Ba
陶义,赵x,王x,hathorn a,hunt c,cruickshank aw,等。高粱大规模GWAS揭示了谷物中粒度的常见遗传控制。植物Biotechnol J. 2020; 18(4):1093-105。gydF4y2Ba
- 29gydF4y2Ba
Kimani W,Zhang L-M,Wu X-Y,Hao H-Q,Jing H-C。全基因组关联研究表明,不同的途径有助于高粱籽粒品质的变异。BMC基因组学。2020;21(1):112.gydF4y2Ba
- 30.gydF4y2Ba
Lu F, Romay MC, Glaubitz JC, Bradbury PJ, Elshire RJ, Wang T,等。玉米全基因组序列锚点的高分辨率遗传定位。Nat Commun。2015;6(1):1 - 8。gydF4y2Ba
- 31.gydF4y2Ba
Raman H,Raman R,Qiu Y,Yadav AS,Sureshkumar S,Borg L等。GWAS提示开花位点T在油菜开花时间和产量相关性状中的多效性作用。BMC基因组学。2019;20(1):636.gydF4y2Ba
- 32gydF4y2Ba
陆凯,魏莉,李X,王勇,吴杰,刘明,等。全基因组测序揭示了甘蓝型油菜的起源及其改良的遗传位点。自然公社。2019;10(1):1–12.gydF4y2Ba
- 33gydF4y2Ba
Li D,Dossa K,Zhang Y,Wei X,Wang L,Zhang Y,et al.GWAS揭示了芝麻种子萌发期抗旱性和耐盐性的差异遗传基础。基因。2018;9(2):87.gydF4y2Ba
- 34gydF4y2Ba
周R,Dossa K,Li D,Yu J,You J,Wei X等人。芝麻(SESAMUM INDICUM L.)中39种种子产量相关性状的基因组 - 宽协会研究。int j mol sci。2018; 19(9):2794。gydF4y2Ba
- 35gydF4y2Ba
多萨K,李德,周R,于杰,王力,张勇,等。高油作物芝麻抗旱性的遗传基础。植物生物技术杂志。2019;17(9):1788–803.gydF4y2Ba
- 36。gydF4y2Ba
何琦,徐峰,闵明辉,朱绍洪,金国伟,朴永杰。利用芝麻(Sesamum indicum)基因分型进行维生素E全基因组关联研究。基因组的基因。2019;41(9):1085 - 93。gydF4y2Ba
- 37。gydF4y2Ba
Challa S,Neelapu NR.植物非生物胁迫耐受性的全基因组关联研究(GWAS)。植物抗非生物胁迫的生化、生理和分子途径。阿姆斯特丹:爱思唯尔;2018年。135–50.gydF4y2Ba
- 38。gydF4y2Ba
甘蓝型油菜(Brassica napus L.)耐高温性状的全基因组关联研究。作物j . 2018; 6(2): 115 - 25所示。gydF4y2Ba
- 39。gydF4y2Ba
王l,xia q,张y,zhu x,zhu x,li d等。使用新的高密度遗传图谱更新了芝麻基因组组件和植物高度和种子涂层颜色QTL的微图。BMC基因组学。2016; 17(1):31。gydF4y2Ba
- 40gydF4y2Ba
Pritchard JK、Stephens M、Donnelly P。利用多位点基因型数据推断群体结构。遗传学。2000;155(2):945–59.gydF4y2Ba
- 41gydF4y2Ba
Yu J, Pressoir G, Briggs WH, Bi IV, Yamasaki M, Doebley JF,等。关联映射的一个统一的混合模型方法,它考虑了多层次的关联。Nat麝猫。2006;38(2):203 - 8。gydF4y2Ba
- 42gydF4y2Ba
Gupta PK、Kulwal PL、Jaiswal V。作物关联制图:机遇与挑战。遗传学进展。阿姆斯特丹:爱思唯尔;2014年。109–47.gydF4y2Ba
- 43gydF4y2Ba
Widmer C,Lippert C,Weissbrod O,Fusi N,Kadie C,Davidson R等人。进一步改善基因组 - 宽协会研究的线性混合模型。SCI批准。2014; 4(1):1-13。gydF4y2Ba
- 44gydF4y2Ba
acta optica sinica, 2011, 32(5): 689 - 693 .李敏,王强,李敏,等。基因组关联与预测集成工具。生物信息学。2012;28(18):2397 - 9。gydF4y2Ba
- 45gydF4y2Ba
康浩民,蔡仁娜,韦德CM,柯比A,赫克曼D,戴利MJ,等。模式生物组合图中种群结构的有效控制。遗传学。2008;178(3):1709–23.gydF4y2Ba
- 46。gydF4y2Ba
张梓,Ersoz E,Lai C-Q,Todhunter RJ,Tiwari HK,Gore MA,等。适用于全基因组关联研究的混合线性模型方法。纳特·吉内特。2010;42(4):355–60.gydF4y2Ba
- 47。gydF4y2Ba
王S-B,Feng J-Y,Ren W-L,Huang B,周L,Wen Y-J等。通过多基因座混合线性模型方法提高基因组关联研究的功率和准确性。SCI REP。2016; 6:19444。gydF4y2Ba
- 48。gydF4y2Ba
Buzdugan L、Kalisch M、Navarro A、Schunk D、Fehr E、Bühlmann P。多变量全基因组关联分析的统计显著性评估。生物信息学。2016;32(13):1990–2000.gydF4y2Ba
- 49。gydF4y2Ba
布什WS,摩尔JH。全基因组关联研究。《公共科学图书馆学报》,2012;gydF4y2Ba
- 50。gydF4y2Ba
TAMBA CL,NI Y-L,张Y-M。迭代肯定独立筛选eM-Bayesian套索算法,用于多基因座基因组 - 宽协会研究。PLOS计算BIOL。2017; 13(1):E1005357。gydF4y2Ba
- 51.gydF4y2Ba
加文达一世,托沃思P,甘瑟T,奥尔顿F,施密德KJ。利用单标记和单倍型方法对德国冬大麦优良品种进行全基因组关联研究。植物品种。2015;134(1):28–39.gydF4y2Ba
- 52.gydF4y2Ba
关键词:大麦,单snp,多snp,单倍型,关联研究植物基因组。2019;12(3):1 - 14。gydF4y2Ba
- 53.gydF4y2Ba
Bansal V、Libiger O、Torkamani A、Schork NJ。涉及罕见变异的关联研究的统计分析策略。纳特·雷夫·吉内特。2010;11(11):773–85.gydF4y2Ba
- 54.gydF4y2Ba
李丙,傅Y,孙R,王Y,王Q。陆地棉(Gossypium hirsutum L.)纤维品质性状遗传分析的单位点和多位点全基因组关联研究。前植物科学。2018;9:1083.gydF4y2Ba
- 55.gydF4y2Ba
崔勇、张芳、周勇。多位点GWAS在水稻耐盐基因座检测中的应用。前植物科学。2018;9:1464.gydF4y2Ba
- 56gydF4y2Ba
李茹,唐W,张Y-W,陈凯恩,王C,刘y等。基因组 - 高粱五种牧草质量相关性状的基因组协会研究。前植物SCI。2018; 9:1146。gydF4y2Ba
- 57gydF4y2Ba
Segura V,Vilhjálmsson BJ,Platt A,Korte A,SerenÜ,Long Q,et al.一种用于结构化群体全基因组关联研究的有效多位点混合模型方法。纳特·吉内特。2012;44(7):825.gydF4y2Ba
- 58gydF4y2Ba
任W-L,Wen Y-J,Dunwell JM,张Y-M。PKWMEB:kruskal-wallis试验与多基因型基因组协会研究中的多基因背景控制下的经验贝叶斯。遗传。2018; 120(3):208-18。gydF4y2Ba
- 59gydF4y2Ba
张勇,刘平,张×,郑Q,陈M,葛F,等.玉米抗倒伏相关性状的多位点全基因组关联研究揭示了玉米抗倒伏相关性状的遗传结构。前植物科学。2018;9:611.gydF4y2Ba
- 60。gydF4y2Ba
后gwas基因组学时代植物的关联作图。正确答案:遗传学进展。阿姆斯特丹:爱思唯尔;2019.p . 75 - 154。gydF4y2Ba
- 61gydF4y2Ba
Klasen JR,Barbez E,Meier L,Meinshausen N,Bühlmann P,Koornneef M,et al.无需群体结构校正的全基因组关联研究的多标记关联方法。自然公社。2016;7(1):1–8.gydF4y2Ba
- 62gydF4y2Ba
张j,冯j,ni y,wen y,niu y,tamba c等人。PlarmeB:对多层基因组关联研究的经验贝叶斯最小角度回归的整合。遗传。2017; 118(6):517-24。gydF4y2Ba
- 63gydF4y2Ba
TAMBA CL,张Y-M。多基因座基因组关联研究的快速MRMLM算法。生物XIV。2018:341784。gydF4y2Ba
- 64gydF4y2Ba
艾尔斯KL,科德尔HJ。全基因组和候选基因研究中单核苷酸多态性的惩罚logistic回归选择。基因流行病学。2010;34(8):879–91.gydF4y2Ba
- 65gydF4y2Ba
Cordell HJ,Clayton DG。一种统一的逐步回归过程,用于评估使用病例/对照或家族数据的基因内多态性的相对效果:在1型糖尿病中施用HLA。我是j嗡嗡声的遗传。2002; 70(1):124-41。gydF4y2Ba
- 66gydF4y2Ba
关键词:混合模型,结构群体,相关性状,关联研究Nat麝猫。2012;44(9):1066 - 71。gydF4y2Ba
- 67gydF4y2Ba
Turley P,Walters RK,Maghzian O,OkbayA,Lee JJ,Fontana MA,等.使用MTAG进行全基因组关联汇总统计的多性状分析。纳特·吉内特。2018;50(2):229–37.gydF4y2Ba
- 68gydF4y2Ba
詹X,赵N,Plantinga A,Thornton TA,Conneely KN,Epstein MP,等。高维结构性状常见或罕见变异的强大遗传关联分析。遗传学。2017;206(4):1779–90.gydF4y2Ba
- 69gydF4y2Ba
马磊,刘明,颜勇,青聪,张×,张勇,等。玉米胚性愈伤组织再生能力的多位点全基因组关联研究。前植物科学。2018;9:561.gydF4y2Ba
- 70。gydF4y2Ba
徐勇,杨涛,周勇,尹S,李平,刘杰,等。玉米淀粉糊化特性的全基因组关联图谱研究。前植物科学。2018;9:1311.gydF4y2Ba
- 71。gydF4y2Ba
苏杰、马Q、李明、郝夫、王聪。中国早熟陆地棉纤维品质相关性状的多位点全基因组关联研究。前植物科学。2018;9:1169.gydF4y2Ba
- 72。gydF4y2Ba
常飞,郭超,孙飞,张杰,王智,孔杰,等。夏播大豆主茎动态株高与节数的全基因组关联研究。植物科学与技术,2018;gydF4y2Ba
- 73。gydF4y2Ba
彭y,liu h,chen j,shi t,张c,sun d等。面包小麦中六种多基因座模型的自由氨基酸水平的基因组 - 宽协会研究。前植物SCI。2018; 9:1196。gydF4y2Ba
- 74。gydF4y2Ba
Gan X,Stegle O,Behr J,Steffen JG,Drewe P,Hildebrand KL等.拟南芥的多个参考基因组和转录组。自然。2011;477(7365):419–23.gydF4y2Ba
- 75.gydF4y2Ba
Goff Sa,Ricke D,Lan T-H,Presting G,Wang R,Dunn M等。水稻基因组的草稿序列(Oryza sativa L. ssp.paponica)。科学。2002; 296(5565):92-100。gydF4y2Ba
- 76.gydF4y2Ba
国际,R.G.S.P.基于地图的水稻基因组序列。大自然。2005;436(7052):793。gydF4y2Ba
- 77.gydF4y2Ba
于静,胡静,王静,王国胜,李静,刘波,等。水稻基因组序列的初步研究。单一共享平台。印度)。科学。2002;296(5565):79–92.gydF4y2Ba
- 78.gydF4y2Ba
Paterson AH、Bowers JE、Bruggmann R、Dubchak I、Grimwood J、Gundlach H等。高粱双色基因组与禾本科植物的多样性。自然。2009;457(7229):551–6.gydF4y2Ba
- 79.gydF4y2Ba
Schnable PS,Ware D,Fulton RS,Stein JC,Wei F,Pasternak S等.B73玉米基因组:复杂性,多样性和动态性。科学。2009;326(5956):1112–5.gydF4y2Ba
- 80.gydF4y2Ba
王X,王H,王J,孙R,吴J,刘S,等。中多倍体作物油菜的基因组。纳特·吉内特。2011;43(10):1035–9.gydF4y2Ba
- 81.gydF4y2Ba
I.B.G.S.大麦基因组的物理、遗传和功能序列组装。自然。2012;491(7426):711–6.gydF4y2Ba
- 82。gydF4y2Ba
Mayer Kf,Martis M,Hedley Pe,Šimkováh,刘H,Morris Ja,等。通过染色体和比较基因组学解锁大麦基因组。植物细胞。2011; 23(4):1249-63。gydF4y2Ba
- 83。gydF4y2Ba
张刚,刘旭,权哲,程胜,徐旭,潘胜,等。谷子(狗尾草)基因组序列提供了深入了解草的进化和生物燃料潜力。生物科技Nat》。2012;30(6):549 - 54。gydF4y2Ba
- 84。gydF4y2Ba
联盟后卫。块茎作物马铃薯基因组序列分析。大自然。2011;475(7355):189。gydF4y2Ba
- 85gydF4y2Ba
财团TG。番茄基因组序列提供了肉质果实进化的见解。自然。2012;485(7400):635.gydF4y2Ba
- 86gydF4y2Ba
陶Y、赵X、梅斯E、亨利R、乔丹D。探索和利用泛基因组学进行作物改良。摩尔植物。2019;12(2):156–69.gydF4y2Ba
- 87gydF4y2Ba
Tettelin H、Masignani V、Cieslewicz MJ、Donati C、Medini D、Ward NL等。无乳链球菌多重致病分离物的基因组分析:对微生物“泛基因组”的意义。国家科学院学报。2005;102(39):13950–5.gydF4y2Ba
- 88gydF4y2Ba
王W,Mauleon R,Hu Z,Chebotarov D,Tai S,Wu Z等。3,010种亚洲栽培水稻种类变异的基因组变异。自然。2018; 557(7703):43-9。gydF4y2Ba
- 89gydF4y2Ba
赵Q,冯Q,鲁H,Li Y,Wang A,Tian Q等。泛基因组分析突出了栽培和野生稻种的基因组变异程度。NAT Genet。2018; 50(2):278-84。gydF4y2Ba
- 90。gydF4y2Ba
Gage Jl,Vaillancourt B,汉密尔顿jp,曼里克 - 克利佩托尼卡州,Gustafson TJ,Barry K,等。多种玉米参考基因组对不同近亲组合的基因组关联研究鉴定变体。植物基因组。2019; 12(2):1-12。gydF4y2Ba
- 91。gydF4y2Ba
李yh,周g,ma j,姜w,金l-g,张z等。DE Novo组装大豆野生亲属,用于多样性和农艺性状的泛基因组分析。NAT BIOTECHNOL。2014; 32(10):1045-52。gydF4y2Ba
- 92。gydF4y2Ba
胡戈宾,高利奇,拜耳,陈CKK, Tirnaz S, Dolatabadian A,等。同源交换是双二倍体甘蓝型油菜基因存在/缺失变异的主要原因。植物生物学学报;2018;gydF4y2Ba
- 93。gydF4y2Ba
黑山JD,Golicz AA,拜耳PE,Hurgobin B,Lee H,Chan CKK等。六倍体面包小麦的基因组。植物J。2017;90(5):1007–13.gydF4y2Ba
- 94。gydF4y2Ba
余杰,高立芝,卢克,多萨克,张勇,陈杰,等。芝麻地方品种和现代品种全基因组组装的进化和功能特征。植物生物技术杂志。2019;17(5):881–92.gydF4y2Ba
- 95。gydF4y2Ba
Contreras-Mietira B,Cantalapiedra CP,García-Pereira MJ,Gordon SP,Vogel JP,Igartua E等。Get_homologue-EST的植物泛基因组和转录om,植物的分析,一种相同物种序列的聚类溶液。前植物SCI。2017; 8:184。gydF4y2Ba
- 96。gydF4y2Ba
宋建民,关Z,胡J,郭C,杨Z,王S,等。8个高质量基因组揭示了甘蓝型油菜的泛基因组结构和生态型分化。天然植物。2020;6(1):34–45.gydF4y2Ba
- 97。gydF4y2Ba
赵杰,拜耳PE,鲁珀罗P,萨克森纳RK,汗AW,戈利茨AA,等。木豆基因组中的性状关联。植物生物技术杂志。2020;18(9):1946–54.gydF4y2Ba
- 98。gydF4y2Ba
阿斯加尔A,马吉德。巴基斯坦不同芝麻品种的化学特性及脂肪酸特征。Am J Sci Ind Res. 2013;4:54 - 5。gydF4y2Ba
- 99.gydF4y2Ba
贝达尔H。为芝麻理想株型的改良进行育种。植物品种。2005;124(3):263–7.gydF4y2Ba
- One hundred.gydF4y2Ba
Kobayashi T、Kinoshita M、Hattori S、Ogawa T、Tsuboi Y、Ishida M等。芝麻金属燃料性能代码的开发。Nucl技术。1990;89(2):183–93.gydF4y2Ba
- 101。gydF4y2Ba
小林T。芝麻的细胞遗传学。植物遗传育种进展。阿姆斯特丹:爱思唯尔;1991年。581–92.gydF4y2Ba
- 102。gydF4y2Ba
Nayar NM,Mehra K。芝麻:它的用途,植物学,细胞遗传学和起源。经济机器人。1970:20–31.gydF4y2Ba
- 103。gydF4y2Ba
PHAM TD,Thi Nguyen T-D,Carlsson As,Bui TM。不同起源的芝麻形态学评价('芝麻型'l。)品种。Aust J Croc SCI。2010; 4(7):498。gydF4y2Ba
- 104gydF4y2Ba
魏伟、张勇、王力、李德、高勇、张×。芝麻10个农艺性状的遗传多样性、群体结构和关联图谱。作物科学。2016;56(1):331–43.gydF4y2Ba
- 105gydF4y2Ba
魏鑫,龚华,于军,刘鹏,王磊,张勇,等。芝麻功能基因组学集成数据库。Sci众议员2017;7(1):1 - 10。gydF4y2Ba
- 106gydF4y2Ba
关键词:芝麻,核心种质,遗传多样性,表型,分子标记,微核心种质BMC麝猫。2012;13(1):102。gydF4y2Ba
- 107gydF4y2Ba
张一X,张一R,华W,王一H,车Z。中国芝麻核心种质地方品种遗传多样性的SRAP和SSR分析。基因基因组学。2010;32(3):207–15.gydF4y2Ba
- 108gydF4y2Ba
Dossa K,Wei X,Zhang Y,Fonceka D,Yang W,Diouf D等人。非洲和亚洲芝麻遗传多样性和人口结构分析,作为其培养的主要中心。基因。2016; 7(4):14。gydF4y2Ba
- 109gydF4y2Ba
Cho Y-I,Park J-H,Lee C-W,Ra W-H,Chung J-W,Lee J-R,等。利用微卫星标记评价芝麻(Sesamum indicum L.)的遗传多样性和群体结构。基因基因组学。2011;33(2):187–95.gydF4y2Ba
- 110。gydF4y2Ba
Yepuri V、Surapaneni M、Kola VSR、Vemireddy L、Jyothi B、Dineshkumar V等。利用EST衍生的SSR标记评估芝麻(Sesamum indicum L.)基因型的遗传多样性。作物科学与生物技术杂志。2013;16(2):93–103.gydF4y2Ba
- 111。gydF4y2Ba
Park J-H,Suresh S,Cho G-T,Choi N-G,Baek H-J,Lee C-W等。利用简单重复序列标记评估芝麻核心种质的分子遗传多样性和群体结构。植物基因资源。2014;12(1):112–9.gydF4y2Ba
- 112。gydF4y2Ba
悦武,魏路,张T,李C,苗族,张H.SSR标记物质中芝麻种质资源的遗传多样性和人口结构。Acta Agron Sin。2012; 38(12):2286-96。gydF4y2Ba
- 113。gydF4y2Ba
魏伟,张Y,吕H,王L,李D,张X。芝麻种质群体结构及含油量的关联分析。科学农业罪。2012;45(10):1895–903.gydF4y2Ba
- 114。gydF4y2Ba
Wei W,张Y,吕H,Li D,Wang L,张X.中国芝麻多样化面板质量特征的关联分析(芝麻型L.)种质。J Intent Plant Biol。2013; 55(8):745-58。gydF4y2Ba
- 115。gydF4y2Ba
吴克,杨梅,刘H,陶Y,梅J,赵Y。利用插入缺失(InDel)和简单序列重复(SSR)标记对芝麻品种进行遗传分析和分子生物学特性研究。BMC基因。2014;15(1):35.gydF4y2Ba
- 116。gydF4y2Ba
Akbar F、Rabbani MA、Masood MS、Shinwari ZK。利用RAPD标记研究巴基斯坦芝麻种质资源的遗传多样性。Pak J机器人。2011;43(4):2153–60.gydF4y2Ba
- 117。gydF4y2Ba
Al-Somain BHA,Migdadi HM,Al-Faifi SA,Alghamdi SS,Muharram AA,Mohammed Na等。利用序列相关的扩增多态性标志物评估从不同生态区域收集的芝麻遗传多样性。3生物技术。2017; 7(1):82。gydF4y2Ba
- 118.gydF4y2Ba
Arriel NHC,Di Mauro Ao,Arriel EF,Unêda-trevisoli Sh,Costa MM,BárbaroIM,等。基于形态学和农艺性状的芝麻遗传分歧。作物品种苹果生物科技。2007年:253-61。gydF4y2Ba
- 119.gydF4y2Ba
Basak M、Uzun B、Yol E。利用双消化RAD-Seq开发的全基因组SNPs研究地中海芝麻核心种质的遗传多样性和群体结构。公共科学图书馆一号。2019;14(10):e0223757。gydF4y2Ba
- 120.gydF4y2Ba
贝迪亚D.芝麻的演变重新审视:驯化,多样性和前景。遗传资源作物evol。2003; 50(7):779-87。gydF4y2Ba
- 121gydF4y2Ba
贝迪亚D,Smyth C,Harlan JR。形态变异insesamum indipum的模式。econ bot。1986; 40(3):353-65。gydF4y2Ba
- 122.gydF4y2Ba
Cui C,Mei H,Liu Y,张H,郑Y遗传多样性,人口结构和芝麻基因组SNP标记揭示的结合映射面板的衔接性和联系不平衡。前植物SCI。2017; 8:1189。gydF4y2Ba
- 123.gydF4y2Ba
Dar AA,Mutigunda,Mittal PK,Arumugam N.患有RAPD和SSR标记的芝麻型遗传多样性的比较评估。3生物技术。2017; 7(1):10。gydF4y2Ba
- 124.gydF4y2Ba
de Sousa Araújo E、Arriel NHC、dos Santos RC、de Lima LM。利用SSR标记和形态农艺性状评价芝麻种质的遗传变异。澳大利亚作物科学杂志。2019;13(1):45.gydF4y2Ba
- 125。gydF4y2Ba
Dossa K,Wei X,Li D,Fonceka D,Zhang Y,Wang L,等.芝麻AP2/ERF转录因子超家族的研究及干旱胁迫下DREB亚家族的表达谱分析。BMC植物生物学。2016;16(1):171.gydF4y2Ba
- 126。gydF4y2Ba
ERCAN AG,Taskin M,Tutgut K.使用RAPD标记的土耳其芝麻(SesaMum Indicum L.)群体遗传多样性分析gydF4y2Ba⋆gydF4y2Ba.遗传资源作物evol。2004; 51(6):599-607。gydF4y2Ba
- 127。gydF4y2Ba
Gebremichael DE,Parzies香港。埃塞俄比亚芝麻地方品种的遗传变异。作物科学杂志。2011;19(1).gydF4y2Ba
- 128。gydF4y2Ba
Hika G,Geleta N,Jaleta Z。埃塞俄比亚芝麻(Sesamum indicum L.)群体表型性状的遗传变异性、遗传力和遗传进展。科技艺术研究杂志。2015;4(1):20–6.gydF4y2Ba
- 129gydF4y2Ba
Pandey SK,DAS A,RAI P,Dasgupta T.形态学和遗传多样性芝麻(芝麻标记L.)征地的索引。physiolmol biol植物。2015; 21(4):519-29。gydF4y2Ba
- 130gydF4y2Ba
关键词:芝麻,农艺性状,ISSR,遗传变异李建平。2011;47(3):314。gydF4y2Ba
- 131gydF4y2Ba
Pham TD、Geleta M、Bui TM、Bui TC、Merker A、卡尔森AS。越南和柬埔寨芝麻遗传多样性的农艺形态学和分子标记比较分析。遗传病。2011;148(1):28–35.gydF4y2Ba
- 132。gydF4y2Ba
魏旭,王璐,张勇,齐旭,王旭,丁旭,等。芝麻(Sesamum indicum)基因组简单重复序列(SSR)标记的建立。分子。2014;19(4):5150–62.gydF4y2Ba
- 133。gydF4y2Ba
Wei X,Zhu X,Yu J,Wang L,Zhang Y,Li D等。鉴定兰斯和品种基因组比较的芝麻基因组变异。前植物SCI。2016; 7:1169。gydF4y2Ba
- 134。gydF4y2Ba
Woldesenbet DT、Tesfaye K、Bekele E。芝麻种质资源的遗传多样性:保存、改良和利用的意义。国际生物技术分子生物学研究杂志,2015年;6(2):7–18.gydF4y2Ba
- 135。gydF4y2Ba
Asekova S,OH E,Kulkarni Kp,Lee Mh,Kim Ji,Pae S-B等。逐步QTL映射和基因组关联分析的组合方法鉴定了芝麻植物的植物基因。生物XIV。2020;gydF4y2Bahttps://doi.org/10.1101/2020.03.18.996637gydF4y2Ba.gydF4y2Ba
- 136。gydF4y2Ba
梅H,崔C,刘Y,刘Y,崔X,杜Z,等。芝麻种皮颜色的全基因组关联研究。公共科学图书馆一号。2020gydF4y2Bahttps://doi.org/10.21203/rs.2.18296/v2gydF4y2Ba.gydF4y2Ba
- 137。gydF4y2Ba
郑秀荣,郑英忠,郑勇,郑祥云,郑庆元,郑明德,等。中国芝麻种质核心种质的建立。基因资源作物进化。2000;47(3):273–9.gydF4y2Ba
- 138。gydF4y2Ba
张慧,缪慧,王玲,曲玲,刘慧,王强,等。重要油料作物芝麻的基因组测序。基因组医学杂志。2013;14(1):401。gydF4y2Ba
- 139。gydF4y2Ba
Kitts Pa,Church Dm,Thibaud-Nissen F,Choi J,Hem V,Sapojnikov v等。组装:NCBI在组装基因组的资源。核酸RES。2016; 44(D1):D73-80。gydF4y2Ba
- 140.gydF4y2Ba
关键词:芝麻,基因组学,遗传学,比较基因组学,Sinbase植物生理学杂志。2015;56(1):e2。gydF4y2Ba
- 141.gydF4y2Ba
TAM V,Patel N,Turcotte M,Bosséy,Parég,Meyre D.基因组协会研究的益处和局限性。NAT Rev Genet。2019; 20(8):467-84。gydF4y2Ba
- 142.gydF4y2Ba
李宁,郑宏,崔杰,王杰,刘宏,孙杰,等。粳稻种质苗期耐碱性全基因组关联研究及候选基因分析。米饭。2019;12(1):24.gydF4y2Ba
- 143.gydF4y2Ba
张平、钟克、钟Z、童H。水稻核心种质重要农艺性状的全基因组关联研究。BMC植物生物学。2019;19(1):259.gydF4y2Ba
- 144.gydF4y2Ba
Hyten DL,Choi I-Y,Song Q,Shoemaker Rc,Nelson RL,Costa JM等。多种大豆群中的连锁不平衡的高度可变模式。遗传学。2007; 175(4):1937-44。gydF4y2Ba
- 145gydF4y2Ba
李敏,刘勇,陶勇,徐超,李旭,张旭,等。通过全基因组关联研究确定大豆开花相关的遗传位点和候选基因。BMC基因组学。2019;20(1):987。gydF4y2Ba
- 146.gydF4y2Ba
吴卓,王斌,陈鑫,吴杰,王光军,肖勇,等。利用ddRAD测序评价甘蓝型油菜种子含油量连锁不平衡模式及关联研究。《公共科学图书馆•综合》。2016;11 (1):e0146383。gydF4y2Ba
- 147.gydF4y2Ba
Rashid Z,Singh PK,Vemuri H,Zaidi PH,Prasanna Bm,Nair Sk。亚洲适应热带玉米的基因组关联研究揭示了高粱稳定抗性的新颖和探索的基因组区域。SCI批准。2018; 8(1):1-12。gydF4y2Ba
- 148.gydF4y2Ba
杜萨K,周R,李德,刘A,秦L,马马迪,等。一个孤儿基因5'-UTR中的新基序gydF4y2Ba大根生物量的gydF4y2Ba在芝麻中调制根生物量。植物Biotechnol J. 2020。gydF4y2Bahttps://doi.org/10.1111/pbi.13531gydF4y2Ba.gydF4y2Ba
- 149。gydF4y2Ba
苏锐,周锐,马曼迪,李丹,秦磊,刘安,等。芝麻(Sesamum indicum L.)根系多样性:形态、解剖和基因表达谱的洞察足底。2019;250(5):1461 - 74。gydF4y2Ba
- 150gydF4y2Ba
关键词:芝麻,种皮颜色,遗传分析,QTL定位,QTL定位8。2013;(5):e63898。gydF4y2Ba
- 151gydF4y2Ba
一个新的渗透素样蛋白基因(SindOLP)的过度表达赋予了芝麻对生物和非生物胁迫的耐受性。植物科学与技术,2017;gydF4y2Ba
- 152gydF4y2Ba
Martins PK,Nakayama TJ,Ribeiro AP,da Cunha BADB,Nepomuceno AL,Harmon FG,et AL.狗尾草花浸:一种简单快速的农杆菌介导的转化方法。生物技术代表,2015年;6:61–3.gydF4y2Ba
- 153gydF4y2Ba
Gomes C,Dupas A,Pagano A,Grima-Pettenati J,Paiva Jap。毛状根转化:探索基因功能和Salix SPP中表达的有用工具。转型顽固。前植物SCI。2019; 10:1427。gydF4y2Ba
致谢gydF4y2Ba
本文汇总的数据已经通过了几位作者的作品产生了我们要感谢他们不断努力的芝麻作物的持续努力。我们也很感谢Muhammad Amjad Nawaz博士在绘制芝麻厂的帮助下。gydF4y2Ba
基金gydF4y2Ba
本研究得到武汉省科技创新基金(2018020401011303)、湖北省科技创新项目(201620000001048)、中国湖北自然科学基金(2019CFB54)、中央非营利科研机构基础研究经费(1610172019004)的资助。中国农业科学院农业科技创新项目(CAAS-ASTIP-2013-OCRI)和中国农业研究系统(CARS-14)。资助者在研究设计、数据收集、分析和解释以及手稿撰写方面没有任何作用。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
M B、K D、L W构思设计了本论文;M B、K D、L W、J Y、D D、X Z收集并分析文献;K D和M B进行了多位点GWAS分析;P A M, I N D, K D, D D设计开发SiGeDiD;M B和K D起草论文并编制数据;L W、J Y、D D、X Z已修改稿件。所有作者都已阅读并批准了稿件的最终版本。gydF4y2Ba
通讯作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用gydF4y2Ba
同意出版物gydF4y2Ba
不适用gydF4y2Ba
利益争夺gydF4y2Ba
作者声明没有利益冲突gydF4y2Ba
附加信息gydF4y2Ba
出版商的注意gydF4y2Ba
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1gydF4y2Ba
:gydF4y2Ba表S1gydF4y2Ba. 利用多位点和单位点模型在GWAS中鉴定出芝麻根长和种皮颜色的qtl总数和候选基因一览表。gydF4y2Ba表S2gydF4y2Ba. 每个GWAS模型检测到的QTL和候选基因的总结。gydF4y2Ba表S3gydF4y2Ba.每个模型中的每个LG检测到的候选基因。gydF4y2Ba
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba. 知识共享公共领域放弃(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非另有用入数据的信用额度。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用本文gydF4y2Ba
Berhe, M., Dossa, K., You, J。gydF4y2Ba等等。gydF4y2Ba全基因组关联研究及其在非模式作物中的应用gydF4y2Ba芝麻纪录gydF4y2Ba.gydF4y2BaBMC植物BIOL.gydF4y2Ba21,gydF4y2Ba283(2021)。https://doi.org/10.1186/s12870-021-03046-021-03046-0.gydF4y2Ba
收到gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
内政部gydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-021-03046-021-03046-0.gydF4y2Ba
关键词gydF4y2Ba
- GWASgydF4y2Ba
- 芝麻gydF4y2Ba
- 统计模型gydF4y2Ba
- 基因组学辅助育种gydF4y2Ba