摘要
背景
利用水稻杂种优势是提高水稻产量的主要途径之一,对粮食安全做出了重大贡献。优良亲本基因型的鉴定和选择,特别是雄性不育系中的优良亲本基因型的鉴定和选择,是利用杂种优势的关键步骤。双系杂交体系是在光敏和温敏核敏雄性不育(PTGMS)材料的发现和应用基础上建立起来的。从一个共同的基因库发展广泛的雄性不育系,导致遗传多样性的狭窄,这是脆弱的生物和非生物胁迫。因此,研究PTGMS系的遗传背景,了解它们之间的关系,对今后的育种策略的选择和设计具有重要意义。
结果
评估了来自中国主要水稻生长区的118条雄性无菌水稻线和13条常规育种线,并针对光敏(项目管理系统3)和温度敏感型雄性不育(TMS5.)基因。总基因库划分为4个主要群体,P1群体拥有项目管理系统3,P2拥有tms5,P3拥有两者项目管理系统3和TMS5.P4含有不含任何雄性不育等位基因的传统育种系。所有群体的纯合等位基因均显示出较高的遗传纯度。种群混合、主成分和系统发育分析显示P2、P3与P4关系密切。种群分化分析表明,P1的分化系数最高。在系统进化树中,P1系与其他3个居群的亲缘关系较好,P2系和P3系与传统居群的亲缘关系较好。构建居群内和居群间遗传多样性最大的前10%的核心种质群,为今后的研究和育种工作提供依据。
结论
P2、P3和P4群体中PTGMS系间的遗传多样性低,亲缘关系密切,可能是与包括P1纯系在内的共同祖先回交的结果。PTGMS小组的核心种质更新了新的多样性种质,将为进一步的两系杂交育种提供最佳服务。
背景
米 (栽培稻L.)是超过50%的全球人口的最重要的谷物和主食,亚洲的90%的大米在亚洲种植[1]. 预计到2050年,世界人口将增至100亿,为养活这一人口,农业生产需要扩大70%。据估计,到2020年,世界人口将需要7.63亿吨大米,到2035年将需要8.52亿吨大米[2那3.].利用杂种优势是提高水稻产量的主要方法之一,并对中国的粮食安全作出了巨大贡献,以及许多其他国家[2那4.].三系杂交水稻(细胞质雄性不育,CMS)和二系杂交水稻(光温敏感核雄性不育)的生产体系是基于雄性不育的。anongs -1是第一个indic我国育成的温敏核不育系开辟了水稻杂种优势利用的新途径[5.].二系杂交体系是在环境敏感核不育(EGMS)材料的发现和应用基础上建立起来的。与三系杂交系统相比,它具有明显的技术优势,容易恢复肥力,在选育杂交稻系时所需的时间和劳力更少[4.那6.那7.].十多年来,光敏或温度敏感的遗传雄性不育(PTGMS)株系占据了中国数百万公顷的稻田[4.].尽管中国的PTGMS线路广泛使用,但缺乏其遗传组成部分的知识[4.].因此,研究PTGMS系的遗传背景,了解它们之间的关系,对今后的育种策略的选择和设计具有重要意义。
优良亲本基因型的鉴定和选择,特别是雄性不育系中的优良亲本基因型的鉴定和选择,是利用杂种优势的关键步骤。根据以往的研究,大多数栽培水稻品种因杂交水稻组合而导致野生脱绿细胞质,分别占中国和印度稻米耕地面积的69%和6.8%[6.那8.那9.].单晶性细胞质可能导致遗传脆弱性。因此,具有新细胞质和更广泛的遗传多样性的优异雄性无菌系的鉴定和利用应该是实现的主要任务。标记辅助选择(MAS)可以是基于其基因型选择植物的最佳工具。
下一代测序(NGS)技术现在可用于植物生物学研究和植物育种[10]. 利用不同的基因分型平台和标记密度,对水稻种质(包括自交系)的遗传多样性、亲缘关系、群体结构、杂种优势群、基因和数量性状位点(QTL)定位进行了研究[11那12那13那14那15].近年来,对水稻等核异质雄性不育系的各种性状,包括雄性不育性、农艺性状、细胞质效应以及对不同胁迫的抗性等也进行了研究[8.].但是,很少有报告基于核基因组的分化。
与其他分子标记相比,单核苷酸多态性芯片技术是目前最快、最准确的植物基因分型方法之一。SNP标记非常丰富,可用于高通量基因分型,在作物育种和遗传应用中非常有用,包括水稻[16].它使研究人员能够方便地选择信息丰富的SNPs子集,用于更小的内部平台,立即应用于各种基因组和分子方法,包括标记辅助或基因组选择,高通量QTL的全基因组多态性和关联作图。它也被用来选择高目标的SNPs集,用于高分辨率单倍型分析和基因发现。几种中密度和高密度芯片阵列已被用于水稻[17].这些SNP检测在不同的密度下得到了发展,例如50 K-SNP芯片[18],C6空气[19, RICE6K [20.],44 k-SNP芯片[21],和700 K-SNP高密度水稻阵列[22]. 水稻中满足这些标准所需的单核苷酸多态性密度是~ 6-7000个标记,这是由于SNP分布和频率的显著差异,这些差异表征了o.苜蓿[19]. 水稻高密度单核苷酸多态性芯片的可用性使得进行大规模、高通量的种质鉴定成为可能,从而提高了世界主要种质资源库中现有遗传资源的价值。
在本研究中,在无菌基因的基础上聚集131根两线PTGM稻米。通过56k全基因组水稻SNP芯片进一步基因分型进一步基因分型,并分析了材料之间的遗传关系。筛选这些材料的核心育种集合,并研究了在育种过程中发生的选择性扫描。结果为可用PTGMS基因库的狭隘遗传基础提供了重要的见解,是未来两线杂种育种和水稻研究计划的参考。
结果
筛选光谱和温度敏感的核基因
评价131个双线雄性无菌系的收集并筛选光敏雄性不育(项目管理系统3)和温度敏感型雄性不育(TMS5.)以确定它们之间的相似性。整个基因库分为四个主要群体。在群体1(P1)中有9.16%(12)个品系,具有较高的亲缘关系项目管理系统3表示为光敏核不育群体。群体2(P2)共有77%(101)株系TMS5.并标记为温敏核雄性不育群体.只有3.82%(5)条线项目管理系统3和TMS5.基因并被归类为人群3(p3)。它们表示为照片/热敏基因雄性无菌群体。其他9.92%(13)条线是没有任何PTGMS基因的正常育种线,分类在群体4(P4)中,并表示为常规育种人群(额外表1).
基因组变异的基因分型和评估
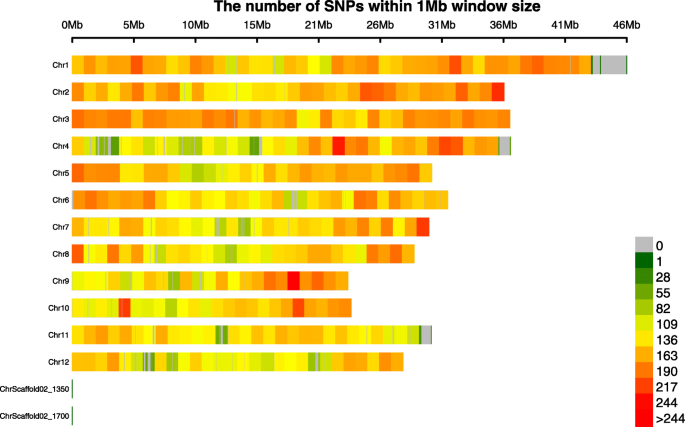
本研究采用56 K SNP标记芯片对131个品系进行基因分型。平均SNP密度为每10 kb基因组区域1.52个SNP,范围从12号染色体的1.32个SNP到3号染色体的1.74个SNP。经过过滤的平均SNP密度为每15.5 kb基因组区域一个SNP,从P4的每9.7 kb一个SNP到P3的每26 kb不等(图1)。1).1号染色体的基因组覆盖率为91%,其余大多数染色体的基因组覆盖率为100%。在基于基因间SNP的检测中,标记密度远远高于12个SNP / Mb [19]但相对低于先前报告的基因单拷贝芯片中每KB 50 KB密度0.745的0.745 [23].因此,本研究中的标记密度适合于研究基因型之间的遗传多样性。
遗传纯度
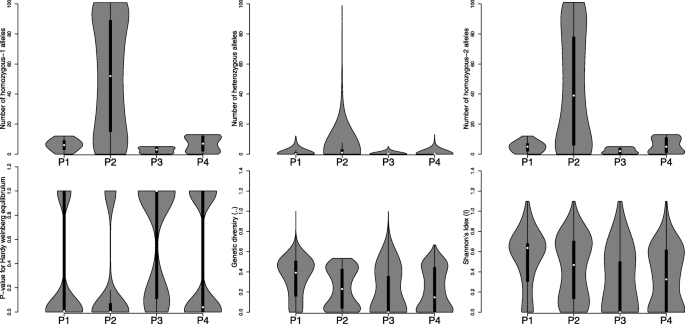
基因型中的遗传纯度可以通过可用的纯合或杂合等位基因评估。在目前的研究中,我们评估了每个基因座的纯合和杂合等位基因的频率,并估计了核苷酸多样性和香农的指数(i)。所有四种种群的基因组高纯合,纯合等位基因93%至97%。在P4(2.66%)中观察到等位基因中最低群体杂合子,然后在P3(3.14%)中,而P1(6.80%),观察到最大杂合子,然后观察到P2(3.70%)(图。2,附加表格1).
全基因组核苷酸多样性
对118个雄性不育系和13个常规育种系的遗传多样性进行了评价。群体的核苷酸多样性(π)较低,为7.54 × 10- 8到4.29 × 10- 4..它表明基因型之间的紧密相关性,并提出了种质中的有限数量的可用签名基因。在所有四个种群中,我们观察到最低的基因组πP4(男性可育)基因型的-值(π= 0.0000339),而遗传多样性最高(π= 0.0000503)在P1。P2 (π= 0.0000348)和P3 (π= 0.0000347)大致相同(图。2,附加表格1). 在每个群体中,P3的遗传多样性范围最广,而P2的遗传多样性最低。
基因组遗传分化
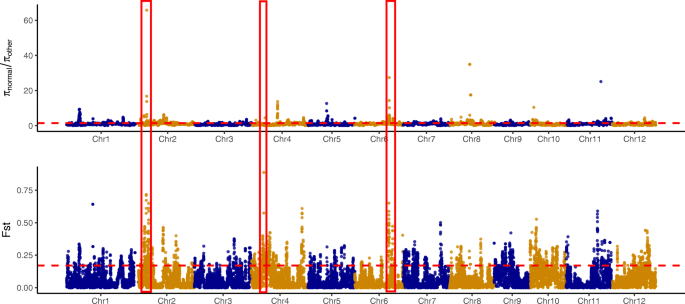
整个雄性不育群体(P1、P2、P3)与常规系的遗传分化程度不高(加权Fst = 0.078),但P1与其他群体的遗传分化程度最高(平均加权Fst > 0.2)。P4(加权Fst = 0.252)最大,其次是P3(加权Fst = 0.178)和P2(加权Fst = 0.108)。3., 桌子1).P3和P4之间的遗传分化水平与P2的P1之间的遗传分化水平类似。另一方面,P3和P4的P2的遗传分化不是非常高(重量FST = 0.045和0.099)。它表明了一个驱动力项目管理系统3和TMS5.在形成遗传变异模式的基因,表明p2和p3的遗传相似性。估计联动不平衡(LD)R2对于位点周围30kb距离内< 1kb的距离类项目管理系统3和tms5。平均R2阈值达到r阈值2在3.9 kb的距离处标记< 0.5TMS5.轨迹虽然这个值保持高于阈值项目管理系统3轨迹(额外的无花果。5.).
四个水稻PTGMS系群体的系统发育聚类分析
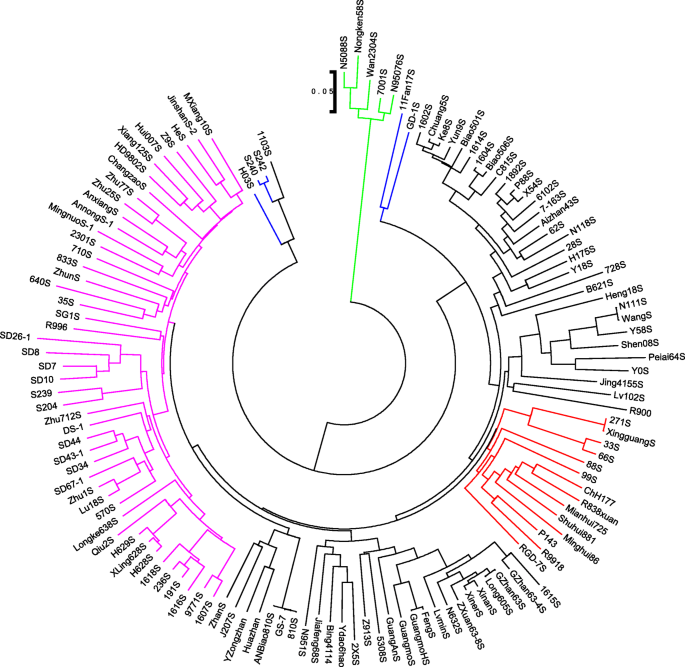
对118个雄性不育系与13个常规育种系进行系统发育分析。在0.05个遗传距离处,根据邻接树(NJ)可将所有株系划分为4个分枝(图1)。4.).其中,第一支包括5个株系(N5088S、农垦58s、Wan2304S、7001S和N95076S)。这些品系的最大遗传距离为0.29,是系统发育树的根。这些品系可能是其他雄性不育系的祖先。第2支由6个基因型组成,其中5个基因型(GD-1S、H03S、S242、S240和1103S)属于P1, 1个基因型(11Fan17S)属于P2。4个基因型(H03S、S242、S240和1103S)聚在一起,与其他基因型的距离较高,而P1 (GD1S)和P2 (11Fan17S)的2个基因型构成其余基因型的根。这些基因型可能是1支系的祖先,也可能是其他基因型的祖先。P2和P3的其余基因型与P4的基因型分组,表明它们的遗传相似性。
稻瘟病四条群体的主要成分分析
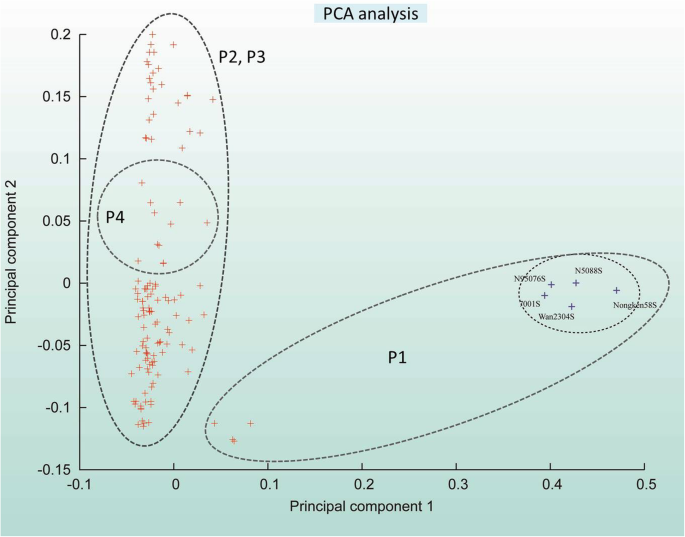
通过主成分分析(PCA)进一步支持结果。PC1和PC2的前两个主要成分分别解释了总变异的19.77%和7.62%,并将种质分为两大类。接下来的两个PC,如PC3和PC4分别解释了总变化的6.23和3.82%(附加图。1).两种簇中的一种与来自其他落后的遗传距离的宽遗传距离分组了两种繁殖线,而其他簇具有来自p2,p3和p4的所有基因型,遗传距离窄。作为其他人的独立集群,群集1遭到了最高级别的遗传分化(表1, 图。5.,附加图。1). 其独特的遗传变异模式也可以在前两个PC中得到证明。随后的集群可以进一步分为三个重叠的组(图。5.).
稻米PTGMS系列群体的混合物聚类分析
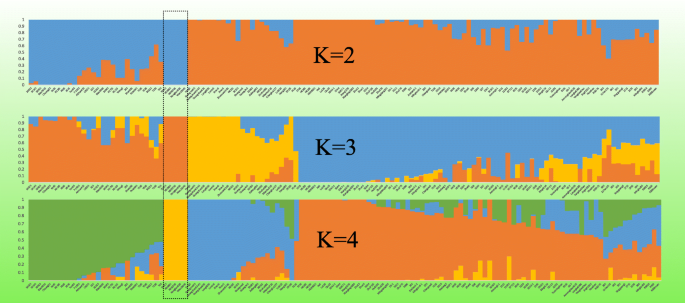
为了推断在131个样品上的混合物程度,我们进一步进行了无监督的混合物分析,基于2-4的k运行,我们发现在K = 2时,P1基因型之间发生遗传发散及其密切亲戚,而K = 3和k = 4分划分组。在k = 4时,将整个种质分别分为四个(S1,S2,S3,S4)28,5,23和75个基因型组。除了在结构分析中被分组的P1中的基因周到五种基因型,在结构分析中,从k = 2至k = 4观察到从p4到其他群体的常规育种线的潜在广泛的育种血液渗入(图。6.). S1、S3和S4共有10个、10个和13个基因纯系,其余均表现为基因组渐渗。这些结果加强了以前纯系和混合基因组系的分析[21那24].在P1,P2和P4的群体中,5,31和1个基因型分别被观察到纯度。相比之下,P4中的所有常规育种线具有狭窄的基因组成分。纯线特别是S2中的基因型可能是剩余种质的祖先。
基因组选择性扫描信号,它们的分子功能和验证
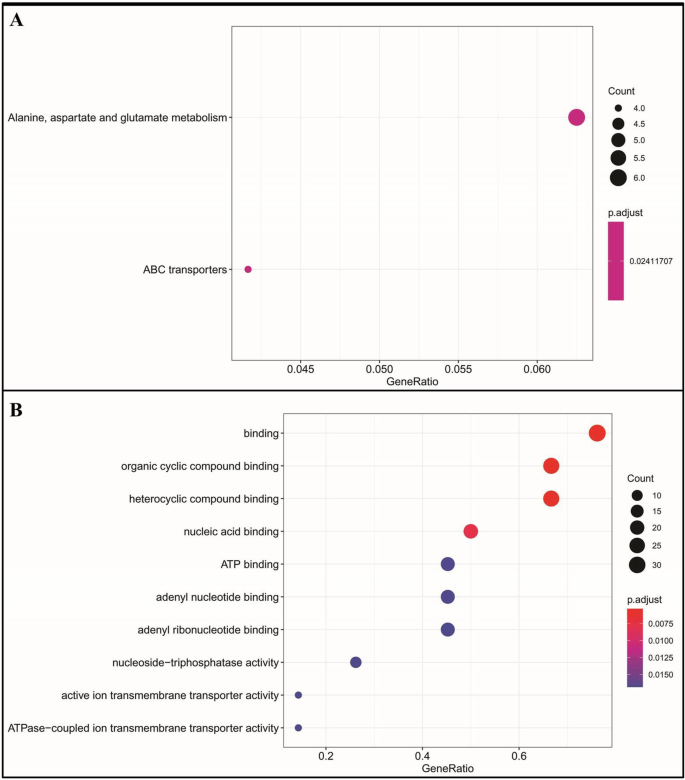
为了更好地检测基因型中与雄性不育相关的全基因组选择信号,我们将群体分为雄性不育(P1,P2,P3)和雄性可育(P4)组。高Fst值(最高1%,Fst值> 0.34)作为选择性扫描的分类标准。在第3、5、8号染色体上没有选择扫描,在其他染色体上共发现1044个候选基因。其中一些基因可能与不育有关(另见表)3.). 位于第1、2、4、6号染色体上的5个扫描点具有较高的Fst值(0.888、0.718、0.652、0.650、0.643),表明雄性不育群体与可育群体之间存在明显的遗传分化。最大的基因组区域为2 在2号染色体和1号染色体上观察到含有376个候选基因的Mb 包含174个候选基因的4号染色体Mb区(附加表2).基因和基因组(Kegg)途径富集分析显示,选择扫描中的候选基因主要涉及“丙氨酸,天冬氨酸和谷氨酸代谢”和“ABC转运蛋白途径”(图。7.A).基因本体(GO)分析发现113个GO项,其中分子结合被确定为最富集的“分子功能”(Fig. 1)。7.B) 是的。
为了进一步验证全基因组选择性扫描信号,根据水稻育种历史,从水稻育种数据库中获得了A、B和C三个家系群(附加图)。2).在血统群A中,有7,15和1个基因型含有项目管理系统3那TMS5.和pms3.tms5.而B组和C组的基因型分别具有不同的基因型TMS5.基因。研究了3个类群的基因组多样性,并与传统基因型(P4)进行了遗传分化。在前5%选择性扫描信号的Fst阈值下,与常规谱线相比,家系A、B和C组共观察到185、182和181个选择扫描信号(附加图。3.,附加表格4.).在前1%的选择性扫描中,我们发现了与雄性不育系(P1、P2和P3)和常规育种系(P4)的顶级遗传分化命中相同的基因组区域。对候选区域的基因进行GO分析。“细胞核”和“膜复合体”中参与“代谢”和“信号转导”过程的“结合”类型的分子功能是最重要的(附加图。4.).
核心种质的选择
根据遗传多样性对所有基因型进行排序,首先选择前30%的基因型。所有聚类分析和雄性不育等位基因的评价将该种质分为4类。根据各群体的有效不育等位基因和基因型间的遗传多样性,对各群体的基因型进行排序。从这两种方法中通常选择的前10%的基因型导致选择13个基因型来发展一个核心集合。其中GD1S、N5088S基因型2个,ZhunS、标506s、申08s、金山、2301、S204、Ke8S、Long605S、66S、Longke638S、99S基因型11个。核心标本中还包括1个P3基因型(S239)和1个常规基因型(曙恢881)。
讨论
作物育种计划的目标是利用遗传多样性获得理想的表现型,以满足人类的需求[25].为了实现理想的基因型,水稻育种计划中精英品种表型选择的瓶颈效应大大缩小了它们的遗传多样性[26].然而,有关产生精英水稻品种所需表型变化的基因的信息是有限的。甚至一些基因也可能导致PGMS线的转变为TGMS线[27].光敏(项目管理系统3)和温度敏感型雄性不育(TMS5.)基因可以将131个双线雄性无菌线的收集分为本研究中的四个主要种群。有关可用PTGMS基因和标记的信息不仅有助于直接选择混合育种的基因型,而且还可以帮助未来的研究计划。基于这些遗传标记,还可以评估和操纵其他种质资源以增强遗传多样性。此外,各种水稻种群的全基因组标记分析表明,遗传群体结构的变化发生了多次历史[28]. 在水稻育种中,利用人类所需的种质资源,可以控制当地基因库中遗传多样性的转移[28].
遗传纯度
通过减少剩余杂合度(异质性)来保持自交系的遗传纯度对优质种子生产很重要[29.]. 阈值可能因品系发展计划的目的和近亲繁殖水平而异。在目前的研究中,所有四个群体的基因组都显示出高水平的纯合性,有93%到97%的纯合等位基因(图。2,附加表格2).通常,雄性无菌线被回复繁殖许多世代。因此,它们可能表现出形态差异,但对核基因组的差异很小。这种级别的异质性可能是由于使用不同方法进行线维护或通过穿越的自然变化。目前,使用遗传纯父母线,特别是双倍单倍体线的均匀杂种,尤其是双倍单倍体线[29.].因此,稻育鳄在新的血统中使用固定线路启动并通过自行进步而不是SIB交配。在改善均匀性的长期解决方案中,建议使用双倍的单倍体(DH)技术在开发遗传纯DH线路中,该纯纯DH线可以在短时间内衍生[29.].
全基因组核苷酸多样性
所有四个种群的核苷酸多样性(π)都很低(图。2,附加表格2),其表明基因型之间的密切关系,并提出了种质中有限数量的可用签名基因。P4中最低的遗传多样性揭示了传统的育种线面临比PTGMS线的密集选择压力。对于P3,观察到最广泛的遗传多样性,而P2中最窄。在雄性无菌群中,在类似水平中选择P2和P3,并且显示比P1更高的选择性扫描。通常,SSR标记观察到亚洲栽培水稻和常见野生稻内的平均遗传分集值为0.67和0.90 [30.]. 对4408份中国栽培稻种质资源的12个同工酶位点进行了分析,结果表明,中国栽培稻种质资源的平均基因多样性在0.012~0.547之间[31.].在我们的实验中,结果表明,测试材料的遗传多样性非常低。它表明基因型和类似血统之间的遗传相似性,导致核基因组多样性降低。
两系杂交稻系间的亲缘关系及群体结构
为了了解遗传混合物,相对血缘关系系数被用作基因型对之间的遗传关系指标。缺乏与更高的关系的较高值缺乏关系的零的值。本研究中的混合物和PCA分析的结果表明,遗传分歧首先发生在P1基因型和近亲之间。每个亚组中纯线的存在强化了人口混合物。通过系统发育分析加强了混合物结果,并揭示了稻米线中的祖先关系(图。4.).总的来说,大多数基因型不能以细胞发育和混合物分析所示,暗示复杂的遗传结构的表达。此外,很难将不同种群的水稻繁殖线分离,表明它们之间的高掺合症。
基因组遗传分化
通过种群分化分析可以评估由于诸如选择或遗传漂移的进化力的完全或部分分离的群体之间的等位基因频率的累积差异。通过FST值研究人口分化。P1人口遗传地与其他人群分化。P4的P3与P2的P1之间没有显着差异。它表明了一个驱动力项目管理系统3和TMS5.在形成遗传变异模式的基因,并表明p2与p3的遗传相似性。结果可能不会完全支持[32.那33.在形成基因组变异时,基于距离的隔离可能已经发挥着关键作用。因此,可以提出强大的人工选择作为成型水稻基因组变异的主要驱动力。
基因组选择性扫描信号和候选基因的作用
通过比较雄性不育组(P1、P2、P3)和雄性可育组(P4),观察到与基因型雄性不育相关的全基因组选择信号。植物中有许多性状,如株高、种子颜色和茎角,都会受到选择性扫描的影响[29.那34.].稻米明显表型评价的研究揭示了选择扫描对水稻育种的生理形态学作用[29.那34.].本研究发现的候选基因在遗传分化和雄性不育诱导中的功能特征。
如曼哈顿图所示(图。3.,附加表格3.),在染色体4(4:7920001-8,040,000)的120 kB间隔内观察到最高的FST分数(0.888)。该地区有19个蛋白质编码基因,包括(i)RPM1抗病蛋白,其促进氧化突发和过敏细胞死亡所需的细胞溶质钙的快速和持续增加([35.], (ii) traf型锌指家族蛋白,其功能非常多样,包括DNA识别、RNA包装、转录激活、凋亡调节、蛋白质折叠和组装以及脂质结合[36.],(iii)壁相关的受体如激酶(奥斯瓦克)蛋白质,它在抵抗一系列真菌和细菌疾病中的中心作用[37.].
两种选择性扫描染色体2(2:4860001-5,080,000&6,080,001-6,600,000)分别包括48和103个候选基因。候选基因的Amon,我们有(i)AP2结构域含有花卉发育,干细胞维持,种子发育和非生物应激抵抗所需的基因[38.那39.](ii)MYB蛋白,它是控制发育、代谢和对生物和非生物胁迫反应的调控网络中的关键因子[40](iii)GDSL样脂肪酶,具有抗菌活性,通过乙烯信号传导调节病原体抗性[41.],(iv)F盒域(OSFBX39.)据报道,这是有助于调节植物中气孔闭合的耐旱性和调节[42.[多种耐药性蛋白质。上述候选基因可能是潜在的影响水稻适应的性状。
通过对P4常规品系与A、B、C三个家系群体的遗传分化的估计,进一步验证了选择扫描的结果。“农垦58”与项目管理系统3,并'安康s-1'和'zhu1s'与TMS5.在A、B和C组中,基因分别被观察为所有其他基因型的根祖先(附加图。2).据报道,安农S-1是第一个indicTGMS线[5.].它在育性表达、不育遗传和生理生化特性方面对温度和光的显著反应也有报道[5.].研究中的双线稻混合系统繁殖面板中的大多数PTGM线都具有TMS5.基因可能是由于安康S-1对其发展的主要贡献产生的。
核心种质的选择
通过鉴定PTGMS株系间的遗传相似性和遗传多样性,设计具有种群间所有可能遗传变异的遗传多样性样品是十分重要的。在大样本中选择一个具有代表性的多样性样本将有助于在育种计划中加强种质资源的利用[43.].选育两系PTGMS核心种质有助于加快种质资源评价和新育种计划的制定。PTGMS资源的开放共享也将有助于减少环境对双系杂交种种子生产的不利影响[44.]. 这样的样品有利于优良的开发和利用的多样性,并将具有成本效益和易于维护的个体育种者[43.].一个核心集合是一个子集,包含总基因型的~ 10%,它们之间捕获了整个集合的大部分可用多样性,几乎与整个集合的多样性相同[24].因此,本研究设计的核心PTGMS集在4个群体中具有最大的多样性,可以使水稻双系PTGMS种质资源得到更高效的利用。这个核心收集应该定期更新新的PTGMS株系和其他生物信息。这些遗传多样性信息将有助于选择适合水稻可持续制种的亲本,并为评价各种农艺上重要的数量性状和质量性状提供依据。
结论
本研究对131个水稻基因型(118个雄性不育系和13个常规育种系)的多样性、亲缘关系、选择扫描和育种推理进行了研究。根据雄性不育基因的存在,这些收集物被明显地分为4个基因库。基于群体内和群体间的遗传多样性,构建了群体内和群体间遗传多样性的核心集合。含有感光基因的群体(项目管理系统3)高度分歧,并建议成为其他人群的祖先。在选择性扫描中鉴定了几种候选基因,并且可以将未来研究的目标解开它们对水稻适应,遗传分化和雄性不育的贡献。
方法
植物材料与基因分型
要深入了解雄性无菌种质的遗传关系并更好地利用水稻育种计划,我们使用了131个基因型,其中包括118个2线PTGMS稻米和13条常规育种线(附加表1).种质是从湖南杂交水稻研究中心,湖南杂交水稻研究中心获得,并在同一研究院的实验农场中生长。采用了所有标准培养实践。收集每种雄性无菌线的至少六种植物的新鲜叶子,并通过简单的CTAB方法提取总DNA,细微修饰[45.].在Huazhi Biotech Co. Ltd.的高密度Illumina 56k Infinium SNP芯片,在中国长沙生物技术有限公司,制备了96孔板格式的DNA样品(每样50-100ng /μl)。单数标记物,具有缺失的值<20%,具有次要等位基因频率<5%,并且从分析中排除不确定的SNP。过滤的基因型数据用于随后的分析。
PCR反应和基因组筛选
两个雄性无菌基因,项目管理系统3和TMS5.对该种质进行了鉴定。采用20 μL(含3 μL引物)、2 μL 10 × PCR缓冲液(含15 mmol/L MgCl)进行聚合酶链反应(PCR)2),0.3μLDNTPS(10mmol / L),50-100ng模板DNA,1 U.Taq酶。反应程序在94°C预变性5 min, 94°C 1 min, 55°C 1 min, 72°C 1 min共35个循环,最后在72°C延长5 min。然后,扩增产物在6%变性聚丙烯酰胺凝胶上进行电泳。在雄性不育基因的基础上,将该种质分为4类,分别为携带“项目管理系统3”、“TMS5.“, 两个都 ”项目管理系统3和TMS5.没有任何雄性不育等位基因的常规育种品系(附加表1).
统计分析
为了研究基因型之间的关系,使用基于模型的程序structure v2.4.2中所有筛选的SNPs计算群体结构[46.]. 每K(种群数量)从1到6进行10次独立的模拟。对于每个模拟,在50000个Markov链montecarlo复制的老化长度之前进行10000次迭代,选择混合物和相关的频率模型。LnP(D)值和最佳K值用Evano's 1估计 K法[46.在线工具结构收割机[47.].并进行了主成分分析。利用VCFtools版本0.1.14计算每个群体内的全基因组多样性(π)和每个群体之间的成对遗传分化[48.],窗口大小为100 kb,步长为20 kb [49.].通过r估计联动不平衡衰减率估计2距离LOCI距离的值。使用30kB距离的SNP对的<1 kB距离窗口。基于Nei基因型遗传距离的NJ方法[50.]使用Mega X [51.[后面是用于种质的系统发育聚类分析。通过Evolview在线工具可视化和编辑树[52.].Powermarker v3.25 [53.用来鉴定育种核心种质。基于PTGMS基因的种质簇在七种可能的组合中比较;1)'pms3'对的tms5”(2)“pms3”vs.'PMS3 + TMS5',3)'PMS3'vs.正常,4)'tms5”vs.' pms3 + tms5 ', ' tms5 'vs.正常,6)'PMS3 + TMS5'vs.7) ' pms3,tms5,pms3 + tms5 '和正常的。将正常基因型与其他(含PTGMS基因)品系的比较结果用于筛选筛选中的候选基因及其GO [54.]和KEGG富集分析[55.].最后,在两种方法中反复发现的总种质的前10%被鉴定为未来水稻育种的核心种质。
可用性数据和材料
植物材料可以在中国湖南杂交水稻研究中心获得。DNA测序数据在登录号下沉积在DDBJ / ENA / Genbank下:QWGC02800000。数据将在出版本手稿后公开发布。
缩写
- CMS:
-
细胞质雄性不育
- DH:
-
双单倍体
- EGMS:
-
环保敏感的遗传性雄性无菌
- 开始:
-
基因本体论
- KEGG:
-
京都基因和基因组百科全书
- 马斯:
-
分子标记辅助选择
- ngs:
-
下一代测序
- NJ:
-
邻居加入
- 主成分分析:
-
主成分分析
- 聚合酶链反应:
-
聚合酶链反应
- PTGMS公司:
-
PhotoCeriod-和热敏遗传敏感雄性无菌
- QTL:
-
定量特质基因座
- SNP:
-
单核苷酸多态性
- TGMS:
-
温度敏感核雄性不育
参考文献
- 1.
脱落酸与非生物胁迫耐受性-不同层次的调控植物生理学杂志。2014;171(7):486-96。
- 2.
程绍华,庄建勇,范莹莹,杜建华,曹丽颖。中国超驯化杂交水稻研究与开发进展。安机器人。2007;100(5):959 - 66。
- 3.
Ullah A,Sun H,Yang X,张X.干旱应对棉花的策略:每滴增加作物。植物Biotechnol J. 2017; 15(3):271-84。https://doi.org/10.1111/pbi.12688.
- 4.
孟辰Z,山W,建坊Y,水勇S,Xin X,QUN X等。中国籼稻P / TGMS线的分类与鉴定。米科。2019; 26(3):195-8。https://doi.org/10.1016/j.rsci.2019.01.003.
- 5。
邓华,舒峰,袁丹。安农S-1型植物的研究与利用概况。杂交水稻。1999;14:1-3。
- 6。
辛格,辛格p,singh dk,singh ak。杂交稻田开发:两行和三线系。j biologix。2013; 178:178-95。
- 7。
徐继,王B,吴y,du p,王j,王m等。PTGMS2-1,水稻光周期热敏基因雄性无菌基因的精细映射和候选基因分析(Oryza Sativa L.)。j理论苹果遗传学。2011; 122(2):365-72。https://doi.org/10.1007/s00122-010-1452-0.
- 8。
刘建强,蔡绍兴,魏龙,程国平。水稻等核异质雄性不育系及其保持系遗传多样性的比较。中国科学(d辑:地球科学)(2007);
- 9。
罗特D,杰娜D,辛格V,阿赫拉瓦特MK,阿索德P,辛格P,等。杂交水稻研究:现状与展望。水稻研究新进展:猪肠粉;2020
- 10.
王伟,毛利安,胡志强,切博塔ov D, Tai S,吴志强,等。3010份亚洲栽培水稻品种的基因组变异。J Nat。2018;557(7703):43-9。https://doi.org/10.1038/s41586-018-0063-9.
- 11.
Marulanda JJ,Mi X,Melchinger Ae,Xu J-L,WürschumT,Longin CFH。利用小麦,玉米,黑麦,大麦,水稻和小麦杂交育种基因组选择的最佳育种策略。j理论苹果遗传学。2016; 129(10):1901-13。https://doi.org/10.1007/S00122-016-2748-5.
- 12.
李杰毅,王杰,蔡格勒.水稻基因组3000计划:未来水稻研究的新机遇与挑战。Gigascience杂志。2014;3(1):2047-2217X-2043-2048。
- 13。
李张拉希德MAR,赵Y, H, J,李z核苷酸多样性,自然变异,进化culm-1灵活和强大culm-2住宿在水稻抗性基因。基因组。2016;59(7):473 - 83。https://doi.org/10.1139/gen-2016-0019..
- 14。
赵y,赵文,江c,王x,xiong h,todorovska eg,等。非SYN GWAS揭示水稻深播种耐受遗传建筑与候选基因。前植物SCI。2018; 9:332。https://doi.org/10.3389/fpls.2018.00332.
- 15.
俞军,熊辉,朱欣,张辉,李辉,苗军,等。通过ho-LAMap,我们发现了OsLG3对水稻粒长和产量的贡献。BMC医学杂志2017;15(1):28。https://doi.org/10.1186/s12915-017-0365-7.
- 16。
Morales-KY,Singh-N,Perez-FA,Ignacio-JC,Thapa-R,Arbelaez-JD等。一个改进的7K单核苷酸多态性阵列C7AIR为水稻育种和遗传学研究提供了大量有效的单核苷酸多态性标记。公共科学图书馆一号。2020;15(5):e0232479。https://doi.org/10.1371/journal.pone.0232479..
- 17。
Singh S, Mahato AK, Jayaswal PK, Singh N, Dheer M, Goel P等。62K基因snp芯片阵列用于鸽子豌豆的遗传研究和育种应用(Cajanus Cajan.L. Millsp。)。SCI代表2020; 10(1):4960。
- 18。
Singh N,Jayaswal PK,Panda K,Mandal P,Kumar V,Singh B,等。基于单拷贝基因的50 K单核苷酸多态性芯片在水稻遗传研究和分子育种中的应用。Sci代表2015;5(1):1–9.
- 19。
Thomson Mj,Singh N,Dwiyanti Ms,Wang Dr,Wright Mh,Perez Fa,等。用于遗传和育种应用的大米6 K SNP阵列大规模部署。米饭(纽约,纽约)。2017; 10(1):40。
- 20.
Yu H,谢W,李继,周F,张Q.一种全基因组SNP阵列(RISH6K)在水稻中的基因组育种。植物Biotechnol J. 2014; 12(1):28-37。https://doi.org/10.1111/pbi.12113.
- 21。
赵克,东CW,埃义达GC,Wright MH,Ali ML,Price Ah等。基因组 - 范围的协会映射揭示了奥里佐苜蓿中复杂性状的丰富遗传建筑。NAT Communce。2011; 2(1):467。https://doi.org/10.1038/ncomms1467.
- 22.
董春华,李永强,李永强,等。水稻全基因组关联图谱的开放获取资源。Nat Commun。2016;7(1):10532。https://doi.org/10.1038/ncomms10532.
- 23.
Singh N, Jayaswal PK, Panda K, Mandal P, Kumar V, Singh B等。50 K单拷贝SNP芯片用于水稻遗传研究和分子育种。Sci众议员2015;5(1):11600。https://doi.org/10.1038/srep11600.
- 24.
张H,张D,王M,Sun J,Qi Y,Li J等。奥雅萨·萨蒂瓦L.在中国的核心收集和迷你核心集合。标记理论和应用的遗传学Theoretische und Angewandte Genetik。2011; 122(1):49-61。https://doi.org/10.1007/s00122-010-1421-7.
- 25。
卢瓦尔群岛。植物育种与多样性:一种纠结的关系?Euphytica杂志。2018;214(7):114.https://doi.org/10.1007/S10681-018-2192-5.
- 26。
藤野K、平山Y、卡吉R。北海道水稻育种中的标记辅助选择。繁殖科学杂志。2019;69(3):383–92.https://doi.org/10.1270/jsbbs.19062.
- 27。
张洪林,陈晓燕,黄建忠,易志国,龚建勇,舒庆勇。中国两系杂交水稻光/温敏感核不育基因的鉴定与转化分析。中国农业科学,2015;48(1):1 - 9。
- 28。
Shinada H,Yamamoto T,Yamamoto E,Hori K,Yonemaru J,Matsuba S等人。水稻栽培北方育种计划中人口结构的历史变迁。j理论苹果遗传学。2014; 127(4):995-1004。https://doi.org/10.1007/s00122-014-2274-2.
- 29。
Wegary D、Teklewold A、Prasanna BM、Ertiro BT、Alachiotis N、Negera D等。适应非洲高原的玉米自交系的分子多样性和选择性扫描。Sci代表,2019年;9(1):13490.https://doi.org/10.1038/s41598-019-49861-z.
- 30.
Beyene Y, Botha A, Myburg AA。通过简单序列重复(SSR)标记评估的传统埃塞俄比亚高地玉米玉米遗传多样性。遗传资源作物evol。2006; 53(8):1579-88。https://doi.org/10.1007/s10722-005-8509-y.
- 31.
Beyene Y, Botha A, Myburg AA。利用AFLP标记和形态性状分析埃塞俄比亚高原玉米传统材料的遗传多样性Biodivers Conserv。2006;15(8):2655 - 71。https://doi.org/10.1007/s10531-005-5407-8.
- 32.
陈玲,王鑫,程丹,陈凯,范勇,吴刚,等。在全球鸡种背景下七个中国本土鸡种的种群遗传分析。动物遗传学报。2019;50(1):82-6。https://doi.org/10.1111/age.12732.
- 33。
关键词:鸡,全基因组,单核苷酸多态性,杂种鸡,中国基因组生物学进展。2019;11(7):1847-56。https://doi.org/10.1093/gbe/evz128.
- 34.
袁勇,张强,曾生,顾磊,司伟,张旭,等。显著正选择的选择性扫掠是粳稻和籼稻品种分化的驱动力。BMC基因组学。2017;18(1):307。https://doi.org/10.1186/s12864-017-3702-x..
- 35.
Grant M, Brown I, Adams S, Knight M, Ainslie A, Mansfield J. The RPM1 plant disease resistance gene促进细胞质钙快速和持续的增加,这是氧化破裂和超敏细胞死亡的必要条件。植物j . 2000; 23(4): 441 - 50。https://doi.org/10.1046/j.1365-313x.2000.00804.x..
- 36.
俗人JH, Lee BM, Wright PE。锌指蛋白:结构和功能多样性的新见解。中国生物医学工程学报。2001;11(1):39-46。https://doi.org/10.1016/S0959-440X(00)00167-6.
- 37。
李H,周茜,赵WS,苏SC,彭YL。一个新的细胞壁相关受体样蛋白激酶基因OsWAK1在稻瘟病抗性中起重要作用。植物分子生物学。2009;69(3):337–46.https://doi.org/10.1007/s11103-008-9430-5.
- 38。
谢y,yu x,姜s,小k,王y,li l等。OSGL6是一个保守的AP2结构域蛋白,促进水稻中的叶片毛状体引发。Biochem Biophys Res Communce。2020; 522(2):448-55。https://doi.org/10.1016/j.bbrc.2019.11.125.
- 39。
谢Z,诺兰TM,江H,尹Y。拟南芥激素和非生物胁迫反应中的AP2/ERF转录因子调控网络。前植物科学。2019;10:228.https://doi.org/10.3389/fpls.2019.00228.
- 40
Dubos C、Stracke R、Grotewold E、Weisshaar B、Martin C、Lepiniec L。拟南芥中的MYB转录因子。植物科学趋势。2010;15(10):573–81.https://doi.org/10.1016/j.tplants.2010.06.005.
- 41.
Lee DS, Kim BK, Kwon SJ, Jin HC, Park OK。拟南芥GDSL脂肪酶2通过对生长素信号的负调控在病原菌防御中发挥作用。生物化学与生物物理学报,2009;https://doi.org/10.1016/j.bbrc.2009.01.006.
- 42.
j,李q,杨j,张g,赵z,吴y等。小麦F箱蛋白质TAFBA1积极调节植物干旱耐受,但负面调节气孔闭合。前植物SCI。2019; 10:1242。https://doi.org/10.3389/fpls.2019.01242.
- 43.
关键词:芝麻,核心种质,遗传多样性,表型,分子标记,微核心种质BMC麝猫。2012;13(1):102。https://doi.org/10.1186/1471-2156-13-102.
- 44.
司慧敏,傅永平,刘文忠,孙志祥,胡国才。光温敏核不育水稻的家系分析。作物学报,2012;38(3):394-407。
- 45。
Eslami G,Khalatbari-Limaki S,Ehrampoush MH,Gholymrezaei M,Hajimohammadi B,Oryan A.三种不同DNA提取方法的语言静脉曲菌作为食品出生病原体的比较。J伊朗J寄生醇。2017; 12(2):236。
- 46。
Evanno G,Regnaut S,Goudet J.使用软件结构检测个人的簇数:模拟研究。Mol Ecol。2005; 14(8):2611-20。https://doi.org/10.1111/j.1365-294X.2005.02553.x.
- 47.
艾尔达。结构收割机:一个网站和程序的可视化结构输出和实现埃文诺方法。保存基因资源。2012;4(2):359–61.https://doi.org/10.1007/s12686-011-9548-7.
- 48.
关键词:数据挖掘,数据挖掘,数据挖掘,数据挖掘变体调用格式和VCFtools。生物信息学。2011;27(15):2156 - 8。https://doi.org/10.1093/bioinformatics/btr330..
- 49。
人口结构分析的f统计估计。J进化。1984; 38:1358 - 70。
- 50
平均杂合度的估计和遗传距离的一小部分个体。遗传学。1978;89(3):583 - 90。https://doi.org/10.1093/genetics/89.3.583.
- 51
Kumar S,SteCher G,Li M,Knyaz C,Tamura K. Mega X:计算平台的分子进化遗传分析。mol Biol Evol。2018; 35(6):1547-9。https://doi.org/10.1093/molbev/msy096..
- 52
陈卫华,高森,胡森。Evolview v3:一个可视化、注释和管理系统发生树的web服务器。中华人民大学学报(自然科学版);https://doi.org/10.1093/nar/gkz357.
- 53.
刘凯,缪斯。PowerMarker:野生大麦核心种质资源遗传多样性的综合分析环境。spontaneum。Hereditas。2005;136:67 - 73。
- 54.
Harris Ma,Clark J,Ireland A,Lomax J,Ashburner M,Foulger R等人。基因本体(GO)数据库和信息学资源。核酸RES。2004; 32(数据库问题):D258-61。https://doi.org/10.1093/nar/gkh036.
- 55.
Kanehisa M, Sato Y, Kawashima M, Furumichi M, Tanabe M. KEGG作为基因和蛋白注释的参考资源。核酸杂志2016;44(D1): D457-62。https://doi.org/10.1093/nar/gkv1070.
致谢
不适用。
资金
中国国家自然科学基金(中国科学院博士后科学基金资助项目31000125号)(批准号2016)资助的课题。 湖南省自然科学基金(批准号2019JJ40117)、湖南省重大科技项目(批准号2018NK1020—1)M600第627号授权书。资助者在本研究和相关数据的设计、分析或解释中没有发挥任何作用。
作者信息
隶属关系
贡献
x.z和H.D.构思并设计了实验。陈晓珠、秦洪华、王志志、F.S、王伟、洪兴、J.P.和张洪华进行了实验,并对实验数据进行了分析。X.Z.写了这篇论文。H.D.监督了这项研究,提供了资金,并修改了手稿的初稿。所有作者都已阅读并批准了稿件的最终版本。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
出版许可
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图1
.131米基因型的主要成分分析,PC1,PC2 PC3和PC4将整个种质分类为簇。图2..根据其繁殖历史从水稻繁殖数据库中获得的血统组A,B和C,顶部是小组中另一个的祖先。可用的雄性无菌基因在括号中表示,具有每个祖先基因型的名称。图3..前1%的选择扫描九个染色体,指示在四个群体中的FST值评估的遗传分化,染色体2中的红色盒子通常鉴定和验证的顶部选择性扫描。图4..富集的基因本体学功能揭示了常规育种系的选择性扫描分析与血统组A(A),B(B)和C(C)的选择性扫描分析的基因本体数据库。
附加文件2:图5
.连接不平衡衰减曲线图,成对LD值(R2)绘制抵靠物理距离。
附加文件3:表1
.118个两系杂交水稻环境敏感基因雄性不育(ESGMS)品系。
附加文件4:表2
.4个群体不同遗传多样性参数的染色体明智值。
附加文件5:表3
.对9条染色体的前1%选择扫频表明4个居群间的遗传分化程度。
附加文件6:表4
.通过Fst值在前5%的阈值,发现传统育种品系与家系A、B和C之间存在遗传分化。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用本文
张,X.,何,Q.,张,W。等等。水稻光温敏感核不育系核心种质的遗传关系及鉴定。BMC植物BIOL.21,313(2021)。https://doi.org/10.1186/s12870-021-03062-x
收到了:
公认:
出版:
关键词
- 雄性不育
- 两系法杂交
- 遗传多样性
- PTGMS.