- 研究文章
- 开放获取
- 发表:
RNA-Seq分析揭示了蕾丝植物细胞程序性死亡和叶片重塑的潜在调控因子(Aponogeton madagascariensis)
BMC植物生物学体积21,文章号:375(2021)
摘要
背景
花边植物(Aponogeton madagascariensis)是一种水生单子叶植物,通过一种被称为程序性细胞死亡(PCD)的发育调节过程,发育出具有独特形状穿孔的叶子。花边植物叶片穿孔形成过程可细分为穿孔前、窗口期、穿孔形成期、穿孔扩展期和成熟期。前三个出现的“无穿孔叶”不形成穿孔,而所有随后的叶子通过发育调控的PCD形成穿孔。PCD在被称为“PCD细胞”的细胞中具有活性,这些细胞不将抗氧化花青素保留在窗期叶叶脉构成的气孔中。靠近静脉的细胞被称为“NPCD细胞”,从花青素中保留红色色素沉着,不进行PCD。虽然在PCD过程中发生的细胞变化已经得到了很好的研究,但这些变化背后的基因表达模式以及在叶片形态发生过程中驱动PCD的基因表达模式大多是未知的。我们试图鉴定介导花边植物叶片重塑和PCD的差异表达基因(DEGs)。这是通过使用转录组学进行基因表达分析,并比较不同叶片发育阶段的deg,以及通过激光捕获显微解剖分离的NPCD和PCD细胞之间的deg来实现的。
结果
转录组从不穿孔、穿孔前、窗口期和成熟叶期,以及从窗口期叶片分离的PCD和NPCD细胞进行测序。数据的差异表达分析揭示了不同的基因表达谱:穿孔前和窗口期叶片的特征是花青素生物合成、植物蛋白酶、膨胀素和自噬相关基因的较高表达。成熟叶和闭孔叶上调与叶绿素发育、光合作用和PCD负调控因子相关的基因。PCD细胞具有较高的乙烯生物合成、油菜素类固醇生物合成和水解酶活性相关基因的表达,而NPCD细胞具有较高的生长素转运、生长素信号、天冬氨酸蛋白酶、半胱氨酸蛋白酶、Bag5和花青素生物合成酶的表达。
结论
RNA测序用于生成从头转录组答:madagascariensis并揭示了许多可能参与PCD和叶片重塑的deg。本研究所得的数据将为今后花边植物叶片发育和PCD的研究提供参考足底.
背景
程序性细胞死亡
程序性细胞死亡(PCD)是一个必要的发育过程,它确保细胞的移除,以进行组织重塑或对环境诱导的应激作出反应[1,2,3.,4].植物发育PCD受内部和外部信号控制,导致发育组织的组织[5,6,7].例子包括胚胎胚柄缺失[8]、通气组织的形成[9],以及木质部分化[10,11].由于在植物模型系统中物理分离PCD目标细胞的实验挑战,目前对控制发育PCD的遗传机制知之甚少。在叶片形态发生过程中,穿孔形成的不同细胞命运相邻区域的植物系统可以为PCD过程提供独特的见解[12].
花边植物模型系统
发育过程中叶片穿孔的形成是独特的,已在天南星科和苦苣苔科等植物科中研究过[12].蓬莱蕉obliqua,m . deliciosa,Aponogeton madagascariensis利用PCD在早期芽发育期间分解和清除指定的细胞,在叶片上产生穿孔。
花边植物答:madagascariensis是一种水生单子叶植物,由于PCD在叶片发育过程中的可获得性和可预测性,最近已成为研究PCD的模型系统。蕾丝植物的叶子也薄而半透明,使它们成为活细胞显微镜的理想选择。最后,整个蕾丝植物在无菌环境下的无菌繁殖为药理学研究创造了机会[3.,12].
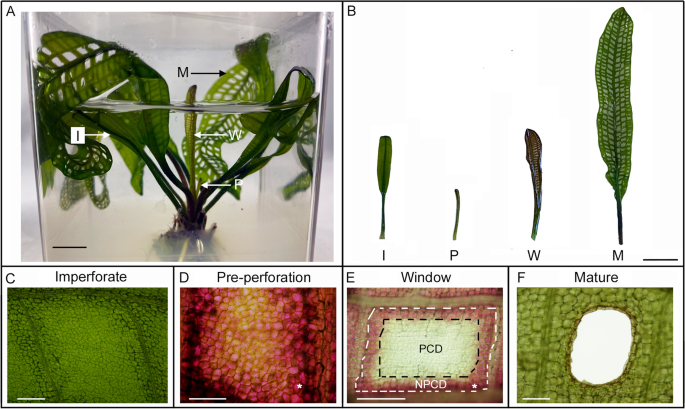
花边植物产生的叶子具有穿孔的形态,其中特定的细胞结合在脉管系统通过发育调节的PCD被去除。1A).花边植物的叶子从地下球茎中以异母细胞系列出现,而前3-4片叶子(称为无孔叶,Fig。1B, C)在发育过程中完全不形成穿孔,所有连续的新生叶都成为穿孔(称为成叶)。早期成虫叶片处于“穿孔前阶段”(图;1B, D)从球茎中呈卷曲状,花青素含量丰富,叶脉形态完整[12,13].当穿孔前叶片展开时,它们过渡到“窗口期”(图2)。1B, E)。当位于纵脉和横脉之间的网状孔中心部分的细胞发生PCD,失去花青素和叶绿素色素沉着时,即达到窗口期(“PCD细胞”,图。1E).靠近静脉的细胞同时保留花青素和叶绿素色素沉着,不发生PCD;因此被称为“非pcd细胞”(NPCD细胞,图。1E).穿孔的大小不断增大,直到停止从静脉到4-5个细胞层。一旦穿孔形成接近完成,穿孔的NPCD细胞边缘的叶肉细胞转分化为表皮细胞。一旦穿孔扩张停止,NPCD细胞达到稳态,叶片就进入成熟阶段。1B, F)。
花边植物程序性细胞死亡(PCD)模型系统。一个-B紫红箱培栽培的蕾丝植物,其发育阶段为预穿孔期(P)、开窗期(W)、成熟期(M)和无穿孔叶(I)。C无孔叶从缺乏花青素的球茎中出现,形成无孔的气孔。D连续的穿孔前阶段叶片从球茎中出现,花青素色素沉着(由星号).EPCD可以在发展的窗口阶段看到活跃的发生。在纵脉和横脉之间,在称为气孔的空间中,可以观察到细胞死亡的梯度。非pcd细胞(NPCD;有界的白色虚线)细胞在成熟后仍然存在。PCD细胞(以黑色虚线)已经失去了花青素,变成半透明的,濒临死亡。成熟叶的PCD和穿孔形成已完成(F),花青素色素沉着明显减少,NPCD细胞达到稳态。比例尺:A = 1厘米;B = 2厘米;C = 200 μm
花边植物PCD机理
窗期叶片气孔内可见的PCD梯度代表了一个强大的模型系统,用于研究发育过程中PCD梯度上的细胞变化[12].在蕾丝植物中,发生在PCD梯度上的细胞变化已被很好地描述,一些蕾丝植物PCD的早期事件包括肌动蛋白细胞骨架的重组、细胞核周围叶绿体环的形成、线粒体聚集和跨空泡链数量的增加[12].在膜结合的囊泡中,细胞器被带入液泡的方式提示了自噬的参与[14].细胞变化的其他例子包括线粒体膜电位的丧失、DNA片段化以及空泡塌陷、质膜收缩和细胞壁降解前caspase样蛋白酶的激活[15,16].
尽管植物PCD的发生过程具有很好的特征,但我们对控制植物PCD调控和执行的分子机制知之甚少[17,18],部分原因是缺乏Aponogetonaceae家族的分子信息。在其他植物模型中,PCD和npc样细胞之间的比较RNA测序(RNA- seq)分析的进展有助于描述类似PCD调控因子的差异表达基因(deg)。分离胚块和胚柄细胞的RNA-Seq分析挪威云杉研究表明,云杉bax抑制剂-1转录同源物在早期PCD胚柄细胞中上调,并在胚柄细胞中调节空泡细胞死亡中发挥作用[19].此外,对早期和晚期形成洞穴的叶通气组织细胞进行了RNA-Seq分析香蒲angustifolia揭示了与溶生通气组织诱导直接相关的膨胀素、钙调素样蛋白和果胶酶转录物[20.].
迄今为止,只有一项转录组研究,Rantong等人(2016)[18],利用互补dna扩增片段长度多态性(cDNA-AFLP)研究了蕾丝植物的叶期。本研究鉴定出79个已注释的deg,这些deg参与光合作用、胁迫反应、病原体防御和PCD等过程。重要的是,他们的结果表明泛素-蛋白质体机制可能参与了蕾丝植物的PCD。然而,由于cDNA-AFLP仅捕获转录组的一小部分,因此整个转录组的表达模式如何答:madagascariensisPCD期间的基因组变化仍然未知。
在本研究中,我们使用高通量RNA-Seq比较了蕾丝植物发育不同阶段以及PCD和NPCD细胞的整体转录组表达谱。我们通过激光捕获显微解剖分离窗口期叶片中的PCD和NPCD细胞,使我们能够识别可能参与PCD和存活的deg。通过RNA-Seq数据的差异表达分析,我们还确定了PCD和NPCD细胞中高表达的基因是潜在的PCD诱导子、执行子和/或调节子。为了确定花边植物叶片重塑的关键调控因子,我们还测试了在穿孔叶和非穿孔叶中高度表达的基因。
我们的目标是识别和比较不同叶片发育阶段以及PCD和NPCD细胞之间的DEGs。我们假设,未穿孔叶和成熟叶中参与光合作用和PCD负调控的基因表达水平明显较高,而穿孔前叶和窗口叶中负责花青素生物合成、caspase样活性、细胞壁组织和PCD前调控的基因表达水平明显较高。
结果与讨论
RNA-Seq数据概述
为了确定蕾丝植物发育PCD和叶片重塑的潜在调控因子,我们生成了一个新的蕾丝植物转录组,并利用RNA-Seq在穿孔前、窗口期、成熟期和不穿孔期以及NPCD和PCD细胞类型的比较中鉴定了deg。从无穿孔、穿孔前、窗口期、成熟叶期、NPCD细胞和PCD细胞的三个生物重复中生成了18个配对端的RNA-Seq文库。使用Illumina (San Diego, CA, USA) NovaSeq6000测序平台在Génome Québec (Montréal, QC, Canada)进行配对端测序,读取长度为100 bp。共生成了1,320,261,351个reads,各个生物文库的数据被存入NCBI SRA数据库,并使用以下SRA登录id: SRR10524134-SR10524151和BIOPROJECTID: PRJNA591467。在读取过滤之后,保留了1,288,318,561个读取,1,102,201,639个读取对齐一致(附加文件1).
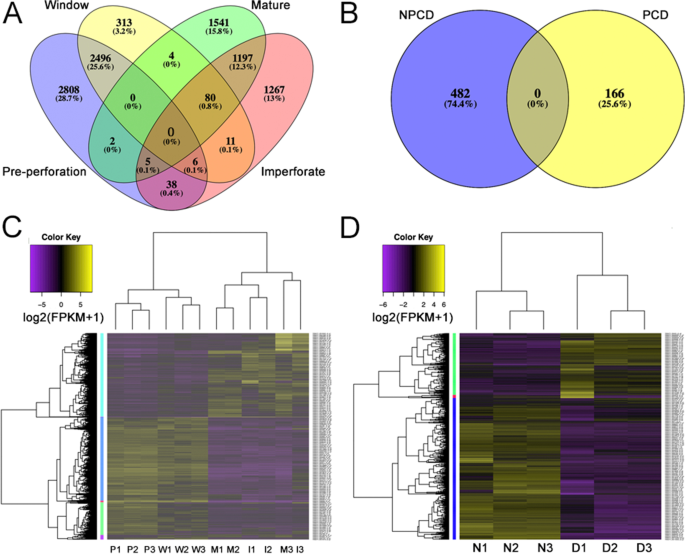
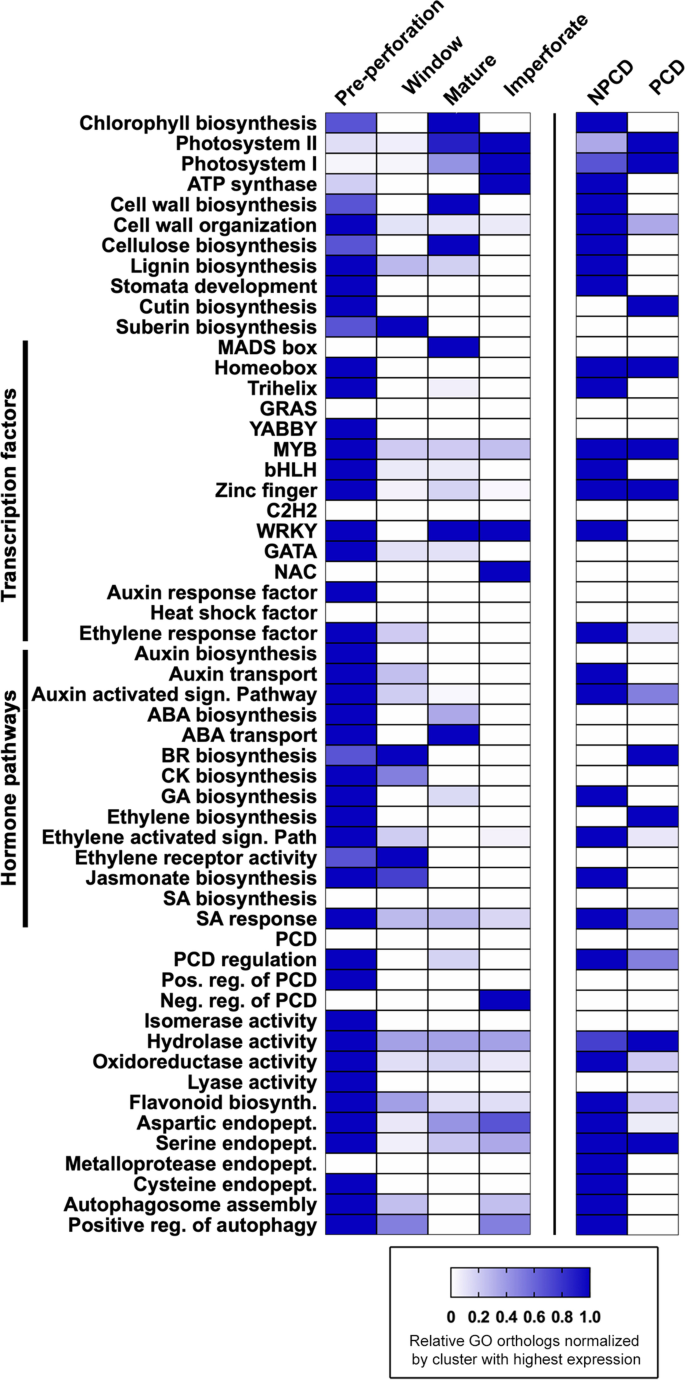
我们从18个RNA-Seq文库中收集了908119个N50长度为1041 bp的转录本,GC含量为49.9%。这些转录本翻译成437,825个蛋白质编码基因(表1).基因本体(GO)跨叶期和细胞类型标注的DEGs占106,222个中的4339个(4.08%)。答:madagascariensisGO在所有叶片阶段和细胞样本中注释了1%的基因P-value cut-off(图;2在10416个DEGs中,分别有2808、313、1541和1267个基因只在穿孔前叶、窗口叶、成熟叶和不穿孔叶中上调(图4)。2A).在细胞类型之间,PCD和NPCD细胞中分别有482和166个基因被独家上调(至少两倍)。其余的DEGs在不同叶期组合之间相互表达(图2)。2).
花边植物发育叶期的转录组谱
基于blast的转录本比对得到了106,222个GO注释基因,这些基因与公共数据库中的序列同源。在注释的基因中,30932个(29.12%)最相似拟南芥, 6,509人(6.12%)至栽培稻692 (0.65%)玉米.
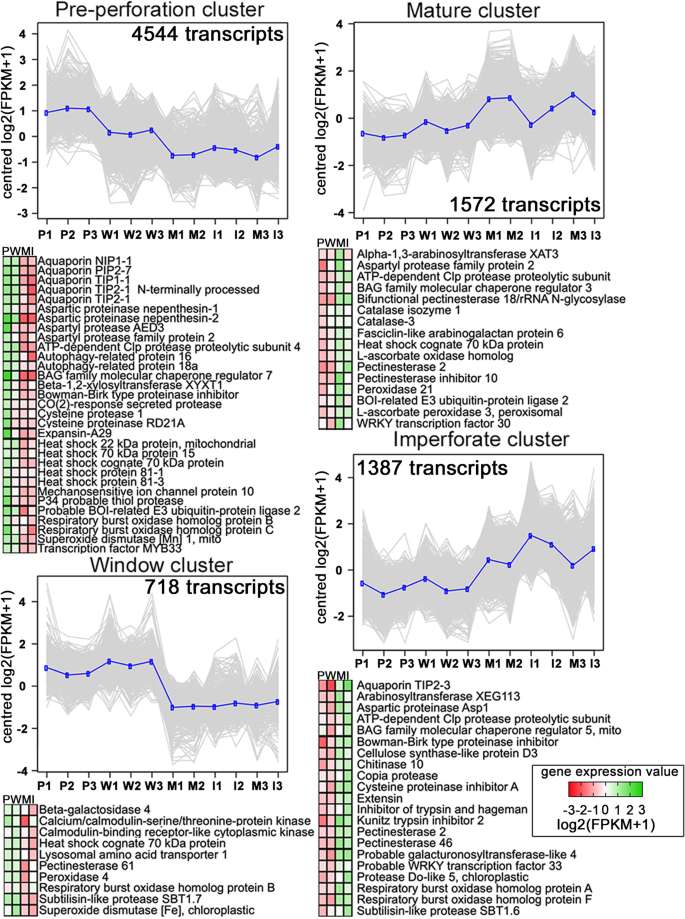
答:madagascariensis通过在四个叶片发育阶段的表达模式的树切割方法,将叶片DEGs分为四个主要簇(图2)。3.,附加文件3.).RNA-Seq数据分为穿孔前簇(4544个DEGs)、窗口簇(718个)、成熟簇(1572个)和非穿孔簇(1387个)(Fold change > 2.0,P< 0.01,错误发现率(FDR) = 1%;额外的文件3.).氧化石墨烯富集分析根据叶片发育四个阶段的表达模式确定了四个主要聚类中富集的生物功能(FDR = 1%,附加文件3.).
花边植物叶片发育阶段的转录组分析。上:按穿孔前(P)、窗口期(W)、成熟期(M)和不穿孔期(I)生物重复的最高表达量分组的四个主要簇(n= 3)。对于每个集群,单独的DEG表达式值(显示为转换后的日志)2(FPKM + 1)值)绘制为灰线,平均表达剖面以蓝色突出显示。每个簇的deg总数显示在每个图的下方(P< 0.01,折叠变化> 2.0)。下图:所示蛋白质的复合基因表达热图,绿色和红色分别对应高和低基因表达。支持数据可在附加文件中找到3.
穿孔前簇,代表穿孔前叶片高表达、随后阶段表达降低的基因,包括编码与类黄酮生物合成、花青素生物合成、乙烯激活信号通路、内肽酶活性、自噬体形成和PCD调控相关的蛋白质。窗期叶片簇是指在穿孔前和窗期表达量最高,在随后阶段表达量降低的基因。该簇包括编码与atp结合、atp酶活性、离子结合、对生长素的反应、对含氧化合物的反应、过氧化物酶活性和水解酶活性有关的蛋白质的基因。成熟簇和闭孔簇均为叶片发育后期表达增加的基因,均含有与光系统I和II、叶绿素结合、光收集复合物组织、催化活性、离子结合和细胞壁生物合成相关的蛋白质编码基因。
综上所述,聚类数据支持以下假设:生长和组织过程在穿孔前叶和窗口期叶的发育中得到了丰富,代谢过程必须发生以促进发育。许多与能量相关的代谢过程发生在成熟和不穿孔的叶子中,这些叶子的发育接近完成,类黄酮的合成减少(图2)。3.,附加文件3.).在叶片簇中选择生物学功能的基因表达的一般模式表明,参与光合作用和PCD负调控的基因在PCD不活跃且达到稳态的成熟和不穿孔叶片中表达水平较高。这表明,未穿孔叶和成熟叶是光合作用的主要部位,而穿孔前叶和窗口叶则专门用于生长、响应激素和执行PCD。所有簇显示高表达基因涉及细胞壁修饰酶,如果胶酯酶。然而,穿孔前叶和窗叶比成熟叶和不穿孔叶具有更多的高表达膨胀素、果胶酯酶和枯草菌素样蛋白酶。穿孔前和窗期叶片代表了许多发育过程的顶点,如PCD的调节、细胞壁组织、木质素和气孔的发育。参与激素合成和运输的蛋白质编码基因在叶期之间有差异表达。穿孔前和窗期叶片生长素生物合成和转运、脱落酸(ABA)生物合成、油菜素类固醇(BR)生物合成、细胞分裂素(CK)生物合成、赤霉素(GA)生物合成、乙烯生物合成、乙烯受体活性、乙烯信号通路、茉莉酸盐生物合成和水杨酸(SA)反应的基因表达量较高。成熟叶和不穿孔叶与穿孔前叶和窗期叶相比,ABA转运基因表达水平相似。总的来说,早期发育的叶片表现出与其他单子叶类植物相似的叶片发育表达模式龙舌兰deserti而且z梅斯[21大多数转录因子(tf)和激素生物合成的表达往往处于最高水平。同样,成熟的叶子表达与光合作用相关的基因[22].
来自NPCD和PCD细胞比较转录组学的见解
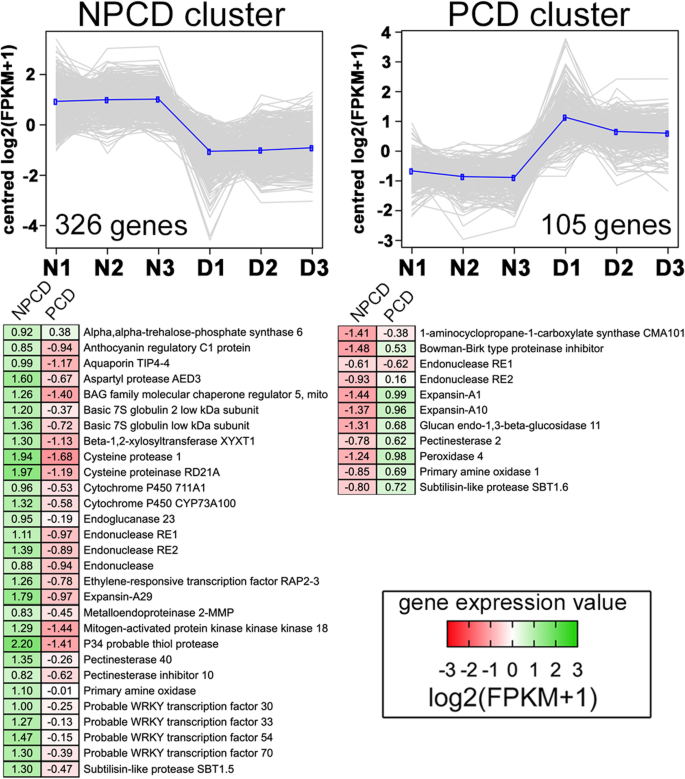
采用激光捕获显微解剖分离NPCD和PCD细胞答:madagascariensis通过RNA-Seq分析揭示PCD的潜在调控因子。基于聚类分析表达模式,将NPCD和PCD转录组分为两个主要聚类(图2)。4)用砍树的方法。该聚类分析确定了大约431个基因在NPCD(326个DEGs)或PCD(105个DEGs)样本中差异表达,且最少发生两倍变化(P< 0.01)。与叶期发现的上调基因相比;这代表了转录组的一小部分,这可能是本实验中使用的有限生物复制的结果。我们测试了氧化石墨烯的富集(FDR = 1%;额外的文件5)。NPCD簇包含编码与呼吸爆发活动、叶片衰老(包括衰老的负调控)、蛋白质自双素化和乙烯激活信号通路有关的蛋白质的基因。PCD簇中含有编码乙烯生物合成、细胞壁修饰剂、蛋白酶抑制剂、ROS生成和PCD调控相关蛋白质的基因(图2)。4).
蕾丝植物NPCD与PCD细胞的转录组学分析。上:在NPCD和PCD细胞生物学重复中按最高表达分组的两个主要簇(n= 3)。对于每个集群,单独的DEG表达式值(显示为转换后的日志)2(FPKM + 1)值)绘制为灰线,平均表达剖面以蓝色突出显示。每个簇的deg总数显示在每个图的下方(P< 0.01,折叠变化> 2.0)。下图:所示蛋白质的复合基因表达热图,绿色和红色分别对应高和低基因表达。支持数据可在附加文件中找到5
比较NPCD和PCD集群之间的相对GO计数(图2)。5)发现PCD细胞上调了更多与类黄酮生物合成、半胱氨酸蛋白酶活性、金属蛋白酶活性、自噬正调控、PCD调控和细胞壁组织相关的蛋白编码基因。相反,PCD细胞上调了更多涉及乙烯生物合成、光系统II和I、BR生物合成和角质生物合成的基因。NPCD和PCD细胞在同型异构体、成髓细胞病(MYB)、锌指TF家族和丝氨酸内肽酶活性的GO类别中表达的基因数量相似,这表明这些家族在分化过程中在两种细胞类型中都起着调节元件的作用。
此外,选择了5个deg,通过定量逆转录聚合酶链式反应(qRT-PCR)在叶期和细胞类型上进行折叠变化表达验证(附加文件)4).的成绩单Bag5,棒曲霉素29,水通道蛋白4 - 4,花青素调节蛋白C1,核转录因子YC-1,α微管蛋白被选为验证目标。所选基因的折叠变化表达归一化α微管蛋白在叶片发育的不同阶段和细胞类型之间的聚类分析中,遵循了类似的模式(附加文件)4),并被视为有效。
转录因子
大多数编码tf的deg在早期发育的叶片中被发现(图2)。5,附加文件3.).在穿孔前叶簇中鉴定出的转录因子包括YABBY、MYB、bHLH (basic Helix-Loop-Helix)、Zn指、GATA、C2C2 CO (constans)、homeobox、DOF(单指dna结合)、TCP (teosinte、branched1、cycloidea和PCF)和三螺旋家族(图2)。5,附加文件3.).这些已确定的家族可能支持对早期叶片发育,特别是轴向极性的建立,气孔发育和维管组织重要的一系列基因的表达,如所见在O. sativa而且答:芥[23,24,25,26,27,28,29,30.,31].成熟叶和无孔叶比穿孔前叶和窗叶表达了更多编码MADS-box和NAC(无顶端分生组织)家族转录因子的基因,这可能是促进光合作用发育、木质素、蜡和次生细胞壁发育的基础,这些在后期发育的叶片中得到加强。
在NPCD和PCD细胞DEG表达谱中也检测到编码tf的DEG(图2)。5,附加文件5).三螺旋和WRKY家族在NPCD细胞中大量表达。这些确定的tf最有可能负责转录参与应激反应的基因,并且已知在发育中的叶肉细胞中上调o .漂白亚麻纤维卷叶子(21].两种constan家族tf在PCD细胞中高表达。Constan TF家族可能参与介导PCD细胞崩溃或抑制抗PCD基因。NPCD和PCD细胞上调MYB、锌指和同源盒子家族tf的数量相同。两种类型的细胞中都可能存在MYB tf,但这两组细胞在促进和抑制类黄酮生物合成方面具有相反的职责。穿孔和非穿孔花边植物叶片基因表达转录调控的差异可能控制了导致差异生长的关键编程事件。
植物激素
我们在穿孔前簇中检测到生长素、ABA、CK、GA、乙烯和茉莉酸激素生物合成基因的较高表达模式(图2)。5,附加文件3.).窗口期簇含有BR生物合成和乙烯受体活性的最高表达水平。在成熟和穿孔前阶段簇中ABA激素转运的表达水平最高(图2)。5,附加文件3.).这些结果支持了先前关于几种植物特异性激素参与PCD信号传导的发现,包括SA、茉莉酸、ABA、GA和乙烯[32,33,34].在蕾丝植物发育早期观察到的激素生物合成模式类似于单子叶沙漠酒,龙舌兰酒而且z梅斯[21,22].
迄今为止,一些药理学全植物实验已经揭示了花边植物穿孔的形成是如何依赖于生长素和乙烯生物合成的[35,36,37].除了已被广泛研究的乙烯外,还需要进一步的工作来解开每种植物激素在调节花边植物叶片发育和穿孔形成中的作用。
与PCD细胞群相比,NPCD细胞群的表达模式包括更多与生长素转运、生长素信号通路、GA生物合成、乙烯激活信号通路、茉莉酸盐生物合成和SA反应相关的高表达基因。5,附加文件5).PCD细胞相对于非PCD细胞上调了1个乙烯生物合成基因编码。乙烯生物合成和乙烯受体参与促进蕾丝植物叶片气孔中注定死亡的细胞的PCD [35,37].此外,与非PCD细胞相比,PCD细胞上调了更多与BR生物合成相关的基因。BRs被认为是调节ROS产生的时间,反过来,在绒毡层细胞中PCD的执行茄属植物lycopersicum[38].BR也可能在PCD细胞触发中发挥类似的作用,这表明与BR合成相关的基因表达较高。与PCD细胞相比,NPCD细胞上调了乙烯反应因子(ERF) RAP2-3。ERF-RAP2-3在乙烯介导的缺氧胁迫中起着重要作用答:芥苗(39],并可能在PCD执行过程中发挥保护NPCD细胞免受PCD细胞积累超氧化物的作用。总之,我们的结果支持了植物激素参与PCD的假设,需要有针对性的方法来解开它们的具体作用和功能。
花青素生物合成酶
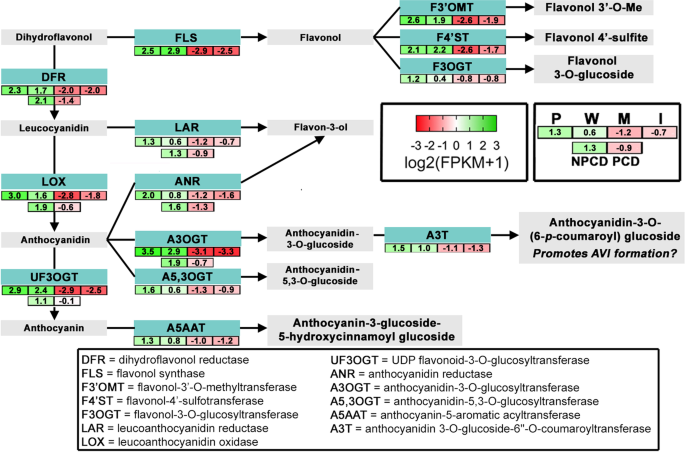
在穿孔前叶簇中,有46个基因被归为具有类黄酮生物合成酶、异构酶、水解酶、氧化还原酶或裂解酶活性的酶,而在窗口叶簇、成熟叶簇和不穿孔叶簇中,这些酶的基因均小于10个。5,附加文件3.).穿孔前簇中上调的8个基因编码类黄酮生物合成途径。穿孔前叶和窗叶上调了促进早期和晚期黄酮类化合物生物合成的基因以及下游转移酶,促进花青素、黄酮醇和花青素的合成。与PCD细胞相比,NPCD细胞还上调了5个编码类黄酮生物合成的基因(图2)。5,附加文件5).只参与类黄酮生物合成的基因黄酮3-双加氧酶2,产生二氢黄酮醇[40],在成熟叶和闭孔叶以及PCD细胞中表达上调3.而且5).上调基因编码的酶,促进花青素生物合成总结在图。6.
花边植物叶片发育最显著的特征是叶片早期PCD过程中可见的花青素色素梯度。前人研究了外源ROS和抗氧化剂在花边植物叶片发育中的作用,发现它们是花边植物模型系统中穿孔形成的关键调节因子[13].与未穿孔和成熟叶片相比,穿孔前和窗期叶片具有最高水平的花青素[13,42].我们的结果与这些发现一致,穿孔前叶、窗叶和NCPD细胞表达了编码酶的基因,这些酶可以促进葡萄糖苷构成的花青素的生物合成(图2)。6).花青素-3- o -葡萄糖苷转移酶(A3OGT)、花青素-5,3- o -葡萄糖苷转移酶(A5,3OGT)和花青素-5-芳香烃酰基转移酶(A5AAT)的上调表明,花青素-3- o -葡萄糖苷、花青素-5,3- o -葡萄糖苷和花青素-3-葡萄糖苷-6-羟基肉桂基葡萄糖苷在早期叶片中被积极合成,导致了花青素的积累。窗期叶片中粉红色花青素液泡的存在与液泡pH值为~ 5.5 pH值的假设相一致,这有利于黄酮阳离子的形成[43].
最近人们注意到窗期叶片中花青素液泡包体(AVIs)的形成,然后随着叶片进入成熟而消散(Gunawardena实验室,2020年未发表数据)。Kallam等人(2017)[43]最近报道了花青素取代基或装饰的组成决定了AVIs在烟草系中的溶解度。花青素3- o -葡萄糖苷-6″- o -香豆油酰转移酶(A3T)对花青素3- o -葡萄糖苷的芳香酰化促进了AVIs的形成,而鼠李糖修饰则通过竞争减少了AVIs的形成。在早期叶片中缺乏上调的芳香鼠李糖转移酶或丙二酰转移酶DEGs(抑制AVI的形成),这表明AVIs在调节正常叶片发育中的作用[43].NPCD细胞上调了花青素3- o -糖基转移酶编码基因,这与促进ROS清除剂和保护PCD一致。花青素化合物在发育叶片阶段和NPCD细胞的鉴定目前正在使用全球质谱法进行研究。
细胞程序性死亡的潜在调节因子
细胞壁修饰酶和水通道蛋白
花边植物穿孔的形成依赖于薄角质层的侵蚀、多糖的去除和纤维素的降解[44来调节射孔扩张。当邻近的PCD细胞崩溃时,木脂素的沉积对于防止NPCD细胞中的感染和营养物质的损失是必不可少的。我们发现窗口期簇具有最多数量的木脂素生物合成相关基因的高表达(图2)。5,附加文件3.),这与绒垫层沉积在发育窗口期最为活跃的假设相一致[12].
穿孔前和窗口期簇分别包含67个和7个基因的高表达,分别属于细胞壁组织或细胞壁生物合成。9、5基因分别高表达。NPCD聚类中有4个基因高表达,PCD聚类中有1个基因高表达(图2)。5,附加文件5).所有聚类均具有较高的水解酶、果胶酯酶、葡萄糖苷酶和木葡聚糖糖基转移酶活性的同源物表达。成熟和无孔叶没有上调编码膨胀酶的基因。
促进细胞扩张和细胞壁松动不仅对发育中的蕾丝植物叶片的生长有重要作用,而且对PCD细胞的执行和NPCD细胞的重组也有重要作用。穿孔前和窗口期叶片上调了几个编码木葡聚糖内转糖基转移酶、扩张素(expansin-A29qRT-PCR检测,附加文件4)和果胶酯酶(图。3.),它们在生长过程中负责细胞壁的松动和重组[45,46,47].成熟叶和闭孔叶遵循类似的模式,但没有上调基因的膨胀素(图。3.).这可能表明,一旦叶子成熟,这种活动就不再需要了。
水通道蛋白(Aquaporins)通过控制吸水在细胞扩张中发挥重要作用[48].穿孔前和窗期叶片中数个编码液泡体固有蛋白(TIP)、结节固有蛋白(NIP)和质膜固有蛋白(PIP)水通道蛋白的基因上调。NPCD细胞上调了TIP4-4水通道蛋白基因。4).TIP4-4qRT-PCR检测(附加文件4).PCD细胞上调一个TIP2-3基因,成熟叶和闭孔叶均上调。所有样品中水通道蛋白基因的表达表明,在叶片发育过程中,通过扩大液泡来进行细胞扩张是必要的,在NPCD和PCD细胞的分化中也需要通过扩大液泡来控制细胞扩张或细胞破裂的进程[48].
热休克蛋白
热休克蛋白(Hsps)是为了应对压力而合成的,在蛋白质发生不可逆变性之前,通过重新折叠蛋白质来维持稳态[49,50].穿孔前叶簇包含25个基因,窗口叶簇包含2个蛋白质折叠基因。成熟叶簇和闭孔叶簇在未折叠蛋白折叠下分别含有1个和2个基因(图2)。3.,附加文件3.).NPCD细胞簇包含1个基因,PCD细胞簇包含0个未折叠蛋白折叠基因(附加文件)5).atp依赖分子伴侣如Hsp81-1、Hsp81-3和Hsp70-15在穿孔前和窗口期叶片中表达上调。
在o .漂白亚麻纤维卷在原生质体模型中,mtHsp70过表达可防止氧化应激下原生质体的ROS爆发和PCD [51].与抑制应激诱导的PCD相反,Hsp70在甜椒促进被感染者的过敏反应烟草benthamiana叶子(52通过效应蛋白的核定位。Hsp70s及其各自的bcl -2相关致病蛋白(Bag)可调节动物PCD和许多细胞过程[1],为他们在蕾丝植物PCD方面的研究提供了依据。Hsp70抑制剂氯苯乙基磺酰胺(PES-Cl)的实验处理显著降低了窗期叶片的穿孔数量、类caspase活性和花青素水平[42],表明Hsp70在花边植物PCD中起介导作用。Hsp70蛋白在穿孔前和窗口期叶片中受到发育调控并显著升高,这与Hsp70-15基因的表达模式一致(图2)。3.).虽然我们的研究结果支持了Hsp70活性在阈值水平上影响蕾丝植物叶片发育的假设,但它的定位仍然未知。
蕾丝植物同源的基因o .漂白亚麻纤维卷Hsp81-1和Hsp81-3在穿孔前期叶片中转录上调(图;3.,附加文件3.).Hsp81s促进植物耐盐性o .漂白亚麻纤维卷和过表达实验答:芥显示它促进耐热性[53,54].蛋白质组学模型预测AtHsp81可以与AtHsp70形成复合物[55]最有可能用于蛋白质质量控制,我们的研究结果表明,这两种植物都是Hsp70的同源物[42]和Hsp81的合成是为了维持叶片早期发育过程中的蛋白质稳态。此外,还需要进行全株药理学实验,以改进蕾丝植物发育过程中Hsps和Bag蛋白功能的表征。
蛋白质包
Bag蛋白家族最近在植物发育生物学领域受到了关注,因为它们在几种植物系统中介导PCD、衰老和自噬[1,55].袋蛋白是Hsp70的主要核苷酸交换因子[56]并有助于加速动物和植物中的Hsp70蛋白折叠循环和调节PCD途径[57,58].Hsp70共同伴侣Bag7在穿孔前叶和窗叶中表达较高。成熟叶和不穿孔叶中Bag3和线粒体(mt) Bag5表达上调(mtBag5qRT-PCR检测,附加文件4).与PCD细胞相比,NPCD细胞中mtBag5的表达值显著升高(图2)。4,附加文件5).
在花边植物中,我们发现穿孔前和窗口期叶片上调了编码同源基因的基因答:芥对于Bag7(图;3.,附加文件3.).Bag7被er定位在答:芥参与未折叠蛋白反应,并可定位于细胞核与多种蛋白质相互作用以调节PCD途径[59,60].
成熟和不穿孔花边植物叶片相对于穿孔前和窗叶上调了Bag3和Bag5同源物(图2)。3.),而与PCD细胞相比,NPCD细胞上调了一个Bag5同源物(图。4).Bag3参与人类货物介导的自噬使其成为潜在的癌症治疗靶点,但其在植物PCD中的作用尚不清楚[61].在答:芥在叶片系统中,Bag5已被发现与热休克同源蛋白70 (Hsc70)结合并定位于线粒体,以促进ROS的生成和叶绿素的清除,而叶片处于衰老状态[58].Bag5基因在NPCD细胞中上调的事实为研究该基因在NPCD细胞与PCD细胞分离或防止线粒体爆裂的可能作用提供了机会[62].植物Bag5在Ca调控中起作用2 +可能是线粒体应激反应的结果。Bag family蛋白似乎在植物PCD中起着重要的调节作用,但其在植物中的确切生化作用尚不清楚[61].
Autophagy-related蛋白质
Dauphinee等人(2019)发现自噬在细胞存活中起主导作用,并没有明确证据表明自噬和PCD在花边植物叶片穿孔形成过程中直接参与[14].只有穿孔前和窗口期叶片被发现上调自噬体组装的GO类基因,如自噬相关蛋白16 (Atg16)和Atg18a的同源物(图。3.而且5,附加文件3.).穿孔前簇包含一个snf1相关蛋白激酶催化亚基α KIN10和一个溶酶体氨基酸转运体1,两者都参与自噬调节。成熟叶,闭孔叶和NPCD细胞(与PCD细胞相比,图。3.而且4)上调参与自噬正向调控的转录因子WRKY33 [63].WRKY33在NPCD细胞中相对于PCD细胞上调,这表明NPCD细胞在穿孔发育过程中需要抗逆性。WRKY33对植物抵抗坏死性病原体很重要,并与核中的Atg18a相互作用[64来应对生物压力。
我们检测到Atg18a和Atg16基因在穿孔前和窗口期叶片中有较高的表达,这表明在植物发育PCD过程中,自噬体的形成和有丝分裂的调节发生了变化。有八个成员AtATG18基因家族(AtATG18a- - - - - -AtATG18h) [63];每个成员都有不同的表达式模式,而且只有AtATG18a显示在饥饿条件下和衰老过程中转录水平增加答:芥.AtATG18a在氧化、盐和渗透胁迫过程中,表达也上调,是自噬所必需的。RNA干扰(RNAi)AtATG18a使植物自噬缺陷,对各种胁迫条件更加敏感[63,65,66,67].Atg16寡聚形成Atg12-Atg5·Atg16复合物,这对自噬至关重要[68].
的答:芥蛋白激酶SNRK KIN10已被证明可以诱导几种自噬基因,如Atg8,一种在蕾丝植物叶片中被发现发育调控的蛋白质[69,70].在叶片发育早期,与成熟叶片和闭孔叶片相比,参与光合作用的基因表达水平较低。长期的压力通常会导致光合作用减少和ROS积累增加,从而引发PCD途径[71].自噬作用在叶片发育早期提供能量,而光合作用和叶绿素基因直到成熟和达到最佳光合作用活性才上调。WRKY33很可能在叶片发育早期转录增加自噬体的形成,并促进应激NPCD细胞的生存。
程序性细胞死亡的调节因子
属于GO类“PCD调控”的基因在蕾丝植物叶片阶段和NPCD和PCD细胞之间存在差异表达。我们发现穿孔前叶片簇含有l型凝集素结构域激酶IX的同源物。I和机械敏离子通道蛋白10 (MSL10)。MSL10促进PCD,以应对病原体的入侵答:芥[72,73,74].此外,穿孔前簇包含编码boi相关e3 -泛素连接酶2的基因。BOI e3 -泛素连接酶能够通过限制α-吡啶酸的生成和凋亡抑制剂的泛素化来抑制PCD [75,76].穿孔前叶和窗期叶中MYB33的上调(图;3.,其他文件3.)对进一步的花边植物研究特别有兴趣,因为它在花药和其他单子叶植物的种子中促进PCD的作用大麦芽而且o .漂白亚麻纤维卷[77].
成熟叶簇和闭孔叶簇分别含有BOI相关的E3泛素连接酶2和呼吸爆裂氧化酶同源物- f (Rboh-F)基因。BOI相关E3泛素连接酶2基因通过抑制ROS产生负向调控PCD [75,76]这与成熟叶和不穿孔叶相一致,这些叶的穿孔形成和超氧化物积累较不活跃[13,42].Rboh-F主要通过ABA信号通路参与ROS的生成答:芥系统(78]并且与免疫有关。
我们还观察到PCD调节基因在NPCD和PCD细胞中的差异表达模式。5,附加文件5).NPCD上调了天冬氨酸蛋白酶AED3和ERF-RAP2-3的编码基因,这些基因之前被认为是pcd的亲贡献者。PCD细胞相对于非PCD细胞上调了一个编码初级胺氧化酶1 (PAO1)的基因。PAOs已被证明在器官发育和PCD期间分化组织时产生ROS中发挥作用[79,80].PCD前基因如天冬氨酸蛋白酶AED3在NPCD中上调是一个意想不到的结果,可能是这些基因在PCD激活期间具有促进NPCD细胞应激反应或衰老的功能。
植物蛋白酶和程序性细胞死亡
穿孔前簇、窗口簇、成熟簇和不穿孔簇均含有上调的内肽酶基因编码。穿孔前的叶片簇显示了59个编码天冬氨酸、丝氨酸和半胱氨酸内肽酶活性的酶的基因。5,附加文件3.).NPCD细胞上调了27个编码天冬氨酸内肽酶活性的基因(PCD细胞上调了2个),1个编码丝氨酸活性的基因(PCD细胞上调了0个),4个编码半胱氨酸活性的基因(PCD细胞上调了0个),1个编码金属蛋白酶的基因(PCD细胞上调了0个;无花果。4而且5,附加文件5).
先前的研究已经确定了类caspase酶在植物发育中作为PCD启动器或执行器的作用[81,82,83].枯草菌素样蛋白酶具有潜在的PCD调节作用,其含有自催化活性的结构域在去除前域后重新进入细胞执行PCD,所有的lace植物叶期簇都上调了几个枯草菌素样蛋白酶。类似的表达式subtilisin-like蛋白酶不同叶期在叶片重塑和内稳态中的作用是一致的。所有叶期和NPCD细胞都上调了一种Bowman-birk丝氨酸蛋白酶抑制剂。丝氨酸蛋白酶的抑制活性可能表明丝氨酸内肽酶活性的协调抑制对正确的PCD执行的作用。
多种半胱氨酸蛋白酶在植物PCD发育中的作用已被证实。例如,半胱氨酸蛋白酶1介导绒毡层PCD答:芥[82]和半胱氨酸蛋白酶R2D1A可增强小鼠先天免疫中的PCD答:芥[83,84].半胱氨酸蛋白酶活性可被serpin和Kunitz蛋白酶抑制剂的活性严格控制(可逆抑制[83]),在成熟叶和闭孔叶中以Kunitz蛋白酶抑制剂2和半胱氨酸蛋白酶抑制剂a的形式上调,蛋白酶级联可引发花边植物叶片PCD。成熟叶片中的Kunitz和半胱氨酸抑制剂等抑制剂可在阻止半胱氨酸蛋白酶活性的效应期和防止PCD再次活跃中发挥作用[85].我们发现前文提到的半胱氨酸蛋白酶1和RD21A基因在NPCD细胞中转录上调。这些蛋白酶的mRNA在注定要生存的细胞中积累的原因仍不确定,这可能表明它们在介导PCD细胞崩溃中起着重要作用。
天冬氨酸蛋白酶也调节植物发育PCD;具体来说,生殖组织的缺失[86,87].在花边植物中,穿孔前、窗期叶片和NPCD细胞都高表达编码天冬氨酸蛋白酶AED3的基因,该基因与PCD有关(图2)。3.而且4;额外的文件3.而且5).在PCD进展过程中,天冬氨酸蛋白酶AED3可能上调使NPCD细胞转分化为内胚层。
与PCD细胞相比,NPCD细胞中金属蛋白酶2-MMP基因上调。4).SI2-MMPs已被发现抑制表皮细胞死亡番茄相反,2-MMP促进早期衰老答:芥[88,89].的上调2-MMP在NPCD细胞中可能提示它们在区分NPCD与PCD细胞或抑制PCD执行方面的作用。
蛋白酶底物磷酸烯醇丙酮酸羧激酶1 (PEPCK1)可以被裂解答:芥metacaspase 9 (MC9)和MC9反过来促进根木质部组织的清除[90]和可能的糖异生[91].基因编码答:芥peppc1同源物[85]在我们的无孔叶簇中表达上调(图;3.,附加文件3.).MC9对PEPCK1的切割可能促进了不穿孔叶的糖异生,支持了不穿孔叶在快速衰老前为lace植物提供能量的假说。
有趣的是,我们的RNA-Seq分析没有揭示任何蕾丝植物空泡处理酶(VPEs)编码基因在叶片阶段或细胞类型之间的差异表达,而VPEs在蕾丝植物叶片的发育PCD中起着重要作用[35,92].Rantong和Gunawardena(2018)使用qRT-PCR方法显示了蛋白的转录水平我VPE-1和-2在早期发育叶和窗叶中显著上调(归一化至我肌动蛋白)。先前的工作强调了VPE活性对lace PCD细胞液泡塌陷很重要的事实。我们和Rantong & Gunawardena(2018)的研究结果中没有这一过程,这表明VPE蛋白的自动加工和翻译后修饰可能解释了其功能活性,而不仅仅是mRNA的更高积累[92].
结论
花边植物叶片PCD的细胞动力学和时间事件有很好的记录。在这里,我们通过描述叶片发育的不同阶段以及从窗口期叶片中分离出的PCD和NPCD细胞的转录组谱来研究这一过程的分子基础。为了总结控制发育性PCD和叶片重塑机制的基因,我们对DEGs进行了分析。7).
基于比较转录组学,我们的结果支持了这样的假设,即NPCD和PCD细胞分化是由植物激素和TF活性的差异平衡介导的,这些激素和TF活性既促进又限制PCD途径。GO富集分析表明,自噬、细胞扩张、蛋白酶活性、ROS生成和类黄酮生物合成共同作用,确保了蕾丝植物叶片发育过程中穿孔扩张的促进。参与这些不同生物学功能的基因的高水平表达在蕾丝植物叶片发育早期和晚期之间存在显著差异,表明它们参与调节穿孔的起始、执行和叶片生长。我们对蕾丝植物转录组和表达模式的研究结果揭示了多种可能参与蕾丝植物叶片细胞死亡起始和进展的候选基因,提出了新的假设,并为植物发育PCD提供了新的见解。未来需要对候选DEGs进行实验,以进一步表征和确认lace植物叶片穿孔形成过程中的蛋白质功能。
综合考虑,这个答:madagascariensis在此生成和分析的转录组数据说明了de novo illumina RNA-Seq的强大功能。我们的转录组既是高质量的基因发现资源,也是通过基因表达谱检测生理变化的框架。结合额外的转录组注释工具,来自模式植物物种的实验观察无疑将促进未来对PCD生物学和蕾丝植物叶片重塑的更深入的了解。
方法
植物组织培养
如Gunawardena等人(2006)所述,蕾丝植物培养物无菌繁殖[93].将Lace植物球茎置于Magenta GA-7箱中培养,埋入由1.5%植物组织培养琼脂(w/v, Phytotechnology Laboratories)组成的100 mL MS固体培养基中,液体MS[3%蔗糖(w/v), 0.01%肌醇(w/v), 0.215% MS基盐(w/v, Phytotechnology Laboratories), 0.0025%硫胺- hcl (v/v), pH 5.7],然后浸泡在150 mL液体MS下,24℃下培养,125 μmol m光照下暴露−2年代−112小时光/暗周期,日光豪华荧光灯灯泡(飞利浦)。答:madagascariensis布鲁根球茎最初购买自The PlantGuy (AB,加拿大)。
培养30天或直到每株植物长出3个穿孔成熟叶。从整个植株培养物中分离出每一个未穿孔、预穿孔、窗口期叶片和一个最近发育的成熟叶片,用蒸馏水彻底清洗,然后从中脉中切除叶片组织并快速冷冻以进行下游分子工作。进行了三个独立的实验。在每个实验中,每个叶期都从整个植株培养物中取一个。
PCD和NPCD细胞制剂
使用蔡司PALM激光捕获显微解剖和成像系统(North York, ON, Canada)分离PCD和NPCD细胞。细胞从窗期叶的气孔中收集,在气孔中可以通过颜色明显区分每种类型。在快速冷冻前,从每个窗口期叶片的4个独立的气孔(每个气孔约2000个细胞)中收集细胞类型群体样本。每种细胞类型分别进行三次独立实验。在每个实验中,从整个植株培养物中取一片窗口期叶片。附加文件中提供了叶片组织样本的激光捕获和弹射过程的样本图2.
RNA提取及质量控制
从四个发育阶段的叶片和两种不同的细胞类型(NPCD和PCD细胞)中提取RNA。按照ReliaPrep RNA试剂盒(Promega)的说明,从3种不同的全株培养物中每一个无孔叶、穿孔前叶、窗口叶或成熟叶的40 mg快速冷冻、无中脉叶板组织中提取RNA。RNA样本用DNAse I(赛默飞世尔)处理。使用Nanodrop分光光度计(Thermo Fischer)估计洗脱RNA的数量,使用生物分析仪(Agilent Technologies Inc., Santa Clara, CA, USA)确定RNA完整性数(RIN)。只有RIN≥6.5的RNA样本被批准进行cDNA转换。
cDNA文库制备及Illumina测序
cDNA文库的制备和测序由Génome Québec (Montréal, QC, Canada)进行。使用特定于链的Trueseq协议,在Illumina NovaSeq6000 (CA, USA)上生成了18个长度为100 bp的配对端RNA-Seq文库。获得的原始读取数据被存入NCBI,并可根据SRA访问。SRA接入id: SRR10524134-SR10524151和BIOPROJECTID: PRJNA591467。首先通过FastQC分析FastQ文件对数据进行质量检查[94].使用Trimmomatic v.0.35修整质量较低且含有适配器污染的读数[95] k-mer大小为25,参数:ILLUMINACLIP:TruSeq3-PE。fa:2:30:10滑动窗口:5:0 MINLEN:50。使用FastQC v0.11.2评估修剪reads的质量(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/).
转录组从头组装
使用Trinity v2.3.1构建一个从头组装的高质量适配器免费读取[96]使用默认设置。利用转录本平均长度、GC%和N50值等主要生物信息学指标来评估基因组合的质量(表2)1).三位一体管道集群从头将转录本组装成基因和异构体,我们只使用“基因”数据集,选择表达量最高的异构体作为每个基因的代表。转录组霰弹枪组装项目已存入DDBJ/ENA/GenBank,编号为。GJFM00000000。本文描述的版本是第一个版本,GJFM01000000。
差异表达基因的转录定量和鉴定
每个基因的丰度是通过期望最大化(RSEM)将每个样本的reads与我们的从头转录组对齐来计算的[97]。的修剪后均值米-values (TMM)方法[98]用于计算归一化因子(一个计算NPCD与PCD细胞的比较,一个计算叶期之间的比较)。使用Trinity [99],使用TMM进行表达归一化,计算外显子模型每百万reads (FPKM)的每千碱基片段数。利用数字基因表达实证分析(Empirical Analysis of Digital Gene Expression, edgeR)统计包[One hundred.) (http://bioconductor.org/packages/release/bioc/html/edgeR.html)执行R (v3.3.2;R Core Team 2015)。差异表达超过两倍且FDR为1%的基因被定义为差异表达[101].
聚类分析
利用聚类分析的方法,分离了叶片期样品及NPCD与PCD细胞之间的基因表达模式。采用集中和日志两种方法实现归一化基因表达的层次聚类2(FPKM + 1)转换[99]和40%深度的树木切割,使用R和包Superheat进行热图可视化[102].
注释和GO富集分析
为了识别与每个转录本相关的基因和功能,组装的转录本使用Trinotate进行注释[103以及公共基因组和功能存储库。我们基于公共数据库(包括GO、京都基因和基因组百科全书(KEGG)、真核同源组数据库(KOG)、瑞士蛋白质资源(Swiss- prot)和Panther数据库)中最高匹配序列相似度对转录本进行了注释,使用了带截断的BLASTX方法E-value为10−5[104,105,106].为了消除来自外来生物和实验室污染物的转录本,去除非植物来源的基因。选择的注释数据包含在附加文件中3.而且5.
go富集分析使用植物转录因子数据库v5.0 [107基于Fisher精确测试的程序,多次测试校正FDR = 1%。GO分析通过将测试样本中的GO术语与所有样本生成的整个蕾丝植物新生转录组背景参考中的GO术语进行比较。
qRT-PCR验证
五个选定的发展类别(Bag5,棒曲霉素29,水通道蛋白4 - 4,花青素调节蛋白C1,核转录因子YC-1,α微管蛋白)用ΔΔC对RNA-Seq的表达结果进行验证T方法。从蕾丝植物叶片阶段和分离的细胞类型中收集和提取RNA,如上所述。使用SuperScript®III First-Strand Synthesis System for qRT-PCR (Invitrogen, Burlington, ON, Canada)和oligo dT合成单链cDNA20.遵循制造商的说明。qRT-PCR采用tor- gene RG-3000系统(Corbett Research, Sydney, NSW, Australia),以0.5 μl cDNA为模板,添加0.4 mmol l−1所有选定基因的引物(附加文件6)在以下条件下:94°C 5 min, 94°C 30 s 35个周期,54°C 30 s, 72°C 1 min,然后72°C。qPCR使用QuantiFast®SYBER®Green PCR试剂盒(Qiagen, Mississauga, ON, Canada)进行。采用Rotor-Gene 6软件进行熔体曲线分析,并使用至少90%效率的实验进行分析(Corbett Research)。实验分为3个重复,分别为未穿孔、穿孔前、窗口期和成熟期叶片以及NPCD细胞和PCD细胞。五个选定基因的cDNA拷贝数(附加文件4)由Ct值的标准曲线(R2> 0.99),并针对α微管蛋白同种型(37].
图像分析与处理
叶片布局图像使用尼康L110数码相机获得。Photoshop和Illustrator (Adobe Creative Cloud;(Adobe Systems Inc.)被用来准备图像,并去除脱落叶子的背景,以便发表。尼康AZ100显微镜获得叶片阶段的显微照片。图像调整是均匀的内部和一致的数字,包括去除背景,以及使用Photoshop对亮度、对比度和色彩平衡进行微调。
统计分析和数据表示
采用单因素方差分析(One-way ANOVA)和Tukey检验(Tukey test)来确定qRT-PCR验证实验的叶片期均值与学生的叶片期均值之间的显著差异t-test用于识别细胞类型之间的显著差异(附加文件4).所有数据均以均数±标准误差表示。使用GraphPad Prism 5软件(GraphPad software Inc.)进行分析。
数据和材料的可用性
原始测序数据文件可在NCBI SRA数据库中获得,项目编号为。PRJNA591467。转录组霰弹枪组装项目已存入DDBJ/ENA/GenBank,编号为。GJFM00000000。本文描述的版本是第一个版本,GJFM01000000。
缩写
- 度:
-
差异表达基因
- 英国石油公司:
-
碱基对
- 走:
-
基因本体论
- nt:
-
核苷酸
- FPKM:
-
每千碱基的外显子模型的片段数
- RNA-Seq:
-
RNA序列
- 纤毛运动:
-
程序性细胞死亡
- NPCD:
-
Non-PCD
- ROS:
-
活性氧
- 存在:
-
定量逆转录聚合酶链反应
参考文献
- 1.
Kabbage M, Kessens R, Bartholomay LC, Williams B.植物细胞的生与死程序性细胞死亡(PCD):单细胞和多细胞生物都采用的一种基因调控的细胞自杀程序。植物生物学,2017;68:375-404。https://doi.org/10.1146/annurev-arplant-043015.
- 2.
Kacprzyk J, Daly CT, McCabe PF.植物死亡之舞。植物细胞程序性死亡。Adv Botanical res 2011; 60:169-261。https://doi.org/10.1016/B978-0-12-385851-1.00004-4.
- 3.
李文华,李志强,李志强,等。植物细胞程序性死亡的研究进展。方法Mol生物学,2016;1419:145-60。https://doi.org/10.1007/978-1-4939-3581-9_12(纽约:Humana出版社)。
- 4.
范豪特格姆T,沃特斯AJ,古德里奇J,诺瓦克MK.只有在死亡中,生命:植物发育过程中的程序性细胞死亡。植物科学进展,2015;20(2):102-13。https://doi.org/10.1016/j.tplants.2014.10.003.
- 5.
高志刚,李志刚,李志刚。细胞程序性死亡在植物发育中的作用与调控。细胞发育生物学,2016;32:1-7。https://doi.org/10.1146/annurev-cellbio-111315-124915.
- 6.
Gunawardena AHLAN, McCabe PF。可汗:施普林格;2015.https://doi.org/10.1007/978-3-319-21033-9.
- 7.
栗山,福田。植物发育性细胞程序性死亡。植物学报,2002;5(6):568-73。https://doi.org/10.1016/s1369 - 5266 (02) 00305 - 9.
- 8.
Bozhkov PV, Filonova LH, Suarez MF, Helmersson A, Smertenko AP, Zhivotovsky B,等。VEIDase是参与植物细胞程序性死亡的一种主要类caspase活性,对胚胎模式的形成至关重要。细胞死亡差异。2004;11(2):175-82。https://doi.org/10.1038/sj.cdd.4401330.
- 9.
陈晓明,陈志伟,陈志伟,等。乙烯和缺氧诱导玉米根通气组织形成过程中细胞程序性死亡的研究。足底。2001;212(2):205 - 14所示。https://doi.org/10.1007/s004250000381.
- 10.
韩俊杰,林伟,小田勇,崔克明,福田华,何小强。蛋白酶体在木质部发育过程中负责caspase-3样活性。植物学报,2012;29(1):344 - 344。https://doi.org/10.1111/j.1365-313X.2012.05070.x.
- 11.
大桥东K,田田Y,福田H。拟南芥维管相关的NAC-DOMAIN6直接调控木质部分化过程中调控程序性细胞死亡和次生壁形成的基因。植物科学进展。2010;22(10):3461-73。https://doi.org/10.1105/tpc.110.075036.
- 12.
Gunawardena AHLAN。植物细胞程序性死亡和组织重塑。中国科学(d辑),2008;https://doi.org/10.1093/jxb/erm189.
- 13.
Dauphinee AN, Fletcher JI, Denbigh GL, Lacroix CR, Gunawardena AHLAN。蕾丝植物叶片的重塑:抗氧化剂和ROS是程序性细胞死亡的关键调节因子。足底。2017;246(1):133 - 47岁。https://doi.org/10.1007/s00425-017-2683-y.
- 14.
Dauphinee AN, Denbigh GL, Rollini A, Fraser M, Lacroix CR, Gunawardena AHLAN。自噬在植物细胞程序性死亡中的作用。前沿植物学报2019;10:1198。https://doi.org/10.3389/fpls.2019.01198.
- 15.
Dauphinee AN, Warner TS, Gunawardena AHLAN。花边植物叶片诱导细胞死亡与发育细胞死亡形态的比较。中国生物医学工程学报。2014;14(1):1 - 13。https://doi.org/10.1186/s12870-014-0389-x.
- 16.
岑勋爵,安太子妃,瓦茨RL,古纳瓦德纳·阿兰。揭示了植物程序性细胞死亡(PCD)过程中线粒体、类caspase蛋白酶和肌动蛋白骨架之间的相互作用。PLoS One, 2013;8:3。https://doi.org/10.1371/journal.pone.0057110.
- 17.
Rantong G, Gunawardena AHLAN。程序性细胞死亡:植物和动物中参与信号传递、调控和执行的基因。植物学。2015;93(4):193 - 210。https://doi.org/10.1139/cjb-2014-0152.
- 18.
兰彤,范·德·凯伦,范·布鲁塞姆,古纳瓦德纳。花边植物叶片发育中差异表达基因的鉴定。中国植物科学,2016;26(5):529 - 529。https://doi.org/10.1086/684748.
- 19.
Reza SH, Delhomme N, Street NR, Ramachandran P, Dalman K, Nilsson O,等。挪威云杉胚胎结构域的转录组分析揭示了胚柄细胞死亡的潜在调控因子。PLoS ONE。2018; 13 (3): e0192945。https://doi.org/10.1371/journal.pone.0192945.
- 20.
杜晓明,倪晓林,任晓林,辛桂兰,贾桂兰,刘海清,等。用从头转录组学分析方法鉴定风蒲叶片通气组织形成过程中的差异表达基因。基因。2018;662:66 - 75。https://doi.org/10.1016/j.gene.2018.03.099.
- 21.
李鹏,Ponnala L, Gandotra N,王玲,司杨,Tausta SL,等。玉米叶片转录组的发育动态。中国科学。2010;42(12):1060。https://doi.org/10.1038/ng.703.
- 22.
Gross SM, Martin JA, Simpson J, abraam - juarez MJ, Wang Z, Visel A.耐干旱CAM植物、沙漠龙舌兰和龙舌兰的转录组组装。BMC基因组学杂志,2013;14(1):563。https://doi.org/10.1186/1471-2164-14-563.
- 23.
水稻叶片发育所需的YABBY基因表达被一个wuschellike HOMEOBOX基因抑制。植物科学进展。2007;44(1):366 - 366。https://doi.org/10.1104/pp.107.095737.
- 24.
郭z, Fujioka S, Blancaflor EB, Miao S, Gou X, Li J. TCP1通过调控拟南芥关键生物合成基因DWARF4的表达调控油菜素类固醇的生物合成。植物学报。2010;22(4):1161-73。https://doi.org/10.1105/tpc.109.069203.
- 25.
丈夫们AY, Chitwood DH, Plavskin Y, Timmermans MCP。信号和预模式:植物器官极性的新见解。基因开发2009;23(17):1986-97。https://doi.org/10.1101/gad.1819909.
- 26.
Kaplan-Levy RN, Brewer PB, Quon T, Smyth DR.三螺旋转录因子家族-光,应激和发育。植物科学进展,2012;17(3):163-71。https://doi.org/10.1016/j.tplants.2011.12.002.
- 27.
金海生,金世杰,阿巴西,Bressan RA,尹金杰,俞sd,等。DOF转录因子Dof5.1通过促进转轮转录影响拟南芥叶片轴向格局。植物学报,2010;36(3):344 - 344。https://doi.org/10.1111/j.1365-313X.2010.04346.x.
- 28.
刘涛,大桥伊藤K, Bergmann DC。拟南芥气孔bHLH基因同源物及其对草类气孔发育的调控。发展。2009;136(13):2265 - 76。https://doi.org/10.1242/dev.032938.
- 29.
Reyes JC, Muro-Pastor MI, Florencio FJ。拟南芥和水稻中的GATA转录因子家族。植物科学进展。2004;34(4):344 - 344。https://doi.org/10.1104/pp.103.037788.
- 30.
Sawa S, Watanabe K, Goto K, Kanaya E, Morita EH, Okada K.丝状花,拟南芥分生组织和器官识别基因,编码具有锌指和hmg相关结构域的蛋白质。基因工程学报1999;13(9):1079-88。https://doi.org/10.1101/gad.13.9.1079.
- 31.
宋永华,李毅,李世善,今泉T,洪继昌。拟南芥光周期开花过程中,CONSTANS和不对称叶片1复合体参与开花位点T的诱导。植物学报,2012;26(2):332 - 332。https://doi.org/10.1111/j.1365-313X.2011.04793.x.
- 32.
转录组足迹揭示了拟南芥活性氧信号的特异性。植物科学进展。2006;26(2):339 - 344。https://doi.org/10.1104/pp.106.078717.
- 33.
郭晓峰,陈志伟,陈志伟,陈志伟。活性氧对植物胁迫响应和细胞程序性死亡的调控作用。BioEssays。28 2006;(11): 1091 - 101。https://doi.org/10.1002/bies.20493.
- 34.
杨晓明,王晓明,王晓明,等。植物ros介导的非生物胁迫诱导细胞程序性死亡。植物学报2015;6:1。https://doi.org/10.3389/fpls.2015.00069.
- 35.
Dauphinee AN, Wright H, Rantong G, Gunawardena AHLAN。乙烯在蕾丝植物重塑过程中参与细胞程序性死亡和气候样行为(Aponogeton madagascariensis树叶)。植物学。2012;90:1237-44。https://doi.org/10.1139/b2012-093.
- 36.
Denbigh GL, Dauphinee AN, Fraser MS, Lacroix CR, Gunawardena AHLAN。生长素在花边植物发育调控细胞程序性死亡中的作用。中国科学(d辑),2010;26(4):366 - 366。https://doi.org/10.1002/ajb2.1463.
- 37.
兰彤,埃文斯R,古纳瓦德纳。蕾丝植物乙烯受体AmERS1a和AmERS1c在叶片形态发生过程中调控乙烯诱导的程序性细胞死亡。中国生物医学工程学报,2015;29(3):344 - 344。https://doi.org/10.1007/s11103-015-0356-4.
- 38.
闫美美,谢德龙,曹俊杰,夏晓军,石凯,周永华,等。油菜素类固醇介导的活性氧在番茄绒毡层降解和花粉育性中起重要作用。植物学报,2010;36(5):344 - 344。https://doi.org/10.1111/tpj.14672.
- 39.
李志强,李志强,李志强,等。低氧胁迫下植物氮代谢的研究进展。足底。2014;239(3):531 - 41。https://doi.org/10.1007/s00425-013-2015-9.
- 40.
卜超,张强,曾杰,曹旭,郝忠,乔东,等。真菌sydowii曲霉H-1花青素合成新途径的鉴定。BMC基因组学。2020;21(1):29。https://doi.org/10.1186/s12864-019-6442-2.
- 41.
Argout X, Fouet O, Wincker P, Gramacho K, Legavre T, Sabau X,等。为了理解可可转录组:制作和分析来自不同组织和不同条件下的可可(Theobroma cacao L.) est详尽数据集。BMC基因组学。2008;9(1):512。https://doi.org/10.1186/1471-2164-9-512.
- 42.
Rowarth NM, Dauphinee AN, Denbigh GL, Gunawardena AHLAN。Hsp70在花边植物(Aponogeton madagcariensis)叶片重塑过程中在程序性细胞死亡中发挥作用。中国科学(d辑:自然科学),2010;https://doi.org/10.1093/jxb/erz447.
- 43.
张海龙,张海龙,张海龙,等。芳香装饰决定花青素液泡包体的形成。中国动物医学杂志,2017;27(7):945-57。https://doi.org/10.1016/j.cub.2017.02.027.
- 44.
古娜瓦德纳娜·阿兰,格林伍德·JS,邓勒·NG。马达加斯加苦苣苔科植物细胞程序性死亡过程中细胞壁的降解和修饰。中国科学(d辑),2007;https://doi.org/10.3732/ajb.94.7.1116.
- 45.
杨晓明,李志刚,李志刚,杨晓明。植物细胞生长的分子网络调控。植物学报,2017;35:98-104。https://doi.org/10.1016/j.pbi.2016.11.010.
- 46.
Landi S, Hausman JF, Guerriero G, Esposito S.禾科与非生物胁迫:关注干旱和盐胁迫,最近的见解和观点。中国植物科学,2017;8:1214。https://doi.org/10.3389/fpls.2017.01214.
- 47.
非生物胁迫下细胞壁重塑。植物科学进展(英文版);https://doi.org/10.3389/fpls.2014.00771.
- 48.
王志刚,王志刚,王志刚。植物水通道蛋白:具有扩展作用的多功能水和溶质通道。植物学报,2002;25(2):173-94。https://doi.org/10.1046/j.0016-8025.2001.00791.x.
- 49.
Kaimal JM, Kandasamy G, Gasser F, Andréasson C.协同Hsp110和Hsp104活性在酿酒酵母中的能量蛋白分解。中国生物医学工程学报,2017;37(11):e00027-e117。https://doi.org/10.1128/MCB.00027-17.
- 50.
沈勇,张志刚,张志刚,张志刚。热休克蛋白70相互作用蛋白(CHIP)的Co-chaperone羧基端起介导作用α-突触核蛋白降解在蛋白酶体和溶酶体途径之间的决定。中国生物医学工程学报,2005;29(4):528 - 528。https://doi.org/10.1074/jbc.M503326200.
- 51.
齐颖,王辉,邹艳,刘超,刘颖,王艳,张伟。水稻线粒体热休克蛋白70过表达抑制细胞程序性死亡。中国科学(d辑),2011;https://doi.org/10.1016/j.febslet.2010.11.051.
- 52.
金新华,黄bk .辣椒热休克蛋白70a与III型效应物AvrBsT相互作用引发植物细胞死亡和免疫。中国生物医学工程学报。2015;37(2):357 - 357。https://doi.org/10.1104/pp.114.253898.
- 53.
达梅托A,布冯G,布拉西ÉA dos R, Sperotto RA。泛素化途径作为水稻非生物胁迫耐受性的靶标。植物信号行为研究。2015;10(9):e1057369。https://doi.org/10.1080/15592324.2015.1057369.
- 54.
杨晓明,杨晓明,张晓明,等。拟南芥hsp90家族基因HSP81的组织特异性表达分析。植物生理学报。1994;35(8):1207-19。https://doi.org/10.1093/oxfordjournals.pcp.a078715.
- 55.
陈娟,高涛,万生,张勇,杨娟,余勇,王伟。茶树HSP基因超家族的全基因组鉴定、分类及表达分析。国际分子化学杂志,2018;19(9):2633。https://doi.org/10.3390/ijms19092633.
- 56.
Behl C. Breaking BAG:健康与疾病中的联合伴侣BAG3。药理学进展,2016;37(8):672-88。https://doi.org/10.1016/j.tips.2016.04.007.
- 57.
BAG蛋白:一个无处不在的伴侣调节家族。细胞生物学与生物工程学报,2008;26(2):344 - 344。https://doi.org/10.1007/s00018-008-7535-2.
- 58.
李林,邢勇,常东,方松,崔斌,李强,等。CaM/BAG5/Hsc70信号复合物动态调控叶片衰老。科学通报2016;6(1):1 - 2。https://doi.org/10.1038/srep31889.
- 59.
李艳,Williams B, Dickman M.拟南芥B细胞淋巴瘤2 (Bcl-2)相关的athanogene 7 (BAG7)介导的耐热性需要易位、sumoylation和与WRKY29结合。中国生物医学工程学报,2017;26(2):366 - 366。https://doi.org/10.1111/nph.14388.
- 60.
Williams B, Kabbage M, Britt R, Dickman MB. AtBAG7是拟南芥bcl -2相关的致病因子,存在于内质网中,参与未折叠蛋白反应。自然科学进展,2010,26(3):344 - 344。https://doi.org/10.1073/pnas.0912670107.
- 61.
唐瑞杰,贾恩·S, Bhowmik SD, Ferguson BJ, Kabbage M, Mundree S, Williams B.植物PCD、胁迫响应和寄主防御中的bag中心性。植物科学,2020。https://doi.org/10.1016/j.tplants.2020.04.012.
- 62.
CEN勋爵,Wertman JN, Lane S, Gunawardena AHLAN。在程序性细胞死亡过程中,线粒体在蕾丝植物叶子的重塑中起作用吗?植物学报。2011;11(1):102。https://doi.org/10.1186/1471-2229-11-102.
- 63.
熊勇,Contento AL, Bassham DC。AtATG18a是拟南芥在营养胁迫和衰老过程中自噬体形成所必需的。植物学报,2005;42(4):535 - 538。https://doi.org/10.1111/j.1365-313X.2005.02397.x.
- 64.
赖志,王峰,郑志,范斌,陈志。自噬在植物抗真菌坏死病原菌中的重要作用。植物学报,2011;26(6):344 - 344。https://doi.org/10.1111/j.1365-313X.2011.04553.x.
- 65.
花冈H,野田T,白野Y,加藤T,林林H,柴田D,等。拟南芥自噬基因的破坏加速了叶片衰老和饥饿诱导的褪绿。植物科学进展。2002;29(3):379 - 379。https://doi.org/10.1104/pp.011024.
- 66.
刘勇,熊勇,Bassham DC。自噬是植物耐受干旱和盐胁迫所必需的。自噬。2009;5(7):954 - 63。https://doi.org/10.4161/auto.5.7.9290.
- 67.
熊勇,Contento AL, Bassham DC。在拟南芥中,自噬的破坏导致了本构性氧化应激。自噬。2007;3(3):257 - 8。https://doi.org/10.4161/auto.3847.
- 68.
杨震,Klionsky DJ。自噬的分子机制综述。正确观点:感染和免疫中的自噬。柏林:施普林格;2009.p .学会年会。https://doi.org/10.1007/978-3-642-00302-8_1.
- 69.
Baena-González E, Rolland F, Thevelein JM, Sheen J.植物胁迫和能量信号转录网络的中央集成商。大自然。2007;448(7156):938 - 42。https://doi.org/10.1038/nature06069.
- 70.
刘勇,Bassham DC。自噬:植物细胞自食的途径。植物科学进展。2012;https://doi.org/10.1146/annurev-arplant-042811-105441.
- 71.
王晓明,张志刚,张志刚,张志刚。复活植物耐干燥的分子机制研究。中国生物医学工程学报(自然科学版),2012;https://doi.org/10.1007/s00018-012-1088-0.
- 72.
黄萍,鞠华文,闵俊华,张欣,金世胜,杨建基,等。l型凝集素样蛋白激酶1的过表达赋予拟南芥对病原体的抗性并调节盐度反应。植物科学进展(英文版);https://doi.org/10.1016/j.plantsci.2012.12.019.
- 73.
Maksaev G, shots JM, Ohri S, Haswell ES。机械敏感通道MSL10的假定孔衬螺旋中的非极性残基影响通道行为并建立非导电函数。植物导刊,2018;2(6):e00059。https://doi.org/10.1002/pld3.59.
- 74.
王艳,李志刚,王志刚。l型凝集素受体激酶:植物免疫中的新力量。公共科学图书馆,2017;13(8):e1006433。https://doi.org/10.1371/journal.ppat.1006433.
- 75.
罗海红,李丽娟,宋飞,李丽娟。拟南芥葡萄孢菌易感基因RING E3连接酶是调控病原菌和胁迫反应的一个亚类。植物科学进展。2010;34(4):379 - 379。https://doi.org/10.1104/pp.110.163915.
- 76.
杨玉玲,李小明。IAP家族:具有多种生物活性的内源性caspase抑制剂。细胞科学,2000;10(3):169-77。https://doi.org/10.1038/sj.cr.7290046.
- 77.
拟南芥gamyb样基因MYB33和MYB65是microrna调控基因,可冗余促进花药发育。植物学报。2005;17(3):705-21。https://doi.org/10.1105/tpc.104.027920.
- 78.
郭建明,莫里IC,裴志明,Leonhardt N, Torres MA, Dangl JL,等。NADPH氧化酶AtrbohD和AtrbohF基因在拟南芥ros依赖ABA信号通路中的作用。中国生物医学工程学报。2003;22(11):2623-33。https://doi.org/10.1093/emboj/cdg277.
- 79.
含铜胺氧化酶和依赖于fad的多胺氧化酶在植物组织分化和器官发育中起着关键作用。前沿植物科学,2016;7:824。https://doi.org/10.3389/fpls.2016.00824.
- 80.
Galsurker O, Doron-Faigenboim A, Teper-Bamnolker P, Daus A, Fridman Y, Lers A, Eshel D.与洋葱皮形成相关的细胞和分子变化提示参与程序性细胞死亡。植物科学学报,2017;7:2031。https://doi.org/10.3389/fpls.2016.02031.
- 81.
Sueldo DJ, van der Hoorn RAL。植物生命需要细胞死亡,但植物细胞死亡需要Cys蛋白酶吗?中国生物医学工程学报,2017;26(3):344 - 344。https://doi.org/10.1111/febs.14034.
- 82.
张东,刘东,吕旭,王勇,荀智,刘忠,等。半胱氨酸蛋白酶CEP1是绒毡层程序性细胞死亡的关键执行子,调控拟南芥花粉发育。植物学报。2014;26(7):2939-61。https://doi.org/10.1105/tpc.114.127282.
- 83.
Rustgi S, box - fontvieille E, Reinbothe C, Von Wettstein D, Reinbothe S. Serpin1和WSCP在拟南芥植物发育过程中对半胱氨酸蛋白酶RD21活性的差异调控。中国科学(d辑),2017,26(3):344 - 344。https://doi.org/10.1073/pnas.1621496114.
- 84.
Shindo T, Misas-Villamil JC, Hörger AC, Song J, van der Hoorn RAL。拟南芥半胱氨酸蛋白酶RD21在免疫中的作用,番茄免疫蛋白酶C14的同源物。PLoS ONE。2012年,7 (1):e29317。https://doi.org/10.1371/journal.pone.0029317.
- 85.
Balakireva AV, Zamyatnin AA。切断蛋白酶和程序性细胞死亡之间的间隙。中国植物科学,2019;10:704。https://doi.org/10.3389/fpls.2019.00704.
- 86.
高红,李荣,郭颖。拟南芥天冬氨酸蛋白酶A36和A39在植物繁殖中的作用。植物信号与行为研究。2017;12(4):e1304343。https://doi.org/10.1080/15592324.2017.1304343.
- 87.
高红,张勇,王伟,赵凯,刘超,白林,等。两种膜锚定的天冬氨酸蛋白酶有助于花粉和胚珠的发育。中国生物医学工程学报,2017;29(1):344 - 344。https://doi.org/10.1104/pp.16.01719.
- 88.
李东,张华,宋强,王玲,刘松,洪勇,等。番茄Sl3-MMP是基质金属蛋白酶家族的一员,是抵抗灰霉病和丁香假单胞菌所必需的。番茄DC3000。中国生物医学工程学报。2015;15(1):1 - 8。https://doi.org/10.1186/s12870-015-0536-z.
- 89.
Zimmermann D, Gomez-Barrera JA, Pasule C, Brack-Frick UB, Sieferer E, Nicholson TM,等。基质金属蛋白酶控制细胞死亡。中国生物医学工程学报,2016;29(2):344 - 344。https://doi.org/10.1104/pp.16.00513.
- 90.
Bollhöner B, Zhang B, Stael S, Denancé N, Overmyer K, Goffner D,等。AtMC9在木质部导管元件中的死后功能。中国农业科学,2013;32(2):498-510。https://doi.org/10.1111/nph.12387.
- 91.
齐亚齐亚尼L, Timmerman E, De Bock PJ, Vercammen D, Stael S, Van De Cotte B,等。拟南芥metacaspase9降解体。植物学报。2013;25(8):2831-47。https://doi.org/10.1105/tpc.113.115287.
- 92.
Rantong G, Gunawardena AHLAN。液泡加工酶AmVPE1和AmVPE2是乙烯调控植物程序性细胞死亡的潜在执行者。植物学。2018;96(4):235 - 47岁。https://doi.org/10.1139/cjb-2017-0184.
- 93.
Gunawardena AHLAN, Navachandrabala C, Kane M, Dengler NG, Teixeira da Silva JA。花边植物:一种研究发育性程序性细胞死亡的新系统。花卉栽培,观赏和植物生物技术:进展和热带问题卷1。2006年,全球科学图书有限公司,米德尔塞克斯,157-62。
- 94.
Andrews S. FastQC:高通量序列数据的质量控制工具。网上。http://www.bioinformatics.babraham.ac.uk/projects/fastqc
- 95.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学。2014;30(15):2114 - 20。https://doi.org/10.1093/bioinformatics/btu170.
- 96.
葛瑞哲,哈斯,李志刚,李志刚,李志刚,李志刚。来自RNA-Seq数据的全长转录组组装,没有参考基因组。中国生物工程学报。2011;29(7):644-52。https://doi.org/10.1038/nbt.1883.
- 97.
李B,杜威CN。RSEM:从RNA-Seq数据中精确的转录物定量,有或没有参考基因组。生物信息学杂志。2011;12(1):323。https://doi.org/10.1186/1471-2105-12-323.
- 98.
Robinson MD, Oshlack A. RNA-seq数据差异表达分析的标度归一化方法。中国生物工程学报。2010;11(3):1-9。https://doi.org/10.1186/gb-2010-11-3-r25.
- 99.
李志强,李志强,李志强,等。利用Trinity平台从RNA-seq中重建转录本序列,用于参考生成和分析。网络学报。2013;26(8):1494-512。https://doi.org/10.1038/nprot.2013.084.
- One hundred.
罗宾逊M,麦卡锡DJ,史密斯GK。edgeR:数字基因表达数据的差异表达分析。生物信息学,2010,26(1):139 - 40。https://doi.org/10.1093/bioinformatics/btp616.
- 101.
一种基于错误发现率控制的简单前向选择程序。生态学报,2009;3(1):179-98。https://doi.org/10.1214/08-AOAS194.
- 102.
Barter RL, Yu B. Superheat:一个为复杂数据可视化创建漂亮和可扩展热图的R包。中国计算图形学报,2018;27(4):910-22。https://doi.org/10.1080/10618600.2018.1473780.
- 103.
Bryant DM, Johnson K, DiTommaso T, Tickle T, Couger MB, Payzin-Dogru D,等。组织映射的蝾螈新生转录组能够识别肢体再生因子。Cell Rep. 2017;18(3): 762-76。https://doi.org/10.1016/j.celrep.2016.12.063.
- 104.
UniProt财团。UniProt:通用蛋白质知识库。中国生物医学工程学报,2018;46(5):2699。https://doi.org/10.1093/nar/gky092.
- 105.
KEGG:京都基因和基因组百科全书。核酸研究,2000;28(1):27-30。https://doi.org/10.1093/nar/28.1.27.
- 106.
塔图索夫RL,库宁EV,李普曼DJ。蛋白质家族的基因组视角。科学。1997;278(5338):631 - 7。https://doi.org/10.1126/science.278.5338.631.
- 107.
金J,田F,杨,孟YQ,香港L,罗J,高g . PlantTFDB 4.0:对中央在植物转录因子和监管中心交互。核酸研究,2017;45:D1040。https://doi.org/10.1093/nar/gkw982.
确认
我们要感谢Michaela Kember(达尔豪斯大学)对手稿进行了严格的审查,并帮助提供了蕾丝植物叶子用于RNA提取。
资金
这项研究由加拿大自然科学与工程研究委员会(NSERC)的发现拨款(# 2017-04299)和授予AHLANG的加速器补充(# 2017-507825)资助。NMR得到了NSERC研究生奖学金、新斯科舍省研究生奖学金和AHLANG的发现助学金的支持。资助机构在研究设计、数据收集、分析和解释以及撰写手稿方面没有任何作用。
作者信息
从属关系
贡献
构思研究,参与设计和协调,获得资金并监督所有实验工作:AHLANG。完成所有实验,参与设计,分析数据,起草第一份手稿:NMR。帮助生物信息学:BAC。参与研究设计,协助生物信息学:ALE。稿件修订:NMR, ALE, BAC, JMA, CRL和AHLANG。所有作者审阅并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者没有声明与作品有任何竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:数据集S1。
三位一体装配的映射率。
附加文件2:图S1。
蔡司激光捕获显微解剖实验。(A)激光分离的细胞群,(B)细胞群提取后剩余的乳晕组织。
附加文件3:数据集S2。
叶期聚类注释、中值表达值、GO项计数和GO富集结果。
附加文件4:图S2。
叶期和细胞类型实验的qRT-PCR验证。所有检测基因的拷贝数以α-微管蛋白的拷贝数归一化。
附加文件5:数据集S3
.NPCD和PCD细胞簇注释、中位表达值、GO项计数和GO富集结果。
附加文件6:数据集S6。
qRT-PCR引物信息。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
罗沃斯,新硕士,柯蒂斯,文学学士,艾因费尔特,A.L.et al。RNA-Seq分析揭示了蕾丝植物细胞程序性死亡和叶片重塑的潜在调控因子(Aponogeton madagascariensis).BMC植物生物学21日,375(2021)。https://doi.org/10.1186/s12870-021-03066-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-021-03066-7
关键字
- 花青素
- 发展纤毛运动
- RNA序列
- 激光捕获显微解剖
- 转录组