摘要
背景
比诺家族的葡萄品种代表了具有主要表型和生理差异的克隆繁殖突变体,如不同的颜色或成熟时间的转移,以及重要的葡萄栽培性状的变化。具体来说,品种“黑比诺”(PN)和“黑比诺早熟”(PNP,早熟)在同一时间开花,但在浆果成熟的开始(版本)和收获时间上有所不同。除了基因型外,季节性气候条件(即高温)也会影响成熟时间。为了揭示可能影响变异发生时间的调控基因,我们用紧密网格的时间序列和两年多的时间,研究了在浆果发育过程中PN和PNP之间的基因表达谱的差异。
结果
在应用的生长条件下,使用已证实的PN和PNP克隆关系的植物材料,PN和PNP之间浆果形成时间的差异被量化为大约两周。共表达基因簇和差异表达基因簇反映了变异发生时间的变化。这些DEGs的功能注释与浆果发育过程中观察到的表型和生理变化相吻合。在PN和PNP之间,我们在2014年共观察到3342个deg, 2017年观察到2745个deg,这两年共观察到1923个deg。其中,388个基因被鉴定为变种特异性基因,12个基因被认为是浆果成熟时间调控候选基因。表达谱显示了我们指定的两个成熟时间控制的候选基因VviRTIC1而且VviRTIC2(分别为VIT_210s0071g01145和VIT_200s0366g00020)。这些基因可能促成了PN和PNP之间观察到的表型差异。
结论
如果按照发育阶段进行排列,1923个DEGs中的许多表达谱在两个品种中高度相似。在我们的工作中,确定了PNP和PN之间差异表达的控制成熟时间的基因以及变体特异性基因。我们指出了这些基因在浆果发育过程中与分子事件的联系,并讨论了可能控制成熟时间的潜在候选基因。其中两个候选基因在浆果发育早期被观察到表达差异。浆果成熟过程中几个下调的基因被注释为生长素反应因子/ ARFs。可想而知,生长素信号的普遍变化可能导致PNP早熟表型的提前。
背景
vinifera subsp;酿酒用葡萄(小道消息)属于这个家族葡萄科.它有6000到11000个品种,是世界上最重要的多年生作物之一[1].葡萄果实发育可分为两个生理阶段,浆果形成和浆果成熟。变异是指浆果从形成到成熟的过渡,这两个阶段中的每一个都用发育的s型生长曲线表示[2].在发展过程中所取得的进展被称为“BBCH阶段”(源自参与阶段定义的协调机构名称的首字母缩写),这些阶段已被定义为包括葡萄在内的几种作物[3.,4].第一个生理阶段被描述为浆果形成(浆果开始和细胞分裂生长),从开花结束(BBCH71)持续到大约60天后,当大多数浆果彼此接触(BBCH79)。变异(BBCH81)的发育阶段是浆果形成的结束和浆果成熟的开始[2].表型上,变异是浆果开始变软时的发育开关,伴随着苯丙素的积累。在红葡萄品种中,变异还表现为浆果的颜色变化,这是由花青素(苯丙素的主要类)开始积累引起的。已得到充分研究的R2R3-MYB转录因子(tf)超家族的成员被认为主要负责控制花青素的积累[5,6,7].浆果的成熟开始于版本,一直持续到收获(BBCH89),这一阶段包括细胞增大,糖分积累和酸度下降。
在控制这一性状的遗传位点和基因组区域的水平上,也研究了变异的时间。由于它是一种受多个或多个遗传位点影响的数量性状,因此对其进行了定量性状位点(QTL)分析。这些研究在16号染色体上发现了一个主要的QTL,并在18号染色体上发现了一个次要的QTL [8].通过整合一些QTL研究,发现了几个与遗传控制的变异时间相关的元QTL,其中最相关的位于染色体14、16和18上[9].
人为气候变化导致葡萄提前成熟,对浆果质量产生重大影响,进而影响理想葡萄酒风格的预期风味[10].此外,由于地区和/或特定年份的天气条件差异,一个特定品种的变异和收获时间可能相差很大。显然,这需要对葡萄成熟时间的控制有更好的分子理解。
比较在相同环境条件下生长的不同葡萄品种,通常可以发现浆果形成时间、变异时间、浆果成熟时间和一般成熟时间的差异。然而,潜在的遗传因素大多是未知的。以往的研究已经阐明了内部和外部因素对成熟时间的影响。例如,植物激素对浆果成熟的影响已被广泛研究[1].一般来说,水果的生长被认为是由几种植物激素控制的,它们在触发或延迟成熟过程中起着至关重要的作用[11].葡萄是一种非更年期水果,脱落酸(ABA)的影响已在许多研究中进行了研究,因为ABA被认为是触发成熟的[12,13,14].此外,ABA还参与控制叶片衰老[15,对干旱的反应[16和病原体防御[17].在葡萄藤中,虽然不像在番茄等更年期水果中那样重要(茄属植物lycopersicum),植物激素乙烯参与控制浆果的成熟[1,13,18,19],而生长素已被证明会导致成熟的延迟[20.,21].
人们对干水果和肉质水果的发育都进行了非常深入的研究,原因很明显,水果对人类营养至关重要[22,23].多肉水果研究的主要模式系统是番茄,因为已有的遗传学和分子生物学、突变体的获取和非常先进的转基因方法可用于基因功能鉴定[24,25].葡萄果实的发育也得到了深入的研究[1,26,通常在转录组水平上。在相当多的研究中,主要是对浆果发育后期进行采样,以使变异的发育阶段成为关注的焦点[9,27,28,29].此外,用粗时间点分布研究了整个浆果的发育[30.,31,32,33,34].
为了在葡萄果实发育过程中,从开花到成熟,以高分辨率监测基因表达的变化,我们从两个皮诺品种中采样了两年的综合时间序列。这些样品是从葡萄品种“黑比诺”(PN)和相对较早的成熟品种“黑比诺早熟”(PNP)中收集的,该品种预计与PN密切相关。品种PNP被列在葡萄属国际品种目录(V印度河流域文明;[35]),并被描述为与PN同时开花,但达到版本明显早于PN [36].转录水平的定量数据,解释为基因表达的值,由RNA-Seq生成。我们研究了在年份和品种的浆果发育过程中基因表达模式的一般过程,并在PN和PNP之间识别了许多差异表达基因(DEGs)。这些deg可以被认为是延迟或推动浆果发育的重要候选者。我们的主要目的是识别控制生长速度的基因,为描述葡萄的相关分子功能提供一个切入点,并促进未来的育种策略,以解决与气候变化相关的性状和受气候变化影响的性状。
结果
两个皮诺品种的表型比较
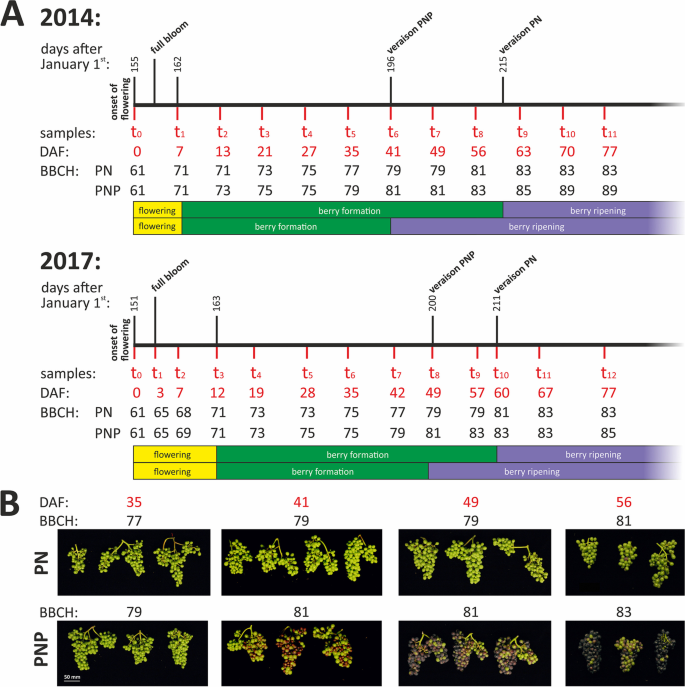
为了研究成熟的变化,我们使用了两个密切相关的葡萄品种的样本。品种PNP是其祖先PN的一个较早成熟的无性系变异。PN和PNP的克隆关系由一组24个SSR标记证实,所有标记在两个品种中显示出相同的等位基因状态(附加文件1:表S1)。为了确认和验证PN和PNP之间的表型差异,确定并记录详细的BBCH发育阶段(图。1).PN和PNP在发育和开花(BBCH65)过程中同时表现出相似的表型特征。然而,PNP的版本(BBCH81)提前了约2周,并且在四个不同的年份观察到类似的变化(表1).此外,图。1A显示了采样时间点的概况。PN和PNP之间的表型差异可以在浆果形成开始和变异期间所拍摄的发育浆果的图像中得到说明(图。1B和附加文件1:表S2、表S3)。Veraison (BBCH81)在花青素积累开始时可见,在PN中检测到的时间比PNP晚约2周。
基因表达模式的全球视角
我们在2014年和2017年从开花到改版期间收集了三个重复样本(时间点见图)。1和附加文件1:表S2和表S3),单个收获被称为以下的子样本,并通过RNA-Seq进行分析。在对原始数据进行预处理之后(参见材料与方法),从每个子样本派生的reads被映射到来自PN40024的参考序列,并相对于CRIBI V2.1注释数据集进行分析。2014年,从72个图书馆中每个子样本获得了大约1970万次阅读。与葡萄参考基因组序列的整体比对率为79%。2017年,从78个图书馆中每个子样本获得了约4350万次阅读。从这些数据中,计算出与参考点的总体对齐率为92%。表达值最初被检测为转录物每千基百万(TPM),并在每个样本的三个子样本上平均。分别考虑两个年份,共检测到28,692个在两个品种和两个年份表达的基因。相比之下,有2152个CRIBI V2.1基因未表达。
基因表达数据之间的相关性,确定为每个样本的TPM值,从两个年份的数据集的所有基因rPN和的皮尔逊相关系数= 0.5095rPNP分别为0.6557。PN和PNP在2014 - 2017年间分别有10205和16226个基因表达值显著相关(p值< 0.05)。附加文件中提供了与BBCH阶段相同的八个时间点的相关强度列表1:表S4。
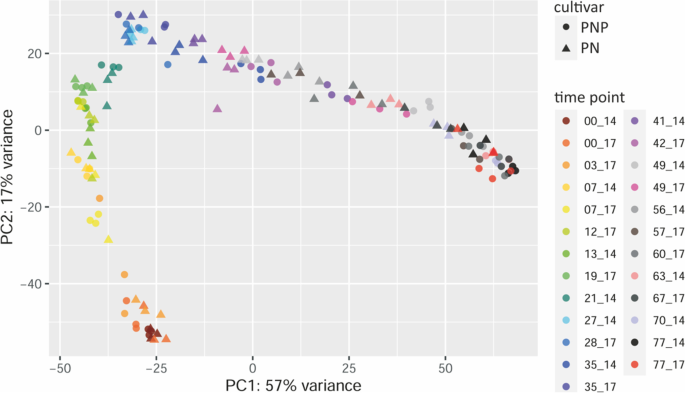
为了可视化从所有子样本中获得的基因表达值的全球趋势和相似性,在保持子样本分离的情况下,进行了两年的主成分分析(PCA)。第一个成分PC1解释了57%的方差,而第二个成分PC2解释了17%(图。2).几乎所有这两年的子样本(一个样本内的三个副本)的数据点都彼此聚集在一起。数据以近乎连续的方式跟随时间的轨迹。影响数据中大部分方差的主要因素是与细胞壁修饰、次生代谢、损伤反应和激素信号转导相关的基因。在PC1和PC2中负责大部分变异的前500个基因列在附加文件中1:表S5和表S6,以及来自MapMan/Mercator和RefSeq的每个列出基因的功能信息。
共表达基因的聚类分析
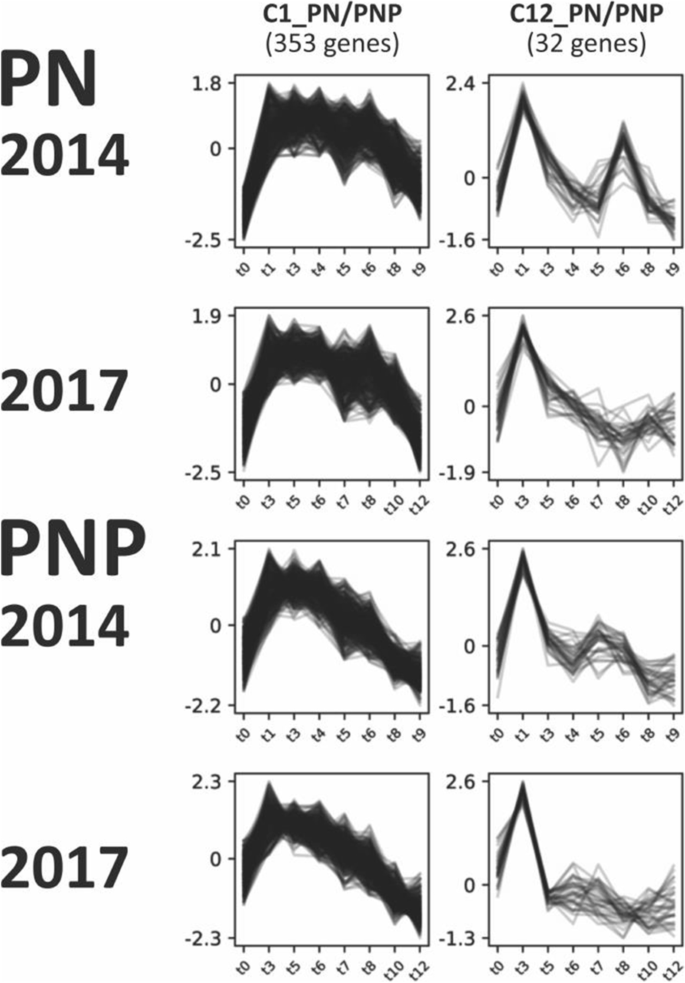
基因表达时间序列谱结合了两种品种的子样本/三倍体在两年内每个时间点的数据。使用聚类工具CLUST比较表达谱。目的是在整个浆果发育过程中,描述不同年份和品种之间基因表达的相似性和/或差异。在所有四个数据集中,获得了13个具有相似基因表达模式(C1-C13)的PN/PNP基因集群(附加文件)2:图S1A)。在这些集群中,在果实发育过程中表达的27139个基因中,有3316个(12.2%)在年份和品种之间共同表达。应该注意的是,CLUST使用标准来定义表达和未表达的基因与上面应用的不同(参见材料与方法).观察到的表达谱在集群之间差异明显,这部分是CLUST提取的集群数量有限的结果。人工检测显示,在特定年份或品种的每个个体簇内,个体基因表达谱几乎没有偏差。所有的聚类基因成员关系,以及额外的特定品种聚类分析的成员关系(见下文),可在附加文件中获得1:表S7-S9。
PN/PNP集群C2、C5、C6、C12(以C12_PN/PNP为例,见图。3.)显示两个采样年份的基因表达谱有微小但可检测的差异,但两个品种的基因表达谱几乎相同。PN/PNP集群C1、C7和C11(以C1_PN/PNP为例,如图所示。3.)在两年内表达谱相似,但因表达峰移位而突出,从而区分了PN和PNP。
为了描述包含在给定聚类中的共表达基因的潜在功能,计算了生物过程的氧化石墨烯术语富集。所有集群的丰富GO术语的完整列表列在附加文件中1:表S10-S12。氧化石墨烯术语出现的两个例子是C11_PN/PNP集群中的“氧化应激反应”(术语GO:0051276)和C5_PN/PNP集群中的“防御反应调节”(术语GO:0031347)。
进行了两个额外的聚类分析,一个是对两年的PN数据进行的2:图S1B)和两个年份的PNP数据(附加文件2:图S1C)。这些分析显示,在C0_PNP和C6_PN簇中有大量表达相似的扩展蛋白基因家族基因。C16_PN簇被发现对“营养向繁殖阶段过渡”(GO:0010228)有高度显著的富集。品种特异性聚类的聚类基因成员关系可在附加文件中找到1:表S8-9,对应的GO术语富集在附加文件中总结1:表S11-12。
差异表达基因的分析
分析了PN和PNP两个品种间差异表达基因(DEGs)在果实发育过程中的表达时间序列。筛选表达差异显著的基因,筛选方法为:筛选filter - adjusted p-value (PADJ) < 0.05和log2fold change (LFC) > 2。结果汇总见表2并在每个时间点的基因水平上进行详细的比较,在附加文件中1:表S13。
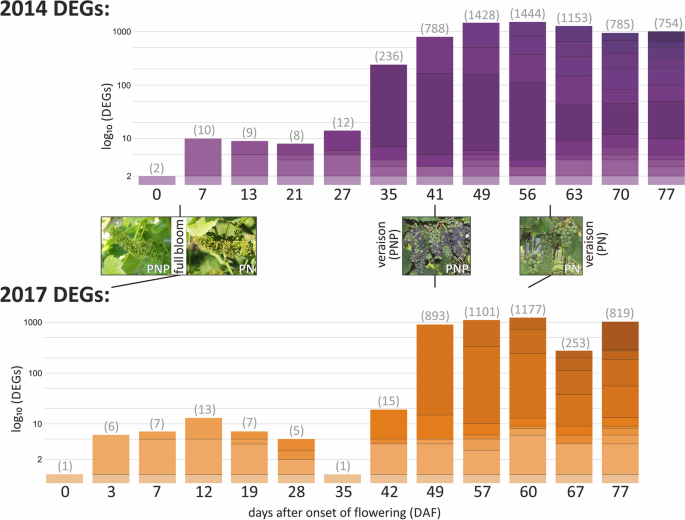
2014年和2017年共鉴定出PADJ大于0.05的deg分别为8206和4419个。与2017年相比,2014年样品中初步检测到的deg数量几乎是2014年的两倍。通过对表达水平差异至少2倍(LFC > 2)的过滤器应用,显著差异的DEGs数量减少,主要是在2014年以来的PN/PNP时间序列中。在这两个时间序列的开始,在开花期间(BBCH61 - 69)仅观察到少数PN和PNP之间的差异(见图)。4).在浆果形成阶段(BBCH71至79),检测到的DEGs数量向变异(BBCH81)增加,因为两种基因型在生理阶段的变化越来越大。与PNP相对于PN的时移版本平行观察到的deg的最高数量。这一时间点也是两个品种表型差异最大的时间点(见图)。1而且4).通过从2014年的时间点DAF35和DAF41中筛选出与2017年基因表达数据中DAF42和DAF49表型相一致的差异基因,定义了一组版本特异性基因。这些标准确定了388个特定于版本的deg。这组388个基因与类似研究的结果进行了比较,发现结果一致(例如81% [27]和52% [28];这388个基因的id,与其他研究结果相匹配的基因及其功能注释都包含在附加文件中1:表S14)。在随后的浆果成熟阶段(BBCH81 - 89,在2014年的DAF56左右和2017年的DAF60左右之后),检测到的deg数量减少。
以对数尺度(log .)显示PN和PNP之间检测到的deg数量10).2014年的结果显示为紫色,2017年的结果显示为橙色。两年来的时间序列在PNP版本中对齐;时间线以开花后的天数(DAF)给出。分别对每个时间点的一对PN/PNP样本进行deg计数,每列上面的数字表示在各自时间点检测到的deg数量。相对于较早的时间点,新出现的deg组由每个时间点的列(栏)中的新颜色阴影表示。对于给定的deg组的成员,为后续的时间点(列/样本)保留属性色度。两柱系列之间的图片显示BBCH65(盛开,50%的花冠掉落[3.])和BBCH81(版本)的PNP和PN
我们对检测到的deg的数量和在时间序列中的给定时间点(样本对PN/PNP)新出现的差异表达基因的变化进行了可视化(图1)。4).新出现的deg组在浆果形成早期只包含少量基因,而PNP变体的数量增加。PN改版后,deg数量减少。如果只计算一次出现在几个时间点的差异基因,则从2014年和2017年分别检测到3342和2745个独特的差异基因(不同基因)(对比表2).
为了进一步增加所选差异分析的可靠性、可重复性和相关性,我们计算了研究两年所确定的差异分析之间的交集。总共获得了1923个唯一的deg (Table2).为了揭示可能参与控制成熟时间的基因,即可能参与区分PN和PNP的主要性状的基因,只选取在PNP变异前的时间点出现的交叉的DEGs。这就产生了一个可能控制成熟时间的12个deg的列表。应该注意的是,这些假定的调控deg应该在变种特异性基因集实现变种表型变化之前是相关的。deg的完整列表、它们的标识和注释信息,以及它们从所有(原始)deg到潜在监管deg的过程中对选择标准的适合度,都在附加文件中详细说明1:表S13。12个假定的成熟时间控制基因的id,与相关研究结果相匹配的基因(7 DEGs [27, 4 DEGs [34和3个DEGs [28)和它们的函数注释包含在附加文件中1:表S14,最相关的数据汇总于表中3..
DEGs的功能分类
为了用葡萄藤的功能信息来补充基因列表,这些信息可能对浆果的发育有潜在的帮助,我们对1923个交叉的deg进行了分析,这些基因被分配到已经为葡萄藤建立的生物通路中(见材料与方法).在247条已确定的葡萄路径中,有46条显著富集(排列)p-value <0.1)被检测到。最可靠的预测p-value <0.001)表示可能相关的途径为光合作用天线蛋白(vv10196;9度);氮代谢(vv10910, 19 DEGs);苯丙类生物合成(vv10940, 66 DEGs),酪氨酸代谢(vv10350, 33 DEGs);输运电子载流子(vv50105, 18 DEGs);苯丙氨酸代谢(vv10360, 33 DEGs);油菜素内酯生物合成(vv10905, 8 DEGs)和类黄酮生物合成(vv10941, 30 DEGs)。浓缩结果在附加文件中提供1:表S15。同样的分析也进行了12假定的成熟时间控制(附加文件1表S16)和388个特定于版本的deg(附加文件1:表S17)。
对1923个交叉的deg进行检查,发现包含141个TF基因。其中48种deg在时间序列中首次出现时明显上调,93种下调。与PN相比,在PNP中高表达(上调)或在PNP中低表达(下调)的TF编码基因的完整列表显示在附加文件中1:表S18。
为了更详细地了解所选TF编码基因的表达模式,我们生成了141个TF基因中丰度最高的TF基因家族,即MapMan功能分配中22例的r2r3 - myb型TF的表达热图(附加文件2:图S2)。因此,VviMYB24(VIT_214s0066g01090),与At3g27810/相关AtMYB21At5g40350 /AtMYB24和At3g01530 /AtMYB57根据TAIR/PhyloGenes,它被确定为一个早期出现的DEG,在开花时表达量最高(BBCH61)。著名的R2R3-MYB已知与花青素积累相关的基因VviMYBA1(VIT_202s0033g00410),VviMYBA2(VIT_202s0033g00390),VviMYBA3(VIT_202s0033g00450)和VviMYBA8(VIT_202s0033g00380)在两个品种中均从版本(BBCH81)开始表达,并在PNP中向较早的表达时间转移。一个额外的R2R3-MYB具有相似表达模式的基因是VviMYB15(VIT_205s0049g01020)。其他R2R3-MYB基因在浆果形成的早期就表达出来了,包括VviMYBF1(VIT_207s0005g01210,与At2g47460/相关AtMYB12/AtPFG1)及VviMYBPA5(VIT_209s0002g01400)和VviMYBPA7(VIT_204s0008g01800,都与At5g35550/有关AtMYB123/AtTT2).根据他们在热图中可视化的相关表达模式(附加文件2:图S2),R2R3-MYB基因分为三组,大致符合图中所示的三个阶段。1B,即开花、浆果形成和浆果成熟讨论).
成熟时间控制基因的假定候选基因
如前所述,我们选择在PNP变异前的两年时间点检测到的DEGs作为控制成熟时间的假定基因(表3.,附加文件1:表S13)。对这12个候选基因进行的VitisNet富集分析结果显示,2个通路中有两个基因显著富集(排列p值< 0.05):生长素信号通路(vv30003 withVviEXPA5(VIT_206s0004g00070)和VviEXPA14(VIT_213s0067g02930))和细胞壁(vv40006 withVviPL1(VIT_205s0051g00590)和VviGRIP28(VIT_216s0022g00960));参见附加文件1:表S16)。
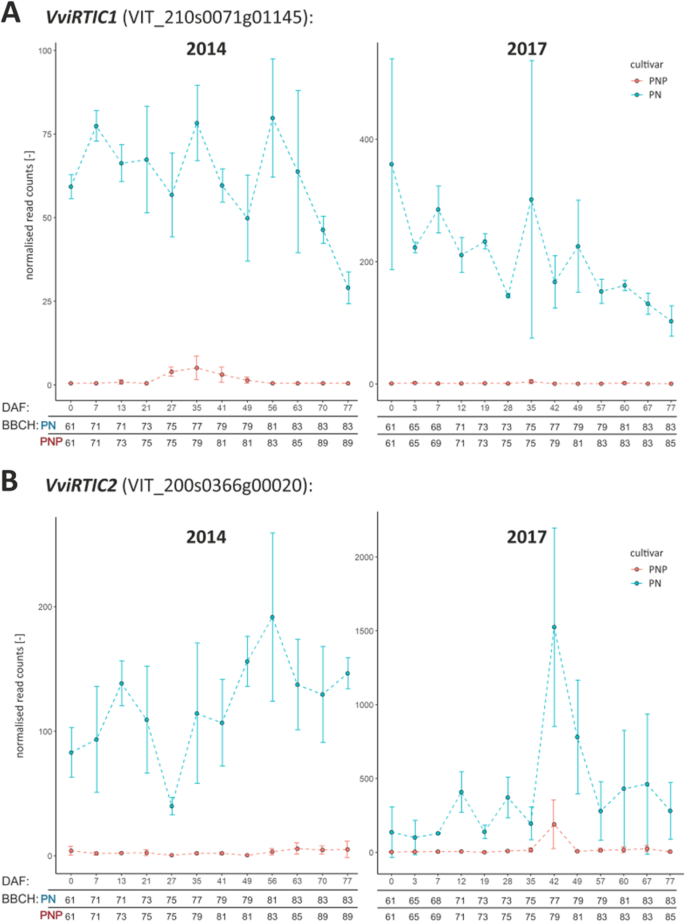
图中所示数据的详细检查。4,以及这些假定的成熟时间控制的deg的结果,导致识别出两个从整个deg列表中脱颖而出的deg。这两种基因在早熟品种PNP中几乎完全缺乏表达,而在PN中有明确表达。因此,这两个基因在这两年的整个时间序列中都被检测为DEGs。两个中的第一个,指定的VviRTIC1对于“成熟时间控制”(VIT_210s0071g01145,编码一种类似于“功能未知的DUF789蛋白”的蛋白),在开花期间表达(BBCH61 - 65),且在PN中或多或少随着时间的推移持续下调(图1)。5A)两个中的第二个,指定的VviRTIC2(VIT_200s0366g00020,编码一种类似于“富半胱氨酸受体样蛋白激酶”的蛋白),在浆果形成和浆果成熟过程中表达,在2017年变异前达到峰值(图1)。5B).假设成熟时间控制基因集的其余10个deg的表达模式见附加文件2:图S3。
12个假定的成熟时间控制基因集合的数据收集在表中3.,主要关注PNP变异前表达的相对上调或下调。此外,还对PN和PNP版本前后表达模式的差异进行了简短的描述。
来自RNA-Seq的表达模式相互确认,也允许对低转录本积累水平进行数字量化。尽管如此,我们还是选择了三个基因通过qRT-PCR进行确认,即VviRTIC1,VviRTIC2而且VviERF027(维特216年s0100g00400)。VviERF027包括在时间序列的不同样本中显示差异表达以及PN和PNP中同样高表达的基因。qRT-PCR的结果与RNA-Seq的数据完全一致(见附加文件)2:图S4)。因此,RNA-Seq和qRT-PCR检测了发育模式和基因型特异性的差异基因活性。
讨论
“Pinot Precoce”品种最早被发现与同义词“(德语)Früh Burgunder Traube,翻译为:早期勃艮第葡萄”(PNP)有关,是在法语书《Ampelographie retrospective》中[37].PNP被认为与黑比诺(Pinot noir)葡萄(PN)密切相关,在这里,我们通过24个分布良好的基因组SSR标记确定了PN和PNP的克隆关系。虽然这并不能证明PN是祖先,但很有可能PNP是通过体细胞突变从PN品种中衍生出来的,如前所述[36].我们使用了这两个以浆果形成表型持续时间为特征的等基因品种,来分析整个浆果发育过程中的基因表达变化。目的是确定控制浆果发育速度和变异时间的候选基因。从开花开始到2014年和2017年PN和PNP版本之后,从花序以及形成和成熟的浆果中收集样本。这些样品在两个良好分辨率的时间序列中进行了RNA-Seq分析。
PN和PNP品种间的表型差异
对同一地点的PN和PNP的BBCH发育阶段进行了4年仔细评估的数据证实了早期葡萄栽培的观察结果[36这导致了PNP作为一个独特的葡萄品种在北欧葡萄酒种植国家的建立。PNP中浆果形成的时间要比PN短约两周,但明显比PN更快,PNP比PN提前约两周进入版本(图1)。1一、表1).可以合理地假设,这种加速影响浆果的形成,从坐果后立即到变异。从功能上讲,这一假设意味着,负责控制浆果发育时间以及建立PN和PNP之间表型差异的基因,应该在浆果发育的早期就开始起作用,至少在开花后不久开始,甚至在BBCH61到BBCH79之间开始。在成熟(收获)结束时,与PN相比,PNP的浆果在季节内更早达到高糖含量。
RNA-Seq数据集的一般验证
为了估计总体数据质量,我们对2014年和2017年这两个采样年份的PN和PNP表达谱进行了相关分析。皮尔逊相关是适度的,但考虑到自由场环境的条件,这是预期的。葡萄藤暴露在诸如生物或非生物压力源等外部因素下,包括年份间显著不同的天气条件,也会影响转录组,从而降低相关性水平[38].在主成分分析(PCA)中,几乎所有的数据点都位于相同的预期轨迹上,来自这两年的生物重复(子样本)彼此靠近。主要影响数据集方差的主要因素是与细胞壁修饰、次生代谢、损伤和激素信号相关的基因。这些基因类别符合逻辑预期,因为已知浆果的发育(i)由激素控制,(ii)需要新的细胞壁,以及(iii)伴随着次生/特化代谢物的积累[24,26].这些初步结果验证了数据集的质量,并清楚地表明,子样本/三副本的生物密切相关材料的采样是成功的。
共表达分析表明,不同品种和年份间的基因表达簇相似
为了进一步验证两年间数据的可比性和可重复性,通过对来自四个不同时间序列的数据进行聚类,我们在所有基因中识别了相关的基因表达谱。一般来说,随着时间的推移,在两个品种和两个年份中都观察到具有相似表达模式的相同基因簇。此外,对2014年和2017年仅PN和仅PNP的基因表达谱进行聚类分析,证实了这两年获得的基因表达谱的可比性。详细的检查发现了有或没有环境影响的表达谱簇(和基因簇)。其中以C1_PN/PNP、C7_PN/PNP和C11_PN/PNP品种特异性集群最为突出。比较这些集群中PN和PNP的表达谱,发现了一个相似的模式,在PNP中移动到不同的时间。这些结果与上述两个品种的成熟时间转移物候学相一致。相比之下,C2_PN/PNP、C5_PN/PNP、C6_PN/PNP和C12_PN/PNP群体在两年内的模式相似性高于两个品种。因此,这四个集群中的基因可能在它们的表达模式中显示出对环境因素的依赖,这可能是由于研究的两年之间的天气条件的差异。在意大利北部的11个不同环境(葡萄园)中,也描述了强烈的环境对基因表达模式的影响[39].其余的PN/PNP簇C0, C3, C4, C8, C9和C10在所有四种情况下表现出高度相似的表达谱。这些聚类中的基因可能较少受环境因素和/或PN和PNP之间的基因型差异的影响。我们的结论是,我们的RNA-Seq结果和两年间的表达水平比较是基于有效的数据。
当DEGs的基因组位置被分析时,第16号染色体上的基因组区域成为焦点。在这个区域,从1923个交叉的差异集合中选出54个差异(表2)。其中28种编码二苯乙烯合成酶[40],它们在PNP (BBCH83)版本后都上调。二苯乙烯是一组苯丙类化合物(包括白藜芦醇),存在于许多植物中,经常在生物和非生物胁迫下积累,是由关键酶二苯乙烯合成酶形成的一种基本结构。该基因组区域与第16号染色体上“变异发生时间”的一个主要QTL (Ver1)相吻合[8].deg的聚集是否偶然还有待确定。由于大量紧密相连的二苯乙烯合成酶基因的共同调控,观察结果可能存在偏差。
在浆果发育过程中差异表达的基因和veraison特异性基因的鉴定
差异基因表达分析和随后的筛选发现PNP和PN之间有1923个DEGs。DEG检测是基于DAF非常相似的两个品种的样品的比较。正如所预期的那样,当PN仍然处于浆果形成阶段时,PNP达到了变异。因此,与滞后PN相比,PNP的不同发展阶段导致了PNP前不久和PNP版本中检测到的DEGs数量的强烈增加。随后,当PN也进入版本时,deg的数量下降(注意图。4使用对数刻度)。我们提取了这两年中出现的388个基因,并将其与已发表的结果进行了比较。有趣的是,在鲜果葡萄品种‘8612-CTR’(野生型)与其早熟芽突变体‘8612-ERN’之间的4,351个差异表达基因中,也描述了约81.5%的388个PN/PNP变异特异性基因[27].此外,通过RNA-Seq对赤霞珠和黑比诺两种品种的浆果进行分析,发现了一个由5404个基因组成的基因集,标志着浆果成熟的开始[28].这个集合涵盖了388个PN/PNP基因特异性基因的51.5%1:表S14)。几个“开关基因”被认为编码发育过渡的关键调控因子[34,41]也包含在388版本特异性基因集中(附加文件1:表S14)。我们得出结论,由388个PN/PNP变异特异性基因组成的基因集代表了一组与浆果形成到成熟转换相关的核心基因集。一个相对较小的基因集被检测到,但与其他解决类似生物学问题的研究发现的基因集仍然有很高的重叠,这表明这里使用的特定实验设置和实现过滤器适用于删除不相关的基因。在这里,“野生型到突变型”RNA-Seq的比较导致了PN和PNP之间的等基因背景,通过在相同的葡萄园/位置采样,减少了环境控制的转录组差异,密集的时间过程采样和较高的RNA-Seq读覆盖使得良好的分辨率。
为了检查veraison特异性基因集中潜在的共表达基因,研究了这些基因在PN/PNP聚类分析中的成员关系。在C6_PN/PNP(包含914个基因)聚类中共检测到48个变种特异性DEGs。这48个基因包括几个显著的与成熟相关的基因,比如VviGRIP61(VIT_201s0011g05110),VviMYBA8,VviMRIP1(VIT_205s0049g00760 [42]),VviGRIP4(VIT_205s0049g00520)和VviGRIP28.~ 20VviGRIP通过差异cDNA筛选,在葡萄[43].另一个相关的集群是C5_PN/PNP(包含263个基因),其中包括388个变体特异性deg中的37个。其中包括VviMYBA1,VviMYBA2,VviMYB15而且VviGRIP22(VIT_206s0004g02560)。两个集群C5_PN/PNP和C6_PN/PNP显示出非常相似的模式(附加文件2:图S1A)。一开始并不明显,是哪个差异迫使CLUST将一个给定的基因放在C5_PN/PNP或C6_PN/PNP中。然而,比较的表达模式VviMYBA2(在C5_PN/PNP)和VviMYBA8(在C6_PN / PNP型;参见附加文件2:热图图S2)表明,对于分配给C5_PN/PNP或C6_PN/PNP,存在边界情况。
在1923个交叉的deg中,共发现22个编码R2R3-MYB tf的基因。根据PN和PNP表达的时间,22R2R3-MYB基因可分为三组(附加文件2:图S2)。第一组用VviMYB24该基因在开花早期(BBCH61)表达,但在开花到浆果形成的过渡阶段(BBCH71)已经关闭。VviMYB24是否可能与三个人的组合相对应拟南芥R2R3-MYB基因(AtMYB21/24/57)在花中表达,并在茉莉酸作用的背景下冗余地调节雄蕊发育[44].这很容易让人猜测VviMYB24在葡萄中也有类似的功能。
第二组约有15人R2R3-MYB在浆果形成和预变异期间表达的基因(BBCH71 - 77)。这一组包括VviMYBF1调节黄酮醇生物合成[45),而VviMYBPA5以及VviMYBPA7属于的支系AtTT2控制原花青素(PA,黄烷-3-醇)生物合成的相关基因[7,46,47,48].另一个R2R3-MYB这一组基因的特征不太清楚,尽管对其中一些基因的功能进行了描述。VviMYBC2-L3(VIT_214s0006g01620)作为苯丙素通路特异性分支的阻遏剂[49].
第三组R2R3-MYB基因从变异(BBCH81后)开始活跃,涵盖了大约6个基因。其中包括花青素积累控制基因,VviMYBA1,VviMYBA2, VviMYBA3而且VviMYBA8有充分证据表明它们能引发花青素的生物合成[50].由于PN和PNP是红莓的栽培品种,引导花青素积累的TF基因的活性是值得期待的。此外,该组还包括VviMYB14而且VviMYB15它们被认为是调节二苯乙烯生物合成途径的51].关于热图(附加文件2:图S2)和本研究中deg的分析,需要注意的是,虽然在发育程序和时间内的分辨率相当好,但我们的数据并不能解决基因表达的确切位置。因此,尚不确定检测到的表达是否来自于浆果皮、果肉、种子或其他组织/细胞。
推测成熟时间控制基因在浆果发育早期起作用
为了集中研究促进PNP中浆果形成加速和/或控制变异时间的基因,我们选择了在PNP变异之前的时间点检测到的deg(表2,无花果。4).这导致一组只有12个基因可能参与调控成熟时间(表3.).根据我们的假设,PNP中加速果实形成的相关基因,导致PNP早熟,应该至少在开花后不久起作用,我们将这组基因指定为“成熟时间控制”。然而,编码各自调控网络组成部分的基因和调控因子的靶基因(包括继发性影响的DEGs)肯定也包括在内[52].这12个假设的成熟时间控制基因,在PNP版本之前检测到的DEGs,编码与生长素作用相关的蛋白质,与细胞壁修饰相关的果胶加工酶,HD-Zip中的tf以及NF-Y/LEC家族,一种富含半胱氨酸的受体样蛋白激酶,一种油糖蛋白,以及功能结构域未知的蛋白质。
这组差异表达基因的表达模式,以及检测到的完整的DEGs数据集(表2),筛选出PNP中表达高于PN的基因。然而,尽管在较早的变异背景下,有许多基因在PNP中表现出比PN中更早的表达上调,但12个“早期差异”假定的成熟时间控制基因中,没有一个在PNP中明显高于开花时或开花后早期PN中的表达(表2)3.).此外,在其他DEGs中,未检测到具有这种差异表达模式的基因。与PN相比,PNP中的这种早期基因激活可能暗示有一种促进快速成熟的主导调控因子,但数据更倾向于快速成熟抑制因子的丢失。
VitisNet (vv30003)分配给生长素信号的两个基因编码扩展蛋白(VviEXPA5而且VviEXPA14, (53])。膨胀素通过细胞壁扩张和细胞增大参与果实成熟[54].生长素可以延缓葡萄变异和成熟过程的发生[19,20.,21].生长素信号通路中基因表达的减少可能表明生长素水平降低导致生长素作用降低,因此PNP进入转基因的加速可能是生长素水平降低引起的。此外,基因VviPL1(果胶解胶酶1 [55]),VviPME10(果胶甲基酯酶10,VIT_206s0009g02560)和VviGRIP28(编码果胶甲基酯酶抑制剂前体样蛋白)也与细胞壁过程有关,这表明细胞壁修饰也是一个重要的靶过程,也早于版本[54].的基因VviGRIP28在指定为ver/ph16.1的特定于veraison的元qtl中也检测到[9].这种关联是否有功能基础还有待确定。
编码tf的12个基因中的两个是VviHDZ28(VIT_216s0098g01170 [56])和VviLEC1(VIT_200s0956g00020 [57])。的诉酿酒用葡萄基因VIT_216s0098g01170VviHDZ28也被认为是AtHB12(At3g61890),但似乎VviHDZ07(VIT_202s0025g02590)和VviHDZ27(VIT_215s0048g02870)更类似AtHB12.在这些情况下,缺乏明确的可分配的同系物,功能信息的转移达到了其极限,可能被限制于下结论VviHDZ28对器官发育很重要葡萄属.的基因VviLEC1有三个基因中的一个吗诉酿酒用葡萄的同源词是什么AtLEC1(At1g21970, NF-YB9)AtL1L(LEC1-like, At1g21970, NF-YB6)。LEC1和L1L是胚胎和胚乳发育的中心调节因子。除其他过程外,它们控制胚胎形态发生和储存储备的积累[58].人们很容易猜测,探测到的原因VviLEC1在12个假定的成熟时间控制基因中,PNP的种子发育也需要比PN更快。这就解释了早期和较高的表达VviLEC1在PNP中与PN相比所观察到的(附加文件2:图S3G)。因此,VviOLE5(VIT_216s0013g00880,编码一种参与油体形成的油苷[59])与种子发育过程中的脂质储存有关。根据VIT_209s0018g01490编码的参与挥发性酯形成的蛋白质所提出的酶作为醇酰基转移酶的功能[60,这个基因可能起着类似的作用。VIT_205s0077g01980基因没有功能注释(未特征蛋白),尽管在木兰植物中存在同源性。
解释PN和PNP差异的因果基因候选
在12个假定的成熟时间控制基因中,有10个已经在上面讨论过,其中两个特别有趣。对整套deg的详细分析,如图所示。4,导致识别VviRTIC1而且VviRTIC2与PN相比,PNP可能与果实发育加快和成熟开始早有关。两种基因表达模式的特殊特征(图。5、表3.)是在第一个被选为BBCH第61期(盛花前开花,DAF为0(0))的分析时间点,两者已经存在差异表达。此外,这两种基因在研究的两年中仅在PNP中表达很少,而在PN中的表达几乎在所有时间点都很高。VviRTIC1被注释为编码含有未知功能结构域(DUF789)的蛋白质,而VviRTIC2被注释为编码“富含半胱氨酸的受体样蛋白激酶”(简称CRK)。最好的BLASTp击中答:芥蛋白序列显示其与At4g23180/AtCRK10相关,但进一步观察发现其与At4g05200/AtCRK25、At4g23160/AtCRK8和At4g23140/AtCRK6的相似性几乎一样高。这种模糊性,以及诉酿酒用葡萄基因组包含几个与之相关的基因VviRTIC2(例如VIT_210s0071g01200, VIT_202s0087g01020或VIT_203s0017g01550如PhyloGenes所列),使功能信息的传递复杂化。CRKs是植物受体激酶的一个亚群[61,由一个由44个基因组成的家族编码答:芥.在系统分析的功能答:芥CRKs,参与植物发育控制、生物和非生物胁迫响应、光合作用以及气孔调节的证据被收集到[62].对大量T-DNA插入突变体的系统表型筛选揭示了各种CRK基因的不同表型,但在未知配体识别和磷酸化信号传递之外,为单个CRK分配分子功能仍然是一个巨大的挑战。
正如上面所指出的,我们已经确定的基因很可能是控制浆果发育时间的遗传途径的一部分诉酿酒用葡萄但我们击中了该通路下游部分的基因。两个候选基因在PN和PNP之间的因果遗传差异中的相关性有待确定。品种的相分离基因组序列将需要解析两个等位基因的基因组结构VviRTIC1而且2PN和PNP基因的信息性比较。在未来的研究中,我们将解决这个问题,例如通过长读DNA测序。
结论
本研究共检测出PN和PNP品种间的1,923个DEGs。两个克隆品种在浆果发育时间上表现出表型差异,PNP比PN更快地从开花到变异。我们定义388个deg为变种特异性,12个deg为假定控制成熟时间。相对较少的变体特异性基因与发表的类似研究结果有很高的重叠(见附加文件)1表S14),可用于研究在PN和PNP中作用相似,但在PNP中加速的植物激素网络。此外,在这里确定的成熟时间控制基因可能提供了一组基因的访问,认为这对触发或延迟葡萄果实的成熟开始很重要。需要进一步的研究来阐明基因组的结构差异,所观察到的DEGs的功能,以及它们在改变葡萄成熟起始期中的作用。
材料与方法
植物材料及其克隆关系分析
葡萄藤(葡萄无性系种群。酿酒用葡萄L.)品种PNP (Pinot Precoce Noir, VIVC No. 9280)早熟,已被描述为与品种PN (Pinot Noir, VIVC No. 9279)有关[35比PNP成熟得晚。为了证明克隆关系,利用24个多态SSR标记(VVS2、VVMD7、VVMD5、VVMD32、VVMD28、VVMD27、VVMD25、VVMD24、VVMD21、VVIV67、VVIV37、VVIQ52、VVIP60、VVIP31、vvip73、VVIN16、VVIH54、VVIB01、VrZAG83、VrZAG79、VRZAG67、VrZAG62、VMC4F3.1、VMC1B11)对两个品种的DNA进行了基因分型[63].这两个品种经鉴定分别为DEU098_VIVC9280_Pinot_Precoce_Noir_DEU098-2008-076和DEU098_VIVC9279_Pinot_Noir_DEU098-2008-075。用于收割的组织如下图所示。1.这两个品种都不属于濒危物种,并根据德国立法获得和种植。
植物材料的表型表征和取样
植物材料来自在格架中培育的PN和PNP葡萄。这些植物生长在位于德国西贝尔丁根的JKI Geilweilerhof葡萄园(北纬49°21.747,东经8°04.678)。南北向种植,行距2.0 m,行距1.0 m。用于RNA提取的PNP和PN的花序、发育和成熟浆果样本在两年内收集,每个样本有三个独立的生物重复(子样本)。采样在系统时间点(2014年12个时间点,2017年13个时间点)和大约在。每天早上8点。2014年,每7天定期进行一次收割,只有两次例外(一天偏差,DAF13和DAF27)。2017年,采收适应了BBCH阶段(图5)。1A).这两年的时间线用开花期(DAF)后的天数来描述,开花期定义为10%的单花脱落花冠的那一天(BBCH61 [3.])。对于三个副本中的每个子样本,选择来自两个相邻葡萄的材料。根据BBCH分期对葡萄植株进行周表型分析[3.,4].反复进行表型分析,以确保从相同发育阶段(如开花期间开放的花朵百分比或浆果发育阶段)的葡萄中取样,以达到一致的子样本。表型观察总结在附加文件1:表S2、S3。从这些数据中,计算出开花、果实形成和果实成熟的持续时间,以及由此产生的品种间的变化1).此外,分别于2014年对35、41、49和56个DAF的果实发育阶段进行了拍摄。样品直接冷冻在液氮中,-70°C保存,直到RNA提取。
RNA提取及cDNA文库构建
生物重复,即子样品,分别在液氮下研磨。根据供应商的说明,使用RNA分离试剂盒(Sigma-Aldrich Spectrum™植物总RNA)提取总RNA。质量控制,RIN编号的确定[64]并在生物分析仪2100(安捷伦)上使用RNA Nano 6000芯片对所有RNA样本的浓度进行了估计。对于RNA- seq,每个子样品使用500 ng总RNA根据Illumina TruSeq RNA样品制备v2指南制备测序文库。对2014年和2017年的子样本分别构建了72和78个文库并进行了测序。使用连接在Illumina试剂盒中的磁珠上的poly-T寡核苷酸,对含有mRNA的poly-A进行了两次富集。在第二次洗脱聚a + RNA的过程中,RNA被碎片化并启动cDNA合成。cDNA合成完成后,对片段进行末端修复并进行a尾尾处理。将多个索引适配器连接到cDNA片段的末端,经过10次PCR循环对连接的适配器片段进行富集。在使用生物分析仪2100 hs - chip(安捷伦)进行质量检查和在FLUOstar Optima Plate Reader (BMG LABTECH)上使用定量it PicoGreen dsDNA测定进行精确定量后,等量地将文库池。
RNA-Seq
用Illumina HiSeq1500在高输出模式下对2014年的合并条形码库进行单端(SE)测序,产生100 nt reads。对于2017年的样本,使用Illumina NextSeq500测序,产生83 nt SE reads;使用2017年的相同条形码库池进行了两次运行。
处理RNA-Seq读取数据
使用Trimmomatic(0.36版本)修剪原始读取[65].对于2014年的原始读数,使用了以下设置:LEADING:10 trails:10 SLIDINGWINDOW:4:15 MINLEN:50。此外,还提供了所有可用的Illumina适配器序列的集合,用于删除参数2:30:10内的匹配。对于2017年的原始读数,修剪设置设置为LEADING:6 trails:6滑动窗口:4:15 MINLEN:36。通过FastQC(0.11.8版本)对所有裁剪的读取进行质量检查[66].因此,可能的适配器序列和低质量的碱基被删除。所有通过QC的裁剪reads被映射到参考基因组序列PN40024(版本12Xv2) [67]使用基于图形的对齐工具HISAT2(2.1.0版本)[68,69没有额外的软裁剪。然后,所有标记的基因(结构基因注释:CRIBI v2.1)使用featucounts (Bioconductor软件包Rsubread版本3.8 [70])。为了估计作为基因表达度量的转录本丰度,计算每千碱基百万转录本(TPM, [71)的决定。
基础基因表达分析
来自不同样本的TPM计数用于手工基因表达检查,以确定表达和未表达基因的数量,并计算两年基因表达值之间的相关性。一年内所有样本中TPM值为>的基因均为表达基因,反之,TPM值为0的基因均为未表达基因。利用SciPy python包(v. 1.2.3)中的函数pearsonr应用了一个自定义python脚本[72],计算所有年度比较的皮尔逊相关系数和p值。使用了两个采样年份每个基因TPM计数的表达数据对,每个样本的三个子样本的平均值。为了测试来自两年的表达值之间的相关性和关系,其中采样模式略有不同的样本(见图。1A),在每年的品种之间选择八个具有相同BBCH阶段的等效时间点(见附加文件)1:表S4)。
主成分分析
为了探索数据的相似性,主成分分析(PCA)计算了所有子样本的所有基因表达值,从年份和品种。使用R包DESeq2 (v. 1.12.4)中的方差稳定转换函数'vst'对所有数据点进行归一化处理[73].随后,使用R包stats中的'prcomp'生成主组件(第3.5.2节)[74].生成的PCA对象显示了主要成分PC1和PC2,绘制并导出。另外,分别提取对PC1和PC2方差贡献最大的基因。
基因的功能注释
主要是从其他植物物种的注释信息转移答:芥,使用MapMan的序列注释工具Mercator (v. 3.6)计算[75,76].此外,所有开放的阅读框架(cd从诉酿酒用葡萄/葡萄藤基因)序列与非冗余蛋白序列数据库RefSeq [77]与基本的蛋白质局部对齐工具BLASTp [78) (e-value≤0.001)。提取基因功能的简短描述并添加到附加文件中的基因列表中1:表S5、S6、S13、S14。
通过R包“topGO”计算生物过程的氧化石墨烯术语富集(第2.38.1节)[79].随后,采用fisher精确检验计算统计信度。所有基因id及其对应的GO术语从CRIBI数据库中提取(http://genomes.cribi.unipd.it/DATA/V2/annotation/bl2go.annot_with_GO_description.txt).GO术语浓缩的所有结果都保存在附加文件中1:表S10-12。
聚类分析
为了揭示所有四个数据集中的共表达基因,使用工具CLUST (v. 1.10.8)和默认参数[80].作为输入,使用了来自8个时间点的原始读取计数。选择这些时间点是为了覆盖2014年和2017年PN和PNP的相同BBCH阶段(附加文件1:表S4)。首先,按照CLUST手册中的描述对所有数据进行预处理。将对应的子样本(三个重复)的值合并并取平均值。为了过滤掉信息不丰富(非常低)的基因表达值,我们使用了一个额外的过滤器:在至少三种条件下,在一年的至少一个品种中没有达到样本表达值> 1的基因被丢弃(-fil-v 1 -fil-c 3 -fil-d 1)。然后,根据CLUST的RNA-Seq默认值对数据进行分位归一化。采用默认设置过滤掉表达谱平坦的基因[80].
差异基因表达分析
对于差异表达基因的分析,DESeq2 (v. 1.12.4;采用R Bioconductor)。为了测试两个样本之间的基因表达是否有显著差异,使用了DESeq2包中包含的似然比检验nbinomLRT。使用来自所有时间点的原始读取计数作为输入。标准化因子和离散度估计的使用如所述[73].输出表中包含了所有差异表达基因(DEGs)和baseMean、log2FoldChange (LFC)、lfcSE (LFC标准误差)、stat(简化模型与完整模型的偏差差)、p值和PADJ(调整后的p值)的对应值。为了关注DESeq2分析中显著差异表达的基因,使用截止过滤器PADJ≤0.05和LFC > 2。
qRT-PCR证实差异基因表达
为了验证RNA-Seq结果,选择2017年的4个时间点(DAF 0(0)、DAF28、DAF57和DAF77)进行qRT-PCR。用First Strand cDNA Synthesis Kit (ProtoScript®II;NEB)根据制造商的说明。qRT-PCR检测采用Luna Universal qPCR Master Mix (NEB),总体积为20µl。所使用引物的序列列在附加文件中1:表S23。反应产物/扩增物采用CFX96实时PCR检测系统(Bio-Rad)检测SYBR绿。对于每个时间点,测量三个生物重复和三个技术重复(即三个重复中的每个子样品)。初始变性后的循环条件:95°C变性5秒,60°C退火/延伸30秒,循环35次。对于QC,每个反应都通过产品熔体分析控制(65°C - 95°C)。作为阴性对照,无模板对照(NTC)和无逆转录酶对照(-RT)也采用3个技术重复进行测定。泛素基因VviUbiquitin1(VIT_216s0098g01190)进行归一化[46].使用CFX Maestro V.4.1.2433.1219 (Bio-Rad)通过相对定量ΔΔCt方法归一化分析测量结果。
选择可能与成熟时间相关的基因集并与文献数据进行比较
为了从与控制和实现成熟相关的差异基因中识别基因集,我们建立了在两年间所有时间点检测到的差异基因的交集。为了确定假定的成熟时间控制基因的子集,使用了覆盖发育阶段BBCH61(开花开始)到BBCH79(变异前的一个发育阶段BBCH)的两个年份之间的交集(时间点2014:DAF 0-35;2017年:德国世界杯0-42)。此外,从发育阶段BBCH79(变异前的一个发育BBCH阶段)到BBCH81(成熟/变异开始;时间点2014:DAF35-41;2017年:DAF42-49)。为了检验亚群的生物学相关性,对所有的deg进行了筛选,使其在类似的相关研究中出现[9,27,28,30.,34,41,81,82].
新出现的基因数量的可视化差异表达
为了使deg随时间的外观可视化,使用R包' plotly '建立了一个堆叠条形图脚本(4.9.2.1节)[83].每个条形表示给定时间点或条件的deg的数量。为了在以后的时间点中跟踪在给定时间点新出现的deg组,表示这组deg的色度保持不变。
途径富集分析
为了在已知的葡萄藤路径中寻找可能的靶点,使用VitisPathways工具进行路径富集分析[84]被执行。为了达到可靠的富集,设置了1000个排列,Fisher精确检验p < 0.05和排列p值< 0.1。因此,所有重要的富集途径基因及其关系都可以在VitisNet中显示[85,这是葡萄的一种特殊分子网络。
的热图
作为基因分配到生物合成途径的延伸,基因也被过滤注释为转录因子(tf)的编码。该过滤器基于从Mercator和RefSeq传输的注释信息(见上文)。看看整个家庭R2R3-MYBTF基因,列表MYB通过MapMan识别的基因通过额外的葡萄藤进行了扩展R2R3-MYB基因家族成员[6,7].的R2R3-MYB利用R包'pheatmap' (v. 2.1.3)在四个个体时间序列(2个品种,2年)的热图中显示交叉deg中检测到的基因[86].对系统发育关系的预测来自于PhyloGenes v. 2.2 [87].
数据和材料的可用性
包含所有RNA-Seq读取的FASTQ文件(PN2014, PNP2014, PN2017和PNP2017)已根据INTEGRAPE指南存放在欧洲核苷酸档案馆(ENA),注册号分别为PRJEB39262, PRJEB39261, PRJEB39264和PRJEB39263(见附加文件)1:表S19至S22)。为本研究开发的所有脚本都可在GitHub上获得[https://github.com/bpucker;https://github.com/jenthein].
缩写
- 阿坝:
-
脱落酸
- CRK:
-
富含半胱氨酸的受体样蛋白激酶
- DAF):
-
开花后的几天
- 度:
-
差异表达基因
- PNP型:
-
黑皮诺早熟
- PN:
-
黑皮诺
- 利物浦:
-
Log2fold变化
- PADJ:
-
假定值调整
- 主成分分析:
-
主成分分析
- QTL:
-
数量性状位点
- TF:
-
转录因子
- TPM:
-
转录本每千基百万
参考文献
- 1.
库恩N,关兰,戴志伟,吴BH,劳维盖特V, Gomès E,李士生,戈多伊F,阿塞-约翰逊P,德尔罗特S.浆果成熟:最近通过小道消息听说。中国生物医学工程学报2014;65(16):4543-59。
- 2.
狭谷BG。葡萄果实发育与成熟的研究。中华医学杂志。1992;43(1):101-10。
- 3.
Lorenz DH、Eichhorn KW、Bleiholder H、Klose R、Meier U、Weber E.葡萄的物候生长阶段。-根据扩展的BBCH量表的编码和描述。德国葡萄酒杂志1995;1(2):100-3。
- 4.
Bleiholder H, Weber E, Feller C, Hess M, Wicke H, Meier U, van den Boom T, Lancashire PD, Buhr L, Hack H等人。单子叶和双子叶植物的生长阶段。BBCH专著:联邦农林生物研究中心;2001.
- 5.
拟南芥中MYB转录因子的研究。植物学报,2010;15(10):573-81。
- 6.
对葡萄MYB R2R3亚家族的分析揭示了葡萄和拟南芥基因组中与葡萄酒品质相关的扩展分支和保守的基因结构组织。BMC Plant Biol. 2008;8:83。
- 7.
王dcj, Schlechter R, Vannozzi A, Holl J, Hmmam I, Bogs J, Tornielli GB, Castellarin SD, Matus JT。对葡萄R2R3-MYB转录因子家族的系统导向分析揭示了对二苯乙烯积累调控的新见解。DNA Res. 2016;23(5): 451-66。
- 8.
Zyprian E, Ochßner I, Schwander F, Šimon S, Hausmann L, bonowo - rex M, Moreno-Sanz P, Grando MS, Wiedemann-Merdinoglu S, Merdinoglu D,等。影响葡萄抗病和成熟的数量性状位点。中国生物医学工程学报,2016;29(4):359 - 359。
- 9.
Delfino P, Zenoni S, Imanifard Z, Tornielli GB, Bellin d通过整合元qtl和转录组数据选择葡萄变异时间的候选基因。中国生物医学工程学报2019;20(1):739。
- 10.
Delrot S, Grimplet J, Carbonell-Bejerano P, Schwandner A, Bert P- f, Bavaresco L, Costa LD, Di Gaspero G, Duchêne E, Hausmann L,等。葡萄适应气候变化的遗传和基因组方法。正确的做法:编辑科尔·C。,et al。,Genomic designing of climate-smart fruit crops. Cham: Springer; 2020. p. 157–270.
- 11.
Fenn MA, Giovannoni JJ。植物激素在果实发育和成熟中的作用。植物J. 2020。
- 12.
杨文华,杨文华。果实成熟的分子调控。植物科学,2013;4:198。
- 13.
孙亮,张敏,任军,齐军,张刚,冷平。果实成熟前后脱落酸与乙烯的相互作用。植物生物学杂志2010;10:257。
- 14.
Pilati S, Bagagli G, Sonego P, Moretto M, Brazzale D, Castorina G, Simoni L, Tonelli C, Guella G, Engelen K,等。脱落酸是葡萄果实成熟启动的主要调节因子:ABA信号网络的新认识。植物科学进展2017;8:1093。
- 15.
李朱Z, G,燕C,刘L,张问,汉族Z,李b . DRL1编码NAC转录因子,参与在葡萄藤叶衰老。国际分子科学杂志2019;20(11):2678。
- 16.
Rossdeutsch L、Edwards E、Cookson SJ、Barrieu F、Gambetta GA、Delrot S、Ollat N. aba介导的水分亏缺反应将葡萄基因型根据其遗传背景进行分离。BMC Plant biology, 2016;16(91): s12870-12016-10778-2874。
- 17.
科埃略J,阿尔梅达-特拉普M,皮门特尔D,苏亚雷斯F,赖斯P,雷戈C, Mithöfer A,福特斯AM。通过对Trincadeira和Syrah品种激素代谢的研究,揭示了水杨酸、茉莉酸、ABA和IAA在葡萄成熟过程和灰葡萄孢感染过程中的新作用。植物科学2019;283:266-77。
- 18.
Chervin C, El-Kereamy A, Roustan J- p, Latché A, Lamon J, Bouzayen M.乙烯似乎是葡萄果实发育和成熟的必要条件。植物学报(英文版);2006;
- 19.
Böttcher C, Burbidge CA, Boss PK, Davies C.乙烯与生长素的相互作用对葡萄(Vitis vinifera L.)果实成熟的控制至关重要。植物生物学,2013;13:222。
- 20.
Böttcher C, Harvey K, Forde CG, Boss PK, Davies C.预版本葡萄(Vitis vinifera L.)浆果的生长素处理既延迟了成熟,又增加了糖积累的同步性。2011;17(1):1 - 8。
- 21.
Ziliotto F, Corso M, Rizzini FM, Rasori A, Botton A, Bonghi C. pre-véraison NAA处理诱导的葡萄果实成熟延迟与生长素和乙烯相关基因表达模式的改变平行。植物生物学杂志,2012;12:185。
- 22.
西摩GB, Ostergaard L,查普曼NH, Knapp S,马丁C.果实发育和成熟。植物学报,2013;64:219-41。
- 23.
乔凡诺尼,阮超,李晓燕,钟松,费卓。果实成熟的表观基因组与转录动力学。植物生物学杂志2017;68:61-84。
- 24.
Karlova R, Chapman N, David K, Angenent GC, Seymour GB, de Maagd RA。肉质果实发育和成熟的转录控制。中国生物医学工程学报2014;65(16):4527-41。
- 25.
Wang R, Tavano E, Lammers M, Martinelli AP, Angenent GC, de Maagd RA。用CRISPR/ cas9诱变技术重新评价番茄果实发育和成熟过程中转录因子的作用。科学通报2019;9(1):1696。
- 26.
Serrano A, Espinoza C, Armijo G, Inostroza-Blancheteau C, Poblete E, meer - regueiro C, Arce A, Parada F, Santibanez C, Arce- johnson P.用于理解葡萄浆果发育的组学方法:与内源性过程和环境响应相关的调控网络。植物科学进展2017;8:1486。
- 27.
马强,杨军。葡萄果实发育和成熟相关功能基因的转录组分析与鉴定。基因。2019;680:84 - 96。
- 28.
法索里M,里克特CL,泽诺尼S,贝尔蒂尼E,维图洛N, Dal Santo S,杜库兹连N,佩佐蒂M,托尼利GB。标志葡萄果实成熟开始的分子事件的时间和顺序。植物生理学报,2018;
- 29.
孙锐,何峰,兰艳,邢锐,刘锐,潘强,王杰,段晨。不同气候地区赤霞珠葡萄浆果的转录组比较。植物生理学报,2015;48(3):447 - 447。
- 30.
Zenoni S, Ferrarini A, Giacomelli E, Xumerle L, Fasoli M, Malerba G, Bellin D, Pezzotti M, Delledonne M.葡萄果实发育过程中转录复杂性的RNA-Seq分析。植物生理学报,2010;32(4):369 - 369。
- 31.
褪黑激素通过增加ABA、H2O2、特别是乙烯的含量促进葡萄果实成熟。Hortic Res. 2018;5:41。
- 32.
Griesser M, Martinez SC, Eitle MW, Warth B, Andre CM, Schuhmacher R, Forneck A.成熟失调浆果枯萎影响Zweigelt葡萄果实花青素的生物合成和糖代谢。足底。2018;247(2):471 - 81。
- 33.
Zarrouk O, Brunetti C, Egipto R, Pinheiro C, Genebra T, Gori A, Lopes CM, Tattini M, Chaves MM.葡萄成熟受灌水不足/温度升高的调节,根据葡萄在冠层中的位置。植物科学进展2016;7:1640。
- 34.
红、白葡萄品种的成熟转录组程序与果实皮花青素积累相关。植物生理学报,2017;32(4):473 - 476。
- 35.
Maul E, Sudharma KN, Ganesch A, Brühl U, Hundemer M, Kecke S, Mahler-Ries A, Marx G, Schreiber T, Walk M等。30年VIVC -葡萄国际品种目录(www.vivc.de).参加:第十一届国际葡萄育种与遗传学会议;延庆,北京,中国,2014。
- 36.
Robinson J, Harding J, Vouillamoz J.葡萄酒葡萄:1368个葡萄品种的完整指南,包括其起源和口味。伦敦:企鹅书店;2012.
- 37.
罗伊- chevrier J. ampelograph回顾展。巴黎,1900年。
- 38.
Pucker B, Schwandner A, Becker S, Hausmann L, Viehover P, Topfer R, Weisshaar B, Holtgrawe D.葡萄芽发育的RNA-Seq时间序列揭示了表达模式与当地温度分布的相关性。植物。2020;9(11):1548。
- 39.
Dal Santo S, Tornielli GB, Zenoni S, Fasoli M, Farina L, Anesi A, Guzzo F, Delledonne M, Pezzotti M.葡萄果实转录组的可塑性基因组生物学杂志,2013;14(6):R54。
- 40.
Vannozzi A, Dry IB, Fasoli M, Zenoni S, Lucchin M.葡萄二苯乙烯合成酶多基因家族的全基因组分析:生物和非生物胁迫下的基因组组织和表达谱。BMC Plant Biol 2012; 12:30 30。
- 41.
Palumbo MC, Zenoni S, Fasoli M, Massonnet M, Farina L, Castiglione F, Pezzotti M, Paci P.综合网络分析确定战斗俱乐部节点是一类集线器,包含了在葡萄生长过程中诱导主要转录组重编程的关键假定开关基因。植物细胞学报,2014;26(12):4617-35。
- 42.
Burger AL, Zwiegelaar JP, Botha FC。编码梅洛成熟诱导蛋白1 (mrip1)的基因特征:证据表明这种假定的蛋白质是植物富含脯氨酸蛋白家族的一个独特成员。植物科学学报2004;37(5):369 - 369。
- 43.
戴维斯C,罗宾逊SP.差异筛选表明在葡萄果实成熟过程中mRNA谱发生了显著变化。编码细胞壁和应激反应蛋白的cdna的克隆和鉴定。植物生理学报,2000;22(3):366 - 366。
- 44.
齐涛,黄宏,宋松,谢东。bHLH-MYB复合体对拟南芥茉莉素介导的雄蕊发育和种子产生的调控。《植物细胞》2015;27(6):1620-33。
- 45.
Czemmel S、strackr、Weisshaar B、Cordon N、Harris NN、Walker AR、Robinson SP、Bogs J.葡萄R2R3-MYB转录因子VvMYBF1调控葡萄果实发育过程中黄酮醇的合成。植物生理学报,2009;29(3):369 - 369。
- 46.
Bogs J, Downey MO, Harvey JS, Ashton AR, Tanner GJ, Robinson SP.葡萄浆果和葡萄叶发育过程中原花青素的合成及白花青素还原酶和花青素还原酶编码基因的表达。植物学报,2005;39(2):652-63。
- 47.
Bogs J, Jaffe FW, Takos AM, Walker AR, Robinson SP.葡萄转录因子VvMYBPA1在果实发育过程中调节原花青素合成。植物物理学报,2007;43(3):347 - 361。
- 48.
山山强,新田芳,中岛1,后藤山本N,松村H,田中N。葡萄MYB转录因子的功能特征及其对葡萄原花青素生物合成的调控。中国生物医学工程学报(英文版);2014;
- 49.
卡瓦里尼E,马图斯JT,费内佐L,泽诺尼S,洛约拉R,古佐F,施莱克特R,阿尔吉奥斯A,阿斯-约翰逊P,托尔尼利GB。在葡萄中,苯丙类途径由一组R2R3-MYB C2阻遏子控制在不同的分支。植物生理学报,2015;37(4):449 - 457。
- 50.
Matus JT, Cavallini E, Loyola R, Holl J, Finezzo L, Dal Santo S, Vialet S, Commisso M, Roman F, Schubert A,等。位于14号染色体上的一组葡萄MYBA转录因子控制营养器官花青素合成,与浆果颜色位点相比具有不同的特异性。植物学报(英文版);2017;
- 51.
Vannozzi A, Wong DCJ, Holl J, hmmmam I, Matus JT, Bogs J, Ziegler T, Dry I, Barcaccia G, Lucchin M. WRKY和MYB转录因子对葡萄二苯乙烯合成酶基因的组合调控。植物细胞学报,2018;59(5):1043-59。
- 52.
杨志强,王晓燕,王晓燕。果实成熟:激素、细胞壁修饰的作用及其与病原体的关系。中国科学(d辑:自然科学版);
- 53.
Dal Santo S, Vannozzi A, Tornielli GB, Fasoli M, Venturini L, Pezzotti M, Zenoni S。对扩展蛋白基因超家族的全基因组分析揭示了葡萄特有的结构和功能特征。科学通报,2013;8(4):e62206。
- 54.
杜·M,斯伯丁EP,格雷WM。生长素介导的细胞快速膨胀。植物学报2020;71:379-402。
- 55.
Guillaumie S, Fouquet R, Kappel C, Camps C, Terrier N, Moncomble D, Dunlevy JD, Davies C, Boss PK, Delrot S葡萄果实晚成熟期转录分析。植物生物学,2011;11:165。
- 56.
李智,张超,郭颖,牛伟,王颖,徐颖。HD-Zip基因家族在葡萄(Vitis vinifera L.)胚胎流产调控中的进化和表达分析。BMC基因组学,2017;18(1):744。
- 57.
崔敏,王伟,郭芳,范旭,关磊,郑涛,朱旭,贾宏,方杰,王超,等。赤霉素诱导葡萄无籽果实发育过程中LEC1基因的表达及时空分析。植物生长学报,2020;90(3):585-96。
- 58.
Jo L, Pelletier JM,原田JJ。叶子叶1转录因子在种子发育中的中心作用。植物生物学杂志,2019;61(5):564-80。
- 59.
刘强,孙勇,苏伟,杨军,刘旭,王赟,王芳,李红,李欣。被子植物中油脂苷的种特异性大小扩展与分子演化。基因。2012;509(2):247 - 57。
- 60.
钱旭,刘勇,张刚,闫安,王红,王鑫,潘强,徐辉,孙亮,朱斌。醇酰基转移酶基因和酯前体在3个葡萄种间杂种挥发性酯组分中的差异。浆果发育期的Vinifera。食品化学2019;295:234-46。
- 61.
梁旭,周建明。受体样细胞质激酶:植物受体激酶介导信号转导的中心角色。植物生物学,2018;69:267-99。
- 62.
Bourdais G, Burdiak P, Gauthier A, Nitsch L, Salojarvi J, Rayapuram C, Idanheimo N, Hunter K, Kimura S, Merilo E,等。大规模表型学确定了CRKs在与氧化应激相关的反应中的主要和微调作用。科学通报,2015;11(7):e1005373。
- 63.
Dallakyan M, Zyprian E, Maul E, Yesayan A, Hovhannisyan N.亚美尼亚油菜品种的遗传特征。葡萄。2015;54:23-6。
- 64.
Schroeder A, Mueller O, Stocker S, Salowsky R, Leiber M, Gassmann M, Lightfoot S, Menzel W, Granzow M, Ragg t。RIN:为RNA测量分配完整性值的RNA完整性编号。分子生物学。2006;7:3。
- 65.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活微调器。生物信息学。2014;30(15):2114 - 20。
- 66.
FastQC,用于高通量序列数据的质量控制工具。[http://www.bioinformatics.babraham.ac.uk/projects/fastqc/].已于2018年11月16日访问。
- 67.
Jaillon O, Aury JM, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C等。葡萄基因组序列提示主要被子植物门的祖先六倍化。大自然。2007;449(7161):463 - 7。
- 68.
Kim D, Langmead B, Salzberg SL. HISAT:一种低内存需求的快速拼接对准器。Nat方法。2015;12(4):357-60。
- 69.
Kim D, Paggi JM, Park C, Bennett C, Salzberg SL.基于图谱的HISAT2和hisat基因型基因组对齐和基因分型。中国生物技术学报2019;37(8):907-15。
- 70.
廖颖,Smyth GK, Shi W. featucounts:一个高效的通用程序分配序列读取基因组特征。生物信息学。2014;30(7):923 - 30。
- 71.
赵松,叶铮,Stanton R.在比对样品和测序协议时RPKM或TPM规范化的误用。RNA。2020年,26(8):903 - 9。
- 72.
Virtanen P, Gommers R, Oliphant TE, Haberland M, Reddy T, Cournapeau D, Burovski E, Peterson P, Weckesser W, Bright J,等。SciPy 1.0: Python科学计算的基本算法。Nat方法。2020;17(3):261-72。
- 73.
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和色散估计。基因组生物学杂志,2014;15(12):550。
- 74.
R:统计计算语言和环境[https://www.r-project.org].已于2018年11月29日访问。
- 75.
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M. MAPMAN:一个用户驱动的工具,将基因组数据集显示在代谢途径和其他生物过程的图表上。植物学报。2004;37(6):914 - 919。
- 76.
Lohse M, Nagel A, Herter T, May P, Schroda M, Zrenner R, Tohge T, Fernie AR, Stitt M, Usadel B.用于植物序列数据基因组级功能注释的快速、简单的web服务器。植物细胞环境学报,2014;37(5):1250-8。
- 77.
NCBI参考序列(RefSeq):基因组、转录本和蛋白质的非冗余序列数据库。核酸Res. 2007;35(数据库问题):D61-65。
- 78.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ。基本的本地对齐搜索工具。中华分子生物学杂志1990;215(3):403-10。
- 79.
Alexa A, Rahnenfuhrer J, Lengauer T.通过去关联GO图结构从基因表达数据中改进官能团评分。生物信息学。2006;22(13):1600 - 7。
- 80.
Abu-Jamous B, Kelly S. Clust:从基因表达数据中自动提取最佳共表达基因簇。《基因组生物学》2018;19:172。
- 81.
Zenoni S, D 'Incà E, Tornielli GB。葡萄果实成熟控制的遗传解剖:确定NAC转录因子的作用。学报2019;1248:387-402。
- 82.
魏亮,曹勇,程杰,向杰,沈斌,吴杰。对鲜食葡萄“夏黑”及其早熟突变体“天宫墨玉”的转录组比较分析,发现了可能参与果实发育和成熟的候选基因。植物学报,2020;15(1):213-22。
- 83.
Sievert C.用R进行交互式的基于web的数据可视化,绘图化和闪亮。博卡拉顿:CRC出版社;2020.
- 84.
柳条MV。葡萄途径:葡萄属植物的基因途径分析。葡萄。2016;55:129-33。
- 85.
格里姆特J,范·赫默特J,卡伯内尔-贝杰拉诺P, Díaz-Riquelme J,迪克森J,芬内尔A,佩佐蒂M, Martínez-Zapater JM。葡萄全基因组基因预测、功能注释、分类和预测基因序列整合的比较分析。BMC研究笔记,2012;5:213。
- 86.
pheatmap:漂亮的热图。R包版本1.0.12 [https://CRAN.r-project.org/package=pheatmap].已于2019年12月16日访问。
- 87.
张鹏,Berardini TZ, Ebert D, Li Q, Mi H, Muruganujan A, Prithvi T, Reiser L, Sawant S, Thomas PD,等。系统基因:一个用于植物基因功能推断的在线系统发育和功能基因组学资源。《植物指南》2020;4(12):e00293。
确认
我们要感谢Boas Pucker提供了处理大型数据集的几个python脚本以及对手稿的有益评论,Katharina Frey-Sielemann帮助可视化DEG分布,Wiebke Halpape提供了GO注释查询。我们还要感谢Kyle J. Lauersen为我们提供了非常有价值的语言编辑,以及Willy Keller为我们提供了出色的技术支持。我们非常感谢CeBiTec的生物信息学资源设施支持团队提供的计算基础设施和优秀的技术支持。本出版物部分基于COST Action CA 17111 INTEGRAPE的贡献,由COST(欧洲科学技术合作)支持;作者感谢BMBF/PtJ在IPAS(项目首字母缩写novisys)背景下的资助。UniBi的作者也要感谢植物遗传学和基因组学主席的其他成员的支持。我们还要感谢比勒费尔德大学的开放获取出版基金提供的出版费用。
资金
由Projekt DEAL支持和组织的开放获取资金。该项目由BMBF/PtJ (FKZ 031A3496)资助。
作者信息
从属关系
贡献
在JKI (Julius Kühn-Institute, Geilweilerhof, Siebeldingen, Germany)进行采样和表型分析。测序和数据分析在比勒费尔德大学生物学院和生物技术中心(CeBiTec)进行。比勒费尔德大学的组织、监督和部分抽样工作由卫生部完成。KH, AK和FS协调每周采样,表型,并为文档捕获图像。后勤工作由KH负责。实验设计由FS、DH和KH三部分组成。采用DH和PV法进行RNA分离和cDNA合成。PV完成了文库的制备和测序。RT-PCR用JT和DH进行定量pcr。RT和KH监督了JKI Geilweilerhof的工作。 LH performed the SSR analysis. BW supervised the work at Bielefeld University. FS, KH, RT and BW acquired project funding and wrote the project proposal. All bioinformatic data analyses, creation of figures, tables and writing of the manuscript were performed by JT with the help of DH. JT and BW drafted the manuscript. All authors have read and approved the final manuscript.
相应的作者
道德声明
伦理批准和同意参与
不适用
发表同意书
不适用
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:表S1。
利用24个SSR标记证实了PN和PNP的克隆关系。表S2。2014年抽样和表型观察(BBCH)。表S3。2017年采样和表型观察(BBCH)。表S4。两年间基因表达值随时间变化的pearson相关性。表S5。影响PC1主成分的前500个基因。表S6。影响PC2主成分的前500个基因。表S7。从聚类分析PN/PNP中获得的集群的集群成员关系。表S8。从聚类分析PN中获得的簇的簇成员关系。表S9。从聚类分析PNP得到的聚类的聚类成员关系。表S10。PN/PNP聚类分析中聚类的GO术语富集。表S11。PN聚类分析中聚类的GO项富集。表S12。PNP聚类分析中聚类的GO项富集。表向。PN和PNP在年份和过滤步骤中差异表达的基因。表S14系列。12个假定的成熟时间对照deg(时间点:2014年的DAF 0-35相交于2017年的DAF 0-42)和388个版本特异性deg(时间点:2014年的DAF35+41相交于2017年的DAF42+49)与之前的转录组葡萄研究重叠。表S15。交叉deg的VitisNet富集。表S16。VitisNet富集的调控deg。表肌力。VitisNet富集veraison特异性deg。S18表美国。deg中转录因子的上调和下调。表S19。与2014年PNP RNA-Seq reads研究相关的ENA样本标识符和元数据(PRJEB39262)。S20表。与2014年PN RNA-Seq reads研究相关的ENA样本标识符和元数据(PRJEB39261)。表S21。ENA样本标识符和与2017年PNP RNA-Seq读取研究相关的元数据(PRJEB39264)。S22表。ENA样本标识符和与2017年PN RNA-Seq读取研究相关的元数据(PRJEB39263)。表S23。qRT-PCR引物序列。
附加文件2:图S1。
两年度PN/PNP的聚类分析(A)、两年度PN (B)和两年度PNP (C)。图S2。两年间PN和PNP的DEGs中所有myb - tf基因表达的热图。图S3。来自成熟调控基因集的基因表达模式。图S4。用qRT-PCR方法确认RNA-Seq的表达数据
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
泰因,Holtgräwe, D.赫尔佐格,K。et al。对皮诺家族两个葡萄品种之间浆果发育的时间变化的转录组学分析揭示了潜在的控制成熟时间的基因。植物生物学21日,327(2021)。https://doi.org/10.1186/s12870-021-03110-6
收到了:
接受:
发表:
关键字
- 葡萄
- 黑皮诺
- 黑皮诺早熟
- 小道消息
- 浆果成熟
- 水果发展
- 差异基因表达
- 转录组分析
- 成熟时间控制
- Veraison