抽象的
背景
气候危机威胁着全世界农作物生产的可持续性。作物多样化可能会提高粮食安全,同时降低气候变化的负面影响。Proso millet(Panicum milaceum.L.)是一种次要谷物作物,其占多样化和适应不同环境条件的潜力。在这项研究中,我们组装了由88个品种和体力成分组成的Proso Mill系列,以研究其种子特征的基因组和表型多样性,并鉴定标记性关联(MTA)。
结果
限制性位点相关的DNA片段的测序产生了494万读数和2,412个高质量的单核苷酸多态性(SNP)。SNP用于研究收集中的多样性,并进行基因组宽协会研究(GWAS)。基因型多样性分析分离源自西欧,东亚和美洲的南亚,亚洲,西亚,西亚和非洲的加入。贝叶斯结构分析报告了四个神秘的遗传群,表明车站脱俗的含量较为巨大的混合物,并且大多数改进的Proso Mill材料与地形分开聚集。该系列对种子特征高度多样化,颜色从白色变化到深棕色和宽度为1.8至2.6毫米。用于种子形态特性的GWAS研究确定了10微米。此外,我们确定了三个MTA用于以前在收集上测量的农艺性状。
结论
使用基因组学和自动化种子表型,我们阐明了全球小米集合中的系统发育关系和种子多样性。总体而言,我们确定了13个MTA,用于关键农艺和种子特征,表明存在具有在ProSo育种计划中申请潜力的等位基因的存在。
介绍
小米是最早的经济上重要的驯养作物中[1那2]仍然是半干旱热带的重要主食,特别是针对种植主要食物作物所需的投入有限的小农农民[3.].术语“小米”是基于其生产和使用的广泛定义;然而,它们是具有不同起源和分类物种的异种物种。由于传统农业的产生潜力有限,小米已经收到了吐温的育种关注[4.]. 然而,谷子是最有前途的被忽视和未充分利用的作物之一,如果适当的价格化,可能会改善粮食安全和营养质量[5.那6.].到目前为止,研究努力主要集中在珍珠米上(Pennisetum blaucum.)和狐尾米勒(Setaria Italica.)二者均建立了国际半干旱热带作物研究所(ICRISAT)的种质资源和完整的基因组序列[7.那8.].其他小米物种具有有限的资源,直到最近被现代研究方法忽视。
Proso millet(Panicum Miliaceum.是二倍体(2n) = 36)一年生草本植物,生长于欧亚大陆、大洋洲、北美,在非洲更为罕见。它对不同环境条件有很好的适应性,对降雨量要求低,主要在干旱气候条件下种植[9.],并且具有大约12周的短候循环,使其成为多重旋转的良好资源[10].在许多农产病学中,Proso Mill主要用作牲畜饲料,但潜在作为乙醇的来源,作为食物谷物[11].实际上,Proso Millet面粉富含蛋白质,维生素,矿物质和微量营养素,包括铁,锌,铜和锰[12].它的谷物比小麦富含必需氨基酸[13]. 然而,尽管proso对健康有益,营养成分宝贵,但将其融入食品工业所需的知识仍有很大差距[14].
谷子在形态特征上表现出高度的变异性[15那16].早期研究将Proso Millet Germplasm分为五场比赛,基于穗形态:米纳瓜那patentissimum那挛缩那紧统,和ovatum.[17]. 最近的研究基于形态农艺性状评估了谷子的多样性,显示出很高的育种潜力[9.]和对温度和干旱胁迫的高弹性[18]. 谷子种质的分子多样性研究有限,很少采用下一代测序技术:谷子遗传多样性的研究主要依赖于RAPD技术[19],AFLP [20]和SSR标记[21]. 虽然这些标记中的一些可以支持标记辅助育种[22],基于NGS的标记现在可以用有限的成本和努力来显著增加任何种质收集的分子特征的定义[23].NGS为基因分型铺平了道路,测序方法旨在从头鉴定单核苷酸多态性(SNPs),加速了NUCs的鉴定。近年来,NGS技术已被应用于谷子等位基因库的表征,包括基因组广泛多样性的研究[24]基因表达特征[25]更进一步的遗传方法来识别数量性状位点(QTL)和标记性状关联(MTA)[26].最近发表的Proso序列和染色体组装[27那28]将这个物种投射到现代基因组学中,揭示了基因挖掘的巨大潜力[29甚至大规模基因分型应用,包括基因组宽协会研究(GWAS)。大规模的GWAS研究依赖于NGS数据有可能通过将异常的等位基因变化带入主流研究来加速NUC育种[30].
基因分型技术的开发是通过靶向制备种质收集的综合和精确表征数据的表型方法补充。这些方法包括自动化表型,可用于用高精度复杂性格的农艺相关性,包括根特征[31],水果特征[32]和种子特征[33].然后可以将任何这些特征与SNP数据组合以识别支然兴趣基因组的MTA [34]从而将它们的相关性投射到育种决策中。例如,种子性状与产量表现有关,可以作为繁殖更理想品种的替代品[35那36]. 这些方法可应用于未开发的NUC等位基因多样性集合,加速新品种的开发[37那38]. 事实上,GWAS以前已经应用于其他谷子,增强了对基因型变异及其与表型关系的理解[7.那39那40].
在这项研究中,我们在世界收藏中表征了遗传多样性和种子特质多样性P粟粒这是高度多样化的农艺性状[18].我们利用这些信息描述了种子的多样性,并进行了GWAS,鉴定了13个与种子和农艺性状相关的mta。我们的研究结果支持NGS技术和自动表型技术在支持NUCs育种方面的潜力。
结果
核心种质的选择和表型多样性
88份核心种质是从一个较大的普通谷子种质中挑选出来的,代表了不同的地理起源和良好的田间表现。核心种质来自以下大陆:东欧23份,西亚16份,东亚14份,美洲12份,南亚6份,西欧8份,非洲5份,大洋洲4份。其中地方品种45份,品种16份,野生型2份,鉴定为育种材料1份。关于遗传物质的全部信息在附加文件中提供1:表S1。核心集合以前的特点是其农艺表现[18].
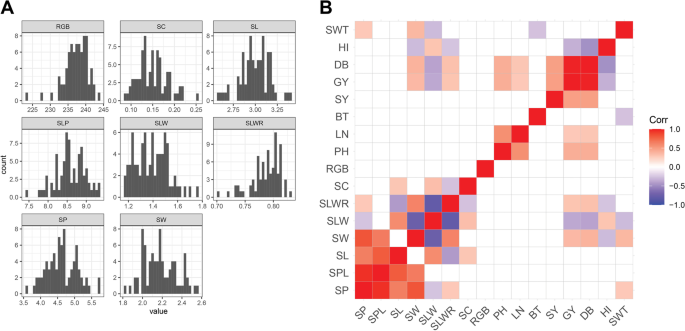
我们选择了对每次加入相同收获的健康种子,我们在种子措施中进行了自动表型,包括种子长度(SL,MM),种子宽度(SW,IN mM),种子周长(SP,MM),种子周边长度(SPL,IN mm),种子长度宽度(SLW,MM),种子长度为宽比(SLWR),种子圆形(SC)和种子颜色(RGB)。我们发现对分析的所有种子特征的广泛变化(表1). 表型频率的分布基本正常(图。1A) ,即使SLWR、RGB和SC由于种子形状在集合中的不均匀分布而显示出过度峰度(附加文件)2:图S1)。在所有测量的性状中进行相关分析。正如预期的那样,大多数种子尺寸的性状高度相关,但种子尺寸和颜色之间没有观察到相关性(图。1b)。当种子特征与先前在收集上测量的农艺性状相关时[18[我们鉴定了SW,SWT和干生物量(DB)和谷物产量(GY)之间的显着正相关,这意味着具有较大种子的屈服率高(图。1B) 是的。方差分析(ANOVA)结果表明,美洲材料的SW最大,SLW和SLWR显著降低;所有这些材料都是改良材料(附加文件)2:图S2)。SW据报道,西亚的种子加入显着较小,大部分都是Landrace材料。基于种子颜色的可见差异,该收集可分为五组:52(59.09%)黄色,15(17.04%)白色/灰色,7(7.95%)绿色,7(7.95%)橙色/红色,和7(7.95%)黑色。扫描图片中的RGB值证实了较轻的较轻种子种子,西部和南亚与其余的RGB值具有明显不同的RGB值(附加文件2:图S2)。
种子及形态农艺性状分析。一种种子特征的直方图。B.种子性状与农艺性状的相关性。关联的方向和强度由图例中的瓷砖颜色表示。空白贴图表示没有显著相关性。SP、种子周长;SPL:种子周长;SL,种子长度;西南、种子宽度;SLW,种子长度与宽度;SLWR,种子长宽比;SC、种子循环; RGB, seed color; PH, plant height; LN, leaf number; BT, basal tiller number; SY, seed yield; GY, grain yield; DB, dry biomass; HI, harvest index; SWT, seed weight
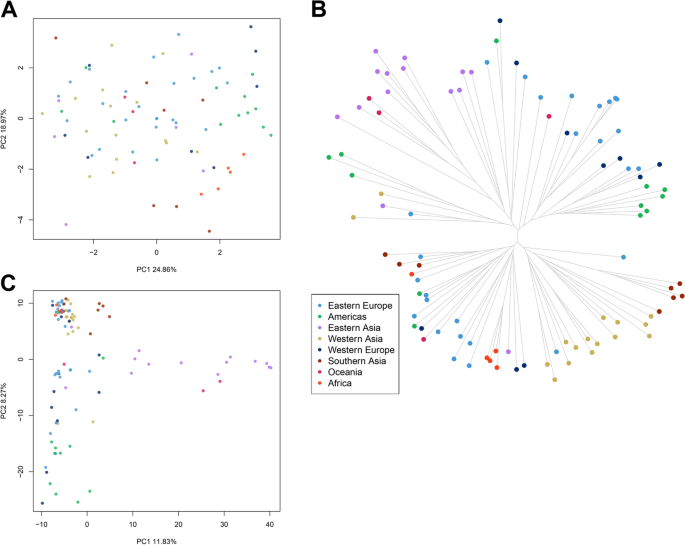
对种子和农艺性状进行了主要成分分析(PCA),以验证核心集合中的任何结构的存在。PC1和PC2分别解释了25%和19%的表型方差,并且没有任何与地理出现相关的分组(图。2大多数地方品种和改良材料几乎没有重叠(附加文件2:图S3A和S4)。与PC1最多相关的变量分别是SLP,SP,SW(分别为0.24,0.35和0.44)和负面的SLW,而PC2主要由SLP和SL(分别为0.15和0.46),并通过pH负面贡献(附加文件2:图S5)。
测序和基因型多样性
从核心收集的幼苗中提取DNA,并用Illumina技术测序。测序共产生494.2米的原料读数,其中0.67%在适配器移除和质量修剪步骤期间掉落。每个样本的保留读数的平均值约为5.14米。读取与Proso小米参考基因组对齐,获得高比例的映射读取(附加文件1:表S2)。SNP调用导致4907个优质标记分布在所有染色体上,经过严格筛选后,这些标记进一步减少到2412个优质SNP,以提高等位基因调用的可靠性。从一组高质量的SNPs得到的系统发育树可以分为两个主要的簇(以下称为簇I和簇II)(图。2b)。集群我将来自东亚,美洲和大洋洲的样本分组。Cluster II将广大院长的大部分加入来自西亚,南亚和非洲。来自东欧和西欧的进入在两个集群之间相当分布。当我们与遗传物质类型的原产地的信息进行比较时,大多数改进的进程分组到集群I中,除了东欧的三种载体(附加文件)2:图S3B)。被指定为未知的申报几乎总是与两个集群中的背板紧紧地聚集;大多数都被分组在集群II中。在群集II中聚集的两个野生access(附加文件2:图S3B)。
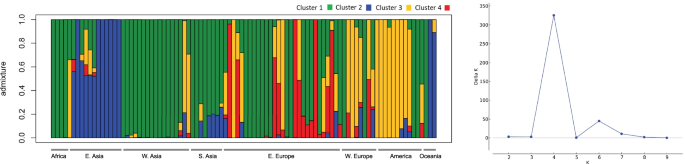
为了更好地观察个体间的遗传关系,我们对分子数据进行了主成分分析(PCA)。2C;额外的文件2:图S3C)。第一个和第二个PCs分别占方差的11.83%和8.27%,报告了数据集中的地理结构,特别是来自东欧、美洲和东亚的材料,这三个PCs几乎没有重叠(补充文件)2:图S6)。来自南亚和西亚的样本一直被分组(图。2C)。贝叶斯结构分析表明,收集中存在的最可能数量的K遗传簇为4(图。3.).总体而言,结果表明,加入有各种混合物。西亚,美洲,南亚和西欧的进入各个地区的背景混合。大洋洲样品的背景类似于西亚,非洲进程没有显示出混合物,在东欧和西亚的一些途中观察了类似的遗传背景(图。3.).
种子和农艺性状的全基因组关联
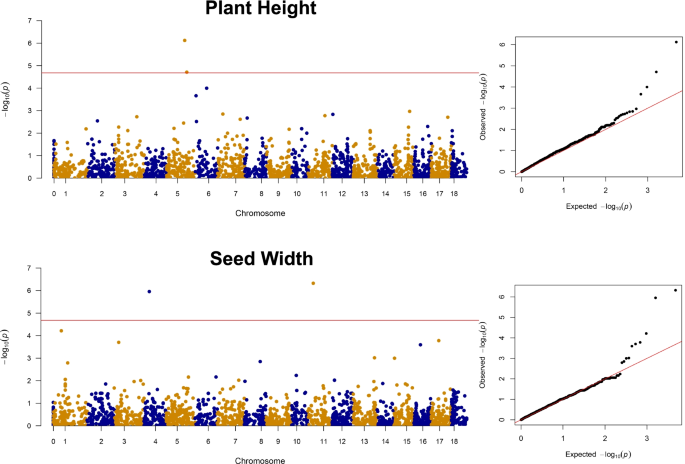
结合表型数据和SNP数据进行GWAS(附加文件)1:表S3)。结果表明,13个mta超过多重检验校正显著性阈值,10个mta超过种子相关性状,3个mta超过农艺性状。SP在6号染色体(Chr)和8号染色体(Chr)上分别鉴定出43.4 Mb和22.4 Mb的mta。在Chr 4和Chr 11上为SW发现了两个mta,分别为10.5 Mb和11.1 Mb。两个用于SL的mta在Chr 8上为31.6 Mb,在Chr 13上为26.2 Mb。另外两个mta在Chr 5上为SLWR映射为44.08 Mb,在Chr 6上为43.5 Mb。RGB与两个mta相关联,在Chr 1上为54.1 Mb,在Chr 8上为372 Mb。对于PH,我们在5号染色体上发现了两个MTA,分别为35.9 Mb和40.1 Mb。LN在Chr 16上发现了一个MTA,为1.03 Mb(表2; 图。4.;额外的文件2:图S7)。LN、RGB和SP的MTA具有最高的显著性(表1)2).mta对PH的影响最大,分别为9.6 cm和7.89 cm。
讨论
在这项研究中,我们组装了来自八个世界地区的88个ProSo小米的集合,以将他们的农艺和种子特征与GWAs的遗传多样性相结合。我们发现了具有一些特征的高变异和强大的信号,支持使用基因组学和表型筛选来快速检测MTA,以加速NUCS育种。
为了我们的表型特征,我们关注种子大小、形状和颜色的数量性状。这些性状具有很高的育种相关性,因为它们反映了超过种子形态的遗传、生理和生态变异[41那42].实际上,种子形态可以影响萌发生理学,营养质量和产量[43那44]. 种子性状可能与成株性状有关,成株性状在作物生命周期和环境适应性中起着重要作用,是产量潜力的组成部分[45那46那47].产量组分是繁殖努力的有价值的目标,特别是对于遗传改善故事有限的NUC。定位这些特征提供了几个优点,包括它们通常具有比产量更简单的遗传测定的事实,更容易测量,受环境的影响较小,导致更高的遗传性[48].这些功能使这些特征有价值的标记辅助选择目标。最后,由于自动化方法的可用性,种子特征易于表型,使得大量出位收集的表征可以在不需要现场实验[49].开放领域的表型受影响基因型表现的环境因素的影响,使GWA更具挑战性。例如,Proso Mill中的花序和尖峰数的大小受到光周期的高度影响,短期诱导减少了两种特征的[50].虽然量化环境效应是育种的有价值的工具,但种子的成像的表型可以提供优先级遗传物质的关键信息。实际上,先前已在包括稻米的其他谷物中鉴定出与晶粒尺寸相关的基因组区域[51]玉米[52] 小麦 [53],还有大麦[54].
使用NGS的核心系列的基因分型使我们能够开发一种高质量的基因组宽SNP,脱落在全球Proso Mill中的系统发育关系上。很少有地体集聚集在改进的材料中,提出了对它们衍生的传统品种的最小偏离偏离。根据其基因型多样性分组acade(图。2C;额外的文件2:图S3C)基于其表型多样性,不对应任何清晰分组的摘要(图。2一种;额外的文件2:图S3A)。尽管如此,在proso谷子收集报告的表型和分子多样性表明在未来的育种计划中有很高的潜力。我们的结果表明,东亚(中国、韩国、日本和台湾)的材料与西亚(阿富汗、伊朗、伊拉克、吉尔吉斯斯坦和哈萨克斯坦)和南亚(印度和尼泊尔)的材料不同(图。2b)。这一发现对应于以前的研究报告来自亚洲的波斯诺小米中的两个主要群体:一个集团在内的东部亚洲国家,包括中国,包括西亚洲国家在内的另一组[55].虽然繁殖材料经常在国家之间交换,但实地性地区不太可能转移巨大的地理距离。他们与特定地点的历史关系可能会及时延迟数百甚至千年[56那57],可以重建由过去过程产生的分化。关于粟的起源和传播,可以提出两个假说;一种是(1)在中国的单一起源中心,然后向西传播,普罗索在到达东欧之前到达中亚;另一种是(2)多个驯化中心[55].众所周知,在BC三千年千禧年之后,内部亚洲山路走廊的迁移可能导致几种谷物的蔓延,包括Proso Millet [58]. 我们的基因组学数据支持这一假设,将西亚种质与欧洲种质分为第二类,尽管需要更多考虑更广泛种质收集的研究来全面揭示谷子的进化历史。
GWA提供QQ图报告的良好模型适合支持的强大关联信号(附加文件2:补充S8)。以前为Proso Mill开发的遗传地图是破裂的,并且在染色体中没有组装[26]限制了我们的mta与先前结果的共映射的可能性。然而,我们报告的关联的强度和程度暗示了mta与proso谷子遗传改良的相关性。大多数加入(~ 76%)的种子有较浅的阴影。GWAS结果显示,在chr1和chr8上分别有两个RGB的mta,分别为54.1mb和37.8mb。以前的研究表明,小杂粮种子颜色的差异可能是由于谷壳中的单宁成分造成的,普罗索谷子种子颜色较深,单宁含量最高[59那60].单宁是具有高抗氧化潜力的酚类化合物[61那62]这意味着颜色较深的种子可能具有更好的营养成分。不管怎样,消费者通常更喜欢黄色谷物[63],这反映在我们的收藏中这种颜色的流行上。有趣的是,谷子种皮颜色与种子发芽率和生活力有关[64那65],表明该研究未考虑其他重要性状的多效性。
自然选择有利于野生亲属的圆形和小尺寸,但繁殖一直专注于增加种子大小[66].我们确定了八种MTA用于种子尺寸Traits SP,SL,SW和SLWR。我们发现这些种子尺寸特征之间的正相关(图。1b)为这些特征观察到的MTA中的有限重叠。我们在CHR 8,9.17 MB分开的SP和SL上的同一染色体上发现了MTA,SP和SP和SP和SLWR ON CHR 6,0.14 MB分开,表明存在控制这些性状的链接基因组基因座。除Proso以外的小米中,基因组的研究已经确定了种子尺寸特征的QTL,包括谷物产量和种子重量的CHR 4,5 [67] Chr 1,2,3,6,7 [68] Chr 2,3,6,8,9 [69]和Chr 3,4,5 [70].种子长度,宽度和周长方面的晶粒尺寸对种子重量直接影响,因此谷物产量;我们发现SP,SW与种子重量的正相关(SWT)(图。1B) 但没有重叠的MTA。在显著相关的单核苷酸多态性中,我们在chr6和chr8上鉴定了4个粒径性状的mta。在谷子中,chr6和chr8上的信号与千粒重(TGW)和产量(GY)等粒重性状有关[69那71].
我们发现LN具有pH,GY和DB的正相关(图。1B)和一个MTA对Chr 16,估计影响1.1额外叶LN。叶片性状的QTL已广泛应用于其他作物,包括Triticum aestivum.[72],Elaeis Guineensis.[73]以及Hordeum Vulgare.[74]作为改善植物架构的门户。我们收藏中的植物高度是非常多样化的,范围为30到100厘米[75].pH的育种至关重要,因为这种特性与繁殖,种子质量,产量和成熟度强烈相关[76].在本研究中,我们发现两个MP,位于CHR 5的pH值,分开4.15 MB,并解释了特征的大变化(表2).谷子的株高曾在谷子的5 Chr (Setaria Italica.) [70那77那78],其基因组对其他小小米的可转移性估计> 85%的可转移性[6.].叶数在植物的光合能力中起着直接作用,因此有助于收益[79那80].
结论
我们的表征揭示了Proso Millet种质中的种子和分子特征的广泛多样性,这是一种有价值的资源来加速本物种的繁殖。虽然本研究中使用的人口的大小很小,但是即使在实现严格的多次测试校正之后,我们也识别出显着的MTA。进一步的研究可以专注于较大的收集和多年的表型实验,以增加这里报告的表征的深度。关于Millet上可用的基因组学工具的信息层的积累将进一步提高我们的结果的可利用性。这包括开发可容易地用于支持在育种中的标记辅助选择的KMPETITIVE等位基因特异性PCR(KASP)。为Proso Millet MTA开发的Kasp可以用于筛选大小米集合中这些基因座的存在。随着更多信息将可用,分子研究可以专注于MTA的验证,以了解营养种子的分子机制和农艺性状。目前,我们的成果报告了ProSo Mill植物遗传资源中繁殖的高潜力,支持使用NGS和自动表型以推动该NUC的育种努力。
材料和方法
核心藏品选择
300年的种子P粟粒(L.)加入从美国农业部(USDA)种质银行获得。收集样品没有必要特殊权限。2016年,全收集播种为种子乘法和筛选在San Piero A Grado,Pisa,意大利(43.6797°N,10.3468°E),使用50cm分开的植物,以避免交叉授粉。核心集合从完整的集合中得出,以便得到的样本池将总结世界ProSo Mill多样性。从USDA获得每个加入的护照数据。优先考虑具有i)已知的地理原产地,ii)Passport数据中的至少部分信息,iii)在2016年的放大现场实验期间显示的良好表现。根据这些标准,核心收集为88种附加标准被选中以进行种子表型和基因分型(附加文件1:表S1)。样品的正式鉴定由通讯作者进行。未存放凭证样本。
种子表型
每次加入的十五种健康种子选自2016年收获种子表型。种子被安排在纸板框架内并放置在EPSON完美3170照片图像扫描仪的扫描表面上。在3200DPI扫描图像并使用数字图像分析软件SmartGrain进行分析[81].在软件指南之后,在每次加入时手动策划种子检测。根据图像定义设置的比例,并且种子表征以批次运行。对于每次加入的15种种子中的每一个,程序测量种子周长(SP,MM),种子周长长度(SPL,IN mM),种子长度(SL,MM),种子宽度(SW,IN MM),种子长度宽度(SLW,MM),种子长度为宽比(SLWR)和种子圆形(SC)。使用imagej软件版本1.52a测量种子颜色(RGB)索引[82].该软件批量运行,以计算为每个扫描图像中的每个像素发出的红色,绿色,蓝色(RGB)光量。随着表型室是恒定的,RGB的任何差异都归因于种子颜色的差异。得到的RGB值用作表型测量。
在以前的实验中,在意大利(40°58'n,14°14'e)和农艺性状的农业研究所种植了相同的ProSo access,并记录了农艺性状:这些包括植物身高(pH,cm),叶子数(LN),基底分蘖数(BT),种子产量(SY,KG HA)-1),谷物产量(GY,KG HA-1),干生物量(DB,KG HA-1),收获指数(HI,谷物/生物质)和种子重量(SWT,G / 100)。完整细节在[18].
基因分型
2017年,来自成像分析的种子在培养皿中发芽,收集绿叶并从五个幼苗中汇集。在制造商的说明之后,使用基因株植物基因组DNA Miniprep套件(Sigma Aldrich,Germany)提取基因组DNA。基因组DNA完整性在1%琼脂糖凝胶中评价并使用Qubit荧光计(Invitrogen,Thermo Fishific,US)定量。在IGA技术服务(Udine,意大利)进行测序。根据限制性位点相关的DNA标记(Rad)方案制备测序文库[83]使用HindIII限制酶。在Illumina Hiseq 2000机器上复用并测序RAD库,以产生短读取,然后通过FASTQC进行质量检查并检查质量[84].简而言之,使用ERNE-FILTER V.2.1.2进行过滤原始读取(http://erne.sourceforge.net/) [85].过滤标准遵循标准程序,以确保仅保留高质量读取。使用BWA-MEM算法v0.7.17(NCBI标识符:PRJNA431363)映射过滤的读取映射了PROSO小米参考基因组装配版本GCA_003046395.2(https://github.com/lh3/bwa/releases/tag/v0.7.17)使用默认参数。
SNPs调用是使用单倍型调用程序进行的[86]从GVCF模式的基因组分析工具套件包版本4.2.0.0(https://github.com/boadinstitute/gatk/releases),遵循最佳实践。只有高质量(质量 > 30.0)双等位基因SNPs被保留。最后,用流苏5.0去除次要等位基因频率(MAF)低于1%的snp[87]和r [88自定义脚本。所有原始数据均可在国家生物技术信息中心(NCBI)数据库(https://www.ncbi.nlm.nih.gov)在Bioproject ID Prjna726150下。
数据分析
种子表型数据用R [88]使用自定义脚本。对于每个测量的种子特征,除去每种进入分析的15种种子中的三种最高和三个最低措施,并且在其余的那些中计算平均值。这旨在通过成像软件从子最优种子识别中消除可能的异常值。R / Corrplot [89[用于研究种子和植物性状的相关性。进行PCA以估计不同特征在捕获集合的变化中的相对重要性,并建立在研究下的所有变量之间的关系。进行ANOVA(R / GGPLOT2)以确定不同地区种子性状表型数据中多样性的统计学意义。
遗传多样性分析采用R [88]调查ProSo收集中分子多样性的不同方面,并识别群体。用R / AdegeNet制备邻近的系统发育[90]对单核苷酸多态性数据集进行主成分分析(PCA),以了解样本中遗传分支的存在和距离。结构2.3.4[91]通过检测最能描述数据的簇数,将个体分配到隐遗传簇中。结构以掺合料模型运行,经过1万次老化迭代和10万次MCMC重复。参数从K = 1到K = 10进行检验,其中K为遗传组数,每个重复10次。运行程序后,结果数据被加载到Structure Harvester [92]根据Evanno方法制作临时统计,以识别最可能的K [93].
将表型和基因型数据合并到GWAS分析中,并加入先前报道的形态-农艺多样性数据[18].使用固定和随机模型循环概率统一(FARMCPU)模型进行GWAS [94]在r / gapit中实现[95]使用基因型数据计算的第一个PC作为协变量。mta被定义为超过错误发现率(FDR)显著性阈值的snp < 0.05,根据楼层法[96]. 用R软件包qqman绘制GWAS结果[97];曼哈顿绘图显示与标称值相对应的严格onferroni阈值P.-value = 0.05,以帮助识别最显著的SNPs。
可用性数据和材料
本研究期间生成或分析的所有数据都包含在本公布的文章中作为补充材料。原始读数在国家生物技术信息中心(NCBI)数据库中提供(https://www.ncbi.nlm.nih.gov)在生物项目ID PRJNA726150下。田间表型来源于参考文献[18].使用的所有数据座对的数据访问都是打开的。脚本可以联系相应作者。
缩写
- SL:
-
种子长度
- SW:
-
种子宽度
- SP:
-
种子周长
- SPL:
-
种子周长长度
- SLW公司:
-
种子长宽比
- SLWR:
-
种子长度与宽比
- SC:
-
种子圆形
- RGB:
-
种子颜色
- 酸碱度:
-
株高
- LN:
-
叶数
- BT:
-
基底分蘖
- SY公司:
-
种子产量
- 灰色:
-
粮食产量
- D B:
-
干生物量
- 你好:
-
收获指数
- SWT:
-
种子重量/100
- nuc:
-
被忽视和未充分利用的作物
- CHR:
-
染色体
参考文献
- 1.
杨X,WAN Z,Perry L,Lu H,Wang Q,Zhao C等人。早期的小米在中国北方使用。Proc Natl Acad Sci U S A. 2012; 109(10):3726-30。
- 2.
鲁H,张j,刘kb,吴n,李y,周k等。最早的融合普通小米(Panicum Miliaceum.)在东亚扩展到10,000年前。Proc Natl Acad Sci U S A. 2009; 106(18):7367-72。
- 3.
达斯·伊克,拉克斯。第一章-谷子,它们的重要性和生产限制。编辑:Das IK,Padmaja PG,编辑。谷子的生物抗逆性。阿姆斯特丹:学术出版社,爱思唯尔出版社;2016年。3–19.
- 4.
LáglerR,戈努利G,Humphreys M,SzabóZ,霍尔瓦特,Bittsánszkya等。普通小米的形态学和分子分析(P粟粒)品种与15世纪(匈牙利)的ADNA样本相比。Euphytica。2005; 146(1):77-85。
- 5.
Li X, Yadav R, Siddique KHM。被忽视和利用不足的作物品种:改善亚洲和太平洋地区饮食多样性和消除饥饿和营养不良的关键。减轻前面。2020;7(254):593711。
- 6.
Vetriventhan M、Azevedo VCR、Upadhyaya HD、Nirmalakumari A、Kane Potaka J、Anitha S等。遗传和基因组资源,以及加速小谷子改良育种:现状和未来干预措施。细胞核。2020;63(3):217–39.
- 7.
Varshney RK,Shi C,Thudi M,Mariac C,Wallace J,Qi P等人。珍珠小米基因组序列提供了改善干旱环境中农艺性状的资源。NAT BIOTECHNOL。2017; 35(10):969-76。
- 8.
张克,刘X,Quan Z,Cheng S,Xu X,Pan S等人。Foxtail小米的基因组序列(Setaria Italica.)提供了对草的进化和生物燃料潜力的见解。生物科技Nat》。2012;30(6):549 - 54。
- 9.
Vetriventhan M、Azevedo VCR、Upadhyaya HD、Naresh D。全球谷子产量的变异性(Panicum Miliaceum.保存在ICRISAT基因库的种质收集。农业。2019;9(5):112.
- 10
Habiyaremye C, Matanguihan JB, D 'Alpoim Guedes J, Ganjyal GM, Whiteman MR, Kidwell KK等。Proso millet(Panicum Miliaceum.L.)及其在太平洋西北部种植的潜力,U.S .:审查。前植物SCI。2017; 7:1961。
- 11
高粱和小米的新型食品和非食品用途。中国粮食科学,2006;44(3):252-71。
- 12
Parthasarathy Rao P,Basavaraj G.印度利用的现状和前景和全球场景。在:小米:促进食品,饲料,饲料,营养和环境安全。全球咨询关于Millets Research促进Millets Research,Icar Indian of Millets Research,Hyderabad。2015. p。197-209。ISBN 8189335529。
- 13
Kalinova J,Moudry J. Proso Millet中蛋白质的含量和质量(Panicum Miliaceum.L.)品种。植物食物嗡嗡声Nutr。2006; 61(1):45-9。
- 14
萨利赫·阿斯姆、张琦、陈杰、沈琦。小米:营养品质,加工和潜在的健康效益。食品科学杂志。2013;12(3):281–95.
- 15
Trivedi Ak,Arya L,Verma M,Verma Sk,Tyagi Rk,Hemantaranjan A. ProSo Millet的遗传变异性(Panicum Miliaceum.)喜马拉雅中部地区种质资源的形态生理特性和分子标记研究。生理植物学报。2015;37(2):23.
- 16
刘米,徐y,何J,张某,王Y,鲁普遗传多样性和武装结构结构(Panicum Miliaceum.L.)基于微卫星标志的中国品种和地体。int j mol sci。2016; 17(3):370。
- 17.
德湿JMJ。次要谷物的起源、演化与系统。见:Seetharam A, Riley KW, Harinarayana G,编辑。全球农业中的小小米。1986年10月29日至11月2日,印度班加罗尔,第一届国际小小米研讨会论文集。
- 18.
Calamai A、Masoni A、Marini L、Dell'acqua M、Ganugi P、Boukail S等。80份谷子种质农艺性状的评价(Panicum Miliaceum.地中海土壤气候条件下。农业。2020;10(12):578.
- 19.
M'Ribu香港,Hilu千瓦。植物种间和种内变异的检测panicum.谷子通过随机扩增多态DNA。理论应用基因。1994;88(3):412–6.
- 20。
Karam D, Westra P, Niessen SJ, Ward SM, Figueiredo JEF。谷子DNA多态性银染AFLP标记的评价(Panicum Miliaceum.L.)。Rev Bras机器人。2006;29:609–15.
- 21。
Rajput S,Plyler-Harveson T,Santra D.基于Switchgrass基因组学的ProSo Mill中SSR标记的开发和表征。am j植物sci。2014; 05:175-86。
- 22。
Collard BCY,麦吉尔DJ。标记辅助选择:21世纪植物精密育种的一种途径。Philos Trans R Soc Lond B Biol Sci.博士。2008;363(1491):557–72.
- 23。
他J,Zhao X,Laroche A,Lu Z-X,Liu H,Li Z.基因分型逐序列(GBS),终极标记辅助选择(MAS)工具加速植物育种。前植物SCI。2014; 5:484。
- 24。
Johnson M, Deshpande S, Vetriventhan M, Upadhyaya HD, Wallace JG。三种小小米的全基因组群体结构分析:科多小米、小小米和普罗苏小米。植物基因组。2019;12(3):1 - 9。
- 25。
yue h,wang l,刘h,yue w,du x,song w等。De Novo组装和扫帚米兰体变量组的特征(Panicum Miliaceum.基因发现和标记开发。前植物科学。2016;7:1083.
- 26。
谷子形态农艺性状的qtl定位Panicum Miliaceum.L.)。mol品种。2016; 36(4):37。
- 27。
邹聪,李力,米基德,李德,唐Q,肖力,等。黍米基因组。自然公社。2019;10(1):436.
- 28
石杰,马×,张杰,周勇,刘明,黄磊,等。染色体构象捕获解析了谷子近全基因组组装。自然公社。2019;10(1):464.
- 29
单Z,江Y,李H,郭J,董M,张J,等。黍NAC转录因子家族的全基因组分析(Panicum Miliaceum.干旱胁迫下的表达分析。BMC基因组学。2020;21(1):96.
- 30
Yabe S,Iwata H.基因组学辅助繁殖的次要谷物和伪谷物。品种sci。2020; 70(1):19-31。
- 31
Iyer-pascuzzi as,symonova o,mileyko y,hao y,belcher h,harer j等。植物根系自动表型化和特质排名的成像与分析平台。植物理性。2010; 152(3):1148-57。
- 32
刘文,刘C,金吉,李德,傅y,袁X.使用X射线计算机断层扫描的形态种子和果实特征的高通量表型。前植物SCI。2020; 11:601475。https://doi.org/10.3389/fpls.2020.601475。
- 33
杜库诺等[1]分析了该方法的应用前景。甜菜种子和幼苗基因型相关性状的高通量表型数据集。数据简单。2020;29:105201。
- 34
Lo S,Muñoz Amatriaín M,Hokin SA,Cisse n,Roberts PA,Farmer AD等。全基因组关联和荟萃分析揭示了与豇豆种子大小相关的区域[Vigna Unguiculata.(L.)沃尔普]。Al Appl Genet。2019; 132(11):3079-87。
- 35
闫S,zou g,li s,王h,刘h,zhai g等。种子大小由控制水稻中不同种子特征的基因的组合来确定。Al Appl Genet。2011; 123(7):1173-81。
- 36
Kesavan M, Song JT, Seo HS。种子大小:谷类作物的优先特性。杂志。2013;147(2):113 - 20。
- 37
Alqudah Am,Haile JK,Alomari Dz,Pozniak CJ,Kobiljski B,BörnerA。基因组 - 范围和SNP网络分析揭示小麦小穗的尖刺菌和产量相关性状的遗传控制。SCI代表2020; 10(1):2098。
- 38
Hamblin MT, Buckler ES, Jannink JL。基于基因组学的作物改良方法的群体遗传学。趋势麝猫。2011;27(3):98 - 106。
- 39
Upadhyaya HD,Vetriventhan M,Deshpande Sp,Sivasubramani S,Wallace JG,Buckler Es,等。全球福克斯米尔种质收集的人口遗传学和结构。植物基因组。2015; 8(3):EPLANTENOME2015.07.0054。
- 40。
Sharma D, Tiwari A, Sood S, Jamra G, Singh NK, Meher PK等。谷子品种间农业形态性状的全基因组关联图谱(Eleusine coracanaL.)基因型使用SNP标记。Plos一个。2018; 13(8):E0199444。
- 41。
岩田H,Ukai Y。形状:一个基于椭圆傅立叶描述子的定量评估生物形状的计算机程序包。杰瑞德。2002;93(5):384–5.
- 42。
卜海英,王学杰,周学海,齐伟,刘凯,葛文杰,等.青藏高原东部383种植物种子形态和体积对萌发的生态学和进化意义。福利亚·杰博特。2016;51(4):333–41.
- 43。
Adewale BD, Kehinde OB, Aremu CO, Popoola JO, Dumet DJ。非洲山药豆种子的遗传和形状测定(蝶形花科).AFR J植物SCI。2010; 4(4):107-15。
- 44。
Cervantes E,Tocino A.乙烯,自由基和植物形态稳定状态的转变。植物信号表现。2009; 4(5):367-71。
- 45。
关键词:小麦,产量,qtl,染色体定位中国生物医学工程学报,2000;
- 46。
Maccaferri M, Sanguineti MC, Corneti S, Ortega JL, Salem MB, Bort J等。硬粒小麦产量和适应性的数量性状位点(Triticum杜伦姆广泛的水资源可利用性。遗传学。2008;178(1):489–511.
- 47。
王旭,罗庚,杨伟,李勇,孙杰,展凯,等。野生二倍体小麦农艺性状和籽粒性状的遗传多样性、群体结构及标记性状关联乌拉尔小麦. BMC植物生物学。2017;17(1):112.
- 48。
告诉我啊。安大略省西南部冬小麦群体产量、株高和容重的遗传力。理论应用基因。1984;68(1–2):21–3.
- 49。
Rolletschek H,Fuchs J,Friedel S,BörnerA,Todt H,Jakob PM,等。主要种子特征的高通量筛选新型非侵袭性程序。植物Biotechnol J. 2015; 13(2):188-99。
- 50。
Tayal MS, Nanda KK。光周期对水稻茎尖发育的影响Panicum Miliaceum.李建平。植物生理学。1998,23:1 - 9。
- 51。
Takano-Kai N, Jiang H, Kubo T, Sweeney M, Matsumoto T, Kanamori H等。水稻粒长基因GS3的进化历史。遗传学。2009;182(4):1323 - 34。
- 52
李伟,白Q,詹W,马C,王S,冯勇,等。玉米粒重主效QTL qhkw5-3的精细定位及候选基因分析。理论应用基因。2019;132(9):2579–89.
- 53
Su Z,Hao C,Wang L,Dong Y,Zhang X.鉴定和开发与面包小麦的粒重相关的Tagw2功能标记(Triticum aestivum.L.)。理论应用基因。2011;122(1):211–23.
- 54
(1)国家自然科学基金项目,国家自然科学基金项目,国家自然科学基金项目,国家自然科学基金项目,国家自然科学基金项目。大麦的表观遗传染色质修饰:IV.大麦Polycomb group (PcG)基因在种子发育过程及对外源ABA响应的研究。中国生物医学工程学报。2010;10(1):73。
- 55
Hunt HV,Campana Mg,Leyes Mc,Park Y-J,Bower Ma,Howe CJ等。武装群小米的遗传多样性和帖子摄影(Panicum Miliaceum.L.)欧亚亚洲。Mol Ecol。2011; 20(22):4756-71。
- 56
琼斯H,Lister DL,Bower Ma,Leigh FJ,史密斯LM,琼斯MK。使用现有地兰和现存植物材料了解史前农业蔓延的方法和约束。植物遗传群。2008; 6(2):98-112。
- 57
Lister DL,Bower Ma,Jones Mk。Herbenium标本扩展了Phylopeography研究中的种质数据的地理和时间范围。分类。2010; 59(5):1321-3。
- 58
Miller NF,Spengler RN,Frachetti M。欧亚大陆谷子种植:起源、传播和季节性气候的影响。全新世。2016;26(10):1566–75.
- 59
călinoiulf,vodnar dc。全谷物和酚酸:关于生物活性,功能,健康益处和生物利用度的综述。营养素。2018; 10(11):1615。
- 60.
洛伦兹K。谷子中单宁和植酸含量的研究(Panicum Miliaceum.).谷物化学。1983; 60(6):424-6。
- 61.
Sripriya G,Chandrasekharan K,Murty VS,Chandra Ts。ESR光谱研究手指小米的自由基猝灭作用(Eleusine coracana).食品化学。1996年; 57(4):537-40。
- 62.
Hedge PS,Chandra TS.ESR光谱研究显示,小米具有较高的自由基猝灭潜力(Paspalum scrociculatum.)与其他小米相比。食品化学。2005; 92(1):177-82。
- 63.
王R,Hunt HV,Qiao Z,Wang L,Han Y.武装群的多样性和培养(Panicum Miliaceum.L.)在中国:审查。econ bot。2016; 70(3):332-42。
- 64。
Khan M、Cavers PB、Kane M、Thompson K。谷子色素种皮的作用(Panicum Miliaceum.L.)在吸收,萌发和种子持久性。res j种子sci。2008; 7(1):21-6。
- 65。
洞穴PB,Marguerite K,James Jot。种子银行为建立新推出的杂草的重要性:ProSo Millet的案例研究(Panicum Miliaceum.).杂草科学。1992年; 40(4):630-5。
- 66。
acta photonica sinica, 2012, 38(5): 762 - 768 .范超,邢莹,毛慧,陆涛,韩斌,徐超,等。GS3是水稻粒长和粒重的主效QTL和粒宽和粒厚的次效QTL,编码一种跨膜蛋白。理论与应用,2006;112(6):1164-71
- 67。
方旭,董凯,王旭,刘涛,何静,任锐,等。谷子农艺性状和产量性状的高密度遗传图谱及QTL分析[Setaria Italica.(L.)P. Beauv。]。BMC基因组学。2016; 17(1):336。
- 68。
Kumar S,Hash CT,Nepolean T,Satyavathi CT,Singh G,Mahendrakar MD等。映射QTLS控制珍珠小米中的开花时间和重要农艺性状。前植物SCI。2017; 8:1731。
- 69。
Jaiswal V,Gupta S,Gahlaut V,Muthamilarasan M,Bandyopadhyay T,Ramchiary N等人。Gextail Millet主要农艺性状的基因组关联研究(Setaria Italica.L.)使用DDRAD测序。SCI批准。2019; 9(1):5020。
- 70。
张克,风扇G,张X,赵F,魏W,Du G等。鉴定QTL的14个农艺上重要特征Setaria Italica.基于高吞吐量测序生成的SNP。G3-基因基因类型。2017; 7(5):1587-94。
- 71。
贾庚,黄×,志H,赵Y,赵Q,李W,等。谷子基因组变异的单倍型图及农艺性状的全基因组关联研究(Setaria Italica.).NAT Genet。2013; 45(8):957-61。
- 72。
刘k,徐h,刘g,关p,周x,peng h等。Qtl映射小麦旗叶相关性状(Triticum aestivum L.)。Al Appl Genet。2018; 131(4):839-49。
- 73。
Babu BK, Mathur RK, Ravichandran G, Anitha P, Venu MVB。油棕叶面积、轴长和总干重的全基因组关联研究(Eleaeisguineensis.)通过测序进行基因分型。公共科学图书馆一号。2019;14(8):e0220626。
- 74。
杜斌,刘玲,王强,孙刚,任鑫,李超,等。大麦不同叶片叶片长度和面积的QTL鉴定。Sci众议员2019;9(1):4431。
- 75。
Baltensperger DD. Progress with proso, pearl and其他小米。选:Janick J, Whipkey A, editors。新作物和新用途的趋势。亚历山大:灰机;2002.100 - 3页。
- 76。
鼹鼠,沃顿二,温暖,斯威芳NG,Laffan SW,Zanne AE等。植物高度的全球模式。J ECOL。2009; 97(5):923-32。
- 77
倪x,xia q,张h,cheng s,li h,风扇g等。通过重新排列RIL群体更新了粪尾小米基因组组装和九个主要农艺性状的基因映射。傻瓜。2017; 6(2):1-8。
- 78
王智,王军,彭军,杜鑫,蒋敏,李勇,等。基于谷子F2大群体全基因组bin图的11个农艺性状QTL定位(Setaria Italica.(L.)P。博夫)。摩尔品种。2019;39(2):18.
- 79
王福明,黄建峰,楼志华。利用最优高光谱波段估算水稻叶面积指数的三种方法比较。摘要阿格利司。2011;12(3):439 - 47。
- 80。
Pérez-PérezJM,Esteve-Bruna D,MICOL JL。叶架QTL分析。int j植物res。2010; 123(1):15-23。
- 81。
七夕T,柴谷T,堀K,叶花K,矢野M。智能谷物:通过图像分析测量种子形状的高通量表型软件。植物生理学。2012;160(4):1871.
- 82.
Schindelin J,Rueden CT,Hiner MC,Eliceiri Kw。imagej生态系统:生物医学图像分析的开放平台。mol sold dev。2015; 82(7-8):518-29。
- 83.
Baird Na,Etter Pd,Atwood Ts,Currey MC,Shiver Al,Lewis Za等人。使用测序的RAD标记的快速SNP发现和遗传映射。Plos一个。2008; 3(10):E3376。
- 84.
Andrews S. FASTQC:高吞吐量序列数据的质量控制工具。2010.在线提供:http://www.bioinformatics.babraham.ac.uk/projects/fastqc。
- 85.
Del Fabbro C,Scalabrin S,Morgante M,Giorgi FM。对Illumina NGS数据分析的读整理效应进行了广泛的评价。Plos一个。2013; 8(12):E85024。
- 86.
Poplin R、Ruano Rubio V、DePristo MA、Fennell TJ、Carneiro MO、Van der Auwera GA等。将精确的基因变异发现扩展到数万个样本。生物十四。2018:201178.https://doi.org/10.1101/201178。
- 87.
Bradbury PJ、Zhang Z、Kroon DE、Casstevens TM、Ramdoss Y、Buckler ES。流苏:用于在不同样本中绘制复杂性状关联图的软件。生物信息学。2007;23(19):2633–5.
- 88。
团队RC。R:用于统计计算的语言和环境。维也纳:R基础统计计算;2013.
- 89。
太原W,Simko V. R包“Corrplot”:相关矩阵的可视化(版本0.84)。2017.可获得https://github.com/taiyun/corrplot。
- 90。
Jombart T,Ahmed I. Adegenet 1.3-1:用于分析基因组SNP数据的新工具。生物信息学。2011; 27(21):3070-1。
- 91。
Pritchard JK,斯蒂芬斯M,Donnelly P.使用多点基因型数据的群体结构推理。遗传学。2000; 155(2):945-59。
- 92。
伯爵大,瓦尔德特BM。结构收割机:一个网站和程序可视化结构输出并实现Evanno方法。保守遗传资源。2012; 4(2):359-61。
- 93。
关键词:个体聚类,结构模型,个体聚类摩尔生态。2005;14(8):2611 - 20。
- 94。
刘X,黄M,范B,巴克勒ES,张Z。迭代使用固定和随机效应模型进行有效的全基因组关联研究。公共科学图书馆Genet。2016;12(2):e1005767。
- 95。
叶丽佳,田芙,王琦,裴飞J,李明,布拉德伯里PJ,等.GAPIT:基因组关联与预测集成工具。生物信息学。2012;28(18):2397–9.
- 96。
楼层JD,Tibshirani R. Genomewide研究的统计学意义。Proc Natl Acad Sci U S A. 2003; 100(16):9440。
- 97。
特纳SD。QQMAN:使用Q-Q和曼哈顿情节可视化GWAS结果的R包。生物XIV。2014;https://doi.org/10.1101/005165。
致谢
作者谨此感谢USDA提供研究材料。作者还要感谢Paola Ganugi,Stefano Benedettelli和MarioEnricoPè的见解和富有成效的讨论。
基金
这项工作得到了VARITOSCAN-CLIMA欧洲农业基金会(FEASR)和意大利比萨圣安娜高等农业生物多样性博士学院的资助。
作者信息
隶属关系
贡献
MD设计了这项研究并监督研究。SB进行了实验和数据分析。MeMa, MaMi管理分子数据并进行数据分析。AM, AC和EP参与了数据收集和实验设计。MeMa, MD, SB起草了手稿并制作了数字。所有作者均已阅读并批准稿件。
通讯作者
道德宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明没有相互竞争的利益。
附加信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
补充文件1:补充表1。
本研究中使用的摘要摘要。表报告了样本名称,材料的类型和地理信息。访问ID和Name将样本的国际标识符报告在美国农业部护照信息中报告的。补充表2。概述RAD测序数据产生和比对参考序列。对于每个样本,表报告了通过质量阈值的总读量、映射到基因组上的总读量以及两者的比例。补充表3。gwas摘要数据。对于每个SNP,表格向参考基因组报告染色体和位置。对于每个特征-SNP组合,表报告了关联的p值和估计效果。特质名称被编码为主文本。
附加文件2:补充图S1。
本研究表征的Proso Mill种子颜色类的代表性照片。补充图S2。地理区域横跨地区地区的种子特质分布箱。使用ANOVA分析差异。补充图S3。谷子种质的表型和分子分析(A) 种子性状和农艺性状表型多样性的主成分分析(B) 基于SNPs数据的系统发育树(C) 基于SNPs数据的主成分分析。不同的符号表示遗传物质的类型,如图例所示。补充图S4。由PC1、PC2和PC3报告的集合中的表型多样性。面板上的不同颜色和符号表示遗传物质的起源地区和类型,如图2和补充图S3所示。补充图S5。种子和农艺性状的主成分分析得分和载体负荷。每个轴(PC1 = Dim1, PC2 = Dim2)解释的方差百分比在轴标题中表示。根据右边的图例,向量颜色表示给定变量在前两个维度上的总贡献。补充图S6。PC1,PC2和PCA3报告的收集中的基因型多样性。面板上的不同颜色和符号表示如图2所示的原产地和遗传物质类型。2和补充图S3。补充图S7。Gwas的曼哈顿图谱在种子特征和农艺性状。该图显示了每个SNP关联(Y轴)的所有染色体(x轴)和-log10 p值的单个SNP。不同的颜色表示Proso Mill的18个染色体。水平线根据严格的Bonferroni方法显示多个测试阈值。请注意,与每个GWA的最高意义相对应的不同Y轴尺度报告不同的曼哈顿图。补充图S8。用于农田模型的分位数(Q-Q)绘图,显示估计与观察到的 - 种子特征和农艺性状的估计 - 乐谱值的分布。
权利和权限
开放存取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/. 知识共享公共领域放弃(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
布卡尔,玛卡里亚,M。et al。基因组广泛协会在世界馆藏上的农艺和种子特征研究(Panicum Miliaceum.L.)。BMC植物生物学21,330(2021)。https://doi.org/10.1186/s12870-021-03111-5.
收到:
公认:
发表:
关键字
- Proso millet.
- 种子形态学
- 群体遗传学
- 农业异地
- 格干
- n