抽象的
背景
枸杞林恩。茄科(Solanaceae)是一个重要的经济植物属,果实和叶子具有很高的营养价值和药用价值。然而,由于缺乏足够的分子标记,限制了该植物的遗传分析和品质改良的分子育种。
结果
在这项研究中,两个父母菌株,'宁奇1'(枸杞子L.)和'云南苟Q'(枸杞yunnanensekuang等。lu),和200 f1杂交个体已重新开始遗传分析。总共开发了8,507份良好的SNP,并且构建了高密度遗传图(NY地图),总遗传距离为2,122.24厘米。通过将纽约地图和先前公布的遗传图(NC MAP)集成含有15,240个SNP的遗传率(NC MAP)来建立共识遗传图,其总遗传距离为3,058.19cm,平均地图距离为0.21厘米。12的12个伪变质体枸杞参照基因图谱进行锚定,锚定率为64.3%。此外,该图谱与辣椒、马铃薯和番茄基因组之间存在弱共线性关系。共鉴定出25个稳定的与叶、果相关表型相关的qtl,包括果重、果度、叶长、果指数和叶指数;这些稳定的qtl定位于4个连锁群,LOD评分为2.51 ~ 19.37,表型变异解释量为6.2% ~ 51.9%。通过RNA-seq分析,188个果实相关性状稳定qtl的预测基因中有82个发生了差异表达。
结论
染色体水平的组装可以为枸杞功能基因组学的进一步研究奠定基础。这些稳定表达的qtl的基因组区域可作为进一步精细定位和开发分子标记辅助选择(MAS)的靶点。本研究为水稻产量和形态性状相关功能基因的图位克隆提供了有价值的饱和SNP标记和可靠的qtl信息枸杞spp。
背景
枸杞林恩。(茄科)为多年生灌木属,全世界有> 80种,主要分布在南美洲、北美西南部、非洲南部和东亚[1].七个物种和三个变种枸杞在中国出现[2];其中,枸杞子('Goji Berry'或中国枸杞)一直在中国西北部驯化和广泛培育> 600岁[2那3.].可食用的水果和叶子l . barbarum在中国被用作功能食品和传统中草药[4.那5.].许多化合物l . barbarum据报道,果实和叶子,包括黄酮类,类胡萝卜素和多糖,与该物种的健康增强效果密切相关[5.那6.].然而,提高质量是困难的枸杞因为其背后的分子遗传机制尚不清楚枸杞果实和叶子特征。
耦合的下一代测序(NGS)与越来越多的参考基因组序列提供了重新设计基因分型策略以进行更有效的遗传映射和基因组分析的机会[7.],可导致超高密度连锁图谱的构建和多个性状qtl的定位[8.那9.].作物全基因组序列的重测序和高密度基因定位天heading8(Dth8) 和LAX Panicle1.(LAX1)作为水稻的候选基因[10.和一种新的离子转运基因的序列改变(GMCHX1)诱导野生大豆耐盐性[11.].此外,Allotetroploid棉中报告了结构变异[12.].随着测序技术成本的降低,全基因组测序被应用于越来越多的植物物种;此外,通过将重测序数据与相应的参考基因组进行比对而开发的众多单核苷酸多态性(SNP)标记可以为破译复杂性状的遗传基础和大规模基因发现提供一种强有力的方法[13.].
第一个基于测序的连杆图枸杞使用二倍体F的特定长度扩增片段测序(SLAF-SEQ)构成1衍生自“宁奇第1”(NN)和“中国GOUQI”和18颗稳定的叶子和水果QTL之间的群体映射到所得遗传地图上[14.].最近,1,891 MB枸杞基因组序列(Cao et al.,未发表于2021年)为开发用于群体基因分型的SNP标记提供了机会。在本研究中,我们使用F1人口(图。1)枸杞与共享亲本‘宁七1号’进行农艺性状qtl定位。通过重测序和SNP鉴定进行基因分型。所得SNPs用于构建高密度连锁图谱和整合共识图谱。利用这些图谱,我们可以绘制与产量相关的qtl枸杞。这些qtl和紧密连锁标记可用于分子育种改良枸杞产量和品质。
结果
叶子和果实特质变异
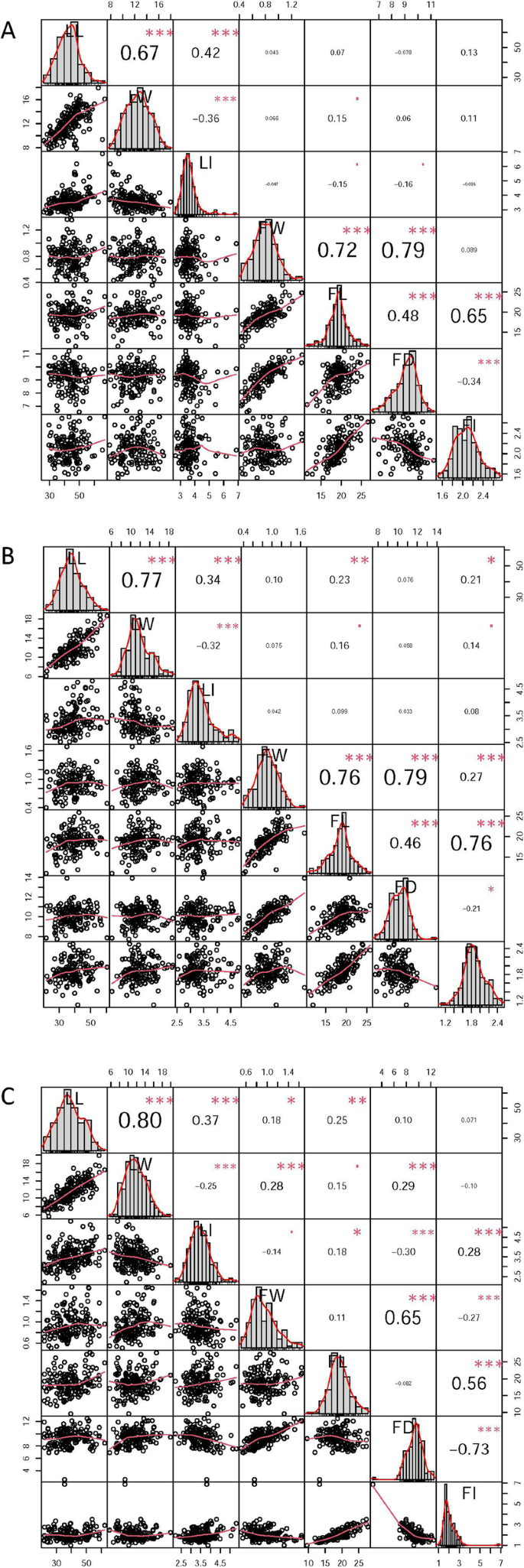
从2016年到2018年,连续三年测量了7个叶片和果实相关的表型性状。多数表型性状的变异系数为> 10%,其中2018年果实指数(FI)最高(30%),2016年果实直径(FD)最低(9%)(表1)1),表明所有性状均表现出自然变异1人口。7个性状均为正态分布或部分正态分布(表)1和图。2a - c)。相关分析表明,叶宽(LW)与叶长(LL)、叶指数(LI)与叶长(LL)、果实经度(FL)与叶长(LL)、果实经度(FI)与叶长(LL)、果实经度(LI)与果长(FI)呈显著或极显著正相关。P. < 0.05) and between single-fruit weight (FW) and FL, FW and FD, and FI and FL (P.< 0.01),而LI对与LW、LI对与FD呈极显著或极显著负相关(P.< 0.05)和FI与FD之间的差异(P.< 0.01)。2a - c)。
变量调用和注释
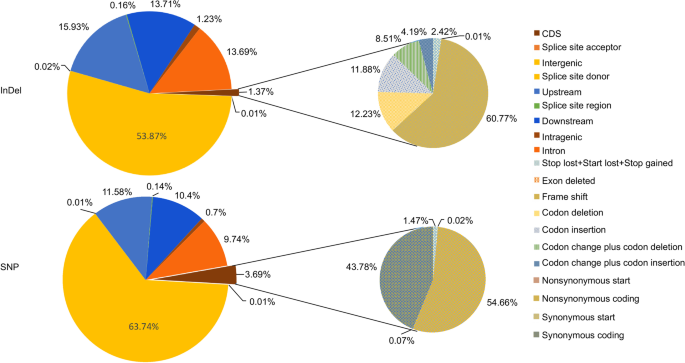
全基因组重构共计共有45.49亿干净的配对读数,女性和男性父母的23,968和227,408,000,000,000,000千升,Q30值为92.06%,表明产生了高质量的源数据。女性和男性父母的平均深度分别为34×和18倍,后代的平均深度为2.58×(补充表1).所有干净的读数都被标记在支架上枸杞参考基因组,9,015,078个SNP和1,317,594名委员会在父母之间召集。SNP和吲哚的变形图如图2所示。3.。SNP密度为4880个/ Mb, InDel密度为714个/ Mb。大多数注释的SNPs(63.74%)位于基因间区域,而在编码序列(CDS)区域,大多数SNPs是非同义的(54.66%)。与SNPs相似,超过一半的InDels(53.87%)位于基因间区域,而1.37%位于基因间区域。在CDS InDels中,60.77%出现移帧突变(图2)。3.).其中,8734,495个snp被成功划分为8个基因分型模式,3451,010个snp (aa × bb型除外)被用于构建高密度的遗传图谱枸杞。
建设超敏遗传地图和共识遗传图
为了保证一套高质量的SNP标记,SNP具有深度的父母<10×,SNP具有完整性≤70%,而且具有SD的高度显着的SNPS(P.< 0.01)。最后,利用10446个SNPs构建了枸杞高密度遗传图谱,成功整合了8507个SNPs(补充图)。1A).整合的遗传图谱包括12个连锁群(LGs),总遗传距离为2,122.24 cM,平均图谱距离为0.25 cM。LG07标记数最多,共1035个,平均遗传距离为0.18 cM,总遗传距离为182.59 cM。LG03区标记数最少(510个),长度为119.69 cM,平均距离为0.24 cM。最大的差距在LG11中达到19.41 cM。LG05和LG02中相邻标记< 5 cM的遗传距离比分别为98.19% ~ 99.83%2).进一步整合我们已出版的地图[14.]构建共有遗传图谱,其含有15,240个SNP,总遗传距离为3,058.19cm,平均地图距离为0.21厘米(表2和补充图。1B).与NY图谱相比,该图谱的SNPs多6733个,平均距离少0.04 cM,分辨率较高,可能是木质植物中SNP标记数量最多的遗传图谱。此外,最大差距也有所缩小。协商一致的地图代表了全面的改进。
遗传地图辅助基因组组装
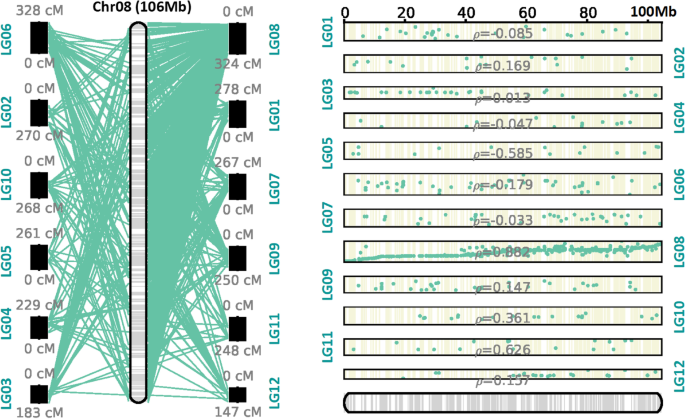
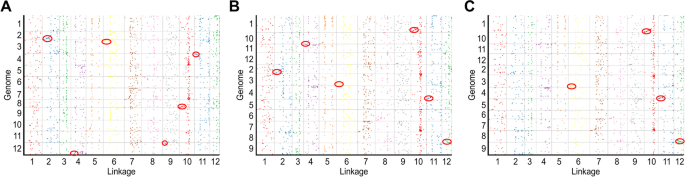
高密度连杆地图可以有助于染色体级基因组组件。组装基因组枸杞在染色体水平的基础上,我们使用了所有的高密度共识遗传图。最后,将〜1.21 GB的支架安装在12个伪染色体中枸杞,占基因组序列的64.3%,其中51.3%为定向序列(图。4.;补充图。2;表格3.).最长的假染色体为LG10,总长度为132.33 Mb,而LG05和LG01上的假染色体分别只有57 Mb和57.69 Mb,与遗传图谱SNP数趋势一致。未安装支架较多(783,254),其中< 1 kb支架占99.3%(777,737/783,254)。ALLMAPS支架是通过推断和最大化基因图谱与scaffold /contigs之间的共线性来进行的。通过比较所有LG染色体和假染色体之间的共线,我们发现每个LG染色体和相应的假染色体之间存在一定的共线。假染色体04最低P.价值。而且,这是P.共线拷贝瘤瘤素01,02,08,09和11的值均为0.8(补充图。2).
同线性分析
我们使用共识遗传图谱与辣椒,马铃薯和番茄的参考基因组进行联合性分析。在枸杞和三种溶那种物种之间发现了薄弱的共同性,并且位于染色体末端的大多数区域显示出高的共同性。枸杞和辣椒之间的共线段对是Lg02-染色体02,Lg04-染色体12,Lg06-染色体03,Lg09-染色体12,Lg10-染色体08和Lg11-染色体04.枸杞和马铃薯的共线段对是Lg02-Chromosome 02,Lg06-染色体03,LG04-染色体11,G10-染色体01,LG11-染色体04和LG12-染色体08.枸杞和番茄的共线段对是LG06-染色体03,LG10-染色体01,LG11-Chromosome 04和LG12-染色体08(图。5.).其中,枸杞-辣椒对和枸杞-番茄对在第02、04、06、10和11号染色体上存在重叠,而枸杞-番茄对和枸杞-马铃薯对在第06、10、11和12号染色体上存在重叠。此外,在枸杞10号染色体上,枸杞-番茄对和枸杞-马铃薯对之间可能存在染色体倒转。
QTL定位
从2016年到2018年使用Resequeccing Genetic Map和连续表型数据,映射了大量负责七个农艺性状的QTL。QTL分布在除LG08之外的所有LGS的枸杞中,具有从6%至73.6%的表型方差解释(PVE)值,最大LOD值为21.39。果实相关的QTL主要映射到LG06,LG07,LG09,LG10和LG12,而叶片相关的QTL主要位于LG01,LG06,LG09和LG10(补充表2).
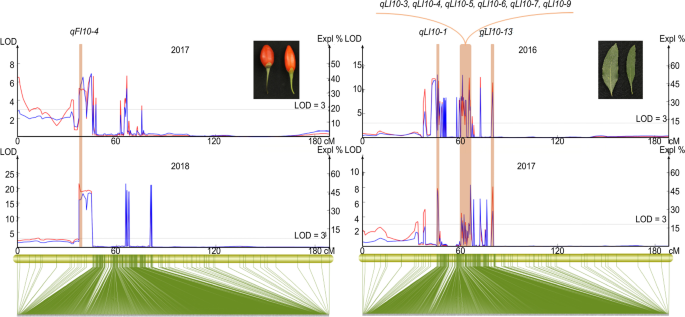
我们进一步筛选了至少2年检测到的稳定qtl,共鉴定出25个qtl (Supplementary Table)3.).共鉴定了13个与叶指数(LI)相关的qtl,这些qtl均位于45.967 ~ 73.823 cM (27.856 cM)的LG10区,其中44个snp位点。LOD最大(13.13),PVE最大(50.5%)QLI10-4和qLI10-12分别(图。6.).LG10中的一些LI QTLs被紧密地收集,平均间隔小于0.63cm,指示这些QTL可能属于相同的QTL(图。6.).对于LL,两个QTL(qLL9-1和qLL9-2)被定位到LG09,有16个snp支持。1个稳定的FW QTL锚定在LG10上,PVE值为59.2%,但有一个标记支持。两个稳定的FL qtl分别锚定在LG10和LG12 (QFL10和QFL12.)九个显着相关的SNP,解释表型变异的7.3%至36.9%(补充表3.).五法(qFI10-1~qFI10-5)在LG10的37.344〜66.309厘米处占PVE的15.8%至51.9%,在这些QTL区域中具有最高的LOD值(超过5.0)。尤其,qFI10-4在2017年和2018年多次检测到(图3)。6.).两个稳定的QTL(qFI7 - 1,qFI7-2)对于LG07的60.634〜61.916厘米的FI,分别占PVE的8.5%和19.3%,平均接口距离为0.18厘米。在稳定的QTL中,我们在标记上游和下游的150 kB内提取预测基因。为了进一步探讨果实相关性状的稳定QTL的表达,我们进行了RNA-SEQ,发现188个预测基因中的82例显示了差异表达(补充表4.).这些基因为进一步的基因克隆和标记辅助选择提供了宝贵的资源。
讨论
一个高密度的遗传图谱可以为破译重要和复杂农艺性状的遗传基础提供有价值的信息。随着测序技术的快速发展,SNPs和InDels在高密度连锁图构建、QTL解剖、候选基因发现和育种应用等方面得到了广泛的应用[10.那15.那16.].双亲本分离群体SNP基因分型全基因组重测序已成功应用于植物[17.那18.那19.].因为我们完全重新组装的基因组序列枸杞(Cao等人,未发表的2021),已经可以使用全基因组重构用于基因组的SNP发现和高密度遗传地图建设枸杞。本研究通过全基因组重测序,共获得9,015,078个SNPs和1,646,131个InDels,构建了包含8,507个SNPs的高密度遗传图谱,分布范围为2,122.24 cM,相邻标记间平均距离为0.25 cM1人口(纽约地图)。而lg11的最大间隙为19.41 cM。这表明该区域存在重组或未发育的标记[20.].与第一个序列的基于遗传图谱相比枸杞我们构建了[14., F的个数1个人减少了100(第一次含有302个人的人口中的数量),而平均距离与公布的遗传图谱非常相似。然而,基于ReseRequeccing的连杆地图的SNP总数和总图距离较高,表明重新排序的较高分辨率比降低表示测序更高。通过整合这两个高密度图开发的共识图表明已经获得了饱和的遗传图枸杞。
高密度遗传图可以改善染色体水平的Contig /脚手架组件[21那22那23].一般来说,遗传图谱可以将所有组装的contigs/scaffold的60-90%分布在假染色体上[24那25那26].随着标记密度的增加,共识遗传图谱的分布率也会相应增加[23那27].在这项研究中,一个高密度的基因图谱枸杞是在重测序的基础上构建的,将高密度基因图谱与之前发表的图谱进行整合,形成共识图谱[14.].最终的遗传图谱包含12个LGs中的15240个标记。根据这张基因图谱,支架枸杞基因组(CaO等,未发布的2021)安装在12个假瘤组上,分配速率为64.30%,相对较低。有783,254个未安装的支架,锚固支架的数量为476.72倍。此外,未取消的支架的平均长度为0.86kb。因此,我们推测碎片化的基因组组件是分配率低的原因。为了枸杞在本研究中使用的基因组,分配率可以受益于PACBIO长读测序的使用[28],Bionano光学图[24]或Hi-C技术[29].
为了深入了解枸杞的进化史,我们对枸杞、番茄、辣椒和马铃薯的基因组进行了共线性分析。与先前的结果相似[14.]枸杞的遗传图谱总体上与这三种基因组显示出低的共同性,表明枸杞的更大分化。然而,在某些情况下观察到高共同性,例如酚醛树脂和枸杞对枸杞和枸杞 - 番茄对的染色体02,04,06,10和11种的区段,表明这些间隔在不同种类的Solanaceae中受到高度保守。此外,随着更多标记物意味着与相应的参考基因组序列对准的标记的更大可能性,标记密度与共同性有关。高密度遗传图上的标记通常通过减少陈述测序来开发[14.那30.或重新排序[17.那18.那19.那31,可以产生数千个随机分布的标记。但植物的参考基因组中总有至少数百个Mbp。因此,利用基于测序的高密度遗传图谱只能检测到保守区域的共线性,我们认为利用枸杞基因组组装可以实现全面的基因组共线性。
果和叶是主要的药用和食用部分枸杞spp。32].在我们的研究中,许多QTLS(补充表2),根据3年多的表型数据,对果实和叶片性状进行了鉴定。多数性状定位在2个以上的qtl上,FI的qtl定位在LG04、LG09、LG11和LG12的不同位置。在LG10中,FW、FL和FI的qtl定位在37.344 cM, LOD和PVE分别达到16.02和62.5%。这些结果表明,果实和叶片的性状枸杞是由多基因座和具有多效效应的单基因座控制的[33].
稳定的qtl对于基于mass的育种计划是有价值和有用的,并且已经在多年生植物中被鉴定出来[34].本研究在LG07、LG09、LG10和LG11中鉴定了25个与2个叶片和3个果实性状相关的稳定qtl3.),其中4个在2个LG10区(38.6 cM和66.3 cM), LOD值较高(15.7 ~ 45.3),与3个性状(LI、FW和FI)相关。此外,在之前的一项研究中[14.[FW的两个稳定QTL位于与FD的两个稳定QTL相同的两个LG区域(133.6厘米和146.4cm),这可能表明这些稳定的QTL(qLI10-10那qFW10那qFI10-2,和qFI10-5)在不同的环境中显示出高可靠性,并且应该被视为主要的QTL。注意,鉴于这些主要QTLS,FW和FI之间的表型相关性存在遗传基础,这与表型分析中的两个特征之间观察到的强相关(图。1a - c)。因此,我们推测LG10中有两个区域(38.6 cM和66.3 cM)在调控中起着关键作用枸杞果实生长发育。这些qtl的snp位点可以用剩余的100个F1这些紧密连接的SNPs可以转化为竞争性等位基因特异性PCR (KASP)标记,并有可能作为早期选择标记。
方法
定位群体构建和表型
由“宁杞1号”(NN)衍生的杂交枸杞群体(l . barbarumL.)和云南勾棋(YG) (枸杞yunnanensekuang等。Lu)于2014年8月生成。女性父母NN是中国西北部的一个主要的人工育种品种。它的果实是鲜艳的红色和椭圆形,味道鲜艳,叶子是披针形。雄性父母Yg是一种野生型枸杞,具有深红色,长,椭圆形,苦味的果实(图。1).F的种子1杂交和两名父母在宁夏农业和林业科学院校(38°38°N,106°90 e),中国宁夏,中国宁夏,中国宁夏,中国,中国宁夏院校播种和播种。总共,300 f1个体被培养,随机选择200个来建立地图种群。水肥管理与生产现场相同。杂草是手工管理的。
叶片和果实相关性状在F1人口(纽约)和两个父母。LL是叶底座和尖端之间的最大距离。LW是叶子的最宽的距离。FW是一个成熟果实的重量。FL是水果顶部和底部之间的最大距离。FD是水果中最宽的距离。根据其他地方描述的方法测量LL,LW,FW,FL和FD [35].LL、LW、FL和FD使用游标卡尺测量,而FW使用电子天平(SE602F, Ohaus, NJ, USA)。LI和FI根据式(i)和式(ii)计算:(i) LI = LL/LW [35(ii)fi = fl / fd [35].利用连续3年(2016 - 2018年)从每棵树采集的30片叶子和果实获得表型数据。每个性状的平均值用于QTL分析。采用SPSS V17.0软件(SPSS Inc., Chicago, IL, USA)进行复杂方差分析、方差分析和相关分析。
群体重测序和基因分型
从父母和200岁的幼叶中提取基因组DNA1植物使用超级植物基因组DNA DP360试剂盒(天根生物技术,北京,中国)。用NanoDrop分光光度计(ND2000, Thermo Fisher Scientific, USA)测定DNA浓度,用0.85%琼脂糖凝胶电泳监测DNA质量。基因组DNA用S2/E210超声仪(Covaris, USA)剪切成350bp的片段。然后将纯化后的产物连接进行末端修复,添加3'A和适配器,并根据碎片大小在1%琼脂糖凝胶上进行选择。通过PCR构建最终文库。文库定量使用Agilent 2100 Bioanalyzer (Agilent Technologies, Palo Alto, CA, USA),文库片段根据制造商的建议,使用Illumina XTen平台(Illumina, San Diego, CA, USA)进行配对端测序(PE125)。支持本研究结果的数据已存入中国国家基因数据库CNGB序列档案(CNSA),登录号为CNP0001536。
过滤原始读取以产生高质量的清洁读取(i)去除适配器序列,(ii)用> 10%未识别的核苷酸滤波读数,(iii)除去具有低验证质量得分的> 50%碱基的读数(≤ 10). Burrows-Wheeler Aligner [36]用来将干净的读数与枸杞基因组(Cao等人,未发表于2021年),使用Picard识别重复(Picard:http://sourceforge.net/projects/picard/).SNPs和插入和删除(InDels;使用GATK软件调用[37],然后由SnpEff注释[38].Circos绘制了SNPs和InDels的基因组变异图[39].采用双等位基因编码规则和8种基因分型模式(aa × bb、ab × cd、ef × eg、hk × hk、lm × ll、nn × np、ab × cc、cc × ab)对SNP基因型进行编码。选择除aa × bb外的所有模式构建杂交授粉(CP)群体高密度遗传图谱。
遗传联系地图结构和QTL映射
进一步筛选所得的SNP。具体而言,HK×HK和NN×NP分离型SNP,父母的深度<6倍,完整性<60%,和显着的分离失真(SD)(Chi-SquareP. < 0.05) were filtered out, whereas the remaining segregation types with a depth < 10 × in the parents, an integrity < 70%, and highly significant SD (chi-squareP.< 0.01)。SNP标记根据两两修正对数比值(MLOD)评分被排列成连锁组(LGs)。MLOD评分> 5的标记被分配到单个LG。平滑算法(40用来纠正基因型,然后k-collect邻近方法[41]用于基因型的输入。利用JoinMap软件(V4.1)的CP种群类型映射功能,在LGs内进行线性排列。使用Kosambi地图功能估计地图距离[42].利用R (www.r-project.org/).QTL分析采用MapQTL V6.0结合区间映射(IM)模型[43, LOD阈值设置为2.5。临界值为> 3.0和PVE > 10%的qtl为主效qtl。
基因图谱辅助的基因组支架组装和基因组同源性分析
已发表的遗传图(NC地图)的SLAF标记[14.[本研究中鉴定的SNP对齐枸杞脚手架级基因组装配(Cao等,未发表的2021)使用blat软件[44].根据SLAF和SNP标记的位置提取这两个LG的相应关系和共享标记。使用BioMercator V4.2构建综合遗传图[45].这个整合的基因图谱被用来固定支架枸杞在染色体水平上使用ALLMAPS软件的基因组[46].将snp整合的遗传图谱与茄科近亲辣椒(http://peppersequence.genomics.cn/page/species/download.jsp.jsp.jsp.#5),番茄(ftp://ftp.solgenomics.net/tomato_genome/assembly/build_2.50/)和土豆(http://solanaceae.plantbiology。msu.edu /pgsc_download.shtml),使用BLAT [44]和同源序列的物理位置在R中生成共线性图。
RNA-SEQ分析
两种父母的果实在授粉后36-40天进行了一式三份,用于总RNA提取[RNAPREP纯植物套件(天根生物技术,北京,中国)]。RNA-SEQ图书馆根据[47];然后将库加载到群体生成的CBOT中,并在Illumina Novaseq平台(Illumina,San Diego,Ca,USA)上进行150-BP配对读取测序。在内部Perl脚本中删除了低质量读取[> 20%的Q值≤20或模糊序列内容('n')的碱基含量超过5%]。然后使用Star映射到Wolfberry(Cao等,未发表的2021)的参考基因组的清洁读数使用默认设置[48].StringTie被用来收集转录本[49].每百万映射的读数(FPKM)方法每千碱基的片段用于定量转录物表达水平,并且使用DESEQ R包来检测差异表达的基因(DEGS)。使用调整后识别出显着的DEGP.值< 0.05,折变≥2。
数据和材料的可用性
所有测序干净数据上传到中国国家基因库数据库(CNP0001536)。然而,这些数据将于2022年4月1日公布。在2022年4月1日之前,通信作者可根据合理要求提供数据集。
缩写
- 单核苷酸多态性:
-
单核苷酸多态性
- QTLS:
-
数量性状位点
- LOD:
-
概率的对数
- 神经网络:
-
'宁奇第1号'
- YG:
-
“云南枸杞子”
- NC:
-
“宁棋1号”ד中国围棋”
- 纽约:
-
'宁齐第1号'×'yunnan gouqi'
- 马斯:
-
分子标记辅助选择
- ngs:
-
新一代测序
- 二:
-
叶片长度
- LW:
-
叶宽度
- 李:
-
叶指数
- 弗兰克-威廉姆斯:
-
单瓜重
- fl:
-
水果经度
- FD:
-
水果直径
- FI:
-
水果指数
- cd:
-
编码序列
- LGs:
-
联系团体
- SD:
-
隔离失真
- 通过:
-
表型方差解释了
- 凯斯克:
-
Kompetitive allele-specific PCR
- CNGBdb:
-
中国国家Genebank数据库
- CP:
-
交替地
- MLOD:
-
修正的几率对数
- FPKM:
-
每百万百万映射的成绩单每千碱基映射的碎片
- 可见:
-
差异表达基因
参考文献
- 1。
王志强,王志强,王志强,等。龙葵属植物的系统发育与生物地理学枸杞(Solanaceae):叶绿体DNA序列推断。mol phylocyet evol。2001; 19:246-58。https://doi.org/10.1006/mpev.2001.0921。
- 2.
赵XQ,郭某,闫H,陆,张F,Qian DW等。叶片中酚酸和黄酮类化合物的分析枸杞子通过超高效液相色谱法从不同的栖息地加上三重四极杆串联质谱法。生物化色谱。2019; 33:E4552。https://doi.org/10.1002/bmc.4552。
- 3.
陈静,刘旭,朱丽,王艳。核基因组大小估算及核型分析枸杞物种(Solanaceae)。SCI Hortic。2013; 151:46-50。https://doi.org/10.1016/j.scienta.2012.12.004。
- 4.
肖旭,任伟,张宁,刘晓涛,赵志,等。对黄芪果实、叶、根皮提取物的化学成分及生物活性进行了比较研究枸杞子。分子。2019;24:1585。https://doi.org/10.3390/molecules24081585。
- 5.
王传聪,张SC, Inbaraj BS,陈炳辉。类胡萝卜素、黄酮类化合物和多糖的分离枸杞子L.抗氧化活性的评价。食品化学。2010; 120:184-192。https://doi.org/10.1016/j.foodchem.2009.10.005。
- 6。
Pedro AC, Maurer JBB, Zawadzki-Baggio SF, Ávila S, Maciel GM, Haminiuk CWI。有机枸杞(枸杞子L.)防止大豆油的氧化劣化。indacrops prod。2018; 112:90-97。https://doi.org/10.1016/j.indcrop.2017.10.052。
- 7。
黄X,冯Q,钱Q,赵Q,王L,Wang A等。全基因组重新排列的高通量基因分型。Genome Res。2009; 19:1068-76。https://doi.org/10.1101/gr.089516.108。
- 8。
周强,苗华,李胜,张胜,王勇,翁勇,等。基于测序的黄瓜连锁图谱。摩尔。2015;8:961-3。https://doi.org/10.1016/j.molp.2015.03.008。
- 9。
邹刚,翟刚,冯强,严胜,王安,赵强,等。基于高通量测序的高粱8个重要农艺性状的qtl定位J Exp Bot. 2012; 63:5451-62。https://doi.org/10.1093/jxb/ers205。
- 10。
高志勇,赵长生,何文敏,郭立波,彭玉玲,王建军,等。通过重组自交系和改进亲本基因组序列分析超级杂交稻产量相关基因座。美国国立科学院。2013; 110:14492-7。https://doi.org/10.1073/pnas.1306579110。
- 11.
齐X,Li MW,谢米,刘X,Ni M,Shao G等。全基因组测序鉴定野生大豆中新型盐耐受基因。NAT Communce。2014; 5:4340。https://doi.org/10.1038/ncomms5340。
- 12.
王胜,陈建军,张伟,胡勇,常磊,方磊,等。基于序列的超密遗传和物理图谱揭示了异源多倍体棉花基因组的结构变异。基因组医学杂志。2015;16:108。https://doi.org/10.1186/s13059-015-0678-1。
- 13.
Han K,Lee Hy,Ro Ny,Hur Os,Lee Lj,kwon JK等。QTL映射和GWAS揭示控制胶囊素内容的候选基因辣椒。植物生物技术学报2018;16:1546-58。https://doi.org/10.1111/pbi.12894。
- 14.
赵军,徐勇,李华,尹勇,安伟,李勇,等。基于snp的枸杞叶片和果实相关数量性状高密度遗传图谱(枸杞林。)。前植物SCI。2019; 10:977。https://doi.org/10.3389/fpls.2019.00977。
- 15.
Pandey Mk,Monyo E,Ozias-Akins P,Liang X,GuimarãesP,Nigam Sn,等。花生改善的阿拉氏基因组学进展。Biotechnol Adv。2012; 30:639-51。https://doi.org/10.1016/j.biotechadv.2011.11.001。
- 16。
番茄性状的发现与编辑。植物j . 2019; 97:73 - 90。https://doi.org/10.1111/tpj.14152。
- 17。
李旭,魏勇,阿查雅,姜强,康健,Brummer EC。同源四倍体紫花苜蓿(Medicago Sativa.L.)利用基因分型测序开发的,与Medicago truncatula基因组。G3-Genes Genom Genet. 2014; 4:1971-1979。https://doi.org/10.1534/g3.114.012245。
- 18。
胡z,邓g,mou h,xu y,chen l,yang j等。基于重新排序的超密集遗传图谱揭示了一种胶粘茎枯燥的抗性相关基因Cucumis Melo.。DNA研究》2018;25:1-10。https://doi.org/10.1093/dnares/dsx033。
- 19。
王洪波,王洪波,王洪波,等。利用全基因组重测序的高密度遗传图谱精细定位和候选基因发现花生抗病。植物生物技术学报2018;16:54 - 67。https://doi.org/10.1111/pbi.12930。
- 20.
王勇,陆军,于建军,于飞。在群体NGS数据中精确的基因型/单倍型推断的综合变异分析管道。基因组研究》2013;23:833 - 842。https://doi.org/10.1101/gr.146084.112。
- 21.
赵立,Yuanda L,Paiping C,Xiangchao T,Xiangdong C,Wei Z等。朝向同种异体四倍体棉基因组组装:具有DNA序列信息的高密度分子遗传键合图的整合。BMC基因组。2012年; 13:539。https://doi.org/10.1186/1471-2164-13-539。
- 22。
王磊,夏强,张勇,朱旭,朱旭,李东,等。更新的芝麻基因组组装和利用新的高密度遗传图谱精细定位株高和种皮颜色qtl。BMC染色体组。2016;17:31。https://doi.org/10.1186/s12864-015-2316-4。
- 23。
Bernhardsson C,Vidalis A,Wang X,Scofield DG,Schiffthaler B,Baison J等人。一种用于评估挪威云杉的高碎片基因组组装的超致密单倍体遗传图(Picea amies).G3-基因基因群。2019; 9:1623-1632。https://doi.org/10.1534/g3.118.200840。
- 24。
杨建军,刘东,王旭东,纪超,程飞,刘波,等。异源多倍体的基因组序列芸苔属植物juncea分析差异同源性基因表达影响选择的分析。NAT Genet。2016; 48:1225-32。https://doi.org/10.1038/ng.3657。
- 25.
Hane JK,Ming Y,Kamphuis Lg,Nelson Mn,Garg G,Atkins Ca等。羽扇豆的全面基因组序列草案(卢比斯angustifolius),一种新兴的保健食品:深入了解植物-微生物相互作用和豆科植物进化。生物技术学报2017;15(15):318-30。https://doi.org/10.1111/pbi.12615。
- 26.
Lee by,Kim Ms,Choi Bs,Nagano Aj,Au DWT,Wu RSS等。基于高分辨率的基于Rad-SEQ的连杆地图,锚定参考基因组和QTL在海绵中染色体的QTL映射oryzias melastigma.。G3-Gene Genom Genet. 2019; 9:3537-3545。https://doi.org/10.1534/g3.119.400708。
- 27.
薛H,王某,姚杰,邓Ch,王L,Su y等。染色体水平高密度综合遗传图改善了Pyrus Bretschneideri.'dangshansul'v1.0基因组。BMC基因组。2018; 19:833。https://doi.org/10.1186/s12864-018-5224-6。
- 28。
Hazzouri公里,Gros-Balthazard M,鲜花JM,Copetti D,Lemansour A,Lebrun M等人。枣椰子果实性状的基因组关联映射。NAT Communce。2019; 10:4680。https://doi.org/10.1038/s41467-019-12604-9。
- 29。
张QJ,Li W,Li K,Nan H,Shi C,Zhang Y等。茶树的染色体级参考基因组推出了促进基因组大小演化中的非自主LTR回收分支突发。莫尔植物。2020; 13:935-8。https://doi.org/10.1016/j.molp.2020.04.009。
- 30.
Geleta M,Gustafsson C,Glaubitz JC,Ortiz R.高密度遗传联系映射Lepidium基于基因分型逐序列的SNP和分离的Contig标签单倍型。前植物SCI。2020; 11:448。https://doi.org/10.3389/fpls.2020.00448。
- 31.
童志,周军,修志,焦飞,胡勇,郑飞,等。高密度基因图谱的构建与全基因组测序烟草l .基因组学。2020;112:2028-33。https://doi.org/10.1016/j.ygeno.2019.11.015。
- 32.
董建忠,卢元元,王艳。栽培叶中黄酮类化合物的分析枸杞子acta botanica sinica(植物营养学报),2009;54(4):489 - 494。https://doi.org/10.1007/S11130-009-0128-X.。
- 33.
Eduardo I, Pacheco I, Chietera G, Bassi D, Pozzi C, Vecchietti A, et al.;两个桃种内群体果实品质性状的QTL分析及成熟期多效效应的重要性树geneet Genom. 2011; 7:323-35。https://doi.org/10.1007/s11295-010-0334-6。
- 34。
潘y,梁x,gao m,liu h,孟h,weng y等。Wi7239黄瓜中的圆形果实由两个相互作用的定量特性基因座控制,一个巨大的编码番茄太阳同源物。Al Appl Genet。2017; 130:573-86。https://doi.org/10.1007/s00122-016-2836-6。
- 35。
Shi Z,Du H,Men H. Goji种质资源描述标准化和数据标准。北京:中国林业出版社;2012年。
- 36。
基于burrowwheeler变换的快速、准确的短读对齐。生物信息学。2009;25:1754-60。https://doi.org/10.1093/bioinformatics/btp324。
- 37.
等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20 l1297 - 1303。https://doi.org/10.1101/gr.107524.110。
- 38.
王丽丽,王丽丽,王丽丽,等。用于注释和预测单核苷酸多态性的影响的程序,SnpEff:在基因组中的SNPs黑腹果蝇应变w1118;iso-2;iso-3。飞(奥斯丁)。2012; 6:80 - 92。https://doi.org/10.4161/fly.19695。
- 39.
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D等人。电讯:对比较基因组学的信息审美。Genome Res。2009; 19:1639-45。https://doi.org/10.1101/gr.092759.109。
- 40.
van Os H, Stam P, Visser RG, van Eck HJ。SMOOTH:一种从高密度遗传连锁数据中成功去除基因分型错误的统计方法。Theor Appl Genet. 2005; 112:187-94。https://doi.org/10.1007/s00122-005-0124-y。
- 41.
黄X,赵y,魏x,李c,王a,zhao q等。全球稻草种质收集开花时间和粮食产量的基因组关联研究。NAT Genet。2012; 44:32-9。https://doi.org/10.1038/ng.1018。
- 42.
范Ooijen JW。JoinMap 4:计算实验群体遗传连锁图的软件。3 .王志刚,2006;33:10-1371。
- 43.
范Ooijen JW。MapQTL 6.0。二倍体种实验居群数量性状位点作图软件。卡亚兹玛BV,瓦赫宁根,NL, 2009,第59页。
- 44.
肯特WJ。Blat-爆炸式对准工具。Genome Res。2002; 12:656-64。https://doi.org/10.1101/gr229202。
- 45.
Arcade A, Labourdette A, Falque M, Mangin B, Chardon F, Charcosset A等。BioMercator:整合遗传图谱和QTL以发现候选基因。生物信息学。2004;20:2324-6。https://doi.org/10.1093/bioinformatics/bth230。
- 46。
唐华,张旭,苗超,张军,明锐,Schnable JC,等。ALLMAPS:基于多个映射的健壮脚手架排序。基因组医学杂志。2015;16:3。https://doi.org/10.1186/s13059-014-05731.。
- 47。
关键词:匍匐茎;转录组分析郁金香属可食的。前植物SCI。2016; 7:409。https://doi.org/10.3389/fpls.2016.00409。
- 48。
张志强,张志强,张志强,等。STAR:超快通用rna测序仪。生物信息学。2013;29:15-21。https://doi.org/10.1093/bioinformatics/bts635。
- 49.
ertea m,pertea gm,antonescu cm,chang tc,mendell jt,salzberg sl。Stringtie使得能够从RNA-SEQ读取改进转录组的重建。NAT BIOTECHNOL。2015; 33:290-5。https://doi.org/10.1038/nbt.3122。
资金
这项工作由中国国家自然科学基金会赞助(32060359,31660220);宁夏辉自治区科技创新领先人才项目(KJT2017004);宁夏自治区农业育种专项基础(2013英亩)。
作者信息
从属关系
贡献
ZJ和AW构思和设计了实验;LH,YY,HT和ZB执行了f1种群建设与表型;WY和LY进行RNA分离和RNA-seq数据分析;XY进行了植物群体遗传分析、遗传图谱分析和QTL定位;ZJ和CY撰写稿件,ZJ对稿件进行了修改。所有作者都已阅读并批准了最终稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
赵,J.,Li,H.,徐,Y.et al。一个一致和饱和的遗传图谱提供了深入了解茄科和枸杞叶和果实相关qtl的基因组锚定、共同性(枸杞林)。BMC植物杂志21,350(2021)。https://doi.org/10.1186/s12870-021-031151.
收到了:
公认:
发表:
关键字
- 遗传地图
- 重新排序
- 与叶子和果实相关的性状
- QTL
- Genome Synteny.
- 枸杞Linn.