摘要
背景
常见的bean (菜豆L.)是一种豆类作物,其谷物可以储存数月,这是巴西种植者的一种常见做法。随着时间的推移,种皮的颜色会变深,也更难煮熟,这对消费者来说是不受欢迎的,他们认为颜色越深的豆子越老。像商业品托豆和蔓越莓豆品种一样,在收获季节和储藏后种子颜色较深的可可豆,其市场价值会下降。
结果
我们的研究目的是鉴定与收获(HL)的种子涂色的轻度相关的遗传控制,并通过基因组 - 宽的协会研究对收获后种子涂层变暗(PHD)的耐受性。为此目的,先前验证用于关联映射研究的Carioca多样性专家组与138种基因型和1,516个高质量的SNP一起使用。使用色度计和CIELAB规模在两个环境中评估面板。30天的货架储存具有最有表现力的结果,L *(亮度)参数导致最大的基因型辨别。用于HL,两种染色体PV04和PV10上的QTL鉴定了三个QTL。关于PHD,结果表明,30天后L *的遗传控制与ΔL*(最终L * -INITIAL L *)不同;只有ΔL*能够正确表达博士学位。提出了四种表型类,通过六个重要的SNP来确定五个QTL。
结论
收获的种子涂层的亮度显示通过中等宽视遗传性和实验中的高基因型相关性进行了抑制性遗传。只有三个QTL对于这种特性很重要 - 两个在PV04上映射到PV10上。考虑到ΔL,六个QTL被映射到四种不同的PHD染色体上。PV10开头的相同HL QTL也与ΔL*相关联,并且可以用作标记辅助选择的工具。鉴定了几种候选基因,可用于加速遗传育种过程。
背景
常见的bean (菜豆L.)被认为是生产食用干燥种子的最重要的种类,用于人类饮食中的直接消费[1那2].这种谷物的固有营养价值及其潜在的健康益处解释了这种豆类作为碳水化合物、纤维、维生素和矿物质来源的营养价值[3.],以及具有抗氧化特性的多酚化合物[4.].在一些非洲和美洲国家,豆类平均提供每日总热量的15%和蛋白质消耗的36% [5.].
自2012年以来,全球干豆产量增长了77.8%,2017年达到创纪录的3150万吨[6.].人口研究表明世界人口的大幅增长,预计未来几年将继续增加豆消费[2].世界干豆生产主要集中在亚洲和美洲国家,合计约占世界干豆总产量的75% [6.].巴西目前是该粮食的第三大生产商,负责南方国家生产的75%,占世界生产的10.4%。此外,巴西被认为是这种食物的最大消费者,平均消费18千克−1人均(7.].
在大多数国家,对某种谷物的需求因地区文化的不同而不同。产于巴西的一种可可豆,其特点是种皮呈奶油色,带有棕色条纹。8.,属于中美洲基因库[9.,占所食用豆类种类的70% [10.那11].据报道品托和蔓越莓品种[12那13, PHD被认为是收获后最有可能使产品贬值的特性[14那15].巴西“里约卡”品种在收获时种皮颜色较深的品种附加值低于种皮颜色较浅的品种,因为消费者会将颜色较深的种皮与较长的烹饪时间和老豆联系起来[8.那16那17].然而,这种普遍的观念是错误的,因为较深的谷物并不总是意味着较长的烹饪时间和/或较老的豆子[15那18].
种子涂层颜色和图案化特征由复杂的遗传网络控制[19].普通豆类颜色多样,由于上位性相互作用、多效性效应、多等位性和连锁基因的存在,控制遗传是困难的[20.].PHD通常分为三类表型:(1)非变暗(ND),(2)缓慢变暗(SD)和(3)常规变暗(RD)。Junk-Knievel等人[21发现单个基因控制了在分型豆类中的基因型是否是SD或Rd,具有Rd的优势。由于SD特征在种子涂层中表达,即母体组织,应考虑母体效果,具体取决于表型的产生[13].根据Elsadr等人[22),J基因决定谷物是否变暗(J) 或不 (JJ.),sd基因预测变暗发生的速度(隐性上位性)。Erfatpour等[12)映射sd和nd位点在蔓肾红莓豆类染色体PV07和PV10上。
早期的晶粒变暗与几种遗传,环境和后收获因子相互作用,并且由于湿度条件,谷物的干燥时间和尤其是储存条件而变暗可能会加剧[15那23那24].PHD归因于原花青素积累及其随后的种子氧化[25那26].这些化合物氧化形成活性醌,沉积在细胞环境中,导致这一层变暗[25那27].它们在豆类中的较高浓度呈正常变暗,而不是在变暗缓慢的人中[27].
关于carioca品种,Silva等人[16]提出了一种评价种子变暗的量表,并认为PHD的控制似乎是单基因的。Silva等人[10.发现该特征受基因型×环境相互作用的强烈影响。作者还表明该特征在弥合性控制之下;然而,观察到的分离模式的不一致性显示了更大程度的复杂性。Alvares等。[28]确认基因型×环境相互作用,发现评估的基因型在测试的各种环境中没有表现出恰逢反应。库托等人[29在分离Carioca群体中鉴定了与PPD QTL相关的三种微卫星标记物。Alvares等。[23鉴定了先前映射的PVSD-1158微卫星标记链接sd轨迹在斑豆中[13,紧密相连轨迹控制可可豆的缓慢变黑。然而,在全基因组标记的定位研究中,没有包括对可可豆品种PHD的有效评价,也没有报道收获时谷物颜色的亮度。
因此,目前研究的目的是使用基因组宽联型方法鉴定与PPD和HL耐受相关的Carioca基因组区域。为此目的,以前验证的GWAS的CARIOCA多样性面板(CDP)在两种环境中表现出了两种环境和基因分型,使用高通量基因分型技术。
结果
表型数据
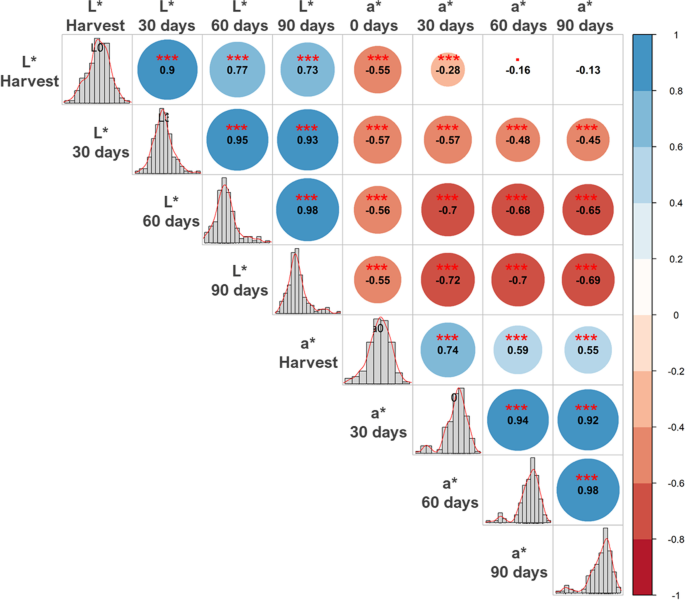
从第一实验的三个评估周期获得的L *和*参数的表型数据用于选择与其他实验的最高相关性的最短储存时间。Pearson相关性分析显示所有参数和时间之间的相关性,除了L * Harvest VS A * 60天和A * 90天之间(图。1).如预期的那样,同一参数的次数之间的相关系数高于不同参数的次数之间的相关系数。L*和a*的相关性虽然显著,但均为负相关,因为L*和a*的值越高,表示样本颜色越淡,红色越少。
在贮藏期方面,30天的贮藏时间最短,相关性均大于90%,为减少第二次试验所需的天数,选择30天的贮藏时间。在相同的贮藏时间下,L*与a*的相关值随时间的增加而增加,从收获时的55%开始,90天后达到69%。然而,数值显示,虽然相关性中等到较高,但这两个参数的区分能力并不相同。
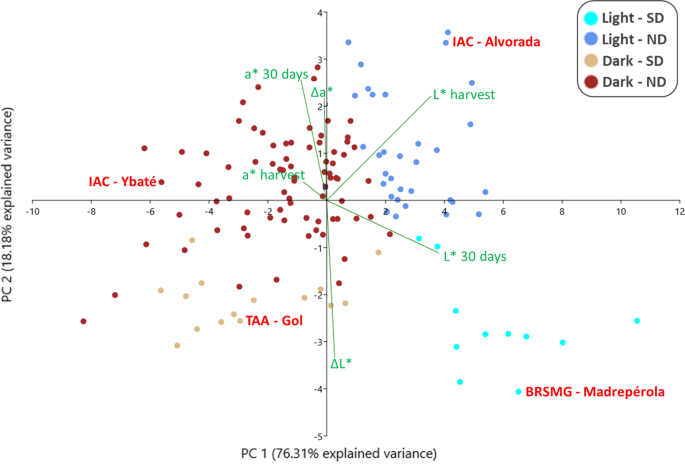
由于L*和a*参数之间有适度的相关性,我们用PCA双图测试了两者的鉴别能力(图2)。2),以选择最佳的遗传作图参数。第一个分量解释了76.3%的观测方差,清楚地区分了收获时最亮和最暗的基因型,而第二个分量区分了SD和RD基因型。考虑到载体,与a*参数相比,L*参数在三个变量(即收获、储存后和delta)中表现出更大的载体,并且因为它解释了更高的表型变异百分比,所以在其他分析中选择该参数。
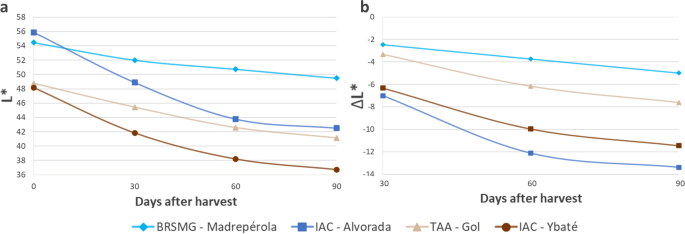
L *的PCA双针(图。2)选取每个表型组(即ac - alvorada [Light RD], BRSMG-Madrepérola [Light SD], IAC-Ybaté [Dark RD], TAA-Gol [Dark SD])的标准品种,使建议组之间的差异更加清晰(图。3.a).在收获期,IAC-Alvorada和BRSMG-Madrepérola种皮颜色的亮度差异仅为2.6%,贮藏90天后,这种差异增加到14%(图2)。3.a).然而,IAC-Alvorada与TAA-Gol相比,收获时的差异为13%,贮藏后的差异仅为3%。因此,结果表明,仅考虑SD和RD性状进行基因型分离,并不能提供充分的分类,因为TAA-Gol将被分类为RD,但随着时间的推移,仅比BRSMG-Madrepérola多损失-2.63 L*。在考虑ΔL*(图。3.b)是,不像L*(图。3.a)随着时间的推移,ΔL*地块的基因型排名没有变化(如IAC-Alvorada在收获时L*最高,但在储存30天后移至第二位)(图2)。3.b)。
在2020年在2018年进行的实验中进行的实验中的平均亮度值较高3%。然而,考虑到整体平均值,基因型在第二个实验中比在第一次实验中变暗了39%(表1).在考虑两种实验时,偏差分析对基因型效应和基因型×环境相互作用具有更高的意义。In spite of that, the genetic correlation for the three traits was superior to 80%, indicating that although the environment influences the lightness of seed coat color of the genotype, the position of the genotype in the variation is not strongly affected (i.e., lighter-colored genotypes are always lighter colored). Moreover, the restricted variance interaction was low for all the traits evaluated, with the lowest being observed for ΔL*.
如预期的那样,基因型效应的偏差分析也具有很高的显著性,L* Harvest (HL)和L* 30天后(L30)的遗传方差占表型方差的65%以上,验证了关联作图的有效性。两个性状的变异系数均小于2.5%,准确率均大于90%。delta L* (ΔL*)表现出中等变异系数值(14.5%),而ΔL*依赖于HL和L30,这导致观测到的变异最大。同样的事实也解释了ΔL*性状的广义遗传力较低的原因,它是0.5,而其他性状是0.7。
格干
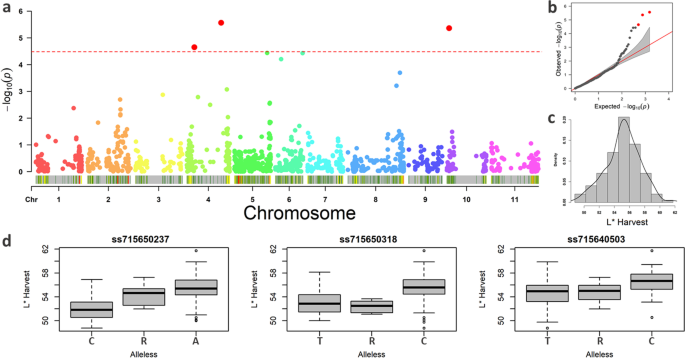
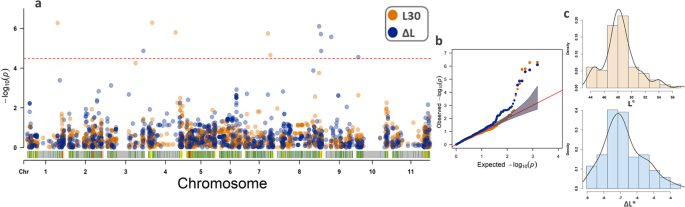
对于关联映射,选择后,共选出1,516个高质量的SNP,分布在物种的基因组上SNPCalling使用138个CDP基因型。根据Bonferroni测试[30.[FarmCPU是否确定了3个HL的显着QTL(图。4.a),与所使用的模型吻合良好(图。4.b)表型数据呈正态分布(图。4.c).两个QTL映射到Pv04上(表2),与SNP SS715650237(位置:8.31 MB)相关的染色体的开头,与SNP SS715650318相关的染色体结束时(位置:38 MB)。
第三个QTL在Pv10的起始位置2.56 Mb,与SNP位点ss715640503相关。在snp中,Pv04的snp均表现出较大的效应;因此,备选等位基因(即SNP ss715650237的‘A’等位基因和SNP ss715650318的‘C’等位基因)贡献了更大的基因型轻度(图。4.d).另外2个QTL (Pv05末端和Pv06上的)的遗传差异均为显著性p-值非常接近显著性极限,即显著性为6%。然而,为了避免I型错误,进一步的分析中没有考虑这两种错误。
为了识别与PPD公差相关的QTL,GWAS分析用来自L30和ΔL的数据进行。考虑到这两个特征,总共11个QTL非常重要,但没有对两个特征都很重要(图。5.一种)。使用的模型适合HL(图。5.b),两种表型数据集还显示正常分布(图。5.C)。
通常,在每种染色体PV04和染色体PV07上的L30,两个QTL具有重要意义,以及在PV01上的一个。对于HL所鉴定的PV04的相同SNP SS715650318显示出L30的显着性,并且PV04上的第二SNP SS715648328对L30的显着性,来自SNP SS715650237的2.81MB与HL相关。PV07上的SNP彼此定位在3.38mb,SNP SS715647732的参考等位基因'G'占储存后谷物的较大亮度,而SNP SS715645851的替代'G'等位基因与较低的亮度有关(桌子2).
ΔL的6个QTL经Bonferroni检验显著性较高[30.].第一个与Pv03上51.28 Mb位点的SNP ss715645575相关。Pv08上还鉴定出3个QTL,第一个与SNP ss715646652相关,位点为58.97 Mb,另外两个与SNP ss715646104和ss715646121相关,位点相差0.44 Mb。在Pv09染色体上,与SNP ss715645744相关的一个QTL被定位在7.81 Mb位点上。最后一个与SNP ss715640510相关的QTL被定位在Pv10染色体上,位于SNP ss715640503的0.02 Mb位点上,对HL有显著影响。
候选基因
考虑到每个显著QTL的置信区间窗为0.59 Mb,所有SNPs都定位在大量基因附近(表1)2).基因数最少的QTL与HL和L30的SNP ss715650318相关,在1.18 Mb范围内共有38个基因。与ΔL*相关的SNP ss715646104和ss715646121定位在同一个QTL中,是基因数量最多的QTL,共235个,置信区间为1.62 Mb。
候选基因的可能作用特征评价,三份的基因编码的NAD (P)绑定rossmann-fold总科蛋白质的发现平均距离0.55 Mb的SNP ss715650318与霍奇金淋巴瘤和翻译(即Phvul.004G112100 Phvul.004G112400, Phvul.004G112501),一个拷贝位于0.24 Mb,来自L30相关SNP ss715646867(即Phvul.001G169500),另一个拷贝位于0.35 Mb,来自ΔL*相关SNP ss715646652(即Phvul.008G237900)。基因编码MYB转录因子也确定为0.27 Mb的SNP ss715650237与霍奇金淋巴瘤(即Phvul.004G057800),从与翻译相关的SNP ss715648328 0.01 Mb(即Phvul.004G046000)和0.13 Mb的SNP ss715645851与翻译(例如,Phvul.007G231800)有关。与ΔL相关的SNPs ss715646104和ss715646652也分别含有编码0.55和0.49 Mb相同蛋白的基因拷贝。
在与ΔL相关的SNP ss715646121位点上分别鉴定到编码‘寡肽转运蛋白’的Phvul.008G280400和编码‘糖转运蛋白’的Phvul.008G280700基因,分别位于0.25和0.28 Mb。此外,在与HL相关的SNP区间ss715640503中发现了8个编码细胞色素P450的基因拷贝。同样的8个基因在SNP区间ss715640510中对ΔL具有显著意义,另外4个编码相同蛋白的基因对相同性状进行了鉴定,一个位于SNP ss715645575的侧边,另外3个位于SNP ss715646104和ss715646121中。对于L30,在0.30 Mb的SNP ss715645851(即phvull . 007g229700)中鉴定出一个拷贝。表中给出了GWAS各显著SNP置信区间内存在的所有基因S2.
讨论
就像斑豆、蔓越莓和红豆品种变黑一样,收获后导致可可豆贬值的最重要因素之一是种皮变黑,因为消费者认为,它们年龄较大,更难烹饪[31].然而,与其他品种不同的是,可可豆不仅会随着时间变黑而贬值,而且对于生产者来说,收获时种皮亮度较低的可可豆(即较黑的可可豆)也会贬值,原因与上述相同[28那32].因此,巴西大豆育种计划不仅集中于培育耐PHD的可可豆品种,而且还集中于那些颜色最浅的谷物[16,品种如BRSMG-Madrepérola [33]和iac-polaco [34].
收获时种皮轻
关于Carioca品种,HL和对PHD的耐受性必须被视为不同的特征,并且在文献中存在一定的误解。从这个意义上讲,映射基因座籽粒轻度的控制和与轻度性状相关的分子标记的鉴定对可可豆育种具有极其重要的意义。
已经进行了一些研究,旨在绘制对PHD耐受性的遗传控制[10.那21那28那31]和该物种的粒色[35那36那37].种皮颜色的表达是由一个多等位基因序列控制的P轨迹显示等位基因与V轨迹[38那39],它们与七种其他基因的等位基因相互作用(GY.那C那R那J那G,B.那和Rk) [35那36].巴塞特(37]提出了一份可以影响种皮颜色特征的24个基因的清单,以可可豆为例,它的特点是有棕色条纹的奶油色种子,条纹和着色的模式是由基因控制的C轨迹[40].
我们的结果表明,HL性状具有寡基因遗传,通过观察到的中等遗传力和实验之间的高基因型相关性来证实。仅有3个QTL具有显著性,其中2个QTL定位于Pv04。在同一染色体上,McClean等人[36]确定了G轨迹连接到OU14900RADP标记,以前被报道为控制籽粒颜色中的黄褐色因子[35].OU14的前向顺序对齐900对参考基因组,菜豆v2.1 [5.,该标记为7.26 Mb,来自SNP ss715650318。虽然距离被认为很大,但McClean等人使用的小地图种群(n80) [36标记的数量少可能导致制图分辨率低。
本研究首次鉴定了与HL相关的QTL,通过对ss715650237、ss715650318和ss715640502 snp的鉴定,对于在育种过程的早期筛选和选择淡色粒的可可豆普通品系具有相当大的潜力。
对PHD的表型耐受
Previous studies classified the genotypes as SD and RD based only on evaluation of the color trait (i.e., diagrammatic scoring scale) or measurement of color (i.e., digital analysis and colorimeters) after a determined storage period, without considering the initial color of the grain [10.那16那29那31那32].从根本上说,SD性状应该与一个特定基因型随着时间的推移颜色亮度下降的事实有关,而不是与它在储存期后颜色较浅的事实有关。我们的结果不仅表明这种分类是不充分的,如IAC-Alvorada和TAA-Gol的对比反应所示(图。3.),但使用不考虑籽粒初始颜色条件的最终评价,在鉴定耐PHD相关的QTL时也会产生不同的结果。
除了SD和RD表型类别,Elsadr等人。[22]提出了斑蝥豆的第三个表型类,ND(不变暗)类。Erfatpour等[12,使用相同的分类,映射ndQTL染色体PV10伴有蔓越莓等基因型。在Carioca品种的情况下,在评估的138个基因型中,所有品种在30天后损失L *值超过两点,显示出多样性的ND表型类。此外,该品种被认为是先前研究中SD的标准[10.那23那33那41]对PHD耐受性次之(-2.46 L*)。
涉及评估Carioca谷物的研究使用L *参数进行育种分类和选择[14那16那42].考虑到其他豆类品种,如斑托豆,一些研究采用了a*参数[12那21那43].虽然在文献中没有共识,但我们的结果表明,对于可可豆,L*参数具有更大的区分能力。关于货架贮藏法对PHD耐受性评估所需的时间,有研究报告需要90天[28那42,而其他人则在收获后6个月评估[44].
结果表明,L*和a*的贮藏期均为30天,与90天的贮藏期相关性均大于90%。Silva等人[16]和Silva等人。[10.研究还指出,相对于60天和90天贮藏期,30天贮藏期是选择颜色较浅的豆科品种的最佳贮藏期,这主要是因为贮藏期相关指数高,评价所需的时间较短。
博士学位的遗传控制
GWAS结果表明,贮藏期后种皮亮度(L30)和耐受性(ΔL*)的QTL存在差异,值得注意的是,Pv07染色体上接近贮藏期的QTL仅L30显著sd轨迹(即,SNP SS715647732由Felicetti等报告的PVSD-0028标记定位2.5 MB。[13与斑豆的SD性状相关。罗德里格斯等[32发现了同样的结果轨迹负责耐受Carioca Beans的博士学位和Alvares等人。[23]在储存后验证了用于选择较轻的线条的标记。所提到的所有研究只使用了存储信息后的颜色,忽略了初始颜色。相比之下,我们的结果表明sd轨迹对PHD的耐受性不显著(即考虑ΔL*特征)。虽然L30性状可能与较浅或较深的颗粒有关,但该性状并不表示颜色的变化(即在贮藏过程中亮度的变化)。
结果表明:ΔL*是最有效表达耐PHD的QTL, 6个QTL分别定位在4条染色体上。这是第一个涉及GWAS对PHD耐受性的研究,不同于之前报道的单基因控制性状的研究[16那21那22那32,我们的结果显示寡基因控制。广义遗传力估计最低(即0.5),且基因型与环境交互作用极显著,证实了该性状更为复杂的遗传谱。Silva等人[16, Silva等人[10., Siqueira等[15, Alvares等[28]也报道了该性状的基因型×环境相互作用,证实了寡基因谱。
定位在与HL相关的PV10染色体开始时的相同QTL对于ΔL显着显着,虽然QTL不是具有最大效果的那个,但它与各种极端重要性的两个特征有关。Erfatpour等[12]也在Pv10上定位到一个与不变暗性状相关的QTL;然而,作者所报道的蔓越莓样豆基因型的QTL不能被认为是与carioca品种相关的同一个QTL,因为距离太远基因座从对方。关于Pv03上发现的QTL,目前尚无文献报道a轨迹映射在这种染色体上。然而,SNP SS715645575置信区间的大量基因强调了未来研究以识别候选基因的研究。
在Pv08上鉴定的QTL均在2.51 Mb,第二个QTL与两个显著的SNPs关联在0.02 Mb范围内轨迹与该特征相关的。库托等人[29]首次鉴定出与可可豆耐PHD QTL相关的分子标记,并报道PVESTBR-98标记位于唯一定位的QTL的侧边。参照基因组(即,菜豆v2.1,[5.在Pv08上所鉴定的第二个QTL中,SNP位点ss715646104与SNP位点ss715646104的位点比对结果为0.06 Mb。此外,McClean等人[36)映射C轨迹与同一染色体末端种皮的图案有关的。作者还确定了T轨迹与染色体PV09上的标记OM19400连接的种子涂层的图案相关。标记OM19400的前向序列的BLASTN显示,它是SNP SS715645744的3.87 MB。
我们的结果表明,可能C和T LOCI.据报道,控制种子涂层的图案可以与Carioca豆类的耐受性有关。需要未来的研究来实现对两者的影响更好的理解基因座随着时间的推移,可可豆浅色的控制。
育种候选基因
PHD特性依赖于一系列的化学过程。以蔓越莓豆品种为例,研究表明RD基因型的总酚含量显著高于ND基因型[45那46].Myers等人[47报道了编码“NAD(P)结合rosssmann -fold超家族蛋白”的基因,该基因与总酚含量有关,因为它参与黄酮类化合物的生物合成。因此,在与ΔL*相关的Pv08第一个QTL区间中鉴定的Phvul.008G237900基因具有相当大的差异表达研究潜力,因为它与Myers等人报道的基因具有相同的注释[47].
在各种多酚化合物中,类黄酮是最常见的,如黄酮醇、花青素和原花青素[12].根据Freixas-coutin等。[25,类黄酮生物合成途径的前体由细胞色素P450 (Cytochrome P450)酶、类黄酮3'-羟化酶和类黄酮3'- 5'-羟化酶催化。Pv10,我们确定了七份基因编码细胞色素P450酶相关的QTL与霍奇金淋巴瘤和ΔL,使这些基因(Phvul.010G013100、Phvul.010G013000 Phvul.010G012900, Phvul.010G012700, Phvul.010G019100, Phvul.010G019600,和Phvul.010G022400)潜在候选人遗传育种。
此外,HL和ΔL*中发现的myb样基因也显示出巨大的潜力,因为苯基丙烷通路基因被认为是由MYB-bHLH-WDR复合物调控的[48那49].MYB蛋白是调控发育代谢(包括花青素合成)的调控网络中的关键因子[50].Erfatpour等[12在鉴定与博士学耐受相关的基因时,还将MyB样基因作为主要候选者之一。
最后,酶,调节分子的运输等“寡肽转运体”和“糖转运体”也可以积累proanthocyanidin起到至关重要的作用,考虑到更大的这些基因的表达与浅色系谷物在所有阶段的蔓越莓豆品种,如Freixas-Coutin等人指出的[25].因此,在ΔL*显著SNP ss715646121置信区间中鉴定的phvull . 008g280400和phvull . 008g280700基因也是改良该性状的潜在候选基因。
结论
这是第一次对收获时种皮颜色的亮度和对PHD的耐受性进行关联定位研究,这两个性状对普通豆类的主要商业品种极为重要。结果表明,可可豆HL性状的qtl均达到了高显著性,3个qtl均达到了高显著性,为可可豆HL性状的辅助选择和种质筛选奠定了基础。在评价对PHD的耐受性方面,我们的表型结果表明,参数L *具有更大的判别力,战略性地认为贮藏30天是评价PHD的最佳时期。在表型分类方面,我们的结果显示存在4个不同的表型分类,为了正确评估对PHD的耐受性,有必要考虑初始和最终种皮颜色(ΔL*)。利用ΔL*,我们在4个不同染色体上鉴定了6个显著的QTL,其中Pv10 QTL最有趣,因为它同时与HL和PHD性状相关。此外,sd轨迹在之前的研究中报道的,显示出只考虑L*参数在贮藏期后使用的carioca多样性面板的意义,因此,不代表对PHD的耐受。
材料和方法
植物材料、基因分型和SNPCalling
本研究采用巴西可可豆多样性面板(CDP),该面板由农艺研究所(IAC)普通豆育种组(Common Bean Breeding Group)的种质库中选择的138份可可豆种质组成,以代表巴西可可豆的遗传多样性。利用Illumina BeadChip BARCBean6K_3高通量基因分型技术对供体进行基因分型[51].Almeida等人验证了CDP对GWAS的有效性[9.),包括巴西的主要商业大豆品种,从1971年发布的第一个品种(即“carioca comum”)[52]在2019年发布的更多现代品种,如IAC-1850 [53].该组合还包括在以前的研究中发现的种皮变暗率不同的基因型,包括RD品种BRS-Cometa、BRS-Estilo、BRS-Pérola、BRS-Pontal、brs - requante、ac - alvorada和BRS-Majestoso [10.那16那17那23那28那29那32]和SD品种BRS-Madrepérola、ANFC-9、Branquinho和TAA-Dama [28那32那33].
使用Tassel 5.0软件进行基因型数据的质量分析[54,剔除小等位基因频率< 3%,杂合度> 5%,缺失数据> 10%的snp。将高质量的基因型矩阵转化为HAPMAP文件格式,参考等位基因用“A”表示,备选等位基因用“G”表示,杂合等位基因用“R”表示,缺失数据用“N”表示。BARCBean6K_3是基于第一个普通豆子基因组(即,菜豆v1),因此,每个SNP的侧翼序列(即BLASTN)针对最新的参考基因组进行blast,菜豆v2.1 [5.[获得每个SNP的位置。除去基因组中未知位置的标记,使用Beagle 5.0软件进行“N”标记的归纳[55].关于CDP的所有信息都在表中给出S1,包括表型和基因型数据。
HL和种皮PHD的评价
试验是在Fazenda Santa Eliza实验站(IAC, Campinas, SP,巴西)进行的,为期两年,第一次试验于2018年8月播种,第二次试验于2020年7月播种。试验小区采用完全随机区组试验设计,行1 m,株系10株,行距0.5 m, 3个重复。人工收获,豆荚在温室中保存10天,以使谷物完全干燥和颜色标准化。谷物过筛后装入纸袋内,置于10±2℃干燥阴暗的贮藏室内。
从每种图中的大约80种种子的随机样品,足以填充6cm的培养皿,用于用比色计(Konica Minolta Cr-410,Osaka,Japan)的分析,其是由五种技术重复的平均值表示的每个样本的测量值。根据CIELAB系统用比色仪测定颜色参数[56,其中三个轴用于描述颜色:在z轴上的L*参数(即光度),即黑暗(即,L* = 0的值为完全黑色)到亮度(即,L* = 100的值为完全白色)分量;x轴上的a*参数,它是一个绿色(即,a* = -60的值是完全绿色)到红色(即,a* = + 60的值是完全红色)分量;以及z轴上的b*参数,这是一个蓝色(即,b* = -60的值是完全蓝色)到黄色(即,b* = + 60的值是完全黄色)组件。L* a* b*颜色系统以与人眼相似的方式工作,除了具有统一的颜色尺度,还能在不同样品之间比较颜色值[57].
在收获时测色后,样品装入自封袋中(8.5 × 12 cm,每袋80 g),在控制温度(26±2°C)和光周期(12 h)的条件下,在装有荧光灯的房间中储存。每周都要重新定位样品,以确保样品上的光照均匀。第一次试验分别在贮藏30、60、90天测定各小区的颜色,以选择最佳的时间进行第二次试验的评价,第二次试验采用贮藏30天的时间进行变暗评价。
表型统计分析
随着时间的推移,豆种涂层趋于变暗和红色,因此仅考虑L *和*参数进行初始分析。为了确定代表所采用的总时间的最短存储时间,Pearson相关值(R2)的L*和a*参数,利用R包Corrplot从第一次实验的表型数据估计[58].用r表示存储时间2值大于90%,对于L*和a*参数,第二次实验采用颜色分析。
为了选择最能解释表型变异的参数,需要在收获和储存30天后的色度计数据,以及这两个参数的delta(即ΔL* =最终L* -初始L*;Δa* = final a* -initial a*),通过PAST4.05程序用于主成分分析(PCA)的双图[59].选择具有三个变量最大载体的参数(即,收获,30天后,δ)用于分析关联映射和遗传参数的估计。为了最佳可视化数据,将基因型分成4个表型类(图。6.):
表型分裂成四个对比等级,第一划分与收获(浅×暗)的种子的亮度(L *)和第二,与收获后种子涂层变暗的耐受性(慢× regular darkening). The genotypes MRSMG-Madrepérola, IAC-Alvorada, TAA-Gol, and IAC-Ybaté were selected to represent the phenotypic classes of Light Slow Darkening, Light Regular Darkening, Dark Slow Darkening, and Dark Regular Darkening, respectively
LIGHT SD:基因型为收获时种子颜色较浅(L*≥53),变暗缓慢(ΔL*≥-4),标准品种为BRSMG-Madrepérola。
理查德·道金斯:光在收获的基因型(L *≥53),具有常规变暗(ΔL*≤-4.1),IAC-Alvorada作为标准品种。
黑暗SD:以TAA-Gol为标准品种,收获时种子颜色较深(L*≤52.9),并缓慢变暗(ΔL*≥-4)。
黑暗的采访:基因型,收获时种子颜色较深(L*≤53.9),并有规律变暗(ΔL*≤-4.1),以IAC-Ybaté为标准品种。
为了评估基因型×环境相互作用并验证GWA的表型数据,通过限制最大似然/最佳线性无偏析预测器(REML / BLUP)估计偏差分析(ANADEV),广义遗传性和方差分量Selegen软件[60].
基因组关联模型
固定与随机模型循环概率统一- farmcpu [61]在rMVP R包中实现[62]由于其统计能力强,对效应较小的QTL敏感性高,因此被用于关联作图。该包探索了多轨迹混合模型并以两个交互步骤执行分析:首先应用固定效果模型,然后是随机效应模型。两种模型相互作用,直到未检测到显着标记。为避免I型错误(即,误报),根据Schwarz的贝叶斯信息标准(BIC)测试测试了结构化矩阵[63],对于常规混合线性模型[64]的前五个成分的主成分分析(PCA,表3 s补充材料)。根据施瓦茨[63]最高的BIC揭示了模型的最佳协调因子。如Almeida等人所示。[9., CDP不需要使用结构矩阵来纠正I型错误(即假阳性),因为集合中没有子组。的p -模型中每个SNP的阈值由Bonferroni确定[30.]阈值法(截断α = 0.05)。所使用的表型矩阵由使用lmerTest R包的限制性最大似然/最佳线性无偏估计(REML / BLUE)估计的基因型值给出[65].
候选基因和基因注释
利用所有重要SNPs的物理位置,通过遗传注释彻底搜索候选基因Jbrowse源自Phytozome v11.0 [66和参考基因组菜豆v2.1 [5.].对于搜索,我们考虑了0.59 Mb的置信区间窗,这是Almeida等人确定的平均距离[9.为CDP(即,到LD衰减的距离= r20.2)。LD衰减使用平方等位基因频率相关染色体内对估计,R包LDcorSV [67],以说明亲属关系(亲属关系[68])。所有染色体的LD衰减曲线(图S1,使用山丘和堰提出的非线性模型解释了补充材料[69,正如Diniz等人所描述的[70].
可用性数据和材料
本研究中产生或分析的所有数据均包含在本文中。
参考文献
- 1。
Broughton WJ,Hernándezg,布莱尔M,Beebe S,Gepts P,Vanderleyden J. Beans(菜豆-示范食物豆类。土壤植物。2003;252:55 - 128。
- 2.
Bellucci E,Bitocchi E,Rau D,Rodriguez M,Biagetti E,Giardini A等。寻常时期的原产地,驯化和演化的基因组学。在:Tuberosa R.,Graner A. Fe,编辑。植物遗传资源基因组学:第1版。DONDRECHT:斯普林克荷兰;2014. p。483-507。
- 3.
皮雷CV,奥利维拉MGA,克鲁兹GADR,门德斯FQ,德雷森德ST,莫雷拉MA。Composição físico-química de differentes culares de feijão (菜豆l .)。反应e nutr araraquara。2005; 16:157-62。
- 4.
植物生物活性化合物的生化目标:作用位点和生物效应的药理学参考指南。纽约;2003.
- 5.
Schmutz J,McClean Pe,Mamidi S,Wu Ga,Cannon Sb,Grimwood J等人。一种参考基因组,用于常见的豆类和基因组的双重驯化分析。NAT Genet。2014; 46:707-13。
- 6.
FAOSTAT。联合国粮食及农业组织。统计数据库,粮食和农业,2019。http://www.fao.org/faostat/en/#data/qc/visualize..2020年7月6日。
- 7.
de Carneiro JES, de Paula TJ Jr, Borém A. Feijão: do plantio à colheita。2015.
- 8.
Chiorato AF, Carbonell SAM。O Melhoramento Genético do Feijoeiro no Instituto Agronômico IAC (1932 a 2014)。坎皮纳斯:O AGRONOMICO;2014.
- 9.
de Almeida CP, de Paulino JFC, Morais Carbonell SA, Chiorato AF, Song Q, Di Vittori V,等。巴西普通豆遗传育种半个世纪后的遗传多样性、群体结构和安第斯渐渗。基因(巴塞尔)。2020; 11:1298。
- 10。
Silva FC,Melo PGS,Pereira HS,Melo LC。Carioca豆种子涂层遗传参数的遗传控制与估计。genet mol res。2014; 13:6486-96。
- 11.
Pereira HS, Mota APS, Rodrigues LA, de Souza TLPO, Melo LC。基于农艺性状和分子标记的普通豆品种遗传多样性及其在亲本推荐中的应用。Euphytica。2019;215:1-16。
- 12.
Erfatpour m,navabi a,pauls kp。从“Wit-Rood Boontje”中映射非变暗的特质(菜豆).Theor Appl Genet. 2018; 131:1331-43。
- 13.
费利塞蒂E,宋琪,贾刚,Cregan P, Bett KE, Miklas PN。通过单核苷酸多态性分析和全基因组测序发现斑豆慢变暗性状的简单重复序列。作物科学。2012;52:1600-8。
- 14.
Ribeiro ND, Storck L, Poersch NL。Classificação de lotes comerciais de feijão por meio da claridade do tegumento dos grãos。Cienc农村。2008;38:2042-5。
- 15.
Siqueira Bs,Pereira WJ,Batista Ka,Oomah Bd,Fernandes Kf,Bassinello Pz。储存对慢速和常规黑暗的Carioca Bean变暗和硬化的影响(菜豆L.)基因型。j农业螺柱。2014; 2:87。
- 16.
席尔瓦GS,拉马略MAP,阿布瑞乌ÂFB,席尔瓦FB。可可豆籽粒早期变黑的遗传控制。作物育种应用生物技术。2008;8:299-304。
- 17.
等。是酶促还是非酶促途径导致了可可豆采后表皮变暗的现象?食品科学技术。2016;69:593-600。
- 18.
Alvares Rc,Pereira HS,Melo LC,Miklas Pn,Melo PG。常见豆类种子涂层变暗的诱导(菜豆L.)和储存后烹饪时间的关联。Aust J Croc SCI。2020; 14:21-7。
- 19.
Bassett MJM。普通豆种子颜色与图案的遗传学。在:植物育种评论。Wiley;2007. p。239-315。
- 20.
mclean PE, Bett KE, Stonehouse R, Lee R, Pflieger S, Moghaddam SM,等。小豆的白色种子颜色(菜豆)P(颜料)基因的收敛演变产生。新植物。2018; 219:1112-23。
- 21。
Junk-Knievel DC, Vandenberg A, Bett KE。花豆种皮的缓慢变暗是由一个单一的主基因控制的。作物科学。2008;48:189 - 93。
- 22。
Elsadr HT, Wright LC, paul KP, Bett KE。小豆种皮采后变黑的特征(菜豆l .)。Theor Appl Genet. 2011; 123:1467-72。
- 23。
Alvares RC, Stonehouse R, Souza TLPO, Melo PGS, Miklas PN, Bett KE,等。慢变暗种皮性状玉米干豆基因型选择的遗传标记的生成与验证。Euphytica。2019;215:1-11。
- 24.
Demito A,Ziegler V,Goebel JTS,Konopatzki EA,Coelho SRM,Elias MC。制冷对贮藏期间Cariocass生物化学,消化率和技术参数的影响。J Food Biochem。2019; 43:E12900。
- 25.
Freixas Coutin JA, Munholland S, Silva A, Subedi S, Lukens L, Crosby WL, et al.;蔓越莓豆种皮中原花青素的积累和转录反应(菜豆对采后暗沉的敏感性不同。BMC Plant Biol. 2017;17:89。
- 26.
erfatpour m,pauls kp。PIGTO和蔓越莓豆类非变暗种子涂层性状的基于R2R3-MYB基因的标记物(菜豆L.)源自“witrood boontje”。Theor Appl Genet. 2020;133:1977-94。
- 27.
贝宁格,顾林,先验RL,垃圾DC,范登堡A,贝特KE。花豆种皮中多酚类物质在变暗过程中的变化。中国农业科学(英文版)。
- 28.
Alvares RC, Silva FC, Melo LC, Melo PGSS, Pereira HS, Mol G.高产直立种皮慢变暗普通菜豆系的遗传参数估计和选择。植物学报2016;15(4):1-10。
- 29.
库托,多斯桑托斯,拉马略,达席尔瓦。Identificação de marcadores microssatélites relacionados ao escurecimento de grãos em feijão。Pesqui Agropecu Bras. 2010; 45:1268-74。
- 30.
Bonferroni C. Teoria statisticsdelle classi e calcolo delle probabilità。《费伦泽超级科学经济学家》1963;8:3-62。
- 31.
pinto bean种皮的缓慢暗化与原花青素生物合成相关的代谢产物和转录本差异相关。BMC基因组学。2018;19(1):260。
- 32.
Rodrigues ll,rodrigues la,souza tlpo,melo lc,pereira hs。三个市场班级普通豆品种遗传防治。农作物科学。2019; 59:2046-54。
- 33。
de Carneiro JES, de Abreu AFB, Patto Ramalho MA, de Paula TJ, Del Peloso MJ, Melo LC等。BRSMG Madrepérola:具有晚变暗的里约卡籽粒的普通豆品种。作物育种应用生物技术。2012;12:281-4。https://doi.org/10.1590/S1984-703320120004000000..
- 34。
Chiorato AF,Carbonell Sam,Bezerra LMC,De Esteves JAF,GonçalvesJGR,Da Silva Da,等。IAC Polaco:Carioca常见的豆品种具有早期成熟和种子变暗的耐受性。作物品种苹果生物科技。1849; 2020(20):1-4。
- 35。
颜色的继承菜豆l . III。关于红种皮颜色的基因和一般合成。瓦赫宁根,1972;29:1-82。
- 36.
麦克林PE,李RK,奥托C, Gepts P,巴西特MJ。菜豆种皮花色调控基因的分子定位和表型定位(菜豆l .)。J在这里。2002;93:148-52。
- 37.
Bassett MJ。基因列表 -菜豆《豆子代表即兴表演》2004;47:1-24。
- 38.
爱默生RA。豆类斑驳的因素。Z Indukt Abstamm Vererbungsl. 1910; 4:372-372。
- 39.
兰普雷希特·祖尔·冯菜豆十二。Über die Vererbung der Blüten- und Stammfarbe。Hereditas。1936;21:129 - 66。
- 40。
颜色的继承菜豆“复合位点c”内的IV重组。
- 41.
席尔瓦FC,佩雷拉HS,甜瓜PGS,甜瓜LC。高农艺潜力种皮变暗慢的普通菜豆亲本及分离群体的选择。2018; 48:75-82。
- 42.
Spitti AMDS, Carbonell SAM, Dias CTDS, Sabino LG, Carvalho CRL, Chiorato AF.通过自然和加速方法耐谷物变暗的可可豆基因型。农业技术。2019;43:12519。
- 43.
Junk-Knievel DC, Vandenberg A, Bett KE。在不同环境下生长的平托豆的加速采后种皮暗化方案。作物科学。2007;47:694 - 702。
- 44.
Arns FD, Ribeiro ND, Mezzomo HC, De Marco Steckling S, Kläsener GR, Casagrande CR.混合选择的可可豆粒度大小,缓慢变暗和储存时间后快速烹饪。Euphytica。2018;214(66):1 - 12。
- 45。
陈PX,唐y,marcone mf,pauls pk,zhang b,liu r等人。常规和非变暗的蔓越莓豆类中自由,共轭和结合酚类和亲脂性抗氧化剂的表征(菜豆l .)。食品化学。2015;185:298 - 308。
- 46。
陈px, Bozzo GG, Freixas-Coutin JA, Marcone MF, Pauls PK, Tang Y, et al.;普通和不变色蔓越莓豆中的游离和共轭酚类化合物及其抗氧化活性(菜豆l .)种子的外套。功能性食品。2015;18:1047-56。
- 47。
Myers Jr,Wallace Lt,Moghaddam Sm,Kleintop Ae,Echeverria D,Thompson HJ等。改善豌豆的健康益处:总酚类含量的基因组关联研究。营养素。2019; 11(10):2509。
- 48.
刘家,奥巴顿A,MA P. MYB转录因子作为植物苯丙醇代谢调节因子。莫尔植物。2015; 8:689-708。
- 49.
徐W,Dubos C,Lepiniec L. myB-BHLH-WDR复合物对类黄酮生物合成的转录控制。趋势植物SCI。2015; 20:176-85。
- 50。
杜华,杨树生,梁铮,冯伯林,刘林,黄玉波,等。大豆MYB转录因子超家族的全基因组分析。BMC Plant Biol. 2012;12:106。https://doi.org/10.1186/1471-2229-12-106.
- 51。
宋强,贾刚,Hyten DL, Jenkins J,黄安英,Schroeder SG,等。在普通菜豆中建立连锁图谱、锚定全基因组序列和其他遗传和基因组应用的SNP分析发展。G3 Genes, genome, Genet. 2015; 5:2285-90。
- 52。
de Almeida LD, Leitão Filho HF, Miyasaka S. Características do feijão Carioca, um nôvo品种。Bragantia。1971;30:33-38。
- 53。
Carbonell Sam,Chiorato Af,Bezerra LMC,GonçalvesJGR,Da Silva Da,De Esteves JAF等。IAC 1850高产Carioca常见豆种。作物品种苹果生物科技。2019; 19:378-81。https://doi.org/10.1590/1984-70332019v19n3c53.
- 54.
张志,kon DE, Casstevens TM, Ramdoss Y, Buckler ES。TASSEL:用于不同样本中复杂性状关联制图的软件。生物信息学。2007;23:2633-5。https://doi.org/10.1093/bioinformatics/btm308.
- 55.
勃朗宁BL,周颖,勃朗宁sr。美J Hum Genet. 2018; 103:338-48。
- 56.
猎人R.出现的测量。纽约:Wiley;1975年。
- 57。
马库斯。R.颜色的测量。In: Nassau K,编辑。科学、艺术和技术的色彩。Armsterdam:爱思唯尔科学;1998.31 - 96页。
- 58。
Wei T,Simko VR。包“Corrplot”:相关矩阵的可视化。2017年(版本0.84。2017)。
- 59。
锤子Ø,哈珀dat,ryan pd。过去:古生物学统计软件包,用于教育和数据分析。Palaeontol电子。2001; 4:178http://palaeo-electronica.orghttp//palaeo-electronica.org/2001_1/past/issue1_01.htm.
- 60。
Resende MDV。Selegen-Reml / Blup软件:植物育种的一个有用的工具。作物品种苹果生物科技。2016; 16:330-9。
- 61。
刘X,黄M,粉丝B,贝勒ES,张Z.迭代使用固定和随机效果模型,以实现强大高效的基因组关联研究。Plos Genet。2016; 12(3):E1005957。https://doi.org/10.1371/journal.pgen.1005957.
- 62。
殷磊,张慧,唐志平,等。rMVP:一种记忆效率高、可视化增强、并行1加速的全基因组关联研究工具。2020;s1672 - 0229(21): 00050 - 4。https://doi.org/10.1016/j.gpb.2020.10.007.
- 63。
估计模型的维数。安Stat。1978;6:461-4。https://doi.org/10.1214/AOS/1176344136..
- 64。
余建平,王志强,王志强,等。一种用于关联映射的统一混合模型方法,该方法考虑了多个层次的相关性。Nat麝猫。2006;38:203-8。https://doi.org/10.1038/ng1702.
- 65。
库兹涅佐娃,布罗克霍夫,克里斯坦森。lmerTest软件包:线性混合效应模型的测试。[J] .软件学报,2017;82(13):1-26。
- 66.
等。植物群落:绿色植物基因组学的比较平台。核酸Res. 2012;40(数据库版):D1178-D1186。https://doi.org/10.1093/nar/gkr944.
- 67.
(1)基于群体结构和相关性的连锁不平衡度量方法。遗传(Edinb)。2012; 108:285 - 91。https://doi.org/10.1038/hdy.2011.73.
- 68.
VanRaden点。计算基因组预测的有效方法。中国乳业科学(英文版)2008;91:4414-23。https://doi.org/10.3168/jds.2007-0980.
- 69。
Hill WG, Weir BS。有限总体中连锁不平衡平方的方差和协方差。《大众生物学》1988;33:54-78。https://doi.org/10.1016/0040 - 5809 (88) 90004 - 4.
- 70.
Diniz AL, Giordani W, Costa ZP, Margarido GRA, Perseguini JMKC, Benchimol-Reis LL等。栽培普通豆类中亲缘关系对连锁不平衡程度影响的证据。基因(巴塞尔)。2019; 10(1): 5。https://doi.org/10.3390/genes10010005.
确认
不适用。
资金
该研究得到了FundaçãodeAmparoàPesquisa(Fapesp)的支持,为研究项目提供资金(FAPESP第2017/4711-4)和奖学金(FAPESP编号2019/19670-2和2018/20992-1)使这项研究能够进行,并向CoornaçãodeherfeiçocoventodePessoaldeNível上级(Capes)获得奖学金。
作者信息
从属关系
贡献
L.L.B-R, c.p.a.和I.L.S.设计了这项研究。C.P.A.构思了手稿的结构,C.P.A.、C.C.F.B、I.L.S、J.F.C.P、C.C.A.P和g.m.c.g进行了实验。Q.S.进行分子基因分型,C.P.A.和G.M.C.G.进行统计学分析。A.F.C, s.a.m., C.R.L.C和L.L.B-R。支持的数据管理和审查的手稿。L.L.B-R。负责该项目的资金。所有作者阅读并批准了最终版本。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
de Almeida, c.p., Santos, i.l., de Carvalho Paulino, J.F.等等。全基因组关联图谱揭示了新的信息基因座与收获时的种皮颜色较浅和可可豆的种皮颜色缓慢变暗有关。BMC植物杂志21,343(2021)。https://doi.org/10.1186/s12870-021-03122-2
收到了:
公认:
发表:
关键词
- 菜豆l
- 晚种皮变暗
- 种皮轻
- CIELAB规模