- 研究gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
多组学分析gydF4y2BaKandelia obovatagydF4y2Ba揭示其对移栽和群体间遗传分化的响应gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba21gydF4y2Ba、物品编号:gydF4y2Ba341gydF4y2Ba(gydF4y2Ba2021gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
通过种植恢复是保护红树林生态系统的主要策略。然而,许多种植园未能生存下来。种植的地点和种子选择问题。gydF4y2Ba
植树造林的过程,即个体被种植在一个新的环境中,本质上是人类控制的移植事件。为了加深和扩大对移栽对植物影响的认识,我们进行了一项为期7年的互惠移栽实验gydF4y2BaKandelia obovatagydF4y2Ba沿纬度梯度的gydF4y2Ba
结果gydF4y2Ba
结合表型分析和下一代测序,我们发现了普通花园不同群体个体间的表型差异和群体间的遗传分化。中心居群遗传多样性丰富,表型可塑性高,可种植范围广。但转移到其他纬度后,其生物量有所减少。RNA-seq显示木质素合成基因的表达受到抑制是导致生物量减少的原因。此外,利用全基因组亚硫酸盐测序,我们观察到MADS-box基因中参与开花时间调控的DNA甲基化修饰,这可能有助于适应新环境。gydF4y2Ba
结论gydF4y2Ba
利用经典生态学实验和多组学分析,观察了不同种群间的形态差异和遗传分化gydF4y2Bak . obovatagydF4y2Ba为制定长期有效的恢复策略提供科学建议,并探讨了植物对不同纬度移栽事件的表型、转录和表观遗传响应。gydF4y2Ba
背景gydF4y2Ba
红树林是生长在热带和亚热带地区陆地和海洋交界地带的木本植物[gydF4y2Ba1gydF4y2Ba].它们具有形态和生理特征,以适应高盐度、强风、潮汐、高温、厌氧土壤等应激条件。[qh]gydF4y2Ba1gydF4y2Ba].红树林在保护和稳定海岸线、生产商业林产品和支持沿海渔业方面发挥着重要作用,具有巨大的生态价值[gydF4y2Ba2gydF4y2Ba].作为最具生产力的生态系统之一,红树林在产生有机碳方面表现突出,并对全球碳循环作出重大贡献[gydF4y2Ba3.gydF4y2Ba].然而,全世界红树林生态系统前所未有的损失引起了科学界的许多关注[gydF4y2Ba4gydF4y2Ba].在20世纪80年代和90年代,据估计,由于森林砍伐,世界上35%的红树林已经消失[gydF4y2Ba5gydF4y2Ba].Hamilton和Casey的估计显示,2000年至2012年间,全球红树林年平均损失为0.26-0.66% [gydF4y2Ba6gydF4y2Ba].根据中国国家林业局的一项调查,到2002年,中国50年代的原始红树林只剩下44% [gydF4y2Ba7gydF4y2Ba].虽然从2000年到2015年,中国已经恢复了12%的红树林[gydF4y2Ba8gydF4y2Ba],红树林的保护和恢复仍面临许多挑战[gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba].gydF4y2Ba
Kandelia obovatagydF4y2Ba是中国分布最广泛的抗寒红树林之一[gydF4y2Ba11gydF4y2Ba].以前的研究观察到的营养形态的变化gydF4y2Bak . obovatagydF4y2Ba不同的纬度[gydF4y2Ba12gydF4y2Ba];在高纬度地区发现侏儒表型[gydF4y2Ba13gydF4y2Ba].自然选择的力量导致基因型和环境的相互作用,从而达到达尔文的适应度,这应该会导致当地种群进化出在当地环境条件下具有优势的性状[gydF4y2Ba14gydF4y2Ba].在没有其他力量和约束的情况下,每个地方种群的常住基因型在其当地栖息地的平均相对适合度高于来自其他栖息地的基因型。这种模式,包括导致这种模式的过程,就是局部适应[gydF4y2Ba14gydF4y2Ba].利用互反移植实验(检测局部适应性的“金标准”),在45%的植物研究中发现了互反适应性[gydF4y2Ba15gydF4y2Ba].在研究中gydF4y2Ba拟南芥gydF4y2Ba,移栽个体存活率和繁殖力较低,而且开花时间也有变化[gydF4y2Ba16gydF4y2Ba].gydF4y2Ba
植物也能适应环境变化并迅速作出反应。表型变化涉及基因表达的修饰。DNA甲基化是一种向胞嘧啶碱基添加甲基的过程,它是一种表观遗传修饰,在不影响潜在DNA序列的情况下构成基因表达的额外调节水平[gydF4y2Ba17gydF4y2Ba].越来越多的证据表明,DNA甲基化在调节植物发育和形态中起着重要作用[gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba].对环境波动和表型变化的快速反应的表观遗传调控也在许多生物中被观察到,例如礁珊瑚[gydF4y2Ba23gydF4y2Ba]和鳄鱼草[gydF4y2Ba24gydF4y2Ba].对非生物应激反应的早期研究表明,应激诱导的DNA甲基化和/或去甲基化模式要么是全基因组的,要么是特定位点的[gydF4y2Ba25gydF4y2Ba].DNA甲基化变化也可能与转录调控有关[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba].这些结果都表明表观遗传修饰对植物的环境敏感性和灵活性起着重要作用。gydF4y2Ba
据我们所知,目前还没有研究红树林表观遗传调控与局部适应之间关系的实验。这里,我们取样了种子gydF4y2Bak . obovatagydF4y2Ba在中国沿纬度梯度的3个种群中进行了7年的互栽试验,并结合田间普通园林试验。测量表型,包括高度、冠宽和基茎直径。利用全基因组重测序(WGRS)、转录组测序(RNA-seq)和全基因组亚硫酸盐测序(WGBS),我们的研究旨在探讨三者是否存在适应性分化gydF4y2Bak . obovatagydF4y2Ba并研究如何gydF4y2Bak . obovatagydF4y2Ba对移植引起的环境变化有反应。gydF4y2Ba
结果gydF4y2Ba
不同物种的形态差异gydF4y2Bak . obovatagydF4y2Ba来自不同种群及其表型可塑性gydF4y2Ba
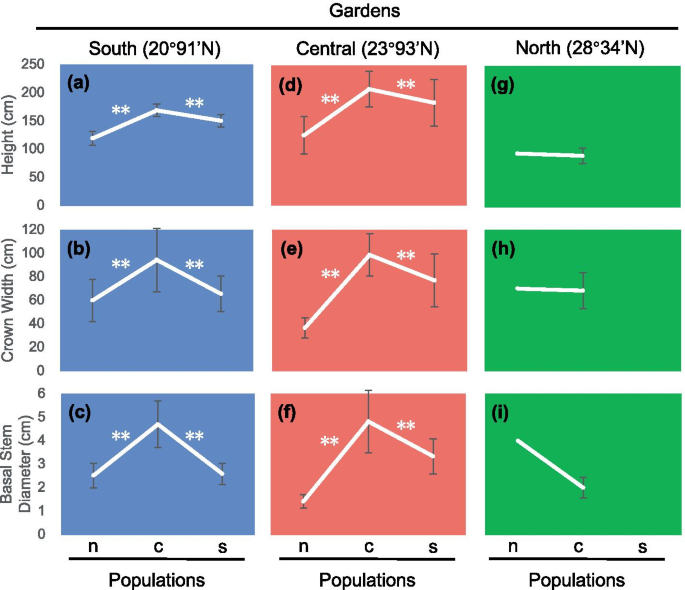
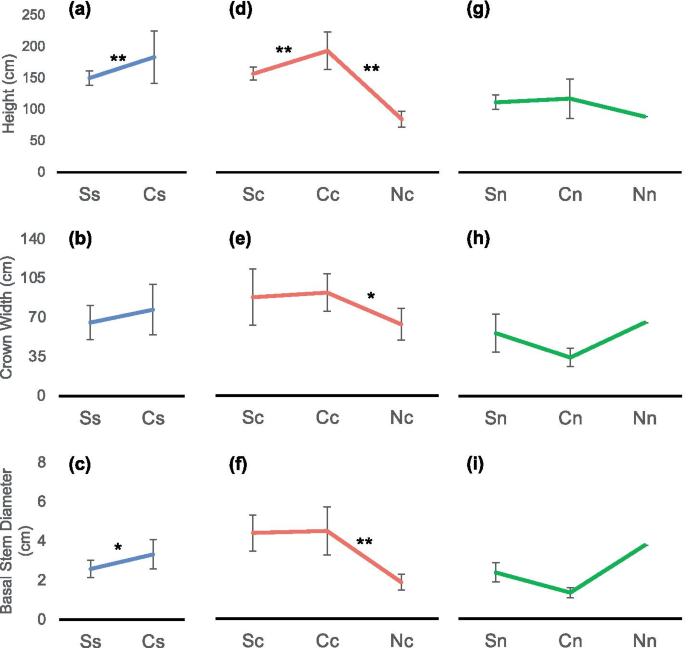
在我们的研究中,我们进行了为期7年的普通园林实验和互栽实验。在南部和中部花园中,不同种群的个体在形态上存在显著差异(图2)。gydF4y2Ba1gydF4y2Baa-f):中心群体的植株生长速度快于其他两个群体,在高度上优势明显(图2)。gydF4y2Ba1gydF4y2Baa, d),冠宽(图2)gydF4y2Ba1gydF4y2Bab, e)和茎基直径(图2)。gydF4y2Ba1gydF4y2Bac, f)。而在北园,来自南方种群的植物没有存活下来,来自中心种群的植物表现出明显的生长抑制(图2)。gydF4y2Ba1gydF4y2Bai).在中心园,本地个体在高度、冠宽和茎粗上表现出显著优势(图2)。gydF4y2Ba1gydF4y2Bad-f)。此外,中心群体在移植后表现出生长抑制(图2)。gydF4y2Ba2gydF4y2BaD-f),也表明当地适应。而在南园,来自南方种群的植物没有表现出任何地方优势(图2)。gydF4y2Ba1gydF4y2Baa - c)。此外,南方种群的植物移栽到中心园后,生长速度有所提高(图2)。gydF4y2Ba2gydF4y2Baa - c)。gydF4y2Ba
群体间的表型差异。高度平均值()gydF4y2Ba一个gydF4y2Ba), (gydF4y2BadgydF4y2Ba), (gydF4y2BaggydF4y2Ba))、冠宽(gydF4y2BabgydF4y2Ba), (gydF4y2BaegydF4y2Ba), (gydF4y2BahgydF4y2Ba))及茎基直径(gydF4y2BacgydF4y2Ba), (gydF4y2BafgydF4y2Ba), (gydF4y2Ba我gydF4y2Ba)),分别在南花园(蓝框)、中央花园(红框)和北花园(绿框)种植时从北部、中部和南部种群采集的植物。条形图表示标准误差。有显著差异* (gydF4y2BaPgydF4y2Ba< 0.05)或** (gydF4y2BaPgydF4y2Ba< 0.01)gydF4y2Ba
为gydF4y2Bak . obovatagydF4y2Ba上述各种群移植到其他纬度后,观察其形态变化(图2)。gydF4y2Ba2gydF4y2Ba).种群在表型可塑性方面存在差异(表2)gydF4y2Ba1gydF4y2Ba):中部种群在高度上、北部种群在冠宽上和基部茎粗上具有最大的表型可塑性。来自中心群体的植株的平均表型可塑性最大。gydF4y2Ba
遗传分化gydF4y2Bak . obovatagydF4y2Ba人口gydF4y2Ba
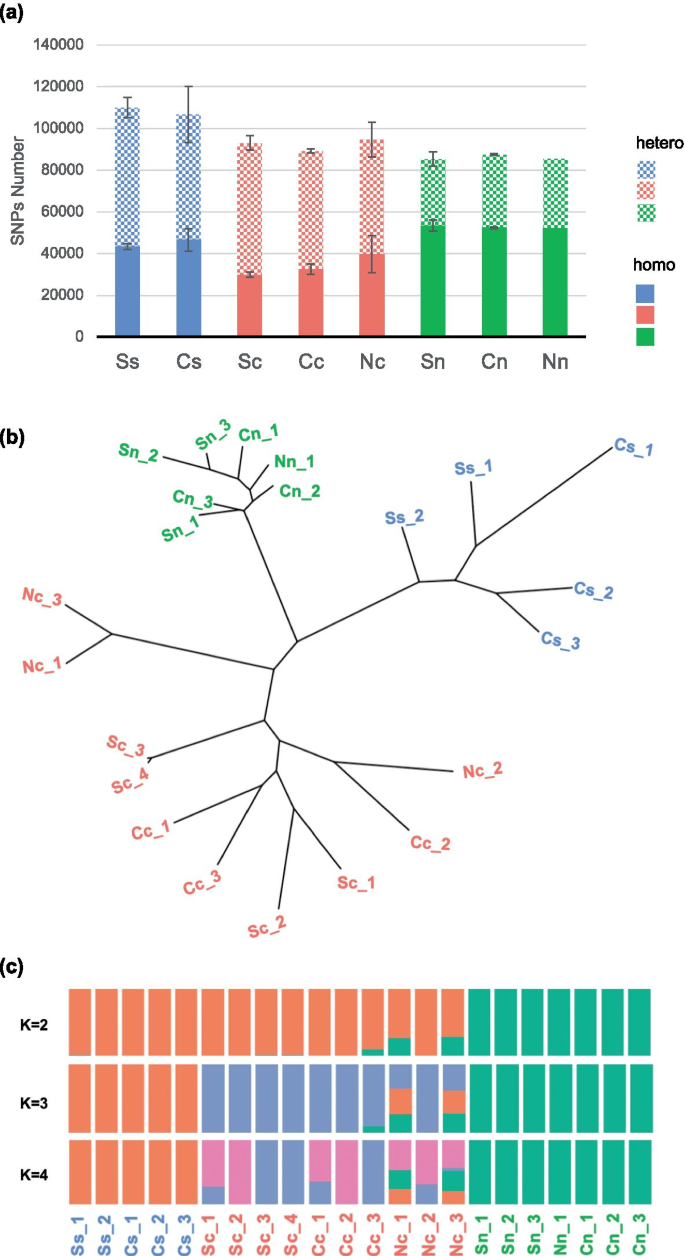
观察植物的形态分化gydF4y2Bak . obovatagydF4y2Ba在三个纬度的种群中,我们利用全基因组重测序的方法进一步探索了其遗传基础gydF4y2Bak . obovatagydF4y2Ba从这三个人群中。将修剪后的高质量reads映射到参考基因组后,总结单核苷酸多态性位点(SNPs)gydF4y2Ba1gydF4y2Ba).对于单个个体,从至少6100万次清洗读取中鉴定出84,264至112,414个snp。南方种群样本中发现的snp最多,而北方种群样本中发现的snp最少。在北方种群样本中发现的平均超过60%的snp是纯合的。中部种群纯合snp仅为36%,南部种群纯合snp仅为42%(图2)。gydF4y2Ba3.gydF4y2Ba一个)。gydF4y2Ba
单就snp数量而言,种群间的差异大于种植环境间的差异(图2)。gydF4y2Ba3.gydF4y2Baa).因此,我们预计在gydF4y2Bak . obovatagydF4y2Ba来自不同纬度的人口。22个个体的最大似然基因组谱系证实了我们的假设。gydF4y2Ba3.gydF4y2Bab).系统发育分析聚类gydF4y2Bak . obovatagydF4y2Ba个体分为三类,其分布与其种群相对应。此外,北方种群的个体之间的聚类更紧密,表明北方种群的多样性较少。gydF4y2Ba
我们进一步使用admix进行种群结构分析,该分析假设K个祖先种群,估计个体祖先和混合比例(图2)。gydF4y2Ba3.gydF4y2Bac).在K = 2时,北部种群出现为单个集群。在K = 3处的admix分析按种源分组。随着K值的增加,在中心群体中观察到相当比例的其他祖先,反映了这些群体之间的历史基因流动。gydF4y2Ba
通过系统发育和admix分析,我们发现南、中、北居群之间存在明显的遗传分化,这可能导致之前观察到的形态分化。遗传多样性在中部种群中最高,在北部种群中最低。gydF4y2Ba
移植物诱导的木质素生物合成和生物量减少的抑制gydF4y2Ba
在向北和向南的移植中均观察到移植物诱导的生物量减少(图2)。gydF4y2Ba4gydF4y2Baa).北移后单株平均生物量由2.26 kg下降至0.16 kg。对9个中心群体的叶片进行转录组测序,其中3个移栽到南园(Sc), 3个移栽到北园(Nc), 3个移栽到中心园(Cc)。表达数据的主成分分析(PCA)显示出与植物种植位置对应的明显分离(图5)gydF4y2Ba2gydF4y2Ba),表明转录组谱受到移栽的影响。相对于Cc, Sc和Nc分别显示了611和2376个差异表达基因(deg)(图2)。gydF4y2Ba4gydF4y2Bab).基因向北移植后表达异常,可能是由于中央花园与北方花园的环境差异较大。昼夜节律、类黄酮和苯丙素生物合成相关基因在DEGs富集的KEGG途径顶端占主导地位(图2)。gydF4y2BaS3-S4gydF4y2Ba).苯丙氨酸解氨酶(gydF4y2Ba朋友gydF4y2Ba在Sc和Nc中,催化苯丙素途径第一步的蛋白(pnas)均显著下调(表5)gydF4y2Ba2gydF4y2Ba,图5gydF4y2Ba5gydF4y2Ba).木质素是由苯丙类化合物合成的。此外,4-香豆酸辅酶a连接酶(gydF4y2Ba4 clgydF4y2Ba)和阿魏酸-5-羟化酶(gydF4y2BaF5HgydF4y2Ba)作为重要的木质素生物合成酶也显著下调(表5)gydF4y2Ba2gydF4y2Ba).与之前的研究一致,我们的转录组分析表明木质素的生物合成抑制和生物量减少可能是通过降低木质素的表达引起的gydF4y2Ba朋友gydF4y2Ba,gydF4y2Ba4 clgydF4y2Ba,gydF4y2BaF5HgydF4y2Ba。gydF4y2Ba
DNA甲基化组gydF4y2Bak . obovatagydF4y2Ba移栽后保持健壮,局部修饰涉及转录和翻译调控gydF4y2Ba
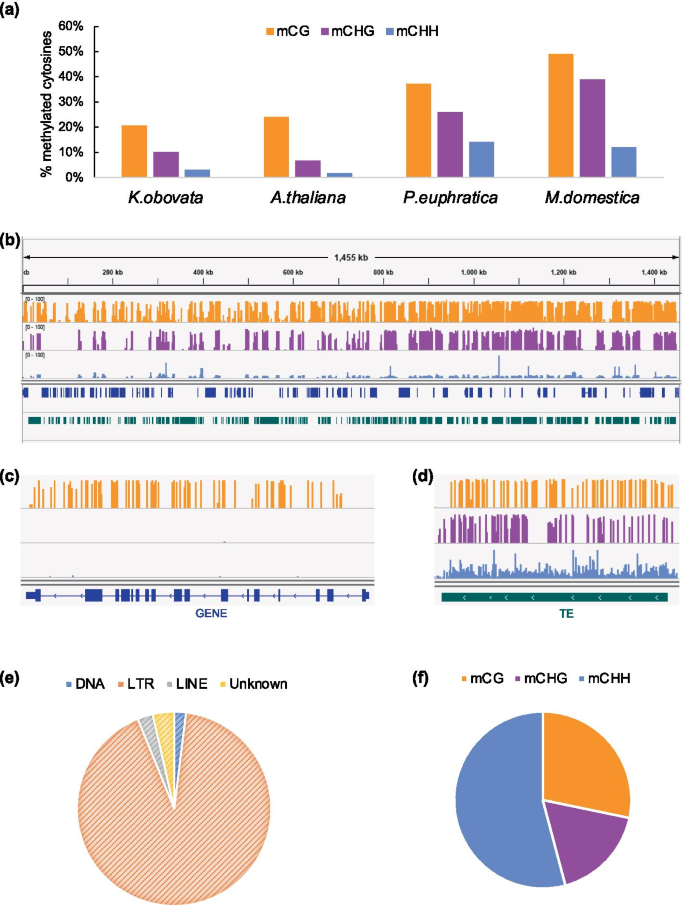
我们测序了大约7亿个对端150-bp (~ 112 Gb)的亚硫酸转化DNA(转化率> 99.8%),平均每条链的深度为~ 30 ×。在全基因组水平上,我们发现CG、CHG和CHH(其中H是腺嘌呤、胸腺嘧啶或胞嘧啶)的DNA甲基化水平分别为20.70%、10.17%和3.07%。DNA甲基化位点分布不均匀,甲基化的胞嘧啶往往位于转座子富集区域。在转座子较少的基因富集区,只观察到CG中甲基化的胞嘧啶(图2)。gydF4y2Ba5gydF4y2Bab).我们进一步研究了基因区和转座因子(TE)区域的甲基化水平。TEs如预期的那样被广泛甲基化(图2)。gydF4y2Ba5gydF4y2Bad),而只有CG中的胞嘧啶高度甲基化,而CHG和CHH的甲基化在基因体中几乎被耗尽(图2)。gydF4y2Ba5gydF4y2Bac)。gydF4y2Ba
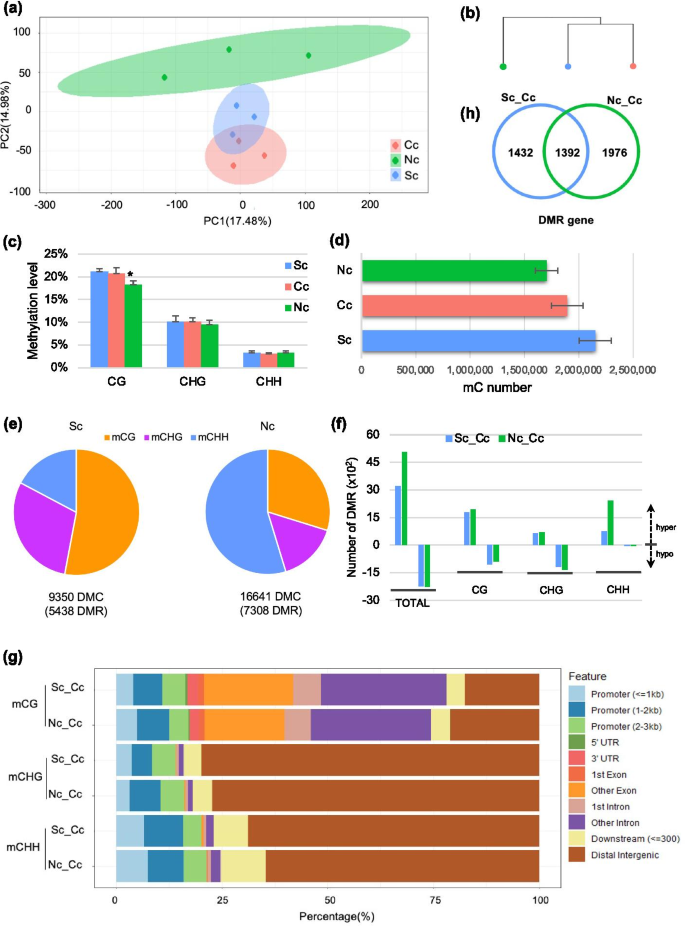
DNA甲基化水平及分布gydF4y2Bak . obovatagydF4y2Ba。(gydF4y2Ba一个gydF4y2Ba)在CG(橙色)、CHG(紫色)、CHH(蓝色)序列背景下的平均甲基化水平gydF4y2Bak . obovatagydF4y2Ba,gydF4y2Ba答:芥gydF4y2Ba,gydF4y2Ba胡杨gydF4y2Ba,gydF4y2Bam .释放有gydF4y2Ba。(gydF4y2BabgydF4y2Ba) Integrative Genomics Viewer描述了mC在Contig64中的甲基化水平和分布。(gydF4y2BacgydF4y2Ba) Integrative Genomics Viewer描述了基因上mC的甲基化水平和分布。(gydF4y2BadgydF4y2Ba) Integrative Genomics Viewer描述了mC在转座元件上的甲基化水平和分布。(gydF4y2BaegydF4y2Ba)饼状图显示了不同类型转座子对mC的偏好。(gydF4y2BafgydF4y2Ba)饼状图显示了mC对不同序列上下文的偏好gydF4y2Ba
我们将至少有5个甲基化胞嘧啶位点定义为甲基化位点(mC位点)。在整个基因组中总共检测到近200万个mC位点,覆盖了2.52%的胞嘧啶gydF4y2Bak . obovatagydF4y2Ba基因组。在这些mC位点中,约9%位于基因中,但几乎88%位于转座子中,特别是在LTR反转录转座子中(表5)gydF4y2Ba2gydF4y2Ba和无花果。gydF4y2Ba5gydF4y2Bae).至于上下文偏倚,在CHH上下文中检测到超过一半的mC位点,其中CG约占29%,CHG约占17%(图2)。gydF4y2Ba5gydF4y2Baf)。gydF4y2Ba
为了了解DNA甲基化对移植的反应,我们比较了9个中心群体样本(Sc, Nc和Cc)的甲基化组。基于甲基化谱的主成分分析(PCA)结果显示,不同纬度的甲基化谱存在明显的分离。gydF4y2Ba6gydF4y2Baa). Cc的甲基化谱与Sc的相似性高于Nc(图2)。gydF4y2Ba6gydF4y2BaB),表明甲基化谱可能是由种植环境决定的。mCGs、mCHGs和mCHHs的全基因组甲基化水平在不同样本中是相似的,除了CG水平在向北移植后有所下降(图2)。gydF4y2Ba6gydF4y2Bac),表明移植后没有发生完整的全细胞DNA甲基化重编程。纬度与mC位点的数量呈负相关,在高纬度种植的样品中检测到的mC位点较少(图2)。gydF4y2Ba6gydF4y2Bad)。gydF4y2Ba
移植诱导的DNA甲基化谱变化。(gydF4y2Ba一个gydF4y2Ba)对9个中心群体的DNA甲基化谱进行PCA分析,包括向南移植(Sc,蓝色)、本地移植(Cc,红色)和向北移植(Nc,绿色)。(gydF4y2BabgydF4y2Ba)移植和未移植的甲基化谱的相关性。(gydF4y2BacgydF4y2Ba)柱状图显示移植诱导的平均甲基化水平差异。(gydF4y2BadgydF4y2Ba)移植和未移植的甲基化胞嘧啶数。(gydF4y2BaegydF4y2Ba)移植诱导的DMCs的总数和序列上下文分解。(gydF4y2BafgydF4y2Ba)甲基化的数量和方向在CG, CHG, CHH发生变化。(gydF4y2BaggydF4y2Ba)堆叠条形图显示了与各种注释基因区域重叠的DMRs的百分比,包括启动子、utr、外显子、内含子和远端基因间区,相对于在Nc或Sc的每个序列上下文中与这些基因特征相关的DMRs的总数。(gydF4y2BahgydF4y2Ba) Sc(蓝圈)和Nc(绿圈)的dmr基因数目gydF4y2Ba
为了研究DNA甲基化在局部发生的动态调控程度,研究人员进一步鉴定了差异甲基化胞嘧啶(DMCs)和差异甲基化区域(DMRs)。我们发现向北移栽(Nc)比向南移栽(Sc)诱导的DMC更多。gydF4y2Ba6gydF4y2Bae)。在CHH序列背景下,Nc中的dmc主要由胞嘧啶组成,而在Sc样品中,CG序列背景下的dmc最丰富(图2)。gydF4y2Ba6gydF4y2Bae)。我们在所有序列背景下观察到DMRs的高甲基化和低甲基化(图2)。gydF4y2Ba6gydF4y2Baf)。此外,我们发现CHG和CHH序列背景下的DMRs在基因间区域强烈富集(图2)。gydF4y2Ba6gydF4y2Bag),而CG序列背景下的DMRs主要发生在基因体(外显子和内含子)。gydF4y2Ba
对移植诱导的DMRs进行注释后(图2)。gydF4y2Ba6gydF4y2Bae)根据其遗传位置,我们分别在Nc和Sc中获得了3368和2824个DMR基因(图2)。gydF4y2Ba6gydF4y2Bah).为了探索这些差异甲基化基因的生物学作用,我们进行了KEGG富集分析。对于dmr基因,在Nc和Sc中,“剪接体”和“RNA运输”途径都显著富集(图2)。gydF4y2BaS6-S7gydF4y2Ba),提示移植诱导的DNA甲基化的局部修饰gydF4y2Bak . obovatagydF4y2Ba参与转录和翻译的调控。gydF4y2Ba
移植后基因表达变化与DNA甲基化修饰的相关性gydF4y2Bak . obovatagydF4y2Ba。gydF4y2Ba
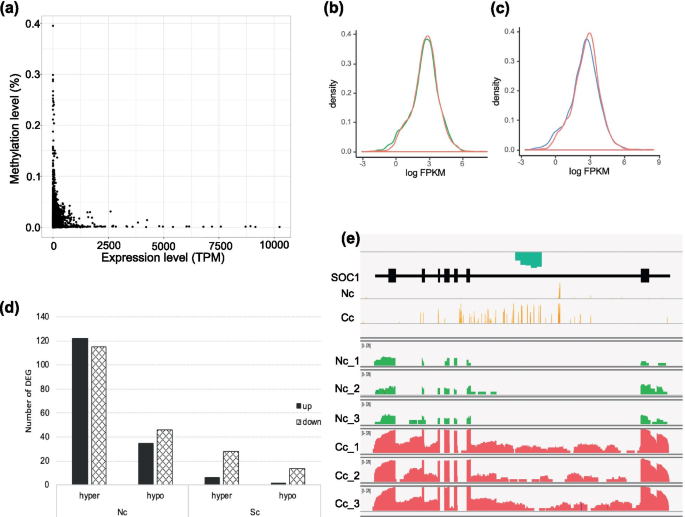
在23683个基因中gydF4y2Bak . obovatagydF4y2Ba在基因组中,高度甲基化的基因很少表达;高表达基因通常甲基化水平很低(图2)。gydF4y2Ba7gydF4y2Baa).移植诱导的DMR基因的转录谱发生了变化(图2)。gydF4y2Ba7gydF4y2Bab, c)。此外,我们注意到超过80%的deg甲基化水平极低(图2)。gydF4y2BaS8-S9gydF4y2Ba),表明DNA甲基化在维持基因表达稳定性中的作用。在2759个移植诱导的deg中,只有347个基因检测到DMRs。本研究未观察到dmr和deg的强烈重叠(图2)。gydF4y2Ba7gydF4y2Bad),表明DNA甲基化可能以一种相当复杂的方式先于转录变化。gydF4y2Ba
DNA甲基化与基因表达的相关性。(gydF4y2Ba一个gydF4y2Ba)中所有基因的DNA甲基化水平和表达水平gydF4y2Bak . obovatagydF4y2Ba。(gydF4y2BabgydF4y2Ba) dmr基因在Nc(绿线)相对于Cc(红线)的表达量密度曲线。(gydF4y2BacgydF4y2Ba) dmr基因在Sc(蓝线)相对于Cc(红线)的表达量密度曲线。(gydF4y2BadgydF4y2Ba) DEGs与DNA甲基化修饰的相关性。(gydF4y2BaegydF4y2Ba) SOC1在Cc和Nc中的表达水平和甲基化水平可视化。橙色:CG环境中的甲基化水平。蓝框:低甲基化区域。绿色峰:RNA-seq在Nc的读取覆盖率。红色峰:RNA-seq reads coverage in CcgydF4y2Ba
在347个差异表达和差异甲基化基因中,有27个被认为是转录因子(表1)。年代gydF4y2Ba3.gydF4y2Ba).值得注意的是,MADS-box家族的转录因子在花的形成和开花时间的控制中最为丰富。这5个MADS-box基因在向北移植后均显著下调。此外,在这五个基因的内含子中观察到CG背景下的去甲基化(表S)gydF4y2Ba4gydF4y2Ba,无花果。gydF4y2Ba7gydF4y2Bae)。gydF4y2Ba
讨论gydF4y2Ba
k . obovatagydF4y2Ba是中国分布最广泛的红树物种之一。的中心gydF4y2Bak . obovata的gydF4y2Ba地理分布位于中国云霄附近。南(雷州)和中(云霄)种群的植物在中心园(云霄)的生长速度比南园(雷州)快(图2)。gydF4y2Ba2gydF4y2Baf)。对于北方种群,种植在北园的个体生物量高于种植在中心园的个体(图2)。gydF4y2Ba2gydF4y2Ba胃肠道)。此外,北方种群在北方园林中表现出优势,说明北方种群的耐寒能力较强gydF4y2Bak . obovatagydF4y2Ba来自北方的人口。在南部、中部和北部人群中发现了遗传分化(图2)。gydF4y2Ba3.gydF4y2Ba),这可能是这三种人群对不同纬度的不同偏好的原因。北方居群遗传多样性相对较低。世界自然保护联盟认为,遗传多样性是值得保护的生物多样性之一。gydF4y2Ba30.gydF4y2Ba].此外,遗传多样性低的红树林物种更容易受到破坏[gydF4y2Ba31gydF4y2Ba].北方种群规模小,抗寒能力强,需要对北方种群进行有效的保护。gydF4y2Ba
的过程gydF4y2Bak . obovatagydF4y2Ba植树造林,将个体移植到一个新的环境中,本质上是人类控制的迁移事件。表型可塑性为植物提供了一种适应广泛环境变化的机制[gydF4y2Ba32gydF4y2Ba].遗传变异有助于提高存活率[gydF4y2Ba32gydF4y2Ba].有许多研究表明,遗传多样性和表型可塑性有助于植物在新环境中生存[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].高遗传多样性和表型可塑性(图2)。gydF4y2Ba2gydF4y2Bad-f,无花果。gydF4y2Ba3.gydF4y2Ba)使中部(云霄)种群的种子成为种植的最佳选择。然而,北方(宁德)人口耐寒性强,可能更适合种植在高纬度地区。gydF4y2Ba
迁移到其他纬度后,云霄种群的个体生物量呈下降趋势(图2)。gydF4y2Ba4gydF4y2Baa).对…的研究gydF4y2Ba杨树gydF4y2Ba观察到的负相关(gydF4y2BargydF4y2Ba=−0.48;gydF4y2BaPgydF4y2Ba值< 0.0001),生物量和木质素含量[gydF4y2Ba35gydF4y2Ba].白杨的突变体(gydF4y2Ba美洲山杨gydF4y2Ba),木质素减少45%,显示4-香豆酸辅酶A连接酶(gydF4y2Ba4 clgydF4y2Ba)基因。木质素是由植物苯丙素途径产生的。在我们的例子中,苯丙氨酸解氨酶(gydF4y2Ba朋友gydF4y2Ba),它催化苯丙素途径的第一步[gydF4y2Ba36gydF4y2Ba],在其原生栖息地外种植后显著减少。此外,单脂醇类生物合成酶、gydF4y2Ba4 clgydF4y2Ba和gydF4y2BaF5HgydF4y2Ba,两者都显著降低。抑制gydF4y2Ba朋友gydF4y2Ba,gydF4y2Ba4CL或F5HgydF4y2Ba导致植物中木质素含量降低[gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba];和gydF4y2Ba朋友gydF4y2Ba敲除突变体发育迟缓且不育[gydF4y2Ba36gydF4y2Ba].在植物中,木质素是次生细胞壁的一种成分[gydF4y2Ba41gydF4y2Ba].树木重量的很大一部分是由木质素组成的,因为树木的木材主要由维管组织的次级细胞壁和伴随的纤维组成[gydF4y2Ba42gydF4y2Ba].我们推测移植诱导的生物量减少与木质素生物合成基因表达的抑制有关,如gydF4y2Ba朋友gydF4y2Ba,gydF4y2Ba4 clgydF4y2Ba,gydF4y2BaF5HgydF4y2Ba。gydF4y2Ba
DNA甲基化是一种保守的表观遗传修饰,在基因调控中起重要作用[gydF4y2Ba25gydF4y2Ba].DNA甲基化模式可以广泛反映植物物种的进化和生活史[gydF4y2Ba43gydF4y2Ba].在被子植物中,DNA甲基化存在广泛的差异[gydF4y2Ba43gydF4y2Ba].gydF4y2Bak . obovatagydF4y2Ba与其他木本植物相比,甲基化水平相对较低(21%,10%,3%)。受欢迎(37%,26%,14%)[gydF4y2Ba44gydF4y2Ba或苹果(49%,39%,12%)[gydF4y2Ba45gydF4y2Ba],但其水平与gydF4y2Ba拟南芥gydF4y2Ba(24%, 7%, 2%) [gydF4y2Ba46gydF4y2Ba].先前的研究表明,基因组甲基化与基因组大小有关,即基因组大小较大的被子植物,其基因组甲基化的比例较高[gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba].苹果的基因组大小(651 Mb) [gydF4y2Ba45gydF4y2Ba]及杨树(485 Mb) [gydF4y2Ba49gydF4y2Ba比人类的基因组还要大gydF4y2Bak . obovatagydF4y2Ba(278 Mb),这可能解释了gydF4y2Bak . obovatagydF4y2Ba(无花果。gydF4y2Ba5gydF4y2Baa).越来越多的证据表明,植物DNA甲基化在单个位点或整个基因组上发生了环境响应性改变,但DNA甲基化修饰仅解释了有限的基因表达变化[gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba].DNA甲基化和基因表达之间的关联是复杂的,并且依赖于上下文[gydF4y2Ba53gydF4y2Ba].在植物中,DNA甲基化的作用取决于潜在的序列和基因组中的位置,而不是简单地打开或关闭[gydF4y2Ba54gydF4y2Ba].我们在移植诱导的MADS-box下调基因中观察到CG去甲基化,且去甲基化均发生在内含子中(表S)gydF4y2Ba4gydF4y2Ba).发现DNA甲基化在春化和开花过程中是必不可少的[gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba].MADS-box基因gydF4y2Ba富尔语gydF4y2Ba,gydF4y2Ba高级副总裁gydF4y2Ba,gydF4y2BaSOC1gydF4y2Ba,据报道涉及调节开花时间[gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba].在植物的一生中,开花是一个关键的发育阶段,非常容易受到环境压力的影响。调整开花时间是植物的进化策略[gydF4y2Ba65gydF4y2Ba].此外,gydF4y2BaSOC1gydF4y2Ba已经被证明可以解释冷反应和开花之间的串扰。的gydF4y2Basoc1gydF4y2Ba突变体的耐寒性增强,开花时间延迟[gydF4y2Ba66gydF4y2Ba].有利于生长和繁殖的季节在地理上是可变的。一般来说,开花时间遵循纬度梯度。例如,gydF4y2Ba拟南芥gydF4y2Ba北方开花较晚,南方开花较早[gydF4y2Ba67gydF4y2Ba].当移植到其他纬度时,DNA甲基化修饰可能会有所帮助gydF4y2Bak . obovatagydF4y2Ba通过调整开花时间来适应新的环境。我们确实注意到了gydF4y2Bak . obovatagydF4y2Ba在某些情况下,南方普通花园的植物开花较早。然而,我们通常每月一次参观花园,时间跨度太长,无法准确确定三个花园中植物的实际开花时间。具体的调控机制还需要进一步的研究和实验验证。gydF4y2Ba
结论gydF4y2Ba
在本研究的基础上,我们观察了不同品种间的形态差异和遗传分化gydF4y2Bak . obovatagydF4y2Ba来自不同的人群,呼吁更多的关注和保护gydF4y2Bak . obovatagydF4y2Ba特别是来自北方(宁德)种群,遗传多样性低,种群规模小,但抗寒性突出。此外,该研究可为人工林选择合适的播种方式以实现有效恢复提供科学依据。通过对移栽事件的表型、转录和表观遗传反应的探索,我们的工作揭示了移栽事件的分子机制gydF4y2Bak . obovatagydF4y2Ba应对环境变化。gydF4y2Ba
方法gydF4y2Ba
植物生长和表型测定gydF4y2Ba
这项研究是在中国南部沿海的开阔泥滩上进行的。沿纬度梯度选择雷州(北纬20°91)、云霄(北纬23°93)和乐清(北纬28°34)3个种植地点。下胚轴的gydF4y2Bak . obovatagydF4y2Ba于2011年春季在雷州(北纬20°91′)、云孝(北纬23°93′)和宁德(北纬27°28′)的天然红树林中采集,分别在选定的种植地点依次移植到潮间带泥滩的普通园林中(图5)gydF4y2Ba1gydF4y2Ba).我们的研究样本来自野外,不需要获得许可。张一辉教授对样品进行了正式鉴定,并提供了存放凭证标本的详细信息。我们的植物实验符合机构、国家或国际准则。我们的实地研究是按照当地法律进行的。所有必要的种植许可均从中国厦门大学获得。gydF4y2Ba
2018年3月下旬至4月上旬,我们在每个站点随机选择来自三个不同纬度的个体,每个纬度有三个生物重复。由于标签错误和自然灾害造成的死亡,获得了22人(表S)gydF4y2Ba1gydF4y2Ba),包括3个c(采自南方种群种植在中心园)、2个s(采自南方种群种植在南园)、4个Sc(采自中心种群种植在南园)、3个Cc(采自中心种群种植在中心园)、3个Nc(采自中心种群种植在北园)。3个Sn(采集自北方种群,种植于南园),3个Cn(采集自北方种群,种植于中心园),1个Nn(采集自北方种群,种植于北园)。在每个花园中,新鲜的叶子被收集起来,并在9点到11点之间迅速冷冻在干冰中。在现场测量表型,包括株高、基茎直径(距土壤表面30 cm)和冠宽(冠层长轴和短轴长度)。为了比较移植引起的表型变化程度,计算了各种群和各变量的表型可塑性指数,即三种种植环境中最大和最小平均值之差除以最大平均值。gydF4y2Ba
为了研究移植引起表型改变的机制,我们重点研究了9个中心种群个体(表S中的黄色个体)gydF4y2Ba1gydF4y2Ba)和地上生物量(gydF4y2BawgydF4y2BaTgydF4y2Ba),根据树高(H)和茎粗(D) [gydF4y2Ba68gydF4y2Ba]:gydF4y2Ba
文库制备及测序gydF4y2Ba
对于每个样本,从同一片叶子中获得dna和rna。总rna采用RNAprep Pure Plant Plus Kit (TIANGEN, Beijing, China, DP441)提取。基因组总dna通过DNAquick Plant System (TIANGEN, Beijing, China, DP321)提取。gydF4y2Ba
对于RNA-seq,按照制造商的建议使用NEBNext®UltraTM RNA Library Prep Kit For Illumina®(NEB, USA)构建文库,并在每个样品的属性序列中添加索引代码。简单地说,利用聚t寡聚磁珠从总RNA中纯化mRNA。在NEBNext第一链合成反应缓冲液(5X)中,用二价阳离子在高温下进行裂解。用随机六聚体引物和M-MuLV逆转录酶(RNase H)合成第一链cDNA,随后用DNA聚合酶I和RNase H合成第二链cDNA,剩余的悬垂部分通过核酸外切酶/聚合酶活性转化为钝端。DNA片段3′端腺苷化后,连接具有发夹环结构的NEBNext适配器,准备杂交。250 ~ 300 bp的片段用AMPure XP系统(Beckman Coulter, Beverly, USA)纯化。然后用3 μl USER酶(NEB, USA)与大小选定的接头连接cDNA在37℃下孵育15 min, 95℃下孵育5 min,再进行PCR。然后用Phusion高保真DNA聚合酶、通用PCR引物和Index (X)引物进行PCR。最后,用AMPure XP系统纯化PCR产物,并在Agilent Bioanalyzer 2100系统上评估文库质量。gydF4y2Ba
每个样本的dna被分成两部分,分别用于全基因组重测序(WGRS)和全基因组亚硫酸盐测序(WGBS)。WGRS文库采用Truseq纳米DNA HT样品制备试剂盒(Illumina USA),以1.5 μg DNA为输入,按照说明书进行制备。简单地说,通过超声波将DNA片段化至350bp,然后将DNA片段末端抛光,a尾,与全长适配器连接,用于Illumina测序并进一步进行PCR扩增。最后用AMPure XP系统对PCR产物进行纯化。gydF4y2Ba
对于WGBS库,采用了与WGRS类似的策略。但与WGRS不同的是,DNA片段化到200-300 bp,条形码被胞嘧啶甲基化。使用EZ DNA甲基化- goldtm Kit (zimo Research)用亚硫酸氢盐处理DNA片段两次,然后使用KAPA HiFi HotStart Uracil + ReadyMix (2X)扩增亚硫酸氢盐转化的DNA片段。gydF4y2Ba
使用Agilent2100生物分析仪分析所有文库的大小分布,并使用实时荧光定量PCR进行定量。随后,在Novogene公司(北京,中国)使用Illumina Hiseq X平台对文库进行测序,生成了150 bp的成对末端reads (Raw reads被放置在GEO中,提交号为SUB7839708)。gydF4y2Ba
测序数据分析gydF4y2Ba
对没有测序适配器的reads和Trimmomatic [gydF4y2Ba69gydF4y2Ba].从reads的两端丢弃低质量核苷酸(Q < 20),并去除短于50 bp的reads (SLIDINGWINDOW 5:20, LEADING 5, MINLEN 50)。gydF4y2Ba
对于每个个体,修剪后的读取被映射到gydF4y2Bak . obovatagydF4y2Ba参考基因组与Burrows-Wheeler Aligner (BWA v.0.7.17) [gydF4y2Ba70gydF4y2Ba],使用默认参数。使用GATK (v.4.0.10.1)标记重复读取并执行变体调用[gydF4y2Ba71gydF4y2Ba].然后使用GATK对变异进行筛选,并使用SNPhylo使用最大似然法构建系统发育关系[gydF4y2Ba72gydF4y2Ba].使用带有Admixture的合格snp估计个体的混杂[gydF4y2Ba73gydF4y2Ba].gydF4y2Ba
修剪后的RNA-seq reads被映射到gydF4y2Bak . obovatagydF4y2Ba参考基因组使用HISAT2 [gydF4y2Ba74gydF4y2Ba].定位后,使用StringTie将RNA-Seq比对组合成潜在转录本[gydF4y2Ba75gydF4y2Ba].用DEseq2测定差异表达基因[gydF4y2Ba76gydF4y2Ba在R中实现。gydF4y2Ba
对于修剪后的WGBS reads,使用Bismark (v.0.20.0)进行定位和甲基化调用[gydF4y2Ba77gydF4y2Ba].对于每个样本,读取与样本特异性修改的伪参考对齐,其中纯合snp插入到dna序列中gydF4y2Bak . obovatagydF4y2Ba参考基因组。使用methylKit (v.1.10.0)鉴定差异甲基化胞嘧啶(DMCs)和差异甲基化区域(DMRs) [gydF4y2Ba78gydF4y2Ba].在鉴定DMRs时,对基因组进行了100 bp长度和100 bp步长窗口的平铺,并总结了这些平铺上的甲基化信息。此外,对于CG、CHG和CHH上下文序列,最大和最小甲基化水平差异分别小于40%、20%和10%的DMC/DMR候选序列被丢弃。gydF4y2Ba
基因表达的qRT-PCR验证gydF4y2Ba
采用qRT-PCR法检测PAL、4CL、F5H的表达水平。使用SMARTScribe逆转录酶(Clontech)合成第一链cDNA,然后根据制造商的说明,在Bio-Rad CFX 96 (Bio-Rad, Inc.)上使用SYBR预混料进行qRT-PCR。三个生物重复的相对定量归一化到内参基因18S rRNA(加入号:18S rRNA)。AY289625),用2−ΔΔCt方法计算。所有引物序列见表SgydF4y2Ba5gydF4y2Ba。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
本研究产生的测序数据已存入NCBI,并可在gydF4y2Bahttps://dataview.ncbi.nlm.nih.gov/object/PRJNA648812?reviewer=90naarf2cn2ua81g2r0aec5m3agydF4y2Ba。gydF4y2Ba
缩写gydF4y2Ba
- k . obovata:gydF4y2Ba
-
Kandelia obovatagydF4y2Ba
- 水气比:gydF4y2Ba
-
全基因组重测序gydF4y2Ba
- WGBS:gydF4y2Ba
-
亚硫酸盐全基因组测序gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- 度:gydF4y2Ba
-
差异表达基因gydF4y2Ba
- KEGG:gydF4y2Ba
-
京都基因和基因组百科全书gydF4y2Ba
- 朋友:gydF4y2Ba
-
苯丙氨酸ammonia-lyasegydF4y2Ba
- 4 cl:gydF4y2Ba
-
4-Coumarate:辅酶a连接酶gydF4y2Ba
- F5H:gydF4y2Ba
-
Ferulate-5-hydroxylasegydF4y2Ba
- TE:gydF4y2Ba
-
转座因子gydF4y2Ba
- 主成分分析:gydF4y2Ba
-
主成分分析gydF4y2Ba
- DMC:gydF4y2Ba
-
差异甲基化胞嘧啶gydF4y2Ba
- DMR:gydF4y2Ba
-
差异甲基化区gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
热带红树林生态系统。第1版。美国地球物理联合会;1992.doi:gydF4y2Bahttps://doi.org/10.1029/CE041gydF4y2Ba。gydF4y2Ba
- 2.gydF4y2Ba
红树林生态学、造林与保护。施普林格科技与商业传媒;2002.gydF4y2Ba
- 3.gydF4y2Ba
[3]李建平,李建平。红树林生态系统的研究进展。见:海洋生物学进展。爱思唯尔;2001.p . 81 - 251。doi:gydF4y2Bahttps://doi.org/10.1016/s0065 - 2881 (01) 40003 - 4gydF4y2Ba。gydF4y2Ba
- 4.gydF4y2Ba
李建军,李建军,李建军,等。恢复红树林的功能研究进展。水生生物学报,2008;89:251-9。gydF4y2Ba
- 5.gydF4y2Ba
Valiela I, Bowen JL, York JK。红树林:世界上受威胁的主要热带环境之一:在过去的二十年中,至少有35%的红树林面积已经消失,其损失超过了热带雨林和珊瑚礁这两个众所周知的受威胁环境。生物科学。2001;51:807-15。gydF4y2Ba
- 6.gydF4y2Ba
Hamilton SE, Casey D. 21世纪高时空分辨率全球红树林连续覆盖数据库(CGMFC-21)的建立。生态学报,2016;25(5):729 - 738。gydF4y2Ba
- 7.gydF4y2Ba
贾敏,王忠,李磊,宋凯,任超,刘斌,等。基于陆地卫星图像面向对象分类的中国红树林制图。湿地。2014;34:277 - 83。gydF4y2Ba
- 8.gydF4y2Ba
贾米、王Z,张Y,毛D,王c .监测损失和恢复红树林在42年:在中国红树林保护的成就。[J] .地球物理学报,2018;33(3):533 - 536。gydF4y2Ba
- 9.gydF4y2Ba
埃里森。红树林恢复:我们知道的够多吗?中国生物医学工程学报,2000;8(1):391 - 391。gydF4y2Ba
- 10.gydF4y2Ba
世界红树林的现状和未来。环境保护。2002;29:331-49。gydF4y2Ba
- 11.gydF4y2Ba
费军,王莹,程华,苏艳,钟艳,郑丽。低温胁迫下红树甘花库斯莫汀的克隆及特性研究。植物化学学报,2013;21(1):1 - 10。gydF4y2Ba
- 12.gydF4y2Ba
陈刚,戴超,李勇,徐军。福建沿海地区红树林植物形态特征比较。安徽农业科学,2016;44:178-182 +253。gydF4y2Ba
- 13.gydF4y2Ba
石霞,潘丽,陈强,黄磊,王伟。红树矮化原因分析gydF4y2BaKandelia obovatagydF4y2Ba在山城湾。湿地科学,2016;14:648-53。gydF4y2Ba
- 14.gydF4y2Ba
李建军,李建军,李建军,等。生态学报。2004;7(1):1 - 4。gydF4y2Ba
- 15.gydF4y2Ba
雷木,李建平,李建平,等。植物本地适应性的meta分析。PLoS ONE。2008; 3: e4010。gydF4y2Ba
- 16.gydF4y2Ba
Ågren J, Schemske DW。互惠移植表明模式生物拟南芥在其原生范围内具有很强的适应性分化。新植物学报,2012;19(4):1112 - 22。gydF4y2Ba
- 17.gydF4y2Ba
A. DNA甲基化景观:来自表观基因组学的挑衅性见解。宗教信仰。2008;9:465-76。gydF4y2Ba
- 18.gydF4y2Ba
引用本文:Ronemus MJ, Galbiati M, Ticknor C, Chen J, Dellaporta SL.拟南芥去甲基化诱导发育多效性。科学。1996;273:654-7。gydF4y2Ba
- 19.gydF4y2Ba
Burn JE, Smyth DR, Peacock WJ, Dennis ES。拟南芥开花晚的基因。遗传。1993;90:147-55。gydF4y2Ba
- 20.gydF4y2Ba
李文杰,李文杰,李文杰。拟南芥DNA甲基化降低导致植物发育异常。科学通报,1996;33(3):849 - 854。gydF4y2Ba
- 21.gydF4y2Ba
Kooke R, Johannes F, Wardenaar R, Becker F, Etcheverry M, Colot V,等。拟南芥形态变异和表型可塑性的表观遗传基础。植物细胞。2015;27:37 - 48。gydF4y2Ba
- 22.gydF4y2Ba
钟松,费震,陈永荣,郑勇,黄明,Vrebalov J,等。番茄果实发育的单碱基分辨率甲基组揭示了与成熟相关的表观基因组修饰。生物医学工程学报,2013;31(1):559 - 559。gydF4y2Ba
- 23.gydF4y2Ba
李建平,李建平,李建平,等。珊瑚礁珊瑚生殖系DNA甲基化:对环境变化的响应模式和潜在作用。中国生物医学工程学报,2016;25(5):591 - 591。gydF4y2Ba
- 24.gydF4y2Ba
李高L,耿Y, B,陈J,杨J .全基因组DNA甲基化改变其中的、在自然和操纵的栖息地:影响表观遗传调节的快速响应环境波动和表型变异。植物学报,2010;33(3):1820 - 1827。gydF4y2Ba
- 25.gydF4y2Ba
张宏,郎志,朱建科。植物DNA甲基化的动态和功能。中华生物医学工程学报,2018;19(1):489 - 496。gydF4y2Ba
- 26.gydF4y2Ba
徐锐,王勇,郑华,陆伟,吴超,黄军,等。盐诱导的转录因子MYB74在拟南芥中受rna导向的DNA甲基化途径调控。[J] .中国生物医学工程学报,2015;33(1):591 - 598。gydF4y2Ba
- 27.gydF4y2Ba
张斌,铁满德明,焦超,徐勇,陈坤,费震,等。冷藏诱导的番茄风味损失与挥发性合成的改变和DNA甲基化的短暂变化有关。《美国国家科学院学报》上。2016; 113:12580-5。gydF4y2Ba
- 28.gydF4y2Ba
李建军,李建军,李建军,等。冬小麦春化处理诱导春化a1位点的DNA超甲基化。植物生理学报,2013;13(3):591 - 591。gydF4y2Ba
- 29.gydF4y2Ba
Yong-Villalobos L, González-Morales SI, Wrobel K, gutisamurez - alanis D, cervantes - persamuz SA, Hayano-Kanashiro C,等。甲基组分析揭示了表观遗传变化在拟南芥对磷酸盐饥饿反应的调控中的重要作用。美国国家科学促进会。2015; 112: e7293 - 7302。gydF4y2Ba
- 30.gydF4y2Ba
李建平,李建平。遗传多样性与适应性的关系。生态学报,2003;17(2):393 - 398。gydF4y2Ba
- 31.gydF4y2Ba
郭忠,李旭,何忠,杨勇,王伟,钟超,等。红树林分类群极低的遗传多样性反映了过去的海平面变化,并暗示未来的反应很差。地球变化学报,2018;24(4):1741 - 1741。gydF4y2Ba
- 32.gydF4y2Ba
张建军,张建军,张建军,等。生物多样性与生物多样性研究进展。生态学报,2002;12(2):1652 - 60。gydF4y2Ba
- 33.gydF4y2Ba
Frenot Y, Aubry M, Misset MT, Gloaguen JC, Gourret JP, Lebouvier M.亚南极Crozet和Kerguelen群岛黄花蒿(Poa annua L.)表型可塑性和遗传多样性。极地生物学报。1999;22:302-10。gydF4y2Ba
- 34.gydF4y2Ba
耿勇,van Klinken RD, Sosa A,李斌,陈健,徐春云。遗传多样性和表型可塑性在决定美国和中国无性系杂草入侵成功中的相对重要性。植物学报,2016;7。doi:gydF4y2Bahttps://doi.org/10.3389/fpls.2016.00213gydF4y2Ba。gydF4y2Ba
- 35.gydF4y2Ba
李建军,李建军,李建军,等。木质素对树木生物量的影响。植物生理学报。2010;24(4):555 - 561。gydF4y2Ba
- 36.gydF4y2Ba
黄军,顾明,赖志,范斌,石凯,周永华,等。拟南芥PAL基因家族在植物生长发育和环境胁迫响应中的功能分析植物生理学报。2010;33(3):526 - 538。gydF4y2Ba
- 37.gydF4y2Ba
Sewalt V, Ni W, Blount JW, Jung HG, Masoud SA, Howles PA,等。转基因烟草木质素含量降低,木质素组成改变,l -苯丙氨酸解氨酶和肉桂酸4-羟化酶表达下调。植物生理学报。1997;15(1):41 - 50。gydF4y2Ba
- 38.gydF4y2Ba
Shafrin F, Das SS, Sanan-Mishra N, Khan H.人工mirna介导的两个单酚类生物合成基因(C3H和F5H)的下调导致黄麻木质素含量降低。植物化学学报。2015;39(1):511 - 527。gydF4y2Ba
- 39.gydF4y2Ba
Elkind Y, Edwards R, Mavandad M, Hedrick SA, Ribak O, Dixon RA,等。含异源苯丙氨酸解氨酶基因转基因烟草植株发育异常及苯丙氨酸生物合成下调。《美国国家科学院学报》上。1990; 87:9057 - 61。gydF4y2Ba
- 40.gydF4y2Ba
陈芳,刘建军,刘建军,刘建军,刘建军。靶向下调紫花苜蓿细胞色素P450酶对饲料品质改善的影响《美国国家科学院学报》上。2005; 102:16573-8。gydF4y2Ba
- 41.gydF4y2Ba
Ralph J, Lundquist K, Brunow G, Lu F, Kim H, Schatz PF,等。木质素 :天然聚合物氧化偶联的4-羟基苯基丙烷。植物化学评论Vol 3 (2004): p 29-60。2004.gydF4y2Bahttps://www.fs.usda.gov/treesearch/pubs/22070gydF4y2Ba。2021年4月19日访问。gydF4y2Ba
- 42.gydF4y2Ba
李建平,李建平。木质素生物合成的基因型与表型的关系。科学通报。2010;44(4):337 - 63。gydF4y2Ba
- 43.gydF4y2Ba
Niederhuth CE, Bewick AJ, Ji L, Alabady MS, Kim KD, Li Q,等。被子植物DNA甲基化的广泛自然变异。中国生物医学工程学报。2016;17:194。gydF4y2Ba
- 44.gydF4y2Ba
苏勇,白霞,杨伟,王伟,陈志,马军,等。胡杨DNA甲基化与盐胁迫的关系基因工程学报,2018;14:86。gydF4y2Ba
- 45.gydF4y2Ba
Daccord N, Celton J-M, Linsmith G, Becker C, Choisne N, Schijlen E,等。苹果基因组的高质量从头组装和早期果实发育的甲基组动力学。生物医学工程学报,2017;49:1099-106。gydF4y2Ba
- 46.gydF4y2Ba
Schmitz RJ, Schultz MD, Urich MA, Nery JR, Pelizzola M, Libiger O,等。群体表观基因组多样性模式。大自然。2013;495:193-8。gydF4y2Ba
- 47.gydF4y2Ba
陈建辉,高永平。基因DNA甲基化的进化模式因陆地植物而异。植物学报,2016;2:1-7。gydF4y2Ba
- 48.gydF4y2Ba
阿隆索C, psamez R, Bazaga P, Herrera CM。全球DNA胞嘧啶甲基化作为一种进化特征:被子植物的系统发育信号及其与基因组大小的相关进化。5 .《前沿杂志》,2015;doi:gydF4y2Bahttps://doi.org/10.3389/fgene.2015.00004gydF4y2Ba。gydF4y2Ba
- 49.gydF4y2Ba
杜思康,杨建军,李建军,等。黑棉杨(Populus trichocarpa)的基因组。&灰色)。科学。2006;313:1596 - 604。gydF4y2Ba
- 50.gydF4y2Ba
吴建军,李建军,李建军,等。巴西橡胶树DNA甲基化模式与非生物胁迫的关系。植物学报,2011;4:996-1013。gydF4y2Ba
- 51.gydF4y2Ba
梁东,张志,吴辉,黄超,帅鹏,叶春英,等。毛杨DNA甲基化与干旱胁迫的关系生物医学工程学报,2014;15:9。gydF4y2Ba
- 52.gydF4y2Ba
李欣,朱军,胡峰,葛松,叶敏,向辉,等。栽培和野生水稻甲基组的单碱基分辨率图及DNA甲基化在植物基因表达中的调节作用。基因工程学报。2012;13:300。gydF4y2Ba
- 53.gydF4y2Ba
Gutierrez-Arcelus M, Lappalainen T, Montgomery SB, Buil A, Ongen H, Yurovsky A,等。被动和主动DNA甲基化及其与基因调控中遗传变异的相互作用。eLife。2013; 2: e00523。gydF4y2Ba
- 54.gydF4y2Ba
李建平,李建平。将DNA甲基化置于环境中:从基因组到植物的基因表达。生物化学与生物物理学报(英文版);2017;19(6):1409 - 1409。gydF4y2Ba
- 55.gydF4y2Ba
Kakutani T.拟南芥DNA低甲基化突变诱导晚花性状的遗传特征。植物学报。1997;12(1):47 - 51。gydF4y2Ba
- 56.gydF4y2Ba
Sheldon CC, Burn JE, Perez PP, Metzger J, Edwards JA, Peacock WJ,等。FLF MADS Box基因:春化和甲基化调控的拟南芥开花抑制因子。植物学报。1999;11:45 - 58。gydF4y2Ba
- 57.gydF4y2Ba
刘国强,刘国强,刘国强,刘国强。DNA甲基化,春化,和开花的开始。《美国国家科学院学报》上。1993; 90:287 - 91。gydF4y2Ba
- 58.gydF4y2Ba
李建军,李建军,李建军,李建军。DNA甲基化与春化促进开花。《美国国家科学院学报》上。1998; 95:5824-9。gydF4y2Ba
- 59.gydF4y2Ba
Pabón-Mora N, Ambrose BA, Litt A.罂粟花瓣/果同源物控制开花时间、分枝、花被同质性和果实发育。植物生理学报,2012;38(4):591 - 591。gydF4y2Ba
- 60.gydF4y2Ba
徐娥,李辉,全健,朴慧,金健,卢云生,等。开花时间基因SOC1及其上游负调控因子FLC介导拟南芥冷响应与开花之间的串扰植物学报。2009;21(3):385 - 97。gydF4y2Ba
- 61.gydF4y2Ba
郑建辉,周勇,徐鹏军,李建辉,朴昌明。SOC1-SPL模块集成光周期和赤霉素信号来控制拟南芥的开花时间。植物科学学报,2012;39(1):577 - 588。gydF4y2Ba
- 62.gydF4y2Ba
李建辉,朴善,李继生,安建辉。短营养期在芸苔属植物开花时间调控中的保守作用。生物化学与生物物理学报(BBA); 2007; 1769:455-61。gydF4y2Ba
- 63.gydF4y2Ba
布里尔EM,沃森JM。巨桉SVP同源基因异位表达改变拟南芥开花时间。植物学报,2004;31(1):517 - 524。gydF4y2Ba
- 64.gydF4y2Ba
李建辉,刘世杰,朴诗,黄毅,李继生,安建辉。SVP在拟南芥环境温度对开花时间控制中的作用。基因工程学报,2007;21(1):397 - 402。gydF4y2Ba
- 65.gydF4y2Ba
Kazan K, Lyons R.花期与抗逆性的关系。[J] .中国生物医学工程学报,2016;37(2):457 - 457。gydF4y2Ba
- 66.gydF4y2Ba
李建军,李建军,李建军,等。拟南芥中GNC和GNL/CGA1基因的交叉抑制作用。植物生理学报,2013;32(2):391 - 391。gydF4y2Ba
- 67.gydF4y2Ba
Debieu M, Tang C, Stich B, Sikosek T, Effgen S, Josephs E,等。拟南芥种子休眠、生长速率和开花时间随纬度变化的共变PLOS ONE。2013; 8: e61075。gydF4y2Ba
- 68.gydF4y2Ba
李春华,李春华,李春华,等。红树地上生物量和叶面积估算的异速生长关系[j]。日本冲绳岛万谷湿地的黄豆树。树-结构-函数。2005;19:266-72。gydF4y2Ba
- 69.gydF4y2Ba
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学。2014;30:2114-20。gydF4y2Ba
- 70.gydF4y2Ba
李宏,杜宾。基于Burrows-Wheeler变换的快速精确短读比对。生物信息学。2009;25:1754-60。gydF4y2Ba
- 71.gydF4y2Ba
麦凯纳A,汉纳M,班克斯E, Sivachenko A, Cibulskis K, Kernytsky A,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。中国生物医学工程学报,2010;20(2):1297 - 1293。gydF4y2Ba
- 72.gydF4y2Ba
李廷辉,郭宏,王旭,金翀,Paterson AH。SNPhylo:从大量SNP数据构建系统发育树的管道。BMC Genomics. 2014;15:162。gydF4y2Ba
- 73.gydF4y2Ba
Alexander DH, nove J, Lange K.基于快速模型的非亲属个体祖先估计。基因组研究。2009;19:16 555 - 64。gydF4y2Ba
- 74.gydF4y2Ba
李建军,李建军,李建军,等。一种低内存要求的快速拼接对准器。Nat Methods. 2015; 12:357-60。gydF4y2Ba
- 75.gydF4y2Ba
Pertea M, Pertea GM, Antonescu CM, Chang T-C, Mendell JT, Salzberg SL. StringTie可以改进RNA-seq读取的转录组重建。生物工程学报。2015;33:290-5。gydF4y2Ba
- 76.gydF4y2Ba
Love MI, Huber W, Anders S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组生物学。2014;15:550。gydF4y2Ba
- 77.gydF4y2Ba
Bismark:一种灵活的对准器和甲基化调用器,用于亚硫酸氢盐-序列应用。生物信息学。2011;27:1571-2。gydF4y2Ba
- 78.gydF4y2Ba
李建军,李建军,李建军,李建军,李建军,李建军,刘建军,等。基因组DNA甲基化分析的研究进展。中国生物医学工程学报,2012;33(4):391 - 391。gydF4y2Ba
致谢gydF4y2Ba
我们感谢陈新聪、林泽阳、王林在田间取样和表型测量方面的帮助。gydF4y2Ba
资金gydF4y2Ba
国家自然科学基金(No.31671318)、国家重点科技发展计划(No. 2016YFC0502901)、福建省自然科学基金(No. 2015j05074)和中央高校基本科研业务费专项基金(No. 20720150068、No. 20720190106)资助。资助机构没有参与研究的设计和数据的收集、分析和解释,也没有参与撰写论文。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
YJS, YHZ和YZZ构思和设计了这项研究。YJS, YHZ, YZZ和YFZ参与了实验。YZZ分析数据并撰写文章。YFZ、CTY、PPL、YJS、YYZ参与数据分析。YJS和YHZ对文章进行了修改。XBP通过qRT-PCR证实了关键基因的表达水平。所有作者都对草稿做出了重要贡献,并最终批准出版。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们没有相互竞争的利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。创作共用公共领域免责声明(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非在数据的信用额度中另有说明。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
赵毅,钟毅,叶昌。gydF4y2Baet al。gydF4y2Ba多组学分析gydF4y2BaKandelia obovatagydF4y2Ba揭示其对移栽和群体间遗传分化的响应。gydF4y2BaBMC Plant BiolgydF4y2Ba21日,gydF4y2Ba341(2021)。https://doi.org/10.1186/s12870-021-03123-1gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-021-03123-1gydF4y2Ba
关键字gydF4y2Ba
- DNA甲基化gydF4y2Ba
- 遗传分化gydF4y2Ba
- Kandelia obovatagydF4y2Ba
- 纬向梯度gydF4y2Ba
- 种群互移植gydF4y2Ba
- 转录组gydF4y2Ba