摘要
背景
小rna是一种短的非编码rna,是控制真核生物各种生物过程的关键基因调控因子。植物可能调节离散的srna集以应对病原体的攻击。菌核病sclerotiorum一种经济上重要的病原体会影响数百种植物,包括经济上重要的油籽吗显著.然而,关于srna是如何在细胞中发生调控的研究还很有限美国sclerotiorum而且显著pathosystem。
结果
我们发现了不同种类的sRNAs显著在接种后24小时(HPI)对复制的模拟样本和感染样本进行高通量测序。总的来说,3999个sRNA位点高表达,其中730个在感染过程中显著上调。这730个上调的sRNAs靶向64个基因,包括抗病蛋白和转录调控因子。在我们的数据集中,共鉴定出73个保守miRNA家族。降解组测序从联合模拟和感染样本中鉴定出2124个从这些miRNAs中分离的mRNA产物。其中,有50个基因与感染有关。总共有20个保守的mirna被差异表达,8个转录本被差异表达的miR159, miR5139和miR390切割,这表明它们可能在美国sclerotiorum响应。mir1885触发的抗病基因衍生的二级sRNA位点也被鉴定出来,并用降解组测序进行验证。我们还通过5 ' -RACE和RT-qPCR发现了一种新型sRNA沉默植物免疫相关的乙烯反应因子基因的进一步证据。
结论
本研究的发现扩展了理解分子机制的框架美国sclerotiorum而且显著在sRNA水平上的病理系统。
背景
小rna (Small RNAs, sRNAs)是一种短的非编码rna,大小在18到30个核苷酸(nt)之间,对真核生物的基因表达调控和基因组稳定性非常重要[1].sRNA主要有三类:microrna (miRNAs)、短干扰rna (sirna)和p元素诱导弱imp相关rna (PIWI);而后者只发生在动物身上[2],前两者存在于植物中。不同类型的sRNAs具有不同的生物发生途径[3.].
植物中的srna通过RNA干扰(RNA interference, RNAi)途径抑制基因表达。RNA依赖的RNA聚合酶(RdRps)、dicer样蛋白(DCLs)和Argonauts (AGOs)是主要的RNAi途径酶。RdRps促进dsrna的形成,dsrna被dcl加工成srna。一般来说,AGO被认为引导dicer处理sRNAs的一条链(导链)沉默互补靶标[4而另一条链(乘客' sRNA* '链)通常会迅速降解。然而,最近的研究表明,客链在植物中也有重要的基因沉默作用[5,6,7].除了与转录物的互补配对外,sirna还可以通过rna定向的互补DNA甲基化在表观遗传学上调节基因表达[1].
植物有许多种类的siRNA [1,8,9,10].siRNA的主要种类有发夹型siRNA (hp-siRNAs)、自然反义siRNA (natsinas)、次级siRNA和异色siRNA (hetsiRNAs) [1].这些类具有不同的生物发生途径,可能涉及DCL蛋白介导的从非常长的发夹(hp- sirna)或由RdRp酶从单链前体产生的双链rna的双链切割[1].除了沉默信使RNA, mirna介导的信使RNA或非编码RNA前体的切割也可能产生次级siRNAs。这些通常被描述为相控sirna (pha- sirna),因为它们出现在距离miRNA裂解位点精确的21-22个核苷酸间隔处。产生pha- sirna的位点称为“PHAS”位点[11].由PHAS基因座产生的次级rna可能使其衍生的基因沉默,也可能起作用反式抑制其他基因的表达;后一种次生siRNAs被称为反式作用siRNAs (ta-siRNAs),其生物发生位点通常被称为“TAS”基因。
虽然在植物基因组中已经发现了大量mirna引发的次生sirna,但只有少数得到了实验验证[12,13,14,15].在模型植物中鉴定了4个mirna触发的ta-siRNA家族答:芥[13].其中,mirna390触发的TAS3基因被发现在各种植物物种中都是保守的。pha- sirna的形成依赖于几种蛋白质成分,包括抑制基因沉默3 (SGS3)、RDR6和DCL4 [13].对不同植物中PHAS基因座的研究表明,mirna可从多种转录本中触发pha-siRNA的产生,包括非编码rna、抗病和戊三肽重复基因的mrna [16].核苷酸结合位点富含亮氨酸重复序列(NBS-LRR)基因是迄今为止发现的最大的一组基因,可在与特定miRNAs结合时产生pha- sirna [17].
植物中的小rna调节与各种生物过程相关的基因,如种子萌发[18,器官发育[19]和成熟[20.,信号转导[21],以及应激反应[22].受病原菌侵袭的植物可能采用多种srna调控的免疫途径[23,24].例如,在研究小麦品种的sRNAome小麦锈菌Gupta等人(2012)报道miR408在易感和抗性品种中经过2天的感染后表现出不同的表达模式[25].一些与免疫相关的sRNAs也已被功能鉴定。例如,在模型工厂中答:芥, miR393针对不同的生长素信号基因授予抗菌抗性[22miR408是植物色素和漆酶基因的负调控因子[26].后一种基因在应激反应、细胞间信号传递和维持细胞壁的可塑性和活力方面发挥作用。此外,miR7695的过表达导致水稻对稻瘟病菌的抗性增加Magnaporthe oryzae[27].在病毒侵染植物过程中,mirna积累的变化导致不同的pha- sirna的产生[28].在豆类和番茄中,许多miRNA家族通过与NB-LRR基因的转录本结合,参与触发pha- sirna [16,29].在番茄中,在细菌和病毒感染过程中,来自抗病基因的次生sirna的丰度较低,这表明phai - sirna的产生对微调防御反应很重要[29].最近Cui等人(2020)证实了mir1885介导的ta-siRNA表达在维持植物生长和免疫中的作用显著有关病毒感染[30.].pha- sirna在植物对细菌和病毒感染的反应中的作用已经在一些研究中进行了研究,但对它们在对病原真菌的反应中的作用知之甚少。Wu等人(2017)对这一主题进行了为数不多的研究。本研究对番茄产生的pha- sirna的特性进行了研究葡萄孢菌感染(14].研究发现,许多对病害有反应的番茄pha- sirna下调了转录因子,这提示在基因表达调控中具有广泛的作用[15].
油菜(显著)是一种经济上重要的油料作物,在世界各地都有种植[31].菌核茎腐病(SSR),由真菌引起菌核病sclerotiorum是一种重要的疾病,会给油菜籽造成巨大的经济损失[32].已经进行了一些研究芸苔属植物识别植物特异性miRNAs [33,34,35在生物和非生物的压力下。有两个研究显著microrna的表达上美国sclerotiorum感染(36,37].然而,这些研究是在接种后3、12和48 h使用单个sRNA文库进行的,没有任何重复,这限制了对小rna在细胞中差异表达的正确理解显著应对美国sclerotiorum.此外,与其他植物在miRBase中沉积的成熟miRNAs相比m . truncatula,o .漂白亚麻纤维卷而且答:芥,mirna的数量显著的含量很低,说明显著尚未被发现。最后,我们对PHAS基因座的触发机制知之甚少显著基因组及其在应对病原体的基因调控中的功能。
为了评估sRNAs的差异表达,识别新的病原体响应miRNAs,并描述次要sRNAs在肿瘤中的作用显著应对美国sclerotiorum,我们开发了复制的sRNA库模拟接种和美国sclerotiorum接种24 h后的叶片来表征不同种类的sRNAs。为了确定这些sRNAs的靶点,我们还进行了高通量降解组测序,并对一个基因进行了5'RACE和RT-qPCR。
我们第一次发现了大量的显著sRNAs对美国sclerotiorum24 HPI后感染。我们还发现了新的PHAS基因座的病原体响应激活的证据,可能涉及到抗病蛋白的调节。我们后续的5 ' -RACE和qPCR研究提供了进一步的证据,证明srna对乙烯信号通路相关基因的调控作用。
结果
测序结果概述
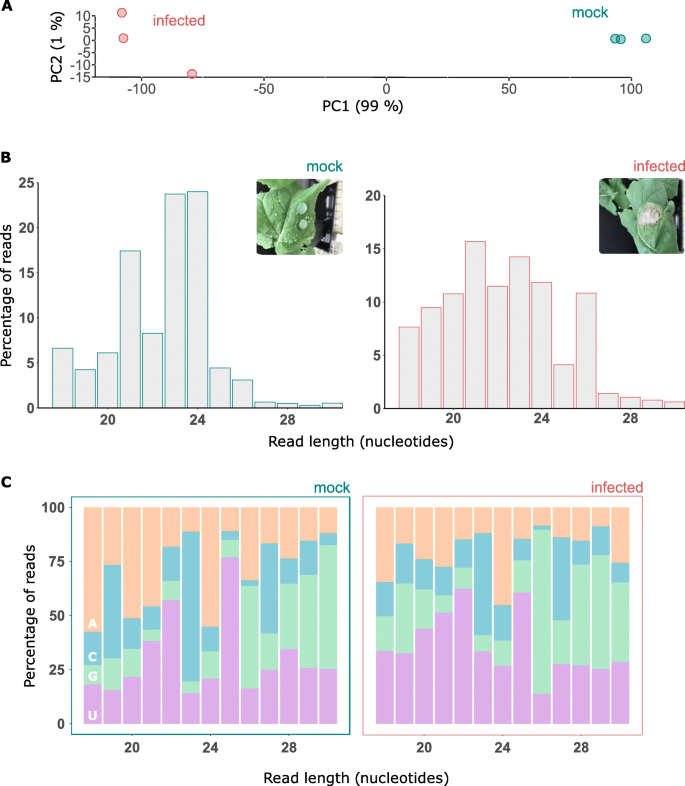
确定…的作用显著srna期间美国sclerotiorum当SSR症状在叶片上出现时,我们从模拟和感染样本的3个重复中分别在24 HPI对Illumina平台上的6个sRNA文库进行了测序。从六个图书馆共获得152,090,773份原始资料。经过适配器调整和长度过滤(18-30 nt)后,我们从这六个库中保留了126,887,984个高质量读取(83.24%)1).分配和删除模棱两可的reads(植物和真菌基因组的映射)导致41,797,278个唯一的显著sRNA读取最匹配的显著所有文库的基因组。可能来自结构rna (rrna, snrna, snorna)的读取占总数的5%左右。然后将干净的、高质量的可映射读取对齐到显著基因组。总比对率为88.7%,其中模拟样本的比对率最高(超过98%),而感染样本的比对率平均为78.9%,重复之间的比对率从77到86.3%不等。在被映射的reads中,约86%被映射到一个以上的基因组位点,这表明这些sRNAs起源于基因组重复。从我们的数据集中,我们发现14%的sRNA是唯一映射到单个基因组位点的。表格1提供测序数据的总体摘要。
为了确定受感染库和模拟库的分组,我们对DESeq2的平均归一化计数进行了主成分分析(图1)。1A).主成分分析显示复制数据集对两个治疗组(即模拟和感染)进行了很好的分组,表明这些治疗之间存在很大的总体差异。模拟样本和感染样本沿主成分1分离,解释了99%的方差。在主成分2沿线感染样品之间有一定的扩散。然而,沿着这条轴,这些样本之间的方差可以忽略不计,因为只有1%的方差可以用PC2解释。
大小类和5 '核苷酸的变化显著响应的sRNAs菌核病sclerotiorum感染。一个基于DESeq2的归一化读计数的主成分分析。x轴表示主成分1,解释了99%的方差,y轴表示主成分2,解释了1%的方差。感染样本用红圈表示,模拟样本用绿松石表示。B从池模拟和感染样本中读取大小的直方图。插图:有代表性的图片显著两种处理(接种后24 h (HPI))的叶片。左:对于模拟样本的池复制,y轴表示所有三个重复的读取百分比,x轴表示核苷酸的读取长度。右:对于受感染样本的池复制,信息与模拟样本相同。C来自模拟(左)和感染(右)的池复制的结果。显示每个大小类(x轴)在5 '位置上有四个核苷酸(AGCU)的读的百分比(y轴)
的特点显著小RNA人口
为了确定哪些sRNAs在感染反应中被诱导,我们分别从模拟和感染样本中进行了三个测序重复。以下指标基于每种治疗、模拟和感染的生物复制池。大小类分布和5 '核苷酸偏倚是决定sRNAs起源和活性的两个重要特征。为了确定模拟和感染样本中sRNA起源的组成是否可能存在差异,我们分析了这些sRNA的核苷酸长度和5 '核苷酸偏性。有趣的是,我们发现模拟样本和感染样本之间的长度分布存在差异(图1)。1B),表明在感染时,sRNA的生物发生机制发生了改变。在模拟样本中,近50%的总reads属于24和23个核苷酸(nt) sRNAs,其次是21个nt。腺嘌呤作为5 '核苷酸在24个nt sRNAs中富集,胞嘧啶在23个nt sRNAs中富集。在22个nt sRNAs中,5 '核苷酸偏向于尿嘧啶。1C)。
在感染样本中,大小分类比模拟样本更加一致(图。1B)。最丰富的reads size是21nt,有轻微的5 '尿嘧啶偏倚,其次是23nt,有轻微的胞嘧啶偏倚(图。1C)。我们还发现感染样本中鸟嘌呤偏向5 '的峰值为26 nt。同样,18、19、20和22 nt大小类在感染样本中也更为丰富。然而,在两个样本的非冗余读取中,大多数为24 nt。除了大小分布之外,总读与唯一读的比率也是sRNA库的一个重要特征[38].与24-nt序列相比,21 -nt序列的复杂性较低,说明21 -nt的少数唯一reads高表达,而不同的24-nt序列较多。这些特征被归因于24 nt异染色质sRNAs [39].模拟和感染样本中非冗余读取的小RNA大小分布显示在补充图中。1.
以前的报道也显示了类似的数据,21个nt的尿嘧啶偏倚和24个nt的腺嘌呤偏倚。24nt 5 '腺嘌呤偏倚的sirna先前已被证实参与了腺嘌呤的RNA依赖性DNA甲基化答:芥优先加载到AGO4,而21 nt 5 '尿嘧啶偏向的srna优先加载到AGO1 [40].总的来说,我们的结果表明srna的表达类型从模拟到感染有显著的变化显著叶子。
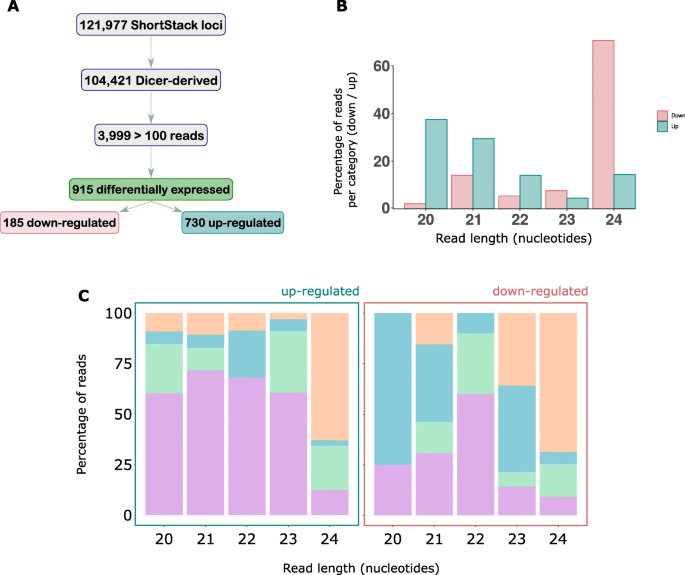
共有730个独特的显著小rna会对菌核病sclerotiorum感染
评估什么显著sRNAs在感染的反应中积累美国sclerotiorum,我们用DESeq2进行了差异表达分析。我们不仅考虑了mirna的差异表达,而且考虑了整个sRNA-ome显著.ShortStack预测了121977个sRNA位点,其中104,421个可能是dicer派生的。在这些基因座中,有3999个高表达,至少有100个原始的主要RNA测序reads(补充表1).如果这些sRNAs对感染有反应,我们假设与模拟样本相比,它们可能在感染样本中表达更多。我们发现915个基因座在感染样本中的表达与模拟样本相比发生了显著变化。其中730例上调B.后显著美国sclerotiorum感染;根据ShortStack预测的主要sRNAs,这些位点产生了565个独特的sRNAs(图1)。2一个)。
这730个上调的sRNAs大多在20和21个nt序列中被富集,而185个下调的主sRNAs在24个nt序列中被富集(图。2B).除了24 nt外,上调的srna的所有大小级别的5 '端尿嘧啶都被富集,24 nt具有5 '腺嘌呤偏性(图)。2C)。然而,下调的srna在20 nt和21 nt表现出5 '胞嘧啶偏性(图。2C). 22号和24号尺码在两组样本中尿嘧啶和腺嘌呤均有5 '偏倚。总之,我们的数据增加了一个假设的权重,即具有不同生物发生和靶向途径的sRNA类在应答中表达美国sclerotiorum挑战。
应激反应基因被上调的小rna靶向
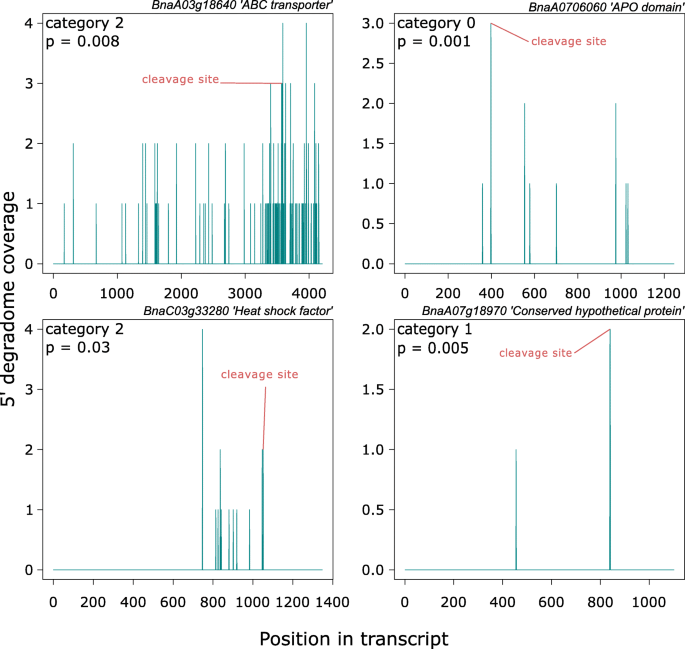
我们使用降解组测序数据来研究感染过程中上调的sRNAs的靶点。通过这种方法,从上调的sRNAs中共鉴定出64个靶基因(表2).图中显示了在感染文库中确定的4个基因的代表性t图,这些基因被分配到不同的PARESnip2置信类别。3..在这64个目标中,在两个库中都找到了10个目标,从而分别为模拟样本和受感染样本提供了29个和15个独特的目标。在感染过程中可能被上调的小rna调控的基因被转录因子相关的InterPro术语注释,如“ABC转录因子”、“热休克反应”、“抗病蛋白样”、“锌指l域”和“亮氨酸拉链域”。这表明,显著转录调控网络可能被感染过程中特异性诱导的srna所修饰。
基因中保守miRNAs的鉴定显著
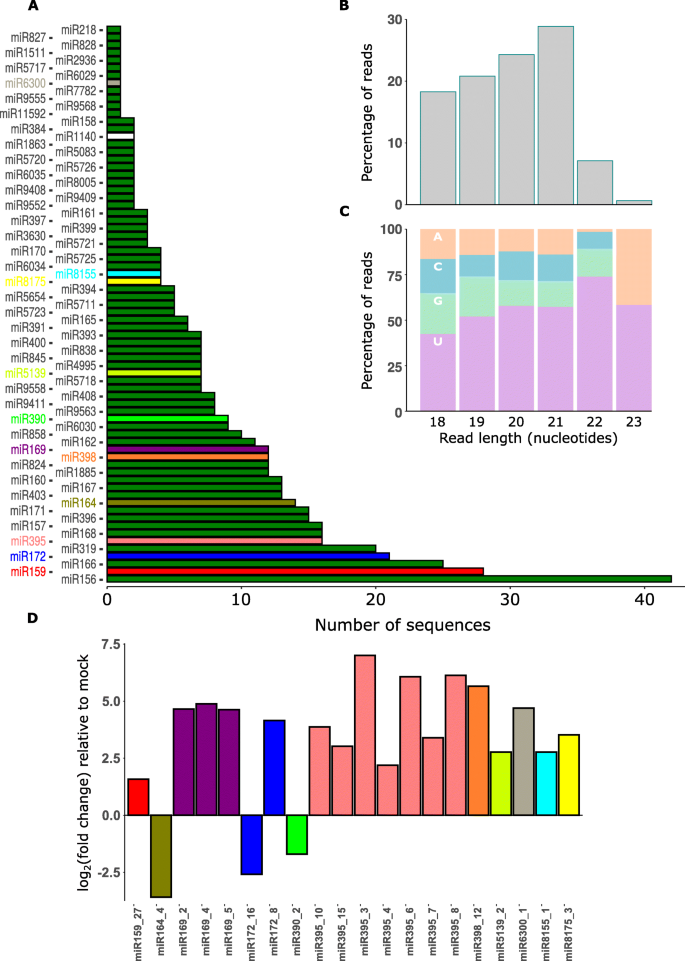
在植物王国中,有几种miRNAs在进化上是保守的[41].我们评估是否保守显著miRNAs在美国sclerotiorum感染。因此,在miRBase(版本22.1)中搜索了所有6个干净的库。从我们的文库中,我们鉴定了73个保守miRNA家族和529个成熟miRNA序列。我们发现61个miRNA家族有一个以上的序列,而12个miRNA家族只有一个成熟序列(图。4A).在这些miRNA家族中,miR156有42个同源序列,其次是miR159, miR166分别有28个和25个同源序列。这些miRNA序列大部分为21 nt长,其次为20、19和18 nt长。4B).在18-22 nt长的miRNA序列中存在5 '尿嘧啶偏倚,这与之前报道的不同植物miRNA研究结果一致(图1)。4C).使用降解组测序,我们发现在73个保守miRNA家族中,有718和1406个裂解产物(补充表2)分别从感染库和模拟库中获取。基于read mapping特征,降解组裂解位点置信度的四个级别描述在[42,43].第0类是最自信的,其次是第1、2和3类。根据文库中片段转录本的丰富程度,在感染样本的718个裂解事件中,0类裂解事件为507个,其次为2、3和1类裂解事件,分别为90个、87个和34个。因此,大多数通过降解组测序鉴定的保守miRNA靶点具有较高的置信度[44].多个靶基因可能被多个miRNA家族沉默。在这些靶基因中,在感染样本中发现了158个非冗余转录本。在受感染样本的158个靶点中,miR160、miR164、miR167和miR396预测每个靶点都超过10个基因。同样,miR156, miR6030, miR400, miR393, miR172和miR171被预测靶向8个基因(补充表3.).我们发现了另外43个保守miRNA家族显著miRbase中记录的miRNAs。这些miRNAs在生物和非生物胁迫下调控植物基因表达的研究中已有报道[45,46,47,48].
预测感染反应的microrna显著基因组。一个73个保守miRNA家族的直方图。y轴表示已识别的保守miRNA家族名称,x轴表示为每个miRNA家族识别的序列(异构体)数量;红色条是表达差异显著的miRNA家族。同分异构体是一类高度相似的miRNA序列家族中的一种,来源于导链或客链。绿色miRNA家族无差异表达。其他颜色的含有差异表达的mirna,其颜色与之对应D.B529个保守mirna的读取大小的直方图。y轴表示核苷酸中读取的百分比,x轴表示核苷酸中读取的长度。C529个mirna的直方图,显示了每个大小类(x轴)的每个核苷酸(AGCU)在5 '位置上的reads的百分比(y轴)。D20个差异表达的miRNAs2(折量变化)在y轴上。颜色与miRNA家族相对应一个
病原体应答mirna在受感染的样品中分裂植物免疫应答基因
为了确定转录物降解是否针对受感染的样本,我们从受感染的样本中过滤出所有在模拟样本中也被靶向的基因,得到50个靶点(补充表4).在这些基因中总共发现了172个Interpro结构域。这些基因具有转录调节、抗病和转录后基因沉默等功能。我们还从这组中发现了一些miRNAs,它们在植物和病原体的相互作用中也有作用。
9个miRNA家族中的17个miRNA在感染过程中显著上调
我们使用miRprof分析的原始读取计数对单个保守miRNAs进行差异表达分析。在保守的miRNAs中,只有20个miRNAs差异表达(图。4D;补充表5).其中,9个miRNA家族的17个miRNA在感染过程中上调,3个miRNA下调。在上调的9个miRNA家族中,miR395有7个异构体,miR169有3个异构体,miR159、miR172、miR398、miR6300、miR8155、miR8175和miR5139各有1个异构体。上调的miRNA序列表现为对数2(倍数变化)值介于1.58到6.13之间。17个mirna中有16个有日志2(翻倍变化)大于2。
这三个下调的miRNA属于miRNA家族miR164、miR72和miR390。这些miRNAs显示日志2(倍数变化)在−1.7到−3.59之间的感染值;两个展示日志2(倍数变化)值低于−2。有趣的是,miR172有两个表达模式不同的异构体,在感染过程中一个表达上调,另一个表达下调。
在17个上调的miRNAs中,我们发现5个基因可能被miR159家族成员miR159_27 (log2(变化倍数)= 1.56;p校正= 0.034),miR5139家族成员miR5139_2 (log2(倍数变化)= 2.77;p校正= 0.0042),在感染样品中。令人惊讶的是,我们发现3个转录本被miR390下调成员miRNA390_2 (log2(fold change) =−2.94;p校正= 0.04),在感染样品中,而在模拟样品中没有这些基因的切割。
RNA结构辅助预测算法识别出135部小说显著微RNA基因座
在过滤掉保守miRNA与miRBase的精确匹配后,从miRBase中识别出135个新的miRNA产生位点显著基因组。在这些miRNA中,有67个位点同时具有乘客链(miRNA*或“星型”)和成熟链reads,根据注释标准揭示了这些新miRNA的可信度。新miRNA基因座的详细描述见补充表6.
在我们的研究中,我们没有发现miRDeep2预测的新miRNAs的任何裂解信号。我们使用相同的mirna集合,通过psRNA目标服务器来预测目标。从psRNA靶中,推测有12,104个基因被这些miRNAs靶向。一些psRNA靶点可能是假阳性,因为它完全基于硅的理论过程,而降解体信号更好地反映了生物裂解。这些新的miRNAs是否具有真正的靶标还有待证实。
九个显著PHAS基因座对菌核病sclerotiorum感染
PHAS基因座尚未被很好地描述显著.因此,我们旨在识别表达的PHAS基因座显著基因组来自我们的测序数据集。我们发现26个PHAS基因座显著基因组。通过将PHAS位点序列与NCBI核苷酸集合(nr/nt)进行比对,对预测的PHAS位点相关基因进行注释。在26个PHAS基因中,约一半与抗病蛋白(5个)、非编码rna(5个)和叶绿体相关(3个)。此外,还发现了金属耐受、五肽重复、cop9信号体复合体亚基和光系统II蛋白D1的单基因。9个PHAS基因与NCBI中的任何序列均无同源性。从这些位点共产生了182个pha- sirna。这些sirna中有41个高表达,reads丰度超过100个。
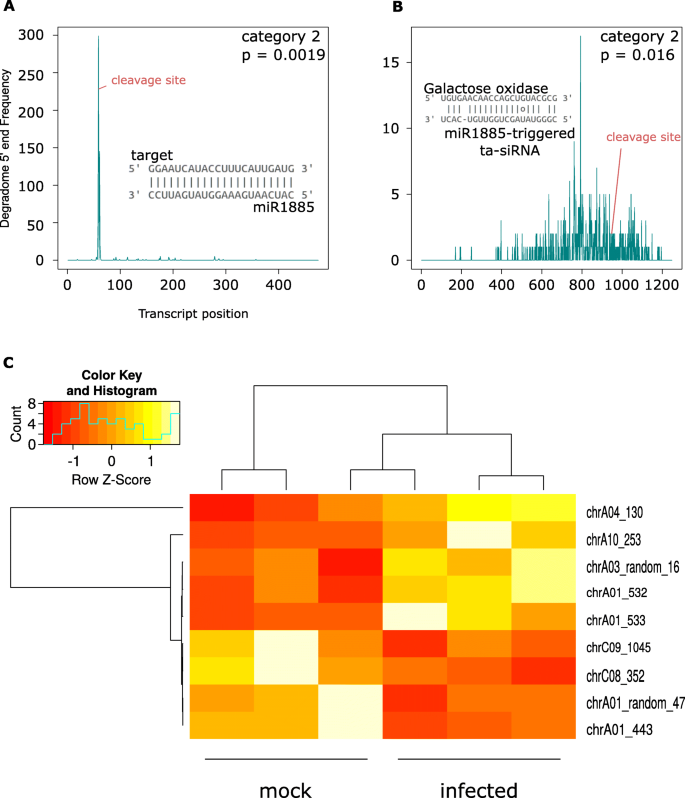
由于mirna是pha-siRNA表达的关键触发器,我们使用psRNA目标服务器从我们识别的保守mirna中找到PHAS基因座中的切割位点。我们发现了6个可能由保守miRNAs触发的PHAS基因座(表3.).所有被切除的PHAS集群及其对应的pha- sinas显示在补充文件中1.mir390触发的PHAS基因TAS3被发现在不同物种中都是保守的。在本研究中,我们发现了两个miR390可能靶向的基因,其中一个与miR390有序列相似性TAS3在答:芥.
我们能够通过降解组测序(第2类,P= 0.0019;无花果。5A).我们没有发现psRNA目标服务器预测的另外5个mirna触发位点的降解信号。我们能够验证的位点很可能是由保守的miRNA miR1885靶向的。最近,实验表明miR1885在靶向位于NBS-LRR基因内的PHAS位点触发ta-siRNA生成方面发挥了关键作用[30.].据此我们发现该位点与NBS-LRR蛋白具有同源性。我们从这个PHAS位点上鉴定出10个可能的ta- sirna。从降解组信号中,只有一个靶标BnaC05g49720D,一种半乳糖氧化酶,β -螺旋桨,从降解组测序中有裂解信号(2类,P= 0.016;无花果。5B)在受感染的样本中。可能的话,显著miRNAs调节基因表达响应美国sclerotiorum通过miRNA的产生引发ta-siRNAs的感染。
miRNA 1885触发TAS基因的降解组验证。一个miRNA 1885确定的TAS基因裂解位点的靶区(t图)。靶基因的转录本位置在x轴上,y轴表示每个位置上的5 ' read coverage;PARESnip2预测的裂解位点标记为红色。的类别和p图的左上角给出了PARESnip2测试的值。B半乳糖氧化酶基因上裂解位点的t图,推测是由miRNA 1885触发的TAS基因产生的一种ta- sirna所靶向。x轴表示目标基因中的转录本位置,y轴表示每个位置上的read覆盖范围为5 ';PARESnip2预测的裂解位点标记为红色。的类别和p在图的右上角给出了PARESnip2的值。C9个差异表达的PHAS位点的热图,用DESEq2的归一化计数绘制
PHAS基因座差异表达分析显示,在感染过程中,有5个基因座上调,4个基因座下调。在上调的基因座中,3个与抗病相关的基因座变化为log2倍,变化范围为0.78 ~ 1.87。其余2个基因分别为叶绿体和光系统II蛋白D1,变异量分别为0.57和1.73的对数倍。在下调的基因座中,两个基因座为非编码rna,变化为−0.7和−0.96的log2倍,一个基因座与COP9信号体复亚基相关(变化为−0.82的log2倍),其余一个基因座未被表征。数字5C为9个差异表达的PHAS位点的热图。
为了获得pha- sinas靶向基因的全局概述,我们使用psRNA靶服务器找到41个高表达pha- sinas的靶。我们发现了5918个可能受这类sRNA调控的转录本。我们对这些靶标进行了GO术语富集分析,发现了几个生物过程的调控(补充表)7).“转录后基因沉默”(GO:0035194)、“细胞钾离子平衡”(GO:0030007)、“ARF蛋白信号转导调控”(GO:0032012)和“苏酰- trna氨基酰化”(GO:0006435)、“氧化-还原过程”(GO:0055114)、“转录dna模板化调控”(GO:0006355)、“碳水化合物代谢过程”(GO:0005975)”等术语显著丰富。
我们还专门研究了mir1885触发的ta-siRNAs的靶标。我们发现了1601个带有psRNA靶标的srna靶标。GO术语富集分析显示,这些ta- sirna可能调节蛋白质磷酸化(GO: 0006468)、转录因子(GO: 0045944)、囊泡介导的转运蛋白(GO:0016192)和聚焦代谢途径基因(GO:0006004)(补充表)8).
利用cDNA末端5 '快速扩增和定量PCR进一步分析sRNA靶向
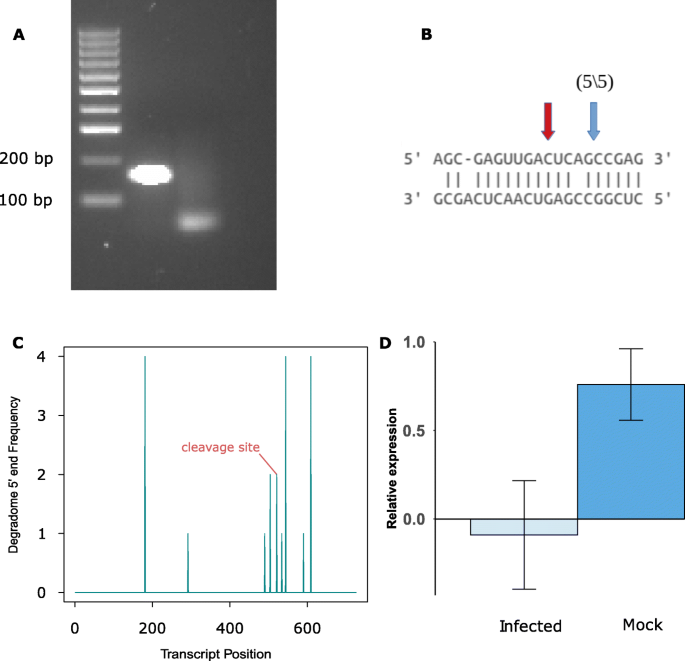
使用5 ' RACE在新的sRNA靶基因中寻找假定的裂解位点BnaA01g27570D,这是一个乙烯响应因子。乙烯信号传导在植物对病原体的免疫中有很好的作用[42],虽然乙烯反应因子在对美国sclerotiorum[49],对于它们可能是如何被srna调控的还知之甚少。从A01染色体上表达的新siRNA长22 nt,既不保守也不特征性,既不是pha-siRNA,也不是miRNA。数字6A表示从感染样品中预测的裂解位点的5 ' -RACE产物;完整的凝胶图像在补充图中。2.解理信号的t图如图所示。6C.我们还用RT-qPCR检测了该靶基因在感染过程中的表达。平均对数(2——∆Ct)的值分别为0.76和- 0.09,标准差分别为0.35和0.53。6C.结合降解组、5'RACE和qPCR结果显示一种新的显著sRNA可能调节乙烯反应因子基因美国sclerotiorum感染,导致其表达减少。综合降解组、5'RACE和RT-qPCR结果显示,一种新型siRNA调控了植物免疫应答基因。
讨论
小rna组学研究揭示了植物中宿主免疫通路的严密调控[23,50,51].在我们的研究中,我们发现了不同种类的sRNAs显著对感染有反应的植物美国sclerotiorum并展示了它们如何通过降解组测序参与调节不同的基因集。
我们发现srna介导的乙烯反应因子基因调控(BnaA01g27570D),我们进一步使用RT-qPCR和5 ' -RACE进行研究。通过降解组测序和5 ' -RACE确定的可能的切割位点,以及该基因在感染期间的表达减少,表明它在植物对病原体攻击的反应中有作用。乙烯产量的增加是植物对病原体攻击感知的早期反应,导致防御系统的诱导[52].在显著据预测,乙烯响应元件结合因子可控制与防御信号、次生代谢物产生和氧化还原调节相关的生物过程。的最接近的同系词BnaA01g27570D在答:芥RAP2.3已被证明与另一个乙烯反应因子ORA59在细胞核中共同定位,以介导对细菌病原体的防御p . carotovorum[53].由于RAP2.3在抗病方面具有已知的积极作用,而乙烯通常被发现对防御有积极贡献,我们的研究显示RAP2.3的表达减少,这是有趣的BnaA01g27570D是由感染过程中潜在的srna介导的调控引起的。可以提出几种假设来解释这一现象。例如,病原体可以引起植物的反应,抑制乙烯反应免疫。或者,乙烯反应系统的某些成分可以负向调节免疫,如其他一些系统所示[42].需要进一步的研究来确定这种特定乙烯反应因子的sRNA裂解的生物学意义美国sclerotiorum感染显著.
总sRNAs的大小类分布在模拟样本和感染样本之间不同。srna的大小分类可以帮助我们了解它们的生物起源,例如,21个nt sRNAs是由DCL1和DCL4处理的,而22个nt sRNAs是通过DCL2的作用形成的;24和26个nt sRNAs由DCL3形成[54].类似地,含有5 '尿嘧啶、腺嘌呤或胞嘧啶的sRNAs分别装入AGO1、AGO2和AGO4或AGO5 [55].此外,此前有报道称,sRNA长度的变化也会影响sRNA的下游功能[56].在模拟样本中,冗余和非冗余分布均在24 nt达到峰值。然而,在感染样本中,冗余分布在21 nt有一个主峰,非冗余分布在24 nt有一个主峰。总的来说,我们的数据表明,一组不同的sRNA生物发生途径在感染美国sclerotiorum.
共有730个sRNA位点被上调显著为了应对美国sclerotiorum感染。上调的srna大多属于20和21 nt的大小类,尿嘧啶偏倚为5 ',而下调的srna在24个nt的偏倚为5 '的序列中过度表达。这表明,在病原体感染后,宿主会招募不同的DCL和AGO蛋白用于后续的基因沉默。从降解组数据中可以确定,上调的sRNAs可能使许多与应激信号相关的基因沉默。这提示疾病症状的增强可能伴随着植物免疫基因的负调控美国sclerotiorum感染显著.
miRNAs是由短发夹前体产生的sRNAs。基于这些标准,已经从许多植物中鉴定出数千个miRNAs并将其沉积在miRBase中。这些miRNAs大多具有高度的保守性。然而,只有92人成熟显著到目前为止已经发现了mirna。它们的数量比答:芥(428),m . truncatula(756)和o .漂白亚麻纤维卷(738)揭示了一些miRNAs尚未在该物种中被发现。
之前的两项研究进行了评估美国sclerotiorum响应显著HPI时间点3,12和48的mirna [36,37].共有227个[36]和77 [37]保守的miRNA序列在这些研究中被鉴定出来。这两项研究都是在每个样本中使用单个sRNA测序库进行的。微阵列miRNA表达分析检测出68种感染应答miRNA, [36],其中10个用茎环qPCR方法进行进一步分析。同样,Jian等人也发现了10种miRNAs的差异表达。[37],而相对于Cao等人的研究,只有一个miR166被发现共同差异表达[36].在这里,根据复制差异表达分析,我们发现了529个成熟的保守miRNA,属于73个miRNA家族,以及20个感染诱导的miRNA。在我们的研究中miRNAs数量的增加可能是由于数据集的重复和测序深度的增加。与之前的两项研究相比,我们没有发现任何共同差异表达的miRNAs [36,37].这可能是由于不同的时间点,显著品种,组织收集和美国sclerotiorum压力我们使用.然而,降解组数据表明,这些miRNAs调节与发育和防御反应相关的转录因子的表达,这证实了之前的发现。
此外,发现了135个新的miRNA基因座,其中67个基因座既有成熟的,也有明星(乘客链)读数。虽然这增加了显著发现了mirna,我们无法识别这些mirna的任何可能靶标。这表明它们要么没有靶点,要么通过一种非切割机制(如翻译抑制)调节基因[43]..
大量转录因子和生长素信号通路基因的表达可能受到这些基于受感染样品降解组数据识别的保守miRNAs的调控。其中一些miRNAs在特定的类中有多个靶标。我们从感染样本中发现了50个模拟样本中没有的独特的裂解产物。这些基因与转录因子、抗病蛋白和转录后沉默有关。例如miR824、miR390/miR5083、miR403/miR838、miR5139、miR1885分别裂解了含有富亮氨酸重复序列、锌指转录因子、蛋白激酶和抗病蛋白编码基因的转录本。同样,miR166和miR858分别分裂了同源结构域和sant/myb结构域基因的转录本。MiR824先前在十字花科植物中被证实在热应激反应中发挥作用[57].值得一提的是,miR858已被证明负向调控MYB转录因子,从而控制对病原菌感染的抗性答:芥[58].同样,本研究中可能受到miR166调控的植物同源结构域蛋白控制着病原防御相关基因的转录调控[59]。这些发现表明,miRNAs参与调节免疫反应的多个方面显著来美国sclerotiorum.
在我们的研究中,我们发现了20种感染诱导的mirna。这些miRNAs先前被证明在不同病理系统的应激反应中发挥作用[50,60,61,62].然而,我们只发现了五个基因转录本的降解证据,BnaA03g22590D,BnaA05g27620D,BnaCnng31260D,BnaCnng49390D,BnaA02g31560DmiR159_27和miR5139_2是被感染样品中上调的miRNAs所靶向。这5个基因中的4个可能被miR159_27切割,其余的基因可能被miR5139_2切割。这些基因包含SANT/myb结构域、同源结构域和锌指结构域等Interpro结构域。miR390_2的表达为~ 69% (log2(0.31) = - 1.7)的低基因中有三个靶基因,它们的mrna只在感染样本中被裂解(BnaA06g09370D,BnaC05g49670D,BnaC08g16190D).这表明这些基因在模拟样本中没有表达。
另一方面,我们在miR395上调的不同异构体的模拟样本中发现了11个转录本被裂解的基因,而在感染样本中没有发现这些裂解的基因。这些数据表明,sRNAs对基因的调控是相当复杂的,而分裂事件并不总是依赖于表达池,其他变量也可能起作用。
MiR390已被证明在ta- sirna的形成和调节生长素反应因子基因中发挥作用[63而miR395在硫同化中发挥重要作用[64].MiR159存在于大多数陆地植物中,它调节编码R2R3 MYB结构域转录因子的基因,该基因可转导gibberlin (GA)信号[65在生长、发育和生物应激反应中发挥作用。最近表明,棉花和拟南芥植物积累了更多的miR159水平,以应对真菌黄萎病dahliae[66].miR159也被报道在拟南芥根结线虫对根结线虫的侵袭,缺乏miR159-Gibberlic acid MYB通路的品系对根结线虫的抗性较好。此外,miR159-GA MYB通路已在答:芥促进细胞程序性死亡反应[67].我们发现miR159裂解的4个MYB结构域基因表明miR159在显著反应美国sclerotiorum.然而,还需要进一步的研究来了解这一途径的确切作用。
在之前的研究中,miR5139在花瓣生长过程中被乙烯调节,并首次在多年生草本植物中检测到[68]但没有分配给这个miRNA特定的功能。后来发现它在小麦中也有组织特异性表达[69].然而,miR5139的同源性基因未见报道显著到目前为止。在这里,我们发现miR5139的表达水平接近7倍(log2(6.96) = 2.8),可使该基因的转录本发生裂解BnaA02g31560D编码锌指结构域的基因。据报道,锌指结构域存在于植物抗性相关蛋白中,这些蛋白参与效应器触发的免疫反应[70].我们的数据显示在美国sclerotiorum攻击,显著部署miR5139负调控锌指结构域基因作为防御反应策略[70].
尽管我们没有发现其他差异表达的mirna的任何降解体切割信号,但在这里发现了一些有趣的mirna,它们在植物对抗病原体的反应中发挥作用。miR164被证明操纵细胞程序性死亡答:thalina。miR164靶基因过表达增强疾病症状[71].miR169在稻瘟病菌侵染过程中负调控水稻免疫基因[50].miR6300和miR8175在链格孢属与原木处理的番茄植株相比2(倍数变化)为3.13或2.65。在这里,我们发现这两种miRNAs在之后分别增加了3.5和4.7倍美国sclerotiorum感染(61].
在降解组文库的帮助下,我们发现miR1885可以触发抗病助教该基因随后产生10个用于基因调控的ta- sirna。这种特殊的miRNA最近被证明可以直接抑制显著TIR-NBS-LRR耐药基因BraTNL1和TAS基因BraTIR1,其裂解产生调控光合作用相关基因的sRNAsBraCP24[30.].通过调节免疫和基础生长,该miRNA可能对优化发育过程中的资源配置至关重要。该miRNA已被证明在大多数条件下表达丰度较低,除了花期,当植物需要协同降低植物合成水平和病原体反应。在我们的研究中,我们没有观察到miR1885表达的变化美国sclerotiorum虽然我们观察到半乳糖氧化酶、β螺旋桨蛋白编码转录本(BnaC05g49720D)由它可能激活的TIR-NBS-LRR TAS基因衍生的一个小rna引起。然而,这些蛋白质的作用相当不同[72,73],因此不可能就这一观察结果的生物学意义得出任何结论。识别和阐明pha-siRNAs的调控网络非常重要,这样就可以通过改变其miRNA触发器的表达来控制这些pha-siRNAs的表达。该策略可用于调节内源和外源靶基因的沉默程度。
结论
总之,我们的综合数据集使我们能够研究宿主转录本的整体病原体响应RNA干扰调控,以及特定小RNA类对病原体攻击的反应。我们的数据表明,包括miRNAs在内的感染诱导sRNAs的靶点可能与应激反应基因相关。显著当受到坏死性病原体的挑战时,植物可能同时表达pha- sirna和保守的miRNAs。综合分析表明,a显著sRNA在病原体攻击过程中调节乙烯反应因子基因。乙烯反应因子调节几种茉莉酸(JA)和(ET)途径,是植物先天免疫的关键因子[51].我们的降解组、5 ' RACE和qPCR结果表明,乙烯反应基因之一的表达被抑制显著后美国sclerotiorum感染。该基因的沉默是由一种新的sRNA介导的,这是以前没有发现的。
方法
生物材料
一个澳大利亚年代.sclerotiorum分离物(CU8.24)最初从南斯特灵澳大利亚州采集,用于感染测定[74].将成熟菌核切成两半,置于含有马铃薯葡萄糖琼脂(PDA)的9厘米培养皿中。菌丝从菌核中萌发后,继代培养到新鲜的PDA培养基上。培养48 h后,将菌丝栓置于一个月大的完全展开的第二或第三叶上显著植物(AV石榴石)。av -石榴石种子最初从澳大利亚谷物基因库(accession AGG95718BRAS1)获得。植株在光照16小时、光照8小时的生长室内生长一个月。感染后,用塑料袋小心地覆盖植物,以增加湿度,从而促进感染过程。24株HPI,在感染叶上观察到特征性坏死病变。用消毒剪刀小心切除感染组织,立即冷冻在液氮中,保存在−80°C,直到RNA提取测序。对于模拟样品,PDA仅使用琼脂插头,不使用任何真菌菌丝体。来自三种不同植物的三片叶子被放在一起进行每次复制。对于小RNA测序,我们分别对3个生物重复进行测序,同时对2个降解组文库进行测序,将所有感染的重复作为一个文库,所有模拟的重复作为另一个文库。
总RNA提取和测序
使用TRIZOL试剂按照制造商的协议(Invitrogen Carlsbad, CA, USA)提取总rna。提取后,使用纳米滴分光光度计和量子比特定量总rna。用琼脂糖凝胶电泳检测RNA样品的完整性。将总RNA的3 ~ 5 μg和25 ~ 30 μg分别送到Novogene(新加坡)进行小RNA测序和降解组测序。根据制造商的协议,使用Illumina®NEBNext®Multiplex小RNA文库准备试剂盒进行sRNA测序,单端读取50 bp。降解组测序如[75].总之,从降解mRNA的降解位点(含单磷酸基)开始构建降解组文库。测序适配器分别添加到降解文库的两端,文库大小选择在200-400 bp左右。测序在Hiseq 2500 SE50上进行。
小RNA测序数据分析
使用cutadapt软件(版本1.15)对原始读取进行剪裁,该软件针对单端读取进行优化,设置为cutadapt -a(通用True Seq适配器)-m18 -M30 [76].通过运行FastQC检查过滤读取的质量[77].长度在18-30 nt范围内的读数被保留。修剪过的感染reads分配给真菌[78和植物[79]在bbmap中使用bbsplit软件引用序列[80程序将歧2选项设置为toss。选项歧2删除映射到两个引用的所有读取操作,具有相同的置信度。这些解读是独一无二的显著保存基因组进行预测显著srna。
的预测显著sRNA生物发生位点,干净的reads与参考基因组对齐显著允许最多两次不匹配。我们使用了ShortStack [81,以获得sRNA产生基因座的总体概念显著并对感染后的高表达sRNA位点进行表征。每个库都是作为一个单独的实体使用的,不会塌陷以输入到ShortStack中。对于保守的miRNA预测,我们将清洁读取与miRBase(版本22)进行匹配(http://www.mirbase.org)使用miRProf程序在UEA小RNA工作台[82].miRbase数据库中与成熟miRNAs匹配且不匹配为0的reads被认为是保守miRNAs。其余与miRBase不匹配的reads被分析,使用miRDeep2程序预测新的mirna产生位点[83].使用R中的Bioconductor包DESeq2进行差异表达分析,估计方差-均值依赖关系。我们使用ShortStack中6个独立库的原始簇丰度来寻找差异表达的sRNA位点。带有Benjamini-Hochberg修正的sRNAsp-值< 0.05被认为是差异表达sRNAs [84].对于保守miRNA的差异表达分析,我们使用了从miRprof程序中识别的单个miRNA序列的原始计数。
利用PHAS坦克软件(1.0版本)的Perl脚本预测位点的相位模式[85].为了找到miRNA触发的阶段性启动位点,使用psRNA目标服务器搜索PHAS位点上预测miRNA的互补切割位点,假设miRNA上的第10个核苷酸位置是其目标PHAS位点的切割起始位置[15].
sRNA靶向在硅靶预测和降解组测序数据中的应用分析
为了确定reads是来自植物还是来自真菌,我们使用了bbsplit [80将受感染的降解组reads分类为真菌或植物特异性reads。通过过滤RFAM数据库,将过滤后的reads从潜在的结构rna中分离出来[86]使用程序Infernal(1.1.3版本)[87].
我们使用psRNA目标服务器(默认设置和期望得分为5)对sRNA目标进行计算预测[88或PARESnip2 [89]根据Fahlgren和Carrington的规则验证降解组数据中的裂解位点[90].我们保留了0-3类目标。0类是降解产物具有多个降解峰的靶标,而转录本上只有一个最大值。第1类是那些降解峰大于一个读数的,是转录本上的最大值,但有多个最大值。第2类峰值是那些有多个阅读,并且高于成绩单上平均片段丰度的峰值。第3类信号是那些有多于一个读取,并且低于或等于平均片段丰度在转录本。使用带有PARESnip2的降解组测序标记进一步验证了特定miRNAs激活PHAS位点。为了更详细地了解不同sRNA类的沉默,我们使用了四个不同的数据集:ShortStack程序中每个位点的高表达主RNA、miRbase中的保守miRNAs、miRDeep2中注释的新miRNAs和PHAS tank中识别的phasiRNA。
基因本体富集分析
利用r3.6.1 Bioconductor包中的topGO程序对sRNA靶转录本进行基因本体富集分析。氧化石墨烯术语富集试验分别在模拟样品和感染样品上进行。在每一种情况下,背景集都是GO项显著基因组和前景集是任何有降解组靶向证据的基因。将模拟样本和感染样本进行比较,以确定哪些基因在模拟样本中富集而在感染样本中缺失,或者反之亦然。与…谈判p-value < 0.05被认为是显著富集或消耗[91].
裂解靶cDNA末端的5 '快速扩增
我们对降解组数据集中的一个乙烯响应因子基因进行了5 ' -RACE实验,该基因可能被植物sRNA切割。选择这个基因的原因是根据以前的文献,这些类型的基因被证明在植物抵御病原体攻击的防御反应中至关重要[92]和该siRNA与目标基因之间的高置信度互补位点根据psRNA目标服务器。此外,针对该基因的sRNA迄今尚未被鉴定,它不是miRNA或phasiRNA。我们使用从独立感染检测中收集的两个独立样本,按照制造商的协议(美国应用生物系统公司),使用首选RACE试剂盒进行5’RACE,不添加犊牛肠道磷酸酶。其中一个样品与用于降解组测序的样品相同,而另一个样品则不同。
简单地说,5 ' RACE适配器连接到5 μg总RNA,使用试剂盒中提供的通用RT引物和MMLV转录酶进行逆转录。第一次PCR是用1 μL cDNA和5 '外RACE引物和基因特异性外引物进行的。第二次嵌套PCR使用第一个PCR产物,内含5 ' RACE引物和内嵌套PCR引物。PCR产物显示在2%琼脂糖凝胶上。将扩增的DNA片段经凝胶纯化后克隆到TOP TA载体中,对5个独立克隆进行Sanger测序。
定量聚合酶链反应
RT-qPCR分析sRNA靶基因的表达水平。从模拟和感染的rna中提取1到5ug显著使用MMLV逆转录酶试剂盒(Sigma-Aldrich)将叶片样本转化为cDNA。在qPCR前将cDNA样品稀释1/20。根据制造商的说明,使用Bio-Rad Taq通用SYBR绿色Supermix进行qPCR分析。热循环器设置为95°C加热2分钟,然后95°C加热15秒,60°C加热30秒,72°C加热15秒,循环40次,然后72°C加热2分钟。每个样品使用三个生物重复和三个技术重复。相对表达式按log(2)计算——∆Ct方法归一化为显著管家肌动蛋白基因。5 ' RACE和qPCR实验所用引物和转接物见补充表9.
数据和材料的可用性
小rna和降解组测序数据已保存在GenBank中,项目名为BioProject PRJNA678586。
改变历史
2021年8月26日
缩写
- ta-siRNA:
-
反式作用短干扰RNA
- 核:
-
短RNA干扰
- sRNA:
-
小核糖核酸
- pha-siRNA:
-
阶段性短干扰RNA
- microrna的:
-
丙肝
- DCL:
-
Dicer-like
- natsiRNA:
-
天然反义短干扰RNA
- hetsiRNA:
-
异色短干扰RNA
- 苏维埃社会主义共和国:
-
菌核病茎腐病
- RDR:
-
依赖RNA的RNA聚合酶
- RISC:
-
RNA-induced沉默复杂
- NBS-LRR:
-
核苷酸结合位点富含亮氨酸重复
- 种族:
-
cDNA末端的快速扩增
- qPCR:
-
定量聚合酶链反应
- nt:
-
核苷酸
- 走:
-
基因本体论
参考文献
- 1.
博尔赫斯F,马丁森RA。植物中不断扩大的小rna世界。中国生物医学工程学报2015;16(12):727-41。https://doi.org/10.1038/nrm4085.
- 2.
奥扎塔DM,盖内迪诺夫I,佐奇A,奥卡罗尔D,扎莫尔警局。piwi相互作用的rna:功能大的小rna。中国生物技术学报2019;20(2):89-108。https://doi.org/10.1038/s41576-018-0073-3.
- 3.
王晓燕,王晓燕,王晓燕,等。植物小rna在非生物胁迫中的作用及其生物机制研究。蛋白质组学。2011;9(6):183-99。https://doi.org/10.1016/s1672 - 0229 (11) 60022 - 3.
- 4.
普拉特AJ,麦克雷IJ。rna诱导沉默复合体:一种多功能基因沉默机器。中国生物医学工程学报,2009;28(6):694 - 694。https://doi.org/10.1074/jbc.R900012200.
- 5.
刘伟伟,孟J,崔健,栾玉生。MicroRNA的特性和功能∗年代的植物。植物科学进展2017;8:2200。
- 6.
杨建军,史伯杰,王晓燕,等。大麦中磷调控的miR399和miR827及其异构体的鉴定。植物生物学杂志,2013;13(1):1 - 17。
- 7.
Trevisan S, Nonis A, Begheldo M, Manoli A, Palme K, Caporale G,等。玉米幼苗根系中硝酸盐响应mirna的表达及组织特异性定位。植物细胞环境学报,2012;35(6):1137-55。https://doi.org/10.1111/j.1365-3040.2011.02478.x.
- 8.
郑颖,王颖,吴娟,丁斌,费志军。植物阶段性小干扰rna的动态进化和功能景观。BMC医学杂志。2015;13:15。
- 9.
谢明宇:植物sirna定向的DNA甲基化。咕咕叫基因组学。2015;16(1):23-31。https://doi.org/10.2174/1389202915666141128002211.
- 10.
Howell MD, Fahlgren N, Chapman EJ, Cumbie JS, Sullivan CM, Givan SA,等。对拟南芥中RNA依赖的RNA聚合ase6 /DICER-LIKE4通路的全基因组分析揭示了mirna和tasirna定向靶向的依赖性。植物细胞。2007;19(3):926 - 42。https://doi.org/10.1105/tpc.107.050062.
- 11.
郑颖,王颖,吴娟,丁斌,费铮。植物相位小干扰rna的动态进化和功能景观。BMC医学杂志。2015;13(1):32。https://doi.org/10.1186/s12915-015-0142-4.
- 12.
de felppes FF, Marchais A, Sarazin A, Oberlin S, Voinnet O.一个miR390靶向事件足以触发拟南芥中TAS3-tasiRNA的生物发生。核酸学报,2017;45(9):5539-54。https://doi.org/10.1093/nar/gkx119.
- 13.
拟南芥中tasiRNAs的形成:microRNA miR173的作用。EMBO众议员2009;10(3):264 - 70。https://doi.org/10.1038/embor.2008.247.
- 14.
吴峰,陈勇,田旭,朱旭,金伟。番茄茄响应灰葡萄孢感染阶段性小干扰RNA基因的全基因组鉴定与鉴定。Sci众议员2017;7(1):1 - 10。
- 15.
李张C、G,王J,方J .识别trans-acting siRNAs及其监管瀑布的小道消息。生物信息学。2012;28(20):2561 - 8。https://doi.org/10.1093/bioinformatics/bts500.
- 16.
翟静,郑D-H,德保利E, Park S, Rosen BD, Li Y,等。MicroRNAs作为植物NB-LRR防御基因家族的主调控因子,通过产生阶段性的反式作用的sirna。基因Dev。2011;25(23):2540 - 53年。https://doi.org/10.1101/gad.177527.111.
- 17.
邓鹏,穆世盛,曹敏,吴磊。植物阶段性小干扰rna的生物发生及调控层次。植物学报。2018;16(5):965-75。https://doi.org/10.1111/pbi.12882.
- 18.
建辉,王娟,王涛,魏亮,李娟,刘磊。盐胁迫和干旱胁迫下油菜种子早期萌发相关microrna的鉴定。植物科学进展2016;7:658。
- 19.
阿查德P,赫尔A,鲍尔科姆DC,哈伯NP。赤霉素调控的microRNA对花发育的调控。发展。2004;131(14):3357 - 65。https://doi.org/10.1242/dev.01206.
- 20.
郭宏生,谢强,费建峰,蔡乃华。在拟南芥侧根发育过程中,MicroRNA可引导转录因子NAC1的mRNA切割下调生长素信号。植物细胞。2005;17(5):1376 - 86。https://doi.org/10.1105/tpc.105.030841.
- 21.
刘强,张永春,王春燕,罗永春,黄秋杰,陈绍勇,等。植物激素调控的microrna在水稻中的表达分析及其在植物激素信号传递中的调控作用。2月。2009;583(4):723 - 8。https://doi.org/10.1016/j.febslet.2009.01.020.
- 22.
Navarro L, Dunoyer P, Jay F, Arnold B, Dharmasiri N, Estelle M,等。植物miRNA通过抑制生长素信号通路促进抗菌素抗性。科学。2006;312(5772):436 - 9。https://doi.org/10.1126/science.1126088.
- 23.
杜军,吴刚,周铮,张杰,李明,孙敏,等。烟草卷曲芽病毒与倍卫星共感染在烟草中调控的microRNAs的鉴定。性研究j . 2019; 16(1): 30。https://virologyj.biomedcentral.com/articles/10.1186/s12985-019-1234-5.
- 24.
Islam W, Qasim M, Noman A, Adnan M, Tayyab M, Farooq TH,等。植物微rna:对抗入侵病原体的第一线玩家。活细胞Pathog。2018;118:9-17。https://doi.org/10.1016/j.micpath.2018.03.008.
- 25.
Gupta OP, Permar V, Koundal V, Singh UD, Praveen S. MicroRNA调控小麦锈病侵染过程中小麦的防御反应。Mol Biol journal, 2012;39(2): 817-24。https://doi.org/10.1007/s11033-011-0803-5.
- 26.
梁刚,艾青,于东。拟南芥营养缺乏间串扰的mirna的发现。Sci众议员2015;5(1):11813。https://doi.org/10.1038/srep11813.
- 27.
Sánchez-Sanuy F, Peris-Peris C, Tomiyama S, Okada K,邢永义,San Segundo B,等。osos - mir7695增强了水稻稻瘟病菌防御反应的转录启动。植物生物学杂志2019;19(1):1 - 16。
- 28.
Peláez P,桑切斯F.病毒和细菌相互作用期间植物防御反应中的小rna:相似和不同。植物科学进展2013;4:343。
- 29.
Shivaprasad PV,陈h - m, Patel K, Bond DM, Santos BA, Baulcombe DC。microRNA超家族调节核苷酸结合位点-富含亮氨酸重复序列和其他mrna。植物细胞,2012;24(3):859 - 74。https://doi.org/10.1105/tpc.111.095380.
- 30.
崔超,王建军,赵建华,方莹莹,何晓峰,郭慧生,等。芸苔属的一种miRNA通过不同的作用模式调节植物生长和免疫。摩尔。2020;13(2):231 - 45。https://doi.org/10.1016/j.molp.2019.11.010.
- 31.
Carré P, Pouzet A.油菜市场,全球和欧洲。OCL。2014; 21(1): 1 - 12。
- 32.
Del里约热内卢L, Bradley C, Henson R, Endres G, Hanson B, McKay K,等。油菜菌核茎腐病对油菜产量的影响。工厂说。2007;91(2):191 - 4。https://doi.org/10.1094/PDIS-91-2-0191.
- 33.
付勇,Mason AS,张勇,林斌,肖敏,付东,等。甘蓝型油菜MicroRNA-mRNA表达谱及其在镉胁迫响应中的潜在作用植物生物学杂志2019;19(1):1 - 20。
- 34.
王魏W,李G,江X, Y, Z,妞妞Z,等。小RNA和降解组在甘蓝型油菜种子发育和油合成中的作用。《公共科学图书馆•综合》。2018;13 (10):e0204998。https://doi.org/10.1371/journal.pone.0204998.
- 35.
沈丹,Suhrkamp I,王宇,刘s, Menkhaus J, Verreet JA,等。以油菜AA (Brassica rapa)和甘蓝CC (Brassica oleracea)为参考基因组,鉴定油菜(Brassica napus)对病原菌长孢黄萎病(Verticillium longisporum)感染反应的microRNAs并进行表征。新植醇。2014;204(3):577 - 94。https://doi.org/10.1111/nph.12934.
- 36.
曹建勇,徐一平,赵亮,李士生,蔡晓铮。甘蓝型油菜与菌核菌相互作用在microRNA水平上的严密调控。植物分子生物学杂志,2016;92(1-2):39-55。https://doi.org/10.1007/s11103-016-0494-3.
- 37.
简华,马军,魏磊,刘鹏,张安,杨斌,等。综合mRNA, sRNA和降解组测序揭示油菜对菌核病(lib.)感染的复杂反应。Sci众议员2018;8(1):17。
- 38.
Omidvar V, Mohorianu I, Dalmay T, Fellner M.在番茄7B-1雄性不育突变体花药发育和雄性不育调控中的潜在mirna的鉴定。BMC基因组学。2015;16(1):1 - 16。
- 39.
Schwach F, Moxon S, Moulton V, Dalmay T.解读植物中小rna的多样性:长与短。蛋白质组学。2009;8(6):472-81。https://doi.org/10.1093/bfgp/elp024.
- 40.
马X, Wiedmer J, Palma-Guerrero J.小麦与发酵酵母互作过程中的小RNA双向串扰。植物科学,2020;10:1669。https://doi.org/10.3389/fpls.2019.01669.
- 41.
罗曦,高铮,石涛,程铮,张铮,倪铮。桃子mirna及其靶基因的鉴定(英文)碧桃L.)使用高通量测序和降解组分析。8。2013;(11):e79090。
- 42.
李晓明,李晓明。植物免疫激素调节的研究进展。中国生物医学工程学报2012;28(1):489-521。https://doi.org/10.1146/annurev-cellbio-092910-154055.
- 43.
李松,刘亮,庄旭,于艳,刘旭,崔旭,等。MicroRNAs抑制拟南芥内质网靶mrna的翻译。细胞。2013;153(3):562 - 74。https://doi.org/10.1016/j.cell.2013.04.005.
- 44.
Addo-Quaye C, Miller W, Axtell MJ。CleaveLand:一个使用降解组数据来寻找分裂的小RNA靶点的管道。生物信息学。2008;25(1):130 - 1。https://doi.org/10.1093/bioinformatics/btn604.
- 45.
崔健,孙铮,李娟,程东,罗晨,戴晨。耐盐条件下甜菜幼苗miRNA160/164的鉴定及其靶标表达。植物分子生物学杂志,2018;36(5-6):790-9。https://doi.org/10.1007/s11105-018-1118-7.
- 46.
王燕,李凯,陈琳,邹燕,刘红,田燕,等。大豆根瘤和侧根发育需要microrna167调控生长素响应因子GmARF8a和GmARF8b。植物杂志。2015;168(3):984 - 99。https://doi.org/10.1104/pp.15.00265.
- 47.
陆勇,冯铮,刘旭,卞林,谢辉,张超,等。MiR393和miR390在不同条件下协同调控水稻侧根生长。植物生物学杂志,2018;18(1):261。https://doi.org/10.1186/s12870-018-1488-x.
- 48.
吴刚,Park MY, Conway SR, Wang J-W, Weigel D, Poethig RS. miR156和miR172的顺序作用调控拟南芥发育时间。细胞。2009;138(4):750 - 9。https://doi.org/10.1016/j.cell.2009.06.031.
- 49.
甘蓝型油菜RNA测序揭示细胞氧化还原控制菌核病感染。中国生物医学工程学报2017;68(18):5079-91。https://doi.org/10.1093/jxb/erx338.
- 50.
李勇,赵树林,李建林,胡晓辉,王辉,曹晓林,等。Osa-miR169负调控水稻对稻瘟病菌的免疫力。植物科学进展2017;8:2。
- 51.
Soto-Suárez M, Baldrich P, Weigel D, Rubio-Somoza I, San Segundo B.拟南芥miR396介导病原相关分子模式触发的针对真菌病原的免疫反应。Sci众议员2017;7(1):44898。https://doi.org/10.1038/srep44898.
- 52.
van Loon LC, Geraats BP, Linthorst HJ。乙烯作为植物抗病调节剂。植物科学进展;2006;11(4):184-91。https://doi.org/10.1016/j.tplants.2006.02.005.
- 53.
金宁,张玉杰,朴玉。AP2/ERF家族转录因子ORA59和RAP2。3在原子核中相互作用并在乙烯反应中共同起作用。植物科学,2018;9:1675。
- 54.
Ossowski S, Schwab R, Weigel D.利用人工微rna和其他小rna对植物进行基因沉默。植物j . 2008; 53(4): 674 - 90。https://doi.org/10.1111/j.1365-313X.2007.03328.x.
- 55.
金正日VN。分类小rna。细胞。2008;133(1):下手为强。https://doi.org/10.1016/j.cell.2008.03.015.
- 56.
吴亮,周浩,张强,张杰,倪峰,刘超,等。由microRNA途径介导的DNA甲基化。38摩尔细胞。2010;(3):465 - 75。https://doi.org/10.1016/j.molcel.2010.03.008.
- 57.
Szaker HM, Darkó É, Medzihradszky A, Janda T. Liu H-c, Charng Y-y, Csorba T: miR824/ agamish - like16模块集成了反复出现的环境热应力变化,以微调后应力发展。植物科学2019;10:1454。https://doi.org/10.3389/fpls.2019.01454.
- 58.
Camargo-Ramírez R, Val-Torregrosa B, San Segundo B. mir858介导的黄酮特异性MYB转录因子基因调控拟南芥对病原感染的抗性。植物细胞学报,2018;59(1):190-204。https://doi.org/10.1093/pcp/pcx175.
- 59.
Korfhage U, Trezzini GF, Meier I, Hahlbrock K, Somssich IE。植物同源结构域蛋白参与病原体防御相关基因的转录调控。植物细胞。1994;6(5):695 - 708。https://doi.org/10.1105/tpc.6.5.695.
- 60.
张强,李勇,张勇,吴超,王松,郝磊,等。Md-miR156ab和md-miR395靶向WRKY转录因子影响苹果叶斑病抗性。植物科学进展2017;8:526。
- 61.
Sarkar D, Maji RK, Dey S, Sarkar A, Ghosh Z, Kundu P.综合miRNA和mRNA表达谱分析揭示了番茄品种对早期枯萎病的反应调控因子。DNA研究》2017;24(3):235 - 50。https://doi.org/10.1093/dnares/dsx003.
- 62.
高峰,王楠,李辉,刘杰,付超,肖铮,等。利用高通量测序技术鉴定沙冬青干旱响应的microRNAs及其靶点Sci众议员2016;6(1):1 - 16。
- 63.
夏荣,徐军,梅耶斯。miR390-TAS3-ARF通路在陆生植物中的出现、进化和多样性植物细胞。2017;29(6):1232 - 47。https://doi.org/10.1105/tpc.17.00185.
- 64.
Matthewman CA, Kawashima CG, Húska D, Csorba T, Dalmay T. Kopriva S: miR395是拟南芥硫酸盐同化调节网络的一般组成部分。2月。2012;586(19):3242 - 8。https://doi.org/10.1016/j.febslet.2012.06.044.
- 65.
王晓燕,王晓燕,王晓燕:植物miR159基因的克隆及功能研究。植物2019年,8(8):255。
- 66.
张涛,赵艳玲,赵建辉,王松,金勇,陈志强,等。棉花植物输出microRNAs来抑制真菌病原的毒力基因表达。Nat植物。2016;2(10):1 - 6。
- 67.
Alonso-Peral MM, Li J, Li Y, Allen RS, Schnippenkoetter W, Ohms S, White RG, Millar AA: microrna159调控的gamyb样基因抑制生长和促进程序性细胞死亡。植物杂志。2010;154(2):757 - 71。https://doi.org/10.1104/pp.110.160630.Epub 2010 8月10日。
- 68.
杨颖,陈鑫,陈娟,徐辉,李娟,张铮:连作地黄植株中miRNA的差异表达。植物生物学杂志,2011,11(1):1 - 11,53,DOI:https://doi.org/10.1186/1471-2229-11-53.
- 69.
潘dey R, Joshi G, Bhardwaj AR, Agarwal M, Katiyar-Agarwal S.小麦组织特异性和非生物胁迫特异性mirna的全基因组研究。《公共科学图书馆•综合》。2014;9 (4):e95800。https://doi.org/10.1371/journal.pone.0095800.
- 70.
卡伯特C, Martos S, Llugany M, Gallego B, Tolrà R, Poschenrieder C.锌在植物防御病原体和食草动物中的作用。植物科学进展2019;10:1171。https://doi.org/10.3389/fpls.2019.01171.
- 71.
李mh,全浩,金汞,朴玉。拟南芥NAC转录因子NAC4在microRNA164负调控下促进病原菌诱导的细胞死亡。新植醇。2017;214(1):343 - 60。https://doi.org/10.1111/nph.14371.
- 72.
陈建明,陈娜丽,王亚杰。β-螺旋桨蛋白的许多叶片:保守但多功能。生物化学进展,2011;36(10):553-61。https://doi.org/10.1016/j.tibs.2011.07.004.
- 73.
Šola K, Gilchrist EJ, Ropartz D, Wang L, Feussner I, Mansfield SD,等。RUBY是一种半乳糖氧化酶,可影响拟南芥种皮表皮的果胶性质并促进细胞间粘附。植物细胞,2019,31(4):809 - 31所示。https://doi.org/10.1105/tpc.18.00954.
- 74.
Denton-Giles M,德比郡MC, Khentry Y, Buchwaldt L, Kamphuis LG。甘蓝型油菜对来自澳大利亚的高攻击性和遗传多样性菌核菌分离株的部分茎秆抗性。植物病理学杂志2018:1-11。
- 75.
Ma Z, Coruh C, Axtell MJ。拟南芥小RNA:拟南芥属中的瞬时MIRNA和小干扰RNA位点。植物细胞。2010;22(4):1090 - 103。https://doi.org/10.1105/tpc.110.073882.
- 76.
Martin M. Cutadapt从高通量测序读取中移除适配器序列。EMBnet日报》。2011; 17(1): 10 - 2。https://doi.org/10.14806/ej.17.1.200.
- 77.
FastQC:用于高通量序列数据的质量控制工具。在.:英国剑桥巴巴拉罕研究所巴巴拉罕生物信息学;2010.
- 78.
德比榭M,丹顿-贾尔斯M, Hegedus D, Seifbarghy S, Rollins J, van Kan J,等。植物病原真菌的全基因组序列菌核病sclerotiorum揭示了对广泛宿主病原体基因组结构的洞察。基因组生物学进展2017;9(3):593-618。https://doi.org/10.1093/gbe/evx030.
- 79.
后新石器时代甘蓝型油菜油籽基因组的早期异源多倍体进化(vol 348, 1260782, 2014)。科学。2014;345(6202):1255。
- 80.
Bushnell BBAF,精确,拼接感知对齐器。美国。: BBMap:一个快速,准确,拼接感知对齐器。美国。。2014.
- 81.
Axtell乔丹。ShortStack:小RNA基因的综合注释和量化。Rna。2013;19(6):740 - 51。https://doi.org/10.1261/rna.035279.112.
- 82.
Mohorianu I, Stocks MB, Applegate CS, Folkes L, Moulton V. UEA小RNA工作台:一套用于小RNA分析的计算工具。In: MicroRNA检测和目标识别:Humana出版社,纽约;2017.p . 193 - 224。
- 83.
Friedländer MR, Mackowiak SD, Li N, Chen W. Rajewsky N: miRDeep2准确识别7个动物分支中已知的和数百个新的microRNA基因。核酸学报,2012;40(1):37-52。https://doi.org/10.1093/nar/gkr688.
- 84.
Love MI, Huber W, Anders S.用DESeq2调节RNA-seq数据的折叠变化和色散估计。基因组医学杂志。2014;15(12):550。https://doi.org/10.1186/s13059-014-0550-8.
- 85.
郭强,曲旭,金伟。PhaseTank:全基因组phasiRNAs及其调控级联的计算识别。生物信息学,2015,31(2):284 - 6。https://doi.org/10.1093/bioinformatics/btu628.
- 86.
Kalvari I, Nawrocki EP, Argasinska J, Quinones-Olvera N, Finn RD, Bateman A,等。使用Rfam数据库进行非编码RNA分析。生物信息学杂志。2018;62(1):e51。https://doi.org/10.1002/cpbi.51.
- 87.
Nawrocki EP, Eddy SR. Infernal 1.1: RNA同源性搜索速度快100倍。生物信息学)。2013;29(22):2933 - 5。https://doi.org/10.1093/bioinformatics/btt509.
- 88.
戴X,赵PX。psRNATarget:植物小RNA目标分析服务器。核酸,供应2,W159。2011年,39:W155。
- 89.
Thody J, Folkes L, Medina-Calzada Z, Xu P, Dalmay T, Moulton V. PAREsnip2:利用可配置的靶向规则从降解组测序数据中高通量预测小RNA靶点的工具。核酸学报2018;46(17):8730-9。https://doi.org/10.1093/nar/gky609.
- 90.
法尔格伦N,卡灵顿JC。植物miRNA靶点预测。In:植物MicroRNAs:施普林格;2010.51-7页。
- 91.
Alexa A: Rahnenfuhrer, J. topGO:基因本体论的富集分析(R包版本2.40。0) Bioconductor;2020.
- 92.
黄鹏云,王晓燕,王晓燕,等。拟南芥乙烯免疫应答因子的研究。中国科学(d辑:自然科学版)2016;https://doi.org/10.1093/jxb/erv518.
确认
这项工作得到了Pawsey超级计算中心提供的资源的支持,由澳大利亚政府和西澳大利亚政府提供资金。作者要感谢Yuphin Khentry博士提供的技术援助。
资金
这项工作是在作物和疾病管理中心(CCDM)内进行的,该中心是科廷大学和谷物研究与发展委员会共同投资的(GRDC项目编号为CUR00023)。RR由澳大利亚政府研究培训计划和联邦科学与工业组织(CSIRO)的奖学金资助。资助者没有参与计划或进行研究;他们唯一的作用就是提供资金。监管
作者信息
从属关系
贡献
RR设计并分析了实验,并撰写了第一份手稿草稿。TEN和LGK参与了高水平和技术的实验讨论,并帮助开发手稿。MCD帮助设计、分析和展示实验数据,监督实验并参与撰写手稿。所有作者都阅读并认可了最终版本的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
这篇文章的原在线版本进行了修改:作者发现文章标题有两个错误。原文和勘误表的标题已经从“鉴定对坏死性病原体感染有反应的甘蓝型油菜小rna”改为“鉴定对坏死性病原体感染有反应的甘蓝型油菜小rna”。
补充信息
附加文件1:补充文件1
.显示候选PHAS位点的软件PHAS Tank的输出。
附加文件2:补充图1
.独特srna的大小分布。
附加文件3:补充图2
.裁剪凝胶的完整图像出现在图中。6.
补充文件4:补充表1
.使用程序Sho:rtStack识别的所有小RNA生物发生位点。
补充文件5:补充表2
.73个miRNA家族的裂解产物。所有使用降解组测序在感染和模拟样本中识别的靶点都包括在内。
补充文件6:补充表3
.基于受感染样本的降解组测序数据的保守microRNAs靶点。该表包括多个miRNA靶向的基因和同一miRNA靶向的多个基因。只包括在受感染样本中发现的目标。
补充文件7:补充表4
.仅在感染样本中被保守mirna靶向的基因。
补充文件8:补充表5
.保守的微RNA序列在感染过程中有差异表达的证据。
补充文件9:补充表6
.在这项研究中发现了已知的和新的miRNAs。
补充文件10:补充表7
.基于psRNA靶分析的可能受pha- sirna调控的5918个转录本的GO术语富集分析。
补充文件11:补充表8
.1601个mir1885触发的ta-siRNAs的psRNA靶预测靶的GO术语富集分析。
补充文件12:补充表9
.本研究中使用的引物。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
瑞米,R,纽曼,t.e.,坎普赫伊斯,L.G.et al。的识别芸苔属植物显著对坏死性病原体感染有反应的小rna。BMC植物杂志21日,366(2021)。https://doi.org/10.1186/s12870-021-03148-6
收到了:
接受:
发表:
关键字
- 小核糖核酸
- 微RNA
- Degradome
- 真菌病原体
- 显著
- pha轨迹
- 助教基因
- Ta-siRNA
- Pha-siRNA