摘要
背景
石榴是一种具有营养、药用、观赏和生态价值的优良树种。研究证实SPL因子在花的转变和花的发育中起着重要作用。
结果
利用生物信息学方法,从“泰山红”石榴中鉴定并分析了15个SPL (SQUAMOSA启动子结合蛋白样)基因。p . granatuml .)基因组。系统发育分析表明,PgSPLs可分为6个亚家族(G1 ~ G6)。PgSPL启动子序列包含多个独联体与非生物应激或激素反应相关的作用元素。根据转录组数据,不同组织和不同发育阶段的表达谱表明PgSPL基因具有明显的时空表达特征。小RNA测序结果中miR156的表达分析表明,miR156负向调控靶基因的表达。qRT-PCR分析显示PgSPL2,PgSPL3,PgSPL6,PgSPL11而且PgSPL14叶中含量显著高于芽和茎中含量(p< 0.05)。表达水平PgSPL5,PgSPL12而且PgSPL13花蕾中显著高于叶和茎中(p< 0.05)。的编码序列的全长PgSPL5而且PgSPL13通过同源克隆技术获得。全长PgSPL5是1020 bp,和PgSPL13为489 bp,分别编码339和162个氨基酸。进一步调查显示PgSPL5而且PgSPL13蛋白质位于细胞核内。外源植物生长调节剂诱导实验表明PgSPL5在叶和茎中表达上调。PgSPL13在叶片中表达上调,在茎中表达下调。分别喷洒6-BA、IBA和PP333,PgSPL5而且PgSPL13在两性和功能雄花P2期(花芽垂直直径为5.1 ~ 12.0 mm)上调最为显著。
结论
我们的研究结果表明PgSPL2,PgSPL3,PgSPL6,PgSPL11而且PgSPL14对石榴叶片发育有一定作用。PgSPL5,PgSPL12而且PgSPL13在石榴花发育中发挥了作用。PgSPL5而且PgSPL13参与了石榴发育过程中不同植物激素信号转导的响应过程。本研究为进一步的功能分析提供了坚实的基础SPL石榴中的基因。
背景
转录因子可以特异性结合独联体启动子的作用元件,其激活或抑制下游靶基因的表达。参与花器官发育的转录因子主要有MADS-box [1], bHLH [2], yabby [3.]和SPL [4].植物特异性SPL (SQUAMOSA启动子结合蛋白样)转录因子参与花的形成和发育、叶片形态发生等生物学过程[5,6].已知SPL家族成员具有高度保守的SBP结构域,由79个氨基酸组成,并具有C3H (C-C-CH)和C2HC (C-C-H-C)两种典型的锌指结构[7].SPL蛋白的c端与第二锌指结合,促进其与DNA结合以发挥功能[8].
SPL家族基因最初是从答:majus[9].它们通过与MADS-box家族基因的SQUAMOSA启动子结合来调控植物的早花[9].在拟南芥, 17SPL基因已被分离出来并命名为SPL1~SPL16.AtSPL13是一对副同源基因,命名为SPL13A而且SPL13B[10].近年来,许多研究者相继在其他物种中鉴定出SPL家族基因,如28SPL杨树的基因(杨树trichocarpa) [11],番茄含15粒(茄属植物lycopersicum) [12],大米19粒(o .漂白亚麻纤维卷) [13], 27名苹果(m .释放有Borkh。)14]和18SPL葡萄的基因(葡萄l .) [15].
MicroRNAs (miRNAs)是一类内源性非编码rna,长度约为20-24 nt,来源于含有茎环二级结构的primary miRNA转录本(pri-miRNAs) [16,17].已知它们通过mRNA切割或翻译抑制在转录后水平抑制靶基因的表达[18,19].在植物中,miRNAs在植物器官发育中起着至关重要的作用[20.]、应力耐受性[21],植物激素信号[22],生长阶段变化[23],以及抗病能力[21].miR156是一类高度保守的mirna,参与调控植物从营养阶段到生殖阶段的转化[24,25,26].在17日SPL基因在拟南芥, 11个成员有miR156结合位点[27,28,29,30.].根据SBP结构域的大小,可以得到SPLmiR156调控的基因可分为两类。一个包括SPL3,SPL4,SPL5它们编码更小的蛋白质,促进花朵形成;另一个SPL9而且SPL15编码较大的蛋白质,主要促进叶片形成和开花[31,32].
之前的研究发现,SPL2通过激活调节花器官发育和植物育性不对称叶2(AS2) [33].SPL3/4/5能激活表情吗多叶的(LFY),FRUITFULL(富尔语),APETALA1(AP1)在花分生组织中[34,35].作为一个不受miR156调控的基因,SPL8与其他靶向mir156的蛋白起冗余作用SPL调控小孢子囊发育、大孢子发生、萼片毛体形成、雄蕊花丝伸长,从而控制植物雄性育性的基因[36].SPL9而且SPL10通过直接激活miR172来控制开花时间[28].SPL15是花期的积极调控因子,它与constans抑制因子1(SOC1)诱导拟南芥花卉发育[37].SPL2,SPL8,SPL9而且SPL15有保持男性生育能力的作用吗拟南芥[38,39].
石榴是一种具有营养、药用、观赏和生态价值的优良树种[40].研究证实SPL因子在花的转变和花的发育中起着重要作用[32,34,35,37,41].它是否在石榴的发育中起作用还没有报道。以‘泰山红’石榴全基因组数据为基础,采用生物信息学方法对SPL家族成员进行了鉴定。对其理化特性进行了分析。的进化关系、基因结构、保守基序、差异独联体探讨了各成员的作用元件及基因组织特征的表达。预测miR156的靶位点。的时空表达特征PgSPL5而且PgSPL13对克隆、亚细胞定位和生长调节剂处理下的基因进行分析,为揭示其在石榴花诱导中的生物学功能提供参考。
结果
SPL转录因子的鉴定与鉴定
从“泰山红”石榴基因组中鉴定出15个SPL基因家族成员。为了便于后续分析,将它们命名为PgSPL1~PgSPL15(表1).分析了PgSPL成员的蛋白质序列和理化性质。研究结果表明PgSPL基因发生了巨大变化。最短的蛋白质编码162个氨基酸(PgSPL13),最长为1049个氨基酸(PgSPL9),分子量为18563.02 ~ 115,942.26 kDa。理论等电点为5.38 ~ 9.39PgSPL13低至5.38PgSPL14高达9.39。含有10种酸性蛋白和5种碱性蛋白。这些结果为PgSPL蛋白的进一步纯化、活性和功能研究提供了理论基础。亚细胞定位结果显示,除PgSPL4,PgSPL6而且PgSPL12在细胞核和细胞质中。
系统发育分析
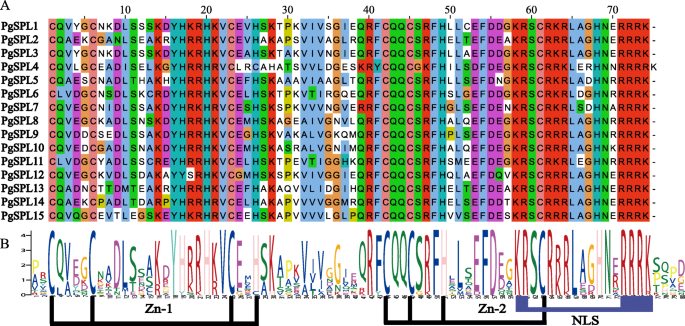
多序列比对结果(图;1)表明,每个SPL转录因子家族成员都含有一个高度保守的SBP结构域,由79个氨基酸残基组成。它们都包含两个锌指结构cys - cys - his (C3H), Cys-Cys-His-Cys (C2HC)和一个核定位信号(NLS)。的n端锌指结构PgSPL4是Cys4 (C4),与其他成员不同。
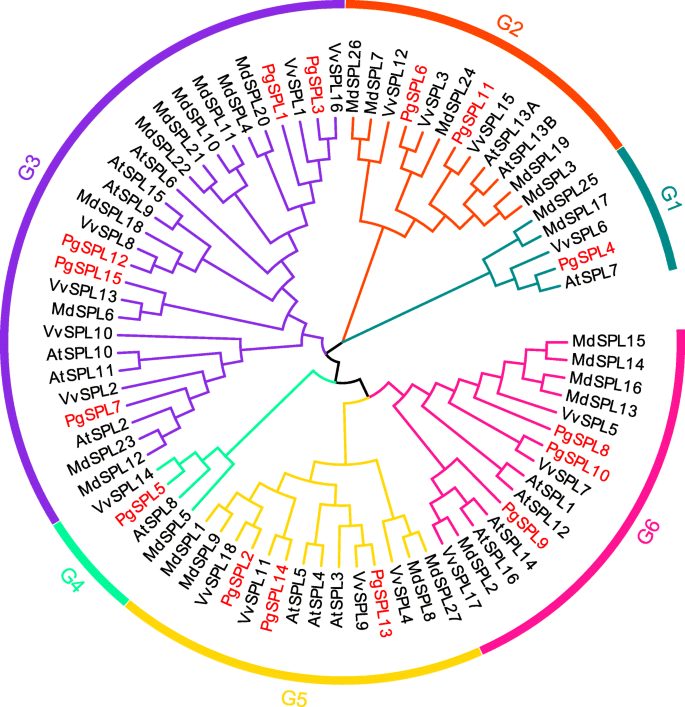
系统发育树结果显示15SPL石榴的基因聚为6个亚家族(G1 ~ G6)。2) [42,43].在这6个亚群中,G1亚群与其他亚群的遗传关系较远,形成了一个相对独立的分支。进一步分析发现,G1亚组的5个成员SBP结构域的n端锌指结构AtSPL7,PgSPL4,VvSPL6,MdSPL17,MdSPL25为C4,而其他子群中所有成员的值为C3H。SPL转录因子家族成员在G3亚组中分布最广。其中,15个石榴中有5个SPL基因分布在G3亚组。G4子组各有一个石榴,拟南芥、苹果和葡萄的SPL基因。有两个PgSPL在G2亚群中有3个基因PgSPLG5和G6基因。
基因结构分析及基序鉴定PgSPLs
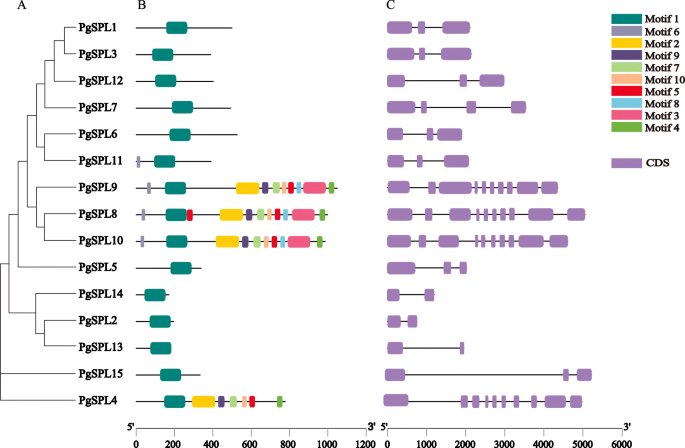
的结果PgSPLs基因结构显示内含子数量为1 ~ 9个(图;3.c). G1和G6的成员包含9个内含子。在G2、G3和G4亚组中,成员除有2个内含子外PgSPL7拥有3个内含子。G5亚群中的PgSPLs仅含有1个内含子。
在PgSPLs中鉴定出10个保守基序(图2)。3.b).所有PgSPL蛋白均含有SBP结构域Motif1,该结构域由约79个氨基酸组成。同一亚科的PgSPLs成员具有相似的基因结构和蛋白保护基序。在G2 ~ G5亚群中,所有成员均仅包含SBP结构域,除PgSPL11.然而,G1和G6亚群中的成员在其他亚群中存在一些无结构域或非典型结构域,这些保守基序的分布差异可能是造成基因功能差异的原因。
独联体-作用元素位于SPL基因启动子
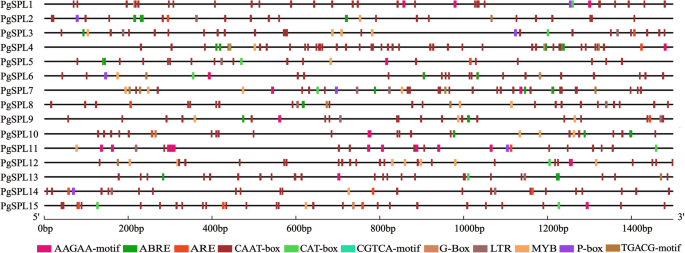
的独联体-作用元件包含了广泛的变化,从23英寸PgSPL14到42英寸PgSPL7(无花果。4).PgSPLs对不同的植物激素和非生物胁迫信号有反应,包括AAGAA-motif和ABRE独联体-作用元件涉及脱落酸反应、赤霉素反应元件P-box、茉莉酸甲酯反应元件CGTCA-motif和TGACG-motif,是必需的独联体-调节元件ARE用于厌氧诱导独联体-作用调控元件CAT-box,与分生组织表达相关独联体在光反应性、冷胁迫响应元件LTR、干旱诱导响应元件MBS和常见的独联体-作用元件CAAT-box在启动子和增强子区域。在15PgSPLs11个基因含有AAGAA-motif和ABRE元件,7个基因含有P-box元件,8个基因含有CGTCA-motif和TGACG-motif元件。10个成员包含ARE元素,9个成员包含CAT-box元素,5个基因包含G-Box元素,9个基因包含胁迫相关元素LTR和MBSPgSPL含有CAAT-box增强子的基因。
的预测PgSPLmiR156靶向的基因
许多研究表明,大多数SPL基因受miR156调控,miR156靶位点位于编码区或3'UTR区。为了确定石榴转录后调控机制,在石榴的编码区和3'UTR区寻找miR156靶位点PgSPLs.结果显示PgSPL1,PgSPL2,PgSPL3,PgSPL6,PgSPL7,PgSPL11,PgSPL12,PgSPL13,PgSPL14而且PgSPL15是miR156的潜在靶点,这些基因聚集在G2、G3和G5亚组。的PgSPLs编码区有潜在miR156靶点的基因属于G2和G3,而PgSPL2,PgSPL13而且PgSPL143’utr位点属于G5亚群。PgSPL基因序列与成熟的比较PgmiR156结果表明,PgSPL中超过一半的PgSPL含有互补的序列PgmiR156序列不超过两个不匹配的碱基(图;5).这一结果与其他物种相似[43,44].
转录组数据的表达模式
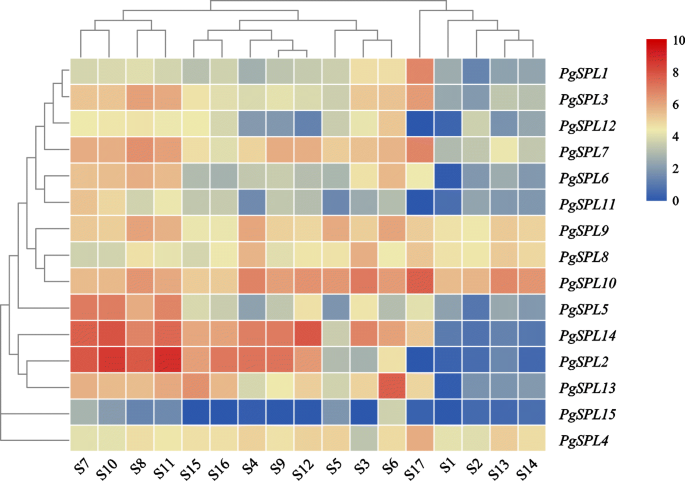
如图所示。6,在不同组织中进行转录组比较分析。结果表明PgSPL1而且PgSPL3在果皮中表达量较高,花中表达量较高,种皮中表达量较低。PgSPL2花中表达量较高,种皮内外表达量中等,而奇妙果皮中表达量不高。差异无统计学意义PgSPL4所有组织的表达水平。PgSPL5两性花I期(3.0 ~ 5.0 mm)、功能雄花I期(3.0 ~ 5.0 mm)和II期(5.1 ~ 13.0 mm)表达量较高,内种皮表达量较低。PgSPL6在外种皮中表达量低。PgSPL7外种皮和两性花II (5.1 ~ 13.0 mm)表达量较高。PgSPL8而且PgSPL9表现出相似的表情模式。PgSPL10在“奇妙”果皮中高度表达。PgSPL11而且PgSPL12并没有在“美妙”的果皮中表达。PgSPL13在叶片中表达量最高,在两性花和功能性雄花的I、II期表达量较高,但在外种皮中表达量较低。PgSPL14功能性雄花I和III的表达量最高。最高水平PgSPL15在叶片中有表达,而在其他组织中低表达或不表达。
的表达热图SPL石榴不同组织中的基因。S1:外种皮;S2:内种皮;S3:果皮;S4;花;S5:根;S6:肉叶;S7:两性花(3.0 ~ 5.0 mm);S8:两性花(5.1 ~ 13.0 mm); S9: Bisexual flowers (13.1 ~ 25.0 mm); S10: Functional male flowers (3.0 ~ 5.0 mm); S11: Functional male flowers (5.1 ~ 13.0 mm); S12: Functional male flowers (13.1 ~ 25.0 mm); S13: Inner seed coat of ‘Tunisia’; S14: Inner seed coat of ‘Baiyushizi’; S15: Mix of leaves, flowers, fruit and roots of ‘Black127’; S16: Mix of leaves, flowers, fruit and roots of ‘nana’; S17: Peels of ‘Wonderful’ (cultivars S1 ~ S6 are ‘Dabenzi’, cultivars S7 ~ S13 are ‘Tunisia’)
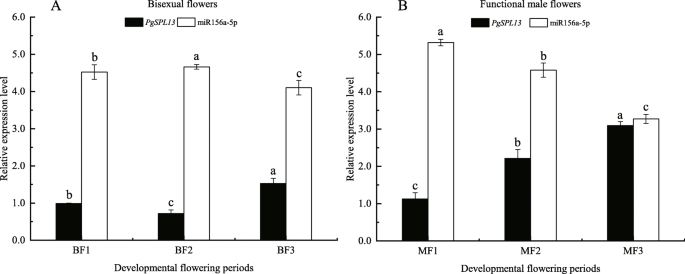
我们也验证了miR156a-5p而且PgSPL13在石榴花发育的不同阶段有不同的表达模式(图;7).研究发现,在两性花的发育过程中(图;7A),表达miR156a-5p的表达先增加后减少,而PgSPL13先减少后增加。并且在每个时期的表达都有显著差异(p< 0.05)。在功能性雄花发育过程中(图;7B),的表达miR156a-5p的表达显著降低,而PgSPL13显著增加(p< 0.05)。
qRT-PCR分析PgSPLs
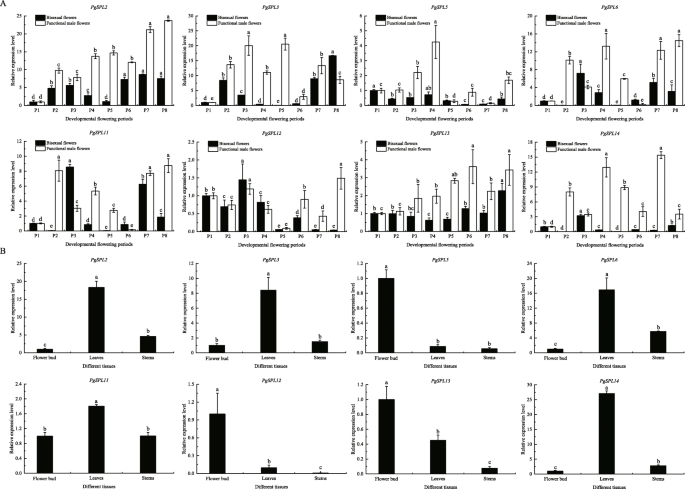
从无花果。6, 8个差异表达PgSPLs筛选基因,得到基因的表达模式PgSPL采用qRT-PCR方法对石榴不同组织中的基因进行分析。结果如图所示。8的表达水平PgSPL2两种花的P7期和P8期表达量最高,两性花P5期表达量显著低于其他期(p< 0.05)。我们发现的表达水平PgSPL3两性花P4 ~ P6期显著低于其他期。P6 ~ P8期显著升高(p< 0.05),且P2 ~ P5期功能性雄花表达较高。PgSPL5在功能性雄花中表达量高于两性花,且在两性花发育过程中表达量呈上升-下降-上升趋势。在功能性雄花P2 ~ P4期,基因表达水平明显降低PgSPL5显著增加(p< 0.05), P4期达到最高水平,之后略有下调。表达水平PgSPL6两性花P2期和P5期显著低于其他期(p< 0.05)。表达水平PgSPL6功能雄花以P8期最高,P4、P7期次之,P6期次之。大量的PgSPL11在两性花P6期均有表达,而在其他阶段均无表达(p< 0.05),且在P5期表达量最低。在功能性雄花的P2、P7和P8阶段,基因表达水平明显降低PgSPL11均显著高于其他各阶段(p< 0.05)。表达水平PgSPL12两种花在P2 ~ P4期均较高,且趋势一致。两性花P5、P7和P8期的表达量显著低于其他期(p< 0.05),在功能雄花P8期表达量最高。表达水平PgSPL13两性花在发育过程中呈先下降后上升的趋势,功能性雄花则相反。PgSPL13在功能性雄花中表达量高于两性花,在两性花的P8期和功能性雄花的P6期达到最大值。表达水平PgSPL14P3期较高,其他阶段较低。在功能性雄花中,表达水平PgSPL14以P4和P7阶段最高,其次为P2和P5阶段。
从图中也可以看出。8表达水平PgSPL2,PgSPL13而且PgSPL14功能雄花P1 ~ P8期的含量高于两性花。表达水平PgSPL2,PgSPL3,PgSPL6,PgSPL11而且PgSPL14幼叶中显著高于花蕾和茎中(p< 0.05)。表达水平PgSPL5,PgSPL12而且PgSPL13花蕾中显著高于叶和茎中(p< 0.05)。
克隆及序列分析PgSPL5而且PgSPL13
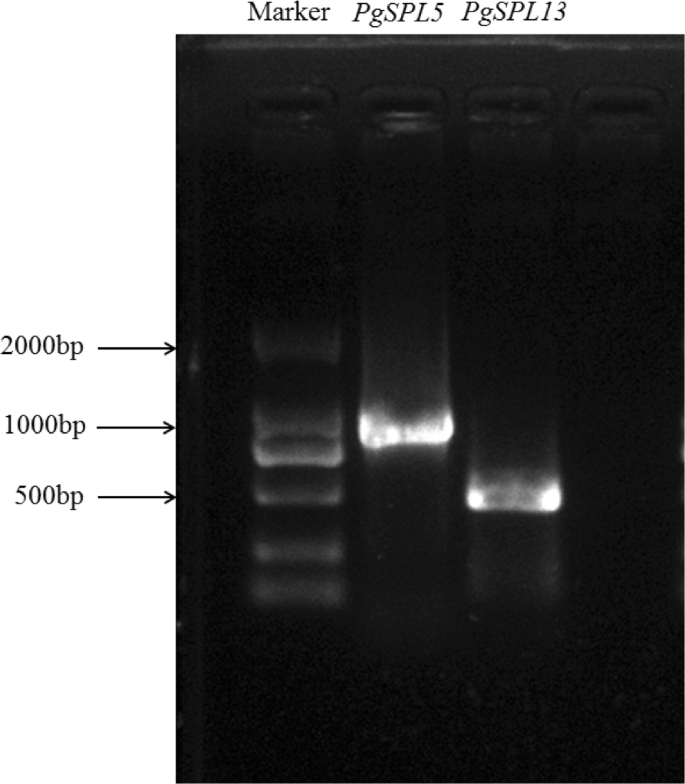
PgSPL5而且AtSPL8在系统发育树中聚为同一类群,重建的系统发育树也表明PgSPL13在同一根树枝上AtSPL3.研究表明AtSPL8在花药发育过程中起重要作用[36),而AtSPL3在?的发展中起到了非常重要的作用拟南芥花(34,35].因此,我们推测PgSPL5而且PgSPL13也在石榴花发育中起作用。数字8的表达水平PgSPL5而且PgSPL13花蕾中含量显著高于叶和茎中含量(p< 0.05),进一步说明这两个基因在石榴花发育中具有重要作用。因此,我们选择了这两个基因进行克隆。以“泰山红”石榴cDNA为模板,PgSPL5引物和PgSPL13引物分别进行PCR扩增。电泳条带与预期靶片段大小一致(图2)。9).的编码区域序列PgSPL5而且PgSPL13测序得到序列分别为1020 bp和489 bp,分别编码339和162个氨基酸。预测蛋白分子量分别为36911.79 kDa和18563.02 kDa。两个基因都包含一个完整的SBP保守结构域(图。10).
亚细胞定位PgSPL5而且PgSPL13
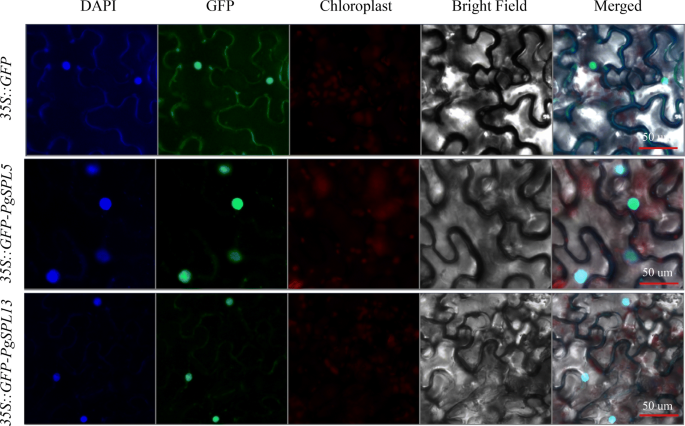
的亚细胞定位PgSPL5而且PgSPL13通过在线工具Cell-PLoc进行预测[45],发现它们都位于细胞核内。为了验证上述预测结果,我们在烟叶中瞬时表达35S::GFP-PgSPL5和35S::GFP-PgSPL13融合蛋白,并发现这两个基因确实位于细胞核中(图。11).据推测PgSPL5而且PgSPL13可能与基因表达的调节有关。
外源激素对细胞表达的影响PgSPL5而且PgSPL13
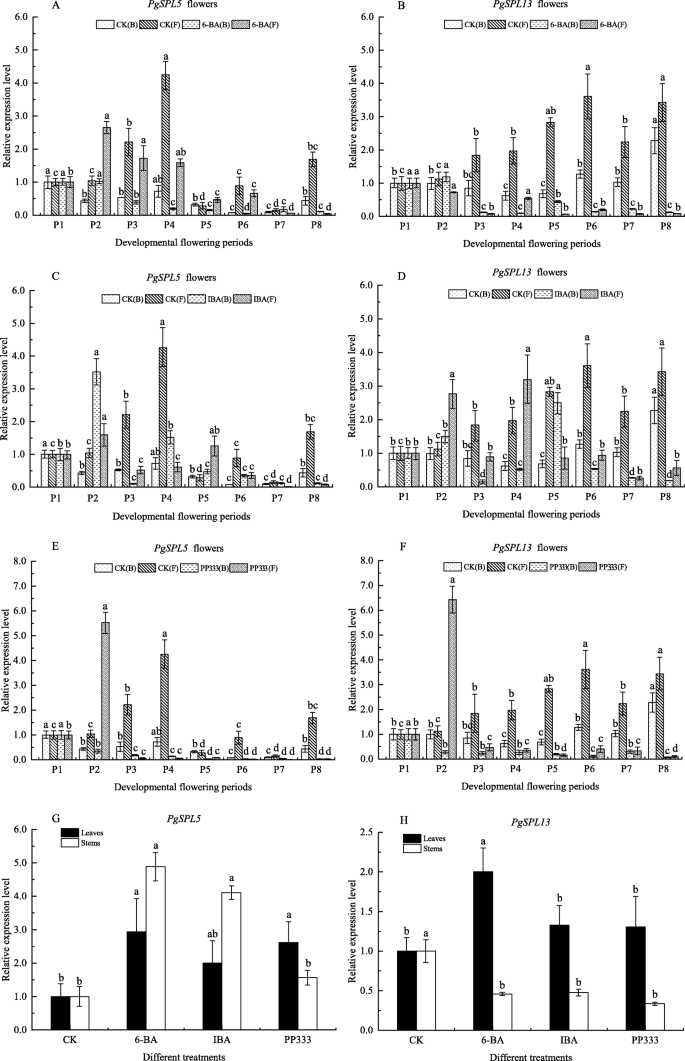
结果表明,适当喷施植物生长调节剂可促进花芽分化,诱导雌花形成,提高植株抗逆性,提高产量。以研究是否表达模式PgSPL5而且PgSPL13基因受不同生长调节剂的影响,通过qRT-PCR分析了不同处理的详细表达模式,不同处理组之间差异较大。
如图所示。12A和b,的表达水平PgSPL56-BA处理下,两种花的P2期表达均显著上调,两性花P7期表达上调但不显著。在两性花的其他发育阶段,表达PgSPL5经外源性6-BA处理后,PgSPL5在功能雄花的P3、P4和P8阶段表达显著下调。表达显著增加PgSPL5在6-BA处理下,叶片和茎部均有显著差异(图;12G),这种变化在茎中更为明显。PgSPL136-BA处理后两性花P2期表达上调,其余各期均下调。的表达式PgSPL13在6-BA处理下,叶片中显著上调,而在茎中显著下调(图2)。12h)。
经IBA处理后(图;12C, d),我们发现的表达式PgSPL5在两性花的P3和P8期表达下调,在两性花的其他发育阶段表达上调,其中P2期表达上调最为显著。在功能性雄花P2和P5期,基因表达水平PgSPL5IBA处理显著上调,其他阶段显著下调。在叶和茎中,表达PgSPL5IBA处理与6-BA处理有相同的变化,但这种变化不如6-BA处理明显。PgSPL13在两性花的P2、P5期和功能性雄花的P2、P4期均显著上调,而在花的其他发育阶段均显著下调。类似地,的表达式PgSPL13IBA处理与6-BA处理叶片和茎的变化相同。
从Fig可以看出。12E和f的表达式PgSPL5而且PgSPL13喷施外源PP333后,在功能雄花P2期表达显著上调,而在花发育的其他阶段均被抑制。PgSPL5在叶和茎中表达上调,而PgSPL13在叶片中表达上调,在茎中表达下调。这与6-BA和IBA处理中观察到的趋势一致。
讨论
近年来,许多研究指出SPL转录因子家族在植物生长发育和调控多种生理过程中起着基础性作用[19,46,47].在本研究中,共有15个SPL采用生物信息学方法,从“台山红”石榴全基因组中鉴定基因。我们从系统发育分析中得到的信息是这15SPL基因被分为六个亚群。这一结果表明p . granatum,答:芥,m .释放有而且葡萄在进化过程中相对保守[48].基因结构和基序组成分析表明PgSPL同一类群内的基因具有相似的基序和外显子-内含子组织,这表明这些基因可能来源于相同的祖先基因,在植物生长发育中发挥相似的功能[49].相对于其他亚群,G1和G6的保守结构域更为复杂,这些亚群的成员可能具有其他功能[50].
的作用独联体影响基因表达的作用元件近年来越来越受到重视[2].本研究发现所有PgSPL启动子均含有多个独联体提示PgSPL可能在植物对非生物胁迫和激素胁迫的反应中发挥作用。特别是激素诱导的生长素、赤霉素等相关元素的发现,提示PgSPL家族基因与石榴的生长发育有关。
SPL家族基因受miRNA156调控。有11个SPL基因在拟南芥由miR156调控,在植物生长发育中发挥多种功能[29,30.,32].本研究预测在石榴中,15个SPL家族成员中,有10个具有miR156结合位点。除了潜在的miR156靶点PgSPL2,PgSPL13而且PgSPL14G5亚组的基因均位于3’utr,其他基因的潜在靶基因均位于编码区。
转录组表达数据显示PgSPL除了极少数外,基因在叶、根、花、种皮和果皮中都有表达PgSPLs在果皮和各种组织的混合物中不表达。这说明PgSPL家族基因参与了石榴叶片发育、相变、花发育和果实成熟等过程。大多数PgSPL基因在两性花和功能性雄花的不同发育时期均有高表达,这些数据支持了该假说PgSPL基因在石榴开花过程中起着重要作用。同时,发现的表达PgSPL4在各组织中差异不显著,这可能与影响其基因功能的基因编码蛋白锌指结构C3H区突变有关。PgSPL8,PgSPL9而且PgSPL10通常在所有组织中都有高表达,这可能与存在其他更保守的基序有关。此外,的水平PgSPL15在所有组织中都相对较低。因此,可以推测其对石榴发育的调节作用较弱。的表达差异也进行了分析miR156a-5p而且PgSPL13在石榴两性花和功能性雄花发育的三个不同阶段,发现它们的表达水平呈相反的趋势,这与miR156负调控其靶基因表达的结论是一致的[29].
八个PgSPLs采用qRT-PCR方法分析石榴不同组织中表达差异显著的基因。结果表明PgSPL2,PgSPL13而且PgSPL14P1 ~ P8期功能性雄花的表达量高于两性花,表现出相似的表达规律。的表达模式PgSPL6而且PgSPL11在两种花中也有相似之处,这可能与它们聚在同一亚群中发挥相似的功能有关[50].石榴发育过程中花芽雌蕊部分流产形成功能性雄花,而功能性雄花雌蕊流产是由于胚珠发育异常,不能形成卵细胞[51,52,53].研究表明花芽纵径5.1 ~ 15.0 mm是石榴胚珠发育的重要阶段[54].PgSPL5在功能性雄花中表达量高于两性花。在功能雄花P2 ~ P4期(5.1 ~ 12.0 mm),蛋白表达水平明显降低PgSPL5显著增加。因此,我们推测PgSPL5可能参与石榴功能性雄花形成的调控。PgSPL12胚珠发育异常前在两种花中表达模式相似,胚珠发育异常后主要在功能性雄花中发挥作用。PgSPL12,AtSPL9而且AtSPL15聚集在同一分支,而AtSPL9而且AtSPL15可调节花期[28]和诱导拟南芥开花[30.].这表明PgSPL12在花的发育中也起着重要的作用。表达水平PgSPL13两种花的发育都相对稳定,这说明PgSPL13对石榴花器官的发育起一定作用。我们还发现PgSPL2,PgSPL3,PgSPL6,PgSPL11而且PgSPL14幼叶中磷含量显著高于花蕾和茎。这说明这些基因在叶片发育中起着重要作用。表达水平PgSPL5,PgSPL12而且PgSPL13花蕾中表达量显著高于叶和茎中表达量,进一步说明这3个基因在石榴花发育中具有重要作用。
在拟南芥,一些AtSPL基因比如SPL3而且SPL8参与植物发育过程中对GA信号转导的响应,如开花转化[34]和花药发展[39].许多研究表明SPL3在?的发展中有非常重要的作用拟南芥花(35,38,55].此外,SPL3,SPL4而且SPL5参与控制开花时间和发育过渡拟南芥[56].研究发现,来自不同物种的同源体通常在生物学过程中具有相似的功能。例如,同源基因OsSPL13,ZmSBP6而且AmSBP1的SPL3涉及花序和花在o .漂白亚麻纤维卷[13],玉米[57),答:majus[58),分别。在这项研究中,我们进行了克隆SPL8而且SPL3同源基因PgSPL5而且PgSPL13通过同源克隆技术从‘泰山红’提取,亚细胞定位验证证明它们在细胞核中发挥作用。我们研究了它们在外源性激素处理下的时空表达特征,发现外源性激素处理后,表达PgSPL5而且PgSPL13被不同植物激素上调或下调。它们在石榴叶、茎、两性花、功能性雄花等组织中均有差异表达,且对各种不同激素的反应也不同。说明这两个基因可能参与了石榴发育过程中不同植物激素信号转导的响应过程。我们发现,在花组织中,这种反应在花蕾幼嫩(P2期)时更强,叶片和茎中这两个基因的水平受到6-BA处理的显著影响。本研究只分析了这两个基因可能的功能PgSPL5而且PgSPL13在石榴的发展中。然而,这两个基因是如何发挥作用的,它们之间是否存在某种机制,还有待进一步研究。
结论
在这项研究中,我们确定并描述了15SPL石榴中的基因。系统发育分析表明,PgSPLs被分为6个亚群,每个亚群成员的保守基序分布和外显子-内含子结构相似。有10个SPL石榴中受miR156调控的基因。根据不同组织的转录组数据,我们发现某些PgSPL基因在石榴生长发育中起着重要作用。qRT-PCR分析显示PgSPL2,PgSPL3,PgSPL6,PgSPL11而且PgSPL14对石榴叶片发育有一定作用。PgSPL5,PgSPL12而且PgSPL13在石榴花发育中发挥了作用。亚细胞定位结果表明PgSPL5而且PgSPL13蛋白质位于细胞核内。此外,外源植物生长调节剂诱导实验表明PgSPL5而且PgSPL13参与了石榴发育过程中不同植物激素信号转导的响应过程。我们发现,在花组织中,这种反应在花蕾幼嫩(P2期)时更强,叶片和茎中这两个基因的水平受到6-BA处理的显著影响。
方法
SPL基因家族的鉴定与序列分析
Pfam SBP/SPL结构域(PF03110)的HMM谱图[8采用HMMER 3.0对“台山红”石榴蛋白数据库(http://hmmer.org/)软件包(Evalue <1e .−5) [1],去除重复,筛选SPL蛋白序列。的SPL蛋白序列答:芥,大肠茅,m .释放有而且葡萄在PlantTFDB数据库中(http://planttfdb.gao-lab.org/index.php) [59]作为查询,对石榴基因组数据库进行BLAST (E-value <1e .−5,身份> 50%)[3.].所有获得的SPL蛋白序列在CDD网站(https://www.ncbi.nlm.nih.gov/cdd) [60],以及SMART (http://smart.embl-heidelberg.de/) [61]来验证SBP域的保守域。
在线工具ExPaSy-Protparam (https://web.expasy.org/protparam/) [62],对石榴SPL家族的分子量、等电点等理化性质进行了预测和分析。亚细胞定位预测使用在线网站(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/) [45].
系统发育分析
为了探索石榴SPL家族基因的系统发育关系,研究了石榴SPL蛋白答:芥(17),m .释放有(27)和葡萄(18)下载进行多序列比对。采用MEGA 7.0, bootstrap = 1000次重复,完全缺失,Poisson模型构建无根邻居连接(unroots Neighbor-Joining, NJ)系统发育树[63].EvolView (https://www.evolgenius.info/evolview/) [64]在线进行系统发育树的美化。
基因结构分析和基序鉴定
利用MEME在线工具(http://meme-suite.org/) [65],得到SPL蛋白的基序特征。外显子-内含子结构SPL根据“泰山红”基因组注释信息确定基因,用GSDS 2.0 (http://gsds.cbi.pku.edu.cn)网上工具[66].
独联体表演元素
从石榴基因组序列中获得了起始密码子上游1500 bp的序列独联体-作用元素分析(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) [67].
的预测PgSPLmiR156靶向的基因
使用psRNATarget网上软件(http://plantgrn.noble.org/psRNATarget/) [68]分析石榴SPL基因的核苷酸序列,预测石榴miR156的靶基因位点。利用DNAMAN软件对PgmiR156和PgSPL序列的反补序列进行多序列比对[43].
转录组数据的表达模式
的表达模式SPL在不同石榴组织和器官中的基因,下载已发表的转录组数据进行表达分析。石榴两性花、功能雄花、叶、根、外种皮、内种皮、果皮的转录本数据来自NCBI数据库(http://www.ncbi.nlm.nih.gov/) [3.].注册号为“大本子”SRR5279388、SRR5279391、SRR5279394-SRR5279397;“突尼斯”SRR5446592、SRR5446595、SRR5446598、SRR5446601、SRR5446604、SRR5446607和SRR5678820;“Baiyushizi”SRR5678819;“Black127”SRR1054190;“nana”SRR1055290和“Wonderful”SRR080723。首先,对所有RNA-Seq数据进行fastp定性控制,获得清洗后的reads。然后用石榴转录组数据对该序列进行索引。通过Kallisto 0.44.0软件[69],并进一步计算和分析基因表达量。的对应表达级别(TPM值)SPL由此得到基因。将TPM值转换为Log2(tpm + 1)。的热图PgSPL用TBtools绘制[1].
同时,分析了石榴两性花和功能雄花3个不同阶段(花蕾垂径3.0 ~ 5.0 mm、5.1 ~ 13.0 mm、13.0 ~ 20.0 mm) miR156的表达情况。
植物材料与外源激素处理
南京林业大学白马教学科研基地调研试验品种为6年生‘泰山红’石榴。选择3种植物生长调节剂,喷施浓度如下(表2) [70].每组3株,每株喷滴处理。单株分别于2019年10月15日(休眠期)、2020年4月15日(春叶展开期)、5月10日(初花期至盛花期)和5月25日(盛花期结束)进行处理。
采集样品不需要权限。从5月10日起,各处理7 d后采集新叶和茎,采集石榴不同发育阶段的两性花和功能雄花。参考Chen和Zhao对石榴花形态和胚胎差异的研究[3.,71],根据花蕾的纵向直径,将花蕾分为8个阶段(图;13): 3.0 ~ 5.0毫米(P1), 5.1 ~ 8.0毫米(P2), 8.1 ~ 10.0毫米(P3), 10.1 ~ 12.0毫米(P4), 12.1 ~ 14.0毫米(P5), 14.1 ~ 16.0毫米(P6), 16.1 ~ 18.0毫米(P7)和18.1 ~ 20.0毫米(P8)。样品用液氮冷冻,保存在- 78°C冰箱中备用。
RNA分离和基因克隆
使用BioTeke植物总RNA提取试剂盒(BioTeke Corporation,北京,中国)提取总RNA,使用反转录试剂盒- primescript™RT reagent kit with gDNA Eraser (TaKaRa生物科技有限公司,北京,中国)制备第一链cDNA。然后以cDNA为模板,对石榴不同发育阶段SPL家族基因进行基因克隆和表达分析。根据DNA和CDS序列,特异性定量引物(表3.)使用Oligo 7软件设计[72].
每50 μL反应液中含有25 μL 2× Taq Plus Master Mix,上下行引物各1 μL, cDNA模板2 μL和21 μL Nuclease-free ddH2O。PCR程序设置如下:95℃预变性3 min;95°C持续15 s, 58°C持续45 s, 72°C持续1 min,共35个循环;72°C 5分钟。首先,PCR产物经1%琼脂糖凝胶电泳分离,凝胶切割回收目标片段;将重组质粒与过表达载体连接,转化为有表达能力的载体大肠杆菌DH5α。然后筛选出PCR检测阳性的单克隆,送到上海生物工程公司进行测序。最后,利用ExPaSy-Translate在线工具将测序得到的序列翻译成氨基酸序列(https://web.expasy.org/translate/) [62].
亚细胞定位分析
确认…的位置PgSPL5而且PgSPL13将无终止密码子的全长编码序列(CDS)克隆到pBI121上,c端与绿色荧光蛋白(GFP)融合。对目的基因两端的酶切位点设计引物(表3.).从重组结扎凝胶中回收产物,通过冻融法将结扎得到的重组质粒导入农杆菌GV3101中。向量GV3101 (农),含有35S::GFP-PgSPL和控制病媒已渗入烟草(烟草benthamiana树叶)。36 ~ 48 h后,使用LSM 710共聚焦激光扫描显微镜(Zeiss, Jena, Germany)捕获荧光图像[72,73].
实时荧光定量PCR分析(qRT-PCR)
的表达式PgSPLs采用qRT-PCR方法分析不同组织中PgSPL5而且PgSPL13在不同生长调节剂下进行了分析。特异性定量引物(表3.)和石榴PgActin基因被用作内部参考。每20 μL反应液中含有10 μL TB Green Premix Ex Taq、0.4 μL ROX参比染料II、0.4 μL上下行引物、2 μL cDNA模板和6.8 μL ddH2O。qRT-PCR采用7500快速实时PCR系统(Applied Biosystems, CA, USA),每个cDNA样本有3个生物重复和3个技术重复。热循环器设置如下:95°C预变性30 s, 95°C预变性5 s, 60°C预变性34 s,循环40次;解离阶段设置为:95°C 15 s, 60°C 60s, 95°C 15 s。2 .对数据进行定量分析——ΔΔCt方法(74].
数据分析
所有qRT-PCR数据均采用单因素方差分析,并采用土耳其试验(p< 0.05)。芝加哥,IL,美国)基于三个完全随机块的值。
数据和材料的可用性
石榴全基因组数据下载自NCBI数据库(https://www.ncbi.nlm.nih.gov/search/all/?term=ASM220158v1),登录号为ASM220158v1。转录组数据来自NCBI (https://www.ncbi.nlm.nih.gov/Traces/study/),注册编号为SRP103147和SRP100581。的SPL蛋白序列答:芥,大肠茅,m .释放有而且葡萄均可从PlantTFDB数据库(http://planttfdb.gao-lab.org/index.php).所有数据库的公开访问都是开放的。支持本文结论的数据集包含在本文(及其附加文件).
缩写
- SPL:
-
SQUAMOSA启动子结合蛋白样
- 答:芥:
-
拟南芥
- 大肠茅:
-
桉树茅
- IBA:
-
Indole-3-butyric酸
- 6-BA:
-
6-Benzylaminopurine
- PP333:
-
摘要
- RNA:
-
核糖核酸
- 互补脱氧核糖核酸:
-
互补脱氧核糖核酸
- 存在:
-
实时定量聚合酶链反应
参考文献
- 1.
赵艳,赵红,王艳,张霞,赵霞,袁震。水稻mikc型MADS-box基因家族的全基因组鉴定及表达分析石榴农学通报,2020;10(8):1197。
- 2.
王颖,赵颖,闫敏,赵红,张霞,袁哲。石榴TALE基因家族的全基因组鉴定与表达分析(石榴l .)。农学。2020;10(6):829。
- 3.
赵颖,刘超,葛东,闫敏,任勇,黄霞,等。花发育过程中YABBY基因家族的全基因组鉴定与表达石榴L. Gene, 2020;752:144784。https://doi.org/10.1016/j.gene.2020.144784.
- 4.
朱涛,刘勇,马琳,王旭,张东,韩勇,等。小麦SPL基因家族的全基因组鉴定、系统发育及表达分析。植物学报。2020;20(1):420。https://doi.org/10.1186/s12870-020-02576-0.
- 5.
普雷斯顿JC,希勒曼LC。植物鳞状突起启动子结合蛋白样(SPL)基因家族的功能进化植物科学进展(英文版);
- 6.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP。植物microRNA靶点预测。细胞。2002;110(4):513 - 20。https://doi.org/10.1016/s0092 - 8674 (02) 00863 - 2.
- 7.
山崎K,木川T,井上M,馆野M,山崎T,矢步T,等。dna结合域的溶液结构揭示了一种新的锌结合基序拟南芥sbp家族转录因子。中华分子生物学杂志,2004;37(1):49-63。https://doi.org/10.1016/j.jmb.2004.01.015.
- 8.
Birkenbihl RP, Jach G, Saedler H, Huijser P.植物特异性sbp结构域的功能解剖:DNA结合和核定位结构域的重叠。中华分子生物学杂志,2005;32(3):585-96。https://doi.org/10.1016/j.jmb.2005.07.013.
- 9.
李志强,李志强,李志强,等。一种新的DNA结合蛋白家族包括假设的转录调控因子金鱼草majus花分生组织鉴定基因。生物信息学杂志,1996;25(1):7-16。
- 10.
Cardon G, Höhmann S, Klein J, Nettesheim K, Saedler H, Huijser p拟南芥SBP-box基因。基因。1999;237(1):91 - 104。https://doi.org/10.1016/s0378 - 1119 (99) 00308 - x.
- 11.
李超,陆松。植物SPL基因家族的分子特征杨树trichocarpa.中国生物医学工程学报。2014;14(1):131。https://doi.org/10.1186/1471-2229-14-131.
- 12.
Salinas M,邢星,Höhmann S, Berndtgen R, Huijser P.番茄SBP-box转录因子家族的基因组组织、系统发育比较及差异表达。足底。2012;235(6):1171 - 84。https://doi.org/10.1007/s00425-011-1565-y.
- 13.
谢凯,吴超,熊玲。水稻SQUAMOSA启动子结合样转录因子与microRNA156的基因组组织、差异表达及相互作用。植物科学进展。2006;42(1):366 - 366。https://doi.org/10.1104/pp.106.084475.
- 14.
李娟,侯辉,李霞,向杰,殷霞,高红,等。苹果SBP-box家族基因的全基因组鉴定与分析(马吕斯×有明显Borkh)。植物化学学报,2013;https://doi.org/10.1016/j.plaphy.2013.05.021.
- 15.
侯华,李静,高敏,辛格思,王华,毛林,等。葡萄SBP-box家族基因的基因组组织、系统发育比较及差异表达。公共科学学报,2013;8(3):e59358。https://doi.org/10.1371/journal.pone.0059358.
- 16.
Bartel DP。MicroRNAs:基因组学,生物发生,机制和功能。细胞。2004;116(2):281 - 97。https://doi.org/10.1016/s0092 - 8674 (04) 00045 - 5.
- 17.
谢峰,王强,孙荣荣,张波。小RNA测序鉴定miRNA在胚珠和纤维发育中的作用。植物学报,2015;13(3):355-69。https://doi.org/10.1111/pbi.12296.
- 18.
李志强,李志强,李志强,等。组织特异性已知和新mirna的高分辨率实验和计算分析拟南芥.基因组学报,2012;22(1):163-76。https://doi.org/10.1101/gr.123547.111.
- 19.
王杰。mir156介导的年龄通路对开花时间的调控。中国机械工程学报,2014;26(3):344 - 344。https://doi.org/10.1093/jxb/eru246.
- 20.
陈霞。microRNA作为肿瘤的翻译抑制因子APETALA2在拟南芥花的发育。科学。2004;303(5666):2022 - 5。https://doi.org/10.1126/science.1088060.
- 21.
Navarro L, Dunoyer P, Jay F, Arnold B, Dharmasiri N, Estelle M,等。植物miRNA通过抑制生长素信号通路促进抗菌性。科学。2006;312(5772):436 - 9。https://doi.org/10.1126/science.1126088.
- 22.
杨晓燕,朱晓燕。植物中新型微rna和小rna的研究进展拟南芥.植物学报。2004;16(8):2001-19。https://doi.org/10.1105/tpc.104.022830.
- 23.
王震,薛伟,董超,金林,边松,王超,等。一项比较miRNAome分析揭示了7个与纤维起始相关的和36个新的mirna。植物学报,2012;5(4):889-900。https://doi.org/10.1093/mp/ssr094.
- 24.
王勇,刘伟,王旭,杨锐,吴震,王宏,等。MiR156通过SPL靶标和其他microrna调控杨树花青素的生物合成。园艺研究2020;7(1):6786-7。
- 25.
叶波,张凯,王娟。miR156在植物再生中的作用拟南芥.中国生物工程学报,2015;29(05):366 - 366。https://doi.org/10.1111/jipb.12855.
- 26.
唐国强,张志刚,张志刚,等。微rna在植物开花过程中的作用。植物学报。2015;8(3):359-77。https://doi.org/10.1016/j.molp.2014.12.018.
- 27.
穆努沙米P,佐洛塔洛夫Y,梅特尼耶LV,莫菲特P, Strömvik MV。植物翻译体非翻译区胁迫相关序列和microRNA靶点的新计算鉴定。科学通报2017;7(1):43861。
- 28.
吴刚,梅杨萍,苏丽娟,王娟,Detlef W, Poethig RS. miR156和miR172的序列作用调控水稻生长发育时间拟南芥.细胞。2009;138(4):750 - 9。https://doi.org/10.1016/j.cell.2009.06.031.
- 29.
于娜,蔡伟,王珊珊,单春,王丽,陈霞。microrna156靶向SPL基因对毛状体分布的时间调控拟南芥.植物学报。2010;22(7):2322-35。https://doi.org/10.1105/tpc.109.072579.
- 30.
邢星,Salinas M, Hohmann S, Berndtgen R, Huijser P. mir156靶向和非靶向SBP-Box转录因子协同作用,确保男性生育能力拟南芥.《植物科学》2010;22:3935-50。
- 31.
张志刚,张志刚,张志刚,张志刚,等。阿拉伯高山多年生开花植物对冬季温度的年龄依赖响应机制。科学。2013;340(6136):1094 - 7。https://doi.org/10.1126/science.1234116.
- 32.
王俊伟,王晓燕,王晓燕,等。mir156调控的SPL转录因子在植物开花过程中的作用拟南芥.细胞。2009;138(4):738 - 49。https://doi.org/10.1016/j.cell.2009.06.014.
- 33.
王志生,王勇,王志强,王志强,王志强,韩丽芳。鳞翅果启动子结合蛋白like 2通过激活不对称叶片2调控花器官发育和植株育性拟南芥.植物分子生物学,2016;9(7):1-14。
- 34.
金俊杰,李俊华,金伟,郑海生,惠泽平,安俊华。miR156-SPL3模块通过FT调控环境温度响应开花拟南芥.中国生物医学工程学报,2012;29(1):466 - 466。https://doi.org/10.1104/pp.111.192369.
- 35.
郑俊华,徐普杰,姜斯克,朴CM。miR172信号被纳入miR156信号通路SPL3/4/5基因在拟南芥发展转变。植物分子生物学杂志,2011;36(1):35-45。https://doi.org/10.1007/s11103-011-9759-z.
- 36.
Unte US, Sorensen AM, Pesaresi P。SPL8是影响花粉囊发育的SBP-box基因拟南芥.植物学报。2003;15(4):1009-19。https://doi.org/10.1105/tpc.010678.
- 37.
李志刚,李志刚,李志刚,李志刚,李志刚SPL15与SOC1合作,整合内源开花途径拟南芥分生组织。生物工程学报。2016;37(3):254-66。https://doi.org/10.1016/j.devcel.2016.04.001.
- 38.
邢世峰,李志刚,李志刚。SPL8mir156靶向的SPL基因冗余调控拟南芥雌蕊分化模式。植物学报,2013;26(4):366 - 366。https://doi.org/10.1111/tpj.12221.
- 39.
张勇,施华泽,张志强,张志强。SPL8在赤霉素介导的发育过程的一个子集中的局部调节因子拟南芥.植物分子生物学杂志,2007;39(3):429-39。https://doi.org/10.1007/s11103-006-9099-6.
- 40.
袁震,方勇,张涛,费震,韩峰,刘超,等。石榴(石榴L.)基因组为果实质量和胚珠发育生物学提供了见解。中国生物工程学报,2018;16(7):1363-74。https://doi.org/10.1111/pbi.12875.
- 41.
杨林,吴国强,吴国强,杨林。微rna调控SBP-box转录因子的研究进展SPL3是leaf, FRUITFULL和APETALA1的直接转录激活因子。生物工程学报。2009;17(2):268-78。https://doi.org/10.1016/j.devcel.2009.06.007.
- 42.
郭安,朱强,顾霞,葛松,杨娟,罗娟。植物特异性SBP-box转录因子家族的全基因组鉴定与进化分析。基因。2008;418(1):1 - 8。https://doi.org/10.1016/j.gene.2008.03.016.
- 43.
熊娟,郑东,朱辉,陈娟,那瑞,程卓。林地草莓SPL基因家族的全基因组鉴定与表达分析草莓属vesca.基因。2018;61(9):675 - 83。
- 44.
牛勇,杨娟,尹刚,李锐,邹伟。参与年龄发育的SPL基因家族的全基因组特征麻风树.BMC基因组学杂志,2020;21(2):585-96。
- 45.
沈华,周凯。Cell-PLoc 2.0:一种改进的网络服务器包,用于预测各种生物中蛋白质的亚细胞定位。自然科学学报,2010;2(10):1090-103。
- 46.
汤姆逊B,韦默F.花发育的分子调控。Curr Top Dev biology 2019; 131:185-210。https://doi.org/10.1016/bs.ctdb.2018.11.007.
- 47.
张东,韩忠,李娟,秦华,周林,王勇,等。茶叶SBP-box基因家族转录因子的全基因组分析及其对非生物胁迫的响应(茶树).基因组学,2020;112(3):2194 - 202。https://doi.org/10.1016/j.ygeno.2019.12.015.
- 48.
李志刚,刘志刚,刘志刚。鳞状突起启动子结合蛋白(Sbp)转录因子家族的全基因组鉴定和比较分析Gossypium raimondii就而且拟南芥.中国科学(d辑),2017;
- 49.
张松,凌玲。通过miR156/529结合位点的改变研究陆地植物SQUAMOSA启动子结合蛋白样基因的多样性。植物学报。2018;14:55-63。https://doi.org/10.1016/j.plgene.2018.04.007.
- 50.
Schwarz S, Grande AV, Bujdoso N, Saedler H, Huijser P. microRNA调控SBP-box基因SPL9而且SPL15控制嫩枝成熟拟南芥.植物化学学报,2008;29(2):344 - 344。https://doi.org/10.1007/s11103-008-9310-z.
- 51.
蔡勇,陆霞,朱磊。粉红石榴花芽分化的初步研究。园艺学报。1993;20(1):23-6。
- 52.
Wetzstein HY, Ravid N, Wilkins E, Adriana PM。石榴两性花型和雄花型的形态学和组织学特征。地球科学进展,2011;26(2):344 - 344。https://doi.org/10.21273/JASHS.136.2.83.
- 53.
陈丽兰,张军,李海霞,牛军,薛辉,刘宝斌,等。转录组分析揭示石榴花雌性不育的候选基因。植物科学,2017;8:1430。https://doi.org/10.3389/fpls.2017.01430.
- 54.
陈琳。石榴调控基因的探索(石榴)基于转录组分析的雌性不育。北京:中国农业科学院;2017.
- 55.
杨林,吴国强,吴国强,杨林。microrna调控SBP-box转录因子的研究进展SPL3是leaf, FRUITFULL和APETALA1的直接上游激活剂。开发细胞。2009;17(2009):268-78。https://doi.org/10.1016/j.devcel.2009.06.007.
- 56.
郑俊华,朱勇,徐普杰。SOC1-SPL模块集成光周期和赤霉素信号来控制开花时间拟南芥.植物J. 2012; 9:577-88。
- 57.
Chuck G, Whipple C, Jackson D, Hake S.玉米穗鞘编码的SBP-box转录因子调控苞片发育和分生组织边界的建立。发展。2010;137(8):1243 - 50。https://doi.org/10.1242/dev.048348.
- 58.
普雷斯顿JC,希勒曼LC。SQUAMOSA-PROMOTE RBINDING PROTEIN 1启动开花金鱼草majus通过激活分生组织身份基因。植物学报,2010;36(4):344 - 344。https://doi.org/10.1111/j.1365-313X.2010.04184.x.
- 59.
金J,田F,杨D,孟Y,香港L,罗JC, et al。PlantTFDB 4.0:朝着植物转录因子和调控相互作用的中心枢纽。核酸研究,2017;45(D1): D1040-5。https://doi.org/10.1093/nar/gkw982.
- 60.
马科勒-鲍尔A,波杨,韩林,何杰,兰奇奇CJ,陆松,等。CDD/SPARCLE:基于亚家族结构域的蛋白质功能分类。核酸学报2017;45(D1): D200-3。https://doi.org/10.1093/nar/gkw1129.
- 61.
Letunic I.博克同行。20年SMART蛋白域注释资源。核酸学报2018;46(D1): D493-6。https://doi.org/10.1093/nar/gkx922.
- 62.
李松,徐旭,陈旭,陈旭,李宏,林勇,等。CK2基因的克隆及表达特征分析花芽龙眼不悦之色。果树学报,2019;36(12):1638-47。
- 63.
Kumar S, Stecher G, Tamura K. MEGA7:更大数据集的分子进化遗传学分析版本7.0。分子生物学杂志,2016;33(7):1870-4。https://doi.org/10.1093/molbev/msw054.
- 64.
Balakrishnan S, Gao S, Lercher MJ, Hu S, Chen W. Evolview v3:一个用于系统发生树可视化、注释和管理的web服务器。核酸研究。2019;47:W270-5。
- 65.
Bailey TL, Johnson J, Grant CE, Noble-William S. MEME套件。核酸工程学报,2015;43(W1): W39-49。https://doi.org/10.1093/nar/gkv416.
- 66.
郭安,朱强,陈霞,罗娟。GSDS:基因结构显示系统。继承。2007;08:1023-6。
- 67.
Magali L, Patrice D, Gert T, Kathleen M, Yves, Yves VP等。PlantCARE,一个植物顺式作用调控元件的数据库和启动子序列硅片分析工具的门户。核酸Res. 2020; 30:325-7。
- 68.
戴霞,庄志,赵平。psRNATarget:植物小RNA靶标分析服务器(2017年发布)。核酸研究,2018;46(W1): W49-54。https://doi.org/10.1093/nar/gky316.
- 69.
陈松,周勇,陈勇,顾杰。fastp:一种超快一体化FASTQ预处理器。生物信息学。2018;34 (17):i884 - 90。https://doi.org/10.1093/bioinformatics/bty560.
- 70.
陈L,妞妞W,刘B, D Jing,罗X,李G, et al。叶面施用不同植物生长调节剂对石榴坐果率和品质的影响。中国植物科学,2020;37(02):244-53。
- 71.
陈玲,李华,牛娟。突尼斯软籽石榴可育花与败育花生长特征及形态差异研究。果树学报。2017;34:80-7。
- 72.
孙文祥,史敏,薛波,夏勇,郭强,王松,等。克隆、亚细胞定位及表达分析EjSPL5三倍体枇杷中的基因。园艺学报,2020;47(02):220-32。
- 73.
祁涛,高敏,杨艳,李敏,马峰,马波MdPH1与酸性有关马吕斯有明显Borkh。植物科学2019;37(06):767-74。
- 74.
Livak KJ, Schmittgen TD。相关基因表达数据的实时定量PCR分析和2——ΔΔCT方法。方法。2001;25(4):402 - 8。https://doi.org/10.1006/meth.2001.1262.
确认
不适用。
资金
南京林业大学人才计划项目(GXL2014070, GXL2018032)、南京林业大学博士奖学金基金、江苏省高校学术发展重点项目(PAPD)、国家自然科学基金(31901341)、江苏省自然科学基金(BK20180768)资助。这些资助机构参与了研究的设计、数据的收集、分析和解释,以及手稿的撰写,以及开放获取付费。
作者信息
从属关系
贡献
BL和YZ参与实验设计,并对系统发育分析进行分析和解释;BL和SW对植物进行表达水平检测;BL、YS、YW提供试剂和分析工具;BL是实验、分析数据和撰写手稿的主要贡献者;YZ, XZ, ZY对稿件进行了修改。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
李,B,赵,Y,王,S。et al。SPL基因家族的全基因组鉴定、基因克隆、亚细胞定位及表达分析p . granatumlBMC植物生物学21日,400(2021)。https://doi.org/10.1186/s12870-021-03171-7
收到了:
接受:
发表:
关键字
- 石榴
- SPL基因家族
- 基因克隆
- 亚细胞定位
- 基因表达