摘要
过去十年的研究表明,单一的参考基因组不能代表一个物种的多样性。MaizeGDB引入了一种泛基因组方法来存储基因组数据,利用大量不同的玉米基因组及其相关数据集,快速有效地跨基因组连接基因组、基因模型、表达、表观基因组、序列变异、结构变异、转座元素和多样性数据,使研究人员可以轻松地跟踪玉米基因座及其同源物的结构和功能差异。我们相信我们的框架是独一无二的,为任何准备托管大规模泛基因组数据的基因组数据库提供了模板。
背景
Zea may ssp。Mays(玉米,玉米)是一种独特的模式生物,因为它作为食物、饲料和纤维产品的广泛重要性,促使土著居民的传统育种做法对其进行了数千年的驯化[1,2,3.],然后是自绿色革命和分子时代以来数十年的定向育种[4,5].在过去的100年里,对玉米的研究也有助于理解植物的生物学、进化、驯化、发育和遗传学[6,7,8,9,10].十多年来,玉米一直是世界上产量最高的粮食作物(http://faostat.fao.org/),主要用于牲畜饲料,最近用于生物燃料[11].
玉米研究数据的收集和共享,如编译基因列表、复合遗传图谱和育种信息,自20世纪初以来一直在进行[9,12].策展玉米研究数据于1991年正式移入数据库(MaizeDB) [13].数据库于2003年扩展至包括序列数据[14]和基因组数据,到2015年已经演变成现在的玉米遗传和基因组数据库(MaizeGDB -)https://www.maizegdb.org)[15].MaizeGDB是玉米社区数据库,为玉米科学家提供数据管理和信息学资源,以支持玉米遗传学、基因组学和育种研究。MaizeGDB也是玉米研究界的中心,为促进合作和数据共享提供支持、推广和培训,并充当玉米遗传和基因组命名的交换中心。
到2019年,MaizeGDB拥有6个玉米自交系和1个大刍草的基因组[16].从那时起,MaizeGDB已经引入了39个额外的参考质量基因组,包括重要的自交系(PH207 [17], Mo17 [18,19]、W22 [20.),一组欧洲线[21,一种甜玉米[22],以及嵌套关联映射(NAM)居群起始系的26个高质量PacBio基因组组合[23].NAM创始人系代表了玉米的大量多样性[24],由此产生的NAM群体已被研究人员广泛用于研究玉米开花时间[25,叶子结构[26,抗病能力[27,以及其他重要的农艺性状[28].NAM创始人基因组的测序、组装、基因模型注释、RNA-seq表达数据、结构变异、转座元件注释和甲基组数据均在相同的实验室中使用相同的协议进行。由于很难从组装和注释质量或技术的差异中梳理出真正的生物学差异,阻碍了基因组组装之间的比较,NAM创始人基因组组装及其相关数据提供了一个独特的机会,以探索单个物种内与生物学相关的基因组多样性。在托管NAM创始人基因组及其数据时,MaizeGDB在单个数据库中更新了26个新的基因组项目/元数据页面;超过100万新的基因模型页面;数百个新的可下载数据集;134个额外的爆炸目标;以及26个新的JBrowse基因组浏览器,拥有超过1000个跨浏览器的总数据轨迹。我们使用这个庞大的黄金标准数据集来开发新的方法,以一种对玉米群落有用和有生物学意义的方式,托管和连接这些基因组及其数据集。
过去十年的研究已经清楚地表明,单一的参考基因组并不能真正代表物种的多样性(见[29])。单个人类基因组的差异可达10% [30.],而在玉米中,只有60%的基因在所有的NAM系中被发现[23].现在,许多具有复杂基因组的品种可以作为群体进行测序和分析,泛基因组数据集变得更加可用(例如在水稻[31,32和番茄[33])。这些泛基因组集对于理解疾病和抗旱性等表型的多样性很有价值。然而,科学用户可能不精通命令行,如何有效地进行泛基因组显示和交互一直是我们试图纠正的一个持续挑战。
在这里,MaizeGDB引入了一种泛基因组方法来管理基因组数据库,利用大量不同的玉米基因组及其相关数据集,快速有效地跨基因组连接基因组、基因模型、表达、甲基组、序列变异、结构变异、转座元素和多样性数据,以便研究人员可以轻松地跟踪玉米基因座及其同源同源物的结构和功能差异。MaizeGDB提供了为每个宿主基因组提供三个视角的工具和资源:(1)基因组可以与相关的基因组特异性数据独立使用;(2)基因组与“代表性的”B73参考基因组和大量伴随数据相关联;(3)基因组在泛基因组框架中呈现,基因注释和序列变异在不同基因组组合之间相互关联。我们相信我们的跨基因组、泛基因组框架在数据库中是独一无二的,但它可以成为任何准备托管大规模泛基因组数据的基因组数据库的模板。
结构与内容
MaizeGDB中的数据被细分为不同的类别,如[16].简单地说,MaizeGDB是根据数据或工具类型(例如,基因组、SNP多样性、BLAST)组织的。玉米基因组列在基因组页面上,有元数据和下载链接;每个注释的基因模型都有自己的基因模型页面。在我们的数据库中,MaizeGDB目前有48个玉米基因组,包括25个NAM创始人系和参考玉米基因组B73的最新版本(版本5)(Zm-B73-REFERENCE-NAM_5.0,在本文中称为B73v5)。下面我们将描述我们如何重新格式化MaizeGDB,使其成为玉米的泛基因组资源,以及这种新格式如何帮助用户将基因组数据连接到功能数据。
实用与讨论
按计划对基因组组合进行分组
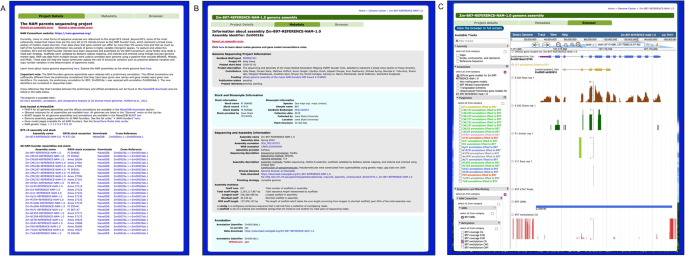
为了方便访问25个NAM创始人基因组,我们重新组织了用户界面,使所有NAM创始人行都可以在一个页面上找到(https://maizegdb.org/NAM_project),再细分为三个选项卡(图。1).“项目详情”标签(图。1A)列出了所有NAM创始人基因组,以及他们的库存链接和访问这些基因组相关数据的下载页面,并包含了NAM项目的描述,与所有NAM线相关。这个标签上的数据对这个基因组集中的所有25个基因组都是通用的。“元数据”选项卡(图。1B)描述了特定于特定基因组的测序、组装、注释和其他数据(在本例中为B97)。第三个标签,“浏览器”,把用户带到该基因组的浏览器实例(图。1C)。
为NAM创始人项目组织的NAM基因组页面(https://maizegdb.org/NAM_project).一个“项目详情”页面描述了NAM基因组项目并列出了与该项目相关的基因组。B基因组B97(正式形式为Zm-B97-REFERENCE-NAM-1.0)的“元数据”选项卡。B基因组B97的“浏览器”选项卡
泛基因组JBrowse
泛基因组可视化的挑战之一是查看和导航多个基因组,独立于参考基因组。通常,泛基因组可视化工具(如IGV或JBrowse)依赖于将其他基因组的数据对准参考基因组,然后从这些对准数据的参考基因组坐标生成轨迹。该技术的一个缺点是很难表示在其他基因组中存在而在参考基因组中不存在的数据。然而,独立于参考基因组坐标系的泛基因组可视化方案,如带有节点和边的图形可视化,用户可能很难解释[34].我们实现的一个解决方案是跨JBrowse实例连接两个或多个基因组之间共享的基因模型、snp或标记。直到2020年,MaizeGDB使用GBrowse(2002年推出的服务器端浏览器软件包)提出基因组组装[35].2020年,MaizeGDB升级到更快、更现代的客户端JBrowse浏览器软件[36].NAM创始人基因组和B73v5现在在JBrowse上。
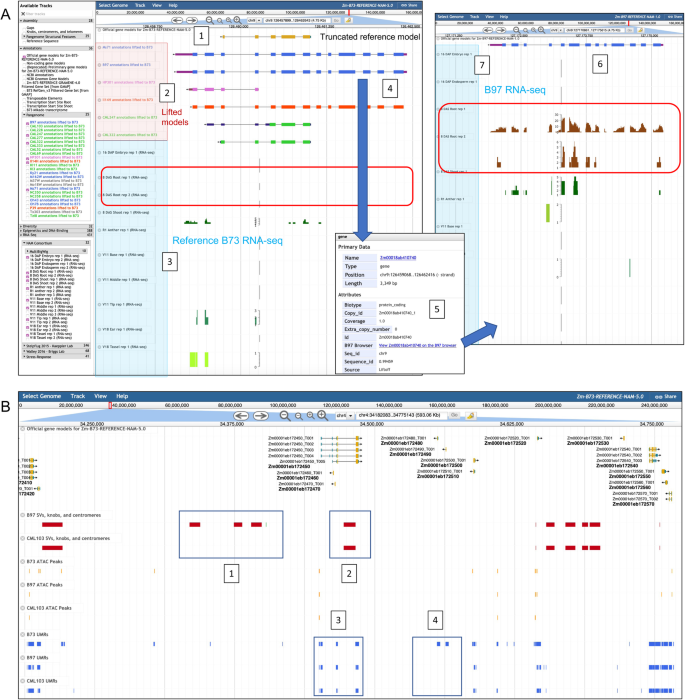
在无花果。2,我们通过连接跨NAM创始人系和B73v5的基因模型来演示我们的JBrowse跨基因组功能。使用注释升降工具Liftoff [37],我们将每个NAM和B73v5基因模型注释集相互提升(总共有676个交叉提升的注释集),并根据结果生成JBrowse轨迹。与其他注释提升工具不同,Liftoff允许跨不同基因组访问提升基因模型注释,因为它考虑到了结构差异,如组装之间的倒置。在任何NAM或B73v5 JBrowse实例中,用户可以选择从任何其他基因组中提取的基因模型注释的轨迹,以确定当前浏览器上的注释是否存在于其他基因组中。如果是这样,用户可以单击被移除的基因模型注释功能,这将打开一个弹出窗口,其中包含一个链接,该链接将把用户带到位于JBrowse基因组实例中对应于被移除注释轨迹的同一基因模型(图1)。2A).这些liftoff生成的轨迹也有助于识别在其他基因组中注释的区域,这些区域可能在目标基因组中被遗漏或截断。一个缺失注释的区域,如果加上其他功能数据,如RNA-seq表达和低甲基化,可以表明该区域可能是功能性的,如果该区域碰巧有一个突变插入或一个用户感兴趣的SNP,这是重要的。图中的例子。2展示了在参考B73v5基因组中被截断的非硬柄温带系B97内的注释。比较两个基因组之间的RNA-seq可以发现,B97在根位点上的注释具有功能支持,而在相应的B73位点上的完整注释则缺乏功能支持。在这个新的跨浏览器功能之前,用户需要推断B73和B97基因模型之间的同步关系,然后分别在各自的浏览器页面上搜索这些基因模型,以便比较这些位点之间的表达差异。
MaizeGDB JBrowse泛基因组优化。一个跨NAM创始人和B73v5的交叉参照基因模型注释。左边的面板是B73v5 JBrowse实例,显示了一个基因模型注释在参考基因组(1)中被截断,但在Ms71和B97的轨迹(2)中完整,这两个都是非硬柄温带系;Il14H,一个甜玉米系,有所有外显子,但没有utr。HP301(爆米花)、CML247和CML322(热带线)也被截断。(3)表明在B73中该位点RNA-seq表达很少,在根组织中无表达(红框)。如果用户点击B97基因模型Zm00018ab410740(4),会打开一个弹出框;点击链接(5),它将把用户带到B97浏览器中Zm00018ab410740的位置(6)。B97 Zm00018ab410740 RNA-seq(7)在根组织(红框)中表现出稳健的表达,支持B97基因模型注释,而与之相关联的B73位点相比,其RNA-seq支持较少,特别是在根中。Ms71位点表现出类似的根表达模式,但Il14H没有根表达(未显示)。BB97和热带系CML103相对于B73v5的大规模结构变异(红色=缺失,绿色=插入),B73v5、B97和CML103的未甲基化区域(UMRs,蓝色)和ATAC-seq峰值(橙色)。所有数据都与参考B73v5基因组进行比对。(1)只在B97中存在的相对于B73的删除(红色)(红色空格表示B73中B97中缺失的区域);这些区域在CML103中没有被删除,正如该基因组中没有红色区域所示。(2) B97和CML103相对于B73共享的缺失。(3)在B73、B97和CML103中存在相同位点的umr。(4) umr仅在B73中存在
这种跨基因组功能也适用于我们从[28],我们已将其映射到NAM的所有创始系和B73 (补充信息),并使用于组装NAM创始人基因组超支架的泛基因组标记成为假分子。通过这种方式,我们可以在所有的NAM基因组和参考基因组中链接标记和重要的表型性状。同样重要的是,我们可以识别在给定的NAM系中,某一特定表型性状的标记缺失的实例。
这种跨浏览器链接是研究人员快速比较两个或多个基因组的基因座之间的RNA-seq、基因模型结构、甲基组、转座元件注释、结构变异和性状标记信息,了解同源区域结构和功能差异的一种有效方法。
扩大玉米的表观遗传和结构关系
MaizeGDB扩大了参考基因组B73v5 JBrowse的轨道数量,包括一个表观遗传图谱[38],包含ChIP-seq(染色质免疫沉淀测序),ATAC-seq(转座酶可达染色质检测),甲基-seq,以及进一步增强参考基因组功能景观的dna结合位点。表观遗传数据可以指示染色质开放区域和功能基因空间,或者反过来可以识别可能被表观遗传沉默的区域。这些信息,连同RNA-seq数据,对于帮助研究人员确定感兴趣的位点是否可能具有功能至关重要。
MaizeGDB还包括NAM联盟生成的表观遗传数据[23包括DNA甲基化、UMRs(未甲基化区域)和映射到所有NAM创始人和B73v5的ATAC-seq数据。此外,NAM创始人数据被映射到B73v5本身,以便同时比较所有NAM创始人与参考基因组的表观遗传景观的差异(图1)。2B)。
结构变异(SV)数据,如旋节、着丝粒、大规模缺失和由NAM联盟生成的每个NAM创始人基因组的其他特征[23在NAM创建者和B73v5 JBrowse实例上显示为跟踪。与表观遗传数据相似,来自其他NAM创始人的SV数据也被NAM联盟预测到B73v5(图1)。2B),使研究人员能够比较所有NAM创始人与参考基因组之间的大规模结构变异的差异。
一个泛基因标签下的基因模型
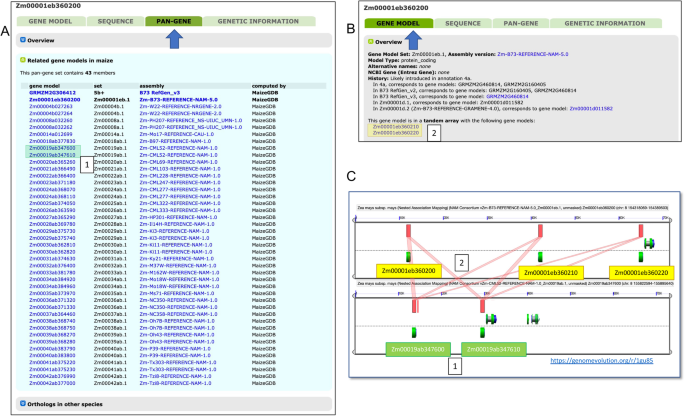
在所有基因组中,注释基因组中的每个基因模型在MaizeGDB上都有一个页面,其中描述了基因组位置、遗传图谱位置、基因模型结构、转录本和蛋白质序列、功能、表达值、突变信息和其他数据(如可用)(图1)。3.).在我们迁移到泛基因组方法之前,这些信息被细分为三个选项卡:一个选项卡用于基因模型和表达信息,一个选项卡用于基因模型的序列信息,还有一个选项卡用于与基因模型相关的遗传信息。随着泛基因组方法的实施,我们添加了一个新的标签,包括一个给定的基因模型和其他玉米基因组中的共tenic基因模型之间的泛基因关系。我们对泛基因的定义是在玉米中包含两个或两个以上基因组的所有同源基因座。MaizeGDB泛基因(图。3.A)通过blastn将所有基因组的主CDS转录本与所有其他基因组的主CDS转录本对齐而生成[39],其次是DagChainer [40],然后使用马尔可夫聚类算法(MCL [41),方法)。泛基因选项卡显示与感兴趣的基因模型相关的泛基因集的所有成员。
基因模型页面上的泛基因组和串联阵列信息。特色是参考B73v5基因模型Zm00001eb360200的信息。一个Zm00001eb360200的泛基因标签(箭头)和大多数由MaizeGDB承载的玉米系的所有同向同源基因。提供了同向同源基因(“基因模型”)的基因模型信息和基因组的名称(“集合”)。可折叠的“概述”(上)和“其他物种的同源性”(下)分别提供了更多关于泛基因关系是如何派生的信息,以及草的同向关系。许多基因组有不止一个代表;例如,“Zm-CML52-REFERENCE-NAM-1.0”集合中有两个基因模型:Zm00019ab347600和Zm00019ab347610;这些是串联复制。B基因模型选项卡(箭头)的部分屏幕截图,其中包含关于基因模型的信息,这些基因模型是参考基因模型Zm00001eb360200 (2) (Zm00001eb360210和Zm00001eb360220)的串行副本,共包含三个基因副本,包括参考基因模型。CA CoGe (https://genomevolution.org/)中B73和CML52的串联阵列的可视化对齐输出一个),B).绿色的是基因模型;红框是两个基因组之间的编码区域的原始排列。此映像支持B73和CML52中串联数组副本的MaizeGDB管道确定
我们还通过300 kb窗口内的自CDS转录片段(方法)识别串联基因阵列,并报告除了基因模型页面中所代表的基因之外,该串联阵列中所有其他的blast片段(图)。3.这提供了一个基因模型在玉米中的保留和它的局部拷贝数的信息。总的来说,这些泛基因标签上的特征允许用户了解玉米中任何给定基因模型与所有其他基因模型的拷贝数和共时关系。
比较玉米基因组与CViTjs的染色体水平结构变异
全基因组水平的基因组特征的宏观视图可以揭示近距离无法检测到的模式。MaizeGDB使用了CViTjs(染色体可视化工具-javascript;https://github.com/LegumeFederation/cvitjs)生成B73v5和NAM奠基人组装体的全基因组视图。CViTjs是一个灵活的Javascript应用程序,它在伪分子、链接组或染色体上使用指定的颜色或热色范围显示特征,分类为点或范围,显示为矩形、圆形或直方图。任何类型的特征都可以有一个附加的标签。CViTjs可用于显示遗传图谱、基因组特征或细胞学特征。CViTjs在B73v5和NAM创始人的全基因组视图上成像层基因和串联重复密度以及着丝粒和旋钮区域。另一种观点显示,在所有26个基因组的10条染色体上,每一条都有相同的特征。数字4显示了NAM始发系Oh7B的一个例子,它有一个从染色体10到染色体9的易位,在CViTjs中优雅地显示出来。
通过qTeller和NAM创始人的RNA-seq可视化
qTeller是一个程序,可视化的RNA表达在给定的基因,基因组坐标,或基因对跨多个RNA-seq数据集(稿件提交)。它允许用户直观地比较所选基因模型在不同组织、时间点和条件下的基因表达,或者比较两个基因模型之间的基因表达。它还接受一个基因模型列表,并输出一个文件,其中包含用户选择的每个组织/条件库的每个基因模型的表达丰度。
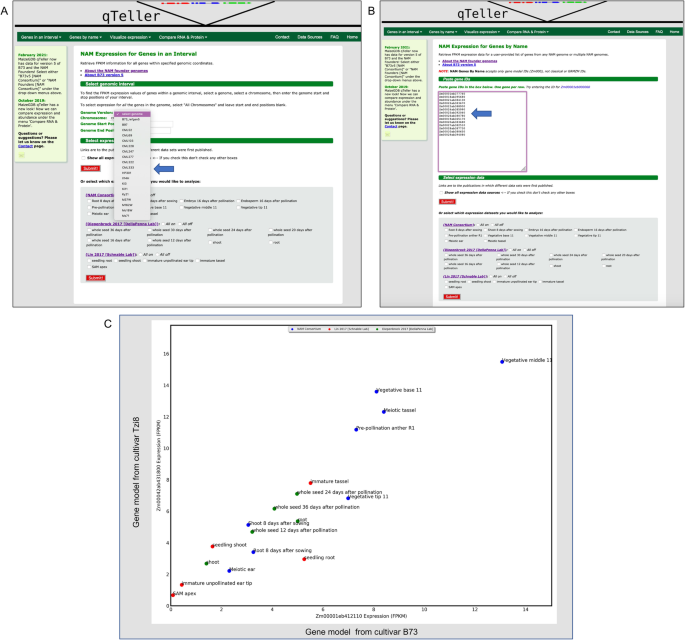
MaizeGDB自2018年以来一直主办qTeller (https://qteller.maizegdb.org/),并更新了该工具,包括一种比较蛋白质丰度和研究多个基因组的方法。最初,只有参考玉米基因组B73的第4版本的基因被表达出来。由于NAM创始人测序项目还对所有NAM创始人系的10个组织的RNA-seq数据进行了测序,我们能够制作一个NAM创始人泛基因组qTeller实例,用户可以将一个NAM基因组中的基因的RNA表达与另一个NAM基因组中的基因的RNA表达进行比较。基因组和RNA-seq数据都是由同一个测序组以完全相同的方式生成的,这使得基因组数据集之间的比较比在不同实验室和不同条件下生成的基因组之间的RNA-seq数据更平等。因此,这个qTeller实例允许用户轻松地比较玉米中共享基因之间的规范化表达谱。5).
MaizeGDB NAM多基因组qTeller实例。一个“间隔中的基因”功能,通过下拉菜单(箭头)选择感兴趣的NAM基因组。B“基因名称”特征,其中基因模型来自几个不同的基因组(由不同的前缀区分:Zm00001eb = B73v5, Zm00018ab = B97等;参见下面的“命名法”部分)可以输入以检索跨基因组的表达结果。CZm00001eb412110 (B73)和Zm00042ab431800 (Tzi8)两个不同基因组的同源基因模型的比较X和Y坐标表示每个基因模型各自的FPKM值。三种不同的RNA-seq表达数据集被表示(蓝色,绿色,红色)。两个基因模型之间的表达值是相当一致的,这通常是同步位点
多基因组的基因组命名法
MaizeGDB与玉米命名委员会合作,作为玉米命名的交流中心,包括基因组组装和注释。对于托管在MaizeGDB中的多个基因组,特别是NAM创建者程序集(需要名称将它们标识为集合的成员),有必要为基因组程序集、注释集和人类和机器都可读的基因模型建立一致的命名约定。这样做还有助于对所有NAM程序集和注释进行计算分析。指南可以在这里找到https://documents.maizegdb.org/nomenclature/maize_assembly_nomenclature_2016_update.pdf.
所有NAM正基程序集都使用模式命名:Zm-[品种]- reference -NAM-1.0,例如,Zm- b97 - reference -NAM-1.0。一个小的例外是B73组合,它是代表性玉米基因组的第5个版本,被命名为Zm-B73-REFERENCE-NAM-5.0。
所有NAM程序集也被分配了形式为Zm[ddddd][l]的编号标识符。对于NAM程序集,它们是Zm00001e (B73v5)和Zm00018a (B97v1)到Zm00042a (Tzi8v1)。这些标识符用作基因模型名称的前缀,再添加一个字母表示注释版本。由于初步注释先于正式注释发布,因此正式注释使用前缀Zm00018ab - Zm00042ab,其中“b”表示第二个注释。基因模型号在染色体上按顺序编号,每隔10。例如:Zm000018ab000100、Zm000018ab000110、Zm000018ab000120等等。染色体编号并不编码在名字中。
结论
基因组测序和组装的效率和质量不断提高,成本呈指数级下降,加快了基因组组装的速度和数量。基因组数据库不仅将储存更多的基因组,而且还将提供资源来整合和比较不同的基因组。MaizeGDB开发了多种方法,利用玉米基因组数量的增长来探索玉米的多样性和复杂性。使用高质量的NAM创始人基因组作为金标准,我们编译了可通过每个基因模型页面访问的泛基因集,使基因组浏览器之间的跳转成为可能,实现跨基因组结构变异比较,并扩展了跨多个基因组的RNA-seq分析。MaizeGDB的这些最新更新可以作为其他数据库管理任何物种的大规模泛基因组的模板。
方法
提升注释:使用Liftoff工具在基因组之间提升注释[37使用默认参数。
MaizeGDB泛基因组:泛基因组和串联复制关系的脚本可以在MaizeGDB GitHub存储库中找到https://github.com/Maize-Genetics-and-Genomics-Database/Pan-Genome.MaizeGDB泛基因组的生成是基于Steven Cannon在Soybase开发的泛基因组管道结构[42]及豆类资讯系统[43]),加上blastn的组合[39, DagChainer [40]和MCL [41].选择每个基因组的主转录本或规范转录本的CDS基因组fasta文件,并从这些文件中生成blast数据库。使用参数blastn -query ${q} -db ${s%运行成对的blastn对齐(包括自对齐)。*} - per_identity 95 - value 1e-10 -outfmt " 6 STD qlen slen qcovs "。输出与每个规范转录本的基因组坐标合并,并为DagChainer格式化。DagChainer参数为perl DagChainer / run_dag_chinner .pl -i ${sample} -D 1,000,000 -g 40,000 -A 5。MCL参数为MCL -I 1.2 -te 20——abc -o。
串联复制关系:来自上述的自-自爆裂比对被过滤,以便在300kb窗口内的非自基因模型被选中。然后使用命令MCL I 2.0 -te 20——abc -o在MCL中运行。
数据和材料的可用性
在这项工作中描述的所有数据都可以在https://maizegdb.org/.所描述的软件可以在https://github.com/Maize-Genetics-and-Genomics-Database而且https://github.com/LegumeFederation/cvitjs.
缩写
- MaizeDB:
-
玉米数据库
- MaizeGDB:
-
玉米遗传学和基因组学数据库
- 南:
-
嵌套关联映射
- 制程:
-
马尔可夫聚类算法
- ChIP-seq:
-
染色质免疫沉淀测序
- ATAC-seq:
-
转座酶可达染色质测定
- SV:
-
结构变体
- CViTjs:
-
染色体可视化工具
参考书目
- 1.
罗梅罗·纳瓦罗JA, Willcox M, Burgueño J, Romay C, swars K, Trachsel S,等。玉米地方品种花期适应性的等位基因多样性研究。Nat Genet。2017;49:476-80。
- 2.
Aguirre-Liguori JA, Gaut BS, Jaramillo-Correa JP, Tenaillon MI, Montes-Hernández S, García-Oliva F,等。在大刍草亚种(玉米(Zea mays parviglumis)和玉米(Zea mays mexicana)中,基因流的差异是由对温度和土壤磷浓度的局部适应所驱动的。Mol Ecol. 2019; 28:2814-30。
- 3.
Gonzalez-Segovia E, Pérez-Limon S, Cíntora-Martínez GC, Guerrero-Zavala A, Janzen GM, Hufford MB,等。大刍草向外渗透的特征。墨西哥高地玉米。PeerJ。2019; 7: e6815。
- 4.
Prasanna BM。全球玉米种质多样性:表征与利用。生物科学学报,2012;37:843-55。
- 5.
安道夫C, Beavis WD, Hufford M, Smith S, Suza WP, Wang K,等。玉米育种的技术进步:过去、现在和未来。《应用科学》2019;132:817-49。
- 6.
Strable J, Scanlon MJ。玉米(Zea mays):植物生物学基础和应用研究的模式生物。2009;2009:db.emo132。
- 7.
小吏GW。玉米- euchlanena杂交种杂交与染色体组合的关系。遗传学。1932;17:481 - 501。
- 8.
Stadler LJ。x射线在玉米中的遗传效应。美国国家科学研究院1928;14:69-75。
- 9.
早年玉米遗传学。1984; 18:1-29。
- 10.
博世M,迈耶CD,库克森A,唐尼森IS。通过玉米伸长和非伸长节间差异基因表达谱鉴定与草细胞壁生物发生有关的基因。J Exp机器人。J Exp Bot;2011年,62年。可以从:https://pubmed.ncbi.nlm.nih.gov/21402660/.[引自2021年3月26日]
- 11.
玉米和高粱:生物能源草的遗传资源。植物学报2008;13:415-20。
- 12.
玉米遗传学的起源。《科学通报》2001;2:898-905。
- 13.
功能基因组学的研究进展(英文)合成功能基因组学。2002;3:128-31。
- 14.
Lawrence CJ, Dong Q, Polacco ML, Seigfried TE, Brendel V. MaizeGDB,玉米遗传和基因组学社区数据库。核酸学报,2004;32:D393-7。
- 15.
哈珀L,加德纳J,安道夫C,劳伦斯CJ。玉米遗传学和基因组学数据库。方法Mol Biol. 2016; 1374:187-202。
- 16.
Portwood JL, Woodhouse MR, Cannon EK, Gardiner JM, Harper LC, Schaeffer ML等。MaizeGDB 2018:玉米多基因组遗传和基因组数据库。《牛津大学学报》2018;47:D1146-54。
- 17.
Hirsch CN, Hirsch CD, Brohammer AB, Bowman MJ, Soifer I, Barad O等。玉米优秀自交系PH207的初步组装,揭示了玉米基因组和转录组的多样性。植物细胞,2016;28:2700-14。
- 18.
杨宁,徐晓伟,王仁仁,彭文林,蔡磊,宋建明,等。墨西哥玉米亚种单倍型对现代玉米的贡献。Nat Commun. 2017;8:1874。
- 19.
孙松,周勇,陈杰,史杰,赵辉,赵辉,等。Mo17与其他玉米基因组之间存在广泛的种内基因顺序和基因结构变异。Nat Genet 2018; 50:1289-95。
- 20.
施普林格王晓燕,李晓燕,李晓燕,等。玉米W22基因组为功能基因组学和转座子生物学提供了基础。Nat Genet。2018;50:1282-8。
- 21.
Haberer G, Kamal N, Bauer E, Gundlach H, Fischer I, Seidel MA,等。欧洲玉米基因组强调重复和基因含量的种内变异。Nat Genet。2020;52:950-7。
- 22.
Hu Y, Colantonio V, Müller BSF, Leach KA, Nanni A, Finegan C,等。基因组组装和群体基因组分析为现代甜玉米的进化提供了新的视角。Nat Commun. 2021;12:1227。
- 23.
Hufford MB, Seetharam AS, Woodhouse MR, Chougule KM, Ou S,刘杰,等。26种玉米基因组的从头组装、注释和比较分析。bioRxiv。2021.可以从:http://biorxiv.org/lookup/doi/10.1101/2021.01.14.426684.
- 24.
于J, Holland JB, McMullen MD, Buckler ES。玉米嵌套关联映射的遗传设计与统计力。遗传学。2008;178:539-51。
- 25.
Buckler ES, Holland JB, Bradbury PJ, Acharya CB, Brown PJ, Browne C等。玉米花期的遗传结构。科学。2009;325:714-8。
- 26.
田芳,Bradbury PJ, Brown PJ, Hung H,孙强,Flint-Garcia S,等。玉米嵌套关联定位群体叶片结构的全基因组关联研究。Nat Genet, 2011; 43:159-62。
- 27.
波兰JA, Bradbury PJ, Buckler ES, Nelson RJ。玉米北叶枯病定量抗性的全基因组嵌套关联定位。中国科学(d辑:自然科学版)2011;
- 28.
张宁,张志强,张志强,张志强,张志强,张志强。许多性状的关联映射揭示了玉米的功能变异模式。PLoS Genet, 2014;10:e1004845。
- 29.
Bayer PE, Golicz AA, Scheben A, Batley J, Edwards D.植物泛基因组是新的参考。Nat Plants。2020;6:914-20。
- 30.
《人类基因组时代的泛基因组学》。Nat Rev Genet自然出版集团。2020;21:43 - 54。
- 31.
赵强,冯强,陆宏,李勇,王阿,田强,等。泛基因组分析突出了栽培稻和野生稻基因组变异的程度。Nat Genet自然出版集团。2018;50:278-84。
- 32.
周勇,Chebotarov D, Kudrna D, Llaca V, Lee S, Rajasekar S,等。代表亚洲水稻种群结构的白金标准泛基因组资源。科学数据,2020;7:113。
- 33.
高亮,刚达一,孙浩,马强,鲍凯,铁曼DM,等。番茄泛基因组揭示了调节水果味道的新基因和罕见的等位基因。Nat Genet自然出版集团2019;51:1044-51。
- 34.
Mikheenko A, Kolmogorov M.装配图浏览器:装配图的交互式可视化。生物信息学。2019;35:3476-8。
- 35.
斯坦因LD,孟加尔C,舒S,柯迪M,曼戈尼M, Day A,等。通用基因组浏览器:模型生物系统数据库的构建模块。Genome Res. 2002; 12:1599-610。
- 36.
Buels R, Yao E, Diesh CM, Hayes RD, Munoz-Torres M, Helt G,等。用于基因组可视化和分析的动态web平台。《基因组生物学》2016;17:66。
- 37.
[中文摘要].一种精确的基因注释绘图工具[互联网]。冷泉港实验室,2020年。2020.06.24.169680页。可以从:https://www.biorxiv.org/content/10.1101/2020.06.24.169680v1.abstract.[引自2021年3月26日]
- 38.
韦瑞茜,陆铮,季琳,Marand AP, Ethridge CL, Murphy NG,等。玉米基因组中广泛存在的远程顺式调控元件。自然植物学报。2019;5:1237-49。
- 39.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ。基本的本地对齐搜索工具。中华分子生物学杂志1990;215:403-10。
- 40.
Haas BJ, Delcher AL, Wortman JR, Salzberg SL. DAGchainer:一种挖掘分段基因组复制和共时性的工具。生物信息学。2004;20:3643-6。
- 41.
Enright AJ, Van Dongen S, Ouzounis CA.一种大规模检测蛋白质族的有效算法。核酸Res. 2002; 30:1575-84。
- 42.
Brown AV, Conners SI, Huang W, Wilkey AP, Grant D, Weeks NT,等。美国农业研究署大豆遗传和基因组数据库SoyBase的新数据和新十年。核酸条例2021;49:D1496-501。
- 43.
Dash S, Campbell JD, Cannon EKS, Cleary AM, Huang W, Kalberer SR,等。豆科植物信息系统(LegumeInfo.org):豆科植物联邦数据资源集的关键组成部分。核酸学报2016;44:D1181-8。
确认
一个也没有。
资金
这项研究得到了美国的支持。农业部,农业研究服务处,项目编号[5030-21000- 08-00 - d],通过爱荷华州艾姆斯的玉米昆虫和作物遗传学研究小组。本出版物中提及的商品名称或商业产品仅为提供特定信息的目的,并不意味着美国农业部的推荐或认可。美国农业部是一个机会平等的提供者和雇主。资助机构在研究的设计、收集、分析和解释数据以及撰写手稿方面没有发挥作用。
作者信息
从属关系
贡献
MRW撰写了手稿,设计了JBrowse跨基因组模式的框架,运行Liftoff为被提升的注释,绘制性状数据,为泛基因标签生成泛基因组,更新了qTeller工具,并在数据库重组计划中发挥了重要作用。EKC重组和重新格式化了数据库,特别是NAM基因组页和泛基因页,安装和实现了CViTjs工具,编写了CViTjs部分,贡献了命名部分,编辑了手稿,并在数据库重组计划中发挥了重要作用。JLP安装JBrowse实例并上传、组织和格式化所有数据和元数据。LCH编辑了JBrowse元数据,对JBrowse和数据库模式进行了修正和反馈,为背景部分、命名部分做出了贡献,并编辑了手稿。JMG编辑了JBrowse元数据,对JBrowse和数据库模式进行了更正和反馈,编辑了手稿,并为命名部分做出了贡献。MLS阅读并编辑了手稿,并提供了关于JBrowse和数据库模式的反馈。CMA是主要调查者,监督项目及其规划,帮助指导其设计,并编辑手稿。所有作者均已阅读并认可该手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者称没有利益竞争。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:补充图1。
NAM创始人的SNP数据映射到参考B73基因组上。在浏览器上表示的是来自非硬茎NAM线(蓝色)、爆米花线HP301(粉色)、甜玉米线(橙色)和热带线的snp的子集。snp是基于性状的颜色编码。1)甜玉米系IL14h和P39缺少snp。2)通过点击Il14h中相邻的SNP,弹出框会打开,其中链接将把用户带到Il14h浏览器上的SNP。这个实验可以在下面的链接复制https://jbrowse.maizegdb.org/?data=IL14H&loc=chr2%3A209060001..210188000&highlight=chr2%3A209624165..209624272&tracks=gwas_snps
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
伍德豪斯,医学博士,坎农,e.k.,波特伍德,J.L.et al。以玉米为模型系统的基因组数据库的泛基因组方法。植物生物学21日,385(2021)。https://doi.org/10.1186/s12870-021-03173-5
收到了:
接受:
发表:
关键字
- 数据库
- 基因组
- 玉米
- Pan-genome
- 命名法
- 浏览器
- 不结盟运动的创始人