摘要
背景
大豆是全球重要的豆科作物,是优质植物蛋白和油脂的主要来源。大豆种子蛋白含量是由多基因控制的重要品质性状。
结果
本研究利用大豆蛋白质含量较高的品种南夏豆25号,通过QTL定位、QTL-seq和RNA测序(RNA-seq),揭示了大豆蛋白质含量的控制基因。利用178个重组自交系(RILs),在除4、12、14、17、18和19染色体外的14条染色体上鉴定出50个SPC QTL。在这些QTL中,主要QTL q程控_20-1和q程控20号染色体上的_20-2在6个测试环境中被重复检测到,与基于全基因组测序的QTL-seq检测到的主要QTL的位置相对应。在q的QTL区域内共获得329个候选deg程控_20-1和q程控基因表达谱分析_20-2。其中9个与SPC相关,可能代表候选基因。克隆测序结果显示,高蛋白和低蛋白基因型之间存在不同的单核苷酸多态性(SNPs)和indelsGlyma.20G088000而且Glyma.16G066600可能是这一性状变化的原因。
结论
这些结果为大豆种子蛋白质含量的候选基因和标记辅助选择的研究提供了依据。
背景
大豆的蛋白质平均含量约为40%,g·马克斯(l)Merr.)是植物蛋白最重要的来源,占膳食消费量的71%,且这一比例仍在逐年上升[1].大豆种子蛋白含量存在较大差异。根据美国农业部大豆种质资源数据库(https://npgsweb.ars-grin.gov/),栽培大豆的SPC为31.7 ~ 57.9%,野生大豆的SPC为35.5 ~ 56.9%,表明大豆SPC的遗传改良潜力很大。虽然提高大豆品种的SPC一直是几十年来许多大豆育种计划的主要目标,但SPC与种子含油量和种子产量的强负相关同时改善了三个性状,这是传统育种中具有挑战性的任务[2,3.,4].世界上种植的大部分大豆都是商品大豆,农民按重量而不是成分支付报酬。因此,在品种培育中,育种者通常选择产量潜力最好的种子,而很少关注种子蛋白质或油脂含量[5].因此,大品种大豆普遍追求高产或高含油量。蛋白质含量高的大规模大豆品种很少,蛋白质含量超过50%的品种更是少之又少。考虑到人们对植物蛋白的需求和农民的经济效益,鉴定与控制种子产量、种子蛋白质和含油量的QTL相关的分子标记是打破这些性状之间负相关关系的必要前提。
经典的定量遗传分析表明,大豆的SPC是一个具有加性效应的数量性状,受多个遗传位点的控制,受genotype×environment相互作用的影响[4,6,7,8].连锁分析是在分子遗传学水平上分析植物复杂性状的有效方法。自从Diers等人(1992)首次使用连锁分析在染色体上发现了一个与大豆蛋白和油含量相关的主要QTL (Chr.) 20 [9],大量与SPC相关的QTL相继被报道。在本研究之前,已有241个具有SPC的QTL位点被记录在soybase数据库(http://soybase.org/),涉及双亲群体的每一条染色体。此外,全基因组关联研究(GWAS)已广泛应用于大豆复杂性状的研究,包括SPC [5,10,11,12,13,14,15].Soybase网站列出了2015年至2018年收集的62个与蛋白质含量相关的QTL,涉及自然群体的每一条染色体。虽然在每条染色体上检测到大量与大豆蛋白含量相关的QTL,但大多数QTL检测频率不高,其中只有57个QTL被验证[16].结果表明,无论是连锁分析还是GWAS分析,与蛋白质含量相关的QTL在20号染色体上的频率最高(连锁群I, LG I),其次是15号染色体(连锁群E, LG E),许多候选基因在这些QTL的置信区间内进行了分析,特别是在20号染色体上。Bolon等人(2010)发现,通过Affymetrix®大豆基因芯片和高通量Illumina®全转录组测序平台,鉴定出13个在NILs之间表现出显著的种子转录本积累差异的基因,这些基因被映射到LG I的8.4 Mbp QTL区域[17].Hwang等(2014)通过关联分析进一步将QTL区间降低至2.4 Mbp [4].在这个区域,候选基因的数量减少到6个(Glyma20g19680,Glyma20g21030,Glyma20g21080以及三个不相关的基因)。上述候选基因在物理图上有一定距离,但如果用这些基因发育标记进行遗传分析,在遗传图上的距离可能非常近。因此,该区域可能存在非常重要的候选基因,有待进一步验证。
Bulked secant analysis (BSA)是一种简单快速的目标基因定位方法[18].根据不同的类群材料和实验设计,牛血清蛋白可分为以下几种:(1)QTL-seq适用于主基因显著的品质性状和数量性状[19];(2) MutMap适用于突变系分析[20.];(3) Mutmap(也称为Mutmap +)适用于早期致死或非杂合突变体[21];(4) Mutmap-gap适用于目标基因不在物种参考基因组上的性状[22].近年来,BSA已广泛应用于重要性状的遗传定位,如答:芥[23,24],大米[19,20.]和玉米[25].基于BSA的作图方法也广泛应用于大豆相关性状的研究,包括株高[26,27],开花时间[28]、疫霉抗性[29],种子子叶颜色[30.].与传统的QTL作图方法相比,BSA只需要考虑群体中的少数极端个体,而不是整个群体,简化了测序过程,显著降低了测序和分析的成本[31].随着DNA测序技术的发展,基于下一代测序(NGS)的BSA方法大大加快和改善了因果基因的鉴定过程[32].
从南夏豆25号繁殖的后代60通过多年的系谱选育,利用Coγ辐射诱变了荣县东豆,是中国西南地区广泛栽培的高蛋白大豆品种。该品种的SPC值为50.1%,具有耐遮阴性好、抗倒伏性强、抗大豆花叶病毒株系SC3和SC7等优良特性,适合与玉米间作。在本研究中,我们使用了由“南夏豆25号”和“同豆11号”与F2分离群体是由‘南下斗25’和‘容先东斗’的杂交发展而来的。本研究结合QTL作图、QTL-seq、全基因组重测序(WGRS)和RNA-seq技术对大豆蛋白质含量的QTL和候选基因进行分析,以期拓宽大豆的遗传基础,为作物改良提供依据。本研究的目的是确定SPC的稳定QTL,挖掘相关基因组区域的潜在候选基因,并开发相应的分子标记,最终可用于标记辅助选择(MAS)技术促进高蛋白大豆品系的开发。
结果
SPC的表型特征
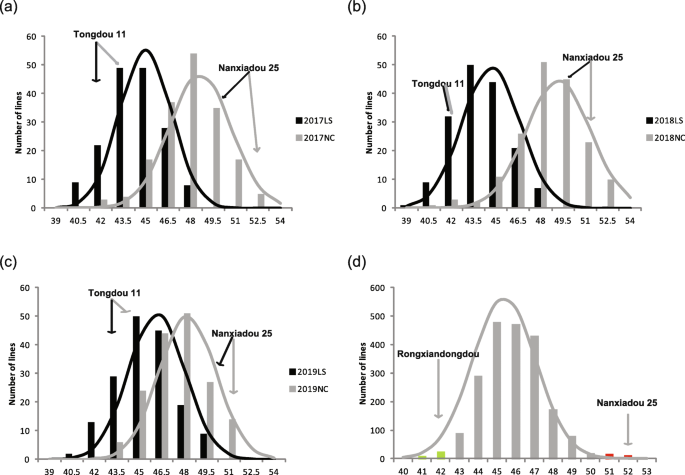
SPC在RIL和F2人口数量见表S1和无花果。1.在RIL群体中,SPC持续变化,并观察到海侵分离。高蛋白亲本南夏豆25号在2个试验点连续3年的SPC均高于同豆11号。“南夏豆25”的SPC平均比“同豆11号”高7.18%。此外,NC组RIL群体的SPC高于LS组。RILs中SPC的变异系数(CV)为3.02 ~ 4.61,广义遗传力高达86.68%。表型数据接近正态分布,表明该定位群体适合进行QTL分析。采用SPSS 20.0软件对RILs群体的SPC进行双向方差分析,基因型(G)、环境(E)和环境相互作用基因型(G × E)对SPC的影响显著(p< 0.001)。对于F2实验结果表明,该粒子的频率近似正态分布,且存在明显的海侵偏析现象。高蛋白亲本“南夏豆25”的SPC值比野生型“容仙东豆”高9.33%。
SPC的遗传图谱及QTL分析
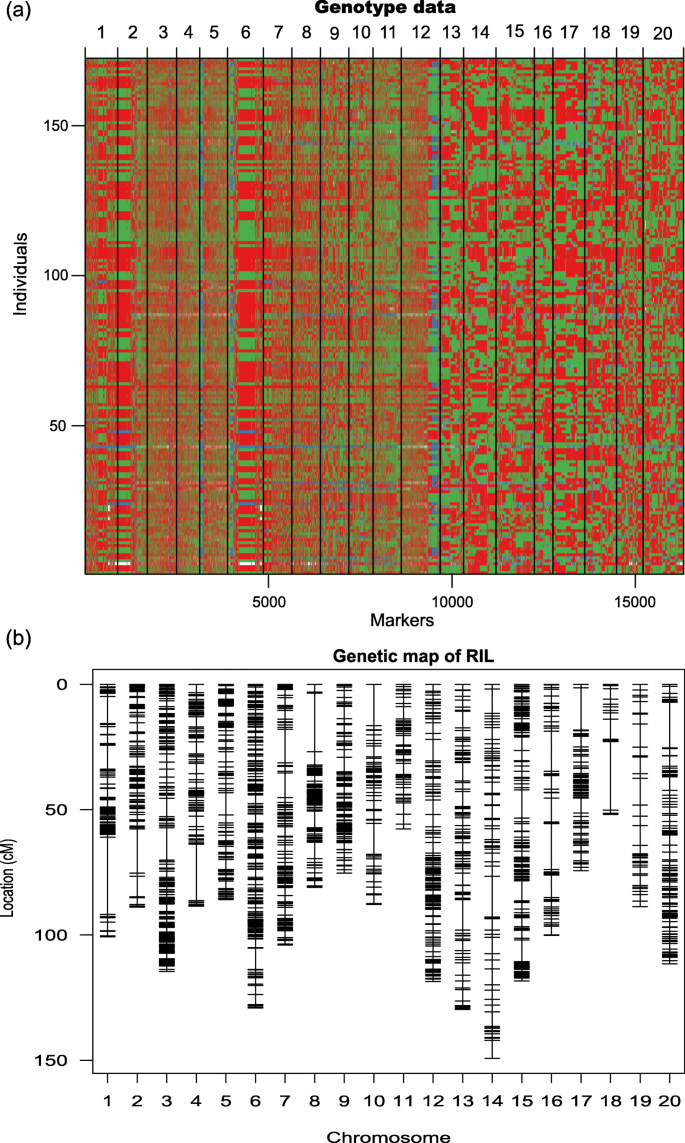
50 K序列共检测到28364个SNP标记在亲本“南夏豆25号”和“同豆11号”之间存在多态性。具有严重偏析畸变(x2测试中,p< 0.05)通过Joinmap 4.0进行去除。其中,在RIL群体中分离比例为1:1的16546个同源SNP标记采用MPR方法进行遗传连锁分析和连锁图构建(图5)。2a).采用R/qtl软件包绘制高密度遗传图谱(图。2b).最终地图包含2072个箱,覆盖1945.09 cM,跨越20个连锁群(LGs),相邻箱间平均距离0.94 cM。每个LG平均有104个箱子,从17个(Gm18)到232个(Gm03)不等(表2)S2而且S3).
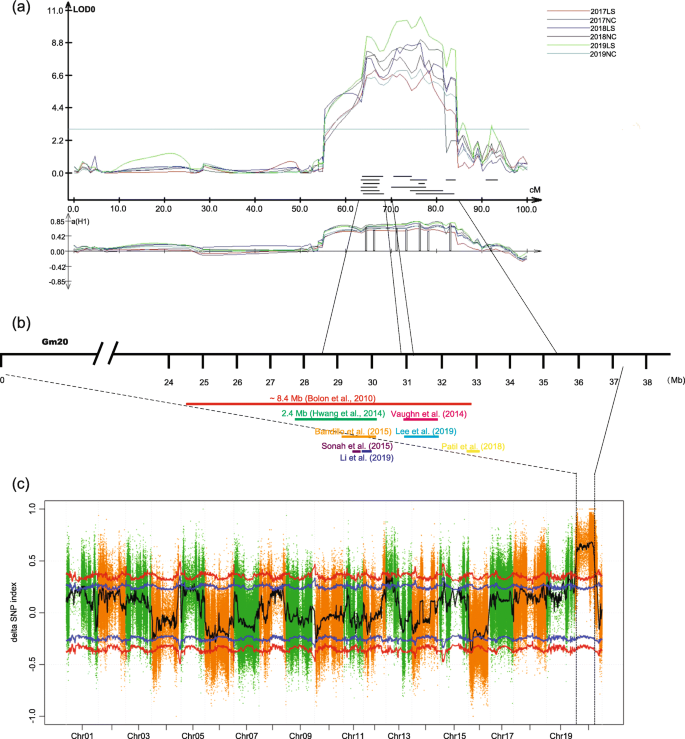
利用遗传图谱,除Chr. 4、12、14、17、18和19外,共在14条染色体上定位了50个QTL。S1).在6个测试环境中,这些QTL解释了1.21-17.50%的SPC表型变异,平均解释了总表型变异的63.16%1).两个紧密相连的QTL q程控_20-1和q程控在连锁组I (Chr 20)中检测到的_20-2解释了6个测试环境中SPC的20.13%至46.88%的表型变异(图2)。3.a),其次是第二大QTL q程控_15 (LOD of 4.06-9.25)1).对于QTL q程控_20-1和q程控_20-2,它的置信区间与许多先前的报道有重叠,这可能是一个主要的QTL(图20 - 2)。3.b). q的等位基因程控_20-1和q程控“南夏豆25”的_20-2对SPC的增加有贡献,而QTL q程控第15染色体上的_15由“同豆11号”贡献。LS环境中检测到的QTL主要集中在第3、15和20号染色体上,NC环境中检测到的QTL分布范围相对较广。在这些QTL中,q程控_16只在Gm16染色体的所有NC环境中检测到,对表型变异的影响为4.67 ~ 4.98%,提示其为环境特异性位点。
QTL-seq在20号染色体上鉴定出一个主要的SPC QTL
根据F2在Illumina X-ten系统上进行全基因组测序,利用两个极端散装和双亲散装构建文库。经过筛选,获得了高质量的测序数据,Q20平均为94.9%,Q30平均为87.6%S4).使用1 kb滑动窗口以1 Mb的间隔计算每个识别SNP的SNP-索引,并绘制HP-bulk(图2)。S2a)和LP-bulk(图。S2b).结合HP-bulk和LP-bulk中SNP-index的信息,计算并绘制了Δ (SNP-index)的基因组位置(图。3.c).在99%的统计水平上,20号染色体上37.88 Mb从0.01到37.89 Mb与SPC显著相关(图;3.c,表S5),命名为qSPC-I。与连锁分析结果比较,该候选区域与QTL q的置信区间重叠程控_20-1和q程控_20-2。这些结果表明,在20号染色体上存在一个与SPC相关的主要QTL。此外,在95%的统计水平上,基因组区域的Δ (SNP指数)值分别在染色体1、2、5、6、10、13、15、16、18和19上检测到与0有显著差异(表S6).对于qSPC-I,在亲本系中鉴定出46,530个snp,其中61个SNP-index在HP-bulk中为1.0,表明reads包含来自'南夏豆25 '的基因组片段)。在所有这些snp中,有778个snp可能导致编码序列的改变S7).
从可靠QTL中鉴定spc相关候选基因
为探索SPC相关的候选基因,采用南夏豆25和容仙东豆生长发育阶段R5、R6和R7种子的RNA进行RNA-seq分析。使用|log2(折叠变化)| > 1.5和aP-value≤0.05,6440、6051、4795三个DEGs在南夏豆25号和容仙东豆之间表达差异显著。其中,有86个基因在三个时间段内表达量显著增加,555个基因在三个时间段内表达量显著降低(图。S3).为了更有效地筛选候选基因,我们从QTL和QTL-seq结果的共定位区间中选择可信位点。因为q程控_20-1和q程控_20-2在6个测试环境中被重复检测到,并与QTL-seq中的qSPC-I重叠,我们分析了q QTL区域内所有基因的表达谱程控_20-1和q程控_20-2,并根据RNA-seq数据去除一些非差异表达基因。共获得329个候选deg(表S8).
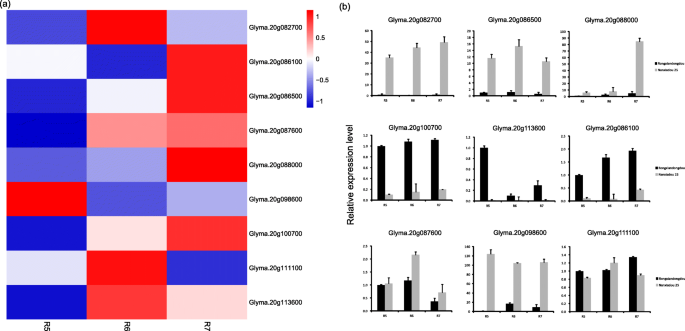
为了缩小SPC的候选基因范围,根据大豆参考基因组的基因注释信息,我们选择了9个最有希望的候选基因,包括结节蛋白MtN3家族蛋白(Glyma.20 g082700)、肌动蛋白相关蛋白C2B (Glyma.20 g086100)、锌节蛋白(Glyma.20G086500),靶向SNARE线圈结构域蛋白(Glyma.20 g087600)、s -腺苷- l-蛋氨酸依赖甲基转移酶超家族蛋白(Glyma.20G088000)、膜体内禀蛋白(Glyma.20 g098600),核酸酶(Glyma.20G100700),是突触breven样蛋白家族的成员(Glyma.20G111100),以及bZIP转录因子家族蛋白(Glyma.20 g113600)(图。4a).我们通过qRT-PCR分析进一步验证了这10个基因在两个亲本之间的表达水平。qRT-PCR结果与RNA-seq分析结果相似,表明我们的RNA-seq结果是可靠的(图。4b)。
SPC筛选候选基因
为了进一步探索SPC的致病基因,我们比较了“南夏豆25”和“容仙东豆”9个最有希望的候选基因的SNP信息。在过滤掉无意义的突变信息后,我们进一步将范围缩小到三个候选基因Glyma.20G088000,Glyma.20G111100而且Glyma.20 g087600.为了消除偶然变异,我们比较了“南夏豆25”和“容咸东豆”的3个潜在候选基因序列和其他4个大豆基因型。由于“南夏豆25”系来自“容县东豆”,我们还从“容县东豆”的2个附加种质(B康57和南豆12)中鉴定了3个潜在候选基因的序列多态性。为Glyma.20G088000在8个大豆基因型中鉴定出47个SNPs和12个indel,其中s -腺苷- l-蛋氨酸依赖甲基转移酶超家族蛋白参与了脂质生物合成过程。在这些SNPs中,有2个SNPs在高蛋白品种中具有特异性:位于32,926,792 bp的一个是T-A取代,导致氨基酸从Asn转变为Lys;另一个位于32,926,782 bp,仅在“南豆12号”和“南夏豆25号”中发现,引起了终止密码子突变。此外,在32,932,285和32,932,328 bp位点有一个44 bp的缺失突变,导致产生一个终止密码子,并且只存在于高蛋白品种中(图2)。5一个)。Glyma.20G111100该蛋白被注释为编码突触brevin样蛋白家族的一个成员,是存储蛋白运输到蛋白质存储空泡(PSV)以及PSV组织和生物发生所必需的。共鉴定出18个SNPs和2个indelGlyma.20G111100在8个大豆基因型中,有2个SNP变异导致了氨基酸的变化(图;5一个)。
8种不同大豆基因型的测序分析和特异性snp鉴定。(a), 8种不同大豆基因型中2个基因的测序分析。(b)自然群体InDel标记多态性的鉴定。M:标记。1-12车道和14-25车道分别对应蛋白质含量差异较大的品种。详细信息见表S9.(c), InDel标记在RILs群体中的多态性鉴定。M:标记。1-12车道和14-25车道分别对应蛋白质含量差异较大的品系。详细信息如表S9所示
Glyma.20 g087600被注释为靶SNARE线圈结构域蛋白,参与ER到高尔基囊泡介导的转运。虽然基因序列没有突变,但其巨大潜力位于多个环境反复检测到的SNP峰值(32,752,215 bp)附近,Lee et al.(2019)报道该SNP与大豆种子蛋白质和含油量有关[5].为了评估该SNP的潜力,我们利用GRIN数据库中的蛋白质含量数据和SoyBase数据库中的基因型数据进一步分析了该位点的自然变异。有趣的是,我们发现在蛋白质含量大于50%的人群中,等位基因“C”的频率为0.61。然而,在蛋白质含量低于42%的群体中,这一比例下降到0.002,该位点只有来自日本的7个品种存在“C”等位基因(图2)。S4a - b)。
此外,qSPC_16只来自NC环境是我们关注的另一个焦点。在这个QTL区域,Glyma.16G066600作为一种参与液泡蛋白加工的α -液泡加工酶引起了我们的关注。在Glyma.16G066600在8个已鉴定的SNP中,只有一个位于6,606,140 bp的SNP是G-T,这导致了氨基酸从Ser到Ile的变化,并不是高蛋白品种所特有的(图。S4c)。
在自然种群和RILs种群中验证变异和表型之间的关联
我们选择Glyma.20 g088000在自然种群和RILs种群中验证变异和表型之间的关联。从自然和RIL群体中随机选择的共96个株系用于扩增具有显著spc相关InDel的DNA片段,该InDel位于的第1外显子内Glyma.20 g088000.PCR扩增结果显示,高蛋白系电泳条带约为160 bp,低蛋白系电泳条带约为204 bp(图2)。5c,表S9).在不同环境的RILs和自然群体中,对两组间的SPC表型进行t检验。结果,含有缺失等位基因的株系表型显著(P< 0.05)均大于在各环境中含有正常等位基因的株系(表S10).
讨论
SPC作为典型的数量性状,受多个小效应基因控制[3.].由于大豆遗传连锁图谱的可用性,近三十年来,通过双亲分离居群的连锁作图和自然居群的关联作图,已鉴定出大量大豆SPC的QTL [33].据不完全统计,这些QTL广泛分布于所有20条染色体的不同区域。其中,20号染色体因其高加性效应(表型变异12-55%)和稳定性而备受关注[34].20号染色体上的基因组区域(24.5 - 32.9 Mb)是SPC的一个特别有吸引力的主要常见QTL [4,10,17].随后,Hwang et al.(2014)进一步将该区域缩小为2.4 Mb区域,位于27.6 - 30.0 Mb之间[4].有趣的是,Vaughn等人(2014)[35]报道了Hwang等人(2014)鉴定的区域下游约1 Mb的QTL [4], Lee等人(2019)进一步通过GWAS在这1 Mb基因组区域中鉴定了一个839 kb的区域[5].然而,Patil等人(2018)使用位于33,975,596-34,027,051 bp的高分辨率bin map报告了一个与SPC高度相关的QTL [34], Li等人(2019)通过多位点全基因组关联研究报告了另一个基因组区域(30995685-31,177,423 bp)与SPC高度相关[33].此外,Chung等人(2013)在20号染色体上发现了长期驯化和选择形成的30.5-32.3 Mb区域[36].Vaughn et al.(2014)根据Tajima’s D值的结果进一步证实了该区域可能与驯化和选择有关[35].一般认为在20号染色体上存在一个控制大豆蛋白质含量的遗传位点,但关于该遗传位点的确切位置存在不同的结论。在我们的研究中,在99%的统计水平上,20号染色体上从0.01到37.89 Mb的一个基因组区域具有Δ (SNP-index)值,该值与SPC显著相关。这一结果进一步证明了SPC在20号染色体上存在一个稳定可靠的遗传位点。有趣的是,我们通过连锁作图在20号染色体上检测到两个稳定的QTL,这两个QTL彼此接近,并且与之前报道的QTL区域重叠(表2 - 4)1和无花果。3.a).结合以往结果的差异,我们推测在20号染色体上可能存在两个或多个控制蛋白含量的遗传位点。
从检测到的QTL中,鉴定出许多SPC的候选基因。Bolon等人(2010)发现,通过Affymetrix®大豆基因芯片和高通量Illumina®全转录组测序平台,鉴定出13个在NILs之间表现出显著的种子转录本积累差异的基因,这些基因被映射到LG I的8.4 Mbp QTL区域[17].Bandillo等人(2017)进一步缩小了这个区域,现在这个区域只包含三个(Glyma20g21030,Glyma20g21040,Glyma20g21080)的原始12个潜在候选基因[10].基于高密度连锁定位,在Chr.20的置信区间QTL中发现了13个基因,标记为bin_20_33,975,596和bin_20_34,027,051 [34),而Glyma20g21693而且Glyma20g21726被认为是另一个区域的重要基因(Gm20_30,995,685-31,177,423 bp) [33].为了在3月20日的主要QTL区域内识别候选基因,我们对南夏豆25号和容仙东豆进行了RNA-seq分析,揭示了一些可能与大豆种子储存白蛋白生物合成有关的关键基因。与高蛋白基因型南夏豆25相比,低蛋白亲本容仙东豆的上调基因多,下调基因少,说明贮藏蛋白积累的高蛋白基因型以下调基因为主,而贮藏蛋白积累的低蛋白基因型以阳性基因为主。我们的实验通过RNA-seq分析确定了9个最有前景的、在主要QTL区域存在显著差异的deg,并通过qRT-PCR和克隆测序分析进一步筛选出9个deg。最后,三个候选基因(Glyma.20G088000,Glyma.20G111100,而且Glyma.20 g087600)被认为是该区域内的重要基因。许多报道表明SPC与含油量和种子产量呈负相关[2,3.],且在20号染色体上有明显的共定位[5,10,33,34].虽然我们在本研究中没有分析种子含油量,但我们认为Glyma.20 g088000高、低蛋白品种序列差异大,在南夏豆25中高表达,可能与南夏豆25的高蛋白形成有关。在多种环境中反复检测到峰值SNP (32,752,125 bp), Lee等[5]报道了该SNP与种子蛋白质和油脂含量有关。根据GRIN数据库的种质资源统计,该SNP位点的罕见等位基因C在低蛋白品种中几乎没有出现。因此,作为最接近SNP的基因,我们推测Glyma.20 g087600具有巨大的SPC潜力。然而,SNP与该基因之间的关系尚不清楚。
除20号染色体外,15号染色体QTL报道最多。在包括第15号染色体在内的其他13条染色体上也检测到若干QTL。作为一个典型的数量性状,我们对仅在NC环境中检测到的QTL比对在多个环境中重复检测但含量低的QTL更感兴趣R2.的问程控_16只在与LS纬度差较大的NC环境中存在,说明q .程控_16是一个环境特定的站点。基于QTL-seq,我们在同一区域发现了一个qSPC-J-1,这进一步表明q程控_16在NC环境下是稳定的。Glyma.16G066600,被认为是该区域内的重要基因,被注释为液泡加工酶(也称为天冬氨酸蛋白酶,APE),在标准生长条件下参与种子贮藏蛋白质加工。综上所述,我们结合QTL作图、QTL-seq、RNA-seq和克隆测序确定了4个潜在的候选基因。然而,我们对上述结果仍持谨慎态度。这些基因参与不同的生物学功能,说明SPC是一个涉及一系列生化通路相关基因的复杂性状。因此,需要更深入的研究来验证候选基因的功能,并深入了解SPC在种子发育过程中沉积的遗传和分子控制机制,以指导作物改良。
结论
本研究利用高蛋白质含量大豆品种南夏豆25号和低蛋白质含量大豆品种同豆11号,进行了数量性状位点(QTL)定位、QTL-seq和RNA测序(RNA-seq),揭示了大豆蛋白质含量的控制基因。利用178个重组自交系(RILs)进行QTL定位,共鉴定出分布在14条染色体上的50个SPC QTL。其中,20号染色体上的主QTL在6个测试环境中被重复检测到,与基于全基因组测序的QTL-seq检测到的主QTL的位置相对应。在q的QTL区域内共获得329个候选deg程控_20-1和q程控基因表达谱分析_20-2。其中9个与SPC相关,可能代表候选基因。克隆测序结果显示,高蛋白和低蛋白基因型之间存在不同的单核苷酸多态性(SNPs)和indelsGlyma.20G088000而且Glyma.16G066600可能是这一性状变化的原因。
方法
植物材料及表型评价
人口1787RILs是由F2品种南夏豆25(高SPC, 50.1%)和品种同豆11(低SPC, 40.5%)的杂交后代。这些RILs及其亲本于2017年夏季、2018年夏季和2019年夏季在中国四川省南充农业科学院(NC;2017年、2018年、2019年海南陵水淡季养殖基地(LS;18.53°N, 110.01°E)。试验采用完全随机区组设计,每组3个重复,每组行长1.0 m,行距0.5 m。总共2097 F2南充市农业科学院于2019年在南充市农业科学院种植了由栽培品种南夏豆25与地方品种容仙东豆(南夏豆25野生型,低SPC)杂交而成的植物。B康57(容县东豆自然突变体,低SPC)、南豆12 (B康57的诱变子代,高SPC)、南夏豆30(高蛋白品种)、石月黄(低蛋白质大豆地方品种)、威氏82来自南充农业科学院保存的种质资源。
采用WinISI II软件(FOSS GmbH,丹麦)的近红外系统6500,用约20克完整种子,在13%的水分基础上测量SPC。波长范围为950 ~ 1650 nm。数据分析采用每个样本三次扫描的平均值。采用SPSS软件对表型数据进行基本统计分析。
图谱构建和QTL分析
合格的文库在Illumina Hiseq Xten平台上进行配对端测序,以获得广泛分布于整个基因组的高质量snp。遗传连锁分析采用软件包MSTmap [37]和Joinmap v. 4.0 [38].首先,利用MSTmap在LOD 5.0处对多态性snp进行分组,然后找到每个连锁群的图的最小生成树,根据成对重组频率确定标记的顺序。采用Joinmap 4.0重新计算并确认各连锁组的标记顺序和距离,LOD评分最低为3.0。
QTL分析采用QTL制图软件WinQTLCart 2.5版本,采用复合区间映射(Composite Interval Mapping, CIM)方法[39].在CIM分析中,选取1-cM的行走速度,回归参数为1000次,显著性水平为0.01。当LOD≥3.0时,该区间可能存在QTL。
QTL-seq
对于QTL-seq,通过混合来自30个高SPC (SPC = 49.72-52.07%)和30个低SPC (SPC = 40.34-42.06%)的等量DNA,分别构建了两个DNA块,高SPC块(HP-bulk)和低SPC块(LP-bulk)2来自2019年实验的个体。由5个随机单株分别构建两个亲本体。用来自两个bulk和两个亲本bulk的约5mg DNA构建了4个配对端测序文库,并在Illumina测序平台上由Genedenovo生物技术有限公司(中国广州)测序。去除适配器和低质量reads后,使用BWA软件将高质量序列对准Williams 82参考基因组[40].基因组分析工具包(GATK)用于调用亲本系和散装的snp和小indels [41].
通过滑动窗口分析计算各SNP位点的SNP-index。去除伪snp, SNP-index < 0.3或> 0.7,读取深度小于7,这是由于测序和/或对齐错误造成的。将两个bulk的SNP指数相减,得到Δ (SNP-index),在10,000个bootstrap重复中,在这些正负峰区域以95%的置信区间确定QTL。然后选取峰值区域的SNPs和indel进行标注,筛选潜在的功能变异。
RNA-seq和候选基因的表达分析
采用RNA-seq和实时定量PCR (RT-qPCR)技术对QTL-seq共定位和连锁定位候选基因的表达模式进行研究。每朵花都标记在花期早期,分别从两个亲本的三株不同植物的R5、R6和R7生长阶段随机收集5个荚果。从不同植物豆荚中提取不同发育阶段的RNA,均匀混合,构建文库。去除低质量的读取,所有清洁的读取都映射到Williams82。a2使用TopHat 2.0版本[42].差异表达基因(DEGs)用R的EBSeq包进行挖掘,FDR(假发现率)< 0.05,|log2(折叠变化)| > 1.5。
选择9个候选基因进行qPCR分析,验证RNA-seq结果的可靠性。根据制造商的说明,1 μg RNA样本使用iScript进行第一链cDNA合成TmcDNA Synthesis Kit (Bio-Rad, Hercules, CA, USA),在CFX96 real-time System (Bio-Rad)上进行实时PCR检测9个候选基因的表达水平。每个样品进行三次技术复制。计算各基因的相对表达量——ΔΔCt方法(43)使用Actin11作为内部控制。qRT-PCR引物采用Primer Premier 5.0设计,列于表中S11.
候选基因序列分析
为了明确两个亲本QTL作图和QTL-seq重叠区域的候选基因的变异情况,对QTL-seq的基因序列进行了分析Glyma.20G111100而且Glyma.20 g087600分别对B康57、南豆12、石月黄和威廉斯82进行测序分析。候选基因采用TransStart®FastPfu DNA聚合酶进行PCR检测,经琼脂糖凝胶电泳后送青岛生物科技有限公司测序。DNA同源性比对采用Invitrogen Vector NTI 11.5.1软件。
数据和材料的可用性
在当前研究中生成和分析的数据集可在NCBI SRA数据库(BioProject ID: PRJNA752801 and PRJNA752694)中获得。在此研究过程中产生的其他数据包括在本文及其附加文件中。本文中基因的序列数据可以在Phytozome 13 (https://phytozome-next.jgi.doe.gov/),以下列登记编号:Glyma.20G088000,Glyma.20G111100,Glyma.20 g087600,Glyma.16G066600.所有实验材料均可按要求提供。
缩写
- 程控:
-
种子蛋白质含量
- QTL:
-
数量性状位点
- 瑞来斯:
-
重组自交系
- MAS:
-
分子标记辅助选择
- 农业部:
-
美国农业部
- BSA:
-
散装隔离分析
- GWAS:
-
全基因组关联研究
- 水气比:
-
全基因组重测序
- 检测:
-
近红外反射光谱
- NC:
-
南充
- LS:
-
陵水
- CIM:
-
复合区间映射
- HP-bulk:
-
蛋白质含量较高
- LP-bulk:
-
蛋白质含量较低
- GATK:
-
基因组分析工具箱
- SNP:
-
单核苷酸多态性
- RT-qPCR:
-
实时定量PCR
- 度:
-
差异表达基因
- 罗斯福:
-
错误发现率
- FPKM:
-
每一百万个片段映射
- LGs:
-
连杆组
参考文献
- 1.
王晓明,王晓明,王晓明,等。大豆蛋白质和油脂含量qtl的元分析[大豆(l)稳定。种子。中国生物化学杂志,2017;18:1180。
- 2.
林克,李志强,李志强,等。美国大豆成熟组II、III、IV的遗传改良。作物科学,2014;54:1419-32。https://doi.org/10.2135/cropsci2013.10.0665.
- 3.
陈鹏,陈晓明,王晓明,陈晓明,等。大豆种子蛋白分子图谱和基因组学研究进展及展望。《理论与实践》,2017;
- 4.
黄怡,宋强,贾国刚,裴志强,刘志强,等。大豆种子蛋白质与油脂含量的全基因组关联研究。BMC Genomics. 2014;15:1。
- 5.
李S, Van K, M Sung, Nelson R, LaMantia J, McHale LK,等。大豆籽粒蛋白质、油脂和氨基酸含量的全基因组关联研究,大豆学报,2019;132:1639-59。
- 6.
刘志强,刘志强,刘志强,刘志强,等。大豆蛋白质、油脂及5种主要脂肪酸含量的QTL鉴定植物科学学报,2014;05:158-67。https://doi.org/10.4236/ajps.2014.51021.
- 7.
李东,赵霞,韩艳,李伟,谢峰。大豆籽粒蛋白质和油脂含量的全基因组关联定位。基因组学。2019;111:90-5。
- 8.
麦肯德里AL,麦克维蒂PBE,沃尔登HD。早熟大豆种子蛋白质和籽油含量的遗传。《中华医学杂志》1985;27:7 3 - 7。
- 9.
迪尔斯BW,凯姆P,费尔WR,鞋匠RC。大豆种子蛋白质和油脂含量的RFLP分析。应用理论。1992;83:608-12。
- 10.
王志强,王志强,王志强,等。美国农业部大豆种质资源的群体结构和全基因组关联分析。植物基因组学报。2015;8:plantgenome2015.04.0024。
- 11.
李丽娟,张辉,李超,陈春春,宋鸿波。野生大豆种子组成性状的全基因组关联研究。BMC Genomics, 2017;18:18。
- 12.
孙丽娟,李志刚,李志刚,李志刚。大豆8个农艺性状的QTL定位和QTL定位分析。中国生物技术学报,2015;13(4):344 - 344。
- 13.
张东,Lü H,褚松,张宏,张宏,杨勇,等。大豆水溶性蛋白质含量的遗传结构及其与总蛋白质含量的遗传关系。科学代表2017;7:5636。
- 14.
张娟,王鑫,陆勇,Bhusal SJ,宋强,Cregan PB,等。对种子组成的全基因组扫描为大豆品质的改善和驯化和育种的影响提供了见解。植物学报。2018;11:460-72。
- 15.
张涛,吴涛,王磊,姜波,甄超,袁松,等。结合连锁和GWAS分析确定了与大豆种子蛋白质和油含量相关的qtl。中国生物化学杂志,2019;
- 16.
李松,李志强,李志强,杨娟,等。基于高密度bin图的多环境下大豆种子蛋白质和油脂含量主效应和上位效应qtl全基因组检测中国药理学杂志2019;20:979。
- 17.
Bolon YT, Joseph B, Cannon SB, Graham MA, Diers BW, Farmer AD,等。互补遗传学和基因组学方法有助于鉴定大豆连锁群I种子蛋白QTL。植物学报。2010;10:41。
- 18.
邹超,王鹏,徐颖。大样本分析在遗传学、基因组学和作物改良中的应用。植物学报,2016;14(4):441 - 441。
- 19.
高木H,安部A,吉田K,小杉S,夏目石S,光冈C,等。QTL-seq:通过对两个大群体的DNA进行全基因组重测序,快速定位水稻数量性状位点。植物学报(英文版);2013;
- 20.
阿部A,小杉S,吉田K,夏目石S,高木H, Kanzaki H,等。利用MutMap测序水稻的重要农艺位点。生物技术学报。2012;30:174-8。
- 21.
费基R,高木H, Tamiru M,阿部A,夏目石S, Yaegashi H,等。MutMap+:水稻无杂交的基因定位和突变体鉴定。科学通报。2013;8:e68529。
- 22.
高木H,植村A,八垣H,田iru M,安部A,光冈C,等。MutMap-gap:突变F2子代体的全基因组重测序结合gap区域的从头组装鉴定了水稻稻瘟病抗性基因Pii。植物学报。2013;200:276-83。https://doi.org/10.1111/nph.12369.
- 23.
Schneeberger K, Ossowski S, Lanz C, Juul T, Petersen AH, Nielsen KL,等。SHOREmap:通过深度测序同时作图和突变鉴定。Nat Methods. 2009; 6:550-1。https://doi.org/10.1038/nmeth0809-550.
- 24.
邹涛,肖强,李伟,罗涛,袁刚,何忠,等。OsLAP6/OsPKS1是拟南芥PKSA/LAP6的同源基因,对花粉外壁的形成至关重要。大米。2017;10:53。
- 25.
李文杰,李志强,李志强,等。扩增分离分析耦合全基因组测序(BSA-Seq)用于玉米基因的快速克隆。G3基因,基因组。麝猫。2018;8:3583 - 92。
- 26.
结合连锁作图和bsa鉴定大豆株高和主茎节数的qtl和候选基因。国际化学杂志,2020;21:42。
- 27.
张旭,王伟,郭楠,张勇,卜勇,赵杰,等。结合QTL-seq和连锁作图对野生大豆大株高等位基因进行了精细定位。BMC基因组学。2018;19:226。
- 28.
Watanabe S, Tsukamoto C, Oshita T, Yamada T, Anai T, Kaga a .大豆开花时间定量性状位点的鉴定。育种科学,2017;67:277-85。
- 29.
钟超,孙松,李勇,段超,朱哲。大豆新疫霉菌抗性基因RpsHC18的新一代测序分析及诊断标记。理论应用,2018;131:525-38。
- 30.
宋杰,李震,刘震,郭勇,邱丽娟。来自大体积分离分析的下一代测序加速了大豆中两个定性基因的同时鉴定。植物科学进展,2017;
- 31.
杨震,黄东,唐伟,郑勇,梁凯,Cutler AJ,等。水稻幼苗抗寒性的数量性状位点的高通量测序。科学通报。2013;8:e68433。
- 32.
Schneeberger K, Weigel D.新测序技术使遗传学快速发展。植物科学进展(英文版);2011;
- 33.
李松,徐辉,杨娟,赵涛。基于多位点全基因组关联研究的长江和淮河流域大豆种子蛋白质和油脂含量遗传结构分析。中国生物化学杂志2019;20:E3041。
- 34.
王晓燕,王晓燕,张晓燕,等。利用高密度连锁作图技术分析大豆种间作图群体中隐含种子蛋白质、油脂和蔗糖含量的基因组热点。中国生物技术学报,2018;16(2):349 - 349。
- 35.
Vaughn JN, Nelson RL,宋q, Cregan PB,李哲。利用多群体全基因组关联扫描方法优化大豆种子组成的遗传结构。G3基因,基因组。麝猫。2014;4:2283 - 94。https://doi.org/10.1534/g3.114.013433.
- 36.
郑文华,郑宁,金杰,李维奎,李永国,李硕,等。韩国栽培大豆和野生大豆基因组深度重测序揭示的群体结构和驯化。DNA决议2014;21:153-67。
- 37.
吴勇,Bhat PR, Close TJ, Lonardi S.基于最小生成树的遗传连锁图谱的高效和准确构建。公共科学学报,2008;4:e1000212。
- 38.
Van Ooijen JW。JoinMap®4软件用于计算实验人群的遗传连锁图:Wageningen;2006.
- 39.
王珊,巴斯滕杰,曾志斌。Windows QTL制图器2.5。罗利:北卡罗莱纳州立大学统计系;2012.http://statgen.ncsu.edu/qtlcart/WQTLCart.htm
- 40.
李华,杜斌。基于钻穴-轮变换的快速精确短读对准。生物信息学。2009;25:1754-60。
- 41.
麦肯纳,汉娜M,班克斯E, Sivachenko A, Cibulskis K, Kernytsky A,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组Res. 2010; 20:1297-303。
- 42.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:存在插入、缺失和基因融合时转录组的精确对齐。中国生物工程学报。2013;14:R36。
- 43.
Livak KJ, Schmittgen TD。利用实时定量PCR和2-ΔΔCT方法分析相关基因表达数据。方法。2001;25:402-8。
确认
我们非常感谢审稿人和编辑对本文提出的建设性意见和建议。
资金
本研究由国家重点研发计划项目(2017YFD0101500)和国家现代农业产业技术体系四川省创新团队(SCCXTD-2021-20)资助。作者声明,资助者在研究的设计、数据的收集、分析和解释以及撰写手稿中没有任何作用。
作者信息
从属关系
贡献
LZL和MRZ设计了研究;JW、ZQZ、XBY、JQL、JF、JGA、WYY进行了研究;JW和LM分析了数据;HYW和MRZ提供资源;LZL为研究和稿件准备提供建议,JW撰写稿件。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明,这项研究是在没有任何商业或金融关系的情况下进行的,这些关系可能被解释为潜在的利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
大豆RIL和F中SPC的描述性统计、广义遗传力和方差分析中的F值2人口
附加文件2:表S2。
大豆染色体/连锁基团上的snp分布表S3。高密度SNP图谱信息,表S4.Illumina测序数据汇总表S6。大豆SPC的QTL定位表S7。基因编码序列中SNP的信息,表S8。qSPC_20-1和qSPC_20-2 QTL区域内基因的RNA-seq数据,表S9。自然居群和RILs居群种子蛋白质含量表S10。P不同环境下spc相关InDel的t检验值,表S11。RT-qPCR候选基因引物信息
附加文件3:表S5。
qSPC-I区域每个SNP的SNP-index和注释的详细信息
附加文件4:图S1。
与蛋白质含量相关的QTL定位。为简单起见,只显示QTL置信区间内的标记,以及包含QTL的染色体两端的末端两个标记
附加文件5:图S2。
HP-bulk(高蛋白块)和LP-bulk(低蛋白块)的单核苷酸多态性(SNP)指数图。(a),表示HP-pool的单核苷酸多态性(SNP)指数图。(b),表示LP-pool的单核苷酸多态性(SNP)指数图
附加文件6:图S3。
RNA-seq数据的维恩图分析显示R5、R6和R7期差异表达基因(南夏豆25/容夏冬豆)。(a),表示上调deg数量的维恩图。(b),表示下调deg数量的维恩图
附加文件7:图S4。
栽培品种SNP的等位基因分类及多序列比对Glyma.16G066600.(a),表示SPC小于42%人群的基因型分布;(b),表示SPC大于50%人群的基因型分布。(c),表示描述氨基酸序列差异的多序列比对Glyma.16G066600
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
王俊,毛丽丽,曾忠。et al。大豆“南夏豆25”高蛋白QTL的遗传定位及候选基因分析。BMC植物生物学21,388(2021)。https://doi.org/10.1186/s12870-021-03176-2
收到了:
接受:
发表:
关键字
- 大豆
- 种子蛋白质含量
- 数量性状位点(QTL)
- QTL-seq
- RNA-seq