抽象的
背景
对花生的过敏反应(arachis hypogaea.L.)会导致严重症状,在某些情况下甚至是致命的,但由于加工食品中花生衍生产品的流行,很难避免。降低花生过敏原性的一种策略是通过诱变改变或消除花生过敏原蛋白。通过改变生物合成酶活性可以改善其他种子品质性状。靶向诱导基因组局部病变(TILLING)是一种反向遗传学方法,用于识别影响花生种子性状的突变。
结果
两个主要过敏原基因的类似副本,ARA H 1,已在四倍体花生中鉴定,一个在每个亚基因中。主要过敏原表明了同样的情况ARA H 2..由于同种异体四倍体花生中同源基因的鉴别困难,采用巢式PCR方法,采用未标记引物对同源基因拷贝进行扩增。随后,使用基因特异性标记引物进行第二次PCR,形成异质双工,CEL1核酸酶切,并对标记片段进行电泳检测。以甲基磺酸乙酯(EMS)为诱变剂,诱变频率为1 SNP/967 kb (3,420 M2个体筛选)。发现的最重要的突变是一个中断的起始密码子ARA H 2.02和一个过早的终止密码子ARA H 1.02. 这些突变的每一代都恢复了纯合子个体,并且确认了Ara h 2.02蛋白的消除。根据2D凝胶分析,几种Ara h 1蛋白亚型被消除或减少。TILLING还被用于鉴定脂肪酸去饱和酶的突变Ahfad2.(还存在于两份拷贝中),一种控制种子中油酸与亚油酸的比例的基因。鉴定了帧突变突变,导致AHFAD2B蛋白的截短和失活。一个突变AhFAD2A预计将恢复到通常无活性酶的功能。
结论
这项工作代表了朝着创造花生品种的第一步,其具有降低的过敏性。在花生中可以扩展到几乎任何基因,并且可用于改变其他特征,例如种子的营养特性,如本研究所示。
背景
花生(arachis hypogaea.L.)是一种重要的石油和蛋白质来源,由于它们的营养益处和多功能性,花生和花生衍生的产品在加工食品中广泛使用。不幸的是,对花生的过敏反应的报道越来越普遍,对花生的严重过敏反应可能是致命的[1].避免是预防过敏反应的最佳策略,但由于食品中花生的患病率,避免可能是困难的。甚至没有特别含有花生产品的食物,但在也用于处理花生的设备上加工,仍可含有大量过敏原,以引发一些患者的过敏反应。根据国际免疫学会(IUIS)的国际联盟,花生含有至少11个潜在的过敏性蛋白质[2].淘汰负责过敏蛋白的产生的基因将是降低花生过敏潜力的一种策略。然而,许多过敏原是种子储存蛋白,其构成相当量的总种子蛋白。例如,主要过敏原ARA H 1占总种子蛋白的12-16%,ARA H 2为5.9-9.3%[3.].虽然Chu等人来说,但是在不牺牲质量或活力的情况下,可以消除其中许多蛋白质。[4.使用转基因沉默以消除花生种子中的ARA H 2和ARA H 6蛋白,并观察到对生存能力的不利影响。虽然这种结果很有希望,但有许多监管障碍必须克服转基因花生作为食物。
另一种策略是使用诱变来淘汰过敏原基因,或者可能改变这些蛋白质中的主要过敏性表位序列。这可以通过耕种(靶向诱导的基因组局部病变)来实现,一种技术可以筛选诱变群体的携带任何已知的感兴趣的基因中的突变。耕作是一种基于PCR的技术,依赖于Cel1核酸酶的错配裂解以鉴定单核苷酸或小插入/缺失突变。倾向于在模型物种中作为反向遗传工具开发拟南芥蒂利亚纳[5.]但已经应用于重要的作物物种,包括米饭(奥雅萨苜蓿l .) [6.],玉米(Zea Mays.l .) [7.]和大豆(大豆(L.)Merr。)[8.],只有几个。
在先前的研究中,我们报告了主要过敏原基因的基因组特征ARA H 2.[9.]编码两种亚型Ara h 2.01和Ara h 2.02的基因很可能是同源基因,代表来自二倍体祖先的同源基因答:duranensis(一个基因组)和A. iPaensis.在这项研究中,我们发现主要的过敏原ARA H 1基因也存在于两份拷贝中,每个副本属于单独的亚因子。开发了基因特异性引物以鉴定两个中的每一个中的突变ARA H 1和二ARA H 2.基因通过耕作。
除了过敏原,种子油组合物是在花生中的另一种质量特征,可以使用耕作方法靶向。单不饱和脂肪酸不易氧化,而不是多不饱和脂肪酸,因此有助于更好的风味和更长的花生油储存寿命[10].此外,单不饱和脂肪酸在营养上是可取的,并被认为有助于心血管健康。亚油酸(18:2)是一种多不饱和脂肪酸,通常约占花生油的15-43% [11].在显影种子中,通过δ的作用,由单一饱和油酸(18:1)产生12脂肪酸去饱和酶(AHFAD2)。两个职业Ahfad2.基因已在花生中鉴定出来,一个来自每个亚群的,指定AhFAD2A和AhFAD2B[12].AHFAD2的活性的减少增加了油酸与亚油酸的比例,但只需要一种功能等位基因来赋予正常的含油表型[13].每个的突变Ahfad2.还使用耕作鉴定基因。
结果
测定基因拷贝数,以及基因特异性扩增
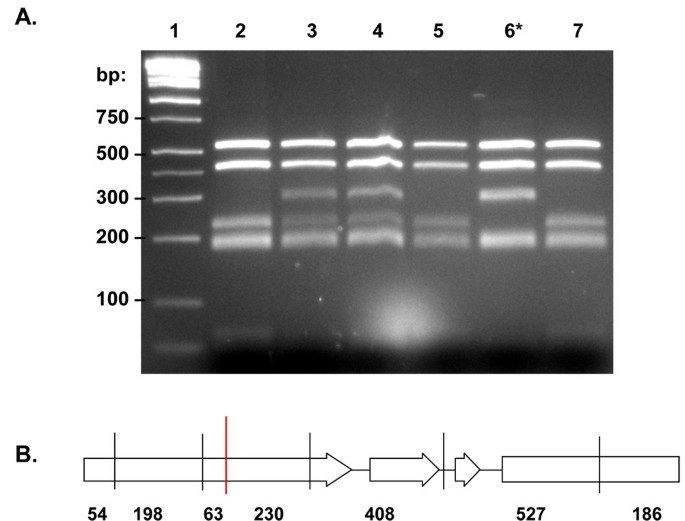
在多倍体(如花生)耕作之前,有必要确定拷贝数并对任何感兴趣的基因进行分子表征,因为大多数基因存在多个拷贝。多个同源序列的共扩增可能会导致TILLING凝胶上的片段过多,难以鉴定突变。此外,当发现突变时,有必要知道哪个基因副本被改变了。花生是一种异源四倍体,编码Ara h 2两种亚型的基因是同源的,代表来自二倍体祖先的同源基因[9.].这两个基因的开放阅读框非常相似,主要区别是在ARA H 2.02,结果插入了12个氨基酸,其中包含一个额外的DPYSPS序列副本,这是一个已知的致敏性ige结合表位[14那15].基因特异性引物对产生1,280bp的扩增子ARA H 2.01和1,227 bp forARA H 2.02(桌子1).每个引物对仅从A-或B-亚基组中扩增一条预期尺寸,以及来自推定的祖细胞答:duranensis和A. iPaensis., 分别 [16].序列分析证实了特异性扩增(数据未显示)。
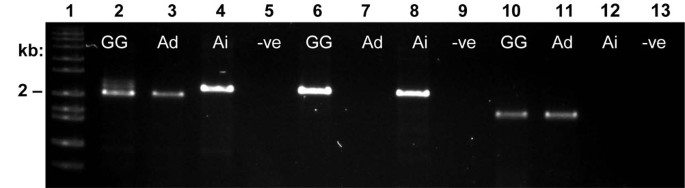
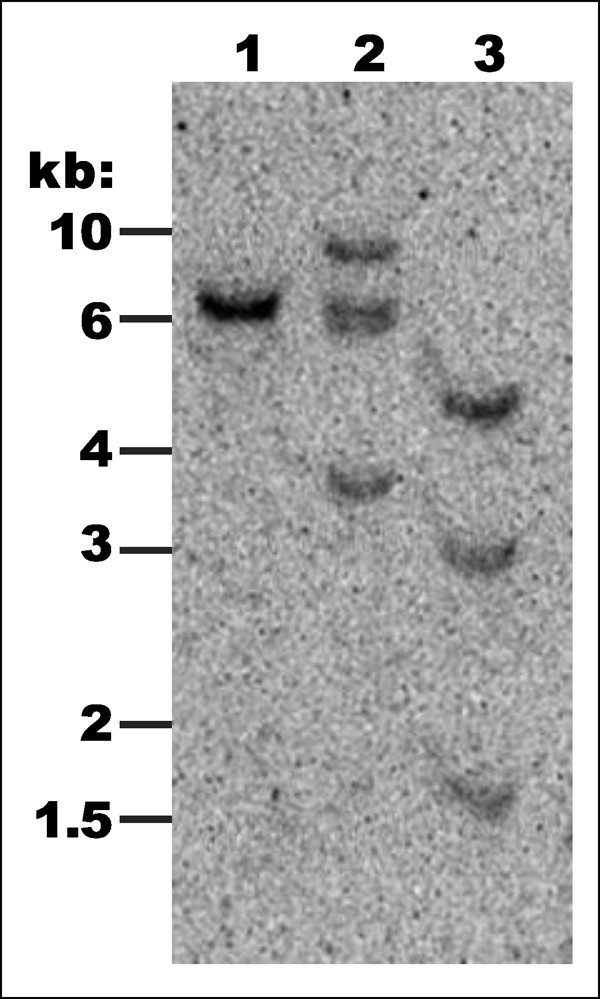
在设计PCR引物之前ARA H 1,两个基因组克隆ARA H 1在GenBank中发现。首次登录[GenBank: AF432231]由Viquez等人报道[17]并且与加入L34402的cDNA序列相同,其编码蛋白通过IUIS指定ARA H 1.0101 [2](同种型ARA H 1.01)。李等人报道了第二个基因组克隆[Genbank:Ay581852]。[18与Chassaigne等人提到的接入蛋白L38853几乎相同[19]作为同种型2.为了清楚起见,我们将把这个同种型称为ARA H 1.02,即使这不是官方的IUIS指定。PCR扩增使用引物1306和1307(表2在琼脂糖凝胶上(2,241 bpARA H 1.01.和2,031 bp forARA H 1.02;数字1).来自基因特异性PCR的扩增子是2,211bpARA H 1.01.1666 bpARA H 1.02(数字1;表格1).分析ARA H 1PCR产物来自答:hypogaea它的二倍体祖先显示了两种基因的存在答:hypogaea,但每个二倍体中只有一份副本。底漆对特定于ARA H 1.01.(1306/1308;表2)仅在答:hypogaea和A. iPaensis.(B基因组),而引物对特定于ARA H 1.02(1306/1309;表2)仅在答:hypogaea和答:duranensis(基因组;图1).使用已知的序列信息,对来自答:hypogaea是为了确认没有额外的副本ARA H 1存在于花生基因组中。基因组DNA用h在任何基因内没有切割位点的DIII在用全长探测时产生两个约6.5kb的几乎重叠的片段ARA H 1.01.探针(引物的PCR产物1306/1308)。DNA也被消化了生态RI,每个副本都有一个切割网站ARA H 1.Southern印迹分析显示了四个碎片,两个来自每个家庭的碎片,如预期的那样。最后,DNA被切割了日月光半导体我,削减ARA H 1.01.(第二个内含子内的两个相邻的切口),但不是ARA H 1.02.正如预期的那样,产生了三个片段(图2).生态携带的Ri消化质粒ARA H 1.01.或ARA H 1.02也装载为控制;探针认识到基因的两份副本(数据未显示)。
耕种的另一个目标,δ12- 脂肪酸去饱和酶基因Ahfad2.在Jung等人的研究中已被描述[12],lópez等。[20., Patel等[21].该基因也存在于两份副本中,每个亚组答:hypogaea.基因序列在两者之间高度保守,除了插入19 bpAhFAD2A(或删除AhFAD2B),起始密码子上游80 bp。利用这种indel的基因特异性引物序列产生大小几乎相同的扩增子:1228 bpAhFAD2A1221 bpAhFAD2B(桌子1).
花生耕种人口和突变频率
使用甲磺酸甲酯(EMS)和一种用二乙基硫酸盐(DES)产生几种群体。选自诱导实验的诱变和治疗时间的浓度,得到30%-50%的种子萌发。从DES治疗的m2为所有六个基因筛选人口,352种植物,没有检测到突变。在该研究中测试了两个EMS诱变处理,1.2%4.5小时,12小时,0.4%。共有3,420个EMS处理的m2对所有6个基因的植株进行筛选(7630 bp/株;表格1).检测到二十七个SNP并确认。EMS的整体突变频率为1 SNP / 967 KB。对于4.5小时,突变率为1.2%EMS为1 snP / 1,067 kB(979株)。突变频率为0.4%EMS 12小时的突变频率在1 snP / 931 kB(2,441株植物)下略高,但这种差异可能不显着。正如EMS诱导的过渡所预期的那样,大多数核苷酸变化为G至A或C至T.发现了几种异常突变AhFAD2A和AhFAD2B,这可能不是EMS处理的结果(表3.).如果是这样,那么平均突变频率将是1 SNP/1186 kb。
ARA H 2.突变
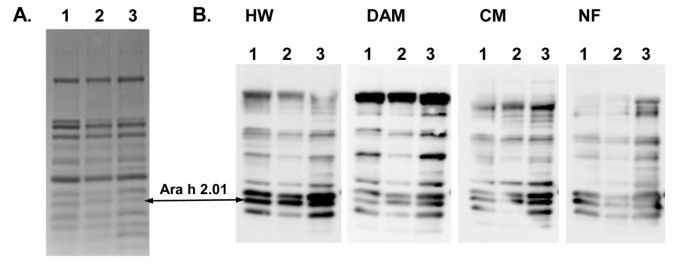
共鉴定出9个SNPsARA H 2.01和五个ARA H 2.02.确定的前两个氨基酸变化ARA H 2.01第20-6行(L49F)和第37-4行(R55H;表3.).第37-4行实际上在这个基因中有两个核苷酸变化,但其中一个是沉默的。这两种突变在M中得到证实3.和M4.几代使用耕种。来自M.的DNA3.或者4.单独分析个体并与野生型DNA混合。当在混合样品中检测到SNP但不在相应的未混合样品中时,鉴定了纯合子。纯合突变体允许在种子提取物中的改变的蛋白质上测试IgE结合。来自纯合的总蛋白质萃取物4.以抗ARA H 2鸡多克隆抗体测量的,20-6和37-4的线被标准化为加载等量的ARA H 2.01,并从4例花生超敏反应患者中测试与血清结合(HW,Dam,cm和nf)。IgE-Immunoblot在花生品种Georgia Green(GG)中存在的天然ARA H 2.01之间没有差异[22]通过耕地,在20-6和37-4行中检测到ARA H 2.01等位基因变体(图3.).虽然突变在品种tifrunner中产生[23] Ara h 2.01蛋白在两个品种间无明显差异。
发现了四种更沉默的突变ARA H 2.01,其中一个与37-4线中的无声突变相同。还鉴定了另一种氨基酸变化(82吨)ARA H 2.01.确定了三种氨基酸变化ARA H 2.02,但其中两个(D 70 N)相同(表3.).这种变化发生在第二个DPYSPS基序中,这是一个已知的过敏原表位[14那15].第三个氨基酸变化(R 62 Q)也位于过敏表位内,就在第一个DPYSPS主题之前(附加文件1).因为纯合子尚未回收,但尚未测试这些突变蛋白结合IgE的能力,尽管这些看起来是降低ARA H 2.02的过敏性的承诺候选者。还在起始密码子上游有315bp的突变ARA H 2.02;然而,它似乎不在任何重要的启动子元件内。
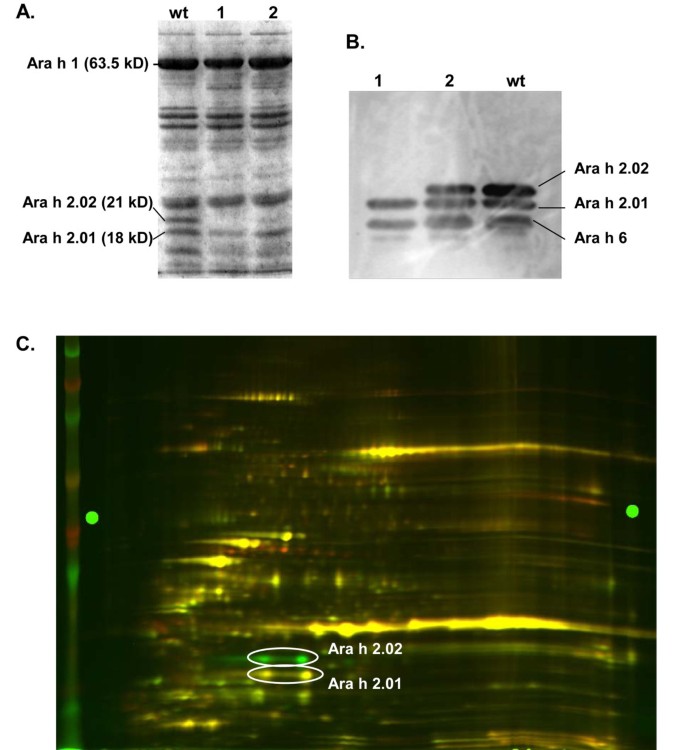
最后,在起始密码子中识别了g到过渡ARA H 2.02.下游ATG超出框架,因此预期蛋白质淘汰赛。两米3.回收种子,从每个蛋白质分析中取出一个小芯片,种植种子。两种种子都含有表型正常的植物。种子蛋白提取物的SDS-PAGE分析证实,其中一种种子确实缺少21kD带,其代表ARA H 2.02蛋白[9.[因此突出的突出(图4).另一种种子Ara h 2.02含量降低;DNA序列分析(数据未显示)证实该植物是杂合子。Western blot分析(图4B.)也证实了Ara h 2.02蛋白在纯合突变体中缺失。通过2d差异凝胶电泳(2d DIGE)进一步分析证实,这两种Ara h 2.02异构体在羧基端有两个氨基酸的截断[24],在纯合的突变线中缺失(图4C.).
ARA H 1突变
在最长的扩增子中,ARA H 1.01.(2,211bp),由于背景和片段长度,Li-Cor凝胶中的来自IRDYE通道的信号有时不可见,但是通过测序验证了从单通道信号识别的SNP。已经确认了四种突变ARA H 1.01.(桌子1)其中一个是在bp位置593处的C到T转换,是沉默的,但其他三个被预测会引起氨基酸变化:R333W、P405L和E437K(表1)3.;附加文件2).在333位的色氨酸变化的精氨酸在表位12中位于表位12 [25].只有一个突变被证实ARA H 1.02;通过C至T突变在BP位置304处产生过早的止挡密码子。这预计将导致102个氨基酸的截短蛋白质(第133线;附加文件2)所有这四种非沉默突变都已在M3.耕种。开发了一种帽(切割的扩增多晶晶序列)标记以检测ARA H 1.02在成功几代截断突变体。野生型扩增子包含六个声波测井I位点,其中一个在突变体中被删除4.生成(图5.).
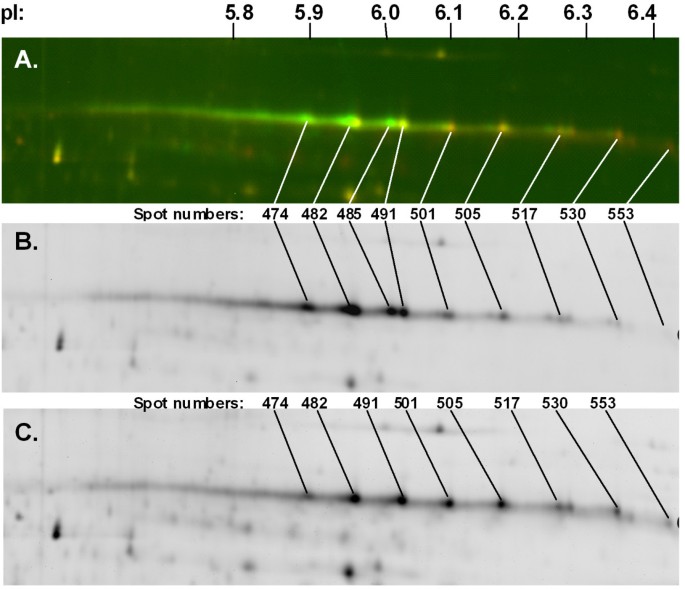
两种Ara h 1蛋白在SDS-PAGE上均显示为约63.5 kD的厚条带[26].虽然这两个基因编码的蛋白质大小略有不同,但我们无法用一维电泳来分辨它们。因此,我们尝试通过2D SDS-PAGE和2D- dige来确认纯合子种子中是否存在该蛋白ARA H 1.02截短突变体。来自2-D页面和2-D Western blot(附加文件3.)不可能仅解决两个不同的ARA H 1同种型,基于ARA H 1的已发表的2-D凝胶分析的预期结果[19].Ara h1的两种亚型会产生多种翻译后蛋白修饰(即各种切割产物或糖基化)。然而,在代表Ara h 1的pI范围为5.9 ~ 6.4的点组中,Cy3(野生型)和Cy5(突变型)的相对信号强度存在一定的差异。从这些数据不可能得出结论,Ara h 1.02亚型已完全消除。然而,定量分析2d DIGE突变体和野生型凝胶显示,三个pI 5.9-6.0点的强度代表Ara h 1(图6A,斑点474,482,485中的斑点474,482,485减少2.4-2.6倍,但具有较高PI的其他似乎增加(1.5-3.5可折叠4.),尽管这些亚型在野生型和突变型中都不如低pI亚型丰富。此外,482和485/491点在野生型中以双峰形式出现(图6B.)在突变体中出现单个斑点(图6C.),表明突变体中确实消除了几种蛋白质产品。
Ahfad2.突变
在每个中发现一个沉默的突变AhFAD2A和AhFAD2B,并发现一个预测的氨基酸变化(p 254l)AhFAD2A.所有三种这些突变是C至T过渡,典型的EMS诱导的突变。在这些不是典型的基因中也鉴定了几种突变:α插入,观察到两次AhFAD2B,三个相同的a到g突变AhFAD2A(桌子3.).这对于ems诱导的突变来说是不寻常的,但这些突变的位置和频率可能是最有趣的。的A-insertionAhFAD2B在起始密码子之后发生442bp,导致架构,并且可能导致由于过早的止脚密码子导致截短的蛋白质(81-4线;附加文件4.).在两种不同的m中鉴定出这种突变2植物在我们的耕种人口中。使用帽标记[27],这种突变已被证明是稳定的遗传3.从我们的耕种突变体中衍生的一代(数据未显示)。在AhFAD2A,三个不同的m2发现植物含有相同的突变,在起始密码子之后的448bp下的A至g转变。预计这是为了将150位从天冬酰胺转变为天冬氨酸的氨基酸(第4-3线;附加文件4.).
讨论
在耕种人口,如高粱等二倍体(高粱双色(L.)Moench)[28] 和莲藕[29,表型突变体经常被观察到。相比之下,在田间或温室栽培的M2这项研究中的花生植物,很可能是由于多倍体引起的遗传缓冲,类似于在耕种人口中观察到的四倍体和六倍倍麦片(普通小麦l .) [30.]在EMS诱变的六倍体小麦中,报道了1个SNP/24kb的突变频率,在四倍体小麦中报道了1个SNP/40kb的突变频率[30.].在本研究中观察到的花生的突变率远低于报道的小麦的突变率,低于拟南芥(1 SNP /〜300 KB [4.]),或大多数豆类,包括大豆(1 SNP/140-550 kb,视处理而定[8.])和豌豆(豌豆l .;1 SNP / 669 KB [31];1 SNP / 200 KB [32])。它类似于或高于大麦的一些人群(Hordeum Vulgare.L.;1 SNP/2500 kb[33],1 SNP/1000 kb[34])和米(1 snp / 1000 kb [35])。与大麦和水稻一样,通过使用不同的基因型、处理条件或诱变剂的选择,突变密度可能会得到改善[6.那36].在DES-诱变群体中没有检测突变,即使这种化学物用于回收花生的高油酸突变体[37].在本研究中,浓度为0.25%的孵育时间为4.5小时,与Moore [37](15分钟为1.5%)。随着4.5小时的较长孵育时间,浓度的浓度大于0.5%。
IgE-Immunoblot在20-6和37-4线中检测到野生型ARA H 2.01和ARA H 2.01等位基因变体之间没有差异(图3.)尽管这两种变化都会影响已知的IgE表位[14那15].尽管在这两个等位变异中没有检测到IgE结合的减少,但已经证明,这种蛋白质的一个小变化确实可以产生预期的效果。在最近的一项研究中,Ramos等人[38在加入中,鉴定了一种天然存在的变体(在73的位置变化)答:duranensis与野生型Ara相比,IgE结合降低56-99% h 2.01。Ara h 1.01中333位的精氨酸到色氨酸的变化属于抗原表位12 [25].尽管这种残基不太可能是IgE结合的关键[25],另外两个氨基酸的变化不在已知的表位内,在对这些蛋白进行检测之前,不能完全排除IgE亲和力降低的可能性。
这ARA H 1.01.和ARA H 1.02蛋白质的基因代码分别为71.3和70.3kd的预测尺寸,但从种子中提取的成熟蛋白在SDS-PAGE上显示为单个63.5kd带[26].纯化蛋白质的N-末端氨基酸序列与预测的N-末端序列不匹配;而是它位于下游的78或84个氨基酸,取决于同种型[39那40].这些前78或84个氨基酸以及包含的25个氨基酸信号肽在翻译后的加工过程中被切断。53或59个氨基酸切割肽含有七种半胱氨酸中的六种半胱氨酸[40]三个过敏症表现[41[假设是否以形成与植物抗真菌肽相似的稳定构象的二硫化物桥[40].在我们的ARA H 1.02截断突变体,截短发生裂解位点的下游,可能使裂解肽完好无损。是否仍然可以看出裂解产物是否在突变体的种子中产生和稳定。
先前描述的突变等位基因AhFAD2B在起始密码子后包含一个a插入442 bp,导致帧移,并可能由于过早终止密码子而导致蛋白质被截断[20.].此突变等位基因已在多种独立衍生的品种中报道,该品种具有高油酸至亚油酸比(高O / L),很可能是由于AHFAD2B的不活动[27].在两个不同的m中鉴定了相同的突变2我们分蘖群体中的植物。虽然这些种子是在USDA-ARS计划启动高O/L性状的广泛育种之前产生的,但该突变等位基因可能在分蘖群体的源种子中以较低频率存在。此外,已报告该突变等位基因的独立世代在中国和美国[27].更令人惊讶的是,三个不同的M2植物被发现含有一种退化到野生型等位基因AhFAD2A而TILLING群体亲本‘Tifrunner’则具有突变等位基因。预计这种逆转将使150位的氨基酸从天冬酰胺转变为天冬氨酸,并恢复去饱和酶的功能。在大多数奔跑型花生品种中,AhFAD2A蛋白被认为是不活跃的,因为在150位置存在天冬酰胺残留[42].天冬氨酸残基可能是酶的活性位点的重要组成部分,并且在来自其他植物的脂肪酸去饱和酶中高度保守,包括答:duranensis,AhFAD2A可能是衍生的[13].基于对美国花生种质收集的迷你核心的调查,Chu等人。[42[发现天冬氨酸残渣还似乎在西班牙和瓦伦西亚市场类型的花生中保存,但发现不活跃的等位基因是弗吉尼亚州和跑步者市场的常见(75%)。在我们三个独立的耕作突变体中,天冬酰胺已被突变回天冬氨酸,最可能恢复AHFAD2A的功能。在重组AhFAD2A蛋白中,通过定点诱变,Bruner等人将天冬氨酸恢复为150。[43]表明它的全功能确实恢复了。这两个突变的频率和性质都是由EMS诱导的非典型的,包括本研究中观察到的其他突变。目前尚不清楚这些突变是否是由于EMS治疗,折叠或遗传杂质在起始种子中,但后者似乎是最可能的解释。如果是这种情况,则在特定基因座的遗传纯度评估可以是另一用于基于不匹配的突变检测的用途。
结论
这些实验代表了低过敏性花生发展的初步步骤。由于花生过敏原的遗传变异有限,必须通过诱变才能产生变异。我们已经证明TILLING是一种有用的技术,用于筛选花生诱变群体诱导的过敏原基因变化。当鉴定出多个IgE结合降低的种子贮藏蛋白,或发现更多的敲除突变时,下一步将是协同育种工作,将这些突变等位基因结合到一株植物中。在育种过程中,TILLING标记、CAPS标记或更有效的SNP检测可以作为跟踪这些等位基因遗传的工具。花生的耕作几乎可以扩展到任何基因,并可以用于帮助修改其他性状,如抗病性、耐受性、早熟,或如本研究所示,种子的营养特性。
方法
Southern Blot分析ARA H 1
从花生(arachis hypogaea.L.)CV。格鲁吉亚绿色[22]使用Sharma等人的Deae-Cellulose的技术。[44].用20微克纯净的基因组DNA消化过夜日月光半导体我生态RI,或h然后将其装载在0.7%琼脂糖凝胶上,并在45 V的TBE缓冲液中电泳约9小时。生态ri -酶切pCR-4 TOPO质粒(Invitrogen, Carlsbad, CA)携带其中之一ARA H 1.01.或ARA H 1.02(引物分别为1306/1308和1306/1309;表格2)也装入相邻的车道中作为阳性对照。使用碱性转移方法将DNA转移至Genescreen Plus尼龙膜(Perkin-Elmer,Boston,MA)过夜[45].用全长基因组片段探测膜ARA H 1.01.,从携带该片段的质粒中进行PCR扩增。探针用α标记32P-dCTP使用随机引物DNA标签试剂盒(罗氏,印第安纳波利斯,IN)。使用Sephadex G-50 (Sigma, Saint Louis, MO)去除未合并标签。杂交和洗涤条件由Sambrook和Russell描述[45].最终洗涤在65℃下进行15分钟。在0.5×SSC缓冲液中(75mM NaCl,柠檬酸钠,pH7.0),0.1%SDS。通过暴露于储存磷光体(Amersham Biosciences,Piscataway,NJ),通过暴露于储存磷光体筛(Amersham Biosciences,Piscataway,NJ)来观察印迹,然后使用风暴840成像系统(Amersham Biosciences)扫描。
突变花生群体
甲磺酸乙酯(EMS)或二乙基硫酸二乙酯(DES)治疗用于诱导花生品种“Tifrunner”中的突变[23].种子在自来水中吸收10-12小时。然后用诱变水溶液替换自来水。测试三种诱变处理:0.4%EMS,12小时,1.2%EMS 4.5小时,或0.25%的4.5小时。将种子浸泡在旋转振荡器上的2L Fernbach瓶中的诱变溶液中,然后在去离子水中洗涤三次。(收集洗奶洗涤)。然后将种子在自来水中漂洗过夜。他们1种子被种植在田里,从每一株植物中收获一个豆荚,产生M2人口。m2种子种植在田野或温室,而m3.从他们那里收获种子来创造永久的耕作人口。
由于种子可用性有限,整个种群将不会分布,尽管可以筛选特定的突变基因和单个品系的分布。
DNA分离和耕种量化
从个体m收集幼叶组织2使用液氮冷冻植物,并在-80℃下储存或直接在96孔收集板中冻干。然后通过用三到四个3mm的3mm不锈钢研磨球在2-ml平底微量离心管中进行涡旋,或使用Genogrinder 2000(OPS Diagnostics LLC,BridgeView,NJ),设置为500英寸/分钟20秒(液氮 - 冷冻组织),或1分钟(冻干组织)。根据制造商的说明,使用DNeasy 96植物套件(Qiagen Inc. USA,Valencia,CA)提取基因组DNA。通过荧光压(Packard / Perkin-Elmer,Waltham,MA)微孔板读卡器,通过Picogreen(Invitrogen,Carlsbad,Ca)或Hoechst 33258染料,通过荧光测定量化DNA。还在琼脂糖凝胶上运行纯化DNA样品以验证质量。将个体DNA样品稀释至5ng /μl的工作浓度。然后单个DNA样品在96孔格式中汇集四倍。为了验证单个突变体,来自“Tifrunner”的基因组DNA作为对照。
底漆设计和PCR
自从ARA H 2.基因较小且无内含子,利用这两个基因上游区域的差异设计TILLING基因特异性引物(引物815和816)。根据GenBank中已有的序列信息,设计引物1306和1307对该基因进行扩增ARA H 1.在开放阅读框架的3'末端附近的印度允许我们设计特定基因的引物1308(ARA H 1.01.)和1309(ARA H 1.02).引物序列1055(AhFAD2A)和1101(AhFAD2B)利用起始密码子上游的indel80bp以扩增一种特定基因拷贝。这些引物与Patel等人使用的引物AF19和BF19相同。[21].对于具有IRDYE标记的引物的扩增,优选较长的寡核苷酸,因此设计引物1458,1459和1460。本研究中使用的所有引物序列如表所示2.
因为花生DNA高度复杂,所以第一轮未标记的PCR用于增加随后标记的PCR的靶序列的浓度。基于可用的序列信息和启动位点的适用性,设计第一轮PCR的引物以扩增两个副本ARA H 2.,两份ARA H 1,或一个特定的副本Ahfad2..第一个PCR在含有10ng GDNA的25μl最终体积中进行,0.5 U跳动Taq.DNA聚合酶1×PCR缓冲液(Sigma,Saint Louis,Mo),0.2mm各数据,DCTP,DGTP和DTTP,各个正向和反向引物的0.2μm,在下列条件下:94℃,1分钟;然后在94℃下进行8个循环35秒,58℃,35秒(-1°C /循环),72℃为100秒。接触循环后,35秒的35秒,50℃,72℃为100秒,达到30℃,72℃的最终延伸7分钟,达到30℃,72℃。使用基因AMP 9700(Applied Biosystems,Carlsbad,Ca)或PTC-200(MJ Research,Waltham,MA)热循环仪进行反应。
从第一PCR产物的1:40稀释的等分试样(2μl)用作第二轮PCR的输入,在10μl终体体积中进行,每次DNTP 0.25毫升,0.25 u exTAQ HS DNA聚合酶(Takara BioInc,Shiga,Japan)与Irdye标记的底漆(MWG Biotech,Huntsville,Al),旨在专门扩增一种基因拷贝。将标记和未标记的引物(100μm股票)以3份IRD-700标记的5'底漆的比例混合到鸡尾酒中:2份未标记的5'底漆:4份IRD-800标记的3'底漆:1部分未标记3'底漆。引物鸡尾酒的浓度,PCR缓冲液和MgCl2针对每个单独的基因进行了优化。触控PCR在PTC-200热循环仪(MJ研究,Waltham,MA)中进行如下:在95℃下变性2分钟,然后在94℃下进行6个循环30秒,58°C为30秒( -1°C /循环),温度斜坡+ 0.5°C /秒至72°C 80秒;然后将45℃的45℃持续30秒,52℃,温度斜坡+ 0.5℃/秒至72℃,80秒。然后将其在72℃下最终延伸7分钟。PCR立即接下来是异络体形成步骤:在99℃下变性10分钟,70℃下的70℃循环20秒,减小0.3℃/循环,最终保持在4℃。
芹菜汁提取物(CEL1核酸酶)的制备
含有Cel1核酸酶的芹菜汁提取物(CJE)由Till等人的纯化方案制备。[46]使用质粒缺口法检测核酸内切酶活性和浓度,如下所示:200 ng圆形质粒与10μl CJE稀释液在1×CELI缓冲液(10 mM MgSO)中孵育4.,10mM HEPES,10mM KCl,0.02%Triton X-100,0.002%的牛血清白蛋白),最终体积为20μL。在45℃温育15分钟后,将样品置于冰上,加入10μl0.15MEDTA以停止反应。在1%琼脂糖凝胶上分析消化产物。将CJE的活性与验船核酸酶(Transgenomic,Omaha,Ne)进行比较,以前通过Ecotilling检测到的已知突变体[38那47].
突变筛选
PCR扩增后,样品(第二次PCR的5 μl)在含有0.03-0.06 μl cce、总体积为10 μl的1 × CEL1 Buffer中消化,在45℃下孵育15 min,如Till等[46].为了停止反应,每个样品加入5μl0.15MEDTA,同时将样品保持在冰上。使用Sephadex G-50(Sigma,Saint Louis,Mo)清洁样品,使用45-μl多屏幕装载机(Millipore,Billerica,MA)均匀装入96孔MultiScreen-HV滤板中,按照制造商的说明。将样品收集在夹板中,转移到96孔PCR板中,并在ISS110速度VAC离心蒸发器(Thermo Savant,Milford,MA)中干燥。将干燥的样品重悬于8μl甲酰胺负载缓冲液(37%甲酰胺,3.75mM EDTA pH 8,00.0075%溴苯酚蓝)中,然后加热至80℃,7分钟,然后加热至92℃,2分钟[30.].然后可以在4℃下在暗中储存样品几天直至分析。在6.5%聚丙烯酰胺凝胶上以1×Tbe加载样品(0.8μl),在Li-Cor 4300 DNA分析仪(Li-Cor Biosciences,Lincoln,Ne)上以1500V,40mA,30m,在45℃下电泳。.目视分析使用Adobe Photoshop(Adobe Systems,Inc,San Jose,CA)和Gelbuddy [48].推测的突变是通过在700和800通道中出现的片段来识别的,其大小加起来等于全长PCR产物。因为最初筛选的DNA是四倍的,然后对每一个个体进行野生型(Tifrunner)筛选,以确定突变体。
使用TOPO-TA cloning Kit for Sequencing (Invitrogen, Carlsbad, CA),按照说明书将pcr扩增的基因或基因片段克隆到pCR4-TOPO克隆载体中,然后用M13正向和反向引物测序,确认突变。突变频率估计为确定的突变总数除以筛选的碱基对总数[(扩增子大小- 200 bp) ×筛选的个体)]。对于每个扩增子,减去200 bp以调整TILLING凝胶图像顶部和底部的100 bp区域,这些区域很难分析[5.].
SDS-PAGE, Western Blots和ige结合分析
如Koppelman等人所述进行种子蛋白萃取。[3.].使用Bio-rad蛋白质测定试剂盒(Bio-rad,Hercules,Ca)和牛血清白蛋白(BSA)作为标准进行蛋白质量化。每条泳道加载10微克总蛋白质,并在12%SDS-PAGE凝胶中分离。然后将蛋白质在30V下电过夜以PVDF膜,0.2μm(Bio-rad)。在PBS-T(137mM NaCl,2.7mM KCl,1.4mm KH中,用5%非脂肪干乳封闭膜。2阿宝4., 4.3 mM Na2HPO.4., 0.1%吐温-20)在室温下轻轻搅拌1小时。Western blot分析:阻断膜后,用鸡抗ara h 2或抗ara h 1(1:8 000稀释)一抗检测1 h,然后用二抗鸡抗辣根过氧化物酶(HRP;1:100,000;Sigma,圣路易斯,MO)在PBS-T中,添加2%脱脂奶粉1小时。为了进行IgE结合分析,从4个被记录为花生过敏的个体(HW, DAM, CM和NF)的血清中获得了阳性食物挑战或令人信服的花生过敏史。过敏个体的血清由阿肯色州医学科学大学(University of Arkansas for Medical Sciences, Little Rock, AR)收集,并根据机构审查委员会的规则和规定获得批准。用1:20稀释的四种血清孵育的印迹在4°C下杂交过夜。每次抗体孵育后,用PBS-T冲洗所有膜3次,5分钟。ECL Plus检测试剂(Amersham Biosciences)用于化学荧光检测,随后在Storm 840成像系统(Amersham Biosciences)上进行扫描。IgE结合分析采用Bio-Rad密度仪对Ara h 2.01蛋白进行相对定量,所有样品归一化至野生型Ara h 2.01/10 μg总蛋白量。进一步使用100 ng Ara h 2.01归一化总蛋白进行IgE免疫印迹分析。
2-D页面和Dige
Ara h 2.02和Ara h 1.02疑似纯合子敲除突变体采用差异凝胶电泳(2d DIGE)进行二维蛋白分析,主要依据Chu等人[4.].使用p解决了用于分析17-19 kd ara h 2(野生型 - cy3;突变体 - cy2)的标记的蛋白质样品一世3-10固定pH梯度(IPG)条。分析~ 65 kD Ara h 50μg Cy3-labeled野生型,Cy5-labeled突变,Cy2-labeled野生型和突变蛋白提取物的混合物混合,集中在一个24厘米pH值5.3 - -6.5在10000 V 121 kVhr IPG地带运行第二个维度Tris-glycine 10% sds - page凝胶。使用Typhoon 9400可变模式成像仪(GE Healthcare)检测标记斑点,并使用samspot Progenesis(非线性动力学)进行数字图像分析。
作者的信息
当前地址:
Jek - USDA-ARS作物遗传和繁殖研究单位,TIFTON,GA 31793,USA
MLR - NIDERA S.A, Venado Tuerto生物技术部门,圣达菲CP2600,阿根廷
AB - 长凳生物技术,VAPI,Gujarat,印度
参考
- 1.
Palmer K,Burks W:花生过敏的当前发展。Curroin Allergy Clin Immunol。2006,6:202-206。10.1097 / 01.ALL.0000225161.60274.31。
- 2.
国际免疫社会联盟:过敏原命名。[http://www.allergen.org/index.php.]
- 3.
Koppelman SJ,Vlooswijk Raa,Knippels LMJ,Hessing M,Knol EF,Van Reijsen Fc,Bruijnzeel-Komen Cafm:主要花生过敏原ARA H 1和ARA H 2在花生品种赛道,西班牙语,弗吉尼亚州和瓦伦西亚,培育在世界的不同地区。过敏。2001,56:132-137。10.1034 / J.1398-9995.2001.056002132.x。
- 4.
zhu Y, fastinelli P, Ramos ML, Hajduch M, Stevenson S, Thelen JJ, Maleki S, Cheng H, Ozias-Akins P:减少IgE结合和不促进曲霉菌曲藤通过同时沉默花生中的阿糖胞苷h 2和阿糖胞苷h 6实现真菌生长。农业食品化学杂志,2008,56:11225-11233.10.1021/jf802600r。
- 5。
格林EA,Codomo CA,泰勒NE,参见Henikoff JG,直到BJ,雷诺SH,恩斯LC,Burtner C,约翰逊JE,Odden AR,Comai的L,参见Henikoff S:从大规模的反向遗传筛选化学诱导的突变的频谱在里面拟南芥.中国生物医学工程学报。
- 6。
直到BJ,Cooper J,Tai Th,Colowit P,Greene Ea,Henikoff S,Comai L:通过耕地发现水稻中的化学诱导突变。BMC植物BIOL。2007年,7:19-10.1186 / 1471-2229-7-19。
- 7。
直到BJ,Reynolds Sh,Weil C,Springer N,Burtner C,Young K,Bowers E,Codomo Ca,Enns LC,Odden Ar,Greene Ea,Comai L,Henikoff S:通过耕地发现玉米基因中的诱导点突变。BMC植物BIOL。2004,4:12-10.1186 / 1471-2229-4-12。
- 8。
Cooper JL, Till BJ, Laport RG, Darlow MC, Kleffner JM, Jamai A, El-Mellouki T, Liu S, Ritchie R, Nielsen N, Bilyeu KD, Meksem K, Comai L, Henikoff S: TILLING技术在大豆诱导突变检测中的应用。植物生态学报。2008,8:9-10.1186/1471-2229-8-9。
- 9。
赵志刚,王志刚,王志刚,等:中华绒螯蟹凝集素基因的染色体和系统发育背景花生基于基因组序列。Mol Genet基因组学。2006,275:578-592。10.1007 / s00438-006-0114-z。
- 10.
圣安吉洛AJ, Ory RL:花生酱中脂肪酸过氧化的原因和预防研究。《华中农业大学学报》,1993,5:128-133。
- 11.
Moore Km,Knauft Da:花生中高油酸的遗传。j hered。1989,80:252-253。
- 12.
Jung S,Swift D,Sengoku E,Patel M,Teuléf,Powell G,Moore K,Abbott A:栽培花生的高油e特征(arachis hypogaea.l .)I.两个编码微粒体油酰pc去饱和酶的基因的分离和鉴定。中国海洋大学学报(自然科学版)。10.1007 / s004380000244。
- 13.
Jung S,Powell G,Moore K,Abbott A:栽培花生的高油e特征(arachis hypogaea.L.)II。分子基础和特质的遗传学。Mol Gen Genet。2000,263:806-811。10.1007 / S004380000243。
- 14.
主要花生过敏原Ara H 2的免疫显性IgE结合表位的鉴定和突变分析。生物化学学报。1997,34(4):444 - 453。10.1006 / abbi.1997.9998。
- 15.
Schein Ch,Ivanciuc O,Braun W:常见的物理化学性质与花生过敏原的IgE表位的类似结构相关。JAgric Food Chem。2005,53:8752-8759。10.1021 / JF051148A。
- 16。
Ozias-Akins P, Ramos ML, Chu Y:婴儿配方奶粉以外的低过敏性食品。食物过敏:理解、治疗和预防。编辑:Maleki S, Burks AW, Helm RM2006, Herndon, VA: ASM出版社,287-308。
- 17。
Viquez OM,Konan KN,Dodo HW:一个主要花生过敏原基因的基因组克隆的结构和组织,ARA H 1.Mol免疫酚。2003,40:565-571。10.1016 / J.MOLIMM2003.09.002。
- 18。
李H-G,王L,张Y-S,林X-D,Liao B,Yan Y-S,Huang S-Z:基因的克隆和序列Ahy -β编码花生征冈犬的亚基。植物SCI。2005,168:1387-1392。10.1016 / J.Plantsci.2004.09.028。
- 19。
Chassaigne H, Trégoat V, Nørgaard JV, Maleki SJ, van Hengel AJ:结合荧光二维差示凝胶电泳、Western blotting和Q-TOF质谱技术,分析和鉴定主要花生过敏原。中国蛋白质组学杂志。2009,31(6):593 - 598。10.1016 / j.jprot.2009.02.002。
- 20。
Lópezy,nadaf hl,smith od,connell jp,reddy,fritz ak:δ的隔离和表征12-花生脂肪酸去饱和酶(arachis hypogaea.L.)并在西班牙市场型线路中寻找高油e特征的多态性。Al Appl Genet。2000,101:1131-1138。10.1007 / S001220051589。
- 21.
Patel M,Jung S,Moore K,Powell G,Ainsworth C,Abbott A:高油eee花生突变体是由螨虫插入到的FAD2基因。中国科学(d辑),2004,28(6):591 - 598。10.1007 / s00122 - 004 - 1590 - 3。
- 22.
分公司WD:'格鲁吉亚绿色'花生的注册。农作物科学。1996年,36:806-
- 23.
Holbrook CC,Culbleath AK:'Tifrunner'花生的注册。J工厂注册。2007,1:124-10.3198 / JPR2006.09.0575CRC。
- 24.
花生过敏原蛋白Ara h 2基因的克隆与表达分析。中国生物医学工程学报。2010,19(4):491 - 498。
- 25.
Shin DS,Compadre Cm,Maleki Sj,Kopper Ra,Sampson H,Huang Sk,Burks Aw,Bannon Ga:ARA H 1上的IgE结合位点的生化和结构分析,一种丰富高,过敏性花生蛋白。J Biol Chem。1998年,273:13753-13759。10.1074 / JBC.273.22.13753。
- 26.
Burks Aw,Williams LW,Helm RM,Connaughton C,Cockrell G,Obrien T:鉴定Atopic-Dercatis和阳性花生挑战患者的主要花生过敏原Ara-H-I。j alerergy inlmunol。1991,88:172-179。10.1016 / 0091-6749(91)90325-i。
- 27.
两个等位基因ahfad2b.控制栽培花生的高油酸性状。农作物科学。2009,49:2029-2036。10.2135 / cropsci2009.01.0021。
- 28。
Xin Z,Wang Ml,Barkley Na,Burow G,Franks C,Pederson G,Burke J:应用基因分型(耕种)和表型分析,以阐明化学诱导的高粱突变群中的基因功能。BMC植物BIOL。2008,8:103-10.1186 / 1471-2229-8-103。
- 29。
Perry Ja,Wang TL,Welham TJ,Gardner S,Pike Jm,Yoshida S,Parniske M:耕作逆向遗传工具和网络可访问的豆类突变体系列莲藕.植物生理学报。2003,31(4):591 - 598。10.1104 / pp.102.017384。
- 30.
基于反基因、非转基因的小麦耕作改良方法。生物工程学报,2005,23:75-81。10.1038 / nbt1043。
- 31。
Triques K, Sturbois B, Gallais S, Dalmais M, Chauvin S, Clepet C, Aubourg S, Rameau C, Caboche M, Bendahmane A:表征拟南芥蒂利亚纳不匹配的特定内切核酸酶:通过豌豆耕作突变发现。工厂J. 2007,51:1116-1125。10.1111 / J.1365-313x.2007.03201.x。
- 32。
Dalmais M,Schmidt J,Le Signor C,Moussy F,Burstin J,Savois V,Aubert G,Brunaud V,De Oliveira Y,Guichard C,Thompson R,Bendahmane A:Utilldb,a豌豆电子正反转遗传学工具。基因组生物学。2008,9:R43-10.1186/gb-2008-9-2-R43。
- 33.
Lababidi S, Mejlhede N, Rasmussen SK, Backes G, Al-Said W, Baum M, Jahoor A:利用TILLING技术在Dhn位点上鉴定‘Lux’大麦突变体。植物学报。2009,32(4):457 - 461。10.1111 / j.1439-0523.2009.01640.x。
- 34.
(1)大麦正向和反向遗传的结构突变群体(Hordeum Vulgare.l .)。植物学报,2004,40:143-150。10.1111 / j.1365 - 313 x.2004.02190.x。
- 35.
Wu JL, Wu CJ, Lei CL, Baraoidan M, boros A, Madamba MRS, Ramos-Pamplona M, Mauleon R, Portugal A, Ulat VJ, Bruskiewich R, Wang GL, Leach J, Khush G, Leung H:化学和辐照诱导的籼稻IR64突变体的正向和反向遗传。植物生态学报。2005,32(5):591 - 598。10.1007 / s11103 - 004 - 5112 - 0。
- 36.
Talame V, Bovina R, Sanguineti MC, Tuberosa R, Lundqvist U, Salvi S: TILLMore,发现化学诱变大麦的资源。生物技术学报,2008,6:477-485。10.1111 / j.1467-7652.2008.00341.x。
- 37.
高油酸花生。美国专利号5945578.1999
- 38.
Ramos ML,Huntley JJ,Maleki Sj,Ozias-akins P:ARA H 2.01的抗过滤性矫形器的鉴定与表征。植物mol biol。2009,69:325-335。10.1007 / s11103-008-9428-z。
- 39.
De Jong Ec,Van Zijverden M,Spanhaak S,Koppelman Sj,Pellegrom H,Penninks AH:花生蛋白多重过敏原的识别和部分表征。Clin Exp Flatergy。1998年,28:743-751。10.1046 / J.1365-22222.1998.00301.x。
- 40
Wichers HJ,De Beijer T,Savelkoul HFJ,Van Amerongen A:主要花生过敏原ARA H 1及其切割的N-末端肽;对花生过敏原检测的可能影响。JAgric Food Chem。2004,52:4903-4907。10.1021 / JF049697O。
- 41
Burks AW, Shin D, Cockrell G, Stanley JS, Helm RM, Bannon GA:花生过敏原和豆类vicilin蛋白Ara h 1的ige结合表位的定位和突变分析。生物化学学报,2003,31(4):457 - 461。10.1111 / j.1432 1033.1997.t01 - 1 - 00334. x。
- 42。
Chu Y,Ramos L,Holbrook CC,Ozias-Akins P:Oleyl-PC-Desaturase中功能突变突变的频率(ahfad2a.)在美国花生种质收集的迷你核心。农作物科学。2007,47:2372-2378。10.2135 / cropsci2007.02.0117。
- 43。
一些花生品种自然产生的高油酸油特性是由于天门冬酰胺150突变降低了油酰pc去饱和酶活性。作物科学,2001,41:522-526。10.2135 / cropsci2001.412522x。
- 44。
Sharma Kk,Lavanya M,Anjaiah V:一种用于分离和纯化适用于分析应用的花生基因组DNA的方法。植物MOL BIOL REP。2000,18:393A-393H。10.1007 / BF02825068。
- 45.
Sambrook J,Russell DW:分子克隆:实验室手册。2001年,冷泉港,纽约:冷泉港实验室出版社,3
- 46.
直到BJ,Zerr T,Comai L,Henikoff S:植物和动物的耕种和生态化的协议。NAT协议。2006,1:2465-2477。10.1038 / nprot.2006.329。
- 47.
Comai L,Young K,Till Bj,Reynolds Sh,Greene Fa,Codomo Ca,Enns Lc,Johnson Je,Burtner C,Odden Ar,Henikoff S通过Ecotilling高效发现自然群体中的DNA多态性。工厂J. 2004,37:778-786。10.1111 / J.0960-7412.2003.01999.x。
- 48.
Zerr T,Henikoff S:使用背景信息的电泳凝胶图像中的自动频带映射。核酸RES。2005,33:2806-2812。10.1093 / nar / gki580。
确认
该工作得到了植物生物技术研究,格鲁吉亚花生委员会,花生基金会,国家花生委员会和美国农业部特定合作协议58-6435-6-050的支持。我们感谢Evelyn Perry Morgan为技术援助和叶楚有用的讨论。
作者信息
隶属关系
通讯作者
补充资料
作者的贡献
jek对南方印迹分析进行了ARA H 1,筛选和鉴定的突变体,并起草了稿件。MLR开发了本研究中使用的耕作方案,制备芹菜汁提取物和筛选和鉴定的突变体。AB辅助筛选和识别突变体。Poa是潜在教调查员,产生诱变的人群,并起草了手稿。YZ,MC和SC执行了2D页面和Dige蛋白质从敲除突变体的提取物。SM对突变体ARA H 2.01蛋白进行IgE结合分析。CCH协助产生,推进和维护诱变的人群。所有作者都读过并批准了稿件。
电子补充材料
12870_2011_867_MOESM1_ESM.PDF
附加文件1:ARA H 2.01和ARA H 2.02野生型蛋白质的序列对准和由耕地鉴定的ARA H 2突变体的野生型蛋白质和预测的蛋白质。WT为野生型蛋白序列。突变ID号用括号表示。(PDF 35 KB)
12870 _2011_867_moesm2_esm.pdf
附加文件2:Ara h 1.01和Ara h 1.02野生型蛋白质的序列比对以及TILLING鉴定的Ara h 1突变体的预测蛋白质。WT表示野生型蛋白质序列。突变体ID号显示在括号中。(PDF 71 KB)
12870_2011_867_moesm3_esm.pdf.
附加文件3:2D页面和Western印迹ARA H 1.02截断突变体。A - Sypro Ruby从野生型(Tifrunner)和纯合ARA H 1.02截断突变体中染色的种子蛋白提取物(1.5mg)的PVDF斑点。首先将蛋白质聚焦在pH5.3至6.5 IPG条带中,然后在10%聚丙烯酰胺三甘油凝胶中分离,然后倒入PVDF膜。B - 使用鸡抗ARA H 1抗体(初级)在面板7a中膜的蛋白质印迹,然后是抗鸡-HRP缀合物(二次),并通过荧光可视化。(PDF 167 KB)
12870_2011_867_moesm4_esm.pdf.
附加文件4:TILLING鉴定的AhFAD2A和AhFAD2B野生型蛋白与AhFAD2突变体预测蛋白序列比对。WT为野生型蛋白序列。突变ID号用括号表示。(PDF 42 KB)
作者的原始提交的图像文件
以下是与作者的原始提交的图像的链接。
权利和权限
开放获取本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
李国强,李国强,曾勇。等等。花生变应原减少和品质性状改良的耕作方法(arachis hypogaea.l .)。BMC植物BIOL.11,81(2011)。https://doi.org/10.1186/1471-2229-11-81
已收到:
公认:
发表:
关键词
- 氨基酸变化
- 过早停止密码子
- arachis hypogaea.
- 甲磺酸乙酯
- 花生品种