摘要
背景
在整个有机体中,从一个基因序列到它的功能,需要一种针对突变的策略,这被称为反向遗传学。逆向遗传学在现代基因组学时代是非常可取的;然而,最有效的方法通常局限于少数模式生物。之前,我们介绍了一种反向遗传策略,它具有普遍适用于缺乏成熟遗传工具的生物体的潜力。我们的TILLING(靶向诱导基因组中的局部病变)方法使用化学诱变,然后筛选单碱基变化,以发现改变蛋白质功能的诱导突变。通过建立高通量TILLING设施和将数百个拟南芥基因中的数千个点突变传递给植物生物学群落的成员,TILLING被证明是一种有效的反向遗传策略。
结果
我们证明,高通量耕种适用于玉米,这是一个具有大型基因组的重要作物植物,但目前可获得有限的逆向遗传资源。我们筛查了来自11种不同基因的1-KB段中的DNA样品的损伤池,获得来自750种花粉诱变的玉米植物的17个独立的诱导突变。靶向的基因之一是DMT102 Chormethyle基因,我们获得了预测强烈有害的三种畸形突变的等位基因系列。
结论
我们的发现表明TILLING是一种广泛适用和有效的反遗传策略。我们正在以现有的拟南芥耕作项目为模型,建立玉米公共耕作服务。
背景
在基因组测序计划正在取得迅速进展提供原料基因功能的潜力的认识,所以有效的反向遗传学策略需求越来越[1].序列信息本身可能足以考虑一个基因是感兴趣的,因为序列比较工具,检测蛋白质序列的相似性,以以前研究的基因,往往允许一个相关的功能推断。以这种方式产生的有关基因功能的假说必须得到经验的证实。基因功能的实验测定在其他情况下也是可取的,例如,当遗传间隔与感兴趣的表现型相关联时。在这种情况下,基因在一段时间内的功能可以从诱导突变的表型中推断出来。此外,基因相互作用的剖析往往需要一系列等位基因类型的可用性。然而,大多数可用的功能推断方法依赖于产生有限范围突变的技术,这些技术是劳动密集型的或不可靠的,或者仅限于已经开发出特殊遗传工具的物种[2].正如诱导突变的发现导致了正向遗传学,快速反向遗传方法的引入也会产生巨大的影响。
几种通用策略已被用于在模型生物中获得功能减少或敲除突变,包括插入突变[3.]和RNA抑制[4],已广泛用于植物。插入诱变现在在很大程度上是一个在网上拟南芥研究人员的程序,因为从T-DNA侧翼序列和转座子插入的可搜索数据库可在线获得[5].目前,RNA抑制需要大量的人工努力,但它有可能减少重复基因的表达,这在植物中特别常见,包括拟南芥。然而,由于这些技术要么依赖于农杆菌T-DNA载体进行传播,要么依赖于内源性标记系统,它们作为一般反向遗传学方法的用途仅限于非常少的植物物种。
在玉米中,大部分反向遗传资源都是利用内源的增变(木)和激活剂(AC)转座子的家庭。突变体转座元件常以高拷贝数存在,并倾向于插入或接近基因[6从而提供了大部分基因敲除和由于插入调控元件而可能丧失功能的等位基因。资源识别亩目标基因的插入包括玉米性状利用系统[7,玉米定向诱变项目[8]和光合突变体狩猎[9].此外,修改突变元素已经被设计成允许质粒拯救元素和侧翼基因组DNA进入大肠杆菌[10,11,以促进标记基因的测序,并允许生成一个插入数据库,类似于目前拟南芥可用的T-DNA数据库。激活剂转座子是低拷贝数的元素,比如亩,优先插入或靠近基因[12].大规模分离和测序交流反向遗传数据库的元素是有利的,因为可以获得单个到低拷贝数的插入。
基于诱导突变的反向遗传策略具有普遍适用性。对植物已经描述了两种这样的方法。一种是使用快中子轰击的缺失突变,这似乎是一种敲除串联重复基因的有效方法[13].另一种是耕种(靶向诱导的基因组局部病变),其中使用传统化学诱变剂的治疗导致点突变,然后使用对单核苷酸突变检测的敏感方法在利益基因中发现的点突变[14].TILLING可以提供一个包括错义和敲除突变的等位基因序列。在传统正向遗传研究中已证实的等位基因序列的效用使TILLING成为一种特别可取的反遗传策略,因为基因组序列越来越容易获得。
我们介绍了一种高通量筛选TILLING方法,基于使用错配裂解内切酶,然后使用LI-COR分析系统在聚丙烯酰胺电泳凝胶上荧光显示裂解产物[15].我们将这种方法系统化,并为一般拟南芥群落建立了公共耕作设施[16].我们的拟南芥TILLING项目(ATP)平均每1 kb筛选3000个EMS诱变的M2拟南芥植株,导致>4000个突变在>400个基因中传递。TILLING数据分析显示EMS是一种近乎理想的诱变剂,99%的情况下产生G/ c到a /T的转变>,只有较小的局部序列偏差[17].我们的分析还表明,基于高通量切割的检测方法是高效的:至少有3/4的突变被检测到,基本上所有检测到的突变都是通过测序确认的。
ATP在生产环境中证明了高通量耕作的实用性。然而,拟南芥的基因组较小,易于培养,这可能使拟南芥比大田种植的基因组较大的作物更容易进行TILL。为了确定开发的ATP程序是否可以推广,我们选择对玉米进行TILL,玉米是一种基因组比拟南芥大20倍的作物植物。我们发现,提供了有效耕作的拟南芥的基本相同的程序也可以应用于玉米,取得了类似的结果。尽管我们诱变的玉米耕作群体规模相对较小,但我们获得了有用的突变。其中包括一个染色甲基化酶基因的有前途的等位基因序列,该基因此前与非cpg DNA甲基化有关,其在拟南芥中的对等物负责表观遗传基因沉默和基因组监测。
结果
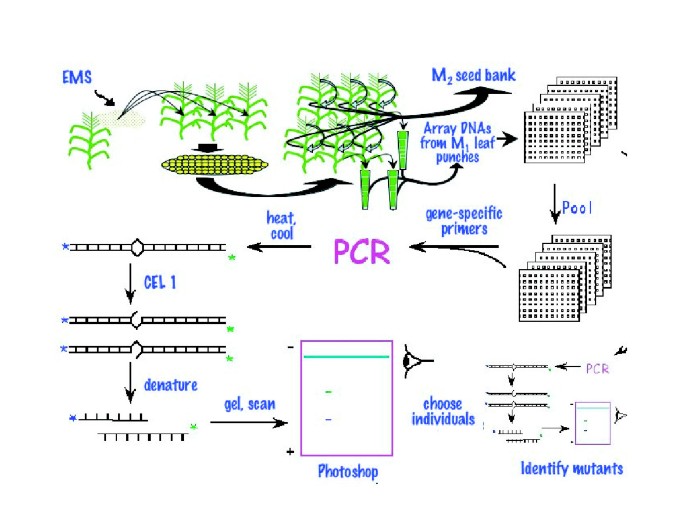
TILLING由一系列步骤组成,从参考个体的化学突变开始,到通过DNA测序确定突变碱基对[14].高通量TILLING利用CEL I错配切割酶对异质双偶,并检测电泳凝胶上末端标记的切割产物[15,18].玉米TILLING的方法是相同的,对于拟南芥,除了而非种子用EMS处理的花粉(图1).
玉米TILLING的示意图。收集新鲜花粉,用甲基磺酸乙酯诱变。然后将花粉应用到来自相同遗传背景的野生型植物丝上。由此产生的穗的种子被培育成M1代植株。这一代植株对任何诱变都是杂合的。组织从每一株M1植物或从大约。M1自交的10 M2兄弟姐妹。M3种子由10-12个M2兄弟姐妹随机交配产生。M3种子作为未来研究的种子储备。从收集的组织中提取DNA,并汇集样本,以增加筛选吞吐量。突变检测采用序列特异性引物,PCR扩增目的位点。 Following amplification, samples are heat denatured and reannealed to generate heteroduplexes between mutant amplicons and their wild-type counterparts. Heteroduplexes are cleaved using CEL I endonuclease and are visualized using denaturing polyacrylamide gel electrophoresis. See reference [34] for further details.
在本研究中,两个单独的诱变的B73玉米群体,分别是UI(384个诱变品系的M2家族)和NS(366个M1单株),以96孔的形式筛选4倍库(4个UI家族或4个NS株系)。为了弥补玉米相对于拟南芥更大的基因组大小,我们将基因组模板DNA扩增量增加了20倍;否则,所有协议和默认参数与拟南芥TILLING相同[16].我们发现14个引物中有11个获得了高质量的产品,而拟南芥获得的成功率约为90%。我们继续在750份DNA样本中对这11个基因片段进行突变筛查,发现21个点状病变(见表)1).所有21都通过测序验证。
21个病灶中,17个是ems引起的。这17个突变为G/C-to-A/T突变,与EMS的预期一致[17].其他四种病变均在同一株植物中发现(见表)1),只有1株为G/ c - a /T转变,表明该植物为非b73污染物,可能是异花授粉所致。在单个植物中,被视为多态性频率过高的污染偶尔被ATP观察到[17,19].在单个植物中检测到的推定EMS诱导的突变,除了DMT102 G878A,其中完全相同的突变是在两个不同的植物中发现;这种情况下,预计和观察到发生的基于诱导的突变的随机分布在拟南芥的时间为4%,基因组和突变的位置的分布的GC含量在片段发现[17].在17个突变中发现一个巧合与预期没有显著差异。此外,发现所有17个突变都是G/ c到a /T的转变,有效地排除了它们是自然发生的多态性的可能性:有4个可能的单碱基变化,观察到所有17个突变都符合EMS突变的期望的概率只有(1/4)。17~ 1/1010.
重要的是,每个群体产生8-9证实新的突变,大约两个突变/兆碱基的突变整体密度。考虑到一个事实,即花粉治疗mutagenizes只有一两个基因组的,而种子处理mutagenizes两者(尽管通过在后者的情况下筛选M2S,突变的四分之一被丢失+ / +分离子),估计的突变密度两个玉米种群是〜3/4的高达我们的平均每诱变基因组拟南芥。
我们之前已经证明,基于atp生成的数据分析,可以可靠地检测8倍池中的突变[17].例如,我们几乎精确地得到了预期的杂合子:纯合子比为2:1的8倍池~1900个突变,这表明1/16的检测与TILLING检测1/8没有区别。为了验证这一检测效率,我们从4个文库中筛选的750份B73 DNA样本中筛选了DMT101和DMT103的引物。在这个测试中,我们只检测到我们通过4倍池化发现的5个突变中的3个。不充分的数据质量不能解释在8倍池中缺失这两个碱基变化(UI20291和NS3471.9),因为凝胶图像是我们ATP操作中所看到的典型。因此,我们考虑到,在这个有限的测试中,5个突变中有2个未能检测到的可能性是由于池中DNA数量的变化造成的。
DNA数量变化的一个潜在来源是降解的DNA在琼脂糖凝胶分析可视化时很难准确测量。我们注意到包括NS3471.9在内的部分基因组DNA样本发生了部分降解。测量样本中DNA数量的不准确会影响到池中的标准化:池中每一株植物贡献的DNA数量的任何变化都会导致一株或多株植物的代表性减少。当来自特定植物的DNA池中DNA的数量减少时,突变检测就变得有限。认识到基因组DNA的质量可能会潜在地阻碍突变检测的吞吐量,我们寻求一种样品定量和标准化的替代方法。我们发现在3%的暗喻上运行示例®(Cambrex)琼脂糖凝胶减少了碎片的“涂抹”,从而便于量化。
虽然较低的DNA质量和不准确的归一化可以解释NS3471.9缺失碱基变化的原因,但这并不能解释缺失UI20291突变的原因,该突变来自一个明显未降解的样本。因此,我们认为样本间变异的另一个来源是对UI系列中M2同胞植株叶片的采样,而不是直接使用M1植株。UI群体的个体DNA样本是通过汇集来自M2家族的大约10个个体生成的。对于新突变,M1杂合子植株产生的M2家系为1:2:1。假设突变等位基因具有良好的生存能力和生育能力,一个大型M2家族的样本应该产生相同部分的野生型和突变等位基因。然而,一个小的M2样本可能会有偏倚,包含了太多的野生型DNA,这将阻止一个给定的目标序列包含突变在8倍的聚合DNA中被检测出来,这取决于健壮突变检测的极限。
直接证据证明来自M2家族的抽样是一个问题来自我们发现,通过我们通常的标准,将植物中发现的两种突变中发现的两种突变分别被评为纯合。花粉诱变只能产生M1杂合子,并且我们根据异质M2植物的有限取样来解释突变体的均匀性。例如,如果只有一个或两个种植的M2种子用于这些家庭,并且限制样品有利于纯合子,然后它们有时似乎在序列迹线中纯粹纯合。同样,相当数量或更多的突变将是持续的,因为野生类型将超过,导致我们错过检测8倍,但不在4倍的池中检测突变。这强调了使用来自M1花粉诱变的个体的DNA的重要性,并注意避免在DNA分离期间能够加剧降解的收集程序。通过测定对隐喻凝胶上的DNA浓度并仅取样M1代,我们应该能够池8倍而不减少检测效率。为了测试这一点,我们筛选了768 M1 W22玉米DNA(花粉诱变)的群体,其具有与上述B73样品相似的降解模式,并使用3%隐喻琼脂糖凝胶标准化。然后,我们从这些768个样品中制作了8倍和4倍的池,并在原始屏幕中用四个引物组中的四个筛选。该筛选为6 MB的总序列导致4倍和8个池中的相同3突变的独立检测(数据未显示)。因此,我们得出结论,即使是来自M1花粉诱变的样品的部分降解的DNA,例如可以从该领域收集的材料中提取,可用于耕种。
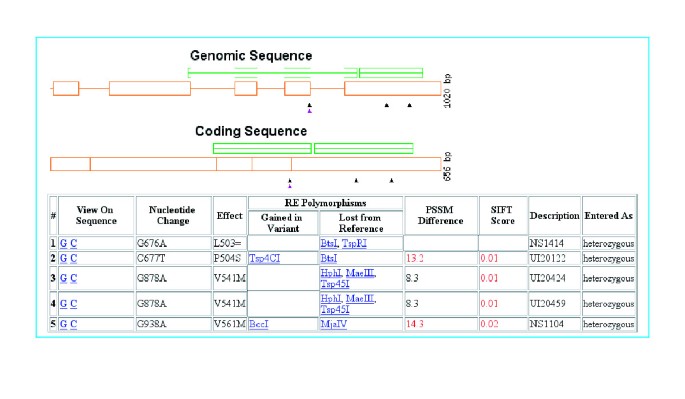
在筛选中发现的17个诱导突变分布与预期一致,其中错义10个,沉默和无截断突变7个,而在~4000 TILLed拟南芥突变中,错义突变51%,沉默突变44%,截断突变5%。考虑到大约一半的错义突变预计会对典型蛋白质造成损害[20.],我们预计,即使一个小的等位基因系列将是表型分析是有用的。事实上,我们发现这三个不同的DMT102错义突变都可能是有害的蛋白质,基于SIFT和PSSM得分的差异(图2).基于对实验突变数据的分析,SIFT算法预测有害错义突变的总体准确率约为75% [21,22]及全面的人类多态及疾病资料[23].因此,DMT102等位基因系列似乎是仅750玉米植物中筛选1-kb的区域之后基本完成。
讨论
我们已经证明,耕作是作物植株反遗传的一种有效方法。我们发现的突变密度似乎只比使用相同方法获得的拟南芥的突变密度略低。对于拟南芥,我们目前筛选了~ 2300m2植株,以获得合适的等位基因序列,平均每1.5 kb片段约有12个突变。基于这项工作,我们估计筛选~4000个玉米植株将提供一个可比较的序列([4000][17个突变][1.5-kb/1-kb]÷([11-kb][750株])= ~12个突变)。自本研究完成以来,我们已经将诱变群体的规模扩大到B73遗传背景中的~2500个M1株和W22遗传背景中的~2000个M1株,准备进行筛选,并正在准备~ 10000个M1株的群体。
为了有效的池化,池中的每个个体必须以与池中的其他成员相等的浓度表示。如果不能做到这一点,可能会导致在检测级别以下的池中表示的突变。目标是通过增加池来最大化吞吐量,同时仍然检测所有可能的突变。我们已经证明,在拟南芥中,杂合突变可以像纯合突变一样在8倍池中被有效发现,从而提供了在生产环境中16分之一的稳健发现的最低估计[16].我们这里所描述的错误可能阻碍的能力,有效样本池的两个可能的来源:准确DNA定量,并抽样误差组织收藏。使用凝胶电泳DNA定量是困难的,当样品被降解,因为DNA的标准带显示为涂片,虽然问题可以用高比例的琼脂糖凝胶被最小化。从单一的诱变的祖先而来的个体的集合中抽样偏差也会导致在池中的突变的非等价表示。本研究中通过在本来应该只产生杂合突变线检测纯合突变揭示正常化不准确或抽样偏差。为了尽量减少抽样偏差,从M1个人只有DNA将被用于创建我们的库玉米TILLING服务。
至少玉米基因之一,我们筛选产生了极好的等位基因系列。我们发现在DMT102 3个错义突变,所有这些预测破坏基于序列保守的蛋白。该分析中使用两个不同的程序:SIFT(年代喜欢运动我ntolerantF只读存储器Tolerant [23,它使用PSI-BLAST对齐和PARSESNPP工程”一个线R得意洋洋的年代影响和E评估SNPS.[24],提供PSSM (P反对- - - - - -年代具体年代取心米ATRIX)的基础上对准块差异得分(图2).DMT102是胞嘧啶-5- dna甲基转移酶中含有染色体结构域的“染色甲基化酶”亚家族的成员。拟南芥CMT3染色甲基化酶是第一个被TILLed基因的例子[14],无义突变导致CpNpG甲基化急剧减少[25].这证实了其中的Mutator插入突变成玉米DMT102显示出降低CpNpG甲基[研究26].植物染色甲基化酶最近受到了广泛的关注。例如,对影响CpNpG甲基化的其他突变的研究揭示了DNA甲基化和组蛋白甲基化之间的第一个联系[27和小干扰RNA (siRNA)机制[28在高等真核生物中。一项甲基化分析研究表明,转座子是体内cmt3依赖甲基化的靶点[29].加上突变体插入突变[26,我们的TILLed DMT102等位基因序列现在可能被用于理解玉米DNA甲基化、染色质结构、sirna和转座子生物学之间的关系。
结论
耕作作为一种一般的反向遗传工具有几个优势,特别是对于其他选择有限的生物。化学诱变产生的高突变密度意味着,相对于插入或缺失诱变,筛选所需的植物数量要少得多,而且可以有效地定位小得多的基因。EMS是一种稳定可靠的突变原,而基于rnai的沉默的稳定性、外显率和准确性尚不确定[30.,31],和插入突变可能导致后续的表型分析[复杂化的染色体重排32].TILLING提供了一系列等位基因突变,并且是唯一可以集中搜索错义突变到蛋白质的一部分的方法,例如在一个多结构域蛋白质的单个结构域。耕作系可以在同质的野生型遗传背景下产生,这避免了插入诱变经常需要的异质性问题,尤其是在玉米中。最后,考虑到与转基因相关的高监管和知识产权成本,以及目前对转基因作物的担忧,在不将任何类型的外源DNA引入植物基因组的情况下产生表型变异可能是农业的兴趣。我们目前正在建立必要的参考群体,为玉米社区提供TILLING服务。
方法
玉米诱变、培养和DNA制备
用EMS诱变B73花粉,对B73穗丝进行诱变处理[33].授粉后5-6周采穗。对于NS群体,种植M1种子,从每株植株中收获整株幼叶并冷冻干燥进行DNA取样。对于UI种群,M1自交生成M2种子,然后种植20个M2亲族,所有家族成员从最年轻的叶片开始打孔共60个叶盘,并将汇集的样本进行冻干。
如上所述,从冻干的叶片组织中制备样品[34],除了干燥的组织在加入缓冲液之前在FastPrep均质器中均质成粉末,并用20毫克这种粉末制备DNA。
高吞吐量耕种
种植拟南芥的程序与种植拟南芥的程序相同[16]仅适用于玉米,只有微小的修饰。设计用于使用CODDLE程序扩增〜1-KB段[35基于已知的玉米基因组序列或来自与无内含子水稻序列同源的玉米cdna。基因选自美国国家科学基金会植物染色质计划网站[36基于基因组序列的可用性或基于至少1 kb大小的外显子区域的预测。由于水稻和玉米通常具有相同的内含子位置,通过将预测的玉米编码序列与水稻基因模型进行比对,我们可以选择只扩增外显子DNA的玉米特异性引物。为了找到这些区域,我们使用改良的BLAST基因对拟南芥和水稻的基因组序列进行了cDNA序列搜索,该基因可以根据水稻和/或拟南芥中相应的外显子区域在玉米中识别出较大的外显子区域。最终,我们从植物染色质基因中设计了14对引物进行筛选,并对引物进行了排序。在其他情况下,玉米基因组序列可从ChromDb获得。96孔板中扩增池和单个DNA样本、退火、CEL I切割、电泳、图像分析、重筛和DNA测序均按所述进行[16].
如前所述,使用LI-COR凝胶分析仪进行突变筛选[34].序列跟踪信息使用Sequencher程序进行分析,如所述[17]
参考文献
- 1.
Nagy A, Perrimon N, Sandmeyer S, Plasterk R:剪裁基因组:遗传方法的力量。自然杂志2003,33增刊。: 276 - 284。10.1038 / ng1115。
- 2.
参见Henikoff S,Comai的L:用于植物功能基因组的单核苷酸突变。植物生物学年度审查。2003,54:375-401。10.1146 / annurev.arplant.54.031902.135009。
- 3.
拟南芥转座子的Ac/Ds标记。方法:中国生物医学工程学报,1998,8:315-328。
- 4.
沃特豪斯,Graham MW,王明波:同时表达反义RNA可以诱导植物抗病和基因沉默。美国国家科学院学报。1998年,95:13959 - 13964。10.1073 / pnas.95.23.13959。
- 5.
kysan PJ, Young JC, Jester PJ, Monson S, Copenhaver G, Preuss D, Sussman MR:拟南芥T-DNA插入位点的特征及其饱和诱变的意义。组学。2002年,6:163 - 174。10.1089 / 153623102760092760。
- 6。
Lisch D:突变转座子。植物科学进展,2002,7:498-504。10.1016 / s1360 - 1385(02) 02347 - 6。
- 7。
Bensen RJ, Johal GS, Crane VC, Tossberg JT, Schnable PS, Meeley RB, Briggs SP:玉米An1基因的克隆与鉴定。acta botanica sinica(云南植物学报),1995,7:75-84。10.1105 / tpc.7.1.75。
- 8。
May BP, Liu H, Vollbrecht E, Senior L, Rabinowicz PD, Roh D, Pan X, Stein L, Freeling M, Alexander D, Martienssen R:玉米定向诱变的敲除资源。中国科学(d辑:地球科学),2003,19(4):541- 546。10.1073 / pnas.1831119100。
- 9。
http://chloroplast.uoregon.edu/:。[http://chloroplast.uoregon.edu/]
- 10.
转基因玉米中RescueMu转座子的体细胞和生发流动性。acta botanica sinica(云南植物学报). 2001,13(4):457 - 461。10.1105 / tpc.13.7.1587。
- 11.
http://gremlin3.zool.iastate.edu/zmdb/RescueMu.html:。
- 12.
郭伯伟,徐志强,闫晓华,王志强,等:转座子Ac在玉米基因组中的应用。acta botanica sinica(云南植物学报),2002,14(6):713-726。10.1105 / tpc.010468。
- 13.
Li X, Song Y, Century K, Straight S, Ronald P, Dong X, Lassner M, Zhang Y:一种基于快中子缺失突变的植物反向遗传系统。植物学报1991,27:235-242。10.1046 / j.1365 - 313 x.2001.01084.x。
- 14.
陈志强,陈志强,陈志强:诱导突变的靶向筛选。生物工程学报。2000,18:455-457。10.1038/74542。
- 15.
Colbert T, Till BJ, Tompa R, Reynolds S, Steine MN, Yeung AT, McCallum CM, Comai L, Henikoff S:诱导点突变的高通量筛选。植物生理学报。2001,26(4):481 -484。10.1104 / pp.126.2.480。
- 16。
Till BJ, Reynolds SH, Greene EA, Codomo CA, Enns LC, Johnson JE, Burtner C, Odden AR, Young K, Taylor NE, Henikoff JG, Comai L, Henikoff S:大规模发现高通量TILLING诱导点突变。基因组杂志。2003,13:524-530。10.1101 / gr.977903。
- 17。
Greene EA, Codomo CA, Taylor NE, Henikoff JG, Till BJ, Reynolds SH, Enns LC, Burtner C, Johnson JE, Odden AR, Comai L, Henikoff S:拟南芥大规模反基因筛选的化学诱变谱。中国生物医学工程学报。
- 18。
Till BJ, Burtner C, Comai L, Henikoff S:单链特异性核酸酶的错配切割。中国生物医学工程学报,2004,32(4):593 - 598。10.1093 / nar / gkh599。
- 19。
Comai L,Young K,Till Bj,Reynolds Sh,Greene Ea,Codomo Ca,Enns Lc,Johnson Je,Burtner C,Odden Ar,Henikoff S:通过Ecotilling高效发现自然群体中的DNA多态性。工厂J. 2004,37:778-786。10.1111 / J.0960-7412.2003.01999.x。
- 20。
马基维兹,克莱娜·LG,克鲁兹·C, Ehret S, Miller JH:乳糖阻遏因子的遗传研究。十四。对4000个改变的大肠杆菌乳糖阻滞剂的分析揭示了必需和非必需残基,以及不需要特定序列的“间隔物”。中国生物医学工程学报,1994,19(4):421- 427。10.1006 / jmbi.1994.1458。
- 21。
吴志强,吴志强:有害氨基酸取代的预测。中国生物医学工程学报,2003,21(4):531 - 534。10.1101 / gr.176601。
- 22.
桑德斯CT,贝克d:对有害突变预测结构和进化的贡献评价。J Mol Biol。2002年,27:891-901。10.1016 / S0022-2836(02)00813-6。
- 23.
伍PC,参见Henikoff S:SIFT:预测影响蛋白质功能的氨基酸变化。核酸RES。2003,31:3812-3814。10.1093 / NAR / gkg509。
- 24.
Taylor N,Greene EA:Parsesnp:一种分析核苷酸多态性的工具。核酸RES。2003,31:3808-3811。10.1093 / nar / gkg574。
- 25.
Lindroth AM, Cao X, Jackson JP, Zilberman D, McCallum CM, Henikoff S, Jacobsen SE: CHROMOMETHYLASE3对CpXpG甲基化维持的需求。科学通报,2001,22(4):417 - 418。10.1126 / science.1059745。
- 26.
Papa CM,Springer NM,Muszynski Mg,Meeley R,Kaeppler SM:CPNPG甲基化需要玉米Chromethylase Zea甲基转移酶2。植物细胞。2001,13:1919-1928。10.1105 / TPC.13.8.1919。
- 27.
KRYPTONITE组蛋白H3甲基转移酶对CpNpG DNA甲基化的控制。自然资源学报。2002,416:556-560。10.1038 / nature731。
- 28。
齐尔伯曼d,曹X,雅各布森SE:轨迹特异性siRNA积累和DNA和组蛋白甲基化的ARGONAUTE4控件。科学。2003,299:716-719。10.1126 / science.1079695。
- 29。
Tompa R, McCallum CM, Delrow J, Henikoff JG, vanSteensel B, Henikoff S:全基因组DNA甲基化分析揭示了CHROMOMETHYLASE3的转座子靶点。生物谷学报,2002,12:65-68。10.1016 / s0960 - 9822(01) 00622 - 4。
- 30。
Chuance,Meyerowitz em:双链RNA在拟南芥中的特定和遗传遗传干扰。美国国家科学院学报。2000,97:4985-4990。10.1073 / PNAS.060034297。
- 31。
杰克逊AL,巴兹SR,SchelterĴ,小林SV,伯查德Ĵ,毛男,李B,CAVET G,林斯利PS:表达谱通过RNAi揭示了脱靶基因调控。纳特生物技术。2003,21:635-637。10.1038 / nbt831。
- 32。
拟南芥t - dna相关的复制/易位。突变体分析和功能基因组学的意义。植物营养与肥料学报。2001,22(2):429 - 434。10.1104 / pp.126.4.1527。
- 33。
冈垣RJ,Neuffer MG,韦斯勒SR:缺失常见于玉米的两个可独立衍生的含蜡的突变。遗传学。1991年,128:425-431。
- 34.
直到BJ,COLBERT T,TOMPA R,ENNS LC,Codomo Ca,Johnson Je,Reynolds Sh,Henikoff Jg,Greene Ea,Stein Mn,Comai L,Henikoff S:高吞吐量耕种功能基因组学。植物功能基因组学:方法和协议。编辑:Grotewalde。2003年,克利夫顿,NJ,Humana媒体,205-220。
- 35.
- 36.
铬DB:。[http://chromdb.org]
确认
该工作得到了国家科学基金会植物基因组研究项目的补助金。感谢Jorja Henikoff for Maize / Reen Sequence比较,Vicki Chandler为底漆设计提供基因组DNA序列,以及来自Weil和Springer Labs的Kelly Marshall,Jianua Yu,Peter Hermanson和Virginia Zaunbrecher在诱变和组织收集中的工作。
作者信息
隶属关系
通讯作者
额外的信息
作者的贡献
月,肯塔基州和EB准备样品,CB, CAC,如冰,ARO进行大规模筛选和数据处理、连续波和NS诱变处理的植物材料,提供各地进行数据分析是机器,LC和SH参与这项研究的设计和执行,写了论文。
作者的原始提交的图像文件
权利和权限
关于这篇文章
引用这篇文章
Till, b.j., Reynolds, s.h., Weil, C。et al。通过耕作发现玉米基因诱导点突变。BMC植物杂志4,12(2004)。https://doi.org/10.1186/1471-2229-4-12
已收到:
接受:
发表:
关键字
- 插入诱变
- 等位基因系列
- 突变密度
- 反向遗传学的方法
- 反向遗传策略