抽象的
背景
伞藻属臼是一个巨大的单细胞绿藻,其规模和复杂的生命周期,使其成为了解形态和亚细胞条块一个有吸引力的模型。这个海洋单细胞的生命周期是由几个发展阶段。少年和成人阶段是按时间排序的,但生理和形态不同。为了鉴定基因的特定于青少年和成人阶段,我们创建了两个相减的cDNA文库,一个成人特异性和一个少年特异性的,并从他们分析了941分随机选择的EST。
结果
聚类分析表明这两个库之间几乎没有重叠。初步的表达数据也表明,我们成功地分离了两个发育阶段之间的差异表达的转录本,许多转录本是特定于一个阶段或另一个阶段的。我们的EST序列与公共序列数据库的比较表明,来自成人和青少年文库的EST分为不同的功能类。3个保守的序列元件在几个ESTs中是共同的,也在来自的碳酸酐1基因的基因组序列中发现A. acetabulum..迄今为止,这些保守的元素特定于A. acetabulum..
结论
我们的数据提供了强有力的证据,表明成人和青少年阶段A. acetabulum.基因表达中显着变化。我们讨论了细胞生长和形态发生的可能作用以及相变。我们还讨论了在转录后调节中的EST序列内发现的保守元素的潜在作用,特别是mRNA定位和/或稳定性。
背景
部分cdna的高通量测序,或表达序列标签(ESTs),提供了相对快速和经济的途径来获取生物体的基因表达谱[1,2].EST文库提供访问转录的基因的人口,使得EST序列的分析信息性在确定哪些基因在特定发育年龄表示,在特定组织,或根据具体的环境条件。
当研究生物时,EST分析特别有用,因为这些生物的序列数据很少,而且基因组测序不是计划好的,或者由于基因组大小而不容易实现。到目前为止,几乎没有关于绿藻(绿藻)的基因组数据,这是一个远比所有陆地植物加起来多样性和进化多样性都要大得多的群体。只能从这个群体中衣藻reinhardtii已成为一项广泛的环境科技项目的目标[3.,4].来自该项目的基因组信息被证明对阐明光合器官的功能、生物合成和调控至关重要[4].
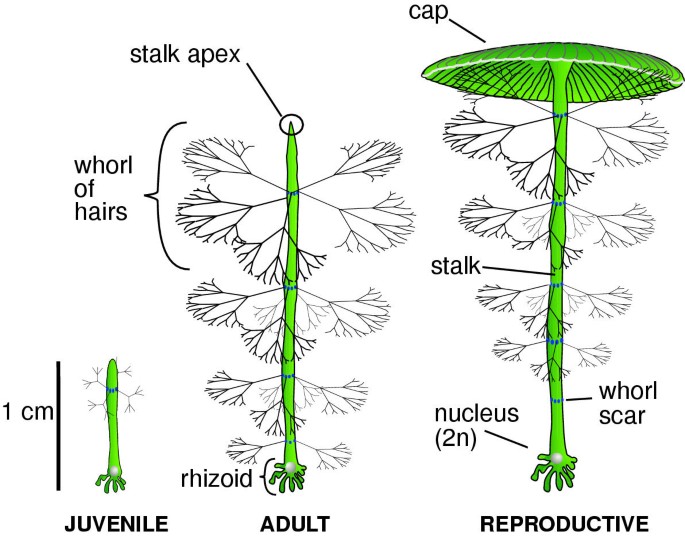
伞藻属臼(无花果。1),又称“美人鱼的酒杯”,是一种巨大的单细胞绿藻,其大小和复杂的生命周期使其成为了解形态发生和亚细胞定位的一个有吸引力的模型系统[5].在到期日达到的高度3厘米,这种单细胞包含只是一个单一的二倍体核其大部分生命周期。它经历了一个复杂的形态发生程序,其中大部分在顶点发生[6厘米远离核心。经典实验A. acetabulum.[7,8为细胞核在形态发生中的作用和“细胞核产物”的存在提供了第一个令人信服的证据,后来被认为是信使rna [9].
生命周期A. acetabulum.由几个发育阶段(图1).像多细胞陆生植物,幼体和成体阶段A. acetabulum.在时间上是连续的,但在形态上是不同的[10].稚虫期包括生长的第一厘米,而成虫期包括其余的2至3厘米[10].幼轮的毛发沿着茎互相堆叠得更近,每个轮内毛发的分支模式比成年的更简单[10].从生理学上讲,这两个阶段也是不同的。例如,幼鱼在拥挤的环境下生长良好,在低种群密度时生长不良,而成鱼只有在低种群密度时生长良好。与陆生植物相似,阶段之间的过渡与顶端生殖能力的变化有关[11,12].在A. acetabulum.(J Messmer和DF Mandoli,未发表)。然而,在分子水平上,成年期和幼年期之间的基因表达模式的差异实际上是未知的。
为了揭示成虫期和幼虫期基因表达的差异,我们构建了两个减去的EST文库A. acetabulum..这些文库被设计用来包含特定于一个阶段或另一个阶段的转录本,推测在涉及形态发生或阶段变化的转录本中富集。我们随机对这两个文库中的941条ESTs进行了测序和分析。我们对这些序列的分析表明,幼期和成虫期的基因表达模式存在显著差异。我们还鉴定了3个主要由成年ESTs共享的共识序列,它们与我们之前克隆的碳酸酐酶基因中的内含子和3' utr具有一致性[13].我们讨论了转录后基因调控,特别是mRNA的定位和/或稳定性,这些保守的元素的潜在作用。
结果
est的一般特性
抑制性消减杂交,或SSH [14的结果是分离和扩增存在于一个群体(测试群体)和不存在于另一个群体(驱动群体)的mrna。使用SSH,我们创建了两个减去的库,一个在青少年特异性转录本中推测丰富,另一个在成人特异性转录本中推测丰富(额外的文件1).从现在开始,我们将分别将这些图书馆称为“少年图书馆”和“成人图书馆”。
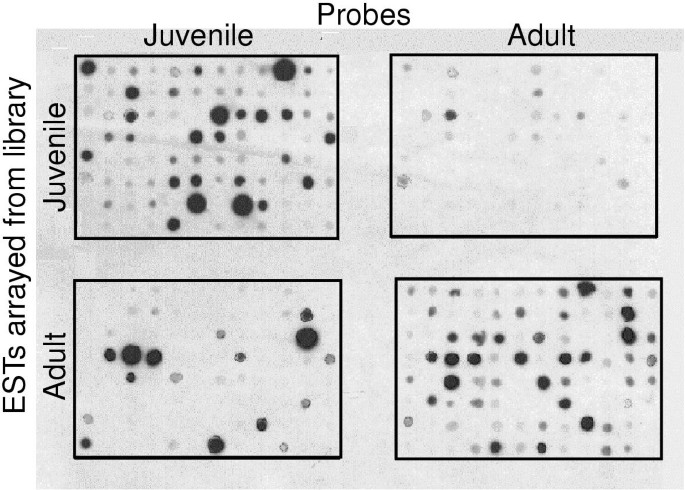
为了检测ESTs的差异表达,从每个文库中随机选择96个克隆,并在两种尼龙膜上以相同的模式进行标记。每个复制膜与两种探针中的一种杂交,这两种探针分别来自成年或幼年mRNA样本(图)。2).出96随机选择的,推定少年克隆,53只在青少年中表达,13在青少年较成人较高水平表达,5在成人相似的水平和少年和25分别表示没有产生任何信号与任一探针(图2,上图)。出96随机选择的,推定的成人克隆,44表达只在成人中,14人在成人比在青少年较高水平表达,10在成人和少年相似水平表达,5在较高水平表达或仅在青少年和23不产生与任一探针的任何信号(图2底部面板)。此外,三个克隆的差异表达被virtual northern blots证实(数据未显示)。Virtual Northern blots与Northern blots的不同之处在于,相特异性cDNA被印迹在尼龙膜上而不是mRNA上[15].这些数据提供了坚实的初步证据表明该SSH是成功的,在隔离许多转录物这两个阶段的差异表达。
总共604和601个EST分别从成人和青少年进行测序。不包括不含插入物或不可靠数据的序列(如序列迹线的证明),从成年文库中留下478个EST,来自少年文库的463个EST进行进一步分析。清洁序列在网上污染片段(载体和引物序列;见材料和方法)。对于87%(411)的成人克隆和83%(392)的少年克隆,该单通次排序提供了插入物的完整序列,即矢量序列与插入物的两端接壤。EST的长度为68个BPS至855个BP。平均而言,少年克隆比成年克隆长,分别平均474个BPS和408个BPS。
由于库的创建方式(额外的文件2),最终文库中的一些est包含polyA或polyT谱束[16].这些片段来源于相应原始mrna的polyA尾部,表明这些ESTs可能含有未翻译区域。由于ESTs不是定向克隆的,所以根据序列的不同,得到了包含polyA或polyT片段的序列。这些通道在青少年文库中的出现明显高于成人文库(分别为40和15%)。
两个库之间的冗余和重叠
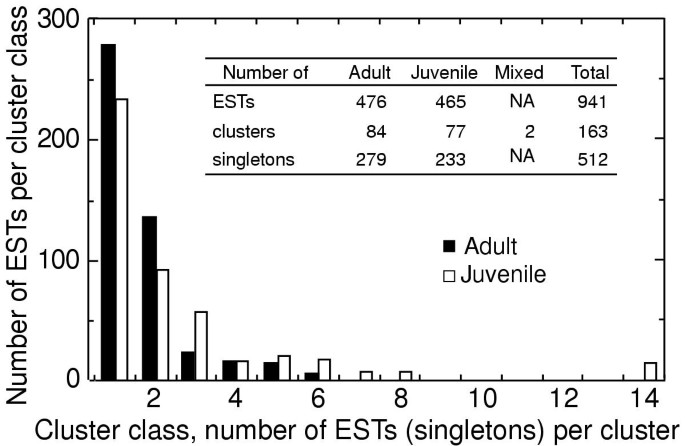
所有EST将所有EST对齐并分配到簇中(详见材料和方法)。所有不是聚类部分的EST仍然是单身人士(279名成人和233名青少年)。共有序列源自含有两个或更多个EST的簇。少年酯形成77个簇,成人酯形成84个簇。簇含有最多14个EST(图。3.).这可能高估了簇的真实数量,因为不重叠的ESTs会被放置到两个或更多独立的簇中,或者即使它们起源于相同的初始mRNA,也会保持单例。另外,可能只是由于测序错误或序列质量差的区域而产生差异的序列没有聚类在一起。
包含来自两个文库的ESTs的簇被标记为“混合簇”。只发现了2个这样的混合集群,仅占集群总数的0.3%(图)。3.).因此,两个库之间的重叠是最小的,提供了额外的证据,即SSH可能成功地分离特定于每个发育阶段的EST。
该EST序列的基因功能
利用BLASTN、TBLASTX和InterPro对所有ESTs或聚类序列进行同源性分析(见材料和方法)。对于BLASTN和TBLASTX搜索,E值<1.00E-06的命中被认为是显著的。总的来说,仅代表162个簇或ESTs的ESTs产生了显著的BLAST命中,其中144个与假定的基因功能相关(见表)1).45%的假定功能是由InterPro搜索独立确认的。有趣的是,仅有的两个混合簇都与大亚基核糖体RNA (rrnL)基因有关,这是在我们的分析中发现的唯一的叶绿体编码基因。
所有的单身人士和集群进行了分析,使用BLASTX对同源性拟南芥蒂利亚纳蛋白质数据库[17],并使用TBLASTX对当前的草案衣藻reinhardtii基因组(18.].对于这两个搜索,E值< 10E-06的命中被认为是重要的。一般来说,相同的序列对每个不同的数据库(表1).这独立地证实了第一次搜索的结果以及在我们的ESTs中编码序列的低百分比。总共有178个est(仅26%)在BLAST搜索中至少有一个产生了显著的命中1).
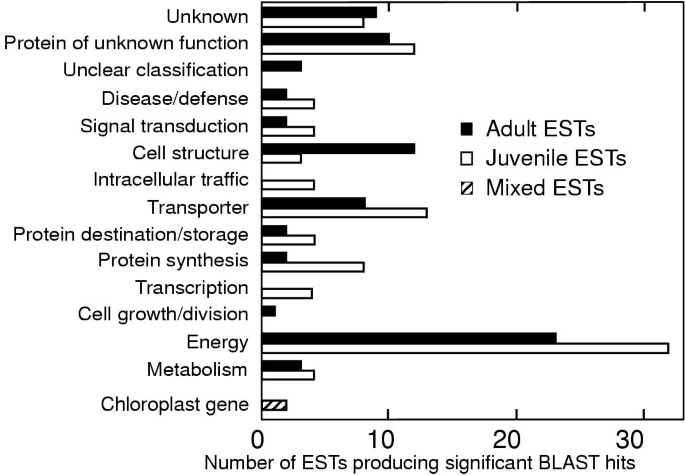
根据为植物开发的分类方案,将产生显着命中的178个EST或群集分类为功能类别[19.] (图。4).这两个文库中最大的功能类包含了与光合作用相关的基因(图中的“能量”)。4).一般而言,青少年的EST更高百分比的具有与转录的功能,和蛋白合成,运输和储存,而成人的EST更高百分比的具有与单元结构(图的功能。4和额外的文件3).
ESTs系统发育分析
评估如果假定的est序列的功能也发生在陆地植物或其他绿藻,我们确定了(为每个178 est序列,产生了巨大的爆炸冲击)序列为最低的E值(“最佳匹配”)在爆炸中搜索和这个序列所属的生物。正如预期的那样,大多数“最佳匹配”序列要么属于衣藻reinhardtii或者Streptophyta(陆地植物)。这并不奇怪,因为大多数绿藻谱系的序列数量仍然非常有限。具体地说,A. acetabulum.属于班级石莼纲只有很少的序列信息可用[20.].
est中的保守序列
一些est在短区域内表现出相似性。这些ESTs分别聚为5、9和2三组序列(图5)。5).序列的公共延伸长度在组1的30 ~ 70 bp,组3的45 ~ 90 bp和组2的170 ~ 250 bp之间变化(图1)。5).在每组内,这些EST在这些地区之外的彼此没有相似,但在这些区域内,身份的水平很高。大多数这些EST属于成人图书馆。这些16个EST中没有任何相关的爆炸击中,使得难以预测它们是否包含编码序列。在共享第二共识序列的序列中(图。5度),除两个外,其余的ESTs均以polyA或polyT束结尾,表明它们可能含有3' uts。所有具有第一或第三条一致序列的est均不存在(图1)。5 b或5 d)末端为polyA或polyT束。这3种保守元素可能是特定于A. acetabulum.,因为它们在Genbank(核苷酸数据库)中除CA1和CA2基因外,没有在其他序列中发现A. acetabulum.[13].所有这三个保守序列的倒在两个CA基因的非编码区,无论是在内含子或的3'UTR(图5).
独立ESTs间的保守序列及其相对位置A. acetabulum.碳酸酐酶(AaCA1)基因组序列.a:结构AaCA1基因组序列。在内含子和3'UTR其中省略序列的位置由倾斜的,重双重线表示。白框表示AaCA1外显子,孵化盒代表3' uts。黑盒子代表了不同EST序列之间的三个强同源区域AaCA1基因和AaCA2信使rna。B, c和d:序列对齐AaCA1基因和不同的est(单粒或簇)在这三个保守区域。每个序列的起源基因或EST的名称显示在序列的前面。这些地区AaCA1和AaCA2括号中显示了排列中的基因。*表示至少70%的序列一致对齐。-表示对齐中有间隙。在EST名称前用“(a /T)”表示EST末端存在多a /T束。
讨论
髋臼棘球蚴成虫期和幼虫期基因表达差异显著
表达式分析(图。2)并且两种库之间几乎没有重叠的事实表明,减法成功地分离差异表达的转录物。
将941个EST组织成675个独立簇或单身。尽管该数量可能是源自同一转录物的高级重叠EST可以分配给不同的簇,或者保持单例 - 这些EST仅代表文库中存在的所有EST的一部分。这些数据提供了成人和少年阶段的强有力证据A. acetabulum.在基因表达和大量基因的显著不同可能是相位特异性的。
两个发育阶段之间的生理差异
以不同的相分配以不同的相分配成功能类的功能(图。4).鉴于库创建使得只有无害环境特定于一个发育阶段将被分离,预计不会基因功能的EST序列之间的分配,以反映典型的光合细胞的,而仅仅是特定于一个发育阶段的功能或其他。
幼鱼似乎将其独特的基因表达大部分用于转录、蛋白质合成、运输和储存,这与幼鱼快速生长、更专注于生长而不是形态发生的一般观点相一致。在幼年期,单细胞的高度在2周内增加了10倍左右(从<1毫米增加到1厘米),不能做帽。而成虫的高度增加了3倍左右,又增加了3厘米。成年细胞比幼年细胞复杂得多,有许多高度分枝的头发螺旋。因此,在成体阶段对细胞壁合成的需求增加。成虫也有能力执行复杂的帽子形态发生,并为核分裂、核运输和配子发生做准备[21.].成虫与幼虫之间的细胞质体积没有很大的增加,因为成虫体内的大部分体积被中央液泡(Ngo等提交)。这种更复杂的成体发育与较低比例的蛋白质合成转录本和较高比例的细胞结构转录本(如细胞骨架蛋白、细胞壁合成或维持酶、组蛋白)相一致。
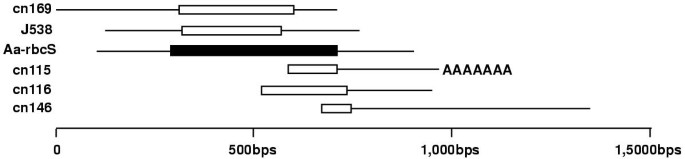
从两个文库的EST的令人惊讶的高数量与相关联的光合作用(额外的文件3例如,5个ESTs被认为是rbcS蛋白的同源物。有可能这些est确实来自不同的转录本。也有可能它们来自同一个转录本,但没有被聚在一起,因为它们不重叠或序列质量较差的区域。为了解决这个问题,我们将这些序列与A. acetabulum.rbcS mRNA序列出现在Genbank中(图。6).我们观察到在每个EST或簇中,都有一个高度序列一致的区域Aa-rbcS序列(在核苷酸水平上,同源性在84 - 89%之间)。其中两个簇(cn115和cn116)除了序列歧义之外完全相同,而这种歧义的频率足以使这两个序列不能聚在一起。除cn116和cn115外,其他序列相互之间以及与该基因有足够的差异Aa-rbc序列,以得出它们实际上不是来自同一转录本的结论。为了支持这一假设,这些高度序列同一性的片段之外的区域无法对齐(<50%同一性)。在氨基酸水平上,这些区域之间的序列一致性(图中的白框)。6)和Aa-rbcS序列几乎完全,在94 - 96%之间。对这些序列的进一步研究证实,orf中的大多数核苷酸差异发生在密码子的第三位,通常导致氨基酸序列的保存。最后,其中一个簇(cn115)以polyA序列结束,表明3'UTR的末端,而生成簇146的转录本似乎拥有一个更长的3'UTR,不包含polyA序列。综上所述,这些结果表明,可能至少有3个不同的基因座编码rbcS蛋白伞藻(CN169,J538和Aa红血球,无花果6).用不同的方法来证实这些令人惊讶的结果,并对不同位点进行详细的表达分析将是非常有趣的。叶绿素A /b结合蛋白的类似分析也可以确定不同的蛋白是否在不同的阶段表达,以及它们的功能是否不同和/或有差异调节。LHC(光收集复合体)结合蛋白形成了一个非常大的家族,在陆生植物中得到了最好的特征[22.].22个EST与身份LHC结合从陆生植物蛋白的分析将提高我们的这个基因家族中的藻类目前知识贫乏。
有特殊意义的假定基因功能
免疫细胞化学已被用于可视化细胞蛋白和肌动蛋白蛋白A. acetabulum.在开发过程中(23.,24.].在各年龄层的菌体中都发现了肌动蛋白微丝,它们沿着整个茎部形成连续的平行束。相反,在减数分裂之前,微管在藻类中无法检测到。在生殖过程中,微管被检测到,在单倍体核被运输到核帽的过程中,微管被包围着。与这些结果一致的是,我们的数据表明,幼鱼表达肌动蛋白转录本(但不表达微管蛋白),而成年鱼同时表达α和β微管蛋白(但不表达肌动蛋白)转录本,可能是为生殖做准备。
我们的两个ESTs是推定的expansin同源物。扩张素促进陆生植物纤维素壁的延伸。典型的“甘露属杂草”,壁复期A. acetabulum.主要是类晶甘露聚糖框架(Dunn等提交)。只有Gametangia封闭在纤维素墙壁中,本身被帽子的甘露墙围绕着。与此符合,我们的结果表明,在成人但不是少年相期间表达扩展基因。到目前为止,扩展素只在陆地植物中发现了[25.在这个过程中,膨胀素在细胞壁内起作用,并被酸性pH激活[26.].如果这些转录本编码的是在细胞壁松动中起作用的膨胀蛋白A. acetabulum.研究它们的作用机制是否与陆生植物相似,以及它们的基质是否也是纤维素壁,将是一件有趣的事情。
A. acetabulum.在它的大部分生命周期中,只有一个核,位于根茎中。这个细胞核在成体末期,在生殖过程中进行复制[27.].在这个关键时刻,需要大量的核苷酸和组蛋白来制造配子发育所需的数百万个单倍体核[21.].与此一致的是,组蛋白mrna仅在成人文库中发现(额外的文件3更深入地研究这些转录本何时表达,以及这种生物如何能在如此短的时间内产生如此大量的组蛋白,将是一件有趣的事情。
最后,其中一个EST对Argonaute蛋白质具有同源性(额外的文件3,11级,E值奥雅萨苜蓿5 argonaute e15汽油)。Argonaute蛋白高度保守,在动物的RNA干扰(也称为真菌的抑制或植物的转录后基因沉默[28.])。这些过程涉及通过双链RNA的特定基因的沉默[29.它们在转录后调控中的重要性才刚刚开始被解读。在陆地植物、鞭毛虫、动物和真菌中已经发现了阿尔古鱼蛋白,但据我们所知,这是第一次发现阿尔古鱼蛋白的藻类序列。
为什么大多数的ESTs不符合任何前面描述的序列
我们可以想到三个原因,为什么只有28.6%的ESTs(这是一个特别低的数字)根据BLAST和InterPro搜索被指定为假定同源物。首先,这些是减去的文库,创建的目的是识别罕见的、特定阶段的转录本或涉及形态发生、顶端生长或阶段变化的转录本。因此,这些ESTs应该包括较少的内务转录本,大量的转录本,或两个阶段或其他生物体共同的转录本。
其次,这些ESTs是由逆转录酶生成的,使用的是poly-T引物,通常不会生成全长cdna。因此,我们的文库往往在转录本的3'端丰富,其中包含非编码序列,在同源性搜索中不会被识别。含有polyA或polyT拉伸的ESTs的高百分比支持这一假设。
最后,A. acetabulum.属于这个秩序DasycladaleS,在绿藻课上石莼纲,目前只有很少的序列数据可用。在加入我们的ESTs之前,只有73个DNA序列来自A. acetabulum.在Genbank中可以找到,只代表37个不同的基因。尽管一些陆地植物和绿藻的完整基因组现在至少部分可用,但似乎大多数A. acetabulum.当进入Blast搜索时,序列从其他藻类或土地植物的植物的植物的常见量太发散[30.].为了验证这一假设,我们将BLASTN和BLASTX在Genbank数据库中的搜索截断值从10E-06提高到10E-03。大多数额外的命中来自藻类或陆生植物序列,而不是Genbank中所代表的生物体的随机分布。这支持了这样的假设,即这些ESTs可能与这些藻类或植物序列同源,但过于分散,无法信任同源性。
成人和青少年的转录本在结构上有区别吗?对转录后调控的洞察
奇怪的是,少年克隆的40%,但只有15%的成年克隆的结束与多聚A或聚T道。如果这些大片对应于mRNA的多聚腺苷酸尾,然后这些EST序列包含一些或所有的转录物从它们起源的3' 非翻译区(3' UTR)的。我们已图解说明假设在成人与少年克隆这些束(图2的差分产生。7).第一个解释推出了用于创建库的技术的工件。来自已消化的cDNA片段的扩增导致Rsa我,每个Rsa我的片段被扩增和克隆的几率是一样的。如果成虫cdna比幼虫cdna消化得更彻底,那么成虫cdna就会产生更多的ESTs,而含有polyA束的ESTs所占比例更低(图2)。7一个).第二种假设假设不同的mRNA长度:如果成年cDNA平均比幼cDNA长,每个成年cDNA将产生更多的ESTs,产生更低比例的含有多A束的ESTs。如果成年cdna的平均编码序列更长,那么它们可能更长(图。7 b)或更长的3'UTRS(图。7 c).如果他们有更长的3'UTR,编码序列的比例,以及无害环境技术与多聚腺苷酸大片的比例将在少年的EST比成人无害环境技术,与我们的研究结果一致较高。
在多聚A的频率之差假想解释/ T的两个库内传导束.黑盒代表假设的转录本的3' UTR。右边是包含polyA或polyT束的ESTs的计算百分比,这可能是由假设的mRNA产生的ESTs产生的结果。a:初始cdna的差异酶切。ESTs来源于被酶切的cDNA片段的扩增Rsa我,每个Rsa我的片段被扩增和克隆的几率是一样的。如果成年cdna比幼年cdna消化得更完全,则成年cdna产生更多的ESTs(在本例中是4个而不是3个),其中含有polyA束的比例更低。b和c:差异mRNA长度在活的有机体内.平均而言,成年cDNA比幼年cDNA长,因此每个成年cDNA产生更多的ESTs(4个而不是3个),产生更低比例的含有多a束的ESTs。成年cdna可能更长,因为它们平均有更长的编码序列(b)或更长的3' utr (c)。
为什么3' utr在成人转录本中比在青少年转录本中更长?在成人A. acetabulum.,生长和形态发生几乎完全发生在茎尖,距离独特的核位于根茎厘米。因此,转录后调控方面,如mRNA的稳定性和mRNA的定位,可能对这些单细胞中的基因表达的调控非常重要。事实上,到目前为止,超过一半的成绩单(9/16)都在研究中A. acetabulum.定位于一个端部或另一个单细胞的,最多至其顶点[31.- - - - - -33.].为了实现这种定位,每个转录本必须在其序列中包含顺式作用的元素,也称为“zipcodes”[34.].在酵母和动物细胞中,'Zipcodes'是局部转录物的3'UTR的一部分[35.].同时,考虑到mRNA分子沿着细胞骨架元素沿茎移动的速率A. acetabulum.[36.,37.,为了达到顶端,任何mRNA都必须至少出生3天,将它们归入“超稳定”mRNA物种[38.].在植物中,负责mRNA分子稳定性的顺式作用元件也位于其3' UTR [38.].因此,转录物3'UTR可能在该物种中基因表达的调节中发挥重要作用,特别是在成人中。
为了实现这些稳定性和定位模式,成体mrna可能在其3' utr中包含几个转录后调控元件,这可能解释了为什么它们会更长。这些监管要素是什么?三个守恒元素(图。5)的几个不相关的EST序列,其中大部分都来源于成人图书馆是大有希望中找到。这些3个元素也似乎是特定于A. acetabulum.并且位于碳酸酐酶基因的非编码区,其转录本位于顶端[13].5个含有第二保守元件的ESTs也含有polyA束,提示这些ESTs可能编码3' uts。第一个和第二个保守元素属于内含子AaCA1(无花果。5).可能的是,这些序列中的元素AaCA1是可替代的内含子的一部分,有时包含在由该基因产生的成熟MRNA中。未来的研究将侧重于阐明这些保守元素的功能及其在开发过程中的空间表达。
结论
这些结果呈现在这里提供了强有力的证据支持这一假设成年和少年阶段中A. acetabulum.基因表达模式差异显著,大量基因具有阶段特异性。我们的下一个目标是在这些基因中识别那些可能涉及形态发生或相变的基因。这两个阶段的ESTs也被划分为不同的功能类,进一步强调了这两个阶段之间的生理差异。最后,我们在EST序列中发现了保守元素。虽然这些保守元件的功能意义仍有待阐明,但这些序列可能参与了这些转录本的转录后调控,可能在亚细胞定位和/或稳定性中。
方法
A.髋臼的文化
单细胞在人工海水中生长,直到它们达到预期的发育年龄。无菌培养是通过净化成熟瓶盖获得的,然后使用无菌配子囊进行交配[21.].受精卵在无菌人工海水Ace27中生长,Ace27与Ace25相同[39.[除了在Chelex-100柱上纯化KCl预涂,含有尿素过氧化氢,最终浓度为10-15年在冷白光下培养,光子通量密度为170 μmol m-2年代-1在14小时光/ 10 H深光周上,在21°C±2°C,并反复稀释以适合其发育年龄[21.].
mRNA提取
幼鱼通过过滤收获,成鱼用无菌的牙科工具收获。单胞菌在Kimwipe上短暂干燥,然后放在铝箔上称重。一袋袋相同年龄的藻类被快速冷冻在液氮中。将7.15克的幼体(约18000个单细胞)和18.2克的成体(约4000个单细胞)在液氮下研磨成细粉。将粉末转移到含有提取缓冲液的Oakridge管中(0.1至0.2 g研磨单细胞细胞/ml提取缓冲液)。按照Chang的方法提取RNA等.[40].
抑制消减杂交(SSH)
cDNA合成和SSH是根据使用PCR cDNA合成试剂盒(Clontech Laboratories公司)和PCR-选择cDNA消减分别试剂盒(Clontech Laboratories公司)制造商的建议进行。所涉及的SSH的步骤和从mRNA中的EST的形成的更详细的图A概要可见于额外的文件1和2.
克隆ESTS制作图书馆
使用标准乙醇沉淀方案沉淀DNA [41.].为了3' A突出端添加到PCR产物用于随后克隆,将DNA重新悬浮于25μl的PCR反应混合物(2.5微升10X缓冲液,1.5μl的氯化镁2,2微升10毫摩尔dNTP,18.875微升水和0.125微升Taq聚合酶(Promega公司))中并在72℃温育8-10分钟。该DNA,再次沉淀[41.然后在TE中重悬至DNA扩增反应的起始体积。根据制造商的建议,使用AdvanTAge™PCR克隆试剂盒(Clontech Laboratories, Inc.,现已停产)和TOPO™-TA克隆试剂盒(Invitrogen)将每个文库克隆到两个不同的克隆载体中。
点印迹和虚拟北方印迹分析的图书馆
减法的质量按照Clontech提供的PCR-Select方案推荐进行控制。从两个文库中随机挑选96个克隆的pcr扩增插入片段,在尼龙膜上进行重复标记,并与两个减号的放射性标记混合杂交。此外,利用SMART cDNA synthesis (Clontech)的virtual northern blots验证了三个克隆的cDNA插入的差异表达[15].在这些点印迹和虚拟北方印迹中使用的克隆没有测序,也不属于以下序列分析的一部分。
EST测序
用无菌牙签从每个文库中随机挑选菌落。从每个菌落中分离质粒DNA,用2 × 40 μl洗脱缓冲液(Plasmid Miniprep Kit, Qiagen)洗脱。
DNA测序的植物基因组学微生物工厂,俄亥俄州立大学进行。测序反应通过混合400质粒DNA和10微升的总体积4皮摩尔引物(M13F(5'-GTAAAACGACGGCCAG-3“)或M13R(5'-CAGGAAACAGCTATGAC-3”)与水的纳克制备。接着,2μl of BigDyeTerminator mixture, version 2 (Applied Biosystems), 4 μl BetterBuffer (The Gel Company) and 4 μl water were added. The cycling parameters were those recommended by the manufacturer except that the reactions were run for 35 cycles instead of 25. The reactions were cleaned up with Millipore Multiscreen/Sephadex columns, according to the manufacturers recommendations (Millipore Technical Note TN053). The resulting 20 μl of clean sequencing reaction product (in water) was placed in an Applied Biosystems 3700 DNA Analyzer for separation and analysis.
序列分析
顺序准备
每个克隆用M13正向引物测序一次。如果序列质量较差,则使用M13反向引物对克隆进行测序。使用Sequencher (Gene Codes Inc.)对每个核苷酸序列进行清洗在网上通过EST序列对准到所述载体的那些引物的在创建库的单独使用污染载体或引物序列的(巢式PCR引物1(5'-TCGAGCGGCCGCCCGGGCAGGT-3' )和巢式PCR引物2(5“-AGCGTGGTCGCGGCCGAGGT-3”)。保险剩余序列是没有可能潜在地产生在BLAST搜索错误的命中[污染性DNA片段的这些步骤16].较高比例的层序还含有多聚A或多聚t束。这些DNA片段也被移除在网上在执行同源性搜索之前的相应序列。
同源性搜索
对每个EST进行如下查询:
- BLASTN查册[42.](数据库查询:nr(非冗余核苷酸序列))[43.].
- TBLASTX查册[42.](数据库查询:nr(非冗余核苷酸序列),遗传密码:6(用于纤毛虫和绒枝藻目[44.]),其余参数使用默认值)[43.].
- InterPro的搜索[45.]查询欧洲生物信息学研究所的蛋白质基序数据库[46.].InterPro的数据被映射到基因本体论[47.].
- BLASTX对搜索拟南芥蒂利亚纳数据库[17].
- 针对的2003年6月草案TBLASTX搜索衣藻reinhardtii基因组(18.].
群体的群集
使用StackPack软件(Electric Genetics,开普敦,南非)对ESTs进行聚类和比对[48.,49.].聚类分为两个阶段。第一阶段使用了“D2”算法,这是'D2'簇的一部分[50,51].第二阶段使用Phrap [52].
该分析采用下列参数运行:
D2_cluster: word_size = 6, similarity_cutoff = 0.96, minimum_sequence_size = 50, window_size = 150, reverse_comparison = 1。
Phrap: old_ace = 1, vector_bound = 0, trim_score = 20, forcellevel = 0, penalty = -2, gap_init = -4, gap_ext = -3, ins_gap_ext = -3, del_gap_ext = -3, maxgap = 30, flags = -retain_duplicates. Phrap: old_ace = 1, vector_bound = 0, trim_score = 20, forcellevel = 0, penalty = -2, gap_init = -4, gap_ext = -3, ins_gap_ext = -3, del_gap_ext = -3, maxgap = 30, flags = -retain_duplicates。
数据的组织
生物信息学的脚本和用于存储和查询序列数据库系统/注释数据是由专门的植物资源信息学和基因组学(小枝)项目提供http://bioinformatics.org/sprig,特别是SPRIG通用EST数据库和支持脚本[53].
序列的可用性
本研究分析的EST序列已提交Genbank的dbEST分区,登录号为CF 258288 ~ CF259228。
缩写
- 3’UTR:
-
3 '非翻译区
- 爆破:
-
基本的局部对齐搜索工具
- SSH:
-
抑制减肥杂交
- AaCA1:
-
碳酸酐酶从1伞藻属臼
- 美国东部时间:
-
表达序列标签
- RTase:
-
逆转录酶。
参考
- 1.
Boguski MS, Lowe TM,托尔斯泰CM:用于“表达序列标签”的dbest数据库。Nat. Genet. 1993, 4: 332-333。10.1038 / ng0893 - 332。
- 2。
Fillingham JS, Chilcoat ND, Turkewitz AP, Orias E, Reith M, Pearlman RE:表达序列标签(est)在纤毛原生动物四膜虫嗜热性。j . Eukaryot。微生物学杂志,2002,49:99-107。
- 3。
Asamizu E,中村Y,佐藤S,福泽H,田畑S:cDNA的在单细胞绿藻,莱茵衣藻规模大结构分析。一,3433非冗余的产生表达序列标签的。DNA水库。1999年,6:369-373。
- 4。
shager J, Hauser C, Chang C- w, Harris EH, Davies J, McDermott J, Tamse R, Zhang Z, Grossman AR:衣藻reinhardtii基因组计划。互补脱氧核糖核酸信息的产生和使用指南。植物营养与肥料学报。2003,31(4):491 - 498。10.1104 / pp.016899。
- 5.
Dumais J,Serikawa K,Mandoli DF:AceTabularia:一种理解亚细胞定位和发生过程中的形态发生的单细胞模型。J工厂增长调节。2000,19:253-364。10.1007 / S003440000035。
- 6.
髋臼柄生长、形态和壁应力之间的关系。植物学报。2001,13:659-666。10.1007 / s004250100538。
- 7.
Hämmerling记者:发展协会UND formbildungsvermogen冯伞藻属地中海。生物学ZENT BL。1932年,52:42-61。
- 8.
Hämmerlingj:优步格式Bei AceTabularia Mediterranea,Ihre Raumliche und Zeitliche Verteilung und Ihre Herkunft。Wilhelm Roux Arch。Entwichklungsmech。org。1934,131:1-81。10.1007 / BF00649809。
- 9.
Hämmerling J:髋臼和其他细胞的核-胞浆相互作用。为基础。启植物杂志。植物生态学报,1996,14:65-92。
- 10.
Mandoli DF:髋臼发展过程中身体计划和阶段变化的阐述:巨型单细胞的复杂结构是如何建造的?为基础。启植物杂志。植物生态学报。1998,49(4):493 - 498。10.1146 / annurev.arplant.49.1.173。
- 11.
琼斯CS:种子植物中的幼苗,相变,异种发病的一篇文章。Int。J.植物科学。1999,160:S105-S111。10.1086 / 314215。
- 12.
陈志强,陈志强,陈志强:玉米早期变化基因的研究进展。acta botanica sinica(云南植物学报),2002,14(4):457 - 461。10.1105 / tpc.010406。
- 13。
髋臼中不对称亚细胞mRNA分布与碳酸酐酶活性相关。植物生理学报。2001,25(4):449 - 454。10.1104 / pp.125.2.900。
- 14。
黄斌,Lukyanov S, Lukyanov K, Gurskaya N, Sverlov ED, Siebert PD:抑制消减杂交:一种产生差异调控或组织特异性cDNA探针和文库的方法。自然科学学报。美国。1996年,93:6025 - 6030。10.1073 / pnas.93.12.6025。
- 15.
弗朗兹,布鲁氏菌I,罗德:用虚拟北方印迹法验证差异基因转录。诊断。中国石油大学学报(自然科学版)。
- 16.
Henry IM, Mandoli DF:珍贵的细胞包含珍贵的信息:从少数细胞表达分析的策略和陷阱。植物功能基因组学:方法和方案第236卷,印社。编辑:E Grotewold ed. Totowa, NJ, Humana出版社;2003.
- 17.
拟南芥蛋白质数据库。[http://www.tigr.org/tdb/e2k1/ath1/].
- 18.
莱茵衣藻基因组的草案。[http://genome.jgi-psf.org/chlre1/chlre1.download.html].
- 19.
Bevan M, Bancroft I, Bent E, Love K, Goodman H, Dean C, Bergkamp R, Dirkse W, Van Staveren M, Stiekema W, Drost L, Ridley P, Chalwatzis N:拟南芥4号染色体1.9 Mb连续序列分析。自然科学。1998,391:485-488。10.1038/35140。
- 20。
NCBI的分类。[http://www.ncbi.nlm.nih.gov/Taxonomy/taxonomyhome.html/].
- 21.
曼多利:髋臼怎么了?将一个曾经经典的模型系统带入分子遗传学时代。Int。中国生物医学工程学报,1998,19(1):1- 7。
- 22.
jansson s:光收获叶绿素a / b结合蛋白。Biochim。Biophys。acta。1994年,1184:1-19。10.1016 / 0005-2728(94)90148-1。
- 23.
menzel d:通过髋关节血管生命周期的脑骨骼变化的可视化。原生质。1986,132:30-42。10.1007 / BF01276373。
- 24。
Menzel D:髋臼的细胞分化和细胞骨架:Tansley综述号77。新植物学报,1994,28(3):369-393。
- 25。
科斯格罗夫D:植物细胞壁因扩张素而松动。自然资源学报,2000,31(4):457 - 461。10.1038 / 35030000。
- 26。
Cosgrove DJ:细胞壁因膨胀而松动。植物生理学报。1998,19(4):437 - 442。10.1104 / pp.118.2.333。
- 27。
Runft LL,Mandoli DF:即先于伞藻属臼生殖发病的细胞事件的协调:在发展一个“循环”的证据。发展。1996年,122:1187至1194年。
- 28.
植物的RNA沉默-防御与反防御。自然科学学报,2001,22(4):427 - 428。10.1126 / science.1061334。
- 29.
Carmell M, Xuan Z, Zhang M, Hannon G: Argonaute家族:触角延伸到RNAi、发育控制、干细胞维持和肿瘤发生。基因工程学报,2002,16:2733-2742。10.1101 / gad.1026102。
- 30.
为什么我们还能找到匿名的ESTs?基因组学杂志。2000,11:689-693。10.1007 / s003350010127。
- 31。
serkawa KA, Mandoli DF: Aaknox1是髋臼中一个类似kn1的同源盒基因,它经历发育调节的亚细胞定位。植物营养与肥料学报,2003,22(4):457 - 461。10.1023 /: 1006387107071。
- 32。
沃格尔H:Untersuchungenn祖spezifischen表达-Gradientenin DER einzelligen,siphonalen Grunalge伞藻属臼(L.)席尔瓦。贾斯特斯-李比希安大学吉森.;1998年。
- 33.
Vogel H, Grieninger GE, Zetsche KH: Acetabularia acetabulum单细胞藻类的差异信使RNA梯度。细胞骨架的作用。植物生理学报。2002,29(4):593 - 598。10.1104 / pp.010983。
- 34.
Bassell GJ, Oleynikov Y, Singer RH: mRNA在所有大小细胞中的传播。王志强,王志强。
- 35.
RNA定位技术的发展。为基础。生物化学学报,1998,32(4):457 - 461。10.1146 / annurev.biochem.67.1.335。
- 36。
Dazy A,博尔吉H,Puiseux-DAO S:RNA迁移在地中海伞藻属:胞菌素B,放线菌酮和延长的暗周期的影响。植物科学快报。1983,30:285-296。10.1016 / 0304-4211(83)90167-0。
- 37。
髋臼的聚腺苷化RNA。分化。1975,4:115-123。
- 38。
植物mRNA稳定性的研究进展:多个水平和调控机制。植物科学进展,1999,4:429-438。10.1016 / s1360 - 1385(99) 01484 - 3。
- 39。
Hunt BE, Mandoli DF:一种新型的人工海水,可促进髋臼(绿藻)大量细胞的生长,减少细胞培养中固有的劳动。[j] .浙江农业学报,1996,32:483-495。10.1111 / j.0022-3646.1996.00483.x。
- 40.
关键词:松树,RNA,分离,生物信息学植物摩尔。生物。代表1993,11:113-116。
- 41.
Sambrook J,Russell D:分子克隆,实验室手册。volume 3.第三版。纽约,冷泉港实验室出版社;2001年:A8.14。
- 42.
Altschul SF, Gish W, Miller W, Myers WE, Lipman DJ:基本的局部定位搜索工具。林志刚,王志强。中国生物医学工程学报。2000,19(4):457 - 461。10.1006 / jmbi.1990.9999。
- 43。
在NCBI爆炸。[http://www.ncbi.nlm.nih.gov/blast/].
- 44。
施耐德苏,裂片MB,杨XP:三种醋酸纤维素的小亚基与两种髋关节症的亚磷酸酶/氧酶之间的强烈同源性和异常密码子使用的发生。摩尔。Gen. Genet。1989,218:445-452。10.1007 / BF00332408。
- 45.
在NCBI爆炸。[http://www.ncbi.nlm.nih.gov/blast/].
- 46.
Apweiler R, Attwood TK, Bairoch贝特曼,伯尼E, Biswas M,布赫P, Cerutti L, Corpet F, cron MDR,杜宾R, Falquet L, Fleichmann W, Gouzy J, Hermjakob H, Hulo N,反观我,卡恩D, Kanapin, Karavidopoulou Y,洛佩兹R,马克思B,穆德N, Oinn T, Pagni M,仆人F, Sigrist C, Zdobnov EM:InterPro数据库,一个蛋白质家族、结构域和功能位点的综合文档资源。核酸学报2001,29:37-40。10.1093 / nar / 29.1.37。
- 47.
Interpro。[http://www.ebi.ac.uk/interpro/].
- 48。
基因本体论财团。[http://www.geneontology.org/].
- 49。
拟南芥蛋白质数据库。[http://www.tigr.org/tdb/e2k1/ath1/].
- 50.
莱茵衣藻基因组的草案。[http://genome.jgi-psf.org/chlre1/chlre1.download.html].
- 51.
Christoffels A, van Gelder A, Greyling G, Miller R, Hide T, Hide W: STACK:序列标签对齐和共识知识库。诊断。植物营养与肥料学报,2001,29(4):434 - 438。10.1093 / nar / 29.1.234。
- 52.
米勒R,Christoffels A,Gopalafrishnan C,伯克Ĵ,Ptitsyn A,Broveak T,隐藏宽:综合方法表达的人基因序列的聚类中的序列标签比对和共有知识基础。基因组研究。1999年,9:1143年至1155年。10.1101 / gr.9.11.1143。
- 53.
DB: d2的生物学评价,一种高性能序列比较算法。中国农业科学,1994,1:199-215。
- 54.
伯克Ĵ,戴维森d,隐藏宽:d2_cluster:用于聚类EST和全长cDNA序列的验证的方法。基因组研究。1992年,9:1135年至1142年。10.1101 / gr.9.11.1135。
- 55。
Phrap和Cross_Match的文档。[http://www.phrap.org/phrap.docs/phrap.html].
- 56。
Open Lab CVS存储库。[http://bioinformatics.org/cgi-bin/cvsweb.cgi/sprig/database/est/MarksDB/].
- 57.
拟南芥蛋白质数据库。[http://www.tigr.org/tdb/e2k1/ath1/].
- 58.
莱茵衣藻基因组的草案。[http://genome.jgi-psf.org/chlre1/chlre1.download.html].
致谢
我们要感谢Harvey Bradshaw博士、Rose Ann Cattolico博士、Richard Olmstead博士和Elizabeth Van Volkenburgh博士提供的有用的建议、评论和鼓励。我们感谢Richard Ivey先生对细胞培养和RNA提取,Eric Blackstone先生对EST文库克隆的帮助,以及生物信息学组(加拿大萨斯卡通大学计算机科学系)Matthew Links先生对EST的聚类和比对。我们的ESTs测序是在植物-微生物基因组学设施(俄亥俄州立大学,哥伦布)进行的。这项工作得到了植物学系奖学金、富布莱特助学金和比利时-美国教育基金会奖学金(全部给IMH)的部分支持,由NSF IBN #9630618,华盛顿大学周五港实验室学徒课程,以及PanWy基金会的拨款(全部给DFM)和德意志Forschungsgemeinschaft SFB 57(给Z.S-S)。
作者信息
隶属关系
通讯作者
额外的信息
作者的贡献
I.H.制备了部分质粒dna,参与了序列的生物信息学分析和研究的设计,分析了结果并起草了手稿。M.W.对序列进行生物信息学分析,建立EST数据库。Z.S-S。创建减去的库。E.G.和J.M.H.协调测序并进行初步分析。参与了这项研究的设计。D.M.构思并发起了这个项目,准备了一些质粒dna,并参与了研究的设计和协调。所有作者阅读,审查和批准的最终手稿。
电子辅料
12870_2003_27_moesm1_esm.pdf.
附件1:减库的建立。参与创作减去库的步骤总结。(PDF 3 KB)
12870_2003_27_moesm2_esm.pdf.
附加文件2:cDNA合成和Rsa我消化。详细描述了从mRNA形成双链cDNA的步骤和cDNA群体的消化过程。这个过程产生了一个ESTs种群,其中一些包含polyA束,因此ESTs的大小取决于位置Rsa我限制的网站。不间断线代表DNA链,虚线代表RNA链。灰色方框表示用于这些扩增步骤的引物。这些步骤的更详细描述可以在亨利和曼多利[16].(PDF 123 KB)
12870 _2003_27_moesm3_esm.pdf
附加文件3:产生显着爆炸点击的EST的功能和名称。根据其功能和遵循对植物开发的分类方案进行分类19.].(PDF 31 KB)
作者为图像提交的原始文件
下面是作者提交的原始图片文件的链接。
权利和权限
关于这篇文章
引用这篇文章
亨利,I.M.,威尔金森,医学博士,埃尔南德斯,J.M.et al。巨型单细胞绿藻幼体和成体阶段ESTs的比较伞藻属臼.BMC植物杂志4,3(2004)。https://doi.org/10.1186/1471-2229-4-3
已收到:
接受:
发表:
关键字
- 陆地植物
- 棒曲霉素
- Argonaute蛋白质
- 抑制差减杂交
- TBLASTX搜索