抽象的
背景
虽然HKT.转运蛋白基因确定作物耐盐机制的一些关键决定因素,II组的多样性和功能作用HKT.面包小麦的基因还不清楚。关于大米的高级知识HKT.因此,全基因组序列用于比较基因分析,以鉴定正交小麦族IIHKT.基因及其在不同盐水环境下具有特质变异的作用。
结果
四组二世HKT.在水稻中发现了两个来自面包小麦的同源基因家族,包括已知的Tahkt2.;1基因家族和新的明显不同的基因家庭指定为Tahkt2.;2.一份Tahkt2.;2各同源染色体臂7AL、7BL和7DL均有表达,各基因在叶片、鞘和根组织中均有表达。由基因编码的蛋白质Tahkt2.;2系列揭示了超过93%的氨基酸序列同一性,但与编码的蛋白质相比,≤52%氨基酸同一性Tahkt2.;1家庭。具体地,已知关键结构域的变化预测了两种蛋白质家族之间的功能差异。类似于染色体6L上的稻米基因,Tahkt2.;1和Tahkt2.;2基因分开在小麦染色体7Al,7BL和7DL上约3kb,在两个物种中形成静态同步块。含有7AL的染色体区域Tahkt2.;1 7艾尔-1与芽Na的QTL共定位+在一些含盐环境中的浓度和产量。
结论
在拷贝数、基因序列和编码蛋白上的差异Tahkt2.;2职业基因和其他第II族HKT.物种内部和跨种的基因家庭可能反映植物中的离子选择性和运输功能多样性。证据表明也没有Tahkt2.;2也不Tahkt2.;1与主根钠+但吸收Tahkt2.;1可能与NA的特征变异相关联+在一些但不是所有的盐碱环境中排除和产出。
背景
对高盐条件的反应是由多个基因控制的几个生物过程相互作用的结果。越来越多的证据表明,Na+从蒸腾水流中排除是与耐盐性有关的一个重要机制[1].Na+作为na测量时排除+和/或k+在组织或器官中的含量,在面包小麦中是一种健壮且高度遗传的特性[2].高亲和力钾转运体(HKT.)基因是植物中最受研究的膜转运蛋白最多的基团之一。HKT.编码na的基因+选择性转运蛋白与盐过度敏感(SOS)途径的内聚力作用[3.确定了Na的主要角色+排除[2,4]小麦和其他物种[5- - - - - -7].几组我HKT.运输商与NA的检索有关+从根茎或鞘内的木耳限制敏感叶组织中盐的运输和积累[1,8,9].草种演化了由II组编码的第二类HKT蛋白质HKT.作为na起作用的基因+和K+可渗透的运输器[10.].这个群体中的一个成员Tahkt2.;1这是在面包小麦中发现的,它编码了一种可能在Na中起作用的蛋白质+通过根部从外部介质吸收[11.,12.].然而,最近的一项研究表明Tahkt2.;1是由四种功能基因和位于家乡7染色体的长臂上的功能基因和伪原组成的多庚烷家族,证据表明个体基因未参与控制NA+但也有可能在排除钠离子方面起作用+或可能参与维持K+工厂中的状态[13.].因此,第二组可能有新的能力HKT.基因已被认识到,有必要进一步研究,以获得额外的见解,以进一步了解这些基因在不同盐水环境下具有特质变异中这些基因的作用和潜力。
第二组最全面的分析HKT.基因已经在米饭中(栽培稻L.)最多四种基因OsHKT2;1;OsHKT2;2,OsHKT2;3.,OsHKT2;4特征在于基因结构,表达和功能。一些功能基因如OsHKT2;2在indic大米Pokkali [14.鉴定为嵌合基因,没有-OsHKT2;2/1,在japonica.米诺岛博克拉[15.和在Nipponbare中截短的假基因[14.为第二组的近期进化变化提供了证据HKT.现代水稻种植中的基因。水稻二世二世二世的系统发育关系HKT.基因显示基因重复和分歧的证据,识别两个不同的簇,其中基因OsHKT2;1和功能OsHKT2;2分开的和聚集的OsHKT2;3.和OsHKT2;4在群区中仅有> 91%DNA序列标识,但簇之间仅为40-50%的同一性[5,16.- - - - - -18.].水稻基因的转录本OsHKT2;1和OsHKT2;2在耐受和敏感水稻品种的不同组织表达的根中检测到[19.),而OsHKT2;3.和OsHKT2;4在拍摄过程中积累的转录本[17.,20.].虽然OsHKT2;1在生理盐水条件下下降[21.[没有证据表明对剩余稻米第II的表达有显着影响HKT.基因[17.].大多数功能族二世HKT.米中的基因作为na+/ K+在维持K+/ Na+植物的内稳态[20.,22.,23.].然而,OsHKT2;1是一个例外并假定用作na+选择性运输器,具有“营养NA”中的推定作用+吸收“K下+饥饿[21.].对水稻II族的结构、表达和功能有广泛的了解HKT.因此,基因可以有效地用于基于对比基因研究鉴定小麦基因脱节论和表征基因。
基因的系统发育相关性和全基因组序列为跨物种鉴定基因同源性提供了机会。水稻可利用的先进基因组资源,包括含有37,544个注释蛋白编码基因的389 Mb基因组中95%的基因组序列数据[24.[允许用户友好地访问基因组数据的集成搜索工具能够在与其他谷物种类的对比基因研究中施加水稻基因组。虽然没有像米饭那么先进,但鉴定了17GB面包小麦基因组的草稿序列> 124,000次注释和有序基因基因座[25.]其中加速了大米和小麦之间的比较基因研究,以鉴定小麦基因及其与控制特质变异的生物过程的关系[26.- - - - - -30.].更具体地说,是高度的序列守恒HKT.基因[10.允许在同一中和多烯家族内部和跨多烯家族的比较基因分析[13.,或不同的草种[31.].因此,可以利用小麦和水稻全基因组序列进行数据挖掘和详细的比较基因分析,以鉴定小麦和水稻的同源性HKT.基因。
虽然大米和小麦的比较基因分析使基因鉴定和表征,但小麦的确定功能HKT.orthologs定义了他们对改善耐盐性的贡献。小麦耐盐性的定量特质基因座(QTL)研究[32.- - - - - -36.可以策略性地用于研究可以在功能上与特性变异相关的基因。特别是,随着父母衍生自小麦品种Berkut和Krichauff的双倍单倍体(DH)映射群体检测了许多与受控和现场盐水环境中的生理和产量相关性状相关的QTL,包括17 QTL的NA+排除量为叶或芽钠+在不同环境中的浓度[33.,37.].有趣的是,一个成员Tahkt2.;1基因家族位于染色体7AL的类似区域[13.]作为拍摄na的qtl+受控(水栽法)盐水条件下的浓度和幼苗生物量和在适用的染色体区域中,用于在适度的盐水现场环境下的产率组分的变化[33.,37.].因此,QTL信息可以用来推测小麦II组的作用HKT.基因同源性在控制不同盐环境下的表型表达,为它们在提高耐盐性方面的可能作用提供了见解。
虽然是一个多基因家族Tahkt2.;1从小麦中表现出色[13.,因为水稻中有四个单独的基因[17.]假设小麦可能已经进化了多个组的多个副本是合理的HKT.基因家庭。因此,这项研究的目的是在比较基因分析中施加全基因组序列,以鉴定和表征水稻二世群体的小麦直脑HKT.基因。利用Berkut/Krichauff DH定位群体,通过QTL分析,研究了不同盐碱环境下小麦基因同源性与性状变异的关联。这项研究的结果将增加我们对第二组的认识HKT.小麦基因及其对不同盐水环境下特质变异的重要性。
结果
小麦基因对稻米组二世稻港币
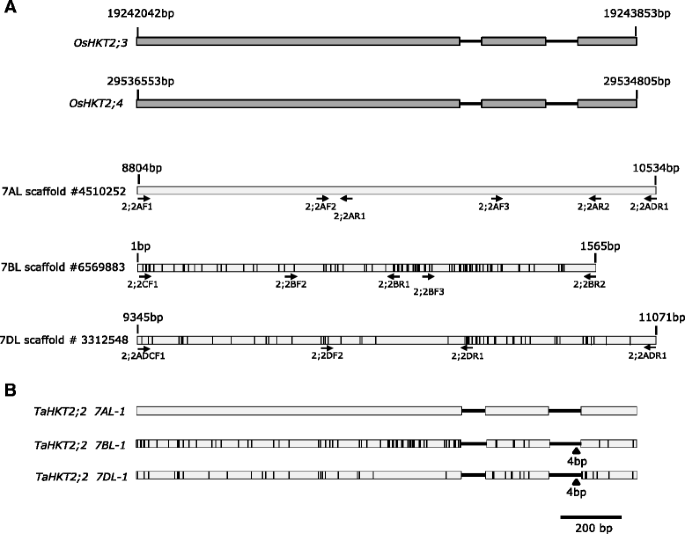
水稻基因全长cDNA(FL-cDNA)OsHKT2;1,OsHKT2;2,OsHKT2; 3和OsHKT2;4作为IWGS调查序列数据库blastn和tblastx搜索的查询序列,以鉴定相关小麦基因序列。4个水稻fl - cdna在7个支架上鉴定出相关的小麦序列(见表)1).密切相关的fl-cDNAOsHKT2;1和OsHKT2;2在6个小麦支架上,3个在小麦7AL染色体上,2个在7BL染色体上,1个在7DL染色体上,DNA同源性高达76%(见表)1),并在已知的小麦属II HKT的同一区域内,Tahkt2.;1. Tahkt2.;1对两者进行了75-76%的DNA同一性OsHKT2;1和OsHKT2;2.同样,FL-cDNAOsHKT2;3.和OsHKT2;4在3个小麦支架上,DNA序列同源性均达到80%1).因此,小麦族IIHKT.基因截然不同Tahkt2.;1有两者的身份OsHKT2;3.和OsHKT2;4.小麦支架具有序列相似性OsHKT2;3.和OsHKT2;4,#4510252;选择在第6569883和#3312548上,分别在染色体7AL,7BL和7DL上进行进一步调查。
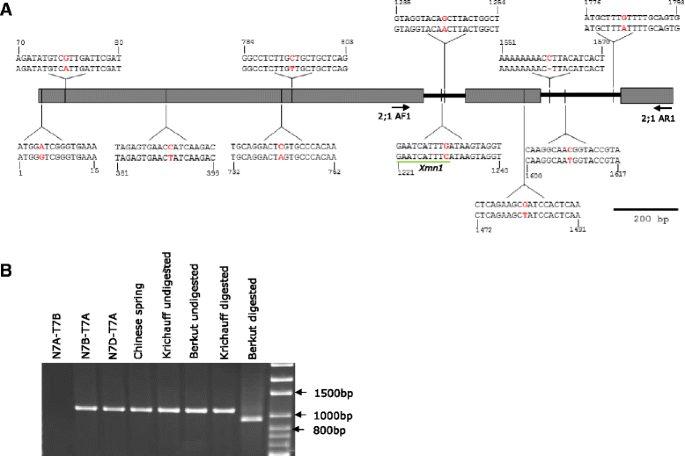
小麦序列具有身份OsHKT2;3.和OsHKT2;4在7AL中,使用该基因在7AL上进行7Al和7DL以7AL的参考作为参考,以确定能够设计基因特异性逆转录聚合酶链反应(RT-PCR)引物对的序列变体(表2)扩增FL-cDNA。RT-PCR引物被战略性地定位在假设的小麦外显子上,假设基因结构与OsHKT2;3.和OsHKT2;4(图。1A).由于7BL上的脚手架没有似乎包含对应于全长序列的序列OsHKT2;3.或OsHKT2;4,利用从7AL和7DL上相似区域设计的引物扩增7BL上基因的3 '区(图3)。1A).显示亚基因组特异性的引物对用于特异性地扩增7A、7B和7D的部分但重叠的基因转录本,并通过无染色体组-四染色体组(NT)分析进行确认(数据未显示)。随后,从根组织中扩增出7AL、7BL和7DL染色体上重叠的基因特异性cDNA,测序并组装(Genbank登录号分别为KR422354、KR422355和KR422356)。7AL、7BL和7DL上每个基因装配的FL-cDNA与支架#4510252同源基因组序列进行比对;#6569883和#3312548,确定内含子-外显子结构(图。1B.).7AL、7BL和7DL染色体上的基因与该基因结构相似OsHKT2;3.和OsHKT2;4包括3个外显子被2内内部中断,其中内含子接头位点分别具有在5'和3'边界处具有保守的基质GT和Ag。每个基因的cDNA具有<64%的DNA序列同一性Tahkt2.;1基因家族证实了这两个基因家族是截然不同的。因此,7AL、7BL和7DL的基因命名为Tahkt2.;2 7-1,Tahkt2.;2 7BL.-1和Tahkt2.;2 7dl.-1, 分别。与之相比Tahkt2.;2 7-1,外显子内的所有变体都是SNP,总计69Tahkt2.;2 7BL.-1和39Tahkt2.;2 7dl.-1相比之下Tahkt2.;2 7BL.和Tahkt2.;2 7dl.确定了76个snp。
小麦基因与水稻同源OsHKT2;3.和OsHKT2;4.一个水稻基因结构OsHKT2;3.和OsHKT2;4显示3个外显子(灰色盒子)被2个内含子(黑线).小麦基因组序列由7AL、7BL和7DL组成,与水稻的FL-cDNA同源性为80%OsHKT2;3.和OsHKT2;4显示在水稻基因结构下方。每个序列顶部的数字表示基因对每个支架上的基因的位置。小麦7bl和7dl相对于7al之间的SNP由黑色垂直线表示。PCR引物扩增小麦FL-cDNA的位置和方向由箭头表示。b小麦基因的结构,Tahkt2.;2 7-1,Tahkt2.;2 7BL.-1和Tahkt2.;2 dl.-1从FL-cDNA序列与同源基因组支架序列的比对中推断。外显子显示在灰色框中,内含子显示在水平条中。SNPs用黑色垂线表示,而黑色三角形表示相对于7AL染色体上的基因的INDELS
编码的蛋白质的分析Tahkt2.;2基因
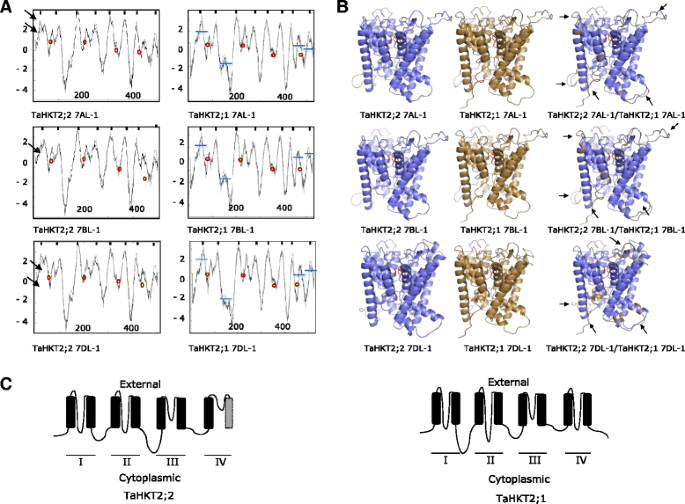
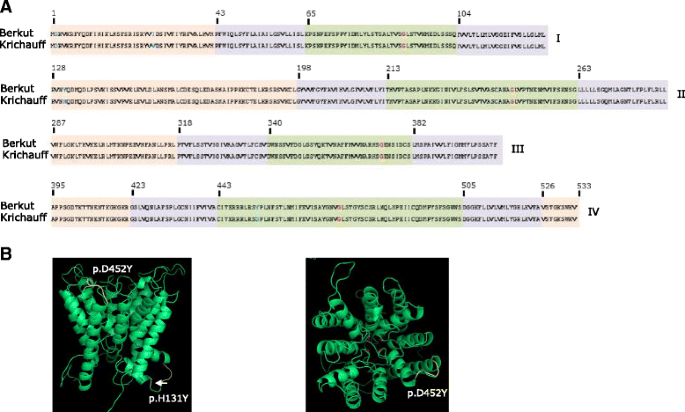
FL-cDNA测序Tahkt2.;2启用预测的翻译和编码蛋白的后续表征。每个Tahkt2.;2染色体上的基因7Al,7BL和7DL编码了508个氨基酸的预测蛋白质。疏水性图的组合分析(图。2A)和预测的3D结构(图。2B.)编码的蛋白质Tahkt2.基因允许鉴定蛋白质折叠模式和蛋白质拓扑的建模。叠加模型鉴定了每个相应蛋白质之间的折叠结构的许多差异Tahkt2.;1和Tahkt2.;2基因(图。2B.).由每个基因编码的蛋白质建模了由类似于HKT蛋白的典型核蛋白结构的四个序列排列的膜 - 倒膜结构域组成的拓扑结构(图1中的域I-IV。2C).它们各自编码的蛋白质Tahkt2.;2在p -loop中,该基因与93 - 97%的氨基酸同源性高度相似,且具有高度的序列保守性(图2)。3.).在每个蛋白质中,Gly80, Gly221, Gly349和Gly453氨基酸位置的保守甘氨酸分子组成假定的阳离子选择性过滤器(图)。3.).然而,氨基酸取代预测NH中蛋白质的物理性质的变化2终点站编码的Tahkt2.;2 7-1和Tahkt2.;2 7dl.-1相比Tahkt2.;2 7BL.-1(无花果。2A和3.).
蛋白跨膜结构域(TM)的预测Tahkt2.;2和Tahkt2.;1基因。一个每个成员编码的蛋白质的疏水性曲线Tahkt2.;2和Tahkt2.;1多基因家族。横轴表示氨基酸位置,纵轴表示疏水性值。TMs在每个图的顶部用黑盒子表示。甘氨酸过滤域的区域用红色圈出。n端物理差异用黑色箭头表示为峰值变化。TaHKT2;1相对于TaHKT2;2蛋白的峰结构差异用水平蓝线表示。b编码的单个蛋白质的3-D蛋白质模型Tahkt2.;1和Tahkt2.;2家族和由家族成员编码的预测蛋白质的叠加三维模型Tahkt2.;2和Tahkt2.;1来自同一染色体。叠加的3-D模型上的黑色箭头代表由编码的蛋白质之间的不同折叠域Tahkt2.;1和Tahkt2.;2每条染色体上的对应物。保守的过滤血清有红色表示。c由编码的蛋白质推导出的预测蛋白质拓扑的一般模型示意图Tahkt2.;2和Tahkt2.;1基因。黑色填充的矩形代表TMs和线胞质,外部和p -环域。灰色矩形表示推定的TM(疏水性值<0)
由Tahkt2.;2和Tahkt2.;1与确定相似之处和差异进行比较。由Tahkt2.;2与所编码蛋白质的氨基酸同源性小于52%Tahkt2.;1(附加文件1:图S1),表明它们有明显的不同。虽然这两个蛋白家族都具有保守的结构核心、蛋白质拓扑结构和重要的功能域,但其编码的蛋白是由Tahkt2.;2相对于Tahkt2.;1有截断NH2-末端,第二胞质结构域和羧基末端,第三胞质结构域插入四个氨基酸(附加文件1:图S1)。在羧基末端中鉴定出显着的差异,其中基于疏水性的分析和基于同源性的3D结构并未预测第4膜 - 孔膜结构的跨膜内螺旋,而是在编码的蛋白质中膜嵌入式羧基末端Tahkt2.;2基因(额外的文件1:图S1)。这与细胞质结合的羧基末端形成鲜明对比Tahkt2.;1.此外,在外部和细胞质的氨基酸变异区域引起了明显的结构差异,如图所示。2C.尽管存在不同的II族HKT蛋白,但具有保守的结构核和G-G-G-G型阳离子选择性滤波器结构域,滤波区中的氨基酸组合物的变化(附加文件1图S1),胞质和外部结构域的差异暗示了功能特征的可变性。
的成员的定量表达Tahkt2.;1和Tahkt2.;2多基因家族
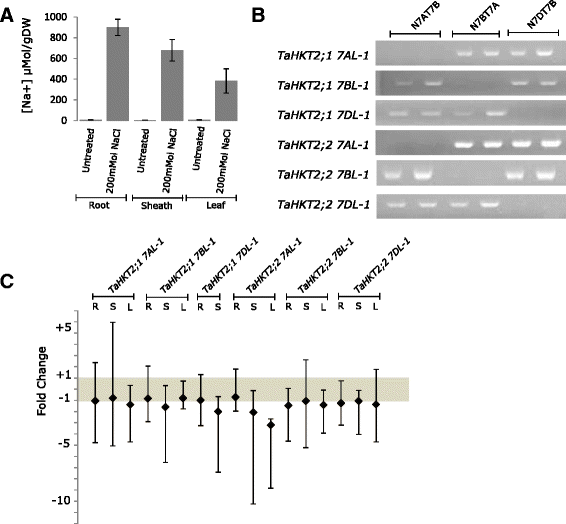
Tahkt2.;1和Tahkt2.;2在小麦幼苗(中国春季)的根,鞘和叶片组织中,分析了染色体7AL,7BL和7DL的基因。na显着更高+叶片水平(CV = 3.26%,P< 0.0001),根(cv = 3.35%,P< 0.0003)和鞘(cv = 7.73%,P与未处理的对照样品相比,200 mM NaCl处理72 h的每个组织均观察到< 0.0015)。4A因此,适用于使用定量RT-PCR(QRT-PCR)的表达。亚基因组特定引物对(表2)基于独特的多态位点设计,NT分析证实了特异性(图。4B.).QRT-PCR分析显示了成员Tahkt2.;2基因家族和Tahkt2.;1 7艾尔-1和Tahkt2.;1 7BL.-1在未处理的根、鞘和叶片组织中表达,在相同的组织中在NaCl胁迫条件下表达(图2)。4C).然而,有一个例外Tahkt2.;1 7 dl-1在鞘和根样品中检测到转录物,但不在对照或NaCl处理的样品中的叶片的QRT-PCR测定的可检测限制中。对照中的表达和200mM NaCl处理的叶片(CV = 11.39%,P> 0.88),根(cv = 13.75%,P> 0.16)和鞘(cv = 15.97%,P > 0.11) tissue samples showed thatTaActin适用于基因表达标准化,是可靠的内部参考基因,用于定量HKT.盐胁迫下的转录本。对照和NaCl处理的褶皱变化(∆∆CT = -1 to +1) usingTaActin作为内部参考表明,个体基因的表达没有差异,这可能是这些基因不是盐反应。然而,一项双重下调的规定Tahkt2.;1 7 dl-1表达和高达三倍下调调节Tahkt2.;2 7-1表达水平表明,这些基因在对照和NaCl处理条件下有差异调控(图2)。4C).
表达式Tahkt2.;2在未治疗和盐水条件下的基因。一个Na+用于qRT-PCR分析的组织样本的浓度。Na+从4个生物重复的未处理(对照)和200mM NaCl处理的组织样品的三种生物重复,测量离子浓度。b琼脂糖凝胶电泳显示II型QRT-PCR的引物对的亚基团特异性HKT.基因构件使用NT线。每一行都有副本(c).表示折叠变化的条形图使用ΔΔc估计T方法。误差条表示相对数量值的可能范围,RQ马克斯和rq.闵,由∆C的标准误差定义TS.QRT-PCR用于II组的转录分析HKT.利用未处理的根(R)、鞘(S)和叶(L)组织的cDNA和200 mM NaCl处理72 h的cDNA,对小麦(中国春季)基因家族成员进行了分析。内部控制基因TaActin用于对每个反应的初始RNA模板的变异性进行归一化。图中的灰色阴影区域突出显示了没有显著差异的折叠范围
小麦小组II的物理映射HKT.基因和启动子区Tahkt2.;2基因家族
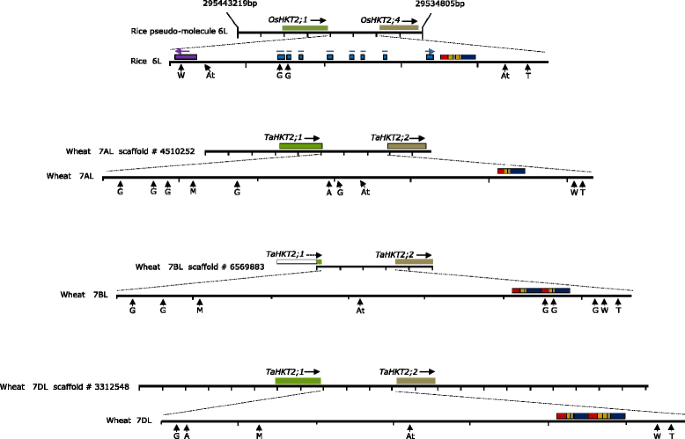
分析水稻染色体6L的假调子确定OsHKT2;1(Ordered Locus Name, LOC_Os06g48810OsHKT2;4(LOC_Os06g48800)通过2441个碱基对的物理距离分离(图。5).同样的,Tahkt2.;1和Tahkt2.;2在7AL染色体(scaffold #4510252)和7DL染色体(scaffold #3312548)上鉴定,基因间距离分别为3154碱基对和3330碱基对(图)。5).Tahkt2.;2 7BL.-1和Tahkt2.;17BL-1基因在3302个碱基对之间分离,只有部分序列Tahkt2.;1 7BL.-1,包括第3外显子、第2外显子和第2外显子的部分序列。5).虽然是第二份Tahkt2.;1 7BL.-2从contig 3599841+中检索到两个不同的支架(#6657249和#16748027),既没有识别的支架Tahkt2.;2 7BL.-1因此,小麦基因组调查序列不含具有整个长度的单个脚手架Tahkt2.;1和Tahkt2.;27bl上的基因。然而,很明显Tahkt2.;1和Tahkt2.;2小麦中的基因在7AL、7BL和7DL上物理连接,基因间距离与OsHKT2;1和OsHKT2;4水稻染色体6L的基因。
比较物理地图OsHKT2;1-OsHKT2;4水稻染色体6L和Tahkt2.;1-Tahkt2.;2小麦染色体7Al,7BL和7DL上的代际区域。序列由染色体区域由来自小麦染色体7Al,7BL和7DL的小麦染色体6L的染色体6L和支架序列表示,其中刻度间隔表示1kb。基因转录的方向由黑色箭头表示。在每个序列或支架下方扩增代际区域,其中刻度间隔代表500个碱基对。每个膨胀代际区域的右侧的串联重复单元由彩色盒子表示,其中每种颜色代表单独的重复基序。PIF / HARBINGER型螨虫元素如紫色所示,仙人掌型转座子“DTC_ISIDOR”的残余物在蓝盒中表示。盐诱导CIS.-作用要素和其他监管主旨OsHKT2;4和Tahkt2.;2启动子区域由箭头表示,包括ABRE(a),Atmyc2(AT),Mycaterd1(M),GT-1盒(G),W字幕(W)和Tata盒(T)
水稻基因间区域的大小相似OsHKT2;1和OsHKT2;4和小麦基因Tahkt2.;1和Tahkt2.;2促使了对核苷酸序列的进一步比较。7AL、7BL和7DL基因间区与基因间区DNA同源性小于40.8%OsHKT2;1和OsHKT2;4在水稻上,显示出该地区显著的多样性。在NCBI中进行的blast和tblastx分析未发现水稻或小麦基因间区域的任何表达基因具有序列相似性。然而,通过自我blast搜索,在基因间区域(翻译起始位点上游约400个碱基对)发现了直接的、串联的不完全重复Tahkt2.;2)米饭OsHKT2;4和小麦Tahkt2.;2-7BL.和Tahkt2.;2-7dl.其中每个重复基序尺寸小于70碱基对(图。5,附加文件2:图S2)。然而,在7AL的代亚基因区域中仅在2个不完美基序的两种拷贝中表示(图。5,附加文件2:图S2)。此外,一个完整的PIF/Harbinger型微型倒置转座子元(MITE),潜_Ors48,在稻米代序序列的5'区域中鉴定为a的残余物直接转矩_andidor.型DNA转座子(TREP accession number TREP3425)在中心区域(图。5,附加文件2图S2),但在小麦7AL、7BL和7DL的基因间区未发现转座子相关元件。
的Tahkt2.;1和Tahkt2.;2代表性地区也代表了启动子区Tahkt2.;27AL,7BL和7DL上的基因,并使能应激调节元素对同源染色体基因的比较分析。小麦Tahkt2.;2启动子区域揭示了同源染色体上基因的启动子之间的67-88%序列同一性(附加文件2:图S2)。守恒的CIS.在小麦染色体7AL、7BL和7DL启动子区发现了3个主要的盐诱导CREs、W-box、GT-box和AtMYC2,水稻启动子区也发现了类似的调控元件OsHKT2;4(图。5,附加文件2:图S2)。此外,这是Tahkt2.;2启动子确定了额外的CRE,包括Mycatred1和7Al,7bl和7dl,但在水稻的启动子区没有明显(图。5,附加文件2:图S2)。因此,CRE元件表明了在调节中的盐活化转录因子(TF)的作用Tahkt2.;2水稻的基因和同源基因,但个体元素的频率可变。
小麦II类的功能关联HKT.基因
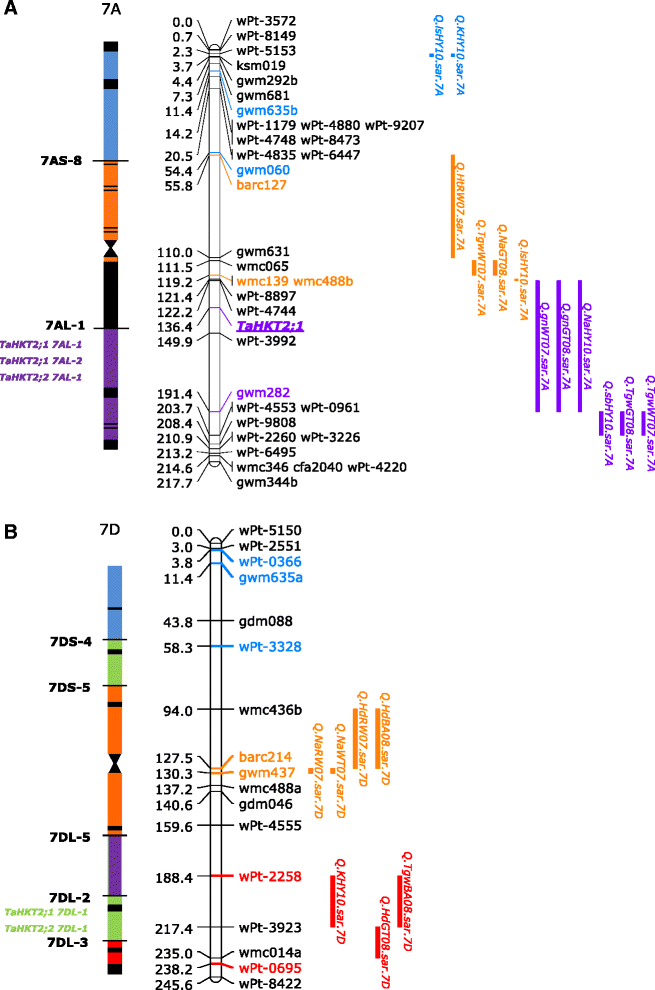
一个协会Tahkt2.;2利用缺失法和遗传作图法对盐胁迫下性状变异的基因进行了研究。缺失线分析表明Tahkt2.;2将基因分配给染色体7Al和7dl的远端,7dl和7bl的近端区域(数据未显示)和与相同的染色体垃圾箱Tahkt2.;1[13.].因此,基于删除线分析的功能分析先前报道了[13.]表示Tahkt2.;2与类似的表型相关联Tahkt2.;1盐胁迫下。为了进一步区分是否Tahkt2.;1和Tahkt2.;2在Berkut/Krichauff DH定位群体中检测到响应低盐和高盐胁迫的QTL [33.,37.基于侧翼标记的染色体箱位置集成到删除图上。总共六个QTL与染色体7Al和相同区域的远端箱对齐Tahkt2.;1 7艾尔-1和Tahkt2.;2 7-1然而,无论是Tahkt2.;1也不Tahkt2.;2与QTL共定位于7DL染色体上的同一染色体库(图。6), 7BL未鉴定QTL。因此,基因和QTL在同一染色体区域的共定位表明Tahkt2.;1 7艾尔-1和Tahkt2.;2 7-1可能有助于叶/芽Na的变异+浓度、千粒重、每米粒数2响应特定盐水环境的7AL和幼苗生物量。
删除垃圾箱和染色体7a的遗传图(一个)及7D (b)和分配Tahkt2.;1和Tahkt2.;2不同盐水环境下类似地区的基因和QTL。分配Tahkt2.;1和Tahkt2.;2在删除箱中,每条染色体的图显示在左边,而染色体连锁图的位置为tahkt2; 1(粗体和下划线)和QTL源自伯克特/ Krichauff DH映射人口[33.,37.]在右边。Tahkt2.;1和Tahkt2.;2基因,QTL和侧翼标记是根据删除垃圾箱内的分配编码的颜色编码[79.,80].在不同盐渍环境下检测到的表型QTL包括芽钠+浓度(Q.NA.)、千粒重(Q.Tgw)、幼苗生物量(Q.SB.),每米粒数2(q.gn.),叶症状(Q.ls)、株高(Q.ht.),航行日期(Q.HD.)然后射杀K+浓度(Q.K)。[37.]
协会Tahkt2.;1 7艾尔-1和Tahkt2.;2 7-1使用遗传映射分析进一步研究了特异性特性变异。Tahkt2.;1 7艾尔-1和Tahkt2.;2 7-1从Berkut和Krichauff进行PCR扩增和测序,以确定多态性,并开发基因特异性标记,用于绘制DH人群的图谱。在Berkut和Krichauff的外显子和内含子中未发现单核苷酸多态性(SNP)或插入-缺失(INDEL)多态性Tahkt2.;2 7-1,说明该基因编码的蛋白差异与7AL染色体上的性状变异无关,故排除在DH群体进一步分析之外。然而,Tahkt2.;1 7艾尔-1在Berkut (Genbank登录号KR422357)和Krichauff (Genbank登录号KR422358)之间鉴定了11个SNPs(图。7A).在翻译起始位点1230个碱基对的SNP位点上发现了一个限制性位点XMN1.贝尔库特的酶,而不是克里肖夫的酶这被用来开发一种标记物Tahkt2.;1 7艾尔-1(图。7A).利用3 ' mismatch PCR引物,2;1AF1和2;1AR1,开发了一个cleaved amplification polymorphic sequence (CAPS)标记2PCR片段的亚基因组特异性通过NT分析得到证实(图2)。7B.).的Tahkt2.;1 7艾尔-1经特异性CAPS标记酶切,鉴定出Berkut亲本的922个碱基对DNA片段,Krichauff亲本的1103个碱基对DNA片段XMN.1(无花果。7B.).对Berkut/Krichauff定位群体(150个DH系)进行基因分型Tahkt2.;1 7艾尔-1将这些数据整合到7A染色体上34个标记的现有遗传图谱中。Tahkt2.;1 7艾尔-1在遗传距离为136.4厘米的染色体7Al上映射到染色体7AL并由标记相下wpt-4744(122.2 cm)和wpt-3992(149.9 cM)+浓度(Q.NaHY10.sar.7A)每米的谷物数量2(q.gnwt07.sar.7a.,Q.gnGT08.sar.7A)。6A).因此,根据遗传作图和QTL分析,有理由认为Tahkt2.;1 7艾尔-1与特定的性状变异有关,但不是在所有的盐碱环境下。
DNA序列变异性Tahkt2.;1 7艾尔-1小麦品种Krichauff和Berkut之间的基因。一个内含子-外显子结构,外显子用灰盒表示,内含子用黑线表示。SNPs用黑色的竖线表示。显示了每个SNP侧的DNA序列和位置(碱基对),上面的序列代表Krichauff,下面的序列代表Berkut。限制性内切酶识别位点的SNP变异,XMN.1,下划线。基因特异性PCR引物对的位置Tahkt2.;1 7艾尔-1, (2;1 AF1和2;1 AR1)扩增1230碱基对SNP的黑色箭头所示。b琼脂糖凝胶电泳Tahkt2.;1 7艾尔-1基因特异性帽标记显示染色体7a的特异性,使用NT分析和大小差异为Berkut和Krichauff父母进行消化XMN.I. DNA梯子被显示在图的右侧
自从Tahkt2.;2 7-1在氨基酸残基中没有显示出任何差异,Berkut和Krichauff预测的蛋白主要集中在Tahkt2.;1 7艾尔-1进一步分析可能与性状变异有关的蛋白质折叠差异。以Chinese Spring的FL-cDNA为参考,从各亲本等位基因序列中预测编码蛋白。在氨基酸残基2、27、131和452处鉴定出了4个Berkut和Krichauff之间的氨基酸替换(图2)。8A).通过叠加来自Berkut和Krichauff的氨基酸序列构建的三维蛋白质模型预测了胞质(p.H131Y)和p-loop (p.D453Y)结构域的差异(图2)。8B.)预期蛋白质之间的功能差异。
讨论
稻米含有四个组二HKT.基因和预期,在某种情况下,总共可以在小麦中鉴定至少12个原序,假设每种正似的一份副本,具有相对相似的序列,但不一定是相同的功能。然而,这项研究中的基因组对比基因分析仅鉴定了两个不同的II族HKT.小麦的基因家庭包括前面描述的Tahkt2.;1家庭(13.]和一个新的基因家族,Tahkt2.;2,这是明显不同的Tahkt2.;1.因此,小麦和水稻似乎属于II类HKT.基因由基因复制进化而来,但它们各自独立地分化,这可能反映了它们参与了跨物种的不同生物过程。
大麦,稻米和拟南芥中的共同结构[10.]使假设是祖先的假设HKT.植物基因由三个外显子组成。本研究中描述的小麦和水稻的比较基因分析支持该假设,随后在两种物种中独立复制并分歧。本研究表明,两稻第II族HKT.基因簇,每一簇由基于系统发育关系的两个基因组成[5,16.,17.],在小麦中有直向性基因家族。小麦Tahkt2.;2基因家族在每种亚基因组上只有一种副本,并且没有显示多倍化之前或之后基因重复的证据。但是,随后的易位和分歧的复制很明显OsHKT2;3.伪分子3和OsHKT2;4在pseudomolecule 6。另一方面,有证据表明,在A和B祖先基因组中的串联基因复制早于多倍体产生多个拷贝Tahkt2.;1小麦染色体臂7AL和7BL的基因家族及其分化和伪基因化[13.类似于同源水稻基因OsHKT2;1和OsHKT2;2在染色体6上。因此,显然是一个共同的祖先的基因重复HKT.基因是II组创建的关键机制HKT.然而,在现代小麦和水稻的驯化过程中,基因拷贝可能受到不同的选择约束,导致分化没有平行进化。
分析与之间的基因区Tahkt2.;2和Tahkt2.;1和同源水稻基因OsHKT2;1和OsHKT2;4为第二组的进化提供了进一步的见解HKT.跨种类的基因。基于基因在基因之间的大型基因组片段内的DNA含量分析,已经鉴定了草基因组中的三种典型结构组织类别[38.].在两种或两种以上禾草物种中,基因间区域的物理大小可能存在显著差异,从几个碱基到几十个碱基大小不等,这主要是由于转座因子和其他重复元素的差异积累[39.].或者,基因组的区域可以具有高基因含量,由此节约地区的编码区和物理距离的尺寸[40[表明基因组区域可能不一定容易易于转换和其他重复元件的增殖。最后,患有跨越不同尺寸的大型基因区域的物种具有类似的基因密度的保守的高基因密度。41.].本研究确定了II族HKT.小麦和水稻的基因在基因间区域具有相似的物理距离,因此,进化机制被认为是跨物种保持基因在物理上相近,可能是保持基因调控、共同表达或共同功能的一种手段[42.,43.].在基因间区发现了螨和转座因子的残体OsHKT2;1和OsHKT2;4在水稻中,这表明逆转录转座事件可能是明显的,但没有持续,因此在进化过程中被消除,留下转座“足迹”。小麦的同源区不存在明显的转座因子残留,但低复杂性不完全重复是小麦基因间区的主要特征Tahkt2.;1和Tahkt2.;2在小麦7bl,7dl和水稻中,但在小麦7al中没有。在染色体和物种之间的短重复图中观察到的差异可以从复制错位和聚合酶滑动中产生,导致重复单元的缺失或添加[44.,45.].因此,显然是那些之间的代际区域Tahkt2.;1和Tahkt2.;2在小麦和局部地区之间OsHKT2;1和OsHKT2;4在水稻中不利于大小的重大变化和保护物种内部的物理距离可能会涉及保存一些基因功能。
这项研究提供了证据,支持水稻和小麦基因相似的物理接近是一种进化手段,以维持相似的基因调节跨物种。Tahkt2.;2基因家族在根、鞘和叶片组织中表达Tahkt2.;1基因在根和鞘中表达,但在叶片中转录量较低。同源水稻基因表现出类似的表达模式OsHKT2;4在根、鞘和叶片组织中表达[20.] 和OsHKT2;1在根和鞘中表达,而在叶片中表达下调[21.]尽管物种之间的核心区中CRE的数量和类型的变异性。因此,在正非之间的类似小物理距离是合理的HKT.基因可能是一个进化机制,以保护大米和小麦中的基因表达。有趣的是,转录物水平的一致表达和维持表明,两只小麦族IIHKT.基因家族可能以协调的方式相互作用,并可能与其他基因(如群I)发生内聚力HKT.基因不受盐环境的影响。在水稻中也观察到类似的基因相互作用,导致对盐胁迫的有组织的反应HKT.基因OsHKT1;4和OsHKT1;5[46.]为小麦提供成绩单协调模型HKT.基因。第二组的方式HKT.基因在生物系统中是如何协调对盐度的反应的,这一问题还有待进一步研究。
两组II之间的代际区域HKT.每条同源染色体上的基因也占据启动子区域Tahkt2.;2基因。除了存在于不同同源基因中的可变数量的CRE外,启动子区域内特定的直接不完全重复基元可以作为基因转录的抑制子或增强子,通常作为推定的转录因子结合域[47.- - - - - -50.].有趣的是,在启动子中发现了对基因表达和调控重要的串联不完全直接重复HKT.基因拟南芥,AtHKT1;1,其中重复的变异或不存在导致对升高的NaCl的显着转录反应[51.].因此,有权进一步研究短重复元素,以确定其在调节中的作用Tahkt2.;2在本研究中分析的不同植物发展阶段的转录和不同的盐水条件。
转录性丰富的折叠差异Tahkt2.;1 7 dl-1和TaHKT2;2 7AL-1可能参与了Na的控制+以组织特有的方式积累。尽管在启动子区存在多种盐诱导的CREs,但大多数II组HKT.在本试验条件下,NaCl处理对根、鞘和叶片的转录没有显著影响。因此,缺乏对盐的反应,使人们对某些基因的转录意义产生了一些怀疑Tahkt2.Na.的基因+在水培条件下的根部,鞘或叶片运输。对I群体观察了类似的结果HKT.会员在那里TmHKT1;4在盐碱条件下,转录本丰度没有变化,但与Na+排除占表型变异的25%,表明转录性丰度可能不仅可以在控制NA中解释基因作用+水平(52.].我们不能排除某些第二组的可能性HKT.s在其他组织和不同环境中盐胁迫下调,或者实际上,在控制NA中起着非转录作用+积累。因此,涉及整个工厂分析的系统和综合研究将更加全面地了解II集团所有成员的协调转录规则和互动塔赫克控制na的基因家族+积累。
两组编码的蛋白质有明显的差异HKT.小麦基因家族预测其功能变异。编码的蛋白质的关键功能域Tahkt2.;1 7BL.-1显示对阳离子选择性和离子传输性能很重要[53.- - - - - -56.]并用作在编码的蛋白质中推断出功能变异的基础Tahkt2.;2.结构建模并叠加着纳的已知功能域+和K+在编码的蛋白质中运输Tahkt2.;1 7BL.-1在被编码的蛋白质中识别出变异Tahkt2.;2.北半球2和羧基末端是HKT蛋白质的基本功能域,影响k+选择性[54.].NH的长度和氨基酸组成2而羧基末端在两个蛋白家族中有明显的差异,截断的结构域可能导致非功能蛋白或具有显著的功能修饰。t4 - p - tm结构域内螺旋中的高度保守的正电荷氨基酸对盐桥和K的形成至关重要+选择性转运仪活动[57.,58.].然而,物种之间的HKT相关蛋白质具有相似的结构芯,并且这些带正电荷的氨基酸不存在于植物K中+渠道(57.].因此,进一步的研究如异源表达将是值得的,以破译截断的羧基端编码的蛋白质Tahkt2.;2具有频道相同的特性的运输扣活动。有趣的是,米汤编码的蛋白质OsHKT2;4在异源表达中表现出不同的动力学,包括钙可渗透的弱选择性阳离子通道,这为II组HKT蛋白的替代功能模式提供了证据[20.,22.,59.].此外,在水稻基因编码的蛋白质中,还发现了等位变异间单个氨基酸替换引起的胞质和外部结构域的差异OsHKT1;4[60.] 和拟南芥AtHKT1;1[61.和类似的变异预测了被编码蛋白质家族的功能差异Tahkt2.;2与编码的蛋白质相比Tahkt2.;1.除了细胞质和外部结构域的一些氨基酸替换外,该蛋白由Tahkt2.;2与由编码的蛋白质相比,鉴定了氨基酸残基216的丝氨酸的丙氨酸取代Tahkt2.;1 7BL.-1这可能对离子传输活动产生显着影响。ala的替代216近孔区的入口降低了na+编码的蛋白质的运输能力Tahkt2.;1 7BL.-1[62.,因此,在这个残基上的一个类似的变化暗示了被修饰的蛋白质的离子转运活性Tahkt2.;2.因此,虽然保守的蛋白质结构核和共享的G-G-G-G-G型阳离子选择性滤波器预测了一些功能相似性Tahkt2.;2和Tahkt2.;1(例如,运输k的能力+和na+),编码的蛋白质可以基于修饰的蛋白质结构域和临界功能域中的氨基酸变化具有离子选择性的多样性。
II组的植物内功能HKT.在小麦的情况下,运输扣难以理解,但盐条件下的Aenuploid系的表型分析为其在植物中控制离子积累方面的潜在作用提供了新的了解。小麦删除线为nullTahkt2.;1在染色体7Al中,7BL和7DL没有显示根NA的显着差异+因此,该基因家族的成员被认为参与了Na+根系向鞘的转运和钾的调控+而不是通过根部从外部介质输入离子[13.].因为Tahkt2.;2基因家族被分配到同一个缺失库中Tahkt2.;1在7AL、7BL和7DL染色体上,两者均不明显Tahkt2.;2也不Tahkt2.;1主要基因为主根钠+尽管两个基因都在根中表达,但从外部培养基中摄取。然而,其中一个或两个基因都可能参与Na的调控+从根到鞘或维持k+在未处理和盐处理条件下的水平。有趣的是,这Tahkt2.;2在大米直接同源,OsHKT2;4介导了一个稳健的低亲和力K+在异源表达系统中运输[59.]然而OsHKT2;4敲除突变体在不同离子条件下并没有产生相似的表型[22.],表明该基因能够但对运输k不重要+而在植物中更可能依赖于其他基因和蛋白质的贡献的一个更复杂的系统。功能联想Tahkt2.;1和Tahkt2.;1因此,进一步研究了性状变异。
删除垃圾箱和遗传映射与QTL分析相结合用作助理组II的指南HKT.盐水环境下具有特质变异的基因。虽然这些资源提供了低分辨率分析,但我们能够证明这一点Tahkt2.;1和Tahkt2.;2基因与染色体7bl和7dl上的盐环境下的一些特征无关。但是,位置Tahkt2.;2和Tahkt2.;1与叶梢Na的QTL在同一个染色体缺失库中被鉴定+不同盐度环境下浓度及产量的测量[33.,37.],表明它们潜在参与表型变异。外显子和内含子Tahkt2.;2 7-1在Berkut和Krichauff之间是相同的,因此,无法分配到DH人口中的遗传地图。然而,结论是,它会过早Tahkt2.;2 7-1由于该基因在植物发育的不同阶段可能存在转录、转录后或翻译后调控,在不同的盐环境下可能影响表型变异,因此不影响特定的表型。内含子和外显子内的DNA序列变异Tahkt2.;1 7艾尔-1允许具有QTL的低分辨率但遗传关联,预测该基因在控制NA方面的作用+排除水培和田间条件下,特定盐渍环境下的粒数和粒重。但是QTL的遗传距离和物理距离较大,我们不能排除除QTL以外的基因Tahkt2.;1 7艾尔-1在间隔中有助于表型变异。良好的分辨率遗传映射将确认作用Tahkt2.;1 7艾尔-1在不同盐水环境下控制一些特征的变化的基因和其他基因。虽然在同一删除箱内检测到另外三个QTL,但是Tahkt2.;1 7艾尔-1不同盐碱条件下千粒重和幼苗生物量的表型变异不太可能与同一作图区间内共定位有关。因此,这些性状的遗传控制很可能是由其他基因引起的Tahkt2.家庭。Berkut等位基因编码的蛋白质的结构模型和变异Tahkt2.;1 7艾尔-1在氨基酸残基452上发现了一个酪氨酸取代,该氨基酸位于第4 P-loop区域的高度保守区域附近,该区域假设一个更大的中性氨基酸比Krichauff编码的蛋白质中带负电荷的天冬氨酸更疏水。生物过程的基因本体论[63., Interpro蛋白结构域搜索[64.]和突变研究[53.]预测第4 p环地区是罗南道的关键地区+小麦的结合、运输和作用HKT.使用缺失线分析控制从根到叶片到叶片的离子输送的基因[13.,进一步证明Berkut等位基因编码的蛋白质可能控制Na+植物内的运输影响特定但不是所有盐水环境下的一些产量相关的性状。
结论
面包用小麦含有两种明显不同的第二类HKT.基因家庭,Tahkt2.;1和Tahkt2.;2与水稻的四个单独基因相比。因此,在两个物种的进化和驯化过程中,一个共同的祖先基因的重复,包含三个外显子,然后出现独立的差异。小麦集团II的新成员HKT.,Tahkt2.;2从具有职业染色体组7L和距相关基因家族的短(〜3k碱基对)物理距离的特征Tahkt2.;1.的Tahkt2.;2多基因家族由3个推测的功能基因组成,分别在对照和200 mM NaCl胁迫条件下的根、鞘和叶片中表达,与对照和200 mM NaCl胁迫条件下表达差异显著Tahkt2.;1.由Tahkt2.;2基因相似但不完全相同的编码Tahkt2.;1. Tahkt2.;2, 如Tahkt2.;1基因家族,不太可能负责根钠+从外部培养基摄取,但可能参与运输NA+从根到叶片或调节钾+运输也可能在植物中具有更广泛的离子选择性和运输。此外,编码的蛋白质Tahkt2.;1 7艾尔-1在一定的盐碱环境下,可能与产量相关性状有关,但QTL区间内的其他基因也可能参与性状变异。高分辨率的遗传作图和QTL分析将为该基因的作用提供进一步的证据Tahkt2.;1 7艾尔-1在不同盐水环境下控制特质变异的其他基因。
方法
植物材料
面包小麦品种Triticum aestivum.L. cv。中国春季,六种含有六种缺斑 - 四元素线(N7AT7B,N7AT7D,N7BT7A,N7BT7D,N7DT7A,N7DT7B),八条缺失线(7AL-1,7AS-8,7BL-2,7BL-7,7DL-2,7DL-3,美国堪萨斯州立大学堪萨斯州立大学博士Raupp,小麦遗传和基因组资源中心,讨论了7dl-5,7ds-5)和两条dt7bl和dt7bs)。先前报道了由Berkut和Krichuaff之间的F1交叉的DH映射群体是报道的[33.].
水稻ⅱ类小麦同源基因的数据库检索与鉴定HKT.基因
第II族HKT.基因OsHKT2;1,OsHKT2;2,OsHKT2;3.和OsHKT2;4并在NCBI(GenBank登录号AB061311,AB061313,AJ491819和AJ491854)处访问它们的相应的FL-cDNA。米FL-cDNA用作Blastn和Tblastx中的查询序列,以搜索小麦测量序列数据库(https://urgi.versailles.inra.fr/blast/blast.php).blast搜索结果显示与水稻FL-cDNA序列有显著命中(E = 0.0),鉴定出含有相关基因序列的小麦支架。获得了每个小麦支架内与水稻基因FL-cDNA序列高度一致的区域,并将目标序列与小麦II组HKTs的FL-cDNA进行了比较,Tahkt2.;1(Genbank登录号码,KJ540616,U16709,KJ540618)通过局部对齐使用肠道6.0.3中的Clustalw功能[65.].
底漆设计和PCR扩增
小麦序列之间的多个序列使用毒毒性6.0.3对齐[65.]和亚基因组特异性序列变异的人工扫描。针对3 '错配设计PCR引物并定位[66.以获得基因特异性扩增子。NetPrimer (http://www.premierbiosoft.com/netprimer/netprlaunch/netprlaunch.html.)软件用于确认引物兼容性和退火温度。底漆细节在表中提供2.
使用基于苯酚 - 氯仿的方法提取来自小麦间倍增管的基因组DNA [67.使用50ng基因组DNA作为PCR扩增模板。PCR反应含有0.5μm的每个引物(表2), 1× PCR缓冲液,1.5 mM氯化镁2,每个脱氧核糖核酸0.2 mM, 1 UTaq.DNA聚合酶(Biotaq™DNA聚合酶,Bioline,澳大利亚)在20μl反应体积中。标准PCR的循环参数为35个94℃30s,引物退火温度的循环(表2)在72°C时为30 s,72℃45 s和最终延伸部7分钟;对于触摸,PCR是94°C 30 S,55-50°C 30 S,72℃45秒的五个循环。然后在72℃下35个94℃,50℃,50℃,72℃,70℃的循环7分钟。PCR产物在0.5×Tris-醋酸凝胶中分离在0.5×Tris-乙酸盐EDTA的恒定电压(90V),30分钟,凝胶用溴化乙锭染色并使用凝胶DOC系统(意大利Biorad,意大利)在UV光下可视化。通过使用含氟类 - 四元素N7AT7B,N7AT7D,N7BT7A,N7BT7D,N7DT7A,N7DT7B作为模板DNA,通过从基因特异性PCR的基因特异性PCR的扩增子确认PCR引物的亚基因组特异性。
FL-cDNA克隆和测序
植物材料的开发,从根系组织中提取的总RNA提取和cDNA的合成,如下所述进行。设计基因特异性引物被设计成扩增用于全长小麦基因的预测编码区域的多个重叠cDNA片段(表2).基因特异性RT-PCR引物,包括2; 2AF1 / 2; 2AR1,2; 2AF2 / 2; 2R2,2; 2BF1 / 2; 2BR1,2; 2BF2 / 2; 2BR2,2; 2BF3 / 2; 2ADR1,2; 2ADCF1/ 2; 2dr1,2; 2df2 / 2; 2adr1(表2)用于扩增根组织的cDNA。每个引物组合在退火温度下进行RT-PCR(表2),在20 μl PCR反应混合物中加入0.5 μM引物2), 1× PCR缓冲液,1.5 mM氯化镁2,每个脱氧核糖核酸0.2 mM, 1 UTaq.DNA聚合酶(Biotaq™DNA聚合酶,Bioline,澳大利亚)。标准PCR的热循环条件为35次94℃30s,底漆退火温度(表2)在72°C时为30 s,72℃45 s和最终延伸部7分钟;对于触摸,PCR是94°C 30 S,55-50°C 30 S,72℃45秒的五个循环。然后在72℃下35个94℃,50℃,50℃,72℃,70℃的循环7分钟。RT-PCR产物在0.5×Tris-醋酸凝胶中在恒定电压(90V)中在1%琼脂糖EDTA上分离30分钟,并在用溴化乙锭染色后使用GEL DOC系统(Biorad,意大利)在UV光下可视化。的RT-PCR amplicons were excised from agarose gels, purified using Wizard® SV Gel and PCR Clean-Up System (Promega, CA, USA), and cloned into pGEM®-T Easy Vector Systems as per manufacturer’s recommendation (Promega, CA, USA). DNA templates from 3 recombinant bacterial colonies and representing each overlapping cDNA fragment were isolated and purified by Wizard® Plus SV Minipreps as per manufacturer’s recommendation (Promega, CA, USA) and sequenced using BigDye™ sequencing chemistry (Applied Biosystems, Perkin Elmer, Weiterstadt, Germany) using the M13 universal primers Forward primer (5’-CGCCAGGGTTTTCCCAGTCACGAC-3’) and Reverse primer 5‘-TCACACAGGAAACAGCTATGAC-3’). Sequences of overlapping cDNA for each wheat gene were assembled and aligned with the cognate genomic DNA sequence using GENEIOUS 6.0.3 [65.].
硅蛋白分析和3-D型造型
从小麦基因的FL-cDNA推导出蛋白质,使用毒性6.0.3获得预测蛋白质的氨基酸序列比定[65.].利用TopRED2分析预测蛋白的疏水性和膜拓扑[68.]及MPEx 3.2 [69.使用默认参数的软件。通过Phyre 2预测蛋白质的三维结构[70].Pymol Viewer和分子图形系统,1.5.0.4(Schrödinger,LLC)分析了3-D模型。
基因间和启动子区域的硅分析
第二组之间的基因间区域HKT.从水稻基因组浏览器版本中检索基因7(http://rice.plantbiology.msu.edu/)和国际小麦基因组序列(http://www.wheatgenome.org/).以blastn、tblastx和基因间区域为查询工具搜索NCBI核苷酸和蛋白数据库,以e值为0的表达序列标签、partial或FL-cDNA识别DNA序列。使用重复遮罩(http://www.repeatmasker.org/)及EMBOSS倒置[71.] 软件。通过在NCBI Blastn Suite中的“对准序列核苷酸爆炸”中通过自我进行旋转重复序列来鉴定串联重复序列。通过TREP中的BLASTN搜索功能进一步分析横向重复[72.]及p -螨(植物螨)[73.)数据库。C是在Plantcare中的BLASTN搜索鉴定了与促进子区域中与盐应激反应相关的调节元件[74.]并放置[75.)数据库。
用于表达分析的组织样本的水培筛选和开发
面包的种子t . aestivumvar。在0.04%次氯酸钠(42g / L)中洗涤中国弹簧,在暗条件下在漂浮的0.1强度营养溶液中彻底漂洗在去离子水中,在浮动丝网上彻底漂洗。全强度的营养成分为:(mm)k+,3.95;加利福尼亚州2+4;毫克2+0.4;NH4+,0.625;不3.−4.375;所以42−,1.9;HPO.42−0.2;Fe-EDTA 0.05;市场经济地位,1.0;霍格兰溶液中四分之一浓度的微量营养素(使用KOH调整pH至6.5)。3 d后,移栽至0.25强度营养液中光照1 d。然后将4天龄的幼苗转移到加气的、箔覆盖的4.5 L花盆中的全强度营养液中。在花盆盖上的小孔中插入泡沫支架,将幼苗直立放置。采用完全随机设计,每个处理4个重复。实验在温度控制的植物加速器(昼/夜20±3℃/15±2℃)中进行,正午记录的光合有效辐射约为1400 ~ 1500 μmol m−2 s−1。用去离子水灌满锅中以维持溶液浓度。
在实验开始后十天,在2.2千叶发育阶段,200mM NaCl治疗在12小时间隔为50毫米的指定处理罐的治疗,并将植物保持在最终200mM浓度的完整3天(72H)。在对照盆中没有NaCl。72小时后,散装叶片的样品,每株植物的护套和根部被分别对组织NA进行进行+分析。在切除前,根在含有4 mM CaSO的溶液中清洗3次,每次10秒4和368毫米甘露醇(200毫米处理的植物)或在4毫米CaSO4(不添加NaCl的植物)。叶片和叶鞘样品用去离子水冲洗。所有用于离子分析的组织样本都在70°C的烤箱中烘干,称重,然后研磨成粉末。干燥粉末状组织样品(100 mg)提取于5 ml 0.5 M HNO中3.在黑暗条件下机械振动筛3天。组织Na.+使用火焰光度计(Sherwood 410, Cambridge)在技术重复中测量浓度,平均值代表每个处理重复的组织样本。通过对参照组织(花椰菜,亚太植物编号85)的分析,验证了该方法的可靠性。
RNA提取和RT-PCR
小麦未经用盐处理幼苗的发展t . aestivum如前所述,《中国的春天》已完成。处理72 h后,从对照植株和处理植株中收获大量叶片、叶鞘和根系,在液氮中快速冷冻2提取总RNA。通过结合TRIzol协议(Invitrogen, Carlsbad, CA)和ISOLATE II RNA Plant Kit (Bioline USA Inc, Taunton)的改良方案,从冷冻组织中提取总RNA。将大约100mg的组织样本在液态氮中研磨2每100 g研磨组织加入1 mL TRIzol试剂。样品在TRIzol中室温孵育5分钟,每1ml TRIzol用0.2 ml氯仿手用力摇晃15-30 s提取。室温孵育2 min, 4℃12,000 g离心15 min。将水相转移到一个干净的无RNase管中,避开间相,加入0.5 ml异丙醇,室温孵育10 min, RNA沉淀。将异丙醇混合物装入从ISOLATE II RNA Plant Kit (Bioline USA Inc, Taunton)中分离出来的ISOLATE II RNA Plant柱中,按照制造商的建议继续提取。用紫外分光光度法在260和280 nm处测定RNA纯度和数量260/一个280.~ 1.9和A260/一个230 > 2). First strand cDNA was synthesised using SensiFAST™ cDNA Synthesis Kit (Bioline USA Inc, Taunton) using 1 μg of total RNA in a 20 μl reaction following instructions by the manufacturer. Integrity of synthesised cDNA was verified by RT-PCR using primers designed for glyceraldehyde-3-phosphate dehydrogenase (GAPDH) gene (Table2).
定量实时PCR(QRT-PCR)
qRT-PCR反应在Rotor-Gene™3000 (Corbett Research Ltd, UK)中进行,使用SensiMix SYBR®No-ROX混合(Bioline USA Inc, Taunton)主混合。利用叶片、根和鞘组织3个独立的生物重复,分析了对照和200 mM NaCl处理条件下各基因的转录丰度。每个生物样品采用三个技术重复。qRT-PCR反应的最终体积为10 μl,包含5 μl SensiMix SYBR®No-ROX mix (Bioline USA Inc, Taunton),每个引物0.4 μM2)和3μl101010稀释的合成cDNA,并使用以下条件进行分析:在94℃下3分钟,94℃下的40℃,60℃,10s,72℃。在底漆退火步骤完成时获得SYBR信号。通过熔化曲线分析(具有0.5℃/ min的加热速率为0.5℃/ min)和琼脂糖凝胶电泳来证实QRT-PCR的完整性和特异性。CDNA样品用作使用三个内部参考基因的稳定性分析的模板泰法,GAPDH和TaActin使用类似的QRT-PCR反应条件。C.T使用对照和200mM NaCl处理的叶片,根和鞘cDNA的每个基因获得的值进行分析,方差分析,选择样品中稳定表达的基因作为内部参考表达归一化Tahkt2.基因。6个感兴趣基因和内参基因的引物序列列于表中2.通过2估计NaCl处理样品中转录物丰度的相对变化-ΔΔct方法 [76.].标准误差2C -∆∆通过预测RQ闵和rq.最大限度[77.,78.].
删除本映射
使用八条缺失线7Al-1,7As-8,7BL-2,7BL-7,7DL-2,7DL-3,7DL-5和7DS-5和DITELOSOMIC系DT7BL和DT7BS映射小麦基因。通过先前描述的苯酚 - 氯仿的方法提取来自缺失和Diteloomic系的基因组DNA [67.]并用作PCR扩增模板。通过使用引物对2的基因特异性PCR的相应缺失或Diteloomic系或不存在扩增子在每个缺失箱内的基因的位置; 2AF1 / 2; 2AR1,2; 2CF1 / 2; 2BR1和2; 2ADCF1/ 2; 2DR1(表2).PCR反应条件与上述相同。PCR产物在0.5× Tris-acetate EDTA(恒压(90 V) 30分钟)1%琼脂糖凝胶中分析,用溴化乙啶染色后,在紫外光下使用gel Doc System (BioRad,意大利)进行可视化。
Na的侧翼标记的染色体箱位置+在Berkut/ Krichauff DH群体中报道的与盐相关性状相关的qtl和其他qtl [33.的删除库映射得到[79.,80].bin的位置wmc139通过PCR使用缺失系为模板DNA和引物5'-TGTAACTGAGGGCCATGAAT-3'和5'-CatcgactcacaActGGGGT-3'通过PCR进行实验确定的SSR标记物。http://wheat.pw.usda.gov/gg3/).如前所述,在65-55°C下进行着陆PCR。
Cleaved amplified polymorphic sequence (CAPS)标记物的开发
Tahkt2.;2 7-1如前所述,利用基因特异性引物从Berkut和Krichauff基因扩增、克隆和测序[13.].类似的Tahkt2.;2 7-1利用引物2;2AF1/2;2AR1, 2;2AF2/2;2AR2, 2;2AF3/2;采用GENEIOUS 6.0.3对基因序列进行多序列比对,寻找DNA多态性[65.].建立了Berkut和Krichauff亲本的基因特异性CAPS标记多态性Tahkt2.;1 7艾尔-1引物2;1AF1和22)在上述的PCR反应和热循环条件下。PCR产物(10 μl)经10 U酶切XMN.1(Promega,CA,USA)通过制造商推荐(Promega,CA,USA),在37℃下孵育2小时。将消化的产物在0.5%琼脂糖上以1.5%琼脂糖(恒压为90V的30分钟)分离,并在用溴化乙锭染色后使用凝胶DOC系统(Biorad,意大利)在UV光下可视化。
II族的遗传映射HKT.S.
来自Berkut/Krichauff定位群体的150个DH系在温室中生长,并使用一种基于苯酚-氯仿的方法从叶片材料中提取基因组DNA,该方法见[67.],通过Nanodrop_nd_1000 V3.2的光谱仪(Thermo Fisher Inc.,De,USA)和DNA浓度量化至50ng /μl。150次数是基因分型Tahkt2.;1 7艾尔-1基因特异性概述标记和等位基因数据集成到现有的简单序列重复(SSR)和分集阵列技术(DART)标记的现有遗传图中(DART)标记33.].标记等位基因数据Tahkt2.;1 7艾尔-1使用“分发”命令分配在遗传映射中,链接条件= 0.01,映射函数= kosambi,后跟MapManagerQTX版本Qtxb20中的“纹波”命令[81.].使用MapChart V2.2图形呈现染色体连杆和QTL间隔[82.].
提供支持数据的可用性
支持本文结果的数据包含在文章及其附加文件中。
缩写
- 帽子:
-
剪切扩增多态性序列
- CRE:
-
CIS.- 处理监管要素
- 镖:
-
多样性阵列技术
- DH:
-
加倍单倍体
- FL-cDNA:
-
全长cDNA
- GAPDH:
-
甘油醛-3-磷酸脱氢酶
- HKT:
-
高亲和力钾转运体
- INDEL:
-
insertion-deletion
- 螨虫:
-
微型倒置的转座子元素
- NACL:
-
氯化钠
- NT:
-
无损伤 - 四元肿
- 存在:
-
定量逆转录聚合酶链反应
- QTL:
-
定量特质基因座
- RT-PCR:
-
逆转录聚合酶链反应
- SNP:
-
单核苷酸多态性
- 紧急求救信号:
-
盐过度敏感
- 苏维埃社会主义共和国:
-
简单的序列重复
- TF:
-
转录因子
参考
- 1。
植物耐盐机理研究。植物学报2008;59:651-81。
- 2。
James RA, Davenport RJ, Munns R.两个Na基因的生理特性+杜兰姆小麦排除,纳克隆和NAX2.植物杂志。2006;142(4):1537 - 47岁。
- 3。
杨Q,陈祖兹,周XF,尹HB,Li X,Xin XF等。过度表达紧急求救信号(盐过敏的)基因增加转基因拟南芥的耐盐性。莫尔植物。2009; 2(1):22-31。
- 4.
Byrt CS, Platten JD, Spielmeyer W, James RA, Lagudah ES, Dennis ES等。HKT1;5类阳离子转运体与Na相连+小麦中的排除基因座,NAX2和KNA1.植物杂志。2007;143(4):1918 - 28。
- 5。
Hauser F,Horie T.保护的初级耐盐机制介导HKT.运输器:高钾钠和维护的机制+/ Na+盐度应力期间叶子中的比率。植物细胞环境。2010; 33(4):552-65。
- 6。
Moller IS, Gilliham M, Jha D, Mayo GM, Roy SJ, Coates JC,等。细胞型特异性Na改变诱导芽胞排斥Na +和提高耐盐性+拟南芥的运输。植物细胞。2009; 21(7):2163-78。
- 7。
Rus A,Yokoi S,Sharkhuu A,Reddy M,Lee BH,Matsumoto TK等。AtHKT1是控制na的耐盐性决定因素+进入植物根源。PROC NATL ACAD SCI U S A. 2001; 98(24):14150-5。
- 8。
Davenport RJ, Munoz-Mayor A, Jha D, Essah PA, Rus A, Tester M. Na +转运体AtHKT1;1控制钠的提取+从拟南芥的Xylem。植物细胞环境。2007; 30(4):497-507。
- 9。
江江文,菅原俊,奥库诺,中山县,施eder JI, Shinmyo A,等。的功能HKT.钠离子在根中的运输和保护叶免受盐胁迫的转运体。生物科技植物》。2008;25:233-9。
- 10.
Platten JD, Cotsaftis O, Berthomieu P, Bohnert H, Davenport RJ, Fairbairn DJ,等。命名为HKT.转运体,植物耐盐性的关键决定因素。植物科学进展。2006;11(8):372-4。
- 11.
Laurie S, Feeney KA, Maathuis FJ, Heard PJ, Brown SJ, Leigh RA。一个角色HKT1小麦根系对钠的吸收植物j . 2002; 32(2): 139 - 49。
- 12.
Schachtman DP,Schroeder ji。高亲和力钾摄取转运蛋白从高等植物的结构和运输机理。自然。1994年; 370:655-8。
- 13。
Ariyarathna HC,Ul-Haq T,Colmer Td,Francki Mg。多尾家族的特征TaHKT 2;1在面包小麦和基因成员在植物NA中的作用+和K+的地位。植物生态学报。2014;14(1):159。
- 14。
Horie T, Yoshida K, Nakayama H, Yamada K, Oiki S, Shinmyo aHKT.具有不同性质的运输扣+和K+运输栽培稻.植物j . 2001; 27(2): 129 - 38。
- 15.
Oomen RJ, Benito B, Sentenac H, Rodriguez-Navarro A, Talon M, Very AA,等。HKT2.;2/1一个K+通过自然遗传多态性调查鉴定耐盐水稻品种的渗透转运体。工厂j . 2012, 71(5): 750 - 62。
- 16。
钾和钠在非动物细胞中的转运:非动物细胞中的钾和钠转运:非动物细胞中的钾和钠转运:非动物细胞中的钾和钠转运载重汽车/Ktr/HKT.转运体家族。中国生物医学工程学报。2010;32(4):457 - 461。
- 17。
Garciadeblas B, Senn ME, Banuelos MA, Rodríguez‐Navarro aHKT.运输者:大米模型。植物j . 2003; 34(6): 788 - 801。
- 18。
Gomez-Porras JL, Riano-Pachon DM, Benito B, Haro R, Sklodowski K, Rodriguez-Navarro A, et al.;K+苔藓植物、石松和开花植物中的转运体表明维管植物的特化。植物学报2012;3:167。
- 19。
引用本文:马志强,马志强,林德伯格OsHKT1,OsHKT2,osvha.在NaCl胁迫下,盐敏感型和耐盐型水稻(栽培稻L.)品种。J Exp Bot。2006; 57(15):4257-68。
- 20.
兰伟忠,王伟,王思明,李乐国,Buchanan BB,林海霞,等。水稻高亲和钾转运体(HKT.)隐藏一个钙渗透阳离子通道。中国科学院院刊2010;107(15):7089-94。
- 21.
Horie T,Costa A,Kim Th,Han MJ,Horie R,Leung Hy等。米OsHKT2;1转运体介导大钠+将涌入组件进入k+为生长而挨饿的根。中国海洋大学学报(自然科学版);
- 22.
Horie T,Brodsky De,Costa A,Kaneko T,Lo Schiavo F,Katsuhara M,等。K.+运输到OsHKT2;4来自米饭的运输车与非典型na+K的输运性质和渗透竞争+在毫克2+和加利福尼亚州2+离子。植物理性。2011; 156(3):1493-507。
- 23.
姚晓东,薛松,梁海英,Katsuhara M, Brodsky DE,等。水稻钠钾运输选择性的差异OsHKT2;1和OsHKT2;2植物细胞中的转运体。植物杂志。2010;152(1):341 - 55。
- 24.
国际水稻基因组测序P.基于图谱的水稻基因组序列。自然。2005;436(7052):793 - 800。
- 25.
Mayer KF,Rogers J,DoleëelJ,Pozniak C,Eversole K,Feuillet C等人。基于染色体的六倍面包小麦的序列(Triticum aestivum.)基因组。科学。2014;345(6194):1251788。1 - 11。
- 26.
Crawford Ac,Francki mg。类胡萝卜素生物合成途径的小麦基因的染色体位置以及与面粉B *颜色变化相关的染色体7a上的过氧化氢酶基因的证据。Mol Genet基因组学。2013; 288(10):483-93。
- 27.
Francki Mg,Hayton S,Gummer JPA,Rawlinson C,Trengove Rd。小麦非洲素线的代谢组分分析和基因组分析,鉴定粒子中的生物化学途径的基因。植物Biotechnol J. 2015; 1-12。
- 28。
Mackay IJ,Bansept-Basler P,Barber T,Bentley Ar,Cockram J,Gosman N等人。冬季播种小麦的八元多价高级发电人口:创作,属性和验证。基因基因类型。2014; 4(9):1603-10。
- 29。
Rasheed A,Xia X,Ogbonnaya F,Mahmood T,Zhang Z,Mujeeb-Kazi A等。基因组 - 使用数字成像分析的合成六倍体麦子中的晶粒形态结合。BMC植物BIOL。2014; 14:128。
- 30.
Webster H,Keeble G,Dell B,FoSu-Nyarko J,Mukai Y,Moolhuijzen P等人。小麦细胞壁转化酶基因的基因组级鉴定耐旱性研究。Funct植物Biol。2012; 39:569-79。
- 31.
黄志强,黄志强,王志强,等HKT.在小麦、大麦和水稻中,Na的关键决定因素+运输和耐盐性。J Exp Bot。2008; 59(4):927-37。
- 32.
DeleónJLD,Escoppinichi R,Geraldo N,Castellanos T,Mujeeb-Kazi A,RöderMs。与矿物成长面包小麦盐度耐受相关的定量特质基因座。Euphytica。2011; 181(3):371-83。
- 33.
Genc Y, Oldach K, Verbyla AP, Lott G, Hassan M, Tester M,等。盐胁迫下面包小麦苗期生长的QTL分析。Theor Appl Genet. 2010; 121:877-94。
- 34.
Ma L,周E,Huo,周R,王G,佳J.小麦重组近铬群体耐盐性遗传分析(Triticum aestivum.l .)。Euphytica。2007;153(1 - 2):109 - 17所示。
- 35.
等。第11届国际小麦遗传研讨会,小麦耐盐性定量性状位点的定位。2008.
- 36。
Quarrie SA, Quarrie PS, Radosevic R, Rancic D, Kaminska A, Barnes JD, et al。分析小麦产量的QTL,从QTL到候选基因。中国生物医学工程学报。2006;57(11):2627-37。
- 37。
关键词:含盐量,面包小麦,群体,农艺和生理性状,数量性状摩尔品种。2013;32(1):39-59。
- 38。
王志强,王志强,王志强,等。草科植物基因组学研究进展。安机器人。2002;89(1):3 - 10。
- 39。
Apples R,Francki M,Chibbar R.谷物功能基因组学的进步。Funct综合基因组学。2003; 3(1-2):1-24。
- 40.
Feuillet C,Keller B.高基因密度在小型和大草基因组的同步基因座上保守。Proc Natl Acad Sci U S A. 1999; 96(14):8265-70。
- 41.
玉米和高粱同源adh区共线性及其例外中国科学(d辑:地球科学)1999;96(13):7409-14。
- 42.
Chen Wh,De Meaux J,Lercher MJ。邻近基因的共同表达拟南芥:从直接相互作用中分离染色质效应。BMC基因组学。2010;11:178。
- 43.
Rustenholz C,Choulet F,Laugier C,Safar J,Simkova H,Dolezel J等人。3,000型染色体转录图3b的六倍体小麦基因岛的结构和功能特征无意识。植物理性。2011; 157(4):1596-608。
- 44.
重复DNA序列的不稳定性:复制在多种机制中的作用。中国科学院院刊2001;98(15):8319-25。
- 45.
李YC,Korol AB,Fahima T,Beiles A,Nevo E.微卫星:基因组分布,推定功能和突变机制:审查。分子Ecol。2002; 11(12):2453-65。
- 46.
Cotsaftis O, Plett D, Shirley N, Tester M, Hrmova M. Na两阶段模型+3D建模解释的大米中的排除HKT.转运体和可变剪接。公共科学图书馆ne。2012; 7 (7), e39865。
- 47.
Espley RV,Brendolise C,Chagne D,Kutty-Amma S,Green S,Volz R等人。启动子区段的多重重复导致红苹果中的转录因子自动调节。植物细胞。2009; 21(1):168-83。
- 48.
Gowik U,Burscheidt J,Akyildiz M,Schlue U,Koczor M,Streubel M等。CIS.- C中叶肉特异基因表达调控元件4植物Flaveria TrinerviaC的启动子4磷酸丙酸胆碱酸酶基因。植物细胞。2004; 16(5):1077-90。
- 49.
Thurau T,Kifle S,Jung C,CAI D.线虫抗性基因HS1PRO-1的启动子激活糖甜菜中的线虫响应和喂养位点特异性基因表达(Beta寻常魅力l .)和拟南芥.植物学报。2003;52(3):643-60。
- 50。
Villar CBR,Erilova A,Makarevich G,TröschR,KöhlerC.控制PHERES1拟南芥的直接串联重复印迹研究。摩尔。2009;2(4):654 - 60。
- 51。
Baek D,江j,chung js,王b,陈俊,xin z等。监管AtHKT1基因通过远端增强子表达和启动子中的DNA甲基化在耐盐性中起重要作用。植物生理学杂志。2011;52(1):149-61。
- 52。
Munns R,James Ra,Xu B,Athman A,Conn SJ,Jordans C等人。祖先Na改善了盐渍土壤上的小麦籽粒产量+运输基因。NAT BIOTECHNOL。2012; 30(4):360-4。
- 53。
Diatloff E, Kumar R, Schachtman DP。位点定向诱变减少Na+的亲和力HKT1,一个土著+通电高亲和力k+运输车辆。2月。1998;432(1):31-6。
- 54.
Kato Y, Sakaguchi M, Mori Y, Saito K, Nakamura T, Bakker EP, et al.支持四跨膜-孔-跨膜拓扑模型的证据拟南芥Na+/ K+把AtHKT1蛋白质,k的超家族成员+运输车。Proc Natl Acad Sci U S A. 2001; 98(11):6488-93。
- 55.
Maser P,Eckelmana Bv,Vaidyanathan R,Horiea T,Fairbairnb Dj,Kuboc M等。改变射击/根NA+拟南芥Na基因突变对盐敏感性的影响+转运体AtHKT1.费用。2002; 531:157-61。
- 56.
卢比奥F,施瓦茨M,加斯曼W,施罗德JI高亲和力K基因突变的遗传选择+转运体HKT1为简化的Na定义循环站点的函数+渗透性和含量增加+宽容。中国生物化学杂志。1999;274(11):6839-47。
- 57.
曹颖,金旭,黄洪,等。钾离子转运体TrkH的晶体结构。大自然。2011;471(7338):336 - 40。
- 58.
引用本文Kato N, Akai M, Zulkifli L, Matsuda N, Kato Y, Goshima S, et al.带正电荷氨基酸在m2d跨膜螺旋中的作用Ktr/载重汽车/HKT.型阳离子转运仪。渠道。2014; 1(3):161-71。
- 59.
Sassi A,Mieulet D,Khan I,Moreau B,Gaillard I,Sentenac H,等。稻米单价阳离子转运蛋白OsHKT2;4:重新探讨离子的选择性。植物杂志。2012;160(1):498 - 510。
- 60。
任志华,高建平,李乐国,蔡新林,黄伟,超大勇,等。一个水稻耐盐数量性状位点编码一个钠转运体。自然麝猫。2005;37(10):1141 - 6。
- 61。
Berthomieu P,Conéjérog,努格·努,Brackenbury WJ,Lambert C,Savio C等人。功能分析AtHKT1在拟南芥中表明na+韧皮部的再循环对耐盐性至关重要。EMBO j . 2003; 22(9): 2004 - 14所示。
- 62.
卢比奥F,加斯曼W,施罗德植物钾转运体钠驱动钾吸收HKT1以及具有耐盐性的突变。科学。1995;270(5242):1660 - 3。
- 63.
Ashburner M,Ball Ca,Blake Ja,Botstein D,Butler H,Cherry JM等。基因本体:生物学统一的工具。NAT Genet。2000; 25(1):25-9。
- 64.
Hunter S, Apweiler R, atwood TK, Bairoch A, Bateman A, Binns D,等。InterPro:整合蛋白特征数据库。核酸Res. 2009;37增补1:D211-5。
- 65.
Kearse M,Moir R,Wilson A,Stones-Havas S,Cheung M,Sturrock S等人。佐芙基本:用于组织的集成和可扩展的桌面软件平台和序列数据分析。生物信息学。2012; 28(12):1647-9。
- 66.
黄新强,Brûlé-Babel A.异源多倍体植物同源基因的基因组特异性引物的开发:四倍六倍体小麦蜡质和淀粉合酶II基因(Triticum aestivum.L.)作为示例。BMC RES笔记。2010; 3(1):140。
- 67.
Francki M,Crasta O,Sharma H,Sharma H,欧姆H,安德森J. Alien的结构组织Thinopyrum美国软红冬小麦(Triticum aestivum.l .)。40基因组。1997;(5):716 - 22所示。
- 68.
Von Heijne G.膜蛋白结构预测:疏水性分析和正面规则。J Mol Biol。1992年; 225(2):487-94。
- 69.
MPEx:一种探索膜蛋白的工具。蛋白质科学。2009;18(12):2624 - 8。
- 70.
Kelley La,Sternberg MJ。网页上的蛋白质结构预测:使用PHYRE服务器的案例研究。NAT PROTOC。2009; 4(3):363-71。
- 71.
EMBOSS:欧洲分子生物学开放软件套件。趋势麝猫。2000;16(6):276 - 7。
- 72.
Wicker T, Matthews DE, Keller B. TREP:一个数据库麦芽汁重复的元素。植物科学进展,2002;7(12):561-2。
- 73。
陈J,胡问,张Y,陆C,旷h . P-MITE:转位因子对植物微型inverted-repeat数据库。42(数据库版):D1176-81。
- 74。
Lescot M,DéhaisP,Thijs G,Marchal K,Moreau Y,Van de Peer Y等。Plantcare,植物顺式动作调节元件和促进剂序列中硅分析的工具的门户网站。核酸RES。2002; 30(1):325-7。
- 75。
Higo K, Ugawa Y, Iwamoto M, Korenaga T. PlantCIS.- 治疗调节DNA元素(地方)数据库:1999。核酸RES。1999年; 27(1):297-300。
- 76。
史密特根TD。利用实时荧光定量PCR技术分析相关基因表达数据-ΔΔct方法。方法。2001; 25:402-8。
- 77。
eickelberg gj,fisher aj。植物基因表达的环境调节:高级本科生物化学或分子生物学过程的RT-QPCR实验室项目。Biochem mol Biol教育。2013; 41(5):325-33。
- 78.
Schmittgen Ts,Livak JK。用比较CT法分析实时PCR数据。NAT PROTOC。2008; 3(6):1101-8。
- 79.
Francki Mg,Walker E,Crawford Ac,Broughton S,欧姆HW,Barclay I等。六倍体小麦遗传和细胞遗传学图的比较(Triticum aestivum.使用SSR和DArT标记。中国生物医学工程学报。2009;32(2):241 - 241。
- 80.
徐志强,陈志强,陈志强,等。基于微卫星的小麦遗传-物理图关系构建系统(Triticum aestivum.l .)。Funct综合基因组学。2004; 4(1):12-25。
- 81.
Manly KF, Cudmore RH, Meer JM。地图管理器QTX,跨平台的遗传作图软件。Mamm基因组。2014;12(12):930 - 2。
- 82。
Voorrips R. MapChart:图形化表达连锁图和qtl的软件。J在这里。2002;93(1):77 - 8。
确认
HACKA由国际研究费用奖学金(SIRF)和大学国际助学金(UIS)资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
黑客设计并进行了实验,生成和分析了数据,解释结果并写了稿件。KHO提供了遗传地图和映射人口的表型数据,有助于解释数据并编辑稿件。MGF有助于设计所有实验,数据解释和编辑稿件。所有作者均阅读,修订并批准了最终的手稿。
附加文件
附加文件1:图S1。
Tahkt2的氨基酸序列比对; 1和Tahkt2; 2个蛋白质。构成阳离子选择性滤波器域的甘氨酸分子由红线表示。(PPTX 758 KB)
附加文件2:图S2。
DNA序列对准OsHKT2;1-OsHKT2;4代际区域(a)和Tahkt2.;1-Tahkt2.;2代际地区7,7BL.和7dl.(B) PIF/Harbinger型螨的完整序列以紫色突出,CACTA型转座子“DTC_Isidor”的残基以蓝色突出,串联复制的单个基序以红色、黄色、绿色和深蓝色突出。盒状序列代表盐激活CIS.-中作用的调控要素和其他调控母序OsHKT2;4和Tahkt2.;2推动者区域。(RTF 545 KB)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Ariyarathna,H.A.c.k.,Oldach,K.H.&Francki,M.G.与水稻鉴定立体族II的比较基因分析HKT.基因及其与Na的关系+浓缩在面包小麦中。BMC植物BIOL.16,21(2016)。https://doi.org/10.1186/s12870-016-0714-7
收到了:
接受:
发表:
关键词
- 第II族HKT.
- Iwgs.
- 水稻基因组
- Na+排除