抽象的
背景
乙醛酸酶途径由两种酶,乙氧酶I(Glyi)和乙醛酸酶II(Glyii)组成,其将高毒毒性代谢物甲基甘油(Mg)释放到其无毒形式中。Mg可形成具有各种细胞宏观分子的先进糖化末端产物,例如蛋白质,DNA和RNA;最终导致他们的失活。甘氧酶酶的作用在各种植物物种中被广泛研究,其在盐度,干旱和重金属胁迫耐受性中表现出其至关重要的作用。以前在模型植物中进行了对乙醛酸酶基因的全基因组分析拟南芥和米饭,但没有这样的研究是在任何豆类物种中进行的。
结果
在本研究中,进行了大豆的综合基因组数据库分析,并分别鉴定了24和12基因编码的推定的41甘氨酸和23种Glyii蛋白。对这些鉴定的成员进行了详细分析,包括它们的命名和分类,染色体分布和重复,外显子系统组织和蛋白质结构域和术语鉴定。这些基因的表达分析已经在不同的组织和发育阶段以及使用公共rnaseq和微阵列数据的盐度和干旱胁迫下进行。这项研究显示Gmglyi.7和GmGLYII-8已在所有发育阶段和组织中集中表达;尽管Gmglyi.-6,Gmglyi.-9,Gmglyi.-20年,Gmglyii-5和Gmglyii-10是高度非生物的应激响应构件。
结论
本研究鉴定了大豆中最大的乙二醛酶蛋白家族,共有41个gmglyyi成员和23个GmGLYII成员。详细的分析Gmglyi.和GmGLYII基因强烈表明乙醛酶成员全基因组的节段和串联复制。此外,本研究为揭示GmGLYI和GmGLYII成员在大豆生长发育和逆境生理中的生物学作用和功能提供了有力的依据。
背景
乙醛酸酶系统是一种双酶驱动途径,将高氧毒性化合物,甲基乙二醛(Mg)释放到D-乳酸盐中。解毒通过两种依赖性酶的顺序作用来实现;乙醛酸酶(Glyi)和乙醛酸酶II(Glyii)。在降低的谷胱甘肽(GSH)的存在下,Mg被自发地转化为半缩醛(HTA),并且Glyi将该HTA的异构化催化到S-D-乳酰基 - 谷胱甘肽(SLG)中。Glyii将SLG水解成D-乳酸,并回收返回系统的一个分子[1].两者,Mg的形成和乙醛酸酶在所有生物中都存在于来自的所有生物中大肠杆菌来智人[2].
除了在Mg的解毒中提出的作用之外,还据报道乙醛酸酶参与各种其他功能。乙醛酸酶系统通过抵抗MG的增加增加,保护人类免受糖尿病的各种血管并发症,例如肾病,视网膜病变,神经病变和心血管疾病[3.].此外,还显示乙醛酸酶途径涉及人类的不同重要的细胞功能,例如细胞分裂和增殖,微管组装和对氧醛毒性的保护[4.].为此,途径被认为是“细胞生长和分裂的标志物”。类似地,通过许多研究,植物中已经报道了乙醛酸酶的应力耐受性潜力[5.].发现转基因植物过度表达的glyi和/或glyii,对包括盐度,干旱和重金属毒性的多种非生物应力提供显着耐受性[5.那6.].因此,MG和乙二醛酶被认为是植物耐受性的潜在生物标志物[7.].
乙醛酸酶蛋白已被广泛的特征在于不同的属,例如大肠杆菌那智人那酿酒酵母那拟南芥蒂利亚纳和奥雅萨苜蓿[2].与其他生物相比,很少有关于植物糖醛酶的人们所知的。第一种植物甘氧酶活性来自Douglas Fir针,Smits和Johnson [5.].此后,已从各种其他植物物种(如米)中报道乙醛酸酶活性的存在。拟南芥,番茄,小麦,甘蔗,芸苔属植物等等。 [7.].在植物进化过程中,由于基因的扩增和复制,植物的大部分基因以家族的形式存在[8.].全基因组序列的可用性为植物乙二醛酶家族的鉴定和表征开辟了广阔的领域。根据在网上基因组广泛分析拟南芥,有11个潜力GLYI和三glyii.大米的基因;和十一GLYI和五个glyii.基因拟南芥[1].所有这些基因的表达分析已经在不同的发育组织和阶段进行,并且响应于使用公共MPS和微阵列数据库的多重非生物应力。已经观察到这一点阿格利-3那Osglyi-11,Atglyii-2那Atglyii-5那Osglyii-2和Osglyii-3显示所有组织和阶段的组成型表达,而Atglyi-8那Osglyi-3, 和Osglyi-10.仅在种子中表达[1].另一方面,阿格利-7那Osglyi-11.那Atglyii-2和Osglyii-3是最紧张的诱导成员[1].
在这些鉴定的乙醛酸酶成员中,glyii.基因已被广泛研究两种水稻和拟南芥但是关于GLYI仍然非常有限。迄今为止,所有五个阿古莉和三osglyii.基因表征得很好。两个都Osglyii-2和Osglyii-3具有典型的Glyii酶活性和这些基因的过表达在烟草中提供了增强的盐度应力的耐受性[9.那10].然而,osglii -1和atglii -5通过具有硫双加氧酶(SDO)活性而不是GLYII显示出功能上的差异[11].大米Glyi,Osglyi-11.2的一个已被广泛研究,发现具有Ni2+- 具有应力调制潜力的依赖性Glyi活动[12].
大豆(甘氨酸最大[L.] Merr。)是papilionoideae家族的豆科植物[13],植物蛋白质和食用油的主要来源。它还具有通过symbioss来修复大气氮的能力[14].然而,由于干旱,盐度和渗透胁迫等不利的环境刺激,大豆的生产受到威胁受到威胁[15那16].这些胁迫严重影响了大豆从萌发到开花各个阶段的植株发育,降低了大豆的产量和种子质量。据报道,由于干旱,产量减少了约40% [15].因此,迫切需要使用可用的基因组数据库识别新型应力响应大豆基因[14].大豆基因组含有46,430个预测的蛋白质编码基因,比70%更高拟南芥.大约在5900万年前和1300万年前,大豆经历了两次基因组复制事件,导致基因组高度复制(超过75%的基因被复制)[14].大豆中已经研究了许多基因家族,例如ERF,HD-ZIP,WRKY,Burp,Mads-Box,MyB,NAC,CYP [13那17-22].
对乙二醛酶基因家族进行了全基因组分析拟南芥和米[1[尽管在公开可用的数据库中携带了少数基因组序列,但在大豆中没有进行这种分析。在这里,我们介绍了大豆的详细基因组识别GLYI和glyii.基因及其系统发育关系,染色体分布,结构和表达分析。结果表明,大豆基因组中含有41个glyyi蛋白和23个GLYII蛋白,这是目前已知的生物中最大的乙二醛酶家族。基于公开的微阵列数据对这些基因的表达分析表明,乙二醛酶成员在响应各种发育线索和应激处理时的差异调节。特别是Gmglyi.-6,Gmglyi.-9和GmGLYII-5是最上调的应力响应构件,其可能通过与其他成员互动来抵抗压力的累积。该研究将促进进一步调查大豆乙醛酸酶基因的生物和分子官能。
结果
的识别GLYI和glyii.大豆的基因家族
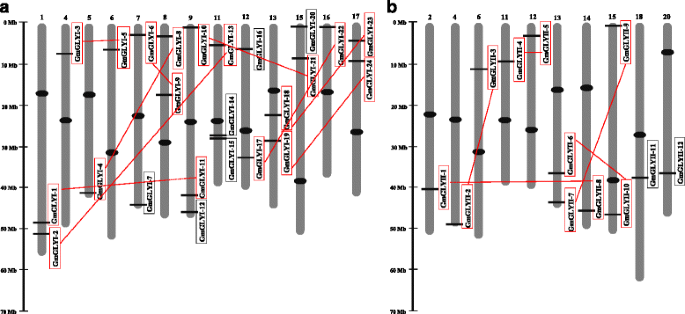
具有LactoylGlutaThione脱氨酶结构域(PF00903)的蛋白质已被分类为Glyi蛋白和金属β-内酰胺酶结构域(PF00753)已被分类为Glyii蛋白[1].此前,乙二醛酶蛋白已经在两个模式植物基因组中被确认,拟南芥和米[1].为了鉴定大豆中所有可能的乙二醛酶蛋白,对大豆基因组数据库进行BLASTP搜索G. Max.wm82.a2.v1(http://phytozome.jgi.doe.gov/pz/portal.html#!earch?show=blast& method=org_gmax.使用以前表征的蛋白质序列作为查询进行。已经使用先前报道的大豆Glyi蛋白(Genbank:NM_00124923.1)主要鉴定了Glyi蛋白。随后,在大豆基因组数据库的BLASTP搜索中,每个新鉴定的Glyi蛋白序列被单独用作查询序列。重复后续搜索过程,直到没有记录新会员。此搜索导致识别共43个独特蛋白质。使用PFAM分析所有这些鉴定的蛋白质,以检查是否存在独特的赤霉素谷胱甘肽裂解酶域(PF00903)。由于缺乏赤霉素凝胶酶域域,该分析丢弃了两个成员,并且最终降落于总共41种大豆蛋白,其大于先前报道的拟南芥(22)和水稻(19)Glyi蛋白。这些41甘氨酸蛋白已被位于13种不同染色体上的24个独特基因进行编码(图。1).他们被确定并命名为Gmglyi.-1到Gmglyi.-24之后以前提出的命名法[1)(表1).
染色体分布Gmglyi.(一种),GmGLYII(B.)基因在不同的大豆染色体。只有具有乙二醛酶基因的染色体被显示出来,它们的数量在上面用罗马数字表示。尺度以兆基(Mb)为单位,中心点区域用黑色椭圆表示。根据序列相似性和差异性分析,红色框表示由红线连接的重复基因片段(表)3.).黑匣子表示未重复的基因
类似地,已经主要用先前表征鉴定了大豆Glyii蛋白Brassica Juncea.Glyii蛋白(Genbank:AaO26580.1)作为查询,其次由新认识的成员。已经鉴定了总共26个独特的蛋白质序列,并使用PFAM存在于存在独特的金属β-内酰胺酶结构域(PF00753)。其中三个没有这个独特的域,并从列表中丢弃。因此,已经确认了总共23种大豆蛋白,其大于先前报道的拟南芥(9)和大米(4)Glyii家庭成员。这些23甘酮蛋白已被位于十种不同染色体上的12个独特基因进行编码(图。1).他们被命名为GmGLYII-1到GmGLYII-12像Gmglyi.基因(表格2).同时Gmglyi.和GmGLYII家族中,蛋白质的数量大于基因的数量(表)1和2);表明大豆乙二醛酶基因存在交替剪接事件。大部分的Gmglyi.基因(24个中的17个)和GmGLYII基因(12个基因中的5个)只有一个产物。然而,其他七Gmglyi.基因形成24个替代拼接产品,而七GmGLYII基因导致了18个蛋白的产生(表1和2).
详细分析识别出的GmGLYI和GmGLYII成员
对所有新识别的GmGLYI和GmGLYII成员进行了详细分析。编码DNA序列(CDS)的长度Gmglyi.成员因333 BP而异(gmglyi - 12.1)至1101 bp(gmglyi-4.1),平均为740 bp。因此,gmglyi-4.1编码家庭最大的蛋白质,多肽长度为366Aa和40.6kDa的分子量;最小的蛋白质(Gmglyi-12.1)是110 AA的长度,重量为12.8kDa(表1).与长度和分子量变异类似,蛋白质在4.86(Gmglyi-9.1)至9.69(Gmglyi-16.3)中显示了它们的等电点(PI)值的广泛偏差。大多数Gmglyi成员均显示酸性PI值(小于或约7),只有七种,例如Gmglyi-1.3,Gmglyi-4.1,Gmglyi-10.5,Gmglyi-11.2,Gmglyi-15.1,Gmglyi-16.1和Gmglyi-16.3表现出基本的pi值(表1).这确保了在某种生理条件下积极和带负电的Gmglyi蛋白的存在。基于两种不同的工具Cello分析所有这些预测的Gmglyi蛋白(41)的亚细胞定位[23]和狼psort [24]并通过氯进一步证实叶绿体定位[25].发现不同的成员在不同的亚细胞室内定位,例如叶绿体,细胞溶质,线粒体,细胞核,细胞外,过氧化物。发现大多数Gmglyi蛋白在胞浆中局部化,其次是叶绿体,线粒体和细胞核(表1).
同样,CDS长度GmGLYII转录物从432 bp变化(gmglyii - 2.4)至1584年的BP(Gmglyii-12.1.)平均为850 bp(表2).最大的Gmglyii-12.1蛋白为527AA的长度,分子量为58.8kDa;最小的蛋白质(Gmglyii-2.4)为143AA的长度,重量为15.9kDa(表2).Gmglyii蛋白还显示出5.62(Gmglyii-1.1)至9.03(Gmglyii-10.1)的PI值的变化。大多数Gmglyii成员(23分)显示出与Gmglyi蛋白相似的酸性PI值,而只有八个Gmglyii成员,例如Gmglyii-2.1,Gmglyii-2.4,Gmglyii-6.1,Gmglyii-7.1,Gmglyii-7.2,Gmglyii-9.1,gmglyii-9.2和Gmglyii-10.1具有基本的PI值(表2).与GmGLYI相似,大多数GmGLYII蛋白定位于胞质,其次是叶绿体(4)、细胞核(3)和线粒体(2)。
染色体分布和基因复制
确定被识别者的确切位置和分布Gmglyi.和GmGLYII构建了不同染色体上的基因,形成了一种详细的染色体图。发现大豆甘油酶基因在整个染色体中不均匀地分布。已经发现24岁Gmglyi.基因位于13种不同的染色体上(图。1).每条染色体的基因密度高度不均匀,其中染色体9和11含有最大的发生GLYI(3)基因。然而,染色体1、7、8、12、13、15、18有两个GLYI基因每个,只有一个GLYI每个基因各自存在于染色体4,5,6和16上。没有GLYI在染色体2,3,10,14,18,19和20上发现基因;此后未在图1中示出。1.同样,12GmGLYII基因被发现位于10个不同的染色体上(图。1 b)每种染色体的基因密度高度不均匀。染色体13和15包含最大值glyii.基因(2),而染色体2,4,6,11,12,14,18和20只有一个glyii.每个基因。不glyii.在染色体的其余部分上发现基因,并且如图2所示。1 b.所有的Gmglyi.和GmGLYII发现基因位于染色体末端(图。1),建议在基因组重复期间不同大豆染色体之间的染色体间遗传重排的可能性。
由于两次复制事件,大豆基因组在一个基因家族中产生了许多同源基因[14].在24个Gmglyi蛋白中(仅在不同交替剪接形式的情况下仅第一成员),20对(10对)聚集(10对),并且八种Gmglyii蛋白成对聚集(4对),其中总共12种Gmglyii蛋白系统发育树(附加文件1:图S1)。所有这些Gmglyi之间的相似性百分比(附加文件2:表S1)和GMGLYII(附加文件2:表S2)蛋白分别合并。观察到Glyi和Glyii系列的所有配对成员(Gmglyi-1 / -11,Gmglyi-4 / -8,Gmglyi-10 / -21,Gmglyi-3 / -5,Gmglyi-14 / -15,Gmglyi-18 / -23,Gmglyi-2 / -13,Gmglyi-17 / -22,Gmglyi-19 / -24和Gmglyi-6 / -9; Gmglyii-4 / -5,Gmglyii-9 / -11,Gmglyii-2 / -3,Gmglyii-6 / -10)具有非常高的级别(超过90%)的序列相似之处。这种高水平的序列相似度表明,在整个进化中,基因分段重复的可能性。此外,在24中Gmglyi.基因一个基因对(Gmglyi.-14和Gmglyi.-15)在染色体11的距离小于5kb(恰好)的距离中连续(没有任何基因)存在于染色体11的距离内。这表明这两个基因可能通过串联复制复制(图。1).为了确定基因重复的时间过程,利用植物基因组重复数据库(http://chibba.agtec.uga.edu/duplication/index/downloads.)[26)(表3.).根据非同义替换与同义替换的比值(Ka/Ks),可以衡量选择作用于不同基因的进化史[17那27].这个比率可以用来解释自然选择强加于各种蛋白质编码基因的方向和程度。一对Ka/Ks < 1的序列意味着提纯选择;Ka/Ks = 1表明两个层序均为中性漂移;最后Ka/Ks > 1表示正向或达尔文选择[17那28].15甘油酶重复基因对的Ka / ks(表3.)被发现小于0.55;表示纯化选择在这些基因对的演变中的影响。考虑到大豆每年同义遗址的6.161029个同义突变的分歧率[29[计算每个基因对的复制时间。观察到所有分段重复的对都显示了3.7和18.8 mya之间的时间帧,但串联重复对发生在33.9 mya以前(表3.).
不同植物乙二醛酶基因的系统发育分析
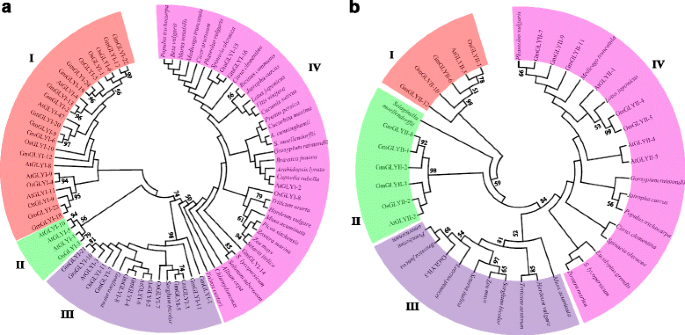
在本研究中,使用Mega 5.2工具构建所有鉴定的Gmglyi或Gmglyii蛋白的系统和来自其他植物物种的其他报道的Glyi或Glyii蛋白的系统发育树(图。2).使用总共83个全长Glyi蛋白序列产生的大豆,水稻和拟南芥GLYI家族,以及来自其他植物物种的蛋白质。树被细分为4个亚科(I到IV),如图所示。2.所有这些Subfamilies都包括来自两个Dicot的代表成员拟南芥和单码米,表明植物的演变GLYI基因发生在分裂在Dicot-Monocot之前。CLADE-IV具有来自不同植物物种的最大的Glyi成员,而Clade-II只有来自的最低数量拟南芥和水稻基因组(图。2).CLADE-I仅包括三种植物的完整基因组数据库的Glyi成员,拟南芥大米和大豆。其中,OsGLYI-10在功能上是水稻GLYI家族的一个分裂成员,可能具有除GLYI以外的其他一些活动(未发表数据)。在演化支iii中,有多个来自拟南芥,大米和大豆;和一个成员每人来自Genlisea Aurea和高粱二色的.其中,三个稻米成员Osglyi-2,Osglyi-7和Osglyi-11;和两个成员拟南芥AtGLYI-3和AtGLYI-6已经被预测为Ni2+-依赖GLYI酶[2].因此,这一思想家的余地将有望拥有ni2+端依赖催化活性。类似地,进化枝iv有来自水稻的成员(osgli -8)和拟南芥(atgly -2)为Zn2+- 依赖性glyi酶[2].因此,来自其他物种的剩余Glyi成员需要Zn2+最适GLYI活性。这表明锌2+- 依赖性Glyi酶更多样化,因为它们存在于许多植物物种中(图。2).
为了阐明Glyii蛋白之间的系统发育关系,我们进一步构建了来自其他植物物种的所有全长序列,Osglyii,Atglyii家族和Glyii序列的所有全长序列构建了另一棵树(图。2 b).这棵树也像之前的树一样被细分为四类(I到IV)。i类蛋白质由三种大豆蛋白质和一种水稻蛋白质组成(OsGLYII-1)拟南芥(AtGLYII-3)。其中,有报道称OsGLYII-1具有硫双加氧酶(SDO)活性,而不是GLYII [11].因此,这种蛋白质的蛋白质可以从Glyii功能多样化。类似地,II类含有米饭(Osglyii-2)的一种蛋白质,拟南芥(AtGLYII-2),Selaginella Moellendorffii.和来自大豆的四种蛋白质。Atglyii-2已被据报道是线粒体局部Atglyii家庭成员[30.].第三级和-IV的划分更有趣,进化得更重要。III类具有来自所有单子植物的Glyii蛋白(米饭,Zea Mays.那Pennisetum.那芸苔属植物那小麦属植物那部落);虽然第四类专门的Dicot成员包括拟南芥、大豆、Medicago.莲花等(图。2 b).除glyyi外,单子叶和双子叶分裂后GLYII蛋白呈现多样化。
基因结构Gmglyi.和GmGLYII基因
详细分析了外显子的内含子结构Gmglyi.(无花果。3),GmGLYII(无花果。3 b)基因在它们之间表现出很大的变化。全部Gmglyi.和GmGLYII基因在其开放阅读框架(ORF)中含有至少一个内含子,这意味着大豆中没有内含子甘氨酸酶基因。在不同的ORF中,内含子的数量从1到9变化Gmglyi.基因(图。3和附加文件3.:表S3)。这Gmglyi.-6.3,Gmglyi.-12,Gmglyi.-17年,Gmglyi.-22在其ORF中包含一个内含子,而在其ORF中发现的内含子数量最多(9)Gmglyi.-4.2转录物。在许多情况下,蛋白质编码序列,5'和3'未翻转区域(UTR)的边界也包含大量内含子[13那31].四分为14.Gmglyi.转录物中,这些基因中的任何3'UTR中没有内含子,其中八个在其5'UTR区域中包含一个内含子。类似地,内含子的数量在不同的不同之处在于1到12之间变化GmGLYII基因(图。3 b和附加文件3.:表S4)。观察到内含子(12)的最大数量GmGLYII-12.1,后面各11GmGLYII-12.2和GmGLYII-12.3。GmGLYII-11.1只包含一个在其ORF中的单个内含子,而其余的内含子数量变化。相似Gmglyi.成绩单,3'UTR中没有内含子GmGLYII转录物。23名转录物中只有六个(GmGLYII-2.2,GmGLYII-2.4,GmGLYII-4.2,GmGLYII-6.1,GmGLYII-7.1和GmGLYII-12.1)在5'UTR区域中有一个内含子。
较长的内含子具有选择性优势,可以平衡突变偏置并提高重组频率[32].通过分析动物,植物和真菌的内含子报道了祖先内含子存在的强大证据[33].此外,在副同源基因中发现外显子和内含子的数量相似(图。3.)聚集在系统发育分析中(附加文件1:图S1)。例如,Gmglyi.-1/-11,Gmglyi.-4 / -8,Gmglyi.-10/-21,Gmglyi.-6 / -9具有相同数量的内含子和外显子。
用于其域架构,催化保护和金属离子依赖性Gmglyi蛋白的分析
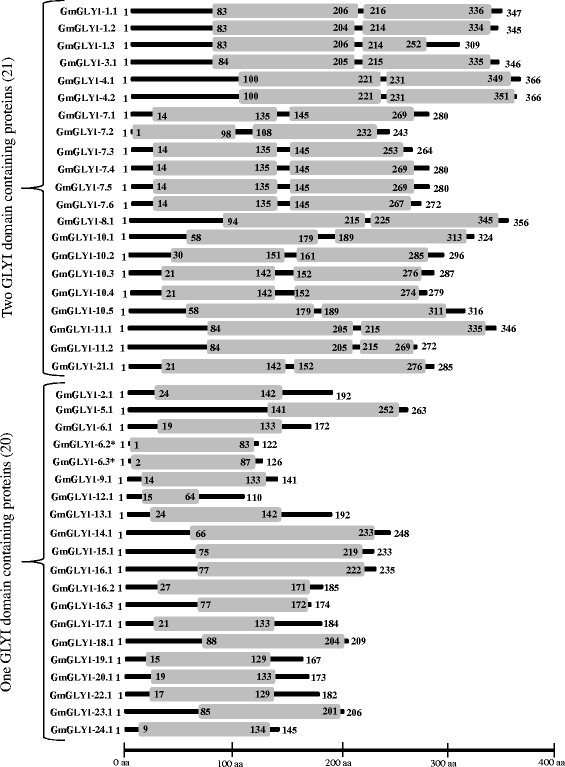
使用Pfam对所有预测的GmGLYI(41)蛋白进行分析,发现其中存在保守的乙二醛酶结构域(PF00903)。对GmGLYI蛋白的分析显示,41个蛋白中有21个含有2个GLYI结构域,而其余20个只有一个GLYI结构域(图)。4.).在一个单一的蛋白中存在两个GLYI结构域的报道以前来自酿酒酵母[34],奥雅萨苜蓿[12] 和疟原虫疟原虫[35].两个结构域的存在在单个单体蛋白质上形成两个推定的活性位点。发现两个有源网站都有功能性,而是体齐地调节疟原虫疟原虫[35,而其中一个活性位点在奥雅萨苜蓿[12].然而,单结构域GLYI蛋白在不同物种中也有报道,如大肠杆菌[36],H. Sapiens.[37]并用作同源二聚体。
Gmglyi蛋白的域体系结构。使用PFAM分析所有四十一种大豆蛋白的功能域(S)(http://pfam.xfam.org/).所有的GmGLYI蛋白都具有用方框表示的乙二醛酶结构域(PF00903)。结构域的位置由方框内的氨基酸数字表示。在41个GmGLYI成员中,20个具有单一的乙二醛酶结构域,其余21个具有两个结构域。完整蛋白质的长度由精确的氨基酸数表示,结构域的相对位置可以用下面给出的比例来解释
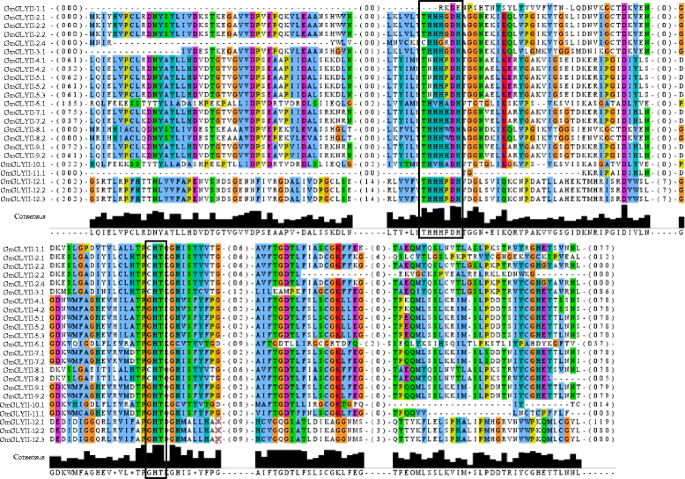
Glyi酶的活性高度依赖于二价金属离子[2].在金属离子特异性的基础上,Glyi蛋白可分为两类;Zn.2+- 依赖或Zn.2+- 独立(主要是NI2+/ co.2+端依赖)。GLYI从智人那酿酒酵母和假单胞菌putida已被报告为Zn2+- 依赖[38-40],而Glyi来自大肠杆菌其中一个水稻GLYI (OsGLYI-11.2)显示Ni2+- 依赖活动[12那36].Glyi酶的金属依赖性可以从Glyi结构域的长度容易地预测,如ni2+-依赖的GLYI的结构域长度为~120 aa和Zn2+- 长度为142 AA的依赖性glyis [2].无论金属离子依赖性如何,Glyi蛋白的活性位点都具有H / QEH / QE的保守基序。其中,谷氨酸残基通过从基材接受质子和这种保守残留物的任何突变而充分丧失活性丧失[12那41].因此,对酶活性和金属离子依赖性的存在进行评论,对准所有推定的Gmglyi蛋白的Glyi结构域(仅在两个结构域内的N-末端)对准(图。5.)与已知的倪2+- 依赖Osglyi-11.2和Zn2+- 依赖Osglyi-8 [2)的蛋白质。所有的金属结合位点都出现在黑盒子和锌的特异区域内2+-依赖的GLYI用黑色箭头表示(图。5.).
基于所有四个保守金属结合位点的存在,预测了推定的Gmglyi蛋白的预期Glyi酶活性(表4.).在总共41个推定的Gmglyi蛋白中,20个具有所有四个保守的残留物,并且预期具有功能性Glyi酶活性(图。5.和表格4.).其中20个预期的功能性Glyi酶,16次预测为Ni2+- 依赖于它们的域长度约为120 AA,并且缺乏特定于Zn的保守区域2+端依赖成员。其余四个GmGLYI-14.1、GmGLYI-15.1、GmGLYI-16.1和GmGLYI-16.2预计为Zn2+- 依赖于其域长度超过145AA并具有保守区域(图。5.和表格4.).
GmGLYII蛋白的结构域和催化效率分析
大豆的基因组揭示了由12个基因编码的23种Glyii蛋白的存在(表2).利用Pfam对所有GmGLYII蛋白进行分析,发现其中存在保守的金属- β -内酰胺酶结构域(PF00753)。对所有GmGLYII蛋白的分析显示,23个蛋白中有12个蛋白只有金属- β -内酰胺酶结构域,其余11个蛋白除了金属- β -内酰胺酶结构域外,还含有额外的羟基酰基谷胱甘肽水解酶c端(HAGH-C)结构域(PF16123)(图)。6.).HAGH-C结构域通常存在于GLYII酶的c端,与催化结构域一起形成底物结合位点(PF00753) [42].然而,来自各种物种的Glyii,如大肠杆菌,S.Cerevisiae,S.Typhimurium,L.Imantum,A. Thaliana,B. Juncea,O. Sativa和智人,含有保守的金属粘合基序(THXHXDH)和活性位点图案(C / GHT)[9.].这两种基序在蛋白质的Glyii酶活性中起重要作用。因此,对推定的Gmglyii蛋白的酶活性的存在进行评论,它们的蛋白质序列通过多序列比对对齐(图。7.).这两个图案都是由黑盒子表示的。7.);他们的存在和缺席列于表中5..在23个推测的GmGLYII蛋白中,只有3个不具有保守的金属结合残基,但所有的蛋白都具有活性位点motif(图2)。7.和表格5.).因此,可以预期所有预测的Gmglyii蛋白质具有除Gmglyii-1.1,Gmglyii-2.4和Gmglyii-11.1(表)之外的功能性Glyii酶活性(表5.).
代表Gmglyi和Gmglyii成员的同源造型
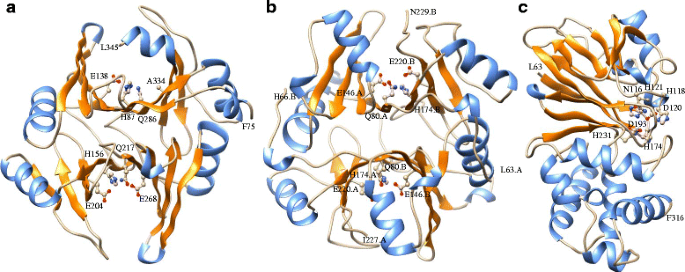
为了解活性位点残基的排列和整体三维配位,构建了GmGLYI-3、GmGLYI-16和gmglyi -5蛋白的同源模型(图3)。8.),基于紧密相关的模板结构Zea Mays.Glyi(PDB:5D7Z)[43],小鼠Glyi(PDB:40PN)和ATGLYII-2(PDB:2Q42)[30.]蛋白质分别。gmglyi-3是ni2+- 依赖单体甘氨酰胺(图。8A),而Gmglyi-16是Zn2+- 依赖同源二聚体酶(图。8B.).Gmglyi-3有两个推定的活跃场所;一个由H-156,E-204,Q-217和E-268组成,另一个由H-87,E-138,Q-286和A-334组成。第二推定的活性位点缺乏高度保守的Glu残基,因此在先前报告的OSGlyi-11.2中可能是无活性的[12].Zn.2+- 依赖Gmglyi-16由单个Glyi结构域组成(图。4.因此,形成同源二聚体以产生两个推定的活性位点(图。8B.).一个假定的活性位点有Q80和E146(来自一个链,A)和H174和E220(来自另一个链,B);另一个是两个链上的相反成员。在这里,两个活性位点都有所有四个保守的残基,因此预测也具有功能活性。另一方面,GmGLYII-5是一个由两个结构方向组成的单体蛋白,一个n端结构域(L63 - D193)具有两个βββαβ拓扑结构,一个c端结构域(T194 - F316)具有五个α-螺旋结构(图4)。8C).金属结合和活性位点残留物是Asn116,His118,Asp120,His121,His174,Asp193和His231(图。8C与模板Atglyii-2蛋白相比,发现被认为是保守的。
表达分析Gmglyi.和GmGLYII不同大豆组织的基因
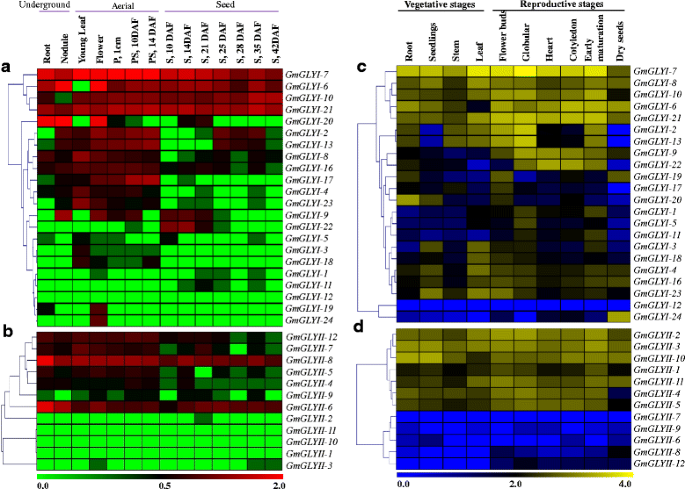
RNA-SEQ图谱甘氨酸最大提供了14种大豆组织的高分辨率基因表达数据,如嫩叶、花、1厘米豆荚(开花后7天,DAF)、豆荚壳(10和14 DAF)、种子(10、14、21、25、28和35 DAF)、根和根瘤。所有这些组织大致可分为三类;如地下、空中和种子。所有RNA-seq规范化表达数据Gmglyi.和GmGLYII从大豆(http://www.soybase.org/soyseq//),除了Gmglyi-14和Gmglyi-15由于缺乏适当的探针(附加文件2:表S5)。分析数据并表示为使用TIGR MEV软件包生成的热图(图。9A和B.).表达分析Gmglyi.基因显示不同成员具有不同的组织特异性表达。在所有22个分析的基因中,Gmglyi.-7在所有组织中显示出最高水平的组成表达,然后是Gmglyi.-21年,Gmglyi.-10和Gmglyi.-6。这种高水平的组成型表达表明它们在所有这些植物组织中的重要作用(图。9A).仅群体在所有地下和空中组织中显示出高水平的表达,然后在种子组织中得到非常低的表达。他们是Gmglyi.-2,Gmglyi.-13,Gmglyi.-17年,Gmglyi.8日,Gmglyi.4和Gmglyi.-16。以前关于稻米的研究拟南芥显示出高度种子特异性GLYI基因如阿古利8日,Osglyi.3和Osglyi.-10 [12].同样,三个Gmglyi.基因如Gmglyi.-1,Gmglyi.-11和Gmglyi.-22仅在不同的种子组织中有中等水平的表达。9A),表明种子特异性存在的进化保守GLYI基因。
大豆乙二醛酶基因在不同发育组织和阶段的分级聚类表达谱分析一种那B.14种发育组织的RNA-SEQ表达数据,如R(根),N(结节),YL(幼牙),F(花),P.1cm(一CM POD),PS.10D(吊舱壳10DAF)),PS.14D(Pod Shell 14DAF),S.10D(种子10dDAF),S.14D(种子14 DAF),S.21D(种子21DAF),S.25D(种子25DAF),S.28D(种子28DAF)),S.35D(种子35DAF),S.42D(种子42DAF)用于分析。归一化数据从大豆(http://soybase.org/soyseq/)并作为附加文件提供2:表S5。使用MEV软件包执行Heatmap生成和分层群集。热图下方的颜色刻度表示表达值;绿色表示低成绩丰富,而红色表示高水平的转录物丰富。C那D.所有的转录组数据Gmglyi.和GmGLYII各种发育阶段的基因(在每个车道的顶部表示)是从国家生物技术信息中心获得的(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163).使用MEV软件包执行HeatMaps生成和分层群集。在热图下面给出的颜色刻度表示蓝色表示低转录物丰度和黄色表示高记录性丰富的表达值
表达分析GmGLYII基因表示两个透明的碎片(图。9B.).在12个被分析的基因中,有5个基因是GmGLYII-1,GmGLYII-2,GmGLYII-3,GmGLYII-10和GmGLYII-11在所有组织中显示出几乎不可检测的表达,少数例外。其中GmGLYII-8在所有组织中显示出最高水平的组成表达,其次是GmGLYII-6.这两个基因可能在所有组织中发挥重要作用。相似Gmglyi.,一群基因(GmGLYII-4,GmGLYII-5,GmGLYII-7,和GmGLYII-12)除种子外,在地下和空中组织中显示出中等表达水平(图。9B.).在情况下观察到没有组织特异性表达模式GmGLYII基因。
从识别的副吉隆对的表达数据分析Gmglyi.和GmGLYII14种大豆组织中的基因显示出高水平的表达分歧。例如,Gmglyi.-6显示高水平的组成型表达,而其副骨Gmglyi.-9在少量组织中显示出可检测的表达。但是,一些副寄生Gmglyi.基因对Gmglyi.-1/-11,Gmglyi.-2/-13,Gmglyi.-4 / -8,Gmglyi.-10/-21,Gmglyi.-19 / -24显示出类似的表达模式。在案例的情况下,发散甚至更差异GmGLYII基因对。例如,GmGLYII-8在所有分析的组织中高度表达,而其副骨对应物GmGLYII-1仍然是不可检测的。如果是,也观察到类似水平的偏差GmGLYII-6 / -10andGmGLYII-7 / -9基因对。
表达分析Gmglyi.和GmGLYII不同发展阶段的基因
的表达Gmglyi.和GmGLYII使用公开可用的基因组的转录数据分析大豆的分析数据分析了不同发育阶段的基因(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163).数据集主要包含两个广泛的发展集,一个在营养阶段(根,幼苗,茎,叶子)和另一个在繁殖阶段(花芽,种子发育的不同阶段 - 球状,心脏,子叶,早期成熟,干燥).如图1所示。9C.,大部分Gmglyi.基因显示出高水平的表达,没有任何明显的表达模式。其中24名Gmglyi.基因,只Gmglyi.-12在所有阶段显示出未检测的表达。其中Gmglyi.-7显示所有发育阶段的最大组成型表达,然后是Gmglyi.-21年,Gmglyi.-6,Gmglyi.-10和Gmglyi.8(无花果。9C.).的两个Gmglyi.成员,Gmglyi.-22和Gmglyi.-9仅表现为生殖期特异性表达。这表明了特定调制的发展Gmglyi.基因表达。
另一方面,观察到一个不同的分裂GmGLYII发育阶段的基因表达(图。9D.).一组基因,如GmGLYII-1,GmGLYII-2,GmGLYII-3,GmGLYII-6,GmGLYII-10,和GmGLYII-11在营养和生殖阶段均表现为阴性或极低水平的表达。然而,其余的GmGLYII成员在所有发展阶段中显示出媒介到高水平的表达(图。9D.).两者的高度表达Gmglyi.和GmGLYII大豆的所有发育阶段的基因表明乙醛酸酶途径在整个植物生命周期中的组成型代谢/细胞作用。
胁迫下大豆乙醛酸酶基因的表达分析
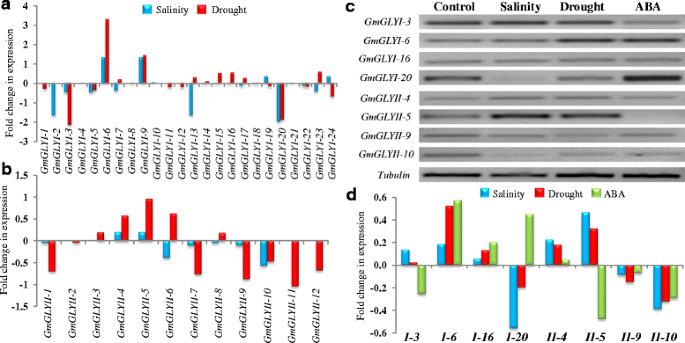
为深入了解乙二醛酶基因在大豆非生物胁迫适应中的功能,对其表达谱进行了分析Gmglyi.和GmGLYII利用公开的微阵列数据分析了响应盐度和干旱胁迫的基因。分别从GSE41125和GSE40627的盐胁迫和干旱胁迫基因表达综合数据库中获得表达数据集。由于各自探针的限制,无法获得所有这些基因的数据。其中有24个Gmglyi.12.GmGLYII基因;数据为19.Gmglyi.和八个GmGLYII分析基因的盐度,而表达数据为21Gmglyi.12.GmGLYII发现了干旱胁迫的基因。不同的乙二醛酶成员对这两种应力的表达有不同的反应(图。10和B.).响应盐度压力,四个Gmglyi.基因和二GmGLYII基因显示出上调,而七Gmglyi.基因和四GmGLYII基因显示下调,其余部分保持不变(图。10和B.).同样,干旱压力导致七个上调Gmglyi.基因和五GmGLYII基因,以及八个基因的下调Gmglyi.基因和六个GmGLYII基因(图。10和B.).在24中Gmglyi.基因;Gmglyi.-6和Gmglyi.-9表达显著上调,而其他2种表达(Gmglyi.3和Gmglyi.-10)在两种应力下均表现为下调(图。10).的情况下GmGLYII;GmGLYII4和GmGLYII-5显示上调,同时GmGLYII-10在盐度和干旱胁迫下显示出显着的下调(图。10 b).两者的其余部分Gmglyi.和GmGLYII家族表现出不同的表达模式。这表明不同的乙二醛酶成员在大豆胁迫调节途径中发挥着不同的作用。
大豆甘氧酶基因响应盐度,干旱和激素治疗的表达分析。所有可用的相对表达数据Gmglyi.(一种),GmGLYII(B.)盐度和干旱胁迫下的基因是从国家生物技术信息地理数据库(http://www.ncbi.nlm.nih.gov/geo/).通过与相应的模拟样本进行比较,表达数据作为折叠改变。蓝色键表示盐度应力的数据,而红颜色表明干旱胁迫。C半定量RT-PCRGmglyi.基因(Gmglyi.-3,Gmglyi.-6,Gmglyi.-16和Gmglyi.-20),四GmGLYII基因(Gmglyi.-4,Gmglyi.-5,Gmglyi-9.和Gmglyi-10)和一个保留的控制基因,管蛋白在不同的条件下,如对照、盐度、干旱和ABA处理。D.通过使用图像J软件测量PCR带强度来分析代表性八种乙醛酸酶基因的相对表达分析,并表示为表达式的相对折叠变化
为筛选大豆乙二醛酶基因在盐、干旱和激素(ABA)处理中的作用,采用半定量RT-PCR方法对4个候选基因进行验证Gmglyi.基因(Gmglyi-3,−6.- -16和 -20.)和四名候选人GmGLYII基因(GmGLYII-4,−5.- -9.和 -10)在微阵列数据分析中非常响应(图。10和B.).为此目的,将15天大豆幼苗进行正常水(作为对照),200mM NaCl(盐度)或取出水(用于干旱)或10mM ABA(用于激素治疗)8小时。将所有候选基因的表达与管蛋白(作为一个管家控制基因)(图。10 c).通过使用图像J软件扫描凝胶图像来计算表达式的凝胶图像来测量所有这些转录物的相对转录性丰度。考虑到表达式的表达相对折叠变化管蛋白作为内部控制(图。10 d).可以从图中清楚地推断它。10 d那GmGLYI -6,Gmglyii-4和Gmglyii-5表现出强大的上调,以应对盐度和干旱,而且GmGLYI -20.Gmglyii-9和Gmglyii-10显示清晰的下调(图。10 d).剩下的两个成员,GmGLYI -3和GmGLYI -16与对照样品相比略有上升/下降。总的来说,这8个候选基因的表达模式(图。10 c)与微阵列数据几乎相似(图。10和B.).
大豆甘氧酶基因启动子区中的顺式元素的鉴定
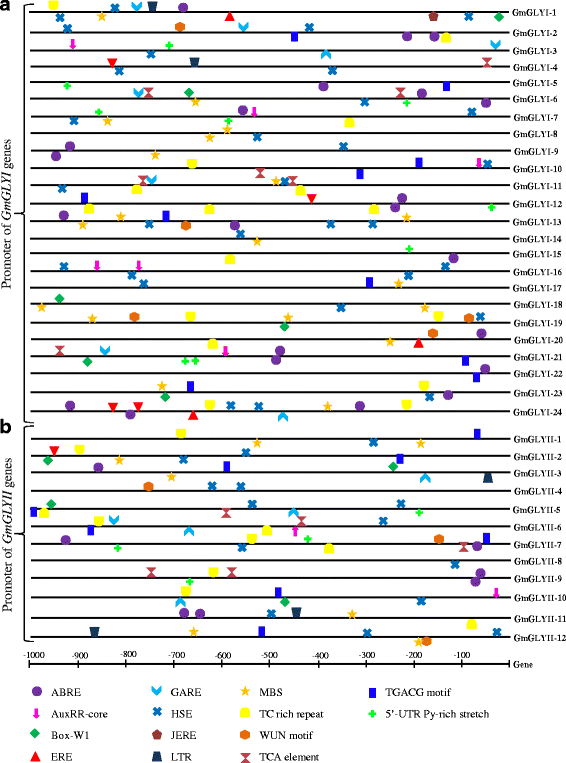
为了评价应激反应的表达gmg基因对盐度、干旱和ABA处理的响应,每个上游1 kb启动子区域Gmglyi.和GmGLYII从大豆(http://www.soybase.org/dlpages/flank/index.php.),并使用PlantCARE分析顺式作用元素的存在[44].通过分析,鉴定了几种胁迫响应的顺式元件,如脱落酸响应元件(ABRE)、生长素响应元件(AuxRR-core)、真菌诱导子响应元件(BOX-W1)、乙烯响应元件(ERE)、赤霉素响应元件(GARE)、热激元件(HSE)、茉莉酸和诱导子响应元件(JERE)、低温响应元件(LTR)、myb结合位点(MBS)、防御和胁迫响应元件(TC-rich)、伤害和病原体响应元件(W-box和wn -motif)、水杨酸响应元件(TCA)、茉莉酸甲酯响应元件(CGTCA box和TGACG motif)、具有高转录水平的元素(5 ' UTR Py-rich stretch)。所有这些基序在植物胁迫调节途径中都是非常关键的,因此在调控各种胁迫响应基因的表达中发挥着重要作用[45那46].所有这些基序均随机分布在启动子序列的正链和负链中(图1)。11).之中Gmglyi.成员,Gmglyi-1和Gmglyi-24具有最大CIS元素(12个元素),而Gmglyi-14启动子在其上至少有两个元素。的情况下GmGLYII成员,GmGLYII-7最多有10个元素,而GmGLYII-8只有一个元素。ABRE、HSE和TGACG基序几乎存在于所有的启动子中gmg少数例外的基因。尽管需要通过实验证实CIS作用调节元件和观察到的转录物丰度之间的相关性,但这些结果表明了应力响应性质gmg基因。
讨论
甲基乙二醛(MG)是所有活细胞自然产生的代谢副产物[7.].但MG的水平会随着植物中各种非生物胁迫的反应而上升[9.].已有文献证实,乙二醛酶途径在MG的解毒中起着重要作用,并提供对多种非生物胁迫的耐受性[5.那7.].单码口种模型植物稻和Dicot模型厂进行了基因组初步分析乙醛酸酶途径拟南芥[1].然而,这个家庭尚未在任何其他物种中进行过研究,包括豆类。在本研究中,我们已经进行了对大豆的基因组分析,以鉴定乙醛酸酶基因家族,包括它们的染色体位置,基因和蛋白质结构,保守的活性位点和催化位点和表达谱。共24个GLYI12.glyii.大豆基因组分别编码41个GLYI和24个GLYII蛋白(见表)1和2).的数量Gmglyi.基因比它的2.2倍拟南芥和米饭(每个11个基因);和GmGLYII显示比大量更多的2.4倍拟南芥(五阿古莉基因),比米饭丰富4倍(三个osglyii.基因)。基因数量显著增加的可能原因可能是大豆的两次复制事件[14[单子叶/双子叶分裂后发生的,或大多数大豆基因以一种特定物种的方式扩展[17].
为了采用不同的不良环境条件,植物倾向于重复基因以产生新的成员或增加数量[17那47].基因复制有三种基本模式,即串联复制、节段复制和转位。在本分析中,共观察到10对重复对Gmglyi.家庭和五分内GmGLYII家庭(表3.).除了一个外,它们都显示出节段性重复,这是植物中基因重复的主要模式。Tandem复制对Gmglyi-14 / -15形成为33.9 mya前;虽然分段重复Gmglyi.3.7 ~ 13.6 Mya之间的基因和GmGLYII发生在6.3和18.8mya之间。这表明在分段复制事件之前发生了串联复制事件。以前针对大豆的HD-ZIP基因报道了类似的重复模式[17].
大豆,包括其他植物被发现拥有更多的GLYI和GLYII基因和蛋白质与动物相比,迄今为止。其中一个可能的原因是植物在进化过程中的基因复制,最终导致基因功能的分化[48].功能分化可能导致亚功能化或新功能化,进而导致新的基因功能[48].在本研究中,在41个预测的Glyi蛋白中,其中20只具有所有四个保守的金属结合位点,并且预期具有功能性Glyi酶活性(图。5.和表格4.).其他蛋白质可能在功能上发散并拥有类似于Glyi的其他活动。结构Glyi是邻氧螯合物(VOC)的成员,包括外氧基酶,Glyi和甲基甘氨酰-CoA映异构酶[49].其中较早预测的GmGLYI (Accession no.;X68819)也被发现具有谷胱甘肽s -转移酶活性[50].除此之外,文献中已经明确指出,GLYIs有两种金属活化类;Zn.2+和非Zn2+(主要是NI2+/ co.2+).这两种课程都具有相同的四个保守金属结合残留物和八面体金属协调;无论金属激活等级如何51].其中一个预测的NI的三维结构2+- 依赖Glyi,Gmglyi-3(图。8A)和Zn.2+-依赖的GLYI, GmGLYI-168B.)确认两者都有相同的活性位点残留物。但是,NI.2+-依赖的GmGLYI-3被发现是由两个GLYI结构域组成的单体,可以折叠形成两个假定的活性位点(图。8A).而含锌的单GLYI结构域2+- 依赖Gmglyi-16需要是创建两个推定的有源网站的同源度(图。8B.).有趣的是,可以基于蛋白质的氨基酸长度和序列容易地预测推定甘氨酸的金属特异性[2那51].Zn.2+- 活化的Glyis在氨基酸长度比NI相对较大2+/ co.2+- 激活的并且序列中具有独特的区域(图。5.).根据这些标准,在20个预测的功能性GmGLYI酶中有16个是Ni2+/ co.2+-活性。NI的同样的统治模式2+/ co.2+在水稻GLYIs中观察到-激活的形式(4个预期有活性的OsGLYIs中的3个)拟南芥glyis(三个预期功能Atglyis中的两个)[2].
相反,Glyii酶含有β-内酰胺酶折叠结构,包括乳酸酶,溴肟:氧氧化还原酶,Glyii,芳基硫酸酶,磷酸二酯酶,羧酸酯酶和TRNA maturase [52].此前,GLYII家族成员来自拟南芥(Atglx2-5)和稻米(Osglyii-1)缺乏Glyii活性,而是它们具有硫醛酶蛋白1(Ethe1)活性的硫酸二氧化酶[11].类似于Glyi,23个预测的Gmglyii蛋白中的三个没有保守的金属结合基质,其可能导致没有Glyii活性并导致功能性分歧。
乙二醛酶基因的表达已被发现对某些组织或发育阶段具有高度特异性拟南芥和米[1].因此,表示模式Gmglyi.和Gmglyi.在不同的发育阶段和组织中分析基因(图。9.).这些数据揭示了大豆中乙醛酸酶基因的组织特异性表达模式。所有之中,Gmglyi.-7,Gmglyi.-21和GmGLYII发现-8是组成型表达大豆甘氧酶系统的成员。一簇Gmglyi.和GmGLYII基因在所有地下和空中组织中保持高水平的表达,然后在种子发育的不同阶段进行低水平的表达(图。9.).这表明在不同组织/发育刺激中的多个成员之间存在功能分布。相反,一些Gmglyi.基因如Gmglyi.-1,Gmglyi.-11,和Gmglyi.-22仅在其他部分中显示出种子组织中的中等表达水平(图。9.).这表明发展的具体过渡/调控Gmglyi.基因。的情况下GmGLYII在它们的表达中观察到基因,在其表达中观察到两个可区分的蛹,其中一个组在所有组织中显示出高水平的表达,另一个组织均无表达。这些低富有症状基因可能具有其他细胞/代谢调节预期发育/组织调节。在非生物胁迫下对大豆乙醛酸酶基因观察到另一个有趣的表达模式(图。10).不同的成员Gmglyi.和GmGLYII家庭差异地反对盐度,干旱和激素(ABA)处理(图。10 c).GmGLYII-9和GmGLYII-10响应所有三种条件而显示出强大的下调GmGLYII-16显示急剧上升(图。10 d).调用推动促进剂序列的各种顺式作用调节元件的存在Gmglyi.和GmGLYII基因可能是这种改变表达后面的可能原因(图。11).以前在水稻中观察到类似的表达模式拟南芥乙二醛酶基因(1[其中每个成员显示针对特定类型的应力处理的特定表达模式。总体而言,本研究中观察到的信息将有助于找出适当的候选基因,用于进一步函数表征和提高应力转基因作物植物。
结论
综上所述,我们进行了全面的在网上大豆乙醛酸酶基因家族分析(Gmglyi.和GmGLYII),并提供有关他们的详细资料。具体来说,我们的结果表明,大豆基因组包含24Gmglyi.12.GmGLYII分别为41种Gmglyi和二十三种Gmglyii蛋白的基因分别代码;最大鉴定的乙醛酸酶基因家族迄今为止迄今为止。目前的研究表明乙醛酸酶基因的基因组重复(两种末端和串联),其导致家族的膨胀。基于保守的主题和序列同源性的存在,我们已经深入了解其推定功能和金属依赖。最后,尽管具有大型多成员家族,表达数据证实了每种基因的开发,组织和应激特异性响应。
方法
的识别Gmglyi.和GmGLYII大豆基因
通过BLASTP搜索对新大豆基因组数据库(WM82.A2.v1)鉴定大豆基因组中的推定的Glyi和Glyii蛋白(http://www.soybase.org/)[53]使用先前报道的大豆Glyi蛋白序列(Genbank:NM_001249223.1)的E值为1。Brassica Juncea.GLYII蛋白序列(GenBank: AAO26580.1)分别查询。随后,每个确定的序列都用作次要查询,以查找其他新成员。所有蛋白序列分别用Pfam (http://pfam.xfam.org/)默认参数和E值1,用于在GlyiI蛋白中存在甘氨酸酶和金属β-内酰胺酶结构域(Metallo-β-内酰胺酶结构域(PF00753)的存在。所有鉴定的推定乙醛酸酶蛋白质是前缀“GM”的命名法甘氨酸最大,随后是根据其染色体位置从1开始串联的Glyi或Glyii和阿拉伯数。通过在“中”之后添加阿拉伯数来表示替代剪接表格。顺序迹象。所有推定的染色体位置Gmglyi.和GmGLYII从大豆酶中鉴定基因(http://soybase.org/gb2/gbrowse/gmax1.01/)[53绘制染色体地图的数据库。使用Prot-Param软件计算所有鉴定的Gmglyi和Gmglyii蛋白的各种物理化学性质(http://web.expasy.org/protparam/).使用Cello V.2.5:子蜂窝定位预测器预测蛋白质的定位(http://cello.life.nctu.edu.tw/)[23]和psort预测软件(http://www.genscript.com/wolf-psort.html.)[24].氯化物进一步证实叶绿体定位(http://www.cbs.dtu.dk/services/ChloroP/)[25].
多序列比对和系统发育分析
为了研究来自各种植物物种的Glyi和Glyii蛋白质中的系统发育关系和保守的基序/金属结合位点,从NCBI下载序列(http://www.ncbi.nlm.nih.gov/),PDB(http://www.rcsb.org/pdb/home/home.do.),水稻基因组数据库(http://rice.plantbiology.msu.edu/),拟南芥基因组数据库(https://www.arabidopsis.org/)和大豆数据库(http://www.soybase.org/).用于研究系统发育分析的蛋白质序列在附加文件中可用4.和5..使用ClustalW进行多序列比对[54]使用Mega 5.2构建系统发育树5.2 [55]带有邻居加入方法和1000个引导程序复制。
基因重复和KA / KS计算
使用植物基因组复制数据库分析基因重复(http://chibba.agtec.uga.edu/duplication/index/downloads.)[26]对于大豆。具有超过90%序列相似性的基因被认为是分段重复,而串联重复基因在100kb区域中的五个或更少基因分离。从植物基因组重复数据库中检索代名词(KS)和非同义替代(KA)率或从Pal2nal程序计算(http://www.bork.embl.de/pal2nal/)[56].以6.1X10的速率计算每个基因对的分化时间(以百万年为单位)-9每年每地点的替代量[17].因此,发散时间(t)= ks /(2x6.1x10-9)x10-6mya。
Glyi和Glyii蛋白的领域建筑,催化保护和金属离子特异性的评估
使用PFAM分析所有预测的Gmglyi(41)和Gmglyii(24)蛋白质,露出保守乙醛酸酶结构域(PF00903)和金属β-内酰胺酶结构域(PF00753)的存在。Gmglyii蛋白的Gmglyi和金属β-内酰胺酶结构域(Metallo-β-内酰胺酶结构域(PF00753)与先前表征的成员分开对准,并用CLUSTAL进行对准并分析保守基序的存在。Glyi具有保守的H / QEH / QE主题用于金属粘合和催化,而Glyii具有两个单独的金属粘合基序(THXHXDH)和活性位点图案(C / GHT)。基于先前的研究预测了Gmglyi蛋白的金属离子特异性[2那51].
各种大豆甘油酶蛋白的基于同源性结构建模
基于GMGLYI-3,GMGLYI-16和GMGLYII-5的基于GMGLYI-3和GMGLYII-5的模型(http://swissmodel.expasy.org/)[57].首先通过模板搜索分析各自的蛋白质序列,然后使用具有最高相似性的最佳模板结构的模型建筑物。Gmglyi-3,Gmglyi-16和Gmglyii-5的结构采用蛋白质数据库(PDB)中获得的大多数类似结构建造。Zea Mays.分别为GLYI (5D7Z)、mouse GLYI (4OPN)和atglyi -2 (2Q42)蛋白。使用UCSF Chimera可视化产生的结构(http://www.cl.ucsf.edu/chimera)[58].基于先前的模板结构分析鉴定并标记活性位点残留物。
使用RNA-SEQ图案的表达分析甘氨酸最大
分析24的组织特异性表达数据Gmglyi.12.GmGLYII基因,它们相应的探针组使用大豆工具识别(http://www.soybase.org/correspondence/index.php).标准化转录数据从soybase下载(http://soybase.org/soyseq/)为14种不同组织,包括根、根瘤(地下组织);叶、花、开花后10天荚果壳(DAF),荚果壳14-DAF, 1厘米荚果(地上组织);种子发育不同阶段(10-DAF、14-DAF、21-DAF、25-DAF、28-DAF、35-DAF和42-DAF种子)。该标准化表达被用于生成热图和使用基因组研究所MeV软件包进行层次聚类[59].
表达分析Gmglyi.和GmGLYII不同发展阶段的基因
表达式模式Gmglyi.和GmGLYII利用公开的转录组数据确定不同发育阶段的基因(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE29163).从NCBI数据库下载了十种不同大豆阶段(根,幼苗,茎,叶,花蕾和种子发育,心脏,子叶,早期成熟和干燥种子的不同阶段)http://www.ncbi.nlm.nih.gov/),登录号为SRX062325至SRX062334。归一化后,利用基因组研究所MeV软件包生成热图[59].
表达分析Gmglyi.和GmGLYII响应盐和干旱胁迫的基因
从国家生物技术信息基因表达综合(Geo)数据库中,检索甘氨酸酶基因响应盐度和干旱胁迫的表达数据[60]分别加入号码GSE41125和GSE40627。相应的探针集Gmglyi.和GmGLYII使用Netaffx分析中心鉴定基因(http://www.affymetrix.com/analysis/infex.affx?navmode=cat530006&aid=netaffxnav.)在线探针匹配工具。具有相同探针集的多个基因被认为是相同的转录谱,而具有多个探针集的基因被认为是最高的转录谱。表达式数据使用mock数据进行规范化,并用条形图表示。
植物材料,应力处理和半定量RT-PCR
大豆(大豆L。在连续30℃温度和12h / 12h的光周期的情况下生长多种苏格兰)幼苗[17].用正常水作为实验控制或200mM NaCl溶液灌溉十五天幼苗,用于盐度应力或10mM ABA溶液,用于荷尔蒙治疗8小时。将幼苗置于滤纸上并暴露于空气中以模拟干旱胁迫。8小时后,从所有这些幼苗收集叶子(用三份酸盐),并使用Trizol®试剂(Thermo Fisher Scientific,USA)提取总RNA。使用Revertaid First Strand cDNA合成试剂盒(Thermo Fisher Scientific,USA)合成第一链cDNA。八个候选基因的基因特异性底漆,在附加档案中列出3.表S6,采用Primer-Blast (http://www.ncbi.nlm.nih.gov/tools/primer-blast/)和大豆管蛋白基因被用作内部对照[22].
推定顺式调节元素的启动子序列分析
以确定启动子序列中各种顺式作用的调控元件Gmglyi.和GmGLYII从大豆基因组数据库中检索基因,1kb 5'上游区域序列(http://www.soybase.org/dlpages/flank/index.php.).使用PlantCare数据库分析启动子序列[44]找出CIS作用调节元素的存在。
可用性数据和材料
关于大豆的所有序列信息可在公共数据库,大豆(http://soybase.org/).除此之外,支持本文得出结论的大多数数据集都包含在附加文件中。系统发育分析中使用的所有蛋白质序列已经沉积在Uniprot(http://www.uniprot.org/),并提供额外的数据。大豆种子(大豆L。孟加拉国农业研究院,孟加拉国邦加尔省的品种索马克。
缩写
- AA:
-
氨基酸
- 阿巴:
-
脱盐酸
- BP:
-
碱基对
- DAF):
-
天开花
- Glyi:
-
乙醛酸酶I.
- glyii:
-
乙二醛酶二世
- GSH:
-
减少谷胱甘肽
- H:
-
小时
- MG:
-
甲基甘油
- mya:
-
万年
参考文献
- 1.
Mustafiz A, Singh AK, Pareek A, Sopory SK, Singla-Pareek SL.对水稻和拟南芥的全基因组分析确定了两个在非生物胁迫中高度表达的乙二醛酶基因。功能整合基因组学。2011;11(2):293-305。
- 2.
kaur c,vishnoi a,ariyadasa tu,bhattacharya a,singla-parek sl,sopory sk。水平基因转移和基因融合的剧集导致不同金属离子特异性乙醛酸酶I. SCI批准的共存。2013; 3:3076。
- 3.
拉巴尼N,索纳利PJ。糖醛酸酶在糖尿病、肥胖及相关疾病中的作用细胞发育生物学杂志。2011;22(3):309-17。
- 4.
Thornalley PJ。乙二醛酶系统:生物生命基础代谢途径功能特性的新发展。j . 1990, 269(1): 1 - 11。
- 5.
kaur c,ghosh a,pareek a,sopory sk,singla-parek sl。植物中的乙醛酸和胁迫耐受性。Biochem Soc Trans。2014;42(2)。DOI:10.1042 / BST20130242.
- 6.
singla-pareek sl,reddy mk,sopory sk。烟草中乙醛酸酶途径的遗传工程引发增强盐度耐受性。Proc Natl Acad Sci U S A. 2003; 100(25):14672-7。
- 7.
乙二醛酶和甲基乙二醛作为植物抗逆性的生物标志物。植物科学进展。2014;33(6):429-56。
- 8。
郭玉玲。绿色植物基因家族进化及其起源与进化拟南芥蒂利亚纳基因。工厂J. 2013; 73(6):941-51。
- 9。
Ghosh A,Pareek A,Sopory Sk,Singla-Pareek SL。一种谷胱甘肽响应水稻乙醛酸酯II,OSGLYII-2,通过维持更好的光合作用效率和抗氧化池来适应盐度适应。工厂J. 2014; 80(1):93-105。
- 10.
Singla-Pareek S,Yadav S,Pareek A,Reddy M,SOHORY S.通过过表达乙醛酸酶II的过表达增强耐药耐药性。转基因Res。2008; 17(2):171-80。
- 11.
水稻非生物胁迫诱导的ethe1样蛋白在根中表达量较高,并受钙的调控。杂志。2014;152(1):1 - 16。
- 12.
Mustafiz A,Ghosh A,Tripathi Ak,Kaur C,Gangquency Ak,Bhavesh NS,Tripathi JK,Pareek A,Sopory Sk,Singla-Parek SL。一种独特的Ni依赖性和甲基甘油诱导的水稻乙醛酸酶I具有单一的活性位点和非生物应激反应的功能。工厂J. 2014; 78:951-63。
- 13.
MainaliHR,Chapman P,Dhaubhadel S.基因组 - 西方细胞苷基因家族分析(甘氨酸最大).BMC Plant Biol. 2014;14:282。
- 14.
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W,Hyten DL,Song Q,Thelen JJ,Cheng J,等。古代聚丙烯大豆的基因组序列。自然。2010; 463(7278):178-83。
- 15.
Manavalan LP, Guttikonda SK, Tran LS, Nguyen HT。提高大豆抗旱性的生理和分子途径。植物生理学报。2009;50(7):1260-76。
- 16.
单例PW,Bohlool BB。盐度对大豆结节形成的影响。植物理性。1984; 74(1):72-6。
- 17.
陈X,陈Z,赵立,赵y,程b,Xiang Y.大豆HD-ZIP基因家族的基因组分析及盐度和干旱处理下的表达剖析。Plos一个。2014; 9(2),E87156。
- 18。
杜华,杨世生,梁铮,冯博良,刘玲,黄玉波,唐应祥。大豆MYB转录因子超家族的全基因组分析。BMC Plant Biol. 2012;12:106。
- 19。
风扇厘米,王x,王yw,胡rb,张xm,陈国,富yf。大豆疯子基因的基因组表达分析显示种子发育中的潜在功能。Plos一个。2013; 8(4),E62288。
- 20.
徐H,李益,严y,王克,高y,胡玉。含大豆突发域基因的基因组鉴定及其在应激处理下的表达。BMC植物BIOL。2010; 10:197。
- 21.
尹g,xu h,xiao s,qin y,li y,yan y,胡y。大豆(甘氨酸最大)Wrky TF家族扩大了分段重复事件,随后在亚组之间进行了不同选择。BMC植物BIOL。2013; 13:148。
- 22。
张克,陈米,陈X,徐Z,关诗,李立立,李安,郭j,茂l,ma y。大豆ERF基因家族的系统发育,基因结构和表达模式(大豆L。).J Exp Bot。2008; 59(15):4095-107。
- 23。
余春春,陈永昌,卢春春,黄家杰。蛋白质亚细胞定位预测。蛋白质。2006;64(3):643 - 51。
- 24。
Horton P, Park KJ, Obayashi T, Fujita N, Harada H, Adams-Collier CJ, Nakai K. WoLF PSORT:蛋白质定位预测器。核酸Res. 2007;35(Web服务器版):W585-7。
- 25。
基于神经网络的叶绿体转运肽及其裂解位点预测方法。蛋白质科学。1999;8(5):978 - 84。
- 26。
李th,唐嘿,王x,帕特森啊。PGDD:植物中基因和基因组重复数据库。核酸RES。2013; 41(数据库问题):D1152-8。
- 27。
李海,枸杞T,Nei M.Pseudogenes作为中性演化的范式。自然。1981; 292(5820):237-9。
- 28。
牧师N,Hoen Dr,Huynh Ml,Harrison PM,Bureau Te。大米宿主基因片段宿主基因段的进化命运。Genome Res。2005; 15(9):1292-7。
- 29。
林奇M,Conery JS。作用的进化命运和二份基因的后果。科学。2000; 290(5494):1151-5。
- 30.
Marasinghe GP, Sander IM, Bennett B, peryannan G, Yang KW, Makaroff CA, Crowder MW。线粒体乙二醛酶的结构研究II。中国生物化学杂志。2005;280(49):40668-75。
- 31。
关键词:真核生物,mRNA,非翻译区,结构与功能基因。2001;276(2):73 - 81。
- 32。
Carvalho AB, Clark AG。内含子大小与自然选择。大自然。1999;401(6751):344。
- 33。
动物、植物和真菌基因内含子位置的大规模比较。中国科学院院刊2002;99(25):16128-33。
- 34.
Frickel Em,Jemth P,Ladersten M,Windervik B.酵母甘油酶I是具有两个活性位点的单体酶。J Biol Chem。2001; 276(3):1845-9。
- 35.
DEPONTE M,Sturm N,Mittler S,Harner M,Mack H,Becker K.两种不同功能活性位点的单次不同功能活性位点疟原虫疟原虫乙醛酸酶I. J Biol Chem。2007年; 282(39):28419-30。
- 36.
他MM,Clugston SL,Honek JF,Matthews BW。确定结构大肠杆菌乙醛酸酶I表明差异金属活化的结构基础。生物化学。2000; 39(30):8719-27。
- 37.
Cameron Ad,Olin B,Ridderstrom M,Manianvik B,Jones Ta。人乙醛酸酶I的晶体结构 - 基因重复和3D域交换的证据。Embo J. 1997; 16(12):3386-95。
- 38.
Aronsson AC,MARMSTAL E,ManiaMyvik B.甘油酶I,哺乳动物和酵母的锌金属酶。Biochem Biophys Res Communce。1978; 81(4):1235-40。
- 39.
ridderstrom m,industvik b.优化的人锌酶甘氧酸酶I. Biochem J.1996; 314(Pt 2):463-7的优化异诺表达。
- 40。
Saint-Jean AP, Phillips KR, Creighton DJ, Stone MJ。的活性单体和二聚体形式假单胞菌putida乙醛酸酶I:3D域交换的证据。生物化学。1998年; 37(29):10345-53。
- 41.
王志刚,王志刚,王志刚。一种新型的Zn2+配体在人乙二醛酶催化中的应用[J] .中国生物化学学报,1998;33(4):427 - 434。
- 42.
camposs - bermudez VA, Leite NR, Krog R, Costa-Filho AJ, Soncini FC, Oliva G, Vila AJ。生物化学和结构表征Salmonella typhimurium.乙醛酸酶II:新的洞察金属离子选择性。生物化学。2007; 46(39):11069-79。
- 43.
Turra Gl,Agostini RB,Fauguel Cm,Presello Da,Andreo Cs,Gonzalez JM,Campos-Bermudez VA。新型单体乙醛酸酶I来自的结构Zea Mays..Acta Crystallogr D Biol Crystalogr。2015; 71(PT 10):2009-20。
- 44.
Lescot M,Dehais P,Thijs G,Marchal K,Moreau Y,Van de Peer Y,Rouze P,Rombauts S. Plantcare,植物顺式行动监管元素数据库和工具门户网站在网上启动子序列分析。核酸学报2002;30(1):325-7。
- 45.
陈W,荣耀NJ,Glazebrok J,Katagiri F,Chang Hs,Eulgem T,Mauch F,Luan S,Zou G,Whitham Sa等。拟南芥转录因子基因的表达谱矩阵表明其响应环境应激的推定功能。植物细胞。2002; 14(3):559-74。
- 46。
Yamaguchi-Shinozaki K,Shinozaki K.渗透性和冷应力响应性启动子的顺式作用调控元素组织。趋势植物SCI。2005; 10(2):88-94。
- 47。
顾z,steinmetz lm,gu x,scharfe c,戴维斯rw,李海。重复基因在零突变遗传鲁棒性中的作用。自然。2003; 421(6918):63-6。
- 48。
谷欣。蛋白质(家族)序列进化的功能差异。遗传。2003;118(2 - 3):133 - 41。
- 49。
Pakhomova S,Rife Cl,Armstrong RN,Newcomer Me。转座子TN2921中氟霉素抗性蛋白FOSA的结构。蛋白质SCI。2004; 13(5):1260-5。
- 50。
Skipsey M,安德鲁斯CJ,镇杰克,杰森I,Edwards R.来自大豆的甘氨酸酶I的克隆和鉴定。拱生物学生生物糖。2000; 374(2):261-8。
- 51。
Suttisansanee U,Lau K,Lagishetty S,Rao Kn,Swaminathan S,Sauder JM,Burley SK,Honek JF。细菌乙醛酸酶I酶的结构变异:对金属酶乙醛酶I的研究acetobutylicum梭状芽胞杆菌.中国生物医学工程杂志。2011;28(4):497 - 497。
- 52。
Limphong P,Nimako G,Thomas PW,Fast W,Makaroff Ca,粉碎MW。Arabidopsis Thaliana Mitochondrial乙醛酸酶2-1表现出β-内酰胺酶活性。生物化学。2009; 48(36):8491-3。
- 53。
格兰特D,纳尔逊RT,Cannon SB,Shoemaker RC。大豆,USDA-ARS大豆遗传学和基因组学数据库。核酸RES。2010; 38(数据库问题):D843-6。
- 54。
Larkin Ma,Blackshields G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,Valentin F,Wallace Im,Wilm A,Lopez R等人。Clustal W和Clustal X 2.0版。生物信息学。2007; 23(21):2947-8。
- 55。
田ura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5:利用最大似然、进化距离和最大简约法的分子进化遗传学分析。生物医学进展。2011;28(10):2731-9。
- 56。
PAL2NAL:蛋白质序列比对到相应密码子比对的稳健转换。核酸Res. 2006;34(Web服务器版):W609-12。
- 57。
Biasini M,Bienert S,Waterhouse A,Arnold K,Studer G,Schmidt T,Kiefer F,Cassarino TG,Bertoni M,Bordoli L等。瑞士型号:使用进化信息建模蛋白质三级和第四纪结构。核酸RES。2014; 42(Web服务器问题):W252-8。
- 58.
Pettersen EF,戈达德TD,黄CC,沙发GS,GreenBlatt DM,Meng Ec,Ferrin Te。UCSF Chimera - 一种用于探索性研究和分析的可视化系统。j计算化学。2004; 25(13):1605-12。
- 59.
Eisen MB,Spellman Pt,Brown Po,Botstein D.集群分析和基因组表达模式的显示。Proc Natl Acad Sci U S A. 1998; 95(25):14863-8。
- 60。
Barrett T,Edgar R. Gene表达综合症:微阵列数据存储,提交,检索和分析。方法酶。2006; 411:352-69。
致谢
AG承认Shahjalal科学技术大学,孟加拉国Sylhet,为提供实验室空间提供了相同大学的物流支持和生物化学和分子生物学。达卡大学植物学系植物园植物育种和生物技术实验室,提供实验室设施。作者感谢达卡大学博伊特教授M. Mozammel Haque博士,达卡,德哈卡1000,孟加拉国和雅伊豪斯教授,Shahjalal科学技术大学,Sylhet-3114,孟加拉国,孟加拉国复制编辑稿件的宝贵时间。
作者信息
从属关系
通讯作者
附加信息
利益争夺
作者声明他们没有竞争利益。
作者的贡献
AG设计和执行实验,并分析了数据。Ti进行了半定量的RT-PCR实验。AG和TI写了稿件。这两个作者都阅读了稿件并批准了最终版本。
附加文件
附加文件1:图S1。
gmglyyi (A)和GmGLYII (B)蛋白的系统发育关系。利用24个GmGLYI和12个GmGLYII蛋白的全长氨基酸序列(在多个成员的情况下仅取第一个剪接变体),使用MEGA5.2软件采用1000 bootstrap的Neighbor-Joining方法生成了一棵无根树。分支旁边的数字表示1000次引导重复的结果,以百分比表示,高于50%的分数表示在节点上。(PDF 17 kb)
附加文件2:表S1。
从大豆的副骨对吡啶蛋白之间的成对相同。表S2。来自大豆的所有Glyii蛋白质之间的身份百分比。表S5。大豆表达分析GLYI和glyii.通过RNA-SEQ数据(XLS 40 KB)基因
附加文件3:表S3。
所有剪接变异的外显子和内含子的数量Gmglyi.基因。表S4。所有剪接变异的外显子和内含子的数量GmGLYII基因。表S6。在半定量RT-PCR中使用的引物。(docx 26 kb)
附加文件4:
用于GLYI系统发育分析的蛋白序列。(多克斯21 kb)
附加文件5:
用于Glyii的系统发育分析的蛋白质序列。(docx 17 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
大豆乙二醛酶基因家族的全基因组分析和表达谱分析(甘氨酸最大)表明其发育和非生物胁迫特异性反应。BMC植物杂志16日,87(2016)。https://doi.org/10.1186/s12870-016-0773-9
已收到:
公认:
发表:
关键词
- 乙醛酸酶
- 甘氨酸最大
- 非生物压力
- 功能差异
- 基因复制
- 微阵列
- 金属依赖
- RNA seq-Atlas
- 半定量rt-PCR