摘要
背景
斑斑,由Cochliobolus巨大成功是大麦中传播最广、危害最大的病害之一。与抗性相关的遗传位点鉴定c .巨大成功对未来的标记辅助选择具有重要意义。目前研究的目的是鉴定具有对两种不同病型的幼苗抗性的基因座c .巨大成功在西伯利亚春大麦核心收集。
结果
共有96个春季大麦品种和线在幼苗阶段表现出两种c .巨大成功分离株(Kr2和Ch3)。根据Fetch-Steffenson评分量表,Kr2/Ch3分离株的耐药基因型分别为16%/17%和26%/30%。利用50 K Illumina Infinium iSELECT分析了94个基因型。在44,040个snp中,有40,703个snp可以得分,其中39,140个snp具有多态性。27,319个snp通过了过滤阈值,用于关联映射。GLM数据分析显示Kr2和Ch3分离株分别有48和41个SNPs。经5% Bonferroni倍数试验校正后,仅鉴定出3个snp, 27个snp。共有3个基因组区与抗性相关。3H染色体上与ch3抗性相关的区域在SCRI_RS_97417和JHI-Hv50k-2016-158003之间扩增,共包含11个SNPs,其中JHI-Hv50k-2016-157070、JHI-Hv50k-2016-156842最低p- 值。在KR2分离物的情况下,这两个SNP也是显着的。染色体的区域2h包括16个基因座(其中7个,其中7个p-values紧密链接到BOPA2_12_11504)。与该区域相对应的三个位点具有暗示性p- 在KR2测试的情况下,染色体2h的基因座也可能有助于抵抗KR2分离物。第三个区域具有重要意义p-Value在KR2测试的情况下,在Locus Jhi-HV50K-2016-33568的染色体上鉴定出染色体1H。
结论
三个基因组区域与对一个或两个分离株的抗性有关c .巨大成功经西伯利亚春大麦核心种质筛选鉴定。将其与之前在双亲本定位群体研究中发现的qtl或与不同种质和其他分离株的GWAS进行比较,结果表明Kr2和Ch3的抗性位点是由已知的斑点抗性位点决定的。有关SNP相关的信息可以进一步用于开发DNA标记,方便诊断大麦育种计划中的抵抗相关等位基因的诊断。
背景
斑斑,由Cochliobolus巨大成功是大麦中传播最广、危害最大的病害之一。与抗性相关的遗传位点鉴定c .巨大成功这对诊断性dna标记的进一步发展具有重要意义,可用于抗病品种的加速选育。与斑点病抗性相关的基因组位点检测主要采用两种方法:(1)双亲本作图群体的qtl分析;syn。-关联映射(AM))。
对12个双亲本作图群体的qtl分析揭示了大麦所有染色体抗斑点病的位点[1那2那3.那4.那5.那6.那7.].关联定位法也已成功地应用于新位点的鉴定,利用不同的种质资源如野生大麦[8.],美国大麦育种种质[9.,弗吉尼亚理工大学冬季大麦系[10.],源自拉丁美洲的种质[11.,以及美国农业部大麦核心收集[12.].这些研究积累的数据一方面提供了新的抗斑点病基因的信息,另一方面验证了AM方法,证实了双亲本群体的研究结果。例如,在这些出版物中最早的[8., 13个位点代表c .巨大成功在1H、2H、3H、5H和7H染色体上发现抗性位点,其中7个位点为新位点,6个位点为双亲本定位群体分析确认的qtl。上述最新的AM研究[12.]报道了10条与斑斑相关的染色体区域,其中6条是新发现的,说明不同种质调查和检测效果不同c .巨大成功检测新的斑点病抗性位点的小种。AM方法尚未广泛应用于俄罗斯大麦种质资源的研究,尤其是西伯利亚大麦种质资源的研究。
目前研究的目的是鉴定具有对两种不同病型的幼苗抗性的基因座c .巨大成功利用50 K Illumina单核苷酸多态性芯片对西伯利亚春大麦核心种质(Kr2和Ch3)进行筛选。
结果
表现型
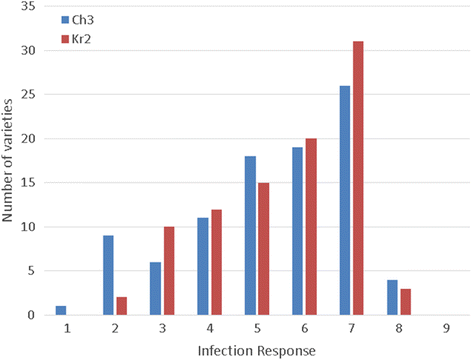
大麦基因型展出的IRS(感染响应)通常在每个实验中的三次重复之间同意。在图2中给出了大麦基因型的平均IRS的频率分布。1.只有3个品种(G-19980、Biom、AC 0760258)表现杂合度,因此排除在进一步分析之外。大部分大麦基因型(43.0%)对两种病均敏感c .巨大成功隔离;53%与Ch3和58%与Kr2 (IRs - 6-9)。9个品种对两种菌株均有抗性:B-1、G-21219、Kolchan、Mutant 68、Omsky Golozyorny 2、Severny、Signal、Svetik和Tanay。16个品种在分离Ch3和15时表现出抗性反应(IRs - 1-3.9)。中等阻力(IRs - 4.0-5.9)至c .巨大成功29个品种分离出Ch3, 24个品种分离出Kr2。品种Emelya、impulse、Kedr、Merit 57和Reyd表现出分离(小种)特异性抗性,对一个分离物敏感,对另一个分离物抗性。11个中抗品种对分离出的Ch3易感,8个中抗品种对分离出的Kr2易感1).
基因分型和GWAS分析
利用50 K SNP iSelect序列对94份春大麦材料进行基因分型,共包含44,040个SNP位点,其中有40,703个位点得分。共有39140个snp(89%)具有多态性。聚类分析的结果显示大麦品种之间的关系在附加文件中2.聚类分析结果显示,供试材料分为4大类,分别为38、7、19、30个基因型。
对基因分型数据集进行质量控制筛选后,共筛选出27,319个snp(62,0%)用于GWAS。通过GLM数据分析,分别检测到48和41个与Kr2和Ch3评分显著相关的SNPs(见表)1和2额外的文件3.).应用Bonferroni多次试验修正后,修正量为5% (p< 1.8302E-6), 48个SNPs中只有3个对分离Kr2评分有显著性(表1),而对于41的CH3 27有重要意义p值(表2).
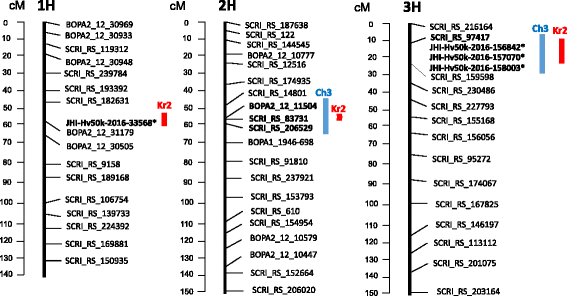
共有3个基因组区域与抗性相关。3H染色体上与ch3抗性相关的区域在SCRI_RS_97417和JHI-Hv50k-2016-158003之间扩增,共包含11个SNPs,其中JHI-Hv50k-2016-157070、JHI-Hv50k-2016-156842最低p值(表2).这两个单核苷酸多态性也与对Kr2分离株的抗性显著相关(见表)1).染色体的区域2h包括16个基因座(表2),其中7个得分最低p-values紧密链接到BOPA2_12_11504。与该区域相对应的三个位点具有暗示性p在Kr2测试的情况下(表1)),因此2H染色体上的位点也可能对kr2分离株产生抗性。第三个区域具有重要意义p- 在染色体上鉴定染色体1H,在轨迹JHI-HV50K-2016-33568中鉴定出,它与KR2抵抗有关(表1).所确定的基因组区域如图所示。2使用iSelect链接映射。
利用Morex / Barke iSelect地图定位与斑点病分离株Kr(红色)和Ch3(蓝色)抗性相关的基因组片段(http://bioinf.hutton.ac.uk/iselect/app/).虚线 - 暗示SNP。* - 标记根据物理位置相对于基因映射的SNPS放置
单核苷酸多态性分析最低p-值作为标记位点转化为方便的诊断标记,表明部分单核苷酸多态性在抗病品种鉴定中具有较高的准确性。例如,19个抗病品种中有14个(74%)在JHI-Hv50k-2016-157070位点具有c -等位变异特征。该等位基因还检测到另外4个品种对一个或两个分离株有中等抗性,但对两个分离株都没有敏感的基因型。
讨论
在94份材料中,47份(50%)是在西伯利亚育种中心培育的品种和品系。其余47份为西伯利亚春大麦种质,但来自其他地区和国家。聚类分析显示了4个主要的加入组(附加文件2).由38个(组I)和30个(组IV)基因型组成的两个大群体中,西伯利亚品种和株系的比例差异显著(分别为73%和17%)。第二组和第三组为“半西伯利亚”(附加文件)2).ⅰ组对两种菌株均有抗性的品种百分率最高c .巨大成功(其他三组18%vs,3和5%),敏感品种的最低百分比(32%vs 47,48和其他组中的50%)到两个分离株(附加文件2).这表明西伯利亚品种在春大麦抗性种质研究中发挥了重要作用。事实上,我们注意到,在对两种菌株都有抗性的材料中,67%来自西伯利亚。
GWAS发现了三个基因组区域(在染色体1H, 2H和3H)与对一个或两个分离株的耐药性有关(表)1和2,无花果。2).尽管本研究的样本量较小,但我们没有发现假阳性基因座。比较在我们的研究中确定的三个基因组区域的位置c .巨大成功前面揭示了电阻QTLS,表明三个区域中的每一个含有已知的斑点斑块电阻基因座。
染色体1 h
SNP与抗c .巨大成功Kr2分离株位于染色体1H遗传图谱的50 ~ 60 cM之间。在50和60 cM范围内,大麦染色体1H存在抗斑点病QTL。1996年,Steffenson等人[1报告了成人抵抗的QTLc .巨大成功染色体1H标记ABG500-ABG494 (53.6-61.2 cM,根据共识图[13.])。2005年,Bilgic等人[2]在同一地区用Steptoe / Morex (R)定位群体发现了主要抗性位点。2010年,Roy等人[8.通过关联作图,在59.7 cM处发现了苗期抗性位点。
随后,在40和50 cM范围内的大麦1H染色体上发现了抗斑点病位点。Gutierrez等人在2013年报道了与该区域的关联[11.他使用am方法利用dart标记。与此同时,周和斯蒂芬森[9.在1H染色体41-43 cM处发现了成苗抗性位点(与BOPA SNP标记11_10764、11_10275和12_30336相关)。2015年,Afanasenko等人[7.]发现了苗期抗性的QTL (snp位点BOPA11_10764,syn。bopa1_5381 - 1950;通过对Zernogradsky 85 (R) / Ranny 1双亲本作图群体的分析,确定其在iSelect map - 41.5 cM中的位置。2016年,Haas等人[6.]发现了苗期抗性的QTL (snp位点BOPA2_12_30404;PI 466423 (R) / Rasmusson双亲本居群分析最近,Wang等人[12.[报道了区域42-44cm(标记SCRI_RS_19392,SCRI_RS_153785,SCRI_RS_170878,SCRI_RS_170869,SCRI_RS_189483,BOPA1_5381_1950)的关联,具有幼苗抵抗力。
本研究在1H染色体上发现的抗性QTL可能与之前在40-60 cM区间内发现的QTL一致[1那2那6.那7.那8.那9.那11.那12.].除了40-60厘米内的染色体区域之外,染色体1H的两个区域与抗点斑点的抗性相关:长臂的远端部分(1HL; QTL在SNP-LOCUS BOPA_11_10433中。syn。bopa1_3201-603;Iselect地图中的位置 - 87.0 cm [7.])和短臂(1HS;基因rcs6.在加利福尼亚州。15厘米[3.那14.], QTL位于dart位点bbp - 9604;16.9厘米(5.])。
染色体2 h
我们在2H染色体上发现的Ch3分离株抗性区域(57-60 cM)有16个SNP位点(表)2).与该区域相对应的三个SNP位点提示p在Kr2测试的情况下(表1),因此2H染色体上的抗性位点也可能对kr2分离株产生抗性(图2)。2).
本研究在2H染色体上发现的抗性QTL可能与之前鉴定的苗期抗性QTL一致[2那7.].2005年,Bilgic等人[2]利用Steptoe / Morex (R)定位群体,在27.1 ~ 46.9 cM (Rbcs-ABG459)区间内检测到QTL。后来,Afanasenko等人[7.]发现snp位点BOPA_11_11015的QTL (syn。bopa1_946 - 2500;通过对Zernogradsky 85 (R) / Ranny 1双亲本作图群体的分析,确定其在iSelect map - 54.2 cM中的位置。
除了这种染色体区域之外,染色体2H的另一部分(长臂的远端部分)与抗点斑的抗性相关。Wang等人。[12.]与SCRI_RS_152664 (138.6 cM)显著相关。
染色体3 h
与ch3抗性相关的3H染色体区域在标记SCRI_RS_97417和JHI-Hv50k-2016-158003之间扩展,包含11个SNPs,其中JHI-Hv50k-2016-157070、JHI-Hv50k-2016-156842最低p值(表2).这两个单核苷酸多态性对Kr2菌株的抗性也很显著(见表)1).遗传标记位置为12.1 cM。
在我们的研究中,在染色体上发现的抗性QTL可能与一些先前识别的QTL相一致。Bovill等人。[4.在3H染色体2 ~ 28 cM范围内,利用不同的定位群体发现了植株抗斑点病的qtl。后来,周和斯蒂芬森[9.在3H染色体上9.6 cM(标记BOPA_12_30818)和19.2 cM(标记BOPA_11_20742、BOPA_11_10565)位点发现了苗期和成虫抗性位点,并利用关联作图法进行了分析。
还发现了染色体上的更多近端位置。Bilgic等人。(2005)揭示了使用STEPTOE / MOTERX(R)映射群体的间隔28.7-42.4(标记ABC171-MWG584)中幼苗抵抗的基因座。GREWAL等人。[5.利用CDC Bold / TR251杂交组合的DH重组品系(R),发现苗期抗性QTL位于24.9-31.1 cM(标记bPb-3565),成株抗性QTL位于23.0-24.9 cM(标记bPb-6127)和31.8-43.3 cM(标记E40M61.1)。12.]报道了3H染色体上的两个区域,分别与25.3 cM处的BOPA1_3906_558和66.2 cM处的BOPA1_5960_1302相关。
结论
三个基因组区域与对一个或两个分离株的抗性有关c .巨大成功经西伯利亚春大麦核心种质筛选鉴定。将其与之前在双亲本定位群体研究中发现的qtl或与不同种质和其他分离株的GWAS进行比较,结果表明Kr2和Ch3的抗性位点是由已知的斑点抗性位点决定的。与Kr2和Ch3菌株抗性相关的SNPs信息和品种抗性评价结果可为进一步的分子标记辅助选择提供依据。snp可以转化为方便的诊断标记(如CAPs)。品种B-1、G-21219、Kolchan、Mutant 68、Omsky Golozyorny 2、Severny、Signal、Svetik和Tanay可作为抗斑点病稳定供体。
方法
植物材料
选取西伯利亚春大麦核心种质96份材料进行表型分析。其中一半是在西伯利亚(阿勒泰、布里亚特、伊尔库茨克、克麦罗沃、新西伯利亚、鄂木斯克、托木斯克、秋明和雅库蒂亚)育种中心培育的品种和品系。另外一半的品种和品系保存在西伯利亚春大麦收集,但来自俄罗斯联邦的其他地区(阿尔汉格尔斯克、车里雅宾斯克、达吉斯坦、基洛夫、克拉斯诺达尔、列宁格勒、莫斯科、奥伦堡、Primorsky、罗斯托夫、萨马拉、斯塔夫罗波尔、斯维尔德洛夫斯克、或其他国家(埃塞俄比亚、德国、哈萨克斯坦、吉尔吉斯斯坦、瑞典、瑞士、乌克兰、美国、也门)。植物生长在塑料托盘(12×17厘米,7厘米深度)装满土壤“Terra维塔”(标准受精(15-27-30,氮磷钾)泥炭和幼苗土壤)的混合物在气候室在20 - 22°C与交替16 h光明/ 8 h的黑暗时期(曝光5000 lx)。采用完全随机设计,3个重复,每盘2株各基因型和品种Harrington作为敏感对照进行抗性评价。
病原菌分离及培养条件
二c .巨大成功利用不同来源的分离株进行幼苗抗性评价:Ch3(俄罗斯西北部列宁格勒地区)和Kr2(俄罗斯欧洲南部克拉斯诺达尔地区)。在以前的研究中,分离物Ch3已被用于广泛的大麦基因型的抗性评价,并对敏感大麦基因型表现出很高的攻击性。选择Kr2菌株是由于其起源和产孢能力强。这些分离株来源于2015年的单个分生孢子,在4°C的CLM培养基(含乳糖和尿素的改良Czapek培养基)中,置于玻璃管中,培养基中(g / 1 l): 0.5 KH2PO4, 0.5 MgSO4, 0.5 KCl, 1.2尿素,20乳糖和20琼脂。
分离菌株在相同的培养基上继代培养,20-22℃,光周期12 h。10-12天后用蒸馏水浸泡培养物,用无菌抹刀去除分生孢子,并通过两层粗棉布过滤。用血球计测定分生孢子浓度,并调整至10000分生孢子/ml。
种植后12 ~ 14 d(2 ~ 3叶期)喷施分生孢子悬液接种植株。每盘(12份大麦)接种约10ml。接种后的植株用塑料袋覆盖,在相对湿度100%的黑暗条件下20-22℃培养24 h,然后在相同的温度下,在70% RH条件下培养16/8 h光暗周期。
感染反应评估
采用Fetch和Steffenson的1 - 9评分标准,记录接种后10天2- 3叶期的感染反应(IRs) [15.].该规模基于病变大小和相关氯化度的程度。低IRS 1.0-3.9(分钟到没有或非常轻微漫射边缘氯仑的小坏死病变)对应于抗性(R),4.0-5.9(中等大小的坏死病变,具有明显但毫无抗体的杂物余量) - 至中等抗性(MR),高IRS - 6.0-9.0(具有不同杂种杂物的大坏死病变和不同程度的扩张扩散氯化) - 对易感性[15.].仅当敏感品种哈林顿表现出高IRs时,才记录反应类型。
DNA基因分型及数据分析
使用DNeasy Plant Mini Kit (Qiagen, CA, USA)从单株幼苗中提取DNA。所有样本均使用tritgenetics GmbH (Gatersleben, Germany)的5万份Illumina iSelect SNP序列进行基因分型。
系统发育分析使用MEGA软件v6.0进行[16.].遗传关系通过最大似然(ML)分析,Tamura-Nei模型计算。引导重复次数为500次。
使用Excel软件过滤SNP数据集。最小等位基因频率≥0.10的标记被考虑为GWAS。删除数据缺失的标记。GWAS是在TASSEL 5包的帮助下使用GLM(广义线性模型)进行的[17.,基于表型(抗性得分为2c .巨大成功对94份大麦材料进行基因型分析(27319个信息snp)。关联分析后使用Bonferroni多重检验进行校正。从资源BARLEYMAP (http://floresta.eead.csic.es/barleymap[18.])及我选择(http://bioinf.hutton.ac.uk/iselect/app/).
参考文献
- 1。
Steffenson Bj,Hayes PM,Kleinhofs A.幼苗和成人植物抗净污染的遗传学(Pyrenophora圆柱状的f。t)斑点斑点(Cochliobolus巨大成功)的大麦。Theor Appl Genet 1996; 92:552-8。
- 2。
综合遗传分析表明,4个大麦群体对斑点斑病抗性存在差异表达。Theor Appl Genet. 2005; 111:1238-50。
- 3.
Bilgic H, Steffenson BJ,海耶斯总理。大麦斑疹病抗性位点的分子定位。植物病理学。2006;96:699 - 708。
- 4.
Bovill J, Lehmensiek A, Sutherland MW, Platz GJ, Usher T, Franckowiak J, Mace E. 4个大麦群体斑点斑病抗性基因的定位。摩尔品种。2010;26:653 - 66。
- 5.
Grewal TS, Rossnagel BG, Scoles GJ。大麦双单倍体群体中与斑点和网状斑点抗性相关的数量性状位点的定位。摩尔品种。2012;30:267 - 79。
- 6.
赵志强,王志强。定位对一种广泛的毒力分离物具有抗性的数量性状位点Cochliobolus巨大成功在野生大麦accession PI 466423。Theor Appl Genet. 2016; 129:1831-42。
- 7.
Afanasenko OS, Koziakov AV, Hedlay PE, Lashina NM, Anisimova AV, Manninen O, Jalli M, Potokina EK。定位控制抗性的基因座大圆蕨和Cochliobolus巨大成功在两个双单倍体大麦群体中。拉斯。j . .Genet。达成。杂志2015;5:242-53。
- 8.
作者简介:王志强(1983 -),男,辽宁大连人,博士,主要研究方向为神经网络。野生大麦抗斑点病的关联作图。摩尔繁殖。2010;26:243-56。
- 9.
周华,Steffenson j。全基因组关联作图揭示了美国大麦耐久斑病育种种质的遗传结构。摩尔品种。2013;32:139-54。
- 10.
Berger GL, Liu S, Hall MD, Brooks WS, Chao S, Muehlbauer GJ, Baik BK, Steffenson B, Griffey CA。Theor Appl Genet. 2013; 126:693-710。
- 11.
Gutiérrez L, Berberian N, Capettini F, Falcioni E, Fros D, Germán S, Hayes PM, Huerta-Espino J, Herrera S, Pereyra S, Pérez C, Sandoval-Islas S, Singh R, Castro A.全基因组关联图谱确定了拉丁美洲大麦种质的抗病qtl。见:张刚,李超,刘旭,编辑。大麦科学的进展。第11届国际大麦遗传学研讨会论文集;2013.209 - 15页。
- 12.
王锐,冷艳,阿力,王敏,钟森。三种不同病型的斑点斑病抗性的全基因组关联分析Cochliobolus巨大成功在美国农业部大麦核心收藏中。摩尔品种。2017;37:44。
- 13。
陈志强,陈志强,陈志强,等。青稞白粉病的基础抗性:数量性状位点与候选基因的连接。MPMI。2010; 23:91 - 102。
- 14.
李Wenzl P, H,联赛J,周M,拉曼H,保罗•E Hearnden P,麦尔C,夏L, Caig V, Ovesna J, Cakir M,保尔森D,王J,拉曼R,史密斯KP, Muehlbauer GJ,查尔默斯KJ, Kleinhofs, Huttner E,大麦Kilian A .高密度共识地图链接飞镖SSR标记,RFLP和STS基因座和农业特征。BMC基因组学。2006;7:206。
- 15.
去找TG, Steffenson BJ。用于评估大麦侵染反应的评定量表Cochliobolus巨大成功.工厂说。1999;83:213-7。
- 16.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物学杂志。2013;30:2725-9。
- 17.
张志,kon DE, Casstevens TM, Ramdoss Y, Buckler ES。TASSEL:用于不同样本中复杂性状关联制图的软件。生物信息学。2007;23:2633-5。
- 18.
坎帕普德拉CP,Boudiar R,Casas Am,Igartua E,Contreras-Moreira B. Barleymap:核苷酸序列的物理和遗传映射和大麦周围基因座的注释。mol品种。2015; 35:13。
致谢
我们感谢ICG collection“GenAgro”(新西伯利亚,俄罗斯)和Tatiana Kukoeva和Yuri Grigoryev,他们形成并提供了春大麦核心收藏。我们也感谢tritgenetics GmbH (Gatersleben, Germany)利用50 K Illumina snp芯片对大麦品种和品系进行基因分型。
资金
出版费用由俄罗斯科学基金会资助(第16-14-00086号)。ICG植物生长核心设施中大麦植株的生长由ICG项目0324-2016-0001提供支持。
数据和材料的可用性
不适用。
关于这个补充
本文已作为一部分发布BMC植物生物学2017年第17卷增刊2:2017年Belyaev会议植物生物学文章选集。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-17-supplement-2.
作者信息
从属关系
贡献
IVB进行DNA提取、种群结构和GWAS分析。NML评估感染反应。VME参与了种群结构和GWAS分析。EKK和OSA进行了实验设计,帮助解释数据和准备手稿。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
西伯利亚春大麦核心种质抗斑点病评估结果表。抗R (1.0-3.9);MR中度耐药- (4.0-5.9);S敏感(6.0-9.0);“——”——失败。(多克斯19 kb)
额外的文件2:
基于50k SNP基因分型的大麦品种间关系研究I、II、III和IV -主要集群。品种名称:易黑一c .巨大成功分离和抗性(中等抗性)到另一个;绿色 - 适度耐用一个隔离和抗性/适度抵抗另一个;绿色下划线 - 抵抗均外分离株;红色 - 易感两种分离株。与群集对应的图表:橙色/蓝色 - 非西伯利亚/西伯利亚品种;绿色/红色/灰色耐抵抗分离株/易受分离物/其他的分离物。(PDF 197 KB)
额外的文件3:
曼哈顿甲基抗斑斑的关联映射研究的绘制研究分离KR2(C)和CH3(D)的GWAs的KR2(A),CH3(B)和定量定量(QQ)曲线。(PDF 307 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
别科娃,静脉注射,拉什娜,新墨西哥州,艾菲莫夫et al。大麦斑点病抗性相关50 K Illumina-chip单核苷酸多态性的鉴定BMC植物杂志17,250(2017)。https://doi.org/10.1186/s12870-017-1198-9
关键字
- 关联映射
- 大麦
- Cochliobolus巨大成功
- GWAS
- 大麦芽
- 电阻
- 单核苷酸多态性