摘要
背景
锌(Zn)是生物必需的微量营养素。锌的缺乏会扰乱植物的重要功能,因为包括转录因子在内的大量酶都依赖于锌的正常发挥。植物对缺锌的反应与高亲和力锌的吸收和转运以及剩余锌的有效利用有关,但在玉米中还没有详细的分子特征。
结果
高亲和力转运体基因Zmzip3,4,5,7和8和尼古拉胺合成酶,主要是ZmNAS5在长期Zn缺乏时被鉴定为玉米根中的初级上调。除了编码参与途径调节活性氧物质和细胞壁相关基因的酶的基因的下调之外,蔗糖流出通道基因的大规模上调SWEET13a c尽管在-Zn中糖是在芽中积累的。此外,参与DNA维持甲基化的酶趋于被抑制,这与缺锌根中DNA甲基化的大量减少相一致。还原硫酸氢盐测序揭示了玉米基因组中14%的碱基特异性甲基化模式,鉴定出-Zn的主要甲基化损失,主要发生在转座元件中。然而,在-Zn中也发现了高甲基化的基因组区域,特别是在对称胞嘧啶环境中。甲基化差异部分与差异表达基因、其启动子或接近调控基因的转座子有关。然而,低甲基化与大约相同数量的上调或下调基因有关,这对基因表达的简单机制关系提出了质疑。
结论
Zn缺陷型根的转录组鉴定了应对缺乏的基因和途径,并对反应性氧代谢的主要下调。有趣的是,营养特异性的DNA甲基化损失,部分地与基因表达以语境特异性的方式,可能在长期应力适应中发挥作用。
背景
锌(Zn)是地球上每一种生物都必需的微量营养素。它是植物中大量转录因子和酶的重要辅助因子[1].锌可以用作结构部件,其助使在适当的蛋白质折叠和作为催化组分中,使能或增强通过酶进行的反应。另外,还需要适当的膜完整性,并参与RNA和DNA代谢以及基因表达调控。它进一步参与超氧化物自由基的解毒和植物激素的合成[1,2,3.,4].考虑到锌在植物中的这些功能,植物长期遭受严重缺锌(-Zn)并发展出一系列应对机制也就不足为奇了。缺锌在钙质、沙质或泥炭质土壤中尤其普遍。由于它们的pH值高,会使锌吸附到粘土或碳酸钙上,根系能吸收的生物有效锌仍然有限,尽管这类土壤中的锌含量很高。在渍水土壤中,锌在氧化的根际形成少量可溶化合物,从而增加了锌缺乏的风险,这影响了全世界的水稻生产[5].总而言之,据估计,约50%的谷类作物农业土壤可能缺锌[5].
在玉米中,缺锌导致净光合作用降低、气孔导度降低等生理变化,包括生长发育不良、维管束比例降低、气孔密度升高等形态异常[6].众所周知,在缺锌情况下,来自ZIP (Zrt/Irt-like蛋白)家族的锌转运基因上调,以促进微量剩余锌的摄取[2,7,8],但在玉米中,这些基因在茎和根中的-Zn含量仅微幅上调[9].此外,在锌耐受基因型中,锌转运基因zmzip1,zmzip4和Zmirt1.锌缺乏可适度上调,但非敏感基因型[10.].
但不仅植物需要足够量的锌,也是动物和人类受缺锌的负面影响。在人类中,特别是发展中国家的普遍存在,缺锌涉及超过20亿人。免疫系统需要人类锌,用于DNA合成和RNA转录,以及细胞分裂和预防细胞凋亡[11.].锌缺乏症的健康后果从体重减轻对免疫系统的减值损害,仅限一些[12.].就此而言,通过作物产品充分摄入锌有助于缓解这种营养障碍。实现这一方法的一种方法是生物侵蚀,通过育种来增加重要作物植物的营养价值。关于使用锌或应对缺锌的植物机制的知识是必要的。关于植物如何处理锌的实质性了解,尽管植物中锌稳态的分子机制仍然是零碎的[2].最近,包括营养压力在内的许多压力都报告了DNA甲基化的变化。在不同的情况下,例如磷饥饿,基因表达变化与DNA甲基化差异相关,特别是在动态的、非对称胞嘧啶环境中[13.,14.].这种甲基化背景,也负责de-novo.DNA甲基化依赖于rna定向DNA甲基化(RdDM)途径,而srna在RdDM途径中发挥重要作用。在这一途径中,植物特有的dna依赖的RNA聚合酶产生短RNA转录本,其中双链RNA (dsrna)由RNA依赖的RNA聚合酶产生。反过来,这些dsrna被一种叫做dicer-like 3 (DCL3)的蛋白质切割,形成21到24个核苷酸srna,然后这些srna被合并到argonaute 4 (AGO4)中。这个复合物然后利用rna的序列信息引导DNA甲基化[15.,16.,17.].
我们假设,在模式植物中被鉴定为受缺锌调控的主要基因类别,在缺锌玉米根系中也受到类似的调控。然而,玉米特有的基因也可能被识别出来。因此,我们对供应充足和缺锌的玉米根系进行了rna测序。虽然鉴定了典型的缺锌反应基因,但我们注意到参与DNA甲基化的基因也受到-Zn的影响,因此量化了缺锌对DNA甲基组的影响。植物的DNA甲基化被认为影响植物的不同功能,如协助病原菌应答、基因组稳定性、杂种优势,以及对转座子(TEs)和基因的抑制[18.,19.].在Zn缺乏症下是否和DNA甲基化与基因表达相关,并讨论了。
结果
玉米植株的表型和养分浓度
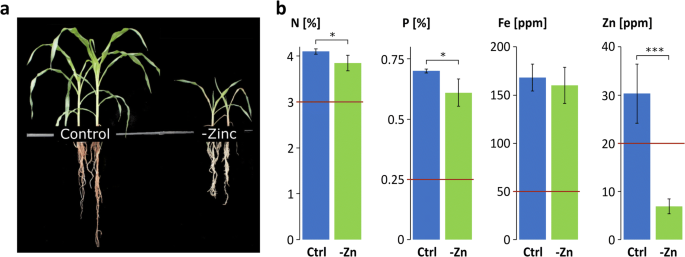
对照植株和-Zn植株表型调查显示,对照植株生长旺盛,而-Zn植株表现出典型的缺锌诱导症状,生长发育严重迟缓。叶和根变小,叶上可见褪绿和坏死的痕迹[3.] (无花果。1a).营养分析证实了-Zn植株的地上部缺锌,而铁则接近于控制水平,铁可以增强活性氧胁迫,在缺锌条件下,铁含量往往更高。1b). Zn含量仅为6.9 ppm,而对照植株Zn含量超过30 ppm,其他所有被测养分均充足,尽管缺锌植株的氮和磷水平略有降低(图2)。1b)。
缺锌了转录组
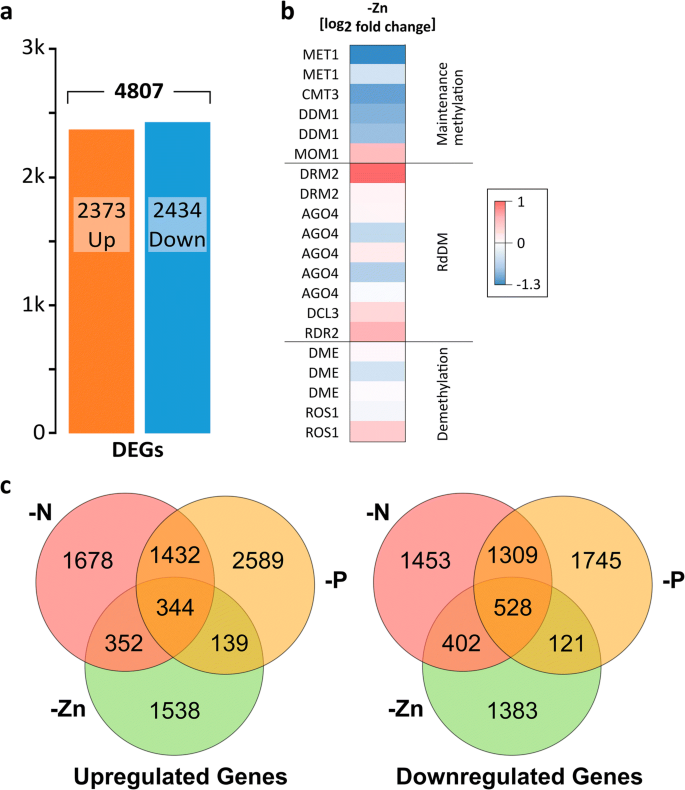
然后获得延长缺锌下的根转录组,并与对照进行比较。在从对照和-Zn根采集的RNA样本中,对照的比对率(91.8%)高于-Zn的79.6%(附加文件1:表S1)。将对齐的转录本组装起来,并对差异表达基因进行分析。共鉴定出4807个显著差异表达基因(DEGs)在对照和-Zn处理之间。上调或下调的基因数量几乎相等,分别为2372和2434个(图。2a).与先前测试的短期或轻度锌缺乏相比[9],几种编码锌吸收系统的基因,ZmZIP3, ZmZIP4, ZmZIP5, ZmZIP7和zmzip8,大幅上调(表1).此外,编码烟胺合酶(NAS)的基因尤其丰富ZmNAS5,在缺锌期间持续上调。NAS合成烟胺,而烟胺又参与了重金属,如锌和铁,在器官之间的转运。锌是通过依赖铜/锌的超氧化物歧化酶来解毒超氧化物自由基所需要的,而这种酶的转录产物在缺锌情况下下调,这与许多其他植物的结果一致[3.].在许多过氧化物酶中也发现了强烈的下调,63个差异表达过氧化物酶中有55个表达水平降低(其中2个为典型,见表)1).碳酸酐酶,众所周知,需要锌作为辅助因子和催化CO2水合,在-ZN中也减少了(表1).紫色酸性磷酸酶有增加,这有助于维持无机磷酸酯代谢,并且可用于动员与磷酸盐复合物的Zn [20.].相比之下,在-Zn作用下,参与植物细胞扩张的Expansins表达下调,这与-Zn作用下器官变小一致。有趣的是,糖外排转运基因的强烈上调Sweet13a.和C,这参与叶子中蔗糖的韧皮含量[21.],被指出(表1).
基因在-Zn中的差异表达(一种)差异表达基因的数量(DEGS,P. < 0.05), either up- (orange) or down-regulated (blue) in Zn deficiency.B.日志2与DNA甲基化和去甲基化有关的推定基因表达的差异。蓝色:下调,红色:上调。(C)不同缺失(-Zn, -N, -P)中上调(左)和下调(右)基因重叠;来自[22.]
基因富集分析显示,参与氧化应激反应的酶的基因编码中有大量下调类别,如前面提到的过氧化物酶和超氧化物歧化酶。下调最显著的三个氧化石墨烯项类别包含参与氧化应激反应的基因(见表)2).尽管上调和下调基因的个体总数相似,但下调基因类别比显著上调基因类别要明显得多,后者只包含一个单独的GO项,这与次级代谢物的产生有关。
有趣的是,一些编码维持甲基化酶的基因也趋向于下调,而一些去甲基化酶则趋向于上调(图)。2b).参与RdDM通路的基因的调控不太一致,但值得注意的是(去)甲基化相关基因的表达总体上相对较低,这在一定程度上质疑了观察到的差异的意义。
比较缺锌下的差异表达量与之前分析的氮、磷缺锌下的差异表达量[22.[我们注意到在所有三种缺陷(344和528分别在所有三种缺陷下调节或下调的基因重叠(344和528。2C)。然而,更大量的基因与个体营养缺陷特异性相关,并且这些包括与上面讨论的-ZN相关的基因。
DNA甲基化对缺锌的适应性
转录数据表明,适应-Zn的根可能被去甲基化,通过还原亚硫酸氢盐测序从相同的根材料中提取的DNA进行了实验检验,并生成了营养分析和RNA-seq谱。这些数据提供了每个样本中14%的玉米基因组(约占所有胞嘧啶的18%)的序列特异性甲基化信息。亚硫酸氢盐处理后的转化率为> 98%(98.41 ~ 99.18%)。大约90%的胞嘧啶在所有情况下至少被5次读取(附加文件1:图S1)和可映射性> 48%(附加文件1:表S2)表示可靠下游分析的足够覆盖和可用性。
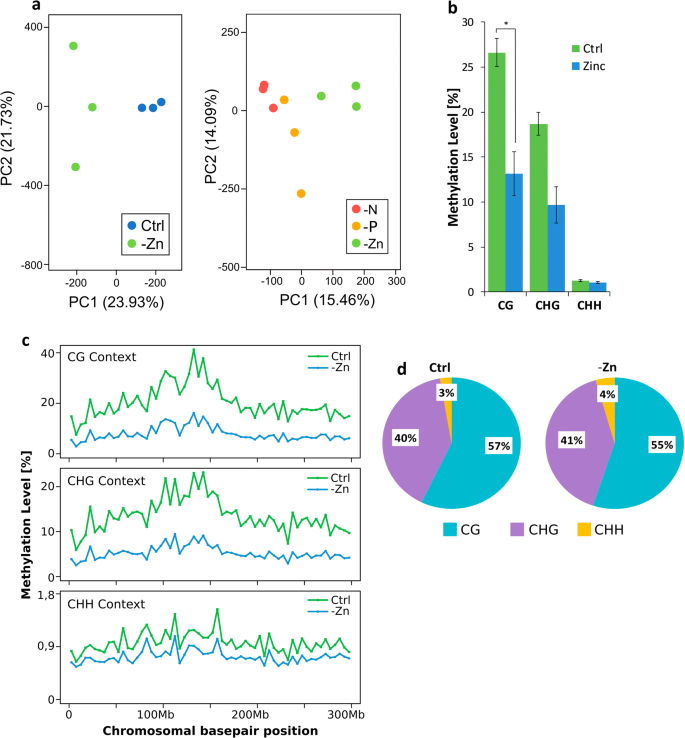
对对照和-Zn样品的胞嘧啶甲基化水平进行主成分分析(图)3.a).主成分1 (PC1)的方差解释了约24%的总方差,并将对照组和-Zn样本分成两组。PC2显然主要是由于-Zn的重复数之间的差异。我们还将我们的数据集与-N和-P处理玉米根系的数据进行了比较[22.[这揭示了每种营养素缺乏的甲基肟样品主要通过PC1分离。这种方差在三种处理之间解释了大约15%的总差。
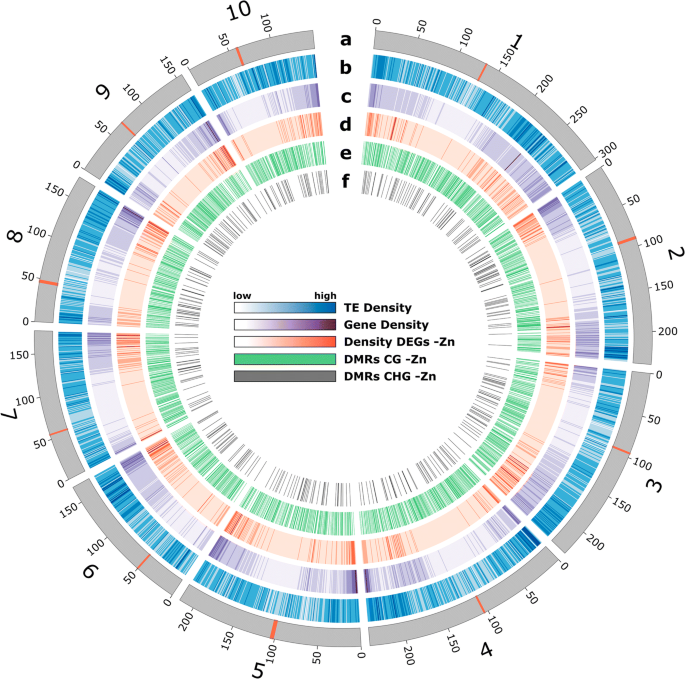
DNA甲基化水平(一种)还原亚硫酸氢盐测序数据的主成分分析(PCA)。左图:控制状态(蓝色)和-Zn(绿色)。右图:锌、氮、磷缺乏,部分数据来自[22.].显示前两个PC。B.所有情况下的总甲基化水平(以百分比计),对照:蓝色;施锌:绿色。C在所有上下文中,平均(跨越500万个基座栅格)染色体1中的甲基化水平。D.每个上下文甲基化对总甲基化水平的贡献,控制:左;-ZN:吧
发现CG和CHG上下文(H = A,T或C)中的大规模甲基化丧失(图。3.b)。在对照中,CG上下文中的26.6%的所有胞嘧啶都甲基化,而在-ZN样品中,只有大约一半的CGS甲基化(13.2%)。在CHG上下文中还存在显着的甲基化酶 - Zn样品,其中9.7%在-ZN中甲基化,相比于对照样品中的18.7%。CHH的背景几乎不受缺陷的影响。在这种情况下,胞嘧啶仅显示出非常低的甲基化,如我们以前的分析中已发现[22.].在-ZN样品中略微进一步降低该甲基化水平,从1.26%的对照中的1.26%达到1.06%。
通过DMRcaller分析整个染色体的甲基化分布(图。3.c). 1号染色体三种不同胞嘧啶环境的甲基化水平是一个代表性的例子。特别是在CG和CHG背景下,甲基化在着丝粒区更高,而在染色体的末端降低。同样,CHH环境形成了一个例外,因为在这里,着丝粒的高甲基化几乎不可见。CG和CHG甲基化的减少相对一致,尽管大多数甲基化似乎在着丝粒区和中着丝粒区丢失,降低了相对较高的着丝粒区整体甲基化水平。在所有染色体中,-Zn的甲基化减少在CHH环境中比在其他两个环境中不明显。
即使在缺乏样品中丢失了大量甲基化,在每个上下文中发现了甲基化胞嘧啶总数的类似部分(图。3.d).这表明,在每种情况下,甲基化的相对损失量是相同的。
目前认为单个碱基甲基化变化与功能关系不大,因此只考虑差异甲基化区域(DMRs)。我们将DMRs定义为50 ~ 500 bp范围内包含至少4个胞嘧啶的区域,每个胞嘧啶需要超过3个reads,其甲基化差异超过40%,p-截断值≤0.01。共鉴定出2762例dmr,大多数dmr在CG环境中(见表)3.).在CHG上下文中,确定了402个DMR。相比之下,在CHH上下文中,发现只有3个DMR。对于DMRS的严格截止值(至少3个胞嘧啶,每胞嘧啶至少3次读数,并且鉴定至少10%甲基化差)41dMR(表3.).
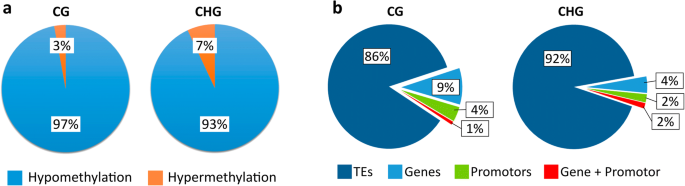
在-Zn中,CG和CHG的甲基化水平大幅下降,DMRs优先低甲基化,但在-Zn中也发现了高甲基化区域。这表明,除了甲基化损失外,-Zn还发生了进一步的从头甲基化。总的来说,大约93-98%的DMRs低甲基化(图。4一个)。
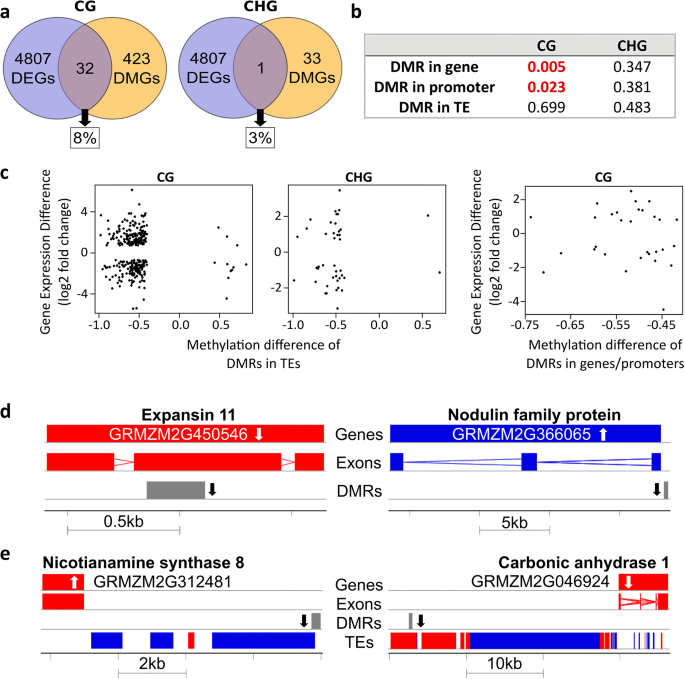
CG和CHG的dmr主要位于te内,CG和CHG的dmr分别超过86和92%。4b).考虑到85%的玉米基因组被认为是由TEs组成的,这表明在CHG环境中DMRs优先针对TEs。这种对dmr的偏好在脑电图中并不明显。同时跨越启动子和基因的dmr相对较少,在两种情况下都只有1-2%。位于启动子中的DMRs也相对少见,根据环境的不同,只有2-4%。在CG和CHG环境中,基因中的DMRs分别达到9%和2%。
DNA甲基化差异与转录变化的相关性
然后分析RRBS和rna测序数据,以确定甲基化与基因表达差异之间可能的相关性。TEs、基因、DEGs和DMRs在玉米基因组全部10条染色体中的分布如图所示。5.中心[23.]标有红线。对每个染色体臂的末端的基因的富集与在-Zn中的更多次数相关。相比之下,CG和CHG DMRS相当均匀地分布在每条染色体上,并且优先于TES局部化。
由于仅在基因组的代表性部分中测量甲基化,但是RNA-SEQ覆盖了所有差异表达的基因,因此我们计算了在其基因体中含有DMR的差异表达基因的百分比。还测定了在基因上游的2kB区域中具有DMRS的差异表达基因的分数,以及附近的DMRS附近(图。6一个)。在CG上下文中,423个差异甲基化基因的32个也差异地表达,含有DMR的8%的基因。在CHG上下文中,仅单个基因差异甲基化并同时差异表达,仅包含3%。
基因表达与DNA甲基化差异的关系(一种)在CG上下文(左)和CHG上下文(右)中,在差异表达基因(DEGS)和差异甲基化基因(DMG)之间重叠。8%或3%的DMG也分别差异表达。B.P.Fisher精确检测基因、启动子和邻近te中DMRs之间的相关性。C差异甲基化基因的散点图也有差异表达。左:CG背景下相邻te的DMRs;中间:CHG背景下相邻te的DMRs;右:启动子或基因体中的DMRs。d、eCG上下文中的差异甲基化基因的实例用差异表达。白色箭头=基因表达上/下,黑色箭头=甲基化上/下,红色=前向股,蓝=反向股线(d)基因体/差异表达基因的启动子DMRS。E.在TEs附近差异表达基因中的DMRs
为了检验上述观察结果的统计学意义,我们只考虑在减少代表性的甲基组中被至少500个碱基对覆盖的基因。相比之下,没有对差异甲基化的te进行长度限制,以测试其对最近基因表达的影响,因为许多te非常短,否则这些会在分析中丢失。在CG环境中,基因表达与启动子或基因本身的甲基化有显著的相关性(5%水平)P.-值分别为0.023和0.005。6b).另一方面,差异甲基化TEs与最近基因的差异表达无显著相关性。
为了进一步检查差分甲基化和基因表达之间的关系,施加散射图,用于差异甲基化的TES,其最接近基因差异表达,以及在其启动子或基因体中具有DMR的差异表达基因(图。6C)。数据点偏向图的左侧部分,因为甲基化主要减少。有趣的是,差异甲基化的启动子/基因体和差异甲基化的TES在上调或下调的基因中,而不偏好进行任一化。因此,即使发现差异表达基因/启动子和基因的表达存在显着的相关性,可以通过去甲基化抑制或刺激基因表达。如果考虑了散点片的所有基因(而不是显着差分表达的,附加文件,则发现了类似的结果1:图S2)。
在所有差异表达的基因中,在其基因体、2 kb启动子或附近的转座元件中具有DMR的基因中,有几个基因可能与锌缺乏反应有因果关系。图中显示了一些差异表达基因的例子,DMRs在基因结构中的位置。6d.在表中讨论的差异表达基因中1,扩展素基因突出且下调在-ZN下。低甲基化DMR位于扩展蛋白11的基因体中,而Nodulin基因在转录起始位点的上游上游升级并使甲基甲基化。在图2中给出了差差异表达基因的差异甲基化TES的两个实例。6e。尼古拉胺合成酶ZmNAS8上调和表达下调节的碳酸酐酶,而每个碳酸酐酶位于甲基化的TE附近。这表明虽然低甲基化是显性的,但在低甲基化时发生基因表达的上调和下调。
由于小RNA在RNA导向的DNA甲基化途径中发挥作用,这对CHH甲基化尤其重要,但对CHG和CG环境也很重要,我们推测,在甲基化大量丢失的-Zn样品中,小RNA可能减少。观察发现,-Zn样本中小RNA占总RNA的比例要低得多,为1.4%,而对照组为9.7%(附加文件)1:图S3)。
讨论
Zn缺乏植物的长期生长引起了根部的一系列变化,包括转录组和表观遗传变化。缺乏植物(6.9ppm)的Zn水平远低于通常被认为足以用于玉米(20ppm)的浓度。这些发育不良的植物具有类似的铁浓度,但它们在n和p略微降低,可能反映了延长Zn缺乏的二次代谢效应,但两种宏观重组都处于足够的范围内。我们进一步的重点是根甲基汞的代表性分数及其整个转录组。
玉米根转录组在延长Zn缺乏时
对于某种营养素的高亲和力转运蛋白的大部分表达是典型的延长缺乏的关键特征,提供了效率吸收剩余的营养素的剩余痕量痕迹。对于Zn缺乏的根,锌跨膜转运蛋白基因的大量上调,以前发现仅被玉米芽和根部的Zn缺乏率略微上调[9),观察。这些包括Zmzip3,4,5,7,8.和一个ZIP1相关基因,在-Zn下也有类似水平的表达,但烟碱合成酶(NAS),其参与合成重金属螯合剂尼古罗胺[7].来自-ZN这个基因家族的最突出的基因是ZmNAS5,而碳酸酐酶基因在-Zn玉米根中轻微下调,与菠菜和水稻植株一致[24.,25.].
根系长期营养缺乏可能会造成次生胁迫,但尽管数千个基因受到不同的调控,-Zn转录组与锌缺乏的许多生理知识是一致的。玉米中-Zn下紫色酸性磷酸酶基因的上调也在小麦中发现[20.].类似于拟南芥的结果[7],一些结核蛋白样蛋白质编码基因在-ZN中进行下调。然而,这里的一些(无表达)Nodulin家族基因也在-ZN玉米根中高度上调。与Zn缺陷类似拟南芥根源,扩张素基因是深刻的下调[7,与较小的根一致。与文献一致的还有超氧化物歧化酶、过氧化物酶和其他活性氧解毒所需的酶基因的强基因表达缺失[3.].在玉米根系中添加锌可改善过氧化氢浓度升高和超氧化物歧化酶活性不足[26.,但目前尚不清楚为什么在-Zn下的活性氧解毒没有被其他不需要Zn的解毒酶的基因上调所补偿。在一些物种中,在缺锌情况下无法维持铜/锌依赖性超氧化物歧化酶活性,并没有通过其他活性氧途径的上调来补偿[3.,导致根组织中超氧化物和过氧化氢浓度增加。有趣的是,众所周知,锌缺乏的植物在枝条中积累碳水化合物,特别是在源叶的韧皮部液中,但尚不清楚这是由于韧皮部输出蔗糖受损还是由于库需求减少[3.].的发现甜点13.蔗糖通道基因在根中特别上调可能表明植物在根中蔗糖流出的大量投资。然后,这些流出通道也可以参与通常在Zn缺陷下发现的增加的根糖渗出[3.].
我们还注意到倾向于减少参与甲基化维护的基因表达。对延长Zn缺乏的转录反应影响了更多的基因而不是拟南芥,但缺锌反应的关键成分高度相似[7,27.].
Deficiency-adapted methylome
从观察到抑制甲基化维持基因的观察开始,我们询问甲基化是否受到-ZN的影响。以前的分析拟南芥表明根系-Zn的甲基化差异和少量甲基化损失[27.].
相比拟南芥在玉米中,甲基化频率的损失非常大,特别是在CG和CHG环境中。总体甲基化的减少,特别是在CG环境下,伴随着DMRs的大部分低甲基化。97%的CG DMRs和93%的CHG DMRs的-Zn甲基化水平低于对照。单个芽之间轻微的视觉症状差异反映了单个植株的轻微变化的胁迫水平,这反映在(池)复制内的一些变化。甲基化的适应性可能直接与锌的缺乏有关,或者与一般的应激反应有关。在磷酸盐饥饿下的水稻中,DMRs优先与饥饿相关基因和邻近基因te相关[13.].典型锌饥饿调控差异基因表达的发生暗示了玉米对锌的特定适应。此外,甲基化与一些差异表达基因的表达显著相关。
de novo更灵活的chh甲基化通常认为对环境条件的反应比对称chg和cg背景更快,但对于我们的意外,CG背景受到影响最大,而ChH的背景几乎没有甲基化,只有一个可忽略的CHH发现了DMR。这与磷酸盐饥饿的米形成相反[13.,即使放宽了dmr的定义标准。Li et al., 2015也在玉米CHH环境中发现了低甲基化,而在5个不同的玉米自交系(包括本文研究的B73)中发现了很少的DMRs。Eichten等人在2013年也发现了同样的发现,他们还发现在自交系B73和Mo17的CHH环境中甲基化非常低(< 10%)。在拟南芥发现了CHH语境中类似的低甲基化水平(1.7%)(法律和Jacobsen,2011),类似于我们实验中的植物(对照植物中的1.3%)。DNA甲基化差异在-4周后记录,但在几天内可能发生变化,如磷酸盐饥饿的水稻[13.].甲基化在发育和治疗过程中的时间进程将有助于更好地理解这些变化的关系。
DNA甲基化与基因表达的相互作用
基因组宽DNA甲基化对营养应激的变化,并且与基因表达的相关性很少在其他植物中研究比稻米或拟南芥.很明显,通过转座子的超甲基化物质沉默的可转换元素的有害活性(Dowen等,2012年; Slotkin和Martienssen,2007; Tan,2010; Tsaftaris等,2003; Yong-Villalobos等,2015).转座子的甲基化变化经常影响相邻基因的表达(Lisch和Bennetzen,2011; Mirouze和Vitte,2014; Slotkin和Martienssen,2007)。这些机制可能不太重要拟南芥,因为这种植物有很少的转座子和低甲基化。在玉米2.3 Gb的巨大基因组中,充满了TEs,基因和基因调控序列都是远距离分布的(Schnable et al., 2009;Tenaillon et al., 2011)。平行代表性的硫酸氢盐DNA测序和RNA-seq鉴定了玉米根的甲基组和转录组的相关变化,并揭示了甲基化的强烈下降。锌缺乏调节的DEGs的上调和下调比例几乎相同(各约50%),这导致了- zn中许多基因类别的显著抑制,包括活性氧处理和细胞壁类别。我们可以证明,在启动子或基因体中含有DMR的几个基因与基因表达改变相关,而在磷和氮缺乏的情况下,没有发现明显的相关性[22.].然而,转换元件中的差异甲基化对P和N缺乏的附近基因的表达具有显着影响,而没有发现Zn缺乏。然而,由于大玉米基因组和可能参与玉米中的调节基因的基因组DNA的大型基因组DNA,我们可能错过了与更远的区域的相关性。相比之下,通过对比,高甲基化的TES通常接近诱导基因[13.),而在拟南芥TEs的高甲基化经常导致附近基因表达减少[28.,29.,30.].在-N -Zn玉米根,大多数TES是如上甲基化的,但是相似数量的近似基因被上调。在-Zn中,观察到与去甲基化基因或启动子相同,它们的表达不一致或减少。因此,高甲基化对基因表达的潜在一般作用仍然尚不清楚。相反,文学以及我们的结果表明强大的物种和营养特异性甲基化和转录适应。启动子和基因体中的甲基化水平与我们的数据进一步支持基因表达相关,但不可能从这些数据中推断因因果关系。
尽管如下假甲基化通常伴随着转换元素的表达增加,但我们的数据集不支持,尽管我们需要注意到TE表达未明确地专注于。虽然在氮气下和磷缺乏下,TES的总体表达至少适度增加[22.],它们在-Zn中的表达甚至略有减少(减少9%)。
小RNA与DNA甲基化相关,特别是在CHH环境中(甲基化很少),我们注意到-Zn样品中小RNA数量的巨大减少。这可能表明了不同甲基化途径之间的进一步交叉,并暗示玉米中-Zn下甲基化的缺失伴随着小RNA量的减少,尽管RdDM途径中涉及的酶的表达没有发生太大的变化。然而,使用生物分析仪定量和减少小RNA也必须谨慎,因为小RNA水平可能会受到人为因素的影响,如RNA降解和不准确的长度估计。
甲基化,其他应力和潜在功能
通常,基因组不稳定性归因于植物、人类、动物和真菌中的DNA低甲基化[31.,32.,33.].人们还经常认为,对压力的适应是由基因组灵活性和不稳定性的增加以及转座子活性的增加带来的[31.,34.,35.,36.].由于我们的结果表明,由于Zn和玉米氮缺乏症,玉米根中的DNA甲基化造成巨大损失[22.,除了基因表达的直接调控外,染色体重排的增加可能是由去甲基化引起的。目前DNA甲基化与基因表达之间的相关性比较有限[37.而关于这些相关性的结论往往是矛盾的,这巩固了DNA低甲基化还有附加功能的假设。
DNA甲基化在体细胞组织中的功能,例如根或叶片,可以与种系中的那些不同,其中DNA甲基化作为表观遗传机制可能有助于转基因遗传。DNA甲基化水平对于调节TE活性,重组事件的频率以及突变的发生是重要的[31.,34.,38.].如前所述,对于其他胁迫,可控的染色体重排增加以及转座元件活性的增加可能增强对-Zn条件的适应过程[31.,34.].
不仅确定基因组的低甲基化,而且发现局部缺失迁移增加基因的重排。发现如下甲基化基因是抗性基因样基因座[39.].这暗示了植物有能力高度控制由基因组不稳定引起的事件的发生,以“控制”遗传多样性的增加,同时平衡基因组不稳定带来的相关风险[34.].DNA甲基化的强烈整体缺失以及着丝粒和中着丝粒区域的强烈缺失可能与此相关,因为te的活性以及着丝粒区域的交叉通常会受到DNA甲基化的抑制[40].为了避免过量基因组不稳定性的不良反应引起的低甲基化(尤其是Centromere,交叉可能导致染色体破裂或损失[41.]),除DNA甲基化之外的表观因子也可能施加控制以平衡基因组不稳定性。因此,全局脱甲酰基化提供了植物的快速基因表达调节和可塑性的能力,以及在某些位置控制的基因组不稳定性的增加。这种假设是通过重组频率的总体增加来证实[40,但由于低甲基化,这些事件的全球重新分布,表明不同(表观遗传)机制的相互作用。甚至有人认为,植物可能能够在逆境条件下“招募”反转录转座子来促进进化[42.].在玉米氮磷缺乏的情况下,TEs的总体表达确实增加了[22.].TE在-Zn下的表达略有下降,可能是由于应力强度较强或表明应力特异性机制。
此外,暴露于电离辐照胁迫的植物是高甲基化的[35.].作者怀疑这些植物的高甲基化可以防止辐射诱导的基因组不稳定性,但仍然认为突变和重组是植物适应辐射胁迫的贡献机制。同样,受病毒感染的烟草植株的后代中普遍的高甲基化和局部低甲基化增加了重组事件的频率[34.].由于渗透压引起的烟草中也发现高甲基化[38.].然而,冷胁迫诱导玉米根的低甲基化[43.重金属胁迫会导致大麻和三叶草的低甲基化[44.].
尽管只有有限的证据表明,由于DNA甲基化变化导致的基因组稳定性变化的遗传性,一些研究人员报告说,至少在应激暴露的植物的直接后代中,重组增加了[34.,35.,39.].此外,基因组稳定性的变化不仅可以通过减数分裂遗传,也可以通过有丝分裂遗传[34.].由此可以通过营养繁殖来增强遗传多样性,例如,通过体细胞重组事件[45.,提供了增强适应性和可塑性的机会,特别是在植物种群水平上。
为了验证DNA甲基化的强烈损失的结论不仅影响基因表达,而且在通过重组和突变频率的控制增加,还在促进遗传多样性方面发挥作用,因此需要进一步的实验。
DNA甲基化缺失可能会增加植物在胁迫下的整体可塑性和反应性,以及增加基因组的不稳定性。其他表观遗传机制,如组蛋白修饰,可能通过抑制过量的染色体重排来平衡甲基化损失的影响。因此,同时研究DNA甲基化和组蛋白修饰,可以更清楚地了解影响基因表达调控和适应策略的不同因素以及基因组不稳定性的微调。
另一个要测试的方面是这种适应的可遗传性。为了考虑减少人民遗传性,应该研究像花粉一样的生成细胞,用于甲基族变化和重组事件的频率以及F1产生的应激父母(例如胚乳和胚胎)的组织。可以在经常通过植物繁殖中经常传播的植物中进行有丝分裂性。
随着进化驱动力的增加,基因组不稳定性的重要性可能对人口水平尤为重要。因此,通过寻找甲基姆和转录组数据的差异,可以通过寻找甲基姆和转录组数据的差异来比较(野生)植物的遗传多样性,并在调查中,调查如果压力的人群显示出对应激反应重要的新特征或基因的差异,则可以进行研究。此外,应测试应激型,应力强度和物种对重组事件,双链断裂或转移元素活性的影响。
结论
本研究证实了营养缺乏条件下根系DNA甲基化的变化。这与基因表达变化有关,这些变化与营养、物种和潜在的器官特异性相关。这是如何通过加速新的、潜在的有益性状的发展来影响植物的可塑性和作为进化驱动程序的功能的,未来应该进行更详细的测试。不同的营养缺乏(包括宏量和微量营养)导致玉米根系的甲基化损失(尽管程度不同),这一事实激发了一系列进一步实验的想法。这不仅可以促进对群体基因组学的理解,而且有助于进化研究。由于在育种过程中,重组是将新的想要的性状引入植物的重要手段,通过甲基化改变重组和TE活性的植物内部机制可以在育种过程中人工诱导,提高育种过程的速度,但需要的是,生殖系中的甲基化对这些刺激的反应与体细胞组织相似[46.,47.].
材料和方法
植物生长和取样
自交系B73的种子Zea Mays.(来自植物育种部的Schipprack博士,Hohenheim大学)首先用10%H表面灭菌2O.2溶液冲洗2分钟,去除H2O.2通过在蒸馏水中洗涤种子。对于24小时,将种子孵育在10mm Caso中4然后放入泡沫板之间浸泡在3mm CaSO4溶液浸泡4天使其萌发。然后,将它们放入2.8 L含0.1 mM K的营养液中2所以40.12 mM MgCl2,0.5 mm ca(没有3.)2和20 μM KH2阿宝4,0.2μmh3.博3., 0.1 μM MnSO4, 0.1 μM ZnSO4, 0.04 μM CuSO42 nM (NH4)6莫7O.24..在3天后,在改进的Zn缺陷型溶液上施加全面供应的玉米水培溶液(2.8L)和-ZN植物。对照植物的溶液含有0.5毫米K.2所以4, 0.6 mM氯化镁2, 2.5 mM Ca(NO3.)2和0.1毫米kh2阿宝4.后者在第3周提高到0.2 mM,第4周提高到0.5 mM。1 μM H3.博3.,0.5μmmnso4,0.5μmznso4, 0.2 μM CuSO4, 0.01 μm (nh .4)6莫7O.24.添加100 μM Fe-Sequestrene。在第一次营养液变化时将其提高到200 μM,在第二次营养液变化时保持在300 μM。缺锌植株也进行相同处理,除了以下修改:KH的数量2阿宝4,但保持在0.1 mM。当Fe-Sequestrene中含有微量Zn时,加入300 μM的Fe-EDTA代替Fe-Sequestrene。溶液中没有锌,但在营养液中总能检测到μ m范围内的锌残留,可能是由于植物生长的塑料容器造成的。在第3周和第4周,0.1 μM ZnSO4给予溶液24小时以防止植物的染色。一周后营养溶液一次交换一次,然后每3天交换一次。
这些植物在气候室中生长,可以控制生长条件。在25℃条件下,昼长16 h,夜长20℃条件下,夜长8 h。光合活性光子通量密度(PFD)为400 μmol m- 2年代- 1并且湿度保持在60-80%。对于每种条件6种植植物,每壶2种植植物。5周(对应于4周的处理),收获根和芽材料,从而合并一个罐中的两种植物,导致控制和-ZN的3种重复(每次重复2株植物)。
营养分析
叶子样品在被微波炉消化之前被磨成细粉。测定锌、铁、磷和氮的浓度。测量后三种物质以确定-Zn样品的营养特异性。锌和铁通过电感耦合等离子体质谱(ICP-MS)测定,磷通过添加钼钒酸盐试剂后的正磷酸盐通过紫外-可见光谱法测定,氮通过凯氏定氮(Kjeldahl)测定[48.].
总RNA样本的RNA测序和转录组分析
根据制造商的手册,通过Analytikjena Innuprep植物RNA试剂盒从对照和-ZN植物的根材料中提取总RNA。用Thermo Scientific Nanodrop 2000c分光光度计和Agilent 2100生物分析仪检查RNA的数量和质量。所有RNA样品均具有OD260 /280≥1.8的纯度。在北京基因组学院(中国BGI)制作了Truseq 160 Basepair(BP)短插入库和100bp配对结束排序。通过FASTQC工具版本0.11.5(Babraham BioInformatics)检查清洁数据,并且读取与Zea Mays.由MaizeGDB(玉米遗传学和基因组学数据库)提供的参考基因组(AGPV3)[49.],选项有——phred64、-dta-袖扣、-no-mixed和-no-discordant。使用了AGPv3注释文件[49.].装配使用工具版本2.2.1的袖扣套件中的袖扣[50]带默认选项和--gtf-guide和 - 无效长度校正。将组件与绒毛胶合并(具有-g和-s选项)后,使用cuffdiff来查找--compatible-hite-norm,-b,-u和否则默认选项来找到差异表达的基因(DEG)。转录量为每百万映射读数(FPKM)的转录物每千碱基的片段给出。根据日志给出条件之间的差异2-fold以log的形式变化2(FPKM施锌/ FPKM.控制).差异表达基因的阈值(在多次检测的Benjamini-Hochberg校正后)设置为P. < 0.05. Overrepresentation of differentially expressed gene categories was determined by a Parametric Analysis of Gene Set Enrichment (PAGE) using the reference genomeZea Mays.AGPv3.30和Agrigo分析工具包版本1.2 [51].使用具有不同表达(P <0.05)的Bonferroni多测试调整值。
还原亚硫酸氢盐测序DNA样本和甲基组分析
在收获玉米根后,将材料研磨成细粉,根据手册通过Qiagen DNeasy植物迷你试剂盒提取DNA。通过Thermo Scientific Nanodrop 2000c分光光度计和Qubit荧光定量检查DNA样品的数量和质量。仅使用具有OD260 / 280更大或等于1.8的样品进行进一步处理。用MSPI消化DNA,然后选择40至220bp之间的片段。这些片段用于构建偏硫酸氢盐测序文库的减少的表示。Illumina Hiseq2000对北京基因组学院(BGI,中国)进行了100辆BP配对结束序列。使用FASTQC版本0.11.5(Babraham Bioinformatics)进行清洁数据的质量检查。为了进一步提高质量,Hannon Lab的FastX-Toolkit版本0.0.13用于切断所有读取的前4个和第6页。通过BS-Seeker2版本2.0.10完成映射[52)使用Zea Mays.参考基因组AGPv3。BS-Seeker2实际上用MspI和所需长度的大小选择序列切割这个基因组。使用的参数是-l 20,−u 400,−-aligner = bowtie2和其他默认选项。通过MspI虚拟消化参考玉米基因组,然后对40 - 220 bp长度的DNA片段进行大小选择,确定了理论上覆盖的基因组片段(14%)。虚拟简化代表基因组的尺寸选择范围(即20-400 bp长度)比已消化DNA样本的尺寸选择范围更广,以解释在消化DNA样本的尺寸选择过程中的不准确性。bowtie2作为短读映射器和默认设置用于对齐和甲基化调用[53].DMRCALLER版本1.2.0 [54在R版本3.2.4中,通过汇集来自所有三个重复的甲基化水平信息来确定差异甲基化区域(DMRs)。平滑是通过带三角核的噪声滤波器完成的[55]用于计算差异甲基化的胞嘧啶。DMRS的特征在50和500bp之间,含有至少4个胞嘧啶(每种胞嘧啶的至少4次读数),与对照和a相比,至少40%的甲基化差异为至少40%P.-Value <0.01。位于基因内的DMRS用来自AGPv3中的玉米注释文件所取的基因信息测定[49.].在基因上游包含2000bp的区域被定义为基因启动子。OrunédeChercheGénomiqueInfo的转算元素(TES)注释[56使用了)。
将转录组数据与甲基化信息相关联
通过R版本3.3.3中的2 × 2列联表Fisher精确检验,研究了基因、启动子或te的差异甲基化与基因表达之间的相关性。为了避免假阴性结果,只有被减少代表性基因组覆盖的至少500 bp的基因被考虑到差异甲基化基因、启动子及其基因表达的量化。更遥远(最近)的te被考虑,如果它们被简化的代表基因组覆盖,以避免非常短的转座元件的损失。BEDTools版本2.26.0 [57]用来确定与每个转座子最近的基因,而不管它是上游还是下游。
量化小RNA
从用于总RNA和DNA萃取的相同根材料中提取小RNA。萃取是根据Analytikjena Innuprep Micro RNA试剂仪手册进行的。在Thermo Scientific Nanodrop 2000c分光光度计中测试样品,纯度,仅使用具有OD260 /280≥2.0的样品。根据Agilent Technologies根据Agilent小RNA试剂盒指南,在Agilent 2100生物分析仪中测定小RNA的量。测定SRNA(15-30nt长度)的量与RNA的总量之间的比率。由于21-24 NT SRNA参与RDDM途径,选择15-30ntSRNA范围以确保考虑所有21-24 NT SRNA。通过R版本3.1.1中的单向ANOVA确定SRNA量的显着变化。
缩写
- 度:
-
差异表达基因
- DMR:
-
差异甲基化区域
- RdDM:
-
RNA依赖的DNA甲基化
- rrb:
-
还原硫酸氢盐排序
- te:
-
可转换元素
- 锌:
-
锌
参考
- 1。
Broadley MR, White PJ, Hammond JP, Zelko I, Lux A.锌工厂。新植醇。2007;173:677 - 702。
- 2。
Assungao Ag,Herrero E,林YF,Huettel B,Talukdar S,Smaczniak C等人。拟南芥转录因子BZIP19和BZIP23调节适应缺锌。Proc Natl Acad Sci U S A. 2010; 107:10296-301。
- 3.
Cakmak I.锌可能在保护植物细胞免受反应性氧气损伤中的可能作用。新植物。2000; 146:185-205。
- 4.
马峰,夏建军,马建军,马建军,等。磷型重金属ATPase OsHMA2介导的锌向水稻发育组织的优先转运。植物杂志。2013;162:927-39。
- 5.
尼尔森跳频。锌在农业中的应用历史。难以减轻。2012;3:783-9。
- 6.
Mattiello EM, Ruiz HA, Neves JC, Ventrella MC, Araujo WL。锌缺乏影响玉米叶片的生理和解剖特性。acta botanica yunnanica(云南植物研究). 2015;
- 7.
van de Mortel JE, Almar Villanueva L, Schat H, Kwekkeboom J, Coughlan S, Moerland PD, et al.;铁和锌平衡、胁迫反应和木质素生物合成基因的表达差异很大,区分了不同的根拟南芥和相关的金属高褶皱菥caerulescens.植物杂志。2006;142:1127-47。
- 8。
GROTZ N,FOX T,CONNOLLY E,PARK W,GueLinot ML,EIDE D.鉴定锌转运蛋白基因的鉴定拟南芥对缺锌有反应美国国家科学学院学报1998;95:720 - 4。
- 9。
黄李,周X, Y,朱L,张年代,赵Y, et al。玉米锌调控转运体、铁调控转运体样蛋白(ZIP)基因家族的鉴定与特性分析。BMC Plant Biol. 2013;13:114。
- 10.
Khatun MA,Hossain MM,Bari MA,Abdullahil Km,Parvez MS,Alam MF等。玉米缺乏耐受性与Zn转运蛋白基因的上调和抗氧化活性有关。植物BIOL(斯图特)。2018; 20:765-70。
- 11.
mochegiani E, Romeo J, Malavolta M, Costarelli L, Giacconi R, Diaz LE等。锌:膳食摄入量和补充锌对老年人免疫功能的影响。年龄(Dordr)。2013; 35:839-60。
- 12.
普拉萨德。锌对人体健康的影响:锌对免疫细胞的影响。摩尔医学。2008;14:353-7。
- 13。
王春华,王春华,王春华,等。应激诱导的基因表达驱动邻近重复元件的DNA瞬时甲基化变化。elife。2015; 4: e09343。
- 14。
Gonzalez-Morales SI, Wrobel K, Gutierrez-Alanis D, Cervantes-Perez SA, Hayano-Kanashiro C等。甲基组分析揭示了表观遗传变化在调控的重要作用拟南芥反应磷酸盐饥饿。Proc Natl Acad Sci U S A. 2015; 112:E7293-302。
- 15.
Lewsey MG, Hardcastle TJ, Melnyk CW, Molnar A, Valli A, Urich MA等。移动小rna调节全基因组DNA甲基化。中国科学院研究生院学报2016;113:E801-10。
- 16.
陈晓东,陈晓东。植物microrna的形成、转化和作用机制。植物细胞。2013;25:2383 - 99。
- 17.
吴L,周H,张Q,张j,ni f,liu c等。DNA甲基化由MicroRNA途径介导。Mol细胞。2010; 38:465-75。
- 18.
植物的表观遗传调控。冷泉Harb perspective Biol. 2014;6:a019315。
- 19。
引用本文:李志刚,李志刚,李志刚。植物中甲基组的进化。基因组医学杂志。2016;17:264。
- 20.
Bharti K,Pandey N,Shankhdhar D,Srivastava PC,Shankhdhar SC。不同锌水平对超氧化物歧化酶和酸性磷酸酶活性的影响和小麦基因型的有机酸渗出。physiolmol biol植物。2014; 20:41-8。
- 21.
杨建平,杨建平,杨建平,等。玉米zmsweet13a,b,c蔗糖转运体三敲除突变体的韧皮部负荷受损。新植醇。2018;218:594 - 603。
- 22.
在缺氮的玉米根系中DNA甲基化的大量缺失与基因表达差异的相关性较差,而在缺磷的玉米根系中DNA甲基化的大量缺失与基因表达差异的相关性较差。植物学报。2018;9:497。
- 23。
Wolfgruber TK,Sharma A,Schneider KL,Albert PS,Koo DH,Shi J等。玉米Centromere结构和演化:焦粒子2和5的序列分析显示主要由回收器塑造的动态基因座。Plos Genet。2009; 5:E1000743。
- 24。
宽广le米,棕色P,Cakmak I,Rengel Z,Zhao F.营养素的功能:微量营养素。在:Marschner的高等植物的矿物营养。3埃德;2011. p。212-22。
- 25。
Randall PJ,Bouma D.缺锌,碳酸酐和菠菜叶片的光合作用。植物理性。1973年; 52:229-32。
- 26。
Moradtalab N,Weinmann M,Walker F,Hoglinger B,Ludewig U,Neumann G.Silicon在玉米的早期生长期间通过对微量营养素稳态和激素余额的影响来提高了耐寒性。前植物SCI。2018; 9:420。
- 27.
Chen X,Schonberger B,Menz J,Ludewig U. DNA甲基化和基因表达在拟南芥缺乏症下的可塑性。植物细胞生理。2018; 59:1790-802。
- 28.
艾哈迈德I,Sarazin A,Bowler C,Colot V,Quesneville H.基因组的局部DNA甲基化从拟南芥中的小RNA靶向序列扩散。核酸RES。2011; 39:6919-31。
- 29.
Eichten SR,Ellis Na,Makarevitch I,Yeh CT,Gent Ji,Guo L等。异铬胺的扩散仅限于特定的玉米回析碳松的家族。Plos Genet。2012; 8:E1003127。
- 30.
霍利斯特·JD,高特·BS。转座元件的表观遗传沉默:减少转座和对邻近基因表达的有害影响之间的权衡。基因组研究》2009;19:1419-28。
- 31.
Iwasaki M,Paszkowski J.植物中的表观遗忘。embo J. 2014; 33:1987-98。
- 32.
陈志强,王志强,王志强,等。拟南芥反转录转位的选择性表观遗传控制。大自然。2009;461:427-30。
- 33.
艾尔·普蒂里,罗伯森·杜兰特。表观遗传机制和基因组稳定性。表观遗传学。2011;2:299 - 314。
- 34.
Boyko A,Kathiria P,Zemp Fj,Yao Y,Pogribny I,Kovalchuk I.转基因感染植物基因组稳定性和甲基化的转基因变化:(病毒诱导的植物基因组不稳定性)。核酸RES。2007; 35:1714-25。
- 35。
Kovalchuk I,Abramov V,Pogribny I,Kovalchuk O.植物适应于切尔诺贝利区的生命的分子方面。植物理性。2004; 135:357-63。
- 36。
Molinier J,Ries G,Zipfel C,Hohn B.在植物中应力的转换记忆。自然。2006; 442:1046-9。
- 37。
Roessler K, Takuno S, Gaut BS。短柄草叶片和花芽组织的CG甲基化与差异基因表达的协方差。《公共科学图书馆•综合》。2016;11:e0150002。
- 38。
Labra M, Ghiani A, Citterio S, Sgorbati S, Sala F, Vannini C,等。豌豆根尖对水分亏缺的胞嘧啶甲基化模式分析。植物医学杂志。2002;4:694-9。
- 39.
Boyko A,Blevins T,Yao Y,Golubov A,Bilichak A,Ilnytskyyy Y等。拟南芥对应激的转基因适应需要DNA甲基化和Dicer样蛋白的功能。Plos一个。2010; 5:E9514。
- 40.
陈志强,李志强,李志强,等。DNA甲基化缺失影响拟南芥的重组景观。中国科学院院刊2012;109:5880-5。
- 41.
Talbert PB,Henikoff S. Centromeres转换但不要交叉。Plos Biol。2010; 8:E1000326。
- 42.
Mirouze M,Vitte C.转置元素,一个用于翻译表观遗传变异的宝库:拟南芥和作物表观症的见解。J Exp Bot。2014; 65:2801-12。
- 43.
STEWARD N,ITO M,YAMAGUCHI Y,Koizumi N,SANO H.通过环境胁迫的玉米核肉和去甲基化的周期性DNA甲基化。J Biol Chem。2002; 277:37741-6。
- 44.
Aina R, Sgorbati S, Santagostino A, Labra M, Ghiani A, Citterio S.重金属诱导白三叶草和工业大麻DNA特异性低甲基化。杂志。2004;121:472 - 80。
- 45.
Boyko A,Kovalchuk I.基因组不稳定性和表观遗传修改 - 对环境压力的遗传响应?CurrOp植物BIOL。2011; 14:260-6。
- 46.
应激的表观遗传反应:三重防御?植物生态学报。2012;15:568-73。
- 47。
潘Q,邓米,闫杰,李L。玉米复合重组不均匀性遗传机制的复杂性。SCI REP。2017; 7:1205。
- 48。
Kjeldahl J. Neue Methode Zur Bestimmung des Stickstoffs在OrganischenKörpern。j分析化学。1883; 22:366-82。
- 49。
Sen Tz,Andorf Cm,Schaeffer ML,Harper LC,火花我,Duvick J等人。MaizeGdb成为以“以依据为中心”。数据库。2009; BAP020:1-9。
- 50.
特拉普奈尔C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ,等。RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28:511-5。
- 51.
杜志华,周旭,凌云,张志华,苏志华。一种面向农业社区的GO分析工具。核酸学报2010;38:W64-70。
- 52.
郭伟,孙旭,张明强,等。BS-Seeker2:亚硫酸氢盐测序数据的通用对齐管道。BMC基因组学。2013;14:774。
- 53.
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。
- 54.
差异甲基化区域调用。R包版本1.6.0。2015.Bioconductor:www.biocondion.org..
- 55.
胡慧敏,胡慧敏。在靶向亚硫酸氢盐测序数据中检测显著差异的甲基化区域。生物信息学。2013;29:1647-53。
- 56.
Jamilloux V, Daron J, Choulet F, Quesneville H. De novo注释转座元件:解决脂肪基因组问题。Proc IEEE。2017; 105:474 - 81。
- 57.
Quinlan AR,Hall Im。Bedtools:灵活的公用设施套件,用于比较基因组特征。生物信息学。2010; 26:841-2。
确认
我们感谢A. Melchinger教授,斯图加特大学植物育种系植物育种部·斯皮帕克博士,为B73种子提供。升值也涉及斯图加特Hohenheim大学营养作物生理学系的工作人员。这项工作得到了Baden-Wuerttemberg(AZ:7533-30-20 / 1)的科学,研究和艺术部的支持。
资金
我们感谢科学和艺术部Baden-Würtemberg在Regio-Researd-Alliance“在动态环境中的产生稳定性”进行资金。资金机构没有参与研究和收集,分析,数据的解释和写作稿件的设计。
可用性数据和材料
这些原始数据已提交给NCBI,可通过以下附录获得:SAMN09464306。
作者信息
从属关系
贡献
SM和UL促成了研究的概念和设计。SM执行了实验和统计分析,并写了稿件的第一个草稿。UL写了稿件的部分。BS有助于数据分析。所有作者均致力于手稿修订,阅读并批准提交的版本。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
同意了。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
额外的文件
额外的文件1:
表S1。rna测序比对率。-Zn的对齐率较低。值在副本之间取平均值。图S1。胞嘧啶在CG, CHG和CHH环境中的覆盖率。在-Zn下观察到较高的胞嘧啶盖度。表S2。RRBS库的对齐输出。RRBS库的控件和-Zn相似的映射能力。数值在重复中取平均值,并以百万表示。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
玉米缺锌根的转录组及其与DNA甲基化损失的关系。BMC植物杂志18,372(2018)。https://doi.org/10.1186/s12870-018-1603-z.
收到了:
接受:
发表:
关键字
- 锌
- DNA甲基化
- 玉米
- 基因表达
- 基因组稳定性
- 转座的元素