- 研究文章gydF4y2Ba
- 开放访问gydF4y2Ba
- 发表:gydF4y2Ba
在Avena(Poaceae)中的重复DNA景观:通过全基因组序列的主要重复类别定义染色体和基因组进化读数gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba19.gydF4y2Ba, 文章编号:gydF4y2Ba226gydF4y2Ba(gydF4y2Ba2019年gydF4y2Ba)gydF4y2Ba
抽象的gydF4y2Ba
背景gydF4y2Ba
重复的DNA主题 - 没有编码遗传信息并重复数百万到数百次 - 构成许多基因组的大多数。在这里,我们确定燕麦中所有重复DNA家族的性质,丰度和组织(gydF4y2BaAvena Sativa.gydF4y2Ba, 2gydF4y2BanGydF4y2Ba = 6xGydF4y2Ba = 42, AACCDD), a recognized health-food, and its wild relatives.
结果gydF4y2Ba
全基因组测序,然后是K-MER和RepectExplorer图的聚类分析,其在共同燕麦和其野生亲属基因组中的重复性DNA组合物的评估。开发了荧光原位杂交(鱼类)的基于核型以了解常见燕麦的染色体和重复序列演化。我们表明,大约200个重复的DNA主题占70%gydF4y2Ba燕麦属gydF4y2Ba基因组,少于20个家庭,占总数的20%。RetroElements代表主要组成部分,TY3 /吉普赛元素代表了所有DNA的40%以上,比TY1 / Copia元素更多的近三倍。DNA转座子约占总量的5%,而Tandemly重复,卫星DNA序列适合55个家族,并且代表约2%的基因组。这gydF4y2Ba燕麦属gydF4y2Ba物种是单系的,但无论是对不同基因组重复序列的生物信息学比较,还是对来自六倍体物种中期染色体的原位杂交,都表明一些重复家族对单个基因组或A和D基因组是特异的。值得注意的是,许多染色体的末端区域显示出不同于其他染色体的重复家族,这表明基因组之间存在易位。gydF4y2Ba
结论gydF4y2Ba
家庭相对较少的重复显示进化限制他们的本性和放大,机制导致同质化,而重复特征是有用的在未来提供基因组标记和协助这个大基因组的组装在二倍体(c。4100 Mb)。基因组间易位的频率表明,利用二倍体燕麦的遗传变异改良六倍体的最佳策略可能与广泛用于面包小麦的策略不同。gydF4y2Ba
背景gydF4y2Ba
基因组进化涉及多个过程,包括全基因组复制(WGDs或多倍体)、片段基因组缺失或复制、染色体重组(融合、裂变、易位和倒转)、基因和重复序列的放大或丢失以及DNA突变[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba].重建真菌的祖先基因组有越来越多的兴趣[gydF4y2Ba3.gydF4y2Ba], 动物 [gydF4y2Ba4gydF4y2Ba植物[gydF4y2Ba5gydF4y2Ba,揭示了基因组进化和多样化的原则,从而导致物种形成和适应。gydF4y2Ba
重复DNA是植物基因组的重要组成部分,通常在25 - 85%之间,可被称为重复组。重复基序在序列和分散模式上差异很大[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba].发现了几组重复性元素:核糖体DNA(RDNA)[45s(18秒-50s-26s)和5S rdNA,具有基因间隔物],端粒重复,I类转回(通过RNA中间体扩增),II类DNA转座子(通过DNA拷贝扩增),并串联重复(通过滑动复制产生/修饰,交叉过渡或滚动圆扩增)[gydF4y2Ba7gydF4y2Ba].它们的存在和相似性、拷贝数和序列的变化对基因组组装和基因注释构成了重大挑战[gydF4y2Ba9gydF4y2Ba].已经假定了重复DNA在基因组中具有多种作用,包括基因组稳定性,重组,染色质调节和基因表达的改性[gydF4y2Ba7gydF4y2Ba].重复序列中的拷贝数变异占人类基因组的5%至10%,对疾病和种群变异非常重要[gydF4y2Ba10.gydF4y2Ba].gydF4y2Ba
在2010年之前的几十年里,重复性知识主要来自于DNA退火实验、随机克隆筛选、限制性片段分析或引物对保守元素的放大。现在全基因组散弹枪测序方法可以用于全基因组的无偏重复分析[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba12.gydF4y2Ba,gydF4y2Ba13.gydF4y2Ba].K-MER分析计数全基因组序列长度读取的k-基座的数量[gydF4y2Ba14.gydF4y2Ba],在不使用参考基因组的情况下识别丰富的图案。基于图形的聚类分析(例如RepectExplorer [gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba15.gydF4y2Ba)是识别和分类原始读重复的另一种方法。这两种方法都是从头识别策略,其结果可用于重复识别或蛋白结构域搜索。由于基因组的多个位置和组装的困难,原位杂交染色体制备是确定基因组位置和重复基序特异性的关键[gydF4y2Ba16.gydF4y2Ba].这些方法已被用于量化香蕉,萝卜,大豆和烟草的基因组重复景观[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21.gydF4y2Ba].gydF4y2Ba
常见的燕麦(gydF4y2BaAvena Sativa.gydF4y2BaL.,2gydF4y2BanGydF4y2Ba = 6xGydF4y2Ba= 42, AACCDD)为温带作物(2017年年产量2300万吨;gydF4y2Bahttp://faostat.fao.orggydF4y2Ba)由于燕麦葡萄糖,批准的健康索赔作为“超级食物”,这有助于降低血液胆固醇水平和心脏病风险[gydF4y2Ba22.gydF4y2Ba,gydF4y2Ba23.gydF4y2Ba].普通燕麦的基因组资源开发,对繁殖和改进重要,落后于其他主要作物[gydF4y2Ba24.gydF4y2Ba,gydF4y2Ba25.gydF4y2Ba].有一些二倍体和六倍体物种的遗传图谱[gydF4y2Ba26.gydF4y2Ba,gydF4y2Ba27.gydF4y2Ba]但没有六倍倍增作物的基因组序列草案(6gydF4y2BaxGydF4y2Ba基因组大小12,600 Mb/1C) [gydF4y2Ba28.gydF4y2Ba]或其二倍体亲属。OAT基因组含有许多重复的家族,并且明显频繁的染色体易位[gydF4y2Ba29.gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba].在最近的系统发育分析中,推测普通燕麦经历了古老的异源四倍体和最近的allohexapploidy事件,涉及C-, A-和d -基因组祖先[gydF4y2Ba31.gydF4y2Ba,gydF4y2Ba32.gydF4y2Ba,而基因组重组掩盖了不同的候选母体a基因组祖先的贡献(双父基因组定义参考[gydF4y2Ba32.gydF4y2Ba])。gydF4y2Ba
在这里,我们旨在阐明二倍体和六倍体燕麦中所有主要重复DNA课程的结构,组织和关系,检查它们的染色体位置,并了解对基因组和染色体演化中的重复的意义gydF4y2Ba燕麦属gydF4y2Ba在基因组、生物信息学和细胞遗传学证据的背景下。重复dna的完整图像为该属的进化和物种形成过程中发生的事件提供了新的证据,包括杂交和染色体易位事件。gydF4y2Ba
结果gydF4y2Ba
基于图的聚类和重复组合gydF4y2Ba燕麦属gydF4y2Ba
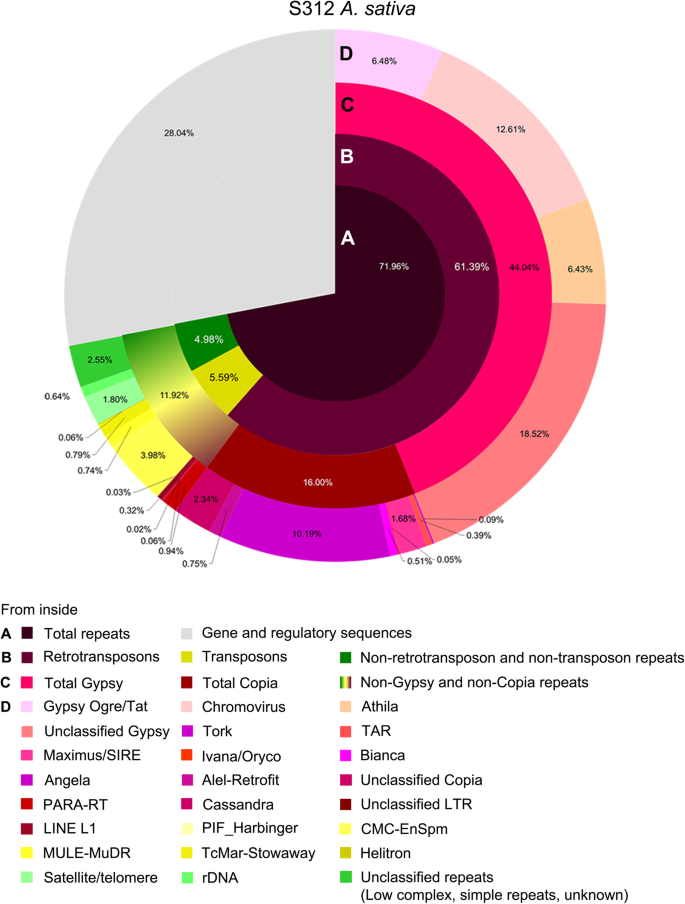
原始序列读取(Illumina 250 BP配对端)gydF4y2BaAvena Sativa.gydF4y2Ba,gydF4y2BaA. Brevis.gydF4y2Ba,gydF4y2Ba答:hirtulagydF4y2Ba,gydF4y2Ba答:strigosagydF4y2BaGC(鸟嘌呤-胞嘧啶)含量平均为43.23%(附加文件)gydF4y2Ba1gydF4y2Ba:图S1,附加文件gydF4y2Ba13.gydF4y2Ba:表S1,附加文件gydF4y2Ba14.gydF4y2Ba:表S2a)。对于基于图的读集群,使用RepeatExplorer分析了1.72到2.87 Gb的子集[gydF4y2Ba11.gydF4y2Ba](附加文件gydF4y2Ba14.gydF4y2Ba:表开通)。总的来说,超过70%的读被分配到200个高度相关序列读的图集群中(附加文件gydF4y2Ba2gydF4y2Ba:图S2,表gydF4y2Ba1gydF4y2Ba),12到18个群集(取决于物种),代表所有读数的超过1%(附加文件gydF4y2Ba15.gydF4y2Ba:表S3b)。gydF4y2Ba
通过检查域同源性或卫星图案进行手动验证(附加文件gydF4y2Ba16.gydF4y2Ba:表S4),65%gydF4y2Ba燕麦属gydF4y2Ba基因组读数与转换元素[TES,包括I类转回(平均60.80%)和II类DNA转座(平均5.78%)]和串联重复[卫星DNA(SATDNA),RDNA和端粒体;平均2.70%](表gydF4y2Ba1gydF4y2Ba,附加文件gydF4y2Ba17.gydF4y2Ba:表S5 [gydF4y2Ba33gydF4y2Ba])。长终端重复(LTR)Rodotransposons在I类Rodrotransposon中占主导地位,具有Ty3-gypsy(Metaviridae;平均42.8%;基因令逆转转录酶-RNAseh-整合酶,RT-RH-int)和Ty1-Copia(Pseudoviridae;平均为16.6%;基因订单Int-RT-RH)超家庭,而非LTR回收碳膜谐波,包括长的间隙核元素(线)表示小于0.5%。II类DNA转座子,包括Helitrons,包括高达5.2%的增强子抑制因子(ENSPM)元件。四个中没有显着的重复性DNA组成差异gydF4y2Ba燕麦属gydF4y2Ba基因组(附加文件gydF4y2Ba17.gydF4y2Ba:表S5)。聚类分析将solo-LTRs和SINEs与亲本元素进行分组;聚类图显示,与编码序列相比,LTRs的丰度更大。gydF4y2Ba1gydF4y2BaH)。爆炸结果鉴定少于20个叶绿体序列簇(丰度排名46-168,从进一步分析中除去),以及大约11个RDNA簇(附加文件gydF4y2Ba16.gydF4y2Ba:表S4)。只有少量比例(2.44-4.49%)群落未分类(附加档案gydF4y2Ba17.gydF4y2Ba:表S5),表现出与特征序列的很少相似,但有些有富含腺嘌呤胸腺嘧啶的域名(附加文件gydF4y2Ba18.gydF4y2Ba:表S6 [gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba])。我们的分析不是设计用于鉴定大多数微卫星阵列(包括端粒序列),通常短于10架。gydF4y2Ba
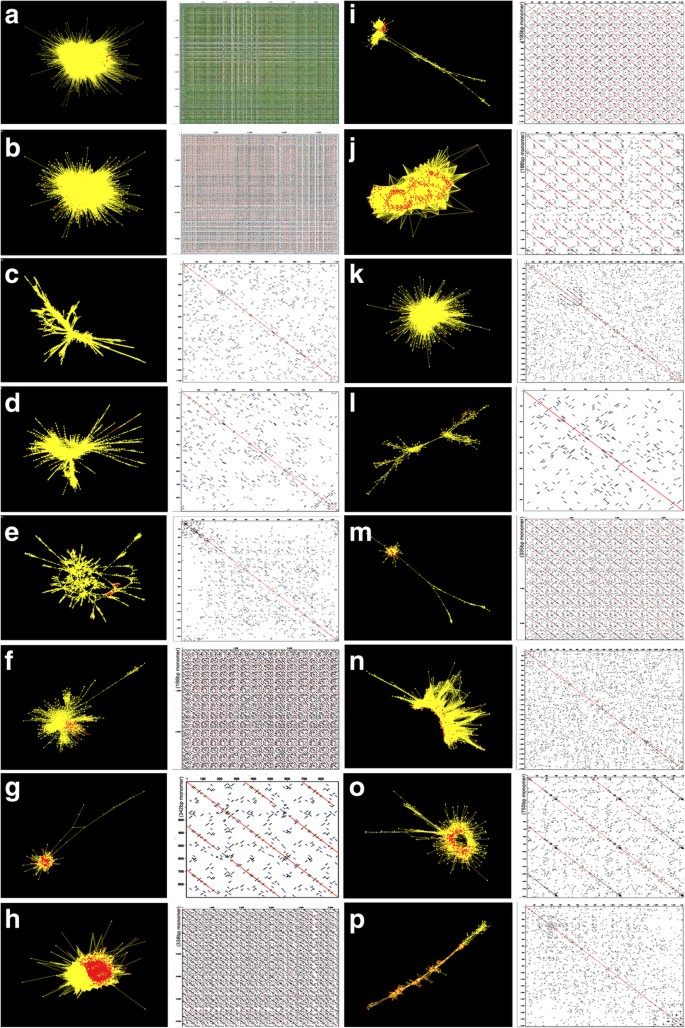
簇显示特征图形图案(图。gydF4y2Ba2gydF4y2BaA-p),我们用来分类重复作案的家庭。例如,0.15%的reads形成了串联重复的卫星DNA As- t119的星形簇图(图2)。gydF4y2Ba2gydF4y2Bah), 0.77%的reads形成射线状逆转录转座子重复Ah-R31(图。gydF4y2Ba2gydF4y2Ban),0.04%的读取形成圆形重复AB-T159(图。gydF4y2Ba2gydF4y2Bao), 0.02%的读取成型线形简单重复Ast-R176(附加文件gydF4y2Ba19.gydF4y2Ba:表S7)。用于原位杂交的8个片段的单体大小为335-360 bp(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a [gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba])。312Cl151C2(没有指定的PCR产品,附加文件20:表S8B)是独特的,因为它显示出由两个密切相关的116bp单体组成的232bp二聚体的高阶重复结构。gydF4y2Ba
k-mer分析gydF4y2Ba燕麦属gydF4y2Ba
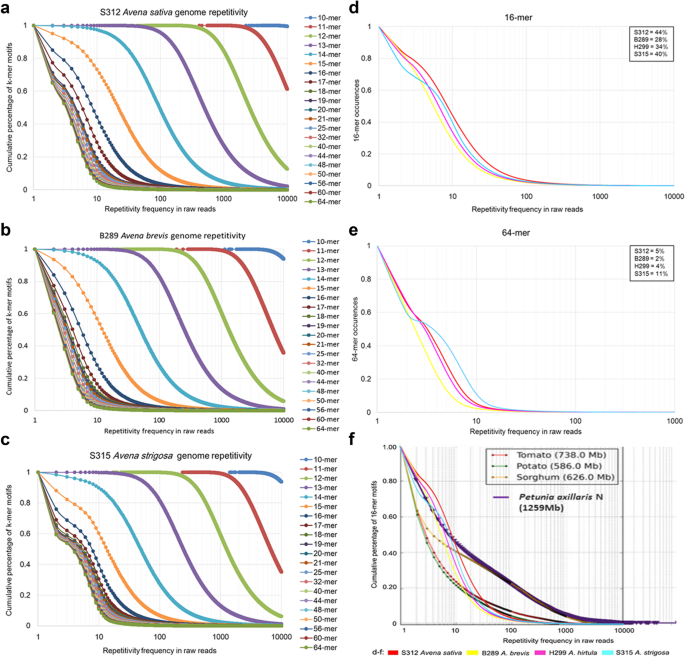
对于10- 64-mers的累积重复频率图,坡度越陡表示累积百分比变化越快,短k-mers (10- 17-mers)的累积百分比变化相对平缓,而长k-mers (18- 64-mers)的累积重复频率图坡度逐渐增大(图2)。gydF4y2Ba2gydF4y2Baa - c)。在相同的重复频率下,较短的k-mer基序具有较高的累积百分比(并且在原始阅读中具有较高的频率;无花果。gydF4y2Ba3.gydF4y2Baa - c)。在四个gydF4y2Ba燕麦属gydF4y2Ba物种,16-MEL主题发生≥10倍占44%gydF4y2BaA.苜蓿gydF4y2Ba基因组,略高于其他物种(28%gydF4y2BaA. Brevis.gydF4y2Ba, 34%的gydF4y2Ba答:hirtulagydF4y2Ba,40%gydF4y2Ba答:strigosagydF4y2Ba;无花果。gydF4y2Ba3.gydF4y2Bad)。64-MEL主题发生≥10倍占11%gydF4y2Ba答:strigosagydF4y2Ba基因组(图。gydF4y2Ba3.gydF4y2Bae)高于其他物种(5%gydF4y2BaA.苜蓿gydF4y2Ba, 2%的gydF4y2BaA. Brevis.gydF4y2Ba, 4%gydF4y2Ba答:hirtulagydF4y2Ba)基因组。总的来说,图表与重复氧化剂分析一致,其中一组非常丰富的序列表示约束的基因组(例如拐点。16-MER图。gydF4y2Ba3.gydF4y2BaD)和其他丰富的序列约占70%,在图形中较平坦的区域,每个基因组的基序所代表的次数少于10次。对于发生≥10次的16个基序,普通燕麦的累积百分率几乎相当于gydF4y2BaPetunia arillaris.gydF4y2Ba,紧随其后的是gydF4y2Ba答:strigosagydF4y2Ba,高粱,gydF4y2Ba答:hirtulagydF4y2Ba,gydF4y2BaA. Brevis.gydF4y2Ba,番茄和土豆(图。gydF4y2Ba3.gydF4y2BaF [gydF4y2Ba21.gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba]);值得注意的是,发生≥1000次的16-mer基序的累积百分比收敛为gydF4y2Ba燕麦属gydF4y2Ba品种,番茄和马铃薯。gydF4y2Ba
高度重复基序的染色体定位和基因组特异性gydF4y2Ba
用作鱼探针的重复片段gydF4y2Ba
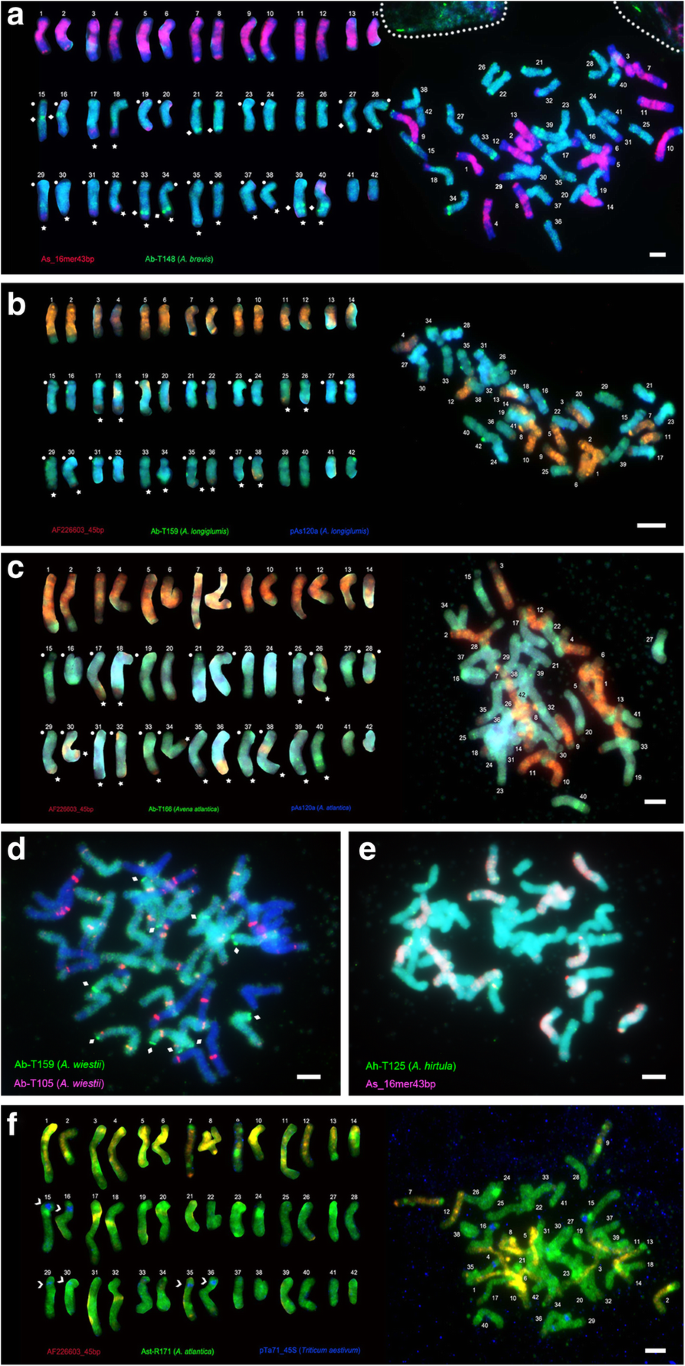
本地化重复gydF4y2BaAvena Sativa.gydF4y2Ba染色体(无花果。gydF4y2Ba4gydF4y2Ba和gydF4y2Ba5gydF4y2Ba,其他文件gydF4y2Ba3.gydF4y2Ba-gydF4y2Ba10.gydF4y2Ba图S3-S10),从k-mer和RepeatExplorer分析鉴定的代表性序列中设计了25个探针用于原位杂交,包括9个卫星,1个DNA转座子,4个LTR-Gypsy反转录元件和11个非机密序列(附加文件)gydF4y2Ba19.gydF4y2Ba:表S7)。除AF226603_45BP和PAS120A外[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba[我们在PCR或克隆策略中选择了几乎没有同源性的序列或没有同源性的序列(TES或串联重复)[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba42gydF4y2Ba].45s和5s rdna [gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba]用于识别某些染色体(附加文件gydF4y2Ba18.gydF4y2Ba:表S6)。gydF4y2Ba
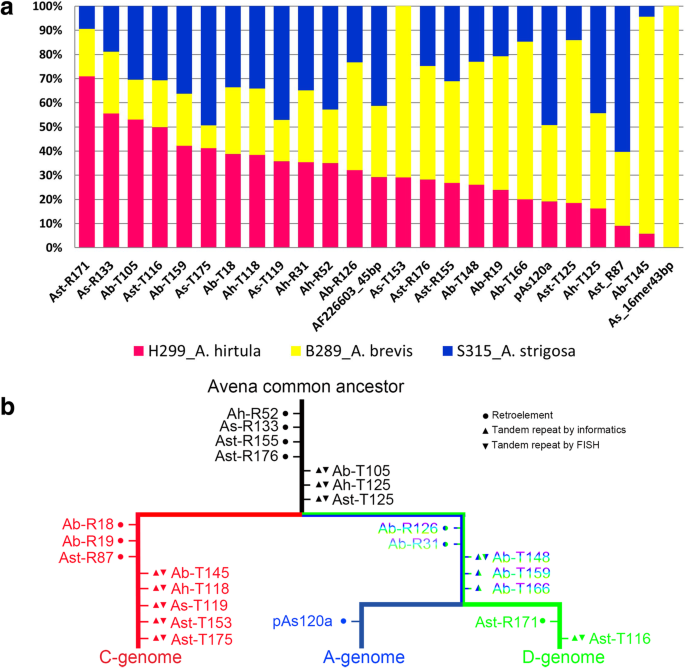
对所选探针的拷贝数和相对比例进行了分析gydF4y2BaA.苜蓿gydF4y2Ba和三个a基因组二倍体(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a)检查丰度和基因组特异性。在所有四个基因组中存在大多数重复(92%)(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a),预期变化从每种基因组超过300万份(AS-16mer43bpgydF4y2Ba一个gydF4y2Ba.gydF4y2Ba苜蓿gydF4y2Ba)几乎无法察觉。用于原位杂交的八个片段具有335-360磅的单体(附加文件gydF4y2Ba18.gydF4y2Ba:表S6,附加文件gydF4y2Ba19.gydF4y2Ba:表S7,附加文件gydF4y2Ba20.gydF4y2Ba:表S8a)。点图显示了单体数(图。gydF4y2Ba2gydF4y2BaF,J,M和O)是单体和聚类算法之间的变异性的结果,与基因组结构无关:串联重复计数需要非常长的读取(例如纳米孔或PACBIO续集)或染色体行走(例如BAC克隆)。在分析的三个二倍体中重复拷贝数,分析的基因组物种不相同(图。gydF4y2Ba6gydF4y2BaA),显示整个分布谱,表明进化分离后的差异扩增或损失。gydF4y2Ba
没有重复是主要的gydF4y2Ba答:strigosagydF4y2Ba,该物种中也没有重复As-T153和As-16mer43bp。一个重复家族(Ast-R171)在gydF4y2Ba答:hirtulagydF4y2Ba4个家族(As-T153、Ab-T166、Ast-T125和Ab-T145)占优势gydF4y2BaA. Brevis.gydF4y2Ba基因组(图。gydF4y2Ba6gydF4y2Ba一个,附加文件gydF4y2Ba20.gydF4y2Ba:表S8a)。As-16mer43bp重复序列仅在gydF4y2BaA. Brevis.gydF4y2Ba缺席,缺席gydF4y2Ba答:strigosagydF4y2Ba只有260份gydF4y2Ba答:hirtulagydF4y2Ba.gydF4y2Ba
原位杂交时,合成序列片段作为末端标记寡核苷酸,或从基因组DNA中进行PCR扩增gydF4y2Ba燕麦属gydF4y2Ba(一个基因组物种gydF4y2BaA. Brevis.gydF4y2Ba,gydF4y2Ba答:hirtulagydF4y2Ba,gydF4y2Ba答:strigosagydF4y2Ba,gydF4y2Ba答:王者世界gydF4y2Ba,gydF4y2Ba答:wiestiigydF4y2Ba,gydF4y2Ba答:longiglumisgydF4y2Ba和C-genome物种gydF4y2Ba答:erianthagydF4y2Ba;附加文件gydF4y2Ba18.gydF4y2Ba:表S6)。从这里鉴定的25个重复序列的原位杂交结果gydF4y2BaA.苜蓿gydF4y2Ba中期染色体使用两个或三个探针同时总结在附加文件gydF4y2Ba21.gydF4y2Ba:表S9, Figs。gydF4y2Ba4gydF4y2Ba和gydF4y2Ba5gydF4y2Ba,其他文件gydF4y2Ba3.gydF4y2Ba-gydF4y2Ba10.gydF4y2Ba:图S3-S10)。正如k-mer和重复开发者分析所预测的那样(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a),所有探头都产生杂交信号,信号强度通常在估计Silico拷贝数中。gydF4y2Ba
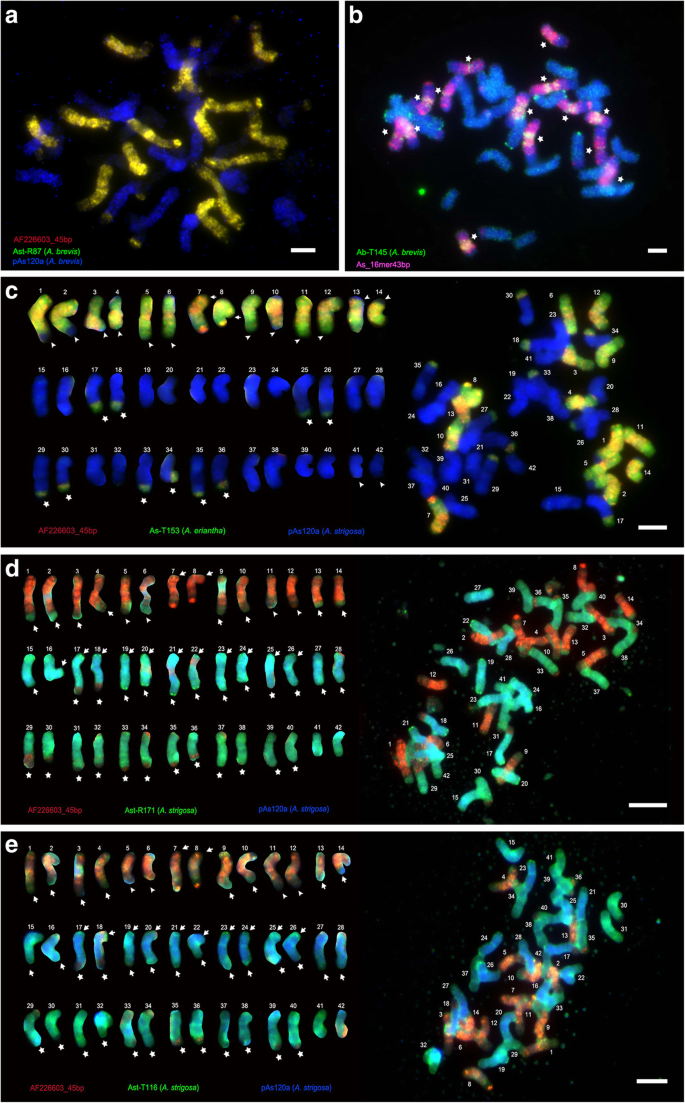
Avena Sativa.gydF4y2Ba染色体根据核型划分为基因组[gydF4y2Ba43gydF4y2Ba]和探针知识(特别是a基因组特异性探针pAs120a [gydF4y2Ba35gydF4y2Ba],c-genome特定af226603_45bp [gydF4y2Ba37gydF4y2Ba],45s和5s rdna)。染色体通过尺寸的降序编号并使用形态和杂交模式成对排列:分别属于C-,A-,D-基因组的染色体1-14,15-28和29-42(图。gydF4y2Ba4gydF4y2Ba汉英和中gydF4y2Ba5gydF4y2Baa - c)。原位杂交模式和生物信息学拷贝数计数使我们能够根据基因组特异性将重复序列分为5类:C-、A-和d -基因组特异性重复显示出对一个基因组的染色体更强的杂交(图)。gydF4y2Ba4gydF4y2Ba安妮,附加文件gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba和gydF4y2Ba6gydF4y2Ba:数字S4-S6);额外的重复是A + d基因组特异性的,或者在所有三个基因组中具有相似的强度(图)。gydF4y2Ba5gydF4y2BaA-E,附加文件gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba和gydF4y2Ba9gydF4y2Ba:数字S7-S9)。gydF4y2Ba
C-基因组特异性重复gydF4y2Ba
在14条c -基因组起源染色体的中间、中端粒和亚端粒区域检测到10个非同源原位杂交信号。我们在5/6和11/12对上看到四个未标记的末端区域,分别表示C-D和C-A易位(图。gydF4y2Ba4gydF4y2BaD-E,附加文件附加文件gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba和gydF4y2Ba6gydF4y2Ba:数字S4-S6)。gydF4y2Ba
重复As_16mer43bp高度丰富,有超过300万个拷贝,占1.45%gydF4y2BaAvena Sativa.gydF4y2Ba基因组(图。gydF4y2Ba2gydF4y2Ba一个,附加文件gydF4y2Ba20.gydF4y2Ba:表S8a;用作探针的合成标记的寡核苷酸,并且显示出强信号,沿着所有C-基因组染色体臂分散,具有较强的信号在最尖端的区域,并且在14 A或D-染色体长臂的远端区域上的弱分散信号(17/ 18和29-40;图。gydF4y2Ba5gydF4y2Baa).较少丰富的重复序列AF226603_45bp (0.33%gydF4y2BaA.苜蓿gydF4y2Ba基因组;附加文件gydF4y2Ba20.gydF4y2Ba表S8a)显示了相似的分布模式:14条c染色体(755,507拷贝;无花果。gydF4y2Ba2 bgydF4y2Ba,附加文件gydF4y2Ba20.gydF4y2Ba:表S8a),除了12 c-染色体1-6&9-14的端部之外,沿着臂和围吻区区域的分散信号(图。gydF4y2Ba4gydF4y2BaC-E)。AF226603_45BP信号还存在于10A + D-染色体的长臂端子,其具有近一半的17/18长臂的独特信号,并且在染色体25 / 26,29-40中的三分之一(图。gydF4y2Ba4gydF4y2BaC-E,附加文件gydF4y2Ba4gydF4y2Ba图S4a-4f,附加文件gydF4y2Ba5gydF4y2Ba图S5a-S5f附加文件gydF4y2Ba6gydF4y2Ba:图S6A-S6 F)。另外,使用探针AB-T148,AB-T159和AST-T116在20A + D-染色体15/16,19-24-38上观察核仁组织者区(NOR)(图。gydF4y2Ba4gydF4y2Bae和gydF4y2Ba5gydF4y2BaA,B,附加文件gydF4y2Ba5gydF4y2Ba:图S5A-S5F,附加文件gydF4y2Ba9gydF4y2Ba:图S9a-S9f)。gydF4y2Ba
几个逆转录转座子重复序列,Ab-R18, Ab-R19和Ast-R87(图2)。gydF4y2Ba2gydF4y2Bac、d和e),但也存在串联重复Ab-T145、As-T153和Ah-T118(图3)。gydF4y2Ba2gydF4y2Baf,g和i)显示出分散的信号,在14 c-染色体上具有高丰度(图。gydF4y2Ba4gydF4y2Bab,附加文件gydF4y2Ba3.gydF4y2Ba图S3a-S3b), A-和d -染色体上很少或没有信号(图gydF4y2Ba4gydF4y2Baa和c)。gydF4y2Ba
其他探针只标记了一些c -基因组染色体,并且在所有染色体上显示了更多的统一信号,表明至少20 kb的大型串联阵列,以看到双或更多点的FISH信号(附加文件gydF4y2Ba3.gydF4y2Ba:图S3D和S3E),例如,AS-T175和AS-T119(图。gydF4y2Ba2gydF4y2Bah和j)。gydF4y2Ba
一个基因组特定的重复gydF4y2Ba
与发布的PAS120A题为已发表的PAS120A的反朗植物相关探针(重复折叠簇289Cl22,299Cl11,312Cl11,312Cl17和315Cl 22gydF4y2Ba36gydF4y2Ba];无花果。gydF4y2Ba2gydF4y2Bak).与所有6个二倍体探针进行原位杂交,所有a染色体上都显示出相似和一致的信号,但有几个末端缺失,但在其他一些染色体的末端有额外的信号,表明A-C (17/18 & 25/26;无花果。gydF4y2Ba4gydF4y2Bac)和A-D (15/16, 19-24 & 27/28;无花果。gydF4y2Ba4gydF4y2Bad, e,附加文件gydF4y2Ba4gydF4y2Ba图S4a-S4f,附加文件gydF4y2Ba5gydF4y2Ba:图S5b-S5c和S5e-S5f)gydF4y2Ba
D-基因组特异性重复gydF4y2Ba
逆转录转座子Ast-R171(无花果。gydF4y2Ba2gydF4y2Bal)和串联重复序列Ast-T116(图1)gydF4y2Ba2gydF4y2Bam)主要在D-染色体上显示出强信号(图。gydF4y2Ba4gydF4y2BaD,E,附加文件gydF4y2Ba4gydF4y2Ba-gydF4y2Ba5gydF4y2Ba:数据。S4a-S4f和S5a-S5f), a染色体上有几个带,c染色体上有最小的信号(附加文件gydF4y2Ba4gydF4y2Ba:图S4B-S4C和S4E-S4F,附加文件gydF4y2Ba5gydF4y2Ba:图S5B-S5C和S5E-S5F)。AST-R171显示D-C在12 D-染色体上具有地板双点信号的D-C易位(29-40;附加文件gydF4y2Ba4gydF4y2Ba:图S4A-S4F),但是缺失的12D-染色体(29-40)缺失终端信号,又显示C基因组重复(图。gydF4y2Ba4gydF4y2Bad,额外的文件gydF4y2Ba4gydF4y2Ba:图S4A-S4F)。同样,12 D-染色体长臂终端(染色体29-40;附加文件)缺少AST-T116gydF4y2Ba5gydF4y2Ba图S5a-S5f)显示A-或c -基因组特异性。Ast-T116表现出典型的串联重复序列信号,即在A-染色体17-26的一个或两个末端出现双点(图17-26)。gydF4y2Ba4gydF4y2BaE,附加文件5:图S5B和S5E-S5F)。有时,在图31/32&37-40的染色体31/32&37-40的长臂终端上观察到弱易位信号。gydF4y2Ba4gydF4y2Bad和附加文件gydF4y2Ba6gydF4y2Ba:图S6a-S6c和图29-34、37/38gydF4y2Ba4gydF4y2Bae和附加文件gydF4y2Ba6gydF4y2Ba:图S6D-S6 F。gydF4y2Ba
A + D-基因组特异性重复并重复标记C-,A和D-基因组gydF4y2Ba
五种原位杂交探针的信号几乎均匀地均匀,但在C-染色体上相对较弱(图。gydF4y2Ba5gydF4y2BaA-D,附加文件gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba和gydF4y2Ba9gydF4y2Ba:数字S7-S9)。反转录转座子Ab-R126, Ah-R31的探针(图。gydF4y2Ba2gydF4y2Ban)和串联重复Ab-T148在A + d染色体上产生分散信号,16个未标记的A + d染色体末端(17/18,25/26和29-40;附加文件gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba和gydF4y2Ba9gydF4y2Ba:图S7-S9),横跨A + D-染色体臂的一些不均匀的闰带(图。gydF4y2Ba5gydF4y2Ba,附加文件gydF4y2Ba7gydF4y2Ba:图S7e,附加文件gydF4y2Ba8gydF4y2Ba:图S8e,附加文件gydF4y2Ba9gydF4y2Ba:图S9c-S9e)。串联重复Ab-T159gydF4y2Ba2 ogydF4y2Ba)和Ab-T166在A + d染色体上表现出均匀分散且弱的中心带信号;在12个A + d染色体终端(17/18、25/26、29-32和35-38)和12个c染色体终端(1-6和9-14)上观察到基因组间易位,A + d染色体上的信号分散,如反转录转座子(图)。gydF4y2Ba5gydF4y2Ba罪犯,附加的文件gydF4y2Ba6gydF4y2Ba:图S6i)。gydF4y2Ba
七个探针显示沿C-,A-和D-染色体的分散信号(附加文件gydF4y2Ba21.gydF4y2Ba:表S9)。Retrotroansposons AH-R52和重复AS-R133,AH-T125,AST-R155和AST-R176在C-,A-和D-染色体上显示了分散的信号(图。gydF4y2Ba2gydF4y2Bap和gydF4y2Ba5gydF4y2Bad e,附加的文件gydF4y2Ba10.gydF4y2Ba:图S10A-S10F)。序列AB-T105和AST-T125显示C-,A-和D-染色体上的间位双点(图。gydF4y2Ba5gydF4y2Bad,额外的文件gydF4y2Ba10.gydF4y2Ba:图S10f)。gydF4y2Ba
rdna网站gydF4y2Ba
45S和5S rDNA序列约占0.5%gydF4y2Ba燕麦属gydF4y2Ba基因组(附加文件gydF4y2Ba17.gydF4y2Ba:表S5)。串联重复45s rdna(9059 bp)含有3197 bp,1811 bp,260 bp,168 bp,216 bp和4407bp的ET,18s,ITS1,5.8s,ITS2和26s,而串联重复5s RDNA(544 BP)包含180bp编码序列和158-206bp interguic spacers(附加文件gydF4y2Ba11.gydF4y2Ba:图S11a-S11b)。在A和d染色体的6个NORs(15/16、29/30和35/36)上分别鉴定出6个45S rDNA位点,在A和d染色体的6个NORs加8个中间位置上分别鉴定出14个5S rDNA位点(15/16、29/30、31/32和35/36)。gydF4y2Ba5gydF4y2Baf,额外的文件gydF4y2Ba11.gydF4y2Ba:图S11c-S11e)。5S rDNA信号与A-和d染色体的6个NORs上的45S信号共定位(图)。gydF4y2Ba5gydF4y2Baf,额外的文件gydF4y2Ba11.gydF4y2Ba:图S11e),加上位于第31/32对的额外5S插入信号(附加文件gydF4y2Ba11.gydF4y2Ba:图S11d-S11e)。在6条NOR A-和d -染色体上可见延伸染色质和杂交信号(附加文件)gydF4y2Ba11.gydF4y2Ba图S11e)表明与c染色体相比,A-和D-染色体上的NOR-rDNA转录更活跃。gydF4y2Ba
讨论gydF4y2Ba
识别和丰富的重复性DNAgydF4y2Ba
基因组宽gydF4y2Ba
分析未加工gydF4y2Ba燕麦属gydF4y2Ba基因组DNA序列阅读采用基序计数(k-mer分析)和基于图的聚类显示,重复的DNA序列约占基因组的72%(图。gydF4y2Ba1gydF4y2Ba, 桌子gydF4y2Ba1gydF4y2Ba).结合原位染色体的分子细胞遗传学和硅分析,我们可以确定基序的性质,并测量它们的丰度,从而对燕麦重复景观的进化关系进行全面的调查。gydF4y2Ba1gydF4y2Ba和gydF4y2Ba6gydF4y2Ba).值得注意的是,这里研究的96%的序列可以被归类为与转座元件或相对少量的串联重复基序相关(图1)。gydF4y2Ba1gydF4y2Ba和gydF4y2Ba2gydF4y2Ba).我们的策略不会指望揭示微卫星基序,用独特的侧翼区域重复二核苷酸或三核苷酸重复,已知在基因组上具有不均匀的分布[gydF4y2Ba44gydF4y2Ba].虽然有越来越多的全基因组重复调查的报告[gydF4y2Ba13.gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,大多数序列程序集崩溃重复到不同程度[gydF4y2Ba21.gydF4y2Ba,gydF4y2Ba47gydF4y2Ba],而库筛选或用引物的PCR扩增是选择性的。因此,我们的结果和许多公开的使用全基因组组件的分析之间的详细比较,参考重复(例如重复掩手机)或有针对性的筛选可能无效。此外,主要群体中的“家庭”的分类是灵活的,具有一些不同的家庭,以及其他序列之间的中间体的其他家庭将被视为截然不同。此处鉴定出在DNA文库的选择性屏幕中鉴定出这里的许多主要系列[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba36gydF4y2Ba]但是这些研究无法量化它们在各个二倍体和六倍体基因组中的丰富。重要的是,与此处未加工随机读数的分析不同,选择性屏幕不能表明已经调查了基因组的所有重复组成部分。gydF4y2Ba
中主要重复DNA类的频率gydF4y2Ba燕麦属漂白亚麻纤维卷。gydF4y2Ba通过基于图形的聚类(重复折叠器)和丰富的K-MEL主题识别重复,并由核苷酸域名命中和数据库BLAST搜索分类。同心圆圈显示来自中心的越来越多的信息;结果与二倍体基因组相似(附加文件gydF4y2Ba17.gydF4y2Ba:表S5)gydF4y2Ba
中选择重复的簇图和点图gydF4y2Ba燕麦属gydF4y2Ba物种。对于每次重复,都会显示RepeatExplorer集群(左;黄色节点表示重复集群内所有组装的contigs;红节点代表更详细分析的contig成员,包括通过扩增和原位杂交。右面板中显示了所选contig的自点图;平行对角线表示串联重复。gydF4y2Ba一个gydF4y2BaAS_16MER43BP,在群集289CL8C154中识别。gydF4y2BabgydF4y2BaAF226603_45BP在群集289CL8C248中。gydF4y2BacgydF4y2Ba群集AB-R18。gydF4y2BadgydF4y2BaAb-R19。gydF4y2BaegydF4y2BaAst-R87。gydF4y2BafgydF4y2BaAb-T145。gydF4y2BagGydF4y2BaAh-T118。gydF4y2BahgydF4y2BaAS-T119。gydF4y2Ba我GydF4y2BaAs-T153。gydF4y2BajGydF4y2BaAs-T175。gydF4y2BakGydF4y2BaPAS120A在Cluster 312Cl17C141中。gydF4y2BalGydF4y2BaAst-R171。gydF4y2Ba米GydF4y2BaAst-T116。gydF4y2BanGydF4y2BaAH-R31。gydF4y2BaoGydF4y2BaAb-T159。gydF4y2BapGydF4y2BaAST-R176。重复名称包括示例性家庭成员的物种来源:AB,gydF4y2BaAvena Brevis.gydF4y2Ba;啊,gydF4y2Ba答:hirtulagydF4y2Ba;Ast,gydF4y2Ba答:strigosagydF4y2Ba;为,gydF4y2BaA.苜蓿gydF4y2Ba重复式:T型,串联式;R,逆转录转座子gydF4y2Ba
通过我们的K-MER分析鉴定的16mer,每个基因组超过10份拷贝对应于来自马铃薯和番茄的图(见图。gydF4y2Ba3.gydF4y2BaF)。16-MERS在24至40%的OAT基因组之间出现小于10次(图。gydF4y2Ba3.gydF4y2Ba),表示重复序列基序内的相对高的变化,并且这些家族可能无法通过重新分配动力学(实验)或图形聚类(生物信息学)来检测。总体而言,16-MERS的比例发生在10次或更少时gydF4y2Ba燕麦属gydF4y2Ba和在gydF4y2Ba佩妮gydF4y2Ba要么gydF4y2Ba高粱gydF4y2Ba(无花果。gydF4y2Ba3.gydF4y2Baf).然而,四之间的变异gydF4y2Ba燕麦属gydF4y2Ba物种(24,28,34和40%;图。gydF4y2Ba3.gydF4y2Ba)很难解释,但可能表明在gydF4y2Ba答:strigosagydF4y2Ba.坡度的变化,如gydF4y2Ba答:strigosagydF4y2Ba对于大于16bp的k-mers(图。gydF4y2Ba3.gydF4y2Ba),可能与不同重复DNA类群的频率或其同质化有关,但在不同重复DNA类群间无显著差异gydF4y2Ba答:strigosagydF4y2Ba和其他二倍体(附加文件gydF4y2Ba14.gydF4y2Ba:表S2)。gydF4y2BaA.苜蓿gydF4y2Ba图表斜率的变化较弱(图。gydF4y2Ba3.gydF4y2Ba),这与添加一个基因的做法一致gydF4y2Ba答:strigosagydF4y2Ba- 麦克风和另外两个物种。LTR回收转换在很大程度上负责相关植物物种之间基因组大小的显着差异,例如,玉米和水稻基因组之间的六倍尺寸差异[gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba所以他们也可以在这里发挥同样的作用。gydF4y2Ba
K-mer重复频率gydF4y2Ba燕麦属gydF4y2Ba基因组原料读。K-MEL主题的累积百分比绘制了不同K-MERS的频率gydF4y2BaAvena Sativa.gydF4y2Ba(gydF4y2Ba一个gydF4y2Ba),gydF4y2BaA. Brevis.gydF4y2Ba(gydF4y2BabgydF4y2Ba) 和gydF4y2Ba答:strigosagydF4y2Ba(gydF4y2BacgydF4y2Ba).比较16-mer(gydF4y2BadgydF4y2Ba)和64-mer(gydF4y2BaegydF4y2Ba频率四gydF4y2Ba燕麦属gydF4y2Ba基因组。Inset Box为每种基因组相当于≥10的百分比百分比。累积16-MER频率(gydF4y2BafgydF4y2Ba)gydF4y2Ba燕麦属gydF4y2Ba基因组比较gydF4y2BaPetunia arillaris.gydF4y2Ba(Bombarely等人[gydF4y2Ba21.gydF4y2Ba];他们的额外文件gydF4y2Ba10.gydF4y2Ba:他们的图S10),土豆(Xu et al. [gydF4y2Ba40gydF4y2Ba),高粱(Paterson等人[gydF4y2Ba39gydF4y2Ba)和番茄(TGC [gydF4y2Ba41gydF4y2Ba];他们的补充图S42)gydF4y2Ba
转座的元素gydF4y2Ba
LTR Retrotransposons的主要系列代表了60%的基因组和85%的零售gydF4y2Ba燕麦属gydF4y2Ba(无花果。gydF4y2Ba2gydF4y2Ba),这个值在不同的物种中有很大的差异。在gydF4y2BaBrachypodium distachyongydF4y2Ba(基因组大小272 Mb)逆转录转座子占基因组的21.6%,在水稻中占26% (430mb [gydF4y2Ba50gydF4y2Ba]),或面包小麦85%(6gydF4y2BaxGydF4y2Ba,16,000 MB [gydF4y2Ba51gydF4y2Ba[])和“超过80%”的大麦报告(2gydF4y2BaxGydF4y2Ba= 14,5100mb [gydF4y2Ba46gydF4y2Ba])。在gydF4y2Ba燕麦属gydF4y2Ba,像LTR-RORTORANSPOSONS(retroElements)这样的TY3-GYPSY比TY1-Copia等序列更高2.3至2.7倍(图。gydF4y2Ba1gydF4y2Ba和gydF4y2Ba2gydF4y2Ba,附加文件gydF4y2Ba17.gydF4y2Ba:表S5)。这些值与三个小麦基因组(比例2.5至2.9)紧密相似[gydF4y2Ba51gydF4y2Ba),尽管在其他物种中有所不同:总重复DNA占总DNA的73%gydF4y2Ba薏苡属aquaticagydF4y2Ba基因组(1.7 Mb),但吉普赛/Copia只有0.74 [gydF4y2Ba52gydF4y2Ba].DNA转座子,元素长度的元素长度缩短,返回的元素长度为5.5%gydF4y2Ba燕麦属gydF4y2Ba基因组,CMC-ENSPM与最丰富的序列一样。这个相对高的值类似于gydF4y2BaBrassica oleraceagydF4y2BaHDEM (4.69% (gydF4y2Ba53gydF4y2Ba])和gydF4y2Ba佩妮gydF4y2Ba物种(4.64 - -5.21%gydF4y2Ba21.gydF4y2Ba]),虽然其他物种较低的包括另一个物种要低得多gydF4y2Ba芸苔属植物gydF4y2Ba,gydF4y2Bab·拉伯gydF4y2BaZ1 (1.72% (gydF4y2Ba53gydF4y2Ba)和茄科植物gydF4y2BaSolanum.gydF4y2Ba和gydF4y2Ba烟草gydF4y2Ba物种(0.6-1.51%[gydF4y2Ba21.gydF4y2Ba])。gydF4y2Ba
所有基因组都具有控制TE扩增的机制。Schorn等人。[gydF4y2Ba54gydF4y2Ba显示在gydF4y2Ba拟南芥gydF4y2BaRNA驱动的DNA甲基化是如何负责沉默的,因为伞菌病毒序列的情况最有可能[gydF4y2Ba55gydF4y2Ba].大的基因组具有更高比例的TE序列,Lyu等[gydF4y2Ba56gydF4y2Ba建议TE负荷减少是美洲红树中基因组减少最重要的驱动因素。这里,燕麦回转回与燕麦糖蛋白相关的重复序列系列在二倍体物种之间的丰富中变化,有些是基本上特异于一个或两个基因组(图。gydF4y2Ba6gydF4y2Ba),暗示特定家庭的损失和收益和定向营业额。gydF4y2Ba
串联/卫星重复gydF4y2Ba
串联重复或卫星DNA是大多数真核基因组的特征。在这里,我们发现12个串联重复家庭,其中八个单体长度为335 bp-360 bp(附加文件gydF4y2Ba22.gydF4y2Ba:表S10)。已经注意到通过可变展开连接接头区域的〜30-60bp的可变展开接头区域包围的两个核体(〜150bp DNA)包围所需的单体长度(〜150bp用于单个核小体)[gydF4y2Ba16.gydF4y2Ba,gydF4y2Ba57gydF4y2Ba].核体和DNA之间的结构相互作用会影响染色质动力学[gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba[串联重复的稳定包裹对于导致沉默的基因组稳定性和甲基化可能是重要的。串联重复探针在共同燕麦染色体臂上显示离散信号(附加文件gydF4y2Ba3.gydF4y2Ba:图S3D-3E,附加文件gydF4y2Ba5gydF4y2Ba:图S5e-S5f),表示至少20kb的大阵列,但也有一些在插层位置显示分散信号(图5kb - 5f)。gydF4y2Ba5gydF4y2BaB-D),可能代表多个较小的数组。gydF4y2Ba
重复家族的亚基序可以作为基因组特异性的探针用于原位杂交,例如gydF4y2Ba芸苔属植物gydF4y2BaC-基因组特异性仙人掌转基因[gydF4y2Ba60gydF4y2Ba].在这里,发现探头AS_16MER43BP主题3,346,757读入gydF4y2BaA.苜蓿gydF4y2Ba,却不在gydF4y2Ba答:strigosagydF4y2Ba(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a)。另一个重复的AF226603_45bp motif首次通过Southern blot分析得到[gydF4y2Ba61gydF4y2Ba丰富的。相比之下,在普通燕麦c染色体上,两个短的45 bp的未知家族基元产生均匀分散的信号(图。gydF4y2Ba4gydF4y2Ba一、汉英)。虽然哺乳动物中约60 bp长的小卫星很常见,但对于植物重复基元来说,它们是相对不寻常的长度[gydF4y2Ba62gydF4y2Ba].gydF4y2Ba
重复序列的本地化gydF4y2BaA.苜蓿gydF4y2Ba通过原位杂交来中期染色体。根尖染色体荧光荧光青色与DAPI(UV激发)污渍;通过红色,绿色和/或蓝色(近红外荧光的假色彩)所示的荧光检测探针杂交位点。(A-C)C-基因组特异性探针;(D-E)D-基因组特异性探针。gydF4y2Ba一个gydF4y2Ba探针AF226603_45BP(红色),AST-R87(绿色;黄色,其中探测重叠)和PAS120A(蓝色)gydF4y2BaA. Brevis.gydF4y2Ba.gydF4y2BabgydF4y2Ba探针AS_16MER43BP(红色)和AB-T145(绿色)。(gydF4y2BacgydF4y2Ba-gydF4y2BaegydF4y2Ba)所在的中源性(右)和染色体被排列为核型显示,显示基因组来源(左),染色体均由探针图案和尺寸对(染色体1-14 c-基因组; 15-28A-基因组; 29-42 d-Genome)。白色星号,箭头和箭头表示值得注意的C-,A-和D-染色体信号。gydF4y2BacgydF4y2Ba探针AF226603_45bp(红色),As-T153(扩增自gydF4y2Ba答:erianthagydF4y2Ba;绿色)和pAs120a(蓝色)。gydF4y2BadgydF4y2Ba探针AF226603_45bp(红色)、Ast-R171(绿色)、pAs120a(蓝色)均从gydF4y2Ba答:strigosagydF4y2Ba.gydF4y2BaegydF4y2Ba探针AF226603_45bp(红色)、Ast-T116(绿色)、pAs120a(蓝色)。比例尺= 5 μmgydF4y2Ba
重复序列的本地化gydF4y2BaA.苜蓿gydF4y2Ba中期染色体。颜色和安排的细节如图所示。gydF4y2Ba4gydF4y2Ba.gydF4y2Ba一个gydF4y2Ba探针AS_16MER43BP(红色)和A + D-基因组特异性探针AB-T148(绿色)。gydF4y2BabgydF4y2Ba探针AF226603_45bp(红色)、pAs120a(蓝色)和A + d基因组特异性探针Ab-T159(绿色)。gydF4y2BacgydF4y2BaAF226603_45BP(红色),A / D基因组特异性AB-T166(绿色)和PAS120A来自gydF4y2Ba答:王者世界gydF4y2Ba.(蓝色)。(F)探针AF226603_45bp(红色),Ab-T148(绿色)来自gydF4y2Ba答:王者世界gydF4y2Ba45S rDNA探针pTa71(蓝色,用白色尖括号表示)。gydF4y2BadgydF4y2BaAb-T159(绿色)和Ab-T105探针gydF4y2Ba答:wiestiigydF4y2Ba.(红色的)。gydF4y2BaegydF4y2Ba探测AH-T125(绿色)来自gydF4y2Ba答:hirtulagydF4y2Ba和AS_16MER43BP(红色)。比例尺= 5 μmgydF4y2Ba
普通燕麦基因组中重复DNA基序的相对比例和进化模型。gydF4y2Ba一个gydF4y2Ba重复的相对比例gydF4y2Ba一个gydF4y2Ba.gydF4y2BaHirtula.gydF4y2Ba,gydF4y2BaA. Brevis.gydF4y2Ba,gydF4y2Ba答:strigosagydF4y2Ba分别用粉色、黄色和蓝色列表示的基因组。gydF4y2BabgydF4y2Ba重复DNA基序的进化模型。红色、蓝色和浅绿色分别表示C-、A-和d -基因组特异重复;蓝绿色的梯度字符表示A + d基因组特异性重复,黑色字符表示标记C-、A-和d基因组的FISH探针。Ab,gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba;啊,gydF4y2Ba答:hirtulagydF4y2Ba;Ast,gydF4y2Ba答:strigosagydF4y2Ba;为,gydF4y2BaA.苜蓿gydF4y2Ba.T,串联;R,逆转录转座子。gydF4y2Ba●gydF4y2Ba:retroelement;▲:通过生物信息学验证串联重复;gydF4y2Ba▼gydF4y2Ba:通过原位杂交验证的串联重复;NCBI串联爆炸结果串联重复家族查看其他文件gydF4y2Ba22.gydF4y2Ba:表S10gydF4y2Ba
RDNA用作染色体特异性探针。基于系统发育证据,gydF4y2Ba燕麦属gydF4y2Ba,每个单倍体染色体组有2个NORs (45S rDNA位点)是祖先性状,而含有4个或更多NORs的染色体是派生性状[gydF4y2Ba63gydF4y2Ba].在结构上,c基因组rdna的消除和a基因组rdna的部分消除在六倍体化事件后gydF4y2BaA.苜蓿gydF4y2Ba表明来自一个祖先的RDNA(父族基因组(见[gydF4y2Ba31.gydF4y2Ba])可能随着六倍体化而沉默和丢失gydF4y2BaA.苜蓿gydF4y2Ba.类似的45S rDNA位点的快速丢失在小麦四倍体中也可见gydF4y2BaAegilops ventricosa.gydF4y2Ba(与DDNN基因组指定),其中d -基因组45S rDNA位点丢失[gydF4y2Ba64gydF4y2Ba].gydF4y2Ba
重复的进化gydF4y2Ba
二倍体物种形成和重复dnagydF4y2Ba
具有重复序列位置的核型为我们理解生物进化提供了新的视角gydF4y2Ba燕麦属gydF4y2Ba.α-基因组特异性PAS120A很久以前分离[gydF4y2Ba36gydF4y2Ba].他们讨论了插入pAs120a内的4个单体的重复长度和存在性,谨慎地认为pAs120a序列可以归类为卫星DNA序列。相反,我们与pAs120a同源的序列与Ogre/Tat和色病毒反转录转座子具有高度的相似性(图。gydF4y2Ba2gydF4y2BaK)表明该重复序列来源于反转录元件,可能是由反转录元件亚区通过滚圈扩增产生的串联重复序列。然而,20年后,我们仍然共享Linares等人的不确定性[gydF4y2Ba36gydF4y2Ba这一序列进化成a基因组特异性的过程。其他序列家族在个体中也表现出差异扩增或减少gydF4y2Ba燕麦属gydF4y2BaA-genomes(无花果。gydF4y2Ba6gydF4y2BaA),并在六倍体与二倍体祖先基因组比较;c染色体特异性基序丰度较高gydF4y2BaA.苜蓿gydF4y2Ba基因组(如As-T153)或d染色体特异性基序的高丰度gydF4y2Ba答:hirtulagydF4y2Ba(Ast-T116;无花果。gydF4y2Ba6gydF4y2Ba一个,附加文件gydF4y2Ba20.gydF4y2Ba:表S8a)。gydF4y2Ba
根据分子年代分析,c基因组二倍体谱系的树冠年龄为20 Mya,大于a基因组多倍体谱系的树冠年龄[gydF4y2Ba31.gydF4y2Ba,gydF4y2Ba32.gydF4y2Ba].这是由更大比例的c -基因组特异性基模支持的,在A-和d -基因组特异性基模辐射之前,c -基因组特异性基模从共同祖先中分离出来,因为A-和d -基因组特异性基模在普通燕麦中独立扩增(图。gydF4y2Ba6gydF4y2Bab). A-和D-基因组或所有三个基因组共同的重复也支持这一进化情景,但没有发现C-和A-或C-和D-基因组特异的重复。反转录转座子可能在基因组行为中发挥作用,作为rna依赖的DNA甲基化的细胞核(如[gydF4y2Ba65gydF4y2Ba]),通过影响相邻基因表达的重复元素周围的异质体化通过异质体化的位置效果差异[gydF4y2Ba66gydF4y2Ba,gydF4y2Ba67gydF4y2Ba].gydF4y2Ba
远端染色体区域和易位gydF4y2Ba
用作探针的重复序列通过共同的燕麦中的染色体易位来显示主要的基因组改变[gydF4y2Ba43gydF4y2Ba].原位杂交是一种独特的方法来显示这些易位的性质和扩展,并使用重复探针(Figs。gydF4y2Ba2gydF4y2Ba和gydF4y2Ba3.gydF4y2Ba)显示三种基因组间易位:gydF4y2Ba
(一)gydF4y2BaC-基因组段易于α和D-染色体gydF4y2Ba- 由AF226603_45BP探针识别的四个A-C易位(对17/18和25/26)(图。gydF4y2Ba4摄氏度gydF4y2Ba,附加文件gydF4y2Ba9gydF4y2Ba:图S9A和S9D-S9E),和由AF226603_45BP探测器识别的12D-C易转置(染色体29-40)(附加文件gydF4y2Ba3.gydF4y2Ba:图S3A,S3C和S3F,附加文件gydF4y2Ba5gydF4y2Ba:图S5A-S5F);gydF4y2Ba
(b)gydF4y2BaA-或D-基因组段易于c-染色体gydF4y2Ba-四个C-A易位(对5/6和11/12)由pAs120a探针识别(附加文件gydF4y2Ba4gydF4y2Ba:图S4C-S4D,附加文件gydF4y2Ba5gydF4y2Ba:图S5C-S5D);和AST-T116探针鉴定的10个C-D易转移(染色体1-4,7-10和13/14)(图。gydF4y2Ba4gydF4y2Bad e,附加的文件gydF4y2Ba5gydF4y2Ba:图S5A-S5C和S5F);gydF4y2Ba
(c)gydF4y2BaA-和D-基因组易位gydF4y2Ba- 通过AST-R171和AST-T116探针识别的10 A-D易位(染色体15/16,19-24和27/28)(附加文件gydF4y2Ba4gydF4y2Ba:图S4B-S4C和S4E-S4F,附加文件gydF4y2Ba5gydF4y2Ba:图S5B-S5C和S5E-S5F),以及由PAS120A探针识别的两个D-A易处值(对41/42)(附加文件gydF4y2Ba5gydF4y2Ba:图S5B和S5F,附加文件gydF4y2Ba7gydF4y2Ba:图S7B和S7F)。gydF4y2Ba
易位可以改变染色体复合频率,并导致新杂种的遗传和进化分离。结果还提出了将遗传变异引入来自野生二倍体的小染色体区段的机会gydF4y2Ba燕麦属gydF4y2Ba物种进入六倍体,具有涉及任何基因组的重组段,或潜在更远的二倍体亲属。与小麦的燕麦骨髓组换算与小麦形成鲜明对比,其中没有关于4a和4b连杆基团的可能出现稳定的基因组易位的报告,其中4a染色体在a和b基因组中的其他染色体中的重复含量基本上不同。在燕麦中,重新排列的染色体可以通过使不同的表达水平和来自同源基因组的基因的表达调节来具有自适应值。gydF4y2Ba
大规模的基因组组织和影响gydF4y2Ba
基因组重复为整合不同基因组区域以协调基因组功能相互依赖的方面提供了物理基础[gydF4y2Ba68gydF4y2Ba].普通燕麦被推断起源于古老的异源四倍体(D-和c基因组)和最近的四倍体(acd基因组)事件在Pooideae亚家族[gydF4y2Ba31.gydF4y2Ba].鉴于A-和d染色体的重复丰度,我们推测gydF4y2Ba答:strigosagydF4y2Ba(D-genome)和gydF4y2Ba答:王者世界gydF4y2Ba-gydF4y2BaA. Brevis.gydF4y2Ba-gydF4y2Ba答:longiglumisgydF4y2Ba-gydF4y2Ba答:wiestiigydF4y2Ba(A-基因组)可能代表A-和d -基因组祖先的进化分离基础,就像其他物种[gydF4y2Ba69gydF4y2Ba])。基因组特异性重复扩增和亚基因组功能差异可能是冷驯化的驱动机制gydF4y2Ba燕麦属gydF4y2Ba[gydF4y2Ba70gydF4y2Ba],而亚基因组优势多倍体化可能支持最终符合农艺性状的抗锈病表型[gydF4y2Ba26.gydF4y2Ba].许多进化模型表明多倍体的形成应该与选择性优势有关,有利于亲代基因组的分化。考虑到普通燕麦中13个反转录转座子中的6个加上3个串联重复序列比二倍体亲本中丰富(附加文件gydF4y2Ba20.gydF4y2Ba:表S8a),推测古代重复相关的基因组重复的爆发是合理的,可以解释燕麦基因组大小的扩张。gydF4y2Ba
结论gydF4y2Ba
在这里,完全重复的DNA含量gydF4y2Ba燕麦属gydF4y2Ba在全基因组数据中进行了调查,重复序列占所有DNA的70%。最丰富的元素之前已经描述过,我们表明,少数重复家族,一些之前没有描述,贡献了所有重复DNA的高比例。很明显,在普通燕麦祖先的进化分离过程中,重复家族的重复扩增和更替已经参与其中,并且,结合频繁的基因组间易位(在其他谷物中没有看到)和进一步的更替事件,导致了六倍体的快速进化。转座因子是基因组的主要贡献者,尽管存在的家族和它们的相对丰度不同gydF4y2Ba燕麦属gydF4y2Ba来自其他物种群。反转录因子作为DNA标记或染色体探针的来源,在作物育种中具有实用价值[gydF4y2Ba71gydF4y2Ba并跟踪杂种和易位系的染色体。随着来自长读技术(包括Nanopore、PacBio Sequel和高通量染色体构象捕获)的数据越来越多,重复序列景观的知识对于优化基因组序列装配方法非常有用,因为它只考虑了少数重复家族的丰度和基因组分布。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
八gydF4y2Ba燕麦属gydF4y2Ba物种(样本来源载于附加档案gydF4y2Ba13.gydF4y2Ba:表S1,染色体和基因组指定参见Liu et al. [gydF4y2Ba31.gydF4y2Ba[[endlzy]],种子均来自加拿大萨斯喀彻温省和美国贝尔茨维尔种质系统)。171.9 Gb的原始序列代表4.58×到7.06×的覆盖率gydF4y2Ba燕麦属gydF4y2Ba基因组(gydF4y2BaA.苜蓿gydF4y2Ba(66.1 Gb),gydF4y2BaA. Brevis.gydF4y2Ba(34.6 GB),gydF4y2Ba答:hirtulagydF4y2Ba(35.3 Gb)gydF4y2Ba答:strigosagydF4y2Ba(35.9 Gb)],采用2 × 250 bp的散弹枪全基因组测序(南京基因先锋生物技术有限公司,Illumina HiSeq2500平台;附加文件gydF4y2Ba1gydF4y2Ba:图S1,附加文件gydF4y2Ba14.gydF4y2Ba:表S2a)。项目数据已在国家生物技术信息中心(NCBI)生物项目PRJNA407595 (SRR6056489-6056492)下保存。gydF4y2Ba
重复发现gydF4y2Ba
基于图的序列聚类gydF4y2Ba
基于相似性的聚类,重复识别和子集合终端原始读取的分类(1.72-2.87 GB占用2.60-8.29%gydF4y2Ba燕麦属gydF4y2Ba基因组;附加文件gydF4y2Ba14.gydF4y2Ba:表S2b)由RepeatExplorer分析执行(附加文件gydF4y2Ba15.gydF4y2Ba:表S3)。它被设置为读重叠,其中包含≥50%的长度,90%的相似性作为边缘,以节省“桥梁”读取的两个不相关的社区之间的部分相似性[gydF4y2Ba15.gydF4y2Ba].通过BLAST搜索针对NCBI数据库分析821个集群中的每一个的最长折叠以检查重复识别(图。gydF4y2Ba1gydF4y2Ba,附加文件gydF4y2Ba16.gydF4y2Ba:表S4)和人工总结的重复DNA组成(补充文件gydF4y2Ba17.gydF4y2Ba:表S5)。引物对是从每个反转录元素或串联重复序列的一个连续体中设计的,这些重复序列属于没有(或更少)一级相邻的簇[gydF4y2Ba72gydF4y2Ba](附加文件gydF4y2Ba12.gydF4y2Ba:图S12A-S12 T,附加文件gydF4y2Ba18.gydF4y2Ba:表S6,附加文件gydF4y2Ba19.gydF4y2Ba表S7),探针名称使用属和种缩写加上T表示串联或R表示逆转录转座子类型,然后是聚类数。克隆pTa71的引物设计[gydF4y2Ba34gydF4y2Ba]及pTa794 [gydF4y2Ba35gydF4y2Ba]分别用于45S和5S rDNA扩增(附加文件gydF4y2Ba18.gydF4y2Ba:表S6)。利用SeqGrapheR v.3.3.1对聚类图、点图和FISH探针拷贝数进行了研究[gydF4y2Ba15.gydF4y2Ba]和Geneious [gydF4y2Ba38gydF4y2Ba(图。gydF4y2Ba2gydF4y2BaA-P,附加文件gydF4y2Ba20.gydF4y2Ba:表S8)。gydF4y2Ba
k-mer分析gydF4y2Ba
通过程序Jellyfish V.2.2.6通过10至64-MER频率评估基因组重复性[gydF4y2Ba14.gydF4y2Ba)四gydF4y2Ba燕麦属gydF4y2Ba物种(图。gydF4y2Ba3.gydF4y2Baa-e)。比较频率gydF4y2BaPetunia arillaris.gydF4y2Ba,马铃薯,高粱和番茄[gydF4y2Ba21.gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba(图。gydF4y2Ba3.gydF4y2Baf). As_16mer43bp共识序列用于设计合成寡核苷酸探针(附加文件gydF4y2Ba18.gydF4y2Ba:表S6),其位置与RepeatExplorer集群图布局对齐(图6)。gydF4y2Ba2gydF4y2Ba一个)。gydF4y2Ba
多色荧光原位杂交gydF4y2Ba
根尖固定在96%乙醇:冰醋酸(3:1)至少为1.5小时,并在固定剂中储存在-20℃过夜。用0.2%纤维素酶Onozuka R10(yakult药物,东京),2%纤维素酶(C1184 Sigma-Aldrich)和3%果胶酶(P4716; Sigma-Aldrich,St Louis,美国)的酶溶液用于消化90分钟的根尖在37°C时。在60%乙酸中浸渍根尖,并在盖玻片下轻轻地压扁根。gydF4y2Ba
在含有50-100 ng基因组DNA、0.2 μM引物、0.4 mM三磷酸脱氧核苷酸混合物、1 × PCR缓冲液、2.5 mM Mg的25 μl反应液中进行PCR扩增gydF4y2Ba2+gydF4y2Ba和0.5 U Taq DNA聚合酶(Kapa Biosystems)。PCR条件为95℃3 min, 95℃30 s, 51.3-68.0℃30 s, 72℃1 min,共35个循环。PCR产物在1.0%琼脂糖凝胶上电泳,使用E.Z.N.A. Cycle Pure Kit (Omega)进行纯化,然后使用生物素阵列比较基因组杂交(CGH)基因组标记系统(Invitrogen)标记生物素-16- dutp或地高黄素-11- dutp(罗氏诊断)。gydF4y2Ba
FISH是由Schwarzacher和Heslop-Harrison描述的[gydF4y2Ba73gydF4y2Ba].34 μl杂交混合物(严格度76%),含50%甲酰胺,2 × SSC(生理盐水柠檬酸钠缓冲液;10%右旋糖酐硫酸盐,0.125% SDS(十二烷基硫酸钠),0.125 mM EDTA(乙二胺四乙酸);每张载玻片上应用1 μg剪过的鲑鱼精子DNA和多达1000 ng的标记探针。72℃变性7分钟后,37℃在ThermoHybaid HyPro-20中杂交过夜,载玻片在42℃0.1 × SSC中洗涤。用异硫氰酸荧光素(FITC)偶联的抗地高辛(200 μg/ml;Streptavidin Alexa Fluor 594 (200 μg/ml;分子探针)或Streptavidin Alexa Fluor 647 (1 mg/ml;4 × SSC, 0.1%吐温20,5% BSA(牛血清白蛋白)。用4 ',6-二氨基-2-苯基吲哚(DAPI; 3 mg/ml)-antifade solution (AF1, Citifluor, London, UK; 50%). FISH images were captured by a Nikon Eclipse 80i epifluorescent microscope fitted with appropriate sets of t band-pass filters to capture Alexa 647, Alexa 594, Tetrachlorofluorescein (TET), FITC and DAPI, a DS-QiMc monochromatic camera, and NIS-Elements v.2.34 (Nikon, Tokyo, Japan). For each metaphase, four single channel images (pseudo-coloured, yellow, red, green, and blue respectively) of 1280 × 1024 pixel size were analysed by Image J v.1.51j8 (Wayne Rasband, NIH, USA) and superimposed in Photoshop CS6 v.13.0 (Adobe System, San Jose, CA, USA). Total 269 slides including 2353 metaphases of 8 species (average 8 metaphases per slide) were observed for in situ hybridization analyses. Probe fragment copy numbers were determined by counting the number of reads (per genome equivalent) mapping to the probe fragment sequence (“Map to Reference”).
缩写gydF4y2Ba
- 阿瑟:gydF4y2Ba
-
Avena Brevis.gydF4y2Ba
- 啊:gydF4y2Ba
-
燕麦属hirtulagydF4y2Ba
- 为:gydF4y2Ba
-
Avena Sativa.gydF4y2Ba
- Ast:gydF4y2Ba
-
Avena Strigosa.gydF4y2Ba
- 爆炸:gydF4y2Ba
-
基本的局部比对搜索工具gydF4y2Ba
- BSA:gydF4y2Ba
-
牛血清白蛋白gydF4y2Ba
- 全息:gydF4y2Ba
-
比较基因组杂交gydF4y2Ba
- DAPI:gydF4y2Ba
-
4',6-二氨基-2-苯基吲哚gydF4y2Ba
- EDTA:gydF4y2Ba
-
Ethylenediamine-tetraacetic酸gydF4y2Ba
- enspm:gydF4y2Ba
-
增强子抑制突变脉gydF4y2Ba
- 鱼:gydF4y2Ba
-
荧光原位杂交gydF4y2Ba
- FITC:gydF4y2Ba
-
荧光素异硫氰酸酯gydF4y2Ba
- GC:gydF4y2Ba
-
Guanine-cytosinegydF4y2Ba
- INT(警官):gydF4y2Ba
-
整合酶gydF4y2Ba
- 线:gydF4y2Ba
-
长时间的核元素gydF4y2Ba
- LTR:gydF4y2Ba
-
长末端重复gydF4y2Ba
- NCBI:gydF4y2Ba
-
国家生物技术信息中心gydF4y2Ba
- 也没有:gydF4y2Ba
-
核仁组织者区gydF4y2Ba
- PCR:gydF4y2Ba
-
聚合酶链反应gydF4y2Ba
- rDNA:gydF4y2Ba
-
核糖体DNAgydF4y2Ba
- RH:gydF4y2Ba
-
rnaseh.gydF4y2Ba
- RT:gydF4y2Ba
-
逆转录酶gydF4y2Ba
- satDNA:gydF4y2Ba
-
卫星脱氧核糖核酸gydF4y2Ba
- SDS:gydF4y2Ba
-
十二烷基硫酸钠gydF4y2Ba
- 锡:gydF4y2Ba
-
短分散核元素gydF4y2Ba
- SSC:gydF4y2Ba
-
盐酸盐钠柠檬酸钠gydF4y2Ba
- TE:gydF4y2Ba
-
可转换元素gydF4y2Ba
- Tet:gydF4y2Ba
-
TetrachlorofluoresceingydF4y2Ba
- WGD:gydF4y2Ba
-
全基因组重复gydF4y2Ba
参考文献gydF4y2Ba
- 1。gydF4y2Ba
植物基因组在染色体中的组织。植物j . 2011; 66(1):像18岁到33岁这样的。gydF4y2Ba
- 2。gydF4y2Ba
Ruprecht C,Lohaus R,Vanneste K,Mutwil M,Nikoloski Z,Van de Peer Y等。在植物中重新审视祖先的多倍体。SCI ADV。2017; 3(7):E1603195。gydF4y2Ba
- 3.gydF4y2Ba
Vakirlis N,Sarilar V,钻探G,Fleiss A,Agier N,Meyniel JP等。祖传染色体建筑和基因曲目的重建揭示了模型酵母属的基因组演变原理。Genome Res。2016; 26(7):918-32。gydF4y2Ba
- 4.gydF4y2Ba
O 'Connor RE, Fonseka G, Frodsham R, Archibald AL, Lawrie M, Walling GA等。猪染色体亚端粒序列的分离用于易位筛选揭示了猪基因组装配的错误。动物麝猫。2017;48(4):395 - 403。gydF4y2Ba
- 5.gydF4y2Ba
Mandáková T,李泽,Barker MS, lyysak MA。在十字花科13个独立的中多倍体事件后不同的基因组组织与基因保留的收敛模式对比。植物j . 2017; 91(1): 3-21。gydF4y2Ba
- 6.gydF4y2Ba
De Koning APJ, Gu W, Castoe TA, Batzer MA, Pollock DD.重复元素可能包含超过三分之二的人类基因组。公共科学图书馆麝猫。2011;7 (12):e1002384。gydF4y2Ba
- 7.gydF4y2Ba
Biscotti MA, Olmo E, Heslop-Harrison JS。真核生物基因组中的重复DNA。Chromosom杂志2015;23(3):415 - 20。gydF4y2Ba
- 8。gydF4y2Ba
Maumus f,Quesneville H.植物基因组中古代重复元素的影响和见解。CurrOp植物BIOL。2016; 30(2):41-6。gydF4y2Ba
- 9。gydF4y2Ba
Treangen TJ, Salzberg SL.重复DNA和下一代测序:计算的挑战和解决方案。中国科学院院刊。2012;13(1):36-46。gydF4y2Ba
- 10。gydF4y2Ba
等。复制和非复制机制形成的集群cnv是由全基因组特征表明。公共科学图书馆麝猫。2018;14 (11):e1007780。gydF4y2Ba
- 11.gydF4y2Ba
Novák P, Neumann P, Pech J, Steinhaisl J, Macas J. RepeatExplorer:一个基于星系的网络服务器,用于从下一代序列读取真核重复元素的全基因组特性。生物信息学。2013;29(6):792 - 3。gydF4y2Ba
- 12.gydF4y2Ba
作者相关论文:张志强,张志强,张志强,等。黑麦全基因组序列(gydF4y2BaSecale Cereale.gydF4y2Bal .)。工厂J. 2017; 89(5):853-69。gydF4y2Ba
- 13。gydF4y2Ba
李志刚,李志刚,李志刚,等。马铃薯与番茄进化枝物种间重复序列的比较分析。安机器人。2019;123(3):521 - 32。gydF4y2Ba
- 14.gydF4y2Ba
Marçaisg,金福德C.一种快速,无锁的方法,可有效地对k Mers出现的速度进行计数。生物信息学。2011; 27(6):764-70。gydF4y2Ba
- 15.gydF4y2Ba
Novák P, Neumann P, Macas J.基于图的下一代测序数据中重复序列的聚类和表征。BMC生物信息学。2010;11(1):378。gydF4y2Ba
- 16.gydF4y2Ba
核小体和着丝粒DNA包装。中国科学院院刊2013;150(50):19974-5。gydF4y2Ba
- 17.gydF4y2Ba
Maças J, Neumann P, Navrátilová A.豌豆中的重复DNA (gydF4y2BaPisum一gydF4y2BaL.)基因组:使用454测序和大豆和大豆的综合表征gydF4y2BaMedicago truncatulagydF4y2Ba.BMC基因组学。2007; 8(1):427。gydF4y2Ba
- 18.gydF4y2Ba
Hřibováe,neumann p,matsumoto t,roux n,macas j,doleželj.banana的重复部分(gydF4y2Ba穆萨acuminatagydF4y2Ba)通过低深度454测序研究的基因组。BMC植物BIOL。2010; 10:204。gydF4y2Ba
- 19。gydF4y2Ba
Renny-Byfield S, Chester M, Kovařík A, le Comber SC, Grandbastien MA, Deloger M, et al.;下一代测序显示异源四倍体基因组缩小gydF4y2Ba尼科尼亚塔哈瓦姆gydF4y2Ba,主要通过消除患者衍生的重复DNA。mol Biol Evol。2011; 28(10):2843-54。gydF4y2Ba
- 20.gydF4y2Ba
何启元,蔡振华,刘宏杰,鲍春林,毛文辉,金伟伟。重复序列和核型分析揭示萝卜中着丝粒相关DNA序列(gydF4y2BaRaphanus SativusgydF4y2Bal .)。BMC Plant Biol. 2015;15:105。gydF4y2Ba
- 21.gydF4y2Ba
致以致法的,Moser M,Amrad A,Bao Mz,Bapaume L,Barry Cs等。从父母基因组中洞察Solanaceae的演变gydF4y2Ba佩妮矮牵牛gydF4y2Ba.Nat植物。2016; 2(6):16074。gydF4y2Ba
- 22.gydF4y2Ba
安通MB,安德森JW。燕麦粥 - 胆固醇联系:10年后。Am J Lifestyle Med。2008; 2(1):51-7。gydF4y2Ba
- 23.gydF4y2Ba
欧洲食品安全局营养产品、营养和过敏专家组(欧洲食品安全局)。根据第1924/2006号法规(EC)第14条,关于燕麦-葡聚糖和降低血液胆固醇和降低(冠心病)风险的健康声明的科学意见。欧洲食品安全署j . 2010; 8(12): 1885。gydF4y2Ba
- 24.gydF4y2Ba
MaçasJ,Neumann P,NovákP,江杰姆。基于大规模测序数据的低聚物频率分析的基于低聚物频率分析的全局序列表征。生物信息学。2010; 26(17):2101-8。gydF4y2Ba
- 25.gydF4y2Ba
罗明杰,顾玉强,普宇东,王浩,Twardziok SO, Deal KR,等。小麦D基因组祖先的基因组序列gydF4y2Ba山羊草属tauschiigydF4y2Ba.自然。2017;551(7681):498 - 502。gydF4y2Ba
- 26.gydF4y2Ba
赵小龙,赵小龙,赵小龙,等。SNP发现和染色体锚定提供了第一个物理锚定的六倍体燕麦图,并揭示了与模式物种的同步性。《公共科学图书馆•综合》。2013;8 (3):e58068。gydF4y2Ba
- 27.gydF4y2Ba
宋国英,霍培杰,吴斌,张志伟。利用SSR标记构建六倍体裸燕麦的遗传连锁图谱。作物j . 2015; 3(4): 353 - 7。gydF4y2Ba
- 28。gydF4y2Ba
Yan Hh,Martin Sl,Bekele Wa,Latta Rg,Diederichsen A,Peng Yy等。属的基因组大小变异gydF4y2Ba燕麦属gydF4y2Ba.基因组。2016; 59(3):209-20。gydF4y2Ba
- 29。gydF4y2Ba
Jellen EN, Gill BS, Cox TS,基因组学gydF4y2Ba原位gydF4y2Ba杂交区分a/D-和c -基因组染色质,并检测多倍体燕麦种(属)基因组间易位gydF4y2Ba燕麦属gydF4y2Ba).基因组。1994年; 37(4):613-8。gydF4y2Ba
- 30.gydF4y2Ba
Katsiotis A,Loukas M,Heslop-Harrison JS。重复的DNA,基因组和物种关系gydF4y2Ba燕麦属gydF4y2Ba和gydF4y2BaArrhenatherumgydF4y2Ba(禾本科)。安机器人。2000;86(6):1135 - 42。gydF4y2Ba
- 31.gydF4y2Ba
刘强,林林,周学勇,温杰。中国古代和近代多倍体化事件的进化动力学gydF4y2Ba燕麦属gydF4y2Ba(禾本科)。SCI批准。2017; 7(1):41944。gydF4y2Ba
- 32.gydF4y2Ba
傅yb。燕麦进化在25岁的母体谱系中透露gydF4y2Ba燕麦属gydF4y2Ba物种。Sci众议员2018;8(1):4252。gydF4y2Ba
- 33。gydF4y2Ba
周XY。a -基因组起源gydF4y2BaAvena Sativa.gydF4y2Ba由主要重复DNA课程定义。M.SC.论文:中国科学院华南植物园;2018. p。131。gydF4y2Ba
- 34。gydF4y2Ba
Gerlach WL,Bedbrook Jr。小麦和大麦核糖体RNA基因的克隆与表征。Nucl酸res。1979; 7(7):1869-85。gydF4y2Ba
- 35。gydF4y2Ba
Gerlach WL, Dyer TA。小麦核内含有5S rRNA基因的重复单元的序列组织。核酸学报1980;8(21):4851-65。gydF4y2Ba
- 36。gydF4y2Ba
6倍体燕麦a和D基因组的鉴别gydF4y2BaAvena Sativa.gydF4y2BaL. Proc Natl Acad Sci U S A. 1998; 95(21):12450-5。gydF4y2Ba
- 37。gydF4y2Ba
AnanieveV,Vales Mi,Phillips RL,Rines HW。a / d和c基因组特异性分散和聚类重复DNA序列的分离gydF4y2BaAvena Sativa.gydF4y2Ba.基因组。2002;45(2):431 - 41。gydF4y2Ba
- 38.gydF4y2Ba
等。Geneious basic:一个集成的、可扩展的用于序列数据组织和分析的桌面软件平台。生物信息学。2012;28(12):1647 - 9。gydF4y2Ba
- 39.gydF4y2Ba
Paterson啊,鲍德·杰,Bruggmann r,Dubchak I,Grimwood J,Gundlach H,等。这gydF4y2Ba高粱二色的gydF4y2Ba基因组和草的多样化。自然。2009; 457(7229):551-6。gydF4y2Ba
- 40.gydF4y2Ba
徐旭,潘胜,程顺福,张博,穆丹斯,倪px,等。块茎作物马铃薯的基因组序列分析。自然。2011;475(7355):189 - 95。gydF4y2Ba
- 41.gydF4y2Ba
番茄基因组联盟(TGC)。番茄基因组序列提供了深入了解肉质水果的进化。大自然。2012;485(7400):635 - 41。gydF4y2Ba
- 42.gydF4y2Ba
本网盖jl,王h。可转换元素对植物基因组学的结构,功能和演化的贡献。Annu Rev植物Biol。2014; 65(1):505-30。gydF4y2Ba
- 43.gydF4y2Ba
一种新的燕麦染色体命名系统(gydF4y2BaAvena Sativa.gydF4y2Bal .,gydF4y2Ba答:byzantinagydF4y2Ba基于单体素线的鱼分析的C.Koch。Al Appl Genet。2010; 121(8):1541-52。gydF4y2Ba
- 44.gydF4y2Ba
Katsiotis A, Schmidt T, Heslop-Harrison JS。属中ty1 -copia样逆转录转座子序列的染色体和基因组组织gydF4y2Ba燕麦属gydF4y2Ba.基因组。1996;(2):410 - 7。gydF4y2Ba
- 45.gydF4y2Ba
王光新,何启勇,马筱敏,Novák P, Neumann P,孟DX,等。串列重复序列的核型和分布gydF4y2Ba芸苔gydF4y2Ba由荧光gydF4y2Ba原位gydF4y2Ba杂化。CytoOgenet基因组Res。2017年; 152(3):158-65。gydF4y2Ba
- 46.gydF4y2Ba
柳条t,schulman啊,丹卡伦j,spannagl m,twardziok s,mascher m等。5100 MBP大麦基因组的重复景观。暴毒DNA。2017; 8:22。gydF4y2Ba
- 47.gydF4y2Ba
Kuhn GCS, Küttler H, Moreira-Filho O, Heslop-Harrison JS。1.688个重复DNAgydF4y2Ba果蝇gydF4y2Ba:不同基因组尺度和与基因相关联的协调演变。mol Biol Evol。2011; 29(1):7-11。gydF4y2Ba
- 48.gydF4y2Ba
国际水稻基因组测序项目。基于地图的水稻基因组序列。自然。2005; 436(7052):793-800。gydF4y2Ba
- 49.gydF4y2Ba
Schnable PS,Ware D,Fulton Rs,Stein Jc,Pasternak S,Liang C等人。B73玉米基因组:复杂性,多样性和动态。科学。2009; 326(5956):1112-5。gydF4y2Ba
- 50。gydF4y2Ba
国际Brachypodium倡议。模型草的基因组测序和分析gydF4y2BaBrachypodium distachyongydF4y2Ba.自然。2010; 463(7282):763。gydF4y2Ba
- 51.gydF4y2Ba
国际小麦基因组测序联盟(IWGSC)。使用完全注释的参考基因组转移小麦研究和育种的限制。科学。2018; 361(6403):AAR7191。gydF4y2Ba
- 52.gydF4y2Ba
蔡志新,刘慧娟,何清云,蒲万伟,陈杰,赖俊生,等。差异基因组进化和物种形成gydF4y2Ba薏苡属lacryma-jobigydF4y2Bal .,gydF4y2Ba薏苡属aquaticagydF4y2BaRoxb。杂交种gydF4y2Ba广西gydF4y2Ba精细核型的重复序列分析揭示。BMC基因组学,2014;15(1):1025 - 40。gydF4y2Ba
- 53.gydF4y2Ba
等。利用纳米孔长读取和光学图的植物基因组染色体级组装。Nat植物。2018;(11):879 - 87。gydF4y2Ba
- 54.gydF4y2Ba
Schorn AJ,Gutbrod MJ,Leblanc C,Martienssen R. LTR-Retrotransposon通过TRNA衍生的小RNA控制。细胞。2017; 170(1):61-71。gydF4y2Ba
- 55.gydF4y2Ba
Richert-Pöggeler KR, Noreen F, Schwarzacher T, Harper G, Hohn T.从矮牵牛内源性原病毒诱导传染性矮牵牛静脉清除(pararetro)病毒。EMBO j . 2003; 22(18): 4836 - 45。gydF4y2Ba
- 56.gydF4y2Ba
吕华,何智,吴慈,石胜。红树林边缘环境的趋同适应进化:卸载转座因子作为红树林基因组的共同策略。新植醇。2018;217(1):428 - 38。gydF4y2Ba
- 57。gydF4y2Ba
Antony Pass D,Sornay E,Marchbank A,Crawford Mr,Paszkiewicz K,Kent Na等。具有尺寸分辨率的基因组染色质映射显示了动态亚核糖组织景观gydF4y2Ba拟南芥gydF4y2Ba.公共科学图书馆麝猫。2017;13 (9):e1006988。gydF4y2Ba
- 58。gydF4y2Ba
Vershinin AV, Heslop-Harrison JS。黑麦、小麦及其亲缘种核小体结构的比较分析。植物学报。1998;36(1):149-61。gydF4y2Ba
- 59。gydF4y2Ba
Kaplan N, Moore IK, Fondufe-Mittendorf Y, Gossett AJ, Tillo D, Field Y,等。真核生物基因组的dna编码的核小体组织。大自然。2009;458(7236):362 - 6。gydF4y2Ba
- 60。gydF4y2Ba
Alix K, Joets J, Dunsdon CD, Heslop-Harrison P. CACTA的转座子Bot1在其中发挥了重要作用gydF4y2Ba芸苔属植物gydF4y2Ba基因组分化和基因增殖。植物j . 2008; 56(6): 1030 - 44。gydF4y2Ba
- 61。gydF4y2Ba
李镭社,罗斯纳格尔BG,Scoles GJ。追踪六倍体燕麦的系统发育gydF4y2BaAvena Sativa.gydF4y2Ba用卫星dnas。农作物科学。2000; 40(6):1755-63。gydF4y2Ba
- 62.gydF4y2Ba
NIWA O,Kominami R.在辐照精子出生的F1小鼠中母体衍生的小鼠过度高度小厕所等位式的未确定突变。Proc Natl Acad Sci U S A. 2001; 98(4):1705-10。gydF4y2Ba
- 63.gydF4y2Ba
Winterfeld G,RöserM.核糖体DNA在多年生燕麦染色体中的处置(Poaceae:Aveneae)。Bot J Linn Soc。2007; 155(2):193-210。gydF4y2Ba
- 64.gydF4y2Ba
Bardsley D,Cuadrado A,Jack P,Harrison G,Castilho A,Heslop-Harrison JS。四倍体小麦中的染色体标记gydF4y2BaAegilops ventricosa.gydF4y2Ba分析了gydF4y2Ba原位gydF4y2Ba杂化。计算机科学。1999;99(1-2):300-4。gydF4y2Ba
- 65.gydF4y2Ba
Matzke Ma,Mosher Ra。RNA定向的DNA甲基化:一种增加复杂性的表观遗传途径。NAT Rev Genet。2014; 15(6):394-408。gydF4y2Ba
- 66.gydF4y2Ba
玉米ltr反转录转座子的研究。In: Bennetzen J, Hake S, editors。玉米遗传和基因组学手册。纽约:施普林格Verlag;2009.307 - 27页。gydF4y2Ba
- 67.gydF4y2Ba
作者简介:李志伟,男,河北人,博士,主要研究方向为生物医学工程。单子叶植物基因组GC含量多样性的生态学和进化意义中国科学(d辑:地球科学)2014;gydF4y2Ba
- 68.gydF4y2Ba
夏皮罗农协。移动DNA与21世纪的进化gydF4y2Ba圣gydF4y2Ba世纪。暴毒DNA。2010; 1(1):4。gydF4y2Ba
- 69.gydF4y2Ba
高重复dna在兄弟物种进化中的平行性。中国生物医学工程学报。2010;27(8):1857-67。gydF4y2Ba
- 70。gydF4y2Ba
钟杰,罗贝特M,Poire A,Preston JC。连续的进化步骤将Poopeae草从热带推向温带地区。新植物。2018; 217(2):925-38。gydF4y2Ba
- 71。gydF4y2Ba
李斯克博士。植物基因调控中的转座子。在:Fedoroff NV,编辑。进化中的植物转座和基因组动力学。新泽西:John Wiley&Sons,Inc。;2013. p。93-116。gydF4y2Ba
- 72。gydF4y2Ba
Rozen S Skaletsky H. Primer3关于一般用户和生物学家程序员的WWW。在:Misener S,Krawetz SA,编辑。生物信息学方法和协议。TOTOWA:Humana媒体公司;1999. p。365-86。gydF4y2Ba
- 73。gydF4y2Ba
Schwarzacher T, Heslop-Harrison P. PracticalgydF4y2Ba原位gydF4y2Ba杂化。英国牛津:BIOS科学出版社;2000.gydF4y2Ba
确认gydF4y2Ba
感谢cns - saskatchewan和usd - beltsville种质系统提供的种子、ELIXIR-CZ项目(LM2015047)提供的基于银河的重复探测资源、广州国家超级计算中心提供的数据分析、J. Bailey博士提供的染色体准备和R.H.M. Salih博士提供的显微镜检查。gydF4y2Ba
资金gydF4y2Ba
J.S. Heslop-Harrison中国科学院海外杰出学者项目(no . Y861041001);中国科学院本科生创新训练计划项目(no . 59);国家科技部基础研究专项(2013FY112100)。资助方在研究设计、数据收集和分析、决定发表或手稿准备方面没有作用。gydF4y2Ba
可用性数据和材料gydF4y2Ba
与本研究相关的所有数据都以表格和/或图形形式包含在手稿中,如果合理要求,作者很乐意分享分析/原始数据和植物材料。支持本文结论的其他数据集包括在本文及其附加文件中。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
QL、TS、JSHH设计实验;QL, XYL, XYZ, MZL, FJZ进行探针设计,FISH并分析数据,QL, TS, JSHH撰写手稿。所有作者均已阅读并批准本稿件。gydF4y2Ba
相应的作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版物gydF4y2Ba
不适用。gydF4y2Ba
利益争夺gydF4y2Ba
作者宣布没有存在竞争利益。gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
附加文件gydF4y2Ba
附加文件1:gydF4y2Ba
图S1。gydF4y2Ba八个取样的小穗形态gydF4y2Ba燕麦属gydF4y2Ba物种。一个gydF4y2Ba答:王者世界gydF4y2Ba:分散单位-上面的小花与下面的小花相连,只有下面的小花断开。bgydF4y2Ba短:gydF4y2Ba小穗显示宿存的小花具双齿外稃尖端在成熟。cgydF4y2Ba答:(hirtulagydF4y2Ba一地中海野生型,外稃刚毛6-10毫米。dgydF4y2BaA. Longiglumis:gydF4y2Ba2-3小花/小穗,每小花断开;外稃背面覆盖着浓密的毛发。egydF4y2Ba答:(strigosagydF4y2Ba2-3小花香/小穗和持久的小花。fgydF4y2Ba答:wiestiigydF4y2Ba:沙漠和草原野生型,带有睫毛刷毛5-8毫米。gGydF4y2BaA. Eriantha:gydF4y2Ba颖片明显大小不等。hgydF4y2BaA. Sativa:gydF4y2Ba尖峰1.5-4厘米,通常在成熟时蔓延。秤杆= 1厘米。(TIF 4210 KB)gydF4y2Ba
额外的文件2:gydF4y2Ba
图S2。gydF4y2Ba基于图形的簇的分布。重复爆发器分析的分层凝聚gydF4y2Ba燕麦属gydF4y2Ba物种基因组显示。一个gydF4y2Ba一个gydF4y2Ba.年代GydF4y2Baativa.gydF4y2BaS312。bgydF4y2BaA. Brevis.gydF4y2BaB289。cgydF4y2Ba答:hirtulagydF4y2Ba.H299。dgydF4y2Ba答:strigosagydF4y2BaS135。彩色条表示群体≥0.01%的基因组:X轴表示累积读数数百分比,而Y轴表示簇中的读数。栏根据重复类型的群集注释(附加文件)gydF4y2Ba15.gydF4y2Ba:表S3)。(TIF 1895 kb)gydF4y2Ba
额外的文件3:gydF4y2Ba
图S3。gydF4y2Ba所选C-基因组特异性重复序列的定位gydF4y2BaAvena Sativa.gydF4y2Ba多色FISH的中期染色体。探测信号分别由黑白CCD相机捕获,然后伪彩色生成叠加图像。有关信号分布的详细描述,请参阅附加文件gydF4y2Ba21.gydF4y2Ba:表S9。AF226603_45BP(杂交位点以红色显示),AB-R18放大gydF4y2Ba答:王者世界gydF4y2Ba(绿色)和pAs120a来自gydF4y2Ba答:王者世界gydF4y2Ba(蓝色)。注意,红色和绿色探测器的重叠信号会给出黄色信号。b Ab-R19(红色)和Ab-R126(绿色),DAPI反染色(蓝色)显示在所有染色体上。注意红色和绿色探针的重叠信号显示为白色,但有几个染色体末端没有被探针标记为蓝色或绿色双点。c AF226603_45bp(红色),Ah-T118 fromgydF4y2Ba答:hirtulagydF4y2Ba(绿色)和pAs120a来自gydF4y2Ba答:hirtulagydF4y2Ba(蓝色)。红色和绿色探针的重叠信号呈现黄色,显示染色体的不均匀标记。在图像底部可见间相核。d As-T119(绿色),在As_16mer43bp的红色信号上方出现黄色双点(星号)。DAPI荧光显示为蓝色。e一个年代-T175 (green, double-dots) and As_16mer43bp (red) showing large blocks of hybridization signal on C genome chromosomes (starred). DAPI fluorescence shown in blue. f TET labeled AF226603_45bp (red), As_16mer43bp (pink), and DAPI (blue). Scale bars = 5 μm. (TIF 6648 kb)
额外的文件4:gydF4y2Ba
图S4。gydF4y2Ba荧光原位杂交(FISH)核型分析gydF4y2BaAvena Sativa.gydF4y2Ba.探针AF226603_45bp(直接TET,红色)代表C基因组,pAs120a(生物素,Alexa 594,蓝色)代表A基因组,Ast-R171(地高皂苷,FITC,绿色)代表D基因组。右边的染色体DAPI图像为白色,中间的同一中期显示了所有三个探针的杂交信号,除了(d)只有黄色的AF226603_45bp和蓝色的pAs120可见。从不同的二倍体物种中扩增出探针。一个gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba.bgydF4y2BaA. atlantica。gydF4y2BacgydF4y2Ba答:strigosagydF4y2Ba.dgydF4y2Ba答:wiestiigydF4y2Ba.egydF4y2Ba答:longiglumisgydF4y2Ba.fgydF4y2Ba答:hirtulagydF4y2Ba.在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色星星、箭头和箭头表示C-、A-和d -染色体区域,易位到一个不同的基因组:在12条d -染色体上有C易位(29-40);4个c染色体易位(5/6 & 11/12)10条a染色体的D易位(15/16,19-24和27/28)。比例尺= 5 μm。(TIF 7816 kb)gydF4y2Ba
额外的文件5:gydF4y2Ba
图S5。gydF4y2Ba荧光原位杂交(FISH)核型分析gydF4y2BaAvena Sativa.gydF4y2Ba.探针是AF226603_45bp(直接TET,红色)的C基因组,pAs120a(生物素,Alexa 594;A基因组为蓝色,D基因组为Ast-T116(地高辛,FITC,绿色)。右边的染色体DAPI图像为白色,中间的同一中期显示所有三个探针的杂交信号,除了(d)和(e),其中只有黄色的AF226603_45bp和蓝色的pAs120可见。从不同的二倍体物种中扩增出探针。一个gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba.bgydF4y2BaA. atlantica。gydF4y2BacgydF4y2Ba答:strigosagydF4y2Ba.dgydF4y2Ba答:wiestiigydF4y2Ba.egydF4y2Ba答:longiglumisgydF4y2Ba.fgydF4y2Ba答:hirtulagydF4y2Ba.在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色圆圈表示核仁组织者区(nors)信号。白恒星,箭头和箭头表示C-,A-和D-染色体地区,转向不同的基因组:10个C-染色体上有D转移(1-4,7-10和13/14);10 d-染色体(29-40)上有C易位;4个c染色体易位(5/6 & 11/12)10条a染色体的D易位(15/16,19-24和27/28)。比例尺= 5 μm。(TIF 8170 KB)gydF4y2Ba
附加文件6:gydF4y2Ba
图S6。gydF4y2Ba荧光原位杂交(鱼)gydF4y2BaAvena Sativa.gydF4y2Ba.远红色(a, d, g),假蓝色(b, e, h)和绿色(c, f, i)基因组染色体的单通道图像由Image J v.1.51j8分析。gydF4y2Ba4gydF4y2BaA (A -c), 4e (d-f)和5c (g-i)(a-c) FISH探针AF226603_45bp(直接TET,红色),pAs120a (biotin, Alexa 594;蓝色)和Ast-R171(地高辛,FITC,绿色)gydF4y2Ba答:strigosagydF4y2Ba:图1的重叠染色体(A)6,(B)21和25,16,23和24,23和24和31&41的分离。gydF4y2Ba4gydF4y2Bad. (d-f) FISH探针AF226603_45bp(远红色)、Ast-T116(绿色)和pAs120a(假蓝色)gydF4y2Ba答:strigosagydF4y2Ba:图1的重叠染色体(D)3,6,12和14,(E)18和27,(F)33和42的分离。gydF4y2Ba4gydF4y2Bae. (g-i) AF226603_45bp(远红色),A/ d基因组特异性Ab-T166(绿色)和pAs120a(假蓝色)来自gydF4y2Ba答:王者世界gydF4y2Ba:重叠染色体的分离(g) 7, 8和14,(h) 18, 23, 24, 25和26,和(i) 31和36的图。gydF4y2Ba5gydF4y2Bac. (TIF 6215kb)gydF4y2Ba
附加文件7:gydF4y2Ba
图S7。gydF4y2Ba荧光原位杂交(FISH)核型分析gydF4y2BaAvena Sativa.gydF4y2Ba.探针是AF226603_45BP(直接TET,RED)用于C基因组,PAS120A(BIOTIN,Alexa 594;蓝色),AB-R126(Digoxigenin,FITC,Green)。右侧为白色的染色体DAPI图像,中间为杂交信号。从不同的二倍体物种中扩增出探针。一个gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba.bgydF4y2BaA. atlantica。gydF4y2BacgydF4y2Ba答:strigosagydF4y2Ba.dgydF4y2Ba答:wiestiigydF4y2Ba.egydF4y2Ba答:longiglumisgydF4y2Ba.fgydF4y2Ba答:hirtulagydF4y2Ba.在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色的星星、箭头和箭头表示C-、A-和d -染色体区域,易位到不同的基因组。比例尺= 5 μm。(TIF 8552 KB)gydF4y2Ba
附加文件8:gydF4y2Ba
图S8。gydF4y2Ba荧光原位杂交(FISH)核型分析gydF4y2BaAvena Sativa.gydF4y2Ba.探针是AF226603_45BP(直接TET,RED)对于C基因组,PAS120A(BIOTIN,Alexa 594;蓝色)用于基因组,AH-R31(Digoxigenin,FITC,Green)。右侧为白色的染色体DAPI图像,中间为杂交信号。从不同的二倍体物种中扩增出探针。一个gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba.bgydF4y2BaA. atlantica。gydF4y2BacgydF4y2Ba答:strigosagydF4y2Ba.dgydF4y2Ba答:wiestiigydF4y2Ba.egydF4y2Ba答:longiglumisgydF4y2Ba.fgydF4y2Ba答:hirtulagydF4y2Ba.在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色的星星、箭头和箭头表示C-、A-和d -染色体区域,易位到不同的基因组。此外,在附加文件中,白钻石表示强烈的绿色波段信号gydF4y2Ba8gydF4y2Ba:图S8e)。比例尺= 5 μm。(TIF 7444 kb)gydF4y2Ba
附加文件9:gydF4y2Ba
图S9。gydF4y2Ba荧光原位杂交(FISH)核型分析gydF4y2BaAvena Sativa.gydF4y2Ba.探针是AF226603_45bp(直接TET,红色)的C基因组,pAs120a(生物素,Alexa 594;蓝色)和Ab-T148(地高辛,FITC,绿色)。右侧为白色的染色体DAPI图像,中间为杂交信号。从不同的二倍体物种中扩增出探针。一个gydF4y2Ba一个gydF4y2Ba.gydF4y2Ba短gydF4y2Ba.bgydF4y2BaA. atlantica。gydF4y2BacgydF4y2Ba答:strigosagydF4y2Ba.dgydF4y2Ba答:wiestiigydF4y2Ba.egydF4y2Ba答:longiglumisgydF4y2Ba.fgydF4y2Ba答:hirtulagydF4y2Ba.在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色的星星、箭头和箭头表示C-、A-和d -染色体区域,易位到不同的基因组。此外,在附加文件中,白钻石表示强烈的绿色波段信号gydF4y2Ba9gydF4y2Ba:图S9C-E)。比例尺= 5 μm。(TIF 8041 KB)gydF4y2Ba
附加文件10:gydF4y2Ba
图S10。gydF4y2Ba荧光原位杂交(鱼)gydF4y2BaAvena Sativa.gydF4y2Ba.生物素标记As_16mer43bp(红色)和荧光原位杂交(FISH)探针标记普通燕麦C-, A-和d基因组染色体的染色体分布(gydF4y2BaAvena Sativa.gydF4y2Ba).从中标记的Digoxigenin标记AH-R52(绿色)gydF4y2Ba答:hirtulagydF4y2Ba.B莫昔芬标记为AS-R133(绿色)来自gydF4y2Ba答:longiglumisgydF4y2Ba.c地高辛标记Ast-R155(绿色)来自gydF4y2Ba答:wiestiigydF4y2Ba.d地高黄素标记Ast-R176(绿色)来自gydF4y2Ba答:hirtulagydF4y2Ba.e Digoxigenin标记为ah-t125(绿色)gydF4y2Ba答:wiestiigydF4y2Ba.f地高辛标记为Ast-T125(绿色)来自gydF4y2Ba答:wiestiigydF4y2Ba.比例尺= 5 μm。(TIF 8865 KB)gydF4y2Ba
附加文件11:gydF4y2Ba
图S11。gydF4y2BarDNA序列组装和FISH。(a-b)对簇连序列进行比对,组装45S (a)和5S rDNA (b)gydF4y2Ba燕麦属漂白亚麻纤维卷。gydF4y2Ba(c-e)鱼核型gydF4y2BaA.苜蓿gydF4y2Ba使用AF226603_45BP(直接TET,RED),RDNA等。右侧为白色的染色体DAPI图像,中间为杂交信号。在核型中(左图),染色体按基因组来源的顺序排列:1-14 c -基因组,15-28 a -基因组,29-42 d -基因组。白色角度括号和鸽子分别表示45s和5s rDNA信号。CIGOXIGENIN标有AB-T148(绿色)gydF4y2Ba答:wiestiigydF4y2Ba生物素标记的pTa71_45S(蓝色)。d地高辛标记的Ab-T148(绿色)来自gydF4y2Ba答:wiestiigydF4y2Ba和生物素标记为pta794_5s(蓝色)。e Digoxigenin标记为pta794_5s(绿色)和生物素标记为pta71_45s(蓝色)gydF4y2Bat . aestivumgydF4y2Ba.比例尺= 5 μm。(TIF 4200 kb)gydF4y2Ba
额外的文件12:gydF4y2Ba
图S12。gydF4y2Ba分析邻近的RepeatExplorer集群。交互图显示了本研究中使用的含有FISH探针的选定簇(红色圆圈)和至少有一个连接的一级邻居(绿色圆圈)。两个相邻簇之间的连接,用灰色和黑色的线表示,并带有相似的读取数。312 cl82。312 cl83 b。c 312 cl119。d 312 cl125。312 cl133 e。f 312 cl151。g 312 cl153。 h 289CL18. i 289CL19. j 289CL93. k 289CL105. l 289CL126. m 289CL145. n 289CL159. o 289CL166. p 299CL31. q 299CL52. r 315CL87. s 315CL116. t 315CL125. Clusters with at least one neighbor shown in circle. Remaining clusters 289CL148, 289CL187, 299CL118, 299CL125, 299CL126, 312CL175, 315CL155, 315CL171 and 315CL176 without any one neighbor. Black letters denote tandem repeats (b, c, f-g, j-k, m-o, s-t) and brown-red letters denote linear cluster graphs of non-tandem repeats (a, d-e, h-i, l, p-r). (JPG 457 kb)
附加文件13:gydF4y2Ba
表S1。gydF4y2Ba本研究中使用的材料。源给出源头和原产地,物种名称,权限,染色体数量和基因组指定gydF4y2Ba燕麦属gydF4y2Ba本研究中使用的样品。还列出了重复群集和尖峰图。(DOCX 27 KB)gydF4y2Ba
额外的文件14:gydF4y2Ba
表S2。gydF4y2Ba全基因组散弹枪测序reads统计和RepeatExplorer分析gydF4y2Ba燕麦属gydF4y2Ba物种。单倍体基因组大小和全基因组霰弹枪测序读取四个统计数据gydF4y2Ba燕麦属gydF4y2Ba物种在(a)和重复折叠式摘要数据中给出(b)。(docx 17 kb)gydF4y2Ba
额外的文件15:gydF4y2Ba
表S3。gydF4y2Ba重复探索器分析四个gydF4y2Ba燕麦属gydF4y2Ba物种。重复DNA组成的聚类、读数和基因组比例(a)和经RepeatExplorer注释的聚类(大于或等于基因组的1.00%)分析(b)的4个gydF4y2Ba燕麦属gydF4y2Ba物种。(多克斯40 kb)gydF4y2Ba
附加文件16:gydF4y2Ba
表S4。gydF4y2BaNCBI爆炸结果为4个821簇gydF4y2Ba燕麦属gydF4y2Ba物种。集群和每个群集列出了每个群集内部,描述,查询覆盖范围和身份的最长折叠。(docx 92 kb)gydF4y2Ba
额外的文件17:gydF4y2Ba
表S5。gydF4y2Ba重复DNA组成四个基因组gydF4y2Ba燕麦属gydF4y2Ba物种。每个物种列出基因组部分(百分比)。还给出了可转换元素I类(Retrotransposons)和II类(DNA转座子)的家庭和亚颗粒,以及串联重复。(DOCX 22 KB)gydF4y2Ba
额外的文件18:gydF4y2Ba
表S6。gydF4y2Ba用于生成FISH探针的寡核苷酸。引物对、退火温度、预期片段大小和从k-mer或RepeatExplorer分析中识别的重复序列的标签gydF4y2Ba燕麦属gydF4y2Ba物种。(多克斯23 kb)gydF4y2Ba
附加文件19:gydF4y2Ba
表S7。gydF4y2BaRepeatExplorer对选定的四个重复集群进行描述gydF4y2Ba燕麦属gydF4y2Ba物种。用作k-mer或regalexplorer分析用作鱼探针的重复片段的表征gydF4y2Ba燕麦属gydF4y2Ba物种。列出了基因组比例、域命中、contig长度和FISH图的参考文献。(多克斯24 kb)gydF4y2Ba
额外的文件20:gydF4y2Ba
表S8。gydF4y2Ba的基因组拷贝数和选择重复的相对比例gydF4y2Ba燕麦属gydF4y2Ba物种。Pairedund Fasta数据用于佐义V.10.0.7(Kearse等,2012)中的“地图以参考”工具。(DOCX 25 KB)gydF4y2Ba
附加文件21:gydF4y2Ba
表S9。gydF4y2BaFISH信号分布模式gydF4y2BaAvena Sativa.gydF4y2Ba使用探针进行选择重复的染色体。根据Sanz et al.(2010)对269张玻片和2353个中期(平均每个玻片8个中期)进行分析,并将染色体分配到基因组C、A或D。10个探针是C基因组特异性的,1个探针是A基因组特异性的,2个探针是D基因组特异性的,5个探针是A和D基因组特异性的,7个探针是与所有三个基因组杂交的。(DOCX 25 KB)gydF4y2Ba
附加文件22:gydF4y2Ba
表S10。gydF4y2Ba串联重复的NCBI爆破结果见图。gydF4y2Ba6gydF4y2Bab.列出了12个串联重复序列的基因组特异性、卫星家族指定、单体长度和NCBI登录。(多克斯28 kb)gydF4y2Ba
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文根据创意公约署署署的条款分发了4.0国际许可证(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条提供的数据,除非另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
刘强,李旭东,周旭东。gydF4y2Ba等等。gydF4y2Ba燕麦(禾本科)的重复DNA景观:全基因组序列中主要重复类定义的染色体和基因组进化。gydF4y2BaBMC植物杂志gydF4y2Ba19,gydF4y2Ba226(2019)。https://doi.org/10.1186/s12870-019-1769-zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-019-1769-zgydF4y2Ba
关键词gydF4y2Ba
- 染色体进化gydF4y2Ba
- 常见的燕麦(gydF4y2BaAvena Sativa.gydF4y2Ba)gydF4y2Ba
- 荧光原位杂交(鱼)基于核型gydF4y2Ba
- 基因组特异性标记gydF4y2Ba
- Intergenomic易位gydF4y2Ba
- 重复dnagydF4y2Ba