抽象的
背景
类黄酮3'-羟基酶(F3'H)是测定黄酮类化合物的B环羟基化图案的重要酶。在单焦点中,先前的研究表明存在两组具有不同酶活性的F3'HS。发现水稻中的一个F3'H显示出新的克莱氏醇特异性5'-羟化酶活性。然而,单焦F3'HS的进化历史和观察到的催化差异的分子基础仍然难以捉摸。
结果
我们对12种常见单子叶植物进行了全基因组调查,共鉴定出44种推测的单子叶植物F3'H基因。结果表明F3'H基因家族在单子叶中经历了挥发性谱系特异性基因复制和基因丢失事件。的扩张F3'H基因家族主要是分散的基因复制。系统发育分析表明,单子叶植物F3'hs.在单子叶植物共同祖先的基因复制后,进化成两个独立的谱系(第一类和第二类)。进化动力学分析在II类动物中发现了积极的自然选择F3'hs.,作用于7种特定的氨基酸位点。蛋白质建模显示这些选定的位点主要位于F3'H的催化腔中。序列对准显示,I类和II类F3'HS在先前发现的两个关键位点显示氨基酸取代,以负责F3'H和黄酮类化合物3'5'-羟化酶(F3'5'H)活性。此外,对于I类和II类,也观察到转录分歧F3'hs.在四种单子叶植物中。
结论
我们的结论是单子叶植物F3'hs.在单子叶植物共同祖先的基因复制之后,已经进化成两个独立的谱系(Mono_F3'H Class I和Class II)。单子叶植物f3’h II类的功能分化受到正向自然选择的影响,这种正向自然选择只作用于特定的氨基酸位点。已确定的关键氨基酸位点极有可能影响II类F3'Hs底物特异性。我们的研究为之前观察到的水稻CYP75B4的5 ' -羟化活性提供了进化和蛋白质结构上的解释,这可能也适用于单子叶植物中其他II类F3'Hs。我们的研究在基因进化水平上为植物-环境相互作用提供了明确的证据,并将指导未来谷类植物中F3'Hs的功能研究。
背景
包括花青素,黄酮和黄酮类的黄酮类化合物是植物的所有器官和组织中存在的普遍存在的次要代谢物[1,2].在过去的几十年里,已经朝着单焦谷物作物中的生物功能引起了巨大的研究注意[1,3.,4],如小麦,大麦,水稻和玉米,这是人类食物的主要来源。从生物学的角度来看,类黄酮生物合成在植物的防御机制中起重要作用,对各种非生物和生物应激因子,包括紫外线 - 辐射,热,重金属离子,干旱,病原体和微生物侵入等。[5,6,7].花和种子中的类黄酮色素是吸引昆虫和动物授粉和传播种子的可见信号[8,9].此外,已显示黄酮类化合物参与花粉萌发[10,11],也可作为生长素运输和分解代谢的发育调节剂[12,13].已经显示出谷物谷物中花青素的黄酮类化合物,从而影响种子休眠,预防预热萌芽[3.,14帮助植物在不利的环境条件下生存。从食物消费的角度来看,类黄酮化合物,由于其抗氧化特性,也被证明对保护退行性疾病如冠心病和癌症有很大的健康益处[15,16,17].
在单子叶植物中,类黄酮生物合成的分子机制已经很好地确立[1].以苯丙氨酸为起点,通过苯丙氨酸途径转化为4-香豆蔻酰辅酶a,进入类黄酮生物合成途径[18].查尔酮合成酶(Chalcone synthase, CHS)和查尔酮异构酶(Chalcone isomerase, CHI)是类黄酮途径中的前两种酶,导致查尔酮和柚皮素的连续生产,作为所有类黄酮的前体[19].基于类黄酮B环的羟基化图案,黄酮类生物合成可以分为三种不同的方向,导致最终产生一种羟基(Pelargonidin型),双羟基(氰化型)和三羟基(Delphinidin-type)花青素[20.].类黄酮b环的羟化模式受两个关键酶类黄酮3 '羟化酶(F3 ' h)和类黄酮3 ' 5 ' -羟化酶(F3 ' 5 ' h)控制。F3’h属于细胞色素p45o依赖单加氧酶超家族的CYP75B亚家族,而F3’5’h属于CYP75A亚家族,与F3’h存在侧位功能差异[21.].在类黄酮途径中,F3'H催化在3'-位置的Naringenin和二氢转蛋白醇的羟基化,导致最终产生Cyanidin的花青素。相反,F3'5'H能够在3'和5'位置羟类化合物B形环,其负责替代蛋白的花青素产生。F3'H活性与F3'5'H活性一起与中央黄酮途径竞争,没有羟化,并导致了黄酮类生物合成途径的大多数[20.].已经提出这种代谢多样化,以在植物在进化期间对各种环境条件的适应中发挥关键作用。
基因复制是植物基因组中的广泛现象。它产生了用于环境选择的原始遗传物质,以对植物多样化起到核心作用,从而促进其环境适应[22.,23.].复制后,基因副本可能丢失或保留,这取决于在蛋白质结构水平和/或基因转录水平是否产生有益功能。物种特异性基因拷贝数的保留被认为是帮助不同植物应对特定环境挑战的重要手段。最近的一项研究报告说F3'hs.(F3'H-1&F3'H-2)在大麦基因组中存在,这是在分化前的一次重复事件的结果麦芽汁部落[24.].组织特异性表达谱也被观察到F3'H-1和F3'H-2.在另一个之前的研究中,3和2份F3'hs.分别在大麦和大米中发现[25.].有趣的是,水稻中两种F3'Hs之一(CYP75B4)被证实在金衣酚上募集了新的5 ' -羟化酶活性,这是tricin生物合成的关键步骤[25.].初步系统发育分析表明,CYP75B4与正常单子叶植物f3’hs不同,属于一个独立的系统发育类群。CYP75B3和CYP75B4在水稻中的底物特异性也不同[26.].这些观察结果表明单子信F3'HS之间的潜在功能性分歧。然而,没有对单子码的F3'H基因家族进行系统和综合的进化分析。蛋白质结构基础下面的CYP75B4的5'-羟化酶活性仍在表征。
在这项研究中,我们调查了在主要谷类植物中假定的F3'H基因的保存情况,这些基因的基因组数据可在公共数据库中获得。通过系统发育和自然选择分析,对单子叶植物f3’h基因的进化史进行了研究。在单子叶植物F3'H基因亚家族的进化过程中,我们发现了植物与环境相互作用的明确证据。我们的研究巩固了类黄酮生物合成途径作为研究植物与环境相互作用的模型,也将为单子叶植物f3’h基因亚家族的功能研究提供指导。
结果
单子叶植物中F3'H的鉴定
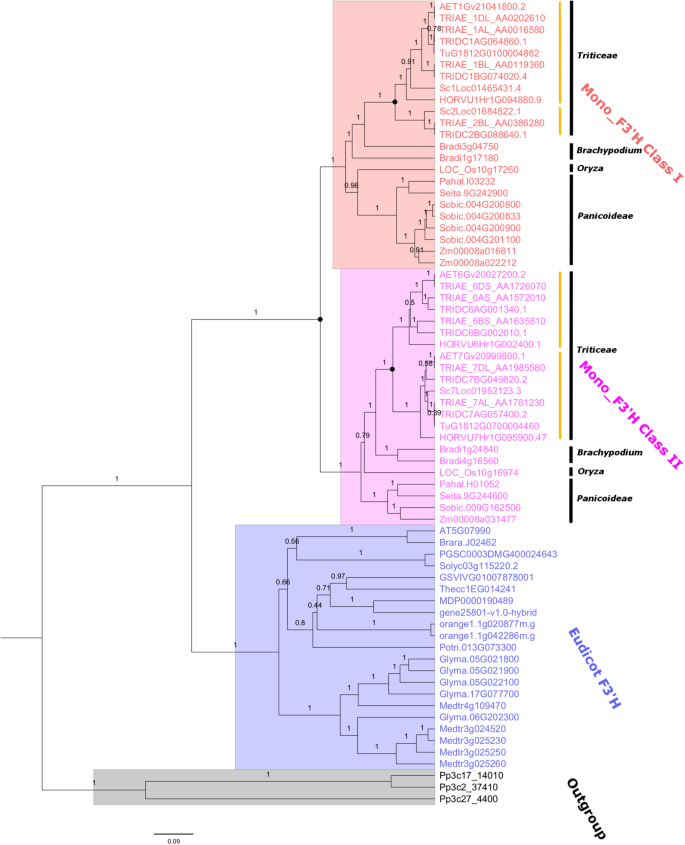
为了鉴定真正的F3'H基因,基于检索到的F3'H同源物的氨基酸序列比对,建立了一个完整的neighbor Joining树。两个不同的分支,包括之前分别鉴定的F3 ' hs和F3 ' 5'Hs。那些在F3'H分支中的同源蛋白被认为是真实的F3'hs.并被选中进行进一步分析。如表所述1,共有44个推定F3'H基因被从12个单子叶中鉴定出来。至少两份副本F3'H存在于每个物种中。最多的F3'H发生在小麦(9),紧随其后小麦属植物dicoccoides(7)麦芽汁亚科。所有其他的麦芽汁作物包括大麦芽,山羊草属tauschii和Secale cereale包含3份F3'H基因,除了Triticum Urartu.,它有2个副本。Broachypodium distachyon.的近亲麦芽汁,载有4份F3'hs..此外,大多数植物在Panicoideae血统,包括Setaria italica和Panicum Hallii.,保留2F3'hs.,同时玉米和Sorhum Bicolor.分别有3和5份。特别是,其他重要的作物Oryta漂白亚麻纤维卷也包含2F3'hs..
发展史推理
为了研究单子叶植物f3’h基因家族的进化史,基于已鉴定的f3’h基因的编码域序列(CDS)比对,建立了贝叶斯系统发育模型F3'hs..EudicotF3'hs.和远程F3'H来自下层植物的同源物Physcomitrella金属盘被纳入外群体。总体而言,系统发生树显示了强大的拓扑支持,表明解析的系统发生是高度可靠的。如图所示。1,目标F3'hs.被分成了两个主要集群,对应于eudicotF3'H和单子叶植物F3'H,分别。单子台F3'hs.进一步细分为两个截然不同的谱系,这里将其分别归类为Mono_F3'H I类和II类。值得注意的是,Mono_F3'H I类和II类分别覆盖了本研究涉及的所有单子叶植物,表明分化发生在物种多样化之前。对系统发育的进一步研究表明,这两种植物具有相同的进化模式,与单子叶植物的系统发育相似。具体来说,在Mono_F3'H I类和II类中,Panicoideae植物包括z梅斯,S. Bicolor.和S. Italica.首先发散,其次是o.苜蓿,这代表了一种进化的中间Panicoideae和麦芽汁.对于其他植物,b . distachyon分化之前麦芽汁.F3'hs.从检索麦芽汁植物簇拥在一起,支撑力强。这些结果表明,I类和II类F3'hs.已经在单子植物植物中垂直进化,提供了进一步的支持,即I类和II类之间的分歧F3'hs.发生在单子叶作物的共同祖先时期。一类和二类的普遍守恒F3'hs.单像表示两者F3'H类是这些植物正常生长所必需的。
的进化F3'hs.在单极管显示一个明确的类和物种特定的配置文件。虽然只有一级I和II级的单一副本F3'hs.被保存在Panicoideae植物美国斜体和p . hallii,S. Bicolor.和z梅斯载有第I类文件4份及2份F3'hs,分别。此外,谱系特异性的扩展F3'H也观察到b . distachyon(一)、(二)两份F3'hs..o.苜蓿res美国斜体和p . hallii一个等级和一等二级F3'H.为麦芽汁,进化F3'H似乎是独立于上述植物的。在Mono_F3'H II类血统中,麦芽汁f3’hs可进一步分为两个分支(黄线;无花果。1),涵盖了所有的麦芽汁本研究包括的植物。这表明II类F3'Hs在共同祖先时期经历了额外一轮的复制麦芽汁,但散之后b . distachyon.值得注意的是,可以观察到类似的扩张模式Triticieae课上我F3'hs.,也演变成两个明显的分支(黄线;无花果。1),指示在事件之前的重复事件麦芽汁多样化也是如此。我的两个子分支之一F3'H(上面的黄线)覆盖了所有麦芽汁另一种只保存在t . turgidum,t . aestivum和美国cereale,但缺席h . vulgare,答:tauschii.次级分支的缺失可能是由于复制后基因丢失造成的。值得注意的是,I类和II类F3'Hs的重复事件麦芽汁可能表明在?的共同祖先中有一个共同的基因组复制事件麦芽汁.需要进一步的研究来证实这一假设。除了麦芽汁,我和II级F3'hs.在b . distachyon倾向于作为独立的谱系独立进化。
基因组结构分析
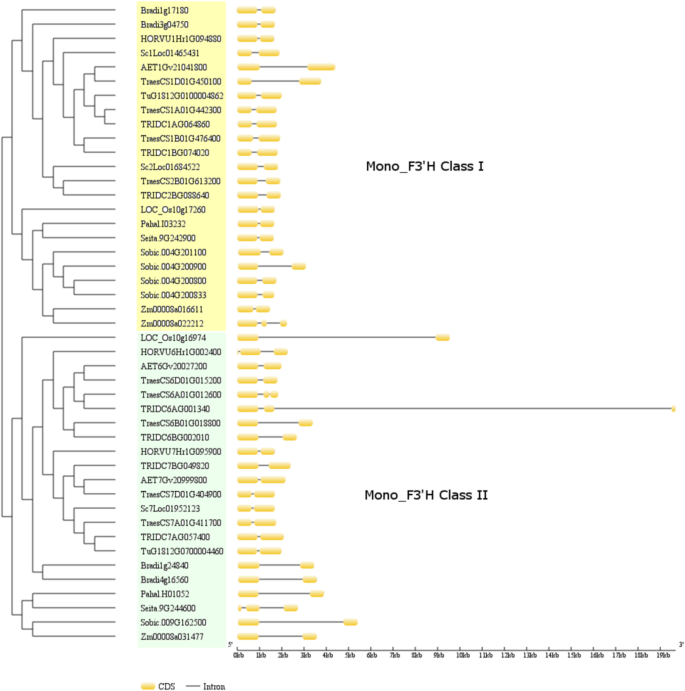
的基因结构特征F3'H及其与单子麦克特F3'H基因亚家族的演化史的潜在关系,单子宫古典的基因结构F3'hs.以发达的系统发育为基础进行分析。如图所示。2,Monocot的大多数(41/45)F3'hs.无论文学发生,含有两个外显子。另外四个推定F3'hs.,分别对应于I类中的Zm00008a022212和II类中的TraesCS6A01G012600、TRIDC6AG001340、Seita.9G244600,保留了3个外显子。大多数情况下,内含子的长度是保守的F3'hs.从不同的系统发育分支,除非麦芽汁二类F3'hs.,内部长度显示出清晰普遍的增加。除了非麦芽汁二类F3'hs.,其他几个F3'hs.来自I类和II类也表现出Intron长度的增加,它对应AET1GV21041800,Traest1D01G450100,Sobic.004G200900(I类)和TRAESK6B01G018800,TridC6BG002010(II类)。在这五个基因中,AET1GV21041800 / TRAESCS1D01G450100和TRIDC6BG002010 / TRAESK6B01G018800是关闭同源物对,可能反映了原点t . aestivumD和B亚基因组答:tauschii和t . turgidum,分别。应注意,这里呈现的基因结构数据是基于植物基因组数据库中的基因注释。需要在各种物种中克隆和测序的实验室基因,以进一步验证这些结果。我们还抑制了将包括5'UTR和3'UTR在内的推定推动者区域进行比较F3'hs.因为缺乏实验资料。
复制模式与共同分析
不同机制引起的基因重复可分为四类:全基因组重复/节段重复、串联重复、近端重复和分散重复。为了进一步研究F3'H家族,基因复制模式确定F3'Hs在9种单码种类(表1).t . aestivum,t . turgidum和美国cereale由于它们的多倍性或缺乏良好的基因组注释信息而被排除在该分析之外。如表所示1,所有的F3'hs.在麦芽汁植物被鉴定为分散的重复。同样的观察是用的F3'hs.在b . distacyon(4份),所有这些都从分散基因重复中出现。F3'hs.在水稻中,位于10号染色体上的两个副本被发现是近端副本。有趣的是,2F3'hs.(ZM00008A016611,ZM00008A022212)来自z梅斯被发现源自全基因组或节段性重复,反映了该物种中F3'HS的不同进化源。另一个F3'H(zm00008a031477)z梅斯也被鉴定为分散的复制品。此外,4F3'hs.在染色体4 inS. Bicolor.被鉴定为串联复制或近端复制,与其他物种相比是非常不寻常的。另一个F3'H(sobic.009g162500)S. Bicolor.被发现是分散的复制品。不像z梅斯和S. Bicolor.,所有的F3'hs.从美国斜体和p . hallii在里面Panicoideae发现部落已导致分散重复。
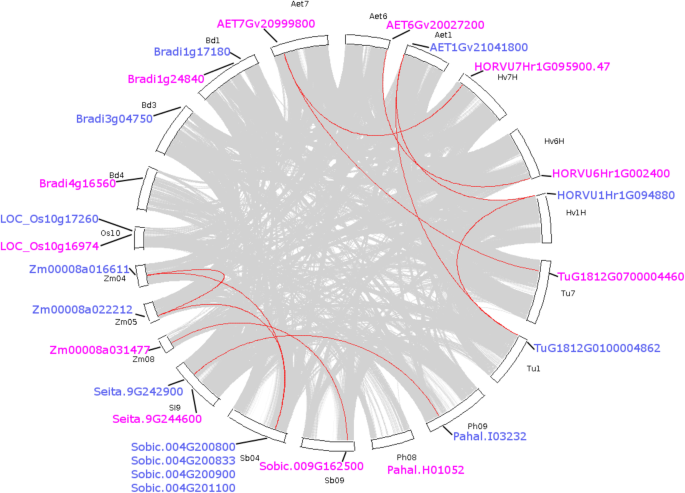
调查同步保护F3'hs.在单焦植物,共线F3'H对基因进行了鉴定。如图所示。3.,共12条共线F3'H对已经被确定F3'hs.在9种单焦种。这些基因对可分为两个簇:麦芽汁- 特价和Panicoideae-具体的。为麦芽汁,F3'hs.位于HV1H,HV6H和HV7H染色体上h . vulgare与?共线F3'hs.在AET1,AET6和AET7染色体上答:tauschii,分别。值得注意的是,TuG1812G0100004862在染色体Tu1中t . urartu位于带有Horvu1HR1G094880和AET1GV21041800的共线区域中,Tug1812G0100004862在染色体TU7上采用AET7GV20999800与Horvu7HR1G095900.47相结合。有趣的是,所有这些基因对都发生在同一类中F3'hs..没有类F3'H一对在麦芽汁.综合起来,确定了F3'HConlinear对反映了密切的关系麦芽汁物种也证明了严格的保护F3'hs.在这些植物。值得注意的是,在Tu6染色体上未发现F3'Ht . urartu,可能在这个物种中丢失了。
其他鉴定的共线F3'H对在Panicoideae(图。3.).对于f3'hs无法识别联接性o.苜蓿和b . distacyon这可能反映了这两个物种中F3'Hs的基因组位置不同。为Panicoideae包括Z. mays, S. italic, S. bicolor和p . hallii,共线性主要存在于II类F3'hs.中鉴定到1对引入种(Zm00008a016611-Zm00008a022212)z梅斯.此外,I类只发现一对共线对(Zm00008a031477-Sobic.009G162500)F3'hs.在Panicoideae.对于我的课程,没有相连的性F3'hs.在S. Italica.(Seita.9G244600)和p . hallii(Pahal.H01052)。同样,第一类和第二类之间没有类间共线性F3'hs.在Panicoideae.
自然选择测试
研究单子叶植物的进化动力学F3'H基因家族,对发达的自然选择测试进行了F3'H发展史。非同义替代和同义替代的比值(ω)是评价进化基因选择压力的重要参数,其中ω < 1、ω = 1和ω > 1分别表示纯化选择、中性进化和正向选择。计算了在不同假设下Monocot F3'H Class I和Class II的分支和氨基酸位点特异性ω值。EudicotF3'H用作参考。对于分支的具体模型,有三个假设(表2)进行了测试。似然比检验(LRTs)表明,双比模型ω(eudi) = ω(mono1)≠ω(mono2)和ω(eudi) = ω(mono2)≠ω(mono1),分别为Monocot F3'H Class I和Class II指定了发散ω值,两者都显著更好(df = 1,p < 0.0001; df = 1,p= 0.0444)比单比模型ω(eudi) = ω(mono1) = ω(mono2).此外,三比值模型ω(eudi)≠ω(mono1)≠ω(mono2),为所有三个分支指定不同Ω值,显着拟合数据集(df = 1,p< 0.0001)优于ω(eudi) = ω(mono2)≠ω(mono1),但不是更好(df = 1,p= 1.0)比ω(eudi) = ω(mono1)≠ω(mono2).这些计算表明ω(mono2)与ω显着不同(eudi)和ω(mono1),ω[eudi]和ω[mono2]彼此没有显着差异。这表明单焦炭类IIF3'hs.与Monocot IIF3'hs.和eudicot.F3'hs..在最佳拟合模型Ω下(eudi) = ω(mono1)≠ω(mono2),ω(mono2)为0.82808,而ω(eudi)和ω(mono1)等于0.11857。这些计算表明,Monocot I类F3'Hs和Eudicot F3'Hs的纯化选择较强,而Monocot II类F3'Hs的纯化选择相对分散。
为了进一步表征单子叶F3'Hs的进化动力学,我们将位点特异性模型应用于同一数据集,该模型允许ω值沿不同氨基酸位点变化。结果(表2)表明选择M1并不好(df = 1,p= 1.0)而不是中性的M1。在选择模型中,未发现任何氨基酸位点处于正选择状态。为了检测ω在特定分支的特定氨基酸位点上是否存在差异,我们还对分支位点模型进行了检测(表2)2).当Monocot II类F3'H的前景分支,选择模型A显示Monocot IIF3'H分支在正面选择(Ω2 = 8.19737;p < 0.05). LRTs showed that Model A was significantly better than its null hypothesis Model A Null, which specified ω2 = 1.0. Comparison of Model A with Neutral M1 (df = 2,p< 0.0001)也支持这些氨基酸位点正选择。相比之下,当Monocot类IF3'H被设定为前景分支,没有氨基酸部位可以在显着水平处被鉴定为正选择(表1).在这种情况下,选择模型A没有更好地适合数据集(df = 1,p= 1.0)比它的null假设模型A null。综上所述,自然选择评估表明Monocot II类F3'Hs处于显著的正向选择中,这只在Monocot II类F3'Hs中检测到,影响了该分支的特定氨基酸位点。
序列对准和蛋白质建模分析
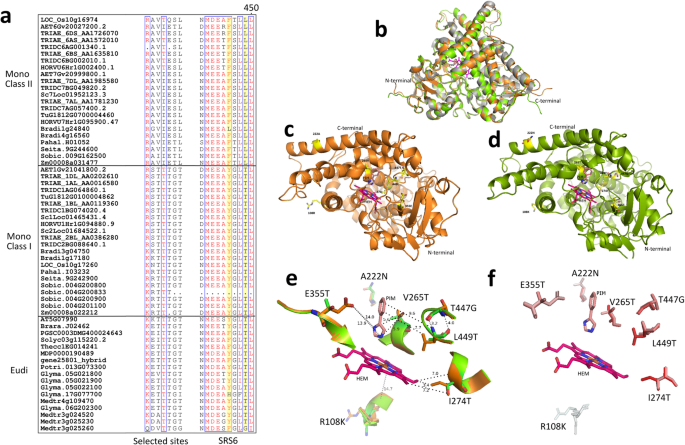
通过序列比对分析I类和II类F3'Hs之间的正面选定位点处的氨基酸取代。如图所示。4a,单子叶植物的I类和II类F3'Hs在7个位点中有6个显示出清晰的氨基酸替换。对于所有这些位点,I类F3'Hs与双二体F3'Hs相似,而与II类F3'Hs不同,表明I类F3'Hs与双二体F3'Hs的关系更为密切。F3'Hs的酶活性受关键氨基酸位点的影响。之前的研究已经鉴定了6个细胞色素p450蛋白的底物识别位点(SRS1-SSR6)。序列比对结果显示,7个氨基酸位点中有4个位于SRS4 (265 V, 274 T)和SRS6 (447S, 449 L),表明对II类f3’hs酶活性有直接影响。此外,还有两个氨基酸位点(图中黄色高亮部分)。4a)在SRS6中,F3 ' h和F3 ' 5'H的活性已被证明是关键的。如图所示。4一个,most Class II F3’Hs contained 446F-449L, whilst Class I F3’Hs displayed 446Y-449 T. Interestingly, the T446F substitution has previously been shown to be able to enable 5′-hydroxylation activity in some eudicot F3’Hs [27.].449位苏氨酸的保留也被认为是F3'Hs中3 ' -羟基化活性的关键[27.].值得注意的是,在第II类f3’hs中,447和449位氨基酸被鉴定为受正向自然选择的影响。综上所述,这些观察结果表明,II类F3'Hs具有很强的功能分化潜力。此外,在Sobic.004G200833中发现SRS6缺失S. Bicolor.,这表明I类F3'H可能无法正常工作。
为了进一步研究自然选择对II类F3'Hs酶功能的潜在影响,采用同源性建模方法建立了水稻II类F3'Hs (LOC_Os10g16974)和I类F3'Hs (LOC_Os10g17260)的三维结构模型。最近检测到的CYP76AH1晶体结构(PDB: 5YM3)与F3'H具有近同源性(约35%的氨基酸同源性),并将其作为模板。选择水稻同源基因是由于该物种的每一类都严格保留一个F3'H拷贝。如图所示。4B,大部分全长米F3'HS可以可靠地建模,除了N和C终端的短片段。预测未模拟的N末端肽被预测为疏水膜结合结构域,并且对酶功能没有直接影响。分析了7个氨基酸位点的空间位置,如图2所示。47个氨基酸位点中有5个被发现位于催化区域(图。4e),形成催化腔的一部分。其他两个氨基酸位点属于N末端区域并且位于外表面上。具体地,催化区中的5个氨基酸位点的侧链朝向结合的基板血红素和PIM,近距离(3.2Å〜14.0埃),并且可以对腔体积和基材结合直接影响。结构叠加显示在所有7个氨基酸位点的LOC_OS10G16974(I类)和LOM_OS10G17260(II类)F3'H之间显示氨基酸取代。值得注意的是,发现Loc_OS10G17260的催化区中的四个氨基酸位点具有苏氨酸残基(中性侧链),其用缬氨酸(疏水),异亮氨酸(疏水),谷氨酸(带电侧链)和亮氨酸(疏水)).所选部位的疏水性谱谱显示在图2中。4f. V265 T、I274T、T447G和L449 T的氨基酸取代不仅使LOC_Os10g16974和LOC_Os10g17260之间的疏水性发生了明显的变化,而且由于侧链的差异,也影响了催化空腔的大小。这些结果强烈表明,单子叶植物中两类F3'Hs分裂后可能出现功能分化,这是由积极的自然选择驱动的。
另外,基于CYP76AH1结构,通过在结束血红素分子和酶抑制剂PIM中选择氨基酸残基并选择氨基酸残基和酶抑制剂PIM来鉴定单焦F3'HS的催化位点。结果,鉴定了总共39个氨基酸位点(附加文件1).序列比对表明,这些推测的催化位点大部分在所有植物F3'Hs中都是严格保守的。然而,在第84位发现了I类和II类F3'Hs之间额外的氨基酸取代,该位置靠近酶底物。
转录分析
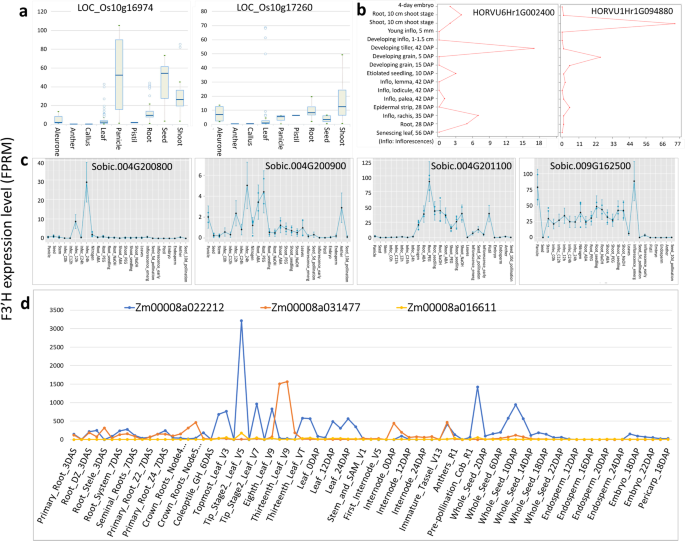
探讨I类和II类之间潜在的转录差异F3'hs.,在公共数据库中搜索单子台F3'HS的转录数据。在水稻中(图。5a),I类F3'H LOC_OS10G16974在穗,种子和芽中高度表达,根部相对较低,并且在其他组织中几乎表达,例如花药,愈伤组织,叶和雌蕊。与LOC_OS10G16974相比,II类F3'H LOC_OS10G17260通常在所有组织中显示出大量的表达,除了雌蕊组织,其中LOM_OS10G17260具有相对较高的转录。在芽和根部发现LOC_OS10G17260的最高表达。应该注意的是,虽然Loc_OS10G17260的中值表达水平在叶中非常低,但其表达在某些条件下可以达到特别高。在大麦中,还观察到I类和II类F3'HS的明显转录分歧(图。5b)。I级F3'H Horvu6HR1G002400被发现主要在开发分蘖中表达。Horvu6HR1G002400的轻微表达也在花序rachis和幼苗根系中观察到。相比之下,II类F3'H Horvu1HR1G094880主要在幼苗芽中表达,其次是早期阶段的显影谷物。Horvu1HR1G094880的少量表达在其他组织中发现。与高粱F3'HS有趣的观察(图。5c)是高粱I类f3’h Sobic.009G162500在花药、种子、胚乳、胚、雌蕊和早期花序等所有生殖组织中几乎没有转录,而出现花序除外。Sobic.009G162500在穗、茎、根、茎等营养组织中大量表达。而II类f3’hs Sobic.004G201100在雌蕊和幼子中表达量中等。Sobic.004G201100在根和茎的营养组织中也有显著的转录。另外两个串联重复的II类F3'Hs Sobic.004G200800和Sobic.004G200900在所有研究组织中表达非常低或几乎没有表达。值得注意的是,虽然Sobic.004G200800在高粱中几乎没有表达,但在病原菌侵染后,其在叶片中的表达量显著增加。在玉米中,I类F3'Hs和II类F3'Hs也表现出明显的转录差异(图。5d). Zm00008a031477 (Class I .)的最高表达F3'H)在叶片中发现,然后在根,细节和减数分裂的中度表达。相比之下,ZM00008A022212(II类F3'H)在授粉后第10天(DAP)在叶尖、丝部和全粒中表达量丰富,在聚叶、顶叶和成熟叶中表达量适中。有趣的是,另一类F3'HZm00008a016611在所有组织中几乎不表达。
讨论
已经表明,复杂代谢途径如黄酮化生物合成的演变,以在植物演化中发挥关键作用,帮助植物适应各种生物和非生物压力源[28.,29.].在Di-cotyledon植物中,类黄酮生物合成途径已被广泛研究,同时仅向单子麦片谷物作物支付了中等注意[1].考虑到小麦,稻米,大麦和玉米等谷物植物的事实,这种观察令人震惊,包括人类和动物最重要的食品和饲料来源。由于植物组织中的黄酮类化合物的普遍存在,并且黄酮类化合物是有效的抗氧化剂,在谷物植物中也建立了黄酮类生物合成和环境适应之间的密切关系[3.].以往关于类黄酮在谷物中的适应性作用的研究主要报道在大麦中[14,30.,31.,32.,33.,34.,35.], 小麦 [36.,37.,38.,39.,40]和米[41.,42.,43.],玉米[44.,45.,46.,47.].这些研究一般可分为两类:在生物或非生物胁迫条件下测定类黄酮含量变化和类黄酮生物合成基因的转录反应。鉴于谷类植物在进化过程中面临着持续的选择压力,在基因分子水平上的环境适应证据理论上也应该在类黄酮生物合成途径中普遍存在。本研究旨在从基因进化动力学的角度探讨禾谷植物与环境之间潜在的相互作用。我们重点研究了F3'H基因亚家族,该基因编码一种关键酶,控制类黄酮b环的羟基化模式。
谱系特异性进化是基因复制进化的重要机制。这是植物进化和多样化过程中常见的现象。本研究利用最新的大麦、黑麦、小麦等近缘小麦全基因组测序数据,对12个单子叶植物的F3'H基因进行了全面的全基因组调查和系统系统发育分析。我们发现假定的分布F3'HS在单子叶间高度不平衡。为麦芽汁,它的范围从2份t . urartu至9份t . aestivum.高波动性的F3'H对非-也观察到数目麦芽汁,S. Bicolor.和b . distachyon分别保留5份和4份。在这项研究中,我们确定了3F3'hs.与Vikhorev [24.],报告只有2份F3'hs..相比之下,我们确定了一个额外的F3'H(HORVU7Hr1G095900.47)与AET7Gv20999800和TuG1812G0700004460共线,位于Aet7和Tu7染色体上答:tauschii和t . urartu,分别。我们的结果与Lam [25.],谁也确定了3F3'hs.在大麦。发现单焦炭中F3'HS的扩展主要归因于分散的基因重复。该发现与从单胞菌植物中的花青素生物合成途径的另外三个基因家族(MYB,Myc和F3'5'H)进行的观察结果一致[48.].值得注意的是,我们发现串联重复和近端复制有助于专门的F3'HS扩展S. Bicolor.和o.苜蓿.此外,ZM00008A016611和ZM00008A022212中观察到一个WGD /节段重复z梅斯.这些观察结果可能反映了物种特有的进化史F3'hs.在这三种植物中。特别是,尽管5F3'hs.出席了S. Bicolor.基因组,一个F3'H由于临界基板识别位点SRS6的丢失,Sobic.004G200833可能不是功能。
本研究的系统发育分析显示单子叶植物F3'Hs已经进化成两个独立的谱系:Mono_F3'H Class I和Mono_F3'H Class II,而此前在水稻中鉴定的CYP75B4被归为II类F3'H。这一发现与Lam [25.],发现CYP75B4属于独立的系统发生支系,并获得了新的5 ' -羟化酶活性。在这里,我们展示了第一类和第二类之间的分歧F3'hs.已经起源于重复事件,谓词单像的物种多样化。此外,还可以在公共祖先期间为I类和II类F3'HS提出另外的复制事件麦芽汁,导致在每个f3’h类中形成2个分支麦芽汁.I类和II类F3'HS的已识别的复制事件麦芽汁可能表明在?的共同祖先中有一个共同的基因组复制事件麦芽汁.有趣的是,I类/ II类F3'Hs的保存和不同的分支麦芽汁两者在不同物种间表现出不均匀分布。而II类F3'Hs的2个分支全部保留麦芽汁在本研究所包括的物种中,发现I类次级分支不存在h . vulgare和答:tauschii.这种基因缺失可能是由于复制后基因丢失,这是植物基因组中常见的现象[22.,23.].在一起,这些发现表明,单子码中的F3'HS已经进行了挥发性谱系特异性基因重复和基因损失事件。
作为本研究的重点,进化动态分析表明,单焦型II F3'HS已被积极的自然选择显然影响。本研究中II级F3'H阳性选择的检测提出了直接证据表明黄酮类化合物生物合成途径的演变受环境选择的影响。F3'H和F3'5'H是Cytochrome P450超家族的近同源物,负责生产红色和蓝色花青素。在单子信F3'5'H基因亚家族中已经进行了类似的观察[48.],已经显示出积极选择来推动单独的出现F3’5是什么血统负责堆积蓝花青素的积累麦芽汁谷物。单子叶植物的选择F3’5是什么这可能是由于植物对强光或热胁迫的适应所致。阳性选择的检测F3'H和F3’5是什么Subfamilies对我们之前的假设提供支持,即基因进化水平的植物环境相互作用的证据应在类黄酮生物合成途径中普遍存在。与单码F3'5'H不同,其显示出增加蛋白质热稳定性的选择,I类和II类F3'Hs之间的分歧似乎与蛋白质结构水平的酶功能更相关。这由本研究中序列对准和蛋白质建模分析的结果支持。最近的报告证实了F3'H和F3'5'H系列中阳性选择的检测,该报告显示,光环境可能导致光保护酚类化合物在长期光谱过程中的差异[49.].
F3'H是单子叶植物中知之甚少的类黄酮生物合成基因之一。F3'H的特性仅在玉米中有报道Pr1[50.,51.],控制红葡萄酒颜色;用于米CYP75B3和CYP75B4 [25.,26.],底层3'-羟基化黄酮和三胞形成;和高粱[52.,53.,54.],参与3-脱氧菁生物合成。有趣的是,Cyp75b3和Cyp75b4分别分别被列为I类和II类,已被证明具有发散的酶活性[25.,26.].特别是,II类F3'H CYP75B4显示出新的金杉醇特异性5 ' -羟化酶活性,并在tricin生物合成中发挥不可或缺的作用[25.].提出了CYP75B4的5'-羟化酶活性的募集是有助于在草地和单子叶中的三胞苷衍生代谢物的患病率有助于在植物防御机制中具有重要功能。这些观察结果表明,CYP75B4由于蛋白质功能分歧,可以获得新的生物学作用。实际上,发现包括CYP75B4的单焦点的II类F3'HS受到阳性自然选择的影响,作用7种特异性氨基酸位点。蛋白质建模和氨基酸性能分析表明,这些选定的位点中的大部分位于F3'H的催化腔中,并且可以对底物特异性进行直接影响。这些发现可以对CYP75B3和CYP75B4之间观察到的酶活性差异提供进化和蛋白质结构解释[25.,26.].值得注意的是,F3 ' h和F3 ' 5'H之间功能差异的分子基础已经被很好地描述[27.].显示SRS6中的两个关键位点的氨基酸取代检测到3'-和5'-羟化酶活性。有趣的是,我们研究中的序列对齐表明,I类和II类F3'HS在这两个地点显示不同的氨基酸取代。有趣的是,II类F3'HS含有位点446的PHE,其通常观察到F3'5'HS。相比之下,I级F3'HS保留了该位置的Tyr,与先前表征的F3'Hs保持一致。这些结果与CYP75B4的观察到的5'-羟化酶活性一致,II类F3'H。相反,对于CYP75B3(I类F3'H)观察到没有5'-羟化酶活性[25.].我们的分析为CYP75B4中的5'-羟化酶活性提供了蛋白质结构解释。这里呈现的结果类似于制定的类似观察结果菊科,其中部分CYP75B蛋白被鉴定为与F3 ' hs聚类,但显示F3 ' 5'H活性[55.].我们的研究表明,整个单码级II F3'HS可能已获得新的克莱氏醇特异性5'-羟化酶功能。
除了在蛋白质结构水平上的差异外,基因复制后的功能差异也可能在转录水平上发生。事实上,基因表达分析已经被广泛应用于研究类黄酮生物合成基因对各种生物和非生物胁迫的响应。在这些研究中,有3个F3'Hs (Class II)在高粱叶片切割胁迫下的差异表达[53.,54.),两个组织F3'hs.(第I及II类)大麦[24.]及米中两个F3'Hs(第I类及第II类)[25.的报道。有趣的是,在所有情况下,I类和II类表现出不同的表达特征。这一观察结果与我们的转录分析结果一致,该分析也涵盖了玉米中的三个F3'H平行对数。通过将这些数据组合在一起,尽管对于Class I和Class II有一个通用的表达式模式F3'hs.跨越这些单子叶植物的物种目前还无法绘制,两者的转录差异较大F3'H血统可能被提出。在许多植物基因家族中已经报道了在蛋白质结构和基因转录水平上观察到的基因功能差异[56.,57.,58.,并通过复制巩固了基因进化理论[22.,23.].在这项研究中,不同F3'h旁边蛋白酶的表达可能已经发展了一种特异性特异性概况,满足不同植物面临的特定环境挑战。例如,我们发现了其中一个我F3'hs.从高粱中鉴定出的蛋白对病原菌感染表现出明显的转录反应,但在正常生长条件下完全不表达。这一结果被先前的研究所证实F3'hs.在高粱52.,53.,54.].除了单子叶植物,也有报道称几种双子叶植物中辅生F3 ' hs和F3 ' 5'Hs的转录差异,如茶树叶中的F3 ' hs [59.,进一步证实了基因转录水平上的功能差异是基因复制进化过程中常见的现象。对这一现象的一个进化解释是,类黄酮生物合成基因复杂表达谱的发展是环境选择压力作用于不同植物的结果,反过来也提高了植物在自然界的生存能力。
结论
基于基因组调查的结果,系统内发生,进化动力学和蛋白质结构建模分析,我们发现单子叶F3'hs.在单胶质植物中接受了特异性特异性基因重复和基因流失事件。我们得出结论,单像F3'HS已经进化为两种独立的衬里(Mono_f3'h等级I和II类),在单码植物的共同祖先期间的基因重复之后。单焦度F3'H类II的功能分歧受到阳性自然选择的影响,作用于几种特定氨基酸位点。这一个mino acid substitutions at these selected sites and other sites in SRS6 displayed high potential to affect the substrate binding of F3’Hs, and may have contributed to the recruitment of chrysoeriol-specific 5′-hydroxylation activity in F3’H Class I, as evidenced by CYP75B4 in rice. In addition, transcriptional divergence between F3’H Class I and Class II have also been observed. Taken together, our study revealed clear evidence of plant-environmental interaction for the flavonoid biosynthetic pathway at the gene evolutionary level.
方法
序列检索和真正的F3'h同源物识别
由于F3 ' 5'H与F3 ' h之间的同源性非常接近,因此我们采用了之前研究中描述的方法来鉴别真正的F3 ' h同源物[48.].包括12种单子叶作物和12种双子叶植物。利用之前在大麦中鉴定的CYP75B3同源物的氨基酸序列查询BLASTP (E-value阈值:1e-30)针对单子叶植物基因组的公共数据库。远程F3'H同源物也从下层植物苔藓(P. Pattens.).整个过程如下:使用MEGA7.0软件开发初步neighbor Joining树,包括上述所有同源物[60.].使用Muscle进行序列比对[61.].使用的替代模型是p距离。为树评估执行了1000次自动启动迭代。选择含有先前报道的F3'HS(CYP75B3&CYP75B4)的不同的系统发育枝被选为真正的F3'HS,用于进一步分析。
系统重建
利用上述鉴定的f3’h的CDS序列和f3’h的远端同源序列进行系统发育重建。使用Muscle对CDS序列进行密码子序列比对,共8次迭代[61.].手动检查所得到的序列对齐以去除显着的对准间隙和5'信号肽。在Beast2中实施的贝叶斯模拟搜查了系统发育[62.]在严格的分子时钟假设下。使用未链接的替代模型Yule + G(5个类别)。单一马尔可夫链 - 蒙特卡罗链条运行1000,000代。每100代采集树木,用1,000代预烧纳直到收敛。通过丢弃的前1000年,TreeNnotator推断出最终的系统发育树。本研究中的所有系统发育树木都是使用FIGTRE的注释(http://tree.bio.ed.ac.uk/software/figtree/).
基因结构分析
CDS和基因组序列F3'hs.从目标单子叶植物的基因组数据库中下载。使用GSDS v2.0在线工具构建基因结构图(http://gsds.cbi.pku.edu.cn/).利用本研究建立的单子叶F3'Hs系统发育子树作为输入,根据系统发育关系对基因结构进行聚类。
基因复制模式和共线性分析
McScanx包[63.]用来描述基因复制模式。从公共数据库下载原始基因组数据,并进行进一步处理,生成MCScanX的输入文件。使用独立的NCBI-BLAST-2.2.29工具进行种内和种间基因组比较E-value阈值1e-05。采用全基因组对全基因组BLAST进行基因复制模式鉴定。为了鉴定共线性基因对,将不同物种的基因组数据进行all-vs-all BLAST组合。
序列比对和蛋白质建模
使用ESPRIPT 3.0进行氨基酸序列对准和注释(http://espript.ibcp.fr/ESPript/ESPript/index.php)以水稻F3'Hs的氨基酸序列为查询源,利用BLASTp对PDB数据库进行同源结构模板鉴定。利用氨基酸同源性最高的CYP76AH1 (PDB: 5YM3)蛋白结构进行模型开发。通过使用modeler服务器进行同源建模,建立了F3'Hs的蛋白质模型。每个蛋白生成5个结构模型。根据离散优化蛋白能量(Discrete Optimized Protein Energy, DOPE)值最低,GA 341评分为1,选择最佳模型,表明模型的可靠性。最终模型通过使用PROCHECK (http://www.ebi.ac.uk/thornton-srv/software/PROCHECK).分子可视化使用PyMOL(版本1.3r1)进行。薛定谔,LLC)。
转录数据挖掘
目标的转录数据F3'H从个体数据库中提取基因:http://expression.ic4r.org/index),大麦(https://apex.ipk-gatersleben.de/apex/f?p=284:10),玉米(https://www.maizegdb.org/)及高粱(http://sorghum.riken.jp/morokoshi/home.html.).
数据和材料的可用性
在当前的研究中生成和/或分析的数据集可在Figshare存储库中找到,网址为https://figshare.com/s/a811638eb6fcd3496d2f.
缩写
- CD:
-
编码域序列
- 气:
-
Chalcone异构酶
- CHS:
-
Chalcone合成酶
- DAP:
-
授粉后的几天
- 涂料:
-
离散优化蛋白质能量
- F3'H:
-
黄酮类化合物3'-羟化酶
- F3’5是什么:
-
类黄酮3 ' 5 '羟化酶
- LRTS:
-
似然比检验
- SRS:
-
基底识别位点
- WGD:
-
全基因组重复
参考
- 1.
黄酮类化合物生物合成途径的研究进展[j]。中国生物医学工程学报。2017;68(15):4013-28。
- 2。
黄酮类化合物的生物合成。遗传学、生物化学、细胞生物学和生物技术的彩色模型。植物杂志。2001;126(2):485 - 93。
- 3.
Khlestkina艾克。类黄酮的适应作用:以谷物为重点。谷物研究。2013;41(2):185-98。
- 4.
Dykes L, Rooney LW。谷物中的酚类化合物及其对健康的益处。中国粮食科学。2007;52(3):105-11。
- 5。
黄酮类化合物在光保护中的作用:新的证据和过去的教训。植物生理学报。2013;72(11):35-45。
- 6。
郭j,汉w,王mh。紫外线和环境胁迫参与诱导和调控蒽皂苷生物合成:综述。AFR J BIOTECHNOL。2008; 7(25):4966-72。
- 7。
古尔德KS。大自然的瑞士军刀:花青素在叶子中的多种保护作用。生物技术学报。2004;2004(5):314-20。
- 8。
Bradshaw HD, Schemske DW。在猴花中,花颜色位点的等位基因替换产生授粉者转移。大自然。2003;426(6963):176 - 8。
- 9。
基因是如何绘制花朵和种子的。植物科学进展,1998;3(6):212-7。
- 10.
莫伊,纳格尔C,泰勒LP。Cholcole合成酶突变体的生化辅作定义了功能性花粉中黄酮的作用。P Natl Acad Sci USA。1992年; 89(15):7213-7。
- 11.
泰勒LP,高手PK。花粉萌发和管生长。annu rev植物phys。1997年; 48:461-91。
- 12.
内质网:生长素生物学中的上升隔间。植物杂志。2010;154(2):458 - 62。
- 13。
Lewis DR, Ramirez MV, Miller ND, Vallabhaneni P, Ray WK, Helm RF, Winkel BSJ, Muday GK。生长素和乙烯通过不同的转录网络诱导黄酮醇的积累。植物杂志。2011;156(1):144 - 64。
- 14。
Himi E,Yamashita Y,Haruyama N,Yanagisawa T,Maekawa M,Taketa S.Ant28基因用于原型霉素合成的编码R2R3 MyB域蛋白(HVMYB10)高度影响大麦的粒度休眠。Euphytica。2012; 188(1):141-51。
- 15。
Hyun JW, Chung HS。从水稻中提取的花青素和malvidin。heugjin朱比通过抑制G(2)/M期和诱导细胞凋亡介导对人单核细胞白血病细胞的细胞毒性。[J] .食品科学,2004;52(8):2213-7。
- 16。
赵c,giusti mm,malik m,moyer mp,magnuson ba。富含种子酵母素提取物对结肠癌和非致盲肠细胞生长的影响。j农业食品化学。2004; 52(20):6122-8。
- 17。
Tsuda T, Horio F, Osawa T. Cyanidin 3-O-beta-D-glucoside抑制大鼠在酵素治疗期间一氧化氮的产生。中国营养科学。2002;48(4):305-10。
- 18。
苯丙素生物合成。摩尔。2010;3(1):2 - 20。
- 19。
Ferreyra MLF,Rius SP,Casati P.Flavonoids:生物合成,生物功能和生物技术应用。前植物SCI。2012; 3:222。
- 20.
Tanaka Y,Brugliera F.花卉颜色和细胞色素P450。Philos T R Soc B. 2013; 368(1612)。
- 21.
Bak S,Beisson F,Bisshop G,Hamberger B,Hofer R,Paquette Sm,Reichhart DW。细胞学p450。拟南芥书。2011; 9:E0144。
- 22.
张生理改变。基因复制的进化:更新。生态学报。2003;18(6):292-8。
- 23.
鞭毛le,wendel jf。基因重复与植物进化新颖性。新植物。2009; 183(3):557-64。
- 24.
Vikhorev Av,Strygina Kv,Khlestkina Ek。在大麦基因组中复制的黄酮3'-羟化酶和黄酮类化合物3',5'-羟化酶基因。peerj。2019; 7:E6266。
- 25.
关键词:水稻,Tricin生物合成途径,细胞色素P450 75B4,金茶酚5 '-羟化酶植物杂志。2015;168(4):1527 - 36。
- 26.
2 .黄酮类化合物3 ' -羟化酶的分子和生物化学分析及其在稻米中合成黄酮类化合物中的作用。中华医学杂志。2016;17(9)。
- 27.
黄酮类化合物3 '-羟化酶与3 ',5 '-羟化酶功能差异的分子基础鉴定。2月。2007;581(18):3429 - 34。
- 28.
翁JK。复杂性的进化路径:新陈代谢的观点。新植醇。2014;201(4):1141 - 9。
- 29。
黄酮类化合物:介导植物对其栖息地适应的代谢网络。植物科学学报。2014;5:620。
- 30.
大麦胚层及其类黄酮含量在抗镰刀菌感染中的作用。Hereditas。1997;126(2):147 - 60。
- 31。
Christensen AB, Gregersen PL, Olsen CE, Collinge DB。一种类黄酮7- o甲基转移酶在大麦叶片中表达,以应对病原菌的攻击。植物学报。1998;36(2):219-27。
- 32.
关键词:UV-B,酚类化合物,青稞,初生叶,DNA损伤植物化学。2003;64(1):243 - 55。
- 33.
关键词:大麦,盐胁迫,差异表达基因,基因测序,基因测序生物技术学报。2009;8(7):1326-31。
- 34.
李志刚,李志刚,李志刚,等。镉对大麦幼苗类黄酮含量的影响[J]。植物生态学报。2005;51(11):513-6。
- 35.
黄秋生,黄秋生,黄秋生,黄秋生,黄秋生,黄秋生。过度光照和干旱胁迫下女贞子叶片中黄酮类化合物和羟基肉桂酸的积累差异。新植醇。2004;163(3):547 - 61。
- 36.
Giovanini MP,Puthoff DP,Nemacheck JA,Mittapalli O,Saltzmann KD,欧姆HW,Shukle Rh,Williams Ce。小麦对黑森州的基因对小麦防御缺乏古典氧化爆发。莫尔植物微生物。2006; 19(9):1023-33。
- 37。
李晓玲,吕旭,王晓华,王丽华,张明敏,任建军。非生物胁迫对紫色小麦花青素积累和粒重的影响作物与牧草学报,2018;69(12):1208-14。
- 38。
水稻籽粒颜色基因(R)对小麦籽粒休眠和胚对ABA敏感性的影响。中国农业科学(英文版)2002;53(374):1569-74。
- 39。
马德元,孙德兴,王春英,李永刚,郭TC。干旱胁迫下小麦叶片中类黄酮生物合成基因的表达及积累植物生理学报。2014;80:60-6。
- 40.
水杨酸对小麦类黄酮合成途径的诱导作用不同。植物学报2016;7:1447。
- 41.
郝振宁,王丽萍,何艳萍,梁建国,陶瑞祥。水稻抗条纹病毒和小褐飞虱防御基因表达及抗氧化酶活性研究植物生理学报。2011;49(7):744-51。
- 42.
thal N, Reddy AR.水稻类黄酮通路基因OsDfr和OsAns是由脱水、高盐和ABA诱导的,它们含有与转录激活因子OsC1-MYB相互作用的胁迫响应启动子元件。植物科学。2004;166(6):1505 - 13所示。
- 43.
籼稻品种中脯氨酸和类黄酮的盐胁迫差异积累。acta photonica sinica, 2009;41(5): 2497-506。
- 44.
Casati P,Walbot V.基因表达分析响应玉米基因型中的紫外线辐射,不同的类黄酮含量。植物理性。2003; 132(4):1739-54。
- 45.
关键词:玉米,低温胁迫,苯丙氨酸,花青素,转录本丰度,色素沉着足底。1994;194(4):541 - 9。
- 46.
Marrs KA, Walbot V.玉米谷胱甘肽s -转移酶Bronze2基因的表达和RNA剪接受镉等胁迫的调控。植物杂志。1997;113(1):93 - 102。
- 47.
陈志强,陈志强,陈志强,等。不同铝胁迫下玉米酚类化合物的结构和铝诱导模式。生物化学学报。2009;30(11):1486-90。
- 48.
贾Y,Selva C,Zhang Y,Li B,Lee Ma,Broughton S,Xiaoqi Z,Sharon W,Penghao W,Cong T等:在谷物中揭开蓝色花青素的进化起源。正在审查,发表。2019年。
- 49.
Oksana S,Marek Z,Susanne N,Peyman Mt,Marian B.不同颜色阴影下的幼苗预测改变了在后期生长阶段的含氮叶片中酚类化合物的积累。Environ Exp Bot。2019年; 165:30-8。
- 50.
Larson R, Bussard JB, Coe EH。玉米中基因依赖的类黄酮3 ' -羟化。生物化学麝猫。1986;24(7 - 8):615 - 24。
- 51.
玉米中3-脱氧类黄酮生物合成调控因子P1调控类黄酮3 '-羟化酶的表达。BMC Plant Biol. 2012;12:196。
- 52。
在高粱中胚轴合成3-脱氧花青素类黄酮3 '-羟化酶的表达。植物学报(自然科学版);2004;32(2):101-13。
- 53。
石昌华,朱立科,叶文康,罗志强。高粱花青素和3-脱氧花青素生物合成中2种黄酮-羟化酶cdna的表达差异。植物生理学报。2006;47(10):1412-9。
- 54。
Mizuno H, Yazawa T, Kasuga S, Sawada Y, Kanamori H, Ogo Y, Hirai MY, Matsumoto T, Kawahigashi H.黄酮合成酶II和类黄酮3 '-羟化酶的表达与高粱褐色受损叶片的颜色变化相关。植物学报2016;7:1718。
- 55。
Seitz C, Eder C, Deiml B, Kellner S, Martens S, Forkmann G.黄酮类化合物3 '-羟化酶和黄酮类化合物3 ',5 '-羟化酶cdna的克隆、功能鉴定和序列分析揭示了黄酮类化合物3 ',5 '-羟化酶在菊科中独立进化。植物学报。2006;61(3):365-81。
- 56。
贾y,黄液,甜蜜曼,福特厘米。植物山梨醇脱氢酶进化史的新见解。BMC植物BIOL。2015; 15:101。
- 57.
li hti-Shiu MD, Uygun S, Moghe GD, Panchy N, Fang L, Hufnagel DE, Jasicki HL, Feig M, Shiu SH.拟南芥全基因组复制转录因子功能分化和衰减的分子证据。植物杂志。2015;168(4):1717 - 34。
- 58.
李志强,李志强。植物基因复制的研究进展。植物杂志。2016;171(4):2294 - 316。
- 59.
Wei K,Wang Ly,Zhang CC,Wu Ly,Li Hl,Zhang H.转录组分析显示关键的黄酮3' - 羟基化酶和黄酮类化合物3',5' - 羟基化酶基因,影响二羟基化与三羟基化的儿茶中的比例山茶花。Plos一个。2015; 10(9):E0137925。
- 60.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。生物化学学报。2016;33(7):1870-4。
- 61.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32(5):1792-7。
- 62.
Bouckaert R,Heled J,Kuhnert D,Vaughan T,Wu Ch,谢D,Suchard Ma,Rambaut A,Drummond AJ。野兽2:贝叶斯进化分析的软件平台。PLOS计算BIOL。2014; 10(4):E1003537。
- 63.
王yp,唐hb,debarry jd,tan x,李jp,王xy,李th,金赫兹,玛勒b,gu h等。McScanx:一种用于检测和进化分析的工具包,对基因同步和共线性分析。核酸RES。2012; 40(7):E49。
确认
我们感谢植物研究团体向公众提供基因组数据和转录数据。
资金
本项目由中国粮食研究开发公司(GRDC)“大麦对酸性土壤改良适应性”项目(项目编号:UMU00046)资助。
作者信息
从属关系
贡献
CL和YX监督了这个项目,并对手稿的发展提供了有价值的意见。YJ构思了这项研究并开发了手稿。YJ和BL检索基因序列。BL和YJ负责基因结构分析。YJ进行了系统发育、进化动态和蛋白质模型分析。YJ, YZ和XZ进行了基因复制、共同和基因表达数据挖掘分析。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
额外的文件1:
植物F3'Hs的氨基酸序列比对。(PDF 50 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
贾勇,李斌,张勇。et al。单子叶类黄酮3 ' -羟化酶基因家族的进化动态分析揭示了植物与环境相互作用的证据。BMC植物BIOL.19,347(2019)。https://doi.org/10.1186/s12870-019-1947-z
已收到:
接受:
发表:
关键字
- 黄酮类化合物
- 黄酮类化合物3'-羟化酶
- 花青素
- 谷物作物
- 演化动力学
- 基因演变
- 积极的自然选择
- 工厂环境交互