摘要
背景
基因组宽协会研究(GWAs)是鉴定与作物物种中各种重要特征相关的定量性状基因座(QTL)和因果单核苷酸多态性(SNP)/基因的强大工具。通常,作物中的Gwas使用自交系面板进行,其中测量相同近交的多重重复,并且将平均表型作为响应变量。在这里,我们描述并评估了用于在单个植物上进行GWA的单株GWA(SP-GWA),这不需要近交联系统。相反,SP-Gwas依赖于从随机交配群体中取样的个体植物的表型和基因型。重要的是,我们展示了SP-GWA如何能够有效地结合批量分析分析(BSA)实验,以便迅速证实有重要的SNP。
结果
在本研究中,我们使用了20世纪60年代从南密苏里州的一个农场收集的Shoepeg玉米地方品种作为开放授粉品种,来评估sp-GWAS与BSA结合是否能够有效和强有力地检测株高(PH)的显著snp关联。植物在8个地点生长了2年,共768个个体进行了sp-GWAS的基因型和表现型。对768个个体的306 k多态性标记进行关联分析,检测到25个显著的SNPs (Pph值≤0.00001)。我们的单株GWAS结果通过ph值的bulk separation analysis (BSA)进一步验证。选择高株和矮株作为单独的群体,对同一群体进行BSA测序。该方法鉴定了37个株高基因组区域。在GWAS的25个显著的SNPs中,有3个最显著的SNPs与BSA鉴定的区域共定位。
结论
总体而言,该研究表明,与BSA耦合的SP-GWA可以是用于检测有效的SNP和识别候选基因的有用工具。该结果对于具有关联面板不容易获得的物种/群体特别有用。
背景
玉米(Zea Mays。L.)是全球最广泛种植的作物之一,因为它对工业食品,饲料,燃料和原料的重要性[1].此外,它也是一个重要的模式物种,具有巨大的表型和分子多样性。在不同的研究中,即使使用适度的种群大小,也观察到数百万分离标记,分子多样性是明显的[2,3.,4].育种家已经取得了显著的成功,捕捉到了这种多样性,开发出了具有更好适应和生产特性的现代玉米品种[5].为了继续开发改良品种,鉴定与重要性状相关的基因或位点是利用这些基因在下游育种中使用的许多步骤中的第一步[6].
株高是玉米等作物的重要农艺性状。育种家已经确定了PH值、粮食产量和生物量之间的相关性[7,8,9].PH值是一种复杂的数量性状,Fisher的无穷小模型对此进行了解释,该模型假定它是由许多影响小的基因控制的[10.,11.].此外,PH值是一种高度遗传性状,尽管只有一个与PH值相关的基因座的子集已被确定[12.,13.,14.,15.,16.].由于株高在农学上的重要性,科学家们经常使用传统的QTL定位方法对其进行研究[17.,18.,19.].已被证明QTL映射是一种强大的方法,以鉴定含有与重要特征相关的基因的基因组区域[20.,21.].例如,一些基于连锁定位的QTL研究已经确定了至少5-12个与PH相关的位点[17.,18.,19.].总体而言,Gramene显示了超过219个与玉米PH相关的qtl (http://archive.gramene.org/qtl/).以前关于pH的研究鉴定了吉伯塞林(GA)和芸苔类药物(Br)作为涉及茎伸长的主要激素[22.,23.,24.,25.].此外,生长素生物合成和信令也在调节茎长中发挥关键作用[26.].然而,QTL作图方法有其局限性,首先需要建立作图群体,这是一个缓慢且资源密集型的过程。此外,绘制图谱的分辨率通常很低,通常包括几个厘米器官,包括几百个基因。另一个限制是QTL作图只能捕捉到许多农艺性状的一小部分表型变异,而这正是将杂交形成作图群体的两个亲本区分开来的原因[27.,28.].
现代高通量基因分型技术使单核苷酸多态性(SNPs)的鉴定更加容易[29.].SNP标记通常用于进行基因组宽协会研究(GWA)以鉴定与包含许多生理,分子和细胞特征的定量性状的变异相关的基因[30.].GWAS通过利用一个物种内的遗传多样性来识别表型。在种群中捕获的历史重组事件大大提高了制图的分辨率。然而,作物中的大多数GWAS以前是使用由在多次重复中表型的自交系组成的群体进行的[31.,32.,33.,34.].采用f - 1关联作图法(FOAM)对4417份玉米地方品种杂合位点进行了GWAS分析。最初的FOAM方法包括一个繁殖步骤,在此过程中,每个地方种的加入都与少量的单杂交杂交雌性杂交,并对每个家庭作为一组重复的后代进行表型分析[35.].在人类和动物GWAS中,个体的非复制表型是常见的,复制基因相同的个体可能是困难的或不可能的[36.,37.].为了减少测量误差而进行重复实验的能力是可能的,而且在自相容的植物中是相对直接的。正因为如此,使用单株表型在作物植物中不是标准做法。但是,如果单独的植物表型可以用于植物的GWAS,这有可能大大减少完成实验所需的时间和资源。
大量分离分析(BSA)是一种替代方法,利用全基因组标记数据来识别复杂性状的随意基因[38.].植物中的BSA最初用于检测分离群体中的标记以鉴定抗病抗性基因[39.].在[33., DNA文库的构建是利用表型差异明显的两个亲本杂交产生的表型极端后代的F2集合样本。然后,对标记进行DNA变异的筛选,这些DNA变异的频率在不同池之间存在显著差异。BSA已经被证明是一种有用的技术,在作物物种中检测大效应的QTL,如抗非生物/生物胁迫或绘制定性突变体[40,41.,42.].类似于早期涉及双亲本或其他结构化种群的BSA研究,通过利用序列数据,BSA的改进实现可以在非结构化种群上执行。这种方法以前是由[43.在那里它被称为xp-GWAS。
在这里,我们使用一个被称为Shoepeg的玉米地方品种进行GWAS,这是一个未改良的群体,随机交配的个体适应环境,并具有该地方品种特有的形态属性。由于分离是任何测绘研究的基本前提,理想的地方品种包含了整个基因组的分离变异,因为地方品种是通过随机交配产生的,通常是异质性的。因此,在任何位点上许多个体都可能是纯合或杂合的。本研究以株高作为中等复杂性状的模型,最终目的是将该方法应用于更困难或更昂贵的表型。我们将GWAS应用于单株基因型和表型,因此将其称为单株GWAS (sp-GWAS),因为在关联分析中,分离的单株植物是分型和表型的。正如我们所展示的,sp-GWAS的一个重要好处是,它可以有效地与BSA结合,快速和独立地确证候选snp。
在此,我们描述了这种SP-GWAS管道作为模型特征的pH的应用。我们证明,由于廉价的基因分型,适度的基因分型和表型个体,以及中度至高遗传性的性状:pH,我们的管道涉及SP-GWAS和基于BSA的SNP证据,可以用于成功和有效地识别候选基因座.我们的管道鉴定的基因座包括先前鉴定的候选基因,通过在同一群体上使用极端表型进行BSA进一步验证。
结果
GWAS和BSA管道概述
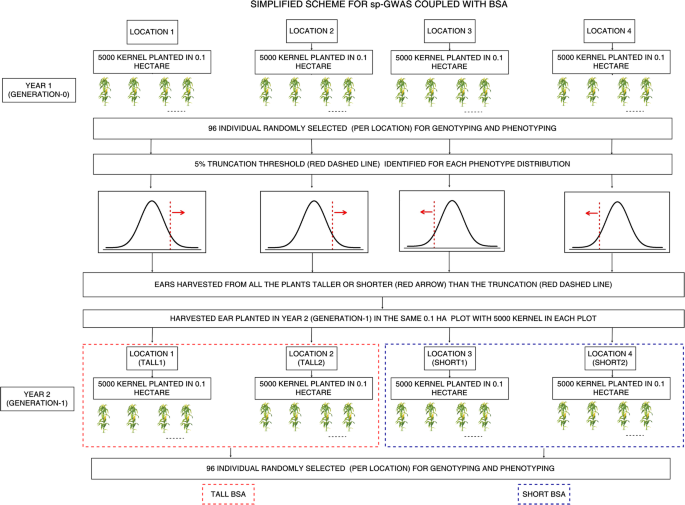
本手稿的方法部分详细描述了我们有效地将sp-GWAS与BSA结合以快速识别和确证候选性状相关SNPs的方法。因此,我们在这里只包含了该方法的概述,以及演示我们的管道的总结性图(图。1).在第0代,我们从Shoepeg种群中分别种植了5000株植物在4个独立的0.1 ha地块(总共20000株)。在每个样地,96株(总共384株)单株采用GBS进行株高表型和基因分型[44.].根据这些植物的表型分布,针对0.1 HA图中的每一个鉴定〜5%截断阈值,并且收获比截断阈值更高(2个图)或更短(2个图)的耳朵的耳朵。在第1代中,来自收获的耳朵的种子再次在四个0.1公顷地块中生长,每个植物中有5000个植物,96个植物/地块进行基因分型和表型(总共384个)。所有768(384×2)表型和基因分型植物用于SP-GWA,从发电-1中的96株/地图中计算的等位基因频率用于表示BSA的表型极端生成-0植物的等位基因频率。在线提供实施我们的管道和分析的脚本(https://github.com/abi01/sp-GWAS).
sp-GWAS与BSA耦合的管道原理图。第1年(0代):在4个地点种植5000株(共20000株)~ 0.1 ha地块,每个地点随机选择96株(共384株)进行基因分型和表型分型。根据96株植物的表型分布,鉴定了每个位点的~ 5%截断阈值。所有高于截断阈值的植株(位置1和2)或较短的植株(位置3和4)的穗被采收。第2年(第1代):从第1年(第0代)收获的种子(5000粒)在同一地点再次种植,每个地点96株(共384株)的基因型和表型与第1年相同。这些种群现在是根据选择制度命名的;第1代tall1,第1代tall2,第1代short1和第1代short2。共768株(384 × 2)表型和基因型植株进行关联分析。从第1年选出的个体的后代被用于改良的整体分离分析,使用高和短群体来定义在硅块
表型评价
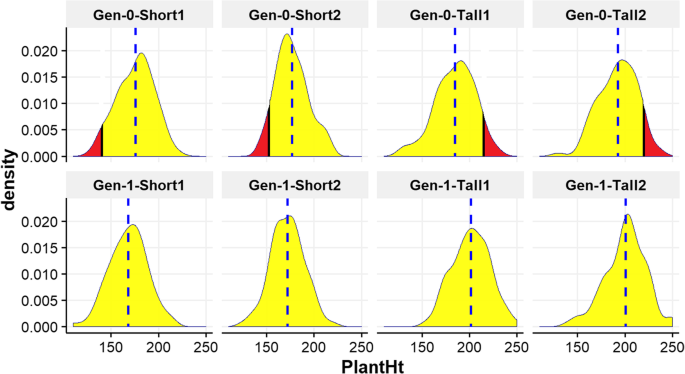
我们在两代和四个地点测量了768个个体的pH值:2016年的第384位,2017年的一代人和384年。在表格中提供了对所有环境和两代人的pH的描述性统计数据1.两代Shoepeg PH在所有四个地点的表型分布均显示出广泛的变异和近似正态分布(图。2).平均遗传率采用GCTA (h2GCTA)估计为0.7463,表明在PH中检测到的主要表型变异是由遗传因素引起的,因此适合进行关联分析。实现的遗传力也使用育种者的方程(h2废话)[45.].如更详细的(附加文件1),不同地点和年份选择环境的环境差异使我们在这种情况下应用育种方程来估计遗传力变得复杂。即便如此,使用这种方法,我们还是保守地估计了一个平均值h2废话为0.31的株高(附加文件1).我们对自己更有信心h2GCTA估计比我们h2废话遗传性估计,尽管两者都显示了可以利用用于映射的基因型和表型之间的关系。其他研究人员在具有类似秘友和样本尺寸的动物群体中成功实施了GWA [46.,47.].
基因组宽协会分析
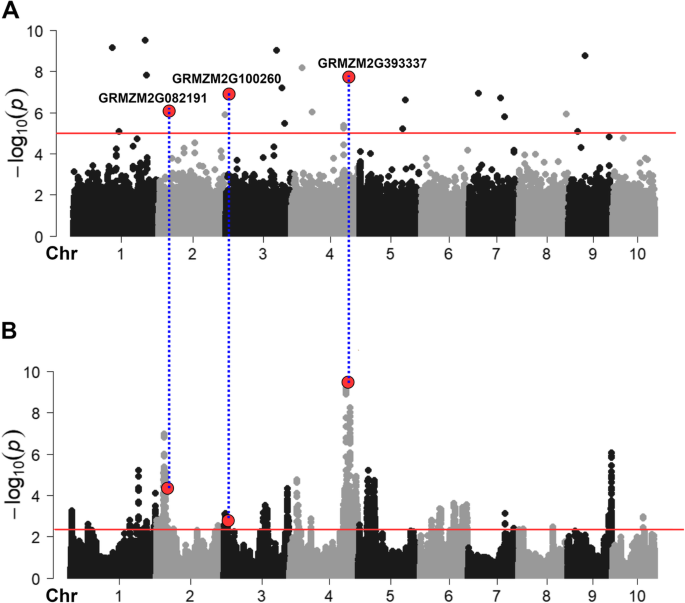
主成分分析(PCA)在归一化后没有揭示整个Shoepeg种群或跨代(0或1)或选择机制(高或短)的实质性种群结构(补充文件)2).这是预期的,因为Shoepeg是一种随机交配人群,因此不应包含主要的混合物特征或反映几代漂移转基因植物。因此,我们仅使用GWAS模型中的协调因子(周期0和循环1)和选择制度(高大和短路)。使用FarmCPU进行GWA。总共25例与植物高度相关的25个重要的SNP(P ≤ 0.00001) were detected by GWAS (Fig.3.一个额外的文件3.、表2).这与之前在玉米中进行的PH相关性研究相比是低的[12.,48.这可能是由于Shoepeg群体的遗传多样性与广泛多样性组相比受到限制。这25个显著的snp分别解释了Gen0和Gen1的48个和36%的方差。在1号染色体上发现了两个最显著的SNPsP分别为3.15e-10和7.17e-10。显著单核苷酸多态性的效应大小为−5.77 ~ 6.47 cm,平均效应大小为0.63 cm。
批量隔离分析
BSA作为我们sp-GWAS管道的一个有价值的验证附加组件,以最小的额外成本提供了最有前途的GWAS snp的确认书。(无花果。1).本研究中的BSA通过选择前5%作为高pH块和底部5%,从GEN-0的短pH块中选择5%。比较了总共243,303个SNP,以在循环1中基因分型的192个个体之间的等位基因频率差异,这代表了循环中最高和最短的个体。等位基因频率估计基于一个在硅片里大部分个人(方法中的细节)。确定了37个地区的1201个重要标记。具有0.05或更小的频率的显着BSA SNP是总体显着的SNP的〜2%。在染色体2和4上发现两种重要区域,这些有关分别包含15.7和28.3 MB(表3.;无花果。3.b)。
候选基因的识别
根据B73参考基因组的信息v3 (49.[25种GWAS鉴定的SNP中的9个位于基因模型中。其中,四个位于翻译区域中,剩下的五个是内含物。基于MaizeSequence提供的基因注释信息(http://ensembl.gramene.org/Zea_mays/Info/Index)及MaizeGDB (http://www.maizegdb.org/gbrowse),我们进一步评估了位于重要位点附近的候选基因的潜在功能。14个候选基因位于25个重要SNPs的150 kb范围内,其中9个功能未知。
我们根据同源性鉴定了几个有希望的候选基因拟南芥蒂利亚纳与植物身材有关的基因.玉米TCP-transcription factor39,GMZM2G170232位于染色体4上的重要SNP下游(位置36,939,527),是矫形器的29kbtcp-transcription-factor1 (tcp1)的A. Thaliana..染色体4上的另一个重要的SNP位于GMZM2G393337内,其垂直于羟甲基戊二酰辅酶a还原酶1 (hmg1/hmgr1)的A. Thaliana.由于抑制细胞伸长率而导致越突变的矮化[50].有趣的是,GRMZM2G393337的SNP对6.4 cm的影响最大。我们鉴定了一个基因GRMZM2G366373,它与A. Thilana Iaa3 - Aux / IAA-转录因子3(IAA3)/短胚轴2(SHY2),位于1号染色体SNP峰下游6.5 kb处(GRMZM2G066234;P= 3.15平台以及)。增加的功能Shy2.突变体呈矮化表型A. Thaliana.[51].在3号染色体长臂(179,174,157位)上游133kb处有一个高度显著的SNPNana plant 1 (na1)当玉米发生突变时,导致矮化的是什么De-eTiolated2(Det2)油菜素内酯合成相关基因A. Thaliana.[52].我们确定了MCF1 - 线粒体载体家族1作为位于染色体5(位置152,583,112)上的重要SNP上游的112kb的候选基因。这是同一等类别蛋白质被鉴定为pH的候选基因[31.].
从牛血清白蛋白中鉴定出其他与PH相关的候选基因。BSA共鉴定出37个区域分布在所有10条染色体上。由于许多BSA区域相对较大(平均大小3.5 Mb),这些区域内的一些候选区域在本次实验中很有可能不是因果关系。然而,BSA区域内的候选基因包括玉米生长素反应因子转录因子2,位于染色体1的16kb内,与假定表达的同源OsARF18。转基因水稻OsARF18等位基因与野生型相比,身高较矮[53].我们还发现了娜娜plant2(钠),玉米的同源词A. Thaliana DWF1基因,在第6染色体上。DWF1植物表现出类似于BR缺陷突变体的严重矮主义。在BSA中确定了参与凝胶酸信号传导的几种GRAs种群转录因子:GRAS45(GRMZM2G02809)和Gras69.(GRMZM2G153333)分别在第9染色体和第6染色体显著的BSA区域内被鉴定。在先前的研究中,gras45被确定为热带线受到严重全球热带风暴袭击[48.].
GWAS与BSA区域重叠
BSA鉴定了37个地区,Gwas确定了与pH相关的25个重要的SNP。具有BSA区域的三个重要的GWAS SNP:GMZM2G082191在染色体2(位置17.4-33.2 MB),GRMZM2G100260,染色体3(位置2.6-12.5 MB),染色体4上的GRMZM2G393337(位置188.4-216.8 MB)。染色体4区的候选者是矫形器羟甲基戊二酰辅酶a还原酶1 (hmgr1)在上面讨论的拟南芥中。第二重叠的SNP /区域位于GRMZM2G082191中的染色体2中,一种受体,如蛋白激酶,纯度(LOC_OS04G42700.1)和拟南芥(AT5G63930.1)。位于GRMZM2G100260中的3型染色体3上的第三重叠SNP /区域与D-Tyr-TRNA(TYR)脱酰酶家族蛋白有关。这些基因中的任何功能都没有根据其基因注释本身与pH相关。然而,两个更可能的候选基因位于GRMZM2G100260附近,仍然在3染色体上的BSA区域内:矮plant1 (d1;GRMZM2G036340)在距GRMZM2G100260 ~ 500 Kb处被鉴定;和IAA8 - AUX / IAA转录因子8(IAA8;GRMZM2G004696),拟南芥(AXR3 / IAA17)的同源物位于GRMZM2G100260上游122 kB。
讨论
全基因组关联研究已被广泛用于识别与复杂性状相关的候选基因[54].株高是一种经常被研究的复杂性状,因为它是一种相对简单的测量表现型,而且由于它与生物量的关系[55],留下抵抗[56,粮食产量[57].利用各种群体和标记集对玉米株高进行了关联研究[12.,13.,14.,15.,31.,48.,58].植物遗传学中的Gwas非常成功地识别复杂定量特征的因果基因,如植物身高,营养架构,生殖架构和代谢过程[30.,59].像GWAS一样,BSA是一种识别与表型相关的标记的技术。下一代测序技术的发展使BSA方法在绘制随机基因图谱方面更加可行[60].最初BSA用于分析模型生物,如拟南芥和酵母[38.,61].最近,这种方法被用于包括水稻在内的重要作物[62,63,大豆64,65]和玉米[66,67,68].这些研究均成功鉴定了与性状相关的QTL和候选基因。
常规GWA用于识别与作物物种中重要特征相关的休闲SNP。但是,几乎每个植物GWA都利用了一组自交系[30.].最近,一种名为FOAM的方法被引入,它涉及到在未重复试验中评估的非近交系地方品种的使用[35.].然而,这种方法仍然需要进行交叉测试来评估关联映射的表型。如果事先没有自交系,使用自交系可以增加研究的长度和费用,因为每个自交系必须分开种植(例如在自己的行/地),以保持其特性。最近的一项关联研究使用了先前研究过的多样性小组中个体的汇集测序,以识别与核行数相关的区域[43.].虽然这种方法减少了基因分型费用,但它仍然需要产生映射人群和大表型试验。相比之下,SP-Gwas依赖于在单一的异构,随机的人群中使用个体植物表型。单人的gwas是植物世界外的常见场所 - 对于人类[69,70[动物[71,72,73单个体表型已经非常成功地用于绘图,因为近交系面板很少可用或不可能创建。尽管如此,为了确保sp-GWAS结果是有效的,在本研究中实施的管道还允许GWAS和BSA的有效结合,以证实结果(图。1).
自孟德尔以来,人们就认识到株高对植物遗传研究的重要性[74].已经进行了许多研究试图阐明解释对pH观察到的宽变异的分子机制。根据我们对使用SP-GWAS和BSA的Shoepeg玉米人群的分析,我们确定了一个主要已知候选基因的pH值为玉米的pH值。然而,我们的研究只确定了有限数量的额外的额外pH相关的SNP。这是一个潜在的原因是我们的研究只能识别在山内人群中进行隔离的因果变形。
使用Dwarf突变体的植物高度和逆遗传学方法的许多先前关联研究已经确定了涉及BR和GA合成或信号传导的基因座。这两种激素都显示出对植物高度或射击长度的直接影响[23.,75].M铃木,等等。[50)表明,hmg1突变体显示出类似的表型对BR缺陷突变体的表型,其中抑制细胞伸长率导致矮化表型。最近的出版物在僵硬的茎秆和非僵硬的茎背景中使用GWA鉴定了使用GWA的PH QTN [76].我们的研究在GRMZM2G082191的基因区域内鉴定了染色体2上的显着重叠的SNP(SP-GWA和BSA),其被HU等人鉴定为候选基因。[76].GRMZM2G082191编码一种类似受体的蛋白激酶,并在水稻中具有推测的油菜素内酯不敏感功能[76].[15.使用联合联系QTL测绘和联合联系GWA,以识别US-NAM和北中央区域工厂介绍站(NCRPIS)AME多样性面板中的pH相关QTL和QTN。我们确定了d1作为我们研究的主要QTN (GWAS和BSA),与玉米NAM群体中鉴定的主要QTN一致[15.].D1ZmGA3ox在玉米中催化GA生物合成,其突变体表现出矮化PH [77,78,79].NA1.是BR合成的另一个重要基因,影响PH [80].重组自交系在PH QTL研究中鉴定为候选基因之一[81].在我们的研究中,na1仅在sp-GWAS中发现,而在BSA中未发现。
重要的是,我们的管道表明,具有非常有限的额外劳动力,BSA可以与SP-GWA合并独立候选SNP奖金。我们的GWA在两年内进行了四个观察部门,并在第一年结束时包括额外的筛选和选择步骤,我们能够在不进行额外测序的情况下包括BSA。值得注意的是,在pH的情况下,这种额外的筛选步骤可以在很短的时间内通过用测量棒(0.5-1小时,为一年的船员有4人的船员)来实现。对于单一的一年地,在0.1-HA地区种植了5000k种子。植物被随机选择,并且对那些随机选择的个体进行表型和基因分型。然而,差异是,在第一年,基于高于蜂鸣块的顶部或底部〜5%进行分歧选择。这种方法使我们能够使用来自两年的基因型和表型数据进行关联分析,而第二年的基因型数据仅用于BSA。我们的实验设计中没有包含空间检查,以防止花粉污染,这对我们的BSA结果有问题。然而,在未来的研究中纳入检查可以代表一种有希望的方法来确认现场均匀性,特别是如果正在评估除pH以外的特性,以便在没有受影响表型的情况下可以进行脱毛。
我们的研究还表明,可以使用异质,随机交配的人群中的SP-GWA来实现显着的关联,例如开放的授粉玉米Landrace。此外,我们能够使用BSA获得所识别的SNP子集的证据,该证据也为pH提供了额外的推定QTL集合。如Dell'Acqua等,在模拟研究中所示。[16.]对于具有70%遗传性的特征,需要至少500个个体来检测标记和特征之间的关联。现场研究还表明,个人数量的增加改善了检测标记特征的权力[82,83].AD LongCH Langley [47.结果表明,标记与性状的关联能力取决于数量性状核苷酸(QTN)和个体数量的变异。在我们的关联研究中,我们使用了768个具有306,522个SNPs (MAF < 0.05)的个体,确定了25个显著的SNPs (P ≤ 0.00001) associated with PH. While 25 associations is not tremendous based on a comparison to other PH experiments (references), a potential reason for this discrepancy, in addition to experimental power considerations, is that Shoepeg is a single populations with limited genetic variation.
作为sp-GWAS管道的附加,BSA通过从第0代中选择不同表型来鉴定与PH相关的位点。利用BSA对群体进行分析,共鉴定出37个ph基因组区,在BSA中鉴定出的QTL数量大于GWAS。这是基于模拟的预期,模拟显示BSA在识别微小和罕见的等位基因,即使影响非常小[38.,84].在37个PH标记的QTL中,3个显著的GWAS关联位于第2、3和4号染色体上的BSA峰内,而其他的BSA峰位于显著的SNPs附近(表)2和3.).
在这项研究中,我们证明sp-GWAS可以有效且经济地产生与传统GWAS实验相媲美的结果。sp-GWAS鉴定的候选基因中,有许多是控制株高的主要数量基因。尽管我们研究的是一个遗传变异有限的玉米地方品种群体,但我们仍然成功地识别了许多与标准GWAS研究有关的候选基因。我们对其中三个snp的关联但独立的BSA结果的确证提供了额外的证据,证明我们实施sp-GWAS是有效的。传统GWAS中的大多数先前的验证工作都是通过连锁映射完成的,而BSA通常用于验证连锁映射或池GWAS [43.,85].然而,BSA已被证明对定位候选qtl有效[43.,69,86,87,88].
有几个潜在的因素有助于SP-GWAS和BSA识别的少量重叠信号。首先,单株植物测量具有比基于曲线的表型更低的遗传性,这肯定降低了我们的方法的力量。此外,BSA分辨率严重依赖于近一项研究生一学的重组模式,而作业研究基于古代重组历史。最后,鉴定BSA中候选基因的力量取决于尾部尺寸(散装中的个体数量)[86].然而,对于确实重叠的三个区域,我们结合sp-GWAS和BSA的管道提供了因果关系的有力证据。在这项研究中,BSA在384个个体中进行(仅从第1代开始,每个个体中有192个),而GWAS在768个个体中进行。
由于在野外环境中测量的植物之间存在宏观和微观尺度的差异,研究人员往往不愿使用单株测量。相反,通常是通过对整个地块的测量值进行平均。我们的结果表明,这种做法可能并不总是必要的,特别是考虑到基于图的实验比单厂测量占用更多的空间、时间和精力。在我们的实验中,种植、表型和收获大约在1小时内完成。每一年的拍摄地点有四名工作人员。值得注意的是,基于地块设计进行研究会引入巷子效应[89],这不存在于例如本文所述的单植物实验中。但是,通过将适当的检查和空间变化纳入我们的模型,我们的设计可以进一步改善。这种方法可能在作物中特别有益,其中关联面板不可用或近亲繁殖是不可行的。
在实际育种中,PH直接表型选择可能比利用QTL标记辅助选择更有效。因此,我们使用PH作为具有中等遗传复杂性的性状的模型,但这些性状可能需要更多的劳动密集型或昂贵的评估。根据育种计划的目标,可以利用基因组选择将PH与其他性状一起作为多性状指数的一部分。在单个地方小种群体中进行关联作图的结果,而不是在一个更多样化的面板中,可能有助于将特定供体群体的遗传变异纳入精英育种材料。此外,在一个环境中识别重要的位点可以在其他群体中识别或产生新的感兴趣的基因变异。即使有了这些进展,鉴定和将来自GWAS的qtl纳入改良性状的标记辅助选择管道之间的差距不大可能受到影响。
结论
总之,在本文中,我们已经证明了一种管道,由此SP-GWA能够有效地耦合BSA,以有效地识别重要的性状相关的SNP。使用这种方法的主要优点是其简单性,时间要求(在场和离田上),以及低成本。我们描述的方法可以与泡沫的概念进行比较[35.,在那里研究了多个地方人种。这两种方法的相似之处在于它们都使用杂合子个体,但不同之处在于FOAM涉及大量取样非常多样化的地方种,并在家族水平上对多个个体进行表型复制,而sp-GWAS涉及对完全未复制个体进行表型。这意味着sp-GWAS的成本非常低,即使在它与BSA结合以实现即时独立的结果确证之后。然而,更大的样本量、更精确的表型复制和更高的标记密度可以进一步提高sp-GWAS的能力。sp-GWAS的力量不太可能与传统的、重复试验的、利用一组自交系的植物GWAS的力量相匹敌。有时,成本效益分析将sp-GWAS作为理想的方法,但当精度至关重要时,更传统的GWAS仍然是有意义的。然而,当研究人员对在没有关联面板或需要花费时间的作物中寻找候选基因感兴趣时,或者当效率和成本非常重要时,sp-GWAS代表了一种识别重要性状候选基因的潜在方法。我们在这里描述的管道的未来研究领域可能会富有成果,包括开发一种策略,在不引入花粉污染的情况下,有效地将实验检查纳入田间计划,评估sp-GWAS和BSA管道是否有潜力在不同的种质集中识别因果位点,除了封闭的种群,如Shoepeg。
方法
植物材料及田间试验
本研究以苏佩玉米地方品种为基础群体。Shoepeg是美国南部一种凹陷的玉米[90,91].从国家植物种质系统获得一百个加入PI 269743(www.ars-grin.gov).这些分离核是在温室中种植的,在那里它们被散装花粉随机交配,以产生对实验的产生-0种子。在2016年夏天,在4〜0.1-HA地块中的每一个(总共20,000株植物中,大约5000种种子。种子在91厘米的行间距分开约15厘米。现场试验是在遗传农场的两块地块中进行,两台在哥伦比亚州的罗林斯农场中进行。从其他玉米场隔离地种植地图,使植物可以在没有来自其他地块或其他玉米领域的交叉授粉的风险的授粉。我们的实验图中没有包括空间检查,因为植物被允许开放授粉,我们不能允许国外花粉污染人口(见批量分析分析部分)。在一年中,在每种曲线中,随机选择5000(96×4 = 384)的96株植物进行基因分型和表型。所有384种随机选择的植物在生殖成熟时单独测量,以从地面到旗叶的轴环以五厘米的增量。根据每个图中的96个单独测量的植物中收集的表型(表1, 图。1).4个样地的5000株植物的每一株都根据它们在截断阈值以上或以下的状态进行表型分析,只有超过截断阈值的穗才会收获。然后从每个地点增加相同数量的种子,形成4个新种群:Generation-1-Tall1, Generation-1-Tall2, Generation-1-Short1和Generation-1-Short2。随机选择这四个地块进行高株或短株的选择。
2017年夏天(第2代第1代),这4个种群又分别种植了大约5000颗种子,在密苏里州哥伦比亚的相同4个近似地点的0.1公顷孤立地块上。基因分型和表型的过程在2016年重复。
基因分型
从96个随机选择的植物从每年的四个位置中的每一个随机选择的植物,并冷冻干燥。从每株植物八到十叶冲孔用于使用QIAGEN DNEASY 96植物试剂盒提取DNA,唯一的修饰是在加入初始裂解缓冲液后用不锈钢珠子短暂地摇动样品。用Promega Quantfluor在Tecan Spark 10M上定量DNA产率。使用100ng DNA和ApeKI基因分型测序(GBS)协议[44.],为每年准备的四个96孔板的每个孔板都准备了库。对方案进行了一些修改,包括将96孔分离成4个池,其中24孔由适配器连接、预聚合酶链反应(pre-PCR)池和使用ThermoFisher Phusion II主混合进行PCR扩增。利用Qubit法测定富集文库的数量,并在Agilent Bioanalyzer高灵敏度DNA芯片上检测文库的大小分布。由于每个样本有384个不同的条形码,所以所有单独的样本库被合并到一个最终的样本库中进行测序。在DeenaBIO上设计条形码适配器,并用IDTdna合成。密苏里大学哥伦比亚分校DNA Core NEXTseq高输出单端75 bp run sequencing reads被定位到玉米B73 reference genome version3 [AGPv3;http://ftp.maizeSequence.org/[49.]]使用流苏5 GBS v2管道[92].这导致414,361初始SNP,平均读取深度为〜2.01x。较小等位基因频率(MAF)<0.05和读数小于40的标记被排除在进一步的分析之外。还筛选了SNP,仅包括Diallelic Loci。使用Beag版本4.1执行缺失标记的归属[93].经过这些过滤和imputation步骤,最终的数据集306,522标记用于下游分析。
表型数据分析
表型数据是用线性模型进行标准化的,该模型对位置进行了固定的处理LM.R中的函数[94].模型的残差被用作GWAS和BSA的响应变量,如下所述。遗传率使用GCTA v1.26.0估算[95].首先,利用所有的基因型snp计算所有768个个体的基因组关系矩阵(GRM)。然后将该GRM作为预测因子来估计遗传力。使用R包adegenet进行主成分分析(PCA),以评估种群结构[96].
关联分析
有许多统计模型用于关联分析,最常见的是混合线性模型(MLM)。将亲属关系和人口结构纳入传销可以控制假阳性,但也可能危及真阳性[97].固定和随机模型循环概率统一(FarmCPU)是一种关联研究模型,与GWAS的其他统计模型相比,该模型已被证明在控制假阳性而不损害真阳性方面是有效的[97].在FarmCPU模型中,控制误报,多个基因座线性混合模型(MLMM)分为两部分:固定效果模型(FEM)和随机效果模型(REM),这些模型迭代地使用[97].FarmCPU中的模型过拟合是通过使用REM中的相关标记来估计亲属关系来避免的,然后FEM使用这些标记作为协变量来检验标记,以控制假阳性和假阴性。我们研究中用于GWAS的FarmCPU模型是使用FarmCPU R包完成的[97].一代和选择制度纳入模型中作为协变量。基于意义阈值来定义有效的SNPP < 0.00001. Since approximately 300,000 SNPs were tested, this threshold means that we expect fewer than three false positives across the entire set of markers. Moreover, this threshold is more conservative than others that have been used for GWAS for plant height in maize [12.,15.,31.].手动筛选150kb内部有效SNP内的基因,以筛选与pH相关的潜在注释。注释从Enerembl下载(http://ensembl.gramene.org/Zea_mays/Info/Index)和MaizeGDB数据库(http://www.maizegdb.org/gbrowse).
批量隔离分析
对第一代观察到的384株植物进行了改进的体分离分析(BSA)。而原方法的RW Michelmore,等等。[39.[在他们的分析中使用了双亲本群体,我们使用了一个隔离群体作为基础,这也类似于一代选择实验。BSA并不是sp-GWAS的固有必需,但我们相信BSA和GWAS的结合为我们确定的候选者提供了强有力的证据,而且这些方法在一条管道中相互补充。在第0代随机选择的384个基因型植物提供了基本等位基因频率的估计。然后,在第一代中随机选择384个基因型的植株,估算出第0代中5%最高和最短植株的BSA等位基因频率。首先筛选> 0.05 MAF标记物,读取计数大于40。过滤后的snp有243,303个用于进一步分析。利用Haase等人的“sm”R-script估计了每个位点参考等位基因的频率[68].通过使用双面Z测试计算每个基因座的意义。要识别重要的SNP,首先确定了重要区域,其中包括所有带有-log10的SNP(p-value)超过离群值阈值0.5% [98].然后将15-SNP滑动窗口施加到平滑的结果[68].
可用性数据和材料
本研究的所有数据和统计数据均以图表的形式收录在本手稿中。原始数据可在figshare上公开获取;https://figshare.com/s/4a9620c8752355a04e2a.我们的分析代码在github上公开;https://github.com/abi01/sp-GWAS.
缩写
- BR:
-
Brassinosteroids
- BSA:
-
批量隔离分析
- Farmcpu:
-
固定和随机模型循环概率的统一
- 遗传算法:
-
吉布林素
- GBS:
-
基因型进行排序
- 克:
-
基因组关系矩阵
- GWAS:
-
全基因组关联研究
- MAF:
-
轻微的等位基因频率
- PCA:
-
主成分分析
- pH:
-
株高
- QTL:
-
定量特质基因座
- 本考察团:
-
定量特征Nucelotide
- SNPS:
-
单核苷酸多态性
- sp-GWAS:
-
单株植物GWA.
参考
- 1。
duvick dn。美国玉米产量遗传进展(玉蜀黍L。).梅迪卡。2005; 50:193-202。
- 2。
孙强,Ersoz ES, Hurwitz BL, Peiffer JA, McMullen MD, Grills GS, Ross-Ibarra J,等。第一代玉米单倍型图。科学。2009;326(5956):1115。
- 3.
贾建民,宋志强,白伯里PJ, Costich D, de Leon N, Doebley J, Elshire RJ, Gaut B, Geller L, Glaubitz JC, et al.;玉米HapMap2从一个不断变化的基因组中识别出现有的变异。Nat麝猫。2012;44:803。
- 4.
王斌,杨斌,徐东,杨斌,谢灿,等。建设第三代玉米单体型图。GigaScience。2018;7(4):GIX134。
- 5.
Wallace JG, Larsson SJ, Buckler ES。进入玉米数量遗传学的第二个世纪。遗传。2014;112(1):30-8。
- 6.
张志强,张志强,张志强。植物遗传改良方法的研究进展。[j] .地理科学进展,2008,29 (6):444-57 .]
- 7.
Sibov ST, Lopes De Souza C Jr, AAF G, Garcia AF, Silva AR, Mangolin CA, Benchimol LL, De Souza AP.利用微卫星标记进行热带玉米(Zea mays L.)分子定位。1.显示扭曲分离的基因座的地图构建和定位。Hereditas。2003;139(2):96 - 106。
- 8.
Lima MLA,De Souza Cl,Dav B,De Souza AP,Carlini-Garcia La。在热带玉米人群中映射QTL以获得谷物产量和植物特征。mol品种。2006; 17(3):227-39。
- 9.
Lübberstedttt,Melchinger Ae,Schöncc,UtzHF,Klein D. QTL在欧洲燧石的Tescrosses中的QTL映射:I。不同测试人员进行牧草收益率的比较。农作物科学。1997年; 37(3):921-31。
- 10.
费舍尔RA。十五。——孟德尔遗传假设下的亲缘关系。中国科学(d辑:地球科学)1919;52(2):399-433。
- 11.
希尔工作组。了解和利用数量遗传变异。中国生物科学(英文版),2010;
- 12.
翁杰,谢c,郝z,王继,刘c,李米,张d,白l,张某,李奇。基因组 - 范围协会研究识别影响中国精英玉米植物高度的候选基因(玉米L.)自交系。Plos一个。2011; 6(12):E29229。
- 13。
Riedelsheimer C, Lisec J, Czedik-Eysenberg A, Sulpice R, Flis A, Grieder C, Altmann T, Stitt M, Willmitzer L, Melchinger AE。玉米复杂性状叶片代谢谱的全基因组关联定位。中国科学院院刊。2012;109(23):8872-7。
- 14。
杨宁,吕永强,杨旭,黄建军,周勇,李飞,文伟,刘军,李建军,闫建军。玉米17个农艺性状的非参数关联研究。公共科学图书馆麝猫。2014;10 (9):e1004573。
- 15.
peffer JA, Romay MC, Gore MA, Flint-Garcia SA, Zhang Z, Millard MJ, Gardner CAC, McMullen MD, Holland JB, Bradbury PJ,等。玉米株高的遗传结构。遗传学。2014;196(4):1337。
- 16.
Dell'Acqua M,Gatti Dm,Pea G,Cattonaro F,Coppens F,Magris G,Hlaing Al,Aung Hh,Nelissen H,Baute J等。魔法玉米人口的遗传特性:ZEA 5月的高清QTL映射的新平台。基因组Biol。2015; 16(1):167。
- 17.
奥斯汀DF,Lee M.玉米重组近交系的开花和植物高度定量特质遗传分辨率和验证。基因组。1996年; 39(5):957-68。
- 18.
基于基因型分析的玉米群体株高数量性状及其与定性遗传位点的关系。中国科学(d辑:地球科学)1991;83(2):141-5。
- 19.
田继华,田文涛,杨建兵,孟西青,李建生,李毅江,李建生。玉米重组自交系株高的分子标记分析。Euphytica。2007;155(1):117 - 24。
- 20。
Mackay TF, Stone EA, Ayroles JF。数量性状的遗传学:挑战与展望。中国农业科学。2009;10(8):565-77。
- 21.
价格啊。信不信由你,qtl是准确的!植物科学进展。2006;11(5):213-6。
- 22.
Peng J, Richards DE, Hartley NM, Murphy GP, Devos KM, Flintham JE, Beales J, Fish LJ, Worland AJ, Pelica F,等。“绿色革命”基因编码突变的赤霉素反应调节剂。自然。1999;400(6741):256 - 61。
- 23。
Monna L, Kitazawa N, Yoshino R, Suzuki J, Masuda H, Maehara Y, Tanji M, Sato M, Nasu S, Minobe Y.水稻半矮化基因sd-1的定位克隆:水稻“绿色革命基因”编码一种参与赤霉素合成的突变酶。DNA研究》2002;9(1):11-7。
- 24。
滕飞,翟磊,刘锐,白伟,王磊,霍东,陶毅,郑勇,张志强。玉米株高QTL qPH3.1的候选基因ZmGA3ox2。植物j . 2013; 73(3): 405 - 16。
- 25。
马凯瑞维奇1,汤普森A,米尔鲍尔GJ,施普林格NM。玉米Brd1基因编码油菜素内酯C-6氧化酶。《公共科学图书馆•综合》。2012;7 (1):e30798。
- 26。
Multani DS,Briggs SP,Chamberlin Ma,Blakeslee Jj,Murphy As,Johal GS。玉米BR2和高粱DW3突变体紧凑秸秆中锰铁转运蛋白的丧失。科学。2003; 302(5642):81-4。
- 27。
导演WD。QTL分析:功率、精度和准确度。在:帕特森,编辑。复杂性状的分子解剖。博卡拉顿:CRC出版社;1998.p . 145 - 62。
- 28.
Schon CC, Utz HF, Groh S, Truberg B, Openshaw S, Melchinger AE。基于重采样的玉米杂交试验数量性状位点定位及其与复杂性状数量遗传的相关性。遗传学。2004;167(1):485 - 98。
- 29.
测序技术-下一代。中国科学(d辑:地球科学)2010;11(1):31-46
- 30.
肖勇,刘洪,吴磊,严建军。玉米全基因组关联研究:赞美和凝视。摩尔。2017;10(3):359 - 74。
- 31。
李X,周Z,丁J,吴Y,周B,王R,MA J,Wang S,Zhang X,Xia Z等。组合的联系和关联映射显示玉米植物和耳高的QTL和候选基因。前植物SCI。2016; 7:833。
- 32。
Buckler ES, Holland JB, Bradbury PJ, Acharya CB, Brown PJ, Browne C, Ersoz E, Flint-Garcia S, Garcia A, Glaubitz JC,等。玉米开花时间的遗传结构。科学。2009;325(5941):714。
- 33.
焦勇,赵华,任磊,宋伟,曾波,郭军,王波,刘铮,陈建军,李伟,等。玉米现代育种过程中的全基因组遗传变化。Nat麝猫。2012;44(7):812 - 5。
- 34.
peffer JA, Flint-Garcia SA, De Leon N, McMullen MD, Kaeppler SM, Buckler ES。玉米茎秆强度的遗传结构。《公共科学图书馆•综合》。2013;8 (6):e67066。
- 35。
Romero Navarro Ja,Willcox M,Burgueno J,Romay C,Swarts K,Trachsel S,PreciaDo E,Terron A,Delgado HV,Vidal V等人。玉米地体面的等位基因多样性研究等位基因。NAT Genet。2017; 49(3):476-80。
- 36。
Pearson TA, Manolio TA。如何解释全基因组关联研究。《美国医学协会杂志》上。2008;299(11):1335 - 44。
- 37。
肉牛屠宰性状的全基因组关联研究。力所能及的科学。2018;213:35-43。
- 38。
Ehrenreich Im,Torabi n,jia y,肯特J,Martis S,Shapiro Ja,Gresham D,Caudy Aa,Kruglyak L.遗传复杂性状的解剖,具有极大的酵母隔层。自然。2010; 464:1039。
- 39。
Michelmore RW, Paran I, Kesseli房车。通过大量分离分析鉴定与抗病基因相关的标记:一种利用分离群体快速检测特定基因组区域标记的方法。中国科学院院刊1991;88(21):9828-32。
- 40.
Quarrie SA, Lazić-Jančić V, Kovačević D, Steed A, Pekić S.分子标记的Bulk分离分析及其在玉米抗旱性改良中的应用。[J] .农业工程学报,1999;50(337):1299-306。
- 41。
Hyten DL,Smith Jr,Frederick Rd,Tucker ML,Song Q,Cregan PB。使用Goldengate测定来定位赋予大豆生锈的RPP3基因座的RPP3基因座,保留所有权利。可以以任何形式或通过任何形式,电子或机械,包括复印,记录或任何信息存储和检索系统的任何形式再现或传输该期刊的一部分,而无需从发布者写入的许可。发行商已经获得了打印和用于转载本文包含的材料的许可。农作物科学。2009; 49(1):265-71。
- 42。
刘某,Yeh CT,唐HM,Nettleton D,Schnable PS。通过堆积偏析RNA-SEQ(BSR-SEQ)进行基因映射。Plos一个。2012; 7(5):E36406。
- 43。
杨军,蒋洪,叶洪涛,于军,杨军,杨建军,杨建军。一种利用基因组序列分析方法鉴定性状相关变异的研究方法。植物j . 2015; 84(3): 587 - 96。
- 44。
Elshire RJ,Glaubitz JC,Sun Q,Poland Ja,Kawamoto K,Buckler Es,Mitchell SE。一种高分辨率物种的鲁棒,简单的基因分序列(GBS)方法。Plos一个。2011; 6(5):E19379。
- 45.
郁郁葱葱的JL。动物养殖计划。Ames,IA:Collegiate Press Inc;1937年。
- 46.
Viana JMS,Mundim GB,Pereira HD,ACB A,FF e S.随机交叉种群的基因组关联研究效率。mol品种。2017; 37(8):102。
- 47。
关联研究在检测候选遗传位点对复杂性状变异的贡献方面的力量。基因组研究》1999;9(8):720 - 31所示。
- 48。
Wallace JG, Zhang X, Beyene Y, Semagn K, Olsen M, Prasanna BM, Buckler ES。撒哈拉以南非洲15个热带玉米种群在管理干旱胁迫和充足水分条件下的株高和开花时间全基因组协会作物科学。2016;56(5):2365 - 78。
- 49。
张建平,张建平,张建平,等。B73玉米基因组:复杂性、多样性和动态。科学。2009;326(5956):1112 - 5。
- 50.
Suzuki M, Kamide Y, Nagata N, Seki H, Ohyama K, Kato H, Masuda K, Sato S, Kato T, Tabata S,等。拟南芥中3-羟基-3-甲基戊二酰辅酶a还原酶1 (HMG1)功能的丧失会导致矮化、早衰和雄性不育,并降低甾醇水平。植物j . 2004; 37(5): 750 - 61。
- 51.
多AUX/IAA-ARF模块调控侧根形成:拟南芥SHY2/ iaa3介导的生长素信号的作用。中国生物科学(英文版);2012;367(1595):1461-8。
- 52.
李建平,李建军,李建军,等。油菜素内酯在拟南芥光依赖发育中的作用。科学。1996;272(5260):398 - 401。
- 53。
黄军,李志,赵东。OsmiR160靶基因OsARF18的调控与水稻生长素信号转导的改变。Sci众议员2016;6:29938。
- 54。
5年的GWAS发现。acta photonica sinica, 2012;90(1): 7-24。
- 55。
Freeman Kw,Girma K,Arnall DB,Mullen RW,Martin KL,Teal Rk,Raun Wr。使用遥感和植物高度在各种生长阶段的玉米饲料生物质和氮吸收的植物预测。Agron J. 2007; 99(2):530-6。
- 56。
K. PICKett L,B.LILJEDAHL J,G.A J H,Ullstrup C.经受横向载荷的玉米茎的流变性质。跨rissae。1969; 12(3):392-0396。
- 57.
杜维克DN:育种对玉米产量进步的贡献(玉米l .)。在:农学进步。卷。86:学术出版社;2005年:83-145。
- 58.
Farfan IDB, De La Fuente GN, Murray SC, Isakeit T, Huang P-C, Warburton M, Williams P, Windham GL, Kolomiets M. Genome wide association study for drought, aflatoxin resistance, and important agronomic traits of maize hybrids in the sub-tropics. PLoS One. 2015;10(2):e0117737.
- 59。
赵成松,杨建明。植物关联图谱研究现状与展望。植物基因组。2008;1(1):5 - 20。
- 60。
Schneeberger K, Weigel D.通过新的测序技术使遗传学快速前进。植物科学进展。2011;16(5):282-8。
- 61。
Schneeberger K, Ossowski S, Lanz C, Juul T, Petersen AH, Nielsen KL, Jorgensen JE, Weigel D, Andersen SU. SHOREmap:通过深度测序进行同步定位和突变鉴定。Nat方法。2009;6(8):550 - 1。
- 62.
Vikram P,Swamy BPM,Dixit S,Ahmed H,Cruz MTS,Singh Ak,Ye G,Kumar A.散装杀菌分析:“一种有效的绘制一致效应干旱谷物产量QTL在水稻中的有效方法”。现场裁剪。2012; 134:185-92。
- 63.
张国良,陈丽艳,肖国勇,肖玉华,陈晓波,张树涛:利用体积分离法检测水稻耐热性相关QTL (奥雅萨苜蓿l .)利用SSR标记。中国农业科学,2009,8(4):482-487。
- 64.
Takagi H, Abe A, Yoshida K, Kosugi S, Natsume S, Mitsuoka C, Uemura A, Utsushi H, Tamiru M, Takuno S, et al.QTL-seq:通过对两个大群体的DNA进行全基因组重测序,快速定位水稻数量性状位点。植物j . 2013; 74(1): 174 - 83。
- 65。
宋军,李智,刘智,郭勇,邱丽娟。下一代大分离分析测序加速了大豆中两个定性基因的同时鉴定。植物学报。2017;8:919。
- 66。
杜华,朱军,苏辉,黄敏,王辉,丁胜,张波,罗安,魏胜,田旭,等。膨化分离RNA-seq揭示了玉米耐涝候选基因的差异表达和SNPs。植物科学学报2017;8(1022)。https://doi.org/10.3389/fpls.2017.01022.
- 67。
Farooqi MQ, Sa KJ, Hong TK, Lee JK。利用SSR标记提高玉米抗寒性的群体分离分析。(4)。https://doi.org/10.4238/gmr15049326.
- 68。
Haase NJ, Beissinger T, Hirsch CN, Vaillancourt B, Deshpande S, Barry K, Buell CR, Kaeppler SM, de LeonG3: | |基因组遗传基因。2015; 5(8): 1593 - 602。
- 69.
Duncan EL, Danoy P, Kemp JP, Leo PJ, McCloskey E, Nicholson GC, Eastell R, Prince RL, Eisman JA, Jones G,等。使用极端截尾选择的全基因组关联研究确定了影响骨密度和骨折风险的新基因。公共科学图书馆麝猫。2011;7 (4):e1001372。
- 70.
Xu J, Zhu K, Xu J, Brown SJ, Mullin S, Tickner J, Pavlos NJ, Dudbridge F, Walsh JP, Wilson SG。人破骨细胞骨密度GWAS变异的表达定量性状位点研究。中国骨矿物质学报2018;33(6):1044-51。
- 71.
Mapholi No,Maiwashe A,Matika O,Riggio V,Bishop Sc,Mackeil MD,Banga C,Taylor JF,Dzama K.南非南非粪便蜱抗性的基因组关联研究。蜱虫蜱传承。2016; 7(3):487-97。
- 72。
彼得斯如此,Kizilkaya K,Garrick DJ,费尔南多RL,Reecy JM,Weaber RL,Silver Ga,Thomas Mg。Brangus Heifers1的遗传性和贝叶斯基因组 - 伟大服务概念与怀孕的研究。j anim sci。2013; 91(2):605-12。
- 73。
Thekkoot DM, Young JM, Rothschild MF, Dekkers JCM。生长结束期剩余采食量不同的约克猪品系母猪泌乳性能的全基因组关联分析1。中国动物科学(英文版)。2016;94(6):2317-31。
- 74.
孟德尔G:植物杂交的实验(1865)。Verhandlungen des naturforschenden Vereins Brünn)www mendelweb org / mendel html(于2013年1月1日访问)1996.
- 75.
王Y,Li J.植物建筑的分子基础。Annu Rev植物Biol。2008; 59:253-79。
- 76.
Hu S, Wang C, Sanchez DL, Lipka AE, Liu P, Yin Y, Blanco M, Lübberstedt玉米l .)。植物科学学报,2017,36(6):591 - 598。
- 77。
喷雾Cr,Kobayashi M,Suzuki Y,Phinney Bo,Gaskin P,Macmillan J.Zea的矮-1(DT)突变体在Gibberellin-Biosynethetic途径中阻断了三个步骤。Proc Natl Acad Sci U S A. 1996; 93(19):10515-8。
- 78。
黄志强,王志强,王志强,等。玉米营养芽赤霉素含量的定性和定量分析。植物生理学报。1988;88(4):1367 - 1366。
- 79。
陈y,侯m,刘l,吴S,沉y,ishiyama k,kobayashi m,mccarty dr,tan b-c。玉米Dwarf1编码胃肠杆菌蛋白3-氧化酶,并且是对细胞核和胞嘧啶的双重定位。植物理性。2014; 166(4):2028-39。
- 80.
陈志强,陈志强,陈志强,等。油菜素内酯对玉米性别的影响。中国科学院院刊。2011;108(49):19814。
- 81.
王波,刘海涛,刘志东,郭建军,李伟,陈建军,高超,朱勇,郑旭,等。利用超高密度基因分型和大重组自交系群体鉴定玉米株型相关性状的小效应qtlBMC Plant biology . 2018;18(1):17。
- 82.
余建军,王志强。玉米遗传关联作图与基因组组织。生物技术学报。2006;17(2):155-60。
- 83.
Flint-Garcia SA, Thuillet AC, Yu J, Pressoir G, Romero SM, Mitchell SE, Doebley J, Kresovich S, Goodman MM, Buckler ES。玉米关联群体:数量性状位点分析的高分辨率平台。植物j . 2005; 44(6): 1054 - 64。
- 84。
孙勇,王静,Crouch JH,徐勇。基因分型在复杂性状遗传分析中的有效性及其在作物改良中的应用。摩尔品种。2010;26(3):493 - 511。
- 85。
Mu J,Huang S,Liu S,Zeng Q,Dai M,Wang Q,Wu J,Yu S,Kang Z,Han D.使用SNP型遗传贴图结合QTL映射和膨胀揭示了小麦条纹铁锈的遗传架构分析分析。Al Appl Genet。2019年; 132(2):443-55。
- 86.
邹超,王鹏,徐勇。遗传、基因组学与作物改良的大样本分析。生物技术学报;2016;14(10):41 - 55。
- 87.
Schlotterer C,Kofler R,Versace E,Tobler R,Franssen Su。结合实验演化与下一代测序:一个强大的工具,用于研究遗传变异的适应性。遗传。2015; 114(5):431-40。
- 88.
Wambugu P, Ndjiondjop MN, Furtado A, Henry R.大量分离菌的测序使水稻直链淀粉含量的遗传控制得以剖析。生物技术学报2018;16(1):100-10。
- 89。
Vincelli P,Lee C.开放小巷在现场试验中的影响评估玉米杀菌剂的产量效应。植物DIS。2015; 99(2):263-6。
- 90。
玉米带玉米的起源:同工酶证据。经济学机器人。1988;42(1):120 - 31所示。
- 91.
Smith CW, Betrán J, Runge ECA。玉米:起源、历史、技术和生产。新泽西州霍博肯:威利;2004.
- 92.
Glaubitz JC,Casstevens TM,Lu F,Harriman J,Elshire RJ,Sun Q,Buckler ES。Tassel-GBS:测序分析管道的高容量基因分型。Plos一个。2014; 9(2):E90346。
- 93.
Browning Brian L,Browning Sharon R.基因型归毒用数百万参考样品。我是j嗡嗡声的遗传。2016; 98(1):116-26。
- 94.
Rstudio:r的综合开发.rstudio,公司,波士顿,马[http://www.rstudio.com/].
- 95.
杨杰,李舍,戈达德我,visscher pm。GCTA:一种用于基因组复杂性状分析的工具。我是j嗡嗡声的遗传。2011; 88(1):76-82。
- 96.
一个用于遗传标记多变量分析的R包。生物信息学,2008;24(11):1403 - 5。
- 97。
刘X,黄M,粉丝B,贝勒ES,张Z.迭代使用固定和随机效果模型,以实现强大高效的基因组关联研究。Plos Genet。2016; 12(2):E1005767。
- 98.
Becker A,Chao D-Y,Zhang X,Salt De,Baxter I.使用单核苷酸多态性微阵列进行散装分析。Plos一个。2011; 6(1):E15993。
致谢
我们还要感谢Alexander Gregory和Husain Agha在表型方面的帮助。我们也要感谢国家植物种质系统对Shoepeg种子的维护和提供。测序是由密苏里大学哥伦比亚分校的DNA核心完成的。
资金
这个项目是由美国农业部农业研究处资助的。
作者信息
隶属关系
贡献
AG,KEG和TMB设计并执行了所有现场实验。柯格进行了基因分型。AG和TMB进行了统计分析并写了稿件。VS和SFG提供科学输出并批判性修订的稿件。所有作者阅读并认可的终稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
.
额外的信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Gyawali,A.,Shrestha,V.,Guill,K.E.等等。单株GWAS结合大量分离分析可以快速识别和确证株高候选snp。BMC植物BIOL.19,412(2019)。https://doi.org/10.1186/s12870-019-2000-y.
已收到:
接受:
发表:
关键词
- 单株全基因组关联研究
- 玉米
- 植物高度
- FarmCPU
- 批量隔离分析