摘要
背景
栽培的花生(arachis hypogaea.)是世界上最重要的油料作物之一,但由于遗传基础狭窄,其改良受到限制。高度变异的野生花生种,特别是在组内。arachis.,可能是花生改良的有利等位基因的丰富遗传来源;教派。arachis.是属中最大的分类学区arachis.它的成员还包括种植的花生。为了更好地利用这些野生资源,对其遗传基础和亲缘关系进行了研究arachis.首先需要更好地理解物种。
结果
在此,在本研究中,我们已经测序和/或组装了十二个arachis.完全叶绿体(CP)基因组(来自派生十一。arachis.).这些cp基因组序列丰富了已发表的文献arachis.CP基因组数据。从十二个获取的CP基因组中,已经确定了大量遗传变化(1368 SND,311 indels),其中与已从相同数据集中识别的69个SSR基因座一起提供了强大的工具,以供未来的探索。我们研究中的系统发育分析已经分组。arachis.物种分为两大谱系(I和II),这一结果与许多早期研究的报告显示,谱系II以AA基因组物种为主,大部分是多年生物种,而谱系I包括基因组类型更多样化的物种,主要是一年生/二年生物种。此外,种植的花生和一个.monticola它们是唯一的四倍体(AABB)物种arachis.这一结果与叶绿体的母性遗传一起表明,这两个四倍体物种来自于一个AA基因组物种。
结论
综上所述,我们获得了12个完全序列arachis.CP基因组,这并不仅仅帮助我们更好地了解培养的花生和其密切的野生亲属是相关的,而且还为我们提供了丰富的遗传资源,可能对未来的花生育种具有巨大的潜力。
背景
属arachis.L.由于其成员种之一,栽培花生(答:hypogaeaL.),是世界上经济上重要的油料作物[1,2].arachis.物种对南美洲有条不紊,它来自那里,耕种花生已经散布,种植在远离其Cardleland的世界许多不同地区,其中中国代表着最大的生产国[1,2,3.].
形态学上,两个亚种已被确定答:hypogaea,可以进一步分化为六种植物品种[4].尽管表型变量,答:hypogaea与其野生亲属相比,具有相对较低的遗传变异级别[5,6,7,8,9,10],这可能是由于驯化,可能是最近的单一多倍体起源,以及栽培花生与野生花生的倍性差异[1,2,11,12].有限的遗传变异答:hypogaea抑制这种作物的进一步改善,特别是从抗病和害虫的角度来看[1,2].幸运的是,之间的间隙杂交arachis.物种是可能的,特别是在现代技术的帮助下,有一个丰富的遗传变异的来源,在密切相关的野生亲缘arachis.可能非常有用,可扩大栽培花生的遗传基础[2,4,12,13,14,15].
到目前为止,已在80多种物种中确定arachis.属(2],它安排在九个分类部分(包括派别。arachis.,教派。Erectoides和教派。平伏的Krapovickas和Gregory [16];栽培花生属于教派。arachis..Many useful resistances to a number of diseases (e.g. early leaf spot, late leaf spot, peanut rust and rosette disease) and pests (such as nematodes, armyworm and corn earworm) that can cause serious yield loss in the cultivated peanut have been identified from the wild Sect.arachis.物种(2].例如,加入一个.Duranensis.Krapov。w。c。格雷戈里和A. Cardenasii.Krapov。& 厕所。属于教派的格雷戈里。arachis.已被发现对12种或更多不同的疾病/害虫具有抗性,是栽培花生新抗性等位基因最丰富的两个来源[2,17].为了更有效地利用这些丰富的遗传资源进行花生育种,更好地了解种内的基因组和系统发育关系arachis.是一个先决条件。
如上所述,属中有九个分类分类部分arachis.它们是根据形态、杂交性、细胞遗传学、杂交活力和地理分布而排列的[2,4,16,18,19然而,该分组已被分子发育研究挑战[20.,21].在九arachis.部分,教派。arachis.包括栽培花生是最大的,最多样化的,最多的一个:它包括超过三分之一的arachis.一年生和多年生物种,它们的染色体数目、倍性水平和基因组类型也可能不同[1,4].许多人试图推断它们之间的系统发育关系arachis.虽然标记和研究之间的不一致是很常见的[8,10,11,14,20.,22,23,24,25,26].快速发展的高通量测序技术可以为我们提供良好的机会,利用叶绿体(CP)基因组数据来帮助改善情况[27,这是由于cp基因组数据在系统发育分析中的几个优势。首先,cp基因组数据包含许多不同的基因位点和非编码区,它们包含相对大量的DNA序列信息,这不仅提高了系统发育推断的分辨率,同时也极大地减少了传统系统发育构建中与单基因有限序列信息相关的随机误差[28,29].第二,作为母系遗传,cp基因组在arachis.为推测栽培花生的母系来源提供了最好的工具。第三,cp基因组的单倍体特性严重抑制了非同源重组事件的发生,这将使cp基因组在系统发育分析中较少遭受重组[30.,31].最后,与核基因组相比,cp基因组的大小相对较小,这意味着它相对便宜、容易测序和分析[27].
除了上述在系统发育分析方面的优势外,cp基因组在开发DNA条形码方面也很有用,例如,在区分类群方面,以及cp基因工程将外源基因转移到cp基因组中[27].与核转基因植物相比,叶绿体基因工程有几个优点,包括转基因表达水平高,转基因不会通过花粉逃逸[27].现在已经有很多cp基因工程的成功案例了[27].此外,cp基因也被发现可能有助于寄主植物对环境胁迫的抵抗[32,33].虽然cp基因组是非常有用的,但是,仍然有非常有限的数量的cp基因组可用arachis.到目前为止的物种。前两个arachis.完成的完整CP基因组来自栽培花生,一个.hypogeae,并被Schwarz等人报告。[34]和prabhudas等。[35].之后,Yin等人[21]获得了七种不同的cp基因组的完整序列arachis.种,包括驯化花生,而Wang等[36.]探索了四个完整的CP基因组序列一个.hypogeae植物品种。总而言之,只有十三个arachis.到目前为止,来自七个不同物种的完整cp基因组已被测序。
在本研究中,我们共组装了12个完整的cp基因组arachis.种,其中11属。arachis.而最后一个来自教团。Erectoides这些数据,这些数据代表了丰富的遗传变异来源,可能对花生改善具有巨大潜力。这些序列与早期发表arachis.Cp基因组数据帮助我们更好地理解arachis.Cp基因组和种内种间的系统发育关系arachis.部分,并提供更多的信息,野生母系来源的栽培花生。
结果
收购的基本特征arachis.叶绿体基因组
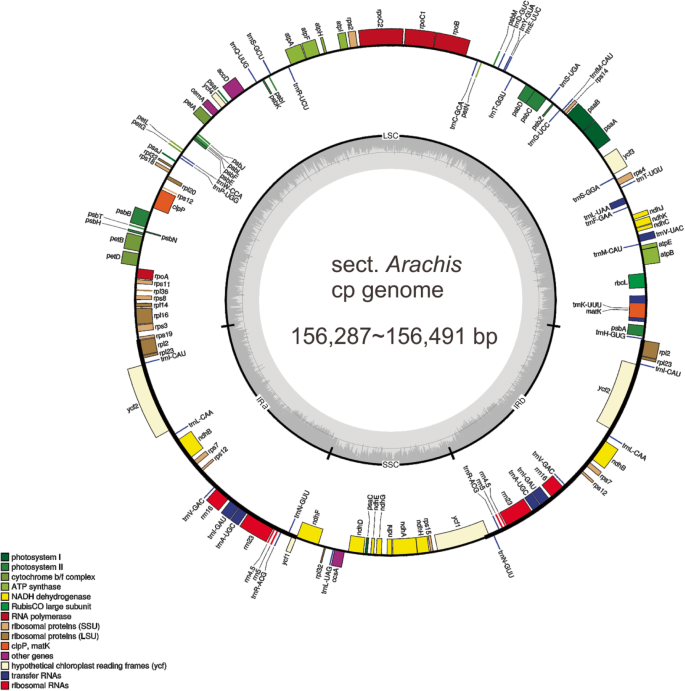
总共十二个arachis.属组的种。阿拉西(A. MonticolaKrapov。& Rigoni [GRIN accession number: PI-219824],A. Duranensis.[pi-263133],答:stenospermaKrapov。& W.C. Gregory [PI-338280],答:batizocoiKrapov。& 厕所。格雷戈里[PI-298639],A. Cardenasii.(π- 262141),A.掌声Martius Ex Krapov。&Rigoni [收集器号:Manso 588],A. Retrentina.(Burkart) Krapov。& W.C. Gregory [PI-331192], a hoehneiKrapov。& W.C.格雷戈里[收藏家编号。KG30006):, a chacoensisKrapov。& W.C.格雷戈里(现在称为答:diogoihoehne)[pi-276235]和A. Villosa.Benth。(π- 210555),答:ipaensisKrapov。& 厕所。格雷戈里[37.])和教派。巴拉圭瓢虫属植物。paraguariensisChodat&Hassl。[PI-331187])具有它们的CP基因组在本研究中测序和/或组装(表1).与核基因组相比,土地植物的CP基因组具有高度保守的圆形DNA分子,其具有两种倒置的重复(IRA和IRB)(相同但相同的方向),由小(SSC)和大(LSC)单拷贝(SC)区域[38.].在本研究中组装的12个cp基因组具有典型的四部分结构(图4)。1和2),总长度从156,287 bp(答:stenosperma)至156,491 bp(A. Cardenasii.)(额外的文件1),与之前发表的类似arachis.cp基因(21,34,35,36.].四种不同的基因组区域范围为85,830 bp(A. Duranensis.)至85,980 bp (A. Villosa.),由18796 bp (A. Retrentina.和答:monticola)至18,942 bp (A. Cardenasii.),由25,776 bp (答:ipaensis)至25,842 bp (答:batizocoi)对于IRA / B(附加文件1).
和之前的报道一样arachis.cp基因(21,34,35,36.],在12个组装的CP基因组中的每一个中发现了110个独特的基因,包括四个核糖体RNA(RRNA)基因,76个蛋白质编码基因和30转移RNA(TRNA)基因(附加文件1和2).对于所有12个组装的CP基因组相同,这些110基因的基因阶相同(图。1),也符合所有已发布的arachis.到目前为止CP基因组[21,34,35,36.].另外,六个鉴定的TRNA基因和11个蛋白质编码基因包含内含子(附加文件2).
与早期研究相似[34,35], 12个cp基因组的总GC含量为36.3 ~ 36.4%,且GC含量在不同基因组区域间分布不均匀:IRs(42.9 ~ 43.0%)的GC含量高于LSC(33.8%)和SSC (29.9 ~ 30.3%)1).IRs的高GC含量主要是由于一个基因区(包括rrn23,的环境a-ugc和的环境l-GAU基因)的GC含量与cp基因组的其他部分相比突出(图。2).
遗传变异和SSRS
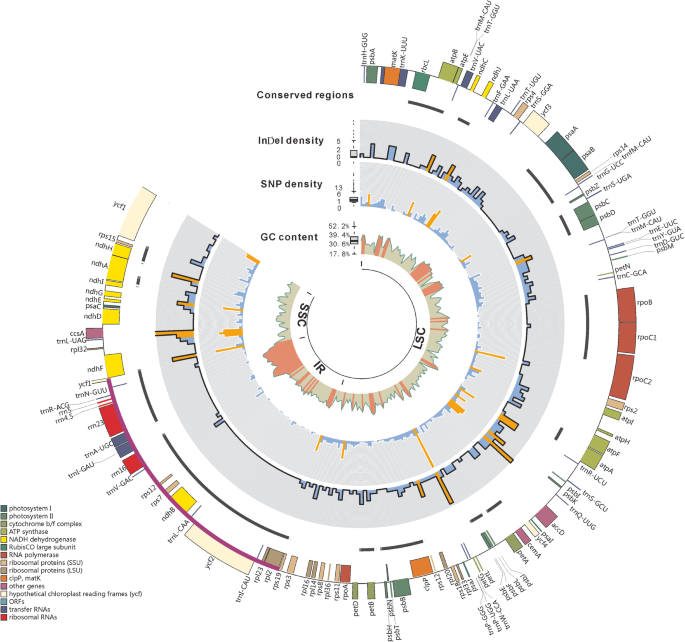
十二cp基因组中被组装在目前的研究中,共有1368个单核苷酸差异(snd)(0.87%),和大多数的snd LSC内分布地区(LSC序列的959人,占1.11%),但是SSC地区包含snd的比例最高(299 1.58%),而IRa/b区SND含量最低(各55,0.21%)2, 图。2).cp基因组中snd密度最高的区域位于两者之间的基因间隔区PSB.e和宠物在LSC内发现了47个snd3.).
在十二个CP基因组中发现的311个插入/缺失(indels)(0.20%),其中90%是短indel(1-10 bp)(表2).这些基因在4个cp基因组区域中的分布与snd非常相似:LSC的数量最多(241个,0.27%),SSC的密度最高(64个,0.34%),而IR区域的数量和密度都最低(每个区域6个,0.02%)2, 图。2).cp基因组中插入量最高的区域位于两个500 bp长的序列区段,其中一个区段包括两个基因组之间的基因间隔的环境q-uug和accD基因在LSC内,而另一个基因在SSC内,由基因间间隔组成rpl32和的环境l-uag基因,的环境l-uag基因以及介质之间的基因间隔物的环境l-uag和ccs一个基因。在这两个区域,分别检测到11个探针(见表)4).Vista分析的结果与SNDS和Indels的结果一致:几乎整个IR地区受到保守,而SCS内的识别的保守区则是短暂的并且散射(表4).
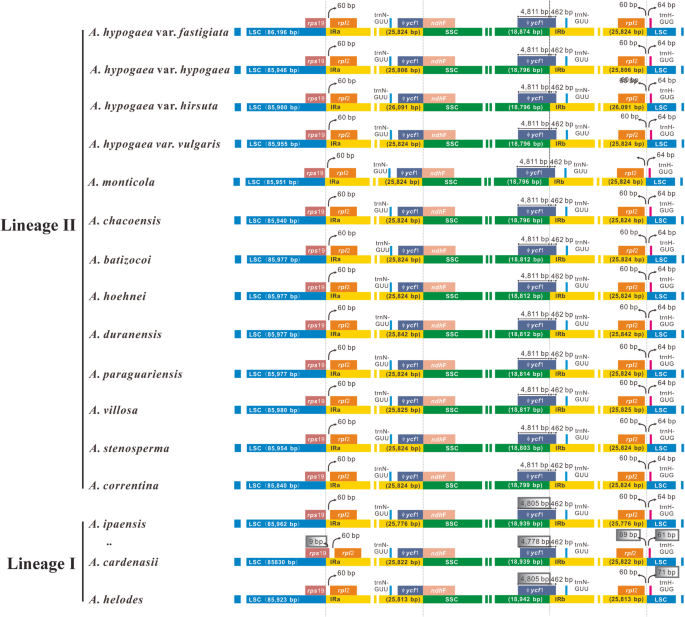
虽然红外地区相当保守,但IR边界甚至在物种中也可能变化[39.因此,为了检测可能存在的IR边界多态性,我们比较了12个组装的基因组之间的4个IR- sc边界,但在IRa-SSC边界未发现差异,而在LSC-IRa、SSC-IRb和IRb-LSC边界仅发现微小差异一个.ipaënsis.,一个.cardenasii.和一个.掌声(图。3.).的石头剪刀IRA-LSC边界处的19个基因从LSC区域扩展到IRA侧的9bp一个.cardenasii.虽然它在其余物种的LSC-IRA结处停止(图。3.).长度ycf.SSC-IRB边界的1基因(SSC)为4805 bp一个.ipaënsis./一个.掌声和4778 bp for一个.cardenasii.,比其他物种短,均为4811 bp(图4)。3.).在IRb-LSC边界的一侧,在rpl2基因(IRB中)和IRB-LSC结长度为69 bp一个.cardenasii.而其他物种的间隔体长度为60 bp。在IRb-LSC边界的另一侧,IRb-LSC连接处与的环境H-GUG基因(LSC中)分别为61bp和71 bp一个.cardenasii.和一个.掌声虽然其余物种的虽然所有64bp都是64 bp(图。3.).
对于MISA分析,在12个组装的CP基因组内检测到69个通用SSR基因座,其中59是单核苷酸,九个是二核苷酸,一种是四核苷酸。大多数已识别的SSRS由A或T组成,这与早期关于其他出株植物的CP SSRS的观察一致[40,41.].在获得的12个SSR位点中,有41个存在变异arachis.CP基因组,其中大部分(75.6%)位于LSC区域,其次是SSC(24.4%),没有在IR中找到(附加文件3.).对41个可变SSR位点分别设计了PCR引物(附加文件)3.).
所研究的系统发育arachis.物种
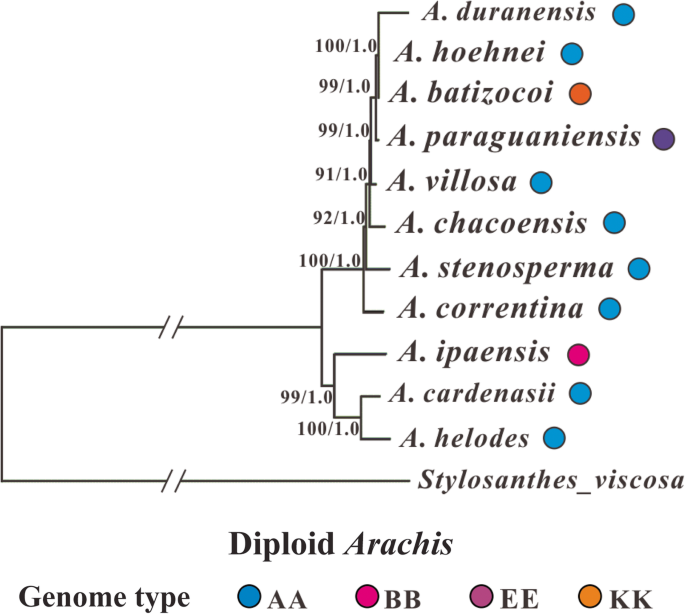
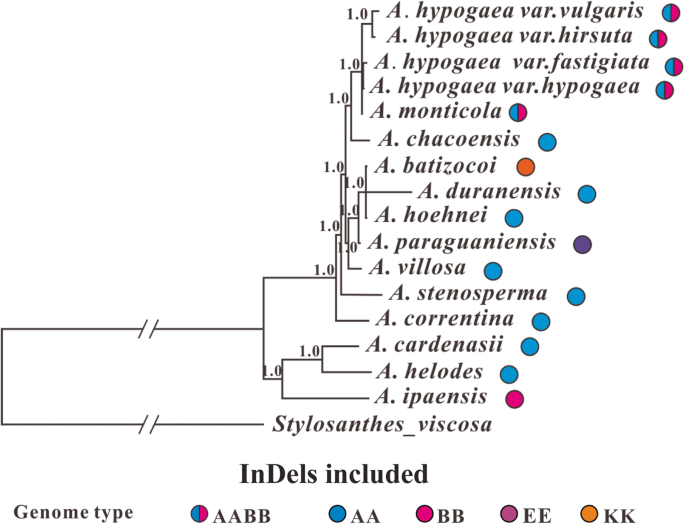
两种不同的系统发育方法(最大可能性和贝叶斯推理)用于推断分析物种之间的系统发育关系,并且两种方法产生几乎相同的树木,因此只显示了贝叶斯推理系统发育树(图。4)(可根据要求提供最大似然树)。考虑到IR区域的取代速度较慢,可能不适合推断密切相关的Sect之间的关系。arachis.物种,因此我们构建了有和没有它们的系统发育。此外,对倍性水平不同的物种进行系统发育分析可能会产生不同寻常的结果,与那些只涉及具有相同倍性水平的物种相比[1,20.],因此我们试图排除两个四倍体物种(一个.hypogeae和答:monticola)仅使用二倍体物种与全基因组数据一起推断系统发育树。此外,在上述系统发育分析中不考虑诱导,然而,嵌入在诱导中的信息可能有助于改善密切相关的物种的系统发育的分辨率[42.].因此,我们表现出从整个CP基因组的indel信息的系统发育的贝叶斯介绍。然而,在我们考虑的所有不同情况中,在所获得的系统发育树中的主要谱系和余序的安排总是保持一致(图。4,5,6和7).下面,我们只会介绍基于整个CP基因组数据的贝叶斯推理树的结果(不包括indel信息)。
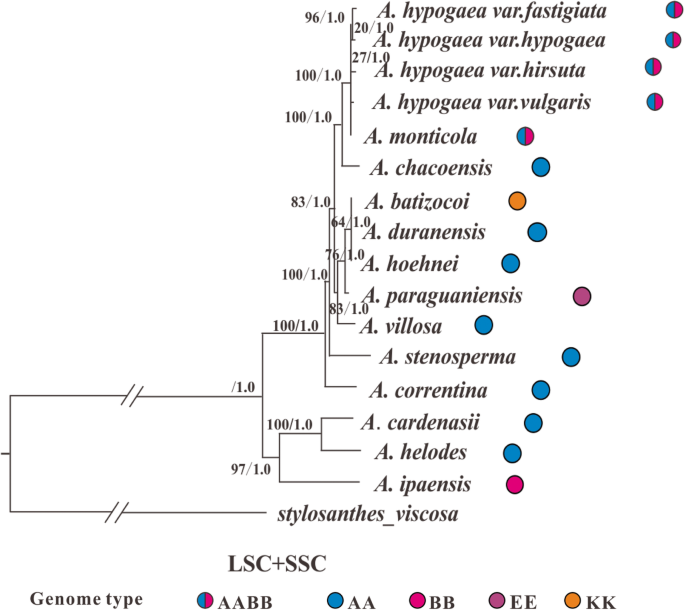
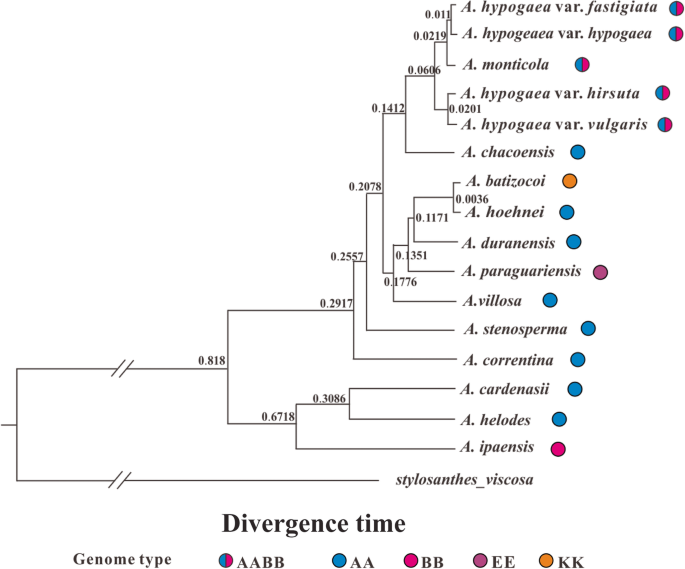
总共16个arachis.基因组(本研究中组装的十二种基因组加四年前发表答:hypogaea基因组织在这种系统发育分析中被列入这部分,并且它们在推断的系统发育树中落入了两个良好的支持主要血谱段(I,II)(自动启动[BS]值:100;贝叶斯后概率[BPP]:1.0) (图。4).血统I由一个BB基因组物种组成(一个.ipaënsis.)和两个AA基因组物种(一个.cardenasii.和一个.掌声),鞋线II包括其余物种(AA基因组种类:一个.Duranensis.,A. Hoehnei.,答:chacoensis,一个.摘要,一个.stenosperma,A. Retrentina.;AABB基因组种:4个答:hypogaea品种和一个.monticola;KK基因组种类:一个.batizocoi;EE基因组种类:A. Paraguariensis.) (图。4).我们的分子年代分析表明,这两个主要的世系在81.8万年前分裂(Mya)(图。8),此发散时间略短于间估计的发散时间一个.Duranensis.(在本研究中属于谱系II)和一个.ipaënsis.(本研究属于I系)(2.16 Mya), Bertioli et al. [43.,考虑到后者是基于双亲本遗传的核基因组数据,而本研究使用cp基因组(单亲本遗传)数据,这可能并不令人惊讶。两种系的GC含量差异不大,I系为35.13%,II系为35.09%。
在谱系I内,两个AA基因组物种(一个.cardenasii.和一个.掌声)紧密地组合在一起(BS: 100;BPP: 1.0),它们最近的共同祖先可以追溯到0.3086 Mya(图。4和8).相比之下,这两个AA物种和BB基因组物种的分裂时间(一个.ipaënsis.)要早得多:0.6718 Mya(图。8).
谱系II到最近共同祖先的时间为0.2917,该谱系内的物种分化迅速(图2)。8).在谱系II中,可以识别出两个不同的upblineages:第一个Sublineage(BS:100; BPP:1.0)由两种AABB基因组物种组成(答:hypogaea和一个.monticola)加上一个AA基因组物种(答:chacoensis),这个分支最近的共同祖先可以追溯到0.1412米亚(图。4和8).在这第一个亚系中,两个AABB基因组物种(栽培花生和一个.monticola)形成一个高度支持枝(BS: 100;BPP: 1.0),估计该演化支到最近共同祖先的时间为0.0606 Mya。第二分支(BS: 82;BPP: 1.0)在谱系II中包含3个AA基因组物种(一个.Duranensis.,A. Hoehnei.,一个.摘要,),一九龙(一个.Bartizocoi.)和一个EE基因组(A. Paraguariensis.).这两个分支之间的分裂可能发生在0.2078 Mya(图。8).
讨论
密切的野生亲戚一直为许多经济上重要的作物促进有价值的遗传资源[44.,45.,46.,47.].本研究成功获得了12个完整的cp基因组arachis.与栽培的花生(答:hypogaea),这是全球最重要的油厂之一。与十二个相关的丰富的遗传资源arachis.CP基因组数据可能对未来的花生品种改善具有巨大潜力。
与其他陆生植物相同[48.,49.,50.),arachis.cp基因组是一个单一的环状分子,显示出一个四方结构(LSC, IRa, SSC, IRb)。此外,基因组大小、基因组成和顺序、GC含量以及IR-SC边界也比较保守arachis.在本研究中获得的CP基因组(附加文件1)和早前出版的13本arachis.cp基因(21,34,35,36.].尽管如此,在获得的12个cp基因组中仍发现了大量的遗传变异(1368个SNDs和311个indel),这些遗传变异与本研究检测到的69个SSR位点可能为今后的研究提供有用的工具。
高度保守的IR地区
在四个cp基因组区域,两个红外区域高度保守的比较两个SC区域,所反映的事实,只有不到8%的新区,indels SSRs,已确定在本研究中是位于红外区域即使爱尔兰共和军和IRb构成约三分之一的基因组(无花果。1、表2).这种低水平的遗传变异在IR区域(与SCs相比)在植物物种中非常普遍[51.,52.,53.].一种可能的解释是,对于单植物细胞中存在的多个拷贝存在的CP基因组,具有针对新突变的微小偏差的基因转化将更有效地减少两种IR(倒置重复)区域的突变载荷而不是SC(单一拷贝)区域由于IRS的重复性质[51.,52.,54.,55.,56.].这种转化偏压可能来自基因转换期间错配修复系统的基础偏好[54.,57.,58.].
教派中的两个主要谱系。arachis.
在教派中有两种主要的基因组类型。arachis.:AA(染色体数字:2n = 20)和BB(2n = 20),这也非常重要,因为四倍体栽培花生具有由它们两者组成的复杂基因组(AABB; 4N = 40)[2].除AA、BB、AABB外,分。arachis.物种也可能具有DD (2n = 20)、FF (2n = 20)、KK (2n = 20)或非整倍体(2n = 18)的基因组类型。我们的系统发育分析表明,所分析的组。arachis.类群分为两个主要的谱系:第一个(谱系I)包括一个.ipaënsis.,这是这里研究的唯一的BB基因组物种,以及两个AA基因组物种(图。4).第二个谱系(谱系II)包含包含两种AABB基因组种,六种AA基因组物种和一个KK基因组物种的分类群。这两个主要谱系在派对内观察到。arachis.通过引导值非常好(图。4),如果没有四倍体答:hypogaea和一个.monticola统一他们;它们可能是合适的分类为单独的分类部分[1,20.].我们的发散时间估计表明,这两个主要谱系的最近共同的祖先被约为0.818 Mya,并且从这个共同的祖先血统上,我最初是衍生的(至少0.6718 Mya),而血统II最近衍生出来(0.2917 mya快速)快速(图。8).cp基因组的大小和结构,基因含量和顺序,以及GC含量在两个谱系之间都很保守。然而,I系物种cp基因组的IR-SC边界区域已经被发现在长度上有所不同,并且与那些完全相同的II系物种不同(图。3.).两个主要教派的存在。arachis.谱系是由基因分型测序(GBS)方法生成的核基因组数据(未发表的观察)确认的。
类似物种在教派中分组(分为两个主要谱系)。arachis.也在其他一些研究中观察到(附加文件4)[11,14,20.,21,22,24,25,59.,60.,61.,62.].这些研究可能有所不同arachis.但在几乎所有这些研究中,II系或类似系都以AA基因组物种为主,并倾向于排除除AA和AABB以外的基因组类型(附加文件)4).相比之下,血统I或类似的更多样化,可以接受任何基因组类型(AA, BB, AABB, DD, FF, KK或非整倍体),已被鉴定在Sect。arachis.但略微喜欢BB基因组种(附加文件4).
有趣的是,AA基因组属属。arachis.都是多年生的(除了一个.Duranensis.,一个.stenosperma和A. Hoehnei.),虽然所有其他各种基因组类型(例如BB,DD,FF,KK和AABB)属于年度/双年度(Sect。arachis.是唯一的两个arachis.有一年生种的部分;另一个是Sect。Heteranthae)[4].一年生/二年生和多年生物种的基因组类型组成可能与较早的发现一致,即一年生物种一般比多年生物种的分子进化速率要高得多[63.,64.,65.].而且,在原产地arachis.,南美洲,每年/两年生物种的分布区域比常年物种的环境条件相对多。在南美洲,教派的整体地理分布。arachis.物种有一个奇怪的形状,有点像大写的T [4].多年生赛。arachis.物种占该T形范围的垂直轴,或多或少地与经络57°和58°符合西部,主要包括巴拉圭和乌拉圭河的流域[4].而一年生/二年生物种主要分布在“T”形地理分布的两个分支:东托坎廷斯河,Mamoré河和Guaporé河,以及西南干燥的“charco”地区(一直到安第斯山脉的山麓)[4].在这些臂区,年/两年生种类通常适应非常紧张的环境条件,例如长期的淹没和周期性干旱[4].
尽管存在两种主要的世系在教派。arachis.这些世系的物种组成的整体模式是明显的,然而,哪个世系是一个教派。arachis.物种应放置并不总是清晰。例如,根据本研究,KK基因组种类,一个.batizocoi然而,大多数分析的AA基因组物种都属于谱系II,一个.batizocoi已被发现出现在三个早期的叶绿体系统发育研究中的血统I-等效的疏水板[14,20.,21].另一个例子是AA基因组种类一个.cardenasii.和一个.掌声然而,在本研究中落入谱系I,经常在早期研究中发现血统II等同的思考[11,20.,21,60.].而且,A. Paraguariensis.(属于教派。Erectoide),这是唯一的非教派。arachis.本研究获得的物种,最初被选为外群,但后来证明是密切相关的一个.batizocoi(kk),A. Hoehnei.(aa)和一个.Duranensis.(AA)在谱系II内(图。4).值得注意的是,相似物种之间的混合arachis.章节并不少见[11,20.,21,24,66.].例如,Yin等人。[21]研究了七arachis.物种,五个属于教派。arachis.虽然其余两个是教派的成员。平伏的;这两个教派并没有合并在一起。平伏的种分别在不同组内嵌套。arachis.物种组。
这些物种关系的这种性能可能是几种不同原因的结果。首先,在不同的情况下报告了高水平的遗传变异arachis.物种(尤其是野生物种)[4,5,6,7,8,9,10,67.].这种高水平的内部侵入性遗传变异可能至少部分地是由于自动生殖系统和地下果实习惯arachis.种间和种内基因流动严重受限[4].因此,同一物种的不同样本可能具有不同的遗传结构(如一个.Duranensis.基于这些不同样本的系统发育推断很可能会导致非常不同的物种关系,因此,在未来的系统发育研究中,需要考虑每个物种更多具有代表性的样本。第二,arachis.物种并不总是完全不相容的,即使在不同的基因组类型或部分之间也有很好地记录了杂交[2,14,15,59.];可能本质上生产的三种杂种可能会使物种边界模糊。第三,考虑到近期的近期分歧。arachis.物种(<1 mya,图。8),祖先多态性也可能保持在远端物种内,并因此使系统发育意义复杂化。此外,在考虑(单一基因或基因组数据)的遗传信息量中,遗传数据类型(AFLP,RFLP和DNA序列等)的分析方法(例如UPGMA和最大可能性)的分析差异也是如此与所获取的数据集的继承模式(双亲子女或单父母)一样,所有人都会对物种关系的推动产生影响[29].
花生的母系来源(一个.hypogeae)
在两个主要教派之间。arachis.从本研究推断的系统发育树中观察到的谱系,栽培花生属于谱系II,在这一组中,考虑了四种一个.hypogeae品种混合在一起一个.monticola.这一结果与先前的观点并不冲突一个.monticola可能是栽培花生的直接野生祖先[11或作为栽培花生和野生花生之间的渐渗衍生物arachis.物种(68.,69.].这些早期的观点是基于各种不同证据的综合。例如,这两个物种是组内唯一的四倍体物种。arachis.它们通常在系统发育分析中组合在一起[2,26,66.].此外,这两个物种的地理分布彼此接近[70它们之间的杂交已经有了很好的记录[14,16].
从上面的讨论中,我们已经知道谱系II或等同物由AA基因组物种,特别是“抗切除”BB基因组物种,尤其如此,这些树木是由现在反映的CP序列推断的系统发育树和三个早期的研究[14,20.,21].实际上在这四项研究中,栽培花生嵌套在血统II或等同物中。考虑到叶绿体基因组是母体遗传的arachis.如早期的研究所示,其中F1杂种之间的两个arachis.物种始终与他们的母体父母一起在基于叶绿体DNA构建的系统发育树中进行[14,本研究结果表明,四倍体栽培花生(AABB)和四倍体野生花生(一个.monticola;AABB)是一个AA基因组物种[14,20.,71,或者可以说A的基因组一个.hypogeae和一个.monticola是他们的母体祖先的贡献。
目前,最受欢迎的观点是哪些物种作为基因组捐赠者一个.hypogeae和一个.monticola是,一个.Duranensis.(aa)和一个.ipaënsis.(BB)分别贡献A和B基因组。这一普遍接受的观点得到了基因组类型、地理分布、杂交性、细胞遗传学分析、分子分析、系统发育分析和基因组序列比较等证据的支持[11,14,43.,60.,70,71,72].然而,在本研究中,一个.hypogeae和一个.monticola与谁都不亲近一个.Duranensis.也不一个.ipaënsis.在推断的系统发育树(图。4),相反,这两个四倍体物种形成了一个有良好支持的亚群答:chacoensis(AA)。然而,我们的结果是不能得出结论答:chacoensis, 代替一个.Duranensis.作为A基因组的供体一个.hypogeae和一个.monticola由于不同原因的组合。首先,一个.Duranensis.有较广的地理分布,而且据报该种内有许多种内变异[8,22,71,72]如果使用具有不同遗传构成的不同样品用于系统发育推理,则结果可能非常不同。下一个,一个.Duranensis.已被证明能够与其他人杂交arachis.物种(14],导致三种基因流动,也可能模糊物种边界。尽管如此,虽然叶绿体基因组在引言中提到的那样具有大量优点,但由于引用的情况相对容易受到迟发的问题和祖先多态性,这些问题在推动密切相关的物种之间的系统发育关系时经常遇到的祖先多态性。他们的母亲继承模式[73,74].
结论
花生是世界上最重要的油料作物之一,野生花生品种的高度变异可能为栽培花生的改良提供丰富的有用等位基因。本研究获得了12个cp的全基因组序列arachis.种,属组11种。arachis.;栽培花生也是派别的成员。arachis..至于其他陆生植物品种[48.,49.,50.], cp基因组的大小和结构,以及基因的含量和顺序在12个获得的cp基因组中高度保守。然而,从获得的基因组中已经鉴定出大量的snd、indels和SSRs,而且这些snd、indels和SSRs大部分分布在两个单拷贝基因组区(LSC和SSC)。两个反向重复基因组区域(IRa和IRb)的遗传变异水平非常低,这可能是由于基因转换偏置造成的。对获得的基因组进行系统发育分析,确定了组内的两个主要谱系(I和II)。arachis..我们的研究结果和早期的许多研究表明,谱系II以AA基因组物种为主,大部分是多年生的,而谱系I物种的基因组类型相当多样化;教派。arachis.与AA以外的基因组类型的物种都是每年/两年期[4].此外,在线植物II中发现四倍体栽培花生,其与叶绿体的母体遗传模式一起表明它是一种AA基因组种类,作为栽培花生的母体供体。总之,本研究中获得的十二个CP基因组不仅有助于我们理解遗传基础和系统发育关系arachis.物种更好,但也为我们提供了大量的遗传资源,可能对未来的花生改良有价值。
方法
植物材料和基因组测序
总共十二个arachis.属组的种。arachis.(答:monticola,A. Duranensis.,答:stenosperma,答:batizocoi,A. Cardenasii.,A.掌声,A. Retrentina.,A. Hoehnei.,答:chacoensis(现在被称为答:diogoi),A. Villosa.,答:ipaensis和教派。Erectoides(A. Paraguariensis SSP。paraguariensis)被纳入本研究(表1).这些野生花生物种的种子(除了答:ipaensis,见下文)最初来自,并被全国野生识别arachis.中国农业科学院苗圃,石油作物研究所,武汉,武汉。所有分析的物种都具有AA基因组类型,除外答:batizocoi(kk),答:ipaensis(BB),A. Paraguariensis.(EE) [2].从单个单月大的植物留下,代表每个物种(除外)答:ipaensis)使用DNeasy植物迷你试剂盒(德国Qiagen,德国)来收集全基因组DNA分离收集的(在温室条件下成长)。使用超级凝胶染料(Usegbright Inc.,Suzhou,China)通过琼脂糖凝胶电泳检查DNA质量。对于所有分析的样品(不同的物种是“用不同的条形码适配器的”索引“索引)的单个成对终端,具有约350bp的平均嵌入式大小的单一末端库,遵循制造商的协议(Illumina,北京,中国)。构造的库首先使用高灵敏度测定套件(Caliber,USA)对CALIPER Labchip GX进行评估,然后在流动单元上杂交和放大,以使用TruseQ PE群集套件V3-在CBOT平台上产生克隆簇。CBOT-HS(Illumina,北京,中国)。通过合成的全CP基因组测序使用Truseq V3-HS试剂盒(Illumina,北京,中国)对Illumina Hiseq Xten平台进行。
基因组组装与注释
为每个样品产生> 1 GB原料配对末端读取的Illumina测序,并且将这些数据沉积到NCBI序列读取存档(SRA)(Bioproject Incession No.Prjna543570)中。这些原始配对结束读取,加上一个原始读取数据集答:ipaensis从NCBI SRA数据库下载(登录号:SRX2701518) [37.]使用NGS QC Toolkit V2.3.3进行分析,用于在质量检查和去除适配器序列后过滤低质量数据[75];分别设定读取长度和PHRED分数百分比的截止值,分别为80和30.每种样品获得305,336-3,503,151个高质量读数,该样品在存在时产生293-3362倍的CP基因组覆盖映射到来自的参考CP基因组答:hypogaea(Genbank [76登录号码KX257487, [35)用领结[77](附加文件1).cp基因组的高质量reads(从上一步获得)被提取,并使用de novo assembler SPAdes v3.9.0组装成contigs [78](具有几种不同的K-ME大小:93,105,117和121)。这些组装的CONDIGS通过Novoplacty V2.6.2进一步组装成完整的CP基因组,这是专门用于组装细胞器基因组的设计[79].使用具有默认参数的Dogma工具注释完整的CP基因组[80].OGDraw v1.2 (81]用来绘制完整的cp基因组图像。
在亮片软件的帮助下,每个组装完成的cp基因组[82],生成SQN文件。然后将SQN文件提交给GenBank(登录号:MK144818答:monticola,MK144823 for.A. Paraguariensis., MK144822A. Duranensis., MK144819答:stenosperma, MK144820答:batizocoi, MK144824A. Cardenasii., MK144826A.掌声, MK144828A. Retrentina.MK144827 for.A. Hoehnei.,MK144821 for.答:chacoensis(现在被称为答:diogoi),MK144829为答:ipaensis和MK144825A. Villosa.).
遗传变异分析
无论是snd和indels检测从测绘(到答:hypogaea参考基因组)使用GATK分析领结的结果(见上)[83](倍增性设置= 1)。Vista Server [84]用于鉴定保守基因组区域。利用Circos方法对整个cp基因组中snd和indels的密度(即每连续500 bp块中统计的snd或indels的数量)和保守区域进行可视化分析[85].使用MISA预测简单的序列重复(SSR)[86]除了单个SSR图案中重复单元的最小计数之外的默认参数:(10)单声道,(6)DI-,(5)三 - (4)TETRA-,(3)PENTA-和(3)六核苷酸重复单位[21].手动检查所有已识别的SSR并删除冗余结果。
系统发育分析和发散时间估计
为了更好地理解物种之间的关系arachis.,特别是在教派内部。arachis.,对cp全基因组数据进行系统发育分析。除了本研究获得的12个蛋白质包体基因组外,先前发表的4个不同的蛋白质包体基因组一个.hypogeae植物品种(Genbank登录号。var mg814006。fastigiataWaldron,MG814007 for Var。多毛的Kohler,Mg814008 for Var。hypogaeaL., MG814009为var。寻常的哈尔茨)[36.]还包括在分析中,因此,总共16种完整的CP基因组来自13arachis.种被认为是进行系统发育推断的依据。此外,一个cp基因组Stylosanthes人造丝(GenBank入库编号。MG735675)作为系统发育分析的外群;该cp基因组与arachis.物种(87在cp基因组中,这些基因组在进行分析时是可用的。在用于系统发育分析之前,17个cp基因组与HolmBlocks管道对齐(默认设置,除非指定)[88[尤其是处理大量分歧的序列数据快速有效,因此适用于克服可以通过相对远远端相关的外谷物种类引入的对准困难。在Holmblocks管道内,Progrestivemauve [89]首先用于鉴定保守基因组区域,然后在此基础上构建初步比对;然后由Gblocks对对齐进行修剪[90消除排列不整齐和分散的区域。
首先使用IQ树中实施的最大似然方法进行系统发育分析[91].执行了10次独立搜索,并使用10,000次非参数bootstrap重复来评估每个预测节点的统计置信度。MrBayes v3.2.5 [92然后用于通过Markov链蒙特卡罗方法进行贝叶斯的贝叶斯推论[92].我们运行100,000个马尔可夫链蒙特卡罗代的推论,抽样频率为1000代。前25%代的结果被丢弃为老化结果,其余的被用来构建50%多数规则共识树。
陆生植物cp基因组有四个典型区域:两个IR区域和两个SC区域,(见结果)[38.].IR区域的核苷酸取代率比SC区域低得多[52.,54.,93],因此可能不适合推断近缘系的系统发育。arachis.本研究分析的物种。这里,我们使用与上述相同的方法重建该系统发生,但不包括IR区域。
与那些只涉及具有相同倍性水平的物种相比,具有不同倍性水平的物种的系统发育分析可能会产生不同寻常的结果[1,20.],为了测试我们的研究是否如此,我们随后排除了两个四倍体物种(一个.hypogeae和答:monticola)仅使用双倍物种来使用全基因组数据推断出系统发育树。
此外,在上述系统发育分析中不考虑诱导,然而,嵌入在诱导中的信息可能有助于改善最近发散物种的系统发育的分辨率[42.].因此,我们对在本研究中观察到的311个indels进行了最后的系统发育分析arachis.全基因组数据考虑。在此步骤中,首先使用简单的Indel编码方法转换为二进制数据[94]使用SeqState软件[95].将获取的二元数据与普通核苷酸替换信息作为混合数据输入MrBayes,进行贝叶斯推断arachis.发展史。
最后,软件BEAST v1.7.2 [96用来估计不同的分歧时间arachis.物种。估计的属间发散时间司机和arachis.来自Saslis-lagoudakis等。[97]作为校准点。
可用性数据和材料
本研究中所使用的植物样品可根据要求从通讯作者处获得。已生成的数据集(组装完整的叶绿体基因组登录号:MK144818 for落花生monticola,MK144823 for.A. Paraguariensis., MK144822A. Duranensis., MK144819答:stenosperma, MK144820答:batizocoi, MK144824A. Cardenasii., MK144826A.掌声, MK144828A. Retrentina.MK144827 for.A. Hoehnei.,MK144821 for.答:chacoensis, MK144829答:ipaensis和MK144825A. Villosa.)或分析(叶绿体基因组的原始读数:SRX2701518答:ipaensis;栽培花生叶绿体基因组:KX257487, MG814006, MG814007, MG814008, MG814009)在GenBank (https://www.ncbi.nlm.nih.gov/genbank).支持本文结论的原始数据、本研究构建的最大似然树、基于基因分型测序(GBS)方法生成的核基因组数据(未发表的观察)的系统发育树(未发表的观察)均可从通讯作者获得。本研究的所有其他数据集都包含在手稿和附加文件中。
缩写
- BPP:
-
贝叶斯后验概率
- BS:
-
引导
- cp:
-
叶绿体
- indel:
-
插入/删除
- 红外光谱:
-
反向重复(地区)
- IRA,IRB:
-
两个相同但方向相反的红外区域
- LSC:
-
长单拷贝(区域)
- mya:
-
几百万年前
- SC:
-
单一副本(地区)
- 新区:
-
单核苷酸差异
- SSC:
-
短单副本(区域)
参考
- 1.
Bertioli DJ,Seijo G,Freitas Fo,Valls JF,Leal-Bertioli Scm,Moretzsohn MC。花生及其野生亲属的概述。植物类型康复。2011; 9:134-49。
- 2.
跟踪狂HT。利用野生植物改良花生。作物科学。2017;57:1102-20。
- 3.
yu scw。花生品种及其在中国的家谱。中国上海:上海科技出版商;2008年。
- 4.
Krapovickas A, Gregory WC, Williams DE, Simpson CE。属的分类学arachis.(豆科)。Bonplandia.2007年,十六7 - 205。
- 5。
利用分子标记检测栽培花生和相关野生种质资源的遗传变异。基因组.1991; 34:1013-20。
- 6。
希卢·KW,潜行者HT。花生与野生种的亲缘关系arachis.教派。arachis.(豆科):rapd的证据。acta botanica sinica(云南植物研究),1995;
- 7.
Kochert G, Halward T, Branch WD, Simpson CE。花生RFLP变异(arachis hypogaea.L.)品种和野生物种。Al Appl Genet。1991年; 81:565-70。
- 8.
Milla SR, Isleib TG, Stalker HT。分类之间的关系arachis.教派。arachis.AFLP标记显示的物种。基因组.2005; 48:1-11。
- 9.
Paik-ro og,smith rl,knauft da。限制片段长度多态性评估六种花生种类Avachis部分。Theor Appl Genet 1992; 84:201-8。
- 10.
de Carvalho MM, Hopkins MS, Mitchell SE, Kresovich S, Valls JFM, Ferreira ME。花生遗传多样性(arachis hypogaea.在基因组高变区分析的基础上,对其和野生近缘种进行了研究。BMC Plant Biol. 2004;4:11。
- 11.
Moretzsohn MC, Gouvea EG, Inglis PW, Leal-Bertioli SCM, Valls JFM, Bertioli DJ。栽培花生(arachis hypogaea.)使用内含子序列和微卫星标记的最密切相关的野生物种。Ann Bot。2012; 111:113-26。
- 12.
Fonceka D, Tossim H- a, Rivallan R, Vignes H, Lacut E, de Bellis F, Faye I, Ndoye O, Leal-Bertioli SC, Valls JF。花生染色体片段替代系的构建(arachis hypogaea.L.)使用野生合成和QTL测绘植物形态。Plos一个。2012; 7:E48642。
- 13.
Leal-Bertioli SCM, Godoy IJ, Santos JF, Doyle JJ, Guimarães PM, Abernathy BL, Jackson SA, Moretzsohn MC, Bertioli DJ。作用中的节段异源多倍体:通过多倍体杂交和同源重组增加多样性。Am J Bot. 2018; 105:1053-66。
- 14。
Tallury SP,Hilu KW,Milla SR,Friend SA,Alsaghir M,Stalker HT,Quandt D.基因组亲缘性arachis.部分arachis.(Fabaceae):分子和细胞遗传学证据。Al Appl Genet。2005; 111:1229-37。
- 15.
Fávero美联社,辛普森CE,瓦尔斯JFM,韦洛NA。通过杂交性研究栽培花生的进化落花生ipaensis,A. Duranensis.,答:hypogaea.作物科学。2006;46:1546-52。
- 16。
Krapovickas A,Gregory Wc。TAXONODIA DEL GENELO“arachis.(Leguminosae)“。Bonplandia.1994年:1-186。
- 17。
Mota APZ, Vidigal B, Danchin EGJ, Togawa RC, Leal-Bertioli SCM, Bertioli DJ, Araujo ACG, Brasileiro ACM, Guimaraes PM。比较野生根转录组arachis.发现NBS-LRR基因与线虫抗性相关。BMC Plant Biol. 2018;18:159。
- 18.
Fernández A, Krapovickas A. Cromosomas y evolution en "arachis.(豆科)”。Bonplandia.1994; 8:187 - 220。
- 19.
瓦尔斯JFM,辛普森CE。新物种的arachis.(豆科)来自巴西。巴拉圭和玻利维亚Bonplandia, 2005; 14:35-63。
- 20.
Friend SA, Quandt D, Tallury SP, Stalker HT, Hilu KW.属的物种、基因组和截面关系arachis.(豆科):一种分子系统发育。acta botanica sinica(云南植物研究),2010;
- 21.
尹冬梅,王勇,张晓刚,马晓丽,何小勇,张建华。花生叶绿体基因组资源的开发(arachis hypogaea.和其他种类的arachis..Sci众议员2017;7:11649。
- 22.
Gimenes MA, Lopes CR, Valls JFM。基因之间的关系arachis.基于AFLP的物种。生物医学杂志2002;25:349-53。
- 23.
Barkley Na,Dean Re,Pittman RN,Wang Ml,Holbrook CC,Pederson Ga。用M13尾的SSR标记和测序评估栽培和野生型花生的遗传多样性。Genet Res。2007; 89:93-106。
- 24.
王春涛,王学忠,唐元元,陈新东,崔发方,张继成,余少林arachis.基于内部转录的间隔序列。遗传资源作物evol。2011; 58:311-9。
- 25.
Bechara MD, Moretzsohn MC, Palmieri DA, Monteiro JP, Bacci M Jr, Martins J Jr, Valls JF, Lopes CR, Gimenes MA。属的系统发育关系arachis.基于ITS和5.8 S rDNA序列。BMC Plant Biol. 2010;10:255。
- 26.
Vishwakarma MK, Kale SM, sriswatthi M, Naresh T, Shasidhar Y, Garg V, Pandey MK, Varshney RK。全基因组范围内插入和删除标记的发现和部署为物种、基因组和属段关系提供了更深入的了解arachis..植物学报2017;8:2064。
- 27.
Daniell H,Lin C-S,Yu M,Chang W-J。叶绿体基因组:基因工程中的多样性,进化和应用。基因组Biol。2016; 17:134。
- 28。
吴志强,葛松。草中BEP分支的系统发育:来自叶绿体全基因组序列的证据。分子系统学进展。2012;62:573-8。
- 29。
Delsuc F,Brinkmann H,Philippe H.语文核核核科和生命之树的重建。NAT Rev Genet。2005; 6:361-75。
- 30.
拉尼尔HC,诺尔斯LL。重组是物种树分析的一个问题吗?系统医学杂志。2012;61:691 - 701。
- 31.
线粒体和叶绿体基因的遗传:定律、机制和模型。《Annu Rev Genet》2001;35:25 - 48。
- 32.
Hao DC,Chen Sl,肖PG。分子演化与氯化体成矿酶泡沫的阳性达尔文选择。j植物res。2010; 123:241-7。
- 33.
PEENG LW,Yamamoto H,Shikanai T.叶绿体NAD(P)H脱氢酶复合物的结构和生物发生。Biochim Biophys Acta-Bioenerg。1807; 2011:945-53。
- 34.
Schwarz en,Ruhlman Ta,Sabir Jsm,Hajrah Nh,Alharbi ns,Al-Malki Al,Bailey CD,Jansen RK。豆类的塑性基因组序列显示平行逆转和多重损失RPS16在papilionoids。中国科学(d辑:地球科学)2015;
- 35.
Prabhudas SK, Prayaga S, Madasamy P, Natarajan P. Shallow whole genome sequencing for assembly of complete叶绿体基因组序列arachis hypogaea.L.前植物SCI。2016; 7:1106。
- 36.
王j,李cj,燕cx,赵xb,山sh。四花生植物品种完全叶绿体基因组序列的比较分析。peerj..2018; 6:E5349。
- 37.
格鲁吉亚大学。DNA-SEQ一个.ipaënsis.: 叶子。2018年。https://www.ncbi.nlm.nih.gov/sra/srx2701518.
- 38。
崔丽丽,Leebens-Mack J, Wang L- s, Tang JJ, Rymarquis L, Stern DB, DePamphilis CW。利用参数自举法推断叶绿体基因组结构的适应进化。BMC Evol Biol. 2006;6:13。
- 39。
Goulding Se,Olmstead Rg,Morden CW,Wolfe Kh。叶绿体倒置重复的潮汐和流动。Mol Gen Genet。1996年; 252:195-206。
- 40.
匡德勇,吴辉,王永林,高利明,张世忠,卢磊木兰kwangsiensis(magnoliaceae):对DNA条形码和人口遗传学的影响。基因组.2011; 54:663-73。
- 41.
黄华,史超,刘勇,毛思宇,高立忠。13茶花高通量测序确定的叶绿体基因组序列:基因组结构和系统发育关系。BMC Evol Biol. 2014;14:151。
- 42.
Redelings BD, Suchard MA。将indel信息纳入快速出现病原体的系统发育估计。BMC Evol Biol. 2007;7:40。
- 43.
Bertioli DJ, Cannon SB, Froenicke L, Huang GD, Farmer AD, Cannon EKS, Liu X, Gao DY, Clevenger J, Dash S, et al.;的基因组序列落花生duranensis和落花生ipaensis是栽培花生的二倍体祖先。Nat麝猫。2016;48:438-46。
- 44.
费塔多A,亨利RJ。作物野生近缘的基因组学:扩大作物改良的基因库。植物生物技术学报2016;14:1070-85。
- 45.
Prohens J,Gramazio P,Plazas M,Dempewolf H,Kilian B,DízusMJ,Fita A,Herraiz FJ,Rodríguez-Burruezo A,Soler S. Introgrartiomics:使用作物野生亲属在育种过程中适应气候变化的新方法。Euphytica.2017; 213:158。
- 46.
邓佩夫夫,鲍特,安德森,基利安,史密斯,瓜里诺。野生近缘植物在作物育种中的应用。作物科学。2017;57:1070 - 82。
- 47.
张H,Mittal N,Lij,Barazani O,Song BH。回到野生亲属的野生遗传多样性,以进行作物改善。evol。2017; 10:5-24。
- 48.
光合真核生物质体基因组结构的研究。生物化学Mosc。2017;82:678 - 91。
- 49.
奥丁索娃等。高等植物和藻类质体基因组的结构和功能。杂志。2003;37:649 - 62。
- 50。
可汗A,可汗IA, Asif H, Azim MK.叶绿体基因组研究的当前趋势。生物技术学报2010;9:3494-500。
- 51。
伍尔夫KH。豆科植物叶绿体DNA中的核苷酸替换率依赖于反向重复的存在。中华医学杂志。2002;55:501-8。
- 52.
朱安,郭伟,顾磊杰,范伟,Mower JP。质体反向重复的进化动力学:扩张、收缩和损失对替代率的影响。新植醇。2016;209:1747-56。
- 53.
周世民,吴春生,吴春生。裸子植物质体基因组的进化。Adv Bot Res. 2018; 85:195-222。
- 54.
Birky CW,Walsh JB。在叶绿体和细菌基因组中偏置基因转化,拷贝数和表观突变率差异。遗传学.1992; 130:677 - 83。
- 55.
李F-W,Kuo L-Y,Pryer Km,Rothfels CJ。转移到塑性倒置的基因中,重复地显示出减速的取代率和升高的GC含量。基因组Biol Evol..2016; 8:2452-8。
- 56.
吴志生,周淑敏。苏铁质体的进化停滞和质体gc偏倚基因转换的第一例。基因组生物学进展。2015;7:2000-9。
- 57.
Nagylaki T,Petes Td。反复基因中的血管囊癌基因转化和维持序列均匀性。遗传学.1982; 100:315-37。
- 58.
沃尔什简森-巴顿。偏倚基因转换在单位点中性理论和基因组进化中的作用。遗传学.1983; 105:461-8。
- 59.
Leal-Bertioli SCM, Santos SP, Dantas KM, Inglis PW, Nielen S, Araujo ACG, Silva JP, Cavalcante U, Guimaraes PM, Brasileiro ACM。花生与栽培花生(a. hypogaea)的关系及其利用诱导的异源四倍体将野生基因渗入花生作物的潜力的研究。安机器人。2014;115:237-49。
- 60。
荣森,泰特PL, Horn R, Kochert G, Moore K, Abbott AG。栽培花生可能祖先的系统发育关系。J在这里。2003;94:334-40。
- 61。
何光华,巴克利娜,赵玉玲,袁敏,普拉卡什。属中种的系统发育关系arachis.基于基因序列。基因组.2014; 57:327-34。
- 62。
da Cunha FB, Nobile PM, Hoshino AA, de Carvalho MM, Lopes CR, Gimenes MA。基因之间的关系arachis hypogaea.L. (AABB)和二倍体arachis.具有AA和BB基因组的物种。遗传资源作物evol。2008; 55:15-20。
- 63。
Bousquet J, Strauss SH, Doerksen AH, Price RA。在进化速度上的广泛变化:种子植物中的基因序列。Proc Natl Acad Sci U S A. 1992; 89:7844-8。
- 64.
Andreasen K, Baldwin BG。花楸一年生和多年生谱系间的不平等进化速率:来自18S-26S rDNA内部和外部转录间隔的证据。Mol Biol Evol. 2001; 18:936-44。
- 65.
Laroche J,Bousquet J. Mitochondrial的演变rps3.多年生和年生学生和同源性的内含子nad5内含子1. mol Biol Evol。1999; 16:441-52。
- 66.
Koppolu R, Upadhyaya HD, Dwivedi SL, Hoisington DA, Varshney RK。属七个部分间的亲缘关系arachis.使用SSR标记研究。BMC植物BIOL。2010; 10:15。
- 67.
沃尔斯特·鲁克,Mahendar T,Aruna R,Nigam Sn,Neelima K,Vadez V,Hoiseton Da。地生中的高水平自然变化(arachis hypogaea.利用选定的信息SSR标记进行种质收集。植物育种。2009;128:486 - 94。
- 68.
尹dm,ji cm,ma xl,李h,张wk,li s,刘fy,zhao kk,li fp,li k。李克。同种异体一体化野生花生的基因组落花生monticola:De Novo集会。傻瓜。2018; 7:GIY066。
- 69.
Grabiele M,Chalup L,Robledo G,Seijo G.驯化花生的遗传和地理起源,如5s RDNA和叶绿体DNA序列所证明。植物系统中的Evol。2012; 298:1151-65。
- 70.
Seijo JG, Lavia GI, Fernandez A, Krapovickas A, Ducasse D, Moscone EA落花生duranensis和一个.ipaënsis.是野生二倍体祖细胞一个.hypogaea(豆科)。中国科学(d辑:地球科学)2004;
- 71.
高志强,李志强,李志强,等。异源四倍体驯化花生的RFLP和细胞遗传学证据。arachis hypogaea.(豆科)。中国科学(d辑:地球科学)
- 72。
Seijo G, Lavia GI, Fernandez A, Krapovickas A, Ducasse DA, Bertioli DJ, Moscone EA.栽培花生(arachis hypogaea.(豆科)及其近缘类群的双GISH揭示。Am J Bot. 2007; 94:1963-71。
- 73。
刘建强。叶绿体与线粒体DNA的基因渐渗越多,基因流动越少Picea Asperata.并与其他针叶树的比较。摩尔生态。2009;18:1396 - 407。
- 74。
Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE. 83个质体基因的系统发育分析进一步解决了eudicots的早期分化。美国国家科学研究院2010;107:4623-8。
- 75。
Patel RK,Jain M.NGS QC Toolkit:用于下一代测序数据的质量控制的工具包。Plos一个。2012; 7:E30619。
- 76.
- 77.
人类基因组短DNA序列的超快和记忆效率比对。基因组医学杂志。2009;10:R25。
- 78.
Bankevich A,Nurk S,Antipov D,Gurevich AA,Dvorkin M,Kulikov As,Lesin VM,Nikolenko Si,Pham S,Prjibelski广告。黑桃:一种新的基因组装配算法及其在单细胞排序的应用。j计算biol。2012; 19:455-77。
- 79.
Dierckxsens N,Mardulyn P,Smits G. novoplasty:新创从全基因组数据组装细胞器基因组。核酸Res. 2016;45:e18。
- 80.
Wyman Sk,Jansen Rk,Boore JL。用教条自动注释细胞细胞基因组。生物信息学.2004; 20:3252-5。
- 81.
Lohse M, Drechsel O, Bock R. OrganellarGenomeDRAW (OGDRAW):一种易于生成高质量定制质体和线粒体基因组图形图的工具。咕咕叫麝猫。2007;52:267 - 74。
- 82.
Sequin, DNA序列提交工具.https://www.ncbi.nlm.nih.gov/projects/Sequin.
- 83。
基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20:1297 - 303。
- 84。
作者简介:陈志伟,副教授,博士,主要研究方向为比较基因组学。核酸学报2004;32:W273-W9。
- 85。
Krzywinski Mi,谢莱茵耶,Birol I,Connors J,Gascoyne R,Horsman D,Jones SJ,Marra Ma。电讯:对比较基因组学的信息审美。Genome Res。2009; 19:1639-45。
- 86。
关键词:大麦,EST, ssr, ssr标记大麦芽L.)。Al Appl Genet。2003; 106:411-22。
- 87。
Lavin M,Pennington RT,Klitgaard BB,Sprent Ji,De Lima HC,Gasson Pe。达伯利亚德豆类(Fabaceae):羽毛单胞外植物的划界。我是J机器人。2001; 88:503-33。
- 88。
毕国强,毛彦祥,邢庆科,曹明。HomBlocks:基于局部共线块搜索的细胞器系统基因组学多序列构建方法。基因组学.2018; 110:18-22。
- 89.
Darling Ac,Mau B,Blattner Fr,Perna nt。紫红色:重排的保守基因组序列的多次对准。Genome Res。2004; 14:1394-403。
- 90。
Castresana J.从多次对准中选择保守块,用于其在系统发育分析中的使用。mol Biol Evol。2000; 17:540-52。
- 91.
Nguyen L-T, Schmidt HA, von Haeseler A, Minh BQ。IQ-TREE:一种快速有效的估计最大似然系统发育的随机算法。生物科学进展。2014;32:268-74。
- 92.
Ronquist F, Huelsenbeck JP。贝叶斯先生3:混合模型下的贝叶斯系统发育推断。生物信息学.2003; 19:1572-4。
- 93.
KH, Li W-H,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸替代率差异很大。美国国家科学研究院1987;84:9054-8。
- 94.
基于序列的系统发育分析。系统医学杂志。2000;49:369 - 81。
- 95.
Müller K. SeqState:系统发育DNA数据集的引物设计和序列统计。: Bioinform。2005;4:65-9。
- 96。
Drummond AJ,Rambaut A.野兽:采样树木的贝叶斯进化分析。BMC EVOL BIOL。2007; 7:214。
- 97。
Saslis-Lagoudakis C,Chase MW,Robinson DN,Russell SJ,Klitgaard BB。核核糖体和塑性DNA序列数据推断出近培养基(Leguminosae:Dalbergieae)的系统学,发散时间和生物地理。我是J机器人。2008; 95:1270-86。
确认
作者谨此感谢荣华堂和梁琼博士的教授,他从广西农业科学院国家外地Genebank南宁分行,以及中国农业科学院石油作物研究所的Huifang Jiang教授帮助我们的野生花生材料。我们还要感谢来自中国孙中山大学的青岛农业科学院博士史博士,中国孙中山大学的吉祥博士为本文的早期版本的良好建议。
资金
山东省自然科学基金项目(no . ZR2017BC082);山东省泰山学者计划项目(no . ts201712080);山东省农业产业技术研究体系(no . SDAIT-04-02);山东省良种繁育工程项目(2017LZGC003, 2017LZGC033)。袁莉感谢Stiftelsen Olle ENGKVIST Byggmästare(2017_185-606)的资金支持。资助方不参与研究的设计、数据的收集、分析和解释以及手稿的撰写。
作者信息
从属关系
贡献
JW和SS构思和设计了这项研究;JW,Cy1(Caixia Yan),QS和CS进行了实验;JW,YL,Cy2(丘陵元),XZ和CL分析了数据;JW和YL准备的人物和/或表格;JW和YL写了稿件;JW,YL和SS批判性地修改了手稿。所有作者阅读并认可的终稿。
相应的作者
道德声明
伦理批准和同意参与
的集合一个.掌声在收集许可的需要之前。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
王军,李勇,李超。等等。野生花生叶绿体的十二个完整基因组:巨大的遗传资源和更好的理解arachis.发展史。BMC植物BIOL.19,504(2019)。https://doi.org/10.1186/s12870-019-2121-3
收到了:
接受:
发表:
关键词
- arachis.
- 花生
- 叶绿体基因组
- 种系发生
- snd
- SSRS.
- 基因组类型
- 遗传变异