摘要
背景
Calligonum(Polygonaceae)通过北方非洲到中亚分发,通常在干旱,沙漠地区发现。以前的研究表明,标准DNA条形码未能歧视Calligonum物种。在本研究中,32种载体的完全塑性基因组(塑料)21Calligonum物种测序不仅产生了属的第一个完整的质体序列Calligonum但还有1)评估综合塑性序列在组内辨别物种的能力,以及2)筛选成本效益的DNA条形码的塑料序列,可用于将来研究中的研究中解析小组内的分类关系。
结果
整个塑料Calligonum物种具有典型的四部结构。的大小Calligonum质体约有161千碱基对(kbp),编码113个基因,包括79个蛋白质编码基因、30个tRNA基因和4个rRNA基因。基于ML系统进化树分析,完整质体的物种识别率(78%)高于标准DNA条形码组合(rbcl. + 马特克 + nrITS, 56%). Five newly screened gene regions (ndhF那TRNS-G.那trnC-petN那ndhF-rpl32那rpl32-trnl.)具有较高的物种分辨率,其中ndhF和的环境-G能够区分最高比例Calligonum物种(56%)。
结论
质体全基因组是该属最有效的条形码Calligonum,而其他基因区域则显示出了巨大的潜力,可作为种识别的分类特异性条形码Calligonum.
背景
CalligonumL.(Kelligoneae,Polygonaceae)是在亚洲,北非和东南部分布的杂草灌木,但中亚是该属的物种多样化中心。许多Calligonum物种是沙漠植被中的主要种类,在那里它们通常减少(或缺席)叶子,幼枝是光合作用的主要器官[1].由于营养器官的极度简化,物种鉴定该属中的四个部分主要基于水果(Achene)形态;CALLIPHYSA.Borszcz。,Calligonum那翼状球菌Borszcz。,和美杜莎SOSK。等亚历山大。通常的特征通常是具有膜质或囊状物的果实,分别在边缘处具有窄翼或刷毛[2那3.那4.那5.].然而,果实形态也可以是高度可变的,使属内的种的界限Calligonum麻烦的[2].估计种类数目视乎处理方法而定:28-80种[6.];174种减少至28种[5.];和35种[3.].
为了帮助物种鉴定,许多分子分析已经实施,但收效甚微Calligonum. 虽然质体基因组的基因区域(马特克那rbcl.那trnH-psbA),以及核糖体内部转录间隔区(nrITS),已广泛用作一般物种鉴定的标准DNA条形码[7.那8.那9.[DNA条形码基于这些标准区域,以及其他塑性DNA序列(ATPB-RBCL.那trnL-trnF那PSBK-PSBI.)未能歧视Calligonum物种(10.那11.那12.].此外,最近的分子序列分析[13.]已经治疗了五种(C. Mongolicum.那C.Pumilum.那c .摘要那c . alashanicum,C. Zaidamense.)作为一个复杂的群体,C. Mongolicum..鉴于这些差异,该属的遗传标记更有辨识力Calligonum需要解决小组内的分类学困惑。
完全塑性序列的产生和利用可能是解决属在属中的分类作业关系的可能解决方案Calligonum.最近,已经提出了完整的塑性基因组作为“超级条形码”以克服与传统DNA条形码相关的固有局限性[14.那15.那16.].通过高拷贝基因组靶标的基因组脱脂方法可以容易地获得完全塑料的遗传序列,其中保守的基因含量,组织和结构易于组装和注释[17.].值得注意的是,除了所有标准的塑性条形码之外,竞争血浆还应为植物物种的遗传鉴定和系统发育分析提供大量信息和可变部位[18.那19.:也见e.g.,榕[20.],百合属植物[21.],人参[22.],大针[23.],水松[24.], 和Diospyros.[25.].
测序后,可以筛选完整的塑料序列,用于潜在的分类群特异性,超可变基因区域,其可能是一个比整个塑料更具成本效益,但有用的物种识别工具的潜在的分类群特异性的超可变基因区域,而不是整个血浆[15.那26.].尽管这一策略已经在一系列的类群(例如,黑死病)的许多基因区域起作用ycf1基因区域内Pterocarpus[27.),李属[28.];的TRNC.-rps16那的环境-trng,Trne.-TNM.基因区域人参[22.];和trnQ-psbK那trnR-atpA那的环境-psbZ和rpl33-rps18为了Oresitrophe[26.])迄今为止,没有报道的任何血浆的序列Calligonum此外,针对该群体的全基因组分类特异性条形码的研究也尚未完成。
测试质体序列在解决属内分类学关系方面的能力和效率Calligonum,我们选择32份材料,代表21个分类群Calligonum,对于基因组撇去。我们解决了以下三个目标:1)为属的完全塑性序列产生Calligonum;2)评价完整的质体序列对类群内物种的识别能力;3)筛选质体序列以获得具有成本效益的条形码,用于进一步研究类群内的分类学关系。
结果
质体系分析
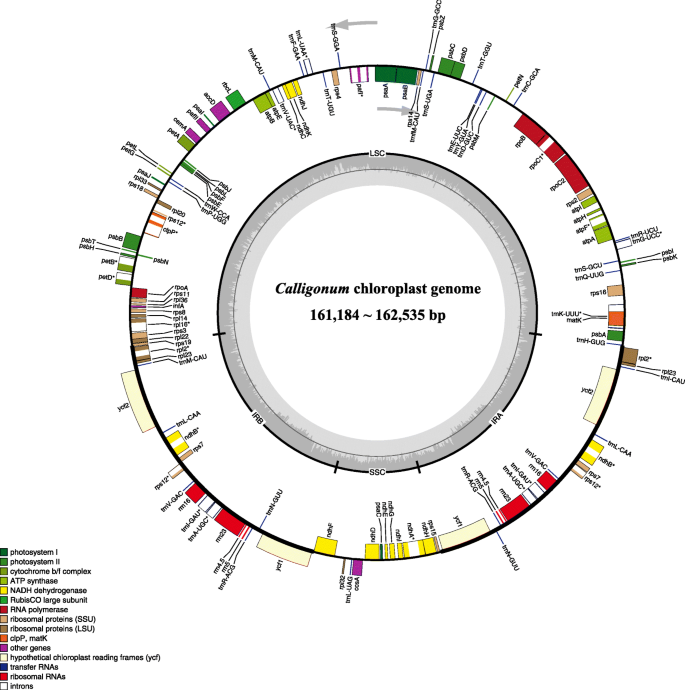
从32份材料中获得完整的质体Calligonum已提交给GenBank(表1).塑料大小从161,184 bp(C. Rubicundum.)至162,535 bp (C. jeminaIncum.).这Calligonum塑料在组织和结构中受到高度保守。它们展示了典型的Quadriparite基因组组织,包括LSC(大单拷贝)区域(86,766-88,160bp)和SSC(小单拷贝)区域(13,286-13,416bp),其被两个红外(倒重复)区域分开(30,468-30,552 bp)(表1,无花果。1).塑料中的GC含量总计为37.50%Calligonum(表1), IR区GC含量(41.30%)高于LSC区(35.60 ~ 35.70%)和SSC区(32.40 ~ 32.70%)。
所有塑料均编码113个unigenes,包括79个蛋白质编码基因,30个TRNA基因和四个具有相同基因令的RRNA基因(表1,无花果。1).没有一个区域被推断为假基因(附加文件1:表S1)。在这些基因中,五种完整的蛋白质编码基因(rpl2那rpl23那ycf2那ndhB那rps7.那ycf1);三个部分蛋白编码基因(rps19那rps12那ndhF)、7个tRNA基因(TNM.标出那TRNL.创新艺人经纪公司那trnV广汽那TRNI.高斯那TRNA.UGC.那trnRACG那Trnn.GUU)和四个rRNA基因(RRN16.那rrn23,rrn4.5那rrn5)在IR区重复(图。1).
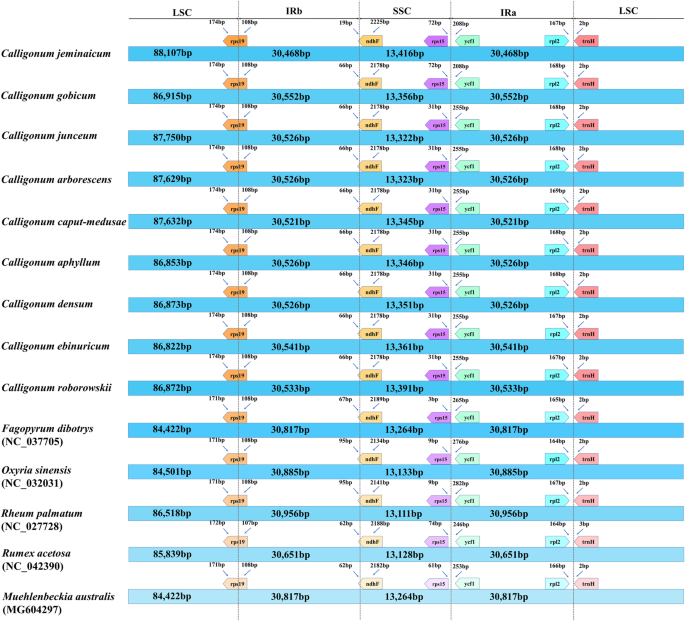
使用C. jeminaIncum.作为参考,21的同源性Calligonum物种被调查以确定序列分化的水平(附加文件2:图S1)。21号染色体的完整质体排列Calligonum物种间没有发生重排事件Calligonum物种(附加文件5.:图S3)。塑性序列在本诸于属中非常相似Calligonum.LSC / IRB和IRB / SSC边界Calligonum塑料位于编码区内rps19(位于IRB的107-108 BP)和ndhF(19–95岁) bp分别位于IRb)基因(图。2).基因间的rps15-ycf1位于SSC/IRa的边界,而基因间rpl2-trnh.古格位于IRA / LSC的边界Calligonum(图。2).基因组大小和IR膨胀/收缩有略有变化(图。2额外的文件2:图S1)。观察到的塑料长度变化是由两个插入物引起的C. jeminaIncum.(附加文件2:图S1),其位于LSC;一(段I:约800 bp)在非基因区域rps16-trnQUug.,另一段(段II,约400 bp)位于基因间区以季戊四醇四硝酸酯-psbM(详见附加文件)2:图S1和附加文件4.:表S2)。
为了估计选择压力,非同义的速率(dN)和同义词(dS)替代品,以及dN/dS(ω)测定79个蛋白质编码基因的比率(附加文件7.:图S4)。在大多数基因中,dS高于dN.这dN和dS值分别为0至0.17和0至0.63。大多数基因显示出来ω比率小于0.5,4个基因(psbI那以季戊四醇四硝酸酯那psbE,psbL)的得分最低(接近于0)ω比率(附加文件7.:图S4)。这ω的比率rpl23那ycf1,ycf2,范围从0.5到1。
整个质体用于鉴别Calligonum
在32的133,980bp矩阵中检测到总1151个多态位点(0.86%)Calligonum登记入册(表2).32之间的序列分歧Calligonum用核苷酸差异和序列距离比较质体。在种间水平,最大分化发生在种间c . taklimakanense和C. jeminaIncum.(P距离= 3.69×10- 3.,不同地点= 2867),而最近的物种为C. Colubrinum.和c . squarrosum(p-距离 = 0,核苷酸差异为1)(附加文件6.).在种内水平,p-距离范围为0.2 × 10- 4.(C无叶)到8.5×10- 4.(C. Roborowskii.),而不同地点的数目由14个(C无叶)到388(C. Roborowskii.)(附加文件)6.).
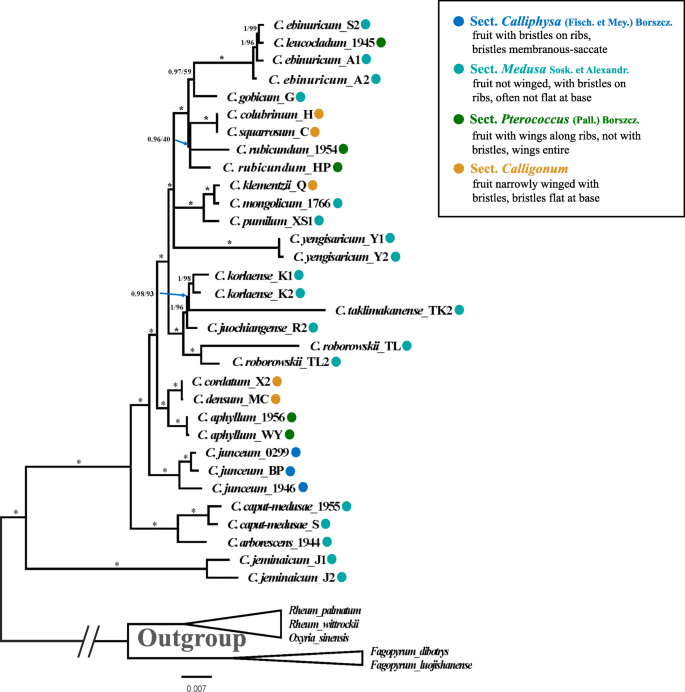
基于塑性基质,获得相同的ML和BI树(图。3.).单系属的单系Calligonum在两种情况下都得到了强有力的支持。低基因系统发育被很好地解析,大多数节点得到了强有力的支持(图。3.).只有两个节点,其中包括一个C. Colubrinum.那c . squarrosum和C. Rubicundum.(BS = 40%,PP = 0.96),另一个包括Cebinuricum公司那c . leucocladum,C. Gobicum.(BS = 59%, PP = 0.97),未得到很好的支持。通过单系调查,评估了质体的区分能力,并在那些多份取样的物种中恢复了分枝支持。9个物种中有7个(78%)具有一次以上的加入,除除以下两种外,其余都是互为单系的Cebinuricum公司和C. Rubicundum.(图。3.). 有一个加入的样本之间的关系得到了很好的支持(BS) > 93%,PP > 0.98),仅限C. Gobicum.(BS = 59%, PP = 0.97)除外。3.).
系统发育树不支持三四个部分的分裂Calligonum[5.那29.].只会。CALLIPHYSA.,包含一个种类(C. junceum.),支持良好(BS = 100%, PP = 1.00)。来自其他部分的种通常形成一个分支。例如,C无叶从教派。翼状球菌形成一个支撑良好(BS = 100%, PP = 1.00)的枝包C. Densum.和c . cordatum他们都来自教派。Calligonum.
对潜在条码的分析
由于ITS PCR失败[12.],我们从基因组浏览数据中重新组装了nrITS,包括ITS1、5.8S和ITS2区域。对32条nrITS序列进行比对和拼接,得到长度为768 bp的矩阵,其中包括22个多态位点(2.92%)2).基于BI方法的歧视性功率分析表现出大多数节点的弱分辨率。仅限九种种类,只有多种换乘Cebinuricum公司(PP = 0.86,图。5.A),有11%的成功率。ITS2(15个多态位点)比ITS1(5个多态位点)具有更强的变异能力,且具有更高的识别能力(见表1)2).
为三个标准质体条形码,完成马特克那rbcl.和trnH-psbA序列具有相同的分辨率(22%)。然而,组合马特克 + 大亚基和那样的trnH psbA公司 + 马特克 + rbcl.略微增加识别力至33%(表2额外的文件9.:图S6C-G)。将质体条形码与nrITS结合,鉴定率提高到44%(TRNH-PSBA + MATK + RBCL + nrITS) and 56% (马特克 + 大亚基+ nrITS)(表2,无花果。5.b,额外的文件9.:图S6H)。这两种组合生成的树拓扑与完整的质体数据集相似,尽管它们的分辨率低于质体基因组(表2).
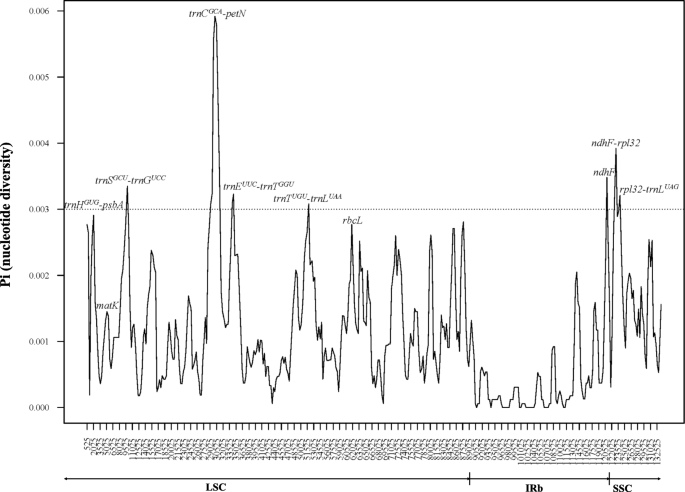
在本研究中,附加的潜在质体区域的可变性通过核苷酸多样性(Pi)来定量,Pi通过滑动窗口(窗口长度= 1000 bp,步长= 300 bp)计算。核苷酸多样性(Pi)取值范围为0 ~ 0.0059。在这些基因组中发现了7个高可变区(Pi > 0.003),其中6个是基因间区(即Pi > 0.003)。TRNS-G.那trnC-petN那trnE-T那TRNT-L.那ndhF-rpl32,rpl32-trnl.).只有一个蛋白质编码区(ndhF,无花果。4.)在内部显示出高核苷酸多样性Calligonum.这些超可变区域都位于LSC和SSC区域中(图。4.).这七个区域中的多态性位点数显着高于标准DNA条形码(rbcl.那MATK,TRNH.-psbAnrITS)(表2). 通过基于树的方法测试了它们作为潜在分类单元特异性条形码的能力。物种鉴别率(从44%到56%,表2,无花果。5.)远高于rbcl.和马特克, 除了TRNT-L.(歧视率22%,表2),trnE-T(歧视率为11%,表2).在这五个地区中,ndhF和TRNS-G.的歧视率最高(56%)(表2,无花果。5.D-e)。五个基因区域的组合(ndhF那TRNS-G.那trnC-petN那ndhF-rpl32那rpl32-trnl.)提高了物种的鉴定率至67%(表2,无花果。5.h)。
讨论
塑料特色
在这项研究中,我们生成了32个完整的Calligonum塑料。塑料Calligonum高度保守,范围为161,184至162,535 bp。与其他多糖糖糖属世属的塑性相比(例如,Fagopyrum[30.],rumex.[31.],胚胎学[32.])在本研究中产生的所有质体均表现出典型的质体结构、基因顺序和含量(图。1).此外,GC含量Calligonum(37.50%)与Fagopyrum(37.80 - -38.0%)30.],rumex acetosa.(37.20%) (31.,并等于胚胎学sinensis(37.50%)[32.].倒置重复(IR)收缩和膨胀是一种常见的进化现象,可能导致塑料长度的变化[33.].尽管如此,红外区域Calligonum在30,468 bp到30,552 bp之间,质粒的大小略有不同(图1)。2).与其他聚面团属相比具有在Enbank中的塑造数据,IR地区Calligonum大单拷贝(LSC)和小单拷贝(SSC)区域更保守,其中大部分差异是在基因间和内含子区域(附加文件3.:图S2)。发现的两个插入(段I)中的一个C. jeminaIncum.也存在于南方木兰贝那胚胎学sinensis,感冒palmatum,而它缺席Fagopyrum那rumex.和Calligonum(除了C. jeminaIncum.那C. junceum.那c . arborescens,c . caput-medusae其他文件2那3.:图S1,S2)。其他插入物(段II)缺席Calligonum(除了C. jeminaIncum.).总的来说,这些结果表明代族和内含子变异是长度变化的重要来源Calligonum,与多边形痤疮中的其他属(图。2额外的文件3.:图S2)。
基于完整质体的分类学解析
完整的质体被认为有可能增加植物物种之间的物种分辨能力[18.那19.],并已被用于在许多难以分辨的分类单元(例如,榕[20.];人参[22.];水松[24.];Diospyros.[25.])。在我们的研究中,9个物种中的7个(78%,表2) 在Calligonum正确识别出多于一个加入的物种。在七种物种中,C. Roborowskii.种内变异最大(388个变异位点),其中两个个体表现出明显的分枝长度差异(图。3.). 以往的研究表明,在不同的种群中,遗传变异很大C. Roborowskii.(阿莫瓦:91.19%,销售税: 0.818),基于cpDNA数据也有显著的系统地理结构[34.].在我们的研究中,我们还发现那些具有单一变异的物种在我们的系统进化树中有很强的支持节点。3.).种群的广泛分布范围、种群的斑块性以及重力作用下种子的短距离传播都可能是造成种子遗传分化的原因C. Roborowskii.[34.].这些结果表明,完整的质体序列是一种有效的物种鉴别工具Calligonum并且与当前的分类学处理方法一致。例如,《中国植物志》[3.],c . juochiangense被简化为C.Pumilum.然而,基于进一步的形态学分析,冯等人。[35.发现两个物种彼此完全不同,并且它们应该被视为两个独立的物种。基于我们的血液发育(图。3.),c . juochiangense形成一个分支C. Korlaense.和c . taklimakanense有了强有力的支持,与分离C.Pumilum..我们的塑料结果支持其实体作为单独的物种分类。虽然C. Colubrinum.和c . squarrosum被视为中国植物群中的不同物种[3.]它们具有非常相似的形态特征,但果实大小、颜色和瘦果上刚毛的位置不同。然而,这些性状在不同的发育阶段可能发生变化,这两个种之间没有不连续的变异。细胞质体间存在单核苷酸位点差异C. Colubrinum.和c . squarrosum,这表明它们确实是同一物种c . squarrosum巴甫洛夫(1933)应该被当作…的同义词C. Colubrinum.E. Borszcow(1860)。
虽然我们的采样仅涵盖了21种Calligonum,这些种代表…分类中的所有科Calligonum[5.](图。3.额外的文件8.:图S5)除了北非和东地中海的物种外,由于采样困难。本研究中提出的塑料数据在本集中提供了进一步描绘了本集团内的分类群。例如,属的Infrogeneric分类都不是Calligonum[5.那29.本研究支持(图。3.额外的文件8.:图S5)。此外,我们的结果与最近的分类治疗相反Calligonum,SOSK。[5.],它描绘了28种,其中许多物种已经减少到同义词:C. Gobicum.那C. Korlaense.那c . yengisaricum,C. Roborowskii.已经减少到了的同义词c . litwinowiiDrob。和C.Pumilum.和C. jeminaIncum.的C. Rubescens..虽然在我们的研究中,完整质体的多态位点比例相对较低(0.86%),但总多态位点数量(1151)相对较高,这表明完整质体可能是解决该类群内分类学问题的有效工具。特别是在有许多近缘种的属中(即那些经历了最近物种形成的属)。
虽然Calligonum质体体具有较高的物种分辨率,约有20%的物种无法被成功识别。Calligonum已知物种在交感器官中杂交[13.那34.那36.]但特别有可能似乎有间隙杂交可能导致缺乏决议Cebinuricum公司和C. Rubicundum..例如,三个Cebinuricum公司accessions成立了一个疏水板c . leucocladum(BS = 100%,PP = 1.00),Cebinuricum公司单独形成一个单位的疏水板,在NRITS系统中具有强载体。已经提出杂交或迟发作为患有患者遗传核基因和母体遗传素血浆基因之间的系统发生模式的原因[37.那38.],因此提供了一种合理的原因为什么c . leucocladum与其共享质体序列Cebinuricum公司.相似地,C. Rubicundum.accessions成了一个单一的疏水板C. Colubrinum.和c . squarrosum(bs = 40%,pp = 0.96),其是已知的混合起源[5.].两者都是这样的事实Cebinuricum公司和C. Rubicundum.在吐鲁番隐生植物植物园的栽培植物中取样,不仅强调了近缘物种在迁地植物收集中发生渐渗的可能性[39.,但也作为一个警告,在某些情况下,利用这些收集来测试物种解决可能是一个问题。
筛选潜在的DNA条形码的整个塑料
筛选完整的塑料序列时Calligonum为了找到合适的条形码区域来鉴定属中的物种,我们首先评估了一套标准DNA条形码的物种分辨率,这些条形码已用于评估其他分类群中的物种分辨率。在我们的研究中,平均而言,所有被筛选的标准DNA条形码的物种分辨率都很低。此外,完整的马特克那rbcl.那trnH-psbA成功地从基因组skimming数据中检索到nrITS序列。作为单一条形码,这些基因区域的物种分辨率很低,从11% (ITS)到22% (ITS)不等(rbcl.那马特克那trnh.-psbA).它们的组合稍微提高了物种分辨率,从33% (rbcl. + 马特克), 33% (rbcl. + 马特克 + trnh.-psbA)至55%(rbcl. + 马特克+)(表2).这些结果证实了先前的研究也显示出相对较低的分辨率[12.那36.],尽管我们能够对标准DNA条形码的较长片段(即完整的基因区域)进行测序和筛选。这些DNA条形码的物种识别失败率高有三个可能的原因Calligonum: 1)目前对该属的分类不准确;2)过去的杂交事件模糊了物种边界;3)最近的物种形成事件导致质体基因组合并失败[13.那34.那38.那40].虽然已确认的品种数量为Calligonum在专着之间变化[3.那5.],该属被认为在中亚西部干旱的沙漠中经历了最近和快速多样化[41.那42.,这可能导致DNA条形码无法区分Calligonum物种。
作为双亲本遗传标记,nrITS(或ITS2)通常比质体DNA条形码具有更高的物种分辨率[9.那43.].然而,nrits在高度保守中Calligonum,具有相对较少的多态性位点(22,2.92%的基因区域)。结果表明,nrITS的物种分辨率(11%)甚至低于3种质体标准条形码的总和。这一结果可能是由于这个属的年龄太小[41.那42.]频繁杂交[13.]和/或迟滞,大多数杂交事件Calligonum在第四纪以来分化的相对年轻的物种之间有记录[5.].例如,实验种间杂交的主要自交不亲和的分类群从组。美杜莎结果表明没有遗传上的生殖障碍[13.].此外,对于这三种合理的生物过程,基于使用基因组撇杀数据的种子和延伸策略,检索和组装研究中的NRITS共有序列。该对齐算法以相对高的频率检索等位基因,因此可能低估与我们的研究所相关的多态性位点的数量[44.](见附加文件10.额外的文件11.:表S3)。总的来说,这些结果表明nrITS不能识别大多数Calligonum在未来的研究中应考虑物种,并考虑这一限制。
筛选其他潜在条码区域
植物的DNA条形码,一般来说,仍然是一个挑战,并且由于标准条形码基因区域的遗传变异缺乏,它是密切相关的,同比物种份额相似的条形码[15.那45.那46.那47.那48.].例如,使用标准DNA条形码的分子分析未能区分物种茄属植物教派.Petota(野生土豆)[49.],柳树[45.],姜黄[50.], 和Euphrasia[16.],命名几个。然而,与使用通常用于宽系统发育分散体的标准DNA条形码相比,它们通常在特定的物种组中提供更多遗传信息,可以提高物种歧视率。此外,与完整的塑料测序相比,使用分类群特定的条形码区域对于对富含物种的大规模评估的大规模评估更具成本效益[15.].在这项研究中,在我们筛选的新区域中Callignoum,五(ndhF那TRNS-G.那trnC-petN那ndhF-rpl32那rpl32-trnl.)的物种分辨率在44 - 56%之间(表2),与发现的结果相当Quercus[51.],Diospyros.[25.], 和人参[22.].在这些地区,ndhF和TRNS-G.有最高的物种歧视(56%),组合(67%)(表2),作为我们研究的分类群。当考虑到与完整的质体测序相关的成本和时间时,这些基因区域很有可能作为一个完整的基因序列Calligonum明确的条形码在未来的研究中。
高通量测序技术的快速和经济有效的发展使得GenBank中可获得的完整质体数量迅速增加(截至2020年2月21日,共有4692种植物;https://www.ncbi.nlm.nih.gov/genome/organelle/).虽然完整的质体测序对许多实验室来说是一个沉重的负担,但我们对这个不断增加的数据集的贡献将使基于质体数据的分类特异性条形码更容易找到。对于GenBank中缺乏质体数据的属,我们建议以相对较低的成本对少数属进行测序,建立质体序列,然后用于筛选分类单元特异性条形码。我们推测,在未来,质体将作为“植物条码2.0”在许多相关领域得到广泛应用[19.那52.].对于那些具有快速辐射或频繁杂交的本身或物种复合物,我们还建议未来的条形码研究用靶向富集方法进行塑性筛选[19.那52.对核基因组中储存的、但相对尚未开发的丰富遗传资源进行采样。
结论
使用标准DNA条形码进行物种鉴定Calligonum是不够的。在本研究中,我们检测了整个质体、标准DNA条形码和超可变的分类单元特异性区域,以确定该属植物的种分辨率。在这些基因工具中,完整的质体大大提高了物种的分辨率Calligonum在未来的研究中,许多基因区域显示出了作为分类特异条形码的高潜力。
方法
分类种类和DNA测序
共有32个样本,代表21种Calligonum[5.那53.从中国西北部收集(表1);本研究中只有三种物种并不包括在内。相关地点/活动不需要具体权限。在21种物种中,九种物种有多个单独的取样。本研究的命名系统遵循植物植物植物胰岛素(FRPS)[54.[中国的植物群(Foc)[3.].凭证标本沉积在新疆生态学和地理研究所(XJBI)和南方植物园(IBSC)的植物管道中储存了俄罗斯省生态地理学研究所的植物管。
总基因组DNA从大约100毫克硅干燥的树枝材料中提取。分离方案采用十六烷基三甲基溴化铵(CTAB)法[55.].提取的DNA片段用于300 bp的短插入文库构建,并在北京华大基因研究所Illumina HiSeq X-Ten仪器上对−2 × 150 bp的对端(PE)序列进行测序。使用FastQC 0.11.5 (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)使用Trimmomatic 0.35编辑[56.移除适配器和低质量的底座。去除低质量reads和适配器序列后,每个样品获得~ 3.0 G bp的对端干净reads。
质体和nrDNA的组装与注释
使用Novoplacty V1.1组装清洁数据[57.的参考基因组Fagopyrum tataricum(Polygonaceae)(GenBank登录号NC_027161)。然后将清洁读数重新映射到初步基因组,使用Bowtie 2 V2.3.4.1调节完整的体积基因组序列[58.]和samtools v1.9 [59.].成品塑体基因组用教条注释[60]和geseq [61],然后使用Geneous v 11.0.2手动调整[62].通过与基因组进行比较来确定基因开始和止扰密码子f . tataricum.最后,将注释过的质体基因组提交到GenBank (Table1)和细胞细胞基因组画[63]用来说明一个圆形的基因组图。
分两步完成nrITS序列重建。首先,核糖体(nr) ITS序列g . junceum(加入基因库。以AB542774为参考,组装nrITS全序列(ITS1、5.8S、ITS2)。序列组装遵循上述相同的程序。每个组装的序列都作为后续步骤的参考序列。其次,使用Bowtie 2 v2.3.4.1将clean reads映射到新的获得的引用[58.]和samtools v1.9 [59.,从而得到一个只有映射读取的BAM文件。然后将BAM文件导入到genius V. 11.0.2中[62]用默认设置提取共识序列。每种共识序列用作最终的NRITS序列,并通过与参考序列进行比较并提交给GenBank(表1).
变异分析
为了说明21个物种之间整个质体序列的种间变异和基因组织,我们使用mVISTA软件和LAGAN模型[64].使用注释的对齐进行可视化C. jeminaIncum.作为参考,在本研究中产生。猛烈v1.1.1(遗传v 11.0.2中的插件)[65]用于比对和检测基因的重排和倒置Calligonum分类赛。滑动窗口分析(DNASP V6 [66)对质体基因组的Pi值进行了测定。利用MEGA X评估32份种质的进化差异(核苷酸差异和p-距离)[67].超可变区定义为具有相对高核苷酸多样性(PI)和高物种分辨率的区域。将步长设定为300bp,用1000bp窗口长度,提取具有pi值> 0.003(最大数量的一半以上的区域)以评估物种分辨率(参见下面描述的歧视性功率分析)。
为了检测质体基因是否受到选择压力,非同义基因的比例(dN),同义词(dS),ω(dN / dS)每种蛋白质编码基因的值Calligonum在PAML版本4.9D中使用Codeml分析塑性基因组[68]采用单比模型(模型 = 0,序列类型 = 1、NSSITE = 0). 如果dS,总结了树的所有分支,是> 0.5 (PAML FAQ,http://saf.bio.caltech.edu/saf_manuals/pamlfaqs.pdf.).
歧视性权力分析
使用基于树的方法来研究物种鉴定的塑料序列的功率和效率。通过单层评估歧视性,分支载体在具有多种加入的物种中回收。使用MAFFT版本7中实施的默认选项对齐完整的体积基因组(除去一个反相重复)和潜在的DNA条形码区域后的DNA序列69]. 根据jModeltest v2.1.10中的Akaike信息标准(AIC),确定了每个核苷酸序列的最合适的核苷酸替换模型[70];结果列于其他文件中12.:表S4。采用MrBayes 3.2.6进行贝叶斯推理(BI) [71[Markov Chain Monte Carlo Simulation算法(MCMC)为1×10.6.几代有四个渐进式加热的链条。每个矩阵被赋予其自己的最佳模型(附加文件12.:表S4)。在RAxML 8.2.10中生成了最大似然树[72] 1000复制。通过Figtree v1.4.3查看和编辑树木(http://github.com/rambaut/figtree/).在所有分析中,5种蓼科植物被选为外类群:感冒palmatum(NC_027728 / AY207370),r . wittrockii(NC_035950 / KF258686),Fagopyrum luojishanense(NC_037706),f . tataricum(NC_027161),F. Dibotrys.(NC_037705 / JN235080)。
可用性数据和材料
本研究中使用的全部质体基因组和nrITS序列均可从美国国家生物技术信息中心(NCBI)获得(见表)1).
缩写
- AIC:
-
Akaike信息标准
- BI:
-
贝叶斯推断
- BS:
-
独自创立
- CTAB:
-
十六烷基三甲基溴化铵法
- 脱氧核糖核酸:
-
DNA序列多态性
- 教条:
-
双细胞器基因组注释器
- 红外光谱:
-
反向重复
- LSC:
-
大单拷贝
- 密度:
-
马尔可夫链蒙特卡罗
- ml:
-
最大似然
- NCBI:
-
国家生物技术信息中心
- nrits:
-
核糖体内部转录间隔
- rrna:
-
核糖体RNA
- SSC:
-
小单副本
- tRNA:
-
转移RNA.
参考
- 1。
刘YX。《中华黄杨荒漠植物志》,第1卷。北京:科学出版社;1985.307 - 16页。
- 2。
毛泽东,Pan Br。属的分类和分布CalligonumL.在中国。Acta Phytotaxonomica Sinica。1986; 24(2):98-107。
- 3.
宝B. Grabovskaya-Borodina AE。CalligonumL. In: Wu CY, Raven PH,编辑。中国植物志,第五卷。北京:科学出版社(北京)和密苏里植物园出版社(圣路易斯);2003.324 - 8页。
- 4.
毛志明,杨刚,王光春。小分枝的染色体数目与解剖学研究Calligonum本属某些种的进化关系。植物分类学报。1983;21(1):44-9。
- 5。
Soskov YD,属Calligonum湖:分类,分销,进化,介绍。Novosibirsk:俄罗斯农业科学院;2011. p。359-61。
- 6.
Sanchez A, Schuster TM, Burke JM, Kron KA。蓼科蓼科分类学:一种新的部落分类。分类单元。2011;60(1):151 - 60。
- 7.
CBOL工厂工作组,Hollingsworth PM, Forrest LL, Spouge JL, Hajibabaei M, Ratnasingham S, van der Bank M, Chase MW, Cowan RS, Erickson DL等。陆地植物的DNA条形码。国家自然科学基金资助项目:国家自然科学基金资助项目。
- 8.
刘杰、严海峰、新大师SG、裴恩、葛学杰。DNA条形码在中国亚热带森林保护生物地理学中的应用。潜水员分布。2015;21(2):188–99.
- 9
中国植物BOL集团,李迪兹,高LM,李HT,王H,GE XJ,刘JQ,陈义,周SL,Chen Sl,等。大型数据集的比较分析表明,内部转录的间隔物(其)应纳入种子植物的核心条形码中。Proc Natl Acad Sci U S A. 2011; 108(49):19641-6。
- 10。
桑切斯,舒斯特·坦雅,凯瑟琳·AK。基于分子数据的蓼科大范围系统发育。植物保护学报;2009;
- 11.
太阳yx,张ml。部落的分子系统发生Atraphaxideae(蓼科)由5个cpDNA基因证明。干旱地区。2012;4(2):180-90。
- 12.
李啊,冯y,王思,刘b,lv gh。DNA条形码在辨别中的失败Calligonum物种。nord j bot。2014; 32(4):511-7。
- 13。
Shi W,Wen J,Zhao Y,Johnson G,Pan B.核核糖体的生殖生物学和变异的核核糖体和ETS序列Calligonum mongolicum复杂的(蓼科)。PhytoKeys。2017; 76:71 - 88。
- 14。
Parks M,Cronn R,Caron A.使用叶绿体基因组的大规模平行测序增加低分类水平的系统发育分辨率。BMC BIOL。2009; 7:84。
- 15。
李X,杨y,亨利rj,rossetto m,王y,陈S.植物DNA条形码:从基因到基因组。Biol Rev. 2015; 90(1):157-66。
- 16。
Wang X,Gussarova G,Ruhsam M,De Vere N,Metherell C,Hollingsworth Pm,Twyford广告。DNA条形码占分类上六种血管基因的偏倍数差异性,但缺乏物种级别分辨率。AOB植物。2018; 10(3):ply026。
- 17
Tonti-Filippini J,Nevill PG,Dixon K,小I.我们可以用1000个塑性基因组做什么?工厂J. 2017; 90(4):808-18。
- 18
Coissac E, Hollingsworth PM, Lavergne S, Taberlet P.从条形码到基因组:延伸DNA条形码的概念。摩尔生态。2016;25(7):1423 - 8。
- 19
Hollingsworth Pm,Li Dz,Van der Bank M,Twyford广告。将植物物种与DNA分开:从条形码到基因组。Philos Transact R Soc B Biol Sci。2016; 371(1702):20150338。
- 20。
Bruun-Lund S, Clement WL, Kjellberg F, Ronsted N.首次质体系统基因组研究揭示了叶绿体进化历史中潜在的细胞核不一致性榕l .(桑科)。分子系统进化。2017;109:93-104。
- 21。
杜玉萍、毕毅、杨福平、张明飞、陈旭、薛杰、张旭。植物叶绿体基因组全序列百合属植物:深入了解进化动力学和系统发育分析。Sci众议员2017;7(1):5751。
- 22。
Manzanilla V, Kool A, Nguyen Nhat L, Nong Van H, Le Thi TH, de Boer HJ。系统基因组学和条形码人参:朝着人参物种的鉴定。BMC EVOL BIOL。2018; 18(1):44。
- 23。
Krawczyk K,Nobis M,Myszczynski K,Klichowska E,Sawicki J.Plastid超级条形码作为羽毛草地歧视的工具(Poaceae:大针).SCI REP。2018; 8(1):1924。
- 24。
傅灿、吴聪、叶立杰、莫志强、刘杰、张宇、李德泽、蔡山明、高林。红豆杉中异构化质体的普遍性和质体超级条形码的有效性(水松)全世界。SCI批准。2019; 9(1):2773。
- 25。
李波,刘y,杨y,xie x,lu y,杨z,金x,dong w,suoz.三种叶绿体基因组序列分类和基因组资源Diospyros.. BMC植物生物学。2018;18(1):210.
- 26。
刘L,王Y,他P,Li P,Lee J,Soltis de,Fu C.雌氯蛋白基因组分析和卓越姐妹属的基因组资源开发Oresitrophe和mukdenia.(Saxifragaceae),使用基因组撇杀数据。BMC基因组学。2018; 19(1):235。
- 27。
焦莉、陆毅、何特、李杰、尹毅。利用三种植物的叶绿体全基因组建立高分辨率DNA条形码的策略Pterocarpus物种。足底。2019;250(1):95 - 104。
- 28.
金浩金,李义明,门j h,金j h。系统发育的分子标记源自比较质体分析李属物种。J Syst Evol。2019; 57(1):15-22。
- 29.
Rechinger KH, Schiman-Czeika H. Polygonaceae。见:《伊朗植物志》第56卷。Graz:维也纳自然历史博物馆;1968.36-46页。
- 30.
王克,丁Mq,zou cy,zhu xm,唐y,周ml,shao Jr。基于形态学的四种荞麦物种和完全叶绿体基因组序列的对比分析。SCI REP。2017; 7:6514。
- 31.
GUI L,Jiang S,Wang H,Nong D,Liu Y.酢浆草完全叶绿体基因组的表征(rumex acetosa.).线粒体DNA部分B. 2018; 3(2):902-4。
- 32.
罗X,王T,胡H,范L,王Q,胡Q。植物叶绿体全基因组的鉴定胚胎学sinensis. 保存基因资源。2016;9(1):47–50.
- 33.
朱艾,郭伟,古普塔,风扇W,割草机jp。塑体倒置的进化动力学重复:膨胀,收缩和替代率损失的影响。新植物。2016; 209(4):1747-56。
- 34。
[15]温志斌,颜玲,张洪祥,孟HH,应峰,魏绍生。中国特有种的种级系统地理史沙拐枣和它的近亲Calligonum部分美杜莎(蓼科)中国西北干旱地区。acta botanica yunnanica(云南植物研究),2016;
- 35。
冯燕,潘br,沈桂根。两种植物的修订Calligonum产于中国西北部的新疆沙漠。J干旱地区。2010;2(3):231–4.
- 36。
Abdurahman M,Sabirhazi G,Liu B,Yin L,Pan B.比较五Calligonum基于形态学和分子数据的塔里木盆地物种。Exp Clin Sci J. 2012; 11:776-82。
- 37。
香QP,Wei R,Shao Yz,Yangzy,Wang XQ,张XC。系统发育关系,可能的古代杂交,以及生物地理史冷杉属(松科)基于核、质体和线粒体基因组数据。分子系统进化。2015;82(Pt A): 1-14。
- 38。
福克拉,小曼德尔,弗洛伊德斯坦合资公司。祖先的基因流动和平行的细胞器基因组捕获导致被子植物谱系中极端的系统基因组不一致。系统生物学。2017;66(3):320–37.
- 39。
Chen J,Cannon Ch,胡H.热带植物园:在原位生态系统管理前沿。趋势植物SCI。2009; 14(11):584-9。
- 40.
Fazekas AJ,Burgess Ks,Kesanakurti Pr,Graham SW,Newmaster SG,Husbany BC,Percy DM,Hajibabaei M,Barrett Sc。来自塑性基因组的多个多层DNA条形码识别植物物种同样良好。Plos一个。2008; 3(7):E2802。
- 41.
Tavakkoli S, Osaloo SK, Maassoumi AA。的发展史Calligonum和Pteropyrum(Polygonaceae)基于核核糖体DNA和叶绿体trnL-F公司序列。中国生物医学工程学报。2010;8(1):7-15。
- 42.
Mabberley D. Mabberley的植物书:一个便携式字典的植物,Teir分类,和用途。剑桥:剑桥大学出版社;2008.p。1021。
- 43。
霍林点。提炼陆地植物的DNA条形码。美国国家科学研究院学报2011;
- 44。
关键词:高拷贝基因座,基因组内多态性,核糖体DNAAsclepias(apocynaceae)。peerj。2015; 3:E718。
- 45。
Percy DM, Argus GW, Cronk QC, Fazekas AJ, Kesanakurti PR, Burgess KS,丈夫BC, Newmaster SG, Barrett SC, Graham SW。了解柳树DNA条形码的重大失败(柳树):这是由跨特定的选择性扫描结果吗?Mol Ecol。2014; 23(19):4737-56。
- 46。
结合分子进化和DNA条形码。摩尔生态。2016;25(9):1908 - 10。
- 47。
王A,Gopurenko D,Wu H,Lepschi B.六个候选DNA条形码基因座的评估鉴定澳大利亚东部五个重要侵入性草。Plos一个。2017; 12(4):E0175338。
- 48。
Collins RA, Cruickshank RH。DNA条形码的七宗罪。Mol Ecol Resour. 2013;13(6): 969-75。
- 49。
DNA条形码在复杂的群体中经常失败:野生马铃薯就是一个例子。中国生物医学工程学报,2009;
- 50。
陈杰,赵杰,夏宁,王文杰。测试近缘物种的DNA条形码姜黄(Zingiberaceae)来自缅甸和中国。Mol Ecol Resour。2015; 15(2):337-48。
- 51。
尹k,张y,li y,du fk。不同的自然选择压力atpF常绿硬光和落叶橡木种类的基因:来自对比较分析的证据完全叶绿体基因组栎属紫外线与其他橡木物种。int j mol sci。2018; 19(4):1042。
- 52.
Alsos Ig,Coissac E,Merkel MKF,Lammers Y,Alberti A,Orvain C,Dossat C,Boyer F,Hollingsworth P,Parducci L等。促进植物条码2.0及其在环境与古代DNA研究中的应用:第8届国际条码的生命会议科学摘要。基因组。2019; 62(6):350-1。
- 53.
Maryamgul A,Gulnur S,Liu B,Yin L,Pan B.分类为两个Calligonum从形态学和分子数据推断的物种。vectoros。2012; 25(2):232-6。
- 54.
Mao ZM,Calligonum L.在:FL。reip。流行音乐。sinicae。在。,卷。25.北京:科学出版社;1998. p。126-8。
- 55.
Doyle JJ,Doyle JL。少量新鲜叶组织的快速DNA隔离程序。Phytochem Buart。1987; 19:11-5。
- 56.
Bolger AM, Lohse M, Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014;30(15):2114 - 20。
- 57.
基于全基因组数据的细胞器基因组重新组装。核酸研究。2017;45(4):e18。
- 58。
Langmead B,Salzberg S.与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。
- 59。
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R, Subgroup GPDP。序列对齐/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
- 60.
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学,2004;20(17):3252 - 5。
- 61.
tilich M, Lehwark P, Pellizzer T, Ulbricht-Jones ES, Fischer A, Bock R, Greiner S. GeSeq -通用和准确的细胞器基因组注释。核酸图谱2017;45(W1): W6-W11。
- 62.
Ripma La,Simpson Mg,Hasenstab-Lehman K.诅咒!系统发育系统研究简化基因组撇渣方法 - 案例研究Oreocarya(紫草科)。应用植物学报,2014;2(12):1400062。
- 63.
Lohse M,Drechsel O,Bock R. Uniterellargenomedraw(OgDraw):一种用于容易产生的塑性和线粒体基因组的高质量定制图形图的工具。Curr Genet。2007年; 52(5-6):267-74。
- 64.
Mayor C, Brudno M, Schwartz JR, Poliakov A, Rubin EM, Frazer KA, Pachter LS, Dubchak I. VISTA:可视化任意长度的全球DNA序列比对。生物信息学。2000;16(11):1046 - 7。
- 65.
亲爱的AE,MAU B,Perna NT。进步:多种基因组对齐与基因增益,损失和重排。Plos一个。2010; 5(6):E11147。
- 66.
Rozas J, Ferrer-Mata A, JC S- db, Guirao-Rico S, Librado P, Ramos-Onsins SE, Sãn-GA。dnasp6:大数据集DNA序列多态性分析。生物学报。2017;34(12):3299-302。
- 67。
陈建平,李建平,李建平。基于遗传算法的分子进化遗传分析。中国生物医学工程学报,2017,33(3):457 - 464。《生物学报》2018;35(6):1547-9。
- 68。
杨卓。PAML 4:最大似然的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。
- 69。
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。
- 70.
Darriba D,Taboada GL,Doallo R,Posada D. JModeltest 2:更多型号,新启发式和并行计算。NAT方法。2012; 9(8):772。
- 71.
Ronquist F,Huelsenbeck JP。mrbayes3:混合模型下的贝叶斯系统发育推断。生物信息学。2003;19(12):1572–4.
- 72。
Stamatakis a . RAxML version 8:用于系统发育分析和大型系统发育后分析的工具。生物信息学。2014;30(9):1312 - 3。
致谢
作者感谢石伟和曹秋梅在物种鉴定方面的帮助。感谢周玉英、赵南、徐勇、肖天文、袁勋、靳路在分子实验和数据分析方面的帮助。高连明教授、闫海飞博士、姚刚博士在论文撰写过程中提供建议。
资金
本研究由国家自然科学基金项目(31770227)、国家基础资源调查项目(2017FY100201)和中国科学院大型科研设施项目(2017-LSFGBOWS-01)资助。资助机构为研究项目提供了资金,但在本研究的设计、收集、数据分析和手稿的撰写中没有起到任何作用。这些都是作者的责任。
作者信息
隶属关系
贡献
XG和YF构思并设计了这项研究。XG、YF和TL采集叶片材料。FS和TL进行了实验并对数据进行了分析。托尔起草了手稿。KB、XG和YF修改了手稿。所有的作者最后都同意了这篇论文。所有作者都阅读并批准了最后的手稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
叶绿体基因组中的基因列表21Calligonum*内含子(S)基因。
附加文件2:图S1。
序列恒等式为21装配Calligonum叶绿体。基因组,C. jeminaIncum.作为参考(左)。特殊插入(或删除)测试结果(右)。段一世:约800 业务伙伴,部门2:大约400 bp。
附加文件3:图S2。
蓼科6个属质体基因组的序列鉴定C. jeminaIncum.作为参考(左)。红框代表两个特殊的插入(或删除)段一世(约800) bp)和2(约400 bp)。
附加文件4:表S2。
特殊插入试验用引物和样品。
附加文件5:图S3。
基因组重新排列事件21组装Calligonum物种。
附加文件6。
Excel电子表格的核苷酸取代数和序列距离在32个完整的CP基因组(表1)中。上三角形显示核苷酸取代的数量,下三角形表示完整CP基因组中的序列距离。
附加文件7:图S4。
这dN/dS(ω)来自蛋白质编码基因的值Calligonum质体基因组。
附加文件8:图S5。
三个部分Calligonum基于伊朗的植物群(Rechinger&Schiman-cazeika,1986)。颜色代表不同的部分。分支上方的数字表示后验概率(PP,左)和ML bootstrap值(BS,右).分支*具有PP = 1和BS = 100%。
附加文件9:图S6。
贝叶斯树从不同的核心条形码推断出来Calligonum.分支上方的数字表示后验概率(一种: its1 + 5.8s;B.:5.8s + ITS2;C:马特克;D.:RBCL.E.:马特克 + rbcl.;F:trnH-psbA;G:马特克 + rbcl. + trnH-psbA;H:马特克 + rbcl. + trnH-psbA + ITS;一世:trnE-T;j:TRNT-L.).
附加文件10。
nrITS序列杂合位点和个体间多态位点的Excel电子表格。*表示Genbank中的序列。△= cggagAtc。
附加文件11:表S3。
nrITS序列装配信息。
附加文件12:表S4。
为所有数据集选择的分子模型。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
宋,福,李,T,伯吉斯,ket al。完整的质体测序解决了物种之间的分类学关系CalligonumL.(Polygonaceae)在中国。BMC植物BIOL.20,261(2020)。https://doi.org/10.1186/s12870-020-02466-5
收到了:
公认:
发表:
关键字
- Calligonum
- DNA条形码
- plastid基因组
- 物种的决议