抽象的
背景
植物产生大量的次生代谢物(SMs),参与植物对生物和非生物胁迫的适应。SMs最显著的特征是其显著的种间和种内化学多样性。细胞色素P450单加氧酶(Cytochrome P450 monooxygenases, CYPs)在SMs的生物合成和化学多样性的进化中起着重要的作用。本文研究了两种CYPs的多样性和进化Jacobaea含有SMS特征组的物种即吡咯烷类生物碱(PAS)。
结果
我们从RNA-SEQ数据中检索了CYPJ. Ventgaris.和j . aquatica,分别得到221个和157个全长的CYP基因。对保守母题的分析证实了这一点JacobaeaCYP蛋白具有保守的基序,包括血红素结合特征、PERF基序、k -螺旋和i -螺旋。KEGG注释显示,被认定为SM代谢途径基因的CYPs均来自CYP71家族,但未被认定为参与生物碱途径。对6个最大CYP家族的全长CYP进行了系统发育分析Jacobaea(CYP71,CYP76,CYP706,CYP82,CYP93和CYP72),并与均芽孢杆菌的另外两个成员的CYP进行比较,向日葵和摘要以,用拟南芥.系统发育树显示CYPs具有很强的谱系特异性多样性,表明CYPs的进化速度非常快,即使在菊科中也是如此。只在亲缘关系很近的物种中J. Ventgaris.和j . aquatica,Cyps通常成对地发现,确认进化历史中的密切关系。
结论
该研究发现有378个全长CYPsJacobaea为进一步研究其功能,包括可能参与植物PA的生物合成和多样性等提供参考。
背景
植物产生各种各样的次生代谢物(SMs),它们参与植物对生物和非生物胁迫的适应[1那2那3.].目前,已分离和鉴定了200,000多个SMS,包括不同的化学类,如硫代葡萄糖苷,生物碱,Terpenes和黄酮类化合物。通常,CLADE中的物种共享类似的SMS类[2].例如,芥菜科、山柑科和Caricaceae中几乎普遍含有硫代葡萄糖苷[4.]而且苄基喹啉生物碱主要发生在罂粟科,兔子群,伯伯科伊和Menispermaceae [5.]吡咯里西啶生物碱(PAs)主要分布在菊科、紫草科、豆科和兰科[6.]. 每一类SMs都包含许多类似的分子,这些分子来自于同一骨架,通过添加一些极性和非极性取代基,在取代基上基本不同。这种结构的多样性是有据可查的PAs在中国Jacobaea奥斯泰西家族的物种。已检测到三十七种结构相关的PASJacobaea寻常的Gaertn.那Jacobaea aquatica(山)G.Gaertn。, B.Mey。& Scherb和他们的混血儿[7.].尚于,尚不完全理解次要代谢物多样性如何以及为什么它保持本质上。
为了了解植物SM多样性的起源,对植物SM生物合成途径的分子研究是很有前途的,因为植物SM多样性是受遗传控制的[8.那9.那10.那11.].涉及SM生物合成的基因通常由基因复制与连续多样化的基因复制(参与所述初级代谢的基因)4.那12.].许多涉及SM通路的基因都属于大基因家族[3.],例如细胞色素p450s [13.那14.].细胞色素P450单加氧酶(CYP)基因在任何给定的植物中都形成一个大家族,并在次级代谢中发挥重要作用[15.].许多CYPs参与各种SMs的生物合成,因为它们利用氧和NAD(P)H催化各种底物的氧化修饰。从结构上讲,目前发现的所有植物CYPs都是膜结合酶,主要通过n端疏水信号序列锚定在内质网膜上[16.那17.]. CYP蛋白具有高度保守的基序,包括血红素结合特征、PERF基序、K-螺旋和I-螺旋,这些基序对催化活性至关重要[18.].Cyps经常在短期生物合成中作为多功能催化剂募集的事实使得这些酶在种类特异性化学多样性的演变中进行了地标[19.].
来自特定物种的良好的CYP基因组对于编码酶的功能鉴定至关重要。近年来,已经进行了来自植物的Cyps的基因组/转录组鉴定,以探讨其参与代谢途径[20.那21.那22.那23.那24.].例如,Liao等人。[23.鉴定了118个全长和175个部分cyp基因红豆杉(REHDER&E.H.WILSON)REHDER转录om,目的是发现候选基因涉及含有紫杉醇的二萜类化合物的生物合成。陈等。[24.]发现了116个全长和135个部分CYP基因萨尔维亚米尔蒂希萨Bunge翻译om与萜类生物合成的候选物。
在我们的研究中,选择PAs启动导致SM多样性的结构基因的发现。到目前为止,唯一确定的PA生物合成途径特异性酶是高亚精胺合成酶,它能将亚精胺和腐胺转化为高亚精胺,这是PA生物合成途径中的第一个特异性中间体[25.].尚不讨论Homospermidine如何转化为Cental PA骨架结构SenecionineN-氧化物。千里光碱N-氧化物以特定位置和立体选择性的方式进行结构转变,导致骨骼结构重排和其氧化修饰[9.].结果表明PAS的多样化Jacobaea物种在射击中发生,而主要pa senecionineN在根中合成氧化物[26.那27.].除苦参碱外,推测PA从苦参碱中分化出来N通过特定的一步或两步反应,包括环氧化、羟基化、脱氢和/或O.乙酰化作用[9.那28.].负责这些过程的酶尚未确定。氧化反应的候选分子是CYP家族的成员。综合研究和比较不同CYPs之间的差异Jacobaea物种将有利于识别潜在的参与PA生物合成的CYP候选者。
我们已经建立了从头转录组组装J. Ventgaris.和j . aquatica建立了全面的CYP家庭信息。这两种密切相关的物种的PA对比已经得到了很好的研究[7.那29.]但是,但有限的基因组或转录组信息可用。我们首先将推定的全长CYP分为不同CYP家族,并提取了保守的主题。此外,我们研究了基于Kegg数据库的各种代谢途径中这些CYP的潜在参与。我们随后进行了最大的CYP家族的系统发育分析Jacobaea种和其他两种来自菊科的CYPs拟南芥(l)Heynh。作为一个外群来探索五种CYPs的亲缘关系和进化。
结果
转录组测序和从头组装
本研究的目的是获得CYPS的系统信息Jacobaea物种,有助于进一步探索Pa代谢中可能的功能。总共获得两组样品J. Ventgaris.(Jv1和Jv2)和j . aquatica(这里和Ja2)。去除适配器序列、二义性reads和低质量reads (Q < 30)后,对端干净reads进行进一步处理。本研究中获得的修剪后的reads已存入NCBI SRA数据库(登录号:SRR10013580-SRR10013584 under The BioProject PRJNA561604)。
对于这四组中的每一组,都有超过2000万条清除后的对端读取被用于与Trinity (Table1).Jv1、Jv2、Ja1和Ja2的组装得到的转录本数量相同,分别包含152,286、142,213、118,936、130365个转录本,平均长度分别为936、1132、1082和1062个核苷酸。为了评估组装的转录本的质量,所有reads都使用Bowtie2重新排列回装配[30.,我们发现83 - 91%的reads被映射回正确的对(表1).这表明,我们的作图率远远高于要求的70-80%,这些组件完全符合进一步挖掘cypp基因的条件。
功能注释和结构分析
四个从头组装转录组的Trinotate注释结果可以在注释报告中找到(附加文件1那2那3.那4.).总共28,192(43.8%),27,255(56.3%),23,023(48.5%),22,077(37.9%)分别用于JV1,JV2,JA1和JA2的Go Database注释。这四个预测基因函数的百分比Jacobaea集合在不同的功能类别中类似地分布。Meja处理的集合(JA2)在类别“细胞分量”中略有较低的百分比,但类别“分子功能”和“生物过程”中的百分比高于其他三个Jacobaea集(额外的文件5.:图S1)。
包括插入,缺失,单核苷酸变体(SNV)和多核苷酸变体(MNV)的诱导和结构变体是基于四个四个的互读映射来检测Jacobaea分别转录om。结果显示在其他文件中1那2那3.那4..此外,从每个组装中获得的转录本被挖掘出它们的简单序列重复(SSRs)。共鉴定出含Jv1、Jv2、Ja1和Ja2 SSR序列13979、14921、13337和12970条。在所有装配中,最丰富的重复类型是二核苷酸,其次是三核苷酸(附加文件1那2那3.那4.).
CYPS的鉴定和分类
总共221个完整长度(附加文件6.:表S1)和323个部分CYP基因J. Ventgaris.,共157个全长(附加文件7.:表S2)鉴定了247个部分CYP基因j . aquatica, 分别。所有全长CYP都被归类为David R. Nelson博士教授所分类和命名。221个全长CYPSJ. Ventgaris.分为八个部落和38个家庭(17个A型家庭,21个非A类家庭),而157个全长CYPj . aquatica共分为8个家系,35个家系(a型16个,非a型19个)(表2).大约是两者全长CYP序列的一半J. Ventgaris.(53.8%)和j . aquatica(46.4%)分配给CYP71,CYP706,CYP76,CYP72,CYP82和CYP93系列,其中CYP72只有非A型。和....相比J. Ventgaris., 为了j . aquatica检测到较少的全长CYP,这可能是由较少数量的基因型和较低的读数引起的j . aquatica样品。然而,全长CYP的比例分布不仅在每个CYP氏族(CHI-Squess = 1.6,DF = 8,NS)中相似,而且在每个CYP系列中(Chi-Square = 18.6,DF = 37,NS)两者Jacobaea物种(表2).
我们比较了检测到的全长CYP的数量J. Ventgaris.和j . aquatica还有其他三种植物,向日葵l摘要以l .,A. Thaliana.. (桌子2).菊科的四种(J. Ventgaris.544(221个全长和323个部分CYPs),j . aquatica404(全长157和部分CYP 247),h . annuus462年,l .漂白亚麻纤维卷374)含CYP基因多于A. Thaliana.(244)。这表明在菊科植物的进化和基因组复制过程中,编码代谢途径的CYP基因在功能上出现了扩展和多样化。
总体而言,五种植物中CYPs在不同族间的分布情况(表52)是可比较的(Chi-Square = 42.0,DF = 32,NS)。但是,不同CYP家族之间的分布显着不同(Chi-Square = 466.7,DF = 212,P.< 0.001)。单家族CYP家族(CYP51、CYP74、CYP97、CYP710、CYP711)的CYP数量相当一致(χ 2 = 11.2, Df = 16, NS)。显著差异是由与陆生植物进化平行的多家系(CYP71、CYP72、CYP85、CYP86)造成的[31.),并且急剧膨胀(卡方= 445.6,Df = 192,P.< 0.001)。按照纳尔逊和WERCK-REICHHART的声明[31.其中,CYP71家族(a型)在5个物种中占主导地位,其数量是5个物种中最多的。菊科中有10个CYP科缺失Jacobaea物种相比,h . annuus和l .漂白亚麻纤维卷,包括CYP703,CYP736,CYP715,CYP734,CYP87,CYP718,CYP724,CYP728,CYP729和CYP733。没有进一步的信息,难以推断缺席/存在是否是进化的后果,或者只是由于在转录组中的全长转录物的不可用Jacobaea.
CYP蛋白的表征
221个全长蛋白的长度J. Ventgaris.(附加文件6.:表S1)范围为460至601氨基酸,平均长度为509氨基酸,以及157个全长蛋白的长度j . aquatica(附加文件7.:表S2)从464到601个氨基酸变化,平均长度为511个氨基酸。提取了血红素结合区、PERF基序、K-螺旋区和I-螺旋区四个典型保守基序的序列标志(图。1). 共同序列的主题J. Ventgaris.和j . aquatica非常相似,也与其他植物物种显示出高度相似性[22.那23.那24.]对于型和非A型CYP蛋白。此外,典型基序(即血红素结合区域,PERC和I-HELIX之间的差异的差异也与其他物种的差异相似。A型CYP的血红素结合区域的共有序列是“PFGXGRRXCP”,而“XFXXGXRXCXG”是在非A型CYPS中发现的。在所有植物P450s中都是保守的F,G和C残基,其中C残留物在跨国跨国的所有P450普遍保守,并将铁坐在血红素中[22.那23.那24.].对于PERF motif, A-type CYPs显示共识“FxPERF”,而非A-type CYPs显示“FxPxRx”,两者都有一个高度保守的“F”存在于大多数CYPs中[22.那23.那24.].A型和非A型CYP的I-HELIX主题分别为“AGXDT”和“AGX [D / E] TT”。αS型Cyps的exxr基序的共识,符合非A型CYPS [22.那23.那24.].与以往的研究结果一致[22.那23.那24.]结果证实,植物CYP蛋白共享血液结合签名,PERF基序,K-HERIX和I-HELIX的保守基序,这对于催化活性至关重要,这是催化活性至关重要的[18.].
KEGG通路分析Jacobaea细胞色素氧化酶
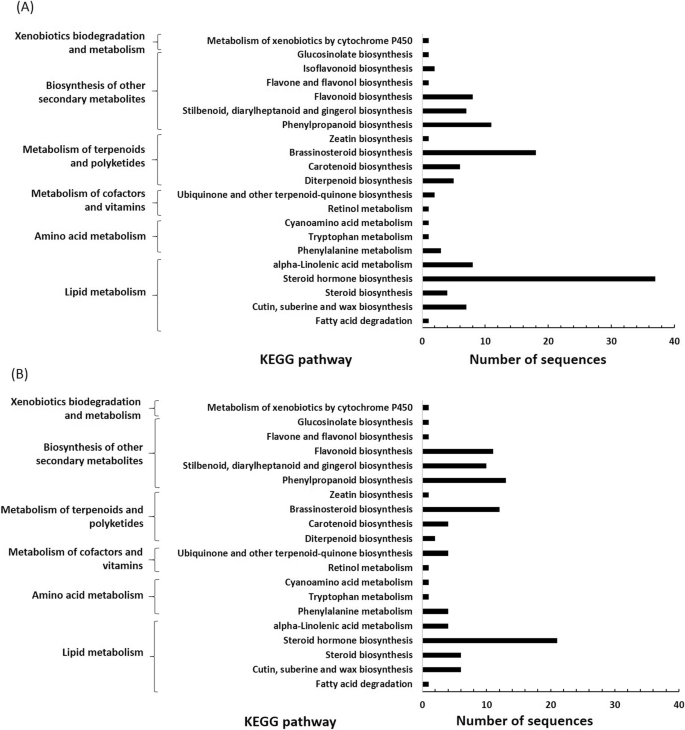
基于KEGG途径的分析是为了了解CYPs在各种生物合成途径中的潜在作用。221个(56.1%)全长CYPs中的124个J. Ventgaris.被指定为37 kegg ortholog(ko)层次结构(附加文件6.:表S1),分布在21个KEGG路径上(图。2a) 是的。对于j . aquatica在157份完整档案中,有91份(58.0%)获委任编入33份KO目录(附加档案7.:表S2),涵盖20个KEGG通路(图。2b).在“其他次生代谢产物的生物合成”类中,21个CYPs参与了苯丙素(K00487, K09754, K09755)、二苯乙烯类、二芳基庚类和姜辣素(K00487, K09754)、黄酮类(K00487, K05280, K09754)、黄酮类和黄酮醇类(K05280)的生物合成,异黄酮(K13260)和/或硫苷(K12153)Jacobaea其中一些基因被分配到一个以上的KEGG通路上。所有与SM相关的CYPs均属于CYP71家族。没有发现参与生物碱合成的基因。这并不一定意味着它们没有参与生物碱的生物合成,因为这可能是因为KEGG数据库中还没有关于PA生物合成基因的信息。
系统发育分析
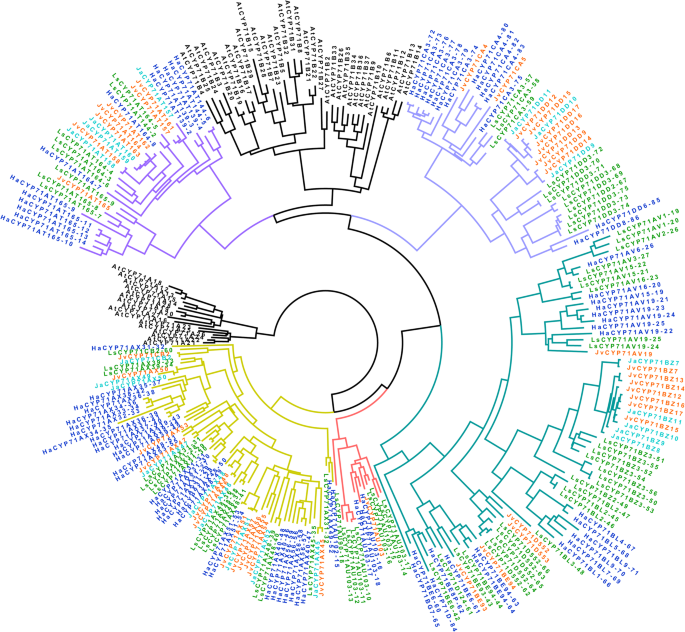
基于进化视角的比较序列分析可以改善功能预测[32.].因此,我们使用最大六个家庭的最大可能性(ml)方法进行系统发育分析雅各布会分别为CYP71、CYP76、CYP706、CYP93、CYP82和CYP72。3.; 其他文件8.那9.那10.那11.那12.:图S2-S6)。功能性分歧经常伴随基因重复,通过我们的研究证实。整体观察到Cyps的谱系特异性扩增(图。3.; 其他文件8.那9.那10.那11.那12.:图S2-S6)。在所有的系统发育树中,来自同一种的CYPs倾向于聚集在一起,形成许多谱系特异性亚科和/或分支。在大多数CYP家族中,CYP在不同物种中的分布并不均匀,表明物种分化后发生了基因复制事件。只有在Jacobaea我们观察到的物种通常是一种思考J. Ventgaris.和一个j . aquaticaCYP。以CYP71家族为例A. Thaliana.陷入了两片植物,而菊科物种的CYP分为五个不同的枝条(图。3.).值得注意的是,果实内CYP的速度非常快,导致物种特异性CYPS。特别是,CYP71AX亚家族的最基本的植物曲线,CYP71AX亚家族已经显着扩增。尽管与树木上的CYPS上的分布相比,与那些相比更加分散A. Thaliana.那Jacobaea物种,h . annuus和l .漂白亚麻纤维卷它们都有自己的谱系特异性亚种。只适用于近缘种J. Ventgaris.和j . aquatica, CYPs经常成对出现,证实了在进化史上的密切关系。对于一些CYPJ. Ventgaris.orthologs缺失j . aquatica(图。3.; 其他文件8.那9.那10.那11.那12.:图S2-S6),可能是由较少可用的全长CYP引起的j . aquatica在本研究中或或者通过进化期间的特定CYP的增益或丧失。
讨论
CYPs在促进化学多样性方面具有重要的功能,而化学多样性是植物的标志[31.].然而,作为从事初级和次生新陈代谢和快速进化的最大酶家族,Cyps对于他们在分类和命名的困难中是臭名昭着的,这阻碍了这些基因的研究。在目前的研究中,获得了具有标准命名的良好的Cyps组J. Ventgaris.和j . aquatica,这对于这些基因的功能表征和比较至关重要。共鉴定出221个和157个全长CYP基因J. Ventgaris.和j . aquatica, 分别。
基于Kegg途径的注释是针对所有全长CYPS进行的,并且没有将CYPS指定为生物合成途径。经验,来自同一家庭/亚家族的CYP通常催化类似/相关反应[31.].例如,参与苄基异喹啉生物碱多样性主要反应的CYPs包括CYP80家族(CYP80A1、CYP80B3、CYP80G2)、CYP719家族(CYP719A20、CYP719A21、CYP719A25、CYP719B1)和CYP82家族(CYP82Y1、CYP82Y2、CYP82N4、CYP82X1、CYP82X2) [33.].尽管如此,相同生物碱途径中的连续步骤也可以被来自不同家族的CYPs催化[31.].例如,一些功能特征的CYPs参与了单萜吲哚类生物碱途径Catharanthus roseus也叫(L.)G. Don来自不同的家庭:CYP71D2,CYP72A1,CYP76B6 [34.那35.那36.那37.].生物碱是特异性的SMS,其特征在于巨大的结构多样性。鉴定特定的生物合成步骤的CYP是由于CYP蛋白共享的同源性和初级结构与催化功能缺乏相关性而挑战[38.),特别是在没有报道CYPs参与PA代谢的情况下。
CYPs是植物进化的优秀报道者,特别是在植物代谢的进化和作用方面。利用系统发生树的进化方法可能有利于CYP功能的预测[31.]. CYPs的多样性对陆地植物进化过程中新代谢途径的出现有着重要的生化影响[39.].在系统发育分析中最丰富的CYP家族Jacobaea,观察到Cyps的快速发展,导致谱系特异性的膨胀。值得注意的是,Cyps并不总是遵循这些模式h . annuus表现出更接近的系统发育相关性Jacobaea物种比l .漂白亚麻纤维卷由菊科植物[40],特别是CYPS在CYP71家族中。经常,CYP71系列中的CYPh . annuus和l .漂白亚麻纤维卷与…的转换的亲缘关系Jacobaea在系统发育树上的物种(图。3.).这表明存在CYP的物种模式。基因复制被认为是进化创新的主要来源之一,导致由于内功能化或副官能化因副葡萄球菌的分歧[41.那42.]. CYP71、CYP72、CYP85等多个家族的CYP成员数量急剧增加,导致基因功能预测困难。然而,那些在系统发育树中以相同分支/亚类结尾的CYPs可能表明与特定类别化合物的代谢或不同底物上的类似反应有关[31.].
在植物进化过程中,单个基因和基因家族面临着通过复制、转座和/或缺失进行拷贝数选择的问题[43.].J. Ventgaris.和j . aquatica是两个系统发育上密切相关但生态上截然不同的物种,它们在组成和浓度上都产生具有巨大种间/种内多样性的PAs [7.].在这两个物种的进化过程中,CYP基因是如何获得或丢失的这个问题对于理解它们的化学多样性至关重要。在我们的研究中,获得了更多的全长CYPsJ. Ventgaris.更常见的是一些cyp orthologsJ. Ventgaris.(如CYP71BZ和CYP71DD亚家族)缺失j . aquatica, (图。3.; 其他文件8.那9.那10.那11.那12.:图S2-S6)。这可能是由于全基因组重复和串联重复事件[44.],尤其是后者,它对于维持大型基因家族以适应动态环境的需求而迅速扩张和收缩是必不可少的[45.].但是,不可能检测到这两个中发生的确切复制/删除事件Jacobaea只有转录组数据的物种。较少的cyp来自j . aquatica也可以解释一种用于的个体植物j . aquatica(7)比J. Ventgaris.(69),导致较少的CYP表达。根据我们的研究,不可能指定参与PA生物合成的CYP候选者。尽管如此,CYP的集合Jacobaea物种可以加快探索以下研究中的功能。只要整个基因组信息Jacobaea种属缺乏,可采用5′小种和3′小种技术获得较完整的全长CYPs。CYP候选基因的预测可以通过将基因表达模式与在产生PA对照的条件下生长的植物或在F2后代分离PA谱。
结论
在这里,我们检测到221和157个全长CypsJ. Ventgaris.和j . aquatica, 分别。五种物种的CYP比较显示了CYP的强大谱系特异性多样化,表明植物科内CYP的快速进化速度。只在密切相关J. Ventgaris.和j . aquatica, CYPs经常成对出现,证实了在进化史上的密切关系。在KEGG数据库中未发现参与生物碱合成的基因。最后,我们的研究首次全面概述了CYPsJacobaea这有利于将来探索其功能,包括可能参与PA生物合成和PA多样性。
方法
植物材料
目的是最全面的CYP基因集,多个个体两者J. Ventgaris.和j . aquatica来自分发范围的不同部分(附加文件13.:表S3)用于转录组测序,因为PA组成和浓度的含有较大的含有含有较大。种子的J. Ventgaris.从德国采集的种子是由Hortus Botanicus Leiden捐赠的j . aquatica来自英国的Kew Gardens捐赠了。所有其他种子主要由K.来自不同地点的vrieling博士收集(附加文件13.:表S3)。不需要特殊的种子收集许可。样品的鉴定是由K.Vrieling博士进行的。从两者起来J. Ventgaris.和j . aquatica物种获得两组样本(附加文件13.:表S3)。首先J. Ventgaris.一组(Jv1)由来自欧洲9个不同种群的59个个体的茎和根组成,其中2个来自组织培养种群,1个来自加拿大种群(附加文件)13.:表S3)。集合Jv1是标准化的。第二个J. Ventgaris.SET(JV2)由多个单独的克隆组成,其中一种基因型被保存在组织培养物中。对于组织培养衍生植物的组合的JV2,5个体J. Ventgaris.用茉莉酸甲酯(MeJA)处理,5只模拟处理个体作为对照。分别从MeJA处理植物和对照植物中获得了cDNA文库。在后面的组装步骤中,生成的读取被集中到矽中。这两个j . aquaticaSets(Ja1和Ja2)源自与两个群体汇集的同一七个人,其中两个人来自组织培养,其中根部包含在JA1中但不在JA2中(附加档案13.:表S3)。Set Ja1在排序前被规格化,而Set Ja2没有。
集Jv1,这里和Ja2,种子发芽湿盆栽土壤表面覆盖的塑料袋和幼苗转移到9×9×10厘米盆满50%沙质土壤(从Meijendel收集),50%的盆栽土壤(Slingerland Potgrond, Zoeterwoude,荷兰)和1.5 g / L Osmocote缓释肥料(斯科特,scott Miracle-Gro, Marysville, Ohio, USA;N: p: k = 15:9:11)。组织培养植物J. Ventgaris.和j . aquatica在Murashige和Skoog(MS)培养基上繁殖,苄基氨基嘌呤为0.44mM。为了诱导根植物,将植物转移到没有激素的MS培养基2周。生根植物以如上所述的如上所述转移到填充有土壤混合物的罐中。将所有植物保存在气候室6周(湿度70%,在20°C的湿度为16小时,在20°C时为暗8小时)。然后将植物分离成芽,根,并用水冲洗根部。将两种完全生长的每种植物完全生长的叶子和¾根缠绕在铝箔中,分别在液氮中冷冻。然后将所有样品分别地研磨成粉末,液氮含量。对于每种植物的比例为3:1的比例将芽粉与根粉末混合,然后合并来自每个个体的相同量的粉末,分别用于JV1和JA1,而仅合并粉末芽为JA2。将所有粉末材料储存在-80℃直至RNA提取。
对于set Jv2,复制J. Ventgaris.组织培养植株在MS培养基上培养2周,在气候室(湿度50%,光照16小时,20℃,暗8小时)培养。在培养基表面加入100微升溶解在10%乙醇溶液(4.5 mmol/L)中的MeJA (Sigma-Aldrich),每管扩散后终浓度为90 μmol/L,而对照组在无菌条件下加入相同体积的10%乙醇。处理后8 d收集5个生物重复的新梢,将其汇集成细粉,分别饲喂诱导组和对照组。所有粉末保存在−80℃,直到提取RNA。
RNA的分离、标准化和转录组测序
用Nucleospin®RNA植物 - 机器人-Choley-Nagel试剂盒提取总RNA,即五个样品,即JV1,Meja诱导的JV2,JV2,JA1和JA2的对照组。使用Agilent 2100 BioAnalyzer评估RNA完整性数(rin)和RNA浓度。使用[中描述的方法生成绞线特异性RNASEQ库。46.[leiden基因组技术中心的微小修改。简而言之,使用寡聚Dynabeads(Ligetech 61,002)将PolyA + mRNA分离为1μg总RNA,并在94℃下在第一链缓冲液中在第一个链缓冲液中缩短至150-200个核苷酸。随机六聚体灌注的第一链在DATP,DGTP,DCTP和DTTP存在下产生。DUTP用于标记第二条股线而不是DTTP。用Kapa HTP文库制备试剂盒进行illumina测序的后续步骤,以轻微的修饰进行。在向DSDNA片段进行分度的衔接子连接之后,将文库用酶(NEB M5505L)进行处理,以便消化第二链衍生片段。在Agilent高灵敏度芯片上量化预扩增的图书馆产率。用双相特异性的热稳定核酸酶(DSN,EVOROGOL)标准化四个(JV1和JA1),以去除旨在提高基因发现率的丰富的库分子。该方案根据JV1和JA1的Illumina指南进行。在DSN处理后,进行第二轮PCR。 All samples were quantified on an Agilent high sensitivity chip prior to pooling in equimolar amounts and sequencing on a HiSeq2500 with 2 × 126 bp paired-end reads in the Leiden Genome Technology Center.
从头组装和评估
去除适配器序列后,使用FASTQC检查原始读取的质量,通过Galaxy平台通过Trimmomatic切断质量低(阈值<30)的碱[47.].配对结束清洁读数用于组装。使用Trinity计划的De Novo装配策略[48.]k-mer大小为32,要报告的最小装配重叠长度设置为300 使用bp组装四组(Jv1、Jv2、Ja1和Ja2)。为了评估四个装配体的质量,Bowtie2将读取序列与转录组进行比对[30.].使用Trinity Suite中嵌入的脚本计算GC内容和基本统计值。
功能注释和结构分析
利用TransDecoder[]对转录组的可能编码区和开放阅读框(orf)进行预测[49.].使用Trinotate管道进行转录组功能注释和分析[50.].具体来说,我们分别使用BLASTx和BLASTp在UniProtKB/Swiss-Prot数据库中搜索转录本和TransDecoder预测的肽段,寻找它们的同源物。此外,HMMER程序鉴定了蛋白质结构域[51.]对pam数据库。用signalP 4.1服务器预测信号肽裂解位点的存在和位置[52.],并使用TMHMM server v.2.0进行蛋白质跨膜螺旋的预测[53.]. 注释输出被加载到每个转录组对应的Trinotate SQLite数据库中,并生成相应的注释报告。GO赋值是使用Trinotate中的脚本“extract\u GO\u assignments\u from\u Trinotate\u xls.pl”提取的。利用WEGO 2.0[54.绘制基因本体(GO)注释结果,并对四种注释结果进行比较Jacobaea在含有转录物同种簇的三位一体的基因水平上设置。
组合解读j . aquatica分别映射到JV1和JV2的转录组,而组合读数J. Ventgaris.分别被映射到JA1和JA2的转录组。使用默认参数,基于CLC基因组学工作台(版本8.5.1)中的读取映射来执行吲哚和结构变体的检测。除了P.-Value threshold设置为0.00001。此外,snv和mnv的检测也基于上述读取图,使用CLC Genomics Workbench中的Variant detector模块的Basic Variant Detection工具,使用默认参数进行检测。使用MISA MicroSatellite识别工具识别SSRs [55.],设置最小重复长度标准为6个重复单位的二核苷酸,5个重复单位的三、四、五、六核苷酸,两个SSR的最大距离为100个核苷酸。
在Cyp基因的硅挖掘中
为了从四个转录om,HMMER程序识别类似CYP的CONTIGS [51.]根据pam数据库的CYP参考文献(PF00067),采用隐马尔科夫模型搜索同源物[56.], e值截止为1e-5。的集合Jv1和Jv2得到了类cypp contigsJ. Ventgaris.合并,通过使用CD-HIT-EST算法(4.6.8版)除去100%相同的转录物[57.那58.].为j . aquatica,样本方法用于组合集Ja1和Ja2中的类cypcontigs。
为了获得额外的CYP样重叠群J. Ventgaris.被映射到j . aquatica在CLC基因组学工作台中使用以下参数:不匹配成本2,插入成本3,删除成本3,长度分数0.8,相似性分数0.97。映射读取的共识序列被保留并与原始CYP样板组装J. Ventgaris.在Semencher(版本5.0)中,使用最小匹配百分比为97%,而最小重叠设定为15%。于是,使用CD-HIT-EST算法检查冗余的CYP样Contigs的SeedInceer组装,其序列同一性为97%作为截止。同样,为了获得额外的CYP样板j . aquatica,CYP样挫伤J. Ventgaris.用作读映射的参考,后面的步骤相同。
通过Transdecoder预测了两种物种的所得CYP样谱的可能编码区域[49.].为了识别全长CYP基因,所有肽序列都针对NCBI喷射,并用于将CYP分类为不同的氏族。在每个氏族内部,在夹层7中进行含有至少400个氨基酸的序列的对准[59.]用于手动策划完整的编码区。根据以下两个标准鉴定推定的全长CYP基因:(1)相应的蛋白质从氨基酸'M'开始并在止挡密码子之前停止;(2)每个氏族内的对齐区域覆盖在NCBI数据库的全长CYP中的大部分长度,高度保守的血红素签名是C-末端的约50个氨基酸。
的分类和表征JacobaeaCYP基因
所有全长CYP蛋白的最终分类和命名由Dr. David R. Nelson教授按照CYP命名原则,通过与包含已发表和保密序列的植物CYP数据库中的参考文献进行比较[60.].家族、亚家族和等位变异的截断值分别为40、55和97%的氨基酸序列同源性。
CYP组件分为a型(仅包含CYP71族)和非a型(包含所有其他植物CYP族)。将a型和非a型序列分别提交给多重期望最大化Motif Elicitation (Multiple Expectation Maximization for Motif Elicitation, MEME)以预测Motif,并提交给Motif Alignment and Search Tool (MAST)以发现同源物[61.]. 主题的logo是使用WEBLOGO创建的[62.那63.]. 此外,利用ExPASy服务器上的计算等电点(PI/Mw)工具对分子量(kDa)和理论等电点(PI)进行了预测[64.],用特异性为> 0.95的TargetP1.1服务器预测亚细胞位置[65.].Kegg自动注释服务器(KAAS)[66.使用具有BLAST程序的SBH(单向最佳击中)方法的Ortholog分配和路径映射。
系统发育分析
CYP蛋白序列h . annuus[67.),l .漂白亚麻纤维卷[68.]从他们的转录组中检索,使用与上述相同的方法Jacobaea基于HMM模型的同源物种。在本研究中选择只有超过400个氨基酸的CYP,作为最可靠的注释CYP的长度A. Thaliana.在不考虑假基因的情况下,氨基酸范围从457到594不等。所有被选中的CYP基因都是根据David R. Nelson教授的最佳blast hits进行分类的。CYP蛋白序列A. Thaliana.从中下载了拟南芥细胞色素P450数据库[69.].分别使用Mega 7封装中嵌入的肌肉模块,分别用于CYP71,CYP76,CYP706,CYP82,CYP93和CYP72系列中推定的全长CYP基因进行多序列对齐。59.]使用默认设置,然后是手动编辑。通过使用Ml方法推断系统发育树。用IQ树获得树木[70那71.]透过“CIPRES科学门户”,介绍XSEDE [72.].使用标准非参数Bootstrap进行引导(BS)搜索,其中包含1000 repicate。
数据和材料的可用性
在研究过程中生成和分析的数据集包含在本文及其补充信息文件中,或可从相应作者处获得。Illumina RNA测序读取可在NCBI序列读取存档数据库中获得(http://www.ncbi.nlm.nih.gov/sra/),注册号为SRR10013580、SSR 100135581、SRR10013582、SRR10013583、SRR10013584,隶属于PRJNA561604生物工程。
缩写
- SM:
-
次生代谢物
- CYP编号:
-
细胞色素p450单氧化酶
- PA:
-
Pyrrolizidine生物碱
- 珍:
-
Jacobaea寻常的
- 是:
-
Jacobaea aquatica
- 哈:
-
向日葵
- LS:
-
摘要以
- :
-
拟南芥
- 梅贾:
-
茉莉酸甲酯
- 走:
-
基因本体论
- SNV:
-
单核苷酸变异
- MNVs:
-
Multi-nucleotide变体
- SSRS:
-
简单序列重复
- ko:
-
Kegg Ortholog.
- DSN:
-
双相特异性热稳定核酸酶
- 小姐:
-
Murashige和Skoog.
- 嗯:
-
隐藏的马尔可夫模型
- MEME:
-
主题诱惑的多重期望最大化
- 桅杆:
-
主题对齐和搜索工具
- KAAS:
-
KEGG自动标注服务器
- ML:
-
最大似然
参考文献
- 1。
Bennett RN,Wallsgrove RM。植物防御机制中的次生代谢产物。新植物。1994年; 127:617-33。
- 2。
生态分子系统发育观点的次生代谢产物的眨眼。植物化学。2003; 64:3-19。
- 3。
Kessler A,Kalske A.植物次级代谢物多样性和物种相互作用。Annu Rev Ecol Evol Syst。2018; 49:115-38。
- 4.
Moore BD, Andrew RL, Külheim C, Foley WJ。在生态学背景下解释植物次生代谢产物的种内多样性。新植醇。2014;201:733-50。
- 5。
齐格勒J,法奇尼PJ。生物碱的生物合成:代谢和运输。植物生物学杂志。2008;59:35 - 69。
- 6。
朗格尔D,奥伯D,佩尔瑟。吡咯里西啶类生物碱的生物合成与多样性。Phytochem启10:3 2011;74年。
- 7。
Cheng D, Kirk H, Mulder PPJ, Vrieling K, Klinkhamer PGL。分离杂种的茎和根中吡咯里西啶生物碱的变异Jacobaea寻常的和Jacobaea aquatica.新植醇。2011;192:1010-23。
- 8。
研究结果表明,吡咯里西啶类生物碱含量的遗传变异Cynoglossum Officinale.L. oecologia。1994年; 99:374-8。
- 9。
Hartmann T,Dierich B.冬季碘型吡咯烷类生物碱的化学多样性和变异:生物需求或巧合?Planta。1998年; 206:443-51。
- 10.
Kliebenstein DJ,Kroymann J,Brown P,Viguth A,Pedersen D,Gershenzon J,Mitchell-Olds T.拟南芥葡萄糖苷碱的自然变化的遗传控制。植物理性。2001; 126:811-25。
- 11.
Macel M,Vrieling K,Klinkhamer PGL。吡咯烷类生物碱图案的变异狗舌草jacobaea.植物化学,2004;65:865 - 73。
- 12.
OBER D.基因重复和此后的时间 - 来自植物次生新陈代谢的实例。植物BIOL。2010; 12:570-7。
- 13。
Bak S, Paquette SM, Morant M, Morant AV, Saito S, Bjarnholt N, Zagrobelny M, Jørgensen K, Osmani S, Simonsen HT等。氰苷:细胞色素P450的进化和应用的一个案例研究。Phytochem启2006;5:309-29。
- 14。
Frey M,Schullehner K,Dick R,Fiesselmann A,Gierl A.Benzoxainoid生物合成,一种植物中次级代谢途径演变的模型。植物化学。2009; 70:1645-51。
- 15.
细胞色素P450多样化对植物代谢的影响。Biol Pharm Bull. 2012; 35:824-32。
- 16。
Werck-Reichhart D, Bak S, Paquette S。细胞色素P450。拟南芥的书。2002。https://doi.org/10.1199/tab.0028..
- 17。
Bak S,Beisson F,Bishop G,Hamberger B,Höferr,paquetts s,Werck-reichhart d. cytochromes p450。拟南芥书。2011年。https://doi.org/10.1199/tab.0144.
- 18。
Paquette Sm,Jensen K,Bak S.拟南芥P450的基于网络的资源,细胞学B.5.、nadph -细胞色素P450还原酶、1家族糖基转移酶(http://www.p450.kvl.dk.). 植物化学。2009;70:1940–47.
- 19。
Hamberger B,Bak S. Plant P450S作为种类特异性化学多样性的演变的多功能司机。Phil Trans R SoC B. 2013; 368:20120426。
- 20.
Hori K, Yamada Y, Purwanto R, Minakuchi Y, Toyoda A, Hirakawa H, Sato F.利用基因组draft序列挖掘加州罂粟中参与生物碱生物合成的未知细胞色素P450基因。植物生理学杂志。2018;59:22 - 33。
- 21.
Ilc T、Arista G、Tavares R、Navrot N、Duchêne E、Velt A、Choulet F、Paux E、Fischer M、Nelson DR等。基因的注释、分类、基因组组织和表达vitis ViniferaCYPome。《公共科学图书馆•综合》。2018;13:e0199902。
- 22.
1 .齐晓,于晓,徐丹,方浩,董坤,李伟,梁超。水稻转录组中CYP450基因的鉴定与分析金银花粳稻生物原合体相关CYP450s的表达分析。peerj。2017; 5:E3781。
- 23.
廖W,赵某,张米,董师,陈y,富c,yu l。转录体组装和新型细胞色素P450s的系统鉴定红豆杉.植物科学与技术,2017;
- 24.
关键词:细胞色素P450,细胞色素P450,细胞色素P450,细胞色素P450萨尔维亚米尔蒂希萨.《公共科学图书馆•综合》。2014;9:e115149。
- 25.
Böttcher F, Adolph R-D, Hartmann T.高亚精胺合成酶(Homospermidine synthase), pyrrolizidine alkaloid生物合成的第一个途径特异性酶。植物化学,1993;32:1373 - 84。
- 26.
Hartmann T,Toppel G. senecionine n-oxide,吡咯嗪生物碱生物合成的初级产物狗舌草寻常的.植物化学。1987;26:1639-43。
- 27.
Hartmann T,Ehmke A,Eilert U,von Borstel K,Reventing C.合成,吡咯烷类生物碱N-氧化物的易分配和积累狗舌草寻常的l .足底。1989;177:98 - 107。
- 28.
PELSER PB,DE VOS H,RESURE C,BEUERLE T,Vrieling K,Hartmann T.频繁增益和吡咯烷类生物碱的进化中的进化千里光部分Jacobaea(菊科)。植物化学,2005;66:1285 - 95。
- 29.
Joosten L, Cheng D, Mulder PPJ, Vrieling K, van Veen JA, Klinkhamer PGL。吡咯里西啶类生物碱作为叔胺的基因型依赖存在Jacobaea寻常的.植物化学。2011; 72:214-22。
- 30.
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。
- 31.
Nelson D,Werck-Reichhart D.以P450为中心的植物演进视图。工厂J1111; 66:194-211。
- 32.
艾森JA,WU M.系统发育分析和基因功能预测:动作中的系统核算。你是popul biol。2002; 61:481-7。
- 33.
【关键词】苯基异喹啉;生物碱;酶;Phytochem启17:249 2018;77年。
- 34.
SchröderG,Unterbusch E,Kaltenbach M,Schmidt J,Strack D,De Luca V,SchröderJ.Int诱导的细胞色素P450依赖性酶在吲哚生物合成中:Tabersonone16-羟化酶。费用。1999年; 458:97-102。
- 35.
Irmler S, Schröder G, St-Pierre B, Crouch NP, Hotze M, Schmidt J, Strack D, Matern U, Schröder J.吲哚生物碱的生物合成Catharanthus roseus也叫:新酶活性及鉴定细胞色素P450 CYP72A1作为Secologanin合酶。工厂J. 2000; 24:797-804。
- 36。
Collu G,Unver N,Peltenburg-Looman Amg,Van der Heibden R,Verpoorte R,Memelink J. Geraniol 10-羟基酶,一种涉及Terpenoid Indole生物碱的细胞色素P450酶。费用。2001; 508:215-20。
- 37。
吉丁斯洛杉矶,利斯康比丹麦,汉密尔顿日本,儿童吉隆坡,德拉潘纳D,贝尔CR,奥康纳东南。生物碱生物合成的一个立体选择性羟基化步骤是由一个独特的细胞色素P450在细胞内进行的Catharanthus roseus也叫. 生物化学杂志。2011;286:16751–7.
- 38。
陆生植物进化过程中P450基因的多样性。植物生物学杂志。2010;61:291-315。
- 39。
Du H,Ran F,Dong HL,Wen J,Li Jn,Liang Z.基因组分析,分类,进化和陆地植物中细胞色P450 93家族的表达分析。Plos一个。2016; 11:E0165020。
- 40。
Funk Va,Anderberg AA,Baldwin BG,Bayer RJ,Bonifacino JM,Breitwieser I,Brouillet L,Carbajal R,Chan R,Coutinho Axp等。在:Funk Va,Susana A,Stuessy TF,拜耳RJ,编辑。“Compositae Metatrees:卷合物中的系统性,进化和生物地理中的下一代”。维也纳:国际植物分类协会(IAPT);2009.747-77。
- 41.
锥形gc,wolfe kh。将业余爱好转化为作业:如何重复的基因找到新功能。NAT Rev Genet。2008; 9:938-50。
- 42.
Nguyen Ba AN, Strome B, Hua JJ, Desmond J, Gagnon-Arsenault I, Weiss EL, Landry CR, Moses AM。通过翻译后调节序列的进化变化检测基因复制后的功能差异。公共科学图书馆计算生物学。2014;10:e1003977。
- 43.
Hofberger Ja,Nsibo DL,Govers F,Bouwmeester K,Schranz Me。串联和全基因组重复的复杂相互作用驱动Brassicaceae中L型凝集素受体激酶基因系列的扩增。基因组Biol Evol。2015; 7:720-34。
- 44.
于杰,特林S,王L,多萨K,张X,柯T,廖B。细胞色素P450基因超家族的进化史和功能分化拟南芥和芸苔物种揭示了全基因组和串联重复的影响。BMC基因组学。2017; 18:733。
- 45.
格雷厄姆GJ。串联基因和聚类基因。J Worl Biol。1995年; 175:71-87。
- 46.
Parkhomchuk D,Borodina T,Amstislavskiy V,Banaru M,Hallen L,Krobitsch S,Lehrach H,SoldaTov A.通过互补DNA的股线特异性测序分析。核酸RES。2009; 37:E123。
- 47.
Afgan E, Baker D, Batut B, van den Beek M, Bouvier D, Čech M等。可访问、可复制和协作生物医学分析银河平台:2016年更新。核酸杂志2016;44:W3-10。
- 48.
哈斯(Haas)等。血液病学杂志,Papanicolaou等。德诺维利用trinity平台从RNA-seq中重建转录本序列,进行参考生成和分析。Nat Protoc。2013;8:1494 - 512。
- 49.
TransDecoder。https://github.com/TransDecoder/TransDecoder/wiki.版本5.5.0。
- 50
Trinotate管道。https://github.com/Trinotate/Trinotate.github.io/wiki.
- 51
HMMER软件。http://hmmer.org.版本3.2.1b2。
- 52
尼尔森H。用signalP预测分泌蛋白。作者:Kihara D,编辑。蛋白质功能预测(分子生物学方法第1611卷)斯普林格;2017年。59–73.
- 53.
kough A, Larsson B, von Heijne G, Sonnhammer EL。用隐马尔可夫模型预测跨膜蛋白拓扑:应用于完整基因组。中国生物医学工程学报,2001;
- 54.
叶j,张y,cui h,刘j,吴y,cheng y,xu h,huang x,李思,周a等。Wego 2.0:2018年更新的用于分析和绘制Go Annotations的Web工具。核酸RES。2018; 46:W71-5。
- 55.
MISA-web:微卫星预测的网络服务器。生物信息学.2017; 33:2583-5。
- 56.
关键词:碳源,碳源,碳源,碳源pam:蛋白家族数据库。核酸杂志2014;42:D222-30http://pfam.xfam.org..访问2020年2月20日。
- 57.
Li W,Godzik A. CD-his:用于聚类和比较大套蛋白质或核苷酸序列的快速计划。生物信息学。2006; 22:1658-9。
- 58.
关键词:CD-HIT,新一代测序数据,聚类,聚类生物信息学。2012;28:3150-2。
- 59.
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870-4。
- 60。
Nelson博士,细胞色素P450主页。哼基因组学。2009;4:59 - 65https://drnelson.uthsc.edu/CytochromeP450.html. 2018年7月15日查阅。
- 61。
Bailey TL,Boden M,Buske Fa,Frith M,Grant Ce,Clementi L,Ren J,Li WW,Noble WS。MEME SUITE:主题发现和搜索的工具。核酸RES。2009; 37:W202-8。
- 62。
Crooks Ge,Hon G,Chandonia JM,Brenner SE。WebLogo:序列徽标生成器。Genome Res。2004; 14:1188-90。
- 63。
Schneider Td,Stephens RM。序列徽标:一种显示共识序列的新方法。核酸RES。1990年; 18:6097-100。
- 64。
Gasteiger E, Gattiker A, Hoogland C, Ivanyi I, Appel RD, Bairoch A. ExPASy:蛋白质组学服务器,用于深入蛋白质知识和分析。核酸杂志2003;31:3784-8。
- 65.
Emanuelsson O,Nielsen H,Brunak S,Von Heijne G.基于其N-末端氨基酸序列预测蛋白质的亚细胞定位。J Mol Biol。2000; 300:1005-16。
- 66.
Moriya Y, Itoh M, Okuda S, Yoshizawa AC, Kanehisa M. KAAS:一个自动基因组注释和路径重建服务器。核酸杂志2007;35:W182-5。
- 67.
Badouin H,Gouzy J,Grassa CJ,Murat F,Staton Se,Cottret L,Lelandais-BrièreC,欧文斯GL,CarrèreS,Mayjonade B等。向日葵基因组提供了对油代谢,开花和分摊演进的见解。自然。2017; 546:148-52。
- 68.
Reyes Chin Wo S,Wang Z,Yang X,Kozik A,Arikit S,Song C,Xia L,Froenicke L,Lavelle DO,Truco MJ,等。基因组组装体外莴苣中的邻近结扎数据和全基因组三次。NAT Communce。2017; 8:14953。
- 69.
Arabidopsis细胞色素P450数据库。http://www.p450.kvl.dk/p450.shtml..2018年6月7日通过。
- 70.
nguyen lt,schmidt ha,von haeseler a,minh bq。IQ树:一种快速有效的随机算法,用于估算最大似然性文学。mol Biol Evol。2015; 32:268-74。
- 71
Kalyaanamoorthy S, Minh BQ, Wong TKF, von Haeseler A, Jermiin LS。ModelFinder:用于精确系统发育估计的快速模型选择。Nat方法。2017;14:587-9。
- 72
Miller Ma,Pfeiffer W,Schwartz T.创建Cipres科学门户,了解大型系统发育树的推断。新奥尔良:网关计算环境研讨会(GCE);2010. p。1-8。
确认
杨安陈谢谢中国奖学金委员会(CSC)的财政支持。我们感谢Kew Gardens and Hortus Botanicus Leiden捐赠种子,Karin Van der Veen-Van Wijk,Onno Schaap,Menno Dijkstra和Issc Helpdesk为他们的技术援助,Paul Van Heusden为他的硬件和软件资源和教授提供支持。博士。David R. Nelson为Cyps的分类和命名。
资金
YC由中国教育部的中国奖学金委员会(CSC)资助。该资助者在研究和收集,分析和数据的设计方面没有作用,并在数据和写作稿件中。
作者信息
从属关系
贡献
KV,JM,YC构思和设计了实验;YC进行所有实验和数据分析,除了制备cDNA文库和转录组测序以及CYP的分类和命名,并准备了稿件;KV,JM和PK修改了手稿。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
额外的文件1。
JV1转录组的功能注释与结构分析。该文件包含基因功能注释,诱导,结构变体和简单序列重复(SSR)的信息。
额外的文件2。
Jv2转录组功能注释和结构分析。该文件包含基因功能注释,诱导,结构变体和简单序列重复(SSR)的信息。
额外的文件3。
JA1转录组的功能注释与结构分析。该文件包含基因功能注释,诱导,结构变体和简单序列重复(SSR)的信息。
附加文件4。
Ja2转录组功能注释及结构分析。该文件包含基因功能注释,诱导,结构变体和简单序列重复(SSR)的信息。
附加文件5:图S1。
Wego直方图表达JV1,JV2,JA1和JA2的转录om。X轴显示用户选择的GO条款;左y轴显示基因的百分比(特定基因的数量除以总基因数)。
附加文件6:表S1。
全长CYP列表J. Ventgaris.在本研究中确定。一种TargetP预测蛋白的细胞定位。“C”叶绿体;“S”分泌;“_”任何其他位置;“*”不得而知。B.无法使用的。C凯格矫形学。
附加文件7:表S2。
全长CYP列表j . aquatica在本研究中确定。一种TargetP预测蛋白的细胞定位。“C”叶绿体;“S”分泌;“_”任何其他位置;“*”不得而知。B.无法使用的。C凯格矫形学。
附加文件8:图S2。
利用最大似然法推断5种植物CYP76家族的系统发育树。CYP450S为不同种类的颜色编码:J. Ventgaris.(橙子),j . aquatica(浅蓝),h . annuus(深蓝色),l .漂白亚麻纤维卷(绿色),A. Thaliana.(黑色)。CYP450的名称h . annuus和l .漂白亚麻纤维卷没有命名的暂定编码。A. Thaliana.用作小组。
附加文件9:图S3。
利用最大似然法推断5种植物CYP706家族的系统发育树。CYP450S为不同种类的颜色编码:J. Ventgaris.(橙子),j . aquatica(浅蓝),h . annuus(深蓝色),l .漂白亚麻纤维卷(绿色),A. Thaliana.(黑色)。CYP450的名称h . annuus和l .漂白亚麻纤维卷没有命名的暂定编码。A. Thaliana.用作小组。
附加文件10:图S4。
从5种的CYP82系列的系统发育树推断出最大似然法。CYP450S为不同种类的颜色编码:J. Ventgaris.(橙子),j . aquatica(浅蓝),h . annuus(深蓝色),l .漂白亚麻纤维卷(绿色),A. Thaliana.(黑色)。CYP450的名称h . annuus和l .漂白亚麻纤维卷没有命名的暂定编码。A. Thaliana.用作小组。
附加文件11:图S5。
CYP93系列的系统发育树从5种,推断出最大似然法。CYP450S为不同种类的颜色编码:J. Ventgaris.(橙子),j . aquatica(浅蓝),h . annuus(深蓝色),l .漂白亚麻纤维卷(绿色),A. Thaliana.(黑色)。CYP450的名称h . annuus和l .漂白亚麻纤维卷没有命名的暂定编码。A. Thaliana.用作小组。
附加文件12:图S6。
CYP72系列的系统发育树从5种,推断出最大似然法。CYP450S为不同种类的颜色编码:J. Ventgaris.(橙子),j . aquatica(浅蓝),h . annuus(深蓝色),l .漂白亚麻纤维卷(绿色),A. Thaliana.(黑色)。CYP450的名称h . annuus和l .漂白亚麻纤维卷没有命名的暂定编码。A. Thaliana.用作小组。
附加文件13:表S3。
详细的样品套J. Ventgaris.和j . aquatica用于深度测序分析。*cDNA文库用双特异性耐热核酸酶进行归一化处理,去除大量文库分子。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Chen,Y.,Klinghamer,P.G.L.,Memelink,J。等等。细胞色素P450s的多样性和进化Jacobaea寻常的和Jacobaea aquatica.BMC植物BIOL.20.342 (2020). https://doi.org/10.1186/s12870-020-02532-y
收到了:
公认:
发表:
关键词
- 化学多样性
- 吡咯烷基生物碱生物合成
- RNA-SEQ.
- 守恒的图案
- 系统发生