- 研究文章
- 开放获取
- 发布:
在极度濒危的半水生有花植物光合关键基因退化Saniculiphyllum guangxiense(Saxifragaceae)
BMC植物生物学体积20., 文章编号:324.(2020)
摘要
背景
质基因缺失pseudogenization已被广泛记载于寄生虫病和植物mycoheterotrophic,这对光合功能轻松选择约束。更神秘的是pseudogenization和谱系中重要的光合作用基因丢失的零星报道认为是完全光合作用。在这里,我们报告的完整基因组的质Saniculiphyllum guangxiense随着叶绿体功能减少的基因组证据,一种极度濒危和系统发育孤立的植物谱系。我们还报道了另外22个质体基因组,代表了其包含的枝Saxifragales的多样性,描述了基因含量,并将变异置于更广泛的系统发育背景中。
结果
我们发现的质体基因组Saniculiphyllum有五个基因经验pseudogenizationNDH.复杂的 (ndhA,ndhB位,ndhD的,ndhF, 和ndhK),以前在水生的习惯,以及两个基因的惊人pseudogenization更多中央光合作用开花植物报道(CCSA和CEMA),与所有其他采样的Saxifragales中的塑性基因含量强的强大系统发育保守效果形成对比。这些基因参与光氧化保护,细胞色素合成和碳吸收。所有七种塑性伪原存在核常规蛋白酶,但这些也不太可能是功能性的。
结论
Saniculiphyllum似乎代表了迄今为止在任何完全光合谱系中观察到的最大程度的质体基因丢失,可能与其极端的栖息地专门化有关,然而质体基因组长度、结构和替代率都在之前报道的光合植物的变异范围内。这些结果突出了质体基因组日益受到重视的活力,否则在10亿年的绿色植物进化中高度保守,在具有高度专一生活史特征的植物中。
背景
质体基因组的结构和含量在约50万种陆地植物及其最近的绿藻亲戚中高度保守[1].光合基因的普遍丧失或pseudogenization是多样的非光合植物谱系的质体熟悉的特征,反映的光合作用基因在异养策略[谱系需要减少2].然而,越来越多的证据证明了在完全光合作用的植物中,只有在胁迫条件下才必需的“辅助”光合基因的假殖化和损失。虽然不是普遍的,但许多伪殖化和损失与高度专一的生活史特征,如水生习性有关[3.,4,5],carnivory [6,7]和肌肤营养阶段[1];这些损失的功能意义仍然是谜[8].
目前最好的质体基因缺失pseudogenization被研究了mycoheterotrophic,主要非光合植物,这是在对光合功能相对宽松的选择。值得注意的是,这些植物谱系往往遵循一个相对可预测的基因丢失序列,与光合附属基因早失,这主要受限于NDH.复杂的(2],它是参与应激条件下光氧化保护[8,9].这是最终其次,在分类单元mycoheterotrophy较长的进化历史,核心光合作用基因的损失。管家基因无关的光合作用,如质体核糖体基因,是损耗非常耐[2].在完全光合作用的生活史中,质体基因含量的研究较少,但迄今为止的例子与这个模型一致。到目前为止,基因丢失仅限于部分NDH.复杂的(3.,8,10,11,12],这似乎是在模型系统中的非必需在不存在非生物胁迫的[9,13].无论是从质体基因组的功能性拷贝的这种损失真正代表的功能丧失仍然不明朗,因为有伴随着功能转移到核基因组的细胞器基因组基因丢失或pseudogenization的很多例子[14,15].
Saniculiphyllum guangxiense吴超英和顾泰昌是一种半水生开花植物,目前仅限于中国云南省的一个极小的区域。它部分生长在小阴影瀑布的水流中,是极度濒危的,在一个约10公里的地区只有4个小种群2以及其他几个已知在过去30年内灭绝的种群[16].与在Saxifragaceae家族内部的这种血管的分离形态和生态特征一致,其系统发育亲情仍然不确定。最近的尝试将此物种放置[16,17,18表现出强烈的反对。Xiang等人[16],使用桑格测序产生的6个基因座,不能肯定地将这一谱系定位于其属于霍瑟勒分支之外,而[17]使用相同的遗传基因座,能够将这种谱系置于0.93-1.0后概率(取决于分析)作为姐妹Boykinia.邓等人[17]属性对准差异在一个单一的快速发展的遗传基因座(ITS)。基于Sanger测序数据这些研究的关系很大的不同在几个方面不同于超过300个核基因[基础上恢复18], 在哪里Saniculiphyllum被放置在适度的引导支持(80%)作为姊妹枝包含Astilbe和Boykinia.团体。
在对Saxifragales的细胞器基因组调查中,我们发现了异常的光合基因序列Saniculiphyllum。在这里,我们报道了新的质体基因组序列的系统进化关键类群,分析了Saxifragales的质体基因进化,并将其放在Saniculiphyllum质体基因组在系统发育背景下评估进化关系和质体进化率。我们想测试一下Saniculiphyllum(1)具有用于经由的附件光合基因pseudogenization,和(2)是否存在用于经由核旁系同源物质假的恢复功能证据降低光合功能的证据。
结果
大会结果
对于所有样本,我们成功地使用novo成形术组装了一个完整的圆形基因组。我们单独确认了所有的序列特征,通过映射到组装,并没有发现错误组装的证据。
基本功能基因组
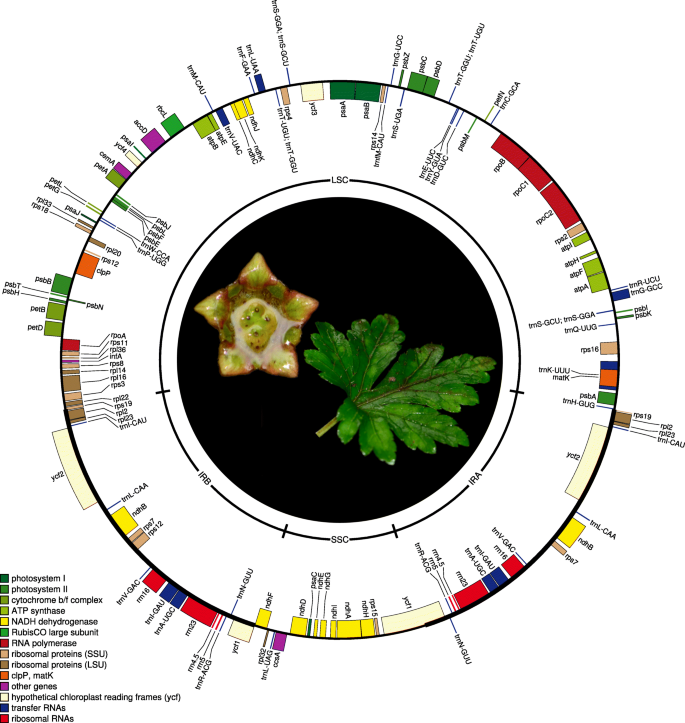
Saniculiphyllum叶绿体基因组151,704 bp长(图。1).该基因组的大规模结构是典型的陆生植物基因组,其大单拷贝区(LSC;84,479 bp)和小单拷贝区(SSC, 15,007 bp)。除推定伪基因外(见下),基因含量与预期一致[20.由73个不同的蛋白编码基因、30个tRNA基因和4个rRNA基因组成。
基因图谱Saniculiphyllum使用OrganellarGenomeDRAW [质体建19];标在圆的外侧面的基因被转录逆时针和那些圆圈内顺时针转录。中心的照片:Saniculiphyllum花和叶;图片来源:C.-L。X
伪析化的证据
我们使用过早终止密码子的存在、框架移位突变、缺失规范终止密码子和非常大的缺失作为我们的伪基因化标准(在[21])。我们使用基因模型在先前公布小花H.var。Saurensis.基因组[22以及在这里提出的Saxifragales基因组的新注释的比较。我们在5个基因中发现了伪基因化的基因组证据NDH.复杂的 (ndhA ndhB ndhD ndhF, 和ndhK),以及两个其它光合基因(cemA, ccsA),总结在表S2中,附加文件1).它们要么是由帧移突变驱动的(CCSA,NDHA,NDHD, 和ndhF),或者通过没有移码提前终止密码子(由于在一个点突变ndhB位和一个短反转ndhK).三个基因(CEMA,ndhD,和ndhF)lack much of the conserved gene sequence due to large deletions > 100 bp. Among these,CEMA没有提前终止密码子,但它具有这样否则示出了在没有虎耳草目尺寸变化的基因的非传统的预测蛋白大小(5个额外的氨基酸);而缺乏这种基因的3'末端的18%,则Saniculiphyllum拷贝具有新颖的终止密码子之前137附加碱基,其序列同源在其亲属相邻间隔区,使得它不太可能,这种序列是功能性的。此外,移导致的保守终止密码子站点丢失ndhA.三个大缺失基因的预测蛋白(CEMA,ndhD,和ndhF)的疏水性超出了其他虎耳草目的变异范围(CEMA50%疏水性氨基酸vs.其他Saxifragales的95%置信区间[50.4,52.2%];ndhD的47.8%对[62.2,63.6%]。ndhF54.9%对[55.6,58.2%])。
证据假的旁系同源
对于这三个基因的大缺失(CEMA,ndhD,和ndhF),我们用Leptarrhena对缺失的DNA进行序列分析,以寻找可能具有功能的核或线粒体旁链;否则我们就用这个分类单元的整个CDS。对于所有7个新的伪基因,我们发现了组装的叶绿体基因组之外的伪基因的证据,其中一些在序列上更保守,缺乏质体伪基因的异常特征(Supplementary Figs。S1-7)。这包括CEMA,ndhD,和ndhF没有质体拷贝中发现的大量缺失。然而,除了部分组装序列ndhF,这些旁系同源物都具有质体复制或新颖提前终止密码子或者相同提前终止密码子和也不太可能是功能性的。这些旁系同源的核可能起源的序列覆盖,这是数量级较低的基础上(黑桃计算k-mer覆盖范围~ 1-5X),比质体或线粒体的预期覆盖范围(k- m覆盖100 - 2000 x)。
除了ndhK,在那里我们找到了4个独立的血统SaniculiphyllumParalogs,基因系术(图S1-7,附加文件1)与最近七个假的旁系同源的起源是一致的。在里面CCSA基因家谱,虎耳草属多茎目柯蒂斯质直系同源物置于内Saniculiphyllum没有支持的进化枝,但其他(CEMA,ndhA,ndhB位,ndhD的,ndhF) 这Saniculiphyllum旁系同源物,为单系。
其他异常特征
在Saxifragales中,一些基因在框架内起始和终止密码子位置上有轻微的变化,但是Saniculiphyllum显示出比我们所取样的任何其他物种更多的变异,其中四个基因显示出独特的CDS终止(ATPB,CEMA,rpl20,ycf2;表S2,附加文件1),其中只有rpl20显示在其他虎耳草目物种的任何大小的变化。同时仍然光合质体基因组的典型长度范围内,所述Saniculiphyllum整体体积基因组显着小于Saxifragales物种的平均值(单尾T检验,p= 1.485平台以及)。
我们通过计算到体积基因组的总基因组DNA百分比,通过计算百分比(大多是核)DNA来间接测量塑性基因组拷贝数。较低的值表明较低的拷贝数,通过每塑性较少的体积或更少的基因组拷贝。有趣的是,这在很小的情况下Saniculiphyllum(3.4%)相比于其他虎耳草目(单尾t检验,p= 1.629 e-07);取样的虎耳草科物种的平均值为10.1%,与在[22].
Saxifragales的塑性基因组多样性
在这项研究中测序的新质体基因组没有任何结构重排的证据,强调质的基因组结构的强烈保守主义虎耳草目[20.,23](罕见的例外是小二仙草科,远亲类群这里讨论;参见[23,24])。以外Saniculiphyllum,我们在DNA序列水平上没有发现除先前记录的Saxifragales以外的伪基因的证据[20.,23]和许多其他被子植物,即ycf15以及由IR(反向重复)区域边界内创建的伪基因ycf1和ycf2(参见表S2,附加文件1).IR区域是质体基因组大小变化的主要贡献者[25],示出了类似的趋势(表1)与以前在Saxifragales中记录到的相似,后者的基因组结构非常保守[20.].但是,家庭虎耳草科朝显示减少的趋势,与一些虎耳草目最小的IR区。
系统发育分析
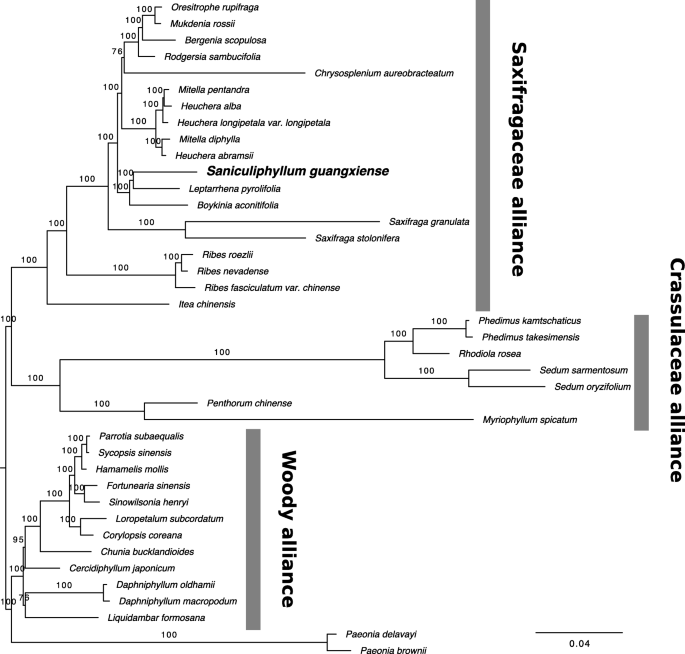
质粒比对长度为172,773 bp,其中间隙性状占比对总数的9.9%,不包括间隙性状的38,332个简约信息性状。叶绿体基因组系统发育中的主干关系与最近的系统基因组研究一致[18(图。2).虽然得到了最大的引导支持,但是Saniculiphyllum我们恢复为以前的所有努力,把这一类群,其中没有一个彼此商定不同,其中没有一个中度以上支持取得了更大的[16,17,18].我们的位置酷似以前的工作[17,18]在安置Saniculiphyllum在包括AstilbeBuch.火腿,Boykinia.英国皇家空军。,和Leptarrhena但这里所报道的新奇安置是姐妹Leptarrhena。
Saxifragales质体基因组的ML系统发育。Saniculiphyllum以粗体显示;标记的分支对应的术语[26].秤条代表每个站点的替代率。分支标签表示引导频率
替代率分析
尽管它的发散质体的特点,全基因组的替代率并没有出现升高Saniculiphyllum系统发育分支长度(图2的基础上。2),提示在核苷酸水平上,阴性选择在这一谱系中并没有明显放松。我们通过明确检测7个质体基因拷贝中是否存在松弛选择,以及假基因化的证据,进一步探索了这一假设Saniculiphyllum.我们通过实现这一目标ω(dN/dS)比率[27].我们对每个基因树采用模型比较的方法来检验Saniculiphyllum分支经历相比,它立即祖先分支选择方式的转变。我们没有发现在选择方案的变化的证据(所有p > 0.05); likewise, we estimated dN/dS < 1 for theSaniculiphyllum跨所有七个假分支(平均0.0319),具有很强的负选择是一致的。
讨论
Pseudogenization
总体而言,我们发现基因组证据在7个推定假Saniculiphyllum质体基因组。其中五个(ndhA ndhB ndhD ndhF, 和ndhK),是基因的NDH.复杂。这些基因在陆地植物和相关绿藻中高度保守[8].质基因功能的大部分损失已经与寄生虫病和植物mycoheterotrophic,这大概对光合作用基因进化功能少约束相关。Pseudogenization或基因的丢失NDH.然而,在一些完全光合的谱系中已经观察到各种各样的通常高度专门化的生活史特征:松科和麻藤属(都是裸子植物)的木本多年生植物,天竺杉科的短命多年生植物(eudiicots: rosids),高山环孢苋科(eudiicots:毛茛科(毛茛科)的肉食性植物和水生植物,兰科(单子叶植物)的各种光合作用植物,泽泻科(单子叶植物)和足藤科(蔷薇科)的水生植物(蔷薇科[1,3.,5,8,10,13,28,29,30.];)。主要功能NDH.复合物被认为是在波动的光条件下减少光氧化胁迫的。虽然这一点NDH.可有可无的温和的生长条件下出现复杂[9,实验证据从单一的击倒NDH.基因表明,一个完整完整的复合物对于有效的光合作用和植物在应激条件下的健壮生长是必不可少的[13].
比损失更不寻常NDH.功能是另外两个光合作用特定基因的明显伪化,这是我们首次报道在完全光合作用的植物中缺失的基因。的基因CEMA编码蛋白质参与碳的吸收;而对于光合作用不是必需的,光合效率下在高亮度环境降低衣藻Ehrenb。缺乏这种基因的突变体[31].的基因CCSA编码参与血红素附着到叶绿体的细胞色素c [蛋白质32].CCSA,至少在衣藻,对系统II光合作用至关重要[32].两个都CEMA和CCSA在初级光合作用的真核生物甚至蓝藻细菌中都是保守的[31,33].
证据在核旁系同源
我们成功地发现并组装了所有7个新的叶绿体伪基因Saniculiphyllum。许多这类同源序列比组装的质体基因组具有更保守的序列;除了ndhK这些出现的分歧之后已经主要起源Saniculiphyllum从这里采集的其他虎耳草科谱系中提取。根据覆盖范围,这些可能代表NUPTs(质体起源的核序列[34];)。我们的研究结果是一个缓慢的转移细胞器基因含量为核基因组[中越来越多的证据一致14,34,这一过程与这些基因组之间频繁的非同源重组修复有关[35].虽然在某些情况NUPTs与恢复功能通过编码已导入官能光合蛋白相关联15,这是不可能的Saniculiphyllum拥有这些基因的任何功能性副本,因为几乎所有的核谬误都显示出伪基因化的迹象。
其他基因组异常
我们还在Saxifragales质体基因组上游或下游的4个基因中观察到异常的CDS终止(表S2,附加文件)1);这些不会导致框架的变化,但预期的蛋白质产品的长度出乎意料。虽然不像我们观察到的假基因化模式那么引人注目,但缺乏长度保护Saniculiphyllum与近亲相比明显更大。同样地,当Saniculiphyllum质体比许多非光合作用植物长得多(在[2),由于整个质体的编码和非编码区域的大量缺失,它是Saxifragales中最短的。
尽管拥有虎耳草目中最不同质体基因组中的一个,对于在高温替代率没有证据Saniculiphyllum基于从整个质体基因组估计的系统发育分支长度(图。2).同样,我们对7个假定伪基因的dN/dS比值进行了测试,证明Saniculiphyllum在密码子水平相比,相关的谱系没有显示出显著不同的选择方案。这些结果表明,Saniculiphyllum其主要差异在于质体基因组的进化通过缺失和罕见的新终止密码子,而在核苷酸水平上没有任何可检测到的纯化选择的全局松弛。质体DNA相对于细胞核的剂量也很低Saniculiphyllum相比于亲戚,可能代表无论是在每个细胞质的减少或每质体基因组拷贝数的减少。
进化关系
这项工作也代表了第一个强大的phylogenomic布局Saniculiphyllum用于在虎耳草解释形态演变[重要组16].我们确认与该公司有密切关系Boykinia.和Leptarrhena组,同它中轴胎座,定聚伞花序,和强根茎习惯。然而,代表Astilbe组和其他几个尚未抽样;需要更密集的分类单元取样来确认这里报告的位置。
结论
虽然Saxifragales中的叶绿体基因组进化先前被理解为非常保守的[20.],还采样已经揭示在其最罕见,不寻常的谱系的一个令人惊讶的质的变化。基因丢失的相似,但不那么极端的模式已经之前为了泽泻目和川苔草科水产成员中观察到,并出现以表示多个独立的进化事件[3.,5],提示与生活史一个可能的关系。像这些谱系,Saniculiphyllum是高度专门化的部分水下阴影瀑布环境,一个稳定的栖息地可能有助于放松选择和光合辅助基因的损失。然而,这种假定的相关性是不完善的;而泽泻目包含了一些最完全适应水生的被子植物,包括水生授粉的唯一例子[3.],Myriophyllum是一种完全水生的Saxifragales谱系,显示出常规基因含量[23,许多其他水生质体基因组也是如此(例如,莲属Adans。[36],睡莲L. [37],浮萍L. [38])。
人们很容易猜测的光合作用基因含量损失的危害保护状况之间的关系Saniculiphyllum由于非生物胁迫反应基因的缺失表明其应对环境变化的能力较差。遗憾的是,我们对模式生物之外的质体基因含量的功能意义了解甚少。我们强调需要对质体基因组进化进行表征,进一步研究其与生活史性状的关系,以及比较系统基因组方法的持续前景[39揭示了这个神秘的模式。
方法
采样
我们测序总共23个plastomes增加亲缘表示。以外Saniculiphyllum,我们抽出了16个萨克菲拉格科的进一步分类群,以涵盖[中的大部分主要公认的参赛因素17],以及另外6只Saxifragales外群,以增加在木本联盟中的代表性(参见[26])。大多数材料来自园艺来源获得;野生材料与来自美国国家森林服务的许可收集。材料Saniculiphyllum不幸的是,目前还没有一个合法的保护名称,它们是从一个未受保护的自然区域收集的。抽样地点见表1.按主要分支名称列出的样本物种总数如下:Darmera团体:木槿花、河豚、三叶木槿花;Boykinia.团体:Boykinia乌头;Leptarrhena团体:Leptarrhena pyrolifolia;乔格拉拿琴团体:毛竹、白竹、毛竹、东叶毛竹、罗汉果毛竹、长叶毛竹var。longipetala, Heuchera墨西哥var。墨西哥菜,小叶小穗,小叶小穗,和臂吊带pentandra;虎耳草团体:虎耳草属多茎目;地位未定:Saniculiphyllum guangxiense.由家庭列出的小组分类群是:Cercidiphyllaceae:连香树。虎皮楠科:交让木;Grossulariaceae:糖茶nevadense和Ribes Roezlii.;天然:中华牛鼻和Sycopsis sinensis.
DNA提取和测序
从硅胶干叶材料(Saniculiphyllum),或者使用经修饰的CTAB提取协议[新鲜材料(所有其它类群)40].虽然我们的目标是塑性DNA,但我们测序总DNA,以使Paragog组装(见下文)和其他未来的关联和核基因组织相关的工作。选择分类群在跨沙克拉塔尔斯代表血统。Sequencing was performed either at RAPiD Genomics (Gainesville, Florida, U.S.A.) with 150 bp paired-end Illumina HiSeq sequencing or with 100 bp paired-end BGISEQ-500 sequencing at BGI (Shenzhen, Guangdong, P.R. China), in both cases with an insert size of approximately 300 bp (summarized in Table1).
基因组组装
我们使用NOVOPlasty诉3.2 [41.为了组装叶绿体基因组的所有测序类群。对于每个样本,我们使用跑了两个组件:和mat从质体基因组的种子的参考基因Heuchera parvifloravar。Saurensis.R.A.民俗[22].读取并没有按照开发人员的建议进行质量过滤https://github.com/ndierckx/NOVOPlasty).我们使用以下参数:K-MER = 39,预期基因组范围为120,000-200,000bp,插入尺寸300bp,插入范围= 1.8,插入范围严格= 1.3。我们还使用INSERT RANSIC SINE-TUNING来计算样本之间的插入大小范围。在整个数据集上运行Novoplatty时,我们发现它仅返回部分塑性基因组组件;使用标准UNIX工具标准化为HiseQ数据的每种样本标准化为每种样本的800万原始读数,用于BGI-SEQ样品(〜100-500x塑性覆盖率),以实现体积基因组的全长组装。大多数植物中的叶绿体基因组在相对于基因组的其余部分的小单一拷贝区域的两种可能取向中存在[42.,在使用geneir9进行序列比对之前,我们在样本间手动标准化了SSC区域的方向。
注释是用在Geneious R9进行Heuchera parviflora参考质体基因组和70%序列同一性的截止值,和质体草案注解的基因组进行比对,并手动检查注释精确度。此外,所有提前终止密码子,倒位,插入缺失移码,以及其它不寻常的特征被单独地在视觉上通过映射原始验证读回使用Geneious读取映射算法[组装质体基因组43.].我们还使用SAMtools计算了这些图谱读取的总DNA中叶绿体序列的百分比[44.].
对于7个假定的质体伪基因,我们使用aTRAM 2在线粒体和核基因组中寻找潜在的伪基因[45.,46.].ATRAM是一种用于迭代的目标组件,它在减少的读取集上实现常用的DE Novo装配模块,其具有与种子序列的序列同源性。种子序列源自我们的分类群中最接近的相对的CDS序列,Leptarrhena pyrolifolia(D.唐)序列十次迭代每组件被使用,和所使用的汇编是黑桃诉3.13.0 [47.];其他选项对应于默认值。对于这些分析,我们提取匹配来自全读Saniculiphyllum数据集(~ 180,000,000读取)。
系统发育
我们进行了最大似然系统发育分析来重新评估两者之间的关系Saniculiphyllum[16,17,18],并评估系统发育上下文质替代的速率。我们从每个基因组(即,与所述反向重复的一个拷贝)分析单拷贝质体序列,与MAFFT v对齐它们。7.388 [48.],并在RaxML v. 8.2.10中进行系统发育分析[49.]下与1000个引导重复(命令“-f a”)的一GTR-Γ模型。位点被划分为两种编码(外显子编码蛋白质的,rDNA的,和tRNA)或非编码。对于这种分析,我们采样22分进一步先前报道的质体基因组(补充表S1,附加文件1),以及从先前报道的短读取数据组件产生质体基因组虎耳草granulataL.([50.];SRA accession SRX665162),全部选择代表Saxifragales的系统发育多样性,共40个类群。我们从16个科中选取了12个,包括虎耳草科联盟的完整代表;cynomoraceae寄生科的质体已被测序,但这是故意排除的,因为它是在一个非常长的分支上[51.].虎耳草科采样盖所确认的10个主要分支的8 [17].树生根如下[18].
在aTRAM的旁系同源物搜索,我们放置在由少于200提取质体序列,用于从质体基因组对准每个基因,修整到叶绿体基因序列的程度,并除去不明确地对准区域,并除去任何序列回收到系统发育背景的序列 bp remaining after these steps. We then built individual gene trees following the RAxML methods above.
测试选择
为了测试在假定的假缓和的选择中Saniculiphyllum我们建立了两个模型,每七个基因树:一个完整的模型允许ω在所有的分支而变化,并且受约束的模型,其中Saniculiphyllum被迫拥有同样的东西ω作为它的直接祖先。我们使用似然比检验来确定约束模型是否可以被拒绝(=沿着这个系统发育分支的选择机制的转移)。由于执行了多次测试,多次比较采用Hochberg方法进行校正[52.].
数据和材料的可用性
支持这篇文章的结论的数据集可在树妖(路线,分区上的文件和树形拓扑结构;https://doi.org/10.5061/dryad.mgqnk98vt),在GenBank(表S1中的登录号;附加文件1).补充的数字是在其他文件中提供1.
缩写
- cd:
-
编码序列
- CTAB:
-
西曲溴铵
- GTR-Γ:
-
伽马一般可逆
- 其:
-
内部转录的垫片
- LSC:
-
长单拷贝[区域]
- NUPT:
-
核质体DNA
- SSC:
-
小单拷贝[区域]
- SRA:
-
顺序读取存档
参考
- 1.
Wicke S,Schneeweiss GM,dePamphilis CW,穆勒KF,在陆生植物质染色体的匡特D.进化:基因内容,基因顺序,基因功能。植物mol biol。2011; 76:273-97。
- 2.
巴雷特CF,Freudenstein JV,李江,Mayfield的琼斯DR,佩雷斯L,皮雷斯JC,等人。调查异养兰花的早期过渡分支质体基因组降解的路径和异养被子植物的影响。mol Biol Evol。2014; 31:3095-112。
- 3.
Peredo EL,王UM,莱斯DH。的质体基因组眼镜蛇flexilis:适应淹没环境是伴随着NDH复合体的水生被子植物的完全丧失。Plos一个。2013; 8:e68591。
- 4.
罗斯TG,巴雷特CF,索托Gomez的男,榄VKY,亨里克斯CL,莱斯DH,等人。质phylogenomics和泽泻目的分子进化。分支系统学。2016; 32:160-78。
- 5。
贝多亚AM,Ruhfel BR,菲尔布里克CT,MadriñánS,波夫CP,MesterházyA,等人。组织结构和金虎尾目比较分析:5种riverweeds的(川苔草科)质体基因组。前植物SCI。2019; 10:1035。
- 6。
Wicke S,SchäferhoffB,dePamphilis CW,穆勒KF。肉食锂致素叶片基因的替代速率和放松纯化选择的分类塑性血液的增加。mol Biol Evol。2014; 31:529-45。
- 7。
Gruzdev EV, Kadnikov VV, Beletsky AV, Kochieva EZ, Mardanov AV, Skryabin KG等。食肉植物的质体基因组Drosera rotundifolia和猪笼草×Ventrata.揭示进化模式类似于那些在寄生植物中观察到。诠释J分子科学。2019; 20。https://doi.org/10.3390/ijms20174107.
- 8。
Martín M, Sabater B. PlastidNDH.基因植物进化。植物生理学生物化学。2010; 48:636-45。
- 9.
Shikanai T,远藤T,桥本T,山田Y,浅田K,烟草的横田A.定向破坏ndhB位基因损害循环电子流围绕照相系统I. Proc Natl Acad Sci U S A. 1998; 95:9705-9。
- 10。
林春生,陈吉文,赵春春,肖华华,杨春娟,金晓辉,等。某些兰花叶绿体和核基因组中伴随NDH复杂相关基因的丢失。植物j . 2017; 90:994 - 1006。
- 11.
Chris Blazier J, Guisinger MM, Jansen RK。质体编码的近期丢失NDH.红念珠菌(天竺葵科)的基因。植物学报。2011;76:263-72。
- 12.
Ranade SS, García-Gil MR, Rosselló JA。非功能性质体NDH.基因片段存在于挪威云杉的核基因组(Picea amiesL.蝎):从核和细胞器基因组的计算机分析见解。摩尔创基因组学。2016; 291:935-41。
- 13.
Ruhlman TA,张W¯¯-J,陈JJW,黄Y型T,陈M-T,张J,等。NDH表达标志着植物进化重大转变,并揭示协调细胞内基因丢失。BMC植物BIOL。2015; 15:100。
- 14.
北江E,莱斯特D. NUPTs在测序真核生物和它们相对于NUMTs基因组组织。mol Biol Evol。2004; 21:1972年至1980年。
- 15.
马吉AM,阿斯皮诺尔S,赖斯DW,库萨克BP,塞蒙男,佩里AS等。局部超突变和豆类叶绿体基因组相关基因的损失。Genome Res。2010; 20:1700至1710年。
- 16。
项C-L,Gitzendanner MA,SOLTIS DE,彭H,雷L--G。神秘和极度濒危属的系统发育位置Saniculiphyllum从质体和核DNA序列的组合分析推断。分子系统学进展。2012;64:357-67。
- 17。
邓J-B,德鲁BT,Mavrodiev EV,Gitzendanner MA,SOLTIS PS,SOLTIS DE。系统发育,分化时间,被子植物家庭虎耳草科的历史生物地理学。mol phylocyet evol。2015; 83:86-98。
- 18。
Folk RA, Stubbs RL, Mort ME, Cellinese N, Allen JM, Soltis PS等。在温带辐射中,生态位和表型进化的速率滞后于多样化。acta Natl academy Sci U S A. 2019; 116:10874-82。
- 19。
洛斯男,Drechsel O,Kahlau S,博克R. OrganellarGenomeDRAW - 用于产生质体和线粒体基因组的物理图谱和可视化表达数据集的工具套件。核酸RES。2013; 41(Web服务器问题):W575-81。
- 20。
董伟,徐超,程涛,周胜景天属植物sarmentosum和叶绿体基因组进化的虎耳草目。Plos一个。2013; 8:e77965。
- 21。
Albalat R,CañestroC.基因损失的演变。NAT Rev Genet。2016; 17:379-91。
- 22。
民间RA,曼德尔JR,Freudenstein合资企业。一种用于最近辐射的含有内含子的序列标记靶向富集方法:从phylogenomic示例乔格拉拿琴(Saxifragaceae)。应用植物学报2015;3:1500039。
- 23。
董W,徐C,吴P,程T,玉Ĵ,周S等人。解决神秘的类群的系统位置:操纵虎耳草目的叶绿体基因组数据。mol phylocyet evol。2018; 126:321-30。
- 24.
廖勇,刘勇,刘旭,Lü T, Mbichi RW,万涛,等。完整的叶绿体基因组Myriophyllum spicatum揭示了一个4-KB的反转和关于小二仙草科质体进化的新见解。11.904↓。2020年https://doi.org/10.1002/ece3.6125.
- 25.
王锐-J,程C-L,昌C-C,吴C-L,苏T-M,曹格S〜M。动态及在单子叶植物的叶绿体基因组的反向重复,大单拷贝路口的演变。BMC生物学EVOL。2008; 8:36。
- 26.
健S,SOLTIS PS,Gitzendanner MA,穆尔MJ,李R,亨德利TA等。解决虎耳草目一个古老的,快速的辐射。系统中的生物学。2008; 57:38-57。
- 27。
杨Z.PAML 4:通过最大可能性进行系统发育分析。mol Biol Evol。2007; 24:1586年至1591年。
- 28。
巴雷特CF,新芬BT,肯尼迪啊。在异养兰属前所未有平行光合损失。mol Biol Evol。2019。https://doi.org/10.1093/molbev/msz111.
- 29。
翁M- l, Blazier JC, Govindu M, Jansen RK。对天竺葵科原始质体基因组的重建揭示了基因组重排、重复和核苷酸替换率之间的相关性。生物科学进展。2014;31:45 - 59。
- 30.
孙勇,邓T,张阿,穆尔MJ,兰迪斯JB,林N,等人。濒危,relictual植物的基因组草图金达尼亚联弗洛拉(CircaEsteraceae,Ranunculales)揭示了进化专业化的潜在机制和危险。生物XIV。2020年https://doi.org/10.1101/2020.01.08.898460.
- 31。
roland N, Dorne AJ, Amoroso G, Sültemeyer DF, Joyard J, Rochaix JD。质体破裂ycf10开放阅读框影响衣藻叶绿体无机碳的吸收。EMBO J. 1997; 16:6713-26。
- 32。
解Z,商家S的质体编码的CCSA血红素附着在叶绿体c型细胞色素上需要基因。美国生物化学杂志。1996;271:4632-9。
- 33。
关键词:组氨酸,色氨酸,氨基酸残基,氨基酸残基CCSA中,一种系统II多面体的细胞色素c的生物合成的蛋白质。J Biol Chem。2003; 278:2593-603。
- 34。
迪米斯JN,Ayliffe MA,黄CY,马丁W.内共生基因转移:细胞器基因组锐意真核生物染色体。NAT Rev Genet。2004; 5:123-35。
- 35。
黄春英,马毅夫,金炯敏。新转移的叶绿体DNA在烟草中的简单和复杂核位点。美国国家科学研究院2004;101:9710-5。
- 36.
吴Z,桂S,泉Z,潘L,王S,柯W,等人。的精确叶绿体基因组莲属椰子(莲科)与桑格评价,Illumina的MiSeq和PacBio RS II测序平台:洞察基础真双子叶植物的质体进化。BMC植物BIOL。2014; 14:289。
- 37.
Goremykin VV,赫希恩斯特KI,WölflS,海尔威格FH。的叶绿体基因组睡莲属阿尔巴:全基因组分析和识别的最基础的被子植物的问题。mol Biol Evol。2004; 21:1445年至1454年。
- 38.
Mardanov AV, Ravin NV, Kuznetsov BB, Samigullin TH, Antonov AS, Kolganova TV等。浮萍的完整序列(浮萍属小)叶绿体基因组:组织结构和其他被子植物的系统发育关系。J Mol Evol。2008; 66:555-64。
- 39。
Smith SD, Pennell MW, Dunn CW, Edwards SV。系统发育学是一种新的遗传学(对大多数生物多样性而言)。0。https://doi.org/10.1016/j.tree.2020.01.005.
- 40。
多伊尔JJ。少量新鲜叶组织的快速DNA隔离程序。Phytochem Buart。1987; 19:11-5。
- 41。
Dierckxsens N,Mardulyn P,Smits G. Novoplasty:来自整个基因组数据的细胞器基因组的De Novo组装。核酸RES。2017; 45:E18。
- 42。
王W,Lanfear R.长读取显示,叶绿体基因组中的两个不同版本的大多数植物存在。基因组生物学EVOL。2019; 11:3372-81。
- 43。
Matthew Kearse,Shane Sturrock和Peter Meintjes。诅咒6.0.3读取映射器。https://assets.geneious.com/documentation/geneious/GeneiousReadMapper.pdf.2019年9月18日。
- 44。
李洪波,李洪波,李洪波,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25:2078-9。
- 45。
艾伦JM,LaFrance R,民间RA,约翰逊KP,Guralnick RP。aTRAM 2.0:一种改进的,灵活的轨迹汇编程序的NGS数据。EVOL生物信息学在线。2018; 14:1176934318774546。
- 46。
黄迪,周永明,黄迪。aTRAM -自动目标限制装配方法:一种从下一代测序数据跨不同类群装配位点的快速方法。BMC生物信息学。2015;16:98。
- 47。
王志强,王志强,王志强,等。一个新的基因组组装算法及其在单细胞测序中的应用。J computer Biol. 2012; 19:455-77。
- 48.
利用MAFFT对DNA序列进行多序列比对。方法:中华医学杂志2009;537:39-64。
- 49.
Stamatakis A. Raxml-VI-HPC:最大基于似然的系统发育分析,有数千个分类群和混合模型。生物信息学。2006; 22:2688-90。
- 50。
Van der Meer S,Van Houtt JKJ,Maes Ge,Hellemmans B,Jacquemyn H.微卫星底漆营养不良草原多年生草地虎耳草granulata(Saxifragaceae)。苹果厂SCI。2014; 2:1400040。
- 51。
贝洛特S,Cusimano N,罗S,太阳G,Zarre S,GrögerA,等人。组装质和线粒体基因组,以及核基因,将寄生虫家庭Cynomoriaceae在虎耳草目。基因组生物学EVOL。2016; 8:2214-30。
- 52。
Hochberg Y.一种锐利的Bonferroni程序,用于多种意义的考验。Biometrika。1988; 75:800-2。
致谢
感谢D. Soltis和G. Wong为获得与10KP项目有关的试点短读数据提供了便利。感谢J. Nelson、J. Xiang和J. v . Freudenstein提供了DNA材料;j·基诺里(J. Ginori)协助测试早期的组装运行,已故的m·惠顿(M. Whitten)在DNA提取协议方面提供了大量建议。
资金
r.a.f由NSF db -1523667资助。资助方没有参与这项研究的设计;收集、分析或解释数据;或者决定将论文提交出版。
作者信息
隶属关系
贡献
皇家空军构想了这项研究;R.A.F.和N.S.进行了分析;b.t. C.-L。X.和R.P.G咨询分析和解释;皇家空军写了第一份手稿草稿;所有的作者都参与了最终的草稿。所有作者都已阅读并批准了本手稿的最终版本。
通讯作者
道德声明
伦理批准和同意参与
作者遵循了所有相关的机构,国家和国际指南,为这项研究收集生物材料。测序材料的收集许可由美国国家林业局颁发。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
从GenBank下载叶绿体基因组序列摘要用于系统发育分析。表S2。提前终止密码子,大/帧移插入缺失,和其它异常的基因组的概要功能独特到Saniculiphyllum。图S1。ML的系统发育基因CCSA,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S2。ML的系统发育基因CEMA,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S3。ML的系统发育基因ndhA,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S4。ML的系统发育基因ndhB位,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S5。ML的系统发育基因ndhD的,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S6。ML的系统发育基因ndhF,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。图S7。ML的系统发育基因ndhK,显示了物种的系统发育位置Saniculiphyllum旁系同源(粗体)质直系同源基因中。这Saniculiphyllum质拷贝标记***。分支标签表示自举频率;那些低于50没有绘出。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用本文
民俗,R.A.,Sewnath,N.,湘,CL。等等。在极度濒危的半水生有花植物光合关键基因退化Saniculiphyllum guangxiense(Saxifragaceae)。BMC植物BIOL.20,324(2020)。https://doi.org/10.1186/s12870-020-02533-x
已收到:
接受:
发表:
关键词
- 质体基因组
- 质体系
- 假基因
- 细胞器
- Saxifragaceae
- Saniculiphyllum