抽象的
背景
主根是萝卜的主要食用器官,决定着萝卜的产量和品质。但在萝卜中,主根增厚的具体分子机制有待进一步研究。本研究采用RNA-seq技术从3个不同根大小的高级自交系中筛选出影响萝卜主根增厚的关键基因。
结果
‘大针状体’和‘nao - yb’之间共有2606个差异表达基因(DEGs),这些差异表达基因在‘苯丙素生物合成’、‘硫代葡萄糖苷生物合成’和‘淀粉和蔗糖代谢’途径中显著丰富。同时,在‘naud - dy’和‘naud - yh’(small circular)之间共有16个差异表达的miRNAs (dm),而在‘naud - dy’中有12个差异表达。关联分析表明miR393a-bHLH77miR167c -ARF8, miR5658APL可能是影响主根类型变异的生物学现象的关键因素,并提出了主根加厚和发育的调控模式。此外,一些关键基因包括SUS1型,EXPB3,CDC5采用RT-qPCR分析。
结论
这种对转录和转录后型材的综合研究可以为综合了解根系植物作物中的根尖增厚的分子调节机制的全面了解提供了新的见解。
背景
萝卜(萝卜L.,2n = 2x = 18)是属于Brassicaceae家族的重要全球根植物作物。肉质的根茎是主要产品器官,并确定萝卜的最终产量和质量。丰富的营养物质存在于包括碳水化合物,粗纤维,维生素C和蛋白质的肉质胶卷中。目前,对根部发育的分子机制的广泛研究已在一系列植物种类如拟南芥中进行[1.),烟草(2.],玉米[3.,4.,5.,大米[6.,7.]. 然而,在萝卜等根类蔬菜作物中,直根增厚的分子机制还远未完全阐明。
在过去的几十年里,随着“组学”方法论的快速发展,RNA-seq已成为系统识别不同植物物种不同组织、器官和发育阶段差异调控基因、mirna和调控通路的一种有价值的策略罗莎中华民神[8.],大豆[9],柑橘[10],杨梅[11],茄属植物lycopersicum[12],和R萨提夫[13]. 此外,现有的萝卜基因组数据库为研究萝卜主根增厚的分子机制提供了一个有用的基因组信息平台[14,15,16].
近年来,已经进行了若干关于在萝卜中噻虫加厚期间基因表达的鉴定和解剖和复杂调节网络的几项研究。使用RNA-SEQ技术,鉴定许多miRNA和转录物在萝卜纺纱加厚期间差异表达[13,17[噻虫加厚期间,在细胞增殖组织中,碳水化合物代谢途径显着激活,特别是在细胞增殖组织中15].然而,以前的研究主要集中在一个萝卜品种上,并且涉及不同根型萝卜基因型中的萝卜根根增厚的关键基因仍然是准确的。增加证据表明,不同品种的根形和尺寸(根型)显着不同,与根系差异相关的基因被认为是培养或抑制萝卜中的Taproot增厚的候选者[15,18,19].到目前为止,没有关于鉴定不同品种中染色体发育的基因的报告,这限制了萝卜栽培品种的遗传改善和种质创新。
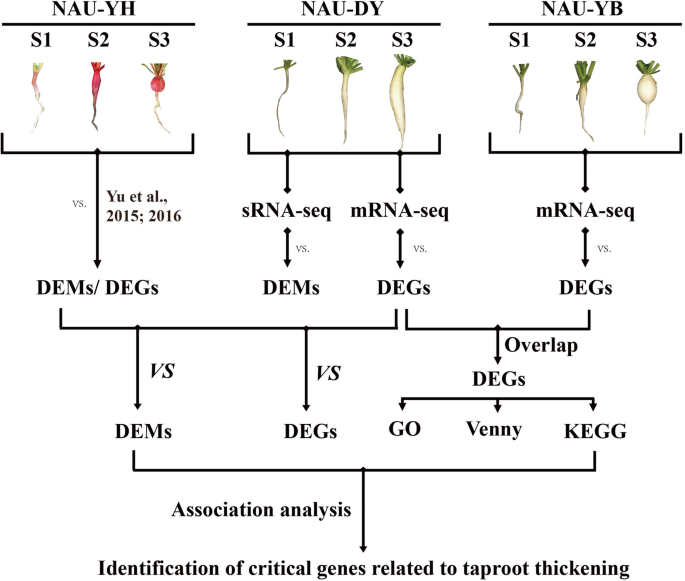
To identify differentially expressed genes (DEGs) and differentially expressed miRNAs (DEMs) involved in taproot thickening, three advanced inbred lines with different root shape and size consisting of radish ‘NAU-DY’ (large acicular), ‘NAU-YB’ (medium obovate) and ‘NAU-YH’ (small circular) were used in this study. An integrated mRNA-seq and sRNA-seq analysis was performed at three development stages of taproot thickening from three advanced inbred lines. Furthermore, RT-qPCR analysis was carried out to validate the expression patterns of several important candidate genes. The outcome of this study could reveal critical genes and miRNAs underlying the taproot thickening, and provide new insights into dynamic regulation of taproot thickening in radish.
方法
植物材料
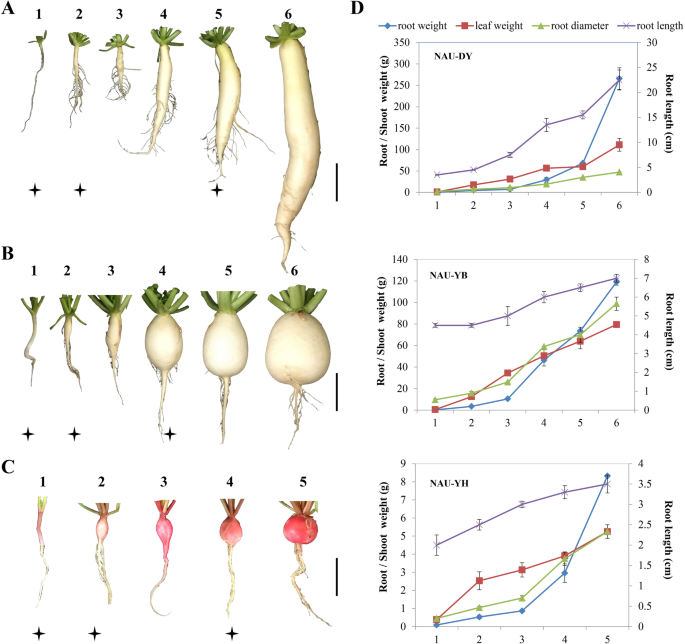
以‘nauy - dy’(大针状)、‘nauy - yb’(中倒卵形)和‘nauy - yh’(小圆形)3个高级自交系为材料,采用南京农业大学园艺学院培育的种子为材料。(无花果。1.).种子在潮湿的滤纸上在室温黑暗中萌发3天,然后种植在塑料盆中,在光照16 h(25℃)和光照8 h(18℃)的生长室内培养。各高级自交系萝卜生长发育特征如图所示。1.两叶一心、皮层分裂和直根增厚率最高的时间点分别作为皮层分裂前期(PSS)、皮层分裂期(CSS)和膨大期(ES)的选择标准。在PSS('NAU-DY',20)处收获直根样品S1、S2和S3 播种后几天,达斯NAU-YB',20达斯;'NAU-YH',10 DAS),CSS('NAU-DY',30 DAS;'NAU-YB',25 DAS;'NAU-YH',20 DAS)和ES('NAU-DY',55 DAS;'NAU-YB',45 DAS;'NAU-YH',40 DAS),分别从五个随机选择的单株植物中提取。从五个个体中收集等量的样本用于文库制备和测序。所有收获的直根样品立即在液氮中冷冻并储存在低温下− 80 °C用于RNA提取。
mRNA-seq和sRNA-seq文库的构建及序列分析
在构建mRNA文库之前,分别按照说明书使用TRIzol试剂(Invitrogen)在主根增稠的三个不同发育阶段提取总rna。详细的cDNA文库构建和测序的实验步骤按照文献报道的方法进行[20.,21]. 相应的mRNA序列文库被命名为S1('NAU-DY',dyu-S1;'NAU-YB',YB\u S1),S2('NAU-DY',DY\u S2;'NAU-YB',YB\u S2)和S3('NAU-DY',DY\u S3;'NAU-YB',YB\u(S3)库。
小RNA文库是根据“NAU-DY”中针对Illumina®(美国内布拉斯加州)的NEBNext®多重小RNA文库制备装置的说明从总RNA中生成的。sRNA文库构建和测序的详细实验操作步骤遵循前面的方法[22[分别命名为DS1,DS2和DS3库的相应SRNA-SEQ库。其中,从我们之前的研究中引用了“Nau-yh”的mRNA-SEQ和SRNA-SEQ数据[序列读取存档(SRA)与GenBank登录号:SRX707630] [13,17].整合mRNA-seq和sRNA-seq分析的技术流程如图所示。2..
基因组映射和差异表达分析
参考基因组序列从萝卜基因组网站下载(ftp://ftp.kazusa.or.jp/pub/radish.),并从我们两篇发表的论文中下载了‘nauy - yh’和‘CKA’两个萝卜转录组序列,这些序列均作为本研究的参考序列[23,24].在RNA-SEQ读取到参考序列的比对之前,通过去除适配器读取和污染物来筛选原始读取以实现高质量的清洁读取。对于mRNA-SEQ数据分析,清洁读数被映射到萝卜的参考序列,不超过两个不匹配的使用TOPHAT2软件[25].采用FPKM方法计算基因表达量。使用TMM方法对读取计数数据进行了规格化处理P-value采用泊松分布模型计算。FDR(错误发现率)是由P-多个测试中的值范围。在本研究中,| log的阈值2.FC(折叠变化)| > 1 withQ- 选择value <0.005作为模拟生物变化,以确定基因是否在Degseq分析中是显着的差异表达[26,27]
对于SRNA-SEQ数据分析,Bowtie软件将独特的小RNA映射到萝卜基因组序列[28].剔除匹配非编码rna的序列,包括RNAs、tRNAs、snrna、snoRNAs。用miRBase 21软件对已知miRNA序列搜索剩余的独特序列,鉴定已知miRNA。然后使用miREvo对剩余的未知rna进行预测[29.]和mirdeep2软件[30.].使用DESEQ软件识别来自突根菌增厚的不同发育阶段的差异表达的miRNA(DEM)[31.].对于所有比较,mirnas与|日志2.FC | > 1和已调整Q-value < 0.01被指定为dem。
miRNA目标预测和注释
psrobot_tar在psrobot中进行miRNA靶基因的预测[32.].去分类(去数据库,http://www.geneontology.org。)和kegg pathway(kegg数据库,http://www.genome.jp/kegg/),分别对不同功能类别的基因进行分配,并预测其生物学功能。校正条件下进行KEGG途径富集和GO富集分析P值< 0.05。
逆转录定量PCR (RT-qPCR)分析
根据制造商的说明,进行总RNA和miRNA提取(天根)和逆转录(Takara)。使用SYBR Primix Ex Taq试剂盒(Takara)进行RT-QPCR,并在罗氏灯芯480仪器上进行扩增反应[33.].这rsactin.分别用5.8S核糖体RNA (rRNA)作为归一化的内参基因。用2计算各基因的相对表达量-△△CT方法。三次重复及邓肯试验(P < 0.05) were conducted, and the Pearson correlation coefficient was calculated by DPS software to evaluate the correlation of gene expression patterns from RNA-Seq and RT-qPCR. Primers were designed by Beacon Designer 7.0 (Additional file1.:表S1)。
结果
SRNA和mRNA测序数据分析
在“Nau-dy”中的Taproot增厚期间,总共14.81米,15.65米和16.95米清洁读数是从三个开发阶段获得的。对于18至30 nt读数的长度,总共10.86米,12.20 m和13.06米清洁读取分别由DS1,DS2和DS3库生成。长度分布的所有三个文库显示出相似的尺寸特性,范围为18至30nt,特别是对于大多数SRNA读数富集在21和24 NT的长度(附加文件1.:图S1)。其中,7.56 M (69.59%) sRNA、9.58 M (78.52%) sRNA和10.55 M (80.79%) sRNA reads分别成功映射到DS1、DS2和DS3库对应的参考序列(附文件)1.:表S2)。
为了鉴定萝卜的Taproot增厚期间调节基因,来自两个高级自交系的六个cDNA文库由植物构建,在Taproot增厚的三个发育阶段(S1,S2和S3)。In the line of ‘NAU-DY’, a total of 34.84 M, 25.42 M, and 28.28 M clean reads were obtained from DY_S1, DY_S2, and DY_S3, respectively, from which 22.62 M (64.91%), 16.19 M (63.69%), and 18.10 M (64.01%) were correspondingly uniquely mapped (Additional file1.:表S3)。而在‘naul - yb’行中,从YB_S1、YB_S2和YB_S3分别生成了32.54 M、33.10 M和31.62 M的clean reads,其中从YB_S1、YB_S2和YB_S3分别唯一映射21.14 M(64.95%)、22.36 M(67.56%)和21.21 M(67.08%)(附加文件1.:表S3)。
直根增厚三个发育阶段的DEMs鉴定
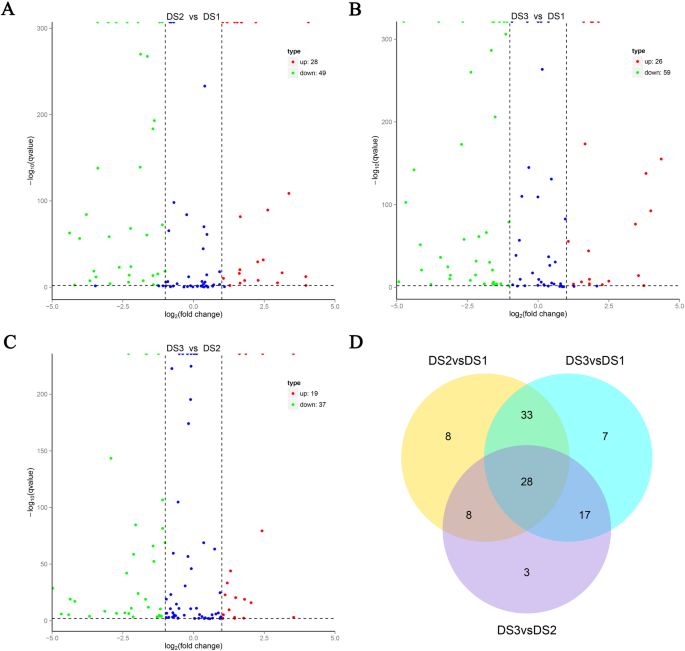
从DS2 VS DS1,DS3 VS DS1和DS3 VS DS2分别鉴定了总共77,85和56个DEM(图。3.a, b, c和附加文件1.:表S4)。与皮层前分裂期相比,皮层分裂期上调mirna 28个,下调mirna 49个。DS3和DS1中分别鉴定出85个dem,其中26个上调mirna, 59个下调mirna。然而,只有56个mirna在DS3和DS2中被鉴定为差异表达,其中19个mirna上调,37个mirna下调。有趣的是,只有8个、7个和3个mirna分别在DS2 vs DS1、DS3 vs DS1和DS3 vs DS2中特异性表达;每两两比较分别有33、17和8个dem;但在三组比较中共用了28个dem(图。3.d)。DEMS的Heatmap聚类显示在附加文件中1.:图S1,结果表明,这些miRNA的表达水平表现出萝卜根眩光加厚期间动态变化的特征。
‘naud - yh’和‘naud - dy’从sRNA-seq数据中总共共享了16个dem(表1.).在这些DEM中,14个DEM是保守的miRNA,两种DEM是非保守的miRNA。有趣的是,MiR394a,mir408-5p和mir828的表达模式在“Nau-yh”和'Nau-dy'之间类似;而MiR395A的“Nau-yh”和“Nau-Dy”的两行相应比较(表1.). 同时,与小尺寸基因型“NAU-YH”相比,在大尺寸萝卜基因型“NAU-DY”的直根增厚过程中,共有12个DEM特异性差异表达(表1)2.).其中,在DS3 VS DS1和DS3 VS DS2对中下调miR165A-3P和MIR165A-5P,并且MIR167C-5P和MIR167D在DS2 VS DS1和DS3 VS DS1对中下调;虽然MiR167A-3P在每个比较对中上调。这些结果表明MiR394a,mir408-5p和mir828可能涉及Taproot增厚,而mir395a和特定于'nau-dy'的dem可能有助于根系差异。
参见鉴定和功能性浓缩分析
共有4131个、4979个和1635个基因分别在YB\u S2和YB\u S1、YB\u S3和YB\u S1以及YB\u S3和YB\u S2中差异表达。同时,从dyu S2与dyu S1、dyu S3与dyu S1以及dyu S3与dyu S2中分别鉴定出2499、3970和1924个deg。在这些DEG中,“NAU-YB”和“NAU-DY”(附加文件)共共享了2606个DEG2.:表S5)。DEGs的热图显示,‘naud - yb’和‘naud - dy’的DEGs表达模式相似1.:图S2);虽然存在更多的Degs,并且他们的表情模式在'Nau-yh'中的表达模式比'Nau-Yb'和'Nau-dy'中的那些13].
对来自‘nauy - yb’和‘nauy - dy’的相同比较对共享的2606个DEGs进行了氧化石墨烯富集分析。“纤维素微纤维组织”(GO:0010215)、“细胞生长”(GO:0016049)、“碳水化合物生物合成过程”(GO: 0016051)和“s -腺苷甲硫氨酸生物合成过程”(GO:0006556)在S3和S1中特别富集,而“碳水化合物代谢过程”(GO:0010215) (GO:0016049)和“s -腺苷甲硫氨酸生物合成过程”(GO:0006556)中富集。0005975)与各比较对在萝卜主根增厚方面相同(表3.).同时,使用Kegg浓缩分析来识别所涉及的基因的临界途径。‘starch and sucrose metabolism’ (ath00500) was shared among three comparison pairs, ‘phenylpropanoid biosynthesis’ (ath00940), ‘glucosinolate biosynthesis’ (ath00966) was shared between two comparison pairs (S2 vs S1 and S3 vs S1), whereas ‘thiamine metabolism’ (ath00730) was specifically enriched in S3 vs S2 (Table4.).结果表明,它是一种物质和能量代谢的过程,促进萝卜的根茎增厚期间细胞生长和器官扩大。
DEM目标预测和关联分析DEG和DEM
利用psRobot软件从NAU-DY(附加文件)的sRNA数据中预测了1280个目标1.:表S6)。在这些靶点中,共有849个靶点和431个靶点分别属于68个已知的miRNAs和36个新的miRNAs(补充文件)1.:表S6)。其中,即使这些miRNA靶向不同的基因,几个靶标的注释属于同一家族,例如,包括miR156a-3p,miR159b-3p,mir159c,mir5658,miR827,miR828,miR858a,mir858b和的若干已知的miRNA属于同一家族。新颖的mirnas,包括novel_119,novel_136,novel_1,novel _37和novel目标多年电价域基因(附加文件1.:表S6)。
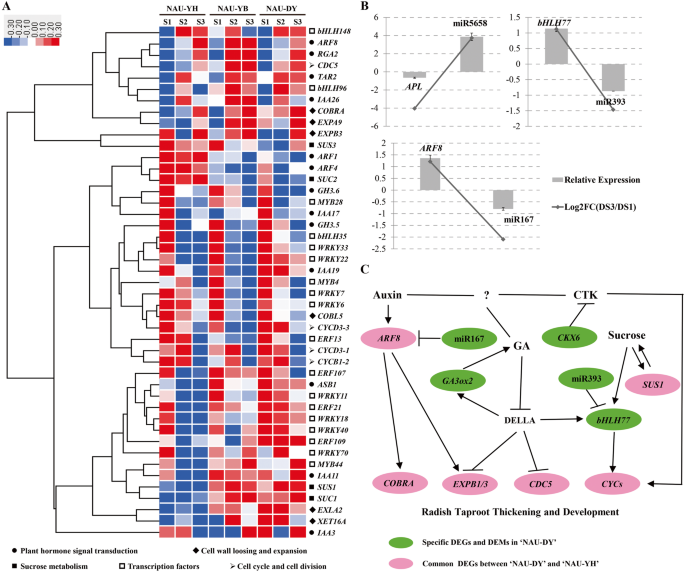
与小尺寸基因型“NAU-YH”相比,大尺寸萝卜基因型“NAU-DY”在直根增厚期间共有12个DEM特异性差异表达(表1)2.). 为探讨miRNA在直根增厚过程中的调控网络,对12个NAU-DY特异性dem进行了miRNA与mRNA的关联分析。结果表明,miR167c-5p(miR167c-5p)具有靶向性ARF8), miR393a-5p(被bHLH77), miR5658(被APL)被鉴定为‘naud - dy’在萝卜主根增厚过程中特异差异表达(附加文件1.:表S6)。
RT-QPCR验证
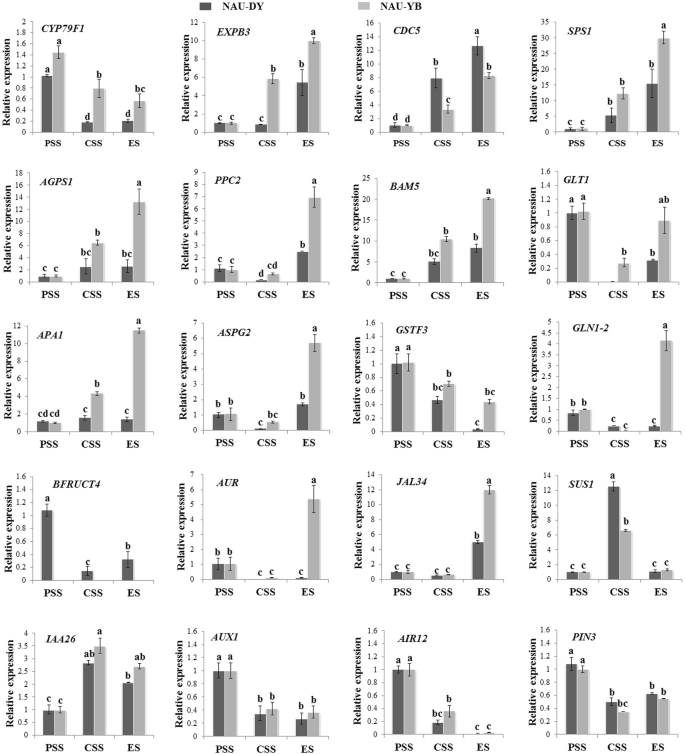
为了探讨萝卜噻虫细节增厚期间的基因表达模式,对来自“Nau-Dy”和“Nau-Yb”的RNA-SEQ数据进行了20个基因进行RT-QPCR分析。RT-QPCR结果表明,根培养蛋白12中养蛋白诱导的表达水平(RSAIR12)、生长素转运蛋白1 (rsaux1.),生长素外排载体组分3(RsPIN3)、二同蛋氨酸n -羟化酶(CYP79F1型)、谷氨酸合成酶1 (GLT1),谷胱甘肽S-转移酶F3(GSTF3.)和酸-果糠糖苷酶4 (BFruct4.), PSS的表达量高于CSS和ES,而生长素响应蛋白(IAA26)和蔗糖合成酶1(SUS1型)在CSS高于PSS和ES(图。4.).同时,扩展蛋白B3(EXPB3)细胞分裂周期5样蛋白(CDC5)、蔗糖-磷酸合酶1 (SPS1)、花椒素相关凝集素34 (JAL34.)、谷氨酰胺合成酶、胞质同工酶1-2 (JLN1-2),保护细胞2中的天冬氨酸蛋白酶2(ASPG2公司),天冬氨酸蛋白酶A1(亚太地区1),β-淀粉酶5(BAM5)、磷酸烯醇式丙酮酸羧化酶2 (PPC2)葡萄糖-1-磷酸腺苷酸转移酶(AGPS1.), ES比CSS和PSS高(图1)。4.).此外,RT-QPCR表达谱表明,与RNA-SEQ数据一致的20个随机选择的基因和两个基因包括JAL34.和Aur.仅在ES和CSS显示差异。总的来说,Pearson相关系数结果显示了RNA-SEQ数据与mRNA水平的RT-QPCR分析之间的正相关(R= 0.78,P = 0.0006), indicating the reliability of the transcriptomic data (Additional file1.:图S3)。
为了验证MiRNA的表达模式及其在萝卜中的相应靶标,选择总共14个DEM和三种差异表达的靶基因进行RT-QPCR分析。如图所示S4,RT-qPCR分析的表达模式与sRNA-seq得到的表达模式一致,但两种方法在表达差异程度上存在差异。这种不一致性可能是由两种不同的计算方法造成的。此外,mirna与其相应靶点(miR167-ARF8,mir393-bHLH77miR5658 -APL)可以在表达水平中发现,表明SRNA-SEQ数据和miRNA介导的基因沉默的可靠性涉及萝卜噻吩加厚(图。5.).
Taproot形成与发展的分子动态调节网络
萝卜主根增稠具有复杂的分子调控机制[13,15,17]. 一般来说,参与萝卜形态发生的物质包括蔗糖、淀粉和蛋白质是萝卜主根形成和发育所必需的。SUS1型,SUS3型,SUC1,SUC2)参与了这些过程。他们之中,SUS1型可能有助于在萝卜中加厚,长而厚的根[15].与植物激素生物合成和信号转导有关的基因包括生长素(如生长素)。索尔40,IAA26,IAA3,IAA7.),CTK(例如LOG8,CKX6),和GA(例如。GA2ox1,GA3OX2,RGA2)可能是萝卜主根增厚信号转导过程中的重要调控因子[34.,35.].细胞周期由F箱蛋白调节(例如,cycu4-1,CYCD3-1,CDC5)和周期素依赖性激酶(例如。KRP5) [36.,37.,38.],这可能会确定萝卜根根的细胞数。细胞膨胀由扩展蛋白调节(例如EXPA9,EXPB1/3),木聚糖内转葡萄糖基酶/水解酶蛋白(例如。XTH9,XTH7)和根细胞伸长因子(例如眼镜蛇) [35.,39.],这可能会在Taproot加厚期间确定细胞尺寸。此外,发现几种转录因子(TFS)参与其他植物的根部发育,包括米饭,拟南芥,一些TFS包括BHLHS(例如MIR393-bHLH77,bHLH96,bHLH148型),mybs(myb4.,MYB28,MYB44)和wrkey(例如:WRKY12型,WRKY19号,WRKY33)在这项研究中被鉴定,表明它们可能在萝卜的直根增厚中起关键作用(附加文件)2.:表S5) [40,41.,42.,43.].
完全,在具有不同根尺寸的三个先进的自交系中,45种共用基因差异表达,并且它们相应的表达模式如图2所示。5.一种。结果表明,大多数Degs共享类似的表达模式,而几次(例如SUS1型,SUC1,EXPB3,EXLA2公司,ERF109型)表现出差异表达模式。有趣的是,嗜酸盐蛋白3氧化酶2(GA3OX2),细胞分裂素氧化酶6 (CKX6)、miR167c-5p(被ARF8),和miR393a-5p(通过bHLH77)在' naud - dy '中特异性差异表达。在此基础上,提出了一个假定的萝卜主根增厚和发育调控模型(图1)。5.b、c)。简而言之,蔗糖(蔗糖代谢的关键基因:SUS1型)作为信号分子可以诱导miR393-bHLH77特定表达[44.,45.],进而调控下游基因(CYCs公司)在萝卜根噻滴加厚期间在细胞分裂中发挥了重要作用[40,43.,46.].参与生物合成的一些基因和生长素的信号转导(mir167-ARF8),CTK(CKX6)和ga(GA3OX2)也调控了一些功能基因(眼镜蛇,EXPB1/3,CDC5),表明它们可能在启动萝卜噻吩增厚和发育中发挥作用(图。5.) [47.].
讨论
综合MRNA-SEQ和SRNA-SEQ方法提供了有价值的工具,用于利用潜在的关键基因和揭示用于特征的复杂监管网络[48.,49.,50.]. 萝卜主根增厚是一个复杂的生物过程,包括一系列物质积累和信号转导途径。迄今为止,萝卜直根形成的分子机制尚未完全阐明。本研究对三个萝卜自交系主根增厚过程中的mRNA-seq和sRNA-seq进行了综合分析,以进一步了解其主根形成的分子机制。以3个具有代表性的萝卜自交系为材料,研究了肉质根大小的差异。“NAU-DY”和“NAU-YB”共有2606个deg,而“NAU-YH”和“NAU-DY”共有16个dem,12个dem在“NAU-DY”中有特异性差异表达。此外,一些关键基因包括SUS1型,EXPB3,CDC5采用RT-qPCR分析。本研究以3个萝卜高级品系为研究对象,采用mRNA-seq和sRNA-seq相结合的方法,对主根形成过程中可能涉及的关键基因进行了系统分析。
与根系差异相关的关键基因
与根型差异相关的基因被认为是候选人,其促进或抑制萝卜中的根尖增厚[51.].利用抑制消减杂交(suppression sub消减hybridization, SSH)技术在瘦根和粗根中分别鉴定出140个和70个特异表达基因[18].瘦根型品种中涉及苯丙素代谢的几个基因都过表达,苯丙素是木质素合成的前体,提示木质素生物合成途径参与了萝卜主根增厚[52.].此外,与乙烯生产和根毛伸长率相关的基因也在瘦性的型根中过表达,并且可能是乙烯和侧面根发育抑制细胞膨胀和主根部的伸长[53.].因此,瘦性和厚根系之间的含量可能涉及根型变异。
In this study, a total of 12 DEMs belonging to miR165, miR167, miR319, miR5658, miR8175, miR857, miR170, and miR393 family miRNAs were specifically differentially expressed during taproot thickening in large acicular radish ‘NAU-DY’, which might be related to root-type variation. On the other hand, many DEGs were identified to be specifically or commonly expressed between ‘NAU-YH’ and ‘NAU-DY’ (Fig.5.).例如,参与淀粉和蔗糖代谢的基因,包括蔗糖合成酶4(苏斯4)、蔗糖-磷酸合酶2 (SPS2)和6-磷酸果糖激酶(FK1型,FK7型)是专门用“naud - dy”表示的,而SUS1型,蔗糖合酶3(SUS3型),蔗糖输送蛋白质(SUC1,SUC2)、蔗糖-磷酸合酶2 (SPS1)和6-磷酸果糖激酶(FK3型,FK6型)在' naud - dy '和' naud - yh '中共表达。mRNA和sRNA的关联分析结果显示,miR167c-5p (miR167c-5p被ARF8), miR393a-5p(被bHLH77), miR5658(被APL在“Nau-dy”的萝卜噻potoot增厚期间,被鉴定为显着表达,表明MiR393-bHLH77,mir167-ARF8, miR5658APL(MYB的成员)可能会在萝卜的根尖增厚中发挥关键作用。
SUS1和CDC5可能是萝卜主根增厚的原因之一
增粗根中碳水化合物代谢被显著激活,尤其是在细胞增殖组织中,其中SUS1型与根系增厚率有关[15].在这项研究中,GO术语“碳水化合物代谢过程”(GO: 0005975)显著富集并被任何比较对共享,不论根的大小,这与之前在萝卜中的报告一致[13,15],暗示碳水化合物代谢可能对噻根增厚至关重要。
有趣的是,在这些参与碳水化合物代谢过程的基因中,SUS1型三个萝卜高级自交系的基因表达模式存在差异。对于较大的针状萝卜,表达水平较高SUS1型(rsa1.0 _00483.1 1_g00003.1)在皮层分裂阶段达到峰值,远远高于中等倒卵形和小圆形萝卜(FD536105)。此外,SUS基因对马铃薯块茎化的发展具有重要意义[54.,番茄坐果[55.]胡萝卜根形成[56.]、玉米籽粒形成[57.]、麦粒形成[58.],水稻籽粒形成[59.].在本研究中,RT-QPCR验证结果表明SUS1型在CSS中显示高表达模式,“Nau-Dy”中的特殊性,表明SUS1型在主根的增厚过程中起着至关重要的作用,特别是对于大的针状萝卜的增厚,而不是小的圆形萝卜(图。4.).
CDC5是一种编码相关蛋白质的细胞周期调节器。最近,越来越多的报道AtCDC5对控制植物生长发育的miRNAs积累起正调控作用[60.].与此同时,CDC5在G2期,基因对细胞周期起着至关重要的作用,G2向M期(G2/M)的转变也受到影响AtCDC5rnai转化株(37.],和AtCDC5-VIGS转化子在脱粒前死亡并加速细胞死亡[38.].有趣的是,CDC5在Taproot增厚期间被识别出上调,无论RNA-SEQ还是ITRAQ-SEQ方法,这将在萝卜中噻虫细胞的细胞分裂中发挥重要作用[13,35.].在本研究中,RT-QPCR验证结果与先前研究的验证结果大致一致,CDC5在ES显示出高丰度表达(图。4.).这些结果初步表明CDC5对萝卜主根增厚的生长发育可能有重要作用。
萝卜根根簇的细胞伸长可能需要眼镜蛇
植物的生长发育是通过一系列有针对性的细胞分裂和细胞扩张来促进的。植物细胞壁为植物体提供了基本的机械支撑,并决定了细胞的大小和形状。纤维素微纤丝作为细胞壁的主要组成部分,其组织结构是细胞扩张的决定因素之一。在这项研究中,GO术语“纤维素微纤丝组织”(GO: 0010215)在“naud - dy”和“naud - yb”的S3对和S1对比对中显著富集,总共有两个眼镜蛇和三个眼镜蛇式基因包括cobl2.,COBL5.和COBL8.被识别出差异表达(附加文件2.).以前的研究表明眼镜蛇参与纤维素合成,控制植物细胞壁中纤维素含量及细胞方向伸长率的函数[61.,62.,63.,64.].有趣的是,在这项研究中,所有眼镜蛇基因被上调,而所有眼镜蛇式基因在“Nau-dy”和“Nau-Yb”之间的S3 VS S1对比较中进行了下调,暗示眼镜蛇可能在萝卜中的根尖增厚的细胞伸长率上发挥着关键作用。
结论
这是三萝卜增厚期间三种萝卜高级自交系中转录组和miRNA一体化分析的第一报告。在“Nau-Dy”和“Nau-Yb”之间共有2606次,这在“苯丙烷化生物合成”,“葡萄糖酸盐生物合成”和“淀粉和蔗糖代谢”途径中显着富集。与此同时,A.total of 16 DEMs were shared between ‘NAU-DY’ and ‘NAU-YH’, whereas 12 miRNAs showed specifically differential expression during taproot thickening in ‘NAU-DY’ with large acicular root when compared with ‘NAU-YH’ with small circular root. Association analysis between DEMs and DEGs indicated that miR393-bHLH77,mir167-ARF8, miR5658APL可能与萝卜的根型变化有关。此外,RT-QPCR验证结果表明,评估的DEGS / DEM与RNA-SEQ数据一致高度协议。这些发现将提供有关综合了解萝卜根茎增厚的分子调节机制的综合了解的有价值的信息,并促进了根植物作物的进一步遗传操纵和质量改善。
数据和材料的可用性
支持本文结果的所有原始数据已被沉积到NCBI序列读取归档中,其中包括六个cDNA文库(对于mRNA-SEQ)和三个小RNA文库,并在加入号SRP158483下的原始测序数据(https://www.ncbi.nlm.nih.gov/sra/?term=SRP158483).支持本文结果的处理数据包括在文章和补充配置文件中。
缩写
- FDR:
-
错误发现率
- 可见:
-
差异表达基因
- 民主党:
-
差异化的mirnas
- Kegg:
-
Kyoto基因和基因组的百科全书
- RT-qPCR:
-
反转录定量PCR
参考
- 1.
Moreno-risueno ma,Sozzani R,YardımcıGG,Petricka JJ,Vernoux T,Blilou I等。根部发育过程中组织形成的转录控制。科学。2015; 350:426-30。
- 2.
宋伟,薛锐,宋勇,毕勇,梁铮,等。两个烟草品种一级侧根伸长对低钾的差异响应与一氧化氮有关。植物生长杂志2018;37:114-27。
- 3.
胡志明,陈志明,陈志明,胡志明。玉米根系发育的蛋白质组学研究植物科学与技术,2018;
- 4.
张震,张欣,林志军,王杰,徐敏,赖杰,等。玉米节根数的遗传结构。植物j . 2018; 93:1032-44。
- 5。
Sanchez DL,Liu S,Ibrahim R,Blanco M,LübberstedtT。玉米根系架构特征的双倍倍增型钝化线的基因组 - 宽协会研究(玉米l .)。植物科学。2018;268:30-8。
- 6。
赵勇,程勇,宋勇,黄勇,周勇,刘旭,等。水稻ERF3与WOX11的互作通过调控细胞分裂素信号转导相关基因的表达,促进冠根发育。植物细胞。2015;27:2469–83.
- 7。
陈H,MA B,周Y,他SJ,TANG SY,LU X等。E3泛素连接酶SOR1通过调节Aux / IAA蛋白的稳定性来调节水稻根系中的乙烯反应。Proc Natl Acad Sci U S A. 2018; 115:4513-8。
- 8。
韩妍,万海华,程聪,王杰,杨文瑞,潘浩特,等。中国植物花瓣发育过程中转录组动态的比较RNA序列分析罗莎中华民神.SCI REP。2017; 7:43382。
- 9。
元股,李河,陈赫,张CJ,陈立,昊QN等。RNA-SEQ对大豆五种不同发育阶段的结节发育分析(大豆)接种Bridyrhizobium japonicum应变113-2。SCI REP。2017; 7:42248。
- 10.
yu k,xu q,da x,guo f,ding y,deng x.转录组果实开发过程中的变化和甜橙成熟(柑橘).BMC基因组学。2012;十三10。
- 11.
冯超,陈敏,徐长军,白玲,尹晓荣,艾伦,等。杨梅转录组学分析(杨梅)使用RNA-SEQ的果实开发和成熟。BMC基因组学。2012; 13:19。
- 12.
Zouari I, Salvioli A, Chialva M, Novero M, Miozzi L, Tenore GC等。从根到果实:RNA-Seq分析表明,丛枝菌根共生可能影响番茄果实代谢。BMC基因组学。2014;15:221。
- 13。
于汝,王杰,徐磊,王勇,王汝,朱×,等.萝卜主根加厚过程中转录组分析揭示了复杂的调控网络(萝卜l .)。正面。植物SCI。2016; 7:1210。
- 14。
Kitashiba H,Li F,Hirakawa H,Kawanabe T,Zou Zw,Hafegawa Y等。萝卜的草稿序列(萝卜l .)基因组。DNA研究》2014;21:481 - 90。
- 15.
三井Y,岛村M,小松K,Namiki N,Shibata Hatta M,Imai M等.萝卜块根形成和发育的基因组和综合基因表达谱。Sci代表2015;5:10835.
- 16。
Jeong YM, Kim N, Ahn BO, Oh M, Chung WH, Chung H,等。通过与芸苔属基因组的染色体水平比较,阐明萝卜的三重祖先基因组结构。理论应用,2016;129:1357-72。
- 17。
俞锐,王勇,徐玲,朱欣,张伟,王锐,等。根microrna转录组分析揭示了萝卜主根增厚的新思路(萝卜l .)。BMC Plant Boil. 2015;15:30。
- 18。
Zaki Hem,Yokoi S,Takahata Y.鉴定萝卜根形状有关的基因(萝卜)使用抑制减去杂交。品种sci。2010; 60:130-8。
- 19。
Zaki Hem,Takahata Y,Yokoi S.三个萝卜根系形态和解剖学特征的分析(萝卜)不同品种的根形状不同。中国生物医学工程学报,2012;
- 20.
孙晓,徐玲,王勇,罗晓,朱晓,Kinuthia KB,等。基于转录组的基因表达谱识别萝卜盐胁迫反应的关键差异表达基因(萝卜l .)。植物细胞代表2016;35:329-46。
- 21.
王锐,梅勇,徐玲,朱欣,王勇,郭静,等。全基因组差异表达基因的鉴定为了解萝卜热应激反应的调控网络提供了线索(萝卜l .)。功能整合基因组。2018;18:25 - 39。
- 22.
徐乐,王勇,翟乐,徐勇,王乐,朱旭,等。萝卜镉敏感微rna及其靶基因的全基因组鉴定与鉴定(萝卜l .)的根源。实验机器人。2013;64:4271-87。
- 23.
于荣刚,徐玲,张伟,王勇,罗晓斌,王瑞荣,等。德诺维Taproot转录组测序和分析萝卜中蔗糖代谢的主要基因(萝卜L.)。前植物科学。2016;7:585.
- 24.
王Y,潘y,刘z,zhu xw,zhai ll,xu l等。德诺维萝卜的转录组测序(萝卜与硫甙代谢有关的主要基因分析。BMC基因组学。2013;14:836.
- 25.
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg SLS。TOPHAT2:在存在插入,缺失和基因融合的情况下,转录om的精确对准。基因组Biol。2013; 14:R36。
- 26.
他g,陈b,王x,li x,李j,他,他。玉米杂交体转录组和外形变异的保护与分歧。基因组Biol。2013; 14:R57。
- 27.
Li Al,Liu Dc,Wu J,Zhao XB,Hao M,Geng SF等。mRNA和小RNA转录omes揭示了新生六倍体麦片中所有多聚倍差杂种优势的动态同源性调节的见解。植物细胞。2014; 26:1878-900。
- 28。
人类基因组短DNA序列的超快速和高效记忆对齐。基因组医学杂志。2009;10:R25。
- 29。
文M,沈Y,史S,唐T。miREvo:一个用于下一代测序实验的整合microRNA进化分析平台。生物信息学。2012;13:140.
- 30.
弗里德兰德Mr,Mackowiak Sd,Li N,Chen W,Rajewsky N.Mideepe2准确地识别七个动物的七分之一的新型MicroRNA基因。核酸RES。2011; 40:37-52。
- 31。
Anders S,Huber W.序列计数数据的差异表达分析。基因组Biol。2010; 11:R106。
- 32.
吴海杰,马玉凯,陈婷,王梅,王旭杰。PsRobot:一个基于web的植物小RNA荟萃分析工具箱。核酸研究,2012年;40:22–8.
- 33.
关键词:萝卜,基因表达,内参基因,内参基因萝卜L.)采用实时荧光定量PCR。生物化学与生物物理研究所2012;424:398-403。
- 34.
李晓军,杨建林,郝斌,吕玉成,钱志良,李勇,等。比较转录组和代谢组分析为主根增厚的分子机制提供了新的见解田七.BMC Plant Bio. 2019;19:451。
- 35.
谢妍,徐丽,王妍,范立新,陈丽尔,唐美杰,等。比较蛋白质组学分析为萝卜直根形成的复杂调控网络提供了新的研究方法(萝卜L.)。Hortic Res.2018年;5:51.
- 36.
Calzada A,Sánchezm,sáncheze,bueno a。CDC6蛋白的稳定性由Cycloromyces Cerevisiae中的细胞周期依赖性激酶/细胞周期蛋白B络合物调节。J Biol Chem。2000; 275:9734-41。
- 37。
林志强,尹克强,朱德立,陈志林,顾海英,曲丽娟。AtCDC5调控细胞周期G2向M的转变,对拟南芥茎尖分生组织的功能至关重要。细胞研究》2007;17:815-28。
- 38。
Lin ZQ,Yin KQ,Wang XX,Liu MH,Chen ZL,Gu HY,等。病毒诱导的AtCDC5基因沉默导致拟南芥叶片细胞加速死亡。植物生理生化。2007;45:87–94.
- 39。
Ko JH,Kim JH,Jayanty SS,Howe Ga,Han Kh。COBRA功能丧失,导向细胞扩张的决定因素,调用细胞防御反应拟南芥蒂利亚纳.实验机器人,2006;57:23 - 36。
- 40.
SRILAKSHMI M,REBECCA SL。BHLH转录因子抹刀通过控制根分生组织的大小来调节根的生长。BMC植物生物。2013;13:1.
- 41.
acta photonica sinica, 2011, 38(5): 732 - 736 .顾敏,张娟,李辉,李锐,戴翔,等。在水稻中,MYB1是一个r2r3型MYB转录因子,它协调调节磷酸盐稳态的维持和根系发育。实验机器人。2017;68:3603-15。
- 42.
Ballachanda ND,Athikkattuvalasu SK,Kashchandra GR.WRKY75转录因子是拟南芥磷酸获得和根系发育的调节因子。植物生理学。2007;143:1789–801.
- 43.
李廷涛,杨世英,康晓坤,雷伟,乔凯,张德伟,等。bHLH转录因子基因AtUPB1公司在拟南芥中通过调节细胞周期进程来调节生长。Biochem Biophys Res Co. 2019; 518:565-72。
- 44.
植物糖信号转导的研究进展。植物医学杂志。2006;57:675 - 709。
- 45.
刘XJ,Xh,刘X,Hu DG,王XF,你CX等。MDSNRK1.1与MDJAZ18相互作用,调节蔗糖诱导的苹果中的花青素和原霉素积累。J Exp Bot。2017; 68:2977-90。
- 46.
杨XM,任业,蔡Y,牛M,冯ZM,静朗,等。过度表达OsbHLH107,一个基本螺旋-环-螺旋转录因子家族的成员,提高水稻的粒径(水稻l .)。米饭,2018; 11:41。
- 47.
胡耶,以色列A,ori n,太阳tp。Della和Arf / IAA之间的相互作用在嗜酸甘油蛋白和蟾蜍蛋白信号传导之间介导串扰,以控制番茄中的果实引发。植物细胞。2018; 30:1710-28。
- 48.
龙JM,刘Z,吴XM,方烨,佳豪,谢ZZ等。基因组测量mRNA和小RNA转录组含有柑橘apomixis的启动。J Exp Bot。2016; 67:5743-56。
- 49.
Sun Q,Du X,Cai C,Long L,张S,Qiao P等人。成为花或结果分支:mRNA和小RNA从不同棉发育阶段转录的洞察力。SCI REP。2016; 6:23212。
- 50.
韩X,尹h,歌曲x,张y,liu m,sang j等。高rNA的整合,DegraDome和转录组测序在超读物器Sedum Alfredii揭示了复杂的调节网络,并向植物植物修复提供了深入的洞察力。植物Biotechnol J. 2016; 14:1470-83。
- 51.
三井Y.块根发育过程中的基因表达谱。在:Nishio T, kitashba H,编辑。萝卜的基因组。Gewerbestrasse, Switzerland:施普林格International Publishing;2017.109 - 19页。
- 52。
沃格特T。苯丙烷生物合成。摩尔植物。2010;3:2–20.
- 53。
Kieber JJ, Rothenberg M, Roman G, Feldmann KA, Ecker JR. CTR1是拟南芥乙烯反应通路的负调控因子,编码一个Raf蛋白激酶家族成员。细胞。1993;72:427-41。
- 54。
Baroja-Fernández E, Muñoz FJ, Montero M, Etxeberria E, Sesma MT, Ovecka M,等。提高转基因马铃薯蔗糖合酶活性(茄属植物tuberosuml)块茎导致淀粉、adp葡萄糖和udp葡萄糖水平和总产量的增加。植物生理学杂志。2009;50:51 - 62。
- 55。
d'aoust ma,yelle s,quoc bn。番茄水果蔗糖合成酶的反义抑制降低了果实的果实环境和蔗糖卸载能力。植物细胞。1999; 11:2407-18。
- 56.
唐国强,Sturm A.胡萝卜蔗糖合酶的反义抑制(胡萝卜胡萝卜L.)影响生长而不是蔗糖分区。植物mol biol。1999; 41:465-79。
- 57.
玉米胚乳中蔗糖合成酶的遗传控制。Mol Gen Genet, 1981; 184:372-6。
- 58.
小麦胚乳中蔗糖合酶活性与最大重量的关系。植物杂志。1986;82:7-10。
- 59.
KATO T.蔗糖合成酶活性在水稻品种胚乳中的变化。农作物科学。1995年; 35:827-31。
- 60.
CDC5是一种DNA结合蛋白,它正向调控初级microRNA转录后加工和/或转录。美国国家科学研究院2013;110:17588-93。
- 61.
Roudier F, Fernandez AG, Fujita M, Himmelspach R, Borner GHH, Schindelman G等。COBRA是一种拟南芥细胞外糖基磷脂酰肌醇锚定蛋白,通过参与纤维素微纤丝的取向,特别控制高度各向异性的扩张。植物细胞。2005;17:1749 - 63。
- 62.
关键词:聚类,聚类,聚类,聚类拟南芥COBRA蛋白促进质膜上的纤维素结晶。生物化学杂志。2014;289:34911-20。
- 63.
戴XX,你cj,陈gx,李xh,张qf,吴迪。OSBC1L4编码一种类似COBRA样蛋白,其影响水稻中的纤维素合成。植物mol biol。2011; 75:333-45。
- 64.
Hochhedinger F,Wen TJ,Zimmermann R,Marolle PC,Silva ODC,Bruce W等人。玉米(玉米L.)Roothairless3基因编码推定的GPI锚定,单子叶特异性眼镜蛇样蛋白质,显着影响谷物产量。工厂J. 2008; 54:888-98。
致谢
不适用。
资金
国家科技支撑计划项目(no . 2018YFD1000800, no . 2017YFD0101806);国家自然科学基金项目(no . 31601766);江苏省农业科技创新基金项目(no . CX (19)3045);江苏省农业产业技术体系专项资金[JATS(2019)428]和江苏省高等学校重点学科发展计划项目(PAPD)。资助机构没有参与这项研究的设计;收集、分析或解释数据;或写手稿。
作者信息
从属关系
贡献
YX,LX和LL设计了实验。YX和YW分析了RNA-SEQ数据并写了稿件。JY,MT和CL帮助分析RT-QPCR数据和收集的样品。LL,YC,JD和EM修订了稿件。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
同意出版
所有作者都同意发布。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
斯普林格自然保持中立,就管辖权的要求,在出版的地图和机构的联系。
补充信息
附加文件1图S1
. 小rna在萝卜主根增厚过程中的序列长度分布。图S2.萝卜主根增厚过程中DEGs和dem的热图。a. ' naud - dy '中dem的热图;b、c分别表示‘naud - dy’和‘naud - yb’中DEGs的热图。图S3.RNA-seq和RT-qPCR基因表达谱的比较分析。通过RNA-seq和RT-qPCR随机选取20个基因,在3个比较组(DS2 vs DS1, DS3 vs DS1, DS3 vs DS2)中表现出不同的表达模式。每个数据点代表日志2.从RNA-seq (x轴)和RT-qPCR (y轴)分析中获得的规范化表达水平。图S4. 萝卜主根增厚过程中14个DEM的RT-qPCR验证。用DS2和DS1库(a)、DS3和DS1库(b)以及DS3和DS2库(c)的相对表达进行分析−ΔΔCT方法。表S1.RT-QPCR测定的引物序列。表S2.小RNA测序数据总结。表S3.mRNA测序数据汇总。表S4.‘naud - dy’萝卜主根增厚过程中dem的详细信息。表S6. 从RNA序列中提取dem及其相应的靶点。
附加文件2表S5
.“Nau-Dy”和“Nau-Yb”之间共享的Degs概述。
权利和权限
开放获取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用额度中另有规定。
关于这篇文章
引用这篇文章
谢,Y.,英,J.,徐,L。et al。全基因组sRNA和mRNA转录组分析对萝卜主根增厚动态调控的研究(萝卜l .)。BMC植物BIOL.20.373 (2020). https://doi.org/10.1186/s12870-020-02585-z
收到了:
认可的:
发表:
关键字
- de
- 民主党
- 萝卜
- RT-qPCR
- 根尖加厚