摘要
背景
通过与参考水稻叶绿体基因组序列对准,从可用的序列数据组装3018种基因型的整个叶绿体基因组,提供高质量的叶绿体基因组,用于分析比以前的任何植物研究更大的规模。
结果
叶绿体基因组的最新注释确认了13个tRNA基因和30个内含子,并确定了更多基因的功能。驯化水稻的叶绿体基因组与野生水稻截然不同。分析证实澳大利亚叶绿体分支是驯化分支的姐妹。所有驯化水稻的基因型都可以归属于两个主要的进化支之一,这表明驯化水稻的两个截然不同的母系基因组进化支在驯化水稻之前就已经分化。这些演化支非常不同的在进化枝1486多态性之间加入一个和所有这些将导致1532年进化枝到达b . 3蛋白的表达改变氨基酸序列和tRNA改变了顺序,可能与两种叶绿体的适应性进化。这些种群的多样性可能是在驯化过程中被捕获的,其中亚分支富集在特定的类群中,如香茅、热带粳稻和温带粳稻。然而,由于现代水稻育种和水稻驯化前的网状进化,叶绿体和核基因组的系统发育存在差异。籼型和aus型基因型在两个叶绿体分支中均有分布,而粳型基因型在同一分支(cladeB)中更有分布。
结论
水稻细胞质和核基因组的不同进化路径导致叶绿体基因组具有明显的功能多样性,这对水稻作物生产性能的影响需要进一步研究。
背景
米为全球粮食安全至关重要。遗传多样性的增强是改善作物适应气候变化的影响的关键策略,并且对多样性的表征在管理粮食安全方面是重要的。世界各地种植的各种水稻类型,以满足不同人口的不同粮食口味。基因组学的进展现在允许对整个基因组进行大规模分析。390 MB核基因组[1,2负责控制水稻籽粒类型差异的性状。然而,135Kb的叶绿体基因组是母系遗传的,并且高度保守,这使它成为一个有用的工具[3.用于追踪驯化基因库中的母系谱系,并将其与野生种群的多样性联系起来。在水稻中,与许多其他植物类群一样,由于来自不同种群的植物的母体基因组的转移,其母体(叶绿体)和细胞核系统发育可能有所不同。
野生稻分析(oryza.物种)在与驯化稻米有关的组中,一种基因组种类揭示了南美洲和非洲的野生窄(oryza longistaminata.和o . glumaepatula)具有最分歧的叶绿体基因组[4,5,6]而澳大利亚人[7](O. Meridionalis.[8]和相关类群)具有最分散的核基因组[9].野生叶绿素基因组oryza.已被广泛研究,并与驯化水稻相关[4,5,6].本文报道了58种野生植物叶绿体全基因组的研究oryza.,根据地理区域划分主要群体,由于亚洲地区没有自然边界和人类流动,这些地区有些重叠[5].
最近报道了超过3018个驯化水稻基因型的DNA序列数据[10.].使用来自水稻总植物DNA的序列数据对水稻证明了对叶绿体的全基因组序列的可靠分析[4].由于叶绿体在细胞内的拷贝数较高,因此从植物总DNA中读取可以高精度地确定整个叶绿体序列。这里使用的管道(并在引用的参考文献中详细解释)依赖于任何NGS全植物DNA分析中真实叶绿体序列的高丰度。在大多数DNA制备中,每一个核基因组拷贝中都有1000个或更多的叶绿体。早期基于PCR或克隆的植物叶绿体基因组序列的研究都是不可靠的,因为这些方法可以优先扩增或克隆叶绿体基因在细胞核或线粒体中的副本。在这里,我们首次使用了大量高质量的完整叶绿体基因组,并完全避免了包括核或线粒体序列的风险。我们报告了3022个水稻基因型的叶绿体基因组分析(见表)1),可以大规模比较完整和准确的叶绿体基因组序列。
结果
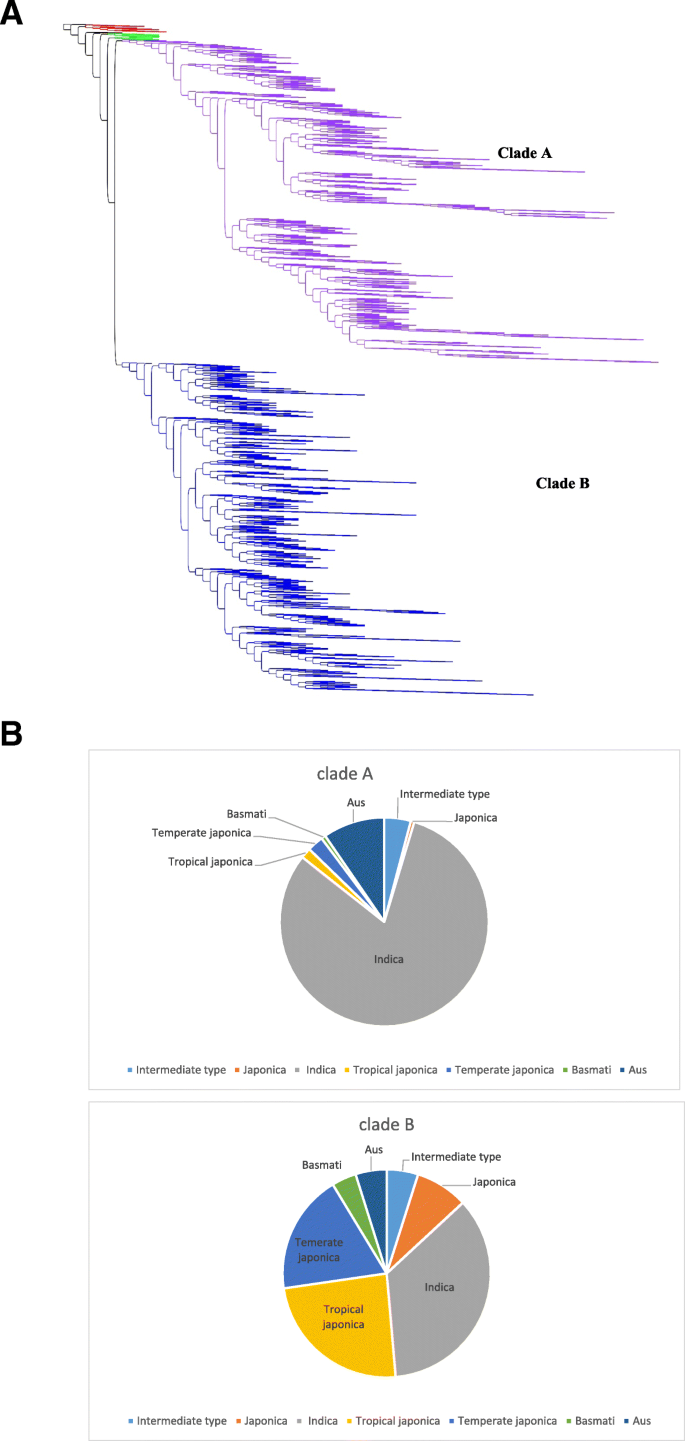
通过对3091个水稻叶绿体基因组的分析,可以分析驯化水稻基因库内的关系以及与主要野生近缘物种的关系。从总DNA序列中生成了可靠的全基因组序列,避免了由于叶绿体插入细胞核或线粒体而导致的错误并发症,这曾困扰早期的叶绿体研究[3.].在分析中,驯化水稻的叶绿体基因组不同于野生水稻的亲缘(图。1A和B)。本研究中包含的亚洲野生河流是驯化丛生的姐姐,澳大利亚野生队姐姐是所有亚洲野生和驯化的罗切尔。将驯化的rics分成两个主要的植物。CLADE A和CLADE B包含一系列水稻类型(表2).主要水稻类型、粳稻(包括热带粳稻和温带粳稻、籼稻、巴斯玛蒂稻和奥斯稻)均存在于两个支系中,但比例不同2).非洲水稻,O. Glaberrima.和野生的非洲米饭,o . bathii在疏枝A内被发现(图。1).
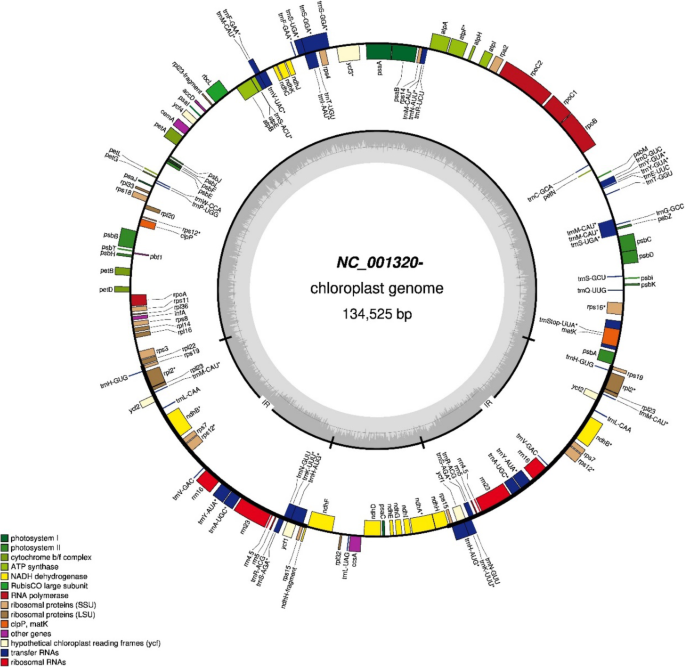
使用较新的分析工具注释叶绿体基因组以产生更完整的注释(图。2)正如最近为其他植物物种开发的[11.].用geseq注释[12.]而不是早期水稻叶绿体基因组注释的教条揭示了51个而不是38个TRNA基因,31个不是1个内含子,并为所有提出的基因提供了基因同一性,而早期的方法表明了许多没有同一性的基因,包括“假设蛋白”(补充数据文件1和2).
这两个分支的序列表明叶绿体功能不同。进化枝A的所有成员与进化枝B的所有成员在4个位置上都不同,这4个位置改变了编码蛋白的氨基酸序列或tRNA的序列(表)3.).两个进化支之间的RNA聚合酶(rpoC2)、核糖体蛋白(rpl20)和一个tRNA具有多态性。光合作用基因psb B也具有多态性,但与其他基因不同的是,这种多态性与两个进化支没有严格的关联。在进化枝A中88个是C, 1398个是T,在进化枝B中全部是C(1532),没有T。
有关两个人的更多细节,请参阅补充文件3..
讨论
早些时候oryza.叶绿体系统发育研究了整个属中不同物种的叶绿体的多样性[6].在亚洲采集的样品中,野生水稻叶绿体基因组序列的范围可能是由于野生种群和驯化到野生种群的基因流的差异,这使得解释关系的基础变得困难。在亚洲品种中可能发现叶绿体与驯化基因库非常相似的水稻[5]但是在没有详细的群体研究的情况下,基因流动的方向可能难以在最近的野生和驯养大麦的研究中揭示的研究[13.].目前的研究明确地扩展了包括亚洲驯养较大的人的关系。目前的系统发育包括比以前的任何研究(超过3000)更多的样品,并且可能更准确地[14.]比早先的研究。现在,驯化数量的驯化和更多的野生骰子现在存在于分析中。
基于核基因组分析的驯养稻米多样性的最近分析表明,粳稻,籼稻和AU型的三种单独驯化[15.]基于核基因组分析。
本研究中的驯化裁判的广泛分析并不反驳这一点,但表明驯化水稻基因型也可以将其分为两个主要的林,强烈暗示驯化两个不同的细胞质基因组的驯化型细胞质基因组的驯化,支持支持单一的多个起源概念的细胞质基因组驯化[16.].这两个分支(A和B)可能在一定程度上与粳稻和籼稻的不同起源有关。然而,由相邻叶绿体连接而产生的两个叶绿体分支随后被独特的多态性所区分(表3.)各种含有各种水稻类型(表2).Clade a包含主要籼稻。大多数japonica rices都是在思工b中,具有不同叶绿体亚片,用于巴斯马蒂,热带粳稻和温带japonica。尽管这些一般分组大多数群体都很混合。籼稻的蛋白质是跨越A的占主导地位,但也广泛分布在包括所有子分支的地图中。Clade A有更多的Aus类型,但许多人也存在于CLADE B中,确认了孔蛋白两种主要类型的孔稻类型的先前报告[5].这些结果表明,细胞质基因组和核基因组被广泛重组以产生当前的驯化水稻基因库。水稻类型可能是由核基因组压倒性的决定,但叶绿体基因组可能往往源自另一种水稻类型。这是现代品种的繁殖或由于驯化事件或驯化之后的遗传重组的产品的程度尚不清楚。
驯养的稻米似乎有共同的驯化基因座[15.而是来自一个以上的祖先群体。一种水稻驯化模式建议从不同的野生种群驯化粳稻,可能还有籼稻O. Rufipogon.和O. Nivara.分别。这需要在一定程度上的渐渗之后,才能产生两种不同类型的水稻,在驯化位点上具有共同的等位基因。这两个种群之间的杂交可以有任何一个亲本作为母本,允许具有两种叶绿体类型的驯化种群进化。最近有证据表明,野生种群通过双向杂交产生杂种[17.].生殖障碍的存在[17.可能导致单向花粉流量和快速核基因组替代或叶绿体捕获。叶绿体基因组类型对植物性能的影响尚不清楚,但可能是显着的。
对作物植物驯化的分析是野生种群源自驯养植物的潜力复杂化,这些植物开发了让他们在外面培养的特征。从驯化到野生种群的基因流量也可以进一步复杂化分析。像米饭一样的早期驯养物可能已经更长的时间在可能发生这些过程。大麦,可能是驯化的第一个植物,在肥沃的新月中驯养。西藏不同的野生大麦种群的存在表明了第二个独立驯化的可能性。然而,最近的基因组分析表明,西藏的野生大麦可能是在过去4000年中由人类引入该地区的驯化大麦[13.].
就水稻而言,野生水稻和驯化水稻之间密切关系的证据已被用作驯化的原始地点的证据[18.].然而,从驯化到野生种群的基因流也可能解释一个密切的关系。发现的两个不同的叶绿体基因组栽培稻基因库仅通过两个不同母体基因组的驯化来容易解释。核基因组驯化史要难以定义,但两种主要类型,籼稻和粳稻可能是单独的驯化产品。大米和现代植物育种的人类运动导致核和母体基因组的广泛复合在此报告。现在可以在现代分享驯化基因座o .漂白亚麻纤维卷基因库,尽管至少有两个独立的主要驯化,捕获了两个不同的母系基因库。籼型和粳稻型早已被公认[1].第三组AUS已被第三种可能的驯化解释。介绍的叶绿体证据表明,AUS基因型均具有两种主要类型的叶绿体基因组,并且不代表单独的母体归类。
叶绿体和核基因组文根在整个方面表现出显着的一种不景气oryza..
O. longistaminata.在非洲和o . glumaepatula南美洲也有类似的叶绿体基因组[4尽管它们的核基因组序列更遥远。o . glumaepatula是家养水稻近亲a基因组分支的一部分,但有一个分化的叶绿体基因组是最分化的a基因组物种。然而,核基因组分析把多样性O. Meridionalis.[19.,20.]从澳大利亚作为一个基因组的最分歧[9].
另一个例子是由澳大利亚人群提供形态相似之处提供O. Rufipogon.在亚洲。该分类群的叶绿体基因组与澳大利亚野生稻的相似[21.]但核基因组与亚洲野生队长群体[9].从这一分析得出的一般结论是叶绿体之间的转移密切相关oryza.分类群一直很普遍。最近的发现[22.,表明这是一个持续的进化过程oryza..
在驯养的大米中发现的两个母系谱系非常截然(表3.)表明这两种祖先类型在很长一段时间内发生了分化。功能多态性的存在,改变编码的氨基酸是重要的,特别是考虑到叶绿体基因组的高度保守性质。这些叶绿体的分化时间可能超过50万年[6].注释工具GESEQ允许检测比早期的工具被广泛用于叶绿体注释的更早的工具。现在正确注释了未知功能(假设蛋白质)的一些基因(补充文件1和2).在叶绿体基因组中发现了额外的tRNA基因。与传统注释方法相比,Geseq具有显著的优势,因为它使用了更多的novo预测器和通过轮廓隐马尔科夫模型(HMM)进行搜索。此外,该工具还在叶绿体基因中发现了更多的内含子[12.].{Tillich, 2017 #2}参考文献13。
功能适应oryza.有人认为是叶绿体。在驯化水稻中发现的光合作用基因psb B具有多态性,与水稻对遮荫和日照的适应有关oryza.在不同生境生长的物种[23.].其他表现出导致蛋白质序列改变的叶绿体基因与基因表达有关;即RNA聚合酶基因(rpoC2)的2个变化和核糖体蛋白(rpl20)的差异。两个叶绿体分支在tRNA序列上也表现出一致的差异。在psb B基因中存在的丙氨酸/缬氨酸多态性表明在两个叶绿体谱系中存在平行适应的可能性。这些差异对水稻在不同环境中的表现的影响值得仔细评估,因为可能使用了来自野生亲缘植物的叶绿体基因组[24.].通过选择母体基因组进行水稻遗传改良的潜力需要仔细分析。这将由于具有接近相同的核基因组和不同的母体基因组的水稻基因型的可用性而变得容易。
结论
本研究表明,水稻遗传学不是一种简单的二分法,具有籼稻和粳稻统治,而是两种不同母体基因组的栓塞,以及来自野生种群的许多基因的血栓增生。这表明驯养的稻米有很多“父亲”,而只有两个“母亲(或母亲群体)”。
进化的演变oryza.属[25.[展示分歧,迟滞和自然和人类选择的模式[26.使得细胞质基因组和核基因组都能够独立进化,特别是对于有可能进行叶绿体转移的近缘类群。由3091个基因型的序列提供的丰富数据集[10.]已被证明是水稻生物学的重要资源,并已成功用于其他研究[27.].
稻育植物未侧重于选择母体基因组。本研究表明,对不同核基因组的不同叶绿体基因组(驯化水稻和其他来自野生种群的两种类型)的分析可能是必要的,以确定叶绿体基因组选择的可能性在不同环境中提高水稻性能的可能性。母体基因组可能影响影响适应特定环境的特征,这可以确定特定区域中的作物性能。鉴于大米作为粮食问题的重要性,这种可能性需要仔细评估。
方法
3018个驯化水稻品种的下一代序列数据来源于NCBI数据库[28.].叶绿体基因组测序和组装的方法已在早期的研究中详细介绍[5].Moner et al.(2018)提供了叶绿体基因组测序和组装的详细讨论[5].通过与水稻叶绿体参考基因组序列对准获得完全叶绿体基因组序列。通过映射的序列组件被用作大量密切相关(驯化)基因组的唯一实用方法,而在早期的研究中,用更加多样化的基因组映射,并且已经手动和解基于Novo的方法以产生基因组序列。对原始读取序列进行质量检查并修剪以获得高质量的读取,使用BWA-MEM版本0.7.13映射到叶绿体参考序列NC_001320.1的修剪读取。29.FastQC工具配合fastqc - o进行整体质量控制,然后cutadapt -m x -q 20用于修剪),通过使用GATK 3.6版进一步的映射回合精制[30.使用BWA-MEM (release 0.7.10)将Reads与Nipponbare参考序列对齐。使用GATK RealignerTargetCreator和IndelRealigner包(3.2-2版本)对IndelRealigner周围的reads进行系统发育分析[9,22.].
3091个样品的叶绿体映射数据在55×(最低样品中,88,478读数为100bp的最低样品)和10,863 x(最高样本,17,382,581读,100bp读数),以确保准确地呼叫共识序列每个样品的叶绿体。所有样品的平均覆盖率为2215 x。
包括澳大利亚和亚洲野生米兰叶绿体序列,并将来自Oryza Officinalis的序列用作out组(表1).使用mafft [叶片序列31使用以下设置的工具(Auto, 1PAM/K = 2评分矩阵,1.53缺口惩罚和0.123偏移值)。为了进行系统发育分析,采用了两种策略:将叶绿体序列、野生近缘和外类群排列在一起,并根据亚种划分类群,而不考虑原产国。系统发育树构建采用Geneious V 9.1,采用neighbor -Joining方法和Tamura-Nei遗传距离模型。注释的叶绿体序列的Fasta序列o .漂白亚麻纤维卷使用网络工具GeSeq对Nipponbare (NC_001320)进行分析,使用默认参数注释普通序列:蛋白质搜索标识=25,rRNA, tRNA, DNA搜索标识=85。该工具在生成高质量叶绿体注释方面的效用最近已得到证实。然后将它们上传到OGDRAW,以可视化叶绿体基因组。将带注释的文件导入Geneious,与原始带注释的文件进行比较。通过对注释表的基因比较,用基因手工评估差异。
数据和材料的可用性
所有原始数据都可以从NCBI数据库中获取[1].本研究中产生的叶绿体基因组数据集可在以下情况下获得:DOIhttps://doi.org/10.6084/m9.figshare.13040462.
参考文献
- 1。
程L等人。粳稻与籼稻叶绿体基因组差异选择的特征。米饭。2019; 12:65。https://doi.org/10.1186/s12284-019-0322-x.
- 2。
matsumoto t等人。基于地图的水稻基因组序列。自然。2005; 436:793-800。https://doi.org/10.1038/nature03895.
- 3.
nock cj等。来自总DNA的叶绿体基因组序列用于植物鉴定。植物Biotechnol J1111; 9:328-33。https://doi.org/10.1111/j.1467-7652.2010.00558.x.
- 4.
Wambugu PW, Nyamongo D, Ndjiondjop MN, Henry RJ。稻属物种间的进化关系。Compend Pl Genome. 2018:41-54。https://doi.org/10.1007/978-3-319-71997-9_3.
- 5.
Moner AM, Furtado A, Henry RJ。水稻AA基因组种的叶绿体系统地理学。分子系统学进展。2018。https://doi.org/10.1016/j.ympev.2018.05.002.
- 6.
Wambugu PW, Brozynska M, Furtado A, Waters DL, Henry RJ。基于叶绿体全序列的野生稻和驯化稻(Oryza AA基因组种)的亲缘关系Sci众议员2015;5:13957。https://doi.org/10.1038/srep13957.
- 7。
亨利rj。澳大利亚野生稻种群:全球粮食安全的关键资源。前植物SCI。2019; 10:Artn 1354。https://doi.org/10.3389/fpls.2019.01354.
- 8。
亨利·罗等。澳大利亚奥雅萨:效用和保护。米饭。2009; 3:235-41。
- 9。
陈志强,等。澳大利亚野生稻基因组测序揭示了与驯化稻的祖先关系。植物生物技术学报2017;15:765-74。https://doi.org/10.1111/pbi.12674.
- 10。
Rellosa MC,等人。3,000个水稻基因组项目。傻瓜。2014; 3:artn 7。https://doi.org/10.1186/2047-217x-3-7.
- 11.
引用本文:王志强,王志强,等。叶绿体基因组注释工具的评价及其在咖啡物种进化分析中的应用。公共科学图书馆。2019;14:ARTN e0216347。https://doi.org/10.1371/journal.pone.0216347.
- 12.
等。GeSeq -细胞器基因组的通用和准确的注释。核酸学报2017;45:W6-W11。https://doi.org/10.1093/nar/gkx391.
- 13。
曾旭,等。青稞在西藏的起源与演变。Nat Commun。2018;9。https://doi.org/10.1038/S41467-018-07920-5.
- 14.
增加的分类单元采样大大减少了系统发育误差。系统医学杂志。2002;51:588 - 98。https://doi.org/10.1080/10635150290102339..
- 15.
Civan P, Craig H, Cox CJ, Brown TA。三种地理上独立的亚洲水稻驯化。Nat Plants. 2015;1:Artn 15164。https://doi.org/10.1038/Nplants.2015.164.
- 16.
蔡继勇,等。水稻悖论:亚洲水稻的多重起源但单一驯化。生物学杂志。2017;34:969-79。https://doi.org/10.1093/molbev/msx049.
- 17.
等。杂交后代中观察到的分离畸变栽培稻和O. Meridionalis.种子发展过程中的堕胎引起。植物(巴塞尔)。2019; 8。https://doi.org/10.3390/plants8100398.
- 18。
黄X等人。水稻基因组变异的地图揭示了栽培水稻的起源。自然。2012; 490:497-501。https://doi.org/10.1038/nature11532.
- 19。
Moner Am,Henry RJ。Oryza Meridionalis NQNG。Compend Pl基因组。2018:177-82。https://doi.org/10.1007/978-3-319-71997-9_16.
- 20。
等。澳大利亚一年生野生稻与两种相关多年生稻的分子关系。2013;6:26(2013年10月28日)。
- 21。
陈志强,等。澳大利亚北部鉴定新型水稻种质的叶绿体基因组。Trop Plant Biol。2014; 7:111-20。https://doi.org/10.1007/S12042-014-9142-8.
- 22。
等。澳大利亚水稻祖先的多样性与进化。生态另一个星球。2018;8:4360-6。https://doi.org/10.1002/ece3.3989..
- 23。
高LZ,等。羚羊叶绿体基因组的演变促进了各种生态栖息地的适应。Communic Biol。2019; 2:套头衫278。https://doi.org/10.1038/s42003-019-0531-2.
- 24。
小杰,等。来自野生水稻的基因提高产量。自然。1996年; 384:223-4。https://doi.org/10.1038/384223a0.
- 25。
Stein JC等人。13个驯养和野生稻豚的基因组突出了矿物质属的遗传保护,周转和创新。NAT Genet。2018; 50:285- +。https://doi.org/10.1038/s41588-018-0040-0..
- 26.
Krishnan SG, Waters DLE, Henry RJ。澳大利亚野生稻揭示了水稻基因组多态性沙漠的驯化前起源。《公共科学图书馆•综合》。2014;9:e98843。https://doi.org/10.1371/journal.pone.0098843.
- 27.
Carpentier MC,等。亚洲稻米的回收展示展示由3000个基因组透露。NAT Communce。2019; 10:artn 24。https://doi.org/10.1038/S41467-018-07974-5..
- 28.
李景云,王杰,齐格勒等。水稻基因组计划:未来水稻研究的新机遇和挑战。Gigascience。2014;3:Artn 8。https://doi.org/10.1186/2047-217x-3-8.
- 29.
李H.探索单样本SNP和Indel与全基因组De Novo集会呼叫。生物信息学。2012; 28:1838-44。https://doi.org/10.1093/bioinformatics/bts280.
- 30.
麦肯纳等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20:1297 - 303。https://doi.org/10.1101/gr.107524.110.
- 31。
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30:772-80。https://doi.org/10.1093/molbev/mst010.
确认
我们感谢昆士兰大学的研究计算机中心,以支持和咨询和澳大利亚的资金研究理事会。
资金
这项研究得到了澳大利亚研究委员会的支持。资助机构在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有作用。
作者信息
隶属关系
贡献
AM, AF和RH对研究的构思和规划做出了贡献。我分析了数据。AM, AF和RH参与了手稿的编写。所有作者均已阅读并批准本稿件。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
在驯化水稻基因库中捕获的两个不同的叶绿体基因组序列可能对水稻生产具有重要意义。BMC植物杂志20,472(2020)。https://doi.org/10.1186/s12870-020-02689-6
收到:
接受:
发表:
关键字
- 大米
- 叶绿体
- 序列
- 驯化