抽象的
背景
属当归由大约60种分布在北半球分布的物种组成。它是Apiaceae中最划分的征征困难的困难之一,主要是由于形态学特征多样。探讨塑性进化和系统发育关系当归,我们确定了八个完整的塑料序列当归物种使用de novo装配方式。
结果
通过综合比较分析,我们发现8个质粒在重复序列、SSR、密码子使用和RNA编辑位点等方面都是相似的。然而,与其他7种相比,L. Delavayi.在基因组大小,基因数,IR / SC边界和序列标识中表现出显着差异。大多数基因仍然在纯化选择下,而四个基因显示放松选择,即CCSA那RPOA.那YCF1, 和YCF2.. 非单系当归从塑料和内转录的间隔物(其)序列系统序列系统发育分析中推断物种。
结论
质体树和ITS树产生不一致的树拓扑,这可能是由于杂交和不完全谱系排序。我们的研究强调了具有大量信息位点的质体在解决系统发育关系方面的优势。此外,结合前人的研究,我们认为目前的分类体系当归需要改进和修改。总之,我们的研究提供了新的洞察塑料进化,系统发育和分类当归物种。
背景
属当归,属于大约60种物种的家庭苹果藻,分布在亚洲,欧洲和北美[1].它有两个分销中心:在喜马拉雅山,另一个在北美[2].我国有该属40种(35种),主要分布在高寒地区[1].
当归是苹果植物中最分散的困难的属之一,主要是由于鲜花,叶子,苞片和mericarps的不同形态特征,这使得难以区分其邻居[1那3.那4.].到目前为止,与附近属的系统发育关系尚不清楚,如Ligutcopsis.那蒂格兰那CNIDIUM.那Selinum.那Hymenidium.那pachypleum.那卢比希拉, 和paraligusticum[1特别是合并蒂格兰和Ligutcopsis.进入当归仍有争议[2].诊断特征蒂格兰是不同的花萼牙齿和携带的mericarp在每个沟槽中轴承vitta [5.,并没有把它和当归.因此,蒂格兰被转移到当归[6.那7.].leute [3.]分开Ligutcopsis.从当归根据突出的萼齿。有些人不支持这种疗法当归物种也有这个特征[2].传统方法以区分这些物种是基于它们的形态特征,而许多上述物种总是表现出类似的特征,导致极度困难的物种分类和通用划界[1那3.那4.].以前,已经使用少数分子标记物研究系统发育当归,如核糖体DNA内部转录间隔区(ITS),质体DNA16卢比那RPS16, 和rpoc1.内含子[4.那8.那9.那10.那11.那12.那13.那14.]但DNA片段未能识别当归作为一个单系群。其中,唐妮等人。[12.]确定了五个片状当归,包括亚焦思工,Conioselinum Chinense.Clade,Pyramidoptereae,Selineae,和Sinodielsia.思工。周等人。[4.]随后划分了属当归进入六分枝和东亚(photoospermopsis.)加入了思克。可以看出属的属当归面临分类和系统发育的一大挑战。因此,重建系统发育关系并重新评估通用限制需要更多的基因组资源当归.
此外,许多当归是珍贵的传统药材,具有很好的药用价值。例如,植物的根茎和根L. jeholense.或者l . sinense被用作名为gao-ben的中医,已被广泛用于治疗感冒,头痛,被困风和风湿关节痛[15.].结果,该草药已经进行了许多关于生物活性,化学成分或药理学的研究[16.那17.].尽管药用价值优异,但缺乏基因组资源,并且物种认证是困难的。因此,必须通过对物种认证的比较塑性方法进行更多的DNA条形码,以确保药物质量。
Plastid是绿色植物的关键细胞器,参与光合作用过程,为植物提供必要的能量[18.].体液基因组(塑料)是在大多数植物中为115至165kb的双链分子[19.].塑性的结构组织,基因布置和基因含量相对节省。典型的塑料含有大的单拷贝(LSC)区域为82-90 kB,小单拷贝(SSC)区域为15-20 kB,两个倒置重复(IRS)区域为22-25 kB [19.].它总是编码110-130个不同的基因,包括蛋白质编码基因(〜80),转移RNA(TRNA)基因(〜30),以及核糖体RNA(RRNA)基因(4)[20.].此外,它通常是装载量的遗传,并且具有低核苷酸替代率[21.].由于这些原因,塑料已成为不同分类水平的植物系统发育研究的工具[22.那23.那24.那25.].目前,体积体内介体分析当归尚未报告。同时,下一代测序和生物信息学技术的持续发展使得比以往任何时候都更便宜,更快地获得完整的塑料序列。因此,我们更愿意使用塑料来推断系统发育关系当归.
在这里,我们新测序了八个塑料当归物种。为了综合了解系统发育关系,我们还使用核的序列来构建系统发育树。我们的目标是(1)推断出塑料的演变当归;(2)为开发候选DNA条形码提供更多基因组资源;(3)试验塑料是否增加了传统DNA标记的分辨率;(4)作为该属的后续文学组织研究的参考。总体而言,这里报道的完整塑料将促进塑料进化,系统发育和分类学研究当归.
结果
特点当归质体系
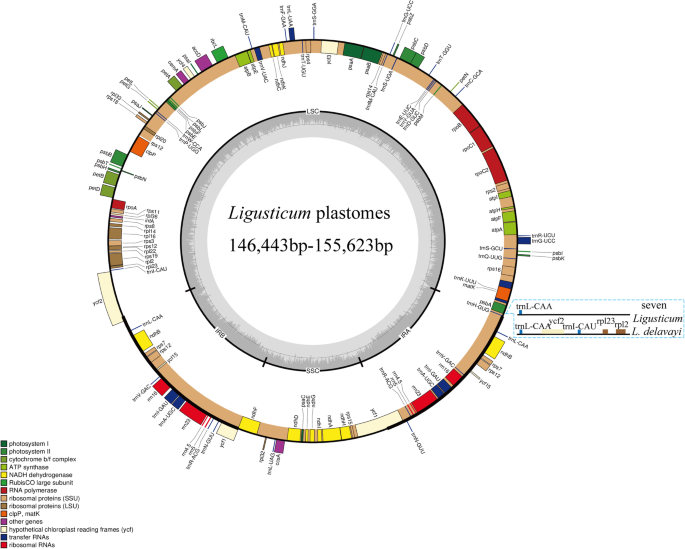
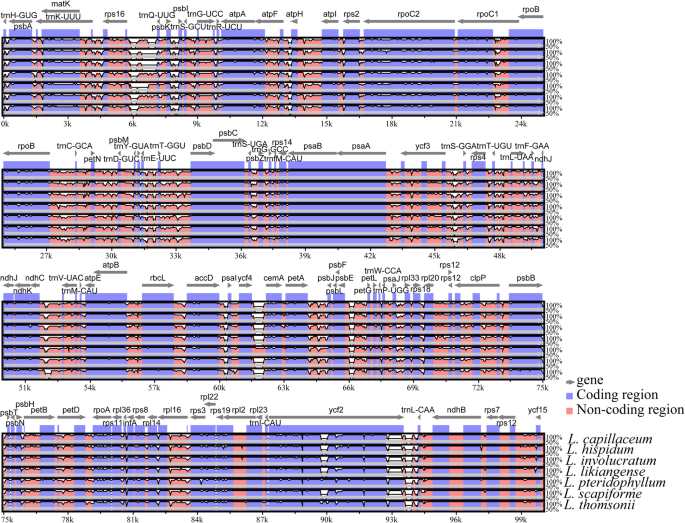
质量控制后,5.76 GB(L. scapiforme.)至7.47 Gb(L. Delavayi.)为八个生成清洁读数当归物种(表1),通过从头组装得到8条完整的质体序列。确定了8个细胞体的完整序列当归种类范围为146443种 bp输入L. Pteridophyllum.155,623 bpL. Delavayi.(桌子1).与大多数有原植物相比,所有这些都在结构方面受到高度保守的,与IR区(18,166-26,908bp),SSC区(16,741-17,591bp)和LSC地区(85,066-93,363bp)分享典型的四胞胎结构。总GC含量在37.3-37.6%之间,而红外地区比LSC(35.7-36.0%)和SSC(30.9-31.2%)地区(表格)更高(42.5-44.8%)(表1).8个质体共含有129 ~ 133个基因,其中85 ~ 88个蛋白编码基因,36 ~ 37个tRNA基因,8个rRNA基因(图1)。1, 桌子1,附加文件2:表S1)。L. Delavayi.含有另外四个基因(YCF2.那rpl23.那rpl2., 和TRNI-CAU.)超过七个当归IRA的物种(图。1,附加文件2:表S1)。这个RPS12.基因分别用5'end致剪液,分别在LSC和IR区域中重复3'end(图。1).这trnk-uuu.拥有最长的内含子(2485-2543 BP)matK基因(图。1).四个rRNA的GC含量(RRN16.那RRN23那RRN4.5, 和rrn5)基因高(55.1-55.3%)(图。1).
密码子使用和RNA编辑网站
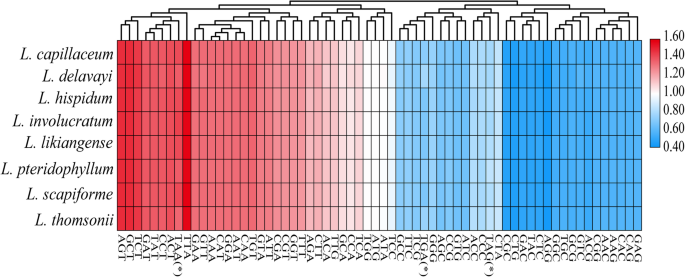
用于密码子分析的蛋白质编码基因的总序列尺寸为八个67,905-68,268英镑当归质体系。这些蛋白质序列编码了22,635-22,756密码子,这些密码子总结在附加文件中3.:表S2。Leu被最高数量的密码子(2382-2419)编码,而Cys是最少(232-241)。热图形式的所有密码子的RSCU值如图4所示。2.红色值表示较高的RSCU值,蓝色值表示较低的RSCU值。热线图表明,大约一半的密码子更频繁地使用。具体地,通常使用RSCO> 1使用30个密码子,并且除了TTG之外的嘌呤(A / T)结束的所有偏置密码子(图。2,附加文件3.:表S2)。第一,第二和第三密码子位置的GC含量的平均值分别为46.0,38.3和29.7%(附加文件4.:表S3)。这一GC含量也表明当归在第三密码子位置有很强的a /T偏倚。两个密码子(ATG和TGG)的使用无偏性(RSCU = 1)(附加文件3.:表S2)。
另外,在8个基因中,有35个基因存在潜在的RNA编辑位点当归质体系。鉴定了共469个RNA编辑部位,其中编辑部位的数量范围为55(L. Delavayi.)至67(L. LIKIANZE.)(额外的文件5.:表S4)。这NDHB.基因在所有八个中有最多的RNA编辑位点(10)当归塑料,而RPS8.基因中也有10个RNA编辑位点L. LIKIANZE..All of the identified RNA editing sites were Cytosine to Uracil (C-U) conversion and most of them were situated in the second codon position (40–51), followed by the first codon position (12–16), but no sites situated in the third codon position (Additional file1:图S1)。氨基酸转化丝氨酸至亮氨酸(S-L)最常发生。此外,质量的RNA编辑位点(420)导致疏水产品的氨基酸变化,例如亮氨酸(L; 219),苯丙氨酸(F; 63),异亮氨酸(I; 58),酪氨酸(Y; 25),蛋氨酸(m; 21),色氨酸(w; 17),缬氨酸(v; 17)(附加文件5.:表S4)。
重复元素分析
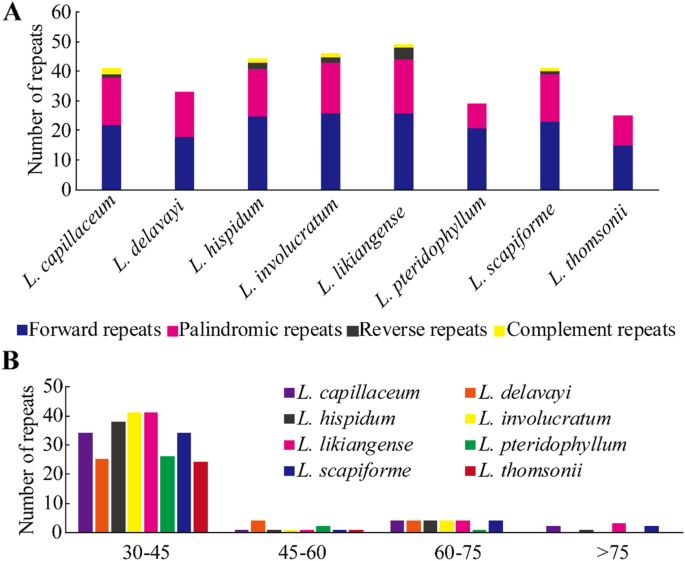
在八个中检测到前方,回文,反向和互补重复当归质体系。总而言之,我们检测到308重复30-82 bp长(附加文件6.:表S5)。向前重复(176)的数量高于回文重复(116),反复重复(10)和回文重复(6)。L. LIKIANZE.含有最多的重复(49),而L. Thomsonii.所包含的最小(25)(图。3.). 根据长度,我们人为地将重复分为四类:30-45 血压,45–60 血压,60–75 英国石油公司,以及 > 75 血压(图。3.).其中,大部分重复(85%)长30-45磅。大部分重复位于非基因或内部区域(70.5%),少数群体位于基因区(29.5%)。检测到603简单的序列重复(SSR),但SSR的数量在八个之间不同当归物种(附加文件)1:图S2,附加文件7.:表S6)。L. scapiforme.包含最多的SSR(82),而L. Delavayi.和L. Hispidum.包含至少(68)。最丰富的单核苷酸重复(54.9%),其次是二核苷酸(25.5%),四核苷酸(12.3%),三核苷酸(3.0%)和五核苷酸(3.0%)。六核苷酸在塑料中非常罕见。SSRS主要分布在LSC(68.3%),IRS较少(16.6%)和SSC(15.1%)(附加档案7.:表S6)。
边界和序列标识的比较
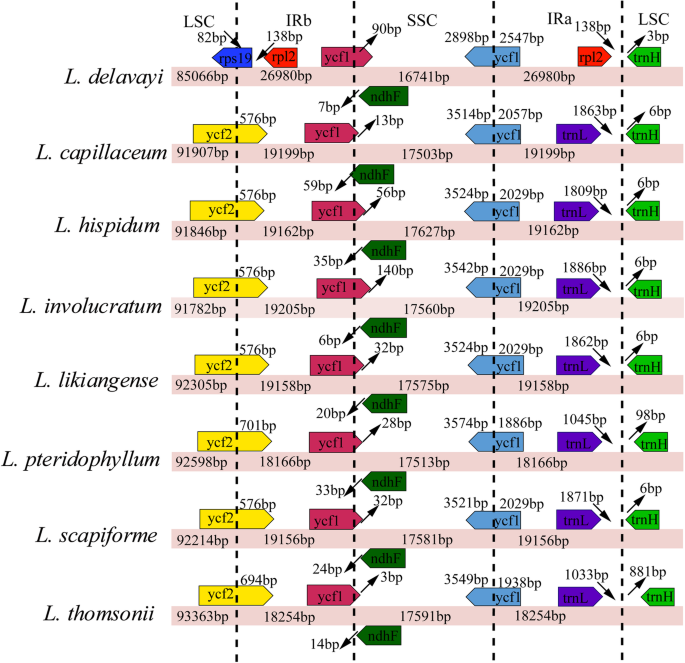
八个中反转重复和单拷贝(IR / SC)边界之间的差异当归检查塑料(图。4.).除了L. Delavayi.,其他七个当归在IR / SC边界处的基因令和基因含量方面被保护。为了L. Delavayi.,LSC / IRB边界是RPS19/rpl2.基因和IRA / LSC边界是rpl2./trnh.基因。另一个呢当归物种,LSC / IRB边界延长了576-701 BP进入了YCF2.基因。SSC / IRB边界延长了3-140 bp进入了YCF1基因,最小和最大的延伸发生L. Thomsonii.(3 bp)和L.劳动力(140 bp)。这NDHF.基因L. Capillaceum.通过59 BP与SSC / IRB边界重叠。这YCF1基因,穿过SSC / IRA边界,位于SSC和IRA区域,3514-3574 BP和1886-2057 BP。这TRNL.和trnh.基因距离IRA / LSC边界1033-1871 bp和6-881bp。
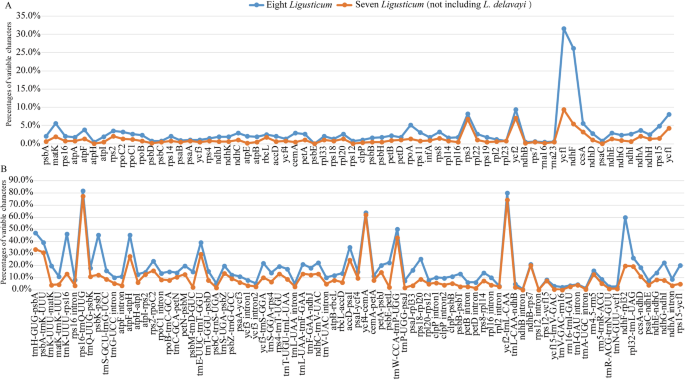
MVISTA程序用于使用序列同一性分析L. Delavayi.作为参考。结果如图所示。5.其中编码区比非编码区具有更高的序列保守性。检测到766个SNPs和351个Indels当归塑料(附加文件8.:表S7)。大多数SNP和Indels(786)来自非编码区,而少数群体(331)来自编码区。我们还确定了149个区域的平均变化百分比(66个编码区,64个代亚基间隔物和19内含子)(图。6.,附加文件9.:表S8)。在这些区域中,非编码区(18.5%)的平均百分比高于编码区域的(3.2%)(附加文件9.:表S8)。十二个非编码区表现出高变异:trnh-gug / psba那PSBA / TRNK-UUU那TRNK-UUU / RPS16那RPS16 / TRNQ-UUG那PSBK / PSBI.那ATPF / Al..那trnE UUC/trnT GGU公司那ACCD / PSAI.那YCF4 / CEMA.那trnw-cca / trnp-ugg那YCF2 / TRNL-CAA, 和ndhF/rpl32型(变异百分比>为30%)。8个编码区表现出高变异:matK那rps3.那YCF2.那YCF1 × 2.NDHF.那RPOA., 和CCSA(变异的百分比> 5%)。七个其他当归物种显示序列差异L. Delavayi.是MVISTA图中的参考(图。5.).因此,我们计算了编码和非编码区域的可变字符的百分比当归物种(不包括L. Delavayi.),以及遗传距离的八当归质体。两两遗传距离为0.0010~0.0239,总平均值为0.0092(另文)10.:表S9)。但是,两者之间的成对遗传距离的值L. Delavayi.和七个其他当归物种更高:0.0234(L. Capillaceum.),0.0236(L. scapiforme.), 0.0234 (L. LIKIANZE.),0.0237(L. Hispidum.),0.0239(L.劳动力), 0.0221 (L. Pteridophyllum.)和0.0232(L. Thomsonii.).数字6.表明七个其他序列差异当归物种没有L. Delavayi..总之,我们的结果表明L. Delavayi.显示比其他七个更高的序列差异当归物种(图。6.,附加文件10.:表S9)。
塑性基因中的选择性压力
计算79个常见蛋白质编码基因的DN / DS比以估计选择性压力(附加文件11.:表S10)。我们的结果中最多基因的DN / DS比例小于0.5,表明它们在纯化选择下。尽管如此,我们还检测到DN / DS的增加,表明九个基因中的放松选择(0.5
系统发育关系
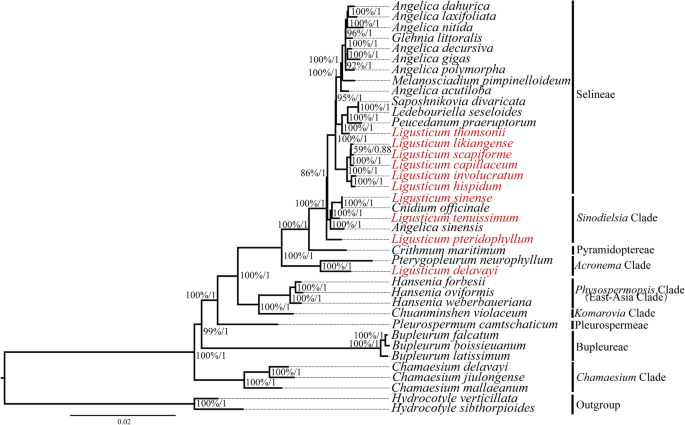
39种完全的塑料和80个核,其序列用于进行系统发育分析(附加文件12.:表S11)。塑料树及其树木产生了不一致的树拓扑,而他们都推断出非单层当归物种(图。7.,附加文件1:图S3)。在塑料树中,L. Capillaceum.那L. scapiforme.那L. LIKIANZE.那L. Hispidum.那L.劳动力, 和L. Thomsonii.属于Selineae。但是,五个其他当归物种形成了一个疏水板,L. Thomsonii.聚集S. Divaricata.那L. Seseloides., 和p . praeruptorum.L. Tenuissimum.和l . sinense曾经属于Sinodielsia.思工,但他们没有形成一个思想家。l . sinense更密切相关C药用植物,然后他们聚集了L. Tenuissimum..L. Delavayi.始终聚集在一起P. neurophyllum, 曾经属于Acronrma思工。L. Pteridophyllum.曾经属于Sinodielsia.思工[4.],虽然它被视为姐妹Sinodielsia.Clade + Selineae。在它的树上,L. Capillaceum.那L. scapiforme.那L. LIKIANZE.那L. Hispidum.那L.劳动力, 和L. Thomsonii.形成了一种疏枝,属于Selineae。L. Tenuissimum.与弱者的Selineae一起解决了(BS = 54%,PP = 0.8)。l . sinense仍然更密切相关C药用植物.的系统地位L. Delavayi.符合塑料树。由ML和BI分析产生的树拓态有些不同。例如,L. Pteridophyllum.聚集l . sinense和C药用植物通过BI分析(PP = 0.57),而这是平行分支的关系l . sinense和C药用植物通过M1分析(BB = 57%)。在Selineae,六当归七当归属于这个部落。就像当归那当归也显示出多重,M. Pimpinelloideum.和G. Littoralis.嵌入其中。Chamaesium.思工是Apioideae的基底分类,而不是Bupleureae。Sinodielsia.思克不是我们的系统发育分析中的单次群体。
讨论
质体基因组进化
质体在基因组结构、基因顺序和基因含量上高度保守[19.那21.那26.].然而,基因组重新排列,基因丧失(伪析化或缺失),结构和尺寸的差异,IR的差异已经证实在塑性进化期间发生了多次[20.那27.那28.那29.那30.].在这项研究中,当归塑料是低保守,在基因组大小(146,443-155,623bp),基因数(129-133)和Ir / Sc边界方面表现出惊显差异。L. Delavayi.质体长度最长,而LSC和SSC区域较其他7个区域短当归物种。长度的差异反映了IRS的扩展和两个SC的收缩。它有四种基因(YCF2.那rpl23.那rpl2., 和TRNI-CAU.)超过七个当归IRA中的物种可能归因于IRS的延伸产生的基因重复。在其他高血管植物血浆中发现了对等内容中的各种基因数[31.那32.].为了L. Delavayi.,IRA / LSC边界是rpl2./trnh.LSC/IRb边界为RPS19/rpl2.基因与七个不同的基因当归物种 (TRNL./trnh.和YCF2.).伊拉区域延伸到YCF1基因是塑性进化中的常见特征[31.那33.那34.].IR区域GC含量较高,为42.5 ~ 44.8%,可能是由于4个rRNA基因GC含量较高[34.].
ssr已广泛应用于植物群体遗传学和进化研究[35.那36.].最丰富的SSR是八个中的单核苷酸当归塑料,其次是二核苷酸,四核苷酸,三核苷酸,戊核苷酸和六核苷酸重复。这种现象已经报告樱草[31.] 和葱属植物[37.].LSC中最大SSRS的最可能解释是LSC比SSC和IRS长。大多数SSRS包含A / T主题,导致整体塑料的丰富性[34.].这里报道的CPSSR是开发遗传多样性研究的分子标记的信息来源当归物种。
AT含量较高的密码子通常用于质体,而a /T在第三密码子位置使用的趋势更为明显[38.].偏见也显示在八个当归质体系。亮氨酸由最高数量的密码子编码,密码子偏好的顺序是TTA> CTT> TTG> CTA> CTC> CTG,其在大多数Geraniaceae物种之后[20.].RNA编辑是调节植物细胞器中术后术语的基因表达的重要过程[39.].除了陆地植物的所有主要谱系中,事件发生在Marchantia polymorpha还有一些绿藻[40].RNA编辑可以在RNA水平校正DNA突变,从而恢复保守的氨基酸残基以维持编码蛋白的功能[41.那42.那43.].大多数编辑站点发生在第二密码子位置,并且在第三密码子位置发生任何网站,也发现了分布模式连翘吊索[44.].像许多其他植物一样NDHB.基因具有最多的RNA编辑位点[40那45.],这表明了NDHB.基因对调节植物生理和生化过程至关重要。
大重复序列被认为是促进塑料重排和序列分歧的主要原因[46.那47.那48.].在鉴定的308重复中,最短重复(85.4%)是最多的,这与许多不附加的塑性相一致[49.那50]. 非编码区的SNPs和Indels分布较多,平均变异率高于编码区。因此,我们的研究表明非编码区比编码区保守。重复序列的分布与突变事件有关,重复序列可能在诱导突变中起作用[30.那51那52].我们的结果还表明,重复分布与突变事件相关,对于主要位于高度可变的非编码区(70.5%)而不是编码区(29.5%)。DNA条形码定义为短DNA序列,具有足够的变化,以鉴定给定的分类学基团中的物种[53].已经描述了八个编码区和具有最高百分比变异百分比的12个非编码区。其中,几个区域已经在其他高昂的植物中确定,例如matK那NDHF.那rps3.那YCF2.那YCF1 × 2.RPOA.那trnh-gug / psba那ndhF/rpl32型那TRNK-UUU / RPS16那PSBK / PSBI.那YCF4 / CEMA.那ACCD / PSAI.那YCF2 / TRNL-CAA那RPS16 / TRNQ-UUG, 和trnE UUC/trnT GGU公司[22.那49.那50那54那55那56].用于草药药物当归,这些地区可以作为候选DNA条形码,用于物种认证,以确保药品质量。
我们估计了79个常见蛋白质编码基因的选择性压力当归质体系。其中大部分为纯化选择,反映了植物质体基因的典型进化保护[57那58].四个基因(YCF1,YCF2那CCSA, 和RPOA.)在放松的选择下。这YCF1和YCF2.在许多事先作品中证明了塑料中最大和第二大基因的基因,已被证明是不存在的或假期化[21.那59].在两种基因上也被观察到了放松的选择Corallorhiza Striata那Lennoa Madreporoides,和Pholisma arenarium.[60那61].基因RPOA.和CCSA编码RNA聚合酶的α亚基和血红素附着到C型细胞色素所需的蛋白质[62那63].他们通常存在于陆地植物中,而它们缺席的塑料Physcomitrella patens.[63].然而,寄生植物的塑料通常是研究轻松选择对光合功能的影响的最佳模型系统[64那65].实际上,一些寄生植物港口大大降低了塑性大小和基因含量,由放松的选择与光合作用相关基因进行了放松[60那64那66].因此,进一步的研究是研究轻松选择的重要作用当归塑性基因。总的来说,这些发现在塑植基因上缩小了新的灯当归物种。
系统发育关系
到目前为止,基于少数分子标记的分子系统发育研究不支持单独的当归物种 [4.那8.那9.那10.那11.那12.那13.那14.].在这里,我们进行了系统发育分析当归使用完整的塑性物及其序列。不幸的是,我们仍未认识到当归作为一个单系群。质体树和ITS树产生不一致的树拓扑。L. Capillaceum.那L. scapiforme.那L. LIKIANZE.那L. Hispidum., 和L.劳动力具有相似的形态特征(如小苞片羽状和茎基部被纤维残鞘覆盖),并在质体树中形成枝支(BS = 100%, PP = 1)。然而,L. Thomsonii.聚集在上述五种物种中,形成其树的疏水板。L. Tenuissimum.曾经属于Sinodielsia.这与Zhou等人的研究结果一致。4.],而它被确定为Selineae的姐妹,其树中的弱载体(BS = 54%,PP = 0.8)。在其他植物谱系中通常观察到核和塑性体积学之间的不统一[67那68那69].这种不协调可能是其不同继承的背景和其血浆DNA的突变率的结果[70那71].核ITS是双亲遗传的,突变率较高,而质体DNA是母体遗传的,突变率较低[70那71].此外,杂交和不完全分选(ILS)可以负责其基于塑性和塑性的文学之间的不一致关系[72那73].l . sinense更密切相关C药用植物,这可以用基因组交叉杂交很好地解释[74].L. Pteridophyllum.聚集l . sinense和C药用植物在它的树中通过BI分析,这与周等人的结果一致。[4.].这些都表明当归物种可能经历过复杂的进化史。多重的当归,以及格里尼亚和Melanosciadium嵌入其中,已被早期的研究记录过[12.那14.那75].Sinodielsia.在其他工作中未观察到的单胞胎,疏浚不会被恢复[14.].Chamaesium.作为近期基于3351个单拷贝基因的研究,思工是APIOIDEAE的基本纳税群[76].
塑料树得到中等至高的支撑,相反地,其树得到了较低的支撑和更平行的分支。因此,我们的结果突出了塑料与质量信息位点来解决解析系统发育关系的优势。本研究也是第一个支持多重的当归基于质体系。进一步的研究包括更大的分类单元取样是必要的,以确定多系的位置当归.此外,结合以往的研究[4.],我们认为目前的分类系统当归需要改进和修改。一词,我们的研究为未来的系统发育,分类和进化历史研究提供了有用的信息当归物种。
方法
分类群抽样和DNA提取
从8种植物的成株上采集新鲜绿叶,然后立即用硅胶干燥,进行下一步。对这些植物取样不需要许可,因为它们不是重点保护植物。采用改良CTAB法从硅片干叶中提取总基因组DNA[77].兴金河(四川大学)开展了植物材料的正式鉴定。凭证标本沉积在四川大学(中国成都)的植物管中(附加档案13.:表S12)。对于ITS分析,我们新测序的17个ITS已提交到NCBI(登录号:MT974009-MT974025)(附加文件12.:表S11)。
基因组测序,装配和注释
八个新序列物种的原始读数是从诺二烯(天津)的Illumina Hiseq x十平台(Cired-End,150 bp)产生的。使用FastP V30.15.0(-N 10和-Q 15)进行原始读取的质量控制[78,对每个物种产生至少5GB的干净读取。然后使用NOVOPlasty v2.6.2进行从头组装[79]带有默认参数。种子序列是rbcl.基因的参照基因组序列L. Tenuissimum.(NC_029394)。程序DOGMA [80]对8个质体进行基因注释,并根据其同属物种的比较,在geneous v9.0.2 (Biomatters Ltd., Auckland, New Zealand)中进行手动调整。所有新生成的8个完整的质体都在NCBI中(登录号:MT409612-MT409619)(表1)1). 使用在线程序OrganellarGenomeDRAW(OGDRAW)绘制圆形塑料体图[81].
密码子,RNA编辑网站和重复序列
从这8个基因中提取蛋白质编码基因当归塑料用于密码子分析。除去所有重叠基因,最终数据集包括每种物种的80个蛋白质编码基因。密码子使用和相对同义密码子使用(RSCU)[82值使用CodonW v1.4.2程序计算[83].来自八个塑料的所有RSCU的热线映射使用TBTOOLS产生[84].蛋白质编码基因的基础组合物通过MEGA6计算[85].植物套件的在线程序预测RNA编辑器[86使用0.8的截止值来预测潜在的RNA编辑位点。
在线声示程序[87用于鉴定重复序列,包括前进,回文,反向和互补重复。根据以下参数:(1)重复大小超过30 bp;(2)两次重复之间超过90%的序列标识;(3)汉明距离= 3。除去所有重叠重复序列。perl脚本misa(http://pgrc.ipk-gatersleben.de/misa//)用于利用简单的序列重复(SSR)。对于单次,二,三,四分之一,四核苷酸,将最小SSR的最小数量设定为10,5,4,3,3和3,分别。
序列分歧
八个全基因组对齐当归使用MVISTA产生并可视化塑性物[88)使用L. Dilavayi.作为参考。八当归塑料在毒血管v9.0.2(BioMatters Ltd.,奥克兰,新西兰)与Mafft v7.221对齐[89,随后,使用“Find variation /SNPs”对Indels和SNPs进行计数和定位。根据Zhang等人的方法计算编码区和非编码区可变字符的百分比[90].八个遗传距离当归用MEGA6计算质体[85].
选择性压力分析
对10种常见的79个蛋白质编码基因进行了选择压力分析当归物种(包括2个发表的塑料)。使用PAML4.9中的Codeml程序使用PAML4.9中的代码型模型(SEQTYPE = 1,型号= 0,NSSITE = 0,1,2,3,7,8)[91那92].密码子频率由F3×4模型确定。我们比较了三组:M0 VS M3,M1 VS M2和M7 VS M8检测所选站点。似然比测试(LRT)用于确认三组的质量。贝叶斯经验贝叶斯(BEB)分析用于统计识别后概率≥95%的选定地点。我们将基因分类为在阳性选择下的发展(DN / DS> 1.0),放松选择(0.5
系统发育分析
早期的分子系统研究确定了五个片状当归,包括亚焦思工,Conioselinum Chinense.Clade,Pyramidoptereae,Selineae,和Sinodielsia.思工[12.].最近,这个属当归分为六个支系,东亚(photoospermopsis.)添加了分支[4.].在这里,我们使用39种完全的塑料和80个核的序列来推断出系统发育关系当归.使用Mafft V7.221实现序列对准[89].然后手动检查并校正对齐的序列。最大可能性(ml)和贝叶斯推理(BI)方法用于推断系统发育关系。raxml v8.2.8 [94使用如建议的1000重复和Gtrgamma模型进行ML分析(见RAXML手册)。MRBAYES v3.2.7 [95]用于执行贝叶斯推断与最佳替代模型由ModelTest V3.7确定[96].BI分析中完整质体和ITS序列的选择模型分别为TVM + I + G和GTR + I + G。马尔可夫链蒙特卡罗(MCMC)算法运行了200万代,每100代采样一棵树。通过计算分裂频率的平均标准差(ASDSF)来确定MCMC收敛性,该收敛性低于0.01。前25%的树被丢弃作为老化树,共识树使用剩余的树生成。使用交互式生命树(iTOL)对ITS树进行可视化和编辑[97]:引导支持低于50%的节点崩溃。
结论
在本研究中,我们测定了8个完整的质体序列当归物种使用de novo装配方式。通过全面的比较分析,观察到与其他七种物种相比,L. Delavayi.在基因组大小,基因数,IR / SC边界和序列标识中表现出显着差异。我们进行了系统发育分析当归使用39个完整的塑料和80个核的序列,发现当归在以前的研究中并不是单系的。杂交和不完全的谱系分类可能是ITS-和基于质体的系统发育不一致的原因。系统发育分析强调了利用具有大量信息位点的质体组解决系统发育关系的优势。我们的研究丰富了植物质体的数据当归并作为该属的后续文学组织研究的参考。
可用性数据和材料
八个注释的塑料和新测序的17款已提交给NCBI(https://www.ncbi.nlm.nih.gov.),注册号分别为MT409612-MT409619和MT974009-MT974025。
缩写
- asdf:
-
分流频率的平均标准偏差
- BEB:
-
贝叶斯经验贝叶斯
- 双:
-
贝叶斯推断
- BP:
-
基对
- BS:
-
分支支持
- CD:
-
蛋白质编码序列
- CTAB:
-
十六烷基三甲基溴化物
- ILS:
-
不完整的谱系排序
- 红外:
-
倒置重复
- 它的:
-
内部转录的垫片
- LRT:
-
可能性比率测试
- LSC:
-
大单拷贝
- MCMC:
-
马尔可夫链蒙特卡洛
- ml:
-
最大似然
- PP:
-
后验概率
- rrna:
-
核糖体RNA
- RSCU:
-
相对同义密码子使用
- SSC:
-
小单拷贝
- SSR:
-
简单的序列重复
- TRNA:
-
转移RNA.
参考
- 1。
PU FD,WATSON MF。当归L.中国植物志. 科学出版社:北京。2005;14:140–51.
- 2。
聚氨酯FD。属的修订当归L.(Umbelliferae)在中国。Acta phytotax sin。1991; 29:385-93。
- 3.
leute gh。UntersuchungenÜberden verwandtschaftskreis der Gattung当归L.(umbelliferae)。Teil I. Ann Naturhist Mus Wien。1969年; 73:55-98。
- 4.
周j,高yz,魏茹,刘泽,唐妮斯。分子系统发育当归(Apiaceae)基于NRDNA其序列:猖獗的多重,中国特有物种的放置,以及大量减少的属性。int j植物sci。2020; 181(3):306-23。
- 5。
Engel E,TiLing H.Forlula Ajanensis。NouveauxMémoiresde lasociétélopérialedes moscous des moscous。1858; 1:97。
- 6.
Kozo Poljansky BM公司。系统性石首鱼。牛Soc Nat Mosc。1916;29:93–222.
- 7.
Hiroe M,康斯坦茨L.Mubelliferae日本。大学加利福尼亚州大学加利福尼亚州1958年; 230:1-444。
- 8.
蜂科蜂亚科的分子系统学:核糖体DNA内部转录间隔区和质体的系统发育分析rpoc1.内含子序列。我是J机器人。1998年; 85(4):563-91。
- 9.
Katz-Downie DS, Valiejo-Roman CM, Terentieva EI, Troitsky AV, Pimenov MG, Lee B,等。蜂科蜂亚科的分子系统发育:核糖体DNA ITS序列的补充信息。植物系统演化。1999;216(3):167-95。
- 10。
老唐尼,卡兹唐尼警长,沃森曼氏。基于叶绿体DNA的开花植物蜂科系统发育16卢比和rpoc1.内含子序列:阿片亚科的超属分类。我是J Bot。2000;87(2):273–92.
- 11.
唐妮斯,沃森MF,Spalik K,Katz-Downie DS。旧世界Apioideae(Apiaceae)的分子系统学:部落Peuceaneae Sensu Lato的一些成员之间的关系,在APIOID超级术中的几种岛屿特有物种的位置和分辨率。可以j bot。2000; 78(4):506-28。
- 12.
唐妮斯,斯宾利克,凯茨 - 唐妮DS,雷隆JP。亚艾肝病亚家族综合症中的主要曲巾,其序列的系统发育分析推断。植物潜水员evol。2010; 128(1):111-36。
- 13。
[10]周健,彭华,唐宁,刘志伟,龚晓。基于核糖体DNA内部转录间隔序列的中国蜂科蜂亚科分子系统发育研究。植物分类学报,2011,30(5):757 - 764。分类单元。2008;57(2):402 - 16。
- 14。
周J,Gong X,Downie SR,Peng H.朝着中国Apiaceae的更强大的分子系统发育亚家族,来自NRDNA和CPDNA内含子的额外证据(16卢比和RPS16)序列。mol phylocyet evol。2009; 53(1):56-68。
- 15.
委员会sp。中华人民共和国的药典I.北京:中国医学科技出版社;2015年。
- 16.
Donkor Po,Chen Y,Ding L,秋F.在本地和传统上使用当归物种 - 对其植物化学,药理学和药代动力学进行综述。J属植物。2016; 194:530-48。
- 17.
张X,韩B,冯Z,姜J,杨Y,张P。生物活性含硫化合物和芳香糖苷Ligusticum chuanxiong..Acta Pharm Sin。2018; 8(5):818-24。
- 18.
Raven JA, Allen JF。基因组学和叶绿体进化:蓝藻细菌为植物做了什么?基因组医学杂志。2003;4(3):209。
- 19.
叶绿体基因组的最新进展。植物系统演化。2008;271:101-22。
- 20。
Guigeer MM,Kuehl JV,Boore JL,Jansen RK。在Agiosperm Family Geraniaceae中的塑性基因组的极端重新配置:重排,重复和密码子使用。mol Biol Evol。2011; 28(1):583-600。
- 21。
陆生植物质体染色体的进化:基因含量,基因顺序,基因功能。中国生物医学工程学报,2011;
- 22。
Parks M,Cronn R,Caron A.使用叶绿体基因组的大规模平行测序增加低分类水平的系统发育分辨率。BMC BIOL。2009; 7(1):84。
- 23。
张SD,金杰,陈SY,Chase MW,Soltis de,Li Ht,等。基于塑体系统核科学的晚期白垩纪以来蔷薇科的多样化。新植物。2017; 214(3):1355-67。
- 24。
李HT,易Ts,高LM,MA PF,张T,杨杰,等。Agenperms的起源和侏罗纪差距的难题。Nat植物。2019; 5(5):461-70。
- 25。
谢德芳,谭建军,于勇,桂丽娟,苏东明,周世德,等。对系统发生、年龄和进化的洞察葱属植物(Amaryllidaceae)基于整个塑料序列。Ann Bot。2020; 125(7):1039-55。
- 26。
Jansen Rk,Raubeson La,Boore JL。depamphilis cw,chumley tw,haberle rc等人。获得和分析整个叶绿体基因组序列的方法。方法苄醇。2005; 395:348-84。
- 27。
Chumley TW, Palmer JD, Mower JP, Fourcade HM, Calie PJ, Boore JL等。的叶绿体全基因组序列天竺葵属植物x hortorum:土地植物最大和最热排列叶绿体基因组的组织和演变。mol Biol Evol。2006; 23(11):2175-90。
- 28。
MartínM,Sabater B. PlastidNDH.植物演化中的基因。植物理性生物化学。2010; 48(8):636-45。
- 29。
Wicke S,Müller KF,de Pamphilis CW,Quandt D,Wickett NJ,Zhang Y,et al.扫帚油菜科光合和非光合寄生植物的功能和物理基因组减少机制。植物细胞。2013;25(10):3711–25.
- 30.
易欣,高玲,王斌,苏勇,王涛。中国水稻叶绿体全基因组序列分析Cephalotaxus Oliveri(头孢噻肟):进化比较头孢菌叶绿体DNA和洞察迷失在裸子植物中倒置重复拷贝的丧失。基因组Biol Evol。2013; 5(4):688-98。
- 31。
任涛,杨勇,周涛,刘志林。比较质体基因组樱草物种:序列分歧和系统发育关系。int j mol sci。2018; 19(4):1050。
- 32。
冯S,Zheng K,Jiao K,Cai Y,Chen C,Mao Y等。完全四个叶绿体基因组酸浆属物种(Solanaceae):灯进入基因组结构,对比分析和系统发育关系。BMC植物BIOL。2020; 20(1):1-14。
- 33。
李震,马X,王代,李y,王cw,金xh。塑体基因组的演变Holcoglossum.(兰科西)近期辐射。BMC EVOL BIOL。2019; 19(1):63。
- 34。
钱J,Song J,Gao H,Zhu Y,Xu J,Pang X等。药用植物的完整叶绿体基因组序列萨尔维亚米尔蒂希萨.Plos一个。2013; 8:E57607。
- 35。
Powell W,Morgante M,Andre C,McNicol JW,Machray GC,Doyle JJ等。高变微卫星提供了叶绿体基因组多态性DNA标记的一般来源。电流生物学。1995;5:1023–9.
- 36。
Roullier C,Rossel G,Tay D,McKey D,Lebot V.结合叶绿体和核微卫星来调查新世界红薯实地的起源和分散。Mol Ecol。2011; 20:3963-77。
- 37。
霍y,gao l,liu b,杨y,kong s,sun y等。完全四个叶绿体基因组序列葱属植物种:比较和系统发育分析。Sci众议员2019;9(1):1 - 14。
- 38。
Morton Br。不同植物和藻谱系中叶绿体和氰基基因的密码子偏差的选择。J Mol Evol。1998年; 46(4):449-59。
- 39。
Wakasugi T,Tsudzuki T,Sugiura M.土地植物叶绿体的基因组学:RNA编辑的基因含量和基因组信息的改变。Photosynth Res。2001; 70(1):107-18。
- 40。
Freyer R,Kiefer-Meyer MC,Kossel H.在土地植物的所有主要谱系中塑体RNA编辑的发生。Proc Natl Acad Sci U S A. 1997; 94(12):6285-90。
- 41。
杨y,lv j,gui b,yin h,wu x,zhang y等。A-to-I RNA编辑改变了高度保守的编码区的较少保守的残留物:对进化中的双重功能的影响。RNA。2008; 14(8):1516-25。
- 42。
Tuckaka M,Zehrmann A,Verbitskiy D,Härtelb,Brennicke A. RNA在植物中编辑及其进化。Annu Rev Genet。2013; 47(1):335-52。
- 43。
王W,张W,吴y,maliga p,乱七八型杂志编辑Spirodela polyrhiza.,一种水生单卵种。公共科学图书馆一号。2015;10(10):e0140285。
- 44。
王W,Yu H,Wang J,Lei W,Gao J,Qiu X等。药用植物的完整叶绿体基因组序列连翘吊索(oleaceae)。int j mol sci。2017; 18(11):2288。
- 45。
KrügerM,Abeyawardana OA,Južíčekm,krügerc,Štorchováh。牙科生物中的塑性基因组的变化Silene Vulgaris..BMC植物BIOL。2019; 19(1):1-15。
- 46。
Ogihara Y,Terachi T,Sasakuma T.叶绿体基因组的分子内重组在小麦物种中短的直接重复序列介导的。Proc Natl Acad Sci U S A. 1988; 85(22):8573-7。
- 47。
Weng ML,Blazier JC,Govindu M,Jansen RK。Geraniaceae中祖先塑性基因组的重建揭示了基因组重排,重复和核苷酸取代率之间的相关性。mol Biol Evol。2014; 31(3):645-59。
- 48。
Timme Re,Kuehl JV,Boore JL,Jansen RK。对比分析Lactuca.和直升机(Asteraceae)塑性基因组:鉴定分歧区域和共同重复分类。我是J机器人。2007; 94:302-12。
- 49。
杨y,周t,段d,杨j,冯l,赵g.比较分析五种完整叶绿体基因组栎物种。前植物SCI。2016; 7:959。
- 50。
张X,周t,kanwal n,zhao y,白g,zhao g.完成八古诺斯蒙B.L.(葫芦科)叶绿体基因组:表征,对比分析和系统发育关系。前植物SCI。2017; 8:1583。
- 51。
Ahmed I,Biggs PJ,Matthews PJ,Collins LJ,Hendy MD,Lockhart PJ。AROIP叶绿体基因组的突变动力学。基因组Biol Evol。2012; 4(12):1316-23。
- 52。
Abdullah Mf,Shahzadi I,Ali Z,Islam M,Naeem M,Mirza B等人。植物家族植物麦芽糖植物叶绿素基因组中寡核苷酸重复,核苷酸取代和插入缺失的相关性。J Syst Evol。2020。https://doi.org/10.1111/jse.12585.
- 53。
李X,杨y,亨利rj,rossetto m,王y,陈S.植物DNA条形码:从基因到基因组。Biol Rev Chab Philos SoC。2015; 90(1):157-66。
- 54。
风扇,吴y,阳j,shahzad k,李Zh。比较叶绿体基因组学川绿断目物种:洞察序列变异,自适应演化和系统发育关系。前植物SCI。2018; 9:689。
- 55。
王X,周t,白克,赵玉。完全叶绿体基因组序列Fagopyrum dibotrys.:基因组特征、比较分析和系统发育关系。Sci众议员2018;8(1):1 - 12。
- 56。
谢DF,yu hx,megan p,谢c,邓yq,陈吉,等。汉语系统发育葱属植物在部分中的物种Daghestanica和自适应演变葱属植物(Amaryllidaceae,丙骨液完全基因组透露的物种。前植物SCI。2019; 10:460。
- 57。
Lee-yaw Ja,Grassa CJ,Joly S,Andrew Rl,Rieseberg LH。评估年向日葵中的广泛性核心核心途径的替代解释(直升机).新植物。2019; 221(1):515-26。
- 58。
张X,Deng T,Moore Mj,Ji Y,Lin N,Zhang H,等。塑料系统的血糖索尿(Asteraceae:Cardueae)。BMC植物BIOL。2019; 19(1):1-10。
- 59。
Timme Re,Kuehl JV,Boore JL,Jansen RK。奥斯泰西的前两个测序叶绿体基因组的比较:莴苣与向日葵。劳伦斯伯克利国家实验室。(LBNL),伯克利,加利福尼亚州(美国)。2009年。https://escholarship.org/uc/item/2kd25122.
- 60。
Barrett CF,Davis Ji。肌雌性养殖的塑性基因组Corallorhiza Striata(兰科)处于相对较早的降解阶段。我是J机器人。2012; 99(9):1513-23。
- 61。
Schneider AC,Braukmann T,Banerjee A,StefanovićS.HoloparasiticLennoaceae的收敛塑料进化和基因流失。基因组Biol Evol。2018; 10(10):2663-70。
- 62。
谢Z,商人S.系塑料编码CCSA血红素附着到叶绿体C型细胞学中需要基因。J Biol Chem。1996年; 271(9):4632-9。
- 63。
Sugiura C,Kobayashi Y,Aoki S,Sugita C,Sugita M.完成苔藓的完全叶绿体DNA序列Physcomitrella patens.:损失和搬迁的证据RPOA.从叶绿体到细胞核。核酸杂志2003;31(18):5324-31。
- 64。
Ravin NV,Gruzdev Ev,Beletsky Av,Mazur Am,Prokhortchouk Eb,Filyushin Ma,等。非光合肌营养营养不良癖的塑性和核基因组中的光合途径丧失单调低价.BMC植物BIOL。2016; 16(3):153-61。
- 65。
Wicke S, Müller KF。植物向非光合作用方式进化速率变化的机制模型。中国科学:地球科学;2016;
- 66。
Barrett CF,Freudenstein JV,Li J,Mayfield-Jones Dr,Perez L,Pires JC等人。研究在异养兰花早期过渡型植物中塑体基因组降解的途径,对异养心血管植物的影响。mol Biol Evol。2014; 31(12):3095-112。
- 67。
Pérez-Escobar OA,Balbuena Ja,Gottschling M.隆隆的兰花:如何评估叶绿体内聚有机体与核主体之间的发散进化。系统中的生物学。2016; 65(1):51-65。
- 68。
胡H,胡Q,al-Shehaz Ia,Luo X,Zeng T,Guo X,Liu J。物种划界和种间关系的属性orychophragmus.(Brassicaceae)从整个叶绿体基因组推断。前植物SCI。2016; 7:1826。
- 69。
江y,杨y,lu z,wan d,ren g.四个划界和四个之间的关系奥斯特里亚基于塑料的物种。BMC Genet。2019; 20(1):33。
- 70。
沃尔夫·哈哈,李夫,夏普PM。核苷酸替代率在植物线粒体,叶绿体和核DNA中变化很大。Proc Natl Acad Sci U S A. 1987; 84(24):9054-8。
- 71。
Koch M,Haubold B,Mitchell-Olds T. Dipsicaceae的分子系统:来自编码塑性的证据matK和核武器CHS.序列。我是J机器人。2001; 88(3):534-44。
- 72。
PELSER PB,KENNEDY AH,TEPE EJ,Shidler JB,Nordenstam B,Kadereit JW等人。塑植与核森西奥(Asteraceae)植物生成的案例和原因。我是J机器人。2010; 97(5):856-73。
- 73。
Suh A,Smeds L,Ellegren H.在新景鸟古代自适应辐射中排序的不完全谱系的动态。Plos Biol。2015; 13(8):E1002224。
- 74。
李世勋、崔世勋、宋杰、邦杰。三种中草药的基因组间关系:CNIDIUM Officinale.那Ligusticum chuanxiong.和Angelica多晶型.基因组织。2010; 32(1):95-101。
- 75。
廖C,唐妮斯,李Q,yu y,他x,周b.新的见解进入系统发育的新见解当归和它的盟友(伞形科)重点是东亚物种,从nrDNA, cpDNA和形态学证据推断。系统机器人。2013;38(1):266 - 81。
- 76。
温茹,余y,谢德夫,彭思,刘q,周SD等。基于转录组的文学性研究和分类论争议亚家族亚家族(Apiaceae)的发育和演变研究。Ann Bot。2020; 125(6):937-53。
- 77。
Doyle JJ。少量新鲜叶组织的快速DNA隔离程序。Phytochem Buart。1987; 19:11-5。
- 78。
陈氏,周Y,陈Y,Gu J. Fastp:超快速的一体化FastQ预处理器。生物信息学。2018; 34(17):I884-90。
- 79。
Dierckxsens N,Mardulyn P,Smits G. Novoplasty:来自整个基因组数据的细胞器基因组的De Novo组装。核酸RES。2017; 45(4):E18。
- 80。
Wyman Sk,Jansen Rk,Boore JL。用教条自动注释细胞细胞基因组。生物信息学。2004; 20(17):3252-5。
- 81。
Greiner S, Lehwark P, Bock R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1:用于细胞器基因组图形化可视化的扩展工具包。核酸图谱2019;47(W1): W59-64。
- 82。
下午好,李先生。单细胞生物同义密码子使用的进化观点。摩尔进化杂志。1986;24(1):28–38.
- 83。
佩戴jf。密码子使用分析。博士论文。诺丁汉:诺丁汉大学;1999年。
- 84。
陈超,陈洪,张勇,Thomas HR, Frank MH,何勇,等。TBtools:一个用于交互分析大生物数据的集成工具包。摩尔。2020;13(8):1194 - 202。
- 85。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析6.0版。中国生物医学工程学报。2013;30(12):2725-9。
- 86。
割草机jp。预备套件:用于植物线粒体基因,叶绿体基因和用户定义的对准的预测RNA编辑器。核酸RES。2009; 37:W253-9。
- 87。
Kurtz S, Choudhuri JV, Ohlebusch E, Schleiermacher C, Stoye J, Giegerich R. REPuter:重复分析在基因组尺度上的多重应用。核科学与技术,2001;29(22):4633-42。
- 88。
Frazer Ka,Pachter L,Poliakov A,Rubin Em,Dubchak I. Vista:用于比较基因组学的计算工具。核酸RES。2004; 32(SUPPLE_2):W273-9。
- 89。
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。
- 90.
张颖,马平,李东。6种竹叶绿体基因组的高通量测序:温带木本竹的系统发育意义。植物学报,2010,49(5):557 - 564。《公共科学图书馆•综合》。2011;6:e20596。
- 91.
杨Z.PAML 4:通过最大可能性进行系统发育分析。mol Biol Evol。2007; 24(8):1586-91。
- 92。
Yang Zh,Nielsen R. Codon-替代模型,用于沿着特定谱系检测各个部位的分子适应。mol Biol Evol。2002; 19(6):908-17。
- 93。
张X,周T,杨杰,孙杰,菊M,赵Y,等.葫芦科植物叶绿体基因组的比较分析:光照的选择压力和系统发育关系。分子。2018;23(9):2165.
- 94。
Stamatakis A. Raxml版本8:一种系统发育分析和大脑发育后的分析。生物信息学。2014; 30(9):1312-3。
- 95。
Ronquist F,Teslenko M,Van der Mark P,Ayres DL,Darling A,HöhnaS等人。MRBAYES 3.2:高效的贝叶斯系统发育推理和跨大型模型空间的模型选择。STYS沸腾。2012; 61(3):539-42。
- 96。
Posada D,Crandall Ka。ModelTest:测试DNA替换模型。生物信息学。1998年; 14(9):817-8。
- 97。
Leatunic I,Bork P.互动生命树(ITOL)V4:最近的更新和新的发展。核酸RES。2019; 47(W1):W256-9。
致谢
不适用。
资金
这项工作得到了中国国家自然科学基金的支持(赠款No.32070221,31872647),国家标本信息基础设施,教育标本子平台(赠款No.2005DKA21403-JK),第四次国家中医资源调查(授予No. 2019PC002)。资助者没有参与数据的设计,收集,分析和解释和稿件写作。
作者信息
隶属关系
贡献
TR和X-JH构思和设计了这项工作。TR,Z-XL和D-FX分析了序列数据。L-JG和CP提供了材料/分析工具。tr写了手稿。JW和X-JH修订了稿件。所有作者都批准了本文。
作者的信息
四川大学生态资源与生态环境的重点实验室,四川大学生命科学,610065,中国成都
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者们宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
八个RNA编辑网站数当归质体系。图S2。分析8种基因的简单重复序列(SSRs)当归质体系。图S3。从最大似然(mL)和贝叶斯推理(BI)分析的39种改造的系统发生树重建,基于核内转录的间隔序列分析。每个节点都列出了引导支持值和后验概率值。
附加文件2:表S1。
八个存在的基因列表当归质体系。
附加文件3:表S2。
八个蛋白质编码基因的密码子使用和相对同义密码子使用(RSCU)值当归质体系。
附加文件4:表S3。
八种蛋白质编码基因的碱基组成当归质体系。
附加文件5:表S4。
RNA编辑网站分析八个当归质体系。
附加文件6:表S5。
重复序列分布在八个当归质体系。
附加文件7:表S6。
八个简单的序列重复(SSRS)分布当归质体系。
附加文件8:表S7。
八个中的indel和snp当归质体系。
附加文件9:表S8。
编码和非编码区域中可变字符的百分比。
附加文件10:表S9。
八个遗传距离当归质体系。
附加文件11:表S10。
与现场特定模型PAML选择性压力分析结果。
附加文件12:表S11。
在系统发育分析中,Genbank中的物种列表及其登记号。
附加文件13:表S12。
收集位置和凭证信息用于八个测序的塑料。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
任涛,李兆祥。DF,谢。等等。八个塑料当归物种:特性,基因组进化,和系统发育关系。BMC植物BIOL.20,519 (2020). https://doi.org/10.1186/s12870-020-02696-7
已收到:
公认:
发表:
关键词
- 当归
- 塑料
- 表征
- 轻松选择
- 进化
- 系统发育关系