摘要
背景
RNA测序已被广泛应用于植物全基因组基因表达谱分析和抗病性等重要性状的候选基因鉴定。非洲菊是全球花卉贸易中最重要的花卉之一,白粉病是非洲菊的主要病害。非洲菊抗药性的遗传改良已成为非洲菊育种的重要目标。深入了解非洲菊对PM抗性的遗传控制,有助于非洲菊抗PM品种的培育。
结果
本研究的目的是鉴定具有对比在PM抗性和序列中的对比表型的非洲菊基因型,并分析它们的叶片转录om以鉴定差异表达和与PM抗性相关的抗病和易感基因。额外目标是识别未来遗传研究的SNP和SSR。我们鉴定了两种非洲菊基因型,UFGE 4033和06-245-03,其分别是耐药和易感的。使用四个互补管道的叶片转录瘤的Novo组装导致1124bp的N50的145,348转录物,其中67,312份转录物含有开放阅读框架,并在两个基因型中表达48,268。对于06-245-03,总共可能参与抗病抗性494种转录物,分别在UFGE 4033中调节17和24个转录物。这些非洲菊抗病转录物最类似于赋予植物中各种病原体的植物抗性基因的NBS-LRR类。四种疾病易感性转录物(枣疯病-like)在06–245-03中仅表达或高表达,为非洲菊抗PM基因编辑提供了极好的候选靶点。非洲菊转录组共发现449897个SNPs和19393个ssr,为开发新的分子标记提供了宝贵的资源。
结论
该研究代表了Gerbera PM抗性的第一次转录组分析,这是全球重要的花卉作物中非常重要而复杂的特征。鉴定的差异表达的抗病和易感性转录物提供了分子标记和遗传图,抗病基因克隆的优异靶标,抗病基因的克隆或靶向疾病易感基因的靶向疾病敏感性抗性。
背景
格贝拉雏菊(非洲菊)因其品种繁多、色彩鲜艳而在全球花卉贸易中广受欢迎。它主要作为切花种植,越来越多地作为花园,床上用品,庭院和室内植物。商业非洲菊起源于两个非洲物种之间的人工种间杂交G. Viridifolia和G. Jamesonii.与家长物种相比,引发新的颜色组合,花卉安排和轻松增长[1].单独削减花大丁草在2014年荷兰拍卖会计约为1.4亿欧元[2];在美国切花大丁鱼在2015年产生了3200万美元的批发价值[3.].世界上已经发布了数千个非洲菊品种,在某些性状,如花的数量和花瓶寿命等方面已经取得了重大的遗传改良[4.].凭借有吸引力和复杂的花卉结构,Gerbera也被用作模型植物,以研究奥斯特科伊家族的花卉发育。它还广泛用于植物继发性植物新陈代谢的研究[5.].
白粉病(podosphaera xanthii.syn。Sphaerotheca fusca)是非洲菊中最常见和最具破坏性的病害。这种病菌感染花、叶和其他植物部位,迅速在植物表面形成难看的白色粉末状亚光。这是由菌丝体和分生孢子结构,可以严重限制植物生长,扭曲花,并导致植物死亡。在受控环境和生产结构(如温室和塑料隧道)中,PM的影响具有高度破坏性,大部分非洲菊生产在这些环境和结构中进行。在商业生产中,PM的控制主要依赖于杀菌剂的频繁使用。然而,当环境条件有利于PM时,使用杀菌剂很难实现完全控制[6.]. 发展和利用寄主植物抗性(HPR)被认为是一种经济有效、环境友好的控制PM的策略。HPR广泛应用于小麦、大麦、番茄、葡萄、黄瓜、向日葵和玫瑰等多种作物[7.那8.那9.那10那11那12那13那14].
在格伯拉,HPR用于PM电阻赋予PMR1.迦特斯曾经报道[15]. 在非洲菊育种系ufge31-19和ufge5-23中发现了一种来源不同但对PM有强HPR的非洲菊,这两个育种系已被用于开发新品种和基因型,包括ufge4033[16那17])。UFGE 4033中的PM抗性(PMR)已在多年和多地点试验中得到证实。之前的一项研究表明,两个连锁基因座,Rpx1和Rpx2型,参与控制UFGE 4033中的PMR [16那18].在这方面取得了重大进展拟南芥以及了解植物抵抗PM和PM抗性基因的克隆和表征的其他植物[11那19那20.那21.那22.].然而,尚未确定在Gerbera中PMR下面的基因或候选基因。
近年来,下一代测序(NGS)技术极大地促进了非模式物种基因组和转录组资源的开发。基于NGS的转录组测序或RNA测序(RNA-seq)已成为了解不同表型基因型间遗传变异的最常用方法之一。以前,RNA-seq研究葡萄孢菌抗植物中的抗性鉴定了苯丙砜,黄酮类化合物,乙烯和茉莉酸途径中涉及的25种同源物[23.]. 通过RNA序列分析和注释,在同一水平上鉴定了两个NBS-LRR转录本Pm21轨迹in.Haynaldia摘要[24.].在玫瑰,在下午的RNA-SEQ(全口足类)-抗性物种罗莎longicuspis,以及易感染pm的物种R巨蜥确定了两个候选基因RgMLO6和RgMLO7参与宿主-病原体相互作用[25.].
非洲菊是高度杂合的,有一个相对较大的基因组(约5.0 Gb),富含重复元素。非洲菊参考基因组尚未见报道。长期以来,非洲菊的基因组资源仅限于一些表达序列标签(ESTs) [26.]. 近年来,对非洲菊花的性状、茎弯曲和叶片弯曲的基因表达进行了研究葡萄球菌抵抗[23.那27.]. 到目前为止,还没有任何研究在非洲菊了解基因表达的反应PM。在这项研究中,我们筛选了7个非洲菊育种系来选择对PM具有不同抗性基因型的基因型,并在PM感染期间对所选基因型进行RNA-seq分析。本研究的目的是:1)鉴定非洲菊叶片转录组并了解其整体基因表达谱;2)进行差异表达分析并鉴定与抗PM相关的转录本;3)鉴定非洲菊叶片转录组Mildew Locus O.4)发现非洲菊转录组中存在的单核苷酸多态性(SNPs)和SSR标记。
结果
非洲菊繁殖线的粉状霉菌易感性
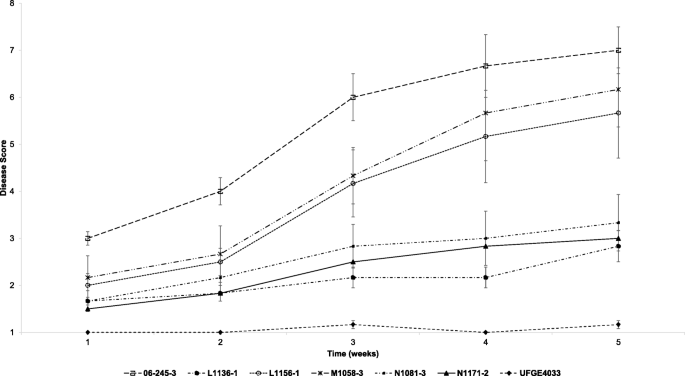
2015-2016年对非洲菊06-245-03、M1058-3、L1156-1、N1081、N1171-2、L1136-1和UFGE 4033 6个非洲菊PM易感育种品系进行抗性评价。06-245-03行显示最高水平的PM易感性和被评为最易感AUDPC选中的行中(疾病进展曲线下面积)每周得分为21.67分,而UFGE 4033一直表现出很少或根本没有点症状AUDPC得分是4.25每星期在研究(无花果。1).线M1058-3,L1156-1,N1081,N1171-2和L1136-1分别易受影响,分别为16.83,15.67,1.50,9.42和8.42的AUDPC评分。基于这些数据,选择繁殖线06-245-03和UFGE 4033作为最易感和最抗性的RNA测序基因型(图S1).图S显示了非洲菊属植物的典型PM症状2.
转录组的组装
对UFGE 4033的三个生物重复(93,675,701 PE reads)和06-245-03的三个生物重复(86,883,908 PE reads)进行测序,共得到54.17 Gb的序列数据,包括180,559,609对端(PE) reads(表S)1).对cDNA片段的两端进行测序得到PE reads;这种类型的读取有助于对齐序列读取,构建更准确的组件,并检测新的转录本。将测序引物和条形码从原始reads中去除并进行质量控制后,将51.80 Gb的测序数据保留下来,其中包含172,658,001条(95.62%)清洗后的reads用于进一步分析。被清理reads的平均GC含量为46.84%。所有被清理的读取,包括来自UFGE 4033的90,476,793和来自06-245-03的82,181,208,被用于从头集会。
最后的汇编由145348份抄本(111409237份)组成 英国石油公司)1124的N50 bp(表1).最小的转录本设置在125 bp,小于200 bp的转录本有4903个(表1)1).转录物的平均长度为761bp,并且最长的转录物含有32,039bp。10个转录物超过10,000bp,31,896个转录物的长度大于1000 bp。有67,312个转录物,开放的读数片段(ORF),组件的GC含量为48.11%(表1).
功能注释
在提交给Blast2GO的145348个转录本中,75390个(51.87%)转录本至少有一个BLASTX命中,66863个转录本被映射,46258个转录本被成功注释到其他植物物种(图S3.). 转录本功能注释数据见表S2.BLAST TOP-HIT分布表明Gerbera转录物最相似Helianthus Annuus.和刺菜蓟var.scolymus(图S)4.).两个都h . annuus和C小豆蔻变量。scolymus属于菊科。映射过程成功映射了66,863(46.00%)转录物。基因本体论映射转录物的注释导致了成功注释的46,258(31.83%)转录物(图S5.).此外,74,195个GO-术语分类为Wego2.0 [28.[其中54,875对应于生物方法,42,566至细胞组分和61,468分子功能。正如植物叶组织所预期的那样,大多数频繁的经常转录注释都提到了主要细胞过程,活动和隔间。有趣的是,大量的转录物获得了向“supplast”(细胞组分分支)的注释,响应刺激(生物过程分支)和“抗氧化活性”(分子功能分支)(图S6.).分配给每个成绩单的多个GO术语导致将每个成绩单分配给多个进程。转录本的酶编码注释在Blast2GO中进行。总共有13085个转录本被分配到特定的酶编码中,其中酶水解酶(5167)是在东非菊叶片转录组中最常见的类群,其次是转移酶3956)、氧化还原酶2334)、裂解酶(647)、连接酶(513)和异构酶(468)(图S7.).
差异基因表达
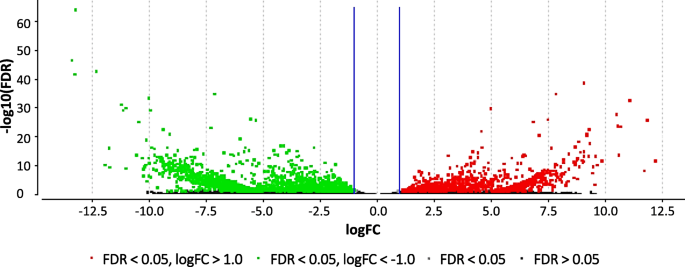
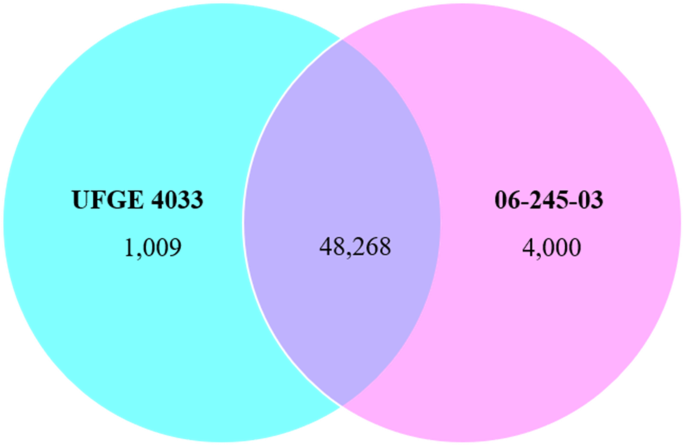
共比对398,015,666条PE reads,其中227,913,186条(57.07%)PE reads成功映射到145,348个转录本,平均映射质量为35.72(表S3.).平均重复率为56.47%,映射reads的平均GC含量为46.51%。使用RSEM对每个样本的映射reads进行计数,用Blast2GO对其进行归一化和差异表达分析。所有组装好的145,348个转录本提交Blast2GO分析,130,858个转录本通过Blast2GO过滤过程(图S2). 在130858份转录本中,Blast2GO对63373份含有ORFs的转录本进行了差异表达(DE)分析。共有3213个差异表达的转录本(FDR) < 0.05)。在去转录物中,1190个表达上调2折叠更改(logFC> 1) ]和2023被下调(logFC<− 1) 在PM抗性非洲菊品系中,分别与PM敏感品系进行比较(图。2). 表S中列出了一个包含所有经过差异表达分析的转录本及其表达值的列表4..从DE分析中观察到12.15 logfc和下调的最高上调和下调-13.42 logfc。育种线中共存在共有48,268个转录物,而1009和4000转录物仅存在于UFGE 4033和06-245-03中(图。3.).在差异表达的转录物中,鉴定出与“氧化还原”,“转录”,“磷酸化”“DNA积分”,“信号转导”和“跨膜转运”的术语鉴定为最代表(图8.).
转录本差异表达并参与抗病
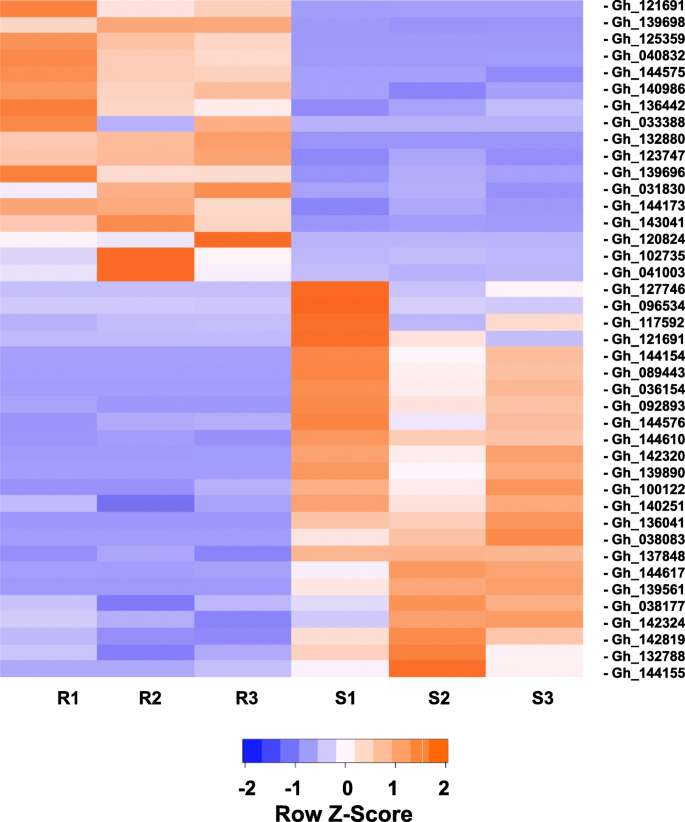
使用关键词“抗病”进行功能注释,从差异表达基因列表中选择与抗病(DR)相关的转录本,结果筛选出494个转录本(表S5.).PM-R细胞系UFGE 4033中有17个DR转录本上调,24个DR转录本下调(图1)。4.,表格2). 四个上调的DR转录物Gh_033388、Gh_120824、Gh_125359和Gh_132880在PM-S系06–245-03中没有表达。两个上调的DR转录物的表达水平较低(FPKM)< 1.0)和更高水平的表达(FPKM 9.18和12.79)在PM-R线(表2).八成绩单,Gh_036154 (logFC =−6.87),Gh_038083 (logFC =−4.81),Gh_089443 (logFC =−5.63),Gh_121691 (logFC =−7.13),Gh_136041 (logFC =−5.09),Gh_139561 (logFC =−6.88),Gh_139809 (logFC =−5.80),Gh_144154 (logFC =−8.25),并不表示在pm r线,但表示在pm s线(表中2).7个转录本在抗性系(FPKM < 1.0)中表达量低,而在感病系(FPKM从4.99到102.13)中表达量高(表1)2).在上调的DR转录本中,有8个与DR转录本最相似AT3G14460, 一个拟南芥基因在抗真菌疾病中发挥着重要作用[29.].有两份抄本类似AT4G27190和RPP13,涉及疾病抗性,七种转录物最相似At1g61180型那At4g27220型那RGA2.那RGA3., 或者马来西亚令吉(表2).在下调的转录本中,有9个转录本与AT3G14460,两个转录本与At4g27220型,四份抄本类似AT4G27190,两个转录本与叶锈病10号抗病受体样蛋白激酶样1.2有关,一个转录本与叶锈病10号抗病受体样蛋白激酶样1.2相似RGA1那RGA3.那RPP13那rmla1.和RGC22.每个(表2). DR转录上调的logFC最高为6.08(ghu 040832和ghu 139712),而下调的logFC最高为ghu 142320和ghu 139712− 8.99(表2).
定量RT-PCR (RT-qPCR)验证差异表达R.-Genes.
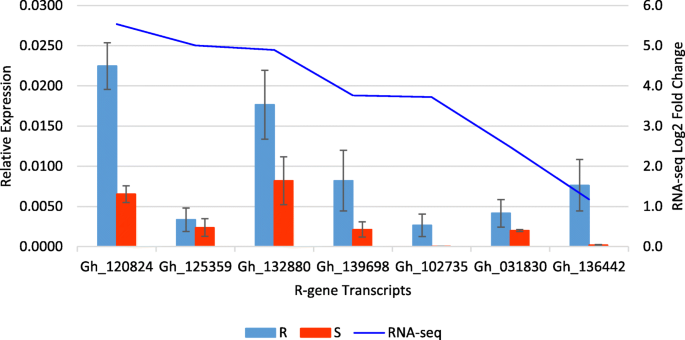
为了验证R.-Genes从RNA-SEQ分析中鉴定,我们随机选择了七个上调R.-Genes并进行RT-QPCR。RNA-SEQ分析结果表明,与敏感线相比,这些基因在抗性管线中差异上调,并且GH_120824,GH_125359,GH_132880,GH_139698,GH_139698,GH_102735,GH_036442具有5.53,5.01,4.89,3.76,3.76,3.76的LOGFC值,3.72,2.47和1.17分别。我们的RT-QPCR数据显示GH_120824,GH_132880,GH_139698,GH_031830和GH_102735的相似较高的表达水平(图。5.).Gh_102735和Gh_136442在敏感系中表达量几乎可以忽略,但在抗性系中表达量高,从而导致其表达量的上调R.-基因转录在RT-qPCR中似乎比RNA-seq分析中更明显。一个七R.-基因转录本gh125359在RNA-seq数据中显示logFC为5.01,但在RT-qPCR分析中抗性品系和感病品系之间具有相似的表达水平。
推定易感性(Mildew Locus O.)的基因在非洲菊转录组
总共有15种假定的疾病易感性(S.)属于的成绩单枣疯病基于与BLAST2GO数据库的注释,在Gerbera转录组中鉴定基因系列(表S.6.).四个S转录本与枣疯病在抗性品系中缺乏表达或表达下调(<−1.0)。Gh_037931、Gh_124463、Gh_124934、Gh_125664近似枣疯病-就像蛋白质8,枣疯病-就像蛋白质6,枣疯病样蛋白12和枣疯病分别 - 蛋白质11(表3.).3个转录本Gh_035301、Gh_116552、Gh_141139相似枣疯病它们的表达水平在抗性线上似乎高于易感线(表S)6.),但其表达量在抗病与感病系之间差异不显著(FDR < 0.05)(表3.).
SSRs和单核苷酸多态性
GATK v1.7、SAMtools v2016.0.109和FreeBayes v1.0.2-6分别鉴定出1,742,092、1,201,429和1,079,141个SNPs(图1)。6.;表S.7.).所有三个管道共鉴定了共449,897个SNP。SAMTOOLS发现了最多的SNP和863,659个SNP,其他两条管道未被其他两个管道识别。Gatk发现了其他两个管道未发现的最低数量的SNP和37,420个SNP。Freebayes检测到其他两个管道未发现的212,262个SNP(图。6.).
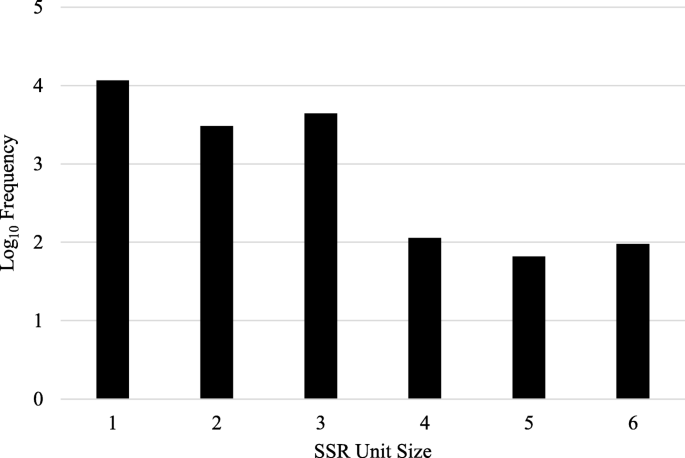
在MISA项目分析的145348个转录本中,14864个转录本包含19393个SSRs(表S8.).有3235种转录物,每个转录物含有多于一个SSR,化合物形成中存在2235个SSR。在Gerbera转录组中鉴定的11,652个单核苷酸Ssrs,3053个二核苷酸ssrs,4413 rinotide ssrs,114四核苷酸ssrs,66个四核苷酸ssrs和95个戊核苷酸ssrs和95个六核苷酸ssrs(图。7.;桌子4.).
讨论
rna测序已被广泛应用于了解基因表达、发现DE转录本和开发植物分子标记。RNA-seq独立于已存在的表达基因数据库,因此可以用于构建非洲菊与PM相关的无偏性基因表达谱。此前,RNA-seq已在非洲菊中用于描述转录组变化,以应对生理紊乱和生物和非生物胁迫,包括茎弯曲,葡萄球菌感染和冷应激[23.那27.那30.]. 利用抗病源UFGE 4033将PM抗性转移到佛罗里达大学开发的新品种,品种开发过程依靠经典育种和PM筛选。利用分子标记辅助育种和新的基因转移方法,鉴定抗稻瘟病基因可以加速抗稻瘟病品种的培育[31.].在这项研究中,转录组测序被用来配置两个非洲菊的叶转录组繁殖,UFGE 4033和06-245-03,不同的抗白粉病和分析基因表达在post-inoculation阶段当大多数基因表达博士的选育品系由于接触点。
非洲菊的记录最类似于h . annuus和C小豆蔻变量。scolymus这两种植物都属于菊科,非洲菊也是。总理在这两方面都是一个具有挑战性的问题h . annuus和C小豆蔻变量。scolymus.研究已经在h . annuus探讨PM抗性和宿主病原体相互作用[32.那33.那34.那35.].
共鉴定出145348个转录组。超过57%的reads被定位到转录组,这可能归因于同一基因不同转录本共享的外显子上的多定位reads和来自未注释转录本的reads仍然未被定位[36.]. 在人类中,70-90%的读取被预期映射到一个参考基因组,而在映射到转录组时,更低的映射率被观察到[36.那37.].UFGE 4033和06-245-03之间的差异表达分析鉴定了差异表达的3213种转录物,其中将1190被上调,并且在UFGE 4033中下调2023.在DEGS中,GO与氧化还原的术语术语:0055114,蛋白质磷酸化GO:0006468,跨膜运输GO:0055085和信号转导GO:0007165富集。氧化还原酶参与反应性氧物质(ROS)的产生,并提高宿主对病原体的抗性。ROS能够通过杀死它们或作为信号分子来限制病原体,以合成抗微生物化合物,并已经在真菌疾病抵抗中进行了研究[38.].GO的富集:00055114在激活苹果抵抗期间发生Botryosphaeria dothidea,导致溃疡[39.].据报道,转录因子的跨膜转运和磷酸化可调节拟南芥的植物防御和真菌抗性[40].信号转导介导的宿主电阻是对生物养真菌病原体的常见电阻机制之一[41.].
该研究确定了41个转录物,其差异表达并参与“抗病性”。三十六个参数包含NB弧域,可以被归类为R.基因转录。铌-电弧畴在其中起着非常重要的作用R.-基因、核苷酸结合位点(NBS)和富亮氨酸重复序列(LRR)基因[42.].这类R.-基因提供对细菌、真菌和病毒植物病原体以及线虫和蚜虫的抗性[43.那44.]. NBS-LRR基因编码的蛋白质主要作为细胞内受体来感知病原体的效应。许多R.-Genes可以在植物中赋予PM电阻[11那45.那46.].例如,在Medicago Truncatula.,Foster-Hartnett等人。鉴定的基因涉及过敏反应(HR)和基底防御erysiphe pisi.[47.].在本研究中,发现与Go-术语相关的转录物:GO55114和GO45454参与氧化还原过程。已知对微生物进行植物防御过程中的氧化还原过程有助于产生反应性氧物质(ROS)并调节HR [48.那49.].在小麦和大麦中,NBS-LRR编码基因,下午和MLA.,赋予PM抵抗Blumeria Graminis.[50].同样地,存在于此的基因运行和仁基因座含有引起PM抗性的NBS-LRR结构域葡萄白粉病在葡萄藤[51].在本研究中,差异表达的丁草“抗病”转录物与前述一些鉴定的高度相似R.基因像RGA、RPP13、RML1A、At3g14460、At4g27190和At4g27220。
在36个丁香NBS-LRR转录物中,八个功能均类似于AT3G14460。基于GO生物学方法分类,通过结合ADP,ATP和腺苷酸环化酶活性,在核中激活AT3G14460蛋白质并在功能上涉及防御响应。有两种类似于AT4G27190的基因。有五个同源物的AT4G27190报告o .漂白亚麻纤维卷还有一个P毛果.它也参与病原体识别和防御反应,属于NB-LRR家族。鉴定出的两个基因与RPP13,这给了抵抗Hyaloperonospora parasitica,一种卵子菌,在包括拟南芥、芸苔和葫芦在内的多种植物中引起霜霉病[52那53].RPP13与易受这种疾病的庭院疫苗相比,也发现在柔软的霉菌新的豚鼠偶然的抗霉菌抗药性转录物中进行了上调[54].RPP13在质膜和细胞质中定位,并赋予防御反应,导致相互作用和过敏反应的不相容[52].有一种类似的基因RGA2.对小麦枯萎病具有广泛的抗性茄属植物bulbocastanum所有种族造成的5种。而RGA2.赋予水平阻力,存在类似的基因RGA3.,如果存在于易感单倍型中,则作为通过缺失和突变产生的伪原。有两个RLM1A样转录本在非洲菊中差异表达,其中一个仅在UFGE 4033中表达。基因RLM1.对半营养性真菌产生抗性,瘦性的黄瓜,在芸苔作物中引起黑腿[55]. 尽管有坏死性反应,L黄斑类似于许多生物营养素疾病,已经建立了与两者的基因型关系芸苔栗鸟和答:芥通过信号转导[55那56]. 有两个基因分别与At1g61180和At4g27220相似。这两个基因都属于NB-LRR家族,都与抗病性有关,但目前对这两个基因知之甚少。
抗病蛋白AT4G27190属于NB-LRR家族[41.].Chen等报道了林地草莓中显著表达的两个at4g27190样蛋白(草莓)在感染期间Phytophthora梵欧姆[57].并不是所有的抗病转录本在非洲菊中都是上调的。有9个与At3g14460相似的转录本,2个与At4g27220相似的基因,4个与At4g27190相似的基因,2个与叶锈病相关的基因10个抗病受体样蛋白激酶样1.2,1个与叶锈病相关的基因RGA1,RGA3,RPP13,RML1A和RGC22.鉴定了在给定的样品中下调了严重疾病载体蛋白中涉及严重疾病载体蛋白的一种基因。需要进一步研究来了解这些转录物的原因是下调。在上调和下调组中存在这种多样性的抗病转录物表明转录物中的功能多样性以防止PM病原体。
霉点O基因编码有7个跨膜螺旋和一个c端钙调素结合域的质膜蛋白,这是PM病原体在许多作物中成功定植的先决条件[58那59那60那61].以前,PM易感性枣疯病据报道,通过促进真菌渗透阶段进入宿主的基因家族可在许多作物中用于PM病原体,包括矮牵牛花和玫瑰等观赏植物,当这些作物发生突变时,可在这些作物中部署广谱、持久的PM抗性[62那63]. 在这项研究中,我们鉴定了四个MLO样转录物,但是这些转录物没有差异表达。这些ghmlo.非洲菊基因组中的基因可以帮助真菌渗透,虽然有必要进一步研究以确认这一假设。参考Gerbera基因组和全基因长度的可用性枣疯病基因将在未来帮助验证这一假说。
来自转录组序列的SNP和SSRS的鉴定可用于构建遗传联系地图,高分辨率基因和基因组测绘,以及标记辅助育种。本研究鉴定了使用SAMTOOLS的1,724,092个SNP,使用GATK和1,201,429个SNPS使用FreeBayes管道和19,393 SSR使用MISA的1,079,141 SNP。这些大量的SNP表示PM抗性和易感非洲菊育种线之间的高水力合酶。此前,龚和邓鉴定了在硅分析中的格伯拉的16,998个EST中的893个EST-SSRS [64].此外,目前尚无利用新一代测序鉴定SSR标记的研究。本研究鉴定的SNPs的准确性可以通过比较不同平台开发的基因分型数据,如测序分型。然而,本研究中没有非洲菊基因组内含子区域的snp。因此,其他方法开发的内含子snp不能与本研究鉴定的snp相比较。在非洲菊中,可以识别和验证可能涉及PM抗性的候选转录本的snp,以开发潜在的PM抗性标记。
总之,我们进行了广泛的分析和表征两种非洲菊雏菊基因型的叶片转录组,其表现出对PM的强抵抗力或极端敏感性,获得145,348个Contig,并且在功能注释的46,258转录物。本研究首次揭示了Gerbera的基因组基因表达差异,响应于PM。我们鉴定了属于的36种抗病基因转录物R.- 基本家庭。这些转录物是显影分子标记的高价基因组资源,以帮助快速筛选PMR的育种群和多系列新的PMR品种。这些转录物可以作为遗传映射和分子克隆的强候选基因R.- 在ufge 4033中控制强抗性的基因。另外,我们识别出来枣疯病仅在pm易感基因型中表达或高表达的类似转录本。这些记录将使我们能够更全面地了解……的作用枣疯病非洲菊中的基因可以作为基因编辑和敲除的强易感基因候选,以提高非洲菊对PM的抗性。本研究所鉴定的ssr和SNPs为开发非洲菊多样性和遗传保真度的分子标记以及在非洲菊品种开发中实施标记辅助选择提供了大量的序列多态性。非洲菊的基因组资源与非洲菊等全球流行的观赏作物相比是贫乏的。在目前的情况下,本研究将为非洲菊的研究增加基因组资源。尽管使用RNA-seq方法获得完整长度的转录本并非易事,但未来参考基因组的可用性将有助于提取具有感兴趣性状的完整基因序列。
结论
非洲菊是世界五大重要花卉作物之一,抗白粉病是非洲菊的重要性状。测序,新创对一对非洲菊抗白粉病育种系的叶片转录组进行了组装和综合分析,鉴定出41个在抗病非洲菊品系中差异表达的抗病基因。这项研究揭示了四个问题Mlo-类似转录本仅在白粉病易感的非洲菊系中高度表达。这些结果代表候选抗性基因和易感基因的第一份报告在非洲菊,提供极具价值的目标发展的分子标记和隔离控制白粉病抗性基因(s)和促进分子标记辅助选择和基因编辑技术的使用在非洲菊。本研究还发现了大量的SNPs和SSRs,这些SNPs和SSRs可用于开发非洲菊全基因组遗传连锁图谱、定位重要园艺基因和研究非洲菊遗传多样性,非洲菊基因组资源在公共领域非常稀缺。
方法
植物材料
从佛罗里达大学非洲菊种质收集的6个具有优良园艺性状的非洲菊育种系进行筛选,以找到最易感的用于本研究的系。选择最敏感的线,这些选择在4英寸从冠塑料锅使用无土盆栽混合物Faffard®3 b(50%加拿大泥炭和50%的蛭石的混合物,松树树皮和珍珠岩)(美国马Agawam)和保存在雾室在墨西哥湾沿岸的温室研究和教育中心,Wimauma,FL,美国。雾室设置为在白天每30分钟运行一次,持续1分钟。两周后,当植物建立起来后,它们被移出雾室。两周后,这些植物被移植到7英寸的塑料盆里,生长一个月。然后对生长中的植物进行PM敏感性筛选。
由佛罗里达大学大学养殖计划开发的格伯拉线UFGE 4033被用作耐受敏感线的耐药控制和生长。UFGE 4033是从PM抗线UFGE 31-19之间的交叉开发的,易感线UFGE 35-4。因此,UFGE 4033是一种杂交,预计将是杂合的[17].UFGE 4033的抗PM性状已转入多个品种[18].因此,该植物用作转录组测序的抗性线。
采用完全随机区组设计,将6个感病品系与抗性对照ufge4033随机分为3个区组。这些植物自然感染了PM,PM广泛存在于温室和设施的其他控制结构中。这种疾病被允许发展一个月,然后每周一次,连续五周,使用1到10的评分量表,其中1 = 无疾病,2 = 跟踪到10%,3 = 10至20%,4 = 20至30%,5 = 30至40%,6 = 40至50%,7 = 50至60%,8 = 60至70%,9 = 70%至80%和10% = 80%至100%的叶片表面覆盖有PM[65].来自实验中最敏感的线条用于转录组分析。
RNA分离和图书馆制剂
选择非洲菊繁殖线UFGE 4033和06-245-03用于转录组测序。ufge 4033和06-245-03如上所述克隆繁殖,以产生三种,其用作三种生物重复。这些个体在白天温度的温室中生长25°C - 32°C,夜间温度为18°C - 22°C的近12小时。植物每周发育,250 ppm为15-16-17个NPK(猫#G99210,Peters Professional,Dublin,Ohio)。在实验期间没有在植物上喷射杀菌剂。当克隆繁殖的植物大约30天时,通过将PM孢子撒在植物冠层上来接种它们。在接种后三到四天后,当在06-245-03出现第一个症状时,将幼叶组织从个体中收集到50ml无菌塑料管中,在液氮中瞬间冷冻并转移至-80℃。叶样品在北京北京北京北京北京时间发货,用于RNA提取和测序。
使用Trizol方法从幼叶组织中提取总RNA [66].在纳米玻璃分光光度计(Thermo Scientific,Waltham,Ma,USA)上测定总RNA的浓度;在1.0%琼脂糖凝胶上评估RNA降解,潜在的污染和纯度; 180V电压16分钟);在Agilent 2100 Bioanalyzer(Santa Clara,Ca,USA)上测定RNA定量和完整性。具有高质量的RNA制剂用于文库制备和测序。
使用NebNettal®UltraRNA库预备套件(新英格兰Biolabs,Ipswich,Ma,USA)进行图书馆结构。使用寡核苷酸(DT)珠子从总RNA富集信使RNA(mRNA)。使用来自Illumina Truseq TM RNA样品制备试剂盒V2(Illumina,San Diego,Ca,USA)的洗脱素片段混合物随机在碎片缓冲液中随机分离富集的MRNA。使用随机六烷烃和使用Developer协议使用illumina Truseq TM RNA样品制备试剂盒(Illumina,San Diego,CA)使用随机六烷烃和逆转录酶合成cDNA。在第一链合成之后,用DNTPS,RNase H和NARSEND合成自定义二链合成缓冲液(Illumina,San Diego,Ca,USA)大肠杆菌聚合酶I通过镍翻译合成互补链形成稳定的双链cDNA。用AMPure XP珠纯化合成的cDNA。进一步纯化cDNA,修复末端末端,加入poly-A尾部制备最终cDNA。测序适配器然后连接到最终cDNA的独特鉴定。然后对这些添加的适配器进行大小筛选,通过聚合酶链反应(PCR)富集,建立待测序的cdna文库。所选cDNA片段大小为250 ~ 300bp,插入到cDNA文库中(无定向)。使用Qubit 2.0荧光计(Life Technologies, carl lsbad, CA, USA)测量浓度,调整库浓度至1 ng/μl,评估库的质量。使用Agilent 2100 (Bioanalyzer)验证插入扩增子的大小,最后通过定量PCR (qPCR)(文库活性> 2 nM)进行更高精度的定量。然后使用质量控制库进行测序。
转录组测序、质量控制和筛选
在Illumina HiSeq 2000平台上使用Illumina flowcell的两条通道对文库进行测序。HiSeq 2000的原始数据通过碱基调用转换为序列读取,并存储为包含序列和相应序列质量描述的fastq文件。FASTQC v0.11.1管道用于执行质量分析,移除适配器并过滤低质量序列。在测序前连接到序列的适配器(RNA 5′适配器(RA5):5′-aatgatacggcgaccagagatctcactcttcctacacgacgccttcgga TCT-3′和RNA 3′适配器(RA3):5′-gatcggagagagagagagagaccgtagagagagacctgactccagtcac(6-核苷酸指数)atctcgtagctccagtctgtgtgtgtgtgtgtgtg-3′) 生成了不包括适配器的bp。序列描述用于评估测序数据的质量。使用Phred评分评估每个碱基的测序错误率。同时对GC含量的分布进行了评价,以检测AT/GC在序列中的分离。在质量评估之后,序列读取要经过不同的过滤。包含适配器序列的读取被丢弃。含有10%以上不确定核苷酸的序列读取(N. > 10%也被丢弃。根据Phred分数,如果测序的读数含有50%以上Phred分数低于20的核苷酸,那么这些读数也被排除。过滤后,含有不到10%未知核苷酸和Phred分数的序列读取≥20个被保留为干净的读数。通过FASTQC方法评估清洁读数的质量,并用于从头组装构建和下游分析。
新创装配
这从头由印第安纳大学国家基因组分析支持中心(NCGAS)开发的组装管道(https://github.com/NCGAS/de-novo-transcriptome-assembly-pipeline)用于从UFGE 4033和06-245-03的已清理的对端读取创建一个程序集。这个管道使用四种不同的汇编程序:Trinity, TransAbyss, Velvet和SoapDenovo [67那68那69那70].Trinity (trinityrnaseq/2.4.0)运行使用默认参数和规范化在50x覆盖(Trinity——normalize_max_read_cov——seqType fq——max_memory 200G——left reads_1.fq.gz——right reads_2.fq.gz——CPU 6) [67].RASICABYSS(TRANSABYSSS / 2.0.1)与K-MERS的K-MERS运行,K35,K45,K55,K65,K75和K85以及以下命令(TRANSABYSS-K 35 - PE $ READS1 $ READS2 -OUTDIR $ OD -NAMEK35.Transabyss.fa --Threads 4 -C 12)[68]. 在一个命令中使用K-mers 35、45和55,在第二个命令中使用K-mers 65、75和85。然后合并每个命令的输出。天鹅绒(Velvet/1.2.10)与35、45、55、65、75和85的k-mers(velveth oases.35-shortPaired-fastq-separate$left$right and)一起使用,插入尺寸为260(velvetg oases.35-read\ trkg yes-ins\ u length 260 and)。程序(sed-i's/>/>velvelet.k35./g'oases.35/transcripts.fa)将不同k-mers(35、45、55、65、75和85)产生的转录本结合起来。Soapdenovo(soapdenovotrans/1.03)以35、45、55、65、75和85的k-mers运行(Soapdenovo-Trans-127mer all-s config_file-k 35-o output35和)。由上述四个装配者组装的转录本被合并,保留产生每一个contig的装配者的名字,并使用Evigene进行BLAST[71]保留高质量的折叠。由单个汇编器生成但不包括在最终合并组件中的折叠被保留但分别分析。
功能注释
使用BLAST2GO版本5进行组装CONTIGS的功能注释[72].在1.0E-03的截止值下,使用BLASTp将非洲菊与NR数据库进行比较,并保留了20个相似性最高的top hits。然后,将BLAST搜索中获得的热门搜索结果提交给基因本体(GO)数据库,以获得BLASTx搜索中搜索结果的GO术语和GO ID。使用E-value-Hit-filter 1.0E-06对指定GO术语的contigs进行注释,注释截止为55,GO权重为5,Blast2GO的其他默认参数[73]. 具有指定GO术语的转录本被提交到Web基因本体注释图(WEGO)2.0[28.]在三个主要GO类别中对GO项进行分类。
量化
读取的量化是通过计算到转录组集合的映射原始读取来执行的。Bowtie2用于使用默认参数执行无间隙映射,RSEM用于量化转录物表达水平[74那75].使用Qualimap和Samtools管道评估读取映射的质量[76那77]. RSEM使用期望最大化来推断转录水平的表达。对映射到每个转录本的序列读取进行计数、标准化并计算为每千碱基转录本片段(FPKM)值。
差异基因表达分析
通过将06-245-03的三个生物学复制作为易感组和UFGE 4033的三个生物学复制作为抗性群体,通过将读取的读数与易感组的三个生物学重复进行读取的读取比较读取。使用软件的内置管道在Blast2Go中进行差异表达。M值(TMM)归一化的修整平均值并使用抗性和易感性进行表达分析,作为实验因素和三种易感和三个抗性样品分别作为参考和对比条件。进行确切的测试以确定显着程度。使用FDR <0.05考虑差异表达转录物。注意到每个转录物的LOG2FOLD变化并用于进一步分析。> 1.0的log2fold变化用于调用上调的转录物,并使用logfc < - 1来调用下调的转录物。具有1至-1之间的表达水平的转录物被认为是差异调节的。
定量rt - pcr分析
使用RNeasy plus mini kit (Cat.)从抗性和易感系的三个生物重复中收集用于RNA-seq(如上所述)的叶组织中提取总RNA。# 74134, Qiagen, Hilden, Germany)使用制造商的协议。用纳米滴分光光度计评价提取的RNA的质量。采用高容量cDNA逆转录试剂盒(Cat.)将每个样本的500ng RNA转化为cDNA。# 4368814,应用生物系统,福斯特市,CA)使用制造商的化学和协议。简单地说,2.0 μL的10x RT buffer, 0.8 μL的25x dNTP Mix (100 mM), 2.0 μL的10x RT随机引物,1 μL的MultiScribe™逆转录酶,4.2 μL的无核酸酶水,500 ng的RNA用于每个样本的cDNA转换。25°C for 10 min, 37°C for 120 min, 85°C for 5 min进行cDNA合成。RT-qPCR反应采用Applied Biosystems (SYBR green, Cat。# 100029284,应用生物系统)。反应混合物为20 μL,其中SYBR green 10 μL,正、反引物各1 μL, cDNA 2 μL,无核酸酶水7 μL。 The PCR was performed with 50 °C for 2 min., 95 °C for 3 min., 95 °C for 30 s., 60 °C for 15 s., 95 °C for 15 s. and 60 °C for 30 s. on a QuantStudio 3 realtime PCR machine (Applied Biosystems). Primers used in the RT-qPCR experiment were designed using primer3 software (https://bioinfo.ut.ee/primer3-0.4.0/)并列于表s中9..从GE等人使用GERBERA中的actin基因的引物。和kuang等人。[27.那78]. 靶基因的表达被标准化为内源性G. Hybrida用2{−Ct(感兴趣的基因)-Ct(肌动蛋白)}[79].
简单重复序列的鉴定
使用MISA程序对转录组中的SSRs进行了鉴定[80].SSRs是根据核苷酸的重复次数来命名的。只有当单核苷酸重复至少10次,二核苷酸重复至少6次,三、四、五、六核苷酸重复至少5次时,才能鉴定出具有单、二、三、五、六核苷酸的SSRs。对于复合ssr,连续ssr之间的最大中断距离设置为100 bp。仅保留重复序列两侧至少100 bp的ssr。
单核苷酸多态性鉴定
SNPs的鉴定采用了GATK v1.7、SAMtools v2016.0.109和FreeBayes v1.0.2-6三个管道[77那81那82].任何质量分数< 20的SNPs都被丢弃。SAMtools的堆积管道及其默认参数用于调用snp。使用faidx命令对参考转录组进行索引,并在运行管道之前对.bam文件进行排序。在带有默认参数的GATK中使用gentypegvcfs命令行,利用该管道并以转录组为参考调用SNPs。使用FreeBayes缺省管道分别调用UFGE 4033和06-245-03中的SNPs,之后共同分析调用共识SNPs。
可用性数据和材料
支持结果和结论的数据图表包含在文章和附加文件中。通过测序为本研究生成的所有序列可以在NCBI短读档案(SRA)Bioproject Prjna647707(https://www.ncbi.nlm.nih.gov/sra//term=prjna647707.).
缩写
- 下午:
-
白粉病
- RNA-seq:
-
RNA测序
- SNP:
-
单核苷酸多态性
- SSR:
-
简单的序列重复
- GATK:
-
基因组分析工具包
- 罗斯福:
-
假发现率
- 飞行里程:
-
每千碱基转录本每百万映射读取片段数
- logfc:
-
日志折叠变化
- RSEM:
-
通过采用RNA-Seq
- NB-LRR:
-
核苷酸结合亮氨酸富含重复
- MLO编号:
-
霉点O
- NB-ARC:
-
核苷酸结合APAF-1、R蛋白和CED-4蛋白
- RT-QPCR:
-
定量逆转录聚合酶链反应
- 人力资源:
-
宿主阻力
- PMR:
-
粉状霉菌抵抗力
- PMS:
-
白粉病敏感
- 澳大利亚石油公司:
-
疾病进展曲线下面积
- HPR:
-
寄主植物抗性
- QC:
-
质量控制
- ngs:
-
新一代测序
- 美国东部时间:
-
表达序列标签
- 开放阅读框:
-
开放阅读框
- pe:
-
Paired-end
- 博士:
-
抗病性
- DEG:
-
差异表达基因
工具书类
- 1.
林奇ri。格贝拉,带有新型杂种的彩色板材。植物群Sylva。1905; 3:206-8。
- 2。
弗洛拉霍兰德。事实和数字2014。https://www.royalfloraholland.com/media/3949227/Kengetallen-2014-Engels.pdf.2014. [2015年12月12日访问]。
- 3.
美国农业部(USDA)。花卉农作物2018年摘要。https://www.nass.usda.gov/publications/todays_reports/reports/floran19.pdf..2019. [2019年11月6日访问]。
- 4.
GPN杂志。https://gpnmag.com/article/new-standards-in-gerberas/.2018.[2020年5月15日通过]。
- 5.
植物多样性研究:非洲菊作为植物发育和生物合成研究的模型系统。BioEssays。2006; 7:756 - 67。
- 6.
Hausbeck MK、Wagstaff SA、Harlan BR、Sconyers LE。非洲菊白粉病防治药剂防治效果评价,2005。真菌内质网试验,2006;植物分布。管理。报告61:OT010。https://doi.org/10.1094/FN61.
- 7.
萧S,卡里斯o,patrick e,张g,charoenwattana p,muskett p,parker je,turner jg。非典型抗性基因,第8页为小麦白粉病抗性的基础防御提供了新的组成部分拟南芥. 植物J。2005;42:95–110.
- 8.
Yahiaoui N,Srichumpa P,Dudler R,凯勒B.不同倍性水平的基因组分析允许克隆粉末状霉菌抗性基因Pm3b型来自六倍体小麦。植物J。2004;37:528–38.
- 9.
Wei F,Wing Ra,Wise RP。基因组动力学和演化MLA.(粉状霉菌)大麦的抵抗轨迹。植物细胞。2002; 14:1903-17。
- 10
Bai Y,Huang Cc,Van der Hulst R,Meijer -Dekens F,Bonnema G,Lindhout P. QTLS用于番茄粉状霉菌抗性(Oidium Lycopersici.) 在Lycopersicon parviflorumG1号。1601与两个抗白粉病基因共定位。植物与微生物相互作用。2003;16:169–76.
- 11
陈建平,陈建平,陈建平,等。一种抗白粉病基因的同源性鉴定。理论应用,2002;104:610-8。
- 12.
库伊斯特拉E。黄瓜对白粉病的抗性。大提琴。1968;17:236–44.
- 13。
Rojas-Barros P, Jan CC, Gulya TJ。从野生植物中转移抗白粉病基因直升机进入耕地向日葵。在:第27届向日葵研究研讨会的诉讼程序。法戈。国家向日葵协会;2005. p。12-3。
- 14。
Linde M,Hattendorf A,Kaufmann H,Debeny T.玫瑰中的粉末状霉菌抵抗:使用选择性基因分型不同环境中的QTL映射。Al Appl Genet。2006; 113:1081-92。
- 15.
陈建平,陈建平,陈建平,等。水稻白粉病抗性的遗传分析。中国农业科学,2003,25 (3):431 - 438非洲菊.Hortscience。2005; 40:1246-51。
- 16.
[16]宋欣,邓铮。非洲菊对白粉病的抗性:遗传模式、数量性状位点鉴定及抗性反应。中国医学杂志,2013;
- 17.
邓Z,Harbaugh BK.UFGE 4141,UFGE 7014,UFGE 7015,UFGE 7023,UFGE 7032,和UFGE 7034:六个非洲菊新品种,用于销售大型容器中的开花植物。霍特科学。2010;45:971–4.
- 18.
邓泽,哈尔波·伯克。UFGE 7031和UFGE 7080丁香品种。Hortscience。2013; 48:659-63。
- 19.
小S,Ellwood S,Calis O,Patrick E,Li T,Coleman M,Turner JG。广谱抗性抗性拟南芥由第8页. 科学。2001;291:118–20.
- 20.
SRICHUMPA P,Brunner S,Keller B,Yahiaoui N.等位基因系列四种粉状霉菌抵抗基因量子化学六倍体面包小麦的基因座。植物生理学。2005;139:885–95.
- 21.
姚G,张J,杨L,许H,姜Y,熊L,张C,张Z,马Z,酸雨我。艾因科恩两个抗白粉病基因的遗传作图(Triticum Monococcum.L.)加入。Al Appl Genet。2007; 114:351-8。
- 22。
陈建平,李建平,陈建平,等。苹果抗白粉病基因的定位Pl1及其与NBS-LRR候选抗性基因的遗传关联。植物品种。2007;126:476–81.
- 23。
Fu Y、Esselink GD、Visser RG、van Tuyl JM、Arens P。基因转录组分析非洲菊包括在硅的硅确认中发现。前植物SCI。2016; 7:247。
- 24。
邢升,胡p,刘j,witek k,周s,xu j,周w,gao l,huang z,张r,王x。Pm21从Haynaldia摘要编码赋予小麦粉状霉菌抗性的CC-NBS-LRR蛋白。莫尔植物。2018; 11:874-8。
- 25。
湘G,张H,剑,闫H,王Q,周N,李S,唐k,邱X.Degogo组装与敏感和抗升玫瑰种类转录组的响应粉状霉菌的转录组合。SCI Hortic。2019; 257:108653。
- 26.
Laitinen Ra,Immanen J,Auvinen P,Rudd S,Alatalo E,Paulin L,Ainasoja M,Kotilainen M,Koskela S,Teeri Th,Elomaa P.花卉转录组的分析揭示了与花有关的器官测定和基因家族的新调节因子器官差异化非洲菊(菊科)。基因组研究》2005;15:475 - 86。
- 27.
ge y,lai q,罗p,liu x,陈w.转录组分析非洲菊说明茎弯曲是水分胁迫和脱落酸调控的结果。BMC基因组学。2019;20:600.
- 28.
叶洁,张勇,崔鸿,刘杰,吴勇,程勇,徐辉,黄旭,李森,周安,张旭。WEGO 2.0:一种用于GO注释分析和绘制的web工具,2018年更新。Nuc Acids Res. 2018;46(W1): W71-5。
- 29.
Deyoung Bj,Bickle Kl,Schrage Kj,Muskett P,Patel K,Clark SE。克拉维多相关的BAM1,BAM2和BAM3受体激酶样蛋白是可以在中拟南芥.植物J. 2006; 45:1-6。
- 30
李士,山q,yu r,siddique a,li x,阳c,阮j,li m。比较转录组分析非洲菊品种表现出耐寒机制。植物组学。2018;十一10。
- 31
邓泽,巴蒂拉伊克。格伯拉。in:van huylenbroeck j,编辑。观赏作物。Cham,Switzerland:Springer;2018. p。407-38。
- 32
一种被白粉病感染、易感、抗性和免疫的向日葵基因型。蛋白质组学。2018;18:1700418。
- 33
Félix-Gastélum R, Olivas-Peraza DD, Quiroz-Figueroa FR, Leyva-Madrigal KY, Peñuelas-Rubio O, Espinosa-Matías S, Maldonado-Mendoza IE。由白粉病引起Golovinomyces spadiceus在墨西哥锡那罗亚的野生向日葵上。植物病理学。2019;41:301–9.
- 34
赵东涛,赵泽,金智怡,蔡奕奕,申德华。首次报道白粉病由Golovinomyces ambrosiae在向日葵salicifolius在韩国。植物分布。2018;102:1453.
- 35
Trigiano RN, Bernard EC, Hadziabdic D, Dattilo AJ, Wadl PA。向日葵白粉病初报(向日葵verticillatus)引起Golovinomyces ambrosiae. 植物分布。2016;100:1017.
- 36。
Conesa A,Madrigal P,Tarazona S,Gomez-Cabrero D,Cervera A,McPherson A,Szcześniakmw,gaffney dj,elo ll,zhang x,mortazavi a。对RNA-SEQ数据分析的最佳实践调查。基因组Biol。2016; 17:13。
- 37。
Dobin A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S, Batut P, Chaisson M, Gingeras TR. STAR:超快通用rna序列。生物信息学。2013;29(1):15至21。
- 38。
Park Ch,Chen S,Shirsekar G,周B,Khang Ch,Songkumarn P,Afzal AJ,Ning Y,Wang R,Bellizzi M,Valent B.的稻瘟病菌效应子AvrPiz-t以环E3泛素连接酶APIP6为靶点抑制水稻病原相关分子模式触发的免疫。植物细胞。2012;24:4748–62.
- 39.
他x,huo y,liu x,周q,feng s,shen x,李b,吴s,陈x。激活疾病抵抗Botryosphaeria dothidea通过下调mdsyp121在苹果。Hortic杂志2018;5:1-6。
- 40。
孟X,徐茹,何y,杨凯,梅德斯基,刘y,张某的ERF转录因子的磷酸化拟南芥MPK3 / MPK6调节植物防御基因诱导和真菌抗性。植物细胞。2013; 25:1126-42。
- 41。
Kliebenstein DJ,Rowe HC。生物营养素与病症的生态成本,过敏反应和信号转导。植物SCI。2008; 174:551-6。
- 42.
陈国强,陈国强,陈国强。植物NBS-LRR蛋白:适应性卫士。基因组医学杂志。2006;7:1-1。
- 43.
迈耶斯BC,迪克曼AW,米歇尔莫尔RW,西瓦拉马克里希南S,索布拉尔BW,青年ND。植物抗病基因编码核苷酸结合超家族中一个古老而多样的蛋白质家族的成员。植物J。1999;20:317–32.
- 44.
Ellis J,Dodds P,Pryor T.结构,功能和植物疾病抗性基因的演变。CurrOp植物BIOL。2000; 3:278-84。
- 45.
Azevedo C, Sadanandom A, Kitagawa K, Freialdenhoven A, Shirasu K, Schulze-Lefert P. The RAR1 interaction SGT1, A essential component ofR.基因触发抗病性。科学。2002; 295:2073-6。
- 46。
Yahiaoui N,Brunner S,Keller B.小麦驯化后的新粉状霉菌抗性基因的快速产生。工厂J. 2006; 47:85-98。
- 47。
Foster-Hartnett DA, Danesh D, Penuela S, Sharopova N, Endre G, Vandenbosch KA, Young ND, Samac DA。的分子和细胞学反应Medicago Truncatula.至erysiphe pisi..Molecular Plant Pathol. 2007; 8:307-19。
- 48。
格林伯格JT,Vinatzer Ba。鉴定植物病原体的III型作用,分析它们与植物细胞的相互作用。Currin微生物。2003; 6:20-8。
- 49。
Nimchuk Z,Eulgem T,Holt III BF,Dangl JL。植物免疫系统中的识别与反应。Annul Rev Genet。2003; 37:579-609。
- 50。
关键词:MLA6, NBS-LRR, NBS-LRR, avrmla6Blumeria Graminis.f . sp。HORDEI.在大麦和小麦。工厂J. 2001; 25:335-48。
- 51。
Goyal N, Bhatia G, Sharma S, Garewal N, Upadhyay A, Upadhyay SK, Singh K.全基因组特征揭示了NBS-LRR基因在白粉病感染过程中的作用葡萄.基因组学。2020; 112:312-22。
- 52
关键词:植物,遗传,物理图谱,生物信息学RPP13轨迹,拟南芥,负责具体识别几个peronospora parasitica.隔离霜霉病。植物与微生物的相互作用,1999;
- 53
Bittner-Eddy PD, Crute IR, Holub EB, Beynon JL。RPP13是一个简单的基因座拟南芥等位基因指定的霜霉病抗性不同无毒决定因素peronospora parasitica..植物j . 2000; 21:177 - 88。
- 54
Bhattarai K,Wang W,Cao Z,Deng Z。凤仙花叶片转录组的比较分析揭示了抗霜霉病的候选基因plasmopara obducens.国际分子科学杂志。2018,19:2057。
- 55
Delourme R,Pilet-Nayel ML,Archipiano M,Horvais R,Tanguy X,Rouxel T,Brun H,雷纳德M,Balesdent MH。一群主要的特异性抗性基因瘦性的黄瓜在里面芸苔栗鸟.植物疗法。2004; 94:578-83。
- 56
斯塔尔J、卡利夫M、博曼S、迪克赛利斯C。海侵分离揭示了两个问题拟南芥ir - nb - lrr抗性基因有效对抗瘦性的黄瓜,黑腿病的病因。植物j . 2006; 46:218-30。
- 57
Chen XR、Brurberg MB、Elameen A、Klemsdal SS、Martinussen I。林地草莓抗性基因类似物的表达(草莓)在感染期间Phytophthora梵欧姆.Mol Gen Genomics。2016; 291:1967-78。
- 58
Büschges R, Hollricher K, Panstruga R, Simons G, Wolter M, Frijters A, Van Daelen R, Van der Lee T, Diergaarde P, Groenendijk J, Töpsch S.大麦枣疯病基因:植物病原体抗性的新型控制元素。细胞。1997年; 88:695-705。
- 59
关键词:白粉病,宿主细胞蛋白,遗传,遗传机制,白粉病Nat麝猫。2006;38:716-20。
- 60.
Devoto A、Piffanelli P、Nilsson I、Wallin E、Panstruga R、von Heijne G、Schulze Lefert P。基因组的拓扑结构、亚细胞定位和序列多样性枣疯病家庭在植物。生物化学杂志。1999;274(49):34993-5004。
- 61.
Kim Mc,Panstruga R,Elliott C,MüllerJ,Devoto A,Yoon HW,Park HC,Cho MJ,Schulze-Lefert P.钙调蛋白与MLO蛋白质相互作用,以规范大麦的防御霉变。自然。2002; 416:447-51。
- 62。
蒋平,陈勇,王尔德。降低矮牵牛花中MLO1的表达增加了对白粉病的抗性。Sci Hortic。2016;201:225-9。
- 63。
邱X,简H,王Q,唐K,鲍M。四种语言的表达模式分析枣疯病玫瑰的基因。美国社会科学杂志。2015;140:333–8.
- 64。
Gong L,Deng Z. Est-SSR标记为格伯拉(非洲菊).mol品种。2010; 26:125-32。
- 65。
Hausbeck Mk,Quackenbush Wr,Linderman SD。非洲菊花抗白粉病的品种评价。B&C测试。2002; 18:O0004。
- 66.
孟,费尔德曼。一种基于TRIzol的两步快速提取DNA游离RNA的方法拟南芥西力克和干种子。生物技术杂志。2010;5:183–6.
- 67.
Haas BJ, Papanicolaou A, Yassour M, Grabherr M, Blood PD, Bowden J, Couger M, Eccles D, Li B, Lieber M, MacManes MD. De novo转录本序列的重建利用trinity平台进行参考生成和分析。Nat Prot。2013;8:1494 - 512。
- 68.
Robertson G,Schein J,Chiu R,Corbett R,Field M,Jackman SD,Mungall K,Lee S,冈田HM,Qian JQ,Griffith M. de Novo集装和RNA-SEQ数据分析。NAT方法。2010; 7:909-12。
- 69.
谢y,吴g,唐j,罗r,帕特森j,刘某,黄某,何g,顾s,李某,zhou x. soapdenovo-trans:de novo转录组组装短的RNA-SEQ阅读。生物信息学。2014; 30:1660-6。
- 70
Zerbino博士,Birney E。Velvet:使用de Bruijn图进行从头短读组装的算法。Genom Res.2008年;18:821-9.
- 71
由mRNA序列而不是基因组DNA构建的基因组。见:第七届节肢动物基因组学年会巴黎圣母院:2013。2013.
- 72。
Conesa A,Götzs,García-gómezJM,Terol J,TalónM,Robles M.Bast2Go:功能基因组学研究中的注释,可视化和分析的通用工具。生物信息学。2005; 21:3674-6。
- 73。
Götz S、García-Gómez JM、Terol J、Williams TD、Nagaraj SH、Nueda MJ、Robles M、Talón M、Dopazo J、Conesa a。使用Blast2GO套件进行高通量函数注释和数据挖掘。核酸研究,2008年;36:3420-35.
- 74。
Langmead B,Salzberg SL.与蝴蝶结2.自然方法的快速间隙读取对齐。2012;9:357.
- 75。
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12:323。
- 76
García-Alcalde F, Okonechnikov K, Carbonell J, Cruz LM, Götz S, Tarazona S, Dopazo J, Meyer TF, Conesa A. Qualimap:评估下一代测序比对数据。生物信息学。2012;28:2678-9。
- 77
Ramirez-Gonzalez RH, Bonnal R, Caccamo M, MacLean D. Bio-samtools: SAMtools的Ruby绑定,一个用于访问包含高通量序列比对的BAM文件的库。《源代码生物医学》2012;7:6。
- 78
关键词:非洲菊,赤霉素,转录组,信号转导引用本文:《公共科学图书馆•综合》。2013;8 (3):p.e57715。
- 79
Livak KJ, Schmittgen TD。采用实时荧光定量PCR和2 (-Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25:402-8。https://doi.org/10.1006/meth.2001.1262.
- 80。
拜尔·S、蒂尔·T、门奇·T、舒尔茨·U、马斯彻·M。misaweb:用于微卫星预测的web服务器。生物信息学。2017;33:2583-5.
- 81。
DePristo MA、Banks E、Poplin R、Garimella KV、Maguire JR、Hartl C、Philippakis AA、Del Angel G、Rivas MA、Hanna M、McKenna A。利用下一代DNA测序数据进行变异发现和基因分型的框架。纳特·吉内特。2011;43:491.
- 82。
加里森E,马特G。基于单倍型的短读序列变异检测。2012年。arXiv:1207.3907。
致谢
我们感谢国家基因组分析中心的支持(NCGAS;https://ncgas.org/),提供多重汇编流水线和转录组分析培训,以及三位审稿人,以表彰他们在改进稿件方面的贡献。这项研究是在佛罗里达大学/IFAS海湾海岸研究和教育中心的设施中进行的,该中心位于美国佛罗里达州威茂马。
基金
该项目的资金,包括助教到KB,由佛罗里达大学/ IFAS植物育种研究生计划(PBGI)提供。美国农业部孵化项目FLA-GCC-005507提供了额外的资金来进行实验和测序.资助机构没有参与这项研究的设计、数据的收集、分析和解释以及手稿的撰写。
作者信息
从属关系
贡献
ZD担保了资金,构思和设计了研究,并批判性修订了手稿。KB进行了实验,分析了数据并写了稿件。SP辅助RT-QPCR实验。AC,SX,NAP和DGC为该研究提供了重要方向,提供了批判性评论并修订了稿件。所有作者均阅读并批准了手稿。
通讯作者
伦理宣言
道德认可和参与同意
不适用。
出版许可
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
附加信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
UFGE4033和06–245-03非洲菊育种系的读取数据、质量控制和GC含量说明。表S2。使用Blast2GO对非洲菊转录本进行功能注释。表S3。利用Samtools和Qualimap对UFGE 4033和06-245-03样本进行重新转录组组装的制图统计。样本R1、R2和R3代表UFGE 4033的三个生物重复,S1、S2和S3代表06-245-03的三个生物重复。表S4。来自粉末状霉菌(UFGE 4033)的Gerbera成绩单列表,来自Gerbera转录组的差异表达分析的差异(06-245-03)育种线。表S5. 非洲菊抗白粉病(UFGE 4033)和易感(06–245-03)育种系的非洲菊转录本列表,功能性注释为非洲菊转录组中的抗病性。表S6。霉变点列表(枣疯病)在使用粉末状霉菌(UFGE 4033)和易感(06-245-03)育种线构建的Gerbera转录物中存在的转录物。表S7。利用RNA-seq和SAMtools、FreeBayes和GATK管道鉴定了抗白粉病(UFGE 4033)和易感(06-245-03)非洲菊育种系之间的单核苷酸多态性。表S8。利用抗白粉病(UFGE 4033)和易感(06-245-03)育种系和MISA工具对非洲菊转录组中鉴定的简单序列重复序列进行统计。表S9。差异上调的RT-qPCR分析引物R.-非洲菊抗白粉病基因的研究。
附加文件2:图S1。
非洲菊育种系06-245-03(左)易感白粉病(PM)和UFGE 4033(右),耐PM用于rna测序。图。S2。粉状霉菌(PM)症状在丁伯拉A)全植物感染PM B)白色真菌孢子在Capitulum D)上的白色PM孢子在Capitulum d)白色PM孢子在花梗和下花表面观察到的白色PM孢子e)pm conidia在带有40倍的显微镜下观察到的。图S3。利用Blast2GO对非洲菊抗白粉病和感病育种系RNA序列进行功能注释分析。图。S4。使用Blast2GO将非洲菊转录本与viridiplantae数据库进行比较,获得非洲菊转录组的Top-hits物种分布。图S5。术语表显示了Gerbera转录物的频率分布,其中基因本体的数量(GO)与关联。该图是使用BLAST2GO分析创建的。图S6。使用wego2.0分析注释分配给Gerbera转录组的基因本体论术语。图。S7。使用Blast2GO检测非洲菊转录本酶类分布的频率分布。图S8。基因本体(GO) - 富含差异表达的Gerbera转录物的频率分布(GO)-Terms。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/. 知识共享公共领域放弃(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Bhattarai,K.,Conesa,A.,Xiao,S。et al。Gerbera Daisy叶片转录om的测序和分析显示差异表达和粉状霉菌抗性抗病性和易感基因。BMC植物杂志20.539(2020)。https://doi.org/10.1186/s12870-020-02742-020-02742-0
收到:
公认:
出版:
关键字
- 差异表达基因
- 抗病性
- 非洲菊
- 粉状霉菌抵抗力
- R.-基因
- 单核苷酸多态性
- 简单的序列重复
- 易感基因