- 研究文章
- 开放访问
- 发表:
靶向扩增子测序+基于下一代测序的批量分离分析鉴定出普通荞麦收获前发芽耐性相关基因位点(Fagopyrum esculentum)
BMC植物生物学体积21., 文章编号:18.(2021)
抽象的
背景
普通荞麦(2n =2X =16)是一种异交伪谷类,其种子含有丰富的营养物质和潜在的抗氧化剂。由于这些有益的化合物会受到收获前发芽(PHS)的破坏,而PHS可能会随着全球变暖而增加,因此寻找有效的方法开发新的耐PHS品系是很重要的。然而,与荞麦小灵通相关的遗传位点和选择标记尚未见报道。
结果
通过对亲本全基因组的下一代测序(NGS),我们开发了一套全基因组的300个标记。通过基于NGS的批量分离分析(NGS- bsa),我们开发了100个与小灵通耐受相关的标记。为了验证从NGS-BSA数据中开发标记的有效性,我们从之前的NGS-BSA数据中开发了100个与自交亲和性(SC)性状相关的标记。利用这些标记,利用AmpliSeq技术构建遗传图谱,利用基于扩增子的多目标NGS快速检测多态性,并结合NGS- bsa进行小穗小穗抗性QTL分析。通过QTL分析,在耐PHS的‘Kyukei 29’和自亲和易感的‘Kyukei SC7’杂交的分离群体中检测到2个主要和2个次要QTL。在耐phs品系‘Kyukei 28’和‘NARO-FE-1’的分离群体中发现了不同的主、次qtl。NGS-BSA检测到的小灵通候选标记均位于这些QTL区域附近。我们还研究了与这些qtl相关的标记在其他分离群体中选择耐phs品系的有效性。
结论
利用AmpliSeq技术和NGS-BSA结合的方法,高效地构建了荞麦耐采前发芽的遗传图谱,并检测了与普通荞麦耐采前发芽相关的qtl。这是首次鉴定荞麦小灵通抗性qtl。我们的标记开发体系将促进普通荞麦的遗传研究和育种。
背景
常见的荞麦(Fagopyrum esculentumMoench;2n =2X =16)是异种花柱自交不亲和(SI)所致的异交假禾谷类。它广泛生长在世界的温带地区。荞麦种子含有促进健康的化合物,具有抗氧化、抗高血压和抗肥胖的特性[1,2],除了高含量的淀粉和优质蛋白质外,还含有平衡的氨基酸[3.,4].然而,这些有益化合物受到外部效应的强烈影响;特别是,植物的发芽,称为预热萌芽(pHS),严重降低了种子质量。
小灵通通常发生在潮湿和温暖的条件下,通常在收获前(图。1).降低了荞麦粉的糊化粘度和品质,降低了淀粉和粗脂肪的总含量[5,6,7].全球变暖可能会扩大小灵通的范围[8].因此,提高荞麦对小灵通的耐受性是世界范围内的重要育种目标。
通过对低phs个体的群体选择,我们开发了4个耐phs的品种/育种系——Harunoibuki、NARO-FE-1 (NF1)、KY28和KY29 [8,9].为了阐明这些品系的PHS耐受性的遗传,我们利用KY28或KY29与自亲和的PHS易感' Kyukei SC7 ' (KSC7) [8];KSC7是通过引入一个野生亲缘植物的自交亲和性(SC)等位基因,F.homotropicum[10.].在F2在KY28 × KSC7的后代中,小灵通频率的分离模式表明,在KY28中有多个隐性基因调控小灵通的耐性。另一方面,在F2结果表明,KY29 × KSC7基因的分离模式符合两个显性基因的预期比例(15:1),表明KY29的PHS耐性受两个显性基因控制[8].由于荞麦是异交植物,很难确定有利的性状,如小灵通的耐性。尽管标记辅助选择(MAS)是一种有效的方法[10.]、数量性状位点(QTL)和荞麦小穗病抗性选择标记等研究尚未见报道。
最近,我们开发了荞麦基因组数据库(BGDB,http://buckwheat.kazusa.or.jp/)[11.]支持荞麦中的遗传分析和标记发育[12.].我们通过下一代测序(NGS)在F中的下一代测序(NGS)中,开发了链接到侧翼基因的区域的分致标记。基于F的下一代测序(NG)2后代[13.].QTL-seq用于BSA全基因组重测序(WGS),是快速检测qtl的强大工具[14.].但是,它需要一个可以绘制物理贴图的长脚手架数据库。不幸的是,BGDB中的可用参考序列短(N50 = 25.1 kB)[11.],因此有必要构建qtl的遗传连锁图谱。
为了有效地构建遗传连锁图谱,我们需要一个全基因组标记集和一个高效的基因分型系统。Ion AmpliSeq靶向测序技术(Thermo Fisher Scientific, Waltham, MA, USA)可以通过基于扩增子的多重靶向NGS快速检测多态性[15.,16.].在这里,我们开发了具有扩增仪的遗传图谱,并寻求NGS-BSA的荞麦的PHS耐受QTL。我们开发了QTL分析的基因组标记,并检测到与PHS耐受相关的几个QTL。此外,我们开发了链接标记,并调查了与标记的选择的效果。此外,我们通过从连接到SC等位基因链接的标记的标记的扩增仪数据,通过开发来自扩增仪数据的联系地图来证明了NGS-BSA的有效性。我们的调查结果和标记开发系统将有助于推进荞麦育种的遗传研究。
结果
三种群体中pHS的分配频率
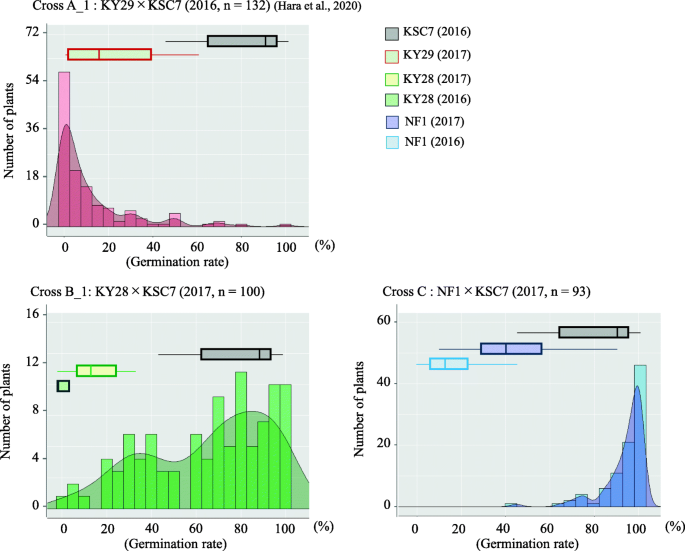
本研究调查了亲本和5个亲本对小灵通抗性的分布频率2每个F1高耐phs品系KY29、KY28和NF1与敏感phs的SC品系KSC7杂交获得的植株(图2)。2;附加文件1和2:表S1, S2)。F2来自KY29 × KSC7 (A_1, A_2)和KY28 × KSC7 (B_2)的居群已经有报道[8].我们在这里调查了KY28 × KSC7 (B_1)和NF1 × KSC7 (C)的衍生物(附加文件)1:表S1)。交叉A_1后代的高率的pHS耐受性表明KY29中的主要显性耐受基因涉及(图。2)[8].杂交B_1后代的耐受率较低,表明KY28的主要隐性耐受性基因参与了这一过程(图2)。2)[8].为了观察新开发的耐phs品系NF1是否具有不同的遗传模式,我们开发了杂交C. Its F2后代表现出对pHs的耐受性很低,表明涉及NF1中的主要隐性耐受基因,如在KY28中那样(图。2).然而,要确定每个群体中还有哪些基因与小灵通耐药相关,还需要QTL分析。
三十字形后代萌发率的分离。一个_1,Kyukei 29(KY29)×Kyukei SC7(KSC7)[8].b_1,Kyukei 28(KY28)×KSC7。cNARO-FE-1(NF1)×KSC7。直方图,植物数量;曲线,核密度估计;盒绘图,父母的发芽率
开发覆盖整个基因组和连锁标记的标记集
为了开发全基因组的AmpliSeq标记,我们首先对Yabe等人的所有微阵列探针序列进行了局部BLAST搜索[17.使用BGDB基因组数据与匹配序列找到脚手架。在所有1129个探针序列中,1063探针匹配八个连杆组中分布的387个支架(附加文件3.:表S3)。我们使用这些387支架作为全基因组支架。从这些SNP位点中,我们选择了300个SNP位点作为全基因组SNP进行AmpliSeq分析(附加文件)4:表S4)。
为了开发pHS连接的标记集,我们比较了在F中的高pHS耐受体和植物的低pHS耐受大部分DNA之间的序列2KY29×KSC7(A_1)的人口,并获得535个具有高值的PHS连接的SNP指数的支架(≥0.700;附加文件5:表S5)。在这里,我们选择了100个支架,并对其连接关系进行了研究。
为了研究NGS-BSA的效果和确定性,我们制作了一个与SC性状相关的AmpliSeq标记集,年代h.我们已经在轨迹处执行了NGS-BSA,并开发了Codominant标记,但我们只使用了前50个候选脚手架[13.].在这里,我们在先前公布的KSC7(LH)和KY29(PIN)的WGS数据中选择了100个具有高SNP索引的脚手架。13.],并研究了它们之间的联系。
最后,我们开发了一个500个标记的定制面板,其放大了100个PHS连接的SNP,100年代h- 链接的SNP和300个基因组SNP(附加文件6:表S6)。
利用AmpliSeq在三个F中构建连锁图谱2人口
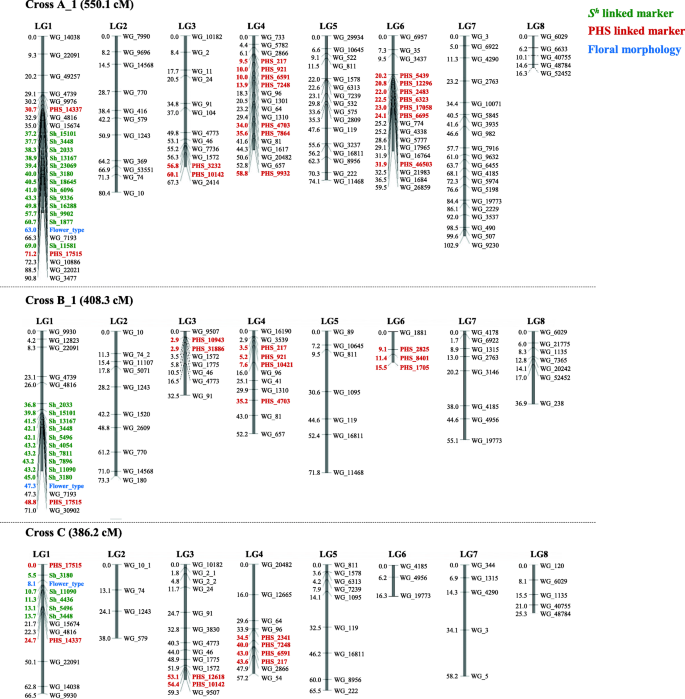
使用该500标记集,我们通过扩增Q衍生自交叉A_1,B_1和C的基因分型后代。通过R / QTL滤出所有基因分型数据,并构建连接图(图。3.).不幸的是,几个标记没有显示SNP,因此不能用于映射。这可能是因为BGDB参考基因组含有大量未识别的核苷酸序列'n',这阻止消除离子扩增设计器中的脱靶杂交(附加文件10.:图S1)。所有检测到的多态性和可用的标记汇总在表中1.所有三个群体都有八个连杆组(LGS),总图长度为550.1cm,在B_1中的408.3cm,3.36.2厘米的C中,C(图。3.).
花的形态(LH/pin)标记,在地图上标记为“Flower_type”,位于LG1(图1)。3.).正如预期的那样,最年代hNGS-BSA开发的-连锁标记在其周围的所有群体中进行了定位。我们省略了45年代h- 在滤波期间,在B_1,B_1,40中的交叉A_1,40中的交叉A_1,40中的标记为38中的标记(表1),所得到的年代h- 链接的标记与之紧密相关年代h结果表明,NGS-BSA检测荞麦单主基因控制性状的有效性和准确性。
用所有标记映射的结果年代h-连锁标记表明,AmpliSeq标记开发系统功能良好,可用于PHS分析。2/ 3(32/48)的phs连锁标记聚类并定位在A_1的LG6上,多个标记分别定位在LG1、LG3和LG4上(图1)。3.;桌子1).此外,杂交A中32个PHS连锁标记中有25个在LG6上重复缺失,表明KY29的PHS耐受主要遗传位点可能位于LG6上。
小灵通抗性QTL分析
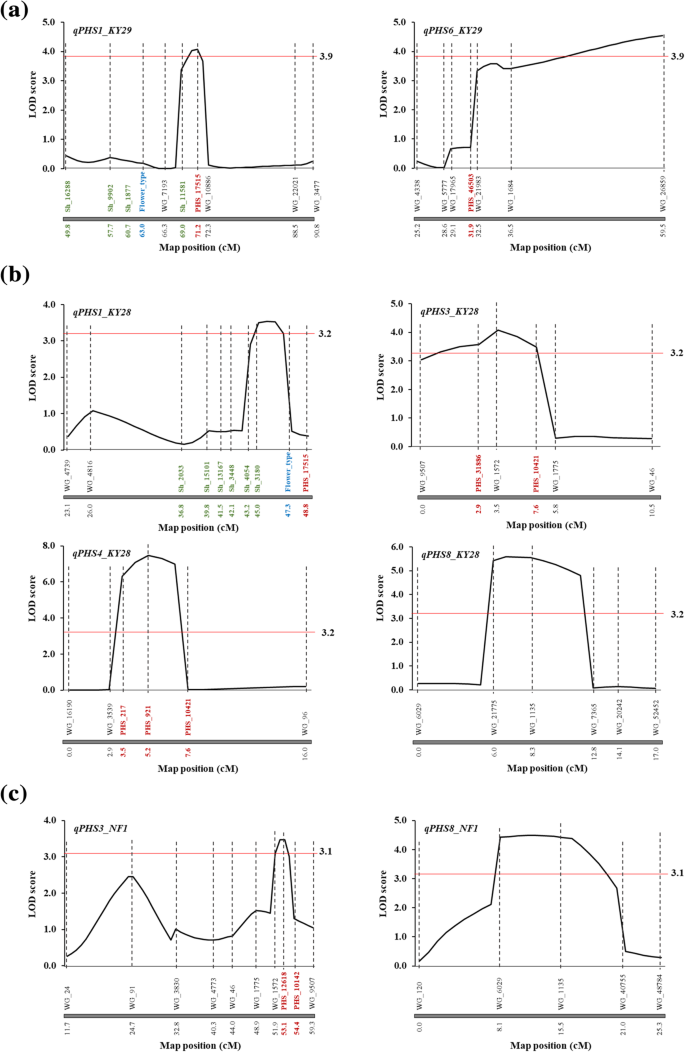
通过复合间隔映射(CIM)对PHS容忍进行了QTL分析。对数似然性的阈值(LOD)意义(P< 0.05), A_1为3.9,B_1为3.2,c为3.1。A_1检测到2个主要qtl和2个次要qtl (LOD阈值±≥2.0)2,无花果。4),并解释了42.2%的表型变异(表2).法QPHS1_KY29,qPHS6_KY29,qPHS4_KY29获得KY29基因型的耐受性qPHS7_KY29具有KSC7基因型的耐受性。qPHS6_KY29影响最大(15.6%),占显著优势。B_1检测到4个主要qtl和1个次要qtl2,无花果。4),并解释了66.4%的表型变异。法qPHS3_KY28和qPHS4_KY28获得KY28基因型的耐受性Qphs1_ky28,QPHS5_KY28,qPHS8_KY28获得KSC7基因型的耐受性。qPHS4_KY28效应最大(22.0%),呈隐性。在C中检测到2个主要qtl和1个次要qtl(见表)2,无花果。4),并解释了38.0%的表型变异。法qPHS2_NF1和QPHS3_NF1授予耐NF1基因型,并且qPHS8_NF1具有KSC7基因型的耐受性。QPHS3_NF1和qPHS8_NF1位于标记区域(WG_1572和WG_1135)附近qPHS3_KY28和qPHS8_KY28(表2).
我们在F2A_1的后代位于LGS 1,3,4和6上(表1).在这些标记附近检测到具有KY29基因型的小灵通抗性qtl(图2)。2、表2), LG3除外。此外,许多phs连锁标记聚类并定位在LG6上,在LG6上检测到效果最大的QTL (qPHS6_KY29;桌子2)在a_1。这些结果表明,由NGS-BSA开发的pHS链接标记有效地检测到pHS耐受性的遗传区域。
利用序列标记位点标记验证qtl对小灵通抗性的影响
为了确定主要qtl与种子发芽率之间的相关性,我们在杂交组合A_1、A_2、B_1和B_2分离群体中,对最接近STS (sequence-tag -site, STS)的标记进行了转化,研究了它们的基因型与种子发芽率之间的关系(表)3.).最接近QPHS1_KY29(lg1)和qPHS6_KY29LG6分别为PHS_17515和WG_26859。3.、表2);转化为STS标记qPHS1_KY29_PHS_17515和QPHS6_KY29_WG_26859.(附加文件7:表S7)。从KSC7和KY29中提取的每个标记的等位基因分别标记为A和B(表1)3.).在A_1,植物纯合QPHS1_KY29_B.和qphs6_ky29_b.萌发率低于纯合的植物QPHS1_KY29_A.和QPHS6_KY29_A.(表3.).虽然A_2的效果不显著,但效果相似(表1)3.).在A_1中,植物的平均萌发率杂合qPHS6_KY29与Ky29等位基因纯合的植物植物几乎是一样的,这表明KY29等位基因在qPHS6_KY29是占主导地位(表3.).另一方面,KY29等位基因在QPHS1_KY29部分占主导地位(表3.).
在B_1和B_2中,最接近Qphs1_ky28(lg1),qPHS3_KY28(lg3),qPHS4_KY28(lg4),和qPHS8_KY28(LG8)被转换为STS标记(附加文件7:表S7)。植物的KY28等位基因纯合在qPHS3_KY28(qPHS3_KY28_B)萌发率低于纯合的KSC7等位基因(qPHS3_KY28_一个)都在B_1和B_2中。KY28等位基因在qPHS4_KY28(qPHS4_KY28_B)的发芽率也低于纯合子植株(qPHS4_KY28_一个在B_1中,但在B_2中没有3.).另一方面,植物纯合qPHS8_KY28_B萌发率更高(表3.).qPHS3_KY28和qPHS8_KY28B_1和B_2的萌发率显著降低(表2)3.).在B_1中,植物杂合子的平均发芽率为qPHS3_KY28和qPHS4_KY28略低于KSC7等位基因纯合植株的等位基因,说明这些位点上的KY28等位基因是隐性或部分显性的(表3.).
讨论
荞麦遗传图谱的快速构建
基因组数据库的发展使得利用BSA (QTL-seq)对水稻、大麦和拟南芥等主要作物和模式植物进行QTL分析变得容易[18.,19.,20.].通过在荞麦中的WGS,我们开发了BGDB [11.[但到目前为止,它的小型脚手架尺寸可防止其在QTL-SEQ分析中使用。此外,为了利用QTL分析的遗传图,目标特征必须在隔离群体中分离。因此,遗传图的构建仍然是荞麦QTL分析的第一步。然而,覆盖整个基因组的地图的构建需要很多努力,特别是在植物种类中。
扩增技术通常用于癌症研究[21.]并开始用于农学。Sato等。[22.]开发了一种高度灵活和可重复的基于ampliseq的全基因组基因分型系统,用于水产养殖研究,以加强种群遗传研究和全基因组关联研究。然而,目前还没有在作物中使用AmpliSeq构建全基因组图谱的报道。本研究利用荞麦杂交A_1 (KY29和KSC7)亲本的WGS数据,构建了一个包含300个标记的全基因组序列。这些标记不仅可以检测到A_1的SNPs,还可以检测到B_1和C的SNPs,这些SNPs均位于三个种群的8个LGs上(见表)1),可能由于栽培荞麦内的高核苷酸多样性(π= 0.0065)[23.].该结果表明该基于扩增基因的基因型基因分型系统可以在荞麦中有效和快速地构建遗传联系地图。
此外,所构建的QTL图谱和标记可用于其他性状的QTL分析,如光周期敏感性和类黄酮含量,尽管这些性状在分离群体中需要分离。此外,通过比较株系和地方品种间的SNP/indel WGS数据,增加标记的数量对精细定位和更详细的QTL分析具有重要意义。
NGS-BSA + AmpliSeq为荞麦qtl的鉴定和选择标记的开发提供了一种有效的方法
由于我们所开发的荞麦草图基因组序列仍然被划分为387,594个scaffold,所以无法在物理图上显示QTL峰。本研究利用NGS-BSA结合AmpliSeq高效快速地识别小PHS的qtl,并建立紧密连接的标记。
由NGS-BSA开发的48个phs链接标记位于LGs 1、3、4和6(图1)。3.、表1).其中2/ 3(32/48)被聚类并定位在LG6上,包括一个与效应最大的QTL (qPHS6_KY29)在交叉A_1(表1和2).由于在F2交叉A_1的后代,我们预计它们将在衍生自KY29的区域中检测到,这对PHS抗性有贡献。在LGS 1上检测到与KY29基因型提供pHS耐受性的QTL(QPHS1_KY29),4(qPHS4_KY29)及6 (qPHS6_KY29)的交叉A_1(表2).另一方面,在LG7上检测到一个对KSC7基因型具有耐受性的QTL (qPHS7_KY29)(表2).因此,QTL分析与NGS-BSA的结果一致,并表明扩增性测序很好地检测源自KY29的pHS耐受性的QTL。如果我们只使用NGS-BSA开发联动标记,我们将不知道这些标记的链接关系,无法选择有效的标记,因为我们无法从链接到不同QTL链接的每个区域中选择标记。
我们还开发了100年代h-连锁标记,并将其定位在与小灵通QTL分析相同的图谱上,研究其遗传区域。所有年代h-连锁标记被定位在LG1的花形态标记附近(图1)。3.、表1).这种NGS-BSA靶向一个主要基因座表明该方法对于开发紧密连接的标记非常有效。因此,具有扩增性的NGS-BSA的组合是识别常见荞麦的定性和定量性状的遗传区域的有效方法。
用于PHS耐受性的新型主导和隐性QTL的检测和起源
我们之前分析了F中的PHS耐受性的分离2源自KY29×KSC7和KY28×KSC7的种群,并确定了KY29和KY28之间的不同继承模式[8].正如我们所预期的那样,KY29和KY28具有不同的QTL,提供pHS耐受性(图。3.、表2).QTL分析和关联分析表明,KY29的小灵通抗性受显性等位基因控制qHS6-KY29和部分主导的等位基因qHS1-KY29,符合我们的期望[8].由于KY29是“Kanoya-Zairai”和“Hitachiakisoba”的杂交产物,它的PHS耐受等位基因可能来自“Kanoya-Zairai”,一个日本的地方品种,具有很高的耐药性[24.].
另一方面,KY28的PHS耐受性受隐性或部分显性等位基因控制qPHS3_KY28和qPHS4_KY28(表2和3.).在杂交组合B_1和C中,在LG3 (qPHS3_KY28和QPHS3_NF1)和LG8(qPHS8_KY28和qPHS8_NF1).KY28是由PHS宽容'HARUNOIBUKI'开发的[9]×'Hitachiakisoba'和NF1是从'Kitawasesoba'中的复合交叉开发的[25.],'yaita-zairai','asahimura-zairai 3','hashikamiwase','hitachiakisoba','chushinkei vii','kyukei 30'和'kyukei 10'。'Harunoibuki'是由“Hashikamiwase”的低位低位人物的大规模选择[9].所以小灵通的耐受等位基因qPHS3_KY28和QPHS3_NF1可能源于“Hashikamiwase”。
我们检测到QTL,其在三个十字架中使用KSC7基因型提供pHS耐受性(qPHS7_KY29,Qphs1_ky28,QPHS5_KY28,QPHS8_KY28,qPHS8_NF1;桌子2).KSC7已从'Norin-PL1'开发[26.)由F. Esculentum.×F.同性恋;后一种物种具有强大的种子休眠[27.].但在KSC7的发育过程中,我们选择了发芽率较低的品系,以避免强的种子休眠以加速世代。在育种过程中,对小灵通发芽率影响较大的遗传区域可能已被消除,但仍可能保留一些弱的小灵通耐受基因。这些qtl可能有助于提高小灵通的耐受性。此外,为了寻找新的小灵通的耐受等位基因F.同性恋,虽然必须去除诸如破碎习惯的不希望的特性,但是必须去除不希望的特征可能是有用的。
小灵通抗性基因在育种中的应用
广泛的工作已经确定了许多植物种类的pHS耐受性的基因或主要显性和隐性等位基因,例如小麦,大麦,水稻,高粱和拟南芥[28.,29.,30.,31.,32.,33.,34.,35.].例如,QPhs.ocs-3A.1(确定为MOTHER-OF-FT-AND-TFL1在小麦、Sdr4(Os07g0585700),和延迟GERMINATION1(AT5G45830)为显性或半显性[28.,29.,30.].另一方面,QSD1.(丙氨酸转氨酶在大麦,QSD2-AK.(丝裂原活化蛋白激酶激酶:MKK3.)在大麦,和PHS1.(MKK3.)小麦是隐性的[31.,36.,37.].在小麦和大麦中QPhs.ocs-3A.1,QSD1.,QSD2-AK.,PHS1.适用于MAS [34.].
使用BGDB,我们调查了我们识别的QTL附近是否存在候选基因(附加文件8和9:表S8,S9)。QTL所定位的所有脚手架都包含几个开放阅读框架(ORFS),除FES_SC0026859.1之外(附加文件8:表S8)。一些ORFS由BGDB注释(附加文件9:表S9),但没有候选基因似乎与PHS直接相关。但是,因为我们无法为该实验中的所有支架开发标记,所以其他支架可能存在于QTL附近。需要进一步研究以识别控制每个QTL的基因及其功能。
由于普通荞麦属单株植物,显性等位基因在选择有利性状方面更有用。另一方面,如果MAS能有效地固定有利基因型,隐性等位基因也能发挥作用。例如,qPHS3_KY28_WG_1572标记可以有效地选择耐phs的后代,从而选择纯合子的KY28等位基因(表)3.).这意味着MAS的金字塔可能是有用的。该信息和标记物可用于加速遗传育种,以改善荞麦中的pHS耐受性。
结论
利用AmpliSeq技术高效构建连锁图谱,并结合NGS-BSA对普通荞麦小穗PHS抗性进行QTL分析。NGS-BSA检测到的小灵通候选标记均位于这些QTL区域附近。此外,所有候选标记都与单一遗传位点相连年代h由NGS-BSA开发的也映射到附近的位置。因此,我们已经表明,NGS-BSA具有扩增性的组合是识别遗传区域的有效方法,以常见荞麦的定性和定量性状。我们在这里检测到的QTL具有具有不同遗传模式的耐受性等位基因:主导,部分主导和隐性。这些等位基因可以用STS标记用MAS有效地选择用于育种计划。这是首次鉴定荞麦小灵通抗性qtl。我们的标记开发体系将促进普通荞麦的遗传研究和育种。
方法
植物材料
荞麦是一种具有两种类型的花卉架构的异常Si种类:Thrum(短风格)和别针(长风格)[38.].这个SI系统由一个单一的遗传位点控制,年代;线头是杂合的(SS.)和pin是纯合隐性的(SS.).我们从普通荞麦,F. Esculentum.,以及一个自相互态的野生相对,F.同性恋[10.].SC线具有由单个等位基因控制的长套装(LH),年代h,在优势关系中年代>年代h>年代[39.].为了开发隔离群体,我们使用了SC线'Kyukei SC7'(KSC7),该组织由我们的SC线'Norin-Pl1'的研究组开发了[26.].
我们选用了3个耐phs的品种/育种系:‘菊系29’(KY29)、‘菊系28’(KY28)和‘NARO-FE-1’(NF1);这些是我们研究小组开发的。KY29和KY28对小灵通的抗性显著高于日本和KSC7的优势品种,评价比较稳定[5,8,24.].
我们开发了五个隔离人群(附加文件15个耐phs系(KY29_1, KY29_2, KY28_1, KY28_2, NF1)与KSC7的独立杂交。Hara等报道了杂交组合A_1 (KY29_1 × KSC7)、A_2 (KY29_2 × KSC7)和B_2 (KY28_2 × KSC7)对小PHS耐性的分离模式。8杂交组合B_1 (KY28_1 × KSC7)和C (NF1 × KSC7)均为新报道。进行连锁图谱构建和QTL分析,分离出F2使用从A_1,B_1和C导出的群体。用于对每个QTL的最近标记的关联测试,分离F.2使用从A_1,A_2,B_1和B_2导出的群体(附加文件1:表S1)。
小灵通公差评价
F2十字架A_1的群体(n = 132), A_2 (n= 106), b_1 (n= 100, b_2 (n= 106), C (n = 93) and each parental line were grown in a field of the Institute of Crop Science, NARO, Tsukuba, Japan, in 2016 (KY28, NF1, KSC7, A_1, A_2) and 2017 (KY28, KY29, NF1, B_1, B_2, C). Because PHS tolerance evaluated in the field is highly correlated with that evaluated in a Petri dish [8],且各品系间值稳定,我们在培养皿中测试PHS耐受性,如所述[8].简单地说,每一种植物的20个新鲜的种子被立即放在培养皿中浸透蒸馏水的滤纸上。培养皿在恒温25°C的黑暗环境下在发芽箱中培养。每天检查一次培养皿,连续7天,清点发芽的种子并取出。结果是百分比。亲本间差异的显著性用Tukey-Kramer检验,采用multicomp R软件包(附加文件)2:表S2)。
用于AmpliSeq分析的全基因组标记开发
用DNeasy plant Mini Kit (Qiagen, Hilden, Germany)从每一株植物的幼叶中分离基因组DNA。我们开发了覆盖整个基因组的全基因组标记、phs相关标记(下一节)和sc相关标记(下一节)。为了开发全基因组标记,我们使用了Yabe等开发的标记的序列信息[17.]在DNA微阵列的高密度连杆地图上。我们对用于构建联动地图作为BGDB参考基因组(FES_R1.0)的查询来执行所有微阵列探测序列的本地BLAST搜索。搜索由Galaxy Blastn v.2.7.1执行默认设置。在确定每种微阵列探针的支架数之后,我们将这些支架评估为基因组宽的支架。从KY29和KSC7(A_1的父母)之间的这些和重新排列数据,我们选择了300个SNP网站。
NGS-BSA为PHS和SI/SC开发标记
为了开发pHS连接的标记,我们将膨胀DNA从46个高度pHS耐受性植物(0%pHS)和来自10个具有低pHS耐受(> 50%pH)的植物中的DNA均来2交叉A_1的总体。在日本Macrogen公司(京都,日本;DDBJ/EMBL/NCBI登录号PRJDB9892)。为了研究NGS-BSA的作用和稳定性,我们制作了一套与SC等位基因相关的标记年代h来自以前发表的KSC7和KY29的WGS数据(accession number DRX178921) [13.].
在TrimMomatic v中修剪低质量读取和适配器(CacgacGctCtCCGATCTCTGTAA)0.32软件[40]带有头像2,SlidingWindow 4:25,前进25,尾随25和Minlen:40。修剪读取被映射到BWA v中的参考序列。0.7.15软件[41.]使用“BWA ALN”选项(-L 32,-K 2,-N 5)和“BWA示例”选项(默认设置)。只有基因组序列[11.选择≥1kb作为参考序列。在SAMTOOLS v中处理映射结果。0.1.18软件[42.].
用GATK v中的统一预替替替换工具检测SNP。3.7软件[43.使用-glm BOTH选项。检测PHS抗性连锁SNPs的标准如下:(1)KSC7和KY29之间的SNPs,深度> 10;(2)高耐phs膨化DNA杂合子;(3)与KSC7核苷酸相同的纯合子,在低ph耐受的膨化DNA中。BGDB中的参考序列被划分为387,594个短支架(N50 = 25.1 kb) [11.,并且很难通过支架描述高ph -耐受性相关SNP位点数量的变化。因此,我们统计了所有参考序列中的SNPs,并计算了PHS-linked SNPs的数量与所有SNPs的比例,作为每个支架的“PHS-linked SNP指数”。利用小灵通和SI/SC候选基因的前100个标记进行定位。一个针对500个区域的定制面板(300个为全基因组,100个为phs连接,100个为年代h基于KSC7定制参考基因组,使用Ion AmpliSeq设计器(https://ampliseq.com/help/startdesign.action.action.)[44.]版本6.0,使用标准DNA (125-275 bp扩增靶尺寸)选项。
扩增库的构建,测序,变体检测和基因分型
AmpliSeq库是用Ion AmpliSeq library Kit 2.0和IonCode Barcode Adapter 1-384 Kit(均来自Thermo Fisher Scientific)制备的,如Ogiso-Tanaka等人所述[15.].在Mosquito HV仪器(TTP Labtech, Royston, UK)上进行多重PCR扩增,总反应量为5 μL手动或4.8 μL机器人[45.],每个基因组DNA的含量为10ng (n= 100在A和B交叉,n = 93 in C cross). The Ion Library Equalizer Kit was used to normalize the library concentration to 100 pM, and libraries were pooled and sequenced on an Ion Torrent S5 system (Thermo Fisher Scientific). Template preparation (emulsion PCR, enrichment of beads containing template, and chip loading) was performed with the Ion Chef instrument and Ion S5 Kit-Chef according to the manufacturer’s instructions. After the preparation of ion sphere particles, sequencing for 500 cycles was performed on an Ion Torrent S5 system using an Ion 540 Chip according to the manufacturer’s instructions. The sequence data were mapped to the KSC7 custom reference genome by the Ion Torrent Mapping Alignment Program v. 5.8.0 in Torrent Suite v. 5.8.0 software. Coverage analysis and variant detection were performed in Coverage Analysis v. 5.8.0.8 and variantCaller v. 5.8.0.19 software with default parameters (Germ Line with low stringency). All detected variants were listed in a hotspot VCF file. Variants at hotspot sites were detected in variantCaller with default parameters. Finally, the genotype file for the R/qtl package (http://www.rqtl.org/)[46.通过IonBreeders ABH插件variantCaller的输出文件转换得到[47.].
遗传联系地图的构建和QTL分析
在构建链接图之前,我们如下预处理数据,如下所示:(1)找到重复标记,在缺少值,除了缺少值外,可以在所有单个中显示相同的基因分型,选择具有最少缺失值的标记,并删除重复标记。(2)用<80基因型滴样品。(3)落下标记子集,其中<90%的人口是基因分型。(4)具有异常基因型分布的滴标记。遗传图在AntMap v.1.2软件中构建48.].QTL分析在WinQTL Cartographer v. 2.5软件中使用复合区间制图(CIM)模型[49.].log likelihood (LOD)评分的显著性阈值基于1000个排列(P = 0.05).
与qtl相关的STS标记的开发
根据KY29和KSC7之间的重置数据或来自SAMPAXISEQ的数据,我们将QTL最近的SMANXISEQ标记转换为STS标记。用基因组DNA扩增作为模板,通过设计的特异性引物进行ExTaq(Takara,Shiga,Japan)如下:30 s,58°C的32个循环30 s,72°C为30 s。通过琼脂糖凝胶电泳证实扩增,用限制酶消化DNA片段(AfaI,alui.,eCORI., 要么MSPI.(所有从豆类))。引物序列和酶的组合列于附加文件7:表S7。
数据和材料的可用性
本研究期间生成或分析的所有数据都包含在稿件及其附加文件中1- - - - - -10..本研究获得的基于下一代测序的散装分离分析的原始reads可从DDBJ/EMBL/NCBI上获得,登录号为PRJDB9892 (http://trace.ddbj.nig.ac.jp/bpsearch/bioproject?acc = prejdb9892.).在本研究中使用和/或分析的数据集可从通讯作者在合理要求。
缩写
- BGDB:
-
荞麦基因组数据库
- BSA:
-
Bulked-segregant分析
- CIM:
-
复合区间映射
- KSC7:
-
'kyukei sc7'
- KY28:
-
“Kyukei 28日”
- KY29:
-
“Kyukei 29日”
- LG:
-
连锁群
- LH:
-
长homostyle
- LOD:
-
对数似
- MAS:
-
标记辅助选择
- NF1:
-
'naro-fe-1'
- 门店:
-
新一代测序
- ORF:
-
开放阅读框架
- PHS:
-
收获前的萌芽
- QTL:
-
定量特质基因座
- SC:
-
自交
- 如果:
-
Self-incompatibility
- STS:
-
Sequence-tagged-site
- WGS:
-
全基因组重新排列
参考
- 1。
陈志强,周敏,郭立峰,等。培育营养品质荞麦。Sci繁殖。2020;70:67 - 73。
- 2。
黄志强,王志强。荞麦中黄酮类化合物的合成与调控。Sci繁殖。2020;70:74 - 84。
- 3.
普通荞麦和苦荞麦麸皮的组成及工艺特性。食品化学。2003;80:9-15。
- 4.
Suzuki T,Noda T,Morishita T,Ishiguro k,Otsuka S,Brunori A.提供荞麦质量育种的现状和未来视角。品种sci。2020; 70:48-66。
- 5。
关键词:荞麦,采前发芽,面粉糊化粘度Fagopyrum esculentumMoench)。植物保护学报2007;10:361-6。
- 6。
Hara T, Sasaki T, Tetsuka T, Ikoma H, Kohyama K.发芽对熟荞麦质地的影响(Fagopyrum esculentumMoench)面条。植物保护学报2009;12:492-6。
- 7.
高L,XIA M,Li Z,王P,王M,高J.萌发过程中常见荞麦淀粉的物理化学特性及相关分析的变化。Fagopyrum。2019; 36:43。
- 8.
Hara T,Takeshima R,Matsui K.具有不同遗传模式的基因调节在预热 - 培养的荞麦线中的种子萌发(Fagopyrum esculentum).JPN Agric Res Q. 2020; 54:137-43。
- 9.
荞麦新品种“春香”。Bull NARO Kyushu冲绳农业Res Cent. 2012; 58:37-47。
- 10.
关键词:荞麦,自交不亲和,遗传,基因组Fagopyrum esculentum).Theor Appl Genet. 2020; 133:1641-53。
- 11.
安井,平川,上野,松井,田中胜,杨世杰,等。荞麦基因组草图的组装及其在鉴定农艺有用基因中的应用。DNA研究》2016;23:215-24。
- 12.
普通荞麦遗传标记系统的发展历程。Sci繁殖。2020;70:13-8。
- 13。
Matsui K,Mizuno N,Ueno M,Takeshima R,Yasui Y.与与自相容性基因座有关的有关的有关的有关的共同主导标记的开发(年代)以荞麦(Fagopyrum esculentum).品种sci。2020; 70:112-7。
- 14。
高木,安倍,吉田,小杉,夏目漱石,三冈,等。QTL-seq:通过对两个大群体的DNA进行全基因组重测序,快速定位水稻数量性状位点。植物j . 2013; 74:174 - 83。
- 15.
高度多路扩增技术鉴定大豆开花时间相关基因的新变异(大豆).DNA研究》2019;26:243-60。
- 16。
Tanaka T,Ishikawa G,Ogiso-Tanaka E,Yanagisawa T,Sato K.通过基于参考的RNA-SEQ分析进行大麦的基因组SNP标记的开发。前植物SCI。2019; 10:1-9。
- 17。
叶彬,田原,上野,Enoki H,木村,西村,等。利用DNA微阵列快速基因分型技术进行普通荞麦高密度连锁定位和QTL定位(Fagopyrum esculentumMoench)。品种sci。2014; 64:291-9。
- 18.
Daware A, Das S, Srivastava R, Badoni S, Singh AK, Agarwal P,等。利用SSR标记和先进的QTL-seq驱动的QTL定位分析水稻籽粒重的候选基因。植物科学学报2016;7:1-17。
- 19.
张旭,王伟,郭宁,张勇,卜勇,赵军,等。结合QTL-seq和连锁作图,精细定位一个具有株高特征的野生大豆等位基因。BMC基因组学。2018;19:226。
- 20.
刘涛,王军,吴超,张勇,张旭,李旭,等。将QTL-Seq与传统连锁分析相结合,对萝卜肉质主根紫色皮的候选基因进行了鉴定。麝猫。2019;10:1-11。
- 21.
Tsongalis GJ, Peterson JD, de Abreu FB, Tunkey CD, Gallagher TL, Strausbaugh LD等。常规使用离子洪流AmpliSeq™癌症热点面板鉴定临床可操作的体细胞突变。临床化学实验室医学。2014;52:707-14。
- 22.
Sato M,Hosoya S,Yoshikawa S,Ohki S,Kobayashi Y,ITou T等人。基于使用下一代测序技术的基于靶扩增子测序的水产养殖研究的高度灵活和可重复的基因分型方法。SCI批准。2019; 9:6904。
- 23。
关键词:基因流动年代栽培荞麦的等位基因区域。BMC Plant Biol. 2019;19:125。
- 24。
Hara T,Tetsuka T,Matsui K,Ikoma H,Sugimoto A,Shiratsuchi H.评估普通荞麦的预见阅览室发芽中的品种差异(Fagopyrum esculentumMoench)。植物刺激科学。2008; 11:82-7。
- 25。
犬山,本田y,弗鲁山s,kimura m,卡萨诺h。荞麦品种的繁殖和特征,'kitawasesoba'。Res Bull Hokkaido Natl Agric Exp Stn。1994年; 159:1-10。
- 26。
Matsui K,Tetsuka T,Hara T,Morishita T.一种新的自相互态荞麦父母线的育种和特征,“荞麦Norin-PL1”。公牛Natl Agric Res Cent Kyushu冲绳厅。2008; 49:1-17。
- 27。
王子,坎贝尔CG。在荞麦2000中打破休眠。Fagopyrum。2000; 17:45-50。
- 28。
关键词:克隆狗,一种控制拟南芥种子休眠的定量特质基因座。PROC NATL ACAD SCI。2006; 103:17042-7。
- 29。
引用本文Sugimoto K, Takeuchi Y, Ebana K, Miyao A, Hirochika H, Hara N, et al.。水稻种子休眠与驯化调控因子Sdr4的分子克隆。中国科学院院刊2010;107:5792-7。
- 30.
中村S,安倍F, Kawahigashi H, Nakazono K, Tagiri A, Matsumoto T,等。FT和TFL1的母系同源物在调控萌发中起作用。植物细胞。2011;23:3215-29。
- 31.
佐藤K,山根M,山二N, Kanamori H, Tagiri A, Schwerdt JG,等。丙氨酸转氨酶控制大麦种子休眠。Nat Commun。2016;7:1-9。
- 32.
Lee Ga,Jeon Ya,Lee Hs,Hyun Dy,Lee Jr,Lee Mc,等。基于大米模型方程的新遗传基因群及其评价。前植物SCI。2017; 8:1-9。
- 33.
Benech-Arnold RL, Rodríguez MV。收获前发芽和粮食休眠高粱二色的:我们学到了什么?植物学报。2018;9:1-8。
- 34.
Nakamura S.谷物休眠基因负责预防大麦和小麦的收获前萌芽。品种sci。2018; 68:295-304。
- 35。
Vetch JM, Stougaard RN, Martin JM, Giroux MJ。六倍体小麦采前发芽的遗传机制研究进展(小麦L.)。植物SCI。2019; 281:180-5。
- 36。
中村村,陈志强,陈志强,等。大麦丝裂原活化蛋白激酶激酶3调控种子休眠。咕咕叫医学杂志。2016;26:775 - 81。
- 37。
等。小麦4A染色体种子休眠的原因基因编码MAP激酶激酶。咕咕叫医学杂志。2016;26:782-7。
- 38。
Garber RJ, Quisenberry KS。荞麦花柱长度的遗传。中国农业科学1927;34:181-3。
- 39。
Woo SH, Adachi T, Jong SK, Campbell CG。种间杂交荞麦自交亲和性和花形态的遗传。植物学报1999;79:483-90。
- 40.
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学。2014;30:2114-20。
- 41.
李洪波,李洪波。基于盲轮变换的短读快速定位。生物信息学。2009;25:1754-60。
- 42.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N等人。序列对齐/地图格式和samtools。生物信息学。2009; 25:2078-9。
- 43.
王志强,王志强,王志强,等。利用下一代DNA测序数据进行变异发现和基因分型的框架。Nat麝猫。2011;43:491-8。
- 44.
离子AmpliSeq设计师。https://ampliseq.com/help/startdesign.action.action..
- 45.
Ogiso-Tanaka E,Kaga A,Hajika M.用于下一代测序的成本效益扩增图书馆准备的小型化技术。J Appl Microbiol Res。2018; 1:43-9。
- 46.
吴海,吴海涛,吴海涛。R/QTL:试验组合的QTL定位。生物信息学。2003;19:889 - 90。
- 47。
Ogiso-Tanaka E,Yabe S,Tanaka T. Ionbreeders:自动化生物信息学辅助育种的基因组学。品种sci。2020;https://github.com/DEMETER298/IonBreeders.
- 48。
IWATA H,Ninomiya S. Antmap:使用蚁群优化算法构建遗传联系地图。品种sci。2006; 56:371-7。
- 49。
王sc,basten j,zeng z-b。Windows QTL Cartographer 2.5。罗利:北卡罗来纳州立大学统计系;2012年。http://statgen.ncsu.edu/qtlcart/wqtlcart.htm..
致谢
我们感谢NARO的吉冈Toru和Aburano Mineko种植荞麦植物并支持提取DNA样本。部分计算是在ROIS国家遗传学研究所的NIG超级计算机上完成的。
资金
这项工作得到了NARO的支持,部分得到了JSPS KAKENHI赠款第18号K14448号给RT,第18H02177号给YY和KM。作者声明,资助机构在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有作用。
作者信息
从属关系
贡献
RT和KM产生分离群体,研究小灵通的耐受性。RT和EOT进行AmpliSeq分析。YY NGS-BSA执行。RT进行QTL分析,研究所开发标记的有效性。RT, EOT, YY, KM撰写稿件。所有作者都已阅读并批准了手稿的最终版本。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
植物材料清单。
附加文件2:表S2。
亲本采前发芽率。
附加文件3:表S3。
与阵列标记序列相关联(Yabe等,[17.)和BGDB支架。
附加文件4:表S4。
在微阵列标记序列之间选择300个SNP位点(Yabe et al., [17.)和BGDB支架。
附加文件5:表S5。
基于SNP指数的基于PHS耐受的支架鉴定。
附加文件6:表S6。
SAMPERSISQ自定义面板的摘要信息。
附加文件7:表S7。
qtl序列标记位点的引物信息。
附加文件8:表S8。
QTL所在的支架和每个QTL最接近的标记。
附加文件9:表S9。
基因在QTL附近注释。
附加文件10:图S1。
靶上速率和均匀性的箱形图。目标上的速率显示为对齐的读的百分比。覆盖率均匀性评分以≥0.2×平均覆盖深度的面积百分比表示。箱形图显示中位数和四分位范围;红色,十字A (n= 94);绿色,十字B (n= 87);蓝色,十字C (n= 84)。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Takeshima,R.,Ogiso-Tanaka,E.,Yasui,Y。et al。靶向扩增子测序+基于下一代测序的批量分离分析鉴定出普通荞麦收获前发芽耐性相关基因位点(Fagopyrum esculentum).BMC植物BIOL.21,18(2021)。https://doi.org/10.1186/s12870-020-02790-w
收到了:
接受:
发表:
关键字
- 配种
- 遗传地图
- 基因组数据库
- 重新排序
- 标记辅助选择
- QTL-SEQ.