抽象的
背景
对比叶绿体基因组学可以在密切相关的物种中建立塑性多样性的主要进化事件中阐明。Polypodeae家族是最丰富的富含物种和望远镜的外延蕨类群体之一。通常认识到,在其组织稳定性方面,息藻膜的塑料非常值得注意。因此,尚未对息藻基肌的基因组结构变异进行研究。
结果
完整的塑料序列Neolepisorus Fortunei.,Neolepisorus ovatus,和Phymatosorus cuspidatus根据下一代测序确定。与已发表的塑料一起,进行了对比较分析的聚吡氏菌塑料的细结构。结果表明,多膜膜的塑料不像先前假定的那样保守。塑料的尺寸在息肉中的尺寸变化很大,并且基因组中存在的大插入片段可能是影响基因组长度的主要因素。塑料雅库什木展示突出的特点,包括超过几kb的大规模红外扩张,也是一个独特的反演。此外,在息藻藻的塑料中鉴定了基因含量,SSR,分散的重复和突变热点区域。虽然分散的重复在粘面膜的塑料中不充分,但我们发现在不同物种中发生的大插入是移动的并且总是与重复的热点区域相邻。
结论
我们的结果显示水龙骨科的质体是动态分子,而不是像以前认为的那样组成静态基因组。在水龙骨科的质体中,插入序列两侧的离散重复序列参与了双链断裂诱导的修复机制,可能是结构进化的主要驱动力。
背景
叶绿体是植物细胞器,其中发生光合作用。每种叶绿体含有自己的基因组(塑料),通常在细胞器内的多个拷贝中发生[1].近年来,叶绿体基因组成为比较基因组学的首选目标,因为它大多是单亲本遗传,紧凑的大小,缺乏重组,和植物细胞中发现的其他两个基因组(核基因组和线粒体基因组)相比,温和的进化速率[2,3.,4].DNA测序技术的发展提供了高效、经济的测序平台,质体的特性使其成为高通量测序和组装的首选。质体现在已被广泛用于探索植物的系统发育关系和理解植物的进化过程[5,6,7].
蕨类植物是维管植物的第二大类群,具有重要的生态作用和系统发育地位[8].蕨类植物塑料的测序极大地提高了我们对这种谱系的质量多样性和演变的理解。蕨类植物塑料的尺寸高度保守,并且它们通常表现出从120〜170 KB的圆形结构[9]. 蕨类植物的质体通常由四部分组成,包括一对大的反向重复序列(IRs)、一个大的单拷贝区(LSC)和一个小的单拷贝区(SSC)。几乎所有的蕨类IRs都包含一个核心基因集,包括四个核糖体RNA(16S、23S、4.5S和5S)和几个tRNA基因(trn公司A-UGC,trn公司I-GAU,trn公司N-GUU,和trn公司R-ACG)。在结构上,蕨类植物的塑料以保守方式演变。大多数蕨类植物塑料很大程度上是线性的,需要几个反转和红外扩展,以考虑主要谱系中观察到的大规模结构重排。例如,可以通过展开IRS和“两个逆转”来解释核心肺孢子蕨类植物蕨类植物蕨类植物(Salviniales,Cyodales和Polypodales)和Schizaeales的独特叶绿体基因组重排。10.],主要影响IRS的方向和基因含量[11.]. 质体组的保守性使得它具有足够的同质性,可以在更高层次的分类群之间进行比较研究,但它也具有足够的差异性,可以捕获各种进化事件。
Polypodeae家族是最丰富的现存蕨类群体之一[12.],显示出色的形态和系统的多样性。在新生代辐射期间在高氮辐射期间在高管植物主导的树冠中多样化的Leptosporangiate蕨类植物,从而建立了息肉(Polypodeae)的多样性[13.].水龙骨科的大多数物种是附生植物,它代表了热带和亚热带森林中最多样和最丰富的泛热带维管附生植物群[14.,15.].息肉膜的塑料在进化时间内经历了各种复杂的基因组重建,使它们与基底蕨类植物(马塔基,Ophioglossales,Psilotales和EquateSales)显着不同。少数研究已经分析了粘蛋白酶的基因含量和细胞体积的细节细节,因为与其他谱系相比,息藻酸薄膜的塑料进化被认为是相对休眠的。大多数相关研究都集中在系统发育主题上。然而,最近的研究表明,塔白德的塑料不仅含有高变区域,而且含有广泛的移动序列[16.,17.].不幸的是,由于该组的可用塑料数据有限,相应的研究很少涉及息糖膜。这表明目前可用的塑料信息可能不足以阐明蕨类基因组的进化模式,并且息肉膜塑料的结构多样性的探索也远未充分。密切相关物种中叶绿体的比较基因组分析可以产生遗传标记,并提供对基因组的进化轨迹的更详尽的理解[18.,19.].然而,低级别分类群之间的质体结构和序列同源性使得识别各种进化事件变得困难。因此,有必要对水龙骨科植物质体的精细结构特征进行比较,以加深对蕨类植物质体多样性和动态演化的认识。在此基础上,我们首次对水龙骨科植物的质体结构和含量进行了家系比较分析。
我们利用高通量测序平台组装了三个新的质体,其中两个来自Neolepisorus和一个来自Phymatosorus并与以前发表的所有水龙骨科质体进行了详细的比较分析,以期:1)描述新测序的质体的基因组结构和基因含量;2) 在精细结构水平上研究水龙骨科质体的变异;3)深入了解水龙骨科质体的演化。
结果
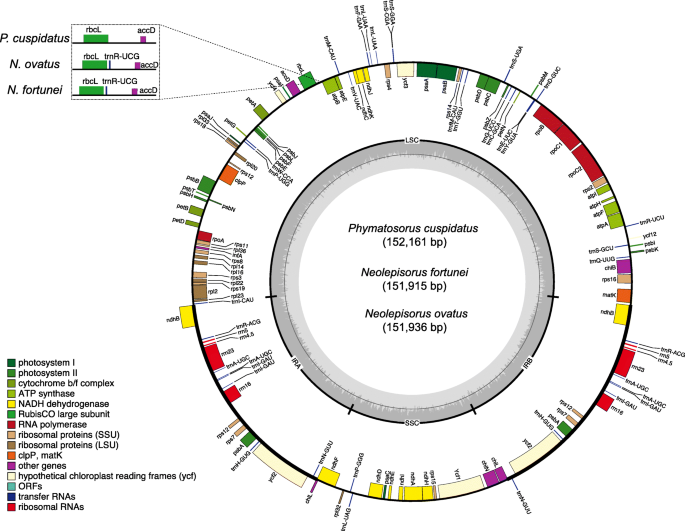
基因组组装和注释
Illumina对端测序产生了6760,171,6821,122和6746140个原始序列Neolepisorus Fortunei.,Neolepisorus ovatus,和Phymatosorus cuspidatus分别(表1).在参考质体上共绘制了635763 ~ 1054231条clean reads, 3个物种共组装了11 ~ 16个contigs,在参考质体上的平均覆盖率超过160×。通风质体N. Fortunei.,N.Ovatus.,和P.Cuspidatus.分别有六个,两个和三个缺口。用相应的引物对pcr测序填充每个质体的间隙区(表1)S1).完全塑性序列的长度范围为151,915至152,161bp,平均GC含量为41.7%(范围41.3-42.3%;表1).所有塑料均表现出典型的四边形结构,覆盖一对大型国税局(24,609-24,756 bp)。两个IR区域将塑料分成LSC区(80,670-81,175bp)和SSC区域(21,601-21,733bp)(图。1、表2). 三个新测序的质体具有相似的基因含量,有一些显著的区别。与其他两种质体相比trnR公司-UCG.观察到在P.Cuspidatus.(图。1).rps16型内含子缺失约470bpN. Fortunei.(图。S1).此外,N. Fortunei.是否有额外的完整副本NDHB.由于其IRA / LSC边界邻近的IRBndh公司B、 鉴于N.Ovatus.和P.Cuspidatus.只包含第二个片段NDHB.由于IRa/LSC边界位于ndh公司B。
水龙骨科全叶绿体基因组比较
三种新获得的粘面藻(N.Ovatus.,N. Fortunei.,和P.Cuspidatus.)与之前发表的水龙骨科3个亚科的9个质体进行了比较2).含有聚微藻膜的塑料似乎在结构上彼此相似,示出了由由LSC和SSC分离的两枚IRS组成的典型的四胞石结构。总的来说,分析表明,塑料组的尺寸在息藻藻之间的广泛变化,范围从151,936 bpN.Ovatus.到164,857 bp雅库什木.大多数物种的LSC和SSC区域的长度分别为81 kB和21kb,但这两个区域Drynaria Roosii.塑料分别较大,达到86 kB和24 kB。此外,塑料Leptochilus hemionitideus也包含较大的SSC (25,492 bp)。与SC区相比,水龙骨科质体的IR区长度在23,416 ~ 32,017 bp之间有显著差异。水龙骨科质体的基本组成相对于其大小变化较为保守。GC含量分布不均,IR区最高(42.1-45.9%),LSC区次之(37.9-43.6%),SSC区最低(34.4-43.0%)(Table2).
粘膜塑料塑料的基因序列几乎是共线。但是,结构的结构S. Yakushimensis.质体与水龙骨科植物的典型结构有很大的不同。两种植物的质体有显著差异S. Yakushimensis.是一个〜6 kB段的横向跨度ndh公司FccsA从SSC到IR;这种反转出现在IRb/SSC的交界处(图。2).在另一个交叉点,SSC / IRA,我们观察了广泛的IR扩展,导致重复ycf.1,的背影L,的背影在爱尔兰共和军的最后。此外,还有一个小反转(~ 2. kb)之间检测到Lepidomicrosorum Hederaceum和D鲁斯塑料,位于rps.15-ycf.1在SSC区域L. Hederaceum.和rrn.L-trn公司在LSC地区rD鲁斯分别(图。2). 发生在基因间区域的基因组结构变化可能起到额外的进化作用,但由于基因间区域没有编码功能,它们很难被检测到。这种独特的反转可能与插入序列有关,或者是其他质体进化的中间形式。随着已发表的水龙骨科质体数量的增加,水龙骨科的进化过程应该变得更加清晰。
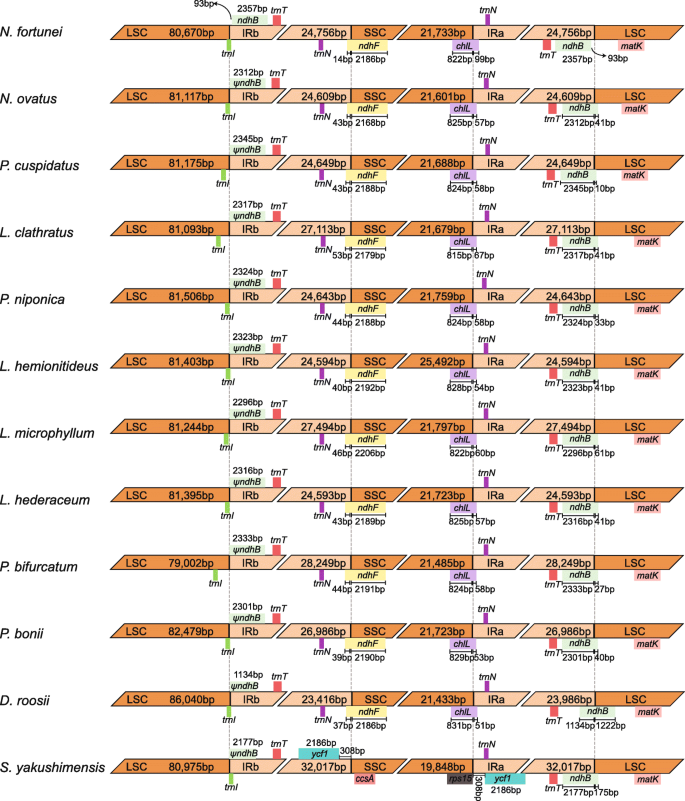
塑料的尺寸变化可能是IR边界的动态变化的结果[20.]. 然而,水龙骨科质体在SC/IR界面上表现出很高的相似性S. Yakushimensis.(图。3.).的trnN公司-瓜族基因位于IR附近NDHF.或者chlL在SSC/IR边界。这个NDHF.和chlL基因分别穿过SSC / IR边界,分别从14至53bp和51-99bp延伸。IRA / LSC边界位于编码区内ndh公司B并在11个塑料中的LSC / IRB边界中产生1,134-2,345bp的伪蛋白,除了N. Fortunei..LSC/IRb边界位于内部或旁边trnI标出基因。相对于红外边界的细微变化,红外S. Yakushimensis.经历了广泛的扩张,捕获了ycf.1,的背影L,的背影在SSC区域的N个基因。这种膨胀,与倒置相结合ndh公司F -ccs一个区域,导致唯一的SSC / IR边界S. Yakushimensis.塑料(图。3.).粘吡膜塑料的SC / IR边界高度相似但不相同,表明IR的膨胀和收缩是一种在演化中的独立和经常性的现象。此外,IR边界的变化不足以引起基因组大小的较大差异,并且我们认为微观结构的变化(例如插入和缺失)可能是差异的负责。
由于不同程度的红外边界变化和倒置,水龙骨科质体中的基因含量存在一定的差异。trn公司除了除以外,r-ucg存在于所有息肉腺痤疮物种中L. hemionitideus,P.Cuspidatus.,和platycerium bifurcatum..trn公司V-UAC也没有在塑料中缺席P. Bifurcatum..另一个不同之处在于塑料N. Fortunei.包含另一个完整的NDHB.IR区域的基因,而只有一个NDHB.碎片存在于其他物种中。此外,对水龙骨科12个质体的基因注释进行了详细的分析rpoc1.基因的L. hemionitideus已经被框架突变伪生化。
含萘糖膜塑料的序列多样性和突变热点
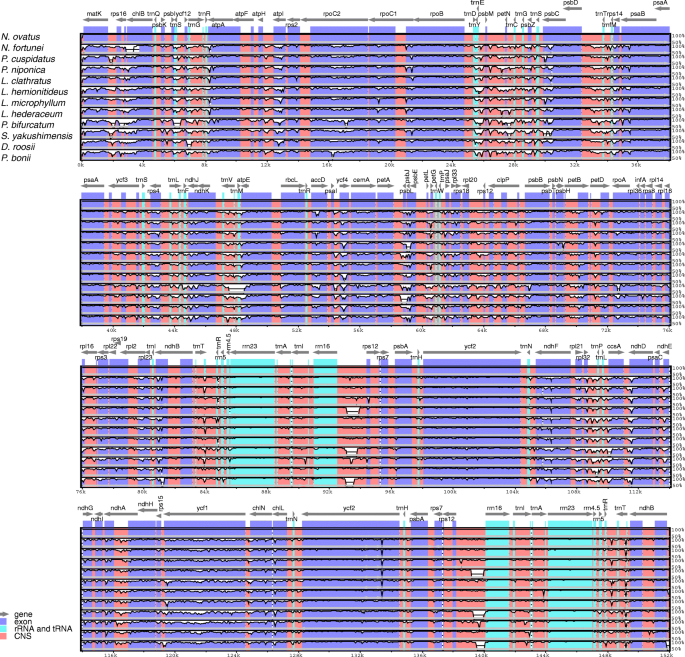
在mVISTA软件中进行的多序列比对显示,所分析的12种水龙骨科植物的质体的全序列具有相似性(图1)。4).在IR和蛋白质编码区中发现较低的分歧,而不是单拷贝和非分量区域。尽管如此,我们发现存在明显的大插入碎片rrn.16-rps.12 IRs在细胞质体中的间隔物Lepisorus clathratus.,Pyrrosia Bonii.,和P. Bifurcatum..为了检测芘的塑料中的突变热点,使用DNASP v5.0对序列的全基因组对准进行滑动窗口分析。结果表明,粘面藻塑料塑料的序列变化相对较低,核苷酸多样性(PI)范围为0.00232至0.20028。总的来说,SSC区域表现出最高的序列变化,平均PI为0.10103,然后是LSC和IR区,平均PI值分别为0.07317和0.02676。在息藻藻的塑料中鉴定了九个高度分歧的基因座,包括垫k-rps.16,rps.16,trn公司C -trn公司克,PSB.Z-psbC,PSB.D级-trn公司T,trn公司P-PSA.j,和rpl.2-trn公司我位于LSC;rrn.16-rps.12,位于IR地区;和一种蛋白质编码基因,ycf.1、位于SSC。的ndh公司F -ccsSSC有一个区域的Pi值高于其他位点,很可能是由于该区域发生了反演,导致了较高的层序变异(图1)。S2).因此,它可能不会被归类为息藻膜中的常见突变热点。
水龙骨科质体的ssr及重复序列分析
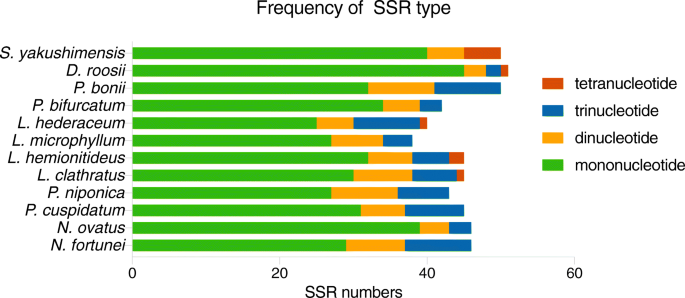
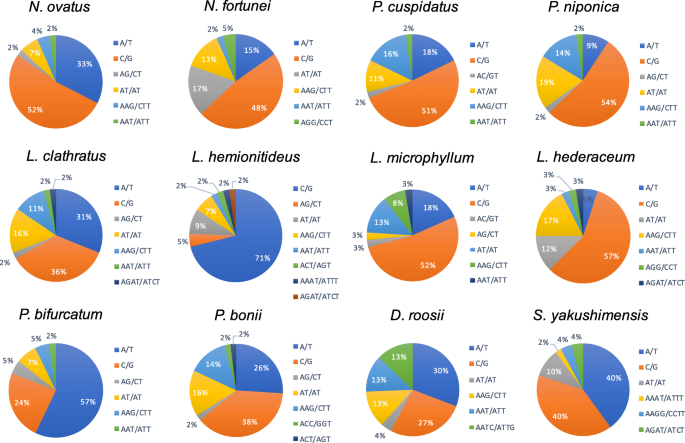
简单的序列重复(SSR)或微卫星短,串联重复的DNA基序为1-6个核苷酸[21.].它们可以表现出高多态性和突变率,这有助于遗传变异的估计[22.].在这项研究中,通过使用MISA从12个谷粉酵e物种的塑料中鉴定出非常相似的潜在CPSSR。息肉中的SSRS总数范围为38至51.检测到四种SSR:单核苷酸,二核苷酸,三核苷酸和四核苷酸(图。5).然而,四核苷酸重复只在染色体的质体中发现L. Clathratus.,L. hemionitideus,L. Hederaceum.,S. Yakushimensis.,和D鲁斯.不同的SSR基元以不同的频率出现在这些质粒中。单核苷酸重复最多,约占总SSR位点的62.8 ~ 88.3%,其次是二核苷酸重复(8.7 ~ 20.9%)和三核苷酸重复(6.6 ~ 22.5%),四核苷酸重复最少(0 ~ 4.4%)。
我们发现在所有被分析的物种中,除了D鲁斯,S. Yakushimensis.,和P. Bifurcatum.是G / C串联基序,其占Polypodeae塑料中的53.3%至100%的单核苷酸重复(图。6).本研究中包含的息肉型群物种在塑料中表现出类似的SSR分布图案。SSRS更频繁地位于LSC地区(48.0-71.1%),而不是IR(10.5-36.0%)和SSC地区(9.3-22.0%)。此外,大多数已识别的SSR都位于非基因间隔物中,占检测到所有SSR的66.7-83.3%。分散在内文区域的SSR是第二个最常见的类别(9.5-28.9%)。最少的SSRS位于编码基因中,占所有SSR基因座的4.0-14.0%(表S2).
在使用声压蛋白酶塑料中检测到具有序列同一性≥90%的高于30bp的转发,回文和反向重复≥90%。结果表明,粘膜膜塑料塑料中的重复数量显着各种各样,范围为9L. hemionitideus到146 in.P. Bifurcatum.. 这些长时间的重复从30到307不等 重复两次。物种在基因间隔区和编码基因的长重复序列数量上存在一定的变异。大多数重复分布在基因间隔区,其余分布在编码基因和内含子区(表1)S3).我们检测到一些具有丰富重复的物种特异性的代际垫片,包括rbcl.-trnR公司-大鼠LSC区的UCG基因间隔区D鲁斯,rps.12-rrn.16个IR区域的代际间隔物L. Clathratus.和P. Bifurcatum.,RPS7-PSBA.基因间间隔区lemmaphyllum脱噬细胞,而且ndh公司内文/的背影L-的背影N晶体SC/IR结的基因间隔区P. Bonii..
串联重复查找器v4.09 [23.还用于鉴定蛋白质塑料中存在的串联重复,重复的最小同一性和尺寸以单位长度设定为90%和15bp。除了除以外部的所有物种中检测到少数串联重复P. Bifurcatum.和L. hemionitideus(表3.). 在检测到串联重复的物种中,L小叶展出了最多的重复和P. Bonii.展出了最少的。代际垫片RRN16.-rps12,RPS7-PSBA.,和rbcl.-trnR公司-UCGG是含有含量丰富的重复序列的区域L. Clathratus.,L小叶,和D鲁斯, 分别。所有检测到的串联重复分布在非分量区域中,位于非基因间隔物中的串联重复的比例高于息肉(表3.).
水龙骨科质体的动态插入序列
通过详细的全基因组比对,我们发现在水龙骨科质体的某些区域,包括rrn.16-rps.12个垫片L. Clathratus.,P. Bifurcatum.,和D鲁斯(~ 2400–3500 bp插入);这个rps.7-PSB.一个垫片L小叶(〜3000 bp插入);这加拿大皇家银行L-trn公司r spacerD鲁斯(〜4000 bp插入);这宠物A -PSA.j spacerP. Bonii.(〜1700 bp插入);和rps.15-ycf.1个垫片L. Hederaceum.(〜3700 bp插入)(表4).插入序列的身份rrn.16-rps.12个垫片L. Clathratus.,P. Bifurcatum.和D鲁斯使用MegAlign v8.1.3进行计算[24.].两两比对显示,3个质体中插入的同源性仅为48.3-50.1%,说明这些插入可能有不同的来源(图1)。S3).
Robison等人。[17.]之前提出了Morffo的概念(蕨间有机细胞器中的移动开放阅读框架),这是一组广泛存在于Fern Interelles中的移动插入序列。为了验证在聚吡铵膜塑料中检测到的插入是否与Morffo序列一致,Morffo序列由当地爆发搜索确定[25.,26.]利用水龙骨科12个质体建立的数据库,获得了一致序列摩福1,摩福2,和摩福3作为疑问。此外,为了检查本研究中识别的插入是否存在移动性质,这些序列受到本地爆炸搜索。我们的结果表明摩福1呈现相似之处宠物A -PSB.J的片段P. Bonii.(71.2%)和rrn.16-rps.12个片段P. Bifurcatum.(70.3%)。摩福2被检测到rrn.16-rps.12P. Bifurcatum.和ycf.1-ccsA.S. Yakushimensis.,身份分别为71.0%和67.9%(表5).
令人惊讶的是,不同物种中包含的插入显示出彼此的显着爆炸率,这表明DNA片段可以从一个塑料转移到另一个塑料。例如,一个片段L. Clathratus.插入显示高序列相似之处rps.7-PSB.位于IR的插入l . microphyllum红细胞L-trn公司R位于LSC的D鲁斯,和rps.15-ycf.1位于SSC的L. hemionitideus.摩福1位于P. Bifurcatum.-P. Bonii.共识插入片段,但摩福2没有显示与本研究中检测到的插入重叠(表5).以往的研究表明,MORFFO-like序列往往与基因组的结构变化有关,可能是质体结构进化的主要驱动力。本研究在水龙骨科相对静态的质体中发现了若干可移动的插入,说明基因组结构进化过程中产生的不同插入片段可能具有不同的功能。
讨论
水龙骨科的质体变异
通过对水龙骨科、桔梗亚科和木梗亚科12种植物质体长度的比较分析,发现水龙骨科的质体长度差异较大,甚至在同一亚科内也存在差异。通常,红外边界的动态膨胀或收缩被认为是导致陆地植物质体大小变化的主要机制[20.].虽然粘膜塑料塑料之间的IR边界存在差异,但它们也表现出明显的相似之处。因此,预计粘膜膜塑料的IR边界中的次要变化将不足以考虑基因组大小的显着差异。例如,在微生物陶器中,我们发现,尽管IR边界深深保守,但IR地区L. Clathratus.和L小叶比其他物种长约2500 bp,L. hemionitideus比其他物种长约3700 bp。对水龙骨科的质体序列数据进行精细分析后发现,在IR或SC区域的特定基因间隔上有几个大的插入,与观察到的基因组大小差异相对应。这种情况与其他两个亚科的种的情况很一致,除了S. Yakushimensis..因此,在大多数物种的息肉藻中,塑料大小差异的主要原因不是SC / IR界限的变化,而是在不同物种中发生的大片段插入。雅库什木Drynarioideae迄今为止展示了蕨类植物谱系中确定的最大IRS,主要是因为它的IR边界延伸到SSC,导致ycf.1,的背影L,ch在IR区域内捕获的LN基因。因此,我们无法从IR边界的扩展和收缩的角度讨论塑料大小的差异,因为塑料本身的保守性质将导致研究人员忽视其他因素。
通常认识到,含有粘膜的塑性塑料的塑料逐渐稳定,并且诸如重排的结构变化很少发生。相比之下,呈现的结果表明了塑料S. Yakushimensis.在某些方面是非常不寻常的,它不仅包含超过几kb的大规模红外膨胀,而且还包含独特的反演。IR的大规模膨胀可能是通过复制过程中IR片段之间的双向重组而发生的[27.].位于原始SSC/IR结的大量分散重复序列(的背影L /trn公司n-guu)被检测到S. Yakushimensis.质体,与红外膨胀机制完全吻合;也就是说,重复序列通过同源重组提供了分子内部或分子之间基因组重排的可能性[27.,28.].总体而言,质体结构变化的同质性相对于层序资料较低。结构上的变化S. Yakushimensis.因此,塑料可以用作物种辨别或系统发育分析中的特定遗传标记。不幸的是,没有办法确定首先发生的事件,因为在SSC / IRA边界处发生IR扩展,而IRB / SSC边界附近发生反转。因此,有必要序列在息藻藻中更加塑性,以改善我们对塑料进化轨迹的理解。
全塑性对齐可以阐明序列发散的水平,并且容易识别大型诱导,这对于系统发育分析和植物鉴定非常有用。在本研究中,我们的结果表明,IRS呈低于SC区域的序列性较低。这种现象被认为是IR序列之间的复制校正和通过基因转换消除有害突变的结果[29.]. 此外,水龙骨科质体之间的序列差异在基因间间隔区表现明显,表明编码区比非编码区更具保守性。水龙骨科植物之间的9个分歧热点,包括垫k-rps.16,rps.16,trn公司C -trn公司克,PSB.Z-psbC,PSB.D级-trn公司T,trn公司P-PSA.j,rpl.2-trn公司我,rrn.16-rps.12,和ycf.1,其中9个位点位于SC, 1个位于IR。虽然ndh公司F -ccs由于该区域的倒置存在,它们还存在较高的PI值,它们可能不适用于息蜡膜的一般突变热点。S. Yakushimensis.质体。因此,在筛选普遍的突变热点时,我们必须进行仔细的数据探索,以避免被质体结构变化所混淆。
在这项研究中,我们发现了许多重复区域,包括前进重复,回文重复和反向和串联结构,这可能是基因组重新配置的重要热点[30.,31.].特别地,在塑性体中发生大重复的发生,例如观察到307bp的回文重复P. cuspidatus,据推测,由于不适当的重排,导致了不稳定的基因组结构[32.]. 此外,这些重复序列为发展系统发育和群体遗传学的分子标记提供了许多信息位点[33.].作为一种非常强大的分子标记,SSR被广泛用于不同的研究领域。它们具有明显的优点,例如高多态性和成本效益[34.].大多数研究表明,陆地植物的主要cpssr与它们的at偏倚质体是一致的。相比之下,水龙骨科的cpSSRs与以往报道的cpSSRs有很大的不同。本研究在水龙骨科的每个质体中鉴定出38个以上的ssr,其中大多数为C/G单核苷酸,分布在非编码区。以前认为cpssr通常由A/T重复序列组成的观点现在受到了挑战[35.].Gao等人[36.]在变性实验中显示,具有较高GC含量的重复结构有助于增加热稳定性Dryophteris雀fr在温度变化时保持其结构。水龙骨科的物种进化出多样化的形态特征和生活方式,这可能是对白垩纪被子植物辐射引起的陆地生态系统变化的响应[37.]. 因此,我们推测这些GC含量高的重复结构可能是水龙骨科植物适应环境的分子基础之一,这也为理解植物的环境适应机制提供了新的思路。此外,本研究开发的cpsrs为研究遗传结构和遗传变异提供了独特的信息。特别是,这些cpsrs将与蕨类植物的核ssr互补和可比。
息糖膜塑料的动态插入
虽然全基因组比对表明水龙骨科质体是相当保守的,但我们发现在某些基因间隔中存在异常大的插入。大片段插入观察RRN16.-rps12基因间的间隔L. Clathratus.塑料与以前的研究结果一致[16.].在一些远距离相关物种的LSC区域和促型有促型物质中检测到类似的插入序列Asplenium nidus.,暗示此类序列可以在基因组隔室之间移动[16.].Robison等人。[17.]通过对蕨类植物的广泛调查发现类似的动态移动元件套件,在Morffo元素的存在相对于反转,代际膨胀和反相重复变化的情况下,脱落灯。在这项研究中,我们在粘面膜的塑料中表征了一种完全不同的插入序列。只有两个插入与...重叠摩福1序列,它们位于宠物A -PSA.J间隔区的LSC区P. Bonii.和rrn.16-rps.12欧林地区的垫片P. Bifurcatum..检测到摩福2序列位于ycf.1-ccs中位于红外/低纬边界的一个区域S. Yakushimensis.和rrn.16-rps.12欧林地区的垫片P. Bifurcatum.,与其他插入没有同源性。我们的结果进一步证实了MORFFO序列在蕨类植物质体中的普遍性,以及这种序列在蕨类植物质体的反转终点的存在S. Yakushimensis.表明Morffo序列可能与反演事件有关。
虽然MORFFO序列在其余的水龙骨科物种中未检测到,但在它们的质体中存在另一组高度可移动的插入物。值得注意的是,质体基因很少获得或丢失,而我们的研究表明,已鉴定的插入序列在水龙骨科质体的进化过程中经常获得或丢失。这种流动性可能表明质体中的这些插入物起到了移动元件的作用。此外,这些插入经常出现在更分散的重复出现的区域附近。许多研究表明,基因组重排与小分散重复序列(sdr)有关,sdr参与了双链断裂的修复机制[38.,39.].sdr通常对高度重排的基因组的重复空间做出重要贡献,甚至在密切相关的谱系中也会增加结构多态性,它们主要存在于非编码DNA片段中,与小发夹结构有关[38.].我们推测,与高度移动插入相结合的丰富重复基序的存在可以构成用于在多泡型物种的塑料中的基因组重排的“触发机制”,这可以在某些条件下诱导塑料的结构变化。用于蕨类植物的当前序列数据的局限性意味着难以确定这些插入序列的精确源。随着未来发表的更多基因组数据,这些插入序列的源和迁移机制应该变得清晰。
结论
通过对水龙骨科其他质体的研究,我们对水龙骨科的质体演化有了更清晰的认识。一般认为水龙骨科质体的进化是保守的,其结构特征在水龙骨科中几乎是不变的。然而,在这种保守的背景下S. Yakushimensis.塑料脱颖而出。IRS的大规模扩张和独特的反演区分S. Yakushimensis.从迄今为止研究的所有其他息肉藻的塑料。此外,在多泡糖岩物种的塑料中发现了许多大型移动插入,其通常在不同的息肉膜塑料中侧翼在分散的重复的元素中。这些不寻常的特征在息蜡膜的结构稳定的塑料中发现,因此可以涉及家庭塑料的动态演变。换句话说,与先前表征的粘膜肌的静态塑料不同,本文表征的塑料在结构上不稳定,如息藻膜中的大型移动插入所证明。
方法
样本收集,DNA提取和测序
在本研究中,鲜叶蕨,卵形蕨,和P.Cuspidatus.采自中国科学院华南植物园的活体标本,液氮速冻,低温超低温冰箱保存− 80 °C直到使用。凭证标本保存于中山大学标本馆(系);代金券:美国刘201,630,美国刘201,654和美国刘201,701蕨,卵形蕨,和P.Cuspidatus.分别)。按照说明书(北京天根生物技术有限公司),使用天根植物基因组DNA试剂盒提取基因组DNA。在0.8%的琼脂糖凝胶中检测DNA质量,并使用NanoDrop分光光度计(Thermo Scientific, Carlsbad, CA, USA)进行DNA定量。质量评估后,用Covaris M220超声仪(Covaris Inc., MS, USA)将500 ng DNA剪切成平均300 bp的片段大小。使用NEBNext, Ultra DNA文库预备试剂盒(New England BioLabs Inc., Ipswich, MA)构建Illumina配对末端(PE)测序文库。测序工作在HiSeq 2500平台上进行(Illumina公司,圣地亚哥,美国)。Illumina测序为每个物种产生了大约2gb的原始数据。
基因组组装和注释
使用FASTQC v0.0.0评估原始读取的质量[40].通过使用Trimmomatic V0.32修剪低质量碱基(Q <20)和适配器序列[41.].清洁读数被映射到参考塑料Lepisorus clathratus.(NC_035739)来过滤叶绿体数据。所有的映射读被从头到尾与天鹅绒(Velvet)组合成contigs [42.],并与参考基因组进一步对齐和定位。剩余的空白被直接PCR填补,使用特定的引物是基于连续序列或同源序列比对设计的。使用DOGMA进行叶绿体基因注释[43.[后面是根据来自其他公布的紧密相关的蕨类植物的同源基因进行手动校正开始和止扰密码子和内含子/外显子界限。使用Aragorn验证转移RNA(TRNA)基因[44.]trnasse可以在组织器搜索模式下使用默认参数[45.]. 用OGDRAW绘制了质体的圆形图[46.].加入号码蕨,卵形蕨,和P.Cuspidatus.分别为MT373087、MT364352、MT364353。
聚吡乙醚塑料的比较分析
来自Polypodiaceae的多种塑料的可用性提供了探讨家庭内基因组的多样性的机会,包括基因组大小和结构,GC含量,基因阶和IR膨胀/收缩。因此,我们对三个组装的塑料序列和氮素聚糖型种类的九种其他可用塑料进行了比较分析,来自Polypodiodes niponica(nc_040221),Lepisorus clathratus.(NC_035739),Leptochilus hemionitideus(nc_040177),lemmaphyllum脱噬细胞(MN623356),platycerium bifurcatum.(MN623367),Lepidomicrosorum Hederaceum(MN623364),Pyrrosia Bonii.(nc_040226),雅库什木(MN623352),和Drynaria Roosii.(KY075853)。塑料D鲁斯使用基因预测工具和手动调整在我们的分析之前进行了成功,因为我们注意到了D鲁斯注释。根据这些质粒的序列和注释信息,比较了它们的基因组大小、基因含量、红外边界和碱基组成。利用Mauve v2.4.0中的ProgressiveMauve算法对12个水龙骨科物种进行全基因组比对,以识别逆序[47.]从每个塑料中除去一个倒置重复(IR)拷贝后。
使用MVISTA绘制12个谷粉膜塑料的总体相似性[48.]带注释的Shuffle-LAGAN模式在线程序N. Fortunei.作为参考。为了估算核苷酸多样性(PI)和突变热点,我们在MAFFT V7.310软件中进行了12种塑料的成对比对[49.],用BioEdit软件手动调整[50.]如有必要。通过使用DNASP V5.0通过滑动窗口分析计算对准序列的核苷酸分集值(PI)[51.,窗长分别为600 bp,步长分别为200 bp。
SSRS的表征和重复序列
用叶绿体序列中发现的重复元素进行息蜡膜膜物种之间的进一步比较。使用Perl脚本MISA检测简单的序列重复(SSR)[52.](微卫星;http://pgrc.ipk-gatersleben.de/misa//)对于单核苷酸,有10个重复基序的最小迭代,对于二核苷酸重复,有5个重复基序,对于三、四、五和六核苷酸,有4个重复基序。利用串联重复查找器v4.09识别了8种水龙骨科植物的串联重复序列[23.[分别在2,7和7处设定的匹配,不匹配和indels。对于最小对准分数和500个,参数设置为90,为最大周期大小。声誉员[53.](http://bibiserv.techfak.uni-bielefeld.de/reputer/)用于可视化分散重复序列(正向重复序列、反向重复序列、互补重复序列和回文重复序列)的位置和大小,最小重复序列大小为30 bp,汉明距离为3。
数据和材料的可用性
塑性塑性的注释序列蕨,卵形蕨,和P.Cuspidatus.已提交基因资料库(https://www.ncbi.nlm.nih.gov/genbank/)分别在MT373087,MT364352和MT364353中分别登录号。本研究期间生成或分析的所有数据都包含在文章及其附加文件中。
缩写
- 内容提供商:
-
叶绿体
- 红外光谱:
-
反向重复
- 核糖体rna:
-
核糖体核糖核酸
- SSC:
-
小单副本
- tRNA:
-
转移RNA.
- LSC:
-
大单拷贝
- 苏维埃社会主义共和国:
-
简单序列重复
- 特别提款权:
-
小分散重复
- 摩福:
-
蕨enterelles中的移动开放阅读框架
参考
- 1。
道格拉斯。质体进化:起源,多样性,趋势。Curr Opin Genet Dev. 1998;8:65 - 61。
- 2。
伍尔夫,李卫华,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸的取代率差异很大。美国国家科学研究院1987;84:9054-8。
- 3.
杜洛因G,达乌德H,夏J。种子植物线粒体、叶绿体和核基因组同义替换的相对比率。分子系统发育。2008;49:827–31.
- 4.
史密斯博士。塑性基因组中的突变率:它们低于您的思考。基因组Biol Evol。2015; 7:1227-34。
- 5。
Palmer JD,Soltis de,Chase MW。生命植物树:概述和一些观点。我是J机器人。2004; 91:1437-45。
- 6。
莱顿南。解决完整的蕨类植物生命树。公共科学图书馆一号。2011;第6章:e24851。
- 7。
陈泽,杨T,林L,Lu Lm,Li HL,Sun M等。生命之树为中国血管植物的属。J Syst Evol。2016; 54:277-306。
- 8.
页面CN。生态学视角下蕨类植物的多样性。正确答案:Dyer AF, editor。蕨类的实验生物学。伦敦:学术;1979.9-56页。
- 9.
割草机jp,vickrey tl。土地植物塑性基因组中的结构多样性。ADV BOT RES。2018; 85:263-92。
- 10。
Wolf PG、Rowe CA、Sinclair RB、Hasebe M。钩端螺旋体蕨类植物叶绿体基因组的完整核苷酸序列,铁线蕨capillus-venerisLDNA Res.2003年;10:59–65.
- 11.
Hasebe M,Iwatsuki K.基因定位在少女毛发蕨的叶绿体DNA上;铁线蕨capillus-veneris.Bot Mag Tokyo。1992年; 105:413-9。
- 12.
PPG I.一种社区衍生的患者源性促进液体和蕨类植物的分类。J Syst Evol。2016; 54:563-603。
- 13。
Schuettpelz E,Pryer Km。在高中蕨类植物中的新生代辐射在高血管型冠层上的证据。Proc Natl Acad Sci U S A. 2009; 106:11200-5。
- 14。
绅士啊,Dodson C.新血管骨骺的多样性和生物地理。安莫僵尸加德。1987; 74:205-33。
- 15.
本津DH。维管真菌。作者:Lowman医学博士,Rinker BH,编辑。森林树冠。圣地亚哥:爱思唯尔;2004年。175–211.
- 16。
Logacheva MD,Krinitsina AA,Belenikin Ms,Khafizov K,Konorov Ea,Kuptsov Sv等人。覆盖蕨(多涂层)塑料的倒置重复的对比分析显示出两个高变区域。BMC植物BIOL。2017; 17:255。
- 17。
rorobinson TA, Grusz AL, Wolf PG, Mower JP, Fauskee BD, Sosa K等。蕨类植物质体演化的运动元素。基因组生物学进化。2018;10:2558-71。
- 18。
Shaw J,Lickey EB,Beck JT,Farmer SB,Liu W,Miller J等人。乌龟和野兔II:21非编码叶绿体DNA序列的相对效用进行系统发育分析。我是J机器人。2005; 92:142-66。
- 19.
Shaw J,Lickey EB,Schilling EE,小RL。整体叶绿体基因组序列的比较选择有罪植物的系统发育研究中的非定量区:乌龟和野兔III。我是J机器人。2007; 94:275-88。
- 20
Kim KJ,Lee HL。从韩国人参完全叶绿体基因组序列(人参schinsengNees)和17种维管植物序列进化的比较分析。DNA研究》2004;11:247 - 61。
- 21
李YC,Korol AB,Fahima T,Beiles A,Nevo E.微卫星:基因组分布,推定功能和突变机制:审查。Mol Ecol。2002; 11:2453-65。
- 22。
Guichoux E,Lagache L,Wagner S,Chaumeil P,LégerP,Lepais O,等。微卫星基因分型的目前趋势。Mol Ecol Resour。2011; 11:591-611。
- 23。
Benson G. Tandem重复发现:分析DNA序列的程序。核酸RES。1999年; 27:573-80。
- 24。
Burland TG。DNASTAR的Lasergene序列分析软件。提名:Misener S, Krawetz SA,编辑。生物信息学方法和协议。风险:胡玛纳;2000.p . 71 - 91。
- 25。
altschul sf,gish w,miller w,myers ew,lipman dj。基本的局部比对搜索工具。J Mol Biol。1990; 215:403-10。
- 26。
Madden T.爆炸序列分析工具。第二届:国家生物技术信息中心(美国);2013年。
- 27。
重复序列介导的重排小球藻艾略薇叶绿体DNA:完成大反向重复序列的核苷酸序列。咕咕叫麝猫。1991;19:139-47。
- 28。
帕尔默JD。叶绿体基因组的比较组织。Annu Rev Genet。1985; 19:325-54。
- 29。
通过基因转换消除质体基因组中的有害突变。植物j . 2006; 46:85 - 94。
- 30
浅野T,月月T,高桥S,岛田H, Kadowaki KI。甘蔗核苷酸全序列(蔗糖officinarum)叶绿体基因组:四种单子叶叶绿体基因组的比较分析。DNA Res。2004; 11:93-9。
- 31
高l,易x,杨yx,su yj,王t.完全叶绿体基因组序列的树蕨alsophila spinulosa:深入了解蕨叶绿体基因组的进化变化。BMC EVOL BIOL。2009; 9:130。
- 32
MaréchalA,Brisson N.重组和植物细胞器基因组稳定性的维持。新植物。2010; 186:299-317。
- 33
Chen C,郑Y,刘S,Zhong Y,Wu Y,Li J等。完全叶绿体基因组樟树并与樟科近缘种进行比较。PeerJ。2017; 5: e3820。
- 34。
王ML,Barkley Na,Jenkins TM。植物和昆虫的微卫星标志物。第一部分苹果Biotechnol G3。2009; 3:54-67。
- 35。
匡王,吴H,王YL,高LM,张SZ,LU L.完全叶绿体基因组序列木兰夸士森(木兰科):DNA条形码和群体遗传学的意义。基因组。2011;54:663 - 73。
- 36。
高河,王W,黄Q,风扇r,王x,feng p等。完全叶绿体基因组序列Dryophteris雀fr(L.)Schott和反复结构对抗热环境。SCI批准。2018; 8:1-11。
- 37。
施耐德H, Schuettpelz E, Pryer KM, Cranfill R, Magallón S, Lupia R.蕨类在被子植物的阴影下多样化。大自然。2004;428:553-7。
- 38。
米利根BG,汉普顿JN,帕尔默JD。叶绿体DNA的分散重复和结构重组。分子生物学。1989;6:355–68.
- 39。
奥多姆OW, Baek KH, Dani RN, Herrin DL。衣藻叶绿体可以利用短分散重复序列和多条途径修复基因组中的双链断裂。植物j . 2008; 53:842-53。
- 40。
FastQC是一个高通量序列数据的质量控制工具。http://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
- 41。
Bolger AM, Lohse M, Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014;30:2114-20。
- 42
Zerbino博士,Birney E。Velvet:使用de Bruijn图进行从头短读组装的算法。基因组研究,2008年;18:821–9.
- 43
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学。2004;20:3252-5。
- 44
拉斯莱特D,坎贝克B。ARAGORN,一个在核苷酸序列中检测tRNA基因和tmRNA基因的程序。核酸研究,2004年;32:11–6.
- 45
Schatter P,Brooks An,Lowe TM。用于检测TRNA和SNORNAS的TRNASCAN-SE,SNOSCAN和SNOGPS Web服务器。核酸RES。2005; 33:W686-W9。
- 46。
Lohse M,Drechsel O,Kahlau S,Bock R. Uniterellargenomedraw-一套用于生成塑体和线粒体基因组的物理图和可视化表达数据集的工具。核酸RES。2013; 41:W575-W81。
- 47。
Darling Ac,Mau B,Blattner Fr,Perna nt。紫红色:重排的保守基因组序列的多次对准。Genome Res。2004; 14:1394-403。
- 48。
Frazer Ka,Pachter L,Poliakov A,Rubin Em,Dubchak I. Vista:用于比较基因组学的计算工具。核酸RES。2004; 32:W273-W79。
- 49。
MAFFT多序列比对软件版本7:性能和可用性的改进。生物科学进展。2013;30:772-80。
- 50.
大厅的助教。BioEdit:一个用户友好的生物序列比对编辑和分析程序,适用于windows 95/98/NT。核酸序列1999;41:95-8。
- 51。
Librado P,Rozas J.DNASP V5:一种用于综合分析DNA多态性数据的软件。生物信息学。2009; 25:1451-2。
- 52。
Beier S,Thiel T,MünchT,Scholz U,Mascher M. Misa-Web:用于微卫星预测的Web服务器。生物信息学。2017; 33:2583-5。
- 53。
Kurtz S,Choudhuri JV,Ohlebusch E,Schleiermacher C,Stoye J,Giegerich R.声誉换句者:重复分析对基因组规模的歧管应用。核酸RES。2001; 29:4633-42。
致谢
不适用。
资金
本工作得到了中国国家自然科学基金的支持[31670200,31770587,31872670和32071781],中国广东省的自然科学基金,中国广东省科技规划项目[2017A030303007 [2016A030313203122] [2017A030303007[中国广东省深圳市科技系项目[JCYJ20160413155520290544990549905349054499054,42090534499054,4119803037070534499054,42011,249037.CHAILOY。该资助者在研究设计中没有作用,对数据的分析也没有写作稿件。
作者信息
从属关系
贡献
TW构思和设计了实验;SL和YS收集和贡献样品;zw执行了实验;SL分析了数据并写了稿件;TW和Ys批判性地审查了手稿。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准并同意参与
不适用。
出版许可
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:表S1。
用于塑料间隙闭合的引物。表S2。粘膜膜不同塑料区SSRS分布。表S3。有关聚吡乙醚塑料中分散重复的信息。
附加文件2:图S1。
对齐的rps.用于3种我们测序的三种塑料的16个外显子/内含子。内含子只删除N. Fortunei..
附加文件3:图S2。
12种水龙骨科植物质体的核苷酸多样性(Pi)。
附加文件4:图S3。
中的大插入片段的标识rrn.16-rps.12在质体中L. clathratus,p. bifurcatum,和D鲁斯.
权利和权限
开放访问本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
刘松,王卓,苏勇。et al。比较基因组分析氯化物叶绿体露出细结构特征和动态插入序列。BMC植物BIOL.21,31日(2021年)。https://doi.org/10.1186/s12870-020-02800-x
收到:
接受:
出版:
关键字
- 塑料
- 水龙骨科
- 插入
- 动态演变