摘要
背景
我们对塑体转录om的理解仅限于一些模型植物,其体液基因组(塑性)具有高度保守的基因阶。因此,关于基因表达如何响应于塑性的基因组重排而众所周知。这在高度重新排列的针叶树塑料中尤为重要。
结果
我们对六种针叶树属植物的质体和质体转录组进行了测序并报道。链特异性RNAseq数据显示了几乎完全的质体链转录,并检测到C-to-U rna编辑位点的意义和反义转录。研究表明,针叶树种间质体编码基因的表达具有很强的功能依赖性。然而,这种关联的强度随着塑料重排数量的增加而下降。这一发现表明,塑性重排影响基因表达。
结论
我们的数据提供了第一个证据,表明塑料重排不仅使塑料结构复杂化,而且驱动了针叶树质体转录组的动态。
背景
针叶树是一组结球果的种子植物。松科(针叶树一枝)和柏树属(针叶树二枝,由五科组成)两个分支约630种。针叶树在温带森林中占主导地位,特别是在北半球,对光合作用和生物量的生产有重要贡献。它们为野生动物提供庇护所,为人类提供重要资源,如实木燃料、珍贵木材、食用种子和精油[1].
Plastid基因转录是一种复杂的方法,涉及原核和真核型系统[2]. 大多数质体基因可能转录为多顺反子mRNAs,然后进行各种转录后修饰[3.].这些过程产生了巨大的详细阐明的转录om,具有前所未有的非编码RNA多样性[4],多个转录起始和终止位点[5,6],基因组的完全或几乎完全转录[7,8],以及不同频率的rna编辑位点[9].
陆生植物质体基因组在基因含量和顺序上高度保守。功能相关的基因通常在集群中发现,并可能作为操纵子共转录[10.].由于选择性约束而不是中性染色体重排的缓慢来保护这些操纵子[11.].然而,越来越多的证据表明,许多类群,包括针叶树(裸子植物的最大类群),都有高度重排的质体[12.,13.,14.].其中一些重排导致规范式操纵子和创建新的共转录单位的破坏。一个例子是中断rps2型操纵子Sciadopitys和Callitris.[15.,16.].我们长期以来对这些发现感到困惑,因为那时还不清楚质体重排是否影响质体基因的转录。如果他们这样做了,这些变化的根本机制和后果是什么?
在这项研究中,我们对现存的六个针叶树科中的一个代表属的质体DNA和RNA进行了测序。链特异性RNA文库的优点是可以区分正反义转录物[17.].我们利用这一点(1)研究了两个质体链的完整转录能力,(2)估计了质体编码和反义转录的相对数量,(3)分别鉴定了针叶树质体中正义和反义转录的C-to-U rna编辑位点。我们还比较了针叶树质体基因表达水平,并证明了基因表达与质体重排之间的强相关性。我们将讨论这种关联的可能机制。
结果
两种塑性链在针叶树中都完全转录
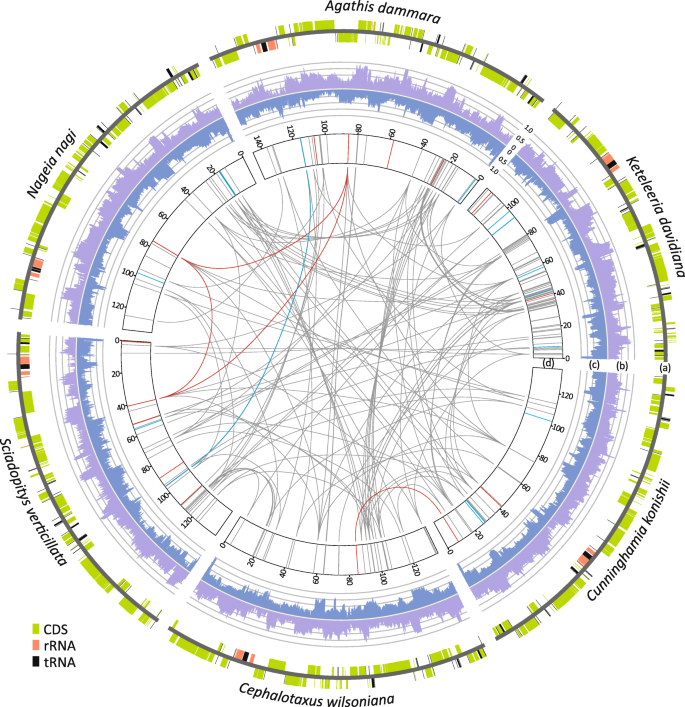
这六个新组装的质体被描绘成线性分子,以方便成对比较(图。1a) 是的。在样品中检测到塑性反转K. Davidiana(NC_011930;无花果。1a).这个多态倒置的两侧是松科I型重复序列[18.],它们能够触发同源重组以产生主要的亚化学计量塑性异构体K. Davidiana(图。S1b) 是的。
六个采样针叶树塑料上的RNASEQ覆盖物在图2中表示为直方图。1.RRNA的RNAseq覆盖率低,表明在测序之前有效地耗尽RRNA转录物。我们还发现,从特定单一DNA链产生的RNA覆盖超过94.2%的塑料序列。从两条链的RNASEQ读数组合覆盖率增加到超过99.9%(图。2).总的来说,我们的数据揭示了两个质体链的几乎全部转录,表明基因间转录、内含子转录和反义转录在针叶树质体中普遍存在。
我们的数据还显示,CDS义转录本通常比反义转录本更丰富,尽管也有几个例外(图S3.).例如,psbN,一个光合系统II基因,位于psbB操作典,一种众所周知的多排序型转录单元,包括四种基因:psbB,psbH,petB,petD[19.].因此,抄本反义psbN可能因为强烈表达而被过度代表psbB操纵子。
塑料重排对CDS表达的影响
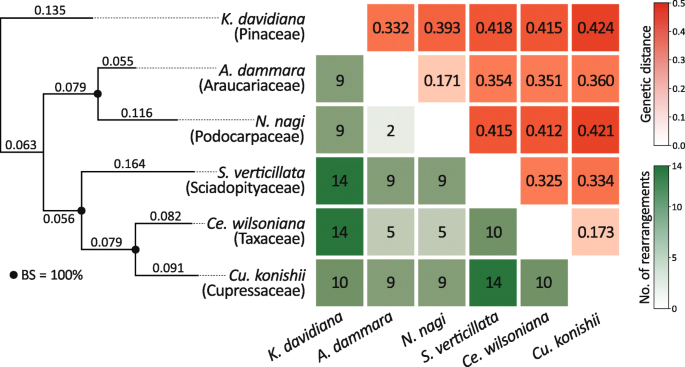
在6个针叶树质体中,鉴别出31个同连体块,以估计塑性重排(图S4一种)。这六个塑料的成对点分析也在图2中示出4b.我们的比较揭示了取样针叶树之间2-14个重排(图。2).为了检验系统发育距离是否与塑性重排的频率有关,我们根据83个同源CDSs的串联推断出的树的分支估计了种间遗传距离(图1)。2).我们没有发现遗传距离和塑性重排计数之间的显着相关性(Pearsonρ= 0.375,P= 0.167;无花果。5).
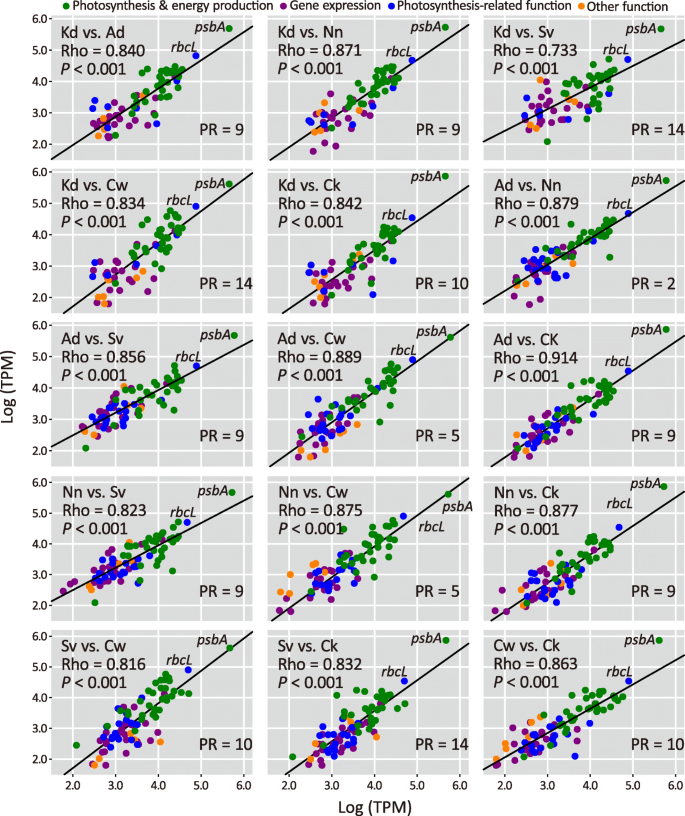
为了使表达水平正常化,收集CDSs上的RNAseq reads,并合并计算每百万转录本(TPM)。数字3.比较不同针叶树种间保持相同功能的同源质体CDSs之间的TPM得分。我们发现(1)psbA和:(2)这些同源基因的TPM得分显著相关(Pearson’sρ= 0.733到0.914P< 0.001),表明它们的表达水平具有很强的功能依赖性。然而,这些相关系数与物种之间的塑性重排(PR)的数量呈负相关(Pearson’s)ρ=−0.626,P= 0.013;无花果。6).我们的结果一起表明,塑性重排减少了塑性基因表达的功能依赖性关系的强度。换句话说,这些重排成为针叶树中的基因表达。
质体RNA编辑既发生在意义转录本,也发生在反义转录本
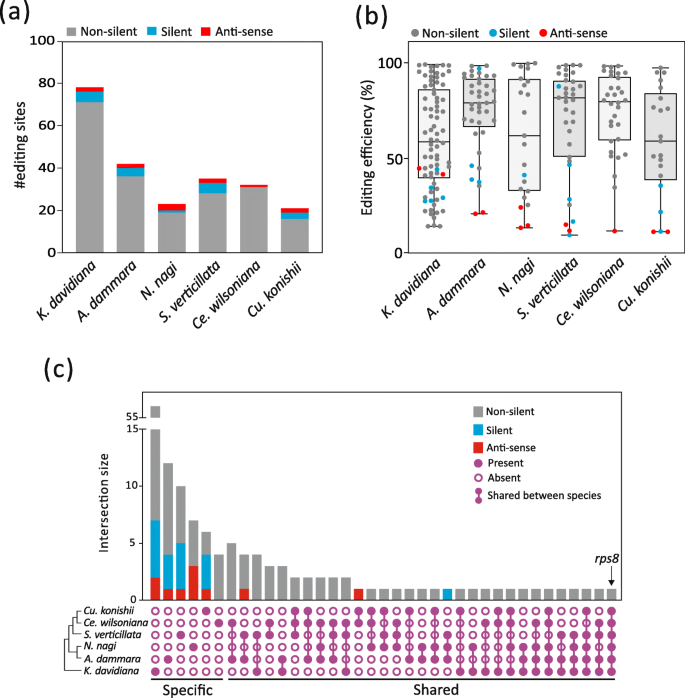
我们检测到78个C-to-U的rna编辑位点K. Davidiana叶绿体,42A. Dammara., 23n nagi, 35美国verticillata, 32Ce。wilsoniana, 21英寸铜。科尼希(无花果。4一个;表的年代1).值得注意的是,这些编辑位点中的大多数(76.2-96.9%)引起非沉默编辑,引入氨基酸序列的非同义变化。相比之下,同义密码子位置的沉默编辑位点仅占0-14.3%。此外,静默编辑站点的编辑效率几乎总是低于50%,只有两个例外:恩德的A. Dammara.和psbA的美国verticillata(无花果。4B;表的年代1).我们还在每个针叶树种的CDSs反义转录本中发现了1 ~ 3个编辑位点。这些站点是部分编辑的,效率低于50%(图。4b) 是的。
我们进一步研究了这些编辑站点之间的交集,基于它们的对齐。在无花果。4C,编辑过的位点在两个或多个物种中出现在相同的对齐位置时被指定为“共享”。那些只在单一物种中发现的被指定为“特定”地点。大多数沉默和反义编辑网站是特定的物种。只有一个地点位于rps8型转录-是所有针叶树共有的,这表明它起源于超过3亿年前所有针叶树的共同祖先[20.].此外,K. Davidiana与我们检测的任何其他物种相比,质体包含更多的物种特异性rna编辑位点,“特异性”位点的比例超过其他针叶树的两倍以上(图S7).该发现意味着Pinaceae在从Cufflecophytes发散后发生了一种独特的塑性RNA编辑位点。
讨论
我们使用链特异性RNAseq数据来探索所有6个针叶树科的质体转录组谱。我们的数据表明,针叶树质体几乎转录了两个DNA链的全部序列,这加强了这样一种观点,即在种子植物中,质体序列的完全转录是常态,而不是例外[8].我们注意到反义文本过多psbN位于高表达的psbB操纵子。这一发现提示质体基因的位置可能影响反义RNA的表达。此外,我们还发现了一些意义和反义转录本的C-to-U编辑位点。这些结果表明,链特异性RNAseq不仅可以消除定位过程中的反义污染,还可以探索反义转录本中的编辑事件,从而提高了rna编辑位点的检测。值得注意的是,所有反义位点的编辑效率都很低,这意味着它们可能是偶然的或组织特异性的[21,22,23].
我们还发现了大量的RNAseq reads定位于内含子、基因间隔子(IGSs)和CDSs反义区域。质体非编码rna被提出调节基因表达[24].在一些模式植物中,质体CDS和IGS转录本具有相似的表达水平[7].我们没有比较CDS和IGS之间的成绩单丰度,因为后者的转录取向是不确定的,使得难以以股线特异性方式识别相应的RNA匹达读数。尽管如此,我们确实观察到CDS的许多转录人反义。在体积中,假设反义转录物以结合mRNA的3'末端并稳定它们[25].这种稳定机制可能对所有CDS转录本都是活跃的,因为它们的反义对等体在针叶树质体中普遍存在。
人们早就知道,大多数质体基因的转录终止是低效的,因为它会导致大量和多样的通读转录,必须进行转录后处理[26].在最近的研究中[27],影响下游基因转录的透读转录机制,导致极端积累accD当上游基因的转录终止时,:,被灭活。在这里,我们建议,通过转录阅读也有助于解释我们的发现,即可塑性重排影响基因表达。重新定位一个基因涉及到重新配置其邻近的基因座,从而改变上游基因的读通转录效应。这最终改变了重新定位的基因及其下游邻近基因的转录本数量。此外,我们排除了系统发育效应可能导致基因表达和可塑性重排之间的关联,因为后者与取样针叶树之间的遗传距离没有显著相关。质体重排可能影响基因表达的发现也使得在高重排质体的转基因实验中确定插入位点变得非常谨慎。然而,如果没有环境胁迫处理,很难将质体重排引起的基因表达改变与生物适应联系起来。幸运的是,在一些针叶树谱系中已经记录了种间和种内的塑性体反转[本研究][18.,28,29,30.,31];为今后研究塑料重排与生物适应性之间的关系提供了理想的材料。
方法
植物材料,DNA和RNA的提取和测序
六种有代表性的针叶树种(即keteleeria davidiana为松科,Agathis dammara南洋杉科,Nageia Nagi.罗汉松科,日本金松Sciadopityaceae,Cephalotaxus wilsoniana红豆杉科对杉木konishii称来自柏科植物)由中国科学院生物多样性研究中心吴仲士博士(Dr. Chung-Shien Wu)采集鉴定。收集这些植物并不需要经过许可。代金券标本保存于台北中央研究院生物多样性研究中心植物标本室。表的年代2).
为了提取DNA和RNA,我们于2019年4月从10 - 30年树龄的树木中收集了约30厘米的新鲜嫩枝。为了降低不同生长条件下的潜在变异性,在生长室内(GC-550R, Yihder公司,新北市),25°C, 100 μmol m的光强下水培生长−2年代−1.24小时后,采集嫩枝上的新鲜叶片,用[32,33], 分别。提取的DNA在Illumina Hiseq 4000系统上以基因组学Biosci&Tech(台湾新台北市)测序。在DNA酶I(Invitrogen)治疗后,使用相同的系统,RRNA耗尽(Illumina Ribo-of rRNA去除试剂盒,植物叶版本)以及具有DUTP和随机六辐射的图书馆建设,使用相同的系统进行链特异性的RNA阵。表的年代2详细介绍了本研究中使用的取样地点、凭单编号、GenBank录入、DNAseq和RNAseq读取计数等信息。
质体组装和RNA定位分析
最初使用黑桃3.13进行塑料组件[34]选择“仔细”和一系列K-MER尺寸(21,33,55,77和99)。使用NCBI-BLAST 2.2.18鉴定塑性葡萄糖[35]针对内部数据库。间隔之间的contigs被关闭使用GapCloser 1.12 [36].这为所有取样的针叶树提供了完整的质体,除了K. Davidiana因为它的长松科I型重复[18.]. 我们随后设计了特异性引物(图1)扩增相应的区域并在后一种物种中进行基因组整理。
对于每个针叶树种,随机抽取2000万条对端RNAseq reads,使用TopHat 2.1.1将其映射到相应的质体上[37参数为:library-type = fr-firststrand, read-mismatches = 15, read-gap-length = 0, read-edit-dist = 15。Samtools 1.9 [38]用于对映射的读取进行排序、筛选和组合。生成的BAM文件被导入Geneous11.1.5(https://www.geneious.com)计算读取计数并进行下游分析。参考每个碱基的映射读数的RNAseq覆盖率使用血液覆盖的100bp非重叠滑动窗口计算,然后用公式转换:日志10.(覆盖率+ 1)/日志10.(最大覆盖范围+ 1)。
rna编辑位点的识别和可视化
为了识别C-to-U rna编辑位点,使用了Geneious 11.1.5中实现的“发现变异”选项,其阈值为:最小覆盖率= 50,最小变异频率= 0.1,最大变异P值= 10−6.编辑效率是通过计算已编辑的碱基与未编辑的碱基在映射读取中的比例来估计的。使用UpSetR评价6种针叶树编辑位点之间的交集[39].使用Circos 0.67可视化质体图谱、转录谱和rna编辑位点[40].
种系发生树结构
从6个组装的质体中手工提取83个同源CDSs(表S3.).使用MUSCLE 3.5对这些CDSs进行序列比对[41使用默认设置。利用RAxML 8.2.10对83个CDSs序列、GTR + G + I模型和1000个bootstrap复制序列进行串联,构建系统发育树[42].
结论
人们早就知道,针叶树中经常发生塑性重排。然而,对重排后质体基因及其下游邻近基因的表达动态尚未进行研究。在这项关键的研究中,我们发现在针叶树中(1)两个质体链都是完全转录的,(2)增加的质体排列减少了功能依赖的质体基因表达的强度,(3)RNA编辑发生在意义和反义转录,(4)松科植物从柏科植物分化出来后,进化出了一套独特的质体rna编辑位点。质体重排与基因表达的紧密联系使我们提出,通读转录可能是这种联系的关键。为了更好地理解针叶树塑料重排的生物适应性,还需要进一步的研究和分子生物学验证。
数据和材料的可用性
本研究分析的6个塑性数据存入NCBI数据库(https://www.ncbi.nlm.nih.gov/nucleotide/),注册号为LC571739、LC571740、LC571741、LC571883、LC572146和LC572147。
缩写
- cd:
-
编码序列
- IGS:
-
基因间的间隔
- TPM:
-
记录每百万
- 公关:
-
Plastomic重组
- 红外光谱:
-
反向重复
- ρ(ρ):
-
皮尔逊相关系数
参考
- 1。
Mofikoya Oo,Mäkenm,jänisj.通过超高分辨率傅里叶变换离子回旋质谱法通过回旋谐振质谱法测定针叶树针精油和溶剂提取物的化学指纹。ACS Omega。2020; 5:10543-52。https://doi.org/10.1021/acsomega.0c00901..
- 2.
Pfannschmidt T, Blanvillain R, Merendino L, Courtois F, Chevalier F, Liebers M等。质体RNA聚合酶:在植物生命周期中,不同进化起源的酶的编配控制着叶绿体的生物发生。实验机器人。2015;66:6957-73。https://doi.org/10.1093/jxb/erv415.
- 3.
叶绿体基因表达的转录后调控。Gene Regul Syst Bio. 2009; 3:31-47。https://doi.org/10.1104/pp.125.1.142.
- 4.
hoto AM, Schmitz RJ, Fei Z, Ecker JR, Stern DB。叶绿体非编码rna的深度测序揭示了叶绿体非编码rna的意外多样性拟南芥转录组。G3。2011; 1:559 - 70。https://doi.org/10.1534/g3.111.000752.
- 5.
Zhelyazkova P, Sharma CM, Förstner KU, Liere K, Vogel J, Börner T.大麦叶绿体的初级转录组:大量非编码RNA和质体编码RNA聚合酶的主导作用。植物细胞。2012;24:123-36。https://doi.org/10.1105/TPC.111.089441.
- 6.
Castandet B, Germain A, Hotto AM, Stern DB。对拟南芥叶绿体转录端进行系统测序,发现了>200转录起始位点和广泛的rna结合蛋白印迹和二级结构。核酸杂志2019;47:11889-905。https://doi.org/10.1093/nar/gkz1059.
- 7.
史超,王松,夏恩恩,蒋建军,曾飞飞,高丽贞。光合作用真核生物叶绿体基因组的全转录。Sci众议员2016;6:30135。https://doi.org/10.1038/srep30135.
- 8.
Sanitá Lima M, Smith DR.线粒体、质体和核型基因组的普遍转录跨越多种质体携带物种。基因组生物学进化。2017;9:2650-7。https://doi.org/10.1093/gbe/evx207.
- 9.
石桥K,小I,石垣T。被子植物质体RNA编辑的进化模式Amborella Trichopoda.植物细胞生理。2019; 60:2141-51。https://doi.org/10.1093/pcp/pcz111.
- 10。
植物叶绿体基因组研究。正确的人选:亨利·R,编辑。植物多样性和进化:高等植物的基因型和表型变异。剑桥,MA: CABI Publishing;2005.45 - 68页。https://doi.org/10.1079/9780851999043.0045.
- 11.
崔力,李本斯.麦克J,王LS,唐J,Rymarquis L,Stern DB,等.用参数自举方法推断叶绿体基因组结构的适应性进化。BMC进化生物学。2006;6:13.https://doi.org/10.1186/1471-2148-6-13.
- 12.
吴淑珍,周思敏。针叶树II支系(柏树)中叶绿体基因组高度重排和大小可变:向更短的基因间隔进化。植物生态学报;2014;https://doi.org/10.1111/pbi.12141.
- 13.
周思明,吴春生,苏甸托。裸子植物质体基因组的进化。入选:chow SM, Jansen RK, editors。植物研究进展。剑桥,MA:学术出版社;2018.p . 195 - 222。https://doi.org/10.1016/bs.abr.2017.11.018.
- 14.
苏甸托E,吴淑珍,周思敏。南半球柏科质体基因组缩小的起源与进化。植物科学与技术,2000,21(1):1 - 7。https://doi.org/10.3389/fpls.2020.00901.
- 15.
恒星,吴cs,chaw sm。在日本伞杉木的四个嵌合体积基因簇的诞生。基因组Biol Evol。2016; 8:1776-84。https://doi.org/10.1093/gbe/evw109.
- 16.
吴淑珍,周思敏。大型对比分析揭示了松柏类植物塑性压实、还原和倒置的驱动机制。基因组生物学进化。2016;8:3740-50。https://doi.org/10.1093/gbe/evw278.
- 17.
Strand-specific RNA-Seq提供了更高的转录组分析分辨率。咕咕叫基因组学。2013;14:173 - 81。https://doi.org/10.2174/1389202911314030003.
- 18.
吴cs,林cp,hsu cy,wang rj,chaw sm。PINACEAE的比较叶绿体基因组:多样化基因组组织机制的见解。基因组Biol Evol。2011; 3:309-19。https://doi.org/10.1093/gbe/evr026.
- 19.
韦斯特霍夫P,赫尔曼RG。叶绿体中复杂RNA的成熟。的psbB操纵子从菠菜。生物化学学报。1988;171:551-64。https://doi.org/10.1111/j.1432-1033.1988.tb13824.x.
- 20.
Leslie AB, Beaulieu J, Holman G, Campbell CS, Mei W, Raubeson LR等。从化石记录看现存针叶树的演化。Am J Bot. 2018; 105:1531-44。https://doi.org/10.1002/ajb2.1143.
- 21。
Sloan DB,MacQueen AH,Alverson AJ,Palmer JD,Taylor博士。快速进化过程中RNA编辑位点的大量丢失silene.线粒体基因组:选择与后处理作为驱动力。遗传学。2010;185:1369 - 80。https://doi.org/10.1534/genetics.110.118000.
- 22。
拟南芥基因家族在RNA编辑中的作用公共科学图书馆麝猫。2013; 9: e1003584。https://doi.org/10.1371/journal.pgen.1003584.
- 23。
陈廷,刘YC,王X,吴思,黄琴,昌蝉。整个塑体转录om揭示了丰富的RNA编辑位点和差分编辑状态蝴蝶兰属阿佛洛狄忒副formosana机器人钉。机器人钉。2017; 58:38。https://doi.org/10.1186/s40529-017-0193-7.
- 24.
Hotto Am,Germain A,Stern DB。塑性非编码RNA:基因调节的新兴候选者。趋势植物SCI。2012年; 17:737-44。https://doi.org/10.1016/j.tplants.2012.08.002.
- 25.
关键词:维管植物,叶绿体,rna,稳定因子学生物化学论文。2018;62:51 - 64。https://doi.org/10.1042/EBC20170061.
- 26.
Stern DB, Goldschmidt-Clermont M, Hanson MR.叶绿体RNA代谢。植物生物学。2010;61:125-55。https://doi.org/10.1146/annurev-arplant-042809-112242.
- 27.
Ji D,Manavski N,Meureer J,Zhang L,Chi W.受管制叶绿体转录终止。Biochim Biophys Acta Bioenerg。2019年; 1860:69-77。https://doi.org/10.1016/j.bbabio.2018.11.011.
- 28.
Guo W,Grewe F,Cobo-Clark A,Fan W,Duan Z,Adams Rp等。2014.体液基因组共存的主要和替代异构体突尼鹦鹉在柏树进化过程中,植物和柏树发生了多次转移。基因组生物学进化。2014;6:580-90。https://doi.org/10.1093/gbe/evu046.
- 29.
苏利文AR, Schiffthaler B, Thompson SL, NR街,王小瑞。种间质体重组反映了中国古代网状结构的进化云杉(松科)。中国生物医学工程学报2017;34:1689-701。https://doi.org/10.1093/molbev/msx111.
- 30.
曲晓军,吴春生,周思明,易世生。柏科中异构体存在的初步认识。植物生理学杂志。基因组生物学进化。2017;9:1110-9。https://doi.org/10.1093/gbe/evx071.
- 31.
付春,吴春生,叶丽娟,莫志强,刘杰,常永伟,等。紫杉中同分异构体的流行率和质体超级条形码的有效性(水松全球)。Sci众议员2019;9:2773。https://doi.org/10.1038/s41598-019-39161-x..
- 32.
Stewart CN Jr, Via LE。快速CTAB DNA分离技术用于RAPD指纹和其他PCR应用。生物学技术。1993;14:748-50。
- 33。
关键词:生物化学,生物化学,生物化学,生物化学,生物化学裸子植物和被子植物优质RNA的分离。生物学技术。2004;36:821-4。https://doi.org/10.2144/04365st06.
- 34。
(1)研究结果表明,该方法具有良好的应用前景。一种新的基因组组装算法及其在单细胞测序中的应用。计算机生物学报。2012;19:455-77。https://doi.org/10.1089/cmb.2012.0021.
- 35。
关键词:聚类,聚类,聚类,聚类BLAST+:架构和应用。BMC生物信息学。2009; 10:421。https://doi.org/10.1186/1471-2105-10-421.
- 36.
罗锐,刘斌,谢勇,李忠,黄伟,袁军,等。SOAPdenovo2:一个经经验改进的高效内存短读从头汇编器。Gigascience。2012;一18。https://doi.org/10.1186/2047-217X-1-18.
- 37.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:转录组插入、缺失和基因融合的精确对齐。基因组医学杂志。2013;14:R36。https://doi.org/10.1186/gb-2013-14-4-r36.
- 38.
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N等。序列对齐/映射格式和SAMtools。生物信息学。2009;25:2078-9。https://doi.org/10.1093/bioinformatics/btp352.
- 39.
Conway JR, Lex A, Gehlenborg N. UpSetR:一个用于交叉集可视化的R包及其性质。生物信息学。2017;33:2938-40。https://doi.org/10.1093/bioinformatics/btx364.
- 40.
Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D等。Circos:比较基因组学的信息美学。基因组研究》2009;19:1639-45。https://doi.org/10.1101/gr.092759.109.
- 41.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸杂志2004;19:1792-7。https://doi.org/10.1093/nar/gkh340.
- 42.
Stamatakis a . RAxML version 8:用于系统发育分析和大型系统发育后分析的工具。生物信息学。2014;30:1312-3。https://doi.org/10.1093/bioinformatics/btu033.
致谢
我们感谢三位匿名评论员的批判性阅读和宝贵评论。感谢台北植物园和台北花卉实验中心提供的植物材料。
资金
本研究由台湾科技部资助(MOST 106-2311-B-001-005),中央研究院生物多样性研究中心资助(s - m.c),部分博士后资助(ES)。资助机构没有参与研究的设计、收集、分析和解释数据,也没有参与手稿的撰写。
作者信息
从属关系
贡献
CSW和SMC构思和设计了这项研究。CSW执行了实验并分析了数据。CSW,es,SMC写了稿件。所有作者均阅读并批准了手稿。
作者的信息
中央研究院生物多样性研究中心,台北11529。
相应的作者
道德声明
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
附加信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
表S1。
本研究检测到质体C-to-U编辑位点。
附加文件2表S2。
本研究中使用的针叶树种类、凭证编号、DNAseq和RNAseq数据。
附加文件3表S3。
本研究利用83个同源cds构建系统发育树。
附加文件4图S1。
Plastomic同分异构体在K. Davidiana.(a)质体比较显示松科I型倒置重复序列(IR)的侧翼是种内倒置。指出了用于检测特定异构体的引物。(b)半定量PCR显示了两个具有不同拷贝数的异构体共存。
附加文件5图S2。
被链RNAseq所覆盖的质体序列的百分比。
附加文件6图S3。
在针叶树质体中CDS转录本及其反义对等体的RNAseq覆盖。使用Log转换覆盖率分数10.(1 +保险)。虚线表示对角线。如果信用违约互换的成绩单的覆盖分数小于反义对应的信用违约互换,则表示信用违约互换。
附加文件7图S4。
针叶树中广泛的塑料重排。(a)在6个取样的针叶树质体中鉴定出31个同生区(色盒)。(b) 6个针叶树质体的点图分析。
附加文件8图S5。
Pearson的相关试验表明,塑性重排与采样针叶树之间的遗传距离没有显着相关。
附加文件9图S6。
Pearson相关检验表明,基因的塑性重排与同源基因表达相关程度呈显著负相关。
附加文件10图S7。
6个代表性针叶树质体中特异性和共享rna编辑位点的比例。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用本文
吴,CS。,Sudianto, E. & Chaw, SM. Tight association of genome rearrangements with gene expression in conifer plastomes.BMC植物杂志21,33(2021)。https://doi.org/10.1186/s12870-020-02809-2
收到了:
接受:
发表:
关键词
- 针叶树
- 质体转录组
- Plastomic重排
- Strand-specific RNAseq
- rna编辑