抽象的
背景
种子的物理休眠是豆科植物驯化的一项重要性状。虽然种子休眠在野生生态系统中是有益的,但由于产量和/或质量的降低,它通常被认为是作物的不良特性。在一些豆科植物中,种子休眠的生理机制和潜在的遗传因素尚不清楚。本文采用综合的方法来了解控制普通大豆种子物理休眠的机制(phoudolusulus vulgaris.l .)。
结果
使用一种创新的CT扫描成像系统,我们能够追踪水在种皮内的运动。我们发现水分的吸收始于大豆种子的晶状体。利用扫描电镜(SEM)进一步鉴定了非休眠大豆基因型晶状体表面的几个微裂纹。对一个双亲本RIL(重组自交系)群体进行了大量分离分析(BSA),用于种子休眠分离。结果表明,种子吸水与Pv03上的一个主效QTL有关。该QTL区位于118kb区间内,共有11个基因。对候选基因的编码序列分析显示,在一个同源基因中有一个5-bp的插入果胶乙酰酯酶8.这导致帧移位,非休眠基因型中的功能突变突变。对比基因型种子涂层中候选基因的基因表达分析表明21倍以下果胶乙酰酯酶8.在休眠基因型。对野生大豆和驯化大豆进行了基因多态性分析。虽然所有的野豆子都具有果胶乙酰酯酶8.在驯化后的大豆中,绝大多数(77%)存在非功能等位基因,表明该变异在驯化过程中面临着很强的选择压力。
结论
在这项研究中,我们确定了物理种子休眠的生理机制,并确定了候选等位基因,导致这种特征变异。我们的研究结果表明,在Ortholog中的5-BP插入果胶乙酰酯酶8.在驯化期间,可能是种子休眠损失的主要致病性突变。虽然目前的研究结果为此方面的作用提供了强烈的证据果胶乙酰酯酶8.在种子休眠方面,似乎有必要利用转基因方法进一步证实。
背景
植物的驯化是农业出现和人类文明兴起的关键一步[1].驯化过程导致植物适应人类管理的农业环境。这些适应发生在野生物种中,用于基因型内的野生物种,具有一套特征,在这些新的栖息地提供了一种选择性优势。在许多情况下,类似的选择实践导致跨物种驯化性状的表型趋同[2].大多数作物常见的驯化性状包括种子失落、种子休眠和萌发更同步[2那3.那4.那5.那6.].虽然驯化特征在农业生态系统中有利,但它们对自然栖息地的健身施加权衡[1那7.那8.].
减少种子休眠是一个重要的过程,通常与驯化有关[2那3.那9.].在自然环境中,种子破裂休眠至关重要的时间,因为错误时间的发芽可能导致生存和健身减少[9.那10.].除了与环境同步外,种子休眠涉及种子分散,并在减少母亲和后代之间的资源冲突中[11.].在这种情况下,休眠较长的种子可以在时间和空间中更广泛地分散,因此减少了母亲和后代和/或后代之间的资源冲突。
尽管在自然环境中具有优势,但延长的种子休眠对作物来说并不是一个理想的特性[2那3.那9.].在栽培的豆类中,种子休眠降低萌发率,导致萌发不均匀,因此降低了产量[12.那13.].此外,种子的物理休眠(见下文)会对种子的水分吸收产生不利影响,而水分吸收在豆科植物的食品加工中很重要[9.].虽然高水平的休眠是不可取的,但低水平的种子休眠可能会给作物带来问题,因为种子在收获前就发芽了,这一过程在谷物中被称为胎生[14.]和豆类[15.].胎生在有利于萌发的环境中是一个主要问题。因此,在快速萌发和种子休眠之间达到最佳平衡对于最大的农业产量和粮食加工至关重要[9.].
几种机制促进种子休眠[16.].在诱导和维持休眠中起着激素相互作用,特别是脱落酸(ABA)和赤霉素(GA)的生理休眠,在诱导和维持休眠中起重要作用,并被认为是种子休眠最常见的机制[16.].种子的物理特性,尤其是种子涂层,也可以通过防止水的吸收来促进休眠较长的休眠。这种物理休眠是最灵感的限制,被认为是栖息地专业化的适应机制[16.].
生理休眠是豆科植物休眠的主要原因,而生理休眠是一些豆科植物和非豆科植物休眠的一个因素[17.].
物理种子涂层不渗透性的潜在机制在豆类物种之间变化。虽然潜在的遗传机制在很大程度上是未知的,但休眠水平的变化由一些豆类物种中的几个基因座控制[4.那18.那19.那20.那21.那22.].从生理角度来看,不透水的种子通常具有硬,果白层的拨打细胞[23.或种皮中木质素含量较高[24.].种子的不透气性也可能与种皮角质层的脂肪酸组成有关[25.].豆科植物种皮起源于母组织,由两个卵珠被(双珠珠胚珠,[26.])。内层在种子发育过程中基本消失[27.].然而,外层发育成种皮,在休眠的种子中变得不透水。种子的物理休眠可以通过种皮上微裂纹的发展来减少[28.].
常见的bean (phoudolusulus vulgaris.L.)是直接人类消费的重要豆科物种。在驯化之前,P.寻常魅力从墨西哥的原产地迁移到中美洲和南美洲。P.寻常魅力然后在中间人和安第斯山脉独立驯化[29.那30.].在不同的大陆的农民成功收养普通豆子,突出了其巨大的遗传多样性和广泛适应。虽然初期的Andean农民(2500年前直到600年前)保持遗传多样性,现代育种实践导致培养的Andeans遗传多样性造成了很大的遗传多样性[31.].
在现代常见的豆类生产中,豆类具有高,均匀的水吸收率被认为是理想的[32.].高水分吸收减少了烹饪时间。此外,水吸收率影响溶胀容量,确定可以从干酪单位生产的罐的数量[33.].
在之前的研究中,我们发现在洪水条件下,种子吸胀率最低的基因型存活率最高[34.那35.].这一发现表明,在种子快速吸水和易受洪水影响之间存在着一种权衡。此外,种子休眠可能有利于自然种群P.寻常魅力通过在不利的环境条件下预防萌发之前驯化之前,在许多其他植物物种中进行萌发[16.那36.].尽管对加工和烹煮豆类有快速吸水的好处,但一些驯化的豆类品种仍然保持着高水平的种子休眠[34.那35.那37.那38.使它们更容忍洪水。
为了研究不同驯化大豆品种种子吸水率差异的遗传和生理基础,我们集中研究了两种具有代表性的常见大豆基因型,它们的种子吸水率差异很大[35.], pr9920 - 171 [39.]及TARS-HT1 [40].PR9920-171是一组Andean基因型中萌发阶段对淹水最耐受的基因型[35.].TARS-HT1是一个深红色的肾,PR9920-171是一种斑驳的浅红肾,随着改善的种质而具有显着耐受性的种质。PR9920-171和TARS-HT1都来自于与不确定的牙买加红色(以下IJR,PI 163122)的交叉(以下是一种缓慢的基因型,具有高水平的洪水和耐热性。
在这项研究中,我们比较了PR9920-171和TARS-HT1的种子特征,并对这两条线之间的交叉进行了遗传分析,以解决四个主要问题:一世)大豆种子休眠的生理机制是什么?2)在发芽期间,如何增加休眠休眠对应于洪水的较高耐受性?III)种子休眠的遗传基础是什么?和4)在驯化大豆种子休眠差异背后的相同遗传机制是否也导致了它与野生群体的差异P.寻常魅力?
结果
种子萌发与吸胀
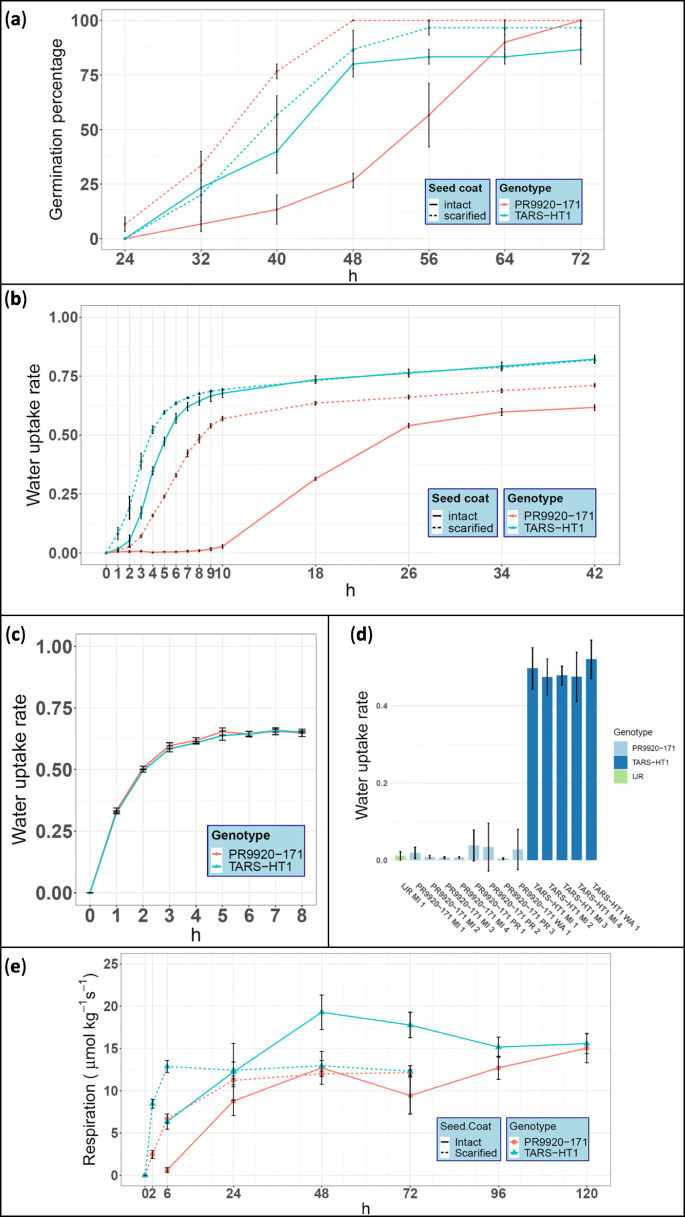
PR9920-171种子萌发试验表明,PR9920-171具有较强的物理休眠,可通过种皮划痕恢复。1一种)。在浸泡后48小时观察到萌发百分比最激烈的差异。在此时间点,虽然PR9920-171种子与完整的种子涂层显示出25%的萌发,但造成PR9920-171至100%的萌发增加。在同一时间点,TARS-HT1萌发〜77%。
TARS-HT1与PR9920-171在物理休眠方面的差异。一种萌发测定对具有完整和划痕种子涂层的两种基因型进行。该实验表明,PR9920-171拥有强大的身体休眠。B.对种皮完好和有疤痕的两种大豆进行了吸吸试验。与PR9920-171大豆相比,tar - ht1大豆吸水速度更快。种皮磨损显著增强了两种基因型大豆的吸胀,PR9920-171表现出较强的物理休眠。C在去除种子涂层后,子叶的吸收测定。对于子叶的吸入率没有检测到显着差异,表明种系为PR9920-171的物理休眠的主要贡献者。D.2019年MI、WA和PR 3个田间环境下种植的PR9920-171和tas - ht1大豆吸水率。在所有位置,PR9920-171的自吸速率都明显低于TARS-HT1。两种基因型亲本IJR均表现出与PR9920-171相似的缓吸表型。每个位置前的数字表示复制编号。E.PR9920-171和TARS-HT1基因型浸泡后不同时间点的种子呼吸测量。与PR9920-171种子相比,TARS-HT1种子中的呼吸率较快。浸泡后,Tars-HT1的完整种子在48小时内达到了最大呼吸能力。相比之下,甚至在浸泡120小时后,PR9920-171中的呼吸速率也不会降低呼吸。在小组中一种那B.那C,E.,红、蓝线分别表示PR9920-171和tar - ht1。实线和虚线分别表示种皮完好和破损的豆子
种皮完好的tar - ht1种子在浸泡2小时内开始吸水(图。1b).相比之下,PR9920-171种子的第一个迹象是在浸种开始后10 h左右观察到的吸胀。对于这两种基因型,我们观察到,与种皮完整的相比,种皮擦伤显著增加了吸胀率。PR9920-171种子的吸胀速率增强尤为明显,表明该基因型的种皮对水分进入种子造成了更大的障碍。在浸泡后8 h和26 h, TARS-HT1和PR9920-171的吸胀速率分别达到稳定水平,但水平差异显著。种子质量的进一步增加与胚根伸长有关,而不是与吸胀有关。
为了测试基因型之间的子宫子摄取率的子叶内的假设,在没有种质涂层的情况下对子叶中进行吸收测定。对于两种基因型,在浸泡的4小时内达到子叶的最大吸收(水吸收)(图。1c)无种皮子叶的吸胀率无显著差异,进一步证实种皮是PR9920-171吸胀率较低的唯一原因。
3个地点的田间评价表明PR9920-171种子休眠的稳定性(图1)。1d)。平均而言,在三个地点上,Tars-HT1种子中的水吸收大于PR9920-171种子吸收超过5倍。此外,PR9920-171吸收类似于IJR吸收。
种子的呼吸作用
我们观察到,种皮完整的tar - ht1种子在浸泡48 h后达到了最大的呼吸量(图。1E),并在96 h试验的剩余时间内下降。PR9920-171完整种子的呼吸速率在48 h内也有所增加,但水平较低,在试验结束时达到与tas - ht1相同的水平(15.0±1.7 μmol kg)- 1年代- 1在120 h)。这表明PR9920-171种子需要更长时间才能达到浸润条件下的最大呼吸能力。与具有完整种子涂层的种子相比,含有划伤的种子涂层的种子的呼吸有很大的轨迹。淹没的tars-ht1种子达到呼吸能力(12.9±0.7μmolkg- 1年代- 1)在6小时内浸泡,留在该高原直至72小时。然而,PR9920-171的呼吸速率增加较慢。
镜片结构是豆种子的潜在部位
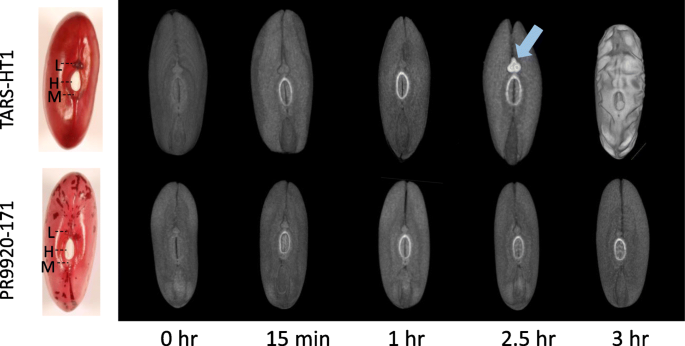
30分钟后,在CT扫描的种子中检测到慢性和快速吸收基因型的CT扫描种子周围的较高密度区域(图。2).然而,这种最初的水分吸收并没有伴随着进一步的水分吸收。浸泡后2.5 h左右,快速吸收基因型的晶状体因较高的水碘吸收而区别于种子的其他部分(图。2).然而,缓慢吸收基因型中的种子在该时间跨度中没有吸水。这表明这些常见的Bean基因型中的吸收与通过透镜结构的初始水吸收相关。透镜或溶液在普通豆睾丸中显示为两个膨胀结构。我们通过指甲油阻止了两种基因型的镜片,并评估了它们的吸收。镜片堵塞似乎似乎无法防止在Tars-HT1种子中吸收。但是,在PR9920-171中,镜片堵塞完全防止种子吸收(图S2).这一结果表明,在具有强生理休眠的基因型中,晶状体对吸胀起着重要作用。
快速吸收基因型在种子透镜表面上具有更多微裂缝
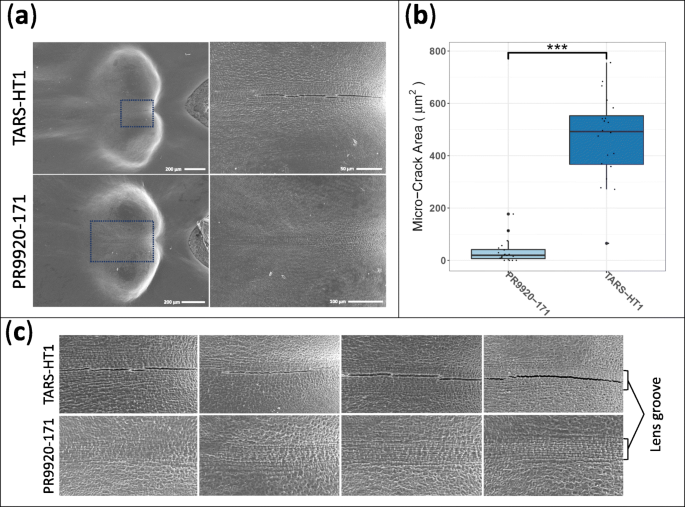
SEM显示快速吸收的种子具有透镜表面旁边的几个微裂纹(图。3.一种)。我们发现微裂纹区域(μm2)在TARS-HT1种子的晶状体表面的含量约为前者的14倍(P. < 0.001,N = 20) than in the slow-imbibing genotype (PR9920–171, Fig.3.b).这些微裂纹出现在tas - ht1的透镜槽旁(图5)。3.C)。
Pv03上的一个主要QTL (P.寻常魅力染色体3是种子休眠变异的基础
我们在RIL群体(PIC-76,有关详细信息的更多细节的方法部分,雇用了膨胀的分析分析(BSA)方法,从慢速(PR9920-171)和快速吸收(TARS-HT1)基因型之间导出,鉴定与种子吸收相关的基因组区域。慢速和快速的吸收池,每个池包含87和90个个体,分别对深度进行测序,分别为116.7倍和110.7倍。总共检测到332,689个SNP。SNP变异在染色体中没有同等分布(表1,图S3).
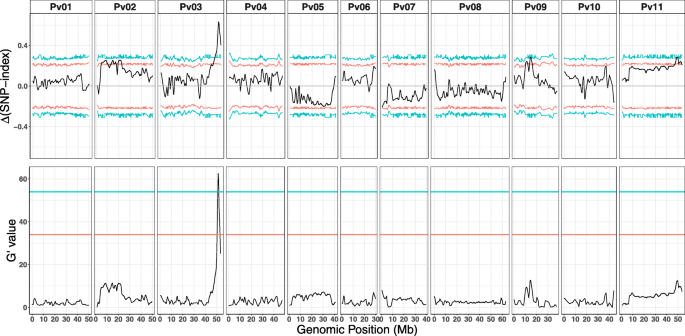
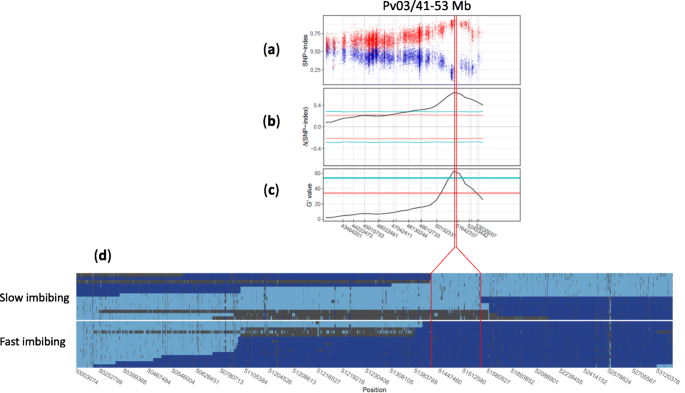
全基因组BSA在Pv03/43.1-53.4 Mb (FDR = 0.05)之间的10mb区间有一个显著的QTL,峰值位于51.73 Mb(图)。4.).该QTL的峰值通过ΔSNP方法的99%置信区间阈值,其g素值为65.9,表明两个库之间的等位基因频率存在较强的差异(图5)S3).进一步的分析表明,在PV03 / 43.1-53.4 MBP上的主要QTL内的TARS-HT1等位基因在快速吸收的池中更频繁地频繁,并且在慢速吸收池中的频率较少。
在PIC-76(源自PR9920-171 × ars - ht1)的一个包含384个ril的亚群上进行QTL精细定位。我们首先将QTL区域缩小到大约1.6 Mb,位于KASP-50.1 Mb和KASP-51.6 Mb之间(图)S4).然后,我们重新测序30个个体,其中在KASP-50.1和KASP-51.6之间发生重组事件。该序列数据允许我们筛选在PV03(53.4Mb)的末端50mbp之间的基因型区域的单倍型组合物。在该地区检测到总共2460个SNP。快速和缓慢的个体的整个基因组测序显示在51,426,054和51,544,057之间的〜118 kB区域(图。5.).在该地区有三种杂合,并用缓慢的个体进行表型分组。在该〜118 kB区域内,鉴定了11个基因 - 包括编码推定果胶乙酰酯酶8蛋白的基因的串联复制(表2).
Pv03末端QTL精细定位。一种QTL区快吸库和慢吸库的等位基因频率差异。每个点代表快速吸收(红色)和缓慢吸收(蓝色)池子中的ars - ht1等位基因频率。B.-C表示检测到的QTL区域,使用ΔSNP-index (B.)和g'(C分别)方法。D.30个体的单倍型图是整个基因组测序以缩小QTL的位置。基于它们的水吸收表型分离各个人。检测到118 kBP区域(被红线包围),其具有缓慢的基因型中的PR9920-171等位基因(浅蓝色)。相同的区域具有快速吸收基因型的TARS-HT1等位基因(深蓝色)。检测到三种杂合子(暗灰色)在118kbp区域中,表达缓慢的表型。红线突出显示初始BSA中的精细映射区域及其对应位置
候选基因的编码序列分析
在精细定位区域的4个基因的外显子区检测到7个变异。所有外显子区域内的点突变都导致非同义置换。的唯一外显子中检测到一个非同义点突变phvvul.003g277400.,它编码一个四肽重复序列。这个点突变导致了一个氨基酸变化(T➔M)在658aa的位置假定的蛋白质。我们将该蛋白与同源蛋白序列进行比对,发现该变化位于非保守区域,因此不太可能产生深刻的功能影响(图)S5).
基因的第3外显子中检测到4个非同义点突变phvvul.003g278200.,它编码长链脂肪醇氧化酶−4 (FAO-4,图S6).第275位(G➔D)和第500位(I➔F)的aa突变均位于该基因较保守的区域。443 aa (I➔V)突变位于保守区域。然而,PR9920-171 (I)和tar - ht1 (V)等位基因在同源基因中分布均匀,表明这两个等位基因都具有功能。我们发现FAO-4在359位的一个aa变化是在基因中较为保守的位置。在这个位置,TARS-HT1编码亮氨酸(L),而蛋氨酸(M)是这个位置的主要aa。然而,这两个aa都具有疏水侧链,这种取代不太可能对蛋白质构象造成重大修饰。
在最后一个外显子(第7)处检测到另一个非同义点突变(P➔A)phvvul.003g278400.位置441 aa。该基因编码细胞色素p450 - 707- a -多肽-1 (CYP707A1)。我们发现该突变位于非保守区域内(图S7).
我们在第6外显子中发现了一个5-bp的插入PHVVUL.003G277600.在tars-ht1中,这是第二个果胶乙酰酯酶8.(PAE-8-2).该插入在tas - ht1 PAE-8的182 aa处引入了一个移码,并导致在199 aa处引入一个未特征的密码子和一个可能被截断的蛋白质(图)S8).该基因在其他物种中与同源基因的对准表明,该帧突变导致近一半的蛋白质损失包括几个保守域(图S8).我们设计了特异性引物,从cDNA模板中扩增了indel位点,证实了该基因在两种基因型中的表达。
种子涂层中候选基因的比较表达分析
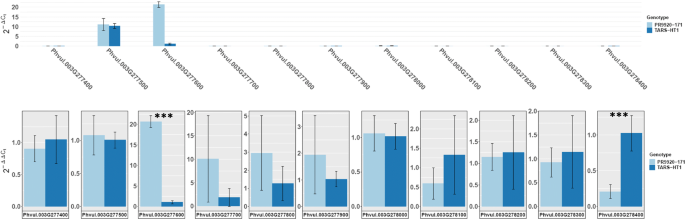
为了评估PR9920-171和TARS-HT1种子涂层之间是否存在候选基因的差异表达,我们进行了RT-QPCR。该分析显示phvvul.003g277500.(基因型)和PHVVUL.003G277600.(在Pr9920-171中,与其他基因模型相比,在种子涂层中基本上表达(图。6.).有趣的是,这两个基因都与拟南芥基因同源AT4G19420编码在细胞外区域中表达的果胶乙酰酯酶8。虽然基因型之间没有检测到表达差异phvvul.003g277500.(PAE-8-1),其表达量在tst - ht1种皮中降低约21倍PHVVUL.003G277600.(PAE-8-2).Tars-HT1中的5-BP插入可能导致转录后基因调节,导致Tars-HT1种子涂层的PAE-8-2转录物的稳定性较低。
PR9920-171和tar - ht1种种皮中11个基因的表达谱。上面的面板显示了使用2的11个基因的表达水平-Δct..高表达phvvul.003g277500.和PHVVUL.003G277600.,编码果胶乙酰酯酶8的编码直肠。底部面板代表2-ΔΔct反映了各基因的相对表达倍数变化。PR9920-171与TARS-HT1之间存在不同的相对表达PHVVUL.003G277600.和phvvul.003g278400.(星号)。的高表达(~ 21倍)PHVVUL.003G277600.在PR9920-171种皮中检测到相对于tar - ht1。而TARS-HT1表达量较高phvvul.003g278400.相比pr9920 - 171。对于两个面板,深蓝色和浅蓝色分别代表TARS-HT1和PR9920-171
野生和驯养豆类PAE-8等位基因频率的差异
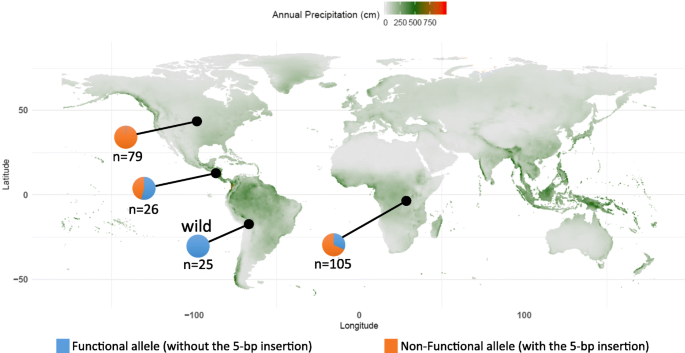
我们预测,如果一个基因参与驯化,我们可以预期,该基因的等位基因频率将在野生和驯化大豆种群之间有很大的差异。为了检验这一预测,我们评估了移码突变等位基因在安第斯多样性小组的一个子集和野生安第斯豆中的等位基因频率分布(图。7.).结果表明,所有野生大豆均具有PAE-8-2等位基因(PR9920-171版本,未插入)。相比之下,大多数(77%)的驯化基因型具有插入5 bp的非功能等位基因(ars - ht1等位基因)。这一插入在地理区域间有差异分布。在北美地区的基因型中,插入5 bp的等位基因几乎是固定的(99%)。然而,在非洲驯化的安第斯豆(68%)和中美洲驯化的安第斯豆(46%)中收集的基因型中,这种等位基因的频率较低。
讨论
在这项研究中,我们发现通过种子上的透镜结构开始进入种子涂层。遗传分析表明,PV03的单个主要QTL在种皮涂层不渗透性下劣化。基于细映射,表达和单倍型分析,我们得出结论,一个5-BP框架突变果胶acetylesterase-8-2是这种种子吸收QTL的候选因果变量。这种框架突变在野生动物豆内不存在,同时在耕种豆内更频繁。总体而言,这些结果表明,普通豆的人类驯化为快速种子饮水的强烈选择潜在地引起了一个非功能变体的等位基因频率的重大变化果胶acetylesterase-8直接同源。
种子休眠在豆类中的重要性
种子休眠在豆类中,特别是在共同的豆中,与他们的演化和驯化是交织的[4.].物理休眠,即种皮不透水,是豆科植物最普遍的休眠形式。在目前的研究中,我们发现在非休眠基因型中缺乏生理休眠与快速的水分摄取和呼吸速率有关。如果淹水(浸泡)持续,非休眠的TARS-HT1种子的呼吸速率较其早期呼吸速率下降,可能是由于早期较高的呼吸速率和O2在环境中。而休眠的PR9920-171种子维持呼吸能力的时间较长,在淹水结束时萌发率较高。说明生理休眠有助于提高水淹胁迫的耐受性。然而,低的种子吸胀率在作物生产中是一个不良的特性,因为它降低了同步萌发,增加了蒸煮时间[32.].因此,Andean多样性小组中大多数驯养豆类缺乏强烈的身体休眠,因此易受繁殖的洪水影响[35.].
种子水吸收从透镜开始
通过共同豆类中的许多不同结构可以发生种子水吸收,种子水的位置在基因型中变化[38.].在这里评估的基因型中,我们发现种子的水分吸收始于透镜。在具有最高水平物理休眠的基因型中,晶体结构对于吸胀尤为重要。此外,透镜槽中的微裂缝可能是水进入这些种子的最初入口点。同样,Hradilova等人[5.]报道更多污水的透镜在非休眠豌豆的镜片(PisumSPP。)。额外的研究发现,镜头是一些豆类物种中的初始进入的初始站点[43.那44.那45.那46.那47.那48.那49.].然而,不同豆科植物的种子吸水机制不同[47.].
种子休眠是驯化综合征的一部分,与Pv03上的一个主要QTL有关
驯化综合症[50.]被定义为栽培作物和野生祖先之间的一组共同的表型差异[3.那4.].在种子作物中,种子休眠的损失通常是在驯化第一阶段选择的第一个特征中[3.].几项研究表明,归统综合征涉及的特性由主要效果的基因率很少控制[4.那19.那51.那52.].
在常见的豆中,统计综合征涉及若干特征,包括种子破碎,种子休眠,测定,种子大小和光周期不敏感性[4.那53.那54.].其中,种子分散(破碎)和休眠是区分野生和驯养豆类的最重要的特征[4.].koinange [4.发现,四种未解释的基因座控制的种子休眠。他们研究中的最大效果(解释的差异的52%)也位于PV03上。我们从Koinange研究中爆炸了QTL侧翼标记,并能够以40.0 MB和53.2 MB的间隔映射。在我们的研究中,在51.4和51.5 MB之间检测到单个QTL,并与Koinange等人报告的主要QTL共同定位。种子休眠。在两个单独的研究中鉴定这种主要QTL控制种子休眠的休眠强烈表明种子水摄取是休眠的关键组成部分。在两个黑豆种群中也研究了种子吸收,并发现了PV07上的主要QTL [32.那55.].这不是出乎意料的,因为多条证据表明,整体驯化与两个基因库中不同基因座的选择相关联[56.],发芽阶段的泛滥耐受由普通豆类的驯养基因库中的不同基因座控制[34.那35.].还观察到两种基因库中不同选择的其他实例用于确定性鳍位点(57.],是颜色表达基因的主调控基因P.[58.].
在我们的研究中检测到的主要QTL位于轨迹附近(〜3.5 MB),该轨迹控制常见豆类中的POD [54.].在扁豆还报道了种子休眠和豆荚粉碎之间的遗传联系[19.].在其他非豆科作物中报道了QTL控制若干驯化性状的QTL的共定位,例如珍珠米[59.]和大米[60.].控制驯化综合征重要性状的基因之间的联系可能是驯化的一个重要特征[3.].
果胶乙酰酯酶5-bp的插入可能是破坏种子休眠的原因突变
在我们的研究中,种子水分吸收QTL精细定位区候选基因编码区中功能代价最大的突变是一个5-bp的移码突变PAE-8-2基因。果胶在胶凝细胞中起着关键作用[61.].基于这一假设,中间层中的果胶是一种可溶性的形式,可以被高温和/或水解聚。当解聚发生时,细胞可以彼此分离。在不溶性的形式下,果胶更耐水吸收,促进中间板层的稳定性和细胞在吸水过程中分离的失败。Kigel [62.建议有几种机制可以有助于果胶不溶性和豆类中的难以烹饪现象。果胶聚合物的乙酰化已被证明通过空间障碍通过钙离子降低果白的交联,因此它们的溶解度增加了[63.].然而,乙酰基可以通过PAE.有助于细胞壁硬质的基因[64.].果胶去乙酰化,通过过表达PAE.,降低果胶酶的果胶聚合物的消化率[65.].此外,已经证明了绿豆的过度表达(豇豆属辐射L.)PAE.在土豆(Solanum Tuberosum.L.)结果与纤巧的细胞壁的马铃薯块茎[66.].乙酰化水平也可能影响疾病的抗性[67.或在食品加工中很重要的果胶的胶凝能力[64.].
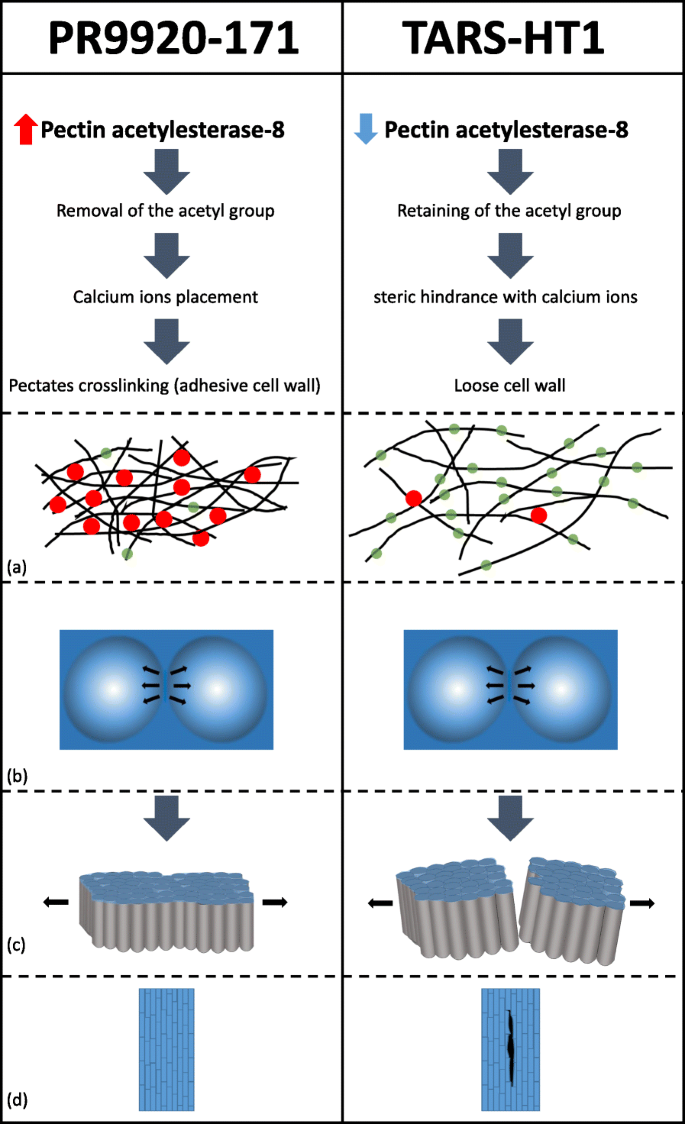
我们假设更高的表达PAE-8在PR9920-171中,与果胶中的乙酰基的除去增加相关,因此细胞粘附的增加(图。8.).该假设与我们的观察结果一致,即Tars-HT1在透镜槽内具有几个微裂缝,可能是由于非功能性PAE-8-2和较低的细胞粘附性。这种机制可能在豆类内普及,因为它被观察到在野生普拉沙德细胞中不溶性果白层PisumSPP。种子与水不渗透有关[23.].此外,提出果胶交联特性是种子休眠在野外的主要贡献者PisumSPP。[5.].
休眠中种子涂层渗透性的假设模型(PR9920-171)和非休眠(Tars-HT1)豆类。在物理休眠种子中果胶乙酰酯酶-8的更高表达导致从果胶聚合物中去除较高的乙酰基速率。这有利于替代钙离子和果胶聚合物的交联,将果胶转化为不溶性和强形式。果胶聚合物的强粘附性抵抗普拉迪斯细胞对镜片结构的施加张力的分离。然而,在非休眠种子(Tars-HT1)中,5bp插入导致果胶乙酰酯酶-8-2中的功能突变突变。这种功能丧失导致剩余的乙酰基与钙离子引起空间障碍。该条件中果胶聚合物中的降低钙介导的交联导致放松的细胞壁,其更容易发生微裂纹。肿胀部分透镜的张力导致镜片槽中的拨型电池彼此分开,并在松散的非休眠种子涂层中发育微裂纹。一种细胞壁内果胶纤维的示意图。黑色实线代表果胶纤维。绿色透明圆形代表乙酰基,红色圆圈代表有利于果胶聚合物的交联的钙离子。B.晶状体上的透镜的示意图。黑色箭头表示透镜槽电池上透镜的膨胀结构施加的可能张力。C位于镜片槽内的拨栅电池。黑线表示这些细胞上透镜的溶胀结构施加的可能张力,这导致在非休眠种子涂层中的细胞分离。D.在非休眠种子中分离的普拉迪斯细胞导致透镜槽内的微裂纹的外观
群体遗传分析表明,PAE-8-2在驯化过程中被选择
高等位基因频率差PAE-8-2在野生和家养的豆类(无花果。7.)建议在驯化期间受人类的强烈选择。我们无法检测到非功能性等位基因的事实PAE-8-2在这个研究中筛选的野豆类表明,这种等位基因产生于驯化期间或驯化后的一个新突变。而非功能等位基因PAE-8-2在驯化的Andeans常见的是,它只靠近美国北方的基因型固定。来自热带地区(非洲和中美洲)的Andeans保留了功能PAE-8-2等位基因适中的频率。该观察表明,通过现代育种计划,北美的非功能性等位基因有更高的选择压力。它可以合理地在高架机械化的农业系统中积极选择更快的吸收和发芽。此外,缓慢吸收可能会对豆质产生不利影响。因此,预计与不溶性果胶物质相关的等位基因将通过育种者从种质中吹扫。
PR9920-171的高水平种子休眠,是在波多黎各开发和测试的繁殖线[39.]源自IJR,这是一种生长在印度热带地区的地方品种。该地方种的功能性等位基因PAE-8-2可能具有较高的环境适应性。PAE-8-2的功能等位基因可能有助于不溶性果胶的形成,这些果胶在温暖潮湿的热带环境中有利于防止收获前萌发和/或促进洪水期间种子的存活。物理休眠也可能与其他热带豆科植物的质量更高有关,因为增加种子休眠被认为是在热带环境中栽培绿豆品种的潜在策略[68.].
结论
身体种子休眠是许多豆类物种的驯化中的重要特征,包括普通豆类。这种特质被认为是农业系统的不良因素。但是,这种教条最近开始受到质疑[9.那68.].这种前景变化的主要原因是气候变化的不可预测后果。最近降水模式的变化与种植时雨水过多相一致,从而影响萌发或植物成熟,从而导致产量和种子质量下降。因此,豆科植物未来的发展轨迹应该考虑保持一定程度的生理休眠,特别是在易受洪灾影响的地区。为了实现这一目标,有必要进行更深入的研究,以揭示这一特性背后的遗传因素。在本研究中,我们发现了一个与种子休眠相关的主要QTL,可能与大豆驯化有关。这一发现与其他研究小组提出的驯化性状是由少数几个主要基因控制的观点一致。我们还发现,促进大豆驯化的可能的诱发突变是一个5-bp的插入果胶acetylestrase-8这可能导致种子失去休眠。虽然我们的研究结果为果胶acetylestrase-8在种子休眠中,通过采用转基因技术,似乎需要进一步的确认。
方法
植物材料
为了鉴定PR9920-171和TARS-HT1之间差异种子休眠的遗传基础,我们使用了来自PR9920-171×TALS-HT1的双亲子生物群(图S1).分离人口及其父母是从蒂莫西门廊博士和菲利普·米卡拉斯博士获得的。来自f的种子1来自PR9920-171和TARS-HT1杂交的植物膨胀,并在每一代中批量前进,直到F5.一代作为开发200多张PIC的一部分(Photoolus改善合作;http://arsftfbean.uprm.edu/bean/?page_id=2)大量繁殖种群。这一种群后来被命名为PIC-76。用于PICs开发的体积化策略的优点是在每一代中推进更多的线路,可以提高捕获多样性的水平。然而,由于选择对种子数量的影响,它会使种群分布发生倾斜。在本文使用的批量分离分析方法中(见下文),这种偏态分布不会影响结果,因为分析使用的是总体的尾部。
种子萌发和吸收测定
建立两种基因型的种子萌发和吸收的速度,PR9920-171和TARS-HT1,植物于2019年7月至10月在密歇根州立大学的温室设施中生长。种子被手动收获,以防止潜在的种子外套损害。分别合并来自每种基因型的十个未公行和划痕的种子。在发芽和吸收测定中评估了三组10种子。为了评估萌发,将种子置于两个湿棉纸之间,在浸泡后24小时开始每8小时评价萌发百分比。为了评估种子吸收,在研究开始时测量干燥的种子重量(时间0),然后煮沸豆。每小时每小时测量水吸收率,然后每8小时到42小时。在每个时间点,排出水,通过用棉纸印刷,然后加权豆类来除去多余的水。计算每个时间点的水吸收率计算为
为了检测PR9920-171和TARS-HT1的子叶吸水率是否不同,我们用刀片小心地去除种皮。暴露种子的子叶与完整种子的子叶进行相同的吸胀试验。用该方法筛选了3组每个基因型10个种子。连续8 h,每小时测定吸水速率。
不同现场环境中表型一致性的评估
要了解如何影响种子的不同环境条件如何影响种子的水平,Pr9920-171和Tars-HT1基因型在2019年的三个不同现场条件下生长(表S1).从每个小区随机抽取4个样本,每个样本包含10颗种皮完好的种子。在水中浸泡6小时后,按上述公式测定相对自吸量。将IJR与PR9920-171和tas - ht1进行了MI现场试验比较。
物理休眠对种子呼吸的影响
使用具有Li-6800便携式气体交换系统(Li-Cor Bio-Sciences,Lincoln,Ne)的昆虫呼吸室附件测量具有完整和严重涂层的种子的呼吸速率。在浸泡之前,每次重复(每次重复五种种子),然后浸泡在蒸馏水中。对于完整的种子,将种子吸收6,24,48,72,96和192h,而吞噬的种子被吸收2,6,24,48和72小时。在每个测量时间点,用纸巾进行涂抹干燥,称重,并放置在昆虫呼吸室内。用以下参数测量呼吸速率:1000μmols- 1流速,70%空气相对湿度,400μmolco2和25°C的空气温度。三个技术测量以30秒进行一次,并平均算作一种生物学测量。在呼吸测量后立即,将种子放回水中以继续处理直至下次测量。
跟踪种子吸收水分的情况
为了鉴定进入种子涂层的初始位置,我们使用CT-Scan用碘溶液作为对比剂的成像。据我们所知,这是第一次CT扫描成像系统用于跟踪种子内的水。虽然以前开发了破坏性染色系统,但CT扫描系统提供了实时无损地跟踪种子内部的水。将来自两个基因型的种子浸泡在Lugol溶液中(1.007g / ml,Sigma-Aldrich,St.Louis,Mo)。在浸泡后30分钟间隔,使用NSI X3000工业CT X射线扫描仪排出并筛选种子。CT扫描仪配有225 kV微焦管,钨靶和8“×10”检测器(VAREX 2520DX),具有1536×1920像素分辨率。获得扫描作为连续扫描,由720个投影和3帧平均值组成。管电压设定为75kV,电子通量为100μA。在成像期间未使用光学过滤器。使用NSI(罗杰斯,明尼苏达州)的EFX-CT软件将投影组合成3D CT图像。 Final voxel resolution was 75.9 μm.
扫描电子显微镜(SEM)样品制备和图像分析
为了检测两种不同基因型的晶状体(见结果)之间的任何潜在的微观结构差异,我们采用了扫描电镜方法。样品制备和扫描电镜在密歇根州立大学高级显微中心进行。每个基因型(PR9920-171和tar - ht1)的20个种子用高真空碳片(SPI供应,西切斯特,PA)安装在铝桩上。在日本大阪Meiwafosis公司的NEOC-AT锇化学气相沉积涂布机中对种子进行~ 10 nm厚度的锇涂层。在JEOL 6610LV(钨发夹发射器)扫描电子显微镜(JEOL Ltd., Tokyo, Japan)中对种子进行成像。ImageJ [69.]来量化两种基因型种子晶状体表面的微裂面积。
膨胀的杀菌分析(BSA)以了解种子吸收的遗传建筑
为了鉴定与物理休眠相关的基因组位点,我们对PIC-76 RIL群体进行了牛血清白蛋白(BSA)检测。为了保证所有实验种子的种皮完好,我们在实验前首先在立体显微镜下对种皮的完整性进行了检测。保留1000颗种皮完好的种子进行吸胀实验。种子在去离子水中浸泡,室温(22°C)保存。5小时后,将PIC-76中的90粒种子种植在Suremix盆栽土壤(SURE, Galesburg, MI, USA),其余种子保持在水中。这90粒被吸收的种子被指定为快速吸收池。1 d后,87粒种子无吸胀现象。这些种子被认为是缓慢吸收的水池。我们把所有的种子从慢慢吸收的水池里割开,然后种在盆栽土壤里。发芽后,从每一个快速和缓慢吸收的豆子中收集叶盘,并将叶盘中的组织聚集在一起。 The genomic DNA was extracted from bulked tissue from both pools and parental lines using a Qiagen DNeasy Plant Mini Kit (Hilden, Germany).
在密歇根州立大学基因组学核心进行了图书馆制备和测序(https://rtsf.natsci.msu.edu/genomics/).简而言之,使用Illumina Truseq纳米DNA文库制备套件制备图书馆后制造商建议。完成的库是QC'd和使用Qubit DSDNA HS(Invitrogen Inc.Carlsbad,CA)和高级分析片段分析仪高敏感性DNA测定的组合量化。使用Illumina Hiseq 4000平台以2×150bp配对端格式进行测序。使用cutadapt修剪原始读取(v1.14 [70];)。首先从配对的reads中去除5 '端15bp的reads,然后从5 '和3 '端分别裁剪质量小于20的碱基。采用FastQC (v0.11.3)来确认reads的质量和适配器的去除。对齐P.寻常魅力参考基因组(v 2.1,[56.)使用BWA-MEM算法[71.].使用samtools对BAM文件进行排序[72.].为了删除副本,我们执行了Picard MarkDuplicates函数[73.].该功能可识别PCR和光学副本。我们遵循GATK4协议调用snp,并测量每个基因库中的等位基因频率。SNPs被使用HaplotypeCaller [74.].只有总reads数大于40且小于500的snp被保留用于下游分析。为了进一步去除不可靠变异,在分析中只保留了在亲本中检测到的两个等位基因(PR9920-171和tar - ht1)的snp。利用GATK包中的VariantsToTable检索每个库中每个SNP的位置和等位基因频率。包含等位基因频率数据的表格被导入到R环境中[75.使用QTLseqr包进行下游分析[76.].采用G '和QTLseq方法进行QTL鉴定。对于这两个函数,窗口大小都被设置为1 Mbp。QTL区间由第一个和最后一个超过0.05 FDR阈值的标记确定。
种子吸胀QTL精细定位
为了精细定位经批量分离分析确定的种子休眠QTL,我们对其周围的基因组区域进行了饱和分析。首先,在Pv03上的QTL边界上设计了两个插入/删除(InDel)标记和两个KASP (komcompetitive allele specific PCR)标记(表3)S2).另外,在QTL峰51.6 Mbp处设计KASP标记,检测可能的双重组基因型。用这三个标记对PIC-76共384个个体进行筛选,确定该区域存在重组个体。鉴定出84个重组个体,并将其种子播种在1加仑的花盆中。另外13个KASP标记贯穿QTL,使该区域饱和(表2S2).成熟时,人工收获84个重组个体的种子。每种植物的6个种子在Lugol溶液(Sigma-Aldrich, St. Louis, MO)中浸泡在22℃下4小时,并在x射线CT扫描仪中显示(见上文)。为了缩小与种子吸胀相关的基因的位置,我们使用QTL-Cartographer [77.].为了进一步缩小QTL的位置,重新测序30重组植物(10x)以识别重组断裂点的确切位置。使用在德克萨斯A&MAGRILIFE基因组学和生物信息学服务中开发的Agseq管道制备样品文库。然后使用德克萨斯A&M Agrlife Research的Illumina Novaseq进行测序文库。使用illumina的NCS 1.0.2和RFV 1.0.2软件实时使用默认参数设置实时完成序列群集识别,质量预滤波,基本呼叫和不确定性评估。
安第斯品种和野生豆类的单倍型调查
细映射和详细序列分析导致鉴定了控制种子吸收变异的可能候选基因(参见结果)。为了测试该基因参与驯化过程的假设,我们比较了野生和驯养的Andeans豆类之间的单倍型频率。驯养的Andean豆类是在密歇根州立大学的干酪育种和遗传计划中获得的。从USDA-AR种质资源信息网络(GRIN)获得野生Andeans。我们首次调查了210个基因型(表S3),属安第斯多样性小组[78.],这对于检测到的5-BP Indel进行基因分型PAE-8-2(见下文)。这些基因型来自三个不同的地理区域;非洲(N = 105), North America (N = 79), and Middle America (Caribbean, Central America and Colombia,N = 26). In addition, we genotyped the same variant in 25 wild beans collected from the Andes region of South America (TableS4).
对比基因型之间候选基因的表达比较
为了比较细映射区域内基因的表达谱,我们进行了表达分析。PR9920-171和Tars-HT1的植物在增长室(分别在生长室(大脚,生物切管人,温尼伯,MB,CANADA)中生长在25/20°C(日夜温度)。在开花阶段,分别为每朵花记录出的假期。在授粉后22天收集种子(早期荚填充阶段)。使用液氮将种子涂层与其他种子份分离,闪蒸冷冻。将五种种子的种子涂层组织合并在一起进行每个生物复制。在该实验中使用了四种生物重复。在制造商协议之后,使用Spectrum植物总RNA试剂盒(Sigma-Aldrich,St.Louis,Mo)提取总RNA。除了去除任何基因组DNA的痕迹,进行柱状DNA酶消化。使用Qubit荧光计(Invitrogen Inc.Carlsbad,Ca)和1%琼脂糖凝胶检查提取的RNA的数量和质量。 Reverse transcriptase and qPCR reactions were performed using Go-Taq 2-Step RT-qPCR System (Promega, Madison, WI) following the manufacturer’s protocol. For each biological replication, −RT reactions were included as negative controls to check for presence of any genomic DNA. Synthesized cDNA was diluted in 1:20 ratio. Eleven primer sets (TableS2)设计用于QTL间隔内的每种候选基因。在两个基因型之间的保守偏振序列设计了引物。actin-11(phvul.008g011000.)作为qPCR表达分析的内参基因。据报道,Actin-11在普通大豆的生物和非生物胁迫下均有一致的表达[79.].使用BIO-RAD CFX384实时系统(HERCULES,CA)进行QPCR。对每个基因型评估了四种生物学和两种技术复制。我们使用了2−ΔΔCt方法以鉴定两种基因型之间的差异表达基因。
数据和材料的可用性
在“当前研究期间生成的序列数据集”在BioProject ID下的NCBI序列读取存档(SRA)存储库中可用:PRJNA666967。
缩写
- AA:
-
氨基酸
- 阿坝:
-
脱落酸
- BSA:
-
膨胀的分离分析
- 图中:
-
电脑断层扫描
- 遗传算法:
-
吉布林斯
- IJR:
-
不确定牙买加红色
- MBP:
-
大型碱基对
- PAE-8-2:
-
果胶乙酰酯酶8.
- SEM:
-
扫描电子显微镜
参考
- 1。
植物和动物驯化的进化、后果和未来。大自然。2002;418:700-7。
- 2。
对植物驯化本质的进化洞见。咕咕叫医学杂志。2019;29:R705-14。
- 3.
Meyer Rs,Purugganan MD。作物种类的演变:驯化与多元化的遗传学。NAT Rev Genet。2013; 14:840-52。
- 4.
普通大豆驯化综合征的遗传控制。作物科学。1996;36:1037-45。
- 5。
Hradilovái,Trněnýo,válkovám,cechovám,janskáa,prokešovál等。两种关键驯化特征的组合的比较转录组,代谢组和解剖学分析:豌豆的荚裂解和种子休眠Pisumsp)。植物科学与技术,2017;
- 6。
RAU D,Murgia Ml,Rodriguez M,Bitocchi E,Bellucci E,Fois D,等。伴随豆类群中泛滥碎片的基因组解析:在豆科物种驯化下的收敛表型进化的基础上的突变。工厂J. 2019; 97:693-714。
- 7。
Simpson KJ, Wade RN, Rees M, Osborne CP, Hartley SE。驯化后还带着武器吗?驯化和农艺选择对谷物硅防御的影响。功能生态。2017;31:2108-17。
- 8。
植物驯化通过对防御性状的直接选择减少了构成性和诱导性化学防御。Sci众议员2018;8:12678。
- 9。
关键词:豆科植物,种子休眠,种子萌发,种皮,休眠机制植物科学与技术,2014;
- 10。
Finch-Savage We,Footitt S.种子休眠循环和可变场环境中时间发芽的休眠机制的调节。J Exp Bot。2017; 68:843-56。
- 11.
Penfield S.种子休眠和发芽。Curr Biol。2017; 27:R874-8。
- 12.
脉冲栽培前驯化。经济学机器人。1987;41:60-5。
- 13。
Abbo S, Zezak I, Schwartz E, Lev-Yadun S, Gopher A.以色列野豌豆的实验收获:对近东农业起源的影响。考古科学,2008,35:922 - 9。
- 14。
Gubler F, Millar AA, Jacobsen JV。休眠释放、ABA和收获前发芽。植物生物学杂志,2005;8:183-7。
- 15。
Ahmad S,Khulbe Rk,Roy D. Mungbean的评估(Vigna Radiata.用于预先收获的散发耐受性的种质。Legum Res。2014; 37:259-63。
- 16。
Finch-Savage我们,Leubner-Metzger G.种子休眠和萌发的控制。新植物。2006; 171:501-23。
- 17。
马丁ac。种子的比较内部形态。我是midl nat。1946; 36:513-660。
- 18。
福布斯我,Wells HD。蓝羽羽的硬和柔软的播种,卢比斯angustifoliusL.:遗传和表型分类。作物科学。1968;8:195-7。
- 19。
Ladizinsky G.属属中硬皮涂层的遗传学镜头.Euphytica。1985;34:539-43。
- 20.
Isemura T, Kaga A, Tomooka N, Shimizu T, Vaughan DA。稻豆驯化的遗传学,豇豆属umbellata.安机器人。2010;106:927-44。
- 21.
Isemura T,Kaga A,Tabata S,Somta P,Srinives P,Shimizu T等人。Mungbean遗传联系地图和驯化相关性状的遗传分析(Vigna Radiata.).《公共科学图书馆•综合》。2012;7:e41304。
- 22.
关键词:码长豆角,驯化,遗传,遗传学豇豆属unguiculata(l)Walp。Ssp。unguiculata cv.-gr。Sesquipedalis。安机器人。2012;109:1185 - 200。
- 23.
Werker E, Marbach I, Mayer AM。种皮的解剖结构、透水性与属中酚类物质存在的关系Pisum.Ann Bot。1979; 43:765-71。
- 24.
Kannenberg LW, Allard RW。青豆种皮中色素和木质素形成之间的联系作物科学。1964;4:621-2。
- 25.
邵氏,Meyer CJ,Ma F,Peterson Ca,Bernards Ma。大豆种子的最外层节:化学成分和在吸收过程中的功能。J Exp Bot。2007; 58:1071-82。
- 26.
Bradford K,Nonogaki H.种子发展,休眠和发芽。牛津:Blackwell Publishing Ltd;2007年。
- 27.
植物解剖学。第二版。纽约:威利;1965.
- 28.
Ma F,Cholewa E,Mohamed T,Peterson Ca,Gijzen M.在大豆种子涂层的Palisade Cullics中的裂缝与它们对水的渗透性相关。Ann Bot。2004; 94:213-28。
- 29。
Bitocchi E, Nanni L, Bellucci E, Rossi M, Giardini A, Zeuli PS等。豆的中美洲起源(phoudolusulus vulgaris.L.)是由序列数据揭示的。美国国家科学院学报2012;109:E788-96。
- 30.
Gepts P,Osborn Tc,Rashka K,Bliss Fa。野生豆类野生形式和体力的样蛋白 - 蛋白质变异性(phoudolusulus vulgaris.):多个驯化中心的证据。econ bot。1986; 40:451-68。
- 31.
Emiliano T, Andrea B, Martina L, Alice I, Stefania V, Laura N等。古老的基因组显示,早期安第斯农民在保留多样性的同时选择了普通豆类。bioRxiv。2019:791806。
- 32.
Cichy Ka,Fernandez A,Kilian A,Kelly JD,Galeano Ch,Shaw S等人。QTL对黑豆罐头质量和颜色保留的分析(phoudolusulus vulgaris.l .)。摩尔品种。2014;33:139-54。
- 33。
Hosfield GL。干豆生产和食品质量因素的遗传控制。食品技术。1991年; 45:98。
- 34。
Soltani A,Mafimoghaddam S,Walter K,Restpo-Montoya D,Mamidi S,Schroder S等人。干豆中美多样性面板洪水耐受性遗传建筑。前植物SCI。2017; 8:1183。
- 35。
Soltani A,Mafimoghaddam S,Oladzad-Abasabadi A,Walter K,Kearns PJ,Vasquez-Guzman J等。干豆的Andean多样性面板洪水耐受遗传分析(phoudolusulus vulgaris.l .)。前植物SCI。2018; 9:767。
- 36。
植物种子的休眠与萌发。植物生物学。2002;5:33-6。
- 37。
斯坦利DW。硬豆——对种植者、加工商和消费者来说都是一个问题。《社会科学》1992;2:37 - 8。
- 38。
Agbo G,Hosfield G,Uebersax M,Klomparens K.种子微观结构及其与中源性线的水吸收的关系及干酪栽培品种(phoudolusulus vulgaris.l .)。食品结构。1987; 6:91-102。
- 39。
B,海狸JS。安第斯起源的普通大豆耐热性的遗传。波多黎各农业大学,2003;87:113-21。
- 40.
Porch Tg,Smith Jr,海狸Js,Griffiths Pd,加拿大Ch。TARS-HT1和TARS-HT2耐热性干豆种质。Hortscience。2010; 45:1278-80。
- 41.
Fick S,Hijmans R. Worldclim 2:全球土地地区的新型1公里的空间分辨率气候表面。in j climatol。2017; 37:4302-15。
- 42.
Wickham H. GGPLOT2:用于数据分析的优雅图形。1 ed。纽约:Springer-Verlag;2016年。
- 43.
关于微生物作用和土壤颗粒磨损导致的物理休眠中断的主张的进化考虑。种子科学,2000;10:409-13。
- 44.
打破种子的物理休眠-聚焦镜头。新植醇。2003;158:229-32。
- 45.
Karaki T,Watanabe Y,Kondo T,Koike T.Strope的黑色蝗虫的种子是水隙。植物物种Biol。2012; 27:226-32。
- 46.
HU XW,王YR,WU YP,Baskin CC。镜片在两种Fabaceae种子中加吸水中的作用(Papilionoideae.)用硫酸和热水处理。种子科学与技术;2009;
- 47.
在澳大利亚本土的豆科植物中,晶体在控制热诱导的雄蕊休眠分解中的作用。安机器人。1998;82:35-40。
- 48.
Jaganathan GK,Li J,Biddick M,Han K,Song D,Yang Y等。支撑种子涂层不渗透性和休眠休息的机制黄芪adsurgens..SCI REP。2019; 9:9695。
- 49.
关键词:种子休眠,种子皮结构,种子休眠银合欢leucocephala和金合欢Nilotica..森林SCI技术。2020; 16:18-25。
- 50.
锤子K.驯化综合症(德语。)。kult。1984; 32:11-34。
- 51.
Fatokun Ca,Menancio-Hautea di,Danesh D,Young Nd。基于RFLP映射的豇豆和绿豆在豇豆和绿豆中的证据。遗传学。1992年; 132:841-6。
- 52.
张建平,李建平,李建平。玉米和大刍草F2群体形态差异的遗传分析[J] .华北农学报,2018,33(2):102 - 105。遗传学。1993;134:559 - 70。
- 53.
Vittori V,Bitocchi E,Rodriguez M,Alseekh S,Bellucci E,Nanni L等。普遍豆类的豆荚与PVMYB26的微调和非功能性脱落层的微调相关。bioRxiv。2020。
- 54.
Parker TA, Palkovic A, Jernstedt J, Gepts P等。豆荚开裂是普通大豆的驯化和抗旱特性。新植醇。2020;225:558 - 70。
- 55.
Sandhu Ks,You FM,Conner RL,Balasubramanian PM,Hou A.遗传分析和QTL在黑色常见豆中种子硬度特征的映射(phoudolusulus vulgaris.)重组自交系(RIL)人口。mol品种。2018; 38:34。
- 56.
Schmutz J,McClean Pe,Mamidi S,Wu Ga,Cannon Sb,Grimwood J等人。一种参考基因组,用于常见的豆类和基因组的双重驯化分析。NAT Genet。2014; 46:707-13。
- 57。
repinski sl,gepts p.常见的豆生长习性基因pvtfl1y是拟南芥tfl1的功能性同源物。Al Appl Genet。2012; 124:1539-47。
- 58。
mclean PE, Bett K, Stonehouse R, Lee R, Pflieger S, MafiMoghaddam S等。豆子种子呈白色(phoudolusulus vulgaris.)的结果是P(色素)基因的趋同进化。新植醇。2018;219:1112-23。
- 59。
PONCET V,Martel E,Allouis S,Devos K,Lamy F,Sarr A等。QTLS影响两种驯化×野珍珠小米驯化性状的QTLS(Pennisetum blaucum.L.,Poaceae)十字架。Al Appl Genet。2002; 104:965-75。
- 60。
Cai H,Morishima H. QTL集群反映了野生和栽培的米饭中的性格联系。Al Appl Genet。2002; 104:1217-28。
- 61。
植酸-植酸-果胶假说与豆科植物在钙溶液中煮熟的品质。中国农业科学,2008;
- 62。
Kigel J.烹饪和营养质量phoudolusulus vulgaris.种子受环境因素的影响。根据。1999年; 3:205-9。
- 63.
罗利特,Crépeau M-J,布赫霍尔特H-C,蒂博J-F。甜菜果胶在甲基化和乙酰化程度不同时的聚电解质行为和钙结合特性。生物化学工程2003;16:191-201。
- 64.
De Souza A,Hull Pa,Gille S,Pauly M.鉴定和功能表征独特的植物果胶酯酶PAE8和PAE9及其缺失突变体。Planta。2014; 240:1123-38。
- 65.
苟j-y,miller lm,hou g,yu x-h,chen x-y,liu c-j。乙酰酯酶介导的果胶的脱乙酰化危害细胞伸长,花粉萌发和植物繁殖。植物细胞。2012; 24:50-65。
- 66.
关键词:绿豆,果胶乙酰酯酶,马铃薯,胞壁聚合物abstract:足底。2012;236:185 - 96。
- 67.
Pogorelko G,Lionetti V,Fursova O,Sundaram RM,Qi M,Whitham Sa等。拟南芥和Brachypodium distachyon表达转基因植物Aspergillus nidulans.乙酰酯酶具有降低的多糖乙酰化和对病原体的抗性增加。植物理性。2013; 162:9-23。
- 68.
Humphry Me,Lambrides CJ,Chapman Sc,Aitken Eab,Imrie Bc,Lawn RJ等。豆丛中硬种植与种子重的关系(Vigna Radiata.)进行QTL分析。植物品种。2005;124:292-8。
- 69.
施耐德CA,Rasband Ws,Eliceiri Kw。nih图像到imagej:25年的图像分析。NAT方法。2012; 9:671-5。
- 70。
Martin M. Cutadapt从高通量测序中删除适配器序列。EMBnet j . 2011; 17:10-2。
- 71.
基于burrows-wheeler变换的快速准确短读对齐。生物信息学。2009;25:1754-60。
- 72.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N等人。序列对齐/地图格式和samtools。生物信息学。2009; 25:2078-9。
- 73.
广泛的研究所。皮卡德工具包。2019.
- 74.
Poplin R, Ruano-Rubio V, DePristo MA, Fennell TJ, Carneiro MO, Van der Auwera GA等。将精确的基因变异发现扩展到数以万计的样本。bioRxiv。2018:201178。
- 75.
R:一种用于统计计算的语言和环境。R基础统计计算;2011.1 2.11.1。
- 76.
Mansfeld BN, Grumet R. QTLseqr:用于下一代测序的批量分离分析的R包。植物基因组。2018;11:180006。
- 77.
王S,Basten CJ,Zeng Z.Windows QTL Cartographer 2.5;2012年。
- 78.
Cichy Ka,Porch TG,Beaver JS,Cregan P,Fourie D,Glahn Rp等。一种phoudolusulus vulgaris.Andean Bean改进的多样性小组。农作物科学。2015; 55:2149-60。
- 79.
Borges A,Tsai Sm,Caldas DGG。生物和非生物应激期间常见豆类RT-QPCR标准化参考基因的验证。植物细胞代表2012; 31:827-38。
确认
我们要感谢Evan Wright管理场情节。我们还要感谢Paul Gept,用于提供种子休眠QTL的侧翼标记序列,在Koinange等人揭示。(1996)研究。
资金
这项研究主要由密歇根州立大学植物恢复力研究所资助。资金用于支持数据收集过程中的不同费用,包括基因分型、表达分析、种植设施等。
作者信息
从属关系
贡献
AS和DBL设计了实验。AS, KAW进行了实验。AS、KAW和ATW有助于DNA提取和KASP标记设计/运行。JPS有助于测量种子呼吸。MQ和DC有助于ct扫描成像。TGP、PM、PEM和JMO发育并提供基因型。AS对数据进行了分析。AS, DBL, PEM, JMO编写稿件。所有作者阅读、编辑和批准手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明他们没有利益冲突。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
PR9920-171和tar - ht1的家系。图S2。镜片堵塞对种子吸收的影响。图S3。快速和慢吸池内SNP标记的等位基因频率。图S4。利用18个KASP和Indel标记对QTL区进行饱和分析图S5。保护非同义点突变phvvul.003g277400.其他物种中同源基因中的编码区域。图S6。非同义点突变的守恒phvvul.003g278200.其他物种中同源基因中的编码区域。图S7。的非同义点突变phvvul.003g278400.编码区。图S8。在6个外显子插入5-BP插入PHVVUL.003G277600.导致tas - ht1的aa序列的移码。
附加文件2:表S1。
现场网站信息
附加文件3:表S2
本研究中使用的引物列表
附加文件4:表S3。
本研究中使用的驯化安第斯基因型列表
附加文件5:表S4。
本研究中使用的野生Andeans列表
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Soltani,A.,Walter,K.A.,Wiersma,A.T.等等。大豆种子休眠的遗传生理是大豆驯化的关键性状。BMC植物BIOL.21日,58(2021)。https://doi.org/10.1186/s12870-021-02837-6.
收到了:
公认:
发表:
关键词
- 菜豆
- 驯化
- 休眠
- 发芽
- 自吸
- 果胶