- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
转录组和基因组 - 范围的协会研究显示玉米氮缺乏症的长度非编码RNAgydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba21gydF4y2Ba, 文章编号:gydF4y2Ba93gydF4y2Ba(gydF4y2Ba2021gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
长链非编码rna (lncRNAs)在必要的生物过程中发挥着重要作用。然而,我们对lncrna作为竞争性内源rna (competing endogenous rna, ceRNAs)及其对氮胁迫的反应的理解仍然有限。gydF4y2Ba
结果gydF4y2Ba
本研究利用lncRNA-Seq和small RNA-Seq对玉米自交系P178苗期高氮(HN)和低氮(LN)条件下叶片和根系的lncrna和mirna进行了调查。共鉴定出894个差异表达的lncrna和38个不同的mirna。共表达分析发现,2个lncrna和4个lncrna靶点分别可以与ZmmiR159和ZmmiR164竞争结合。为了剖析lncrna如何适应有限的氮可用性,用一个包含高密度单核苷酸多态性(SNP)阵列(56110个SNP)和从水培中获得的可变LN耐受性相关表型的关联定位板进行全基因组关联研究(GWAS)。通过结合GWAS和RNA-Seq,筛选出170个显著标记范围内差异表达的lncrna。此外,还鉴定出40个对ln响应一致的基因,包括那些参与谷氨酰胺生物合成和根系氮素获取的基因。中瞬时表达分析gydF4y2Ba烟草benthamianagydF4y2Ba证明了gydF4y2BaLNC_002923gydF4y2Ba可以居住gydF4y2BaZmmiR159gydF4y2Ba引导的乳沟gydF4y2BaZM00001D015521gydF4y2Ba.gydF4y2Ba
结论gydF4y2Ba
这些lncrna含有与性状相关的显著SNPs,可能与根系发育和养分利用有关。综上所述,我们的研究结果可以为lncrna在LN胁迫响应中的潜在调控作用提供新的见解,并为进一步筛选候选基因以及提高玉米对LN胁迫的抗性提供有价值的信息。gydF4y2Ba
背景gydF4y2Ba
目前,玉米(gydF4y2Ba玉米gydF4y2BaL.)是全球人类消费和动物饲料的主要栽培作物。氮素是一种重要的植物常量养分,提高氮素吸收是提高作物产量的关键选择[gydF4y2Ba1gydF4y2Ba].硝酸(不gydF4y2Ba3.gydF4y2Ba)和铵(nhgydF4y2Ba4gydF4y2Ba)分别是植物根系在好氧土壤和淹水土壤条件下吸收和同化的主要无机氮源[gydF4y2Ba2gydF4y2Ba].几乎所有的土壤都缺氮,这直接导致了过量施用氮肥。因此,降低农业成本及其对环境的影响就显得尤为重要。最近,研究人员发现生长调节因子4 (GRF4)和生长抑制DELLA蛋白在调节植物生长和氮代谢中起平衡作用[gydF4y2Ba3.gydF4y2Ba这将有助于我们了解植物的氮代谢,为全球安全食品供应的可持续发展做出贡献。gydF4y2Ba
植物硝酸盐转运蛋白包括低硝酸盐 - 亲和转运蛋白,例如NRT1家族的大多数成员[gydF4y2Ba4gydF4y2Ba],它是硝酸盐吸收和在细胞、组织和器官之间运输的关键转运体。同时,高硝酸盐亲和转运蛋白NRT2家族等gydF4y2Ba拟南芥蒂利亚纳gydF4y2BaNRT2.5 (AtNRT2.5) [gydF4y2Ba5gydF4y2Ba]负责在相对较低的浓度范围内吸收硝酸盐。其他硝酸盐转运体,氯离子通道(CLC)家族[gydF4y2Ba6gydF4y2Ba]可以介导硝酸盐的积累和运输。在这些蛋白质家族中,比如gydF4y2Ba拟南芥蒂利亚纳gydF4y2BaNRT1.1 (AtNRT1.1) [gydF4y2Ba7gydF4y2Ba],扮演着NO3的双重角色gydF4y2Ba−gydF4y2Ba受体和转运蛋白。氨转运体主要位于质膜上。它们负责疏水性NHgydF4y2Ba3.gydF4y2Ba氨的运输和分配等gydF4y2Ba拟南芥蒂利亚纳gydF4y2BaAMT1.1 (AtAMT1.1) [gydF4y2Ba8gydF4y2Ba].gydF4y2Ba
长度非编码RNA(LNCRNA)通常定义为不能编码蛋白质的转录物并且具有从200nt至100kb的序列长度的序列长度。LNCRNA通常调节相邻的靶基因gydF4y2BaCIS.gydF4y2Ba,并调节远处目标基因gydF4y2Ba反式gydF4y2Ba[gydF4y2Ba9gydF4y2Ba].因此,当猜测其功能时,LNCRNA的位置是有用的。包括MRNA,转录的假药和LNCRNA的Cerna假设描述了它们可以通过共享相同的MicroRNA响应元素(MRE)来彼此通信,并且RNA通过竞争MiRNA来影响彼此的表达[gydF4y2Ba10gydF4y2Ba].例如,ceRNA linc-RoR被证明与核心转录因子(TFs)共享相同的miRNA,并阻止这些核心转录因子在自我更新的人类胚胎干细胞中受到miRNA介导的抑制[gydF4y2Ba11gydF4y2Ba].此外,在玉米中,高度丰富的pi缺乏诱导的长非编码RNA1 (gydF4y2BaPILINCR1gydF4y2Ba)可以有效地损害gydF4y2BamiR399gydF4y2Ba磷酸二酯的引导裂解(gydF4y2BaPHO2gydF4y2Ba) 规范gydF4y2BaPHO2gydF4y2Ba将公差降低到低Pi [gydF4y2Ba12gydF4y2Ba].在水稻中,从RNA-seq数据中鉴定出潜在的由376个和511个茎和根的lincRNA组成的ceRNA网络,表明了低Pi胁迫下lincRNA功能的系统调控[gydF4y2Ba13gydF4y2Ba].gydF4y2Ba
玉米自交系B73参照基因组测序完成后,单核苷酸多态性(SNPs)和插入-缺失多态性的发现为定位与玉米农业特性相关的qtl和探索候选基因奠定了基础[gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba].全基因组关联研究(Genome-wide association study, GWAS)是基于连锁不平衡(linkage disequilibrium, LD)来发现群体中客观性状与遗传标记之间的关系[gydF4y2Ba16gydF4y2Ba].使用GWA研究了世界上不同地区的自然变化的玉米线,以找到基因gydF4y2BaZmNAC111gydF4y2Ba,编码位于10号染色体上的NAC转录因子,该转录因子在玉米幼苗耐旱性中起重要作用[gydF4y2Ba17gydF4y2Ba],共对384个玉米自交系进行了681257个snp基因分型,并利用22个幼苗根系构型性状来鉴定与苗期根系发育相关的候选基因[gydF4y2Ba18gydF4y2Ba].在双单倍体玉米中,共鉴定出54个snp与玉米褪绿斑驳病毒和致命坏死的抗性显著相关[gydF4y2Ba19gydF4y2Ba].此外,通过组合代谢物谱和GWA,开采了五种与形态特征和同时参与代谢途径相关的低PI响应的共有基因[gydF4y2Ba20.gydF4y2Ba].gydF4y2Ba
以往的研究主要集中在揭示氮调控的编码基因;相比之下,由缺氮引起的非编码成分如lncrna却很少受到关注。本研究采用高通量测序技术分析了HN和LN条件下玉米苗期lncrna、mrna和mirna的表达谱。潜在lncrna和miRNA的靶基因gydF4y2Ba- - - - - -gydF4y2Ba预测LNCRNA对。我们还基于“Cerna假设”,确定了在共表达网络中的LNCRNA的功能。中瞬时表达分析gydF4y2Ba烟草benthamianagydF4y2Ba证明了gydF4y2BaLNC_002923gydF4y2Ba能抑制裂解吗gydF4y2BaZM00001D015521gydF4y2Ba通过gydF4y2BaZmmiR159gydF4y2Ba.鉴定了含有有明根特征相关的SNP的一百七十个LNCRNA。而且,总共40份始终如一gydF4y2Ba- - - - - -gydF4y2Ba结合GWAS和RNA-Seq筛选应答候选基因。总之,我们的研究结果为理解LN提供了多种见解gydF4y2Ba- - - - - -gydF4y2Balncrna在玉米幼苗中的响应机制,为提高玉米氮素利用效率提供了新的思路。gydF4y2Ba
结果gydF4y2Ba
LNCRNA和小RNA文库的高通量测序gydF4y2Ba
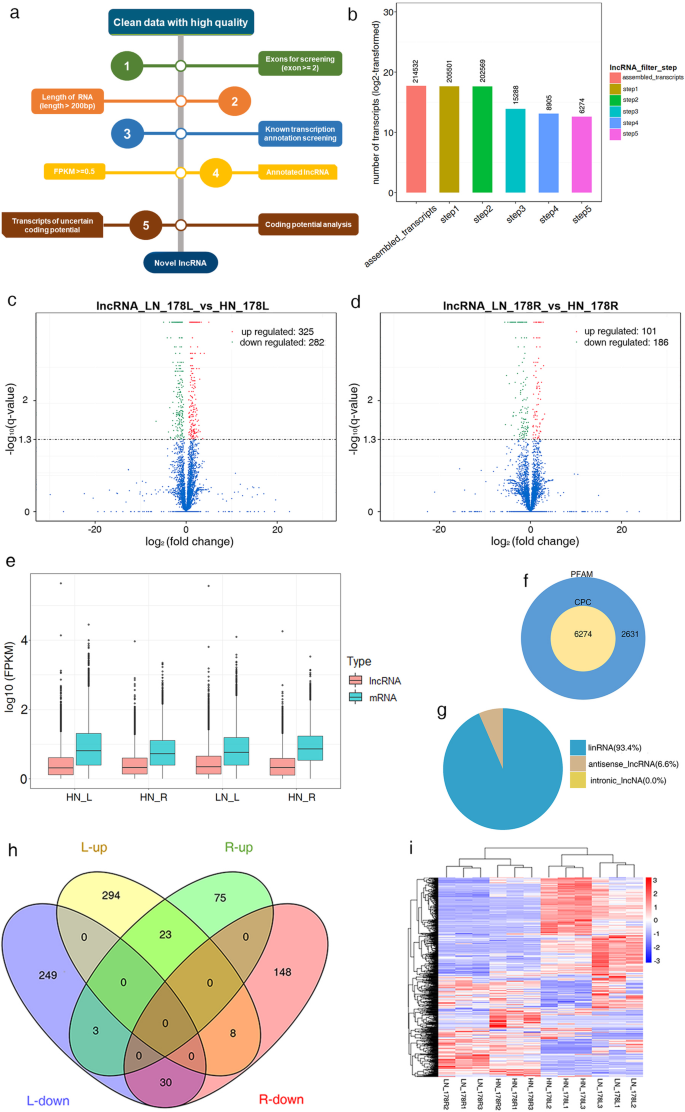
从lncRNAs和小rna文库中分别获得了1,241,588,130和168,839,937条raw reads。后移低位gydF4y2Ba- - - - - -gydF4y2Ba我们从lncRNA和小RNA文库中获得了大约1,175,852,744和165,272,934个clean reads。然后,我们将每个样本的清洁reads映射到玉米基因组(B73 RefGen_V4),并用于进一步分析(表gydF4y2Ba1gydF4y2Ba).在mrna方面,从所有12个RNA-seq数据集中共重建了214,532个转录本,在叶片和根中鉴定出了8836个差异表达基因(图1)。gydF4y2BaS1gydF4y2Baa,b)。对于LNCRNA,我们基于我们对成分剪接结果和结构特征和非编码蛋白的结果分析,建立一系列严格的筛选条件(图。gydF4y2Ba1gydF4y2Baa).共6274个转录本进行差异表达分析(图5)。gydF4y2Ba1gydF4y2Bab).我们在叶片和根中获得了894个可靠表达的lncrna(图)。gydF4y2Ba1gydF4y2BaC,D)。对于miRNA,从清洁读取产生总小RNA的长度分布(范围18-30nt)(图。gydF4y2BaS2gydF4y2Baa). miRNA的TPM (transcript per million)如图所示。gydF4y2BaS2gydF4y2Ba湾然后,使用筛选的SRNA分析参考序列上的分布并鉴定已知和新的miRNA。在叶子和根中发现了总共184名已知的miRNA和106个新的miRNA(表gydF4y2BaS1gydF4y2Ba).在HN和LN条件下差异mrna和mirna表达模式的分析见图。gydF4y2BaS3gydF4y2Ba.gydF4y2Ba
用于lncRNA鉴定的管道(gydF4y2Ba一个gydF4y2Ba).设置5个步骤以过滤组装的转录物(gydF4y2BabgydF4y2Ba).两种氮条件下叶片中差异表达lncrna的火山图(gydF4y2BacgydF4y2Ba)和(gydF4y2BadgydF4y2Ba)根。根据RNA-Seq结果分别计算lncRNAs和mrna的表达值(gydF4y2BaegydF4y2Ba).LncRNA分类(gydF4y2BafgydF4y2Ba),利用蛋白家族数据库(Pfam)和编码潜能计算器(CPC) (gydF4y2BaggydF4y2Ba).叶片和根部之间的差异表达的LNCRNA(gydF4y2BahgydF4y2Ba).lncRNA在HN和LN条件下的表达谱(gydF4y2Ba我gydF4y2Ba).HN,留下氮。ln,低氮。l,叶。r,根gydF4y2Ba
lncrna全基因组鉴定gydF4y2Ba
在玉米幼苗中,lncrna的长度从201个碱基对(bp)到约29,176 bp,平均长度为829 bp,分布在每条染色体上。lncrna的FPKM低于mrna(图1)。gydF4y2Ba1gydF4y2Bae).这些lncRNAs中,93.4%为lincRNAs, 6.6%为反义lncRNAs(图5)。gydF4y2Ba1gydF4y2BaF)。它们的编码电位也通过PFAM和CPC预测(图。gydF4y2Ba1gydF4y2Bag).为了识别参与氮胁迫响应的lncrna,我们选择了HN组和LN组之间q值< 0.05的差异表达lncrna。总体而言,从叶和根中广泛筛选了607个和287个差异lncrna(图1)。gydF4y2Ba1gydF4y2Ba(c, d),其中23个在根和叶中均持续上调,30个在根和叶中均持续下调(图5)。gydF4y2Ba1gydF4y2Ba这表明这些lncrna在不同组织中对LN应激的反应具有相似的功能。通过对不同lncrna的聚类分析,确定HN和LN条件下的聚类模型(图1)。gydF4y2Ba1gydF4y2Ba我);这两种组织的lncrna表达分别上调和下调。此外,还发现了477个可能包含具有一定编码潜能的lncrna子集的不确定编码潜能(TUCPs)差异表达转录本(图1)。gydF4y2BaS1gydF4y2Bac, d)。所鉴定的lncrna特征如图所示。gydF4y2BaS4gydF4y2Ba.在这些结果中,我们获得了大多数的lincrna和反义lncrna,而tucp的比例很低,这表明RNA-Seq策略可以用于这些rna。gydF4y2Ba
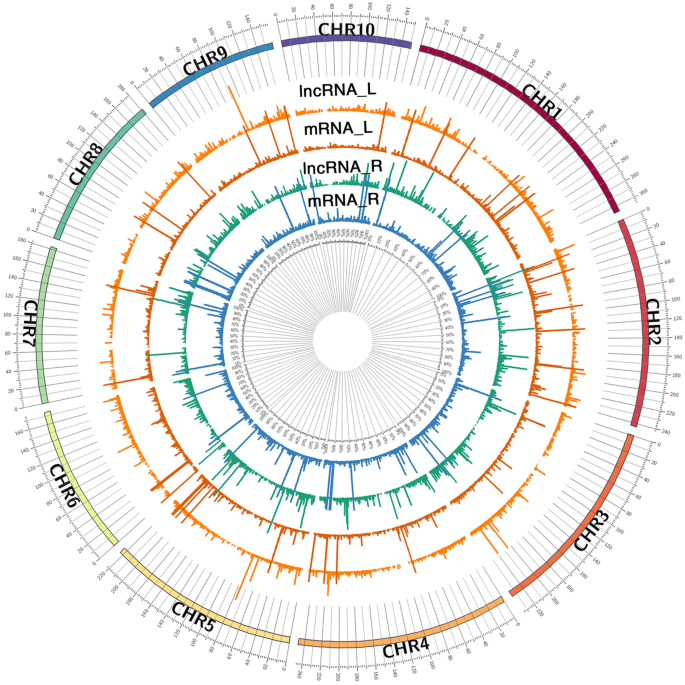
12个文库在10条染色体上的表达水平分布如图所示。gydF4y2Ba2gydF4y2Ba).为了了解lncRNAs和编码转录本之间可能的相互作用,我们鉴定了lncRNAs上下行100kb内的相邻基因(Table .)gydF4y2BaS2gydF4y2Ba).我们还使用Pearson的相关系数来分析LNCRNA和基因之间表达水平的相关性;具有大于0.95的相关值的人进行分析(表gydF4y2BaS2gydF4y2Ba).共同位置和共表达的目标分析提供了一种方法来预测LNCRNA的主要功能。gydF4y2Ba
使用qRT-PCR验证lncRNA、lncRNA靶点和miRNA表达gydF4y2Ba
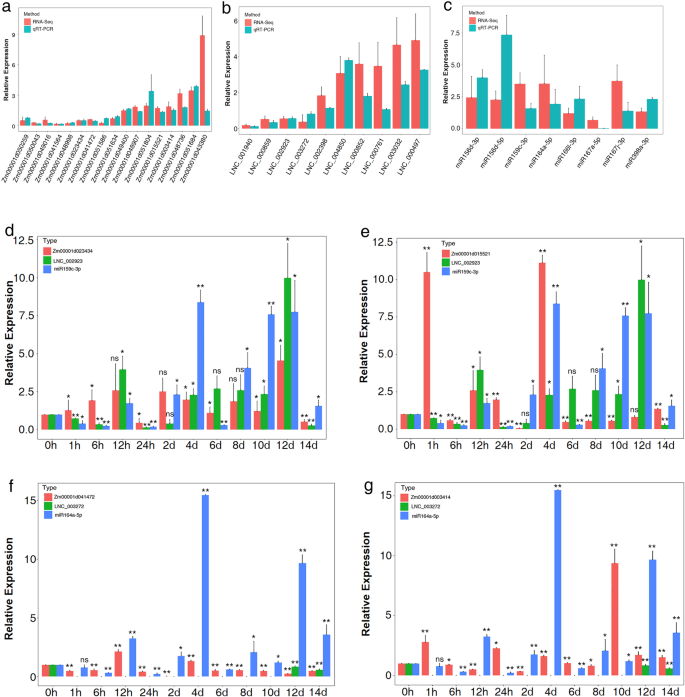
为验证深度测序结果的可靠性,随机选取17个mrna、10个lncrna和8个mirna进行qRT-PCR分析。如图所示。gydF4y2Ba3.gydF4y2Baa-c、qRT-PCR和RNA-seq数据显示出相同的趋势,说明RNA-seq结果是可靠的。gydF4y2Ba
共表达网络分析gydF4y2Ba
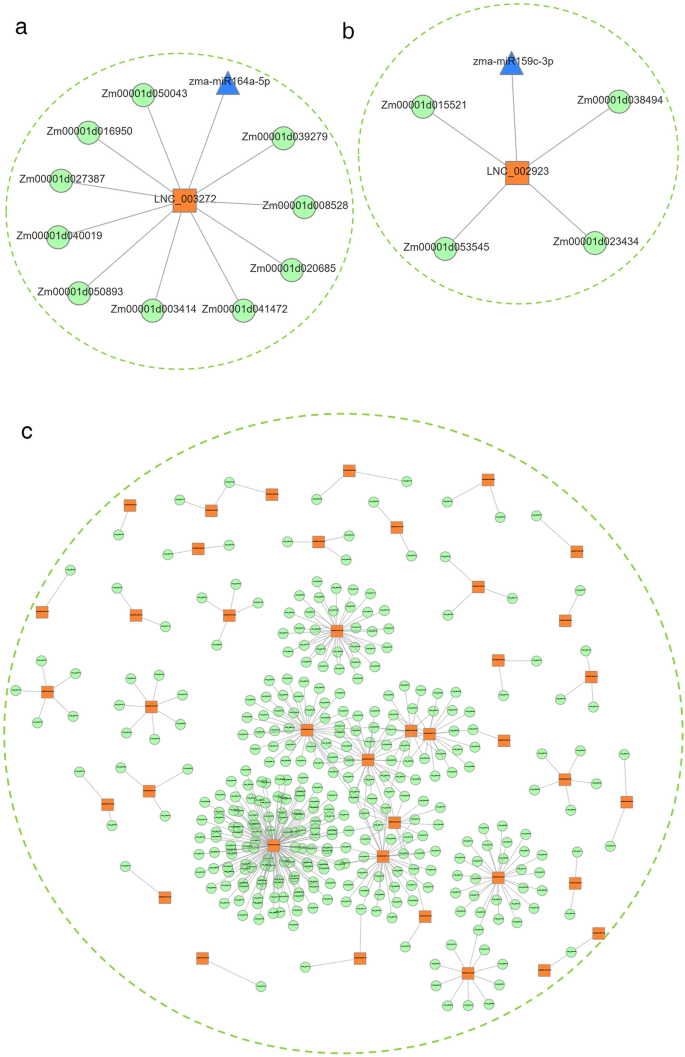
共有来自12个小RNA文库的38种差异表达的miRNA(图。gydF4y2BaS1gydF4y2Bae, f)用于分析它们与lncrna和mrna的相互作用。然后,我们构建了lncRNA的共表达网络gydF4y2Ba- - - - - -gydF4y2Ba基因对通常具有相同的miRNA结合位点[gydF4y2Ba10gydF4y2Ba].如图所示。gydF4y2Ba4gydF4y2Ba这两个共表达网络包括a, b, 2个lncRNAs, 2个miRNAs, 14个mrna,这些lncRNAs作为ceRNAs,通过与特定的miRNAs竞争,与许多mrna通信。这些结果有助于我们了解lncrna在LN胁迫下玉米幼苗中的潜在功能,并揭示lncrna在整个转录组中调控基因表达的机制。gydF4y2Ba
然后我们分析lncRNA-miRNA-mRNA表达水平在不同处理阶段对LN应激的动态响应。对于叶片中的共表达网络,gydF4y2BaLNC_002923gydF4y2Ba,gydF4y2BamiR159cgydF4y2Ba,选择2个mrna进行qRT-PCR。如图所示。gydF4y2Ba3.gydF4y2BaD, e,的表达式轮廓gydF4y2BaZm00001d023434gydF4y2Ba类似于gydF4y2BaLNC_002923gydF4y2Ba,而与miR159c相反。此外,gydF4y2BaZM00001D015521gydF4y2Ba在lncrna相关共表达网络中的表达模式与gydF4y2BaLNC_002923gydF4y2Ba,但类似于gydF4y2BamiR159cgydF4y2Ba在14天。一般来说,水平gydF4y2BaZm00001d023434gydF4y2Ba除24 h和14 d外,在缺氮胁迫下,除24 h和14 d外,其余均上调。gydF4y2BaZM00001D015521gydF4y2Ba除1 h、12 h、24 h和4 d外,其他阶段几乎完全抑制。的水平gydF4y2BamiR159cgydF4y2Ba大部分处理期叶片表达上调,仅在1 h、6 h、24 h和6 d时表达下调。的水平gydF4y2BaLNC_002923gydF4y2Ba在短期内几乎总是被下调吗gydF4y2Ba- - - - - -gydF4y2Ba但从长远来看却被上调了gydF4y2Ba- - - - - -gydF4y2Ba术语。对于根中的共表达网络,表示为gydF4y2BaLNC_003272gydF4y2Ba,gydF4y2BamiR164agydF4y2Ba,两个mrna如图所示。gydF4y2Ba3.gydF4y2Ba的表达式gydF4y2BaLNC_003272gydF4y2Ba仅在12、14 d时表现出下调。的水平gydF4y2BamiR164agydF4y2Ba在LN应激后14天,mrna呈现相反的表达模式。总之,氮响应lncRNAs、miRNAs和mrna在后期的表达谱匹配优于其他短链gydF4y2Ba- - - - - -gydF4y2Ba这表明ceRNA机制对于进一步研究lncRNA在玉米LN条件下的功能至关重要。gydF4y2Ba
ln响应lncrna的靶标分析gydF4y2Ba
检测gydF4y2BaCIS.gydF4y2Ba对叶片和根中607个和287个差异表达的lncrna,分别筛选上下游100kb,并进行lncRNA-mRNA对表达相关性分析。共发现叶片和根中差异表达lncrna的lncRNA-mRNA对分别为2561对和1142对(TablegydF4y2BaS3gydF4y2Ba).GO分析预测了在生物过程的主要类别中以下亚类别中的一些差异基因:氮化合物代谢过程、氧化应激反应和染色质重塑。此外,还富集了氮化物转运、器官氮化物生物合成过程、硝酸盐代谢过程、外部反应调节等GO项(表)gydF4y2BaS5gydF4y2Ba).gydF4y2Ba
另一方面,检测不同生物过程中反式lncrna对基因表达调控的影响。根据lncrna与mrna的表达相关性(Pearson correlation> 0.95),在叶片和根中分别发现了59577和22227个lncrna与ln -响应基因的共表达关系(TablegydF4y2BaS4gydF4y2Ba).分析了GO类别和子类别,结果预测,大多数这些基因是叶和根部的有机物质生物合成,α-氨基酸代谢和光合膜。gydF4y2Ba
我们接下来分析了用差异表达基因的Kegg途径富集分析,并介绍了TOP20富集的途径(表gydF4y2BaS6gydF4y2Ba).结果表明,反式lncrna在与半胱氨酸和蛋氨酸代谢、氰胺代谢相关的几个途径中富集。同时,还发现了一些与氮代谢、丙氨酸、天冬氨酸和谷氨酸代谢相关的富集途径。在gydF4y2BaCIS.gydF4y2Ba-acting,这些基因的结果在叶绿素代谢、次生代谢物的生物合成以及光合生物体的碳固定中起作用。这些富集的生物过程和途径与谷氨酰胺家族氨基酸的生物合成过程和非生物胁迫有关,表明差异表达的lncrna在玉米发育过程中氮素吸收和转运中发挥着重要作用。gydF4y2Ba
LNC_002923gydF4y2Ba抑制裂解gydF4y2BaZM00001D015521gydF4y2Ba通过gydF4y2BaZmmiR159gydF4y2Ba
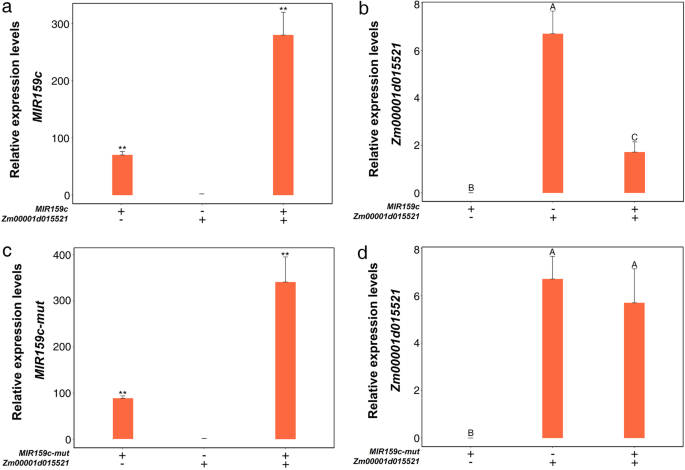
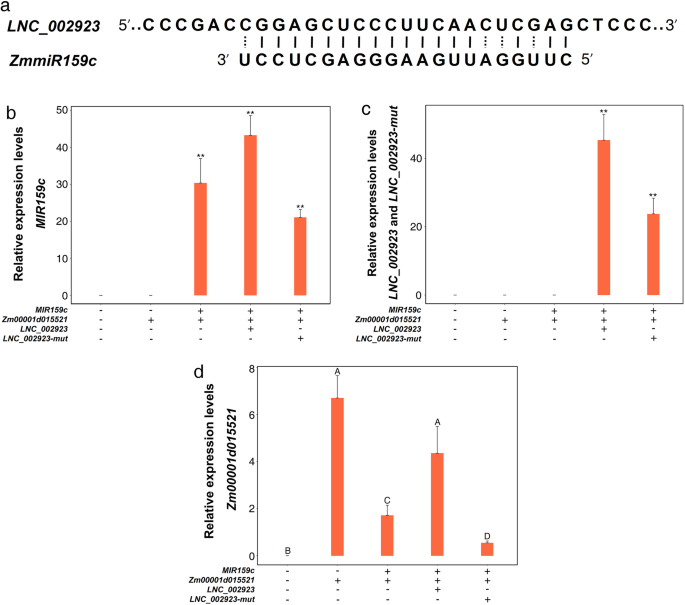
根据我们之前预测的共表达调控网络,我们通过实验来验证lncRNA是否影响了mRNA-miRNA对。的gydF4y2BaZM00001D015521gydF4y2Ba预测的靶基因是gydF4y2BaZmmiR159cgydF4y2Ba.我们使用gydF4y2BaZmmiR159cgydF4y2Ba和gydF4y2BaZmmiR159c-mutgydF4y2Ba构建瞬态表达测定gydF4y2Ba烟草benthamianagydF4y2Ba,其中gydF4y2BaZmmiR159c-mutgydF4y2Ba是六个基因座的设计底漆gydF4y2BaZmmiR159cgydF4y2Ba成熟序列引入目标突变。的表达水平gydF4y2BaZmmiR159cgydF4y2Ba和gydF4y2BaZmmiR159c-mutgydF4y2Ba采用qRT-PCR检测(图。gydF4y2Ba5gydF4y2Baa、c).如图。gydF4y2Ba5gydF4y2BaB, d,表达水平gydF4y2BaZM00001D015521gydF4y2Ba与gydF4y2BaZmmiR159cgydF4y2Ba,然而,表达水平gydF4y2BaZM00001D015521gydF4y2Ba与之共识时没有受到影响gydF4y2BaZmmiR159c-mutgydF4y2Ba.这些结果表明gydF4y2BaZM00001D015521gydF4y2Ba是ZMMIR159C的靶基因之一。先前的研究发现,LNCRNA可以与miRNA结合,从而减轻miRNA对靶基因的抑制作用。在这项研究中,绑定部位gydF4y2BaZmmiR159cgydF4y2Ba被发现在gydF4y2BaLNC_002923gydF4y2Ba序列(图。gydF4y2Ba6gydF4y2Ba一种)。我们还介绍了六个突变gydF4y2BaZmmiR159cgydF4y2Ba的序列gydF4y2BaLNC_002923 (LNC_002923-mut)gydF4y2Ba.然后,我们进行瞬时表达分析gydF4y2Ba烟草benthamianagydF4y2Ba检测是否gydF4y2BaLNC_002923gydF4y2Ba能栖居于乳沟之间吗gydF4y2BaZM00001D015521gydF4y2Ba通过gydF4y2BaZmmiR159cgydF4y2Ba.的表达水平gydF4y2BaZmmiR159c, LNC_002923gydF4y2Ba和gydF4y2BaLNC_002923-mutgydF4y2Ba采用qRT-PCR检测(图。gydF4y2Ba6gydF4y2Bab、c)。如图所示。gydF4y2Ba6gydF4y2BaD,表达水平gydF4y2BaZM00001D015521gydF4y2Ba没有受到影响gydF4y2BaLNC_002923gydF4y2Ba与之联合表达gydF4y2BaZmmiR159cgydF4y2Ba,这表明gydF4y2BaLNC_002923gydF4y2Ba能有效抑制卵裂吗gydF4y2BaZM00001D015521gydF4y2Ba通过gydF4y2BaZmmiR159cgydF4y2Ba.然而,gydF4y2BaZM00001D015521gydF4y2Ba共表达时,表达量显著降低gydF4y2BaLNC_002923-mutgydF4y2Ba.这些结果表明gydF4y2BaLNC_002923gydF4y2Ba能降低抑制作用吗gydF4y2BaZmmiR159cgydF4y2Ba.gydF4y2Ba
共表达组合表达质粒gydF4y2BaMIR159c / Zm00001d015521gydF4y2Ba(gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba),gydF4y2BaMIR159c-mut / Zm00001d015521gydF4y2Ba(gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba)gydF4y2Ban benthamianagydF4y2Ba.的相对表达水平gydF4y2BaMIR159cgydF4y2Ba(gydF4y2Ba一个gydF4y2Ba),gydF4y2BaMIR159c-mutgydF4y2Ba(gydF4y2BacgydF4y2Ba),用qRT-PCR检测,数据用U6基因归一化。的相对表达水平gydF4y2BaZM00001D015521gydF4y2Ba(gydF4y2BabgydF4y2Ba,gydF4y2BadgydF4y2Ba),利用烟草18S基因进行归一化处理。相同的字母在gydF4y2BaPgydF4y2Ba< 0.01。**,重要的gydF4y2BaPgydF4y2Ba < 0.01
互补结合序列图gydF4y2BaLNC_002923gydF4y2Ba和gydF4y2BaZmmiR159gydF4y2Ba在玉米(gydF4y2Ba一个gydF4y2Ba).共表达组合表达质粒gydF4y2BaMIR159cgydF4y2Ba,gydF4y2BaZM00001D015521gydF4y2Ba,gydF4y2BaLNC_002923gydF4y2Ba,gydF4y2BaLNC002923-mutgydF4y2Ba(gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba).的相对表达水平gydF4y2BaMIR159cgydF4y2Ba(gydF4y2BabgydF4y2Ba)用U6基因进行归一化。利用烟草18S基因对mRNA和lncRNA的相对表达量进行归一化处理(gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba).相同的字母在gydF4y2BaPgydF4y2Ba< 0.01。**,重要的gydF4y2BaPgydF4y2Ba < 0.01
HN和LN条件下的表型差异gydF4y2Ba
描述性统计和广义遗传力估计(HgydF4y2Ba2gydF4y2Ba)的17个玉米幼苗性状见表gydF4y2Ba2gydF4y2Ba.通过对均线的分析,可以揭示基因型和对比N浓度对玉米幼苗的意义。除了精细根数(SRN)之外,大多数特征在LN应力和控制之间显示出显着差异。这些发现表明,对于随后的关联分析,对自然群体的收集足够多样化。平均而言,芽增长在LN条件下有限,而根生长得到提高。在HN(36.278)下的芽长(SL)比LN(30.377)处理更大。此外,在LN下的根系干重(RDW)和总根长度(TR1)较高,而不是在HN下。Pearson的相关系数针对表格下列出的氮水平下的17个收集的性状计算gydF4y2BaS8gydF4y2Ba.gydF4y2Ba
全基因组关联分析揭示的候选基因gydF4y2Ba
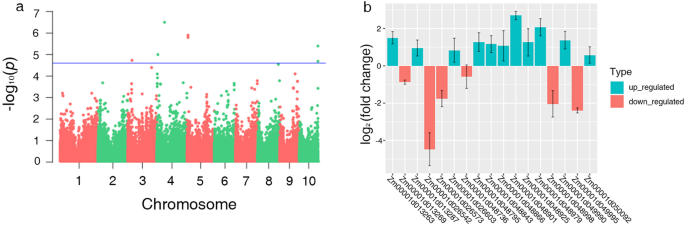
为了在全基因组范围内识别与性状相关的显著SNPs,利用TASSEL 5.0软件,利用混合线性模型(MLM)和标记agydF4y2BapgydF4y2Ba-value <1e−4.6用于候选基因分析。低氮耐受性指数(LNTI)检测到23项显著标记,LN处理下检测到3项显著标记。其中12个snp与冠根数(CRN)相关。7个和3个显著的关联分别与Forks和tip相关,3个标记与crossing和平均根直径(ARD)相关。此外,还发现一个SNP与两个根性状(叉和尖)显著相关。根据定位群体内所有10条玉米染色体的平均连锁不平衡衰减距离(由TASSEL 5.0计算),在这些显著相关的SNP标记附近筛选出1474个候选基因。3个显著标记分别与LN处理下的232个基因相关,分别为put - 161a -13,126,581-167、ze -104092320和ze -109052750,分别与种子根数(SRN)、Crossings和Tips相关,分别位于第4、6和9号染色体上。同时,LNTI值显示了23个与1242个候选基因相关的显著snp。表中列出了所有显著标记的特质关联gydF4y2BaS7gydF4y2Ba.根据鉴定出的lncrna的位置,分别在LN和LNTI下筛选出显著标记范围内不同表达的lncrna分别为36个和134个(TablegydF4y2BaS7gydF4y2Ba).这些LN响应性候选基因和LNCRNA可以在玉米幼苗阶段调节根系发展方面发挥重要作用。gydF4y2Ba
结合GWAS和表达谱挖掘一致的候选基因gydF4y2Ba
结合GWAS和RNA-Seq,我们发现在LN条件下GWAS检测到的232个候选基因中,16个和29个候选基因分别表现出明显的下调和上调模式,其中7个候选基因在根和叶中表达(图1)。gydF4y2BaS6gydF4y2Ba和表gydF4y2BaS7gydF4y2Ba).同时,在LNTI下GWAS检测到的1013个候选基因中,分别有107个和125个基因表达下调和上调,其中38个候选基因在根和叶中表达(图1)。gydF4y2BaS6gydF4y2Bab和表gydF4y2BaS7gydF4y2Ba).此外,整合多个RNA-SEQ和GWA的结果,共有10个重要的SNP(gydF4y2BaPgydF4y2Ba< 1e−4.6)与利用根性状LNTI鉴定出的34个候选基因相关,在LN胁迫下发现了与6个候选基因相关的一个显著SNP(表gydF4y2Ba3.gydF4y2Ba).通过LN胁迫下玉米幼苗根系的RNA-Seq检测,对255个玉米品系中gwass鉴定的候选基因的表达水平进行了评价。这包括在整合LN胁迫下玉米的meta分析和大规模基因表达谱数据中发现两个基因[gydF4y2Ba22gydF4y2Ba].例如,gydF4y2BaZM00001D051804gydF4y2Ba在玉米和拟南芥种子萌发过程中响应外源氮的谷氨酰胺生物合成中起重要作用[gydF4y2Ba26gydF4y2Ba].此外,gydF4y2BaZm00001d048998gydF4y2Ba是一种叶绿素A-B结合蛋白,参与玉米氮营养物的吸收和利用[gydF4y2Ba21gydF4y2Ba]。对于其他基因,gydF4y2BaZm00001d051666gydF4y2Ba和gydF4y2BaZm00001d049380gydF4y2Ba被发现与玉米根系的氮获取有关[gydF4y2Ba23gydF4y2Ba].最后,在玉米根系响应氮胁迫的RNA-seq中发现了6个候选基因[gydF4y2Ba24gydF4y2Ba在比较涉及玉米碳和氮代谢的研究时,始终检测到29个基因[gydF4y2Ba25gydF4y2Ba),分别。fork的曼哈顿图和RNA-Seq的折叠变化值如图所示。gydF4y2Ba7gydF4y2BaA,B。在我们的研究中探索的这些一致的候选基因可以强烈地促进缺乏缺乏耐受性。gydF4y2Ba
为了研究lncRNA在LN胁迫下的玉米幼苗中的调控作用,使用预测lncRNA靶点,潜在的lncRNA可以影响40个一致的lncRNA候选基因gydF4y2BaCIS.gydF4y2Ba或gydF4y2Ba反式gydF4y2Ba的方式确定。如图所示。gydF4y2Ba4gydF4y2Bac,共发现354个lncrna。此外,这些lncrna的靶点包含了显著的多根性状snp (TablegydF4y2BaS9gydF4y2Ba), lncRNA靶点与氮代谢和非生物胁迫响应途径显著相关。在这些lncrna中,我们发现LNC_002984、LNC_002985和LNC_002986位于gydF4y2BaZM00001D051804gydF4y2Ba这可能会调节邻近基因的表达水平gydF4y2BaCIS.gydF4y2Ba方式。gydF4y2BaZM00001D051804gydF4y2Ba据报道,与对低氮素胁迫的反应有关[gydF4y2Ba22gydF4y2Ba].此外,拟南芥的同源基因在缺铵条件下的铵同化和促进根中氮的再转移中发挥了重要的冗余作用[gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba].这意味着这些LNCRNA可以在根系发育中发挥重要作用,特别是在植物中的氮吸收,转移和同化中起作用。本分析提供了解LNCRNA作为与LN压力相关的生长和发展中的重要调节因素的新信息。gydF4y2Ba
讨论gydF4y2Ba
对非编码RNA调控机制的研究越来越多。在哺乳动物和植物中发现了许多潜在的lncrna,并发现它们与人类疾病和植物中参与生物学过程的基因表达水平的调节有关[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].ceRNA假说是解释lncrna和蛋白质编码基因如何通过microRNA响应元件(MREs)相互沟通的新选择。lncrna的全基因组筛选和分析可以为植物应对LN胁迫提供新的思路。在本研究中,我们对12个lncRNA和12个小RNA文库进行了深度测序,这些小RNA文库来自于暴露于HN和LN条件早期的玉米幼苗。我们鉴定了894个可靠的差异表达lncrna和38个mirna,包括19个新mirna。gydF4y2Ba
microRNA在LNCRNA和MRNA之间调解沟通gydF4y2Ba
MicroRNA作为共表达网络的核心负责介导LNCRNA和MRNA。我们确定了gydF4y2BamiR159cgydF4y2Ba和gydF4y2BamiR164agydF4y2Ba可以与单个lncrna通信,这些lncrna通过竞争的特定mirna进行通信。gydF4y2BaMiR159gydF4y2Ba在花药和种子中控制gamyb样基因的表达;这些转录因子参与了ga诱导糊粉层的发育和死亡[gydF4y2Ba32gydF4y2Ba],也被报道调控许多靶基因,如MYB转录因子和保守的R2R3结构域[gydF4y2Ba33gydF4y2Ba].由gydF4y2BamiR159gydF4y2Ba可减少MYB33/65的抑制,在营养发育中发挥重要作用[gydF4y2Ba34gydF4y2Ba].具体地说,gydF4y2BamiR159gydF4y2Ba据报道参与了对磷酸盐缺乏的反应[gydF4y2Ba35gydF4y2Ba].然而,研究gydF4y2BamiR159gydF4y2Ba在LN应激背景下的功能尚未见报道。gydF4y2BaMiR164gydF4y2Ba据报道参与了侧根的形成[gydF4y2Ba36gydF4y2BaLN应激反应[gydF4y2Ba37gydF4y2Ba].在nitrate-starvation条件下,gydF4y2BamiR164gydF4y2Ba可以将其靶基因NAC1在转录后水平的第10和第11个碱基之间削减以抵抗LN可用性[gydF4y2Ba38gydF4y2Ba].总之,在共表达网络下,gydF4y2BaLNC_003272gydF4y2Ba和gydF4y2BaLNC_002923gydF4y2Ba可以在生物过程中发挥枢轴作用,包括根系发展和种子萌发,以及可能参与抑制miRNA响应氮缺乏的影响。gydF4y2Ba
共表达网络在幼苗生长过程中表现出动态的表达和调控模式gydF4y2Ba
结果显示,该网络包含2个lncRNAs、2个miRNAs、14个mrna,形成了两个共表达调控网络(图1)。gydF4y2Ba4gydF4y2BaqRT-PCR分析显示,在LN治疗后的大多数阶段,共表达网络的模式与ceRNA假说相似。对于叶片共表达网络,表达趋势为gydF4y2BaZm00001d023434gydF4y2Ba与之一致吗gydF4y2BaLNC_002923gydF4y2Ba,但与mir159c的相反。在根中,MIR164a的表达显示了相反的图案gydF4y2BaZm00001d041472gydF4y2Ba,但与之相一致gydF4y2BaZm00001d003414gydF4y2Ba在压力14天后。表达gydF4y2BaLNC_003272gydF4y2Ba除10天和14天外,其他阶段均未检出。与此同时,基因gydF4y2BaZM00001D015521gydF4y2Ba据报道,可调节植物激素细胞分裂素的代谢,受叶绿素生物合成基因的影响,增加吸收根的数量,促进幼苗生长[gydF4y2Ba39gydF4y2Ba].此外,gydF4y2BaZm00001d041472gydF4y2Ba被发现参与植物组织中的氮气积聚,并改善玉米的温度和产量[gydF4y2Ba40gydF4y2Ba].综合这些发现,我们提出了这样的建议gydF4y2BaLNC_002923gydF4y2Ba和gydF4y2BaLNC_003272gydF4y2Ba可能参与氮素利用途径调控叶绿体发育、氮素利用,并可能参与抑制mirna响应氮缺乏的作用。gydF4y2Ba
LNC_002923gydF4y2Ba抑制裂解gydF4y2BaZM00001D015521gydF4y2Ba通过gydF4y2BaZmmiR159gydF4y2Ba
以往的研究表明,lncrna可以调控mRNA的翻译,并与mirna竞争,间接影响动植物中编码基因的表达[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].我们预测了两种基于lncrna和mrna具有相同的miRNA结合位点的共表达网络。将基因构建到pCAMBIA2300-35S-OCS载体中,进行不同组合的瞬时表达gydF4y2Ba烟草benthamianagydF4y2Ba.我们证明了gydF4y2BaZmmiR159gydF4y2Ba可以切断目标基因吗gydF4y2BaZM00001D015521gydF4y2Ba并抑制其表达水平。此外,我们发现gydF4y2BaLNC_002923gydF4y2Ba能抑制裂解吗gydF4y2BaZM00001D015521gydF4y2Ba基因指导下gydF4y2Bazmmir159c。gydF4y2Ba的同源物gydF4y2BaZM00001D015521gydF4y2Ba在拟南芥中发现,与根和茎的发育和生长以及干旱和营养等非生物胁迫有关[gydF4y2Ba39gydF4y2Ba与此同时,)gydF4y2BamiR159gydF4y2Ba已报道参与非生物胁迫响应相关通路和植物生长发育[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].根据这些结果,我们得出结论,相对较高的丰度gydF4y2BaLNC_002923gydF4y2Ba能有效地抑制gydF4y2BaZmmiR159cgydF4y2Ba切割潜在的编码基因,并进一步调控gydF4y2BaZM00001D015521gydF4y2Ba在玉米响应低氮胁迫中发挥重要的调控作用。gydF4y2Ba
GWAS和RNA-Seq的低氮耐受性gydF4y2Ba
这里有必要将多种方法组合以获得可靠的信息。一种综合方法,包括GWA,RNA-SEQ和基因组选择与不同环境中收集的表型数据相结合,发现16个基因座,其与田间的白色模具和温室中的11个基因座显着相关,并在温室中具有11个基因座[gydF4y2Ba42gydF4y2Ba].在玉米中,结合代谢谱和GWAS分析了低磷胁迫的响应机制,并在一个重组自交系群体中验证发现了一些与产量相关的候选基因[gydF4y2Ba20.gydF4y2Ba].我们通过整合RNA-Seq谱和GWAS数据筛选了40个一致响应ln的候选基因。值得注意的是,在这12个与5个根系性状相关的显著SNPs中,有7个与第4染色体上的Forks、Crossings、tip、PRL和CRN相关。之前的GWAS研究[gydF4y2Ba43gydF4y2Ba还发现候选基因控制染色体4.此外,一些研究还检测到与氮利用和磷吸收相关的QTL,位于染色体4上[gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba].本研究结果与先前研究结果的一致性表明,LN胁迫下可能影响根系生长和养分利用的候选基因可能位于第4染色体上。gydF4y2Ba
目标分析揭示了玉米中LN响应LNCRNA的潜在监管gydF4y2Ba
我们对lncrna进行了GO和KEGG分析,发现有大量的GO项与组氨酸生物合成过程、硝酸盐代谢过程和染色质重塑相关。我们的研究结果表明,这些lncrna参与调控非生物胁迫和营养代谢[gydF4y2Ba12gydF4y2Ba,gydF4y2Ba46gydF4y2Ba].此外,针对植物生长和营养代谢相关生物学过程的KEGG分析表明,这些lncrna可以与玉米幼苗在LN胁迫下的mrna相互作用,特别是与光合作用和次生代谢物合成有关(gydF4y2BapgydF4y2Ba< 0.05)。综上所述,我们认为这些lncrna参与了包括发育、生物合成和非生物胁迫响应在内的关键生物学过程。gydF4y2Ba
在lncRNA-mRNA对中,预测有44个和310个lncrna调控24个邻近基因和16个远缘基因的转录激活和表达gydF4y2BaCIS.gydF4y2Ba和gydF4y2Ba反式gydF4y2Ba,并被多个lncrna调控。特别是lncrna与gydF4y2BaZM00001D051804gydF4y2Ba,gydF4y2BaZm00001d048998gydF4y2Ba,gydF4y2BaZm00001d051666gydF4y2Ba,gydF4y2BaZm00001d049380gydF4y2Ba,在决定根系生长和响应氮的有效性方面发挥着重要作用gydF4y2BaCIS.gydF4y2Ba和gydF4y2Ba反式gydF4y2Ba态度,值得进一步研究。我们的研究结果对于进一步探索lncrna及其靶基因在LN胁迫下幼苗的功能具有重要意义。然而,到目前为止,尚未有研究将GWAS和lncRNAs结合起来研究玉米幼苗LN反应的机制。gydF4y2Ba
结论gydF4y2Ba
我们在玉米幼苗中鉴定了几百个差异表达的lncRNA,并基于“ceRNA假说”,利用它们构建了两个lncRNA为ceRNA的共表达网络。结合GWAS和表达谱,共鉴定出40个一致响应ln的候选基因和可能与根系性状相关的lncrna。进一步研究lncRNA的生物学功能和调控,包括近、远靶点的识别、GO富集和KEGG分析,将为深入了解lncRNA在玉米幼苗缺氮早期阶段的调控机制提供有用的信息。为提高氮肥利用的育种效率提供了新的见解。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
玉米自交系P178和gydF4y2Ba烟草benthamianagydF4y2Ba在本研究中使用(两者都由四川农业大学玉米研究所提供)。关联映射面板包括362条近交系,具有从西南养殖计划获得的遗传背景上的差异,众所周知,在我们的透水研究中,包括人口结构,遗传多样性和联系不平衡衰减距离[gydF4y2Ba47gydF4y2Ba].gydF4y2Ba
生长条件gydF4y2Ba
以玉米自交系178为材料,进行表面处理gydF4y2Ba- - - - - -gydF4y2Ba用6%次氯酸钠消毒15分钟,用去离子水洗涤两次,浸泡在饱和CaSO中gydF4y2Ba4gydF4y2Ba12 h后在粗石英砂中发芽,直至可见两片叶子。取去胚乳后,将幼苗置于盛有改良的半浓度霍格兰营养液的25 l桶中2天以适应水培环境,然后再提供全浓度霍格兰营养液。对HN处理,营养液由(mM): 4 KNO组成gydF4y2Ba3.gydF4y2Ba4 MgSOgydF4y2Ba4gydF4y2Ba, 5kcl, 5caclgydF4y2Ba2gydF4y2Ba1 KHgydF4y2Ba2gydF4y2Ba阿宝gydF4y2Ba4gydF4y2Ba, 0.1 Fe-EDTA, 0.046 HgydF4y2Ba3.gydF4y2Ba薄gydF4y2Ba4gydF4y2Ba0.009 MnSOgydF4y2Ba4gydF4y2Ba0.0007 ZnSOgydF4y2Ba4gydF4y2Ba0.0003 CuSOgydF4y2Ba4gydF4y2Ba和0.0002(nhgydF4y2Ba4gydF4y2Ba)gydF4y2Ba6gydF4y2Ba莫gydF4y2Ba7gydF4y2BaOgydF4y2Ba24gydF4y2Ba.对于LN的治疗,4mm KNOgydF4y2Ba3.gydF4y2Ba用0.04 mM KNOgydF4y2Ba3.gydF4y2Ba.调整溶液pH至6.0-6.5,营养液每2天更新一次。在光照强度为200 μmol光子m、光照周期为16/8 h(光/暗)、光照温度为25/22℃的温室中生长gydF4y2Ba−2gydF4y2Ba年代gydF4y2Ba−1gydF4y2Ba.相对湿度保持在65%。在HN和LN条件下进行了3次独立的重复,每个实验包括至少3个幼苗。从上到下的第二个完全展开的叶片和整个根系取样,在吸墨纸上干燥,立即在液态氮中冷冻gydF4y2Ba2gydF4y2Ba最后保存在−80℃。gydF4y2Ba
RNA分离,LNCRNA和小RNA测序的文库制剂gydF4y2Ba
在HN和LN条件下,从14日龄的幼苗叶片和根中提取总rna。lncrna和小rna文库在Illumina Hiseq2500平台上对12个标记为HN_178L1、HN_178L2、HN_178L3、HN_178R1、HN_178R2、HN_178R3、LN_178L1、LN_178L2、LN_178L3、LN_178R1、LN_178R2和LN_178R3的样本进行测序。使用NanoPhotometer®分光光度计(IMPLEN, CA, USA)检测RNA纯度。RNA浓度使用Qubit®RNA检测试剂盒在Qubit®2.0 Flurometer (Life Technologies, CA, USA)中测量,完整性使用Bioanalyzer 2100系统的RNA Nano 6000检测试剂盒(Agilent Technologies, CA, USA)进行评估。每个样本共3 μg RNA作为RNA和小RNA库的输入材料。RNA产品纯化后(AMPure XP系统),文库质量在Agilent Bioanalyzer 2100系统上进行评估。文库序列由Novogene公司(北京)在Illumina Hiseq 2500平台上进行测序。gydF4y2Ba
lncrna的生物信息学鉴定gydF4y2Ba
用于lncrna鉴定的管道如图所示。gydF4y2Ba1gydF4y2Ba一种。通过删除包含适配器的读取,包含Poly-n的读取和从原始数据的低质量读取来获得清洁数据。映射到B73 Refgen_v4基因组的高质量的清洁数据直接从基因组网站下载(gydF4y2Bahttp://www.gramene.org/gydF4y2Ba).使用Bowtie V2.0.6建造了参考基因组的指数[gydF4y2Ba48gydF4y2Ba]和对端干净reads使用TopHat对参照基因组进行对齐[gydF4y2Ba49gydF4y2Bav2.0.9。比对后,每个样本的映射reads都由圣经(beta2) [gydF4y2Ba50gydF4y2Ba和袖扣(v2.1.1) [gydF4y2Ba51gydF4y2Ba以参考为基础的方法。这两种方法都使用拼接读取来确定外显子的连通性,但有两种不同的方法。接下来,CPC预测的具有编码潜力的转录本[gydF4y2Ba52gydF4y2Ba]及pam扫描[gydF4y2Ba53gydF4y2Ba],并将无编码潜能的lncrna作为我们的候选lncrna集。Cuffdiff (v2.1.1) [gydF4y2Ba51gydF4y2Ba]计算每个样本中lncrna和编码基因的FPKMs。Cuffdiff提供了统计例程,使用基于负二项分布的模型来确定数字转录本或基因表达数据中的差异表达。经过调整的成绩单gydF4y2BaPgydF4y2Ba< 0.05为差异表达。gydF4y2Ba
小RNA测序数据分析gydF4y2Ba
去除含有poly-N、5 '适配器污染、不含3 '适配器或插入标签的reads,以及原始数据中低质量的reads后,用Bowtie [gydF4y2Ba48gydF4y2Ba不匹配,以便进一步分析。Rfam 11.0 [gydF4y2Ba54gydF4y2Ba]用于去除来自蛋白质编码基因、重复序列、rRNA、tRNA、snRNA和snoRNA的标记。然后,利用映射的小RNA标签寻找已知的miRNAs。参考MiRBase20.0,修改后的mirdeep2 [gydF4y2Ba55gydF4y2Ba]和sRNA-tools-cli获取潜在的mirna并绘制二级结构。miRNA前体的发夹结构特征可用于预测新的miRNA。软件miREvo [gydF4y2Ba56gydF4y2Ba]和mirdeep2 [gydF4y2Ba55gydF4y2Ba通过探索二次结构,加床裂解位点和在前一步骤中未经发挥的小RNA标签的最小自由能来实现以预测新的miRNA。通过以下标准通过每百万(TPM)的转录物估计表达水平[gydF4y2Ba57gydF4y2Ba].使用DESEQ R包(1.8.3)进行两种条件/组的差异表达分析。的gydF4y2BaPgydF4y2Ba使用Benjamini和Hochberg方法调整值。纠正gydF4y2BaPgydF4y2Ba默认设置-value < 0.05为显著差异表达的阈值。gydF4y2Ba
预测靶基因gydF4y2Ba
的gydF4y2BaCIS.gydF4y2Ba-作用机制是lncrna作用于邻近靶基因。为了鉴定这些lncrna,我们搜索了这些lncrna上下游10 - 100kb的编码基因;然后对鉴定出的细胞进行功能分析。的gydF4y2Ba反式gydF4y2Ba- lncrna通过表达水平相互识别的作用机制。我们对来自不同样本的基因进行聚类,寻找共同的表达模块,然后通过功能富集分析对其功能进行分析。通过psRobot_tar在psRobot中预测miRNA的靶基因[gydF4y2Ba58gydF4y2Ba对植物。gydF4y2Ba
实时荧光定量PCR的验证gydF4y2Ba
施用定量实时PCR(QRT-PCR)以验证测序结果。从0小时,1小时,6小时,12小时,24小时,2天,4天,6天,8天,10天,12天和14天,从叶片和根部的叶片和根中提取总RNA。三苯醇试剂(Invitrogen)的氮处理。使用Primescript TM II第1链CDNA合成试剂盒(TAKARA)和SYBR®Primescript™miRNARTT-PCR试剂盒(Takara)进行逆转MRNA和小RNA,并在制造商的说明之后进行。gydF4y2Ba
lncrna、mrna和miRNA的qRT-PCR验证使用罗氏Cobas Z480系统,使用FastStart Essential DNA Green Master(罗氏)和SYBR®PrimeScriptTM miRNA RT-PCR Kit (TAKARA)。数据以myosin (mRNA和lncRNA qPCR)和U6 (miRNA qPCR)基因为标准进行相对定量分析。进行3个独立试验,每个试验在3个技术重复中进行。gydF4y2Ba
与玉米发展相关的共表达网络的构建gydF4y2Ba
基于“ceRNA假说”,MREs可以作为转录本相互影响调节其表达水平的核心。首先,将差异表达lncRNA的靶基因与差异表达mrna结合分析。当lncrna的靶点也存在显著差异时,mrna更容易受到lncrna的调控。然后,根据lncRNA与miRNA前体的同源性,筛选出可能是miRNA前体的lncRNA,利用psRobot软件预测miRNA的靶lncRNA。第三,各组差异表达基因的上调和下调结果,以识别作用于mrna的候选miRNAs。最后构建以lncRNA为诱饵,miRNA为核心,mRNA为靶基因的共表达网络。gydF4y2Ba
标记数据gydF4y2Ba
使用包含56110个SNPs的MaizeSNP50 BeadChip对面板进行基因分型。该芯片的详细资料可从Illumina MaizeSNP50网站(gydF4y2Bahttp://support.illumina.com/array/array_kits/gydF4y2Bamaizesnp50_dna_analysis_kit/downloads.html)和B73 RefGen_v2中snp的位置信息可以从国家生物技术信息中心(NCBI) GEO网站上下载。gydF4y2Ba
表型测量gydF4y2Ba
采用纸辊生长法培养玉米[gydF4y2Ba59gydF4y2Ba].生长条件和营养液与前面描述的相同。然后,用6%次氯酸钠钠对种子进行表面消毒,放在潮湿的滤纸上,在黑暗中发芽。2天后,将6颗萌发的玉米粒置于双层棕色发芽卷纸(Anchor paper, St. Paul, MN, USA)上,该卷纸预先用杀菌剂Captan溶液(2.5 g/L)保湿。发芽纸卷垂直放置在装有5 l营养液(HN和LN)的10 l塑料桶中。调整溶液pH为6.0-6.5。营养液每2天更新一次。gydF4y2Ba
以每卷六株幼苗为实验单元。联想系以完全随机的设计在温室中完成三个独立的复制。14天后,将幼苗从塑料桶中取出,测量表型性状(每卷6株幼苗中取样3株相似的幼苗,以消除行内可能的异常值,并取所有性状的平均值)。如果不能在某一天进行测量,则用30%的乙醇替换营养液,以防止进一步生长。利用WinRhizo程序记录根系性状。测量完成后,分别收集芽和根,在80°C的烘箱烘干机中干燥至少48小时。gydF4y2Ba
表型分析gydF4y2Ba
使用R软件分析表型描述性统计和相关系数。幼苗特征的变化分析和广义遗传性(HgydF4y2Ba2gydF4y2Ba)由SAS执行。采用低氮耐受性指数(LNTI)性状和均值进行GWAS, LNTI为HN处理与LN处理的相对性状值除以LN处理的相同性状值。gydF4y2Ba
耐低氮相关性状的关联分析gydF4y2Ba
使用PowerMarker3.25软件计算255米玉米自交系和人口结构的等位基因频率[gydF4y2Ba60gydF4y2Ba和结构2.3 [gydF4y2Ba61gydF4y2Ba),分别。用基因组关联和预测集成工具-R封装(Gapit)和结构设定为k = 2,根据前一项研究的结果进行测量[gydF4y2Ba47gydF4y2Ba].剔除低次要等位基因频率(MAF) < 0.01和缺失率> 0.2的SNPs,剩下46108个高质量SNPs可供进一步关联分析。所有标记均均匀分布在1 ~ 10条染色体上。TASSEL 5.0软件[gydF4y2Ba62gydF4y2Ba选择使用混合线性模型(MLM)和标记用46,108高质量的SNP(MAF> 0.01)进行GWA进行。gydF4y2BaPgydF4y2Ba考虑> 4.6作为候选基因分析。gydF4y2Ba
瞬时表达gydF4y2Ba烟草benthamianagydF4y2Ba
用特异引物扩增基因全长cDNA和lncRNA。的前体gydF4y2BaZmMIR159gydF4y2Ba和gydF4y2BaZmMIR164gydF4y2Ba从基因组DNA中扩增。作为阴性对照,序列gydF4y2BaLNC_002923gydF4y2Ba,gydF4y2BaLNC_003272gydF4y2Ba,gydF4y2BaZmMIR159gydF4y2Ba和gydF4y2BaZmMIR164gydF4y2Ba使用Mut Express II Fast mutagenesis Kit V2 (Vazyme, Nanjing, CN)进行定点突变。将扩增片段克隆到pCAMBIA2300-35S-OCS载体中gydF4y2Ba萨尔gydF4y2BaI限制站点由In-Fusion (TAKARA)。转化表达质粒gydF4y2Ba农gydF4y2BaEHA105并注射到表皮中gydF4y2Ban benthamianagydF4y2Ba瞬时表达。进行四次独立实验。2天后用上述的Trizol试剂(Invitrogen)从叶片中提取RNA。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
所有原始数据均可从NCBI数据库(登录号:PRJNA661965和PRJNA662035)下载。gydF4y2Ba
缩写gydF4y2Ba
- HN:gydF4y2Ba
-
高氮gydF4y2Ba
- LN:gydF4y2Ba
-
低氮gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- 单核苷酸多态性:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- LncRNAs:gydF4y2Ba
-
长时间的RNAgydF4y2Ba
- 龙头:gydF4y2Ba
-
内源性rna竞争gydF4y2Ba
- 研究硕士:gydF4y2Ba
-
微反应元素gydF4y2Ba
- LD:gydF4y2Ba
-
连锁不平衡gydF4y2Ba
- TPM:gydF4y2Ba
-
记录每百万gydF4y2Ba
- 存在:gydF4y2Ba
-
定量实时聚合酶链反应gydF4y2Ba
- HgydF4y2Ba2gydF4y2Ba:gydF4y2Ba
-
遗传gydF4y2Ba
- lnti:gydF4y2Ba
-
耐低氮指数gydF4y2Ba
- MAF:gydF4y2Ba
-
轻微的等位基因频率gydF4y2Ba
- 传销:gydF4y2Ba
-
混合线性模型gydF4y2Ba
- tucps:gydF4y2Ba
-
编码潜力不确定的转录本gydF4y2Ba
- SRN:gydF4y2Ba
-
的根数gydF4y2Ba
- RDW:gydF4y2Ba
-
根干重gydF4y2Ba
- SL:gydF4y2Ba
-
拍摄长度gydF4y2Ba
- 实验室:gydF4y2Ba
-
总根长度gydF4y2Ba
- CRN:gydF4y2Ba
-
皇冠根号码gydF4y2Ba
参考gydF4y2Ba
- 1.gydF4y2Ba
徐刚,范晓明,徐国良。植物氮素同化与利用效率。植物生物学杂志。2012;63:153-82。gydF4y2Ba
- 2.gydF4y2Ba
关键词:植物,氮,营养,信号转导植物生物学杂志。2017;39:57-65。gydF4y2Bahttps://doi.org/10.1016/j.pbi.2017.05.010gydF4y2Ba.gydF4y2Ba
- 3.gydF4y2Ba
acta photonica sinica, 2011, 38(5): 732 - 736 .李松,田勇,吴坤,叶勇,于军,张军,等。为可持续农业调节植物生长-代谢协调。大自然。2018;560:595 - 600。gydF4y2Bahttps://doi.org/10.1038/s41586-018-0415-5gydF4y2Ba.gydF4y2Ba
- 4.gydF4y2Ba
Chiba Y,Shimizu T,Miyakawa S,Kanno Y,Koshiba T,Kamiya Y等。鉴定拟南芥NRT1 / PTR家族(NPF)蛋白质能够运输植物激素。j植物res。2015; 128:679-86。gydF4y2Ba
- 5.gydF4y2Ba
Lezhneva L, Kiba T, Feria-Bourrellier AB, Lafouge F, Boutet-Mercey S, Zoufan P等。拟南芥硝酸盐转运蛋白NRT2.5在缺氮植物中发挥着获取和再转运硝酸盐的作用。植物j . 2014; 80:230-41。gydF4y2Ba
- 6.gydF4y2Ba
De Angeli A, monachelo D, Ephritikhine G, Frachisse JM, Thomine S, Gambale F,等。硝酸盐/质子逆向转运蛋白AtCLCa介导植物液泡中硝酸盐的积累。大自然。2006;442:939-42。gydF4y2Ba
- 7.gydF4y2Ba
Krouk G, Lacombe B, Bielach A, perrin - walker F, Malinska K, Mounier E等。NRT1.1调控植物生长素的转运,定义了植物营养感知的机制。Dev细胞。2010;18:927-37。gydF4y2Ba
- 8.gydF4y2Ba
Loqué D, Lalonde S, Looger LL, Von Wirén N, Frommer WB。反式激活域对铵的吸收至关重要的胞质反式激活域大自然。2007;446:195-8。gydF4y2Ba
- 9.gydF4y2Ba
柯普F,门德尔JT。长链非编码rna的功能分类和实验解剖。细胞。2018;172:393 - 407。gydF4y2Bahttps://doi.org/10.1016/j.cell.2018.01.011gydF4y2Ba.gydF4y2Ba
- 10.gydF4y2Ba
潘多菲,陈晓明,陈晓明,等。ceRNA假说:隐藏的RNA语言的罗塞塔石?细胞。2011;146:353-8。gydF4y2Bahttps://doi.org/10.1016/j.cell.2011.07.014gydF4y2Ba.gydF4y2Ba
- 11.gydF4y2Ba
王Y,Xu Z,Jiang J,Xu C,Kang J,Xiao L等。内源性miRNA海绵LINCRNA-ROR调节人类胚胎干细胞自更新的OCT4,NANOG和SOX2。DEV细胞。2013年; 25:69-80。gydF4y2Bahttps://doi.org/10.1016/j.devcel.2013.03.002gydF4y2Ba.gydF4y2Ba
- 12.gydF4y2Ba
杜强,王凯,邹超,徐超,李文轩。PILNCR1-miR399调控模块是玉米耐低磷的重要调控模块。植物杂志。2018;177:1743-53。gydF4y2Ba
- 13.gydF4y2Ba
徐XW,周Xh,王rr,彭wl,y,chen ll。竞争内源性RNA网络磷酸盐饥饿水稻中长性非编码RNA的功能分析。SCI REP。2016; 6(1月):1-12。gydF4y2Bahttps://doi.org/10.1038/srep20715gydF4y2Ba.gydF4y2Ba
- 14.gydF4y2Ba
等。关键词:Schnable;玉米B73基因组:复杂性、多样性和动态。科学(80 -)。2009; 326:1112-5。gydF4y2Ba
- 15.gydF4y2Ba
acta photonica sinica, 2011, 40(5): 762 - 768 .焦勇,裴鲁索,史静,梁涛,Stitzer MC,王斌,等。利用单分子技术改进玉米参考基因组。大自然。2017;546:524-7。gydF4y2Bahttps://doi.org/10.1038/nature22971gydF4y2Ba.gydF4y2Ba
- 16.gydF4y2Ba
Edwards AO, Ritter R, Abel KJ, Manning A, Panhuysen C, Farrer LA。补体因子H多态性与年龄相关性黄斑变性。科学(80 -)。2005; 308:421-4。gydF4y2Ba
- 17.gydF4y2Ba
毛辉,王辉,刘松,李忠,杨鑫,闫军,等。NAC基因中一个转座因子与玉米幼苗的耐旱性有关。Nat Commun。2015;6:1-13。gydF4y2Bahttps://doi.org/10.1038/ncomms9326gydF4y2Ba.gydF4y2Ba
- 18.gydF4y2Ba
关键词:玉米,根发育,全基因组关联分析,玉米abstract:BMC基因组学。2015;16;1 - 12。gydF4y2Ba
- 19.gydF4y2Ba
Sitonik C, Suresh LM, Beyene Y, Olsen MS, Makumbi D, Oliver K等。热带玉米种质中玉米褪绿斑驳病毒和致命坏死的遗传结构、连锁分析和基因组预测。Theor Appl Genet. 2019; 132:2381-99。gydF4y2Bahttps://doi.org/10.1007/s00122-019-03360-xgydF4y2Ba.gydF4y2Ba
- 20.gydF4y2Ba
罗斌,马平,聂震,张旭,何旭,丁旭,等。代谢物分析和全基因组关联研究揭示了玉米幼苗缺磷的响应机制。植物j . 2019; 97:947 - 69。gydF4y2Ba
- 21.gydF4y2Ba
关键词:玉米,叶片,光合系统,低氮胁迫,RNA-seq分析国际分子科学杂志。2017;18:1-12。gydF4y2Ba
- 22.gydF4y2Ba
罗波,唐华,刘华,顺宗S,张森,吴磊,等。整合meta分析和大规模玉米基因表达数据挖掘耐低氮基因。Euphytica。2015;206:117-31。gydF4y2Ba
- 23.gydF4y2Ba
zainin L, Zamboni A, Monte R, Tomasi N, Varanini Z, Cesco S等。转录组学分析强调了尿素和硝态氮在玉米根系获取氮素中的相互作用。植物生理学杂志。2015;56:52 - 48。gydF4y2Ba
- 24.gydF4y2Ba
他x,ma h,zhao x,nie s,li y,zhang z等。比较RNA-SEQ分析表明,玉米根部发育的调节网络控制基因的表达响应N应激。Plos一个。2016; 11:1-24。gydF4y2Ba
- 25.gydF4y2Ba
张宁,李鹏,李鹏,等。玉米巢式关联定位群体碳氮代谢的全基因组关联植物杂志。2015;168:575 - 83。gydF4y2Ba
- 26.gydF4y2Ba
Stelpflug SC, Sekhon RS, Vaillancourt B, Hirsch CN, Buell CR, de Leon N等。基于RNA测序的玉米基因表达图谱及其在探索根系发育中的应用。植物基因组。2016;9:1。gydF4y2Ba
- 27.gydF4y2Ba
关键词:小西,石山,beer MP, Inoue E, Kanno K, Yamaya T,等。两种胞质谷氨酰胺合成酶同工酶对拟南芥根系铵态氮同化的贡献。实验机器人。2017;68:613-25。gydF4y2Ba
- 28.gydF4y2Ba
谷氨酰胺合成酶同工酶(Cytosolic glutamine synthease同工酶)在低铵态氮条件下对拟南芥根系的铵态氮同化中起着重要作用。植物生理学杂志。2018;59:01 - 13。gydF4y2Ba
- 29.gydF4y2Ba
Wilusz JE, Sunwoo H, Spector DL。长链非编码RNA:来自RNA世界的功能惊喜。基因Dev。2009;23:1494 - 504。gydF4y2Ba
- 30.gydF4y2Ba
李丽,叶春涛,吴伟,等。玉米长链非编码rna的全基因组发现与鉴定。基因组医学杂志。2014;15:1-15。gydF4y2Ba
- 31。gydF4y2Ba
刘杰,王辉,蔡颖慧。植物长链非编码RNA转录组。植物生物学报2015;13:319-28。gydF4y2Ba
- 32.gydF4y2Ba
李建平,李永平,李永平,等。拟南芥中microrna159调控的gamyb样基因抑制生长并促进程序性细胞死亡。植物杂志。2010;154:757 - 71。gydF4y2Ba
- 33.gydF4y2Ba
Reyes JL, Chua NH。在拟南芥种子萌发过程中,ABA诱导miR159控制两个MYB因子的转录水平。植物j . 2007; 49:592 - 606。gydF4y2Ba
- 34.gydF4y2Ba
李毅,王刚,王明斌,黄国良。MYB33/65在拟南芥莲座中普遍存在miR159的抑制作用是强大的,不会被广泛的胁迫干扰。植物生物学杂志。2016;16:1-13。gydF4y2Bahttps://doi.org/10.1186/s12870-016-0867-4gydF4y2Ba.gydF4y2Ba
- 35.gydF4y2Ba
聂震,任震,王磊,苏胜,魏旭,张旭,等。玉米早期缺磷反应的microRNAs的全基因组鉴定。杂志。2016;157:161 - 74。gydF4y2Ba
- 36.gydF4y2Ba
方Y,谢克,熊L.保守的MiR164靶向NAC基因对水稻的抗旱性负面调节。J Exp Bot。2014; 65:2119-35。gydF4y2Ba
- 37.gydF4y2Ba
徐忠,钟山,李旭,李伟,罗斯坦SJ,张松,等。玉米叶片和根中低硝酸盐利用率的microrna全基因组识别。《公共科学图书馆•综合》。2011;6。gydF4y2Ba
- 38.gydF4y2Ba
Jun W,Junhong Z,Menghui H,Minhui Z,Zaikang T. MIR164及其靶基因NaC1响应于桦木荧光素的低硝酸盐可用性的表达分析。易川。2016; 38:155-62。gydF4y2Ba
- 39.gydF4y2Ba
张国栋,张国栋,张国栋,等。细胞分裂素通过双组分信号系统和叶绿体相关基因的激活调控黄体-叶绿体的转化。植物杂志。2016;172:464 - 78。gydF4y2Ba
- 40.gydF4y2Ba
张j,fengler ka,van hemert jl,gupta r,mongar n,sun j等。一种提高玉米产量的新型温泉QTL的鉴定与表征。植物Biotechnol J. 2019; 17:2272-85。gydF4y2Ba
- 41.gydF4y2Ba
Gong C, Li Z, Ramanujan K, Clay I, Zhang Y, Lemire-Brachat S, et al. .一种长链非编码RNA LncMyoD通过阻断imp2介导的mRNA翻译来调节骨骼肌分化。Dev细胞。2015;34:181 - 91。gydF4y2Bahttps://doi.org/10.1016/j.devcel.2015.05.009gydF4y2Ba.gydF4y2Ba
- 42.gydF4y2Ba
文忠,谭荣,张胜,Collins PJ,袁军,杜伟,等。整合GWAS和基因表达数据,研究大豆抗白霉功能特性。植物生物技术2018;16:25 - 35。gydF4y2Ba
- 43.gydF4y2Ba
关键词:玉米,根发育,全基因组关联分析,玉米abstract:BMC基因组学。2015;16。gydF4y2Ba
- 44.gydF4y2Ba
朱杰,Kaeppler SM, Lynch JP。差异磷供应条件下玉米侧根分枝和侧根长度qtl定位理论与应用,2005;111:688-95。gydF4y2Ba
- 45.gydF4y2Ba
李鹏,陈峰,蔡辉,刘杰,潘强,刘志,等。QTL分析表明玉米氮素利用效率与幼苗根系性状的遗传关系。实验机器人。2015;66:3175-88。gydF4y2Ba
- 46.gydF4y2Ba
关键词:长链非编码rna,核定位,干旱,盐胁迫植物杂志。2017;175:1321-36。gydF4y2Ba
- 47.gydF4y2Ba
张X,张H,Li L,Lan H,Ren Z,Liu D等。用基因组SNP标记表征西南玉米育种种质的人口结构和遗传多样性。BMC基因组学。2016; 17:1-16。gydF4y2Bahttps://doi.org/10.1186/s12864-016-3041-3gydF4y2Ba.gydF4y2Ba
- 48.gydF4y2Ba
Langmead B,Trapnell C,Pop M,Salzberg SL。超快和记忆高DNA序列对人类基因组的对准。基因组Biol。2009; 10。gydF4y2Ba
- 49.gydF4y2Ba
Trapnell C,Pachter L,Salzberg SL。Tophat:发现带RNA-SEQ的接头连接点。生物信息学。2009; 25:1105-11。gydF4y2Ba
- 50.gydF4y2Ba
Guttman M,Garber M,Levin JZ,Donaghey J,Robinson J,Adiconis X等人。错误:鼠标中细胞类型特异性转录组的AB初始重建揭示了Lincrnas的保守的多偏振结构。NAT。Biotechnol。2010; 28:503-10 NAT Biotechnol。2010; 28:756。gydF4y2Ba
- 51.gydF4y2Ba
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, Van Baren MJ等。转录本组装和定量的RNA-Seq揭示了在细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28:511-5。gydF4y2Bahttps://doi.org/10.1038/nbt.1621gydF4y2Ba.gydF4y2Ba
- 52。gydF4y2Ba
孔璐,张勇,叶志强,刘秀琴,赵树青,魏磊,等。CPC:利用序列特征和支持向量机评估转录本的蛋白质编码潜力。核酸杂志2007;345-9。gydF4y2Ba
- 53。gydF4y2Ba
贝特曼(Bateman A, Coin L, Durbin R, Finn RD, Hollich V, griffith - jones S, et al.)pam蛋白家族数据库。核酸研究2004;32(数据库ISS): 138-41。gydF4y2Ba
- 54。gydF4y2Ba
Burge SW, Daub J, Eberhardt R, Tate J, Barquist L, Nawrocki EP,等。Rfam 11.0: 10年的RNA家族。核酸杂志2013;41:26 26 - 32。gydF4y2Ba
- 55。gydF4y2Ba
Friedländer MR, MacKowiak SD, Li N, Chen W, Rajewsky N. MiRDeep2准确识别了7个动物分支中已知的和数百个新的microRNA基因。核酸杂志2012;40:37-52。gydF4y2Ba
- 56.gydF4y2Ba
温敏,沈勇,史胜,唐涛:一种面向下一代测序实验的microRNA进化分析平台。BMC生物信息学。2012;13。gydF4y2Ba
- 57.gydF4y2Ba
周L,陈继,李Z,李X,胡X,黄y等。微小RNA和MRNA的综合分析:MicroRNA位于XQ27.3与透明细胞肾细胞癌相关联。Plos一个。2010; 5; e15224。gydF4y2Ba
- 58.gydF4y2Ba
吴宏杰,马玉坤,陈涛,王敏,王兴军。PsRobot:一个基于网络的植物小RNA元分析工具箱。核酸杂志2012;40:22-8。gydF4y2Ba
- 59.gydF4y2Ba
桑切斯,达琳Lonjas。双单倍体外源渐渗系对玉米氮素利用效率的分子和表型研究。2017;15409。gydF4y2Ba
- 60。gydF4y2Ba
刘凯,Muse SV。PowerMaker:用于基因标记分析的集成分析环境。生物信息学。2005;21:2128-9。gydF4y2Ba
- 61。gydF4y2Ba
关键词:种群结构,多位点基因型数据,遗传算法遗传学。2000;155:945-59。gydF4y2Ba
- 62.gydF4y2Ba
张志明,张志明,张志明。TASSEL:用于在不同样本中进行复杂性状关联映射的软件。生物信息学。2007;23:2633-5。gydF4y2Ba
致谢gydF4y2Ba
感谢李文雪(中国农业科学院作物科学研究所,作物分子育种国家工程实验室)对定点诱变实验的评论。我们也要感谢北京诺金有限公司在RNA-Seq实验和数据分析方面的帮助。gydF4y2Ba
资金gydF4y2Ba
本研究由国家科技支撑计划(no . 2016YFD0100707, no . 2018YFD0200707)资助。四川省科技支撑项目(20121yfyz0027)。国家自然科学基金资助项目(31971955);gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
SG:设计了实验。PM,BL和XZ:进行实验设计和数据分析。ZC,XH,Hz,BL,DL,LW,SG,DG和ZS:执行了数据收集。PM和SG:起草稿件。所有作者阅读并认可的终稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意事项gydF4y2Ba
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
叶和根中HN和LN条件下差异mrna (a, b)、TUCPs (c, d)和miRNAs (e, f)的火山图。gydF4y2Ba图S2。gydF4y2BaTPM(每百万转录本)(a)和18- 30-nt小rna的长度分布(b)。12个文库的长度分布集中,峰值分别为24-nt。gydF4y2Ba图S3。gydF4y2BaHN和LN条件下幼苗mRNA和miRNA的表达谱叶片和根中所有mrna (a)和miRNAs (c)表达的聚类热图。差异表达mrna (b)和mirna (d)的VEEN分析。gydF4y2Ba图S4。gydF4y2Ba玉米幼苗中鉴定的lncrna特征。gydF4y2Ba图S5。gydF4y2Baln响应lncrna的功能分析。丰富的京都基因百科全书(KEGG)途径的叶片(a, c)和根(b, d)。gydF4y2Ba图S6。gydF4y2Ba通过RNA-Seq和GWAS结合,在LN (a)和LNTI (b)条件下检测到45个和232个一致的候选基因。gydF4y2Ba
附加文件2:表S1。gydF4y2Ba
从RNA-Seq中鉴定出的已知miRNAs和新miRNAs。gydF4y2Ba表S2。gydF4y2Ba通过共同位置和共同表达预测的LNCRNA的目标。gydF4y2Ba表S3。gydF4y2BaLncRNA-mRNA双gydF4y2BaCIS.gydF4y2Ba代理。gydF4y2Ba表S4。gydF4y2BaLncRNA-mRNA双gydF4y2Ba反式gydF4y2Ba代理。gydF4y2Ba表S5。gydF4y2BaLNCRNA的去分析。gydF4y2Ba表S6。gydF4y2Balncrna的KEGG分析。gydF4y2Ba表S7。gydF4y2BaGWAS和RNA-Seq鉴定的候选基因。gydF4y2Ba表S8。gydF4y2Ba所有17个性状之间的Pearson (r)相关性。gydF4y2Ba表S9。gydF4y2Balncrna的靶点具有显著的多根性状snp。gydF4y2Ba表S10。gydF4y2Ba本研究所用引物序列信息。gydF4y2Ba表S11。gydF4y2Ba通过手动和WinRizo收集性状的缩写和描述。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.“创作共用公共领域”豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非另有用入数据的信用额度。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
马平,张学军,罗斌。gydF4y2Ba等等。gydF4y2Ba转录组学和全基因组关联研究揭示了长链非编码rna对玉米氮缺乏的响应。gydF4y2BaBMC植物杂志gydF4y2Ba21日,gydF4y2Ba93(2021)。https://doi.org/10.1186/s12870-021-02847-4gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-021-02847-4gydF4y2Ba
关键词gydF4y2Ba
- 玉米gydF4y2Ba
- 氮利用效率gydF4y2Ba
- RNA-SeqgydF4y2Ba
- 全基因组关联研究gydF4y2Ba
- LncRNAgydF4y2Ba
- Co-expressiongydF4y2Ba