摘要
背景
识别基因座和解剖遗传建筑潜在的小麦产量和质量相关性状对小麦滋生至关重要。使用高密度90K SNP阵列进行基因组 - 宽协会研究,以分析543个面包小麦品种的产量和质量相关性状。
结果
在21条染色体上共有11140个多态性snp,其中270个显著snp与25个产量和质量相关性状相关。此外,根据BLUP数据,在显著SNPs附近检测到638个假定的候选基因,包括3个(TraesCS7A01G482000,TraesCS4B01G343700,和纸盘6B01G295400)与每穗小穗数、第一节间直径和粒体积有关。利用RNA-seq分析获得的分期和组织特异性基因表达数据,进一步分析了这三个候选基因。这些基因是提高小麦产量和品质相关性状的有前途的候选基因。
结论
本研究结果为了解小麦产量和品质的遗传基础提供了新的思路。此外,本研究所检测到的标记可用于小麦育种中的标记辅助选择。
背景
面包小麦(小麦L.),这是一种广泛栽培的谷物作物,这是高度适应性的,在人类饮食中提供了总热量的约21%和23%的蛋白质(www.fao.org/faostat/en.).作为全球约35-40%人口的主食,小麦是一种良好的营养来源,并具有独特的麸质特性,可用于生产多种食品[1].全球人口的增长和世界上许多人生活水平的提高迫使育种者不断致力于培育优质和高产的小麦新品种[2].
产量和品质是复杂的性状。此外,面包小麦有限的遗传多样性导致育种瓶颈,应用传统育种方法使小麦产量和品质逐步提高[3.]. 基因组测序和基于高通量芯片的基因分型平台对于阐明小麦产量潜力和品质调控机制以及改进育种方法至关重要[4]. 多个SNP阵列(例如,9 K、 35个 K、 90个 K、 660个 K、 和820 K) 最近发展起来的。它们已被用于分析双亲群体和确定控制产量和品质相关性状的基因座(QTL)[5,6,7,8,9].然而,传统的QTL定位方法通常基于亲本群体的特定特征,费时费力[10].
GWAS在育种计划中很常见,因为与其他研究方法相比,它们在分析各种环境条件下的复杂性状时效率更高,所需的努力更少[11].具体地,Gwass对于在小麦中检测产量相关的基因座,包括植物高度(pH),每个穗(KNP)和千粒重量(TGW)[12,13]. 然而,由于对许多种子的需要以及对某些品质性状进行评估所需的大量时间,有关小麦品质性状(如湿面筋含量(WGC)和籽粒蛋白质含量(GPC))的gwas相对较少[14,15].抗倒伏是影响小麦产量和品质的一个重要因素,而利用GWAS研究抗倒伏的报道较少。
对于GWAS来说,面板的大小和多样性很重要,因为小面板和大连锁不平衡(LD)块可能导致假阳性关联的识别[16].在小麦方面,只有少数产量和品质性状的GWAS涉及大量的自然群体和SNP芯片。此外,小麦在中国已有4000多年的种植历史,目前已在10个主要农业生态区种植[17].由于漫长的进化期,中国小麦种质在不同地区的人工中选择,具有区域遗传特征[18,19]. 因此,不同地区小麦品种改良的目标和要求有很大差异。因此,我们对543个有代表性的面包小麦品种(包括10个省的531个中国小麦品种和90个小麦品种)进行了小麦产量和品质的GWAS分析 在六种环境中进行表型分析的K-SNP阵列。
本研究的目的是鉴定与小麦产量和品质显著相关的稳定位点和候选基因。研究结果可为揭示水稻产量和品质的遗传基础提供依据。所鉴定的相应SNP标记为小麦优质高产新品种的选育提供了依据。
结果
表型变异和相关分析
543个小麦品系在6个环境下的生长发育相关性状、产量相关性状和品质相关性状的表型数据列于表中S1.根据遗传力、变异范围、均数、标准差和变异系数确定基因型间的表型变异。E5环境下各性状变异明显,其中推力变异系数(TH)为49.28%。表格S2提供此综合分析的估计相关系数。广义遗传力(h2)对于大多数特征约为0.80,检测到pH(0.92)和PET(0.70)检测到最高和最低的遗传症。因此,大多数特征是稳定的并且主要由遗传因子决定。湿麸质含量(WGC)和谷物蛋白质含量(GPC)之间的相关系数最高(0.970),但TGW和GPR(0.919)之间的相对较高,湿谷蛋白含量(WGC)和面粉含量(FC)(0.842).
全基因组关联研究
90年代 利用81587个单核苷酸多态性的K小麦iSelect单核苷酸多态性阵列进行基因分型。经过一个质量控制步骤后,11140个SNP标记保留用于关联图谱[20.].鉴定了与产量和质量特征相关的270个重要的SNP基因座(表S3,图。S1).这些snp分布在21条染色体上,占表型变异的1.27 ~ 8.47%。A、B和D亚基因组中分别有94、139和37个SNPs(见表1)S3).在这些SNP的中,在染色体1b,2a,2b,2d,3a,3b,3d,4b,4d,5b,5d,6b,6d,7a和7b上检测到与两个或更多个性状相关的42个与两种或更多种性状相关的磷酸杆菌基因座。公共基因座(表1).
生长发育相关性状
在1B、1D、2A、2B、2D、3A、3B、4B、5A、5B、5D、6B、6D、7A和7B染色体上共检测到28个与旗叶长度有关的显著SNP位点,占表型变异的2.44-4.12%。旗叶宽度(FLW)在13条染色体(1B、2B、2D、3A、3B、4A、4B、5B、5D、6B、6D、7A和7B)上检测到33个显著SNP位点,占表型变异的2.52 ~ 6.92%。在旗叶面积16条染色体(1B、2A、2B、2D、3A、3B、3D、4A、4B、4D、5B、5D、6B、6D、7A和7B)上检测到40个显著SNP位点,占表型变异的2.58 ~ 6.37%。旗叶角(FA)在1B、2B、3A、3B、4B、4D、5A、6A和6D 9条染色体上检测到13个显著SNP位点,占表型变异的2.06-3.35%。在68个旗叶相关性状的snp位点中,11个多效位点与3个性状相关。
对于最大分蘖数(MTN),在14条染色体(1A、1B、2B、2D、3A、3B、4B、4D、5B、5D、6B、6D、7A和7B)上检测到37个显著的SNP位点,解释了约1.80–5.32%的表型变异。抽穗期(HD)的5个SNP位点分布在2A和5A染色体上,占表型变异的2.73~5.94%。在2B、3B、6A和7B染色体上检测到7个显著的成熟期SNP位点,占表型变异的2.70-3.75%。在2A、2B、2D和7B染色体上检测到7个显著的灌浆期SNP位点,占表型变异的1.37~2.99%。在1A、1B、2A、2B、4A、4B和5B染色体上检测到9个与籽粒灌浆速率相关的SNP位点,占表型变异的2.39~3.59%。在1B、2B、3A、3B、4B和6D染色体上检测到8个显著的单核苷酸多态性位点,解释了2.29-3.59%的表型变异。
对于pH,在10个染色体(1a,1b,2b,3a,3b,4a,4b,4d,5a和6b)上检测到20个重要的SNP基因座,并解释了表型变异的约1.96-4.42%。对于第一个(FD)的直径,在九个染色体(1A,1B,2B,3D,4A,4B,5B,6A和7A上)检测16个重要的SNP基因座,并解释了约1.53-4.85%的表型变化。关于第一节末(FIL)的长度,在八条染色体(1A,1B,2A,3A,3B,4D,6B和7A)上检测17个重要的SNP基因座,并解释了表型变异的约2.13-5.76%。对于第二个子体(SD)的直径,在八个染色体(2a,2b,2d,3b,4b,5b,6b和7b)上检测到六种环境中鉴定的18个重要的SNP基因座,解释约2.15-5.02%表型变异。对于第二个子体(SIL)的长度,在七条染色体(1A,1D,4B,4D,5B,7A和7D)上检测到15个有效的SNP基因座,并解释了表型变异的约2.57-5.74%。对于TH,在15染色体(1b,2b,2d,3a,3b,4a,4b,4d,5b,5d,6a,6b,6d,7a和7b)上检测到45个重要的SNP基因座,占大约2.5-7.13%的表型变异。其中,值得注意的是,染色体4B上的三个基因座与FD和SD相关联。
产量相关性状
穗长(SL) 9个显著SNP位点分布在1B、3A、3D、4B、6D、7A和7B 7条染色体上,占表型变异的2.42-4.33%。在穗粒数(SNS)方面,在2A、2B、2D、4B、4D和7A 6条染色体上检测到14个显著SNP位点,解释了约1.27 ~ 4.02%的表型变异。9个显著SNP位点分别位于1A、3A、4A、4B和5D染色体上,占表型变异的2.58 ~ 4.32%。在分蘖百分率(PET)方面,在10条染色体(1A、2A、3A、4D、5B、5D、6B、6D、7A和7B)上检测到11个显著SNP位点,解释了约2.74 ~ 3.55%的表型变异。每亩穗数(EPM)在1B、2A、4B、4D、5A、5B、7A和7B 8条染色体上检测到17个显著SNP位点,占表型变异的2.33 ~ 8.47%。
质量相关的特征
在籽粒体积(GV) 5条染色体(1B、3B、3D、6A和6B)上检测到17个显著SNP位点,占表型变异的2.52 ~ 5.01%。在1A和3D染色体上检测到的2个GPC显著SNP位点占表型变异的2.35 ~ 2.53%。在3D和5D染色体上检测到2个显著的WGC SNP位点,占表型变异的2.43 ~ 2.90%。在1B、4B、5A、5B、6A和7B染色体上检测到10个面粉含量(FC)显著SNP位点,占表型变异的1.80 ~ 3.27%。
推测候选基因分析和表达数据
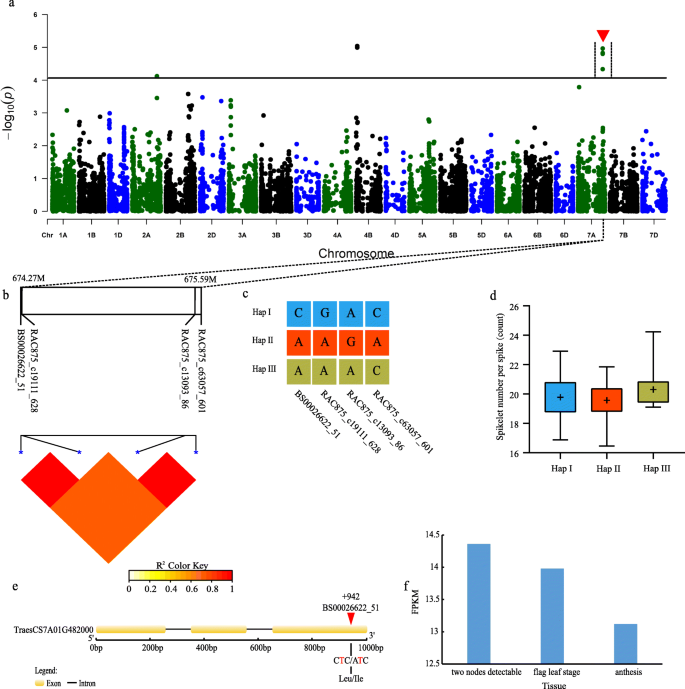
在我们的研究中,分别位于亚基因组A、B和D中相关SNP两侧的200、380和600kb序列被确定为潜在的候选基因区域。通过筛选最近发布的基因组序列(IWGSC RefSeq v1.0)中的注释基因,共鉴定了638个基于BLUP数据的重要SNP侧翼区域的假定候选基因(表1)S4). 我们随后对以下三个关键性状进行了单倍型和表达分析:SNS(图。1)、FD(无花果。2),和GV(图。3.).
关于与SNS相关的SNPs,在染色体7A上检测到一个包含四个显著相关SNPs的关联峰(图。1a, b).根据7A染色体上的SNPs,鉴定出3个单倍型,单倍型II的SNS均值(19.57±1.14)显著低于单倍型I的SNS均值(19.78±1.33)和单倍型III的SNS均值(20.31±1.43)(图1)。1C,D)。四个重要的SNP(BS0002662222_51)中的一种位于基因的第三个外显子的942bpTraesCS7A01G482000.该位点在4种环境和BLUP模型中均有检测。在相关基因组区域的这个位置的SNP (C/A)导致氨基酸从亮氨酸变为异亮氨酸(图。1e) 是的。表达TraesCS7A01G482000根据棘突的RNA序列分析,当检测到两个节点时,候选基因显著上调(图。1f)。
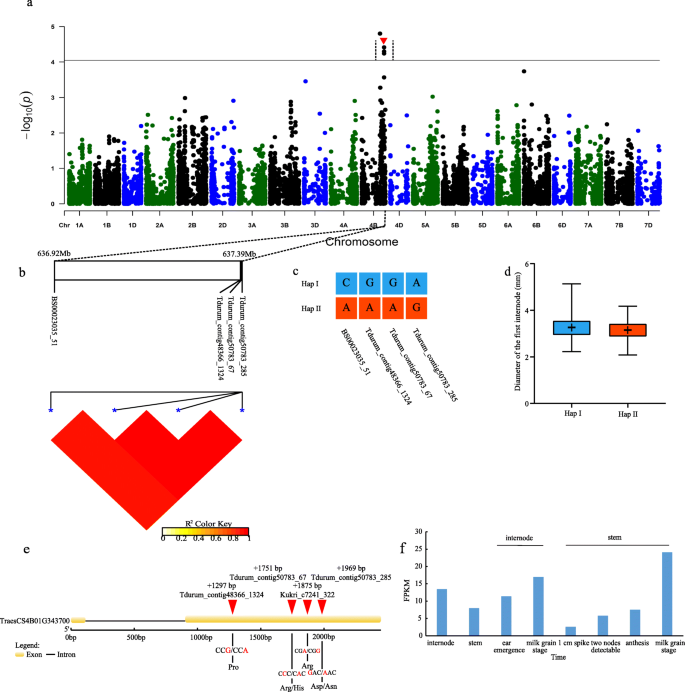
在与FD相关联的SNP中,在染色体4b上分布四种类似的SNP,并且在增孔模型中并在至少一个环境中检测到所有的SNPs(图。2a).这4个SNPs分别为BS00023035_51、Tdurum_contig48366_1324、Tdurum_contig50783_67和Tdurum_contig50783_285, 4个SNPs被分离到470.67 kb(图。2b)。因为他们的亲缘关系密切,显著相关,这些snp是用于随后的单体型分析,显示两个不同的单(I和II)。共有301个小麦材料包括单体型,我的FD 3.25±0.48厘米,而208年小麦品种包括单体型二世,FD为3.17±0.45 cm。2c、 (d)。单倍型I的FD显著大于单倍型II,说明单倍型I小麦品种比单倍型II小麦品种倒伏的可能性小。此外,四个有意义的单核苷酸多态性中有三个在小鼠CDS中被检测到TraesCS4B01G343700.该基因包含两个外显子,CDS包含2450 bp。位于第二外显子的两个snp导致氨基酸从精氨酸转变为组氨酸,从天冬氨酸转变为天冬酰胺(图。2e)。TraesCS4B01G343700在小麦生长发育过程中,茎和节间的表达量增加,在乳汁期达到高峰。这表明该基因有助于调控小麦节间的生长发育。2f)。
GV的变化与6B染色体1.95 Mb基因组区域(528.62-530.57 Mb)的一组snp高度相关(图1)。3.a、 (b)。这些位点在多等位基因和BLUP环境中被检测到。根据基因分型结果,在407个小麦品系中检测到3个单倍型。更具体地说,在339份、40份和28份材料中分别检测到单倍型I、II和III。单倍型Ⅱ平均GV(789.88) ± 8.58)显著低于单倍型I(798.50) ± 8.29)和III(799.19 ± 4.60)(图。3.c、 (d)。此外,在3315处检测到四个重要的单核苷酸多态性中的一个 英国石油公司纸盘6B01G295400编码序列(CDS)。SNP (G/A)导致氨基酸的变化,从精氨酸到组氨酸(图。3.e) 是的。RNA-seq数据显示,该基因在6℃时在发育中的种子中高表达 花后天数(图。3.f)。
讨论
GWAS已经被用来分析包括水稻在内的各种作物的产量和质量[21,大豆22],棉花[23],还有高粱[24]. 研究人群的大小与关联分析的准确性密切相关。Long等人[25据报道,增加人口大小增加了罕见等位基因的个体的数量,从而提高了罕见等位基因定位的准确性和效率。因此,我们的GWA用于产量和质量相关的特性涉及小麦90克SNP阵列,用于多种环境中的543个小麦牧场。
我们完成了一个大范围的比较结果,我们的研究和那些以前的调查。对于PH,一个位点(RAC875_rep_c105718_585)在四个环境中根据BLUP数据被识别,这是一个来自Li等人描述的QTL (qph - caas- 4ds)的LD。26),该QTL在中国两个面包小麦群体中也得到了稳定的鉴定。Gao等人在4种环境中较早检测到4A染色体标记Kukri_rep_c106490_583和RAC875_c29282_566之间的KNPS QTL (QKNS.caas-4AL) [13].与我们研究中透露的KNP相关的两个站点(Excalibur_C9370_966和WSNP_RA_REP_C702333_67968353位于此QTL中。高等人。还报告了一个稳定的QTL(Qtkw.caas-4bs.1),用于在标记Bobwhite_c162_145和Kukri_c66885_230上的Tgw染色体4b。在本研究中,在染色体4B上的两个标记之间检测到预测的TGW相关的SNP基因座(TDURUM_CONTIG97386_207)。此外,Taapo-A1 / WAPO-A1(纸盘7A02G481600)通过图位克隆鉴定为SNS候选基因[27,28,29]. 根据BLUP数据,在四种环境中鉴定出与7A染色体SNS相关的两个基因座(BS00026622_51和RAC875_c19111_628)在1个LD以内TaAPO-A1. Dobrovolskaya等人[30.]克隆的WFZP.(小麦卷曲的圆锥花序)染色体2D上与SNS相关的基因。该基因位于本研究发现的一个稳定的sns相关位点(gene -0787_85)的一个LD内。对于与品质相关的性状,我们的GPC位点(GENE-0411_807)与Kumar等人定位于1A染色体的GPC QTL重叠[31]. Li等人[32]在7B染色体上发现了一个与淀粉粒相关的基因座(IWB41869),与在同一染色体上检测到的FC基因座(RAC875_c26057_370)接近。
此外,我们还发现了影响特定基因的新的稳定snp,并已在多种环境中检测到。例如,TraesCS7A01G482000预测其编码一个含卤酸脱卤酶(HAD)样水解酶结构域的蛋白。在大米中,过度表达oShad1.据报道,在缺磷的转基因植株中,在穗发育早期,磷酸酶活性增强,总磷和可溶性磷含量增加。提高磷的吸收速率可以促进小穗的形成[33,34]. 在线小麦基因表达数据库中的RNA-seq数据表明,当检测到两个节点时,该基因高度表达,这与小麦小穗发育的关键时期相吻合。
抗倒伏性是另一个重要的产量相关性状。在4B染色体上发现了4个显著的节间直径snp。这些SNPs在内部被检测到TraesCS4B01G343700. 在拟南芥中,AtVPS25型(空泡蛋白分选相关蛋白)通过影响特定生长素相关基因的表达来调节生长素的生物合成。小麦植株生长素含量的增加可能导致茎粗增加和抗倒伏能力增强[35,36,37]. 基因转录组水平分析TraesCS4B01G343700在不同的组织和发育阶段证明了该基因在茎和节间发育阶段高表达。表达水平在乳粒期达到高峰,说明乳粒期的变化对小麦抗倒伏有重要意义。
关于质量相关性状GV,我们确定纸盘6B01G295400,作为候选基因。在米饭里,奥斯陆-L1(Lissencephaly型-1样1蛋白)对于雄性配子形成是重要的,对于该基因的突变导致异常发育。另外,通过该基因编码的蛋白质影响谷物特征,与花卉发育和籽粒填充阶段密切相关[38].对公开的小麦RNA-SEQ数据的分析显示,该基因在显影种子中高度表达,特别是在第6天至第14天的淀粉胚乳中,并显示出逐渐降低的趋势。因此,该基因对于早期籽粒灌装阶段很重要。
结论
本研究以小麦90 K SNP序列为基础,对543份主要小麦材料的产量和品质相关性状进行GWAS分析。对所得数据进行分析,以确定相关SNP位点和候选基因。我们目前正在为重要的位点开发竞争性等位基因特异性PCR标记。这些标记将使研究人员和育种人员能够有效地将等位基因转入优质小麦基因型[39].此外,本文描述的候选基因的更全面的特性将有助于我们对调控小麦产量和品质的分子机制的理解。
方法
植物材料和实验设计
以543个基因型(包括品种、区域试验系和引进亲本系)的面包小麦为试材,详细资料已在上一篇论文中发表[20.]. 在这两个生长季节,河北省有三个地方生长着小麦。地理位置为保定(115.5°48′,38°85′),沧州(116°80′,38°58′),邢台(118°9′,39°42′)。六个环境分别为:2016保定(E1)、2016沧州(E2)、2016邢台(E3)、2017保定(E4)、2017沧州(E5)和2017邢台(E6)。现场试验采用完全随机设计。每个地块包含三个1.5 m行,0.25 我在两行之间。株距2.5左右 厘米。小麦是按照当地的正常做法种植的。
表型评价
测定了25个表型性状,包括生长发育相关性状(flll、FLW、FLA、FA、MTN、HD、MP、GFP、GFR、TGW、PH、FD、FIL、SD、SIL和TH),产量相关性状(SL、SNS、KNPS、PET和EPM)和品质相关性状(GV、GPC、WGC和FC)。每个性状记录的数据汇总在表中S1. 在所有6种环境中对表型性状进行了评估。每个环境的表型数据和BLUP数据用于全基因组关联分析。
表型数据分析
使用SPSS 25.0软件完成了表型数据的描述性统计分析和相关分析。Pearson的相关系数被计算为评估特征之间的相关性。
SNP基因分型
小麦90 利用K Illumina Infinium单核苷酸多态性阵列对543份材料的关联面板进行基因分型。使用Illumina BeadStudio基因分型软件(Illumina,美国加利福尼亚州圣地亚哥)对SNP数据进行聚类并自动调用。对数据进行过滤,去除检测率小于0.1和次要等位基因频率小于0.05的等位基因[12].另外,剔除损失率大于10%和杂合频率大于20%的样品。
全基因组关联分析
既往研究对种群结构、亲属关系和LD进行了分析[20.].在目前的研究中,我们使用Gapit包完成了GWA [40]在R软件中。混合线性模型程序(Q + k)[41.],随着人口分层结果和作为协变性的血清关系,用于最小化误报[40].的P基于标记数计算值阈值(P = 1/n号,n = total number of SNPs used) as described by Li et al. [42.]. 关于GWAS结果P取值为1/ 11140(−log . log)10P= 4.05)作为识别显著SNPs的标准。
候选基因预测及表达分析
“中国之春”基因组数据库(IWGSC RefSeq v1.0,http://www.wheatgenome.org/)用于预测全基因组关联分析显示的重要位点的候选基因。根据染色体组间LD衰减距离的差异,确定了显著位点附近的候选基因。利用在线小麦基因表达数据库(http://www.wheat-expression.com/).该数据库包括850个小麦RNA测序样品和注释基因组,揭示了不同组织,发育阶段和品种同源表达水平之间的相似性和差异[33,43.].
数据和材料的可用性
在本研究中产生或分析的所有数据均包含在本文(及其补充信息文件)中,或应通信作者的合理要求向其提供。
缩写
- 按照项目:
-
每亩穗数
- F A:
-
旗叶角度
- 舰队指挥官:
-
面粉的内容
- FD:
-
第一个节间的直径
- 文件:
-
第一个节间的长度
- FLA:
-
旗叶面积
- 飞行高度:
-
旗叶长度
- FLW:
-
旗叶宽度
- 绿色荧光蛋白:
-
灌浆期
- 肾小球滤过率(GFR):
-
籽粒灌浆速率
- GPC:
-
籽粒蛋白质含量
- 全球价值:
-
颗粒体积
- 高清:
-
标题日期
- KNPS:
-
每穗核数
- 身份证号码:
-
连锁不平衡
- 议员:
-
成熟的时期
- MTN编号:
-
最大分蘖数
- 宠物:
-
承载钉分蘖的百分比
- 酸碱度:
-
植物高度
- QTL:
-
定量特质基因座
- SD:
-
第2节间的直径
- SIL:
-
第二节间长度
- SL公司:
-
穗长
- 社交网站:
-
每穗穗尖峰号码
- TGW:
-
千粒重量
- TH:
-
猛推
- 黄金协会:
-
湿面筋含量
参考
- 1.
Shewry PR, Halford NG。谷物种子贮藏蛋白质:结构、特性和在谷物利用中的作用。实验机器人,2002;https://doi.org/10.1093/jexbot/53.370.947.
- 2.
柯蒂斯·T,哈尔福德·吴。粮食安全:提高小麦产量的挑战和不损害粮食安全的重要性。安应用生物学。2014;164(3):354–72.https://doi.org/10.1111/aab.12108.
- 3.
Edae EA, Byrne PF, Haley SD, Lopes MS, Reynolds MP。不同水分条件下春小麦产量及产量构成因素的全基因组关联定位中国生物医学工程学报,2014;https://doi.org/10.1007/s00122-013-2257-8.
- 4.
黄建平,李建平,李建平,等。六倍体小麦基因组测序技术的研究进展。正确答案:Thomas M, Viktor K, editors。瑞斯出版社出版。德国:学术;2019.p.49 - 63。
- 5。
Azadi A, Mardi M, Hervan EM, Mohammadi SA, Moradi F, Tabatabaee MT, Pirseyedi SM, Ebrahimi M, Fayaz F, Kazemi M, Ashkani S, Nakhoda B, Mohammadi- nejad G.正常和盐胁迫条件下面包小麦产量及其产量构成因素的QTL定位(小麦l .)。Plant Mol Biol Rep. 2015;33(1): 102-20。https://doi.org/10.1007/s11105-014-0726-0.
- 6。
崔芙、李杰、丁安、赵总、王丽、王旭、李SS、鲍一格、李新芳、冯德生、孔瑞尔、王海格。小麦穗长和节间长与株高关系的条件QTL定位。理论应用基因。2011;122(8):1517–36.https://doi.org/10.1007/s00122-011-1551-6.
- 7。
崔芙、赵总、丁安、李杰、王丽、李新芳、包一格、李总、王浩。利用3个相关小麦RIL群体构建综合连锁图谱及产量相关性状QTL定位。理论应用基因。2014;127(3):659–75.https://doi.org/10.1007/s00122-013-2249-8.
- 8.
Cuthbert JL,Somers DJ,Brlé-Babel Al,Brown Pd,Crow GH,遗传学A.春小麦产量和产量组分的定量特性基因座的分子映射(小麦l .)。Al Appl Genet。2008; 117(4):595-608。https://doi.org/10.1007/s00122-008-0804-5.
- 9.
贾希,万浩,杨淑,张梓,马梓。我国小麦育种重点亲本重组自交系群体产量相关性状的遗传分析。理论应用基因。2013;126(8):2123–39.https://doi.org/10.1007/s00122-013-2123-8.
- 10
Sukumaran S, Dreisigacker S, Lopes M, Chavez P, Reynolds议员。温带灌溉条件下优质春小麦群体籽粒产量及相关性状的全基因组关联研究中国生物医学工程学报,2015;https://doi.org/10.1007/s00122-014-2435-3.
- 11.
George AW,Cavanagh CJT,遗传学A。植物全基因组关联图谱。理论应用基因。2015;128(6):1163–74.https://doi.org/10.1007/s00122-015-2497-x.
- 12.
李峰,陈by,徐凯,吴建峰,宋伟林,Bancroft I, Harper AL, Trick M,刘世英,高GZ。全基因组关联研究分析了油菜种子重量和质量的遗传结构(芸苔栗鸟L.)。DNA Res.2014年;21:355–67.
- 13.
高福明,文伟,刘建东,拉希德·阿,尹光辉,夏晓春,吴学祥,何志华。中国小麦杂交周8425B/中国春季产量组成、株高及产量相关生理性状QTL的全基因组连锁定位植物科学与技术,2015;
- 14.
陈杰,张文,赵cj,lv gg,太阳cw,潘yb,郭xy,陈文六种品质特征的基因组关联研究揭示了联想焦油13L1基因用面粉颜色在中国面包小麦。植物Biotechnol J. 2019; 17(11):2106-22。https://doi.org/10.1111/pbi.13126.
- 15.
关键词:小麦,产量,品质相关性状,全基因组关联分析摩尔品种。2017;38:1。
- 16。
Turuspekov Y、Baibulatova A、Yermekbayev K、Tokhetova L、Chudinov V、Sereda G、Ganal M、Griffiths S、Abugalieva S。春小麦生育期和产量构成因素的GWAS分析(小麦在哈萨克斯坦的三个地区收获。BMC植物生物学。2017;17:190.
- 17。
庄QS。中国小麦改良与系谱分析。北京:农业出版社;2003年。379–82.
- 18。
郝茜、董YC、王LF、游GX、张HN、葛HM、贾JZ、张XY。中国小麦遗传资源的遗传多样性与核心种质的构建。金牛。2008;53:1518–26.
- 19.
何志海,辛志勇,黄国珍。中国小麦育种的历史。神经学报,2001;523:805-13。
- 20
李赵Y, JH,赵RL,徐K,小岁,张SH,田JH杨XJ。全基因组关联研究揭示了小麦抗寒性的遗传基础。摩尔繁殖。2020;40(4):36。https://doi.org/10.1007/s11032-020-01115-x.
- 21
Yano K,Yamamoto E,Aya K,Takeuchi H,Lo PC,Hu L,Yamasaki M,Yoshida S,Kitano H,Hirano K,Matsuoka M.使用全基因组测序的基因组 - 范围协会迅速识别影响农艺性状的新基因在米饭中。NAT Genet。2016; 48(8):927-34。https://doi.org/10.1038/ng.3596.
- 22
方C、马英明、吴世伟、刘Z、王Z、杨R、胡GH、周ZK、于H、张M。全基因组关联研究剖析了大豆农艺性状的遗传网络。基因组生物学。2017;18(1):161.https://doi.org/10.1186/s13059-017-1289-9.
- 23.
孙志伟、王新芳、刘志伟、顾QS、张勇、李志克、柯海峰、杨杰、吴建华、吴立群、张国荣、张赛、马志。一项全基因组关联研究揭示了陆地棉产量相关性状的新的基因组区域和候选基因。理论应用基因。2018;131(11):2413–25.https://doi.org/10.1007/s00122-018-3162-y.
- 24.
关键词:高粱,籽粒产量,全基因组关联,遗传多样性植物基因组。2016;9:17。
- 25.
长广告,兰利克。关联研究的力量检测候选遗传基因素对复杂性状的变异的贡献。Genome Res。1999; 9(8):720-31。
- 26.
李FJ,Wen W,He Zh,Liu Jd,Jin H,Cao Sh,Geng HW,Yan J,Zhang PZ,WAN YX,夏XC。使用高密度SNP标记的三种中草小麦群中产量相关性状的基因组联动映射。Al Appl Genet。2018; 131(9):1903-24。https://doi.org/10.1007/s00122-018-3122-6.
- 27。
kuzay s,xu yf,张jl,katz a,pearce s,su zq,弗雷泽m,安德森ja,棕色guedira g,dewitt n,peters haugrud a,faris jd,akhunov e,bai gh,dubcovsky J.识别通过高分辨率遗传映射,小麦染色体臂7AL上每穗穗数的QTL候选基因。Al Appl Genet。2019; 132(9):2689-705。https://doi.org/10.1007/s00122-019-03382-5.
- 28。
Muqaddasi QH, Brassac J, Koppolu R, Plieske J, Ganal MW, Roder MS. TaAPO-A1,水稻异常穗型组织1的同源系,与欧洲优质六倍体冬小麦每穗总小穗数相关(小麦l .)品种。Sci众议员2019;9:13853。
- 29。
Voss-Fels KP,Keeble-Gagnere G,Hickey Lt,Tibbits J,Nagornyy S,Hayden Mj,Pasam RK,康德S,Friedt W,Snowdon RJ,Apples R,Wittkop B.每个Rachis rachis节点的高分辨率映射,小麦籽粒产量组分的临界决定因素。Al Appl Genet。2019; 132(9):2707-19。https://doi.org/10.1007/s00122-019-03383-4.
- 30
Dobrovolskaya O, Pont C, Sibout R, Martinek P, Badaeva E, Murat F, Chosson A, Watanabe N, Prat E, Gautier N, Gautier V, Poncet C, Orlov YL, Krasnikov AA, Berges H, Salina E, Laikova L, Salse J. FRIZZY PANICLE在面包小麦中产生额外的小穗。植物杂志。2015;167(1):189 - 99。https://doi.org/10.1104/pp.114.250043.
- 31
关键词:小麦,品质,产量,全基因组遗传分析,粒形,粒大小《公共科学图书馆•综合》。2019;14 (9):e0221826。https://doi.org/10.1371/journal.pone.0221826.
- 32
李JY,拉希德A,郭Q,董Y,刘JD,夏XC,张Y,何ZH。普通小麦淀粉粒径分布的全基因组关联图谱。谷物科学杂志。2017;77:211–8.https://doi.org/10.1016/j.jcs.2017.08.016.
- 33
expVIP:一个可定制的RNA-seq数据分析和可视化平台。植物杂志。2016;170(4):2172 - 86。https://doi.org/10.1104/pp.15.01667.
- 34
Pandey BK, Mehra P, Verma L, Bhadouria J, Giri J。oShad1.,一种类似于卤酸脱卤酶的APase,增强了磷酸盐的积累。植物杂志。2017;174(4):2316 - 32。https://doi.org/10.1104/pp.17.00571.
- 35.
郭MM,陈M,刘RB,马YZ,李LC,许ZS,张XH,西北A。空泡蛋白分选AtVPS25型调节拟南芥的生长素反应。中国农业科学,2014;17:3510-2。
- 36.
马清明,徐云云,赵马,宋学勇,裴永海。玉米茎秆抗倒伏相关生理生化指标及关键酶基因的表达分析植物生理学学报(英文版);
- 37.
任明林,刘文阁,刘涛,杜永利,杨文英。遮荫胁迫下大豆茎形态建成的转录组分析。作物学报,2016;42(9):1319。https://doi.org/10.3724/sp.j.1006.2016.01319.
- 38.
关键词:lis - l1,半脑畸形,半脑-1-like蛋白,转录因子WD40水稻的株高和雄配子体的形成需要重复。足底。2012;235(4):713 - 27所示。https://doi.org/10.1007/s00425-011-1532-7.
- 39。
Rasheed A, Wen W, Gao FM, Zhai SN, Jin H, Liu JD, Guo Q, Zhang YG, Dreisigacker S, Xia XC, He ZH.(中国)面包小麦关键经济性状基因KASP测定方法的建立和验证。《中国科学(d辑)》2016;https://doi.org/10.1007/s00122-016-2743-x.
- 40
李普卡AE,田F,王QS,佩弗J,李M,布拉德伯里PJ,戈尔马,巴克勒ES,张ZW。GAPIT:基因组关联与预测集成工具。生物信息学。2012;28(18):2397–9.https://doi.org/10.1093/bioinformatics/bts444.
- 41
Yu JM,Pressoir G,Briggs WH,Bi IV,Yamasaki M,Doebley JF,McMullen MD,Gaut BS,Nielsen DM,荷兰JB,Kresovich S,Buckler ES。一种统一混合模型方法,用于占多级相关性的映射。NAT Genet。2006; 38(2):203-8。https://doi.org/10.1038/ng1702.
- 42
李浩,彭哲,杨旭,王伟德,付建军,王建军,韩一奇,郭特,杨恩,刘杰,沃伯顿ML,程一博,郝XM,张P,赵JY,刘玉杰,王GY,李JS,严JB。全基因组关联研究剖析了玉米籽粒油脂生物合成的遗传结构。纳特·吉内特。2013;45(1):43–50.https://doi.org/10.1038/ng.2484.
- 43
关键词:多倍体小麦,转录空间分布,转录空间分布,转录空间分布科学。2018;361 (6403):eaar6089。
致谢
不适用。
资金
该研究得到了中国国家自然科学基金(2017YFD0100600),中国国家自然科学基金(31901539)和河北省科技规划项目(16226320D)的支持得到了支持。这些融资机构在研究和收集,分析和解释的设计中没有作用,以及编写手稿。
作者信息
从属关系
贡献
YZ和XJY构思了该项目并制定了科学目标;LG、CSM、TFY、KX、HWC和SHZ进行了实验设计和数据分析;LG和YZ撰写了手稿草稿;YZ和XJY对论文进行了修改。所有作者讨论了结果,并阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
我们声明这些试验符合中国的伦理标准和法律,所有小麦品种都是按照国家标准收集的。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商说明
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
权利和权限
开放获取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用本文
高丽丽,李建平,李国强,李国强。等。全基因组关联研究揭示了小麦产量和品质相关性状的遗传基础。BMC植物BIOL.21,144(2021)。https://doi.org/10.1186/s12870-021-02925-7
收到:
接受:
发表:
关键词
- GWAS公司
- 质量
- 单核苷酸多态性
- 小麦
- 屈服