抽象的
背景
属于的丝氨酸蛋白酶抑制剂Potatoⅱ型在幼儿家庭P旋转速度我抑制剂(Pin-II型pi)是植物必需的防御分子。它们具有多个抑制重复域、保守的二硫键模式和一个三肽反应中心环。Pin-II型pi的这些特性使它们成为蛋白质工程和设计农业和治疗应用抑制剂的潜在分子。然而,由于现有数据库中缺乏注释的蛋白质序列及其功能属性,这些pi的多样性仍未被探索。
结果
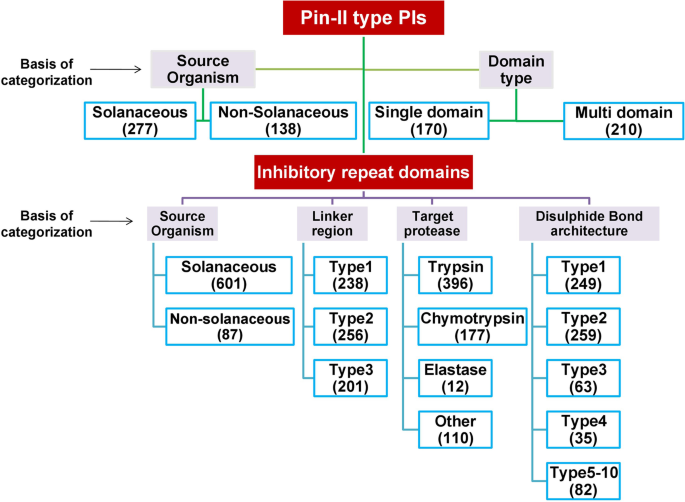
我们开发了一个数据库,PINIR(PIN-II型PIS信息资源),通过系统收集和手动注释415针-II型PI蛋白序列。对于每个PI,特征序列的数量和位置指定:695个结构域,75个接头,63反应性中心环和10个二硫键模式被识别和映射。数据库分析显示了PI的新型子类别,物种相关性发生抑制结构域,反应性中心环和二硫键模式。通过分析连接器区域,我们预测接头区域的替代处理可以在茄科家族中产生PI变体。
结论
PINIR(https://pinir.ncl.res.in.)为浏览和分析PinⅡ型PIs的蛋白质序列提供了一个web界面。提供了有关特征序列、时空表达、生化特性、基因序列和文献参考的信息。PINIR分析描述了PinⅡ型PI蛋白序列的保守物种特异性特征。抑制域和反应环序列的多样性为设计PinⅡ型pi提供了潜在的应用。PINIR数据库将作为进一步研究Pin II型PIs的综合信息资源。
背景
蛋白酶抑制剂(pi)作为植物的内在防御分子,在治疗方面越来越重要[1.,2.].P大田型在Hibitor-II家庭πS(Pin-II型PIS)形成了一个主要的植物PIS系列,这在保护植物免受生物应激的影响中起着至关重要的作用,特别是对抗鳞翅目昆虫害虫[3.].PIN-II型PI是主要在溶律植物中发现的丝氨酸PIS,并在伤害或昆虫侵扰时诱导。它们通过形成蛋白酶-PI复合物,抑制昆虫肠道中存在的消化丝氨酸蛋白酶,这导致蛋白质同化的降低,并最终导致昆虫的生长和发育中的延迟[4.,5.]. PinⅡ型PIs也被用作抗肥胖药物,以减少人类的食物摄入量,因为这些PIs通过抑制胰蛋白酶增加饱腹激素(胆囊收缩素)的水平[6.,7.,8.].PIN-II型PIS的蛋白酶抑制潜力也已用于抗菌和抗真菌剂的发育[9,10].
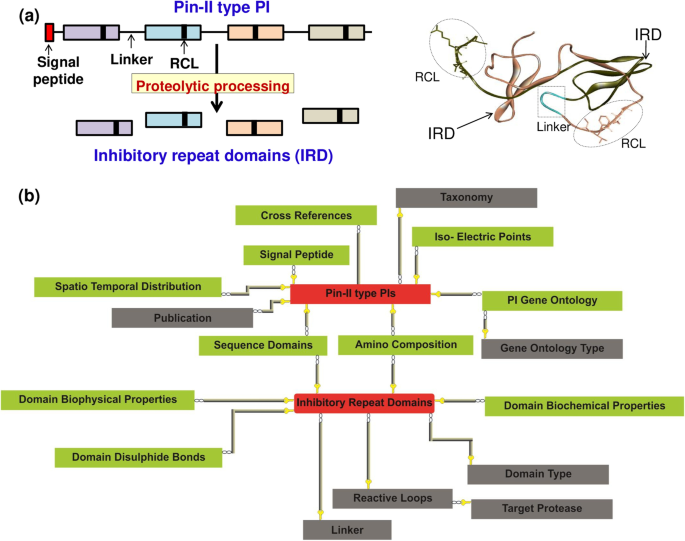
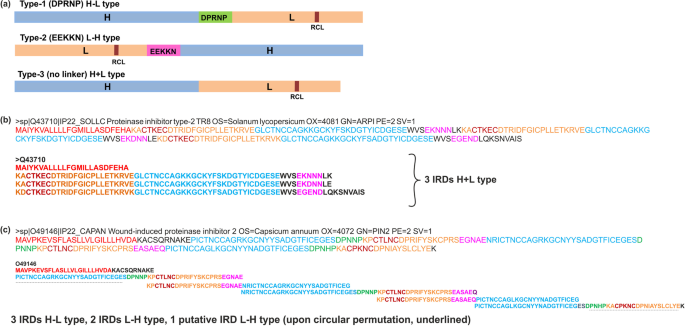
PIN-II型PI的独特特征是存在多个抑制域(IRD)。单个PIN-II型PI由多个IRD组成,该IRD由5至6个氨基酸接头区域连接。IRDS为50至55个氨基酸长蛋白,具有二硫键稳定的独特结构[11,12].接头区域处的蛋白水解加工释放来自其母体PIN-II型PI分子的IRD。然后可以靶向多个丝氨酸蛋白酶[5.,13] (图。1.a) 是的。PI中ird的数目是可变的;同时,IRDs的蛋白质序列也表现出细微的差异,这可能对IRDs的功能产生深远的影响[14]. 例如,IRD-7和IRD-9(PINIR数据库中的IRD-145和IRD-143)来自一年生辣椒蛋白质序列相似性超过90%,但对丝氨酸蛋白酶的抑制活性存在显著差异[15].这种差异归因于IRD-9中的IRD-7中的IRD-7中的两个半胱氨酸残基,这导致两种二硫键的损失。这些变化导致IRD-9的灵活性增加,允许在胰蛋白酶的活性部位中更好地适合IRD-9,因此更高的抑制活性[15,16].蛋白质序列的多样性对于组合抑制很有用,因为IRDs可以针对在适应其他PI亚型时产生的抗性蛋白酶[4.].每个IRD包含一个三肽环称为反应中心环(RCL),它提供了针对Pin-II型pi的靶标特异性[17].RCl是针对靶丝氨酸蛋白酶的主要相互作用位点,并用作独立于天然IRD支架的抑制三肽[18].
PIN-II型PIS提供多级策略来设计用于农业和治疗应用的分子。与短链接器连接的多个IRD的PIS表明了同时的潜在方法体外PIS的交付[19,20.].二硫键本身是人类治疗剂工程PIS的潜在候选者,如循环酯的证明[21,22]和Bowman-Birk蛋白酶抑制剂(BBI)蛋白[23].此外,由于RCL区域定义了Pin-II型pi的反应活性[17]将这些多肽接枝到现有的支架上,可以制备功能发生改变的蛋白质。RCL三肽也可用作小分子PIs,因为它们适于化学合成肽[24].
这些特征序列(IRD,LINKER和RCL)在不同PIN-II型PI蛋白中的数量和序列中变化。因此,必须具有这些PI的专用资源来了解这个家庭的分类和多样化。虽然有几种蛋白质和PI数据库可用,但None为此PIN-II类型PI系列提供了详细的注释信息。具体地,IRDS,接头,RCl,二硫键和生物化学特征的数量和位置未在任何蛋白质数据库中映射。
我们开发了皮尼尔(Pin-II型pi信息资源),可用AT.https://pinir.ncl.res.in.是一种基于Web的信息系统,提供具有用于浏览和分析与PIN-II型PI相关的信息的实用程序的用户友好的Web界面。Pinir的当前释放具有415个PIN-II PI序列的序列注释,其中包含695个IRDS,75个接头和63 RCLS。在数据库中也汇编了表达模式,靶蛋白酶和昆虫等生化特征。此外,我们基于IRDS,连接器和RCLS的位置,数量和序列等注释对PIN-II型PIC进行了分类。在分析PIN-II型PI序列中的链接器区域的PINIR数据库时,我们假设PIN-II型PIS可能经历替代处理以产生各种IRD。对该数据集的进一步解释将突出显示PIN-II型PI序列的分层分类,并用作有用的科学资源。
建筑和内容
数据库开发模式
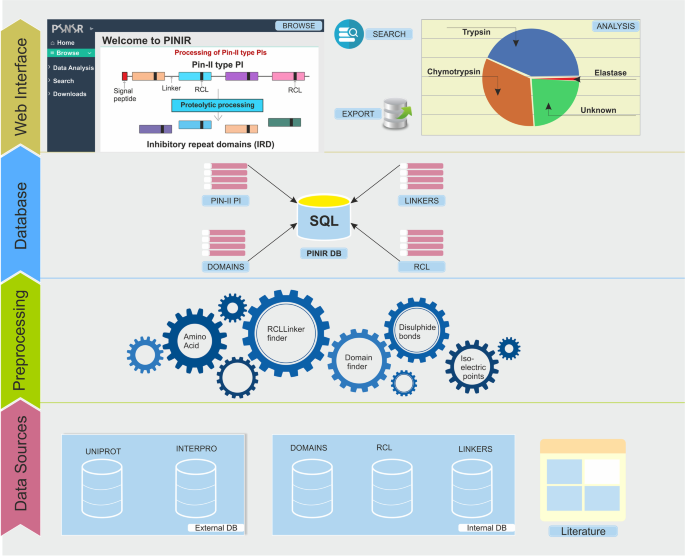
PIN-II型PIS的文献综述和蛋白质数据库的研究导致了PIN-II型PIS各种特征的阐释,该分子确定了我们数据库的各种元素(补充表S.1.). 数字1.b显示Pinir数据库的概念数据模型(CDM)。它识别了他们之间的不同实体和关系。所有信息围绕两个主要实体构建:'PIN-II型PIS'和“抑制重复域”(IRDS /域)。桌子1.列出捕获主要实体和提供支持信息的实体的详细信息的实体。Pin II PI信息资源组装遵循的开发模型如图所示。2..它包括四个开发阶段:(1)数据来源;(2)数据预处理和整合;(3)数据库创建;(4)和PiniR网站。
数据源
PiniR数据库方案的实体的数据由几个来源累积,例如外部数据库,已发布的信息和内部数据编译。
- 一种)
蛋白质序列和关于PIN-II型PIS的相关数据从UNIPROT提取[25]和Interpro [26].使用Interpro ID的PIN-II PI系列的术语查询这些在线数据库:IPR003465和PFAM ID:PF02428。从UniProt下载了共有415个PIN-II PI型记录。在415,15,15中进行了审查,399个未经概览的Uniprot参赛作品。
- (二)
使用带有Pfam域ID PF02428的HMMSEARCH对Pin II型PIs中的IRD进行识别,得到404个结果。还使用UniProtKB中的一些序列(P84813、P01078、P01079、P56615、P83241)执行PHMMER搜索。删除冗余和不完整的序列导致了总共695个ird。
- (三)
通过IRD序列的序列对准手动制备反应性中心环(RCl)和接头的数据。在Pin-II型PI系列中鉴定了75个独特的接头区域和63 RCLS。
- (d)
使用UniProt和Google Scholar的PubMed ID搜索与Pin-II型PI相关的出版物。从制品中手动编制时空分布,结构域结构折叠,生物化学性能和生物物理性质等实验信息。
数据预处理和集成
数据的预处理是手动完成,也可以在许多情况下通过使用Python程序或结构化查询语言(SQL)查询和程序来自动化。以下是执行数据预处理的场景:
- 一种)
从Uniprot导入的PIN-II型PI数据具有诸如PIN-II型PIS冗余和覆盖各种特征的单片记录。为了有效处理和创建简化视图,使用SQL查询和程序将UNIPROT数据分发给不同的表,保持其关系完好无损。
- (二)
通过添加RCl序列的P1,P2和P1的P1,制备了RCl序列及其靶蛋白酶的手动制备的RCl序列列表。此外,生物体植物富含种类和物种数据。这两者都是通过使用Python编写的程序来实现的。
- (三)
编写了Python程序来确定695个IRDs(域)中63个RCL和75个连接器的位置。
- (d)
基于域序列中的RCL和连接器的位置确定的另一个重要属性“域类型”被识别出使用Python程序的所有可用IRDS确定。
- 电子)
编写了一个Python程序来捕获Pin-II-PI序列中ird的位置和出现的数据。
- F)
有关IRDS中二硫键的信息使用在线效用二核编制[27].识别PIN-II型PI序列的等电点,网站的在线效用http://isoelectric.org/[28使用了。
- G)
我们使用Python编程来得出关于PIN-II型PIS和域序列的氨基酸组成的信息。
数据库创建
Pinir是一种基于Web的信息系统,可以让用户轻松访问PIN-II型PI数据库。此信息系统是使用WISA(Windows,IIS,SQL和ASP.NET)堆栈开发的。Windows Server 2012 R2用作后端数据库的SQL Server 2016,它托管在IIS 8.0 Web服务器上,使用ASP.NET(C#)Framework 4.7创建前端。
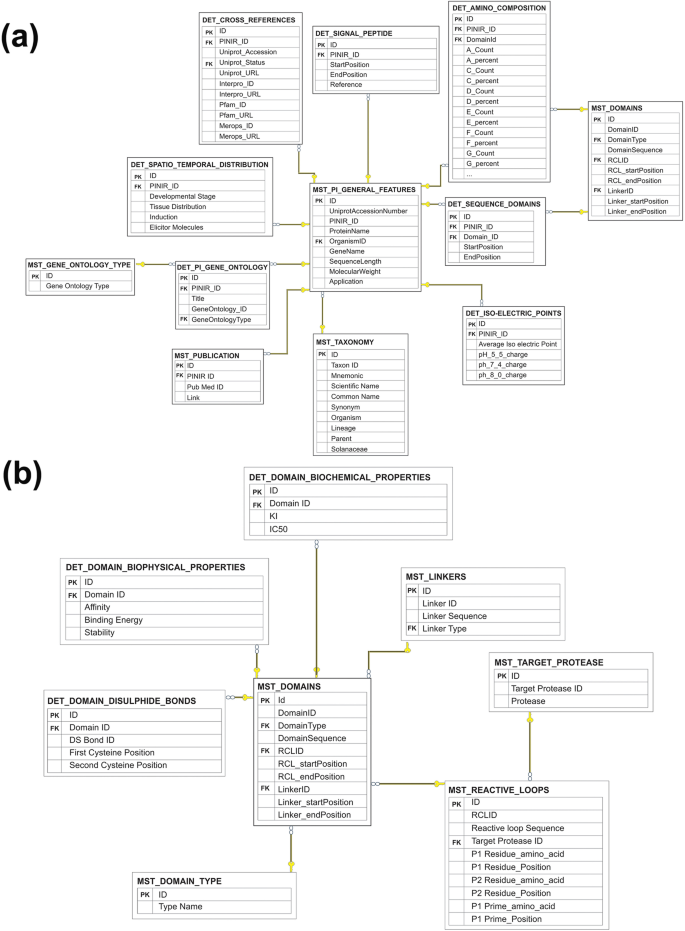
在清洁发展机制层面确定的所有实体(图。1.b)的PINIR DB转换成相应的Table(列于Table2.)在逻辑数据模型(LDM)级别。LDM捕获它们之间的实体和关系,并指定每个实体的属性,主键和外键PIN-II型PIC(图。3.a)和抑制域(图。3.b) 是的。在LDM级别,包含不依赖于其他表的数据的表的名称以Master的缩写“MST”作为前缀。包含有关主表的额外信息的表以缩写“DET”作为前缀表示详细信息。
在LDM中,表MST_PI_GENERAL_FEATURES保存在CDM中标识实体'PIT II型PIS'的信息。如表S所示2.,属性“ID”表示主键,“organimid”是表MST\u分类法的外键。类似地,另一个主要实体“Inhibitory Repeat Domains”由LDM(表S)中的表MST\u Domains表示2.),它保存标识Pin-II类型pi的IRDs的信息。这里的'ID'属性表示主键,有3个外键,'RCLID', 'LinkerID',和'DomainType',分别指MST_REACTIVE_LOOPS, MST_LINKERS和MST_DOMAIN_TYPE表。
最后,LDM转换为物理数据模型(PDM),表示模型是如何构建在数据库中的。在PDM中,将附加字段,即列名称,列数据类型,列约束,主键(S)和外键添加到相应实体中LDM中标识的表中。表S.2.列出了PiniR数据库的各种表结构,它使用Microsoft SQL Server实现。
PINIR网站
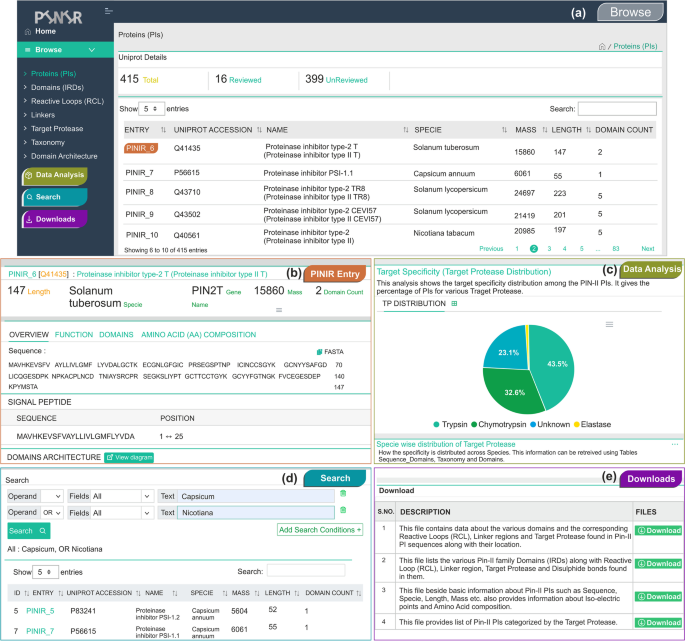
为使Pin II型PI数据库可公开访问,开发了一个基于web的信息系统,可通过以下方式访问:https://pinir.ncl.res.in..PiniR提供直观和用户友好的Web界面来浏览,分析,搜索和下载与Pin-II型PI相关的信息(图。4.).
浏览
Pinir网站的浏览模块提供了一种用户友好的界面,以查看Pin-II型Pissuch的功能注释作为IRDS,RCL,接头区域,靶蛋白酶和生物体。浏览蛋白质,列出了一个带有Pinir条目,Uniprot登录号,名称,物种,物种,长度和域数的表。单击任何PIN-II型PI条目直接指向PIS详细信息页面,由四个部分组成:概述,功能,结构域和氨基酸组成。
概述部分提供有关PIN-II型PI的基本信息,例如长度,质量,物种,基因名称,等电点和域数。它提供了有关PI序列的细节,即氨基酸序列,信号肽,域架构。它还提供了对Uniprot,PFAM,Interpro,EMBL和Merops数据库的交叉引用以及与可用的任何引用的链接。
功能部分提供了关于RCL、靶蛋白酶和与Pin II型PI相关的GO术语的信息。本节还提供了有关时空分布、畴结构折叠、生物化学性质和生物物理性质的详细信息。
域部分列出了在PIN-II型PIS序列中发现的IRD,其中包含RCL和链接器。它还提供有关PI序列的域架构的详细信息。
氨基酸组成部分列出了PIN-II型PIS序列中的每个氨基酸数及其百分比组成。
数据分析
PiniR网站的数据分析模块旨在为用户提供对Pin-II型PI数据库的全面分析。它分为5类:基于Pin-II型PI,结构域(IRD),反应循环(RCl),接头和靶蛋白酶的数据分析。在每个类别下,以各种方式分析数据。对于每个分析,对结果的标题和简要描述具有相应的图表。这些图表可在PNG(便携式网络图形)和SVG(可缩放向量图形)格式中下载,并且以表格格式显示的结果数据可以以CSV(逗号分隔值)文件下载以进行离线分析。
搜索和下载
对于浏览模块中的每个选项,结果以表格格式显示。通过根据用户要求单击列标题,可以对结果数据进行排序。此外,在搜索结果表的右上角提供了快速搜索。此快速搜索是不敏感的,接受用户可以根据显示列的内容过滤结果的任何模糊术语。除了快速搜索外,网站上提供专用搜索模块。此搜索允许使用Uniprot登录,物种,RCL,链接器,靶蛋白酶和基因本体术语的基于文本的搜索。用户还可以通过使用布尔表达式组合多个查询(例如,或者而不是)来创建复杂搜索查询。搜索结果显示在表中,其中每个记录表示PIN-II型PI及其在单独列中的信息。PiniR网站还提供了一个下载模块,以方便其用户在CSV文件中下载Pini数据库中捕获的信息。
实用与讨论
目前可用的蛋白质和PI数据库,如UNIPROT,PFAM,Interpro,Plant PI和Merops具有有限的PIN-II型PI系列蛋白序列的注释信息。因此,我们开发了该系列的全面信息资源,以提供每个可用的Pin-II型PI序列的功能注释。我们已经鉴定了IRDS,RCl,接头和二硫键及其在PI序列中的位置。在数据库中也汇编了物理化学性质,例如等电点和氨基酸组合物。以下部分描述了PiniR数据库的深入数据分析:
PinⅡ型PI序列的出现与域分布
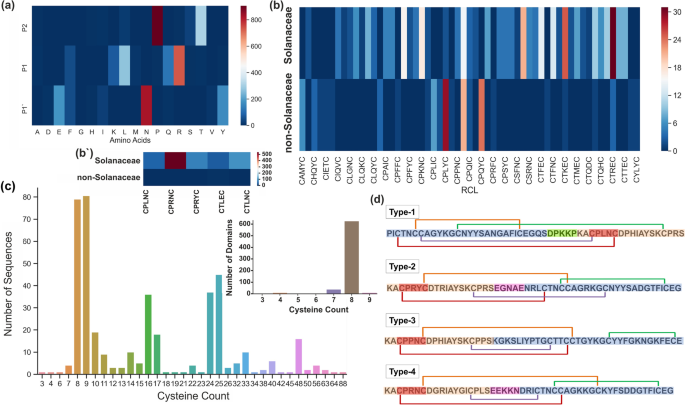
截至2020年10月2日,PiniR数据库封装了109种分布的415针-II型PIS的信息。在415个序列中,277个PIN-II型序列来自溶均植物,138来自非溶性植物。大量的PIN-II型PI序列由溶于溶的植物代表一年生辣椒(N = 99), followed bySolanum Tuberosum.(N = 52) and烟草(N= 22)。我们还在非茄科植物中发现了Pin-II型PI序列江南卷柏,Coffea Canephora.(Rubiaceae),罗莎中华民神(蔷薇科),表明Pin II型PIs的广泛存在(补充图S)1.a,b)。
我们在415个PIN-II型PI中获得了695个独特的IRD序列,表明PIS由IRD的多种组合组成。尽管在属中发现了更多的PIN-II型PIS辣椒比尼古利亚娜,后者中发现的独特IRD的数量越高,如补充图所示1.光盘。这表明属尼古利亚娜具有更大的IRD序列的多样性,而辣椒从有限的IRDS产生更多数量的PIN-II型PIS。大多数IRDS出现在仅一个序列的一部分(数据库中的单一发生)(补充表S3.).这表明反复性的IRD可能更加保守。有趣的是,所有的IRD都对他们的起源属特异;也就是说,仅在单个属中发现每个结构域(一次或重复)。因此,PIN-II型PI由IRDS的组合组成,这可能是由特定属内的序列的限制演变(补充表S.4.).
我们在PINIR数据库中发现了14个PinⅡ型PIs域结构(补充表S)5.).我们将PIN-II型PIS分类为单域(N-DOMAINA = 1)或多域(n个域> 1)。在多域PIS中,大多数由2到5之间的n个域组成,几个n个域大于7.在Interpro中有17个用于Pin-II型PIS的域架构,其中9个由域组成IPR003465(PIN-II型PIS),而8个架构由PIN-II结构域组成,与其他家庭的域组合。因此,我们从PiniR数据库中识别了用于PIN-II型PIS的新型域架构。此外,Solanaceae植物由具有1至21个范围的PIS组成,而非溶性植物仅由单结构域PI组成。在170个中,单结构域PIS,60分布在10种溶那种物种中,而110个单结构域PIS在63种非溶性物种中分布。这表明溶于溶律植物优选表达多域PIS,其可以容纳抑制多种蛋白酶所需的不同/相同的IRD序列。我们也观察到这一点一年生辣椒具有N-Domain = 5的大多数序列,Solanum Tuberosum.具有最大序列N = 2 or 3, and烟草具有n-域的最大序列 > 5表明n-结构域和物种之间存在相关性(补充表S)6.).
PIN-II型PIS中的链接器多样性和分布
PinⅡ型PI家族中存在两种类型的连接区,即Ⅰ型(DPRNP)和Ⅱ型(EEKKN)[29]. 连接区将IRD分为重片段(H)和轻片段(L)。L片段由RCL组成(图。5.a) 是的。根据H和L片段在连接器区域周围的排列,有三种类型的IRD,如图1所示。5.一种。具有类型-I链接器的IRD具有H-L型排列:{H-Linker-L},而具有II型连接器的人具有L-H型:{L-Linker-H}。没有接头的IRDS具有H + L布置(III型)。在手动识别接头区域后,我们发现了75个接头序列,属于I型(n = 24)和II型(N = 51).
我们将IRD序列分为三种亚型,类型是基于IRD中是否存在i型、ii型或无连接区。共鉴定出含IRDs的i型连接子238个(H - L型)、含IRDs的ii型连接子256个(L - H型)和无连接区IRDs的201个(H + L型)。Kong and Ranganathan [29建议,IRD子类型的存在基于多域Pin-II型PI中的域组织。针对Pin-II型PI系列报告了两种类型的域组织:串联重复(串上的珠子)或圆形允许的(扣紧手镯)。串联重复PIS具有L-H型IRD,而在循环允许的PIS中,由N-和C末端序列形成的畴存在于H + L拓扑中,而其他域采用H-L拓扑。然而,在我们的分析中,我们发现L-H和H-L拓扑结构都在相同的IRD序列内存在,这取决于链接器在父PIN-II型PI序列中的位置。我们将IRD序列和接头区域映射到PIN-II型PI序列,发现相同的片段可以是在接头区域的交替加工时是(L-H和H-L)拓扑的一部分。对于H + L型,链接器区域存在于IRD序列之外(图。5.b).在Pin-II型PI中存在重叠的IRD序列,表明PI在连接区域的交替蛋白水解过程可能产生来自同一亲本Pin-II型PI的巨大的IRD多样性。例如,在图。5.C,连接器区域的替代处理可以导致五种IRD变体而不是4个IRD。在不增加蛋白质长度的情况下,可以将PIN-II型PI中的重叠变体序列的存在作为自然排列机制,以产生大量的IRDS。PIN-II型PIS的这种特征在于昆虫蛋白酶的共同演进中可能有益,其中多个蛋白酶可以通过昆虫肠道的几种IRD靶向[30.].与PIS相比,它也可能解释了较高的IRD,如我们分析所示。无论这些替代处理机制是否在自然中发生并发挥PIN-II型PI组织的作用,需要阐明。
Linker区的物种分布表明,来自溶均匀植物的IRDS具有接头区域,而非溶于溶于植物植物的PIS是不含接头的所有类型-III。此外,含有IRD的类型-I连接器主要是存在于辣椒,而II型和III型链接器的IRDS尼古利亚娜和Solanum.属,表明连接子优先出现取决于物种(补充表S)7.).
此外,I类型-I链接器序列DPNNP是独占的辣椒而DPRNP主要分布于尼古利亚娜和dpknp in.Solanum.植物。DPRNP和DPKNP在物种中显示出广泛的发生。在II型接头序列中,EGNAE是独家的辣椒,EEKKN在尼古利亚娜,和EGSPESolanum.物种(补充图2.; 补充表S8.S9). 这表明连接子的有限分布,这表明PinⅡ型PIs可能具有物种特异性的蛋白水解加工机制。
RCL分布和特异性
在IRD序列数据集中,鉴定了63个RCL序列。RCl序列“CPRNC”是PIN-II型PIS中最主要的,在PIN-II型PI序列中发生576次,其次是“CTLNC”(113次)和“CPRYC”(94次)(补充表S10)在IRD数据集中,“CPRNC”出现了291次“CPRNC”在中国广泛存在辣椒,尼古利亚娜和Solanum.物种,并以高数量发现。但是,“CTLNC”和“CPRYC”显示有限的发生,主要是一年生辣椒(补充表S10).此外,RCL的P1',P1和P2位置与靶蛋白酶的活性位点相互作用。这些位置的氨基酸的分布显示出这些关键位置的几个氨基酸的偏好。具体而言,Pro在P2残基下占优势,然后是Thr(图。6.a) 是的。据报道,P2-Pro在决定RCL的效价和特异性方面起着重要作用[18].有趣的是,尽管在结构上与Thr结构相似,但Ser很少在这个职位上找到。在P1位置,这是特异性确定残留物,Arg是显性的,表明大多数是胰蛋白酶抑制剂。接下来是Leu,表明胰凝乳素抑制剂。令人惊讶的是,我们发现GLN也在〜100序列中占据P1位置。在丝氨酸蛋白酶抑制剂P1位点的GLN的存在尚未提前报道。由于GLN,Ser,Thr,Met,Glu和P1残基的氨基酸,23.1%的靶向IRDS的靶标未知。这可能是PIN-II类型PIS的独特功能,导致其目标特异性的多样化。RCls的物种分布表明,溶于溶于溶于植物含有不同的RCl序列,Arg / Lys / Leu / Gln在P1残基。相比之下,非溶性植物主要含有P1位置的Gln / Leu(图。6.b和6 b”)。在P1'残留物中,Asn最常见,其次是Tyr和Glu。这些氨基酸的功能特征有待进一步研究。
PinⅡ型PI家族中二硫键的变化
二硫键对于维持PIN-II型PIS的适当结构折叠至关重要,对IRDS的效力很重要[11,16].二硫键的变化和半胱氨酸残基的数量赋予针-II型PIS的差异效力[14].4二硫键的存在是PIN-II型PIS的IRDS的特征。我们对PIN-II型PI序列进行了氨基酸组成分析,表明CYS是最丰富的残留物,其次是GLY(补充图)3.).其他氨基酸分布在Pin-II型PI家族中。半胱氨酸的数量分析表明,大多数IRDs含有8个Cys残基或4个二硫键。几个序列(N = 4) had 7 Cys residues, indicating disulfide bond variants (Fig.6.C)。
此外,PIN-II型PI序列上的CYS分布表明,大多数序列具有8或9个CYS,对应于IRDS观察到的CYS。此外,具有16,24,32,40和48个Cys的序列突出,显示了多域PI。有趣的是,具有9,10,17,25和33个Cys的序列也具有高数量,表明在PIN-II序列中存在二硫键变体或自由Cys残基(图。6.C)。虽然IRDS由8个Cys残留量组成,但PIN-II型PIS中奇数CYS的存在表明额外的CYS介导的结构支架。
此外,我们还分析了IRD序列中的二硫键。我们在IRD中发现了10种二硫键结构(补充表S)11). 类型1到4是最常见的(图。6.d) 是的。二硫键类型与连接体区域的类型相关,其中1型和2型二硫键结构主要发现于分别具有1型和2型连接体的IRD中(补充表S)12).因此,二硫键连接是守恒的类型的IRDs。此外,辣椒物种包括1型债券架构,常见于2型和3型尼古利亚娜物种,突出了二硫键支架的特异性偏好(补充表S.13).除了ird内键外,确定Pin-II型pi中ird间的二硫键连接也很有趣。
结论
PIN-II型PI具有希望作为蛋白质工程支架的应用。这些蛋白质可用作农业害虫控制剂,因为它们有效降低了农业害虫的生长Helicoverpa Armigera[4.,15].此外,据报道,据据报道,以线性和环状形式合成的RCl肽用于制定环保害虫害虫控制策略[24].PIN-II PIS也被用作抗微生物和抗真菌剂[9].例如,Potamin-i抑制了几种蛋白酶,如胰凝乳素,胰蛋白酶和木瓜蛋白酶,并且是对致病微生物菌株的活性,包括白色念珠菌,辣椒[10].这些PI也用作减少肥胖的制剂[6.,31.].虽然已经研究了来自马铃薯的PIN-II型PI,但是通过利用PINIR数据库,也可以探索来自其他物种的PIN-II型PIS,其提供签名序列,即RCL,链接器和域区域的存储库。这些注释在任何其他公共数据库中都不提供,使PiniR成为针对Pin-II型植物PI系列的独特资源。
PINIR是一个家族特有的数据库,它根据序列特征识别了Pin II型PI家族中的新子类别(图。7.).我们确定了415个PIN-II型PI中的688个IRDS,表明许多PI具有重叠的IRD,并且这些IRD在其氨基酸序列中具有小的变化。对接头区域的研究及其位置以连续的PIN-II型PI序列突出显示H-L和L-H型IRDS。该结果表明植物中的未探明机制,用于从有限的PIN-II型PI蛋白的有限尺寸产生几种IRD组合。我们的目标是通过常规添加PIN-II型PIS的新功能和序列来持续更新和扩展PINIR数据库。
可用性数据和材料
在当前研究期间生成和分析的数据集可在Pinir存储库中提供,https://pinir.ncl.res.in
缩写
- 乘客信息:
-
蛋白酶抑制剂
- PIN-II型PIS:
-
土豆类型II抑制剂家族蛋白酶抑制剂
- PINIR:
-
PIN-II型PIS信息资源
- IRDS:
-
抑制重复域
- RCL:
-
反应中心循环
- BBI:
-
鲍曼伯克蛋白酶抑制剂
- 清洁发展机制:
-
概念数据模型
- SQL语句:
-
结构化查询语言
- LDM:
-
逻辑数据模型
- PDM:
-
物理数据模型
- PNG:
-
便携式网络图形
- SVG:
-
可缩放矢量图形
- CSV:
-
逗号分隔值
参考
- 1.
海林格R,格鲁伯CW。植物肽基蛋白酶抑制剂。今天的毒品交易。2019;24:1877–89.https://doi.org/10.1016/j.drudis.2019.05.026.
- 2.
Srikanth S,Chen Z.治疗植物蛋白酶抑制剂 - 对癌症治疗的关注。前药狼。2016; 7:470。https://doi.org/10.3389/fphar.2016.00470.
- 3.
Tamhane VA,Mishra M,Mahajan NS,Gupta VS,Giri AP。植物PIN-II家族蛋白酶抑制剂:结构和功能多样性。Funct植物sci biotechnol。2012; 1:42-58。
- 4.
Dunse Km,Stevens Ja,Lay Ft,Gaspar Ym,Heath Rl,Anderson Ma。薯类型和II蛋白酶抑制剂的共表达给棉花植物免受昆虫损伤的保护。Proc Natl Acad Sci U S A. 2010; 107:15011-5。https://doi.org/10.1073/pnas.1009241107.
- 5。
Mishra M,Tamhane VA,Khandelwal N,Kulkarni MJ,Gupta VS,Giri AP。重组笨蛋的相互作用Helicoverpa Armigera肠道蛋白酶揭示了它们的加工模式,稳定性和效率。蛋白质组学。2010; 10:2845-57。https://doi.org/10.1002/PMIC.200900853..
- 6。
通过胰蛋白酶依赖机制,马铃薯蛋白酶抑制剂抑制食物摄入并增加循环胆囊收缩素水平。《科学通报》2011;35:36 - 43。https://doi.org/10.1038/ijo.2010192.
- 7。
Peters HPF,Foltz M,Kovacs Emr,Mela DJ,Schury Eah,Wiseman Sa。蛋白酶抑制剂衍生自马铃薯的蛋白酶抑制剂对人类食欲,食物摄入和血浆胆囊蛋白水平的迷你硅酸盐中配制。in j obes。2011; 35:244-50。https://doi.org/10.1038/ijo.2010.136.
- 8。
朱y,拉斯拉多·贾,胡锦涛蛋白酶抑制剂II抑制健康女性的餐后食欲:随机双盲安慰剂对照试验。食物功能。2017; 8:1988-93。https://doi.org/10.1039/c6fo01803c..
- 9。
Bártováv,bártaj,贾罗斯科姆抗真菌和抗菌蛋白和肽的土豆(Solanum Tuberosum.块茎及其应用。应用微生物生物技术。2019;103:5533–47.https://doi.org/10.1007/S00253-019-09887-019-09887-019-09887-019-.
- 10.
Kim JY,Park SC,Kim MH,Lim HT,Park Y,Hahm KS.金英杰,朴英杰,朴英杰,朴英杰,朴英杰。马铃薯胰蛋白酶-糜蛋白酶抑制剂的抑菌活性研究。生物化学Biophys Res Common。2005;330:921–7.https://doi.org/10.1016/j.bbrc.2005.03.057.
- 11.
Gartia J,Anangi R,Joshi Rs,Giri AP,King GF,Barnwal RP等。抑制域重复域变体12的NMR结构和动力学,一种植物蛋白酶抑制剂一年生辣椒与其它植物蛋白酶抑制剂的结构关系。生物分子结构动力学杂志。2020;38:1388–97.https://doi.org/10.1080 / 07391102.2019.1607559..
- 12.
Bartent-NG IH,NG KK-S,Cherney MM,Pearce G,Ghani U,Ryan Ca等。番茄抑制剂的未结合形式-II揭示了反应部位环中的互联柔性和构象变化。J Biol Chem。2003; 278:31391-400。https://doi.org/10.1074/jbc.m304562200.
- 13.
Bartent-NG IH,NG KK-S,Cherney MM,Pearce G,Ryan Ca,James Mng。抑制的结构基础,揭示了双头番茄抑制剂-II和枯草杆菌蛋白酶葡萄糖蛋白的1:2络合物。J Biol Chem。2003; 278:24062-71。https://doi.org/10.1074/jbc.m30202020200.
- 14。
Schirra HJ、Guarino RF、Anderson MA、Craik DJ。马铃薯Ⅱ型丝氨酸蛋白酶抑制剂中二硫键的选择性去除尼古利亚娜alata.揭示了反应性部位环的差异稳定。J Mol Biol。2010; 395:609-26。https://doi.org/10.1016/j.jmb.2009.11.031.
- 15.
乔希RS,古普塔VS,吉里AP。差异抗菌Helicoverpa Armigera的独特抑制重复域一年生辣椒蛋白酶抑制剂。植物化学。2014;101:16-22。https://doi.org/10.1016/j.phytochem.2014.01.011.
- 16。
Joshi Rs,Mishra M,Tamhane VA,Ghosh A,Sonavane U,Suresh CG等。Pin-II蛋白酶抑制剂SAN的显着效率是两个保守的二硫键是由于反应性部位环中的柔韧性和氢键密度增强。J Biomol Struct Dyn。2014; 32:13-26。https://doi.org/10.1080/07391102.2012.745378.
- 17。
Beekwiller J,Schipper B,Bakker P,Bosch D,Jongsma M.马铃薯蛋白酶抑制剂II反应性位点突变体的表征。EUR J Biochem。2000; 267:1975-84。https://doi.org/10.1046/j.1432-1327.2000.01201.x.
- 18。
Saikhedkar Ns,Joshi Rs,Bhoite,Mohandasan R,Yadav Ak,Fernandes M等。来自马铃薯II型蛋白酶抑制剂的反应性中心环衍生自蛋白酶抑制剂的三肽优先抑制中肠蛋白酶Helicoverpa Armigera.昆虫生物化学Mol Biol。2018; 95:17-25。https://doi.org/10.1016/j.ibmb.2018.02.001.
- 19。
Khandelwal N,Doke DS,Khandare JJ,Jawale PV,Biradar Av,Giri AP。生物物理评估和体内递送固定在二氧化硅纳米球上的植物蛋白酶抑制剂。胶体表面B生物界面。2015; 130:84-92。https://doi.org/10.1016/j.colsurfb.2015.03.060.
- 20
Tamhane VA,Dhaware DG,Khandelwal N,Giri AP,Panchagnula V.增强渗透,叶片保留和植物蛋白酶抑制剂活性与双连续微乳液。J COLDOIT接口SCI。2012; 383:177-83。https://doi.org/10.1016/j.jcis.2012.06.025.
- 21
Northfield Se,Wang CK,Schroeder Ci,Durek T,Kan MW,瑞典贝格·杰等。二硫化物富含巨环肽作为药物设计的模板。EUR J Med Chem。2014; 77:248-57。https://doi.org/10.1016/j.ejmech.2014.03.011.
- 22.
关键词:生物化学,生物化学,生物化学,生物化学分子接枝到一个稳定的框架产生新的环肽治疗多发性硬化症。ACS Chem bio2014; 9:156-63。https://doi.org/10.1021/cb400548s..
- 23.
Fittler H,Avrutina o,Rimping M,Kolmar H.基于向日葵胰蛋白酶抑制剂支架的人矩阵-1的有效抑制剂。j pept sci。2014; 20:415-20。https://doi.org/10.1002/psc.2629.
- 24.
Saikhedkar ns,Joshi Rs,Yadav Ak,Seal S,Fernandes M,Giri AP。从植物PIN-II型蛋白酶抑制剂反应性中心回路的植物启发循环肽用于作物保护免受虫害。Biochim Biophys Acta - Gen Subj。2019; 1863:1254-62。https://doi.org/10.1016/j.bbagen.2019.05.003.
- 25.
consortium tu。Uniprot:蛋白质知识的全球中心。核酸RES。2018; 47:D506-15。https://doi.org/10.1093/nar/gky1049.
- 26。
Mitchell Al,Attwood TK,Babbitt PC,Blum M,Bork P,桥A等。Interpro在2019年:改善覆盖范围,分类和访问蛋白质序列注释。核酸RES。2018; 47:D351-60。https://doi.org/10.1093/nar/gky1100.
- 27。
Ceroni A,Passerini A,Vullo A,Frasconi P.二核:二硫键键合状态和半胱氨酸连接预测服务器。核酸RES。2006; 34:W177-81。https://doi.org/10.1093/nar/gkl266.
- 28。
Kozlowski LP。IPC - 等电点计算器。Biol Direct。2016; 11:55。https://doi.org/10.1186/s13062-016-0159-9..
- 29。
孔L,Ranganathan S. Tandem重复,循环排列,分子适应:Solanaceae如何通过抑制剂抵抗害虫。BMC生物信息学。2008; 9(4):S22。https://doi.org/10.1186/1471-2105-9-S1-S22.
- 30
Jongsma Ma,Beekwiller J.昆虫蛋白酶和植物蛋白酶抑制剂的共同演化。Curr蛋白Pept Sci。2011; 12:437-47。https://doi.org/10.2174/138920311796391115..
- 31
Flechtner-Mors M,Thoma U,Wittmann R,Boehm Bo,Mors M,Steinacker JM等。土豆苷蛋白酶抑制剂II对减少体重减轻时胃肠激素和人类饱腹感的影响。糖尿病,Metab Syndr互惠互联网。2020; 13:521-34。https://doi.org/10.2147/dmso.s201853.
致谢
我们感谢Sheon Mary博士,格拉斯哥大学,英国,为修改手稿的英语语言的建议。
资金
该工作得到了科学和工业理事会的支持,下,在印度政府新德里的农作物保护,植物保护肽(NCP)。资金机构提供了财政支持,在设计,收集,分析和解释方面没有作用,以及编写稿件。
作者信息
从属关系
贡献
尼基开发的数据库和网站,准备了稿件;NSS进行分析,解释结果和准备的稿件;APG构思了这个想法,计划和监督整体工作,并用所有作者的评论写了论文。所有作者都读过并批准了稿件。
通讯作者
道德声明
道德认可和参与同意
不适用。
同意出版物
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
斯普林格自然保持中立,就管辖权的要求,在出版的地图和机构的联系。
补充信息
附加文件1:图S1。
PIN-II型PIS的分布。图S2。I型和II型接头区域的发生。图S3。氨基酸在Pin-II pi中的百分比分布。表S1。有关在线数据库中的PIN-II类型PI序列的可用信息。表S2。PINIR数据库中实现的详细表结构。表S3。PiniR数据库中的IRDS发生。表S4。鸟类的物种分布(发生频率>10)。表S5。PIN-II类型PI系列的域架构。表S6。多域PinⅡPIs的物种分布。表S7。连接区域的属级分布。表S8。PIN-II PIS中I型接头区域的物种分布。表S9。PinⅡPIsⅡ型连接区的物种分布。表S10。Rcl的发生和分配。表S11。在IRDS中的二硫键建筑。表S12。根据DSBond类型的分布和与链接器类型的相关性。表S13。鸟类在dsBond型中的属分布。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Yadav,N.K.,Saikhedkar,N.S.&Giri,A.P.Pinir:Pin-II型蛋白酶抑制剂的综合信息资源。BMC植物BIOL.21,267(2021)。https://doi.org/10.1186/s12870-021-03027-0
收到:
认可的:
发表:
关键词
- 蛋白质序列分析
- PinⅡ型蛋白酶抑制剂
- 数据库
- 知识表示
- 注释
- 数据分析