摘要
背景
胡椒 (辣椒L.)含有不同的辣椒素是世界上种植最广泛的香料。然而,物种之间的极端基因组多样性代表了繁殖辣椒的障碍。
结果
在这里,我们报告了从头基因组组装辣椒“早期的卡尔旺德(非刺激性,ECW)”和“小水果(刺激性,SF)”以及他们的注释。总共,我们组装了2.9 Gb的ECW和SF基因组序列,占估计基因组大小的91%以上。这两个辣椒基因组的结构和功能注释分别产生了大约35000个蛋白质编码基因,其中93%被赋予了假定的功能。新发布的辣椒基因注释与公开发布的辣椒基因注释进行了比较,发现既有共享的基因,也有特异的基因。此外,通过全基因组比对,对核苷酸结合和富亮氨酸重复序列(NLR)基因进行综合分析,发现了5个NLR拷贝数变异(CNV)的显著区域。这些区域的详细比较表明,这些CNVs是由特定基因组内的变异产生的,加速了辣椒nlr的多样性。
结论
该研究揭示了辣椒种质资源中nlr拷贝数变异的进化机制,为辣椒抗病功能基因组学和分子育种提供了新的基因组资源辣椒物种。
背景
胡椒 (辣椒辣椒是最重要的蔬菜作物之一,约有35个品种,能产生有益的分子,如维生素C、色素和辣椒素[1那2那3.那4.].2018年,全球辣椒产量约6000万吨,贸易额约160亿美元[5.].最广泛种植的辣椒品种,辣椒(2n = 24)基因组较大,估计长度超过3.0 Gb [2那3.那4.].目前,四个组装的基因组可用C. annuum.[2那3.那4.那6.].基因组资源,包括转录om,变形和蛋白质,也积累在公共数据库中[7.那8.].尽管如此,仍需要更多的资源来识别基因组和遗传特征,以便深入了解农艺性状和表型变异。
下一代测序(NGS)和长读取测序技术的进步加速了植物基因组的测序和组装。迄今为止,已发表了数百种植物基因组,这些序列代表了育种研究的基本资源[9.那10.].具体来说,利用这些参考基因组以及重测序数据进行了群体遗传学研究,以确定与重要农艺性状相关的基因组变异[11.那12.].然而,由于基因组内的极端差异,一个参考基因组不能代表一个植物物种的全基因库[9.那13.].为了克服这些局限性,泛基因组项目已经在主要农作物如水稻,玉米和番茄进行,构建代表目标物种的全基因库[整合基因组序列的目标14.那15.那16.].这些利用多基因组资源的泛基因组分析可以为植物育种相关的农艺性状(如抗生物和非生物胁迫)提供一个平台[13.].
核苷酸结合,富含亮氨酸重复基因(NLRS)已经被迅速放大,并在植物多样化。NLRS的域结构被分为三个主要部分:一个N-末端结构域包括一个收费/白介素-1受体同源性,卷曲螺旋,或白粉病8,中央核苷酸结合(NB-ARC)结构域的电阻和C末端结构域包括富含亮氨酸重复。特别地,所述保守的NB-ARC结构域主要用于限定NLRS [17.].对nlr进行了广泛的种内和种间比较[18.那19.].例如,64年的一项全物种研究拟南芥泛nlrome揭示了NLR进化的过程,包括NLR域结构的多样化及其在物种内的特定选择模式[18.].尽管胡椒有一个很大的nlr扩展池[6.,这些基因在物种内部的复杂性和变异之前从未被研究过。
在这里,我们报告了两个基因组组装和注释C. annuum.入选品种:甜甜椒“Early Calwonder”(ECW)和辣椒“Small Fruit”(SF)。通过对新组装的和公开获得的辣椒基因组的比较分析,揭示了辣椒种质间的进化关系和基因组变异。基于辣椒参考基因组“Criollo de Morelos-334”(CM334),我们对NLR进行了重新注释,并构建了一个物理的NLR地图,其中包括来自本研究的两个基因组(ECW和SF)和其他公开的基因组C. annuum.Zunla-1和Chiltepin。在NLR图谱中,我们发现辣椒品种中NLR基因的拷贝数存在显著差异。这些区域的广泛比较表明,nlr的拷贝数变异可能是辣椒基因组内特定序列突变积累的结果。新构建的基因组组装和注释,以及NLR图谱,为抗病的功能基因组学和分子育种提供了宝贵的资源辣椒SPP。
结果
基因组测序,装配和注释
组装两个辣椒基因组并通过所述管道注释(补充图。1).使用Illumina Hiseq X-Ten和Novaseq 6000平台,我们生成了460.2 GB的原始序列,分别代表了146.8×和145.8×'早期呼叫者(ecw)'和'小水果(SF)'基因组的覆盖率(补充表2).在去除不必要的读取之后,基于19-MEL频率分布,分别估计基因组大小为ECW和SF的3.14和3.18GB(补充图。2和补充表3.).总共83,882和87,732(〜2.84 GB长度),N50长度为114和110 kB的初始调节组装成44,107和44,731个支架,每个2.88 GB长度的支架,每个支架为ECW和SF的预期基因组大小的91.7%和90.6%分别(表1).我们使用BUSCO在ECW和SF基因组组装中检测到1323(96.2%)和1316(95.7%)保守的单拷贝同源基因[20.,表明与其他辣椒基因组相比,组装质量相当(补充表4.).
基因注释预测ECW和SF中蛋白编码基因分别为35355和35158个(表1)1).这些当中,32983(ECW,93.3%)和32838(SF,93.4%)已经在公共数据库中分配推定的功能描述(表1和补充表5.).在ECW和SF基因组与公开可用的胡椒基因注解的基因的比较揭示注释的基因的长度均胡椒基因组(补充图中相似。3.).使用Busco检测到1,254(91.2%)和1,269(92.3%)保守的单拷贝直肠的注释基因分别在ECW和SF中进行验证(表1和补充表4.).考虑到与公开的辣椒基因组相似的基因结构和基因组组装和注释基因的验证,这些结果表明我们的组装和注释的ECW和SF是可靠的。
重复分析表明,2.64(ECW,84.1%)和2.63GB(SF,82.7%)作为重复序列注释,而仅将1-2%的组装基因组分配为基因。LTR / GYPSY元素代表所有注释重复类型的68.8%(补充表6.),与其他辣椒基因组的重复含量一致[3.].
注释基因的聚类和系统发育分析
来自ECW,SF和5公共可用的基因注释辣椒CM334、Zunla-1和ChiltepinC. annuum.;PI159236在c .摘要;和pbc81C. Bacadatum.)分为35,037组。随后,我们根据每一组的基因数量,将基因分为单拷贝(在所有物种中包含一个基因的集群)、多拷贝(在所有物种中包含多个基因的集群)或其他(图。1a及补充表7.).共11419个(32.6%)组包含单拷贝同源基因(图。1a). ECW和SF的未聚类基因最少(分别为541个[1.5%]和627个[1.8%]),说明大部分蛋白序列与其他辣椒基因注释非常相似(图1)。1一种)。
为了进一步验证进化关系,我们使用辣椒注释中的单拷贝直系同源序列串联构建了一个系统发生树(图。1b)。四个加入C. annuum.与CM334相比,Zunla-1、ECW、SF、Zunla-1和Chiltepin之间的亲缘关系更密切(图3)。1b).更仔细地观察基因簇C. annuum.结果显示,在17,202个集群中共有108,533个基因(每个基因组中约21,700个基因[61%])。1c).在核心簇基因的前20个功能域描述中,涉及防御病原体和发育功能的功能域占主导地位(补充图)。4.).
ECW和SF分别含有750个(2.1%)和873个(2.5%)基因,与其他3个种质不聚类(图2)。1光盘)。其中,含有Nb弧,富含亮氨酸的重复和蛋白激酶结构域的基因最丰富(图。1d、补充表8.).的ECW和SF特定基因也表现出疾病抗性相关的蛋白的富集如晚疫病和TMV抗性蛋白和LRR受体样激酶(补充图基因本体论(GO)分析。5.).此外,在CM334、Zunla-1和Chiltepin中,分别有2,204个(6.1%)、1,672个(4.7%)和2,652个(7.7%)基因未被纳入任何组合,同时也包含大量与病原体防御相关的结构域(补充图)。6.).综上所述,这些观察表明,特定基因库,包括抗病基因,如nlr,在辣椒基因组中发生了动态变化。
NLRS的识别和分类
为了阐明NLR序列,我们在五个辣椒基因组中重新注释了NLR(图。2,补充表9.和补充表10.).鉴定了760(在ECW)和972个NLR(在Chiltepin)之间(补充表10.).其中,ECW和SF具有最小的NLR,分别具有760和761,而CM334和Chiltepin分别具有951和972的最大数量的NLR。随后,我们构建了NLR的系统发育树并确定了它们的亚组。分析还揭示了辣椒甲的NLR子组的CNV。特别是G1和G2,Pepper NLRS中最大的亚组[21.],在所有的供试材料中显示可变的拷贝数(补充表10.).另一方面,GT(包括TIR-NLR (TNL))和G10(被称为古老的自主NLR (ANL)) [22.],有中等数量的NLR亚组和中等拷贝数变异。此外,GR,其包括RPW8型辅助NLR(RNL),和G8,其中包含用于细胞死亡(NRC)辅助组所需的NLRS [23.],有NLRS和拷贝数变异最少的小数字。总的来说,这些结果表明,有一个物种在同一NLR组内种质之间以及群体之间的CNV。
辣椒品种nlr的种内变异
为了准确检测辣椒品种间NLR基因的同源性,我们基于CM334参考基因组和其他4个辣椒品种(ECW、SF、Zunla-1和Chiltepin)构建了一个NLR图谱(图2)。3.和补充表11.).由于这种分析,共有4,278个NLR(总共98.2%的4,357个)被分配给NLR地图。在所有载体中注释的核心NLR Orthologs的数量为1,955(391对,44.9%)(图。3.一种)。除了核心NLR之外,共有1,670个可分子NLR在两个或多个换乘(555对,38.3%)之间共用,只有一个加入中存在的特定NLR数量为161(3.7%),43(1.0%)分别为CM334,ECW,SF,ZUNLA-1和CHILTEPIN的44(1.0%),160(3.7%)和245(5.6%)(图。3.b).基于CM334参考基因组的nrs的染色体分布显示,包括功能性抗性基因在内的nrs在染色体两端都富集,并且亚群位于特定的染色体上(图334)。3.C)。例如,G1和G2中的NLR分别在染色体5和9处富集(补充表12.).
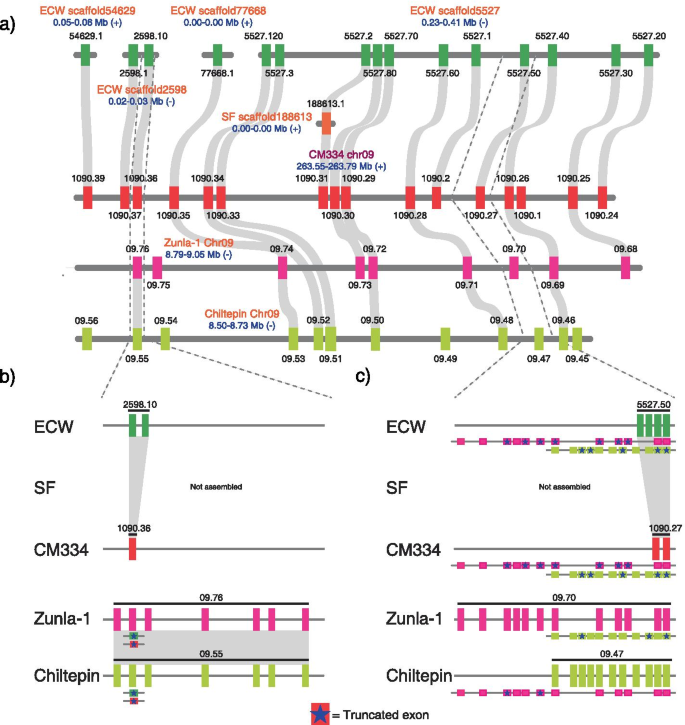
为了识别nrs在辣椒遗传多样性中分布不均匀的区域,我们对nrs基因间区小于50 kb的物理聚类中nrs的数量进行了卡方检验。两个染色体区域和三个支架在NLR中显示出显著的CNVs (Supplementary Table)13.调整后P.值< 0.05)。例如,当我们比较16 NLRs chr09:263.55 - 263.79 Mb的基因组区域丰富的CM334 G2组与相应地区的所有其他登记入册,所有15 NLRs ECW和七NLRs九NLRs Zunla-1直系同源基因在至少一个其他加入。相比之下,我们只在SF基因组中发现了一个NLR,在Chiltepin的12个NLR中有7个在同一区域具有同源基因(图)。4.一种)。这些结果表明,NLR的极端CNV可能是SF和Chiltepin基因组中低NLR拷贝数的结果。
具体而言,CM334中的CA.PGAV.1.6.Caffold1090.36,奇拉-1的ECW,CHR09.76中的ECW.SCOFFOLD2598.10和CHILTEPIN的CHR09.55在SF中的NLR不匹配,因为在此期间省略了相应的区域SF基因组组装。CA.PGAV.1.6.SCAFFOLD1090.36和ECW.SCOFFOLD2598.10的NLRS也与慈悲中ZUNLA-1和CHR09.55中的CHR09.76的NLR匹配,因为7,841bp和8,113bp的大插入位于Zunla-1和Chiltepin基因组分别。因此,这些基因之间的正交关系被破坏(图。4.b)。当我们在CM334和ECW.scaffold2598.10在ECW到Zunla-1和Chiltepin基因组区域映射CA.PGAv.1.6.scaffold1090.36,我们确定了早期终止密码子由于点突变的翻译产生的异常终止(图4.b)。在另一种情况下,我们在CM334和ZUNLA-1中对应于CHR09.70的CM334和ECW基因组区域中鉴定了14个终止密码子(图。4.c).这些终止密码子是点突变和由插入或删除(InDels)组合产生的帧移的结果。将Chiltepin的Chr09.47蛋白与CM334和ECW的基因组区域进行比对,发现了6个点突变和插入导致的终止密码子。相反,每个基因组中的一个NLR被注释在Chr09.70和Chr09.47蛋白被绘制的区域之间。当将Chr09.70和Chr09.47分别定位到每个基因组时,我们分别鉴定出3个和6个点突变的终止密码子。PCR扩增和Sanger测序也证实了小变异介导的早期终止密码子导致nlr蛋白的截断(补充图)。7.).因此,小的基因组变异,如点突变和InDels导致了NLR库的变化。综上所述,这些结果表明,微小的序列变化介导了物种nlr的拷贝数变异。
讨论
最近,植物泛基因组已经取代了参考基因组的作用[13.].然而,高质量的从头基因组组装的数量有限,特别是大型植物基因组,阻碍了泛基因组研究的实施。具体来说,基于低深度测序方法构建的辣椒全基因组[24.在了解辣椒基因组多样性方面仍然有限。在这里,我们通过短读测序提供了两个辣椒品种ECW和SF的基因组片段,其中包含超过100 kb的大型contig N50(见表)1).使用BUSCO对基因组组装和注释的评估表明,两个从头开始的基因组组装和注释的质量足以与其他公开可用的基因组进行进一步分析(补充表)4.).来自单拷贝同源基因的系统发育树与之前的研究略有不同[4.]可能是因为不同的方法,例如最大似然和邻接方法的应用产生了不同的拓扑(图1b)。然而,Zunla-1与其野生祖的最近的关系表明我们的系统发育树代表了合理的拓扑。这些结果表明,这两个新组装的和注释的基因组具有足够的质量,以将基因曲目与其他基因组组件进行比较,并构建泛基因组的染色体水平组装C. annuum.。
通常,可以通过不同的注释方法和资源产生注释偏差,并防止基因的准确对比分析。在该研究中,使用相同的方法进行NLR的重新注释[25.,最终可在5个辣椒基因组中提供改良的NLR资源。系统发育和比较基因组分析表明,在辣椒中复制较多的G1和G2亚群[21.],用大的CNV,不仅形态之后,但也种内分化之后(补充表多元化9.和10.).相反地,GR和G8亚组在所有的供试材料中都被保留(补充表)9.和10.).因为这些组包含与多个传感器NLR相互作用的辅助NLR,以识别病原体效应和介导免疫信号[23.,这些群体中nlr的进化可能受到更严格的调控和保守。此外,我们利用全基因组比对方法构建了NLR图谱,以准确预测NLR的同源关系C. annuum.(无花果。3.).基于NLR地图进行对比分析,确定了NLR拷贝数差异显著的区域C. annuum.。在其他物种中也观察到这种现象,包括拟南芥和番茄18.那19.].然而,CNV区域的详细比较显示,由于基因组序列变化如吲哚和其他突变,NLRS介导的CNVs的截短蛋白质结构(图。4.).这些结果表明,微小的基因组变异对NLR多样化的进化过程至关重要。
根据系统发育树,我们确定了5个具有统计学意义的CNV区域,与NLR组中检测到的更显著的CNV相比,这5个区域似乎是一个小数目(Supplementary Table)10.和13.).这是因为用于构建NLR图谱的辣椒基因组数量有限。通过对NLR基因家族的分析,结合系统发育、同源性和统计检验,可以全面了解NLR的多样性。最近,利用14个多参考基因组和100个不同株系进行了全基因组分析,揭示了番茄基因拷贝数与功能变异之间的关系非烟熏糖基转移酶1(NSGT1),NSGT2[16.].这表明,结合多种策略和更多基因组组合对nrs进行综合分析,可以检测到更多的CNVs,并阐明与基因组变异相关的nrs的进化和功能机制。
结论
综上所述,我们对两个辣椒基因组进行了组装和注释,并利用公开的基因组构建了辣椒NLR基因家族的基因拷贝数。这两个新的辣椒基因组组合、注释和NLR图谱为鉴定功能性抗病基因以及研究辣椒属抗病进化机制提供了宝贵的资源辣椒。
材料和方法
DNA提取和测序
由于辣椒参考基因组(CM334)是一个接近野生种的地方品种,因此选择了2份辣椒材料作为基础资源进行全基因组分析。本研究选用的材料分别是无刺激性的钟形辣椒‘Early Calwonder (ECW, IT158295)’和辣椒素含量较高的‘Small Fruit (SF, IT218615)’。此外,已知ECW品种对细菌斑点病原体(Xanthomonas.SPP。)并用作细菌斑抗性基因的近代源线(Bs1, Bs2, Bs3, Bs4,和BS6)[26.那27.].无论是从国家农业生物多样性中心的RDA-基因库信息中心(NAAS,RDA,韩国)获得。将植物在24℃的16/8小时光照/黑暗周期下在环境控制生长室中生长。从3周龄植物的叶子立即在液氮中冷冻,用于分离基因组DNA。具有高分子量的基因组DNA(gDNA的)从冷冻的叶中提取,和的gDNA的质量通过分光光度分析(DS-11分光光度计; DeNovix Inc.)结果确认和琼脂糖凝胶电泳(1.0%w / v琼脂糖TAE 1X凝胶含1X EcoDye;BIOFACT、大田、韩国)。使用TruSeq DNA Nano Kit(350-、550-和600-800 bp)和Nextera Mate Pair Kit(2-和5-kb)分别构建NGS的配对端(PE)和配对对(MP)文库(Illumina, San Diego, CA, USA)。通过qPCR对各文库的质量进行验证。分别用HiSeqX-ten和NovaSeq6000测序平台(Illumina)对PE和MP文库进行测序。
从头基因组组装
使用“Quality_trim”(-q 20 -m 76)预处理,总共460.2 GB(146.8倍)的ECW和460.2 GB(145.8×)的SF原始数据进行预处理,并在CLC工具V4中实现的“Remove_duppatic”功能。0.6(CLC Bio,Aarhus,丹麦)去除低质量和重复的序列。为了估计基因组大小,使用水母V1.1.5计算19-MER频率[28.]估计基因组大小。在过滤的PE文库中,通过闪光V1.2.2(-m30m-x 0.1 -R 151 -f 300-40)将具有重叠重叠的短读入较长的碎片[29.],然后用Platanus v1.2.4 (-k 71 -c 5 -d 0.3 -t 60 -m 750)组装成初始contigs [30.].随着添加MP文库,支架组件也由Platanus(-L 3-51 -U 0.2-T 30)进行。使用来自两个库的读数,用Platanus(-ed 0.1-T 30)关闭组装间隙。
基因和重复注释
如Kim等人所述,对两个辣椒基因组进行了基因注释[6.,除了SF基因组的转录注释。为了注释ECW基因组,RNA-seq从水果组织中读取[3.]进行比对使用顶帽2.1.1装配基因组[31.和Cufflink v2.2.1 [32.]使用默认设置,以构建转录物,将其用ISGAP [处理33.]鉴定编码序列。公开可用的蛋白质序列C. annuum.CM334 v2.0 (5.),简历。zunla-1 v2.0 [4.), var。glabriusculum.Chiltepin v2.0 [4.],C. Bacadatum.PBC81 v1.2 [6.],c .摘要PI159236 V1.2 [6.),而Solanum lycopersicumItag3.2 [34.]被利用Exonerate v2.2.0定位到ECW和SF基因组[35.].ab-initio.奥古斯都v3.2.3进行了预测[36.使用先前为辣椒基因组构建的训练集[6.].随后,转录组,蛋白质比对和从头开始合并预测,形成最终的ECW基因模型;只有后两者被合并为SF。这些基因模型的功能注释由InterproScan v5.22-61.0 (-f tsv -iprlookup -goterms -appl TIGRFAM, ProDom, SMART, ProSiteProfiles, prositeppatterns, SUPERFAMILY, PRINTS, Pfam)生成[37.]和BLASTp (- value 1e-4 -max_target_seqs 5)使用公开可用的注释数据库,包括RefSeq [38.]和Uniprot / Swiss-prot [39.].为了验证基因组组装和基因注释,我们执行了BUSCO v3.1.0 [20.]在有胚植物1375种保守直向同源物蛋白质(odb10)。我们还比较了基因外显子,内含子的长度分布,并出版辣椒基因注释之间信用违约掉期[4.那6.].
基因注释完成后,使用RepeatMasker v4.0.3 (http://www.repeatmasker.org)默认选项和前一项研究中构建的辣椒基因组重复图书馆[6.].
同源群鉴定及系统发育分析
从七个辣椒聚集蛋白质序列,包括来自这项研究的两个新注释和五个公开的注释C. annuum.cm334 [6.),简历。Zunla-1 [4.],Chiltepin [4.],C. Bacadatum.PBC81 [6.),而c .摘要PI159236 [6.].使用OrthoFinder v2.2.7 (-M msa)连接和对齐单拷贝直方图[40].使用IQ-TREE v1.6.12 (-alrt 1000 -bb 1000 -nt AUTO -safe -blmin 10e-6)导入单拷贝正交序列,构建最大似然树[41.].选择最佳的替代模型作为MOMEFINDER的VT + F + R2 [42在IQ-TREE中实现。正交复制C. annuum.使用TBtools v1.051对物种进行比较,并用维恩图显示[43].中除转座元件相关结构域外,提取了Pfam结构域内容C. annuum.用于功能比较。通过使用BLAST2GO的每次加入中的总基因进行比较来进行那些未灌胃基因的基因本体论(GO)富集分析进行[44].
NLR基因的鉴定与分类
要识别额外的NLS,我们使用TGFAM-Finder V1.20重新注释每个辣椒基因组组件,其中默认参数[25.].简单地说,我们使用了上面描述的相同的基因组组装和注释C. annuum.(CM334, ECW, SF, Zunla-1, Chiltepin)和搜索域。经过六帧翻译后,搜索到包含NB-ARC结构域(PF00931)的100 kb侧翼序列的靶区。Kim等人使用的50种植物的nlr [25.],以及含有NB-ARC结构域各自胡椒注释被用作蛋白质映射资源的蛋白质。RNA-seq的读取由前一报告获得[3.]用于转录组作图。ab-initio.进行基因预测,通过将基因模型与蛋白质取向,组装的转录物组合和从头开始基因预测。
要分配推定的NLR组,由之前研究建立的NLR分类管道[6.那19.那21.在使用时作了一些修改。来自GenBank和植物抗性基因数据库(PRGdb) 3.0版的已知NLR基因(补充表)1)[45,以及Kim等人的NLR组信息[6.],被用作对组分配的参考。使用NLR-PARSER v1.0进行搜索和提取NLR的NB-ARC域(P.-Value Cutoff = 1.9E-5)[46].我们定义了一种完整的NB弧域,其四个主要基序中的至少三个(p环,glpl,Kinase2和mhdv)排列在序列阶和至少160个氨基酸的长度中。使用Mafft V7.407( - - - 曲线1000-globalpair)对齐这些完整的NB弧域[47],并使用trimAl v1.4去除对齐序列中间隙大于92%的位置。rev22 [48].从IQ-Tree V1.6.12推断出最大似然性系统发育关系[41.]具有超快速引导(UFBoot)[49] 1000(-bb 1000 -alrt 1000 -safe)。使用MODEFINDER选择替代模型[42在IQ-TREE中实现。最佳拟合模型是JTT + F + R7。基于已知的NLR基因,UFBoot值> 90%和先前分配的组信息分配了完整NLR组[6.].对于部分无完整NB-ARC结构域的NLR,使用完整NLR组和BLASTp (- value 1e-4)划分推定NLR组。与完整nrs的NB-ARC域相比,匹配数量超过50%相似度和30%覆盖率的组被分配到部分nrs中。
NLR图的构建和CNV分析区域的提取
NLR地图是使用ppsPCP v1.0和默认参数构建的[50].nlr的假定位置由NUCmer的输出和在MUMmer v4.0.0beta2中实现的delta-filter(-1选项)分配,这是ppsPCP管道的一部分。那些既不固定在NLR图谱上,也不特定于每个加入的基因被过滤以进行下游分析。在50kb内重叠的nlr被定义为使用bedtools v2.25.0中实现的“合并”功能的物理集群[51].基于物理聚类,使用Get_homologues-est v1.0的迭代来预测原始关系,具有-m -c -a -t 0选项[52].使用GGPLOT进行可视化框和镦粗地块[53]和tbtools v1.051 [43], 分别。在RSTATIX中的“CHISQ_TEST”函数中,通过CHI-Square测试检测了含有显着CNV的物理NLR簇(标准偏差> 2)。在RSTATIX中的“CHISQ_TEST”函数中进行虚假发现速率(FDR)校正(https://cran.r-project.org/web/packages/rstatix/index.html) v0.6.0和“p。一种D.just” function in R (https://www.R-project.org/)v3.6.3,分别。使用Perl模块进行NLR子群的富集和FDR校正“数学:: GSL :: CDF”(https://metacpan.org/pod/math ::csl:cdf.)和“统计::多元”(https://metacpan.org/pod/Statistics::Multtest),分别。NLR地图使用Circos v0.69-9绘制[54].基于NLR图中的显着CNV区,通过在JCVI包中实施的“JCVI.Graphics.Synte”函数提取并绘制了每个辣椒释放中的染色体或支架中的同期区域的NLR。55]并简化使用Illustrator。
通过PCR扩增和Sanger测序确认nlr中的CNVs
采用CM334、ECW和Zunla-1的gDNA进行PCR扩增。1) CA.PGAv.1.6.scaffold1090.36, ECW.scaffold2598.10, Zunla-1和Zunla-1的同位段,2)Zunla-1的Chr09.70以及CM334和ECW中相应的片段设计了两套引物:5 ' -CAGTTCCCACAAGAAGCTAAAAGAC-3 ', 5 ' - gttaaatgagctaaagctactgagttttg -3 '和5 ' -CAGCAACGTAGAAAACAATACCTAAG-3 '和5 ' - caccatatagttag -3 ';5 ' -CCTTGATTGATGCCGAGATTAG-3 ', 5 ' - gaatagagtgttttctgatc -3 '和5 ' - ccactactaattgactcagaaag -3 ', 5 ' - ccttactctatactcaaatttctac -3 '和5 ' - ccttactactactcaaatttctac -3 '。每个引物的特异性通过BLASTn搜索得到(- value 1)。30 s, 98°C变性步骤;15 s, 58°C退火步骤;70 s, 68°C延伸步骤,35个循环)。PCR产物装入1%琼脂糖凝胶中,使用商品化试剂盒(Cosmo GENETECH®)进行Sanger测序。
数据和材料的可用性
将测序的原始数据沉积到NCBI序列读取存档(SRA)中。登录号为ECW(SRR10007904至SRR10007908)和SF(SRR10007830至SRR10007834)的SRP119199。最终组装的基因组和注释可在Genbank获得,根据“加入号码VYZY01000000(ECW)和VYZZ01000000(SF)”,可以从我们的网站下载(http://peppergenome.snu.ac.kr).额外的数据集和脚本存储在GitHub (https://github.com/sdaf11111/NLR-map-in-pepper).
缩写
- ANL:
-
古代和自主NLR
- CM334:
-
克里奥罗·德·莫洛雷斯- 334
- CNV:
-
拷贝数变异
- ECW:
-
早期的呼叫者
- InDel:
-
插入或删除
- 议员:
-
Mate-pair
- 米娅:
-
几百万年前
- NB-ARC:
-
Nucleotide-binding
- NGS:
-
新一代测序
- NLR:
-
核苷酸结合和富亮氨酸重复基因
- 美国核管理委员会:
-
细胞死亡所需的NLR
- 体育:
-
Paired-end
- RNL:
-
RPW8-type助手NLR
- SF:
-
小水果
- SNP:
-
单核苷酸多态性
- TNL:
-
人数/ il - 1 receptor-NLR
参考文献
- 1。
carzoo - garcia C, Barfuss MHJ, Sehr EM, Barboza GE, Samuel R, Moscone EA等。辣椒的系统发育关系、多样性和扩展(辣椒茄科)。安机器人。2016;118(1):35-51。
- 2。
Hulse-Kemp Am,Maheshwari S,Stoffel K,Hill Ta,Jaffe D,Williams SR等。3.5-GB基因组的参考质量组件辣椒从一个链接阅读库。Hortic杂志2018;彼前。
- 3。
Kim S, Park M, Yeom SI, Kim YM, Lee JM, Lee HA,等。辣椒的基因组序列为辣椒的辛辣性进化提供了深刻的见解辣椒物种。Nat麝猫。2014;46(3):270 - 8。
- 4。
秦超,于春春,沈友,方晓东,陈磊,闵建民,等。对栽培辣椒和野生辣椒的全基因组测序提供了深入了解辣椒驯化和专业化。Proc Natl Acad Sci U S A. 2014; 111(14):5135-40。
- 5.
FAOSTATF。粮食和农业数据。2019.
- 6.
Kim S, Park J, Yeom SI, Kim YM, Seo E, Kim KT,等。新的辣椒参考基因组序列揭示了植物抗病基因通过逆转录复制的大规模进化。基因组医学杂志。2017;18:210。
- 7.
刘福,俞蜂,邓yt,郑俊,刘ml,ou lj等。Pepperhub,辣椒研究界的信息枢纽。莫尔植物。2017; 10(8):1129-32。
- 8.
Kim MS, Kim S, Jeon J, Kim KT, Lee HA, Lee HY,等。辣椒果实器官和病原菌感染的基因表达谱分析辣椒Sci Data. 2018;5:180103。
- 9.
Wendel JF, Jackson SA, Meyers BC, Wing RA。植物基因组结构的进化。基因组医学杂志。2016;17:37。
- 10。
陈飞,董伟,张建文,郭学勇,陈建华,王志军,等。被子植物基因组测序和基因组数据库。植物学报。2018;9:418。
- 11.
杨志强,杨志强,杨志强,等。利用全基因组测序进行全基因组关联研究,快速识别影响水稻农艺性状的新基因。Nat麝猫。2016;48(8):927 - 34。
- 12.
梁志强,段绍生,朱胜,倪旭,邵建军,等。472的全基因组重测序vitis.葡萄园多样性和人口统计历史分析的过程。NAT Communce。2019; 10(1):1190。
- 13。
拜耳PE,Golicz AA,Scheben A,Batley J,Edwards D.植物泛术是新参考。Nat植物。2020; 6(8):914-20。
- 14。
王天宇,王志强,王志强,等。玉米全基因组序列锚点的高分辨率遗传定位。Nat Commun。2015;6:6914。
- 15.
赵Q,冯Q,鲁HY,李勇,王甲,田QL等。泛基因组分析强调在栽培稻和野生稻基因组变异的程度。NAT Genet。2018; 50(2):278-84。
- 16.
王小龙,王小龙,张磊,等。广泛分布的结构变异对番茄基因表达和作物改良的主要影响。细胞。2020;182 (1):145 - 61. - e123。
- 17.
de Araújo AC, Fonseca FCDA, Cotta MG, Alves GSC, Miller RNG。植物NLR受体蛋白及其在生物胁迫持久遗传抗性发展中的潜力。生物技术学报。2019;3(1):80-94。
- 18.
Van de Weyer AL, Monteiro F, Furzer OJ, Nishimura MT, Cevik V, Witek K, et AL .;一个物种范围内的NLR基因和等位基因的清单拟南芥。细胞。2019;178 (5):1260 - 72. - e1214。
- 19.
Seong K,Seo E,Witek K,Li M,Staskawicz B. NLR抗性基因在野生番茄物种中具有非碳N-末端域的抗性基因的演变。新植物。2020; 227(5):1530-43。
- 20。
Simao FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝同源性评估基因组组装和注释完整性。生物信息学,2015,31(19):3210 - 2。
- 21。
徐e, Kim S, Yeom SI, Choi D.全基因组比较分析揭示了茄科植物中核苷酸结合富亮氨酸重复基因家族的动态进化。植物学报2016;7:1205。
- 22。
李海英,孟赫,崔娥,徐烨,金ms,吴胜,等。辣椒免疫受体的全基因组功能分析揭示了种子植物中一个自主的NLR分支。新植醇。2020;229(1):532 - 47岁。
- 23。
Wu CH, Abd-El-Haliem A, Bozkurt TO, Belhaj K, Terauchi R, Vossen JH, et al.;NLR网络介导了对多种植物病原体的免疫。中国科学(d辑:地球科学)2017;
- 24。
OU L,Li D,LV J,Chen W,Zhang Z,Li X等。培养辣椒的泛基因组(辣椒)及其在基因存在-缺失变异分析中的应用。新植醇。2018;220(2):360 - 3。
- 25。
金淑君,郑克,朴J,金ms,金j,徐MK,等。TGFam-Finder:植物靶基因家族注释的新方法。新植醇。2020;227(5):1568 - 81。
- 26。
Stall RE, Jones JB, Minsavage GV。番茄和辣椒对引起细菌性斑点病的黄单胞菌的抗性持久性。植物病理学杂志2009;47:265-84。
- 27.
Parisi M,Alioto D,Tripodi P. Pepper中的生物应激概述(辣椒SPP。):遗传性,分子育种和基因组学的来源。int j mol sci。2020; 21(7):2587。
- 28.
一种快速、无锁的k-mers并行计数方法。生物信息学。2011;27(6):764 - 70。
- 29.
短读序列的快速长度调整以改进基因组组装。生物信息学。2011;27(21):2957 - 63。
- 30.
Kajitani R,Toshimoto K,Noguchi H,Toyoda A,Ogura Y,Okuno M,等。高效的德诺维从组装全基因组鸟枪法高度杂合基因组的短读数。Genome Res。2014; 24(8):1384至1395年。
- 31.
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg SL。TOPHAT2:在存在插入,缺失和基因融合的情况下,转录om的精确对准。基因组Biol。2013; 14(4):R36。
- 32。
特拉普奈尔C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ,等。RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28(5):511 - 5。
- 33。
Kim S, Kim MS, Kim YM, Yeom SI, Cheong K, Kim KT,等。综合结构注释德诺维RNA-SEQ提供洋葱巨大基因组的准确参考基因组(Allium Cepa.L.)。DNA Res。2015; 22(1):19-27。
- 34。
番茄基因组联盟。番茄基因组序列提供了深入了解肉质果进化。自然。2012; 485:635-41。
- 35。
生物序列比较启发式算法的自动生成。BMC生物信息学。2005;航班。
- 36。
斯坦克M,Keller O,Gunduz I,Hayes A,Waack S,Morgenstern B. Augustus:从头开始预测替代转录本。核酸学报2006;34:W435-9。
- 37。
李文智,张海英,李文智,等。InterProScan 5:基因组级蛋白质功能分类。生物信息学。2014;30(9):1236 - 40。
- 38。
O 'Leary NA, Wright MW, Brister JR, Ciufo S, Haddad D, McVeigh R,等。NCBI的参考序列(RefSeq)数据库:现状、分类扩展和功能注释。44(D1): D733-45。
- 39.
Bateman A,Martin MJ,果园S,Magrane M,Alpi E,Bely B等人。Uniprot:蛋白质知识的全球中心。核酸RES。2019; 47(D1):D506-15。
- 40.
Emms DM, Kelly S. OrthoFinder:比较基因组学的系统发育直系学推断。基因组医学杂志。2019;20(1):238。
- 41.
阮LT,施密特HA,冯Haeseler A,胡志明BQ。IQ-TREE:用于估计最大似然系统发育的快速且有效的随机算法。分子生物学EVOL。2015; 32(1):268-74。
- 42.
Kalyaanamoorthy S, Minh BQ, Wong TKF, von Haeseler A, Jermiin LS。ModelFinder:用于精确系统发育估计的快速模型选择。Nat方法。2017;14(6):587 - 9。
- 43.
陈超,陈洪,张勇,何勇,等。TBtools:一个用于大生物数据交互分析的综合工具。摩尔。2020;13(8):1194 - 202。
- 44。
格茨S,加西亚-Gomez的JM,固醇Ĵ,威廉姆斯TD,Nagaraj SH,Nueda MJ,等人。高通量功能注释和数据挖掘与Blast2GO套件。核酸RES。2008; 36(10):3420-35。
- 45。
Osuna-Cruz CM, Paytuvi-Gallart A, Di Donato A, Sundesha V, Andolfo G, Aiese Cigliano R, et al.;PRGdb 3.0:植物抗病基因预测与分析的综合平台。核酸Res. 2018;46(D1): D1197-201。
- 46。
Steuernagel B,Jupe F,Witek K,Jones JD,Wulff BB。NLR-PARSER:植物NLR补充的快速注释。生物信息学。2015; 31(10):1665-7。
- 47。
kaoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。生物医学进展。2013;30(4):772-80。
- 48。
Capella-Gutierrez S, Silla-Martinez JM, Gabaldon T. trimAl:大规模系统发育分析中的自动对齐修剪工具。生物信息学,2009,25(15):1972 - 3。
- 49。
黄德辉,陈瑞敏,冯海泽勒。UFBoot2:改进超快速引导近似。生物医学进展。2018;35(2):518-22。
- 50。
Tahir UL Qamar M,Zhu X,xing F,Chen LL。PPSPCP:植物存在/缺席变体扫描仪和泛基因组建筑管道。生物信息学。2019; 35(20):4156-8。
- 51。
昆兰AR, Hall IM。BEDTools:用于比较基因组特征的一套灵活的工具。生物信息学,2010,26(6):841 - 2。
- 52.
Contreras-Mietira B,Cantalapiedra CP,Garcia-Pereira MJ,Gordon SP,Vogel JP,Igartua E等。Get_homologue-EST的植物泛基因组和转录om,植物的分析,一种相同物种序列的聚类溶液。前植物SCI。2017; 8:184。
- 53.
Wickham H. GGPLOT2:用于数据分析的优雅图形。1 ed。纽约:Springer-Verlag;2016年。
- 54.
王志强,王志强,王志强,等。Circos:比较基因组学的信息美学。基因组研究》2009;19(9):1639 - 45。
- 55.
唐H,鲍尔斯JE,王X明R,阿拉姆男,帕特森啊。同线性和共线性在植物基因组。科学。2008; 320(5875):486-8。
确认
作者欣赏李博士提供GDNA样本C. annuum.简历。Zunla-1。
资金
本研究得到了美国农业部下一代生物绿色21计划农业基因组中心的资助。PJ013153)和韩国国家研究基金会(NRF)授予由韩国政府(2018号r1a5a1023599 (SRC))特区,和2020年首尔大学的研究基金”栏目论文资助机构没有参与研究设计、数据收集、分析,和准备的手稿。
作者信息
隶属关系
贡献
s.k.和D.C.构思了这个项目,设计了内容,并组织了手稿。J.K.和h.g.m.准备了DNA样本。S.K, m.s k, G.Y.C和D.C.进行了从头基因组组装、基因和重复注释和技术验证。m.s k进行了NLR重新注释、分类和其他下游分析。s.o进行PCR扩增和Sanger测序。M.-S.K G.Y.C。,S.O j.k., H.-G.M, S.K,特区写的手稿。所有作者均已阅读并批准本稿件。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称没有相互竞争的利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
勾勒出基因组组装和注释的工作流程。图S2。在两种辣椒品种中分布19-MER频率。图S3。5个不同辣椒品种基因模型的比较。图S4。含有CM334,ECW,SF,ZUNLA-1和CHILTEPIN共享的功能结构域的前20个最高数量的基因辣椒。图S5。辣椒加入中未灌胃基因的基因本体学富集分析。图S6。含有CM334、Chiltepin和Zunla-1特异功能域的基因数量。图S7。nlr CNV的PCR和测序验证。
附加文件2:表S1。
本研究参考抗病基因。表S2。本研究中使用的原始数据的统计数据。表S3。ECW和SF的K-mer频率。表S4。使用Busco验证基因组组装和基因注释。表S5。使用已发表的注释数据库比较预测基因。表S6。重复注释摘要。表S7。每个物种的直系数。表S8。含有ECW和SF功能结构域的特定基因的数量C. Annuum。表S9。分配的NLR组和NB-ACC类型。表S10。分类NLRS的统计数据C. Annuum。表S11。5个样本之间的邻位对nlrC. Annuum。表S12。每1 MB窗口丰富NLR组统计。表向。nlr的重要CNV区域列表。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Kim, MS., Chae, G.Y., Oh, S。et al。De Novo Genomes的比较分析显示辣椒中NLR的动态内差异。BMC植物杂志21,247(2021)。https://doi.org/10.1186/s12870-021-03057-8.
已收到:
公认:
发表:
关键字
- 辣椒
- 基因组
- 抗病性
- NLR进化
- 拷贝数变异