摘要
背景
低温引起的冷胁迫是制约水稻生产的重要因素。鉴定能在寒冷环境中稳定表达的耐冷基因对水稻分子育种至关重要。
结果
在本研究中,我们采用高通量定量性状位点测序(QTL-seq)分析了一个460个个体的F2:3定位群体,鉴定水稻苗期耐冷性的主要QTL基因组区。一个新的主要QTL (qCTS6)在9°C/10 d低温条件下的存活率(SR)被定位在第6号染色体的2.60 mb区间上。设计了27个单核苷酸多态性(SNP)标记qCST6基于重新排序数据的区域,并使用传统的联系分析进行本地QTL映射。最终,我们映射了qCTS6其中7个预测基因包含13个非同义SNP位点。定量反转录PCR分析结果显示,只有Os06g0719500OsbZIP54转录因子,受冷胁迫强烈诱导。单倍型分析证实+376 bp (T >a)在大肠杆菌中OsbZIP54编码区在水稻耐冷性调控中起着关键作用。
结论
我们确定了OsbZIP54作为一种新的水稻冷响应性状调控基因,其Dongfu-104等位基因表现出特异性的冷诱导表达,是水稻改良的重要分子变异。这一结果有望进一步探索水稻苗期耐冷性的遗传机制,并通过标记辅助选择提高水稻品种的耐冷性。
背景
大米(栽培稻是一种适应热带气候的重要粮食作物。因此,高纬度高海拔地区水稻的寒害已成为制约其生产的重要因素[1].特别是在营养生长的早期阶段,冷却直接导致幼苗的生理损害,从而阻碍植物的营养形成并最终影响产量[2].因此,解决这个问题的关键是探讨具有强大优点的抗耐受性(CT)基因,这些优点是有助于最终改善植物CT的等位基因。
水稻中的CT是由多种遗传和环境因素控制的定量特征。在前20年中,使用CT品种开发了许多双倍单倍体线,背交近交系,重组近交系和近代型线,如东乡野生稻[3.],kunmingxiaobaigu [4],丽江内丘[5],Koshihikari [6], M202 [7]和Norin-PL8 [8],并在12条染色体上鉴定出>250个ct相关qtl [9].迄今为止,已鉴定出水稻苗期CT (CTS)的80个qtl [2,10,11,12].此外,与CTS连锁的qtl在原居群中大多定位在10 ~ 30 cM范围内,使得分子标记辅助育种难度较大。因此,解决这一问题的关键是构建精细映射种群。到目前为止,qCTS4[13],qCtss11[14],QSCT1[15],qSCT11[15),而qLOP2 / qPSR2-1[16]已经精细映射,只有七个基因响应CTS被基于地图的克隆(COLD1,qCTS-9,QPSR10,bZIP73,HAN1,OsLTPL159和QCST10).COLD1[17]编码G蛋白信号调节剂,过表达能显著改善水稻CT,而无表达或低表达的水稻系对冷敏感。的启动子区域qCTS-9[18]具有明显与低温耐受性相关的诱导标记,其过表达增加了低温的米差。QPSR10[19]在全基因组关联研究中检测到,其CT单倍型在粳稻资源中发现。bZIP73[1在粳稻和籼稻基因组上有一个单核苷酸多态性(SNP),这个SNP决定了水稻对低温的耐受性。HAN1[20.]调控低温反应和茉莉酸介导的CTS,是CTS的负调控因子。OsLTPL159[21.编码非特异性脂质转移蛋白,其编码序列(CDS)位于映射区域QCST10[22.].这些例子表明,CTS的分子调控极其复杂,尽管这些研究为了解CT在早稻生长中的分子机制奠定了基础,但对水稻CT背后的遗传学的剖析仍处于起步阶段。通过双亲本交联定位鉴定这些qtl是一个耗时费力的过程,包括对分离群体中大量个体的基因型进行定位;因此,要获得可靠的qtl,需要通过精细定位、基因克隆、功能验证,最终应用于分子标记辅助育种来完成CTS改良。
新一代测序(NGS)技术的快速发展,使体积分离分析(BSA)与NGS的结合成为挖掘qtl和基因的重要方法[23.,24.].与传统的连锁作图相比,QTL测序(QTL-seq)可以提高工作效率,提供高密度变异。QTL-seq已成功应用于多种植物,包括黄瓜[25.,大豆26.)、大米(27.,西红柿[28.].虽然该技术能够快速定位主要QTL,但置信区间(CI)分辨率仍低于最佳。因此,研究人员不得不将QTL-seq与精细mapping结合起来[29.]及RNA-seq [30.]为了鉴定候选基因的QTL。例如,CaqSW1.1[31.通过QTL-SEQ和典型的QTL映射从1.37 MB映射到35 kB,以允许识别最终候选基因。另外,四种候选基因(SLCATHB2,SLGST,SLUBC5,和刹车1.)与番茄耐热性相关的QTL-seq和RNA-seq检测[28.].因此,利用用于通过QTL-SEQ识别主要QTL间隔的组合方法和挖掘级靶基因的微型映射提供了新的视角。
本研究采用QTL-seq和精细定位两种方法鉴定水稻CTS基因。F2:3从Dongnong430(冷敏感品种)和Dongfu104(强CT品种)之间的交叉衍生的人口用于QTL-SEQ分析,和qCTS6被定位在第6号染色体的2.60 mb基因组区域。此外,我们使用精细映射策略来锚定qCTS6在这7个基因区间内鉴定了13个非同义SNPs (nSNPs),并预测它们会导致氨基酸变化。定量反转录(qRT)-PCR和单倍型分析表明OsbZIP54+376-bp (T>A)和+376-bp (T>ADongfu104.可以认为是中国北方粳稻罕见的功能变异。这些结果从分子水平揭示了水稻冷敏感转化为CT的机制,提高了对水稻冷敏感转化机制的认识。
结果
CT评分的筛选和评价
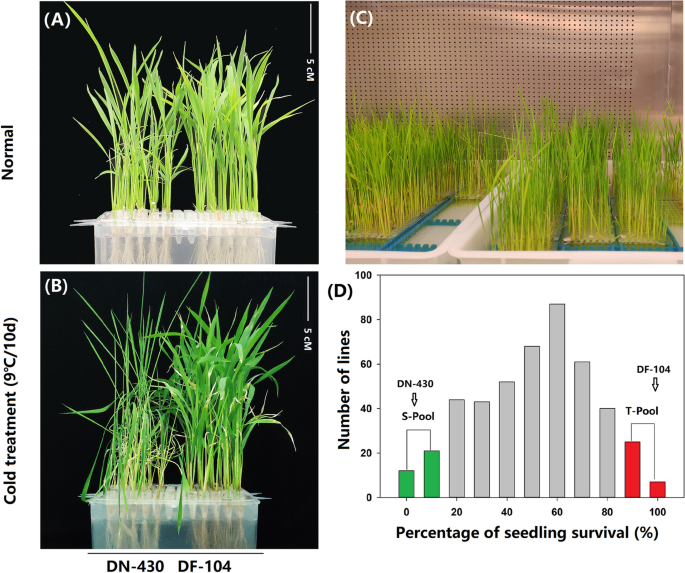
亲本品种DN430和DF104(图2)。1a,b)以及他们的460 f2:3行(无花果。1c),在9°C / 10天的正常和寒冷条件下进行评估。CT-FINDIAL DF104的SR显着高于冷敏感DN430的SR(图。1B),说明DF104的CT强于DN430。在F2:3人口,与SR相关的偏斜和峰度的绝对值接近1(表S1),表示适用于QTL分析的数据。而且,F2:3种群在0 ~ 100之间表现出不同的SRs。1D显示在460个株系中,鉴定出30个CT株系和30个冷敏感株系。然后选择它们分别制备CT (t池)和敏感池(s池),然后用于DNA重测序。
全基因组重测序和QTL-seq分析
亲本和两个库的平均基因组覆盖深度为50×。与‘Nipponbare’参考基因组比较,4个样本共获得786,407个SNPs和133,992个ints。经过修剪和过滤,得到521,630个SNPs和85,824个indelet(表2)S2).从这些单核苷酸多态性中筛选出356,831个亲本纯合且亲本间存在多态性的高质量单核苷酸多态性,用于进一步的QTL-seq分析。
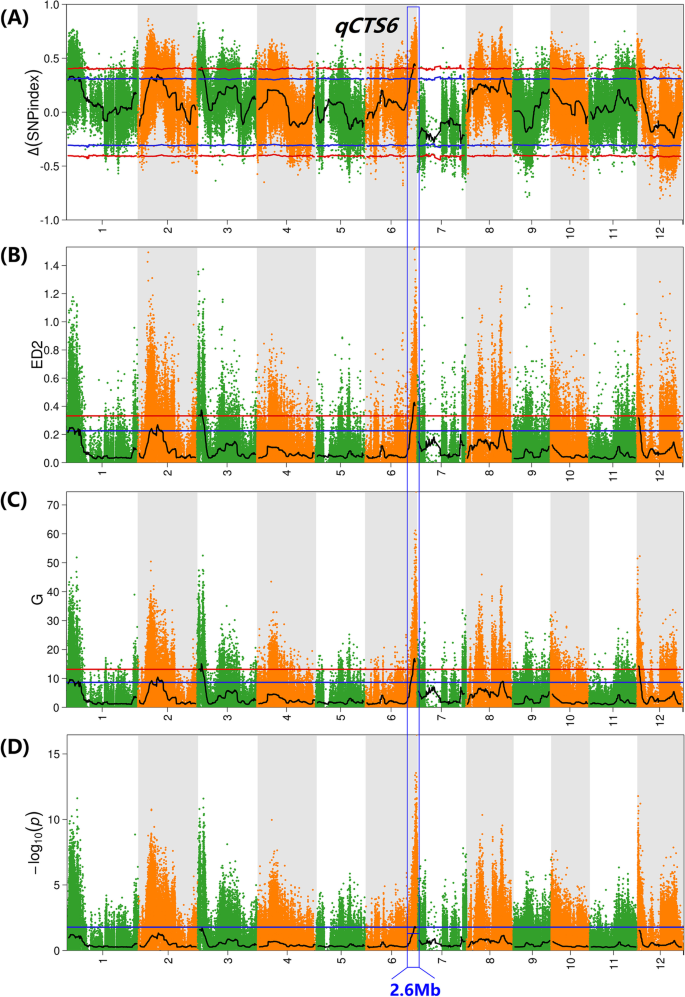
对于所有得到的snp,∆(SNP-Index)(图。2A)、欧几里得距离(ED)(图。2b),g值(图。2C)和Fisher的精确检验(图。2D)用来拦截关联间隔。然后,我们在水稻基因组中鉴定了与CTS相关的候选区域(见表)1).对于∆(SNP-Index), CTS在染色体3和6上有两个主峰(P< 0.01),跨度为2.58 Mb (1.50-4.08 Mb)和3.24 Mb (28.00-31.24 Mb)。ED算法给出的显著峰完全覆盖了∆((SNP-Index)算法返回的结果,随后对两个主要区间进行命名qCTS3和qCTS6.G算法类似地捕获了前两个算法的结果,并在染色体12中识别了另一种新的间隔(0.00-2.42 MB)(命名QCST12).只有qCTS6是四种方法应用时得到的。通过比较四种方法的关联结果,其基因组区域的交点与Fisher精确检验确定的关联区域一致。共发现2.60 MbqCTS6时间间隔。值得注意的是,在三种算法中,由qCTS6比qCTS3(桌子S3),表明两种混合池的等位基因比例存在显著差异。因此,qCTS6被认为是探索候选基因的最重要的CTS靶点。据统计分析,qCTS6编码区含有407个SNPs和18个indeleS4).
缩小的qCTS6到一个优秀的地区
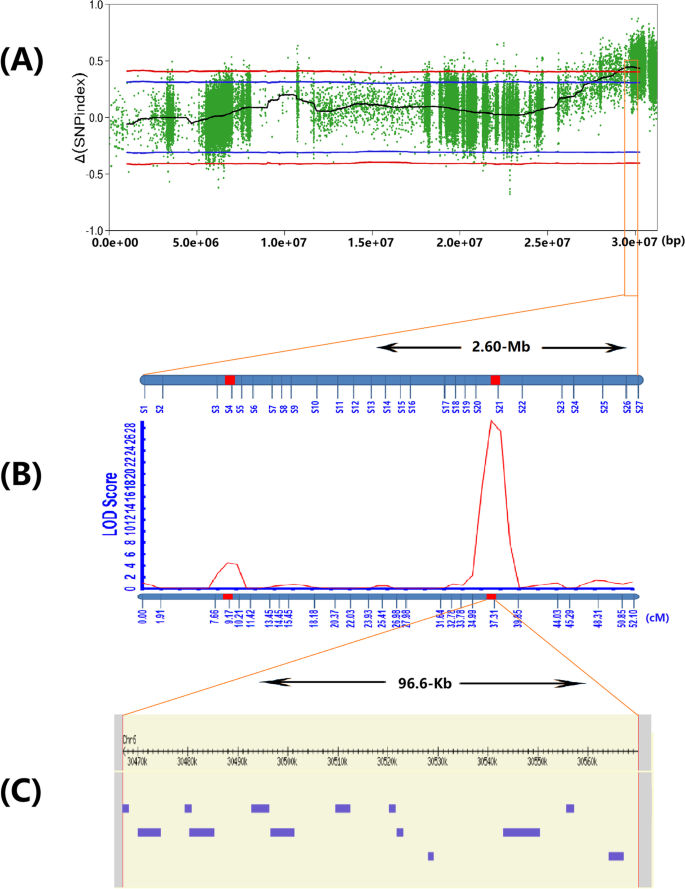
因为2.60-MB区域包含大量基因,所以我们进行了精细的映射qCTS6基于2.60-MB区域的基础变化信息,以识别可能与根据以下标准相关联的有效SNP的有效SNP:Δ(SNP-index)值> 0.48,这将显着高于统计上限“没有QTLS的零点假设”下的CISP< 0.01水平”;所选择的SNP代表了均匀分布在该区域内的非同义突变。我们最终捕获了27个优秀的nsnpqCTS6对460个株系进行基因型扫描,得到aqCTS6联系地图(图。3.b)。我们使用了几个赔率(LOD)值> 3.0的对数作为连续事件的阈值来确定QTL的存在。然后,我们通过连杆测绘在冷应力下分析了460线的幼苗Sr。qCTS6与Sr同时连接并锚定到30,467,391bp和30,563,973bp之间的96.6-kb间隔(图。3.B),从而解释了SR中表型变异的25.83%(表2).正等位基因qCTS6由DF104贡献,CT6优化为96.6 kb的物理间隔。其他效应较小的QTL区间(29,739,644 ~ 29,771,312 bp)由于LOD值较低,未用于后续分析,占表型变异的3.17%。
推测的候选基因qCTS6
从Nipponbare基因组注释信息中截取96.6 kb区间(http://rice.plantbiology.msu.edu/),从而捕获13个注释基因(图。3.C)。除了FON1[32.]或功能基因在克隆花器官的数量,没有评估其他基因。以往的研究报道了重要的基因或基因家族,如编码伪尿嘧啶合酶家族蛋白的基因[33.], C3HC4 [34.]假设蛋白质[18,蛋白磷酸酶2C [35.]和Bzip家族转录因子(TF)[36.,37.,38.,与水稻或其他作物的CT有关。虽然在13个候选基因中包含了相似的基因或蛋白质,但从基因本体论(GO)分析中获得的10个基因并没有标注“对寒冷的反应”。与生物过程、分子功能和细胞组分本体相关的最丰富的术语分别是代谢过程(即GO:0044267、GO:0008152、GO:0034641)、催化活性(即GO:0003824、GO:0140096)和细胞(即GO:0005623、GO:0044464)S5和图S1).此外,在染色体上的96.6-kB区域,位于父母线和δ(SNP-INDEX),G值,ED和双尾渔民的精确测试之间的染色体6的缔蔽24的哈博尔24 SNP。P的值高于统计置信度P< 0.01。此外,89个SNPs和18个插入位点位于基因间区、内含子区、未翻译区、上游和下游区域。13个基因中有7个携带13个nSNPs(见表)S6),以亲本和亲本为参照,鉴定出4个nSNPs,影响4个候选基因编码伪尿嘧啶合成酶(Os06g0717400)、蛋白磷酸酶2C (Os06g0717800)、一个假想蛋白(Os06g0718000)和一个bZIP TF (Os06g0719500)(表3.).如上所述,这些基因可能在响应植物中的冷凝压力方面发挥重要作用。基于该发现,我们使用以下三种方法鉴定了四种可能的候选基因:基因反应和冷应激之间的可能关系,蛋白质编码区域中的不同义突变,以及在特定代谢途径中的基因产物的参与影响ct。
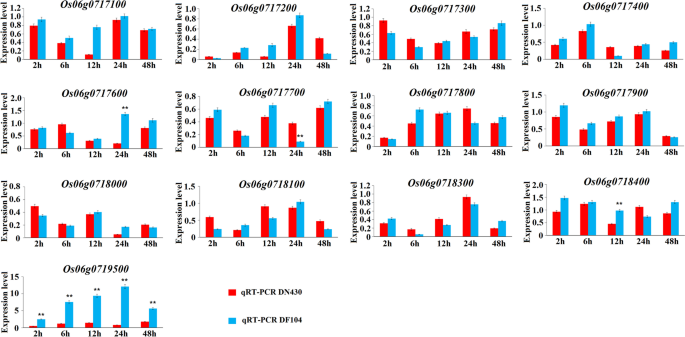
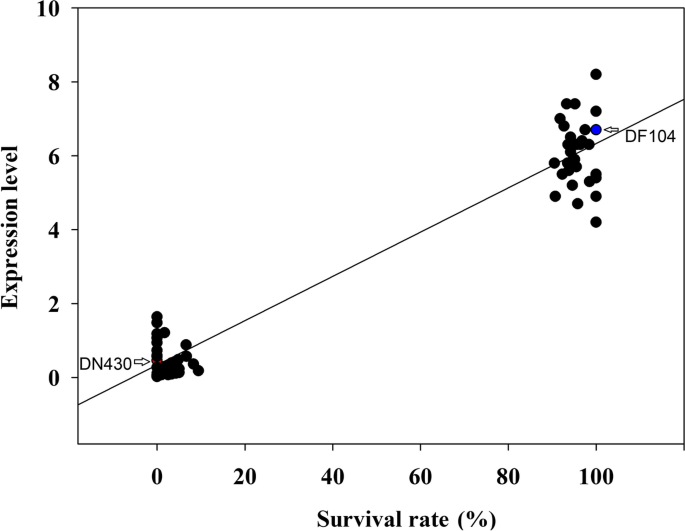
来识别强候选基因qCTS6,我们分析了13个基因在冷胁迫和正常条件下的表达水平,发现有4个基因在冷胁迫下表达水平发生了显著变化。其中,Os06g0761700、Os06g0717700和Os06g0718400在单个时间点上差异显著,只有Os06g0719500在CT极强的亲本中表现出强烈的冷应激反应诱导(图2)。4)并表明OS06G0719500作为可能的候选基因qCTS6.相比之下,在正常条件下,所有13个候选基因显示出表达没有显着变化(图S2).值得注意的是,OS06G0719500编码Bzip系列TF注释为OsbZIP54,带有SNP标记snp - 630563973位于这个编码区域的。此外,这60个细胞系的mRNA水平与CT之间存在较强的相关性OsbZIP54在CT(无花果。5).这些发现表明OsbZIP54作为可能的候选CT基因qCTS6进行进一步分析。
候选基因单倍型分析
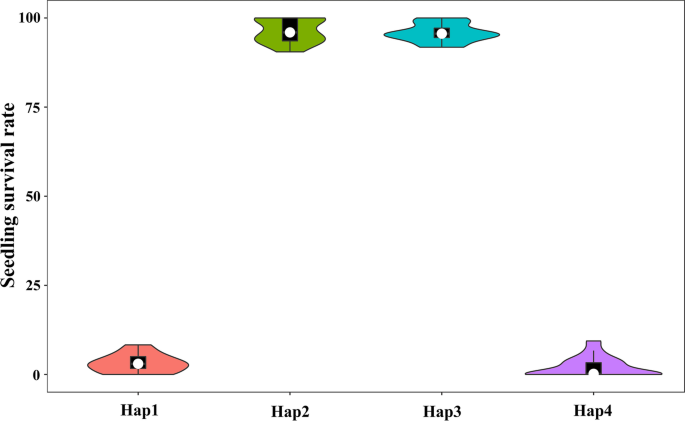
来揭示变化的重要性OsbZIP54编码区域,我们分别从T-Pool和S池中选择了60行,并测序它们OsbZIP54Sanger测序的基因。我们确定了两个SNP(SNP-30563973和SNP-30565007)OsbZIP54外显子和内含子(图S3)分别在两个父母之间的启动子区内没有检测到多态性(图S4).为了确认单倍型与所研究性状的关联,我们生成了小提琴图来评估单倍型群内的性状分布(图1)。630个CT和30个冷敏感品系在+376-bp (T>A)编码区分离一致OsbZIP54(桌子S7),提示这可能导致CT的表型差异。另外,另一个SNP SNP (SNP-30565007)与CT无相关性OsbZIP54从粳稻品种DF-104中分离得到。为了弄清+376-bp (T>A)在水稻中的分布,我们利用3010水稻基因组工程的数据发现,只有275个粳稻基因组携带+376-bp (T>A),而籼稻基因组携带>(1600个)。的比例OsbZIP54籼稻和粳稻的功能变异为5.8:1,表明+376 bp (T>A)在粳稻中较籼稻为罕见变异。我们随后分析了变异类型OsbZIP54在295米北粳稻(表S8),在样本中未发现+376-bp (T>A)的实例。提示+376 bp (T>A)为自然变异;因此,+376-bp (T>A)可作为DF104中一种特殊罕见的功能变异。但是,也不能排除其他的日本OsbZIP54可能存在类似的单碱基变异。
讨论
大米是世界上约50%人口的主食。39.].由于起源于热带和亚热带地区,水稻对冷胁迫比其他谷类作物更敏感。值得注意的是,在温带和亚热带地区以及高海拔地区,水稻幼苗对早春的低温十分敏感,导致幼苗生长迟缓、变黄、枯萎、分蘖减少、生长受阻[2].要找到CT的主要QTL,我们使用了几种方法来获得最可靠的QTL间隔。首先,我们使用大群人在低温条件下识别它们的表型,从而获得每条线的精确CT表型。其次,使用具有严格的CT表型差异的两种DNA池进行QTL-SEQ分析。此外,我们使用了四种生物信息学分析方法来在99%的意义水平下映射QTL区域。最后,两个非常重要的山峰(qCTS3和qCTS6),前者定位在1.50 Mb ~ 4.08 Mb之间,后者定位在28.44 Mb ~ 31.04 Mb之间。QTL的QLVG3.根据‘Nipponbare’参考基因组(第3染色体,5.92-Mb,距离qCTS3)[40].qCTS3位于1,500,000至4,080,000 - BP地区,CDPK13[41.] 和OsCDPK13[42.]在该地区检测到。在染色体6中,位置的位置qCTS6在目前的研究完全重叠的区域,以前的报道QLVG6.并在萌芽期与CT连结[40].此外,qRCT6b,控制生殖期CT的QTL,距离约23.00-MbqCTS6.此外,我们发现没有与染色体6上的幼苗阶段相关的CT QTL,并且在此内没有报告冷响应基因qCTS6时间间隔。值得注意的是,实现三种算法[δ(SNP-index),ED和G值]导致峰值值较高qCTS6(桌子S3)比那还多qCTS3.此外,与qCTS6,qCTS3与费希尔的测试结果无关。这表明在两个混合池之间存在显著的等位基因比例差异。因此,我们认为qCTS6作为CTS和候选基因探索的最重要靶点。
采用传统的QTL作图与QTL-seq相结合的方法,对水稻进行了QTL作图Cf-10基因到790千克地区枝孢属fulvum[43.),而GAFZL.被定位到一个包含7个注释基因的70kb区域[44.].因此,我们认为结合QTL-seq和传统连锁定位来发现非常狭窄的候选区域的策略非常适合快速靶向目标基因。来筛选潜在的候选基因qCTS6间隔,我们组合QTL-SEQ和精细映射,这使得能够降低候选基因的数量qCTS6QTL-SEQ从393到13基因定义的间隔。在这13个基因中,七个具有13个功能基础变化。此外,表达水平OsbZIP54在两个亲本中均表现为冷处理后强烈诱导。表达式的分析OsbZIP54在60个线中,极端CT的差异显示CT和表达水平之间的相关性(图。5).此外,序列分析发现在编码序列区域有两个SNP差异OsbZIP54,其中+ 376-bp(t> a)是功能变体,并且在确定CT中的差异时发挥着重要作用(表S7).然而,作为一种新发现的CT调节剂,其信号转导系统及其下游通路OsbZIP54尚不清楚。DF104携带一种罕见的外显子SNPOsbZIP54在寒冷条件下高度诱导,表明SNP可能参与转录调节OsbZIP54,这项保证进一步探索。有趣的是,尽管表达了强烈的表达OsbZIP54冷应激诱导,DF-104和DN-430的启动子不含多态性。之前的研究报告说bZIP73在编码区域的511-bp位置存在SNP差异,决定了水稻的CT [36.].在本研究中,我们未发现多态性bzip54启动子。然而,另一项研究表明,BZIP73-BZIP71相互作用可能有助于转录激活bZIP73,这就给了水稻一个冷信号开关。这提供了一个可能的原因上调OsbZIP54表情是冷的压力。然而,表达水平CTB4a在冷应激下受启动子突变和功能性SNP的影响[45.];因此,水稻应对冷胁迫的遗传机制是复杂的,基因在冷胁迫下表达的显著变化并不仅仅由其各自的启动子决定。下一步,我们将进一步克隆候选基因的等位基因,构建植物和酵母的表达载体,并在水稻原生质体、酵母和酵母中进行表达拟南芥比较这种天然等位基因变异对冷应力下基因表达和亚细胞定位的影响。生物学功能OsbZIP54其相关的基因组变异需要利用基因编辑和高效过表达转化系统来确认。尽管我们发现了Os09g0444200中的nSNP与所研究的表型性状之间的相关性,但这不足以将一个CT表型归因于单个SNP。相比之下,我们也认为一个关键基因的有利等位变异不足以提供CT,植物的最终CT表型应该被认为是不同关键基因的有利等位变异组合的结果。
CT是由多个基因控制的复杂性状[46.,这些分离出来的基因可以分为两种不同的类型。一种包括具有特定功能的基因,这些基因参与系统代谢以抵御寒冷压力[47.].例如,OsLTPL159增强水稻苗期CT,降低活性氧毒害作用,增强细胞壁纤维素沉积,促进渗透物质积累[21.].此外,CTB4a,是一种富含亮氨酸的重复受体样激酶,通过与ATP合酶β亚基相互作用,正调控ATP活性和含量,从而提高花粉的育性[48.].第二类包括在应激反应中调控基因表达的基因(即信号元件和转录因子)[47.].与其靶基因一起,TFS可以作为信号转导的调节因子,以激活或抑制参与冷应激反应的基因。因此,TFS是用于改性作物复杂性状的优异候选者[49.].相反,在水稻中有91个bZIP家族成员基因[50.,其中几个涉及到CT的调控。值得注意的是,作为一种转录激活剂,其表达OsbZIP52在低温(4°C)下强诱导[51.].其他bzips,如bZIP73,与bZIP71为了调节脱落酸水平和活性氧的平衡,从而提高水稻的耐受性低温[36.].然而,在北方粳稻中,bZIP TFs在CT中的潜力仍有待探索。在本研究中,我们发现了一种罕见的变异OsbZIP54在中国北方粳稻中,295个品种未发生单倍型事件S8).此外,3010米基因组项目数据库显示OsbZIP54与籼稻相比,在粳稻中占有率非常低。一种罕见的胸腺嘧啶变异OsbZIP54在DF104中发现外显子,可作为改进CT的基因组标记。此外,还探讨了分子间相互作用的机理OsbZIP54及其有针对性的启动子元素,下游基因和辅助因子需要进一步调查。
结论
综上所述,研究紫茎泽兰的遗传基础对其育种具有重要意义。在本研究中,我们使用QTL-seq和连锁图谱分析来鉴定qCTS6qtl与米饭ct相关。通过QRT-PCR和单倍型分析进行实验验证OsbZIP54在qCTS6区域作为候选基因。结果表明,该等位基因具有特异的冷诱导表达模式,可作为水稻改良的重要分子标记。
材料和方法
植物材料
以抗寒母本东农430 (DN430)和抗寒父本东福104 (DF104) 2个粳稻品种为亲本,分别配制了460 F2:3人口。这些材料来源于东北农业大学(中国哈尔滨)。
CTS评估
种子在40°C孵育~36 h打破休眠,然后在30°C去离子水中浸泡~72 h发芽。将萌发后的种子移栽到96孔PCR板中,在温室环境(白天26°C /夜间22°C,光照12小时/光照12小时,相对湿度80%)中生长。当幼苗长成三叶时,在培养箱中低温处理9°C/10 d,均为460华氏度2:3幼苗在28°C条件下恢复生长10天,研究每个重复60株幼苗的存活率(测量幼苗总成活率相对于总被试数量的百分比)。进行了3次生物重复。
建造隔离池
所有年轻的叶子为460 f2采用十六烷基三甲基溴化铵法分别采集个体进行总基因组DNA提取[52.,稍作修改。选择30个极CT (ECT)和30个极冷敏感(ECS)个体的基因组DNA作为2个大群体2:3人口(范围:0 - 100%)。[为了简化下面的描述,我们将DF104缩写为T, DN430缩写为S]。使用Nanodrop 2000分光光度计(Thermo Fisher Scientific, Waltham, MA, USA)对分离的DNA进行定量。所有来自30株ECT和30株ECS植物的DNA都在Qubit 2.0荧光仪上进行了定量(Life Technologies, Carlsbad, CA, USA),并将相同数量的来自ECT和ECS植物的DNA混合。
NGS测序和BSA-seq分析
从膨胀的池中萃取总基因组DNA,使用至少3μg基因组DNA构建使用配对末端DNA样品制备试剂盒(Illumina,San Diego,Ca,USA)的嵌件尺寸为500bp的配对末端文库.在Genedenovo(中国广州)的Hiseq X10(Illumina)NGS平台进行测序这些文库。质量修剪是生成高置信变体调用的重要步骤。加工原始读取以根据三种严格的过滤标准获得高质量的清洁读数:1)除以≥10%的未识别核苷酸的读数;2)用> 50%碱基覆盖读取质量评分≤20;3)读取与条形码适配器对齐。
为了识别SNPs和indele,将过滤后的reads与Nipponbare参考基因组序列进行比对[53.]使用带有参数'Mem-M'的挖掘机轮式对准器(V.0.7.16A-R1181);-m是用于标记较短的拆分对齐作为辅校准的选项[54.].使用GATK unifiedgene型进行变异调用(v.3.5;https://gatk.broadintitute.org/hc/en-us/community/posts/360073637011-unifiedgenotyper-in-gatk4.).SNPs和indele使用符合适当标准的GATK variantfiltering功能进行过滤(-Window 4, -filter“QD < 4.0 || FS > 60.0 || MQ < 40.0”,-G_filter“GQ < 20”)。使用ANNOVAR对基因和功能以及基因组区域的所有突变进行注释[55.].使用SNP-Index进行关联分析[23.),∆(SNP-Index) [56., G统计量的计算[57.,58.], ED [59.,以及双尾Fisher精确检验[60.基于单核苷酸多态性。使用步长为20kb的2000kb滑动窗口计算平均值,并丢弃小于10 SNP/Indel的窗口。如果snp数量不足,则将此窗口的结果合并到下一个窗口。将四种方法的重叠区间作为CT的最终QTL区间。
发展SNP标记,缩小候选区间
为进一步筛选QTL-seq序列,筛选出27个峰显著的nSNPsqCST6在四个关联 - 分析算法下的区域[δ(SNP-INDEX),G值,ED和FISHER的精确测试],被认为是用于精细映射的标记。Primer Premier 5.0(Palo Altimity,Palo Alto,CA,USA)用于设计底漆(表S9.).选择所有标记物用于父母之间的多态性,并且使用多晶型标记物进行460个体的基因分型,该多态性标记用于构建连杆地图并使用QTL ICIMAPPT的ICIM模块缩小候选区域(V.4.2;http://www.isbreeding.net).宣布显著QTL存在的LOD评分阈值通过1000个重复的排列检验确定P< 0.01。
CTS候选基因的预测
为了预测CTS可能的候选基因,我们综合考虑了以下三种策略。首先,利用亲本全基因组DNA重测序数据库进行候选基因预测,比较QTL区域内基因的DNA序列。在QTL区域,我们重点研究了两个亲本之间具有非同义突变snp的开放阅读框。其次,根据其功能注释重新选择成功率最高的候选人,用于筛选水稻基因组数据库(http://rice.plantbiology.msu.edu/).第三,GO [61.]基因和基因组的京都百科全书[62.]数据库作为参考,利用BLAST软件评价候选基因[63.].
候选基因表达的验证
在三次重复的冷处理后在2小时,6小时,12h,24h和48小时内收集DN430和DF104的幼苗叶,在液氮中冷冻,并在-80℃下储存直至总RNA提取。还收集控制植物并类似地储存。QRT-PCR用于量化冷处理下候选基因的表达水平。使用Trizol试剂(Thermo Fisher Scientific)从水稻组织中提取总RNA,并用DNase I处理,以消除任何DNA污染。通过电泳评估RNA质量,并在-80℃下储存直至使用。根据Primescript RT Master Mix(Takara BioMedical Technology(北京),北京,中国)的说明,合成第一链cDNA(10μl)。引物设计有引物总理v.5.0(总理Biosoft International),并列于表格中S9..家务基因actin1(Os05g36290)用作内部控制[64.]和QRT-PCR是使用带有2×SYBR Green I PCR主混合物(Thermo Fisher Scientific)的肌腱2.10系统(Roche,Basel)(Roche,Basel)进行QRT-PCR。如前所述进行QRT-PCR分析[65.].
候选基因的多样性分析
对候选基因的单倍型进行了分析,结果如下:30个t库、30个s库和295个中国北方粳稻。295个水稻品种来自中国的黑龙江、吉林和辽宁,以及其他国家,包括日本、朝鲜、朝鲜民主主义人民共和国和俄罗斯[66.].设计候选基因在亲本间SNP位点的引物序列,对60个株系序列的DNA片段进行PCR扩增,以确定目的基因分布的单倍型统计量。所有特异性引物序列见表S2.PCR反应液总体积为20 μL,其中正向引物(10 μm) 1.5 μL,反向引物(10 μm) 1.5 μL,基因组DNA (50 ng/μL) 2 μL, ddH 5 μL2o和10μl的PFU主混音(CWBIO,北京,中国),包括TAQ DNA聚合酶,PCR缓冲液,MG2+和核苷酸。PCR反应在Eppendorf 5333 Mastercycler (Eppendorf, Hamburg, Germany)中进行,使用与qRT-PCR相同的方法[65.].产物经1%琼脂糖凝胶电泳检测。PCR产物由中国深圳华大基因生命科技有限公司进行直接测序。单倍型性状数据绘制在ggplot2 R包中(ggplot2:用于数据分析的优雅图形;美国纽约施普林格)。
统计分析
用SPSS18.0软件检测亲本和子代的CT差异。数据代表平均值±标准差。利用SigmaPlot软件(v.12.5;Systat Software, San Jose, CA, USA)。
可用性数据和材料
本研究的QTL-seq数据可在美国国家生物技术信息中心序列阅读档案中找到,登录号为SSR13319856、SSR13319857、SSR13319858和SSR13319859 (https://www.ncbi.nlm.nih.gov/bioproject/prjna688381.).
缩写
- BSA:
-
膨胀的分离分析
- CD:
-
编码序列
- 置信区间:
-
置信区间
- CT:
-
耐寒性
- CTS:
-
苗期耐冷性好
- 艾德:
-
欧氏距离
- ICIM:
-
包含复合区间映射
- LOD:
-
对数的赔率
- NSNP:
-
非同义单核苷酸多态性
- 存在:
-
定量逆转录PCR
- QTL:
-
定量特质基因座
- SNP:
-
单核苷酸多态性
- SR:
-
存活率
- TF:
-
转录因子
参考文献
- 1.
Liu CT, Schlappi MR, Mao BG, Wang W, Wang AJ, Chu CC. bZIP73转录因子对水稻生殖期耐冷性的调控。植物学报;2019;17(9):1834-49。
- 2.
Andaya VC, Mackill DJ。水稻营养期耐冷性相关qtl的定位J Exp Bot. 2003;54(392): 2579-85。
- 3.
毛德辉,于林,陈德志,李丽丽,朱应祥,肖玉强,等。多个抗寒基因座表明东乡野生稻对高纬度生境具有较高的耐寒性。acta photonica sinica . 2015;128(7): 1359-71。
- 4.
Dai Ly,Lin Xh,Ye Cr,Ise K,Saito K,Kato A等。昆明雄黄云南地兰对生殖阶段耐寒耐寒耐寒地点的定量特质基因座。品种sci。2004; 54(3):253-8。
- 5。
Shirasawa S,Endo T,Nakagomi K,Yamaguchi M,Nishio T.一种QTL区域控制耐寒阶段的QTL区域,在稻米,“丽江欣朱尼格”,米西族苜蓿中的玉米苜蓿。2012; 124(5):937-46。
- 6。
Endo T, Chiba B, Wagatsuma K, Saeki K, Ando T, Shomura A, et al.;水稻品种‘库科奇’耐冷性QTL的检测及QTL积累效应acta physica sinica, 2016;129(3): 631-40。
- 7.
水稻幼苗耐冷性QTL qCTS12的精细定位。应用电子学报。2006;113(3):467-75。
- 8.
Saito K,Hayano Saito Y,Kuroki M,Sato Y.基于地图的水稻冷耐耐菌基因CTB1的克隆。植物SCI。2010; 179(1-2):97-102。
- 9.
杨利民,刘海林,赵宏文,王建国,孙军,郑海林,等。水稻孕穗期耐冷性的数量性状位点定位及meta分析Euphytica。2019;215(5):89。
- 10.
韩L,乔y,张某,张y,cao g,kim j等。鉴定水稻幼苗活力冷应性状的定量特性基因座。j遗传学基因组学。2007; 34(3):239-46。
- 11.
Suh JP, Lee CK, Lee JH, Kim JJ, Kim SM, Cho YC,等。利用粳稻与热带粳稻杂交衍生的RILs鉴定幼苗耐冷性数量性状位点。Euphytica。2012;184(1):101 - 8。
- 12.
楼强,陈林,孙震,邢勇,李军,徐旭,等。一个与水稻苗期耐冷性相关的QTL。Euphytica。2007;158(2):87 - 94。
- 13。
安雅省VC,Tai Th。与稻米幼苗耐寒相关的QCTS4Locus的精细映射(Oryza Satival。)。mol品种。2007; 20(4):349-58。
- 14。
关键词:野生稻,数量性状,苗期耐冷性中国生物医学工程学报。2010;34(1):45-54。
- 15.
Kim S,Suh J,Lee C,Lee J,Kim Y,Jena KK。QTL测绘和开发与水稻幼苗耐寒相关的候选基因衍生的DNA标志物(Oryza Sativa L.)。Mol Gen Genomics。2014; 289(3):333-43。
- 16。
萧,黄某,李啊,gao y,li yh,pan ch,等。QLOP2和QPSR2-1基因座的精细映射与野生米幼苗的冷胁迫耐受性相关。Al Appl Genet。2015; 128(1):173-85。
- 17。
孟云,董晓燕,徐云元,刘伟,张晓明,等。COLD1使水稻具有耐冷性。细胞。2015;160(6):1209 - 21所示。
- 18.
赵继,张某,东俊,杨t,毛X,刘Q等。水稻幼苗阶段耐寒性的一种新型功能基因。植物Biotechnol J. 2017; 15(9):1141-8。
- 19.
肖娜,高原,钱慧娟,高强,吴艳艳,张丹萍,等。与耐冷性有关的基因和具有耐冷性的功能等位基因的鉴定。植物杂志。2018;177(3):1108 - 23所示。
- 20.
毛德,Xin Yy,Tan Yj,Hu XJ,Bai Jj,Liu Zy等。Han1基因的自然变化赋予水稻的耐受性,使适应温和气候。pnas。2019; 116(9):3494-501。
- 21.
赵军,王胜,秦军,孙超,刘芳。脂质转移蛋白OsLTPL159与水稻苗期耐冷性的关系。植物学报;2020;18(3):756-69。
- 22.
刘锋,徐伟,宋强,谭磊,刘军,朱振华,等。水稻耐冷性数量性状的微阵列精细定位。摩尔。2013;6(3):757 - 67。
- 23.
Abe A,Kosugi S,Yoshida K,Natsume S,Takagi H,Kanzaki H,等。基因组测序揭示了使用mutmap中的大米中的复杂重要基因座。NAT BIOTECHNOL。2012; 30(2):174-8。
- 24.
Kadambari G, Vemireddy LR, Srividhya A, Nagireddy R, Jena SS, Gandikota M,等。基于qtl - seq的遗传分析鉴定了水稻矮化的一个主要基因组区。植物学报2018;37(4):677-87。
- 25。
卢海峰,林涛,Klein J,王胜国,齐建军,周强,等。QTL-seq鉴定了一个位于黄瓜开花位点T附近的早花QTL。Theor Appl Genet. 2014; 127:1491-9。
- 26。
宋军,李智,刘志新,郭勇,邱丽娟。新一代基因序列分析加速了大豆中两个定性基因的同时鉴定。植物学报2017;8 - 11。
- 27。
Wambugu P,Ndjiondjop Mn,Furtado A,Henry R.块状粒子的测序允许解剖稻米含有直链淀粉含量的遗传控制。植物Biotechnol J. 2018; 16(1):100-10。
- 28。
温建强,姜福林,翁永强,孙明山,史小平,周永忠,等。通过常规QTL定位、QTL-seq和RNA-seq鉴定番茄耐高温QTL和高温胁迫响应基因。植物学报。2019;19(1):1 - 17。
- 29。
张XL,王文,郭,张yy,bu yp,zhao JM等。将QTL-SEQ和联系映射结合到精细图的野生大豆等位基因较大的植物高度。BMC基因组学。2018; 19(1):1-12。
- 30。
Park M,Lee J-H,Han K,Jang S,Han J,Lim J-H等人。使用QTL-SEQ和RNA-SEQ揭示辣椒囊肿中辣椒蛋白生物合成的主要QTL和候选基因。Al Appl Genet。2019; 132(2):515-29。
- 31。
Das S, Upadhyaya HD, Bajaj D, Kujur A, Badoni S, Kumar V,等。利用QTL-seq快速定位鹰嘴豆主要性状相关QTL的候选基因。DNA研究》2015;22(3):193 - 203。
- 32.
Suzaki T, Sato M, Ashikari M, Miyoshi M, Nagato Y, Hirano HY.花器官NUMBER1基因调控水稻花分生组织大小,并编码一个与拟南芥CLAVATA1同源的富含leucine的重复受体激酶。发展。2004;131(22):5649 - 57。
- 33.
林丹,孔瑞,陈丽,王勇,吴丽,徐建军,等。水稻叶绿体低温发育需要伪尿苷合酶基因TCD3。Sci众议员2020;10(1):1 - 11。
- 34.
荣毅杰,李伊赫,诺思,李KD,拉舒特AM,康KK。BrRZFP1:甘蓝c3hc4型RING zinc finger蛋白,参与冷、盐和脱水胁迫。植物医学杂志。2013;15(2):274 - 83。
- 35.
Tähtiharjus,palva t.反义抑制蛋白质磷酸酶2c的抑制在拟南芥中加速了冷驯化。工厂J. 2001; 26(4):461-70。
- 36.
刘超,欧胜,毛斌,唐建军,王伟,王浩,等。bZIP73的早期选择促进了粳稻对寒冷气候的适应。Nat Commun。2018;9(1):1 - 12。
- 37。
刘超,Schläppi MR,毛斌,王伟,王安,楚聪。bZIP 73转录因子对水稻生殖期耐冷性的调控。植物学报;2019;17(9):1834-49。
- 38。
刘c,吴y,王X. bzip转录因子Risbz5:水稻中寒冷和干旱应激反应的潜在负调节因子。足底。2012;235(6):1157 - 69。
- 39。
国际水稻基因组测序项目:对水稻基因组进行完全测序的努力。植物学报。2000;3(2):138-41。
- 40。
韩LZ,张益,乔YL,CAO GL,Zhang Sy,Jong Hwan K,等。稻米萌发低温活力的遗传和QTL分析。Acta Genet Sin。2006; 33(11):998-1006。
- 41。
杨国兴,杨国兴,杨国兴。钙依赖性蛋白激酶13和钙蛋白互作蛋白1的过表达对水稻抗寒性的影响。中国生物医学工程学报。2007;27(6):713-23。
- 42。
OsCDPKI3是水稻钙依赖蛋白激酶基因,是由低温和赤霉素诱导的。植物学报。2004;55(4):541-52。
- 43。
刘克,赵特,你x,江j,李茹,徐x。Cf-10基因的分子映射通过组合SNP / Indel指数和番茄(Solanum Lycopersicum)的联系分析。BMC植物BIOL。2019; 19(1):1-11。
- 44.
冯X,程H,左张D,张Y,王Q,刘k等。DPL972中无模糊基因GAFZL的精细映射和鉴定(Gossypium arboreum)。Al Appl Genet。2019; 132(8):2169-79。
- 45.
张泽,李杰,潘毅,李杰,周l,施高清,等。CTB4A的自然变化增强了对冷栖息地的水稻适应性。NAT Communce。2017; 8(1):1-13。
- 46.
张强,陈庆华,王少林,洪永红,王志林。水稻与冷胁迫:耐冷性相关数量性状位点的评价与总结方法。大米。2014;7(1):1 - 12。
- 47.
黄光谷,马少林,白立平,张磊,马辉,贾鹏,等。植物在冷、盐和干旱胁迫下的信号转导。acta photonica sinica; 2012;39(2): 969-87。
- 48.
张泽,李杰,潘耶,李杰,施高,曾yw等。CTB4A的自然变化增强了对冷栖息地的水稻适应性。NAT Communce。2017; 8(1):1-13。
- 49。
细胞对脱水和冷胁迫的反应和耐受性的转录调控网络。植物学报2006;57:781-803。
- 50。
Kawahara Y,De La Bastide M,Hamilton JP,Kanamori H,McCombie WR,Ouyang S等人。使用下一代序列和光学图数据改进oryza sativa nipponbare参考基因组。米。2013; 6(1):1-10。
- 51。
刘翠翠,吴彦波,王小平。bZIP转录因子OsbZIP52/RISBZ5:水稻寒冷和干旱胁迫反应的潜在负调控因子。足底。2012;235(6):1157 - 69。
- 52。
默里M,汤普森WF。高分子量植物DNA的快速分离。核酸RES。1980; 8(19):4321-6。
- 53。
Matsumoto T,Wu Jz,Kanamori H,Katayose Y,Fujisawa M,Namiki N等人。基于地图的水稻基因组序列。自然。2005; 436(7052):793-800。
- 54。
Kumar S, Agarwal S. Ranvijay:将NGS读取到参考基因组的快速和内存高效方法。中国生物化学学报。2019;17(02):1950008。
- 55。
Wang K, Li MY, Hakonarson H. ANNOVAR:基于高通量测序数据的基因变异功能注释。核酸学报2010;38(16):e164。
- 56.
高木,安倍,吉田,小杉,夏目漱石,三冈,等。QTL-seq:通过对两个大群体的DNA进行全基因组重测序,快速定位水稻数量性状位点。植物j . 2013; 74(1): 174 - 83。
- 57.
Magwene PM, Willis JH, Kelly JK。使用下一代测序的批量分离分析的统计。公共科学图书馆。2011;7(11):e1002255。
- 58.
Mansfeld BN,Grumet R. Qtlseqr:AN R包,用于批量隔离分析,下一代测序。植物基因组。2018; 11(2):1-5。
- 59.
山JT,Demarest BL,BISGROVE BW,戈恩斯B,SU YC,YOST HJ。MMAKAK:突变映射分析管道,用于汇集RNA-SEQ。Genome Res。2013; 23:687-97。
- 60。
fisher ra。论应急表中的χ2的解释,以及P.J r Stat SoC的计算。1922年; 85(1):87-94。
- 61。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM,等。基因本体论:生物学统一的工具。Nat麝猫。2000;得分上以25 - 9胜过25(1):。
- 62。
Kanehisa M,Goto S,Kawashima S,Okuno Y,Hattori M.用于解密基因组的Kegg资源。核酸RES。2004; 32(1):277-80。
- 63。
张建军,张志强,张志强,等。gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报1997;25(17):3389-402。
- 64。
Siahpoosh MR, Sanchez DH, Schlereth A, Scofield GN, Furbank RT, van Dongen JT,等。OsSUT1基因表达修饰调控水稻的盐反应。台北309。植物科学。2012;182:101-11。
- 65。
张震,李军,唐震,孙旭,张华,于军,等。RAWUL蛋白Gnp4/LAX2通过干扰水稻的OsIAA3-OsARF25相互作用,通过生长素信号通路调控粒长。J Exp Bot. 2018;69(20): 4723-37。
- 66。
李恩,郑h,崔j,王j,刘h,sun j等。幼苗粳稻粳稻种质碱性耐碱性的基因组 - 宽协会研究及候选基因分析。米。2019; 12(1):24。
致谢
我们感谢编辑(www.editage.cn.)作英文编辑。
陈述
我们确保本研究中使用的所有水稻种子起源于中国黑龙江省东北农业大学。这些种子的合法性符合IUCN关于研究涉及灭绝风险的研究的政策声明和濒危野生动物群和植物群的贸易公约。在该研究中收集的稻米种子在中国培养了米饭,而不是濒危野生物种。这些品种通过了中国的法律品种认证程序,并获得了生产,种植和市场业务的许可。此外,这些品种是我们实验室水稻种质资源的成员,并且是常规粳稻类型。可以从稻米数据库中检索农艺特征,培养特征和相关品种类型(https://www.ricedata.cn/variety/).
资金
本研究得到了黑龙江省重大科技项目(批准号:)的资助。基金资助:国家自然科学基金资助项目(批准号:2020ZX16B010);2019 m651249)。
作者信息
从属关系
贡献
L.Y.和D.Z.对研究进行了构思和设计;L.L.、j.w.、L.L.参与数据分析;J.W、H.Z、W.X和L.L.进行了材料开发、样品制备和数据分析;L.Y.写了手稿;H.Z .和D.Z.修改了原稿;最终的手稿经过了所有作者的阅读和批准。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明没有利益冲突。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1
.GO注释基因96.6 Kb区域的聚类图。横坐标为GO类别的内容,纵坐标左侧为基因数量。该图显示了GO二级功能在相关区域所有基因背景下的基因分类。图S2.采用qRT-PCR检测13个候选基因在DN430和DF104正常条件下的表达水平。结果采用学生t检验进行统计分析(**,P< 0.01)。图S3.OsbZIP54的CDS区序列差异分析。Os07g0569700的基因结构及OsbZIP54在DF-104和DN-430之间的序列差异。Ref为Nipponbare基因组的参考序列。图S4.启动子序列差分分析OsbZIP54.启动子结构OsbZIP54和序列差异OsbZIP54在DF-104和DN-430之间。Ref为Nipponbare基因组的参考序列。补充表1.样本中检测到的单核苷酸多态性(SNPs)和InDels的数量。补充表2.冷治疗父母和F2:3种群的生存率。补充表3..由四种算法驱动的显著峰值统计量。补充表4..基于重构数据的QCTS6的2.60 MB间隔变化统计。补充表5..GO注释结果为96.6-KB间隔。补充表6..基于重置数据的96.6KB间隔的变化统计。补充表7..Os06g0719500在T-Pool和S-Pool中的单倍型分析。补充表8..295个华北粳稻qCTS6区间基本变异统计补充表9..本研究使用的引物。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
杨,L.,王,J.,Han,Z.等等。将QTL-SEQ和LINKAGE MAPPETPE结合到精细图候选基因qCTS6对水稻苗期耐寒性的影响。BMC植物BIOL.21,278(2021)。https://doi.org/10.1186/s12870-021-03076-5
收到了:
接受:
发表:
关键词
- 栽培稻L.
- 耐寒性
- QTL-SEQ.
- Linkage-mapping
- 幼苗阶段