摘要
中医(TCM)基因组项目旨在揭示草药的遗传信息和监管网络,并阐明其在预防和治疗人类疾病中的分子机制。此外,TCM基因组可以为在中医中发现活性成分的功能基因和中医的育种和改善提供基础。中医基本局部对准搜索工具(TCM-BLAST)是用于TCM蛋白和DNA序列相似性搜索的Web界面。它含有大约40g的TCMS基因组数据,包括36个TCMS的蛋白质和DNA序列,具有高医学价值。在TCM-BLAST网站上托管的公开可访问的TCM基因组对齐数据库(http://viroblast.pungentdb.org.cn/tcm-blast/viroblast.php.)扩展以查询多个序列数据库以获得TCM基因组数据,并提供用户友好的输出,以便于分析和浏览BLAST结果。TCMS的基因组测序有助于阐明重要的次生代谢物的生物合成途径,并为基因发现研究和分子育种提供了基本资源。TCMS基因组在中医临床实践的指导下提供了对来自这些TCMS的新型生物活性化合物和药物的有价值资源。在确定其基因组数据后,我们的数据库可以扩展到其他TCM。
背景
中药基础植物全基因组测序是中药相关基因发现和培育、合成生物学、药物发现和分子育种的重要手段[1那2那3.那4.].基因组序列不仅为基础和应用研究提供了宝贵的资源,而且也为进化和比较基因组学分析,特别是在中医[5.那6.那7.那8.那9.].
实验和临床研究表明,TCMS具有广泛的药理学性质,如抗炎,抗病毒,抗微生物,抗氧化,抗真菌,抗血栓形成,抗渗透性,镇痛,抗糖尿病,抗抑郁药,抗血症和抗癌活动以及免疫调节,抗糖尿病,胃保护,肝保护性,神经保护和心脏保护作用[10那11那12那13那14那15那16那17那18].基因组测序及其注释为通过分子育种改良中医药提供了重要资源[19那20.那21]以及发现利用合成生物学方法制造生物活性化合物的有用基因[1那22那23那24].这些基因组资源的可用性将促进药物和营养重要基因的发现,TCMS的遗传改善[7.那21那25]和新型候选药物的鉴定[26].
草药OMICS数据库(http://herbalplant.ynau.edu.cn/html/Genomes/)只收集了23个已发表的草药基因组,自2019年以来,这些增加的数据没有继续更新。药用植物基因组资源(http://medicinalplantgenomics.msu.edu).抗植物基因组数据(https://blast.ncbi.nlm.nih.gov/blast.cgi?prov = blastn&_type=blastsearch& blast_spec=plants_mv&link_loc=blasttab&last_page=blastp.)包括少数类型的药用植物,并且提供了最常见的可食用植物的基因组比较)。
建筑和内容
TCMS的基因组数据源自草药OMICS数据库(http://herbalplant.ynau.edu.cn/html/Genomes/)、药用植物基因组学资源(http://medicinalplantgenomics.msu.edu),中国科学院基因组学研究所的大数据中心(http://bigd.big.ac.cn/gsa/statistics).
未标注文献的中药材基因组数据来源于http://medicinalplantgenomics.msu.edu/那http://bigd.big.ac.cn/gsa/statistics.
TCM-Blast的部署策略包括实例化提供的病毒母细胞[27将中药基因组比对的核心组件捆绑在一起。在PHP 7.0.32内,已实现一个方便用户的网页界面,以搜寻资料库(http://www.php.net.),部署在Apache 2.4.18 web服务器(http://www.apache.org/)及MySQL数据库服务器(https://www.mysql.com/)使用Ubuntu 16.04服务器(http://mirrors.aliyun.com/ubuntu-releases/16.04/).TCM-Blast有36个中医基因组数据集。
关于TCM基因组数据集的信息总结在TCM-BLAST网站的在线。从草药OMICS数据库中收集TCM-Blast中使用的TCM基因组数据(http://herbalplant.ynau.edu.cn/html/Genomes/)、药用植物基因组学资源(http://medicinalplantgenomics.msu.edu)、北京基因组研究所大数据中心(http://bigd.big.ac.cn/gsa/statistics)(关于三十六个TCMS的基因组数据来源的进一步细节,见表1).这些数据资源被学术机构或政府部门与植物基因数据库合并,发表在专业期刊和植物基因数据库上,数据来源丰富,数据质量可靠。除其他数据资源外,该数据库在我们研究中具有以下优势:1)该数据库是目前最大的中药基因组数据库;2)该数据库包括中药来源的植物遗传数据;3)该数据库为中药育种、栽培和有效成分的发现提供了支持。
实用与讨论
概述TCM-Blast
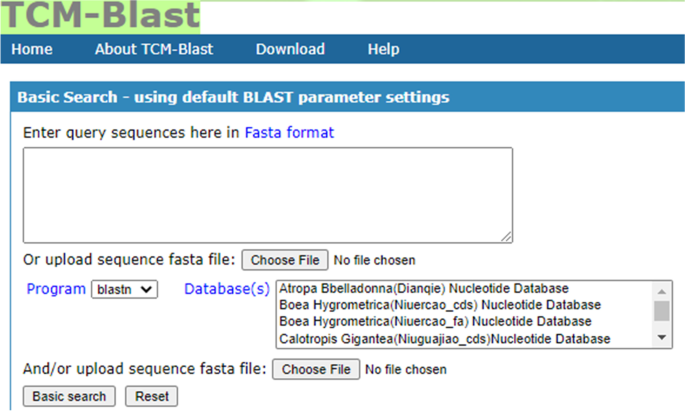
我们已经开发了TCM-Blast,是一种基于Web的TCM基因组对齐数据库(图。1).TCM- BLAST提供了从包括TCM蛋白质和DNA序列数据集在内的TCM基因组数据库中选择的接口,该数据库提供了BLAST实现的查询功能[40].TCM- blast目前包含约40gb的中医基因组数据,包括36种中医的蛋白质和DNA序列。
TCM-BLAST的主电源
用户可以直接将查询序列粘贴到查询框中,也可以将查询序列作为FASTA文件从本地文件上传。TCM- blast提供多个TCM序列数据库。然后用户可以选择特定的TCM基因组数据库来运行不同的程序(blastn, blastp, blastx, tblastn, tblastx)。TCM-Blast包括五种一般BLAST形式[27那41那42那43]对于TCM基因组数据:
blastn:通过核苷酸查询查询TCM核苷酸数据库。
使用蛋白质查询功能搜索中医蛋白质数据库。
blastx:使用翻译的核苷酸查询查询TCM蛋白数据库
tblastn:使用蛋白质查询查询TCM翻译的核苷酸数据库。
Tblastx:使用翻译的核苷酸查询搜索TCM翻译的核苷酸数据库
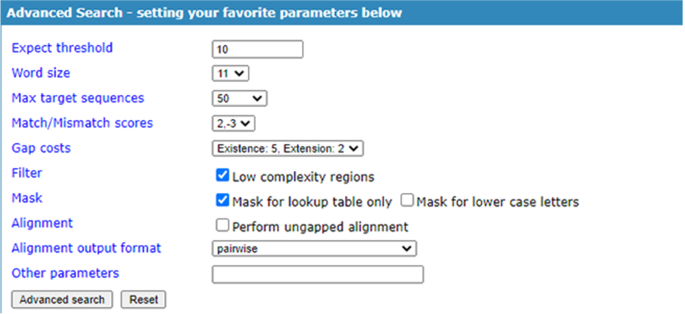
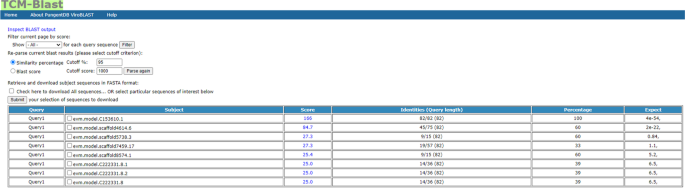
TCM-Blast为需要收集更具体信息的高级用户提供了可选的搜索功能。2),并能够设置不同的参数,如预期阈值、字长、最大目标序列等,以便为用户收集更具体的信息。TCM基因组序列的TCM- blast序列比对结果显示在汇总表中,汇总表中包含查询序列名称、受试者序列名称、受试者来源数据库、位置评分、身份百分比、E值(图1)。3.).
对此数据库的案例研究
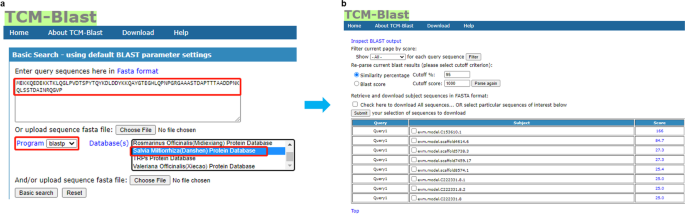
例如,用户可以使用blastp程序选择丹参蛋白数据库,通过输入蛋白序列,得到自己期望的BLAST结果。在无花果。4.,用户已输入蛋白质序列片段:
“Mekkqedekktklqglpvdtspytqykdlddykkqaygtehlqpnpgrgaaastdaptttaaddpnkqlsstdainrqgvp.,在“输入查询序列”框中输入;选择丹参蛋白数据库;,点击“基本搜索”按钮获得BLAST结果。本次搜索得分最高的是“evm.model.C153610.1”主题,说明输入序列片段与丹参蛋白具有很高的相似性。有关此数据库的更详细用例,请参阅补充文件.
未来,我们将收集更多的中医药基因组数据,为中医药研究提供数据支持。
结论
该数据库整合了多种数据库资源,显著提高了中医基因组学研究的效率。该数据库将允许用户对集成的中医基因组序列数据库执行批量序列搜索。因此,TCM- blast为中医药科研提供了全面的中医药基因组资源数据,消除了其他平台存在的潜在冗余。
可用性数据和材料
TCM-Blast是一个免费的数据库和可视化工具,对所有用户开放,无需登录,可以通过以下URL访问:http://viroblast.pungentdb.org.cn/tcm-blast/viroblast.php..这个网络工具在所有现代的网络浏览环境中都可以使用,包括谷歌Chrome, Mozilla Firefox和Safari。所有相关物种的基因组数据都可以下载http://viroblast.pungentdb.org.cn/TCM-Blast/db.
缩写
- 中医:
-
中国传统医学
- DNA:
-
脱氧核糖核酸
- TCM-BLAST:
-
中医基本局部对齐搜索工具
参考资料
- 1.
Mochida K, Sakurai T, Seki H, Yoshida T, Takahagi K, Sawai S, et al.;药用豆科植物甘草(Glycyrrhiza uralensis)基因组组装与注释草案。植物j . 2017; 89(2): 181 - 94。https://doi.org/10.1111/tpj.13385.
- 2.
李志刚,杨涛,杨鹏,等。基于SLAF的枸杞高密度连锁图谱果实大小相关数量性状位点的鉴定植物学报。2020;20(1):1 - 18。https://doi.org/10.1186/s12870-020-02567.1..
- 3.
陈X,李继,王X,钟l,唐y,周x等。全长转录组测序和茉莉酮类诱导的基因的表达谱分析与Pogostemon Cablin的菠萝醇生物合成和调节相关。BMC植物BIOL。2019; 19(1):1-18。https://doi.org/10.1186/s12870-019-1884-x..
- 4.
陈胜,宋建军,孙超,徐建军,朱勇,Verpoorte R,等。草药基因组学:检查传统药物的生物学。科学。2015;347 (6219): S27-S29。https://doi.org/10.17660/ActaHortic.2015.1089.62
- 5.
张关R,赵Y, H,风扇G,刘X,周W, et al。银杏活化石的基因组草图。Gigascience。2016;5 (1):s13742 - 016 - 0154 - 1。https://doi.org/10.1186/s13742-016-0154-1.
- 6.
孙辉,吴胜,张刚,焦超,郭胜,任勇,等。古异源四倍体葫芦属基因组的核型稳定性和无偏分。摩尔。2017;10(10):1293 - 306。https://doi.org/10.1186/s13742-016-0154-1.
- 7.
吴鹏,周超,程胜,吴志,卢伟,韩建军,等。生物柴油植物麻疯树基因组序列及连锁图谱。植物j . 2015; 81(5): 810 - 21所示。https://doi.org/10.1111/tpj.12761.
- 8.
严磊,王旭东,刘洪,田勇,连建,杨锐,等。铁皮石斛(Dendrobium officinale)的基因组揭示了中国重要的传统兰花草本植物的生物学特性。摩尔。2015;8(6):922 - 34。https://doi.org/10.1016/j.molp.2014.12.011.
- 9.
刘勇,曾胜,孙伟,吴敏,胡伟,申旭,等。两种枸杞果实中类胡萝卜素积累的比较分析。中国生物医学工程学报。2014;14(1):1 - 14。https://doi.org/10.1186/s12870-014-0269-4.
- 10。
陈智,曹颖,张颖,乔颖。刺激性中药在特殊器官层面的整体功效的新发现。中华医学杂志。2019;20(3):752。https://doi.org/10.3390/ijms20030752.
- 11.
张F.中医:中国制造。自然。2011; 480(7378):S82-3。https://doi.org/10.1038/480S82a.
- 12.
甘草及其活性成分的药理作用:最新研究进展。Phytother杂志2015;29日(12):1868 - 86。https://doi.org/10.1002/PTR.5487.
- 13.
江W-y。中医治疗智慧:现代科学的视角。趋势Pharmacol SCI。2005; 26(11):558-63。https://doi.org/10.1016/j.tips.2005.09.006.
- 14.
中国计划使传统医学现代化。大自然。2007;446:590-1。https://doi.org/10.1038/446590a.
- 15.
科学AAftAo。传统医学的艺术与科学第一部分:今天的中医——一个整合的案例。科学。2014;346(6216):1569。https://doi.org/10.1126/science.346.6216.1569-D.
- 16.
Xiong X.将中药融入西部心血管医学:一种基于证据的方法。NAT Rev Cardiol。2015; 12(6):374-374。https://doi.org/10.1038/nrcardio.2014.177-c1.
- 17.
田平。会聚:东西方交汇之处。大自然。2011;480 (7378):S84-6。https://doi.org/10.1038/480S84a.
- 18.
赵家,江泽,张W.TCM药理学研究的分子网络。简短生物形式。2010; 11(4):417-30。https://doi.org/10.1093/bib/bbp063.
- 19.
歌,刘y,宋a,董g,赵h,sun w等。菊花Nankingense Genome提供了菊花和药物性状的进化和多样化的见解。莫尔植物。2018; 11(12):1482-91。https://doi.org/10.1016/j.molp.2018.10.003.
- 20。
Da Silva Jat,Jin X,DobránszkiJ,Lu J,Wang H,Zotz G等。石斛分子研究进展:遗传变异,鉴定和育种的应用。mol phylocyet evol。2016; 95:196-216。https://doi.org/10.1016/j.ympev.2015.10.012.
- 21。
徐军,褚勇,廖波,肖胜,尹强,白锐,等。人参皂苷生物合成的基因组检测。Gigascience。2017;6 (11):gix093。https://doi.org/10.1093/gigascience/gix093.
- 22。
Vining KJ,Johnson SR,Ahkami A,Lange I,Parrish An,Trapp Sc等。薄荷龙玉清基因组序列草案以及薄荷品种改善资源的发展。莫尔植物。2017; 10(2):323-39。https://doi.org/10.1016/j.molp.2016.10.018.
- 23。
沉Q,张立,廖Z,王S,yan t,shi p等。Artemisia Annua的基因组提供了洞察Asteraceae家族和青蒿素生物合成的演变。莫尔植物。2018; 11(6):776-88。https://doi.org/10.1016/j.molp.2018.03.015.
- 24.
杨建军,张刚,张建军,刘辉,陈伟,王旭东,等。中草药灯盏花的杂交从头基因组组装。Gigascience。2017;6 (6):gix028。https://doi.org/10.1093/gigascience/gix028.
- 25.
张丽,李旭,马斌,高强,杜华,韩勇,等。苦荞基因组提供了深入了解芦丁的生物合成和非生物胁迫耐受性。摩尔。2017;10(9):1224 - 37。https://doi.org/10.1016/j.molp.2017.08.013.
- 26.
陈W,kui l,zhang g,zhu s,zhang j,王x等。中国草药诺辛中草药的全基因组测序和分析。莫尔植物。2017; 10(6):899-902。https://doi.org/10.1016/j.molp.2017.02.010.
- 27.
邓炜,尼克斯直流,学习GH,玛斯特B,穆林斯吉。Viroblast:一个独立的Blast Web服务器,用于灵活查询多个数据库和用户数据集。生物信息学。2007; 23(17):2334-6。https://doi.org/10.1093/bioinformatics/btm331.
- 28.
武韵T-N,王L,刘H,王X,张L,Bennetzen JL等。耐橡胶树基因组在聚异戊二烯生物合成的演变中提供了洞察力。莫尔植物。2018; 11(3):429-42。https://doi.org/10.1016/j.molp.2017.11.014.
- 29.
秦g,xu c,ming r,唐h,guyot r,kramer em,等。石榴(Punica Granatum L.)基因组和瞳孔生物合成的基因组学。植物J. 2017; 91(6):1108-28。https://doi.org/10.1111/tpj.13625.
- 30.
Tamiru M,Natsume S,Takagi H,White B,Yaegashi H,Shimizu M等。主食食品作物白色豚鼠的基因组测序使得能够开发用于性别测定的分子标记。BMC BIOL。2017; 15(1):1-20。https://doi.org/10.1186/s12915-017-0419-x.
- 31。
肖林,杨光,张磊,杨旭,赵胜,纪志,等。Boea hygrometrica的复活基因组:脱水生存的蓝图。中国科学院院刊。2015;112(18):5833-7。https://doi.org/10.1073/pnas.1505811112.
- 32。
田勇,曾勇,张军,杨超,严玲,王旭,等。辣木是一种潜在的多年生作物。中国生命科学。2015;58(7):627-38。https://doi.org/10.1007/s11427-015-4872-x.
- 33。
张刚,田勇,张军,舒丽,杨胜,王伟,等。中国草本植物丹参(Salvia milorrhiza Bunge)杂交从头基因组组装。GigaScience。4 (1): 2015; s13742 - 015 - 0104 - 3。https://doi.org/10.1186/s13742-015-0104-3.
- 34。
Van Bakel H, Stout JM, Cote AG, Tallon CM, Sharpe AG, Hughes TR,等。大麻的基因组和转录组草图。基因组医学杂志。2011;12(10):队。https://doi.org/10.1186/gb-2011-12-10-R102.
- 35。
刘X,刘y,黄p,ma y,清z,唐q等。药用植物Macleaya Cordata的基因组为苄基异喹啉生物碱代谢提供了新的见解。莫尔植物。2017; 10(7):975-89。https://doi.org/10.1016/j.molp.2017.05.007.
- 36.
Hoopes Gm,Hamilton JP,Kim J,Zhao D,Wiegert-Rininer K,Crisovan E等人。基因组组装和注释药用植物Calotropis Gigantea,抗癌和抗疟药生产商。G3:基因,基因组,遗传学。2018; 8(2):385-91。https://doi.org/10.1534/g3.117.300331.
- 37.
付勇,李丽,郝胜,关锐,范光华,石超,等。藏药红景天基因组序列草案。Gigascience。2017;6 (6):gix033。https://doi.org/10.1093/gigascience/gix033.
- 38.
赵德,汉密尔顿·吉普,Pham Gm,Crisovan E,Wiegert-Rininer K,Vaillancourt B等。Capptotheca Acuminata的De Novo Genome组装,抗癌复合Camptothecin的天然来源。GigaScience。2017; 6(9):GIX065。https://doi.org/10.1093/gigascience/gix065.
- 39.
Kellner F,Kim J,Clavijo Bj,Hamilton JP,Childs KL,Vaillancourt B等。植物天然产物生物合成的基因组导向调查。工厂J. 2015; 82(4):680-92。https://doi.org/10.1111/tpj.12827.
- 40.
张继,田义,闫朗,张克,王x,zeng y等。植物Maca(Lepidium Meyenii)的基因组照亮了中央安第斯山脉高原适应的基因组基础。莫尔植物。2016; 9(7):1066-77。https://doi.org/10.1016/j.molp.2016.04.016.
- 41.
Jones DT, Swindells MB.从PSI-BLAST获得最多。生物化学学报。2002;27(3):161-4。https://doi.org/10.1016/s0968-0004(01)02039-4.
- 42。
Schäffer AA, Aravind L, Madden TL, Shavirin S, Spouge JL, Wolf YI,等。利用基于成分统计等改进方法提高PSI-BLAST蛋白数据库搜索的准确性。核酸学报2001;29(14):2994-3005。https://doi.org/10.1093/nar/29.14.2994.
- 43。
Johnson M, Zaretskaya I, Raytselis Y, Merezhuk Y, McGinnis S, Madden TL. NCBI BLAST:更好的网页界面。核酸Res. 2008;36(suppl_2):W5-W9。https://doi.org/10.1093/nar/gkn201
确认
我们要感谢Xingde Ren,Xiaosa Shi宝贵的建议。该工作得到了中国国家自然科学基金(81430094)和中国博士后科学基金会(No.2020M670236)。
资金
开放收费项目资助:国家自然科学基金项目(No.81430094)和中国博士后科学基金项目(No.2020M670236)。国家自然科学基金项目(No.81430094)对工程的构思和设计做出了重大贡献。中国博士后科学基金资助项目(No.2020M670236)支持对该手稿的工作和写作进行数据分析和解释。
作者信息
隶属关系
贡献
Y.Z.和Y.Q.构思和设计了实验;Z.C, J.L.和N.H.收集数据;Z.C.贡献试剂/材料/分析工具;zc.c建立了数据库并撰写了这篇手稿,Y.Z.和Y.Q.对手稿进行了修改。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
竞争利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1
.通过‘blastp’程序设置与甘草蛋白数据库的蛋白序列比对选项。图S2.BLAST结果与甘草Uralensis蛋白数据库进行序列比对,输入查询的蛋白序列。图S3.用‘tblastn’程序设置甘草核苷酸数据库的蛋白序列比对选项。图S4.BLAST结果用' tblastn '程序与甘草(Glycyrrhiza Uralensis)蛋白数据库进行序列比对。图S5.通过blastn程序设置甘草乌拉尔根核苷酸数据库的核苷酸序列比对选项。图S6.核苷酸序列与Glycyrrhiza Uralensis核苷酸数据库通过“BLASTN”程序的爆炸结果。图S7.通过‘blastx’程序设置甘草蛋白(Gancao)数据库的核苷酸序列比对选项。图S8.核苷酸序列与Glycyrrhiza Uralensis蛋白(Gancao)数据库通过“Blastx”程序的爆炸结果
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
陈,Z.,李,J.,侯,N。等等。中药基因组测序整合资源。BMC植物杂志21,339(2021)。https://doi.org/10.1186/s12870-021-03096-1
收到:
接受:
发表: