摘要
背景
燕麦是北美和北欧的重要作物。在斯堪的纳维亚,产量受到燕麦不能用作冬季作物的影响。为了制定这种作物,需要更多关于燕麦耐寒机制的知识。
结果
从OAT cDNA文库中获得9792单通过EST序列。该图书馆由汇集的RNA样本制备从4周龄的叶子隔离燕麦属漂白亚麻纤维卷(燕麦)植物在+4°C孵育4,8,16和32小时。排除100 bp以下的序列,得到8508条平均长度为710.7 bp的优质est序列。聚类和组装确定了一组2800个不同的转录本燕麦属漂白亚麻纤维卷冷诱导UniGene集(AsCIUniGene集)。利用各种工具和数据库,推测的功能被分配到1620个(58%)这些基因。在剩余的1180个未分类序列中,427个似乎是燕麦特异性序列,因为它们缺乏任何显著的序列相似性(BlastE价值> 10.-10)转换为公共数据库中可用的任意序列。在2800个UniGene序列中,398个序列显示显著同源性(BlastXE值≤10.-10)到先前被报道参与冷应激相关过程的基因。107个新的燕麦转录因子也被鉴定出来,其中51个与之前被证明是冷诱导的基因相似。CBF转录因子在冷驯化中起着重要的调节作用。共发现4条燕麦CBF序列,属于DREB家族ERF/AP2结构域蛋白单子叶簇。最后,在全部EST序列数据(5.3 Mbp)中,大约发现了400个潜在SSRs,其频率与之前识别的频率相似拟南芥ests。
结论
Asciunigene集合现在将用于制造燕麦生物芯片,以在不同温度下孵育的不同燕麦品种进行各种表达研究,以产生分子标记,并为燕麦中的各种遗传转化实验提供工具。这将导致更好地了解这一重要作物的细胞生物学,并将开辟新的方法来改善其农艺性质。
背景
燕麦属漂白亚麻纤维卷(燕麦)属于禾本科家庭。其他谷类有小麦、大麦和黑麦[1].野生燕麦是二倍体,但所有栽培的燕麦都是六倍的六倍,估计的1c基因组大小为13.23 pg,对应于约13000 Mbp [2].燕麦的商业价值来自其高能量谷物和优越的突破作物益处。由于这种作物的高疾病耐受性和低营养要求,燕麦种植园也具有相对较低的杀虫剂,杀真菌剂和肥料的需求[3.].目前,燕麦主要用作动物饲料,但它是功能食品领域最有前景的谷物之一。由于其可溶性膳食纤维和高β-葡聚糖含量,它具有独特的、有充分证据证明的降胆固醇效果[4- - - - - -6].燕麦饮食可大大改善乳糜泻患者的良好感觉,也可降低饮食相关疾病的风险[7- - - - - -9].燕麦富含天然酚醛抗氧化剂[10.- - - - - -14.],防止心血管疾病和某些癌症的发展。在瑞典,大多数收获的燕麦用于动物饲料。每年只有约35000吨用于人类消费。然而,由于其许多健康增强性能,燕麦和处理过的燕麦产品,如燕麦牛奶,用于人类消费迅速增长。
许多欧洲国家在秋天播种的燕麦生长,即燕麦,在野外的冬天幸存下来。因此,与夏季品种相比,冬季燕麦具有更长的生长季节,允许早期收获并提供更高的收益率。然而,固有的燕麦不是冬天耐寒,因为黑麦,小麦和大麦。由于斯堪的纳维亚国家的恶劣气候,因此冬季燕麦在那里没有成长。根据冬季燕麦的英语经验,瑞典冬季燕麦可能会增加收获的收益率至少30%(John Valentine,Iger,英国,个人沟通)。此外,由于燕麦是小麦和油作作物最重要的旋转作物,因此冬季燕麦的早期收获意味着早期播种的旋转作物,导致这些作物也增加了产量。因此,开发适合斯堪的纳维亚气候的冬季燕麦是高度优先(Anders Jonsson,Swedishfarmers供应和作物营销合作,个人沟通)。由于冷耐性是由几种基因控制的定量性状[15.迄今为止,传统的植物育种计划在提高任何重要作物品种的抗寒性方面取得的成功有限[16.]瑞典燕麦饲养员或多或少地允许他们努力生产瑞典冬季燕麦(Rickard Jonsson,SvalöfWeibullAb,个人沟通)。
从具有大,复杂和未知的基因组的生物体获得新数据的成本效率和快速的方法是通过随机选择的cDNA克隆的部分测序。所得到的表达序列标签(EST)的收集反映了采样组织中基因表达的水平和复杂性,并且还将深入了解所选生物的基因结构。这不仅导致鉴定来自具有已知或推定功能的新生物体的许多基因,而且还能识别出令的函数,而且还能对完全新的,以前未知的推定蛋白质的发现。
在这项研究中,我们从9792个EST序列中鉴定了2800个推测的燕麦基因,其中一些基因与之前定义的与寒冷胁迫相关或参与转录调控、信号转导或代谢的基因相似。几个序列可以代表新的,未知的和独特的燕麦基因也被鉴定。这些数据将被用于研究燕麦的冷驯化,识别调节越冬存活的关键基因,产生分子标记以促进抗冬育种,并构建具有更高抗冻性的转基因燕麦。这些实验将增加我们对一般植物,特别是燕麦的冷驯化生理学的一般知识,并在长期内使斯堪的纳维亚冬季燕麦的发展成为可能。
结果
低温驯化
120个人,四周,温室养殖植物冬天的燕麦品种杰拉尔德,83 - 48 CH和spring燕麦品种玛蒂尔达和贝在黑暗中孵化+ 4°C(±0.5°C) 12和24小时,之后搬到-15°C, 1, 2, 4, 8或16 h。120株被直接在-15°C 1 2 4,8小时或16小时。在孵育期后,将植株移回温室,让其恢复一周。然后将可见的冻害划分为1到5,其中1表示没有可见损害,5表示植物死亡(图)1模拟).在冻结之前在+ 4℃下孵育的植物比直接转移到-15°C的植物(表1).因此,燕麦植物如预期的那样能够在一定程度上冷却。夏季和冬季品种之间可以看到明显的差异,但后者在适应之前和之后的冬季脾气暴躁(表1).
燕麦的冷驯化.(A)温室栽培的四周龄的Matilda(1)和Birgitta(2)(春燕麦),Gerald(3)和83-48-CH(4)(冬燕麦)。(B)在温室植物恢复一周适应(24 h + 4°C)和孵化-15°C 3 h。(C)植物恢复一周后的温室适应(24 h + 4°C)和孵化-15°C 6 h。(D)在温室植物恢复一周适应(24 h + 4°C)和孵化12 h。-15°C (E)在a separate experiment four-week old Gerald plants were incubated at +4°C for 0, 4 h, 16 h, and 64 h (indicated below lanes), total RNA was isolated and Northern hybridisations was performed using the oat Cor410 genes as a probe.
为了在分子水平上证实冷驯化,研究了一个已知的冷驯化标记。在+4°C孵育4、16、32和64小时后,从3周龄的冬季品种Gerald植株中分离出总RNA。一种燕麦序列,与小麦的COR410基因相对应,已知在小麦中可冷诱导[17.,通过PCR从燕麦基因组DNA中扩增。northern的一项分析使用从冷诱导植物中分离的RNA和燕麦COR410作为探针,结果表明,COR410基因在4 h后可以被检测到,然后在冷培养64 h后仍然保持强表达(图)1E).
EST测序和UniGene序列构建
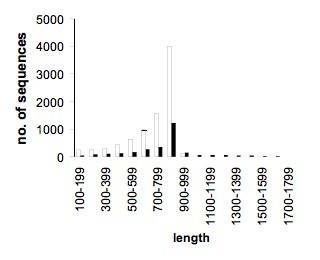
由于我们在EST分析中同时针对早期和晚期的冷诱导基因,因此从不同冷驯化阶段的燕麦植株中分离到的RNA样本中构建了cDNA文库。文库电镀后,随机选取菌落,对这些克隆的质粒中存在的cdna进行9792次单次序列反应。结果共获得8508条100 bp及以上的优质est序列,平均序列长度为710 bp。8508个ESTs被组装成1100个contigs和2616个单重子,得到3716个候选基因。所有包含rRNA、线粒体或叶绿体序列的ESTs均被排除在进一步分析之外。最后,消除候选基因中冗余序列的数量。这是通过将候选基因集与NCBI非冗余(nr)蛋白数据库进行BlastX分析(见“材料和方法”)完成的。这将候选基因集减少到2800个转录本,平均序列长度为800 bp(图)2).这些最终基因表示为asciunigene(燕麦属漂白亚麻纤维卷冷诱导UniGene)设置。
注释和功能分类
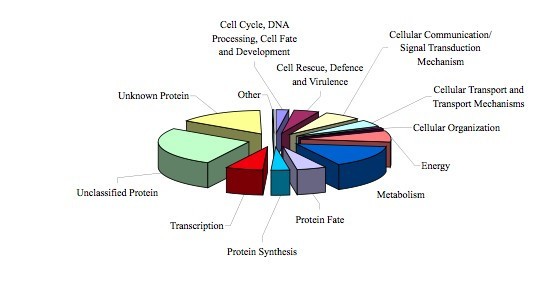
ASCIUNIGENE集的注释基于同源性。Asciunigene集中的每个基因继承了在NCBI对NR蛋白数据库的BLASTX搜索后发现的最佳匹配的注释。期望值(EValue)阈值为10-10是使用。所有的序列,总共427个E高于此阈值的值被标注为未知。
根据慕尼黑蛋白质序列信息中心(MIPS)的蛋白质对AsCIUniGene集合中的每个单独序列进行了功能分类。拟南芥给BlastX评分最高的数据库(MATDB)。由于MATDB中的每个基因都被分配到MIPS功能分类系统定义的28个不同类别中的至少一个,这证明了在AsCIUniGene集合中分配假定功能的有效方法。MIPS分类系统的一个缺点是它有些粗糙,因为许多功能分类是自动分配的。因此,除了自动化过程外,我们还手动检查了所有蛋白质的功能分类分配,并在必要时重新分配。通过这种方式,所有的2373个燕麦序列(其中大约400个是人工分配的)最终被归入前面提到的MIPS类。剩余的427个序列(用E价值> 10.-10在BLASTX与nr的搜索中)不能自动或手动分配,因此归类为未知蛋白(图3.).
最丰富的ESTs
为了分析来自冷诱导的cDNA文库的最丰富的EST,我们通过Kog(蛋白质的真核菌群)数据库分组序列。该数据库目前包括7个真核生物基因组,包括拟南芥.这为基于同源蛋白的序列提供了功能注释[18.].此外,我们使用互补数据库来注释我们的序列,如FOGs (Fuzzy Orthologous Group),其中包含的蛋白质具有混杂的结构域,由于同源关系不清楚而未被分配到KOG身份,TWOGs,包含在两个基因组中表示的临时蛋白质簇,以及LSEs,其中包含的蛋白质是存在于KOG数据库中的同源基因的谱系特异性扩展。分类是基于BlastX搜索的最佳同源匹配拟南芥蛋白质序列的期望值(EValue)阈值为10-10是使用。以这种方式注释的蛋白质被称为“Kog-Twog-LSE”。既然不是全部拟南芥蛋白质在Kog数据库中表示,并非所有EST都可以用Kog-Twog-LSE注释。oat ests在同源匹配中拟南芥但不是Kog-Twog-LSE注释,继承了MIPS注释中的注释。此外,几个序列没有拟南芥同系物与an匹配E值超过阈值。这些序列与NCBI nr蛋白数据库中BlastX搜索结果的同源性最好。同样,一个E价值阈值10-10是使用。
作为一个比较,来自非诱导的燕麦叶库的2189 EST序列[19.以同样的方式分析。可以从表中推断出来2,非诱导和诱导的叶库是完全不同的。在整个文库中,叶绿素A / B结合,氢皂苷1,5-双磷酸羧酸酯/氧酶小链和核苷酸二磷酸羧酸酶/氧合酶活性(Interpro登录号IPR001344和IPR000894)是最表达的基因家族,但虽然以不同的顺序(表2).在非诱导的文库中,核糖糖1,5-双磷酸羧酶/氧酶序列是最常见的,也许反映了这些植物的较高的光合活性。有趣的是,几种感冒相关蛋白质是冷适应文库中最丰富的最丰富的EST中,但在最佳条件下从燕麦种植的最丰富的EST中没有表示。冷诱导的Cor410(Wcor410)是脱氢[17.].在水缺损和冷应激期间表达脱氢。冷响应lea / rab相关的cor蛋白(wrab17)属于Lea-proteins的3组,并先前已被冷却诱导[20.].在此背景下,其他有趣的蛋白质是auqaporin PIP和富含羟脯氨酸的糖蛋白。水通道蛋白是一种促进水通过生物膜的膜蛋白。在拟南芥有13个水蛋白的成员属于质膜内在蛋白(PIP)亚组。最近,一项研究表明,响应于各种非生物应力的倾斜或下调皮蛋白[21.].富含羟脯氨酸的糖蛋白与OSR40蛋白具有较强的同源性奥雅萨苜蓿,此前已被证明是由于盐胁迫而上调的[22.].这些基因在非诱导文库中均不存在,说明诱导文库中与冷胁迫相关的基因大量富集。由于只包含了被标注为冷诱导、冷适应或冷调节的基因,其他间接参与冷胁迫反应的应激相关基因也很可能在这个集合中富集。
冷调基因
在属于四个类别的功能分类序列中;“细胞救援,防御和毒力”,“蜂窝通信/信号转导机制”,“新陈代谢”和“转录”被认为是对冷驯化潜力巨大的兴趣。这些类别在一起由ASCIIGENE集中的931个条目表示,对应于所有基因的超过30%(表3.).为了提高分析的分辨率和改进假定的冷调节基因的鉴定,我们建立了一个单独的数据库,其中包含了以前文献中报道的直接参与冷应力相关过程的蛋白质[23.- - - - - -26.].这个“冷应力数据库”(CSDB)目前包括545个条目。在针对CSDB的AsCIUniGene序列的BlastX搜索中,我们在AsCIUniGene序列中发现了398个具有显著同源性的序列(E≤10-10)在CSDB中的基因(表3.).因此,在AsCIUniGene集合中,14.2%的基因似乎与冷应激有关。只看我们认为在冷适应过程中最重要的两类(细胞拯救、防御和毒性;细胞通讯和信号转导)几乎40%的序列与潜在冷胁迫相关蛋白同源。在“代谢”和“细胞运输和运输机制”类中,与冷胁迫相关的基因也占多数。25%)。
为了分析在AsCIUniGene集合中,冷调节基因占总基因数的高比例是否与来自冷驯化植物的其他EST集合相似,我们下载并分析了来自冷驯化小麦和大麦的EST数据集。使用TGI聚类工具对数据集进行聚类和组装(参见材料和方法)。结果得到2894个TaCIUniGene和3932个HvCIUniGene。此外,作为比较,相同的燕麦est收集来自在最佳条件下生长的植物叶片(表2)聚类并组装成AsNIUniGene的1445条序列。然后根据CSDB搜索这三个UniGene集。由此可见,冷胁迫相关基因在TaCI候选基因中所占比例为9.6%,在HvCI中所占比例为11.1%,而在AsNI UniGene中所占比例仅为5.1%(表2)4).因此,ASCI Unigene集合与先前的OAT EST收集以及含有所有谷物的冷诱导基因比例最高的EST收集。
含AP2蛋白的系统发育分析
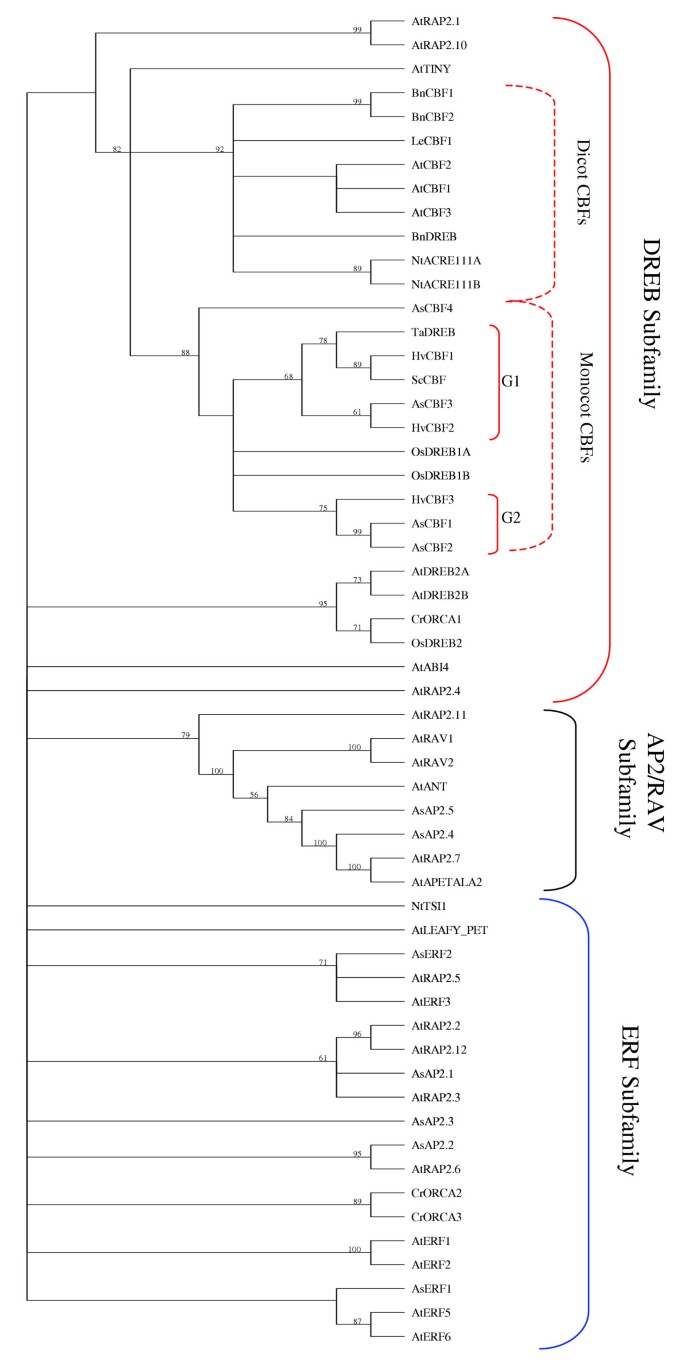
在转录类的190个序列中(表3.),发现107种与转录因子同源。值得注意的是,这107个序列中的51个对CSDB中的基因同源,代表近48%的Asciunigene集中发现的转录因子。要探讨该方法,转录因子进一步分为14个不同的转录因子家族(表5).其中AP2结构域、同源结构域和zink finger蛋白的编码基因占主导地位。CBF/DREB1转录因子家族的蛋白质此前已被证明是大多数冷反应基因的调控因子。CBF/DREB1家族属于AP2/ERF超家族[27.),在拟南芥AP2/ERF超家族由145个蛋白组成。基于其dna结合域的相似性,这些蛋白被细分为AP2、RAV、DREB和ERF亚家族,并与其余的蛋白有一个家族。在AsCIUniGene集合中,我们发现了11个AP2/ERF超家族序列(见表)5).这11个AP2/ERF域燕麦属更接近地分析蛋白质,也与含有蛋白质的前述AP2 / ERF的45个相比[28.].系统进化树显示,燕麦AP2/ERF蛋白中有4个属于DREB亚家族,2个属于AP2/RAV亚家族,其余5个属于ERF亚家族(见图)4和表格6).从分析中还可以推断出燕麦CBF/DREB1蛋白与单子叶CBF/DREB1蛋白关系最为密切(图)4).
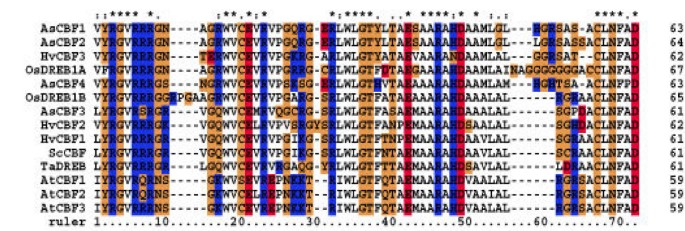
为了进一步分析燕麦与其他单子叶CBF/DREB1蛋白的关系,我们对AsCBF1、AsCBF2、AsCBF3、AsCBF4、OsDREB1A、OsDREB1B、HvCBF1、HvCBF2、HvCBF3、ScCBF和TaDREB的AP2/ERF结构域进行了多位点比对。此外,我们还纳入拟南芥CBF/DREB序列AtCBF1、AtCBF2和AtCBF3,进一步阐明双子叶和单子叶之间的关系(图)5).在先前的研究中,据报道,特别是两个氨基酸,位于19(V19)的缬氨酸,在24(E24)处的谷氨酸(E24)(图5)的AP2/ERF域在确定dna结合特异性方面具有重要作用[27.].ASCBF蛋白在V19位置具有保守的缬氨酸,但不是E24位置中的谷氨酸(图5).相反,缬氨酸在这个位置被保守。此功能实际上是共享的,包括单码CBF / DREB蛋白(图5).单焦CBF / DREB1蛋白可以进一步分为三个亚组(图4).第一亚组(G1)包含AsCBF3、HvCBF1、HvCBF2、ScCBF和TaDREB蛋白,第二亚组(G2)包含AsCBF1、AsCBF2和HvCBF3蛋白,第三亚组剩余的AsCBF4、OsDREB1和OSDREB2蛋白(图)4).该分组基于不同蛋白质之间的位置10,18和39处AA的差异(图4, 数字5).在第10位,G1蛋白有一个基本的精氨酸(R)残基,G2蛋白有一个亲水天冬酰胺(N), G3蛋白有N,亲水丝氨酸(S)或甘氨酸(G)残基(图)5).在第18位,G2和G3蛋白中有一个基本残基,即精氨酸(R),而G1蛋白有一个亲水残基,即谷氨酰胺(Q)(图)5).最后,G2蛋白在39位有一个亲水酪氨酸(Y),而G1和G3蛋白这个位置被疏水苯丙氨酸(F)占据,除了AsCBF4,它有一个碱性组氨酸(H)(图)5).
AP2 dna结合域的聚簇分析.AsCBF1、AsCBF2、AsCBF3和AsCBF4的AP2结构域与其他植物CBF相关蛋白的AP2结构域进行了比对,如左侧文本所示。拟南芥蒂利亚纳ATCBF1,ATCBF2,ATCBF3;Horedeum vulgare.HvCBF1、HvCBF2 HvCBF3;小麦Tadreb;奥雅萨苜蓿osdreb1a,osdreb1b;Secale cerealeScCBF(序列的AC-numbers如表6所示)。氨基酸残基根据功能进行着色,着色模式如下:酸性残基(DE)为红色,碱性残基(RHK)为蓝色,疏水残基(AILMFPV)为白色,亲水残基(NCQGSTY)为橙色。
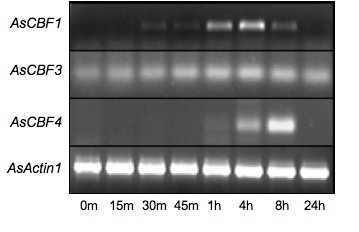
的表达ASCBF.基因
以探究燕麦是否被识别出来ASCBF.用与其他先前描述的CBF基因相似的动力学感应冷诱导基因,进行表达分析。多用RT-PCR在从3周龄植物的叶子中分离的总RNA进行,使用基因特异性引物在15分钟至24小时的时间点的叶片中分离出来的叶片(至+ 4℃)。ASCBF1.,ASCBF2.,ascbf3.和ASCBF4.分别。在同一时间点与未处理的植物分离的总RNA作为比较。燕麦肌动蛋白基因(AsActin)也从与加载和RNA质量控制相同的RNA样品中扩增。为了定义PCR扩增在指数阶段的条件下,进行几个具有30,35或40个循环的实验。这表明不同的ASCBF.所有冷治疗都诱导基因,但它们的差异调节(图6).的ASCBF1.基因在0时未检测到,约30 min后诱导,4 h达到高峰,24 h后完全关闭ascbf3.基因有完全不同的表达模式。它在非诱导植物中也弱表达,但在15 min后,在第一时间点已经迅速被诱导。此后,表达水平继续增加,在约4 h后达到峰值,但在24 h仍然显示出升高的表达ASCBF4.在4小时后,缓慢地诱导基因并未检测到。与其他人不同,其表达达到稍后达到峰值,过8小时后,24小时后完全下降。尽管有几次尝试,使用不同的底漆对我们无法获得可重复的表达模式ASCBF2.基因(数据未显示)。因此,不同的燕麦ASCBF.基因似乎在冷驯化过程的不同阶段是活跃的,因此可能会诱导不同的下游基因程序。我们目前正在更具体地处理这个问题。
鉴定的微卫星
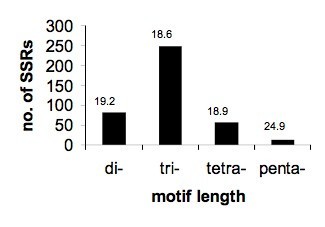
使用Prom方法部分中规定的Sputnik程序和阈值,我们在3716候选基因集中搜索了潜在的微卫星(SSR)序列。鉴定了满足搜索标准的399次偏核核苷酸SSR。这对应于每13 kB序列的大约一个微卫星。使用相同的方法和阈值,Cardle等。(2000)在每14千克EST数据中平均发现一个SSR拟南芥.这表明在燕麦中SSR的频率与在燕麦中相似拟南芥.在燕麦序列收集中,三核苷酸重复是最常见的,然后是二核苷酸重复(图7),再次匹配结果拟南芥[29.].除了用于长度45和55 BP的SSR的两个特殊TA / SSR,SSR长度范围为15到25 BP,16和17 BP是最常见的。
现在正在进行工作以确定这些SSR中的哪一个可以通过PCR再现地扩增,是多态的,并且可以与表型标记连接。在非编码DNA中发现绝大多数燕麦SSR。由于它们不仅代表了积极转录的基因,我们预计其中几种将成为繁殖的有用标记。
讨论
植物表达序列标签(ESTs)已被证明是分子生物学研究中有价值的工具,许多不同植物物种的收藏现已公开[19.].在地球上最重要的粮食供应者谷物中,已经开展了几个主要的EST测序项目。在撰写本文时,共有284 779条公开可用的无害环境技术奥雅萨苜蓿(大米),562 786从小麦(小麦)和367 768来自Hordeum Vulgare.(大麦)。相比之下,只有7个624个条目燕麦属漂白亚麻纤维卷(燕麦),没有来自冷驯化燕麦的序列可用。显然,对这种重要作物也非常需要更多的EST测序。在此,我们向研究界贡献了另外9 792条来自冷适应燕麦的序列。由于我们主要对冷驯化的感知、信号转导和早期调控相关基因感兴趣,所以我们关注的是从几分钟到24小时的短孵育时间。经驯化和非驯化的植物抗冻性存在明显差异,冬季品种的抗冻性强于春季品种(表)1).为了确认冷致诱导基因在这些植物中超越,对对应于先前描述的冷诱导小麦的燕麦基因进行北分析COR410在+4℃低温驯化过程中,从几个不同时间点分离的RNA上的基因。这揭示了诊断COR410基因在燕麦中也是冷诱导的,有趣的是,在冬季品种中表达量峰值更高(图)1和表格1).其他冷诱导基因(数据未显示)也出现了同样的趋势,即诱导较早且表达水平较高。
利用已证实的冷诱导植物的叶片进行cDNA构建和EST序列生成。叶被用作RNA源以来,最常见的est序列集合代表各种基因参与光合作用,叶绿素a / b结合蛋白,核酮糖1,5-bisphosphate羧化酶/加氧酶,二磷酸核酮糖羧化酶/加氧酶活化酶、光系统I反应中心蛋白,fructose-bisphosphate醛缩酶,碳酸酐酶/碳酸盐脱氢酶和光系统II氧进化复杂蛋白。其他具有代表性的序列是那些编码核糖体蛋白的序列(见表)2).然而,在表达最多的20个基因家族中,脱醇也存在,这表明我们的收集确实代表了低温胁迫诱导条件下的植物。通过与非诱导植株叶片衍生的EST序列的直接比较,证实了这一点。在这个集合中,脱水和其他冷诱导基因不在20个高表达基因之列。
从我们的冷诱导ent集合中,确定了2800个基因的ASCI Unigene集。其中,1 726可以置于MIPS定义的官能团(图3.),留下这种分类外部的相对较大的基因(约40%)。感知应激刺激,应力信号的转导和分子响应是必要的活动,如果植物对非生物胁迫反应。然而,在燕麦中,很少有关于这种水平的冷应激反应的知名,尽管所有植物可能相似。为了更好地鉴定涉及感冒响应的燕麦基因,因此我们创建了一种表示的CSDB(冷应激数据碱基)的数据库,其中收集来自公共领域的所有基因和分类为冷应激响应或冷诱导。当将CSDB中的序列与OAT asciunigene集合时,我们发现匹配的398个序列,表明Asciunigene集合中的所有基因的至少14%涉及冷应激。其中,编码与感知,信号转导和转录调节有关的活动的序列是超人的(表3.).我们使用与AsCIUniGene相同的工具,从温室条件下生长3周的燕麦植株叶片生成的燕麦EST收集中创建了一个包含1445个不同转录本的UniGene集合。这种非诱导叶集被称为niunigene。当用AsNIUniGene搜索CSDB时,只有5.1%的基因是相似的4),对Asciunigene集的剧烈差异。这些研究延伸到来自寒冷的小麦和大麦的勘探收集。通过在CSDB中分析这些系列,通过创建未成分套(Taciunigene和Hvciunigene)。然后我们发现,小麦和大麦收集中,寒冷相关基因的量约为10%(表4).普遍性,似乎至少有10%的冷适应植物中表达基因的所有表达基因致力于制备植物到冷冻温度所需的各种细胞反应。冷诱导的燕麦基因集合现在将在进一步分析此类基因中是一个有价值的新资产。
我们对Asciunigene集的功能分析表明,转录因子基因由107个序列表示,属于至少14个不同的家族(表5).其中,51与来自其他系统的冷诱导基因同源。对冷驯化的特殊重要性是CBF转录因子家庭。在该家庭中的基因调节几种不同的下游基因,包括Cor基因[16.,30.].然而,这种调节是复杂的,涉及到几个不同的CBF基因。从AsCIUniGene集合中,我们鉴定了4个燕麦CBF基因ASCBF1.,ASCBF2.,ascbf3.和ASCBF4..我们的系统发育和多重比对分析表明,这四种蛋白都属于ERF/AP2结构域单子叶DREB亚家族。的ASCBF1.和ASCBF2.基因非常密切相关,而ascbf3.和ASCBF4.基因之间的亲缘关系较远,它们也属于不同的支系ASCBF1.和ASCBF2.基因(图4和5).
来研究ASCBF.基因,我们设计了基因特异性引物并通过RT-PCR分析显示,这些基因确实是冷诱导的,但它们的表达模式不同。他们的表情在15分钟后已经从早期诱导范围内(ascbf3.)1小时后诱导(ASCBF4.),并由4小时起峰(ASCBF1.和ascbf3.)在8小时(ASCBF4.)(图6).的ascbf3.特别是有趣的,因为它弱组成型表达,表达了冷治疗后表达明显增加,24小时后表达仍然表达。尽管使用不同底漆对进行了多次尝试,但我们无法获得可重复的表达模式ASCBF2.基因。目前尚未知道这一点的原因。复杂的调节ASCBF.基因与之前描述的内容不同拟南芥[31.),ATCBF1.,ATCBF2., 和ATCBF3.这表明,CBF因子在诱导和维持燕麦冷驯化过程中具有复杂而不同的个体作用。大麦的初步数据证实了这一点。这种谷物至少有10种不同的基因编码CBF因子,它们都受到不同的调节(Eric Stockinger,俄亥俄州立大学,个人交流)。因此,对结构和规制进行了较为详细的分析CBF谷物中的基因可能揭示了低温诱导的新途径,而这种途径在玉米中并不存在拟南芥.
在ASCIIGENE集中鉴定了许多具有迄今未知功能的基因。这些被分成两组,其中存在来自其他系统的同源或类似基因,并且在任何其他序列中没有明显相似性的情况下,即可以是燕麦特异性的基因。为了排除对该组有贡献的小“非真实”肽,仅包括具有100AA或更多的开放阅读帧的序列。现在正在进行中,以阐明由冷应激,干旱胁迫或不同应力因子的组合引起的427燕麦特异性未知基因中的哪一个。假设大约。这些序列中的10%是冷相关的,在该系列中将存在超过40种涉及冷驯化的新燕麦基因。这些基因可能非常有趣,可以编码迄今为止的不起作用的蛋白质或调控因素,参与冷适应和冷冻保护
微卫星(SSRs)是优良的DNA标记用于遗传作图,因为它们具有多态性,丰富,显示共显性遗传,易于用PCR分析[32.].SSRs因此被广泛应用于植物基因组研究[33.- - - - - -36.].当需要在大规模育种计划中追踪理想性状时,当需要为基于图谱的基因克隆策略确定锚点时,它们尤其有利。然而,目前只有少数燕麦ssr可用。在这里,我们鉴定了大约400个潜在的燕麦SSRs,大多数存在于EST序列的非编码部分。目前正在进行的工作是优化这些SSRs的引物,并确定那些提供可靠的PCR产物和多态性的引物。两种二倍体[37.,38.]和六倍体燕麦[39.,但这些地图需要改进[40].因此,最好的SSR标记将被定位到燕麦基因组,并与有价值的遗传标记相连。
AsCIUniGene现在将被用于制造含有所有2800个已识别基因的燕麦生物芯片。此外,通过建立减法库,将产生更多与寒冷有关的无害环境技术。我们将进行各种表达研究,并从我们收集的基因中选择对寒冷或干旱有快速诱导作用的基因进行进一步分析。我们对EST收集的那些基因特别感兴趣,这些基因在+4°C下表现出非常快速的诱导,并具有DNA结合特性。特别有希望的基因将在转基因中进行测试拟南芥燕麦系统[41,42通过选择的补充拟南芥T-DNA敲除突变体。
结论
从冷诱导的OAT cDNA文库中产生UNIGENE为2800个基因。进一步的分析表明,与冷应激相关的基因在该文库中呈现过持续,并且几个基因可以编码迄今未知的功能。RT-PCR分析CBF转录因子基因显示它们在燕麦中差异表达,因此可能调节不同的冷途径。约。在收集中,400个潜在的SSR标记也存在于非编码区中的几个,以及依赖于调节冷适应的基因附近。
方法
植物的生长
燕麦植物,燕麦属漂白亚麻纤维卷v。杰拉尔德,83-48 CH,SW Matilda和SW Birgitta从SW-Collection获得(SvalöfWeibullAb,Landskrona,瑞典)。Gerald和83-48是英国冬季品种,而Matilda和Birgitta是瑞典春天品种。种子在2升罐中萌发,装有受精和压制泥炭。在补充有金属卤素灯的自然光下的温室中培养植物,每米为240μmol的光子通量密度2光周期18 h,昼夜温度20/12℃,相对湿度70%左右。这些植物按需浇水。
冷诱导实验
为了调查所选燕麦品种的冷适应能力,制备了每种杰拉德,83-48克,畸变和Birgitta的24座含有10种种子的盆。萌发后大约三周,当每株植物产生3-4个叶子时,将罐子在+ 4°C(±0.5℃)的暗冷室中移动,并孵育12小时和24小时。在此后,将罐用于-15℃(±1℃)3小时,6小时和12小时。此外,植物从温室直接移动到-15℃,并孵育3小时,6小时和12小时。冷培养期后,植物恢复到温室以进行恢复。一周后,寒冷的伤害在视觉上得分。
总RNA制备
冬季燕麦(杰拉尔德)在温室里萌发并种植了三周。然后将它们在+ 4℃(±0.5℃)的黑暗中温育4,16,32或48小时。在每个时间点,叶子随机从几个个体植物中挑选并汇集。基本上从汇集的叶片中提取RNA,如Chang所描述的et al。(1993)。组织分离株在液氮中研磨,转移到65°C CTAB提取缓冲液(2% CTAB[十六烷基三甲基溴化铵][Sigma], 2% PVP[聚乙烯吡罗烷酮,特性粘度29-32][Sigma], 100 mM Tris-HCl [pH 8.0], 25 mM EDTA, 2.0 M NaCl, 0.5 g / L亚精胺,2% β-巯基乙醇),以等量的chisam(酚:氯仿:异戊醇:1:1:24)萃取两次。加入0.25 v/v 10 M LiCl,在4°C过夜沉淀RNA。将沉淀溶于1 × SSTE (1.0 M NaCl, 0.5% SDS, 10 mM Tris-HCl, 1 mM EDTA), pH 8.0,等体积chisam萃取,两体积99.5%乙醇沉淀,DEPC处理的蒸馏水重新悬浮。在OD值分光光度法测定各样品总RNA260.一点头2601对应于40μg/ ml的RNA。随后,将RNA沉淀并再悬浮在将DepC处理的蒸馏水中悬浮至终浓度为1mg / ml。
北方杂交
10 μg总RNA用乙二醛/DMSO进行变性[43]并在1%琼脂糖凝胶上分离。将RNA呈印迹到尼龙膜(Boorder-Mannheim)上并在教堂杂交缓冲液中杂交[44].与小麦COR410基因相似的燕麦序列作为探针。利用正向引物5'- atggaggatgagaggagac -3'和反向引物5'-TTTCTTCTCCTCCTCGGGC-3'对燕麦基因组DNA进行PCR扩增。引物设计基于小麦序列。扩增得到一个530 bp的序列,经DNA测序验证(数据未显示)。这个片段被贴上了标签32.P-dCTP (Amersham),使用随机的六核苷酸混合物和标记级Kleenow酶(Boeringer-Mannheim)。在65°C下进行严格的洗涤,在2 × SSC, 0.5% SDS中洗涤2 × 5 min,在0.2 × SSC, 0.1% SDS中洗涤4 × 5 min。薄膜暴露于x射线胶片(杜邦医疗斯堪的纳维亚AB)。
cDNA文库构建及EST测序
从+4°C孵育6、12和24小时的植物中分离的总rna进行汇总。将收集的RNA送到德国MWG Biotech,在那里构建cDNA文库,将cDNA克隆到pSPORT1载体中[45]并进行EST测序。
生物信息学工具
使用BlastN、BlastX或TBlastX工具在本地批量执行所有相似度搜索[46],全部包含在BLASTALL计划包中[47].Transeq, EMBOSS软件包中的程序[48,49将DNA序列转化为蛋白质序列。在NCBI对保守域数据库(CDD)进行保守域搜索(CD-search) [47]使用具有翻译EST的反向位置特异性Blast(RPS-Blast)算法。译文[50,51]用于扫描已翻译的ESTs,以获取InterPro成员数据库中的蛋白特征。对于多重对齐,我们使用ClustalW [52],包括在MacVector 7.2.2包(Accelrys Inc)。利用邻域连接(neighbour-joining, NJ)算法,利用MacVector 7.2.2工具包构建系统发育树。编写了适当的PERL脚本,以便按照顺序运行工具、解析结果文件并将结果加载到数据库中。所有数据和结果存储在PostgreSQL数据库中。
数据集和处理
本文开始使用四种不同的主要序列数据集。第一组是来自寒冷适应的燕麦的9792个est,这是表示的燕麦属漂白亚麻纤维卷冷诱导(ASCI)数据集。第二数据集包含2189 EST [53],它来自3周龄燕麦植物的未经处理的绿叶,并表示为燕麦属漂白亚麻纤维卷未引起的(ASNI)数据集。第三个数据集,其中包含4337个序列源自冷适应小麦[53被表示为小麦冷诱导(TaCI)数据集和最终数据集包括5418条来自冷胁迫大麦植株的序列[53,表示为Hordeum Vulgare.冷诱导(HVCI)数据集。
EST聚类和装配
使用ASCI数据集进行过滤,群集和组装,并与PARACEL脚本汇编程序(PTA)程序(PTA)程序(PTA)程序(Paracel,Pasadena,CA)集成到单个管道中的质量过滤,聚类和组装。过滤步骤包括掩蔽载体序列,低复杂度,低质量,重复和多(A)区域。在下一步中执行群集。这里PTA在全面与所有序列比较中使用急速算法。将用于聚类序列的标准设置在一起是至少100个碱基的对准,并且在对准序列之间具有至少93%的相似性。不适合这种簇的序列被定义为单体。在组装步骤中,PTA使用CAP4,这是CAP3算法的改进[54].不符合连续序列的序列也被定义为单线序列。最后,通过过滤器但未屏蔽序列< 100个碱基的单链est被丢弃。得到的单线和contigs代表了AsCI候选基因集。
使用TGI聚类工具聚集和组装成候选基因集的其他数据集[55].群集是通过NCBI的MegaBlast程序的一个稍微修改的版本来执行的[56]使用CAP3组装所得簇。
最丰富的est序列
最佳的Blastx同性恋者首次将个体EST归因于拟南芥蒂利亚纳,一个E值< 10-10是使用。的答:芥从MIPS中检索蛋白质拟南芥数据库(MATDB)。此后,在KOG数据库中给出的注释[18.每个人都被检索答:芥蛋白质。对于那些答:芥未进行KOG注释的同源物,EST序列继承了MATDB的注释。这些序列没有答:芥阈值上方的同源物与NCBI的NR数据库中的最佳同性恋者匹配带来了最佳同源物。再一次E值< 10-10是使用。
Unigene集测定
在两步程序中创建非冗余组基因组单曲和Contigs(Unigene集)。通过与同源相比,鉴定衍生自RRNA,叶片DNA或线粒体DNA的第一序列信息拟南芥序列(登录号分别为X52322、AP000423和Y08501/Y08502)。通过这种方法,包含rRNA或线粒体DNA的序列从基因组序列中分离出来。
第二步是对非冗余蛋白数据库进行BlastX搜索。登录号和E从结果文件中提取最佳匹配的值。用于确定非冗余序列的标准是基于BlastX搜索中获得的登录号的唯一最佳匹配。如果两个或多个查询序列产生具有相同登录号的最佳匹配,则根据它们的登录号对它们进行排序E值。只有最小的序列EUnigene集中包含值。
注释和功能分类
基于NR数据库的BLASTX搜索的结果注释UNIGENE集。喷射匹配的定义线用作Unigene基因推定功能的描述。一个E价值阈值10-10,未满足此要求的UniGene基因被标注为未知。
我们对个体基因的功能分类遵循慕尼黑蛋白质序列信息中心(MIPS)定义的功能分类。拟南芥蒂利亚纳功能目录(MATDB;下载http://mips.gsf.de.).为了创建一个半自动化的功能分类管道,开发了两步程序。首先,使用UniGene集和MATDB执行BlastX搜索,需要一个E值< 10-10.轨迹的名字和E从结果文件中提取每个基因的最佳匹配值。其次,利用位点名称进行搜索,识别功能分类拟南芥功能目录。对于不符合半自动化方法功能分类标准的基因,根据注释和从NCBI网站下载的保守域数据库(CDD)与保守域搜索结果进行人工分类。
微卫星序列鉴定
通过聚类和组装步骤得到的3716条序列,共包含5.3 Mb的序列,搜索微卫星(简单序列重复;>的单核苷酸重复序列为15 bp, >的二核苷酸重复序列为14 bp, >的三核苷酸重复序列为15 bp, >的四核苷酸重复序列为16 bp, >的五核苷酸重复序列为20 bp。29.].为了更好地定位二核核苷酸重复,我们还使用了在华盛顿大学开发的Program Sputnik [57].该程序通过对插入、不匹配和删除实现一个评分系统,允许ssr上有轻微的缺陷。为了定位单核苷酸重复,我们使用了我们自己开发的一个简单的PERL脚本。
RT-PCR.
逆转录酶聚合酶链反应(rt - pcr)进行总RNA准备从三个星期老燕麦植物的叶子(各种杰拉尔德),孵化0分钟,15分钟,30分钟,45分钟,1 h, 2 h, 8 h和24 h + 4°C,使用上标™三世一步法rt - pcr系统(Invitrogene™)。RNA样本首先用dna酶处理Dnase I放大花园从Invitrogene™。来放大不同ASCBF.基因使用了以下引物:
ASCBF1.正向引物5“-CCACAGTCCACCGTATCAGCAAG-3”
ASCBF1.反向底漆5'-cgtctccttgaacttggtgcg-3'
ascbf3.正向引物5“-CGGGCAAAGTTGAGGCAGGC-3”
ascbf3.反向底漆5'-taggctctgcctcgcgcaccttc-3'
ASCBF4.正向引物5“-CCCAGCCTTCAGCAGCGTC-3”
ASCBF4.反向引物5“-TCTCCACAGTCTCCTCCGTGC-3”
为ASCBF1.基因a产物大小为174 bpascbf3.104 bp及ASCBF4.172个基点。的AsActin基因用作对照,并使用正向引物5'-gcgacaatggaactggc-3和反向引物5'-gtggtgaaggagtaacctctctcg-3'扩增。在这种情况下,预期的产品尺寸为580bp。RT-PCR反应根据制造商的说明进行,每次反应中使用100ng总RNA。使用30分钟的逆转录,然后使用具有30,35或40个循环的PCR扩增步骤。为了验证RT-PCR反应的结果,将相应的RT-PCR反应混合物的相应量(30%)施加在含有溴化乙锭(0.5ng / ml)的1%琼脂糖凝胶上。
参考
- 1。
谷类作物和其他禾草的关系。中国科学(d辑:地球科学),1998,95(5):2005-2010。10.1073 / pnas.95.5.2005。
- 2。
贝纳特MD,史密斯·杰布:核DNA在被子培养物中量。Philos Trans R SoC Lond B Biol Sci。1976,274(933):227-274。
- 3。
绿色c:燕麦在一个新的时代。Semundo Limited,49北路,剑桥大北阿宾顿;1999:88。
- 4.
格拉尔SR,梵恩斯·瓦斯·王,公会M:可溶性纤维和血清脂质:文献综述。J AM Diet Assoc。1994,94(4):425-436。10.1016 / 0002-8223(94)90099-x。
- 5.
Brown L,Rosner B,Willett WW,麻袋FM:饮食纤维的胆固醇效果:荟萃分析。AM J Clin Nutr。1999,69(1):30-42。
- 6.
Trowell HC,Burkitt DP:西部疾病:它们的出现和预防。剑桥,马:哈佛大学出版社;1981年。
- 7.
Janatuinen ek,Pikkarainen博士,Kempainen Ta,Kosma VM,Jarvinen RM,Uusitupa Mi,Julkunen RJ:饮食与乳糜泻的成年人中的饮食比较。新英格尔J Med。1995,333:1033 - 1037-10.1056 / NEJM199510193331602。
- 8.
Janatuinen EK, Kemppainen TA, Julkunen RJ, Kosma VM, Maki M, Heikkinen M, Uusitupa MI: 5年摄入燕麦对腹腔疾病无危害。内脏。2002,50:332-335。10.1136 / gut.50.3.332。
- 9.
A:成年腹腔患者确实能耐受大量燕麦。中国临床营养杂志,2003,21(4):427 - 434。10.1038 / sj.ejcn.1601525。
- 10。
燕麦中抗氧化剂的提取与鉴定。美国石油化学学会杂志。1991,68:365-370。
- 11.
燕麦油及其组分的抗氧化作用。《脂肪科学技术》,1990,92:319-321。
- 12.
邢颖,白萍:燕麦壳中抗氧化剂的鉴定及其作用。石油化学学报,2003,31(4):457 - 461。
- 13。
Auerbach RH, Gray DA:燕麦抗氧化剂的提取和测定-迈向商业过程。中国农业科学。1999,79:273-282-10.1002 /(SICI)1097-0010(19990301)79:3<385::AID-JSFA260>3.0 co;2-L。
- 14.
Tian LL,White PJ:大豆和棉籽油中燕麦提取物的抗氧化活性。J AM Oil Chem SoC。1994年,70:1079 -11085。
- 15.
高等植物冷驯化的分子遗传学研究。遗传学的进步。1990年,28日:99 - 131。
- 16.
Sarhan F,Danyluk J:工程冷耐寒作物 - 用于施工主交换机。植物科学趋势。1998,3(8):289-291。10.1016 / s1360-1385(98)01285-0。
- 17.
王志强,王志强,王志强,等。低温敏感和耐低温禾本科植物酸性脱氢酶基因的克隆及表达分析。王志强,王志强,王志强,等。10.1016 / 0014 - 5793 (94) 00353 - x。
- 18.
Tatusov RL,Fedorova ND,杰克逊JD,Jacobs Ar,Kiryutin B,Krylov,Krylov DM,Mazumder R,Mekhedov SL,Nikolskaya An,Rao BS,Smirnov S,Sverdlov Av,Vasudevan S,Wolf Yi,Yin JJ,Natale Da:COG数据库:更新版本包括真核节。BMC生物信息学。2003,4(1):41-10.1186 / 1471-2105-4-41。
- 19.
NCBI:表达序列标签数据库[http://www.ncbi.nlm.nih.gov/dbEST]。2005
- 20。
Tsuda k,Tsvetanovs,Takumi S,森,阿纳萨夫A,Nakamura C:来自普通小麦(Triticum aestivum L.)的冷响应群-3 lea / Rab相关的Cor基因家族的新成员。基因类型系统。2000,75(4):179-188。10.1266 / ggs.75.179。
- 21。
关键词:拟南芥,质膜水通道蛋白,非生物胁迫,表达分析acta botanica sinica(云南植物学报),2004,34(5):713-725。10.1023 / B: PLAN.0000040900.61345.a6。
- 22。
杨志刚,王志刚,王志刚,等。水稻抗脱落酸和盐胁迫基因的克隆及表达分析。植物学报。1997,20(4):443-454。10.1007 / s004250050148。
- 23。
Chinnusamy V,OHTA M,Kanrar S,Lee Bh,Hong X,Zarwal M,朱JK:ICE1:拟南芥冷诱导的转录组和冷冻耐受性的调节因子。基因开发。2003,17(8):1043-1054。10.1101 / gad.1077503。
- 24。
关男,Narusaka男,石田Ĵ,南T,藤田男,大野Y,神谷A,中岛男,Enju A,樱井T,佐藤男,秋山K,塔吉T,山口筱崎K,Carninci P,河合Ĵ,Hayashizaki Y,Shinozaki K:使用全长cDNA微阵列监测干旱,冷和高盐度应力下的7000个拟南芥基因的表达曲线。工厂J.2002,31(3):279-292。10.1046 / J.1365-313x.2002.01359.x。
- 25。
Fowler, Thomashow MF:拟南芥转录组分析表明,在冷适应过程中,除了CBF冷响应途径外,还有多个调节途径被激活。植物学报。2002,14(8):1675-1690。10.1105 / tpc.003483。
- 26。
植物冷驯化:抗冻基因及其调控机制。植物营养与肥料学报,2003,21(5):591 - 598。10.1146 / annurev.arplant.50.1.571。
- 27.
拟南芥dreb转录因子ERF/AP2结构域的dna结合特异性研究。中国生物化学学报。2002,29(3):337 - 341。10.1006 / bbrc.2001.6299。
- 28.
Dubouzet JG,Sakuma Y,Ito Y,Kasuga M,Dubouzet Eg,Miura S,Seki M,Shinozaki K,Yamaguchi-Shinozaki K:Osdreb基因在水稻中,Osyza sativa L.,编码转录激活剂在干旱中起作用 - 高度 -盐和冷响应基因表达。工厂J. 2003,33(4):751-763。10.1046 / J.1365-313x.2003.01661.x。
- 29.
Cardle L, Ramsay L, Milbourne D, Macaulay M, Marshall D, Waugh R:植物物理聚类简单序列重复的计算和实验表征。中国生物医学工程学报,2017,32(2):457 - 461。
- 30.
Stockinger EJ,Gilmour SJ,Thomashow MF:拟南芥CBF1编码AP2域的转录激活剂,其与C重复/ DRE结合,CIS作用DNA调节元素响应低温和水缺损刺激转录。Proc Natl Acad Sci U S A. 1997,94(3):1035-1040。10.1073 / pnas.94.3.1035。
- 31。
Gilmour SJ,Zarka DG,Stockinger EJ,Salazar MP,Houghton JM,Thomashow MF:AP2转录激活剂的拟南芥CBF系列的低温调节作为冷诱导的Cor基因表达的早期步骤。工厂J.998,16(4):433-442。10.1046 / J.1365-313x.1998.00310.x。
- 32。
Weber JL:人(DC-DA)n的信息性。(DG-DT)n多态性。基因组学。1990,7:524 -5530。10.1016 / 0888-7543(90)90195-z。
- 33。
王志强,王志强,王志强:豆科植物微卫星指纹图谱研究。基因组学。1995,38:507 -5515。
- 34。
pcr扩增的微卫星标记在植物遗传学中的应用。植物学报,1993,3(1):175-182。10.1046 / j.1365 - 313 - x.1993.t01 - 9 - 00999. x。
- 35。
荣文J,Akkaya Ms,Bhahwat Aa,Lavi U,Cregan Pb:使用微卫星DNA标志物进行大豆基因型鉴定。理论和应用的遗传学。1995,90(1):43 -448。10.1007 / BF00220994。
- 36。
Sharon D,Adato A,Mhameed S,Lavi U,Hillel J,Gomolka M,Epplen C,Epplen J:使用简单序列重复和小型卫星探头的植物中的DNA指纹。Hortscience。1995,30(1):109 -1112。
- 37。
王志强,王志强,王志强,王志强:燕麦二倍体类群杂交的rflp图谱。基因组学。1992,35:765-771。
- 38。
Rayapati PJ,Gregory JW,Lee M,Wise RP:基于RFLP基因座的二倍体燕麦Avena的连锁地图和赋予普科尼亚冠军抵抗力的基因座。Avenae。理论和应用的遗传学。1995,89:831-837。
- 39.
O'Donoughue LS, Kianian SF, Rayapati PJ, Penner GA, Sorrells ME, Tanksley SD, Phillips RL, Rines HW, Lee M, Fedak G, Molnar SJ, Hoffman D, Salas CA, Wu B, Autrique E, Van Deynze A:栽培燕麦的分子图谱。基因组学。1995,38:368-380。
- 40。
Wight CP,Tinker Na,Kianian SF,Sorrells Me,O'Dooughue LS,Hoffman DL,GROH S,Scoles GJ,Li CD,韦伯斯特FH,Phillips RL,Rines HW,Livingston SM,Armstrong KC,Fedak G,Molnar SJ:在“Kanota”X'ogle'六倍oat(Avena SPP)中的分子标记图。通过附加标记和强大的框架增强。基因组。2003,46(1):28-47。10.1139 / g02-099。
- 41。
Gharti-Chhetri G, Olsson O: 7个瑞典燕麦(Avena sativa L.)品种不同外植体的愈伤组织增殖和植株再生体系的建立。(已提交植物细胞组织和器官培养)。2005.
- 42。
Gharti-Chhetri Olsson, Gharti-Chhetri GB:新型转化方法和转化植物专利申请。2005.
- 43。
Ausubel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA, Struhl K:分子生物学的简短协议。纽约,John Wiley & Sons;1995.
- 44。
Church GM, Gilbert W:基因组测序技术。临床生物学进展1985,177:17-21。
- 45。
Yulan P,Naomi T,Meng KL,Minoru SHK:通过通用PCR扩增方法的总RNA。基因组研究。2001,11(9):1553-1558。10.1101 / gr.185501。
- 46。
Altschul SF,Gish W,Miller W,Myers EW,Lipman DJ:基本的本地对齐搜索工具。J Mol Biol。1990年,215:403-410。10.1006 / jmbi.1990.9999。
- 47。
NCBI:基本的局部对齐搜索工具[http://www.ncbi.nlm.nih.gov/blast]。2005.
- 48。
米P,Longden I,Bleasby A:浮雕:欧洲分子生物学开放的遗传学软件套件趋势。2000,16:276-277。
- 49。
EMBOSS:欧洲分子生物学开放软件套件[http://emboss.sourceforge.net]。2005.
- 50.
Apweiler R: InterProScan - InterPro签名识别方法的集成平台。生物信息学。2001,17:847-848。10.1093 /生物信息学/ 17.9.847。
- 51.
ebi:译员[http://www.ebi.ac.uk/Interproscan/]。2005.
- 52。
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、特定位置间隙惩罚和权重矩阵选择来提高累进式多序列比对的灵敏度。核酸研究,1994,22:4673-4680。
- 53。
NCBI:国家生物技术中心信息信息[http://www.ncbi.nlm.nih.gov]。2005.
- 54。
黄旭,马丹A: CAP3: DNA序列组装程序。中国生物医学工程学报,2019,39(9):1043 - 1046。10.1101 / gr.9.9.868。
- 55。
TGI集群工具[http://www.tigr.org/software/]。2003.
- 56。
一种用于DNA序列对齐的贪婪算法。中国生物医学工程学报,2000,17(1):123 - 127。10.1089 / 10665270050081478。
- 57。
abajan C: Sputnik [http://espressosoftware.com/pages/sputnik.jsp]。1994.
- 58。
ebi:embl-核苷酸序列数据库[http://www.ebi.ac.uk/embl/index.html]。2005.
确认
这项工作得到了VL-foundation、瑞典农民供应和作物营销合作社、瑞典基因组学和生物信息学研究学院以及瑞典研究理事会(VR)的资助。
作者信息
隶属关系
相应的作者
额外的信息
作者的贡献
MB贡献了EST数据分析、系统发育分析、CBF表达实验和论文撰写。AL对表达最多的基因进行了数据分析。GC种植燕麦植株并进行冷冻实验。SZ对燕麦进行冷诱导,制备RNA,并对用于cDNA制备的RNA进行质量和诱导控制。BO做了EST数据分析,SSR的鉴定和书写。OO提供了工作的计划、监督和融资以及论文的写作。
作者为图像提交的原始文件
权利和权限
开放访问本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Bräutigam, M., Lindlöf, A., Zakhrabekova, S。et al。冷适应燕麦9792 EST序列的产生与分析,燕麦属漂白亚麻纤维卷.BMC植物杂志5,18(2005)。https://doi.org/10.1186/1471-2229-5-18
已收到:
接受:
发表:
关键字
- 冷压力
- 低温驯化
- BLASTX搜索
- Avena Sativa.
- 质膜固有蛋白