抽象的
背景
NADP-苹果酶(NAPD-ME)和丙酮酸正磷酸二磷酸二基酶(PPDK)是参与C的重要酶4.光合作用。然而,C基因的进化历史和驱动这些基因进化的力量4.植物并不完全理解。
结果
我们确认162nadp-me和35PPDK对25个种的基因进行了分析,并构建了各自的系统发育树。我们分类nadp-me基因分成四个分支,A1,A2,B1和B2,而PPDK被分为两个分支,其中单焦点在分支机构中,并且Dicots在分支机构中。对选择性压力的分析napd-me和PPDK基因家族在NADP-ME的A5分支中鉴定出四个阳性选择的位点,包括在PPDK的E分支中的94h和196h处,在后概率阈值为95%。正面选择的位点位于螺旋和片状区域中。定量RT-PCR(QRT-PCR)分析显示表达水平为6nadp-me和2PPDK暴露于光线后,来自Foxtail Millet的基因是上调。
结论
本研究表明,在C中肯定选择了NADP-ME和PPDK演变的位置4.植物。它提供了有关植物分类和正面选择的信息nadp-me和PPDK基因,结果应在进一步研究C的进一步研究C4.植物。
背景
光合作用是植物将太阳能转化为化学能的过程。这使他们能够制作自己的发展食物[1].高植物中的光合作用可以分为c3., C4.和古代酸代谢(CAM)基于它们在导致不同初始光合作用产品的过程中固定碳。大多数土地植物使用c3.途径,而c4.和凸轮植物从c演变3.植物(2那3.].C4.植物比C更有效3.植物利用CO2导致亚热带和热带环境的卓越适应性,较低浓度的CO2,更加紧张的环境[4.].许多研究都集中在了解碳的固碳效率和机理上4.植物(5.那6.].
在涉及C的许多酶中4.光合作用途径PPDK和NADP-ME被认为是最重要的[7.那8.].
PPDK是一种控制C中的光合速率的关键酶4.植物(9.].许多PPDK基因C4.并且凸起的凸轮植物被克隆,示例在玉米和玉米和MESEMBRYANTHEMUM CRYSTLINUM[10.那11.].一种系统发育研究表明PPDK高粱和大米的基因是同源的[12.].详细分析PPDKPoaceae和Arabidopsis之间的同种型序列表明它们的序列分享了约20个叶绿体过渡肽(CTP)的氨基酸,证明了PPDK基因在单子叶和双子叶分化之前就已经进化了[12.].
nadp-me基因可分为光合作用基因和非光合作用基因。前者主要在叶绿体中起作用[13.]通过促进CO的释放来提高光合效率2在近端束鞘细胞中的苹果酸脱羧,在c中4.通过提供CO的植物2对碳固定的rubisco [14.那15.].基因组和系统发育分析表明nadp-mePoaceae的基因家族有四个分支,一个分支(nadp-meiv)在塑性体中表达。C4.-具体的nadp-me是否有一些密码子在正选择下被抑制,并且独立于nadp-meIV家族[16.那17.].
自然选择,生物进化中的关键因素,包括阳性选择,净化选择和中性选择[18.].基本替代率(非同义/同义,ω= DN / DS),确定改变后选择压力的索引,通常用于了解编码序列中的进化方向及其选择性强度。如果ω> 1,则基因可能经历阳性选择或存在新的氨基酸提供健身优势;ω= 1表示中性选择;ω<1的值表示净化选择[19.].作为自适应演化的重要依据,通过良好的传播和增加突变等位基因的频率的阳性选择功能[18.].
正面选择通常意味着新功能的出现[19.那20.].在C的转变中3.C4.途径阳性选择主要发生在C中的关键酶4.光环醚,如uphisco,磷酸泛蛋白羧基酶(Pepc),NADP-ME和PPDK [12.那21.那22.那23.那24.那25.那26.].例如,两个正面选择的大亚基(LSU)氨基酸取代,M309i和D149a,区分c4.来自祖先C的rubiscos3.物种 [21.].切换到C4.,21例在阳性选择下演化的21个氨基酸,并在大多数草c中融合到相似或相同的氨基酸4.Pepc谱系[22.].收购C.4.莎草中的PEPC (Cyperaceae.)在至少16个密码子上被阳性选择驱动[23.].以前的研究用来使用氨基酸的变异来研究C中的进化率4.-NADP-ME途径,发现大量残基处于显著正选择状态[24.].在C4.植物强的正面选择导致序列收敛[25.].例如,在C的29个残基之间4.nadp-mes和non c4.鉴定NADP-MES,残基284,450和539在C的演变期间被确定为在阳性选择下4.- 在草丛中,暗示他们在解释C之间的动力学和结构差异方面非常重要4.和非c4.团体[26.].系统发育分析还表明玉米PPDK基因及其高粱ortholog在显着的阳性选择下,暗示可能的功能变化[12.].
C的底层分子机制4.光合作用较差,很少有研究旨在了解阳性选择是否与NADP-ME和PPDK的演变相关4.植物。完成C的全基因组序列4.高粱和玉米等植物[27.那28.]并改善光合途径和进化的知识,为研究键C的演变和表达进行了稳定的基础4.酶基因。比较PPDK和nadp-me基因家庭在c中4.植物可以进一步了解这些基因在光合作用中的进化、功能和代谢作用。本研究探讨了人类的进化过程nadp-me和PPDK在藻类、苔藓、石松植物、单子叶植物和双子叶植物中,提供了C4.光合作用。
结果
的数量nadp-me和PPDK基因在植物
在25种,包括一个藻类,一只苔藓,一个单子叶,10个单子叶(包括6℃),总共162种NADP-ME和35个PPDK序列。(包括6℃4.)和12个Dicot(包括1 c4.)物种(附加文件1S1:表;额外的文件2:表S2)。有14个nadp-me大豆的基因。胡萝卜,棉花和杨树每个人都有9nadp-me基因和Selaginella Moellendorffii.有3个(附加文件1:表S1)。的数量PPDK基因较少,最大数量为香蕉种类Musa Acuminata..大多数其他物种只有1或2PPDK基因(附加文件2:表S2)。
NADP-ME和PPDK蛋白保守氨基酸序列分析
用于分析NADP-ME和PPDK蛋白的保守序列的MEME程序确定了20个图案(附加文件3.:表S3;额外的文件4.:表S4)。之中nadp-me基因,来自藻类的基因(CRE14.G629700.t1.1.那Cre14.g628650.t1.2那CRE14.G629750.T2.1.那Cre01.g022500.t1.2)没有包含主题13、14、15、17和19。亚家族A有两个独特的图案,17和19,而亚家族B有三个独特的图案,13,14和15(附加文件5.:图S1)。这PPDK绿藻基因缺乏主题15,而所有其他PPDK基因拥有所有20个候选主题(附加档案6.:图S2)。
系统发育nadp-me和PPDK
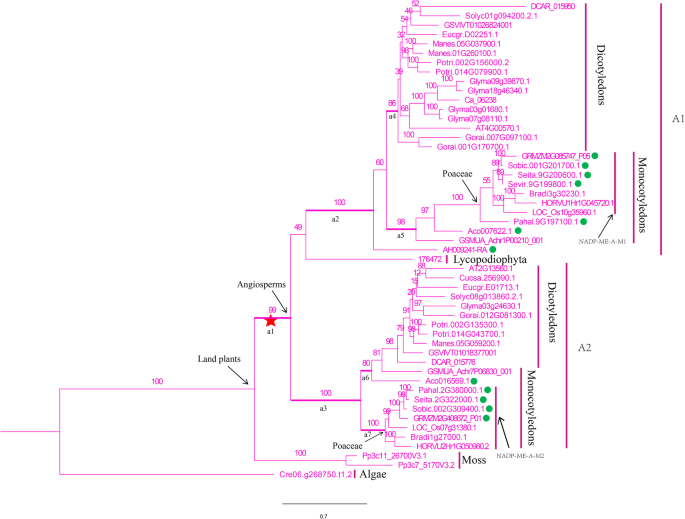
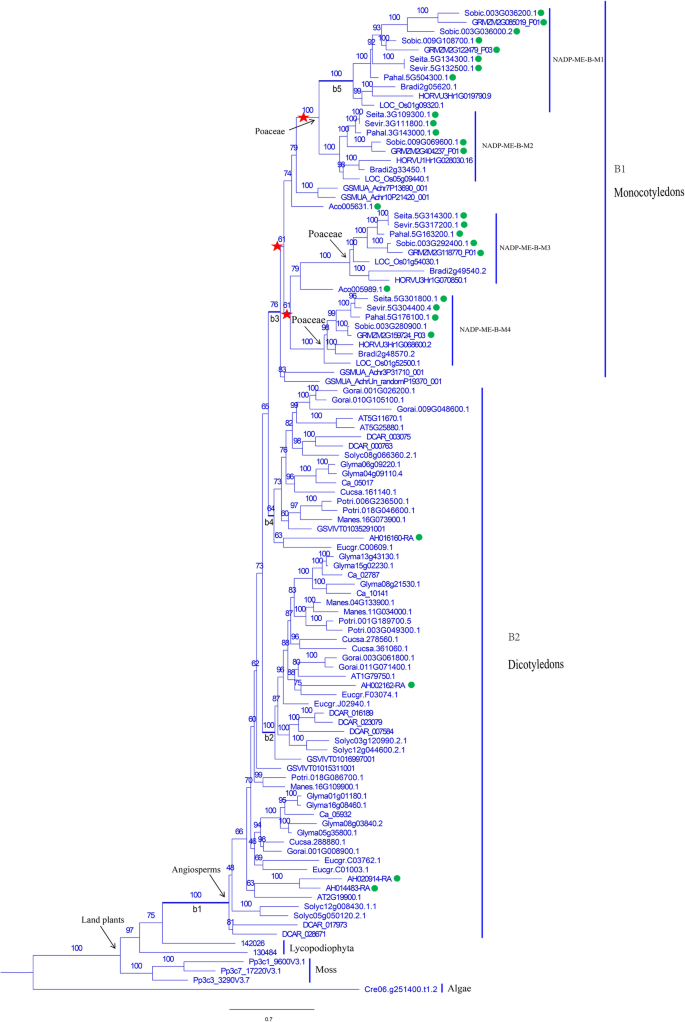
我们为所有162年构建了一个系统发育树nadp-me来自25种的基因,并发现他们分享了一个共同的祖先。藻类nadp-me是最古老的基因,并从其他地区发散。全基因组复制后分离亚属A和B(附加文件7.:图S3)。在亚族A中nadp-me基因首先在藻类中分支,其余的则分为A1和A2亚科。单子叶植物和双子叶植物各亚科间存在明显的聚类现象。在A1和A2单子叶植物分支中,nadp-me在Musa Acuminata.和ananas comosus.在Poaceae之前分支。在Poaceae内,nadp-me基因C4.植物彼此更密切相关(图。1).在B亚家族中,nadp-me藻类基因再次分支,其次是土地植物Physcomitrella金属盘和Selaginella Moellendorffii..在被子植物物种,nadp-me来自Dicots(B2亚家族)首先分支和nadp-me在基因重复后,单焦度分歧并形成B1亚家族,其接受了三种全基因组重复事件。喜欢一个亚家族,nadp-me在Musa Acuminata.和ananas comosus.早于淘eA的对应分支,其中有四个分支,即NADP-ME-B-M1,NADP-ME-B-M2,NADP-ME-B-M3和NADP-ME-B-M4(图.2).我们发现了nadp-me基因被聚集并密切相关的C3.和c4.物种组。
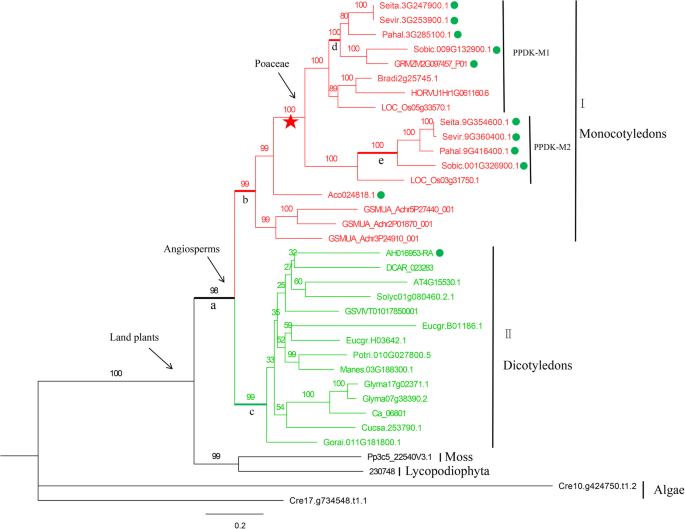
所有35PPDK25种物种的基因用于构建系统发育树。这PPDK绿藻的基因首先是分支,进一步分解为亚属I和II。亚家族,我由单焦点和亚家族组成,由Dicots组成。这PPDK亚家族的基因我第一次出现在Musa Acuminata.和ananas comosus.后来分歧到痘皮。然后在这种分歧后发生全基因组重复,并形成两个主要分支,其中一个分支包括大麦,玉米和Brachypodium distachyon显示损失PPDK基因或缺乏保守的PPDK结构体。还有人发现PPDK基因C4.植物密切相关(图。3.).
对选择压力的分析nadp-me和PPDK基因
在A和B个子美中的每一个内的选择压力nadp-me研究了基因。在亚家族A中,M0和M3模型基于用于计算的现场模型。在M0型号Ω下为0.091,表明它在净化选择下。这P.- 从Chi平方测试比较M0和M3型号的值为0.000,表明ω值在基因座上不是恒定的(表1).对于分支模型,七个分支机构A1-A7被分配为前分支。分支模型结果表明,所有前部分支的Ω值为<1.似然比测试(LRT)显示,分支A1,A2和A3与所有Ω值<1的其他分支显着不同<如此建议净化选择(图。1;桌子1).分支部位模型表明,A1-A5的阳性选择的比例分别为5,0.2,5.8,11和1.5%,而A6和A7的比例接近0. A1的正面选定的位置的数量-A7为8,5,14,5,4,2和2,后概率为0.6。LRT结果表明,分支A1,A3和A5与M1模型显着不同(P. < 0.05). Interestingly, the a1 branch, ancestral to subfamilies A1 and A2, was stabilized after positive selection at both the a1 and a3 branches. On the contrary, a4 and a5 still had positively selected sites following positive selection at the a2 branch. This suggested that subfamily A1 had undergone different levels of positive selection at different branches. The a5 and a7 branches comprised mostly monocots and C4.植物(图。1;桌子1).对于B亚族,在位点模型上ω值与A亚族相似。LRT表明,B亚家族仍在纯化选择中,ω值在位点之间存在差异(表2)2).根据分支和分支位点模型,b1-b5的阳性位点比例分别为5.5、0、0.6、8.9和1.8%,ω值远小于1(表1)2).B1-B5的正选数的数量为8,0,2,0和5,后概率为0.6(图。2;桌子2).得出结论,B1是最古老的分支nadp-me亚家族B中的基因,共有8个正面选择的位点。B3和B5分支具有2和5个阳性选择的位点,而B2和B4分支均包含双斑点,并且在后部概率下没有阳性选择的位点0.6,因此表明B2和B4分支比B3更加保守B5分支,并且亚家族B中B1至B3和B5的进化步骤相当复杂(图。2;桌子2).
为了PPDKM0模型和M3模型的LRT比较结果表明P.- 基于网站模型为0.000。这表明ω值在站点上不是恒定的,类似于nadp-me基因系列结果(表3.).分支机构A-E被分配为分支模型中的前景分支,其Ω值小于1,暗示净化选择。有趣的是,来自A至E的Ω值逐渐增加,具有(0.0006)
NADP-ME和PPDK的蛋白结构特征
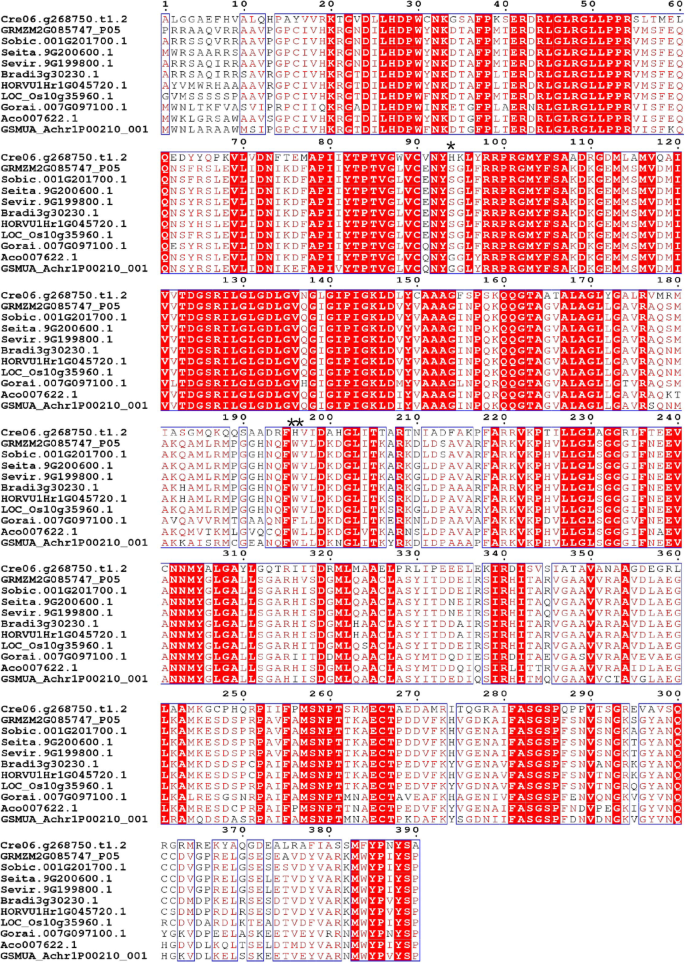
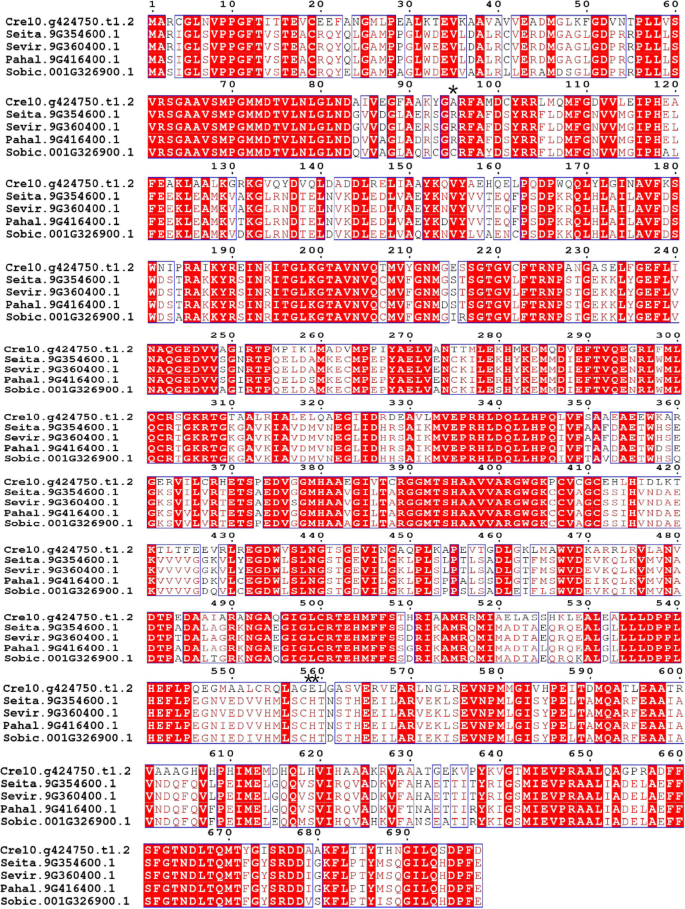
基于上述系统发育关系和阳性选择分析,我们使用NADP-ME的蛋白质序列对准在A5分支和E分支的PPDK中进行了详细的结构和功能研究,该蛋白质序列在E分支中含有单码和C.4.植物分别。Cre06.g268750.t1.2在A5分支机构和CRE10.G424750.t1.2在E分支中用作参考序列以进一步分析。A5分支中的位点94h和196h(图。4.在E分支中,95A和559E(图。5.)在95%的后概率阈值下显着呈正选择。区别和高度保守的地区。
NADP-ME在A5分支中的氨基酸序列的多对准。Cre06.g268750.t1.2那GRMZM2G085747_P05那sobic.001g201700.1.那Seita.9G200600.1那sevir.9g199800.1.那Bradi3g30230.1那HORVU1Hr1G045720.1那loc_os10g35960.1那gorai.007g097100.1.那Aco007622.1, 和gsmua_achr1p00210_001代表nadp-me基因Chlamydomonas Reinhardtii.那Zea Mays.那高粱双色那Setaria Italica.那Setaria Viridis.那Brachypodium distachyon那Hordeum Vulgare.那奥雅萨苜蓿那Gossypium raimondii.那ananas comosus., 和Musa Acuminata., 分别。在上述11个单圈子中的NADP-ME中肯定选择的位点被标记并通过ESPRIPT3.0显示(http://espript.ibcp.fr/espript/cgi-bin/espript.cgi.).Cre06.g268750.t1.2用作参考序列。后概率(P.)被指出:*,P. > 95%; **,P.> 99%。保守区域呈盒状,高度保守位点呈红色
e分支中PPDK氨基酸序列的多对准。CRE10.G424750.t1.2那seita.9g354600.1.那sevir.9g360400.1.那Pahal.9G416400.1.和sobic.001g326900.1.,代表PPDK基因Chlamydomonas Reinhardtii.那Setaria Italica.那Setaria Viridis.那Panicum Hallii., 和高粱双色, 分别。在上述五个C中为PPDK提供正面选择的网站4.植物被标记并通过ESPRIPT3.0显示(http://espript.ibcp.fr/espript/cgi-bin/espript.cgi.).CRE10.G424750.t1.2用作参考序列。后概率(P.)被指出:*,P. > 95%; **,P.> 99%。保守区域呈盒状,高度保守位点呈红色
NADP-ME和PPDK三维结构上正选择位点的分布
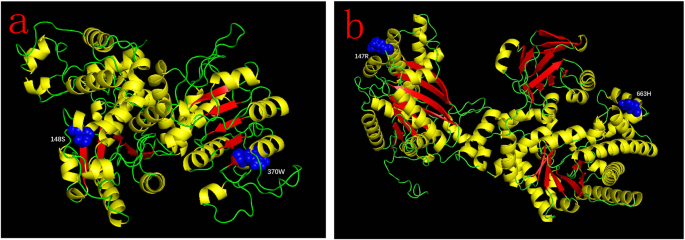
我们采取了三维(3D)模型seita.9G200600.1和seita.9g354600.1.并以此为例进行了实证分析。如图所示。6.A,NADP-ME-A的A5分支中的正面选定的位置94h和196h被映射到位点148s和370 wseita.9G200600.1.类似地,将PPDK的e分支中的阳性选择的位点95a和559e映射到位点147r和663hseita.9g354600.1.(图。6.b)。3D模型中的黄色表示螺旋区域,红色表示纸张区域,蓝色对应于特定的氨基酸。在螺旋区域中,147R,663H位于螺旋区域中,并且位于纸张区域中的370W(图。6.).
Foxtail小米的表达分析nadp-me和PPDK由QRT-PCR测定的基因
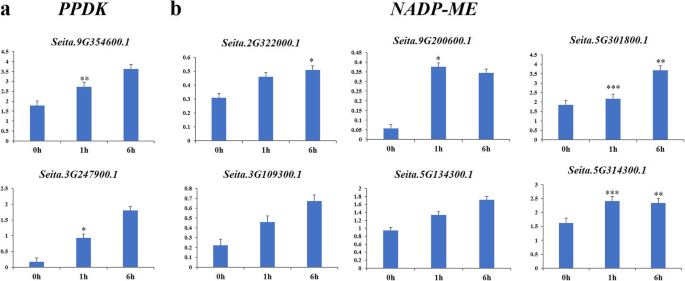
根据系统发育关系(图。1那2和3.),我们选择了6nadp-me和2PPDK谷子基因在光处理后的qRT-PCR。光照射1 h后,这些基因的表达水平均上调。除了nadp-me的基因,seita.5g314300.1.和Seita.9G200600.1,其余经光处理6 h后表达水平较高(图2)。7.;额外的文件8.:表S5)。
讨论
演变的nadp-me和PPDK基因家庭
C4.光合作用大约在3000万年前进化而来[29.].Angiosperm C.4.植物物种随后经过62个独立进化事件[30.].大多数C.4.植物是单码,包括4600种草和1600种莎草种类,而只有1600℃4.来自16个家庭的物种是Dicots,其中75%的家庭在Chenopodiaceae,Amanthaceae,Euphorbiaceae和Asteraceae中有75%[31.].以前的研究得出结论,尽管C的特定细胞结构4.植物酶Pepc,NADP-ME和PPDK对于C至关重要4.光合作用 [32.那33.].有趣的是,数量的增加nadp-me和PPDK基因发生在进化中。各种研究表明,在植物演进过程中发生了多种重复事件,包括分离单焦点和平坦的γ事件[34.]和ρ事件发生在小麦,玉米和米饭分歧之前发生的,但在草地和菠萝分歧后[35.[Poaceae中发生的τ和σ事件[36.].
在这项研究中,14和7nadp-me基因分别在大豆和玉米中被鉴定(附加文件1:表S1)。虽然玉米基因组大小(2300 MB)的大豆(1100 MB)的两倍多(1100 MB)[28.那37.] 的数量nadp-me玉米基因小于大豆,表明膨胀的扩张nadp-me基因家族不是通过基因组重复,但在单子叶和单萝卜物种的分歧后由不同的膨胀模式引起[38.那39.].对于35.PPDK本研究中发现的25个物种的基因中,大多数物种只有一个或两个成员(附加文件)2:表S2)。相比nadp-me数字的数量PPDK在进化过程中,基因数量减少但更加稳定。
nadp-me和PPDK基因广泛存在于光合植物物种中,如藻类、苔藓、蕨类、裸子植物和被子植物[40那41.].从本研究中构建的系统发育树上,我们得出结论nadp-me包含所有双子叶植物的B2分支比包含所有单子叶植物的B1分支进化得更早,这表明B亚家族在单子叶植物和双子叶植物分化后独立进化,这一步被称为γ事件(图2)。2)[34.].系统发育树PPDK基因家族表明,单焦点分枝并形成了亚家族,然后在亚家族II形成的亚家族之前,表明PPDK基因家族在单子叶和双子叶分化后独立进化[34.].在Poaceae中,单焦点和双点内有明确的聚类。例如,NADP.和PPDKC.的基因4.植物比C彼此更密切相关3.植物(无花果。1那2和3.).我们推断出来nadp-me和PPDKPoaceae中的基因家庭在单码本的ρ事件后接受了独立的演变[36.].此外,nadp-me和PPDK在C.4.植物比C更紧密地聚集在一起3.植物物种,可能是由于C的光合效率越高4.植物。
识别肯定选定的网站及其功能意义
本研究使用了网站,分支和分支机构模型来研究选择压力对的影响nadp-me和PPDK基因家庭。站点和分支模型都未能检测到任何正面位点,可能通过净化选择和中性漂移来否定[42.那43.].分支位点模型是最准确的,可以检测到特定分支上罕见的正选择位点[44.].分支位点模型共检测出55个位点,后验概率为0.6nadp-me基因家族(表1和2).在亚家族A中,我们发现总共8,5,14,5,4,2和2个阳性选择的A1-A7分支的位点(图。1;桌子1).在亚家草B中,我们发现B1-B5分支的8,0,2,0和5个阳性选择的位点(图。2;桌子2).分支模型PPDK基因家族揭示了五个前部分支的ω值小于1,表明强净化选择强大(表3.).分支站点模型检测到1,1,0,0和8个用于分支A-E的正面选择的站点(表3.).
两个网站和分支机构模型都建议nadp-me和PPDK基因家族在维持正常基因功能的同时,大多经历了纯化选择。通过更精确的分支位点模型检测少数积极选择的位点,表明在进化过程中,为了适应不断变化的环境,只发生了少数有益的突变[45.].C4.植物能够利用较少量的CO2与他们的c相比3.同行。这可能与在两者中发现的阳性选择位点有关nadp-me和PPDKC.的家庭4.植物。
阳性选择是在群体中的有利突变的保留和传播,并且长期被认为是蛋白质功能的转变的同义[45.].确定阳性选择的量具有广泛的影响,用于了解基因组功能和遗传变异的维护[46.].在该研究中,在PPDK的E分支的NADP-ME和95A和559E的A5分支中,在PPDK的E分支中,在PPDK的e55%的概率阈值下,在95%的e分支中鉴定出四种正选,包括94h和196h。(图。4.和5.).以往的研究表明,初级结构的微小变化是每个NADP-ME和PPDK亚型不同动力学行为的原因[47.那48.].阐明C的肯定选定网站的角色4.植物演化和探索C中肯定地点与高光合速率的关系4.植物,3D模型seita.9G200600.1和seita.9g354600.1.被吸引。如图所示。6.,阳性选择位点148S、147R和663H位于螺旋区,而370 W位于薄片区。这些阳性氨基酸选择位点可能反映了C4.和c3.导致C的植物4.植物具有更高的光合能力。这些结果还表明,NADP-ME和PPDK家族成员的氨基酸位点在植物演变期间发生变化,并且进化率不同。它还为进一步分析了NADP-ME和PPDK的职能提供了优先级。
进一步分析NADP-ME的a5分支和PPDK的e分支的基因,发现C4.在a5分枝的植物包括GRMZM2G085747_P05那sobic.001g201700.1.那SEVIR.9G198800.1.那Pahal.9G197100.1.那Aco007622.1,和Seita.9G200600.1(图。1).以前的研究表明玉米GRMZM2G085747通过叶血管束玉米鞘细胞中的碳固定参与钙普循环(A C4.物种)在光合作用期间[49.].高粱nadp-me基因sobic.001g201700.在C中显示出高记录性丰度4.通路(50.].此外,比较一个c3.和11℃4.草种(Poaceae)表明了成绩单丰富sobic.001g201700.在c中始终升高4.物种 [24.].PPDK成员的E分支都属于C4.植物,包括seita.9g354600.1.那sevir.9g360400.1.那Pahal.9G416400.1,和sobic.001g326900.1.(图。3.).之前的研究报告说Sobic.001G326900.在c中显示出高记录性丰度4.通路(50.].在本研究中,当后验概率阈值为95%时,NADP-ME a5分支中的94H和196H位点和PPDK e分支中的95A和559E位点被确定为阳性选择(图5)。4.和5.).GRMZM2G085747和sobic.001g201700.在NADP-ME的A5分行,和Sobic.001G326900.在PPDK的E分支中都涉及C.4.光合作用 [24.那49.那50.].我们的结果表明,在C期间,这些网站被肯定地选择了高光合速率4.进化。
结论
一百六十二nadp-me和35PPDK特征在25种的基因具有高度相似的亚壳内的基因组合物。系统发育分析表明nadp-me和PPDK基因分别可以分别置于四个和两个分支中。这nadp-me和PPDK基因C4.物种的进化关系比C更近3.物种。对选择性压力的分析napd-me和PPDK基因家族在NADP-ME,95A和559E的A5分支中鉴定出四个正面选定的位点,在PPDK的E分支中,在后概率阈值为95%。正面选择的位点位于螺旋和板材区域。推断出积极的选择正在推动NADP-ME和PPDK的演变4.物种。本研究有助于进一步了解NADP-ME和PPDK在C3.和c4.物种,并向C的进化生物学提供洞察4.植物。
方法
数据集
保守的NADP-ME和拟南芥和稻米的PPDK蛋白序列是从公共数据库UNIPROT获得的(https://www.uniprot.org/)和tair(https://www.arabidopsis.org/).从植物血红素V12获得25种的所有NADP-ME和PPDK蛋白序列和CDS(编码序列),包括藻类,苔藓,血管薄膜,单子叶和双旋肽物种的代表(https://phytozome.jgi.doe.gov/pz/portal.html),并纳入本地数据库。利用阈值为E < 1e-5的blastp将每个序列与其他物种的NADP-ME和PPDK蛋白序列以及拟南芥和水稻的NADP-ME和PPDK蛋白序列进行比较。利用CDD和Pfam分析序列是否含有保守的NADP-ME和PPDK蛋白结构。去除不完整的蛋白质结构。
利用Expasy分析NADP-ME和PPDK蛋白序列的分子量和等电点https://web.expasy.org/compute_pi/).
系统发育树木的构建与保守蛋白序列分析
利用MUSCLE3.8.31软件对候选NADP-ME和PPDK蛋白序列进行多重比较[51.].邻居加入(NJ)树通过软件Mega 7.0使用泊松模型使用1000个引导复制,使用成对方法填补了空白,其他参数基于默认值[52.].使用贝叶斯信息标准(BIC)和1000个引导复制,为NADP-ME和PPDK构建最大可能性(ML)树和使用软件IQ-Tree1.6.5 [53.].使用参数M:仅测试估计ML树的最佳模型。使用图3R的构建系统发育树的可视化。
使用-NMOTIFS使用软件MEME 2.12.0的保守蛋白序列分析:20,--minw:10,maxw:50 [54.].其他参数基于默认值。使用TBtools软件将结果可视化。
自然选择压力分析
使用肌肉3.8.31软件测定来自多个比较分析的NADP-ME和PPDK的蛋白质序列,CD和对齐的蛋白质序列提交给在线工具Pal2nal(http://www.bork.embl.de/pal2nal/)用于密码子对齐。使用软件PAML4.9e计算选择压力,ω<1表示净化选择,ω= 1表示中立,ω> 1表示正选择[55.].采用三种方法计算选择压力:(1)场地模型,试验采用M3和M0模型;(2)比较前景分支和背景分支以检验阳性选择的分支特定模型;(3)分支站点模型(模型A),用于测试积极选定的站点。统计分析采用卡方检验。
蛋白序列的阳性选择和结构分析
排列重排的CDS和氨基酸进入PAL2NAL (http://www.bork.embl.de/pal2nal/),用于执行多个密码子对齐的Web工具。然后通过ESPRIPT V3来观察对齐的序列(http://espript.ibcp.fr/espript/cgi-bin/espript.cgi.).
Foxtail Millet的全长蛋白序列(Setaria Italica.)NADP-ME和PPDK被提交给I-Tasser服务器(https://zhanglab.ccmb.med.umich.edu/i-tasser/)来预测三维结构。在分支位点模型中,以95%的后验概率阈值对积极选择的位点进行测试,并通过PyMol v2.3 (http://pymolwiki.org.).
植物生长和收获
用于qRT-PCR的谷子品种玉谷1号由河南省安阳市农业科学研究所提供。种子表面用0.5% NaClO灭菌1 min,用灭菌蒸馏水清洗3次,然后用gm -琼脂培养基,4℃黑暗分层3天。种子萌发后,在27℃黑暗条件下培养3 d,然后转入27℃光照条件下(600 μmol m .- 2 s- 1).光处理0、1、6 h后采集叶片,立即用液氮冷冻,在−80℃保存,用于RNA分离。所有样本生物复制3次。
QRT-PCR.
由引物3设计的引物,使用cDNA序列Setaria Italica.v2.2(phytozome.jgi.doe.gov.)列在附加文件中9.:表S6。使用SYBR®Green PCR Master Mix Kit (Applied Biosystems, GA, USA)进行3个重复的qrt -PCR。数据采集和分析使用ABI7900系统(应用生物系统)进行。用2——ΔΔCT分析方法。
可用性数据和材料
本研究中产生或分析的所有数据均包含在原稿和补充信息文件中。
缩写
- 3 d:
-
三维
- BIC:
-
贝叶斯信息准则
- 凸轮:
-
景天酸代谢
- CD:
-
编码序列
- 轻轨交通:
-
可能性比率测试
- 路易斯安那州立大学:
-
大亚基
- ML:
-
最大似然
- nadp-me:
-
NADP-苹果酶
- NJ:
-
邻居加入
- Pepc:
-
磷丙酮酸羧化酶
- PPDK:
-
丙酮酸正磷酸盐dikinase
- QRT-PCR:
-
定量RT-PCR
参考文献
- 1。
Wohlfahrt G,GU L.叶片对叶片到全球光合作用研究的许多含义及其对光合作用研究的含义。植物细胞环境。2015; 38:2500-7。
- 2。
Aldous Sh,Weise Se,Sharkey Td,Waldera-Lupa DM,Stuhler K,Mallmann J,等。磷酸的演变enol.丙酮酸羧基酶蛋白激酶系列在c中3.和c4.Flaveria.SPP。植物理性。2014; 165:1076-91。
- 3.
Yin H,Guo Hb,Weston Dj,Borland Am,Ranjan P,Abraham Pe,等。Diel重新兴奋和古植物蛋白的阳性选择使凸轮光合作用的演变龙舌兰.BMC基因组学。2018;19:588。
- 4.
Caemmerer SV,Ghannoum O,Furbank RT。C4.光合作用:50年的发现和创新。J Exp Bot。2017; 68:97-102。
- 5。
王胜,杜立德,朱晓刚。C4.光合作用在C.3.稻米:生物化学和解剖因素的理论分析。植物细胞环境。2017; 40:80-94。
- 6。
尹x, Struik PC。增加叶片光合作用可以转化为更高的作物批量生产?使用作物模范稻米米的模拟研究。J Exp Bot。2017; 68:2345-60。
- 7。
Covshoff S,Szecowka M,Hughes Te,Smith-Unna R,Kelly S,Bailey KJ等人。C4.稻米的光合作用:来自杂草的洞察力echinochloa glabrescens..植物杂志。2016;170:57 - 73。
- 8。
陈YB,LU TC,Wang HX,Shen J,Bu Tt,Chao Q等。玉米叶绿体丙酮酸正磷酸酯直立通酶的后期改性揭示了其酶活性的精确调节机制。植物理性。2014; 165:534-49。
- 9。
Chastain CJ, Baird LM, Walker MT, Bergman CC, Novbatova GT, Mamani-Quispe CS等。玉米叶片PPDK调节蛋白isoform-2特异性于束鞘叶绿体,但却缺乏pi依赖性的PPDK激活活性。J Exp Bot. 2018; 69:1171-81。
- 10。
Fukayama H,Tsuchida H,Agarie S,Nomura M,Onodera H,Ono K,等。C的显着积累4.-特异性丙酮酸,正常磷酸二磷酸酯中的c3.植物,米饭。植物理性。2001; 127:1136-46。
- 11.
Matsuoka M,Furbank RT,Fukayama H,Miyao M. C的分子工程4.光合作用。Annu Rev植物Biol。2001; 52:297-314。
- 12.
王x,gowik u,tang h,鲍德·杰,韦斯特霍夫P,帕特森啊。C的比较基因组分析4.光合途径进化在草丛中。基因组Biol。2009; 10:R68。
- 13。
Arias Cl,Pavlovic T,Torcolese G,Badia MB,Gismondi M,Maurino VG等人。NADP依赖性苹果酶1参与脱钙酸反应拟南芥蒂利亚纳.前植物SCI。2018; 9:1637。
- 14。
遮荫对NADP-ME光合效率的影响小于PEP-CK和NAD-ME C4.草。J Exp Bot。2018; 69:3053-68。
- 15.
尹x, Struik PC。C的能量预算4.光合作用:专用电子传输模型的见解。新植物。2018; 218:986-98。
- 16。
Gerrard Wheeler Mc,Tronconi Ma,Drincovich Mf,Andreo CS,FlüggeUI,Maurino VG。NADP-MOLIC酶基因家族的综合分析拟南芥蒂利亚纳.植物理性。2005; 139:39-51。
- 17。
Christin Pa,Samaritani E,Petitpierre B,Salamin N,Besnard G.进化洞察C4.来自基因组学和系统发育的草丛中的光合亚型。基因组Biol Evol。2009; 1:221-30。
- 18。
从进化遗传学到人类免疫:选择如何塑造宿主防御基因。Nat Rev Genet, 2010; 11:17-30。
- 19。
Rosnow JJ,Edwards Ge,Roalson eh。Kranz和非Kranz C的正面选择4.软蒿科(藜科)磷酸烯醇丙酮酸羧化酶氨基酸。J Exp Bot. 2014; 65:3595-607。
- 20。
Kapralov MV, Smith JAC, Filatov DA。C4.eudicots:Amaranthaceae的分析Sensu Lato..Plos一个。2012; 7:E52974。
- 21。
Kapralov MV,Kubien DS,Andersson I,Filatov Da。C的rubisco动力学在C的演变过程中的变化4.光合作用Flaveria.(Asteraceae)与编码酶的基因的阳性选择有关。mol Biol Evol。2011; 28:1491-503。
- 22。
Christin P-A, Salamin N, Savolainen V, Duvall MR, Besnard G. C4.草类的光合作用是通过平行的适应性基因变化进化而来的。咕咕叫医学杂志。2007;17:1241-7。
- 23。
Besnard G, Muasya AM, Russier F, Roalson EH, Salamin N, Christin PA。Phylogenomics C4.淀粉(Cyperaceae)的光合作用:多种外观和遗传收敛。mol Biol Evol。2009; 26:1909-19。
- 24.
Watson-Lazowski A, Papanicolaou A, Sharwood R, Ghannoum O.研究C4.使用转录物和氨基酸变异的草4.光合基因。Photosynth Res。2018; 138:233-48。
- 25.
王L,Peterson RB,Brutnell TP。C的监管机制C.4.光合作用。新植物。2011; 190:9-20。
- 26.
Saigo M,Alvarez Ce,Andreo CS,Drincovich MF。来自草地的塑性鼻涕酶:解开到C的方式4.特定的亚型。植物生理学报。2013;63:39-48。
- 27.
Paterson啊,鲍德·杰,Bruggmann r,Dubchak I,Grimwood J,Gundlach H,等。高粱双色基因组和草的多样化。自然。2009; 457:551-6。
- 28.
Schnable PS,Ware D,Fulton Rs,Stein JC,Wei F,Pasternak S等人。B73玉米基因组:复杂性,多样性和动态。科学。2009; 326:1112-5。
- 29.
Aubry S, Kelly S, Kümpers BM, Smith-Unna RD, Hibberd JM。基因表达的深度进化比较确定了C的两个独立来源的反式因子的平行招募4.光合作用。Plos Genet。2014; 10:E1004365。
- 30。
Sage RF, Christin PA, Edwards EJ。C4.地球的植物谱系。J Exp Bot。2011; 62:3155-69。
- 31。
gowik u,Westhoff P.来自C的路径3.C4.光合作用。植物理性。2011; 155:56-63。
- 32。
王勇,Bräutigam A,韦伯AP,朱兴国。三种不同的生化亚型C4.光合作用?建模分析。J Exp Bot。2014; 65:3567-78。
- 33。
姜湖,陈烨,郑俊,陈泽,刘y,陶伊等。玉米丙酮酸正磷酸二磷酸氨基酶调节蛋白可逆磷酸化的结构基础。植物理性。2016; 170:732-41。
- 34。
Bowers Je,Chapman Ba,Rong J,Paterson啊。通过染色体复制事件的系统发育分析解开缓解性植物基因组演变。自然。2003; 422:433-8。
- 35。
Ming R,Van Buren R,Wai Cm,Tang H,Schatz MC,Bowers Je,等。菠萝基因组和凸轮光合作用的演变。NAT Genet。2015; 47:1435-42。
- 36。
Mckain Mr,Tang H,McNeal Jr,Ayyampalayam S,Davis Ji,De Pamphilis CW,等。古代古代多倍体和基因组进化的系统核发科学评估。基因组Biol Evol。2016; 8:1150-64。
- 37。
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W等人。古代聚丙烯大豆的基因组序列。自然。2010; 463:178-83。
- 38。
秦Z,王义,王Q,李安,侯F,张L.植物基因组简单序列重复的演化分析。Plos一个。2015; 10:e0144108。
- 39。
D'Hont A,Denoeud F,燕麦JM,Baurens Fc,Carreel F,Garsmeur O,等。香蕉 (Musa Acuminata.)基因组和单子叶植物的演变。自然。2012; 488:213-7。
- 40.
Motti Ca,Bourne Dg,Burnell Jn,Doyle Jr,Haines DS,Liptrot Ch,等。筛选C抑制剂的海洋真菌4.植物酶丙酮酸磷酸盐直立通酶:织布酚作为潜在的新型除草剂候选者。Appl Environ Microb。2007; 73:1921-7。
- 41。
Tronconi MA, Andreo CS, Drincovich MF。植物苹果酶家族的嵌合结构:nad和nadp依赖亚型的不同进化场景。植物学报2018;9:565。
- 42。
Lawrie DS,Messer PW,Hershberg R,Petrov Da。在D Melanogaster的同义网站上强烈净化选择。Plos Genet。2013; 9:E1003527。
- 43。
史密斯MD,Wertheim jo,Weaver S,Murrell B,Scheffler K,Kosakovsky Pond SL。较少的是:一种自适应分支网站随机效应模型,用于高效检测情节多样化选择。mol Biol Evol。2015; 32:1342-53。
- 44。
Gharib WH, Robinson-Rechavi M.阳性选择的分支位点测试惊人地健壮,但在GC中同义替代饱和和变异下缺乏力量。生物学杂志。2013;30:1675-86。
- 45。
钱J,Liu Y,Chao N,Ma C,Chen Q,Sun J等。植物中法呢基焦磷酸盐合成酶基因的正选择与功能分歧。BMC MOL BIOL。2017年; 18:3。
- 46。
Williamson RJ, joseph EB, Platts AE, Hazzouri KM, Haudry A, Blanchette M, Wright SI。广泛的阳性和阴性选择的证据编码和保守的非编码区域Capsella Grandiflora.Plos Genet。2014; 10:E1004622。
- 47。
魏M,李Z,叶D,Herzberg O,Dunaway-Mariano D.识别域名域对接网站Clostridium symbosum.丙酮酸磷酸二激酶由氨基酸取代。生物化学学报。2000;275(52):41156-65。
- 48.
Wheeler MC,Arias Cl,Tronconi MA,Maurino VG,Andreo CS,Drincovitch MF。拟南芥蒂利亚纳NADP-苹果酶同种型:高度的身份,但明显不同。植物mol biol。2008; 67(3):231-42。
- 49.
李成,黄义,黄岚,吴y,王W.玉米核淀粉生物合成的遗传建筑。植物Biotechnol J. 2018; 16(2):688-95。
- 50。
döringf,streubel m,bräutigama,gowik u.大多数光呼吸基因优先在c的束鞘细胞中表达4.草高粱双色.J Exp Bot。2016; 67(10):3053-64。
- 51。
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32:1792-7。
- 52。
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870-4。
- 53。
nguyen lt,schmidt ha,von haeseler a,minh bq。IQ树:一种快速有效的随机算法,用于估算最大似然性文学。mol Biol Evol。2015; 32:268-74。
- 54。
Bailey TL,Boden M,Buske Fa,Frith M,Grant Ce,Clementi L等。MEME SUITE:主题发现和搜索的工具。核酸RES。2009; 37:W202-8。
- 55。
杨志。PAML 4:最大似然法的系统发育分析。生物化学学报2007;24:1586-91。
确认
我们非常感谢来自Robert A. Mcintosh(悉尼大学)的帮助,具有英语编辑。
资金
山西省国家重点研发计划的补助金提供了这项工作(20183D221019-5,2015-TN-09)和山西农业大学科技创新基金会(2016年)。资金机构在研究和收集,分析和解释或写作稿件中没有作用。
作者信息
隶属关系
贡献
JG和PYG促成了实验的整体设计,为数据分析提供了建议,并协助撰写稿件。WPS分析了数据,进行了实验并写了稿件。LQY和JHG有助于进行系统发育,选择压力和蛋白质建模分析。JMW,XYY和SQD参加了实验的设计。所有作者都读过并批准了稿件。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有利益冲突。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Shi,W.,Yue,L.,Guo,J.et al。C4.植物中的光合途径基因。BMC植物BIOL.20,132(2020)。https://doi.org/10.1186/s12870-020-02339-020-02339-020-02339-020-02339-020-02339-0
关键字
- C4.
- 进化
- nadp-me
- PPDK
- 选择积极的网站