抽象的
背景
Pumilio RNA结合蛋白在整个真核生物中进化保守,并参与细胞质中的RNA衰变、运输和翻译抑制。虽然大多数Pumilio蛋白在细胞质中起作用,但已有报道称两种核仁形式在拟南芥的rRNA加工中起作用。属的种轮藻被认为与陆生植物关系最密切,因为它们与现代胚胎植物有几个共同特征。
结果
在本研究中,我们鉴定了两个推定的核仁Pumilio蛋白基因,即,ChPUM2和ChPUM3的转录组轮藻最纯粹.ChPUM2与拟南芥APUM23的氨基酸序列最相似(同源性为27%,同源性为45%),预测的蛋白质结构与拟南芥APUM23最相似;ChPUM3与APUM24相似(同源性为35%,同源性为54%)。的瞬态表达式35 s: ChPUM2-RFP和35 s: ChPUM3-RFP在烟草叶片细胞中显示融合蛋白的核仁定位,表达类似于35 s: APUM23-GFP和35S:APUM24-GFP.而且,35 s: ChPUM2补充了形态上的缺陷apum23表现型,但不是apum24,而35 s: ChPUM3不能补充apum23和apum24突变体。同样,35 s: ChPUM2 / apum23植物救出了前rRNA加工缺陷apum23,但35 s: ChPUM3 / apum24+/-植物也没能挽救生命apum24.与这些互补结果一致的是,APUM23的18S rRNA (5 ' -GGAAUUGACGG)末端有一个已知的靶rna结合序列在拟南芥中被保守c .最纯粹而APUM24的ITS2 pre-rRNA靶区长156 ntc .最纯粹而不是答:芥.此外,预测CHPUM2和APUM23具有几乎相同的结构,但CHPUM3和APUM24在第5个C末端PUF RNA结合结构域中具有不同的结构,其在CHPUM3中具有比APUM3更长的随机线圈。
结论
ChPUM2的c .最纯粹在拟南芥中具有类似APUM23的功能,但ChPUM3在拟南芥中不能替代APUM24。蛋白同源性建模显示APUM23和ChPUM2的覆盖度较高,但APUM24和ChPUM3的结构存在差异。拟南芥中一个短ITS2的pre-rRNA可能与ChPUM3本身的蛋白质结构一起,阻断ChPUM3与3 ' -extended 5.8S pre-rRNA的结合。
背景
Pumilio蛋白是一类rna结合蛋白,在真核生物中进化保守[1].典型的Pumilio蛋白有8个Puf结构域串联重复,识别8个RNA碱基,每个Puf结构域包含35-39个氨基酸,形成3个α-螺旋结构[2,3.].这些蛋白质的RNA识别的基础是新月形结构[4,5].在新月形结构的凹面上保守的芳族和碱性氨基酸与RNA相互作用,而凸侧的氨基酸与伴侣蛋白相互作用。虽然Pumilio蛋白质具有多种生物作用,但它们的主要分子函数是mRNA衰减和定位,平移抑制[6,7, rRNA处理[8].大多数Pumilio蛋白质在细胞质中局部化,并且参与mRNA的后术语调节。然而,这些蛋白质的小子集局部化在核仁中并参与RRNA加工。例如,酵母的核仁NOP9 [9]和锥虫的TbPUF7 [8通过适当的预抗RRNA加工参与18S rRNA生物合成和核糖体成熟。在植物中,两种核碱蛋白质涉及rRNA加工[10,11,12,13,14,15],包括Arabidopsis Apum23,酵母NOP9的同源物,APUM24,人PUF-A和酵母PUF6的同源物。APUM23不仅需要正常生长图案,例如叶片开发和器官极性[10,16]但也参与了ABA信令[17].APUM24对于植物开发至关重要,因为其纯合突变体显示胚胎杀伤性[15].APUM24参与5.8S和25S rnas的成熟,APUM23参与18S和5.8S rnas的加工。
Pre-rRNA是一种长单链RNA,由核仁中的rDNA重复序列转录而成。该转录本随后通过核糖核酸内溶解活性裂解为3种成熟的rrna (5.8S、18S和25S) [18,19].在rRNA处理期间产生的错误加工RRNA副产物在5'-to-3'和3'-to-5'的外核酸核酸活性降解。这两个预rRNA加工活动需要额外的辅助蛋白,例如RNA外出组分,Pumilio蛋白和许多RNA结合蛋白。据报道,拟南芥和水稻显示出类似的预rRNA加工途径,可能是由于ITS1中A2和A3的endocleavage位点周围的类似侧翼序列20.,提示RNA结合特异性对切割位点的选择至关重要。最近,APUM23被发现在18S rRNA的1141-1151位结合11 nt [12] AND和APUM24与包含5.8s和ITS2区域的RRNA段相互作用[15].因此,APUM23和APUM24可能在拟南芥18S和5.8S rnas成熟过程中靶RNA序列的募集和相互作用蛋白中发挥了关键作用。
大约4.5亿年前,陆地植物从淡水中生活在淡水中,适应陆地环境[21,22,23,24].轮藻门与有生命的陆地植物有许多相同的分子和生理特征,而这些特征在绿藻门中是找不到的[25,26].轮藻在质体上与陆生植物具有高度的序列相似性ATPB.和:,线粒体NAD5.,核编码的小亚基rRNA基因[27].
在本研究中,我们发现所有的绿色植物(Viridiplantae)都有两个假定的核仁Pumilio蛋白与拟南芥同源APUM23和APUM24。与此相一致的是,两个核仁的Pumilio基因,ChPUM2和ChPUM3,在轮藻最纯粹通过转录组分析。我们推测,这些基因编码的两个Pumilio蛋白可能在进化和功能上是保守的,因为它们的拟南芥同源物在蛋白质合成所需的rrna前处理中发挥了关键作用。瞬时表达的ChPUM2-RFP和ChPUM3-RFP定位于核仁尼古利亚娜·宾夕法尼亚州叶细胞,表明CHPUM2和CHPUM3的核仁功能。这apum23突变改变了与35 s: ChPUM2将其缺陷的rRNA加工和形态表型恢复到正常水平。然而,rRNA的加工缺陷和胚胎的致死率apum24突变体没有被解救35 s: ChPUM3或ChPUM2.与互补的失败相一致apum24和35 s: ChPUM3,APUM24在C-Terminus的不同域结构来自CHPUM3。此外,Arabidopsis前RRNA的靶标占Arabidopsis的靶标是156ntc .最纯粹并且可能不足以使CHPUM3的结合。
结果
核核苷酸蛋白的系统发育
Pumilio蛋白质在真核生物中普遍存在,尽管不同的数字[1,5].在整个基因组序列可获得的生物体中,高等植物具有比光合单细胞生物和非孕妇的蛋白质蛋白数量较多;例如,发现25种蛋白质拟南芥, 20栽培稻, 14Physcomitrella patens.,5 in.Chlamydomonas Reinhardtii., 11秀丽隐杆线虫7在酿酒酵母,人类2例[10].基于相似性搜索,使用拟南芥核仁Pumilio蛋白(APUM23和APUM24)作为查询,核仁定位信号(NoLS)的存在作为要求[28],其基因组已测序的绿色植物(Phytozome v12.1;https://phytozome.jgi.doe.gov)显示有两个推定的核仁蛋白蛋白。使用PACBIO ISO-SEQ分析,我们还确定了两种推定的核仁蛋白质,其中四种核苷酸中的四种蛋白质最纯粹。与我们的转录组分析一致,预先预测了四种Pumilio蛋白质轮藻基因组数据,包括2个核仁型[26].与拟南芥APUMs相比,由25个Pumilio蛋白组成的ChPUM2和ChPUM3分别与APUM23和APUM24具有较高的同源性,而ChPUM1和ChPUM4属于其他不同的支系(附加文件)1:图S1)。
为了深入了解推定核仁蛋白质蛋白的进化关系,我们构建了一种系统发育树,其具有14种绿色植物的同源蛋白质,其中5种含有NOL(S)[28] (图。1a和b).本研究分析的所有绿色植物都有两个蛋白,属于APUM23和APUM24分支。的ChPUM2c .最纯粹而ChPUM3属于APUM24分支。利用ChPUM2和ChPUM3进行系统发育分析表明c .最纯粹与本研究研究的其他绿藻相比,更接近陆生植物,表明核仁Pumilio蛋白的进化与之前确定的系统发育定位一致[21,22,23,24,25].
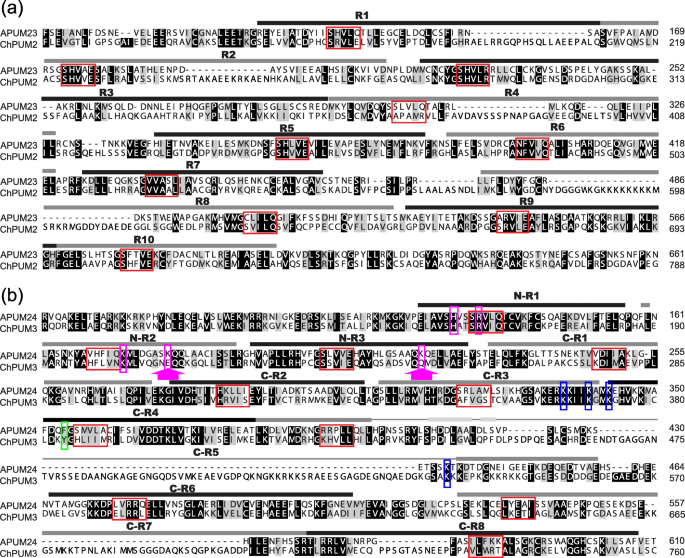
绿色植物代表种两个核仁Pumilio蛋白家族的系统发育树(一个和b)和核仁α和小区的蛋白质结构(c).一个和bAPUM23和ChPUM2家族推定核仁Pumilio蛋白的系统发育关系(一个)及APUM24及ChPUM3系列(b).系统发育树采用MEGA7软件的最大似然LG + G模型构建[29使用1000个引导复制。红藻的两个独立核仁Pumilio蛋白(陨石球粒管和Galdieria sulphuraria),果蝇黑胶基,智人,酿酒酵母作为外群体使用。cAPUM23和APUM24的一级蛋白结构拟南芥ChPUM2和ChPUM3来自轮藻最纯粹.黑色六边形表示Puf rna结合域
然后,我们使用SMART web程序(http://smart.embl-heidelberg.de.) [30.].APUM23和APUM24分别有6个和5个Puf域,ChPUM2和ChPUM3分别有1个和5个Puf域。因为每个Puf结构域都能识别单个RNA碱基[3.[该观察结果提出了CHPUM2可以具有来自APUM23的不同RNA结合性的可能性,并且CHPUM3可以结合类似的,如果不是相同的RNA基序作为APUM24(图。1c)。
ChPUM2和ChPUM3的结构
Pumilio蛋白的经典结构分析显示C末端区域中的8个PUF结构域的串联重复[31,32].然而,最近对人PUF-A和酵母PUF6的分析鉴定了11个PUF结构域,其中包括3个额外的域中的额外域,参与预阵挛加工的蛋白质[14].以前对APUM23执行的类似的分析显示了10个PUF域而不是六个先前已知的域[10,12].与APUM23和ChPUM2之间的系统发育关系一致(图。1a和附加文件1:图S1), ChPUM2还包含10个Puf域,其分布与APUM23相同(图S1)。2a和附加文件2:图S2a)。CHPUM2的每个结构域显示平均26%的同一性和41%的APUM23的域。值得注意的是,在每个PUF结构域的第二α-Helix的第1,第2和第5个残基中发现了高度的同源性(图2中的红色盒子。2a和附加文件2:图S2a)。这三个残基在RNA碱基识别中起着关键作用[14].
使用同样的方法,ChPUM3具有11个Puf结构域(N-R1到N-R3和C-R1到C-R8)。2b和附加文件2:图S2b),在人类Puf-A和APUM24中报道[14].APUM24与ChPUM3的3个n端Puf结构域的氨基酸同源性平均为47%,同源性为67%;8个c端结构域的同源性平均为39%,同源性为55%。在11个Puf结构域中,有两个结构域(C-R5和C-R7)的同一性低于其他结构域(图2)。2b)。
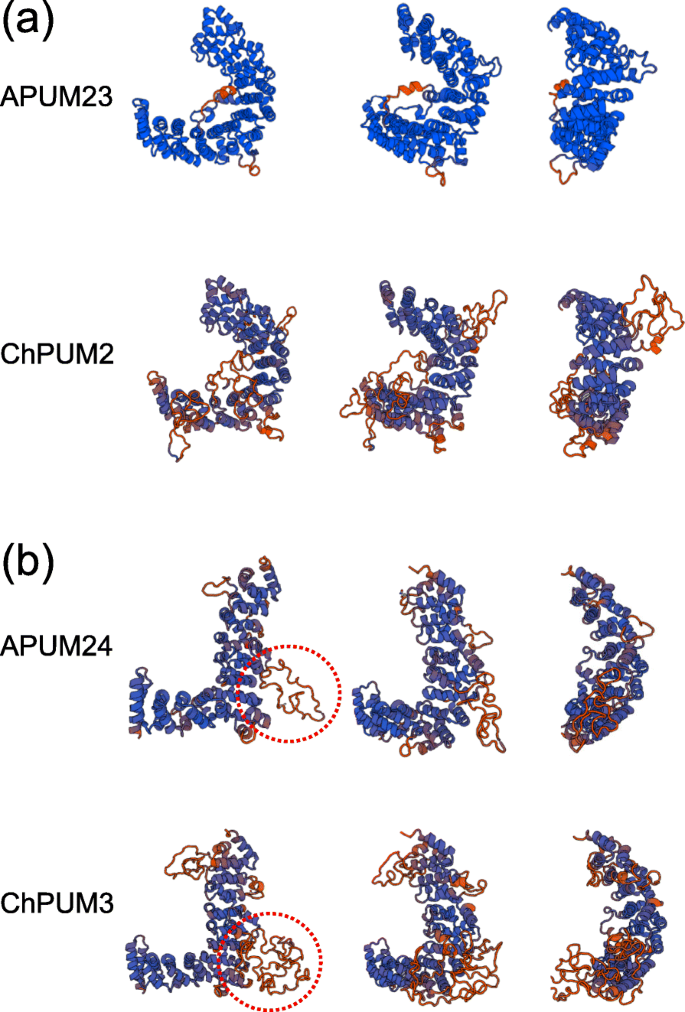
比较Puf结构域之间的氨基酸序列,ChPUM2和ChPUM3可能分别是拟南芥APUM23和APUM24的功能同源物(图2)。2A和b),这与系统发育分析的观察结果一致。但是,由于ChPUM2和APUM23不同于ChPUM3和APUM24,在经典结构域分析中(图2)含有不同数量的Puf结构域。1c),它们可能结合不同的RNA底物。因此,为了确定核仁ChPUM和APUM蛋白之间的结构关系,我们使用SWISS-MODEL web服务器(https://swissmodel.expasy.org/) [33].先前的高分辨率结构研究证明APUM23的C形结构在R3结构域的第2和第3α-Helix之间具有长链,该α-螺旋参与RNA碱基的识别[12].同源造型在三维结构中揭示了CHPUM2和APUM23参考蛋白之间的高相似性,以及类似于APUM23的C形结构(图。3.一种)。与APUM23相比,CHPUM2在R3结构域中具有长随机线圈(图1的底板中的红色彩色线。3),但它在该域中保持不间断的第2和第3α - 螺旋结构,类似于APUM23。因此,共有氨基酸序列和同源性建模的分析表明CHPUM2可以识别类似的,如果不是相同的RNA碱基。
与ChPUM2和APUM23的c型结构相比,ChPUM3和APUM24的结构预测为l型,类似于人类Puf-A参考蛋白[14](图)。3.b). ChPUM3和APUM24在结构上最显著的差异是在C-R5、N-R2和N-R3结构域。ChPUM3在C-R5结构域的随机线圈比APUM24长(图)。2b和图中的虚线圈。3.b).此外,ChPUM3在N-R2和N-R3结构域中含有带负电荷(E210)和不带电荷(Q249)的氨基酸(图2)。2b),而不是已知参与RNA结合并在APUM24和人类Puf-A参考位点发现的基本氨基酸[14].因此,考虑到C-R5的链长和2N末端PUF结构域中的缺乏碱性氨基酸,CHPUM3似乎具有来自APUM24的不同RNA结合特异性。
ChPUM2和ChPUM3的亚细胞定位
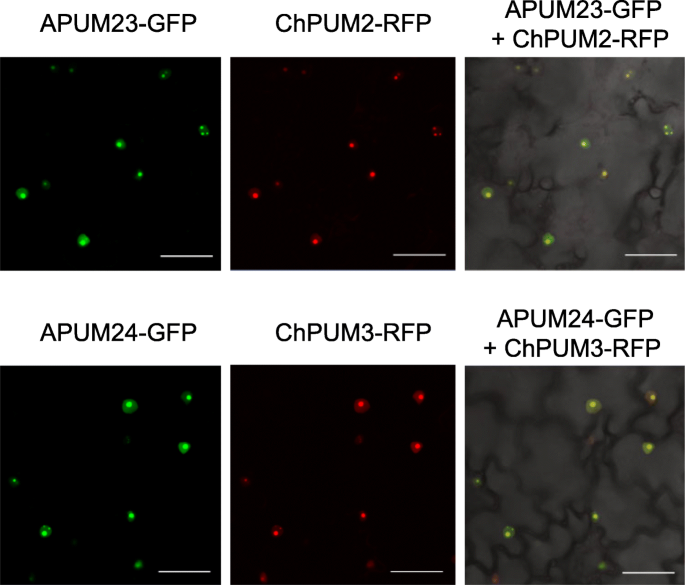
接下来,我们检测了ChPUM2-RFP和ChPUM3-RFP的亚细胞定位。此前,已知APUM23和APUM24的GFP融合优先定位于拟南芥根和烟草叶细胞的核仁[10,15].我们进行了农杆菌介导的共渗n benthamiana叶细胞使用35 s: APUM23-GFP和35 s: ChPUM2-RFP和35S:APUM24-GFP和35 s: ChPUM3-RFP.在核仁中发现所有GFP和RFP标记的Pumilio蛋白质,并且在核桃中弱检测(图。4).分致化结果表明CHPUM2和CHPUM3可以在rRNA识别和处理中发挥类似的作用,以及APUM23和APUM24。
ChPUM2救了apum23突变表型,但ChPUM3没有救援apum24
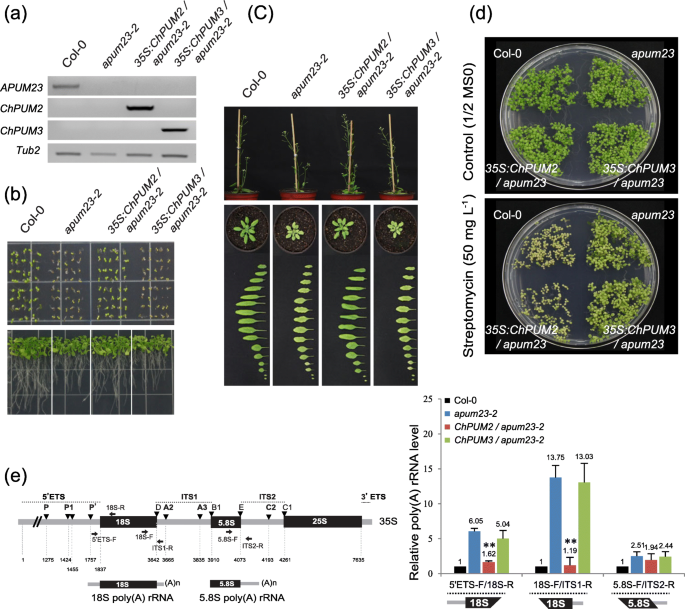
为了评估ChPUM2和ChPUM3是否参与类似APUM23和APUM24的核仁功能,对纯合子进行了互补分析apum23(无花果。5)和杂合的apum24(无花果。6)突变体通过改变35 s: ChPUM2和35 s: ChPUM3构造.虽然这一点apum23-2突变体表现出延迟萌发表型(图。5A和B),短根(图1中的底板)。5b),短花序茎(图1中的上板。5c),浅绿色叶子(图1中的底板。5c)和链霉素耐药性(图。5d),35 s: ChPUM2 / apum23-2−−/植物表现出正常表型(图。5模拟)。值得注意的是,回收的链霉素易感性35 s: ChPUM2 / apum23-2−−/植物表明辅助植物的正常核糖体功能(图。5d).与35 s: ChPUM2 / apum23-2−−/植物,35 s: ChPUM3 / apum23-2−−/植物保持了apum23-2突变表型(无花果。5模拟)。日志含义恢复文件失败apum23-2表型35 s: ChPUM3排除了CHPUM3和APUM23是直向性蛋白质的可能性。
互补的化验35 s: ChPUM2和35 s: ChPUM3转基因植物apum23突变背景。一个确认表达ChPUM2和ChPUM3转基因在apum23-2利用rt - pcr突变。从原始数据处理凝胶图像(附加文件3.:图S3)。b正常发芽和根系生长apum23-2补充与35 s: ChPUM2。c植物高度和玫瑰花属植物在成熟的植物中。从2周龄植物中收集叶子。d链霉素敏感性的恢复apum23-2补充35 s: ChPUM2。eQRT-PCR分析在野生型COL-0中未加工RRNA,apum23-2,35 s: ChPUM2 / apum23-2,35 s: ChPUM3 / apum23-2.2个技术重复和3个生物学重复进行PCR检测。星号表示学生的结果t之间以及apum23-2和转基因植物(**;p < 0.01).值表示平均值±标准差,SDs (n= 3)。左图是poly(a) pre-rRNA的副产物和用于检测pre-rRNA的引物的示意图。未加工的poly(A) 18S (~ 2.6 kt)和5.8S (~ 300 nt) pre-rRNA显示在35S pre-rRNA的下方
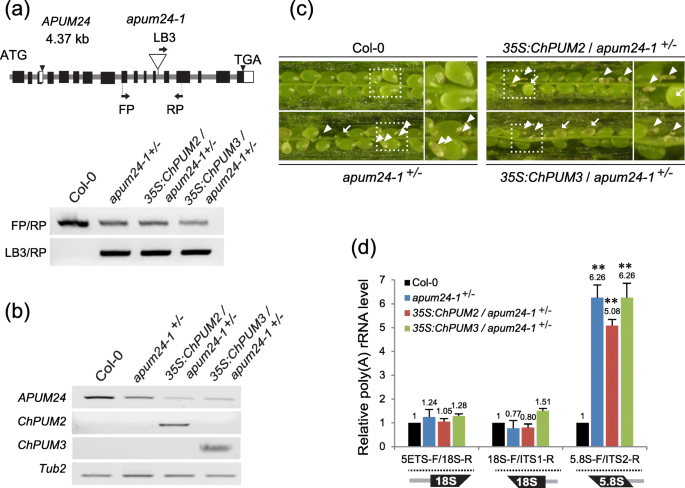
互补的化验35 s: ChPUM2和35 s: ChPUM3转基因植物apum24+/-突变背景。一个T-DNA插入位点APUM24-1突变等位基因(上)和基因分型(下)。用于基因分型的引物用箭头表示。底部面板的原始凝胶图像在附加文件中提供4:图S4a。b确认表达ChPUM2和ChPUM3转基因在APUM24-1+/-利用rt - pcr突变。原始凝胶图像在附加文件中提供4:图S4b。ccol0对照组的sililiques,APUM24-1+/-,转基因APUM24-1+/-表达35 s: ChPUM2或35 s: ChPUM3.每条工厂线的右边面板是方框区域的放大图像。箭头和箭头分别表示未发育的胚珠和败育的种子。值得注意的是,没有一种转基因将异常种子补充到正常水平。d用于分析Col-0对照中相对未加工rRNA水平的qRT-PCR,APUM24-1+/-,35: ChPUMN/APUM24-1+/-使用图1中使用的相同引物。5.2个技术重复和3个生物学重复进行PCR检测。值表示平均值±SDS(n= 3)(**;p < 0.01)
除了恢复形态表型外,35 s: ChPUM2/apum23-2−−/挽救了观察到的缺陷apum23-2−−/突变体累积聚(a)的致氢〜18s-ITS1和5.8S-ITS2预rRNA(图。5e).采用定量逆转录酶- pcr (qRT-PCR)和三种引物组合检测poly (A) pre- rrna。在apum23-2−−/突变体的qRT-PCR产物(5’ets - 18s、18S-ITS1和5.8S-ITS2)均有积累,但在突变体中,qRT-PCR产物的积累量明显增加35 s: ChPUM2/apum23-2−−/与那些相比,植物,18秒-TIS1和5.8S-ITS2预雷纳斯的植物量大大减少了apum23-2−−/,poly(A) 5.8S-ITS2的量略有下降。相比35 s: ChPUM2/apum23-2−−/,35 s: ChPUM3/apum23-2−−/显示了几乎相同的聚(a)预rRNAapum23-2−−/突变体(图。5e,右侧面板)。QRT-PCR结果表明CHPUM2参与了类似的预rRNA处理途径作为APUM23,尽管在去除5.8S前RRNA副产物中没有完全起作用。
与恢复相反apum23由ChPUM2转基因,呢apum24表型没有恢复ChPUM2或ChPUM3转基因产品。纯合子的apum24−−/突变体是致命的[13,15,34),杂合的APUM24-1+/-突变体用于互补分析。这35 s: ChPUM3 / apum24-1+/-植物以类似的速率设定正常和异常的种子apum24+/-突变体(图。6A-C和表格1),显示30.7和33.2%的异常种子35 s: ChPUM3 / apum24-1+/-和APUM24-1+/-植物,分别。正如预期,35 s: ChPUM2 / apum24-1+/-植物产生的异常种子(32.5%)与35 s: ChPUM3 / apum24-1+/-植物。与这种结果一致的形态表型,35 s: ChPUM2/APUM24-1+/-和35 s: ChPUM3/APUM24-1+/-植物累计聚(a) - itailed 18s-ITS1和5.8S-ITS2预rRNA类似于APUM24-1+/-植物(图。6d),表明ChPUM3不是拟南芥APUM24的功能同源物。
ChPUM2恢复了对盐和葡萄糖过敏的表型apum23,但ChPUM3没有恢复apum23和apum24表型
apum23和apum24显示核糖体生物发生相关基因表达水平的变化,进而导致对高浓度盐的过敏反应apum23[17和葡萄糖在一个弱apum24突变体(13].因此我们研究了ChPUM2和ChPUM3可以恢复改变的生理表型apum23−−/和apum24+/-.这35 s: ChPUM2 / apum23-2−−/幼苗表现出与野生型COL-0幼苗相似的抗抗抗性和200mM葡萄糖,而35 s: ChPUM3 / apum23 -2−−/幼苗对NaCl和葡萄糖的敏感性与甘薯相似apum23-2−−/幼苗(图1中的左图)。7然而,a)。35 s: ChPUM2 / apum24-1+/-和35 s: ChPUM3 / apum24-1+/-未能恢复盐敏感性APUM24-1+/-(图1中的右图。7意外)。,35 s: ChPUM2或35 s: ChPUM3是在APUM24-1+/-,他们的转基因幼苗表现出与野生型和APUM24-1+/-幼苗(图1中的右图)。7a).先前有报道说APUM24通过外源供应葡萄糖,野生型植物中基因表达大大增加[13,15].我们的数据显示APUM24COL-0(PB2GW7)控制中的转录物,APUM24-1+/-,35 s: ChPUM2 / apum24-1+/-,35 s: ChPUM3 / apum24-1+/-在200mm葡萄糖的存在下(图。7b)。正常生长APUM24-1+/-提示杂合子表达APUM24对葡萄糖诱导的表型来说可能足够了。类似于APUM24-1+/-,APUM24-1+/-植物转化与35 s: ChPUM2或35 s: ChPUM3在葡萄糖处理下生长正常,可能是由于杂合子的表达APUM24.此外,在200 mM葡萄糖处理下,所有植株未加工的5.8S rrna数量与Col-0 (pB2GW7)对照相似(图2)。7c),未加工的5.8S rrna含量是Col-0 (pB2GW7)对照的5.08 ~ 6.26倍(图2)。6d)。该结果表明葡萄糖补充杂合apum24+/-增加了杂合的水平APUM24直到纯合子APUM24等级。因此,观察到的正常表型和5.8s预rRNA处理35 s: ChPUM2 / apum24-1+/-和35 s: ChPUM3 / apum24-1+/-外源葡萄糖导致拟南芥APUM24水平升高。
盐、糖敏感性恢复分析apum23和apum24表型ChPUM2和ChPUM3.一个apum23-2(左侧面板)和APUM24-1+/-(右图)表达的幼苗35 s: ChPUM2或35 s: ChPUM3在没有或有NaCl和葡萄糖的情况下。Col-0控制(pB2GW7),APUM24-1+/-,35 s: ChPUM2 / apum24-1+/-,35 s: ChPUM3 / apum24-1+/-种子在含10 mg L的1/ 2ms培养基上萌发−1Basta和指示治疗。b表达水平的APUM24Col-0 Control (pB2GW7),APUM24-1+/-,转基因APUM24-1+/-植物表达35 s: ChPUM2和35 s: ChPUM3在没有和存在200mm葡萄糖的情况下。注意类似的表达水平APUM24在200mm葡萄糖的存在下。原始凝胶图像在附加文件中提供5:图S5。cQRT-PCR分析Col-0对照中相对未处理的RRNA水平(PB2GW7),APUM24-1+/-,转基因APUM24-1+/-植物表达35 s: ChPUM2和35 s: ChPUM3在200mm葡萄糖的存在下。使用相同的引物,如图2中所用。5.2个技术重复和3个生物学重复进行PCR检测。值表示平均值±SDS(n= 3)。
讨论
基于我们的转录组数据、公共数据库和以前的结果[10,15,35],我们发现本研究中检测的绿色植物有两个核仁Pumilio蛋白。蛋白质系统发育表现出与多细胞植物的核亮菌蛋白的更密切关系c .最纯粹比单细胞绿藻(绿藻门)多。这一结果与之前的报告一致,表明该属轮藻与土地植物更密切相关,而不是其他绿色植物[27].虽然某些推定的核仁Pumilio蛋白不具有核仁功能的可能性尚未排除,但陆生植物似乎已经进化出两种用于去除异常pre- rrna的Pumilio蛋白。
我们证明了ChPUM2在拟南芥细胞中是APUM23的功能同源体,从缺陷的pre-rRNA处理和形态的恢复中可以明显看出apum23在35 s: ChPUM2 / apum23植物。已知拟南芥核仁蛋白蛋白在识别前RRNA上的靶序列和募集催化蛋白质如外放车酶[10,12,13,15].预rRNA的比较鉴定APUM23的靶序列(5'-GGAAUUGACGG-3')[12的18S rRNAc .最纯粹在职位1148-1158(附加文件6:图S6)。因此,CHPUM2可能会绑定到该序列中c .最纯粹.ChPUM2和APUM23的一级结构支持了这一假设,除了第四个结构域外,ChPUM2和APUM23的Puf结构域在ChPUM2和APUM23之间高度保守(图4)。2a).因此,Puf结构域的共同靶点rRNA序列和相似的氨基酸组成可能使其恢复apum23表型,包括形态和rRNA处理缺陷,以正常35 s: ChPUM2 / apum23.在apum23使用互补分析35 s: ChPUM2,聚(a) - 累积的5.8s预rrnas累积apum23突变体未被完全去除(图。5e).这似乎可能是由于ChPUM2与其他未知蛋白的弱相互作用,这些蛋白属于5.8S rRNA加工所需的内在APUM23伴侣。事实上,ChPUM2的预测结构比APUM23具有更多的展开链(图2)。3.a),可能干扰ChPUM2与其他蛋白质组分的相互作用。为了验证这种可能性在足底在CPUM2中,有必要对与CPUM2相互作用的元件进行识别和比较c .最纯粹和Apum23在拟南芥中。
虽然ChPUM3在结构上与APUM24相似(图。3.b),CHPUM3在拟南芥中没有功能替换APUM24。我们假设该结果是由于CHPUM3和APUM24之间的细结构差异。典型的Pumilio蛋白与Puf结构域的第二α-螺旋结合到特异性RNA碱基,但APUM24及其同源物不能通过该α-Helix结构域的特异性RNA碱进行结合[14,15].ChPUM3不补充apum24突变体可能是由于(1)C-R5结构域中的非常长的随机线圈,(2)两个n末端结构域中的带负电和不带电的氨基酸,和(3)序列c .最纯粹.首先,C-R5域的C-R5结构域的非常长的随机链将中断其他C末端域与RNA碱基的相互作用在拟南芥前rRNA的5.8s-ITS2结中。的确,据报道,人PUF-A及其同源物APUM24在C-R5中具有长随机线圈,可防止C末端PUF结构域与RNA结合[14].ChPUM3有一个比APUM24长80 aa的随机线圈(图。2b);因此,CHPUM3可能无法识别拟南芥前rRNA。其次,在CHPUM3中,贴剂1b的N-R2和N-R3包括带负电(E210)和没有充电的(Q249)氨基酸,与APUM24的两个位置的正氨基酸(k)不同,这可能导致差异结合Apum24对5.8s-its2区域的特征。人PUF-A蛋白的N-R2和N-R3结构域对于RNA结合至关重要[14].第三,ChPUM3可能对长ITS2序列的识别进行了优化。ITS2的c .最纯粹pre-rRNA比拟南芥长156 nt(附加文件7:图S7)。除了ChPUM3中C-R5结构域的长侧链外,拟南芥pre-rRNA相对较短的ITS2序列也可能阻止ChPUM3与底物结合。事实上,ITS2进化迅速,已被用于评估遗传差异[36,37].
结论
在本研究中,我们鉴定了两个核仁Pumilio蛋白,分别是ChPUM2和ChPUM3c .最纯粹在系统发育和结构上分别与拟南芥核仁Pumilio蛋白APUM23和APUM24接近。互补分析使用35 s: ChPUM2和35 s: ChPUM3显示,ChPUM2拯救了有缺陷的表型apum23突变体,但是ChPUM3没有恢复apum24突变体。与这些互补结果一致的是,ChPUM2在一阶氨基酸序列中具有与APUM23相似的Puf结构域,并具有预测的三维蛋白质结构。ChPUM3在C-R5结构域有一个长长的随机线圈,在n端结构域含有与APUM24不同的氨基酸。ChPUM3与APUM24在结构上存在差异,拟南芥pre-rRNA的短ITS2序列可能会阻碍ChPUM3对拟南芥5.8S pre-rRNA的加工。综上所述,ChPUM2在拟南芥中具有功能,与APUM23相似,但ChPUM3不能替代APUM24。进一步研究Charophyta中ChPUM2和ChPUM3的核仁功能将有助于我们了解绿色植物中rRNA加工的进化。
方法
植物材料和生长条件
c .最纯粹在韩国的私人土地(38°33'N'n,128°50'e)收集,具有土地所有者的惯用许可,并在室温下在一个小水族馆的室温下种植。基因组DNA和优惠券标本c .最纯粹国立生物资源研究所植物资源科金敏河博士(https://www.nibr.go.kr/),并以号码NIBRGR0000609814存放。这apum23[10),apum24[15]突变体,从拟南芥生物资源中心获得,之前报道过,用于互补分析。这35 s: ChPUM2和35 s: ChPUM3转基因拟南芥植物apum23和apum24背景是通过使用转换产生的农杆菌肿瘤术GV3101带花浸渍法[38].答:芥野生型Col-0和对照(由pB2GW7转化的Col-0)apum23-2−−/, apum24-1+/-,35 s: ChPUM2,35 s: ChPUM3在16小时光下在22℃下在MS培养基中或在土壤中生长过表达线(120μmol光子m−2 s−2)和8小时黑暗循环。对于抗生素,盐和葡萄糖抗性,种子在补充50mg L的1/2 MS板上发芽−1分别用链霉素、150 mM NaCl和200 mM葡萄糖在生长室内培养10 ~ 12天。播种前将所有种子在4℃下分层3天。
鉴定ChPUM2和ChPUM3成绩单
关键词:Pumilio蛋白c .最纯粹(Chpum.S)通过搜索从全植物产生的PACBIO ISO-SEQ转录组数据来获得,包括Thallus,Rhizoids,小球(Antheridia)和肺炎(Archegonia)。表达Chpum.使用RT-PCR验证S.通过拟南芥核仁蛋白质(APUM23和APUM24)与Pumilio蛋白的比较鉴定了核仁Chpumsc .最纯粹并显示有nol [28].
ChPUM2和ChPUM3同源基因的系统发育分析
为了进行系统发育分析,从Phytozome (v 12.1)的Viridiplantae(绿色植物)的代表物种中获得ChPUM2、ChPUM3、APUM23和APUM24蛋白的氨基酸序列COGs (Orthologous Groups) [35],Klebsormidium flaccidum在klebsormedium基因组数据库(http://www.plantmorphogenesis.bio.titech.ac.jp/~algae_genome_project/klebsormidium/)和Ensembl植物数据库中的两种红藻类(http://plants.ensembl.org).通过使用基于LG + G模型的MEGA7软件使用最大似然方法来构建系统发育树[29,39].使用CLUSTALW进行氨基酸序列比对(http://www.clustal.org/),并使用BioEdit软件编辑(https://bioedit.software.informer.com/).
质粒构建
为了建造35s:chpum.质粒,编码序列(CDSs)ChPUM2和ChPUM3通过RT-PCR扩增使用引物CHPUM2-F和CHPUM2-R1和CHPUM3-F和CHPUM3-R1(参见附加文件8引物序列见表S1)。PCR产物插入到pENTR-D-TOPO载体(Invitrogen),然后通过Gateway™LR Cloase II (Invitrogen)转移到pB2GW7或pK2GW7(根特大学弗拉姆斯研究所voor生物技术)。为了建造35 s: ChPUM-RFP和35S:APUM-GFP质粒,信用违约掉期ChPUM2,ChPUM3,APUM23岁,APUM24通过RT-PCR通过底漆组合CHPUM2-F / CHPUM2-R2,CHPUM3-F / CHPUM3-R2,APUM23-F / APUM23-R和APUM24-F / APUM24-R进行扩增。将PCR产物插入PETR-D-TOPO载体中,然后转移到PB7RWG2或PK7FWG2载体中[40Gateway™LR克隆酶II。
ChPUM和APUM融合蛋白共定位分析
ChPUM2和ChPUM3的c端RFP融合蛋白和APUM23和APUM24的c端GFP融合蛋白瞬时表达n benthamiana利用农业渗透的叶片[41].简单地说,携带融合构建物的农杆菌在固定相收获,并在MMA缓冲液(10 mM MES, 10 mM MgCl)中重悬2和150 μM acetosyringone)的OD值600= 0.8。对于chpumr - rfp和APUM-GFP的共表达,两种农杆菌培养物的体积相同35 s: ChPUM-RFP或35S:APUM-GFP渗透前混合Construct。用无针注射器对烟叶背面进行浸润。将植株置于22℃、高湿度的黑暗环境下保存30-34 h,在荧光显微镜下观察渗入叶片。
定量逆转录酶- pcr (qRT-PCR)用于分析未加工的rRNA
使用RNEasy植物迷你套件(Qiagen,Cat。#74904)分离出总RNA。从100毫克幼苗中,用2个单位的RNase-Fious Turbo™DNase(Ambion,Cat.#AM2238)在37°C下进行处理50分钟。使用寡核苷酸(DT)从5μg的总RNA合成第一链cDNA18引物在20 μL反应,稀释3倍。将1 μL的cDNA与0.6 μL的10 mM引物和10 μL的2 × SYBR®Green Supermix (Bio-Rad, cat。# 172-5261),在20 μL的反应中进行PCR,并按照制造商的说明进行。为检测未加工poly(A) rrna,采用3种不同的引物组合(5’ets /18S、18S/ITS1和5.8S/ITS2)。微管蛋白(Tub4,以At5g44340) cDNA作为内部对照。qPCR检测使用了两个技术重复和三个生物学重复。数据采用2——ΔΔCT方法(42].
可用性数据和材料
的cdna序列数据ChPUM2和ChPUM3在GenBank数据库(https://www.ncbi.nlm.nih.gov/)分别在加入号码MN652915和MN652916下。在识别期间分析的转录组数据集ChPUM2和ChPUM3转录本可在GenBank数据库(NCBI SRA登录号;SRP249259)。
缩写
- C-R:
-
C终端重复
- 它的:
-
内部转录序列
- 女士:
-
Murashige和斯库
- N-R:
-
氨基端重复
- 前rRNA:
-
预核糖体RNA
参考
- 1.
Spassov DS,jurecic R.PUF系列RNA结合蛋白质:是否进化地保守的结构等于保守功能?IUBMB生活。2003; 55:359-66。
- 2.
张斌,张志强,张志强,等。一种保守的rna结合蛋白,调节性命运C. Elegans.雌雄同体生殖细胞系。大自然。1997;390:477 - 84。
- 3.
米勒MT,希金JJ,霍尔TM。通过酵母Puf4p的晶体结构揭示PUF蛋白改变rna结合特异性的基础。Nat Struct Mol Biol. 2008; 15:397-402。
- 4.
Edwards TA, Pyle SE, Wharton RP, Aggarwal AK。Pumilio的结构揭示了RNA和肽结合基序之间的相似性。细胞。2001;105:281-9。
- 5.
Quenault T,Lithgow T,Traven A.Puf蛋白:抑制,激活和mRNA定位。趋势细胞BIOL。2011; 21:104-12。
- 6.
Murata Y,沃顿和母性的结合驼背mRNA在果蝇胚胎的后向模式中是必需的。细胞。1995;80:747-56。
- 7.
Sonoda J,沃顿RP。招募Nanos驼背由Pumilio mRNA。基因Dev。1999;13:2704-12。
- 8.
Droll D,Archer S,Fenn K,Delhi P,Matthews K,Clayton C.锥虫组的Pumilio-Domain蛋白PUF7与核环脂蛋白相关联,并参与核糖体RNA成熟。费用。2010; 584:1156-62。
- 9.
Nop9是一种RNA结合蛋白,存在于40s前的核糖体中,是酵母中18S rRNA合成所必需的。RNA。2007; 13:2165 - 74。
- 10.
Abbasi N, Kim HB, Park NI, Kim HS, Kim YK, Park YI,等。APUM23是一个核仁Puf结构域蛋白,参与拟南芥核糖体前RNA的加工和正常的生长模式。植物j . 2010; 64:960 - 76。
- 11.
Abbasi N, Park YI, Choi SB. .拟南芥中Pumilio Puf结构域rna结合蛋白的研究。植物信号行为。2011;6:364-8。
- 12.
鲍华,王宁,王超,姜勇,刘军,徐磊,等。APUM23特异性识别18S rRNA的结构基础核酸学报2017;45:12005-14。
- 13.
前川,石田,平贺泽APUM24,编码一种新的rRNA加工因子,诱导糖依赖核仁应激和改变糖的反应拟南芥.植物细胞。2018;30:209-27。
- 14.
邱超,McCann KL, Wine RN, Baserga SJ, Hall TM。一个分化的Pumilio重复蛋白家族用于pre-rRNA加工和mRNA定位。中国科学院院刊2014;111:18554-9。
- 15.
Shanmugam T,Abbasi N,Kim HS,Kim Hb,Park Ni,Park Gt等人。拟南芥发散的Pumilio蛋白,APUM24对于胚胎发生至关重要,并且需要忠实的预rRNA加工所必需。植物J. 2017; 92:1092-105。
- 16.
Huang T, Kerstetter RA,爱尔兰VF。APUM23是PUF家族的一种蛋白,在叶片发育和器官极性中起重要作用拟南芥.J Exp Bot. 2014; 65:1181-91。
- 17.
黄KC,林伟伟,程文辉。盐敏感突变体9是核仁APUM23蛋白,与ABA信号通路相关拟南芥.BMC Plant Biol. 2018;18:40。
- 18.
Henras AK, plison - chastang C, O 'Donohue M-F, Chakraborty A, gleises P-E。真核生物核糖体前RNA加工综述。威利跨界Rev RNA。2015; 6:225-42。
- 19.
王志强,王志强,王志强,等。植物核糖体生物学特性的研究进展。植物科学进展。2015;20:729-40。
- 20。
挂r,王z,邓x,刘c,yan b,杨c等。核糖体RNA生物发生及其对寒冷胁迫的反应栽培稻.植物杂志。2018;177:381。
- 21。
黄志强,王志强,王志强,等。绿藻细胞壁的进化与生物多样性研究进展。植物科学学报2012;3:82。
- 22。
Sørensen I, Domozych D, Willats WGT。植物细胞壁是如何进化的?植物杂志。2010;153:366。
- 23。
张志强,张晓东,张志强,等。轮藻类绿藻为植物细胞壁的早期起源提供了深刻的见解。植物j . 2011; 68:201-11。
- 24。
Domozych D, Sørensen I, Popper ZA。编者按:轮藻:植物的进化祖先和植物研究的新兴模式。植物科学学报2017;
- 25。
Lewis LA, McCourt RM。绿藻和陆生植物的起源。[中国科学(d辑:地球科学)]。
- 26。
Nishiyama T,Sakayama H,De Vries J,Buschmann H,Saint-Marcoux D,Ullrich Kk等。这轮藻基因组:植物陆地化的二次复杂性和影响。细胞。2018; 174:448-64 E24。
- 27。
carol KG, McCourt RM, Cimino MT, Delwiche CF.陆地植物的现存近亲。科学。2001;294:2351-3。
- 28.
Scott MS, Troshin PV, Barton GJ。NoD:真核和病毒蛋白的核仁定位序列检测器。BMC生物信息学。2011;12:317。
- 29.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。Mol Biol Evol. 2016; 33:1870-4。
- 30.
Schultz J,Copley RR,Deerks T,Ponting CP,Bork P. Smart:一种基于网络的基因移动域的工具。核酸RES。2000; 28:231-4。
- 31.
Miller Ma,Olivas WM。PUF蛋白在mRNA降解和翻译中的作用。威利跨界Rev RNA。2011; 2:471-92。
- 32.
王X,McLachlan J,Zamore PD,Hall TM。用人的轻型同源结构域的RNA的模块化识别。细胞。2002; 110:501-12。
- 33.
Waterhouse A,Bertoni M,Bienert S,Studer G,Tauriello G,Gumienny R等人。瑞士型号:蛋白质结构与复合物的同源性建模。核酸RES。2018; 46:W296-303。
- 34.
植物核仁胁迫与糖胁迫的关系。植物信号学报。2018;13:e1442975。
- 35.
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40:D1178-86。
- 36.
陈志强,陈志强,陈志强,等。绿藻门(绿藻门、绿藻门)植物ITS2进化与物种形成的研究进展。BMC Evol Biol. 2011;11:262。
- 37.
秦勇,李敏,曹颖,高艳,张伟。ITS2的分子阈值及其在种子植物分子进化和物种鉴定中的意义。Sci众议员2017;7:17316。
- 38.
花浸:农杆菌介导的植物转化的一种简化方法拟南芥.植物j . 1998; 16:735-43。
- 39.
一种改进的一般氨基酸置换矩阵。生物学报2008;25:1307-20。
- 40。
Karimi M,Inzéd,Depicker A. Gateway™矢量农杆菌属介导的植物转化。趋势植物SCI。2002; 7:193-5。
- 41。
古老mm,dietzgen rg,schichnes d,ruzin s,杰克逊alo。PGD载体:用于在农药叶片中的绿色和红荧光蛋白融合的多功能工具。工厂J. 2002; 31:375-83。
- 42。
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据−ΔΔCT方法。方法。2001; 25:402-8。
致谢
我们感谢Jungho Lee博士,了解文化条件的建议。
资金
本研究由下一代BioGreen21计划,农村发展管理局(NO.PJ013663),韩国,向SBC提供资金。资金代理商支持这项研究项目,但在稿件的研究,数据分析,解释和写作的设计中没有发挥作用。这些是作者的唯一责任。
作者信息
隶属关系
贡献
SHP和SBC设计了实验并撰写了文章。SHP生成构建物并进行互补分析。HSK协助亚细胞定位研究。PJK有助于杂合突变体的互补。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
用25个APUMs系统分析了Pumilio蛋白的系统进化树答:芥apums和4个小区c .最纯粹.使用具有1000个引导复制的JTT + F + G模型生成最大似然树。
附加文件2:图S2。
每个Pumilio中的五个RNA识别残留物的对准从核磁甲壳和CHPUM重复。在APUM23和APUM24中的每个Pumilio重复的五个残基分别与RNA识别的典型重要性,分别与其同源CHPP,即CHPUM2和CHPUM3的那些对准。
附加文件3:图S3。
原始琼脂糖凝胶图像的RT-PCR产物为Fig。5.
附加文件4:图S4。
原始琼脂糖凝胶图像的RT-PCR产物为Fig。6a和b。
附加文件5:图S5。
原始琼脂糖凝胶图像的RT-PCR产物为Fig。7b。
附加文件6:图S6。
18S rRNA序列比对答:芥和c .最纯粹.红框为APUM23与之结合的拟南芥rRNA在nt 1141-1151位置的序列,蓝框为18S rRNA在nt 1148-1158位置的相同序列c .最纯粹.
附加文件7:图S7。
5.8S rRNA与ITS2序列比对答:芥和c .最纯粹.(a)对准5.8s rRNA序列。(b)其2序列的对齐。
附加文件8:表S1。
本研究使用的引物。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Park, s.h., Kim, HS。、Kalita P.J.et al。拟南芥和角藻核仁中pilio rna结合蛋白结构和功能的异同轮藻最纯粹.BMC植物杂志20,230(2020)。https://doi.org/10.1186/s12870-020-02444-x
收到了:
接受:
发表:
关键字
- 拟南芥
- 轮藻最纯粹
- 轮藻门
- ITS2
- Puf
- rna结合蛋白
- rRNA