抽象的
背景
植物的固定性意味着它们在生命周期中经常面临各种生物和非生物的胁迫。在各种非生物胁迫下,水分应激,升温,盐度和重金属毒性是挑战整体植物生长的主要非生物胁迫。植物已经进化了复杂的分子机制,以适应给定的非生物胁迫。长期非编码RNA(LNCRNA)-A含有> 200个核苷酸(NT)的不同类别的RNA--在植物适应各种非生物应激中的基本作用。
结果
LncRNAs play a significant role as ‘biological regulators’ for various developmental processes and biotic and abiotic stress responses in animals and plants at the transcription, post-transcription, and epigenetic level, targeting various stress-responsive mRNAs, regulatory gene(s) encoding transcription factors, and numerous microRNAs (miRNAs) that regulate the expression of different genes. However, the mechanistic role of lncRNAs at the molecular level, and possible target gene(s) contributing to plant abiotic stress response and adaptation, remain largely unknown. Here, we review various types of lncRNAs found in different plant species, with a focus on understanding the complex molecular mechanisms that contribute to abiotic stress tolerance in plants. We start by discussing the biogenesis, type and function, phylogenetic relationships, and sequence conservation of lncRNAs. Next, we review the role of lncRNAs controlling various abiotic stresses, including drought, heat, cold, heavy metal toxicity, and nutrient deficiency, with relevant examples from various plant species. Lastly, we briefly discuss the various lncRNA databases and the role of bioinformatics for predicting the structural and functional annotation of novel lncRNAs.
结论
了解应激响应性LNCRNA的复杂分子机制在其婴儿期。在作物植物中的全基因组的全面地图集的可用性,并综合了解调节各种非生物应激反应的复杂分子机制,使我们能够使用LNCRNA作为潜在的生物标志物,用于裁缝中非生物胁迫耐受性植物未来。
背景
植物的固定性意味着它们在生命周期中经常面临各种生物和非生物的胁迫。植物已经进化出多种复杂的机制来识别各种应激因素,产生适当的信号通路,并在转录、转录后和表观基因组水平上通过重新编程多个基因的表达来做出相应的反应,以适应恶劣的环境条件[1那2].研究团队已经成功地确定了一些复杂的机制,植物利用这些机制在遗传、生理、生化和分子水平上在不利的环境下维持“细胞内稳态”[2那3.].MiRNA(21-24nt)-A新类非编码RNA(NCRNA)的发现 - 以及控制涉及各种发育,生物学和应力反应的基因的调节机制,我们对植物基因调控的理解提出了我们的理解[4.那5.].基因组测序的技术创新,特别是下一代测序,RNA测序(RNA-SEQ)和先进的生物信息学工具,改善了转录,转录后,翻译和表观遗传水平的各种基因的功能阐明[6.].这些创新使新的NCRNAS发现,包括LNCRNA,以及它们在调节哺乳动物和植物中的各种生物过程,开发和应力反应方面的作用(有关详细信息,请参阅[7.那8.])。LNCRNA是一个不同类别的RNA,最大的阶级是“生物调节因素”,控制转录调节和基因组印记[9.那10].众多值得注意的LNCRNA调节植物发育,抗病性,营养率采集等通过染色质重塑,组蛋白修饰,PRI-mRNA替代剪接或作为“目标模仿”的抗药性的生物学过程进行了记录[11那12那13那14那15].然而,很少有研究已经进行了对LNCRNA的基因组探索,其复杂的调节分子机制或功能注释[16].在这里,我们解释了LNCRNA的类型和功能,并更新各种LNCRNA,其目标基因的作用以及参与适应植物的复杂操作分子机制,以适应各种非生物应激的挑战性环境。
lncrna的生物起源、类型和功能
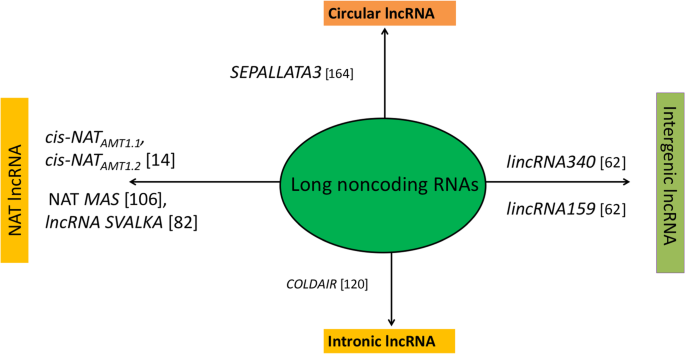
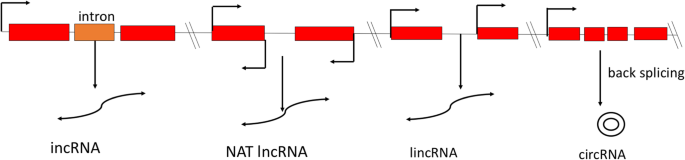
在各类ncrna中,lncrna是一种异构类的RNA转录本> 200 nt,不能编码蛋白质,作为“核糖体调节因子”,位于细胞核或细胞质中,由RNA聚合酶II或III和聚合酶IV/V转录[17那18那19].Pol IV LNCRNA用作小干扰RNA的前体(SIRNA)[19].POL V依赖性LNCRNA有助于调节局部染色质环[20.],从蛋白质编码位点的任何一条链转录,可能有也可能没有5 '帽和3 '尾的聚腺苷酸化,并以“组织特异性”的方式表达[21那22那23].LNCRNA可以广泛地分类为(i)长期性NCRNA(LincrNA),(II)内肾NCRNA(Incrnas),(III)天然反义转录物(NATS),(IV)基于的圆形长度非编码RNA(Circrnas)他们的位置和邻近的蛋白质编码基因[22那24那25那26].LincRNAs起源于基因间区域,具有弱剪接、聚腺苷化的组织特异性表达和执行反式(远端基因)调节功能[27那28那29],而Incrnas从内文区域转录。NAT源自感测编码区域的互补DNA股[26)和特性顺式和反式监管行动30.].然而,Circrnas处于低丰度,源自初MRNA中内外的“背部剪接反应”[29那31],具有共价闭合结构,比线性lncrna具有更高的序列保守性[29那32].各种类型的LNCRNA及其可能的生物发生在图1和2中示出。1和2.它们的功能是“诱饵”或“海绵分子”、“信号分子”、“主干分子”和“引导分子”[25那34那35].此外,lncRNAs可以作为miRNAs和siRNAs的前体,调节pre- mrna的选择性剪接,并作为内源性靶模仿物(eTM)竞争各种miRNAs [20.那36那37].
各种类型的LNCRNA,各种植物中具有合适的实例[164.]
植物lncrna的序列保守、多样性和系统发育特征
lncrna高度进化的特性导致了植物和动物物种之间较低的序列保守性,从而导致较少的系统发育关系[38那39].马奎斯和庞廷[40]的研究结果显示,lncrna的含量低于2%拟南芥蒂利亚纳在整个植物界都是进化保守的,这解释了lncRNA序列的快速进化。来自5个单子叶和5个双子叶物种的lncrna的保护分析表明,在种内和亚种水平上具有高的序列保护[41].在种间水平上,lncrna在核苷酸水平上仍然高度分化,并表现出了不同的调控作用[41那42].穆罕默德汀等人。[43还支持LNCRNA的位置序列保护Athionema Arabicum.和Tarenaya hassleriana在核苷酸水平上使用系统基因组学方法。同样地,Golicz等人[44]证实了大豆、鹰嘴豆、和4个lncrna的序列同源性Medicago truncatula。尽管序列相似,但在众多植物物种中,LNCRNA在其低表达能力,短长度和少于外显子和剪接变异方面相似,包括拟南芥,黄瓜,玉米,鹰嘴豆,大豆[43那44那45那46那47].同样,已经研究了动物和植物物种中LNCRNA的保守功能[38].不断增长的lncRNA数据库和比较基因组学分析可能为lncRNA基因的功能保护及其在各种植物中的作用和功能模式提供新的动力[38].
LNCRNA控制干旱胁迫耐受性
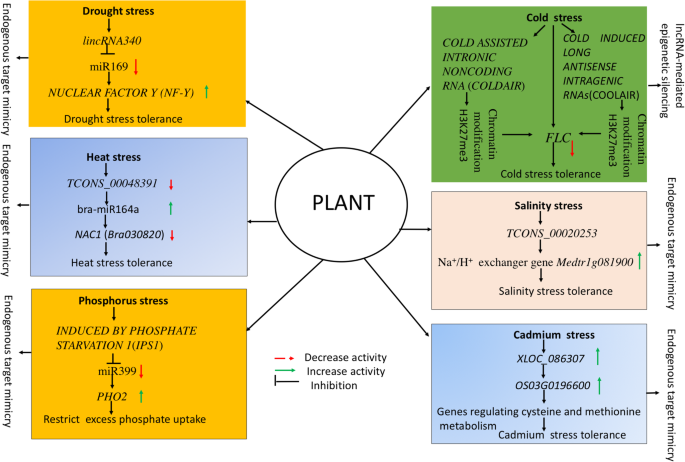
在全球范围内,由于降雨量不稳定的降雨模式,干旱胁迫相关事件的剧集正在增加,这影响了植物增长并对全球粮食安全构成了严峻挑战[48].植物具有多种多样的生理生化和复杂的分子网络,包括各种信号转导通路的级联,以适应干旱胁迫[49].分子生物学的进展揭示了植物干旱胁迫响应的潜在基因/ qtl和各种复杂的调控基因网络和分子信号级联[48那50].随后,在各种植物中发现干旱响应的miRNA及其候选靶基因对干旱胁迫适应所涉及的分子机制(见[51])。同样,新兴的证据表明,LNCRNA在植物中的干旱胁迫下,利用基于LNCRNA,miRNA和蛋白质编码基因的共表达网络以及转录因子的转录因子来揭示了LNCRNA的参与性作用[52那53那54].在各种植物物种-6中据报道了干旱响应LNCRNA的显着实例拟南芥[55],504 in杨树spp。56],98米[57],664在玉米[58],19块狐尾米勒[59],185 in Cassava [60.]和1597在switchgrass中[52].lncrna可以通过募集基于eTM的复杂机制、反义转录介导的调节、染色质调节或直接调控各种干旱应答基因的转录来影响干旱胁迫响应[60.那61.那62.那63.].Doxtail Millet的深度测序提供了探索584 LNCRNA的机会[59[其中17 Lincrnas和两个NAT LNCRNA在干旱胁迫下表现出差异表达。同时,作者发现了20个类似的Lincrnas和一个NAT LNCRNA对高粱的干旱胁迫作出反应[59].谷子中只有一个干旱响应lncRNA与高粱中的干旱响应lncRNA存在序列共线性,说明了lncRNA的低保守性[59].在杨树trichocarpa,系统的RNA-seq分析探索了> 2500 lncrna的综合景观[56[其中504次响应504。使用RT-QPCR 504干旱响应LNCRNA的八种干旱响应LNCRNA的功能验证揭示了六种Lincrnas的上调和下调水分胁迫下的两个Lincrnas。为了在木薯基因组中调查干旱响应性LNCRNA,STRAND特异性RNA-SEQ数据用于鉴定一组318 LNCRNA和153 NAT LNCRNA响应冷和干旱胁迫[60.].在51个干旱特异性的差异表达的LNCRNA(Delncrnas)中,40在干旱胁迫下显示出上调节作用。使用QRT-PCR所选LincrNA的功能验证显示了上调的Lincrna101,Lincrna391,和lincRNA356和下调lincRNA64、lincRNA350 lincRNA182,和lincRNA392在干旱压力下。此外,依靠目标模拟机制增加了表达lincRNA340在干旱条件下,目标miR169的活性降低,最终提高核因子y (nf-y)基因表达(60.)见图。3.].丁等人。[53]在木薯中恢复124次Delncrnas,其中11个,其中11作为MiR156,MiR164,MiR169和MiR172的目标模拟。功能验证显示TCONS_00068353lncRNA作为miR156k和miR172c的靶模拟物,miR156k和miR172c控制着各种非生物胁迫应答基因Tcons_00060863.和TCONS_00097416在干旱胁迫下,LNCRNA分别参与ABA和乙烯信号传导途径[53].
LncRNA模块控制植物的环境非生物胁迫响应。依赖靶模拟机制增加了表达lincRNA340在干旱条件下,目标miR169的活性降低,最终提高核因子y (nf-y)基因表达(60.].lncrnas.Tcons_00048391.和TCONS_00010856充当BRA-MIR164A的内源性目标模拟,其调节热应激反应[64.].镇压镇压FLC.冷应激早期发生的轨迹是由冷诱导的长度反义腺瘤RNA的反义取向转录的另一种剪接的NAT lncRNAFLC.基因通过染色质修饰(降低活性组蛋白标记H3K36me3和增强抑制组蛋白标记H3K27me3)FLC.vernalization期间的轨迹[65.].在盐度压力下tcons_00020253上调的表达Na+/小时+交换者基因Medtr1g081900在根68.].lncRNA的诱导机制磷酸盐饥饿诱导1(IPS1)作为MIR399的ETM或诱饵工作,并调节目标Pho2.基因表达与磷稳态拟南芥(Franco-Zorrilla等。[11和Bari等人[69.])。lncXLOC_086307的显著上调活性表明,XLOC_086307可能通过控制富含半胱氨酸的多肽代谢相关基因参与水稻的Cd响应过程OS03G0196600[88.]
考虑到NAT LNCRNA的调节机制,使用RNA-SEQ分析在水稻中回收98个干旱响应LNCRNA,以及两个重要的干旱响应LNCRNANAT OS02G0250700-01(瞄准后期胚胎发生丰富的蛋白质基因)和NAT Os02g0180800-01(瞄准cinnamoyl-CoA还原酶基因)[57].这两种lncrna的表达与它们对应的靶基因保持负相关。玉米全基因组干旱响应lncrna的研究鉴定出1535个不同发育阶段的lncrna [54].R1阶段(吐丝阶段)捕获的lncrna在干旱胁迫耐受性中起着关键作用。此外,V-ATPase编码基因(vpp4)可能是lncRNA的靶基因MSTRG.6838.1;vpp4并且鉴定的LNCRNA可以作为顺式作用对工作。
除了作为ETM还是NAT,LNCRNA可以调节各种干旱胁迫响应基因的转录[52那62.].lncrna可能在调控干旱胁迫耐受中发挥作用拟南芥,鉴定出一种新的lncRNA干旱诱导lncrna.(DRIR),包含一个755 nt长的lincRNA,它控制多个干旱胁迫应答基因,包括aba信号基因(ABI5,P5CS1,RD29A, 和RD29B)、水通道蛋白基因(NIP1那提示),annexin基因(annat7)那FUCOSYL TRANSFERASE4(FUT4.)基因和转录因子基因(NAC3, WARKY8)在转录水平[37].的drirD.(T-DNA插入突变体)DRIR-overexpressing拟南芥这些基因的高倍数表达表明,这些株系比野生型幼苗具有更高的耐旱性。因此,lncRNADRIR通过用作正调节器赋予水缺陷应力耐受性。
同样地,在水稻中报道,调节参与参与乙烯和信号,钙信号,淀粉和蔗糖合成的各种干旱响应性调节基因以及各种代谢过程[63.),柳枝稷(黍virgatuml .) [52],P..betulifolia[70],木薯[53那71.),而Songorica[62.)(见表1).在柳枝稷不同生长阶段干旱胁迫下鉴定的441个DElncRNAs中,lncRNAsXLOC_053020那XLOC_014465, 和XLOC_033252控制ABA合成,XLOC_074836促进乙烯信号转导XLOC_005809参与海藻糖磷酸合酶代谢的蛋白表达上调,表明其在干旱胁迫耐受中发挥了重要作用[52].已经鉴定了各种LNCRNA及其可能的靶基因和有助于各种作物中的干旱胁迫和其他非生物应激反应的工作机制(见表2).综上所述,各种lncRNAs通过作为各种miRNAs的靶模仿物,通过上调或下调调控各种干旱应答靶基因或转录因子的表达,从而在调控干旱胁迫中发挥作用。这些新出现的lncrna可以在转录、转录后和表观基因组水平上作为调控枢纽,控制各种干旱响应激素信号通路。
lncrna控制热应激耐受性
热胁迫是一种影响植物生长、生理、代谢活性、发育和产量性能的主要非生物胁迫[2].随着全球温度的目前,植物候选和适应过程的变化对作物产量产生负面影响,这是挑战全球粮食安全[107].植物招募各种机制,包括适应性,生化和分子,以应对热应激[2那108].植物产生不同的植物激素、热休克蛋白(HSPs)/伴侣蛋白、抗氧化酶和代谢物,这些在适应热应激中起着关键作用[108那109].在分子水平上,调节通路的激活在植物对热胁迫的适应中起作用[2].有证据表明miRNAs调节各种植物的热应激反应[110].lncRNAs作为一种重要的分子调控层的证据越来越多,这为深入了解作物热应激响应的调控机制提供了依据。为了探索lncrna在热应激反应中的作用,我们使用小麦Affymetrix基因芯片芯片和Solexa测序在小麦中鉴定了54个假定的热应激反应lncrna [75.].在已鉴定的lncRNA转录本中,分别有4个和26个是miRNAs(即miR2004、Ta-miR2010、miR2066)和siRNAs的前体。老年病的lncRNAsTahlnRNA27和talnrna5.并通过QRT-PCR分析证实了它们对应的miR2010和miR2004,表明它们在小麦中的热应激反应中的显着作用。但是,热应激响应仍然保持特定于组织/依赖性talnrna5.在种子组织中表达量高于其他组织[75.].大多数lncrna的序列保守性较弱;它们的表达因组织、发育阶段甚至物种而异[65.].lncrna在玉米中的组织/发育特异性表达已被报道[54那87.]和木薯[53],且物种特异性表达显著胡杨和黄杨变种锥体在盐度压力下。
在各种作物中已经发现了大量差异表达的lncrna及其相应的蛋白质编码热应激应答靶基因和mirna [64.那77.].Wang等人[64.]利用链特异性RNA-seq in,研究了参与油菜素内酯、ABA、生长素、jasmonic acid、水杨酸、乙烯激素信号通路的lncRNAs和差异表达基因(DEGs)的上调和下调,以及编码全基因组各种热休克蛋白的差异表达基因(DEGs)Brassica Rapa.在热应力.三种鉴定的热响应Delncrnas,LNCRNA的差异表达Tcons_00004594在蛋白质编码基因的下游Bra021232通过qRT-PCR提示其独联体- 提升表达[64.].此外,LNCRNA.Tcons_00048391.和TCONS_00010856充当BRA-MIR164A的内源性目标模拟,其调节热应激反应。因此,在热应激下,胸罩的上调和LNCRNA的下调Tcons_00048391.和目标BRA030820(母)“XK”品种耐热性基因([64.),见图。3.).同样的,约束lincRNA159与保守的miR164降低了三个miR164靶向的表达n基因(南、ATAF1/2 CUC2)在冷凝压下的木薯[60.].同样,drought-responsive lncRNAmstrg.42613.1.被确定为保守的miRNA164调节干旱胁迫的目标模拟C. Songorica[62.].将来,操纵靶向基因的过表达或敲除控制热应激反应的基因可以帮助我们为工程耐热作物植物。
控制冷胁迫耐受性的lncRNAs
低温胁迫是一种重要的非生物应力,挑战植物生长和产量[2那111].植物协调若干复杂的c -重复序列结合因子调控基因网络(CBFS.)和冷调基因(cor) [112]而无数的新型监管mirnas [110使它们能够适应冷胁迫。遗传和基因组学方法的进展已经阐明了几个与植物耐寒相关的qtl和可能的候选基因[111].同样,有新发现的LNCRNA证据证据了植物中的冷应激反应[65.那80那103].lncrna调控冷适应的新作用被记录在拟南芥[65.那82.],木薯[60.],Brassica Rapa.[15那82.,香蕉,葡萄藤[81.]和短柄草[104].
在适应寒冷气候的植物中,春化现象已经形成,它在冬季营养生长阶段阻止开花,而在春季有利条件下的生殖阶段允许开花[113].在拟南芥那开花轨迹C(FLC.)是一个众所周知的表观遗传学调控基因位点[114].FLC.也作为一个抑制开花在寒冷中拟南芥[115].在这种情况下,LNCRNA在抑制表达中的参与性作用FLC.通过多胞膜介导的介导的表观遗传调节,通过vernalization在冷应力下是一种用于控制冷适应的良好的机制拟南芥[65.那67.那116].镇压镇压FLC.冷应激早期发生的轨迹是由冷诱导的长度反义腺瘤RNA的反义取向转录的另一种剪接的NAT lncRNAFLC.基因通过染色质修饰(降低活性组蛋白标记H3K36me3和增强抑制组蛋白标记H3K27me3)FLC.vernalization期间的轨迹[65.那66.那67.].有趣的是,Castaings等人。[117]证明了第一类反义COOLAIR控制的进化保守的作用FLC.在春化过程中的抑制拟南芥,阿拉伯人兰迪拉塔,和而阿尔物种。
同样的,冷辅助内肠道非致rna(COLDAIR) [116],从Intron1转录FLC.该基因招募了Polycomb抑制性复合物2 (PRC2),该复合物有助于染色体的修饰(增加H3K27me3)FLC.基因座,从而抑制表达FLC.基因座(见图。3.).随后,Kim等人。建议“polycomb结合lncrna,COLDWRAP“可以进一步合作在稳定的镇压中FLC.在春化期间的轨迹拟南芥.
最近,Kindergren等人。[80]提高了我们对冷驯化机制的认识拟南芥通过说明新的角色SVALKA和神秘的反义CBF1(asCBF1)冷应激诱导的LNCRNA。这些LNCRNA通过抑制转录来调节冷适应CBF1.基因由RNA聚合酶II (RNAPII)碰撞lncrna衍生SVALKA和asCBF1.同样,探讨LNCRNA控制冷应激反应的作用拟南芥,链特异性rna测序(ssRNA-seq)鉴定出4050个NAT lncrna和2460个lincrna为冷响应lncrna [103].其中,作者证实了马斯(NAT LNCRNA_2962.),在冷应激下诱导的顺式作用NAT LNCRNA,其激活了相应的冷响应性的转录影响开花4的疯子(MAF4.)那一个FLC.家庭成员,通过涉及WDR5A复合物,将H3K4ME3存放在MAF4.基因的激活。因此,被激活的基因最终抑制冷胁迫下的开花。同样,在芸苔,三FLC.据报道,作为花卉阻遏物的副病剂据报道[118那119].NATs的参与FLC2轨迹Brassica Rapa.有报道称[120].RNA-seq驱动转录组分析对照和冷处理叶片Brassica Rapa.已识别2088个lncrna [82.],其中三个BrFLC基因座只参与冷应激调节BrFLC2,窝藏NATBrFLC2as(mstrg.2765.),与COOLAIR的转录本有同源性拟南芥蒂利亚纳并在冷凝压下显示上调[82.].在功能上,COOLAIR充当“独联体-NAT关于AtFLC轨迹”67.];但是,行动BrFLC2as作为顺式或trans作用模式有待进一步研究。同样地,考虑到加基因,这Bra024350轨迹(同源AtMAF1) - 一种被称为natmstrg.14523- 在冷凝压下下调。然而Bra024351轨迹(同源AtMAF4) - 一种被称为natmstrg.14524-在冷胁迫下没有下调Brassica Rapa.,说明上述lncrna的作用机制不同于春化作用的lncrna拟南芥蒂利亚纳[82.].此外,在众多差异表达的lincrna中,NAT lncrna鉴定出了lncrnaMSTRG.4795 MSTRG.18513,和MSTRG21908差异和MSTRG.259 MSTRG.491,和MSTRG.17153在不同阶段施加的冷胁迫下均呈下调趋势Brassica Rapa.[82.].
利用RNA-seq分析,对葡萄中冷响应的lncrna进行全基因组调查,发现284个上调的新lncrna, 182个下调的新lncrna, 242个针对326个蛋白编码基因的delncrna,以及各种应激响应基因,包括CBF4.转录因子基因,晚期胚胎素丰富的蛋白质基因,以及WRKY转录因子基因[81.].通过qRT-PCR对选定的lncrna进行功能验证,证实了lncrna上调VIT_200S0179N00030,VIT_207S0141N00070, 和VIT_207s0005n0048和下调VIT_201S0010N00070那VIT_208S0007N00270,和VIT_209s0002n00020,表明他们在调节葡萄中的冷应激耐受性的重要作用[81.].在木薯中,为了揭示全基因组寒冷和干旱响应的lncRNAs,我们捕获了318个lncRNAs [60.].考虑到他们在冷应力耐受性的贡献作用,功能验证lincRNA419, 207,和234揭示了冷胁迫下它们的活性上调。为了破译miRNAs、lncRNAs和控制耐寒性的应激反应基因的调控网络,lincRNA159作为miR164的靶模拟物降低了miR164的表达n寒冷胁迫下的基因[60.].除了这些机制外,lncRNAs和pri-miRNAs的选择性剪接(AS)可能参与控制冷应激反应拟南芥[79.].在135个冷依赖差异表达和差异选择性剪接的lncrna中,诱导tas1a.中发现AS在冷胁迫下调控lncRNA拟南芥.未剪接内含子转录本at2g274001来自tas1a.包含“mir173结合位点和tasiRNAs而剪接的转录本AT2G27400_ID1仍然没有内含子。由于温度的降低,AT2G27400_ID1转录本在冷处理后的前6 h迅速下降,而未剪接的AT2G27400.1转录本在前3 h上升。随后,它在接下来的12小时内下降[79.].因此,lncrna的AS在调节冷胁迫耐受性中起着重要的作用。lncrna可以通过染色质调制/重构、AS机制和转录调节耐寒基因来调控耐寒性。进一步了解lncrna调控低温胁迫的作用机制,为工程抗寒作物提供机遇。
Lncrnas作为施放压力的植物适应的新球员
过度灌溉农田和地下水的迅速枯竭是世界范围内与盐有关的问题增加的主要因素[121].全球范围内,45 Mha的水浇地和32 Mha的耐旱地受到盐碱胁迫的挑战[122那123].因此,土壤盐渍化仍然是对全球粮食生产的日益限制。在盐胁迫下,植物承受过量的有毒离子负荷,会降低植物的生长发育和粮食产量[124].
植物已经进化出几种细胞和生理机制来适应盐胁迫(见[124])。在分子水平上,由基因/ qtl和其他调控基因编码的大量离子转运蛋白在不同植物的盐胁迫控制中起着关键作用(见[121那124])。同样地,lncrna调控植物耐受盐胁迫的证据,也加深了我们对植物盐胁迫响应调控分子机制的理解[37那55].
阐明LNCRNA的功能作用Medicago truncatula在盐胁迫和渗透胁迫下,几种lncrna已经被鉴定出来,包括TCONS_00046739(在根中调节细胞色素P450),TCONS_00097188(通过上调调节光合作用Medtr6g006990基因),Tcons_00047650(上调表达MEDTR3G069280编码磷脂酰肌醇特异性磷脂酶C的基因,TCONS_00116877(会上调Medtr7g094600编码根中谷胱甘肽过氧化物酶的基因),以及TCONS_00020253(上调Na的表达+/小时+交换者基因Medtr1g081900在根)([68.),见图。3.).同样,存在lncrna的存在DRIR调节高表达P5CS1那RD29A那RD29B那AtrbohB那FUT4.那ANNAT7, 和NAC3赋予盐度应力耐受性的基因drirD.突变体和DRIR-Overexpressing Lines拟南芥[37].在棉,盐处理叶组织的深度转录组测序促进了来自1117个独特的LNCRNA的44个差异表达的Lincrnas [86.].通过RT-qPCR对选定的lincrna进行功能验证,揭示了其上调调控作用lnc_388在CIS-Action.富含亮氨酸的重复8(Gh_A09G1182)基因和lnc_883LNCRNA靶向GH_D03G0339MS_CHANNEL蛋白编码基因的盐度应力(表2).作者也证实了lncrna的作用lnc_973和lnc_253作为盐胁迫下ghr-miR399和ghr-156e的靶模拟物[86.].同样,利用叶片和根组织衍生RNA库的深度转录组分析,我们在Lluteño玉米地方品种中探索了1710个响应盐和硼联合胁迫的lncrna [87.].有趣的是,三种普遍玉米品种基因组序列的比较(B73,MO17和Palomero)和LluteñoLandrace确定了955个保守的LNCRNA转录物的存在;然而,755个是LluteñoLandrace独家,这可以解释其盐度和硼胁迫耐受性[87.].为了深入了解lncrna对盐度和硼的响应,对来自848个差异表达的trans-NAT lncrna的12个反式nat lncrna的功能验证表明,它们在控制各种胁迫调节基因的表达方面具有重要作用,包括盐度和硼联合胁迫和烟酰胺代谢过程[87.].因此,所鉴定的lncrna通过调节抗氧化酶编码基因和调节各种Na来控制氧化胁迫,从而授予了盐胁迫耐受性+/小时+与盐度应力相关的交换剂基因和其他调节基因。
LNCRNA在植物中调节营养缺乏
土壤的营养收购仍然是调节植物生长和发展的基本生理过程之一[125].包括许多营养转运蛋白的几种分子机制,积极参与植物营养素稳态[126那127].在各种非编码调节RNA中,在各种植物中发现了MIRNA和LNCRNA调节营养采集的证据[11那14那61.].
在主要营养物质中,磷(P)是促进植物生长发育的基本元素;它也是产生ATP的P源[128].土壤无机磷酸盐(Pi)对植物的有效性受到限制植物生长发育的几个因素的制约[129].在植物中P宿潮的复杂监管网络上有很少的信息[127那130].植物激活了几种分子和生化机制,以改善土壤无机磷酸盐可用性并增加磷使用效率(PUE)[127那129那130].在这种情况下,在各种植物中报道了MiRNA控制磷酸盐可用性的作用[110].同样地,lncrna在植物中调控磷酸盐含量的新作用也已经确立拟南芥[11那93.)、大米(14那131]和模型豆类Medicago truncatula[61.].miR399的工作机制及其靶基因磷酸盐2(Pho2.)是公认的调节磷酸盐含量拟南芥[132那133].随后,Franco-Zorrilla等。[11]揭示了LNCRNA的归纳机制磷酸盐饥饿诱导1(IPS1),可作为miR399的eTM或诱饵,调节靶标Pho2.基因表达与磷稳态拟南芥.因此,考虑到磷酸盐的丰富,lncRNAIPS1与miR399结合并阻止其作用于靶基因Pho2.,这可能是通过泛素化途径取消了磷酸盐转运体的功能,从而限制了根系对过量磷酸盐的摄取([69.),见图。3.).在磷酸盐缺乏条件下,Pho2.基因被抑制,因为miR399降解了转录物Pho2.最终使磷酸盐转运体积累磷酸盐[132].
此外,磷酸盐调节机制为基于“PHR1-mir399-Pho2“途径与磷酸缺乏响应性LNCRNA相关联PDIL1,一个副鸟Mt4 -已经证明Medicago truncatula[61.].作者确定了lncrna的负调控作用PDIL2和PDIL3控制磷酸盐转运基因的表达Medtr1g074930。同样的,工作机制cis-NATPHO1; 2lncRNA作为翻译增强子PHO1; 2在水稻中发现了磷酸盐稳态基因[131].
和P一样,氮(N)是植物生长发育所必需的营养物质,也是植物中氨基酸、ATP和N代谢的N来源[134].据报道,农业重要性各种作物的几个QTL,提高了氮素使用效率(NUE)[134].功能基因组学方法的进展已经确定了几种控制作物氮素利用效率的调控基因和转运基因[135].然而,在植物中不了解n同化的整个分子机制[136].通过RNA-SEQ的最先进的深度转录组测序,进一步推进了对植物中的N encoocation的LNCRNA贡献的理解。在各种植物物种viz中发现了许多N响应的LNCRNA。,米,玉米,杨树[14那95.那96.].LNCRNA的操作机制CIS-NAT.AMT1.1和CIS-NAT.AMT1.2,瞄准这一点AMT1氮稳态基因在水稻中已被广泛识别[14].LNCRNA的研究拟南芥基因组在各种营养缺乏条件下所起的作用trans-acting siRNA3(TAS3.)作为一种重要的靶向于硝酸盐转运蛋白2基因,从而调节氮缺乏环境中的氮转运[99.].
在各种微量营养素中,硼(B)是植物生长发育、细胞膜完整性和细胞壁合成所必需的微量营养素[137那138那139].基因组 - LNCRNA调节B缺乏反应的基因组探索poncirus trifoliata通过链特异性深度转录组分析检测到2101个独特的lncrna [98.].此外,表达谱分析发现在B缺乏胁迫下有729个上调和721个下调的lncrna。通过对所选lncrna的功能验证,阐明了B缺乏胁迫下钙信号通路和植物激素信号转导通路的靶基因poncirus trifoliata[98.].
以上研究结果为进一步深入研究各种lncrna对植物营养缺乏的调控作用奠定了基础。
lncrna在重金属毒性作用下的作用
快速工业化的成果、大剂量化肥的应用以及灌溉用水和耕地中重金属的任意污染对作物产量和人类健康构成了严重挑战[140),尤其是镉。为了减少重金属从土壤进入植物,植物使用了几种调节分子机制[140[-Lncrnas可能在控制植物系统中的重金属的吸收方面发挥至关重要的作用。
RNA-seq分析鉴定出了301个镉敏感lncrna芸苔属植物显著,其中67个是36个cd敏感mirna的etm [141].功能验证Tcons_00091906,Tcons_00033487., 和TCONS_00097191使用QRT-PCR分析的Cd胁迫下的LNCRNA表明了作为EL628609,TC182597和TC203372 MRNA参与CD摄取和排毒的TC203372 MRNA的显着作用[141].同样,Chen等人[88.]利用RNA深度转录组测序对lncrna进行了全基因组研究,提供了lncrna参与Cd响应的上调和下调的证据。此外,通过对DElncRNA的功能分析,进一步了解了lncrna在Cd胁迫下调控半胱氨酸和蛋氨酸代谢相关靶基因的作用(见图)。3.).考虑到水稻砷(As)毒性的证据越来越多,唐等[142]提供了对As响应lncrna以及其他调控水稻As毒性反应的非编码rna的新见解。然而,lncrna对重金属的调控作用机制尚不清楚,有待进一步研究。
lncrna数据库和网络资源
功能基因组学的进展,尤其是RNA-SEQ分析,使得能够发现调节各种生物过程的新型LNCRNA,包括应力反应。然而,LNCRNA的准确预测,它们的结构,基因组内容,保护和功能注释仍然是一个挑战(见[8.])。为了解决这些不足,目前已经开发了一些基于网络的资源和数据库,如NONCODE提供了lncrna全面的生物学功能[143那144那145],plnlncrbase包含来自超过40种的1187种植植物lncrnas [146],植物长链非编码RNA数据库(Plant Long non-coding RNA Database, PLncDB)提供了植物中6480个lncrna的信息拟南芥[147].同样,植物自然反义转录物数据库(PlantnatsdB)提供有关植物NAT的信息,控制各种生理和开发过程[148, Plant ncRNA Database (PNRD)保存了包括lncRNAs在内的25739个非编码rna的记录[149坎塔坦,从10种植物物种中维持45,117个LNCRNA [16), CANTATAdb 2.0。注释植物lncRNAs [155]和PLncPRO提供了水稻和鹰嘴豆中非生物胁迫响应lncrna的信息[72.].Table是植物lncRNA数据库的详细列表3..一些重要的工具,如CPPred [158], REPTree [159],pfamscan [160], 来 [161],plit [156]和cpc2 [162.,可用于区分lncrna和mrna。生物信息学工具和新算法的发展将进一步推动我们在发现新的lncrna及其精确功能注释方面的努力。
结论
植物lncrna数量的迅速增加及其在多种生物过程中的多方面调控作用成为生物学研究的热点[8.那12].然而,在植物物种中,lncrna的全基因组发现、表征和功能注释仍然有限。农作物参考基因组序列的日益普及,将为利用比较基因组分析探索各种lncrna及其序列相似性和“功能保护”提供机会[38].此外,深入的转录组测序,计算生物学的快速进步以及LNCRNA和有效方法/工具的增加数据库可以有助于预测精确的LNCRNA和新型LNCRNA的功能注释。对应于LNCRNA的突变体的缺乏是新型LNCRNA的功能分析的另一个挑战[17].在这种情况下,新型非生物胁迫响应LNCRNA中的CRISPR / CAS9工程突变可以脱落LNCRNA的功能,从而有助于非生物胁迫耐受作物植物的设计[163.].在作物植物中的全基因组的全面地图集的可用性,并综合了解调节各种非生物应激反应的复杂分子机制,使我们能够使用LNCRNA作为潜在的生物标志物,用于裁缝中非生物胁迫耐受性植物未来。
数据和材料的可用性
不适用。
缩写
- lncRNA:
-
长非编码RNA
- miRNA:
-
microRNA.
- ncRNAs:
-
非编码RNA
- RNA-SEQ:
-
RNA测序
- siRNAs:
-
小干扰rna
- eTM:
-
内源性目标模拟
- Lincrnas:
-
长orcencir lncrnas.
- 奈特:
-
自然anti-transcript
- DElncRNAs:
-
差异表达lncRNAs
- vpp4:
-
V-ATPase编码基因
- DRIR:
-
干旱诱导lncrna.
- 可见:
-
差异表达基因
- CBFS.:
-
C-repeat绑定因素
- cor:
-
冷调节基因
- FLC.:
-
开花轨迹C
- 冷空气:
-
冷诱导的长度反义腺瘤RNA
- COLDAIR:
-
冷辅助内肠道非致rna
- RNAPII:
-
RNA聚合酶II
- 病人:
-
磷
- 护士:
-
氮
- B:
-
硼
- IPS1:
-
磷酸盐饥饿诱导1
- 自虐:
-
氮利用效率
参考
- 1.
何敏,何cq,丁nz。非生物胁迫:陆地植物的一般防御和工程多胁迫耐受性的机会。植物科学与技术,2018;
- 2.
[15]张杰,李晓明,李洪祥,崇开。基于温度弹性的作物改良。植物生物学。2019;70:753-80。
- 3.
Kollist H,Zandalinas Si,Sengupta S,Nuhkat M,KangasjärviJ,Mittler R.对非生物应力的快速反应:启动信号转导网络的景观。趋势植物SCI。2018; 24:25-37。
- 4.
植物发育中的miRNAs。1 .植物年报2019https://doi.org/10.1002/9781119312994.apr0649..
- 5.
Wani SH, Kumar V, Khare T, Tripathi P, Shah T, Ramakrishna C,等。miRNA在植物非生物胁迫耐受工程中的应用。Biologia。2020;https://doi.org/10.2478/s11756-019-00397-7.
- 6。
李家,刘C,Sun C等人。植物应激RNA-SEQ Nexus:植物细胞中的应激特异性转录组数据库。BMC基因组学。2018; 19:966。
- 7。
俞y,张y,陈x,陈玉。植物非划分的rnas:隐藏在发展和压力响应中的球员。Annu Rev Cell Dev Biol。2019; 35:407-31。
- 8。
Budak H, Kaya SB, Cagirici HB。参考序列时代植物中的长链非编码RNA。植物科学与技术,2020https://doi.org/10.3389/fpls.2020.00276.
- 9。
Böhmdorfer G, Wierzbicki AT。长链非编码RNA控制染色质结构。趋势细胞生物学。2015;25:623-32。
- 10.
Chekanova农协。长链非编码rna及其在植物中的功能。植物生物学杂志。2015;27:207-16。
- 11.
Franco-Zorrilla JM,Valli A,Todesco M,Mateos I,Puga Mi,Rubio-Somoza I等。目标模仿提供了一种用于调节MicroRNA活性的新机制。NAT Genet。2007; 39:1033-7。
- 12.
Nejat N,Mantri N.长期非编码RNA在植物反应中的新兴作用对生物和非生物胁迫。Crit Rev Biotechnol。2018; 38:93-105。
- 13.
Sun Z,Huang K,Han Z等人。基因组识别拟南芥响应蓝光的长度非编码RNA。SCI代表2020; 10:6229。
- 14.
Shin SY, Jeong JS, Lim JY, Kim T, Park JH, Kim JK,等。水稻转录组学分析(栽培稻使用多个OMIC技术在氮饥饿下的基因和非编码RNA。BMC基因组学。2018; 19:532。
- 15.
Corona-Gomez Ja,Garcia-Lopez IJ,Stadler PF,Fernandez-Valverde SL。植物长期非编码RNA中的拼接保护信号。RNA。2020;https://doi.org/10.1261/rna.074393.119..
- 16.
Szcześniak MW, Rosikiewicz W, Makałowska I. CANTATAdb:植物长链非编码rna的集合。植物细胞生理学。2016;l57:e8。
- 17。
关键词:非编码rna,植物,生物学功能,生物信息学基因组学、蛋白质组学、生物信息学。2015;13:137-47。
- 18。
作者:Ariel F, Jegu T, Latrasse D, Romero-Barrios N, Christ A, Benhamed M,等。非编码转录由替代RNA聚合酶动态调节生长素驱动的染色质环。摩尔细胞。2014;55:383 - 96。
- 19。
AJ先生,Jensen MB, Dalmay T. Baulcombe DC。RNA聚合酶IV指导内源性DNA的沉默。科学。2005;308:118-20。
- 20.
植物的非编码转录与劫持:植物的非编码转录。植物科学,2015;20:362-71。
- 21.
长链非编码rna的进化与功能。细胞。2009;136:629-41。
- 22.
关键词:长链非编码RNA;分类;趋势麝猫。2015;31:239-51。
- 23.
张海燕。长链非编码RNA的生物发生与功能。Nat Rev Genet. 2016; 17:47-62。
- 24.
Mattick JS, Rinn JL。长链非编码rna的发现与注释。Nat Struct Mol Biol. 2015; 22:5-7。
- 25.
关键词:长链非编码RNA,植物,逆境响应,调控机制生物物理学报。2018;503:402-7。
- 26。
切赫TR,施泰兹JA。非编码RNA革命——抛弃旧规则,打造新规则。细胞。2014;157:77 - 94。
- 27。
Ulitsky I, Bartel DP。lincRNAs:基因组学,进化和机制。细胞。2013;154:26-46。
- 28。
王浩,钟培军,刘杰,张智勇,张永明。长非编码自然反义转录本的全基因组识别及其对光的反应拟南芥.基因组研究》2014;24:444-53。
- 29。
吴华,杨玲,陈丽丽。长链非编码rna的多样性及其产生。趋势麝猫。2017;33:540-52。
- 30.
杨L, Froberg JE, Lee JT。长链非编码RNA: RNA世界的新视角。生物化学学报,2014;
- 31。
张曦,王HB,张Y,Lu X,Chen LL,杨L.互补序列介导的外显子化。细胞。2014; 159:134-47。
- 32。
楚q,白p,zhu x,zhang x,mao l等。植物圆形RNA的特征。简短生物形式。2018;https://doi.org/10.1093/bib/bby111.
- 33.
王HV,Chekanova JA。在植物中长的非编码RNA。Adv Exp Med Biol。2017; 1008:133-54。
- 34.
关键词:长链非编码rna;分子机制;摩尔细胞。2011;43:904-14。
- 35.
RNA在意想不到的地方:长链非编码RNA在不同的细胞环境中发挥作用。细胞生物学杂志。2013;14:59 - 712。
- 36.
acta photonica sinica, 2012, 38(5): 736 - 741。番茄lncRNA23468作为内源竞争性RNA进行调节nbs-lrr.在番茄中诱骗miR482b基因-phytophthora infestans.相互作用。Hortic Res。2019; 6:28。
- 37.
秦涛,赵海燕,崔鹏,Albesher N,熊利民。细胞核定位的长链非编码RNA增强了干旱和盐胁迫的耐受性。植物杂志。2017;175:1321-36。
- 38.
Ulitsky I.进化到救援:使用比较基因组学理解长期非编码RNA。NAT Rev Genet。2016; 17:601-14。
- 39.
Simopoulos CMA,Weretilnyk EA,Golding GB。来自不同植物物种的长非蛋白质编码RNA的分子特征表明了系统发育关系的少迹。G3(Bethesda)。2019; 9:2511-20。
- 40.
动物长链非编码rna的分类:适度保护和不完整。基因组医学杂志。2009;10:R124。
- 41.
关键词:植物,长链非编码rna,遗传多样性,保护中国科学:生命科学。2018;61:190-8。
- 42.
马军,白欣,罗伟,冯勇,邵欣,白青,等。两种近缘杨树长链非编码rna的全基因组鉴定及其对盐胁迫的响应麝猫。2019;10:777。
- 43.
mohammad S, Edger PP, Pires JC, Schranz ME。位置保守但序列分化:十字花科和柳科长链非编码rna的鉴定。植物生物学学报。2015;15(5):559 - 562。
- 44.
大豆基因组的长基因间非编码RNA (LincRNA)。植物杂志。2018;176:2133-47。
- 45.
李丽,叶春涛,吴伟,等。玉米长链非编码rna的全基因组发现与鉴定。基因组医学杂志。2014;15:R40。
- 46.
关键词:黄瓜,长基因间非编码rna,全基因组鉴定,进化分析《公共科学图书馆•综合》。2015;10:e0121800。
- 47.
关键词:鹰嘴豆,长基因间非编码rna,花发育,全基因组分析Sci众议员2016;6:33297。
- 48.
Jha UC, Bohra A, Nayyar H.“组学”方法在谷物豆类中应对干旱胁迫的进展。植物品种。2019;139:1-26。
- 49.
Lamaoui M, Jemo M, Datla R, Bekkaoui F.作物的高温和干旱压力及其缓解方法。前化学2018;第一。
- 50。
关键词:小麦,抗旱性,QTL, QTL分析农学。2017;7:5。
- 51。
Ferdous J, Hussain SS, Shi BJ。microrna在植物抗旱性中的作用。植物生物技术学报2015;13:293-305。
- 52。
张c,唐g,peng x,sun f,刘s,xi y.SwitchGrass的长期非编码RNA(黍virgatumL.)在多种脱水应力中。BMC植物BIOL。2018; 18:79。
- 53。
丁震,铁伟,付玲,严勇,刘刚,严伟,等。基于链特异性RNA-seq的木薯干旱响应lncrna鉴定和功能预测。BMC基因组学。2019;20:214。
- 54。
关键词:玉米,长链非编码rna,干旱胁迫,时空动态基因(巴塞尔)。2019; 10:138。
- 55。
Ben Amor B,Wirth S,Merchan F,Laporte P,D'Aubenton-Carafa Y,Hirsch J等人。新的长期非蛋白质编码RNA参与拟南芥分化和应激反应。基因组研究》2009;19:57 - 69。
- 56。
帅P,梁D,唐S,张Z,叶凯,苏y等。基因组识别和新型干旱响应LINCRNA的功能预测杨树trichocarpa.实验机器人。2014;65:4975-83。
- 57.
郑pj,郑h,郑dh, Ha SH, Choi YD, Kim JK。水稻干旱响应非编码rna及其靶基因的转录组分析。BMC基因组学。2016;17:563。
- 58.
张伟,韩志,郭强,刘勇,郑勇,吴芳,等。干旱胁迫下玉米长链非编码rna的鉴定。《公共科学图书馆•综合》。2014;9:e98958。
- 59.
齐X,谢S,刘y,yi f,yu J.基因组的基因分析和福克尾部的非编码RNA响应于深度测序的模拟干旱胁迫。植物mol biol。2013; 83:459-73。
- 60.
李士,俞x,程z,赵p,他,王w等。Cassava中冷和/或干旱反应LNCRNA的基因组鉴定和功能预测。SCI批准。2017; 7:45981。
- 61.
王涛,赵敏,张欣,刘敏,杨超,陈勇,等。豆科模型植物中新型磷酸缺失应答长非编码rnaMedicago truncatula.实验机器人。2017;68:5937-48。
- 62.
闫强,吴峰,严志,李娟,马涛,张勇,等。长链非编码rna和mrna的差异共表达网络Songorica在水分压力和恢复过程中。BMC植物BIOL。2019; 19:23。
- 63.
魏东Q,洪平C等人。长期非编码RNA的系统特征及其对东乡野生稻干旱胁迫的反应。ric sci。2020; 27:21-31。
- 64.
王安,胡娟,高超,陈刚,王斌,林超,等。长链非编码rna的全基因组分析揭示了大白菜耐热性的调控作用(芸苔属rapa ssp。中国人).SCI批准。2019; 9:5002。
- 65.
关键词:拟南芥,冷诱导,Polycomb,长反义转录大自然。2009;462:799 - 802。
- 66.
Sun qw,csorba t,skourti-stathaki k,houthfoot nj,dean c. r环稳定抑制拟南芥FLC基因座的反义转录。科学。2013; 340:619-21。
- 67.
反义COOLAIR介导了春化过程中FLC染色质状态的协调转换。美国国家科学院学报2014;111:16160-5。
- 68.
王tz,刘米,赵mg,陈立,张wh。渗透和盐胁迫中涉及长期非编码RNA的鉴定与表征Medicago truncatula采用基因组高通量测序。BMC植物BIOL。2015; 15:131。
- 69.
Bari R, Datt Pant B, Stitt M, Scheible WR。PHO2、micro- RNA399和PHR1定义了植物中磷酸盐信号通路。植物杂志。2006;141:988 - 99。
- 70.
王健,林健,阚健,王慧,李鑫,杨青,等。中新型干旱响应lncrna的全基因组识别和功能预测Pyrus betulifolia.基因(巴塞尔)。2018; 9:311。
- 71.
吴超,丁铮,陈明,杨刚,等。木薯中lncrna对PEG和ABA处理响应的鉴定和功能预测环境实验机器人。2019;166:103809。
- 72.
关键词:植物长链非编码rna (long non-coding RNAs, lncRNAs), PLncPRO,非生物胁迫响应lncRNAs核酸研究2017;45:e183。
- 73.
Cagirici HB,Alptekin B,Budak H.RNA测序,并在现代和野生麦中的联合非编码RNA。SCI批准。2017; 7:10670。
- 74。
关键词:木薯,褪黑素,干旱胁迫,链特异rna序列,功能预测植物生理学与生物化学,2019;
- 75。
辛敏,王勇,姚勇,宋宁,胡忠,秦东,等。利用微阵列分析和SBS测序对白粉病感染和热胁迫应答的小麦长链非蛋白编码rna进行鉴定和表征。植物生物学杂志。2011;11:61。
- 76。
lncrna在干旱和高温胁迫中调控作用的全基因组研究Brassica Juncea.(印度芥菜)。环境实验机器人。2019;171:103922。
- 77。
宋XM,刘GF,黄ZN,段WK,TAN HW,LI Y等。基因的温度表达模式及其CO表达与RNA-SEQ在非标记大白菜中揭示的LNCRNA。BMC基因组学。2016; 17:297。
- 78。
刘文,程C,林Y,Xuhan X,Lai Z.MRNA和LNCRNA的基因组鉴定和表征在野生香蕉中感冒压力(Musa Itinerans.).《公共科学图书馆•综合》。2018;13:e0200002。
- 79。
acta photonica sinica, 2011, 40(5): 689 - 693 .张锐,张伟,等。拟南芥长链非编码rna的冷依赖表达和选择性剪接。植物科学学报,2019;
- 80。
晶文p,ard r,ivanov m,Marquardt S.转录读取的长型RNA Svalka治理植物冷适应。NAT Communce。2018; 9:4561。
- 81。
王P,Dai L,AI J,Wang Y,Ren F.葡萄葡萄葡萄葡萄与冷与冷离无码RNA(LNCRNA)的鉴定与功能预测。SCI批准。2019; 9:6638。
- 82.
Shea DJ, Nishida N, Takada S, Itabashi E, Takahashi S, Akter A等。长非编码rnaBrassica Rapa.l以下vernalization。SCI REP。2019; 9:9302。
- 83.
[10]冷艳,孙军,王军,刘辉,郑华,等。水稻孕穗期冷响应基因全基因组lncrna鉴定及关联分析(栽培稻l .)。工厂。基因组。2020;13:e20020。
- 84.
赵敏,王涛,孙涛,于鑫,田锐,张文辉。中组织特异性和冷响应lncrna的鉴定Medicago truncatula通过高通量RNA测序。BMC植物BIOL。2020; 20:99。
- 85.
Karlik E,Gözükırmızın.盐度压力中大麦LNCRNA表达分析的评价。Russ J Genet。2018; 54:198-220。
- 86.
邓飞,张旭,王伟,袁锐,沈飞陆地棉盐胁迫下的长非编码RNA(LNCRNA)。BMC植物BIOL。2018; 18:23。
- 87.
Huanca-Mamani W, Arias-Carrasco R, Cárdenas-Ninasivincha S, Rojas-Herrera M, Sepúlveda-Hermosilla G, Caris-Maldonado JC,等。阿塔卡马沙漠超干旱Lluteño玉米对盐和硼胁迫响应的长链非编码rna。基因。2018;9:170。
- 88.
Chen L,Shi S,Jiang N,Khanzada H,Wassan Gm,Zhu C等人。长期非编码RNA的基因组分析,影响镉胁迫下水稻响应的早期阶段的根源发育。BMC基因组学。2018; 19:460。
- 89.
Shumayla SS,Taneja M,Tyagi S,Singh K,Upadhyay SK。高通量RNA-SEQ数据的调查显示了在面包小麦的开发和压力反应期间LNCRNA的潜在作用。前植物SCI。2017; 8:1019。
- 90.
关键词:小麦,根,Ca,长链非编码RNA,全基因组鉴定2+通道阻断剂。植物生态学报,2018;
- 91.
袁军,李军,杨勇,谭超,朱勇,胡磊,等。长链非编码RNA聚腺苷酸化的应激反应调控栽培稻.工厂J. 2018; 93:814-27。
- 92.
Yu F,Tan Z,Fang T,Tang K,Liang K,秋F.综合转录组体分析显示,在玉米涝渍应激伴随湿润的胁迫下,涉及关键代谢途径的长期非编码RNA。基因。2020; 11:267。
- 93.
元j,张y,dong j,sun y,lim bl,liu d等。新型LNCRNA响应磷酸盐饥饿的系统特征拟南芥蒂利亚纳.BMC基因组学。2016; 17:655。
- 94.
任鹏,李斌,马鑫,司恩,赖勇,等。大麦对磷饥饿的适应和恢复的分子机制(大麦芽l .)。前植物SCI。2018; 9:500。
- 95.
陈米,王C,宝H,陈H,王Y.在氮缺乏下杨树新型LNCRNA的基因组鉴定与表征。Mol Genet基因组学。2016; 291:1663-80。
- 96.
吕勇,梁铮,葛明,齐伟,张涛,林飞,等。玉米氮响应基因间和内含子长非编码rna的全基因组鉴定和功能预测(玉米l .)。BMC基因组学。2016;17:350。
- 97.
陈智,姜强,姜平,等。大麦地方品种B968苗期低氮胁迫响应长链非编码rna (lncRNA)的研究植物生物学杂志。2020;20:142。
- 98。
周GF,张LP,Li Bx等。基因组 - 在Trifoliate橙色长期非编码RNA鉴定(poncirus trifoliata(L.)raf)响应硼缺乏症。int j mol sci。2019; 2:5419。
- 99。
Fukuda M,Nishida S,Kakei Y,Shimada Y,Fujiwara T. Gengeome-Intocoding RNA的基因组宽分析对拟南芥的低营养状况 - 反向作用siRNA3响应低氮气的可能参与。植物细胞生理。2019; 60:1961-73。
- 100。
Tan X,Li S,Hu L,张C.在两个染色体油菜籽中长期非编码RNA(LNCRNA)的基因组分析(芸苔属植物显著l)基因型受干旱胁迫和再浇水的影响。植物生物学杂志。2020;20:81。
- 101.
黄瓜热应激响应lncrna、circrna和mirna的共表达和ceRNA网络的系统鉴定和分析(Cucumis sativusl .)。杂志。2020;168:736-54。
- 102.
春化作用通过长非编码rna引发基因内染色质环的形成。Dev细胞。2017;40:302 - 12. - e4。
- 103.
关键词:拟南芥,MAF4, lncrna,反义RNANat Commun。2018;9:5056。
- 104.
关键词:冷诱导,反义转录,反转录开花轨迹C在遥远的草丛中。前植物SCI。2019; 10:72。
- 105.
张欣,等。长链非编码RNA lncRNA973参与了棉花对盐胁迫的响应。中国生物医学工程学报(英文版)
- 106.
Jannesar M, Seyedi SM, Moazzam JM,等。非模式植物中盐相关长链非编码rna的全基因组鉴定、鉴定和功能分析Pistacia Vera.L.使用转录组高通量测序。SCI代表2020; 10:5585。
- 107.
acta photonica sinica, 2011, 41(5): 689 - 693 .朴松,刘强,陈安,杨森I,付勇,戴军,等。植物物候与全球气候变化:当前进展与挑战。2019; 25:22 - 40。
- 108.
Janni M, Gullì M,等。作物植株热应激反应的分子和遗传基础,以及提高弹性和生产力的育种。J Exp Bot; 2020;https://doi.org/10.1093/jxb/eraa034.
- 109.
JHA UC,Bohra A,Jha R,Parida S.综合的“OMICS”方法在热压力下维持全球全球谷物豆类生产力的方法。植物品种。2017; 136:437-59。
- 110.
acta botanica botanica(云南植物研究中心),2011 - 01。植物生物学杂志。2019;70:489-525。
- 111.
低温胁迫下增加作物产量的育种方法和基因组学技术。植物细胞代表2017;36:1-35。
- 112.
Gilmour SJ,Fowler SG,Thomashow MF。Arabidopsis转录活化剂CBF1,CBF2和CBF3具有匹配的功能活动。植物mol biol。2004; 54:767-81。
- 113.
金dh, MR . Doyle, Sung S, Amasino RM。春化:冬季和植物开花的时间。Annu Rev Cell Dev Biol. 2009;25:27 - 99。
- 114.
FLC基因座:一个发现表观遗传学和适应的平台。Annu Rev Cell Dev Biol. 2017;33:55 - 75。
- 115.
Bastow R,Mylne JS,Lister C,Lippman Z,Martienssen Ra,Dean C. vernalization需要通过组蛋白甲基化进行FLC的表观遗传沉默。自然。2004; 427:164-7。
- 116.
HEO JB,SUNG S. vernalization介导的介导的外膜遗传沉默,长内肾内含RNA。科学。2011; 331:76-9。
- 117。
关键词:开花C座,冷诱导,反义rna,进化保护拟南芥蒂利亚纳多年生的亲戚。NAT Communce。2014; 5:4457。
- 118。
油菜科植物农艺性状的表观遗传调控。植物细胞代表2018;37:87-101。
- 119。
Shea DJ, Itabashi E, Takada S, Fukai E,等。花期基因座C在芸苔春化中的作用:面对气候变化春化研究的重要性。农业科学与技术,2018;
- 120.
李X,张某,白j,他y。调整增长周期芸苔属植物作物通过自然反义转录BrFLC.植物Biotechnol J. 2016; 14:905-14。
- 121。
关键词:盐胁迫,组学方法,盐胁迫,豆科植物植物细胞代表2019;38:255-77。
- 122。
Zelm EV,张Y,Testerink C.植物的耐盐机制。Annu Rev植物Biol。2020; 71:403-33。
- 123。
粮农组织。粮农组织土地和植物营养管理服务。罗马:联合国粮食及农业组织;2015.
- 124。
基因组学、生理学和分子育种提高耐盐性的方法。植物生物学。2017;68:19.1-19.30。
- 125.
伊斯兰教女士。水稻植物中氮的感应与吸收分子观。ric sci。2019; 26:343-55。
- 126.
TEGEDER M,Masclaux-DAUBresse C.氮气运输的源和水槽机制。新植物。2017; 217:35-53。
- 127.
关键词:拟南芥,磷胁迫,PHL4,功能特性植物科学与技术,2018;
- 128.
Adnan M, Zahir S, Fahad S, Arif M, Mukhtar A, Imtiaz AK等。在碱性土壤中,溶磷细菌消除了土壤钙化对磷生物有效性的拮抗作用。Sci众议员2018;8:4339。
- 129.
Puga Mi,Rojas-Triana M,De Lorenzo L,Leyva A,Ruyva A,Rubio V,Paz-Ares J.在植物中Pi饥饿反应调控中的新型信号:事实和承诺。CurrOp植物BIOL。2017; 39:40-9。
- 130.
王F,邓米,徐茹,朱X,毛C.磷酸盐输送分子机制及高等植物信号传导。Semin Cell Dev Biol。2018; 74:114-22。
- 131.
李国强,李国强,李国强,李国强,李国强独联体-天然反义RNA作为其同源mRNA的翻译增强子,有助于磷酸盐稳态和植物适合度。植物细胞。2013;25:4166 - 82。
- 132.
邱tj,昂奎,林思,吴传忠,蒋顺芳,苏春林。拟南芥中microRNA对磷酸盐稳态的调控。植物细胞。2006;18:412-21。
- 133.
黄特克,汉族,林Si等。拟南芥蛋白质组学鉴定泛素缀合酶磷酸盐2的下游组分。植物细胞。2013; 25:4044-60。
- 134.
关键词:小麦,氮素利用效率,遗传变异,遗传改良植物生物学杂志。2019;49:35-42。
- 135.
关键词:氮胁迫,小麦,转录组,转录组响应Sci众议员2017;7:1-14。
- 136.
等。玉米氮素利用效率(玉米l):从“组学”研究到代谢模型。实验机器人。2014;65:5657-71。
- 137.
缺硼对植物细胞功能的影响。植物与土壤,1997;
- 138.
Camacho-CristóbalJJ,Anzellotti D,González-Fontes A.短期硼缺乏期间烟草植物酚类代谢的变化。植物理性生物化学。2002; 40:997-1002。
- 139.
Camacho-Cristóbal JJ, González-Fontes A.缺硼降低质膜H+- 酶的表达和硝酸盐摄取并促进烟草根中亚氨酰胺的铵同化。Planta。2007; 226:443-51。
- 140.
关键词:重金属,环境化学,生态毒理学,环境持久性,毒性,生物积累J化学2019;6730305:1-14。
- 141.
冯SJ,张XD,刘奇,谭SK,楚SS,蒙杰,等。甘蓝型油菜中镉毒性反应中长期非编码RNA的表征。rsc adv。2016; 6:82157。
- 142.
唐Z,徐米,ITO H,CAI J,MA X,Qin J,yu D,Meng Y.解密非编码RNA水平对稻米砷胁迫的响应。植物信号表现。2019; 14:1629268。
- 143.
Liao P,Li S,Cui X,郑Y.对植物科学研究的非编码RNA资源全面述评。int j biol sci。2018; 14:819-32。
- 144.
刘C,白B,Skogerbo G,Cai L,Deng W,Zhang Y等。noncode:非编码RNA的集成知识数据库。核酸RES。2005; 33:D112-5。
- 145。
赵颖,惠玲,方松,等。NONCODE 2016:一个信息丰富且有价值的长非编码rna数据来源。核酸研究2016;44:D203-8。
- 146。
宣浩,张磊,刘旭,等。PLNlncRbase:植物中实验识别的lncrna资源。基因。2015;573:328-32。
- 147。
金健,刘杰,王辉,等。PLncDB:植物长链非编码RNA数据库。生物信息学。2013;29:1068 - 71。
- 148。
陈丹,袁超,张杰,张卓,白磊,孟y,等。PlantNATsDB:一个综合性的植物自然反义转录本数据库。核酸研究,2012。2012; 40: d1187 - 93。
- 149.
yi x,zhang z,ling y等。PNRD:植物非编码RNA数据库。核酸RES。2015; 43:S982-9。Szcześniak MW, Rosikiewicz W, Makałowska I. CANTATAdb:植物长链非编码rna的集合。植物细胞生理。2016; 57:E8。
- 150。
PaytuvíGallart A, Hermoso Pulido A, Anzar Martínez de Lagrán I, Sanseverino W, Aiese Cigliano R. GREENC:一个基于维基的植物lncrna数据库。核酸AcidRes。2016年,44:D1161-6。
- 151。
关键词:植物,非编码rna,转座因子,转录因子数据库(牛津大学)。2018; 2018:1-7。
- 152.
周波,赵辉,于军,郭超,窦鑫,宋飞,等。EVLNCRNA:通过低通量实验验证的长期非编码RNA的手动策划数据库。核酸RES。2018; 46:D100-5。
- 153。
周波,赵辉,于军,郭超,窦鑫,宋飞,等。经过实验验证的植物lncRNAs在EVLncRNAs数据库中。方法Mol Biol. 1933; 2019:431-7。
- 154.
陈伟,张刚,李军,张鑫,黄松,向松,等。CRISPRlnc:一个用于lncrna的验证sgrna的手动管理数据库。核酸研究2019;47:D63-8。
- 155.
Szcześniakmw,bryzghalov o,ciomborowska-basheer j,Makałowskai.cantatab2.0:扩大植物长Nonoding RNA的集合。方法Mol Biol。2019; 1933年:415-29。
- 156.
关键词:PLIT,非编码rna,转录组数据,非编码rna引用本文:compput Biol Med. 2019; 105:169-81。
- 157.
孙慧霞,蔡颖慧。植物长链非编码rna (lncRNAs)的生物信息学研究方法:RNA-Seq数据集中lncRNAs的识别和功能解释。方法:《Mol Biol》。
- 158.
童晓,刘绍鹏:基于RNA序列全局描述的编码势预测。核酸研究2019;47:e43。
- 159.
关键词:长链非编码rna,模式识别,模式识别,植物abstract:短暂的Bioinform。2018;20:682-9。
- 160。
Mistry J,Bateman A,Finn Rd。预测PFAM数据库中的活动位点残留注释。BMC生物信息学。2007; 8:298。
- 161。
胡l,xu z,hu b,lu zj。来源:基于多个特征的LNCRNA识别和表征的强大编码潜在计算工具。核酸RES。2017; 45:E2。
- 162。
康永军,杨大川,孔玲,侯敏,魏玲,等。CPC2:一个基于序列内在特征的快速准确编码的潜在计算器。核酸图谱2017;45(W1): W12-6。
- 163。
刘D,墨瓦尔河,胡河r等。新技术加快了园艺植物中非编码RNA的探索。Hortic Res。2017; 4:17031。
- 164。
来自SEPALLATA3的环状rna通过r -环的形成调控其同源mRNA的剪接。Nat植物。2017;3:17053。
致谢
RJ,MK,MZ,中国农业科学院作物科学研究所的支持,中国北京北京。
资金
撰写本文不需要基金。
作者信息
从属关系
贡献
UCJ构思了这个想法并撰写了HN, RJ, NM, MK. MZ, KHMS编辑的MS. UCJ承认来自印度新德里的印度农业研究委员会(ICAR)的支持。作者已阅读并批准稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
Jha, u.c., Nayyar, H., Jha, R.。et al。长期非编码RNA:新兴球员调节植物非生物应力反应和适应。BMC植物杂志20.466(2020)。https://doi.org/10.1186/s12870-020-02595-x
已收到:
公认:
发表:
关键字
- 非生物压力
- 长非编码rna
- 基因调控
- 目标模仿