抽象的
背景
红三叶草(Trifolium praatense.因其营养价值高,改良土壤质量好,在全球范围内被用作饲料植物。红三叶草对刈割的响应表现出特殊的形态特征来弥补生物量的损失。形态反应已经很好地描述了,但其潜在的分子机制和作用的植物生长在田间。
结果
在这里,我们通过比较温室条件下的植物在农业上使用的田地上生长的植物来表征对红三叶草割草的全局转录反应。出乎意料地,我们发现,在现场生长的植物的生物和非生物应激相关变化覆盖其再生相关的转录变化和表征相关蛋白质参与这些过程的转录相关蛋白质。此外,我们可以表明胃纤维素在其他植物激素中也有助于生物量损失后与再生有关的发育过程。
结论
我们的研究结果表明,巨大的生物质损失触发了田间种植植物的转录变化,而不是与生物和非生物胁迫的斗争,并且吉布林素也在割草后割草后与再生后的发育计划发挥作用。我们的结果提供了对割草割草的生理和发育过程的首先见解,可以作为红色三叶草产量改善的基础。
背景
Trifolium praatense.红三叶草是一种重要的世界性饲料作物,具有重要的经济价值。这种多年生植物具有蛋白质含量高、土壤改良特性好等优点,减少了人工氮肥的使用,提高了牲畜的采食量。众所周知,红三叶草的缺点包括在几种土地利用情况下,如放牧或砍伐,持久性差[1,2,3.].T. Praatense.豆科(或豆类)是豆科(或豆类)的一员,由于其经济价值,豆科是植物王国中被研究最多的科之一,其基因组序列可用于像Medicago truncatula三叶草(桶)4],莲花japonicus.(鸟脚三叶草)[5],大豆(大豆)6],菜豆(菜豆)[7],菊苣(鹰嘴豆)8],Vigna Unguiculata.(豇豆)[9],三叶草subterraneum(地下三叶草)[10.]T.媒体(锯齿状三叶草)[11.),而T. Praatense.(红色三叶草)[12.,13.].
面对当今的挑战,如全球气候变化时代对粮食生产的需求增加,以及以环境友好和可持续的方式解决这些问题的目标,需要改善饲料作物,如T. Praatense.[14.,15.].T. Praatense.育种旨在提供改善关键农艺性状的基因型(干物质产量,高品质,抗病和非生物/生物/生物应激,持续性),同时提高其再生能力[2,16.].不幸的是,形态学调查的几个T. Praatense.群体表现出了持久性与不利性状的相关性,如植物的小尺寸和匍匐生长习性[17.].此外,大多数T. Praatense.品种或加入在本地调整,并要求其特定的当地条件显示有利的特征[18.,19.],这降低了育种过程中个体性状的稳定性[20.].T. Praatense.由于高血管分离遗传多样性,表现出显着的内部内部变化,因此,持续和性能响应割草或切割取决于各种损害时的发展阶段[21.,22.,23.,24.].
持久性可以定义为几个生长期间的持续饲养产量[25.]是受多种非生物和生物因素以及植物再生能力影响的复杂性状[26.].具有高再生能力的植物可以存活更频繁和强烈的生物量损失,因此更持久。由于草食病或割草引起的斩首或生物量损失触发受环境条件,植物形态,建筑,发育阶段和基因型影响的复杂反应[21.].在被斩首后,已知的第一个应激反应在其他豆科植物中像M. Sativa.和p .一包括植物激素的产生:细胞分裂素,生长素,和strigolactones[27.,28.,29.].此外,能量储备的动员被激活[30.].可以观察到植物架构的表型可塑性与激素浓度的改变相结合p .一和T. Praatense.斩首后[24.,29.,31.].然而,使植物在巨大的生物量损失后仍能茁壮成长的分子过程仍不清楚,甚至在拟南芥[32.,33.].
在这里,我们比较阳光(切割)与未被淘汰(未切割)的转录组T. Praatense.来自生物多样性探索网站“Heinich-Dün”上两个不同的、经过充分调查的野外地点的植物[34.]以及温室种植的植物。我们的田间样品经过标准的农业处理,因此我们可以区分由非生物因子和田间生物相互作用引起的转录变化与那些调控再生的转录变化。我们提出了在田间和温室割草后再生阶段差异表达的可能有助于植物再生反应的发育调节因子的鉴定和电子表征T. Praatense.并证明吉布林素是红色三叶草中再生形态的具体方面的主要监管机构。
结果
RNA-Seq结果,从头组装,以及连续序列的功能描述
每个文库RNA-Seq产生的总短读数在4470 - 5810万之间,有两个例外(表S4)总计608,041,012个原始读取。参考转录组的从头组装T. Praatense.产生了44,643个Contig,其中41,505个Contigs被注释,29,781种Contigs被鉴定为植物特异性。Contigs的最小长度为124bp,最大长度为15,551 bp(表S5). 在重新组装T. Praatense.每个文库与参考转录组进行比对,确定总体比对率为77.85 ~ 90.32% (TableS6).
44,643个Contig的63%可以映射到已知的轨迹T. Praatense.基因组注释[12.,35.],32%可以映射到未知的轨迹T. Praatense.基因组和5%无法定位到T. Praatense.基因组(图。S2).所有植物特有的contigs都用几个数据库进行了注释(表S3).为了进一步验证我们的复制质量,我们鉴定了两个复制所共享的转录本。我们计算每个转录本的TPM值,以及TPM值< 1的丢弃转录本的TPM值。在所有处理/地方,两个重复的转录本之间共享的比例在90 - 94%之间,表明RNA-Seq数据具有高度的可重复性(表S8).此外,我们通过q-RT-PCR对4个随机选择的contigs tdn_146439 (LTP)、k65_9861 (P5CS)、tdn_69411 (PME44)和tdn_85889 (ENGase85A)进行了RNA-Seq数据的验证。在qRT-PCR和RNA-Seq样本中,这四个基因的表达模式一致。S3).当单独检测单个组织(腋生分生组织(AM)和叶片(L)时,只有LTP表达与RNA-Seq数据相似。
差异表达的基因分析揭示了受到位置和环境条件影响的再生的不同基因子集
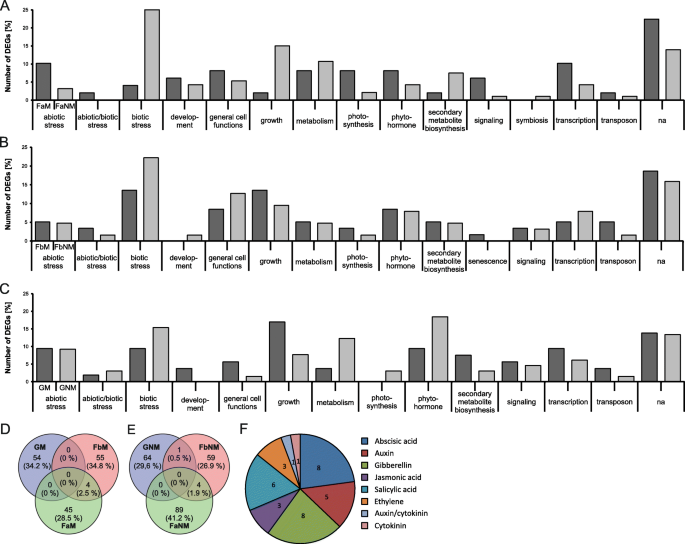
为了确定修剪后再生反应的基因表达响应,我们对a场修剪和a场非修剪进行了数字基因表达分析(FaM和FaNM);场B割vs场B不割(FbM vs FbNM);温室刈割与温室未刈割(GM vs. GNM)的差异(表S9)从修剪过的植物。有趣的是,使用| log2foldchange| ≥ 在所有的比较中,差异表达基因(DEGs)的数量非常相似,从119(GM vs.GNM)到142(FaM vs.FaNM)(表1)1).
去充实
我们对各组的DEG进行GO富集分析,以获得与再生有关的转录变化的差异观点(表1)2,3.,4).然后,将每个样品(FaM、FbM、GM、FaNM、FbNM和GNM)的鉴定GO术语以刈割与非刈割的方式进行比较,以确定各自处理的特异性GO术语。结果表明,GO术语主要涉及一般代谢过程和途径,以及一般反应,在非刈割植物中富集,如“蛋白质代谢过程”、“代谢过程”、“细胞过程”、“分解代谢过程”、“生物合成过程”(图1)。S4和表格第11页).在阳光样本中,我们发现富集的以下GO术语:“核酸结合”(GM);GO与光合作用相关的术语(“光合作用”,“囊体”),细胞成分和蛋白质转运(“Golgi装置”,“细胞质”)和与再生和应激反应相关(“前体代谢物和能量的产生”,“细胞生长“,”细胞通信“)(图。S4和表格第11页).在GO术语“细胞生长”中,存在参与细胞伸长和根毛缺陷3的CONTIGS Gibberellin调节蛋白1,存在于根毛生长中的蛋白质。对于FBM,我们发现与代谢过程(“代谢过程”,“脂质代谢过程”),细胞相关(“细胞质”,“细胞外空间”),酶促和分解代谢过程(“酶调节剂活性”,“催化剂”活动“)和GO术语”结合“,其包括在乙烯生物合成中涉及的蛋白质的CONTIG编码。
有趣的是,大多数官能团在田间和温室位置不同(图。1例如,A-C)例如,在非割草法中,在非阳光下,在非阳光和温室位置上调了与生长有关的更多基因,因此在割草厂中可以上调更多基因。只有与生物应激过程相关的基因在所有未被淘汰的位置上调,并且在割草植物中上调有更多的转座相关基因(图。1a - c)。
转录组分析显示割草后相关的生物学过程。一个-cDEGS与a | log2foldchange | ≥ 2 are shown in percentage share of each class to the corresponding DEG list in bar charts, dark grey bars show datasets from mown, light grey bars indicate datasets from non-mown plants, showing the changing distribution of DEGs in the various biological processes after mowing.一个来自字段a的课程,b来自Field B的Degs类,c温室植物中DEGs的种类。d-eVenn图显示割所样品中的共享上调基因的数量(d)和未被淘汰样本中的共享基因的数量(e)和|log2foldchange|≥2。蓝圈表示温室中基因表达上调,绿色表示田间A基因表达上调,红色表示田间B基因表达上调。f属于“两种领域的DEG列表中的”植物激素“类的次数和温室转录om。饼图显示与不同的植物激素(脱落酸,生长素,植物蛋白和细胞蛋白途径,细胞蛋白,乙烯,赤霉素,茉莉酸和水杨酸)相关的DEG的数量
A地的光合作用和激素相关基因与B地的激素和信号转导相关基因表现出相似的模式。与发育、细胞一般功能和转录相关的基因在A田与温室栽培植物之间也有相似之处,在刈割植物中有更多的转录和发育相关基因上调。出乎意料的是,与衰老相关的基因在b田的修剪植物中上调。然而,由于我们的分析不能区分衰老的激活因子和抑制因子,我们不能从我们的数据中得出结论,修剪植物是激活了还是抑制了它们的衰老程序。
差异表达基因中最大的一组是与生物胁迫相关的基因,在一个位置差异表达的基因高达38%(B区,图。1b).这表明生物胁迫在非刈割植物中发挥了突出的作用。在与生长相关的过程中也可以观察到类似的现象,多达24%的基因在已修剪和未修剪的植物中上调,这表明不同的生长程序在已修剪和未修剪的植物中是活跃的。
我们可以在割草表明他们大规模改变他们的新陈代谢和信号流程后,所有地点都可以说明所有地点的割草厂改变了他们的转录计划。然而,在所有三个位置之间的分子答案到大量生物质损失不同。
为了在处理和/或位置之间找到相似的调控基因,我们生成维恩图来比较修剪样品和非修剪样品中共享的deg的数量(图。1D-E,表十二).在割下的样本中,除了两个转录组(FbM和FaM)中4个上调的deg(图1)外,我们没有发现组间的重叠(图2)。1d).这些基因中有2个没有注释,分别是编码微管蛋白β链2的基因和编码GA反应蛋白GAST1的基因。在非修剪样本中,4个基因在田间转录组(FbNM和FaNM)之间共享。在这些仅在环境胁迫条件下表达并参与植物免疫的几丁质酶a (III类)基因中,有一个富亮氨酸重复序列(LRR)家族蛋白,一个蛋白精氨酸甲基转移酶(ATPRMT6),以及一个我们无法注释的基因。MADS-box转录因子编码基因SEP1.是田间B和温室之间唯一共享的基因。1e).所有三个样本之间没有基因共享,无论是修剪处理还是非修剪处理。
参与发展过程的基因
然后,我们感兴趣的是确定基因的转录变化,指导发育过程所需的再生过程。因此,对DEG分析的结果进行了重组,将DEG划分为16个描述性类别,这些类别由数据库和文献挖掘定义(表S3和S10级). 这些类别(非生物胁迫、非生物/生物胁迫、生物胁迫、发育、一般细胞功能、生长、代谢、光合作用、植物激素、次生代谢产物生物合成、衰老、信号、共生、转录、转座子、,无可用注释)描述主要功能组,并对基因的潜在作用进行广泛分类(表S10级).
我们比较了不割的植物20摄氏度的20摄氏度,并观察到温室植物在再生过程中显示出更多的参数(类生长,转录和植物激素)(图。1c,表S3).前20个DEGs中有3个涉及生长,2个涉及激素基因,2个涉及转录调控基因,10个涉及温室生物和非生物胁迫S3).在田间,植物生长发育相关的deg最多,有4个与生长发育相关,3个与生长发育相关,5个与胁迫相关。田间B区植物的前20个DEGs仅包含2个生长相关转录本和6个应激相关转录本。综上所述,温室栽培的植物表现出了大部分与生长、转录和再生反应有关的DEGs,因为温室栽培的植物在较低的胁迫条件下生长,而与胁迫相关的DEGs在大田栽培的植物中占主导地位(图1)。1A-C /表23,4).
植物激素相关基因
我们感兴趣的是个体植物激素对植物再生反应的贡献T. Praatense.,因为他们众所周知,在调节发展和压力反应中发挥重要作用。我们鉴定了与PhytoOmone合成,稳态,运输和信号相关的Degs,用于数据集中的所有主要类植物激素。具有最多相关的Degs的四种植物激素是:脱离酸(8℃),嗜酸甘油蛋白(8℃,水杨酸(6℃)和唾液酸盐(5℃)(图。1f).众所周知,脱落酸和水杨酸分别参与响应干旱和非生物/生物胁迫。生长素是调节生长和细胞分裂所需的主要植物激素,因此,我们预计在我们的分析中会有大量与这些植物激素相关的DEGs。然而,出人意料的是,发现了8个与赤霉素相关的deg。由于赤霉素在逆境胁迫下调节生长,但迄今为止尚未与生物量损失后的再生有关,我们建议赤霉素作为一种新的候选植物激素来影响再生反应。
在再生过程中,特定的转录调节因子家族在差异上表达
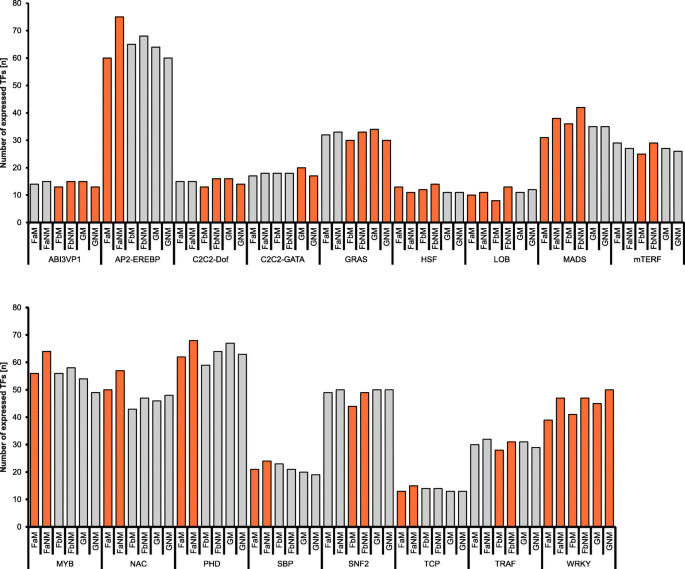
由于应激反应的调节,生长和发育依赖于转录因子的差异活性,我们的目的是通过将转录组映射到PlnTFDB中来识别与刈割后2周发生的生物过程相关的转录[36.].在硅中鉴定了一个特定转录调控家族(TRF)的所有成员,并比较了它们在已修剪植物和未修剪植物之间的表达(表第13页). 图形2在至少10%的成员中,经修剪和未修剪后的TRFs表达显著差异,图。S5和表格第13页还包括那些拥有5%的成员的TRF,在割草中差异化。
鉴定出17个trf,其中至少10%的成员在刈割与未刈割比较中表现出差异表达(图。2):abi3vp1,ap2-erebp,c2C2景深,C2C2-GATA, GRAS, HSF, LOB, MADS, mTERF, MYB, NAC, PHD, SBP, SNF2, TCP, TRAF, WRKY
在FIELD A中,AP2-erebp,LOB,MAD,MYB,NAC,PHD,SBP,TCP和WRKY TRF在未受植物的植物中更加突出,并且只有HSF TRFS对割草更加突出。在割草时,ABI3VP1,C2C2-DOF,GRAS,HSF,LOB,MAD,MTERF,SNF2,TRAF和WRKY TRF减少。在温室生长的植物中,ABI3VP1,C2C2-DOF,C2C2-GATA和GRA的成员,响应割草的数量增加。此外,ARF,C2H2,Homeobox,MyB,NAC和TRF TRFS显示所有位置中表达的变化,尽管仅在5到10%的成员之间进行差异表达(表第13页).
在割草时,两个TRF表现出抑制表达:无论出现,割草植物中的10%的腕子转录物都不那么丰富。此外,发现疯狂盒转录物也在上调,但仅在现场衍生的转录组中。通常,在此分析的17个TRF中只有四种表达在温室衍生的植物中表达的显着变化,这表明它们对比现场衍生的植物造成的强烈反应。六个TRF(AP2-ereBP,MYB,NAC,PHD,SBP,SBP和TCP)显示了对割草的反应转录变化,仅在FIELD a中播种(MTRF,SNF2,TRAF)仅在FIELD B中显示出来,这表明了生物和非生物因子与割割的生物和非生物因子的组合不同于两个现场位置,并且以类似的方式在现场位置和温室之间。
值得注意的是,只有c2C2-GATA TRF至少有10%的成员在温室条件下对割草发生了转录变化,但在大田条件下没有,表明对其他生物和非生物因子的转录变化可能覆盖了再生反应。综上所述,TRF分析显示,刈割反应仅诱导了部分TRF的转录变化,这表明这些转录变化在缓解生物量损失和再生的胁迫中起着主要作用。
在割红色的三叶草割草后,赤虫素也是重要的调节因素
我们已经在前面展示了(图。1g)与赤霉素相关的基因也有差异表达,尽管GA在调节与生物量损失相关的生物过程方面并不为人所知。然后,我们想知道赤霉素是否与红三叶草的再生调控有关,并对红三叶草进行外源赤霉素处理3..
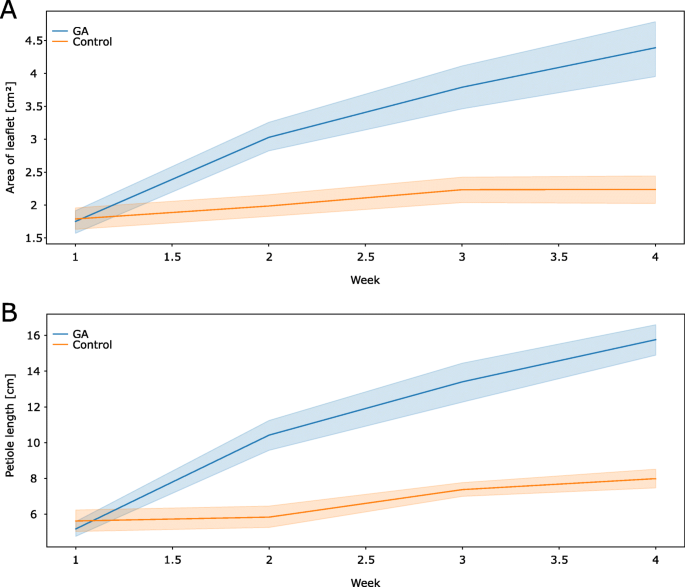
在再生过程中,每周施用赤霉素会导致植株的形态发生显著而特异的变化。3.).以前的工作表明,抛光的植物产生较小的叶片比非割草植物更短而圆角的小叶[24.].因此,在该实验中测量了叶片,芽和花序,叶面积和传单的圆度(图。3.,无花果。S6).在1.5周后,赤霉素治疗的第一个可见效果在1.5周后识别出来,显示胫骨菌蛋白的显着较高的鳞片状面积。后来观察到,经处理的植物的叶柄平均为未处理植物的叶柄(分别为16.7±1.9cm和8±1.2cm)。胃巢素治疗的植物的传单具有4.7±0.9厘米2几乎是未处理植株的两倍(2.4±0.6 cm2).然而,赤霉素处理的植物仅生产总叶面积比对照植物更多。其他形态特征,如花序,叶和芽的数量,胃肠杆菌素治疗不受影响(图。S6). 综上所述,修剪过的植物通常会产生叶柄较短的叶子,限制了它们的小叶面积,叶子变得更圆。赤霉素处理部分缓解了这些发育变化,即刈割后,赤霉素处理的植株叶片较大,叶柄较长,而叶片形状不受赤霉素处理的影响。
讨论
RNA-Seq和组装
DE Novo组件与基于参考的方法进行注释的方法导致了44,643个ContIG,其中29,781个以植物特异性注释(图。S2).与之前的从头组装,4051个附加的contigs没有发现在T. Praatense.1.0(GCA_000583005.2)基因组[12.,35.].估计的基因组大小T. Praatense.是~ 440兆位[27.].这T. Praatense.本研究的转录组数据是~ 大小为55 Mbp,对应于~ 12.5%的转录区在T. Praatense.基因组,其在先前公布的转录om(〜10%(42mbp)范围内[37.]). 有趣的是,我们发现了植物特有的,以前未报道过的contigs,表明T. Praatense.基因组在测序覆盖率和蛋白质编码序列注释方面可能需要改进。
生物和非生物胁迫有助于差异基因表达
与温室植物相比,田间种植的植物面临不同的应激源,我们对田间条件如何影响差异基因表达感兴趣。不同位置的转录组比较显示,刈割温室植物中可能参与再生过程的DEGs所占比例最高。1a-c)。相反,野外转录组显示出非生物和生物胁迫反应的模式。比较未经修饰的田间转录组的前20个DEGs表明,在A和B上生长的植物比在非生物胁迫下面临更多的生物胁迫。A区的一个上调基因是几丁质酶同系物,表明这些植物受到真菌和/或昆虫的攻击。后续分析将环境条件以及生物多样性探险家监测到的生物和非生物胁迫与两个现场位置的差异基因表达相关联,这将是一个有趣的项目,但超出了本工作的范围。相比之下,温室植物前20位的DE转录物包括植物激素和转录相关基因,但也有高比例的生物和非生物胁迫相关基因。这表明这些植物也必须应付压力,但在较小程度上强调它们的再生反应更强烈的20度内。一般来说,未刈割的植物在其生命的某一阶段表现出更多的上调生物胁迫相关基因,当衰老开始时,它们变得更容易受到病原体的攻击。刈割后的植株在再生期没有衰老,其幼嫩器官似乎较少受到生物胁迫的影响。
割草后细胞壁被重塑
在巨大的生物量损失之后,就像割草一样T. Praatense.,植物首先需要密封受伤的组织。已知几种已知在组织核心过程中发挥作用的转录调节剂番茄茄,黄瓜和A。拟南芥(综述[38.])。这些基因的同源基因也被鉴定为差异调控T. Praatense.刈割后的转录组(表第14页),如若干生长素反应因子(ARF)家族成员或无顶分生组织(NAM)家族成员ANAC071,刈割后温室植物中最高上调转录因子的同源物(表第14页)[39.].建议高水平的养肝诱导表达ANAC071通过ARF6和ARF8(在切割茎的上部),同时,切割后直接降低养蛋白水平,激活激活表达RAP2.6 L.此外,通过ARF6和ARF8的生长素信号通过激活茉莉酸的合成爸爸1,因此在一起氧合酶2增加RAP2.6 L在组织重聚期间的表达A. Thaliana.(表第14页)[39.].进一步证明ANAC071可以启动木葡聚糖内转糖基化酶/水解酶家族成员(XTH20和XTH19)的表达,重组半纤维素链,在组织重聚时驱动细胞膨胀[40].有趣的是,我们发现所有细胞壁重塑途径的成员都有不同的表达模式——一些在修剪过的植物中表达上调,比如XTH32(K69_7012,在FBM,TDN_94651中上调,在GM,FAM和FBM中上调),XTH6(tdn_91763, GM上调),XTH8(K71_5058,在GM,FBM中上调),XTH9(TDN_113578,在通用汽车上上调),XTHA(TDN_87930,在通用汽车上上调),氧合酶2(tdn_156279, FbM上调)ARF8(TDN_156886在GM中上调,TDN_156890在GM)上调)(表S9,第13页和第15页).这意味着再生反应中的早期步骤在核心销售中保存,并且细胞壁重塑过程在割草后至少2周继续。
生长素对腋芽的控制损失可在去头后两周检测到
腋芽p .一植物在增长激活时出口毒素,由此介导的过程p .一PIN1 PsPIN1同系物。随着根尖生长素运输的损失,PsPIN1的极化发生变化,这种新的极化导致生长素从休眠的腋芽输出,这是生长素激活所必需的[41.].脱掺杂后PSPIN1的亚细胞靶向和极化在斩波后开始约6小时,然后PSPIN1表达增加。但是,如果升高的PSPIN1表达延长,则仍然不清楚[42.].我们的数据显示,与未修剪的对照植物相比,三个PIN1同源物在温室修剪植物中的表达更高(表S2),表明即使在生物量损失2周后,PIN1的同源蛋白仍在持续表达,这可能有助于激活植物中剩余的休眠芽t . pratense。
与嗜酸甘油蛋白相关的基因会影响再生T. Praatense.与其他植物激素协同作用
伤口诱导第一应力反应,激活植物激素茉莉酸,水杨酸和乙烯的相互作用。这允许个体对各种非生物和生物应力的反应,并且受到物理力,病原体或草食病造成的伤害之间的分化[43.,44.,45.,46.,47.,48.].脱落酸是通过抑制其他植物激素来调节茉莉酸/水杨酸/乙烯诱导的胁迫反应所必需的[49.].在第一次应激反应后,额外的植物激素参与了植物的再生。生长素、细胞分裂素、独脚金内酯和赤霉素参与后期阶段。随着生长素和细胞分裂素诱导的芽生长开始,增加赤霉素浓度可以使芽伸长[31.,50.,51.,52.].此外,生长素、细胞分裂素和水杨酸参与了芽的分枝,高水平的生长素和水杨酸对侧芽的生长有抑制作用。高水平的细胞分裂素促进芽的生长A. Thaliana.,o.苜蓿,和p .一[53.,54.,55.,56.].我们感兴趣的主要是切削过程发生大约2周后,这些激素的作用已经研究过,我们集中在再生期间赤霉素的作用,它还发现最相关的激素基因转录组。赤霉素参与植物发育的多个方面,如细胞伸长、开花时间调节和种子萌发。因此,可以假定,编码各种赤霉素合成、感知和分解代谢的蛋白质的基因会影响植物的形态。我们的RNA-Seq数据显示赤霉素相关基因的高丰度差异表达(图。1F,表S13)),这可能与修剪后植株的形态变化有关,如叶片变圆、暂时的矮化外观和较高的累积生物量产量[24.].
当分析嗜酸甘油蛋白施用到割草植物的形态学效应时(图。3.)),外部胃肠杆菌素应用导致典型的特定性状的消失为割草响应。割草厂开发的叶柄短,叶片尺寸区域较小[24.]但是当用吉布林素治疗时,叶子和叶柄在未被植物中看到的大小。
赤霉素通过刺激抑制生长的DELLA蛋白的降解来促进细胞的扩张和增殖的能力已经建立[57.].吉林蛋白处理的割草植物中叶柄的长度增加与来自非割草的报告数据Pisum sativum.但在这些植物中,叶片大小在赤霉素处理后保持不变[58.],表明在红三叶草生物质损失后的再生反应中的胃肠杆菌蛋白在胃肠杆菌中更具体的作用。而且,它显示在A. Thaliana.升高的赤霉素浓度提高了叶片远端细胞分裂的速率(综述在[59.])。如果这些结果转移到T. Praatense.刈割后的植株经赤霉素处理后,赤霉素处理可使小叶变长。有趣的是,叶片形状没有改变,只是尺寸增加了,这表明生长模式的再生特异性转移不受赤霉素的影响,但类似于幼植物的叶片形状[24.].然而,人们还可以假设在据报道的那个中的红色三叶草叶中的细胞分裂模式A. Thaliana.并且可以更均匀地反应以增强嗜酸甘油蛋白浓度。
有趣的是,赤霉素治疗刈草T. Praatense.由于叶子保留未经处理的割草厂的圆形特性,植物通常不会导致纵向增长更强。这些重生特异性特征也可以在其他物种中找到,例如A. Thaliana.,草莓属ananassa,duchesnea indica.,和G. Max.,赤霉素处理使叶柄拉长,叶片增大,生长习性更加直立[60,61,62,63]. 这可能为增加生物量的积累提供一种新的途径,适合于动物饲料。以前的草实验莱姆萨山和多年生黑麦草显示Gibberellin作用受到施肥的限制[64,65].红三叶草是一种与固氮细菌共生的植物,不依赖于额外施氮,在贫瘠土壤上不施肥可产生高蛋白质含量的生物量。
方法
植物生长条件,嗜酸性植物治疗,组织采样,RNA提取,cDNA文库构建和RNA-SEQ
RNA-Seq的植物材料从3个地点收集(农田和温室,无花果。S1和表格S1).RNA-Seq的现场植物组织于2014年11月6日在长期开放研究平台生物多样性探索“Hainich-Dün”范围内取样[34.],位于德国图林根州。农民和地方当局的收集许可是由生物多样性探索性研究平台集中获得的。T. Praatense.是一个原产地使用的物种,原产于德国,并不属于名古屋议定书。其与后续数据库比较的测序确定了该领域收集的物种。在四个邻点上采样材料;两种阳性牧场(FAM和FBM)和两种非割所(FANM和FBNM)的草地(图。S1和表格S1).在取样的那一年,未割草的牧场和草地没有被放牧或割草。这两种处理具有可比性,因为这是距离最近的试验田,与一个已修剪和一个未修剪的试验田相邻。试验田按照正常的农田进行管理,种植了类似的红三叶草,因为它们是野生的、固定的种群。为温室样品,区域性种子T. Praatense.从Rieger Hofmann Seew Company(Blaufelden,Germany)获得了覆盖着主要覆盖图林荫,萨克森,萨克森 - 安尔哈尔特,德国森林和Uckermarck的区域。温室中的植物在23°C中生长,12厘米的盆中的16小时,每天含水,每10天给予复合肥料(8'8'6'+)。播种122天后,将一半的植物切成5厘米(GM和GNM)。从割草/切割后大约14天取样来自割草植物的材料,以避免与第一应力反应相关的转录物进行测序[38.].采集后,将样品在液氮中快速冷冻。每组(FaM、FbM、GM、FaNM、FbNM和GNM)收集8株植物的茎叶材料。分别的8株植物被分成4株植物的两个生物复制体,这些复制体的RNA随后被汇集到一起。如[38.],组织团聚和伤口闭合的预期时间约为7天(黄瓜和番茄)至14天(A. Thaliana.).基于这些信息,我们假设第一个应激反应和再生的启动T. Praatense.切割/割草后大约2周。
根据制造商的说明,使用NucleoSpin®RNA植物试剂盒(Macherey-Nagel GmbH&Co.KG,德国杜伦)提取RNA。对于每个复制,四种植物的等量RNA汇集在一起。由Eurofins Genomics(Ebersberg,德国)进行cDNA文库的制备和链特异性测序。RNA-Seq文库在Illumina Hiseq2000平台上用chemistry v3.0进行测序,产生2个序列 × 100 bp成对结束读取。
参考转录组和注释的组装
RNA-Seq数据的原始读质量用FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc). Illumina适配器和低质量区域用Trimomatic修剪[42.]使用Illuminaclip,SlidingWindow:5:20和Minlen:50个选项。汇集了质量修剪读取并进行数字归一化[66].使用Trinity计算多个Novo组件[67]和oass [68]所有的k-mer参数都在19到85之间。此外,利用Trinity的基因组草图进行基因组引导组装T. Praatense.1.0(一般条款000583005.2)[12.,35.].使用TransDecoder (https://transdecoder.github.io/).evidalgene管道(http://arthropods.eugenes.org/EvidentialGene/about/EvidentialGene_trassembly_pipe.html)用于基于TransDecoder CDS预测合并和过滤contigs。通过计算非规格化读取到contig的映射率来确定最终的contig的完整性。原始序列的读取可以在NCBI: PRJNA561285上找到。
Contigs上传到“序列分析和管理系统”(SAMS)[69使用SwissProt [70],trembl [71]和植物群落[72](1E-5的电子值截止)数据库。此外,从爆炸点击中提取基因名称或功能描述等属性。contigs被映射到了T. Praatense.使用GMAP参考基因组[73].将所绘制的contigs与草图基因组的参考注释进行比较。将与内参基因具有相似外显子/内含子链的Contigs(包括内含Contigs)划分为内参基因ID。所有非绿色植物群落被丢弃。利用植物转录因子数据库Potsdam (PlnTFDB)对蛋白序列进行blastp搜索,确定了转录因子([36.],版本3.0蛋白质数据库,具有1E-20的电子值截止值。可以在表中找到成绩单的所有功能注释S2).
RNA-Seq的qRT-PCR确认
qRT-PCR样本包括3个来自普通花园实验种植植物的叶片和轴梢分生组织的生物学复制(关于生长条件,请参见Herbert等人(2018)),以及用于RNA-Seq的12个样本。样品处理和RNA提取如上所述。第一链cDNA通过使用RevertAid™h -第一链cDNA合成试剂盒(Thermo Scientific)按照制造商协议合成。
PerlPrimer软件(Marshall,2004)用于设计qRT PCR引物(表1)S6),用相同的循环仪设置进行引物效率测试,标准cDNA稀释系列为模板。RocheLightCycler®480II系统(Roche,Basel,瑞士)用于QRT-PCR。每个样品的总反应体积为20μl,其中5μl1:50稀释的cDNA模板,每次引物(10μm),3μl无菌H组成2o,10μlssybr绿色i master(roche)。QRT-PCR在下列循环仪设置中进行:95℃,5分钟,然后进行45℃,10s,60℃,10s,72℃,10 s。Contig K65_5754用作参考基因[37.]. 每个分析的基因和引物组合使用三个生物重复和两个技术重复。只使用扩增效率在1.8到2之间的引物。对于转录组文库样本,在qRT-PCR分析中仅使用了两个生物复制。按照Pfaffl(2001)所述进行计算。
差异基因表达分析、富集分析和差异表达基因的分类
使用RSEM计算每个样品中最终装配的每个重叠的读取计数[74]与领结映射。鉴别差异表达(DEGs)T. Praatense.基因,使用DESEQ2预先形成所有处理的成对比较[75]工具≤0.01和| log2foldchange | ≥ 2 between FaM and FaNM, FbM and FbNM; GM and GNM respectively. The top 20 DEGs were determined for each comparison based on the expression strength (log2foldchange). Homologs in the next closest species andA. Thaliana.对于每一个人T. Praatense.基于候选基因鉴定T. Praatense.植物染色体组中沉积的基因组序列[72].计算TPM(每百万转录本)值来估计contig表达水平[76] (RSEM读取计数见表第16页).
我们使用从颤抖和SwissProt获得的描述和基因名称来搜索UniProt [77],ncbi [78]和TAIR [79]获得更多信息的数据库(表S3). 原始阅读被组装成重叠群,表现出一种基因结构(ORF),并获得一个假定的注释,以下称为基因。
去浓缩分析和BLAST2GO分析T. Praatense.基因组
为了进一步探索数字基因表达结果并找到更多候选基因/鉴定差异表达的基因簇,具有基因本体学的富集分析(GO)术语[80,81,82]进行了。对于每对成对比较,使用Goseq包筛选上调基因的富集和耗尽术语[83]分别为每种治疗。然后,对每两两比较中确定的氧化石墨烯条件也进行了刈割与非刈割的比较,以显示治疗特定的氧化石墨烯条件。该分析的结果用GOplot程序可视化[84]在RStudio中实现[85]和程序R [86].
两次本地BLAST查册[87, e值为1.0e-3, HSP长度截止为33,使用Blast2GO [88].只有相似性最高的blast hits被用于进一步比较(blast hits的数量= 1),相似性低于50%和e值高于1.0e-4的序列被省略。Blast2GO输出与内部使用NumPy (https://numpy.org/),熊猫(https://pandas.pydata.org/)及海邦(https://seaborn.pydata.org/)应用从PlnTFDB下载的转录因子(TF)列表。我们在Uniprot数据库中搜索了与发育和植物激素相关的基因。随后,我们在我们的注释中寻找giberellin基因的基因idT. Praatense.转录om。基于TPM值过滤匹配,并基于生物合成分类及其调节,分解代谢,激活/抑制或信号/响应,并鉴定转录组内的相应表达模式。
赤霉素治疗
评价赤霉素在黄芩再生反应中的作用T. Praatense.,如[24.].其中7株作为对照,7株喷施100 μM赤霉素3.(荷兰哈勒姆Duchefa Biochemie B. V)如[89].每隔一天每隔4个星期测量每次测量不同的形态特征(叶子数,传单,叶柄长度,花序数量和花序数量和主要芽的数量)。
数据和材料的可用性
在当前研究期间生成和/或分析的数据集可在NCBI数据库存储库中使用,https://www.ncbi.nlm.nih.gov/search/all/?Term=PRJNA561285.
工具书类
- 1。
关键词:科兹洛夫,克里缅科,高斯,伊瓦舒塔红三叶草首个RFLP连锁图谱(Trifolium praatense.红三叶草(红三叶草)的cDNA探针及其在其他红三叶草种质上的可移植性。理论应用,2003;108:105-12。https://doi.org/10.1007/S00122-003-1412-Z..
- 2。
Isobe S,Sawai A,Yamaguchi H,Gau M,Uchiyama K. Trifolium介质×T.Pratense之间的杂交之间的回复后传递的育种潜力。可以j植物sci。2002; 82:395-9。https://doi.org/10.4141/P01-034.
- 3.
Eriksen J,Askegaard M,SøegaardK.红三叶草纳入黑麦草 - 白三叶草喷淋的互补效果,用于放牧和切割。草牧草sci。2014; 69:241-50。https://doi.org/10.1111/gfs.12025.
- 4.
Young ND, Debellé F, Oldroyd GED, Geurts R, Cannon SB, Udvardi MK,等。苜蓿基因组提供了深入了解根瘤菌共生的进化。大自然。2011;480:520-4。https://doi.org/10.1038/nature10625.
- 5。
佐藤S,中村Y, Kaneko T, Asamizu E,加藤T,中尾M,等。豆科植物的基因组结构。Lotus japonicus Theor Appl Genet. 2008; 15:27 - 39。https://doi.org/10.1093/DNARES/DSN008.
- 6.
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W,等。古多倍体大豆基因组序列。大自然。2010;463:178 - 83。https://doi.org/10.1038/nature08670.
- 7.
Schmutz J,McClean PE,Mamidi S,Wu GA,Cannon SB,Grimwood J等。普通大豆的参考基因组和双重驯化的全基因组分析。纳特·吉内特。2014;46:707–13.https://doi.org/10.1038/ng.3008.
- 8.
Varshney RK, Song C, Saxena RK, Azam S, Yu S, Sharpe AG,等。鹰嘴豆(Cicer aritinum)基因组草图为鹰嘴豆性状改良提供了资源。生物科技Nat》。2013;31:240-6。https://doi.org/10.1038/nbt.2491.
- 9.
Lonardi S, Muñoz-Amatriaín M, Liang Q, Shu S, Wanamaker SI, Lo S,等。豇豆(Vigna unguiculata L. Walp.)植物j . 2019; 98:767 - 82。https://doi.org/10.1111/tpj.14349.
- 10
[1]王永平,王永平,王永平,等。三叶车轴草的最新参考基因组揭示了与农艺性状相关的基因。植物生物技术[j];https://doi.org/10.1111/pbi.12697.
- 11
Dluhošová J, Ištvánek J, Nedělník J, ŘepkováTrifolium praatense.)和之字形三叶草(T.媒体) -基因组的异同图。植物科学与技术,2018;https://doi.org/10.3389/fpls.2018.00724.
- 12.
IštvánekJ,Jaros M,Krienek A,řepkováJ.基因组组装和红三叶草的注释(三粒子普拉汀; Fabaceae)。我是J机器人。2014; 101:327-37。https://doi.org/10.3732/AJB.1300340.
- 13。
JJ de V、Ayling S、Hegarty M、Kudrna D、Goicocechea JL、ErgonÅ等,《红三叶草》(Trifolium praatense.(l)基因组草图为性状改良提供了平台。Sci众议员2015;5:17394。https://doi.org/10.1038/srep17394.
- 14。
Jahufer MZZ, Ford JL, Widdup KH, Harris C, Cousins G, Ayres JF等。为大洋洲改良白三叶。作物学报,2012;63:739。https://doi.org/10.1071/cp12142.
- 15.
Barrett Ba,Faville MJ,Nichols Sn,Simpson Wr,Bryan Gt,Conner AJ。通过进料屏障打破:改善饲料遗传学的选择。anim prod sci。2015; 55:883。https://doi.org/10.1071/AN14833.
- 16.
řepkováj,nedělníkj.现代化遗传改进方法Trifolium praatense..中华植物遗传育种杂志。2014;50:92-9。https://doi.org/10.17221/139/2013-CJGPB..
- 17.
DIAY PMB,Julier B,SAMPOUX J-P,BARRE P,Dall'Agnol M.红色三叶草的遗传多样性(Trifolium praatense.L.)通过形态学和微卫星(SSR)标记揭示。Euphytica。2008; 160:189-205。https://doi.org/10.1007/S10681-007-9534-Z..
- 18.
安尼奇亚里科P,普罗耶蒂S。白三叶草的选择提高了竞争能力,扩大了与牧草的相容性,有利于优化豆科植物含量和牧草产量。牧草科学。2010;140:不,不。https://doi.org/10.1111/j.1365-2494.2010.00749.x.
- 19.
福特JL,Barrett Ba。在吃草下改善红三叶草持久性。新西兰草原协会的诉讼程序。2011; 73:119-24。
- 20.
Naydenova G,Hristova T,Aleksiev Y.在临时牧场使用多年生豆类繁殖的目标和方法。生物动漫husb。2013; 29:233-50。https://doi.org/10.2298/BAH1302233N.
- 21.
Tiffin P.耐受草食动物伤害的机制:我们知道什么?EVOL ECOL。2000; 14:523-36。https://doi.org/10.1023/A:1010881317261.
- 22.
Diaz S,Lavorel S,Mcintyre Sue,Falczuk V,Casanoves F,Milchunas DG等。植物特质对放牧的反应?全球合成。Glob Chang Biol。2007; 13:313-41。https://doi.org/10.1111/j.1365-2486.2006.01288.x.
- 23.
关键词:红三叶草(Trifolium pratense),建筑特性,再生,植物abstract:中国草地学报,2015;https://doi.org/10.1111/gfs.12138.
- 24。
Herbert DB、Ekschmitt K、Wissemann V、Becker A。刈割减少了饲料作物生物量生产的变化,并允许低绩效者迎头赶上:一个案例研究Trifolium praatense.l .(红三叶草)。植物生物(Stuttg)。2018; 20:465 - 73。https://doi.org/10.1111/plb.12695.
- 25。
Conaghan P,Casler MD。多年生黑麦草最佳育种系统的理论与实践分析。爱尔兰J农业食品Res。2011; 50:47-63。
- 26。
Ortega F,Parra L,Quiroz A.在智利的持久性改善持久性中育种红三叶草:审查。农作物牧场sci。2014; 65:1138。https://doi.org/10.1071/cp13323.
- 27。
Sato S, Isobe S, Asamizu E, Ohmido N, Kataoka R, Nakamura Y,等。红三叶草(Trifolium praatense.L.)。DNA研究,2005年;12:301–64.https://doi.org/10.1093/dnares/dsi018.
- 28。
Shimizu-Sato S,Tanaka M,Mori H.Auxin-cytokinin射击分支控制中的相互作用。植物mol biol。2009; 69:429-35。https://doi.org/10.1007/s11103-008-9416-3.
- 29。
豌豆芽位置和植株个体发生对枝条枝条形态的影响。阿拉斯加)。安机器人。1995;76:343-8。https://doi.org/10.1006/anbo.1995.1106.
- 30.
Briske DD, Richards JH。植物对落叶的反应:生理、形态和种群的评价。正确答案:Bedunah DJ, Sosebee RE,编辑。野生植物:生理生态学和发育形态学。第1版。牧场管理协会:丹佛,科罗拉多州;1995.p . 635 - 710。
- 31
Kotova LM, Kotov AA, Kara AN。豌豆苗去头后茎和根中植物激素状态的变化。植物生理学杂志,2004;https://doi.org/10.1023/B:Rupp.0000011309.47328.23.
- 32
李某,StridÅ。拟南芥拟南芥中CHS和PR-5基因表达的累积和变化,除去花序茎(斩首)。植物理性生物化学。2005; 43:521-5。https://doi.org/10.1016/j.plaphy.2005.05.004.
- 33
Scholes Dr,Wszalek Ae,Paige Kn。再生模式和ROSette属性有助于拟南芥基因型对顶端损伤的差异补偿性响应。植物生物(Stuttg)。2016; 18:239-48。https://doi.org/10.1111/plb.12404.
- 34
Fischer M,Bossdorf O,Gockel S,HänselF,Hemp A,Hessenmöllerd,等。实施大规模和长期功能生物多样性研究:生物多样性探索。基本应用ECOL。2010; 11:473-85。https://doi.org/10.1016/j.baae.2010.07.009.
- 35
Ištvánek J, Dluhošová J, Dluhoš P, Pátková L, Nedělník J, Řepková J.红三叶草(Trifolium pratense)基因分类与分子标记挖掘。植物科学,2017。https://doi.org/10.3389/fpls.2017.00367.
- 36。
Pérez-Rodríguez P, Riaño-Pachón DM, Corrêa LGG, Rensing SA, Kersten B, Mueller-Roeber B. PlnTFDB:植物转录因子数据库更新内容和新特征。核酸杂志2010;38:D822-7。https://doi.org/10.1093/nar/gkp805.
- 37。
Yates SA、Swain MT、Hegarty MJ、Chernukin I、Lowe M、Allison GG等。基于RNA-Seq数据的红三叶草转录组的从头组装提供了对干旱反应、基因发现和标记鉴定的见解。BMC基因组学。2014;15:453.https://doi.org/10.1186/1471-2164-15-453.
- 38。
asahina m,satoh s。分子和生理机制调节切割植物组织中的组织团聚。j植物res。2015; 128:381-8。https://doi.org/10.1007/s10265-015-0705-z.
- 39。
Pitaksaringkarn W, Ishiguro S, Asahina M, Satoh S. ARF6和ARF8有助于切割的拟南芥花序茎的组织团聚。生物科技植物》。2014;31:49-53。https://doi.org/10.5511/plantbiotechnology.13.1028B..
- 40。
关键词:生物力学,Asahina M, Pitaksaringkarn W, Matsuoka K, Miura K, sagage -Ono K, Ono M, et al.;在生长素流作用下,ANAC071调控的XTH20和XTH19参与了拟南芥切割花序茎的细胞增殖。植物j . 2014; 80:604-14。https://doi.org/10.1111/tpj.12654.
- 41。
摘要/ abstract Balla J, Kalousek P, Reinöhl V, Friml J, Procházka S. Competitive canalization of PIN-dependent auxin flow from腋芽控制豌豆芽的生长。植物j . 2011; 65:571-7。https://doi.org/10.1111/j.1365-313X.2010.04443.x.
- 42。
Bolger AM, Lohse M, Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014;30:2114-20。https://doi.org/10.1093/bioinformatics/btu170.
- 43。
Teichmann T,Muhr M.塑造植物建筑。前植物SCI。2015; 6:233。https://doi.org/10.3389/fpls.2015.00233.
- 44.
Dar TA、Uddin M、Khan MMA、Hakeem KR、Jaleel H。茉莉科植物抗逆性研究进展。环境实验机器人。2015;115:49–57.https://doi.org/10.1016/j.envexpbot.2015.02.010.
- 45.
Schilmiller AL, Howe GA。伤口反应中的全身信号。植物生物学杂志,2005;8:369-77。https://doi.org/10.1016/j.pbi.2005.05.008.
- 46.
Wasternack C.茉莉酸盐在植物压力反应和开发应用方面的作用。Biotechnol adv。2014; 32:31-9。https://doi.org/10.1016/j.biotechadv.2013.09.009.
- 47.
黄H,刘B,刘L,宋S。茉莉酸在植物生长发育中的作用。J实验机器人。2017;68:1349–59.https://doi.org/10.1093/JXB/ERW495.
- 48。
Mur LAJ, Prats E, Pierre S, Hall MA, Hebelstrup KH。将一氧化氮整合到水杨酸和茉莉酸/乙烯的植物防御途径中。植物科学与技术,2013;https://doi.org/10.3389/fpls.2013.00215.
- 49。
李永平,李永平。ABA信号转导在生物和非生物胁迫响应的交叉路口。植物细胞环境。2012;35:53-60。https://doi.org/10.1111/j.1365-3040.2011.02426.x.
- 50.
Kebrom TH, Spielmeyer W, Finnegan EJ。草类为枝条分枝的调控提供了新的见解。植物科学,2013;18:41-8。https://doi.org/10.1016/j.tplants.2012.07.001.
- 51。
王颖,赵杰,陆伟,邓丹。赤霉素在株高控制中的作用:老的作用,新的故事。植物细胞代表2017;36:391-8。https://doi.org/10.1007/s00299-017-2104-5.
- 52。
Morris SE,Cox MCH,Ross JJ,kriantini S,Beveridge CA.断头后的生长素动态与腋芽的初始生长无关。植物生理学。2005;138:1665–72.https://doi.org/10.1104/pp.104.058743.
- 53。
Domagalska MA,Leyser O.信号集成在拍摄分支中。NAT Rev Mol Cell Biol。2011; 12:211-21。https://doi.org/10.1038/nrm3088.
- 54。
Dun EA,de Saint-Germain A,Rameau C,Beveridge CA.豌豆中strigolactone功能和茎分枝反应的动力学。摩尔植物。2013;6:128–40.https://doi.org/10.1093/mp/sss131.
- 55
梅原M,花田A,吉田S,秋山K, Arite T,武田神宫N等。新萜类植物激素对枝条分枝的抑制。大自然。2008;455:195 - 200。https://doi.org/10.1038/nature07272.
- 56
关键词:独脚金内酯,共生信号,枝分枝,调控足底。2016;243:1351-60。https://doi.org/10.1007/S00425-016-2503-9..
- 57
Achard P,Gusti A,Chominant S,Alioua M,Dhondt S,Coppens F等人。吉伯利林信号传导控制拟南芥中的细胞增殖率。Curr Biol。2009; 19:1188-93。https://doi.org/10.1016/j.cub.2009.05.059.
- 58
德马森检察官,切蒂VJ。GA、生长素和UNI表达之间的相互作用控制着Pisum sativum (Fabaceae)的茎发育、叶片形态建成和生长素响应:或者UNI -tac突变体是如何获救的。中国生物医学工程学报2011;98:775-91。https://doi.org/10.3732/AJB.1000358.
- 59
双子叶和单子叶的叶子生长:如此不同却又如此相似。植物生物学杂志。2016;33:72-6。https://doi.org/10.1016/j.pbi.2016.06.009.
- 60.
Guttridge Cg,Thombson Pa。胃纤维素对Fragaria和Duchesnea生长和开花的影响。J Exp Bot。1964年; 15:631-46。https://doi.org/10.1093/jxb/15.3.631.
- 61.
Leite VM, Rosolem CA, Rodrigues JD。赤霉素和细胞分裂素对大豆生长的影响。科学。阿格利司。(电力、布拉兹)。2003; 60:537-41。https://doi.org/10.1590/S0103-90162003000300019.
- 62.
Tsukaya H,Kozuka T,Kim G-T。拟南芥叶柄长度的遗传控制。植物细胞生理。2002; 43:1221-8。https://doi.org/10.1093/pcp/pcf147.
- 63。
拟南芥叶柄伸长与赤霉素20氧化酶基因的关系。植物杂志。2005;138:1106-16。https://doi.org/10.1104/pp.104.059055.
- 64。
Cai Y,Shao L,Li X,Liu G,Chen S.Gibberellin通过调节果酱相关基因的表达抗羊草(Leymus Chinensis)后刺激再生。j植物res。2016; 129:935-44。https://doi.org/10.1007/s10265-016-0832-1.
- 65。
Morvan-Bertrand A,Ernstsen A,Lindgard B,Koshioka M,Le Saos J,Boucaud J,等。Lolium Perenne中的内源性胃蛋白酶和脱落对伸长叶碱和叶护套的裂缝对其内容物的影响。physiol植物。2001; 111:225-31。https://doi.org/10.1034/j.1399-3054.2001.1110214.x.
- 66。
Haas BJ,Papanicolaou A,Yassour M,Grabherr M,血液PD,Bowden J,等人。从RNA-SEQ使用Trinity平台进行参考生成和分析的DE Novo转录序列重建。NAT PROTOC。2013; 8:1494-512。https://doi.org/10.1038/nprot.2013.084.
- 67。
Grabherr Mg,Haas Bj,Yassour M,Levin Jz,Thompson Da,Amit I等。来自RNA-SEQ数据的全长转录组件没有参考基因组。NAT BIOTECHNOL。2011; 29:644-52。https://doi.org/10.1038/nbt.1883..
- 68.
Schulz MH,Zerbino Dr,Vingron M,Birney E. O.:of Novo RNA-SEQ跨越表达水平的动态范围。生物信息学。2012; 28:1086-92。https://doi.org/10.1093/bioinformatics/bts094.
- 69.
Bekel T,Henckel K,Küster H,Meyer F,Mittard Runte V,Neuweger H等。序列分析和管理系统——SAMS-2.0:数据管理和序列分析适应了从传统sanger测序到超快测序技术不断变化的需求。生物技术杂志。2009;140:3–12.https://doi.org/10.1016/j.jbiotec.2009.01.006.
- 70.
Boutet E,Lieberherr D,Tognolli M,Schneider M,Bairoch A. Uniprotkb / Swiss-prot。方法Mol Biol。2007; 406:89-112。
- 71.
baroch A, Apweiler R. SWISS-PROT蛋白序列数据库及其补充数据库TrEMBL。核酸杂志1997;25:31-6。https://doi.org/10.1093/nar/25.1.31.
- 72.
等。植物基因组:绿色植物基因组学的比较平台。核酸杂志2012;40:D1178-86。https://doi.org/10.1093/nar/gkr944.
- 73.
吴泰,瓦丹甲凯克。GMAP:MRNA和EST序列的基因组映射和对准程序。生物信息学。2005; 21:1859-75。https://doi.org/10.1093/bioinformatics/bti310.
- 74.
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12:323。https://doi.org/10.1186/1471-2105-12-323.
- 75。
爱情MI,Huber W,Anders S.使用DESEQ2的RNA-SEQ数据的折叠变化和分散的调节估计。基因组Biol。2014; 15:550。https://doi.org/10.1186/s13059-014-0550-8.
- 76。
Wagner GP,Kin K,Lynch VJ。使用RNA-SEQ数据测量mRNA丰度:RPKM测量在样品之间不一致。理论Biosci。2012; 131:281-5。https://doi.org/10.1007/s12064-012-0162-3.
- 77。
UNIPROT联盟。UNIPROT:通用蛋白质知识库。核酸RES。2016; 45:D158-69。https://doi.org/10.1093/nar/gkw1099.
- 78。
NCBI的资源。协调员。国家生物技术信息中心的数据库资源。核酸杂志2016;44:D7-19。https://doi.org/10.1093/nar/gkv1290.
- 79
Berardini Tz,Reiser L,Li D,Mezherity Y,Muller R,海峡E,Huala E.拟南芥信息资源:制作和挖掘“金标准”注释参考植物基因组。创世纪。2015; 53:474-85。https://doi.org/10.1002/DVG.22877..
- 80.
Camon E, Barrell D, Brooksbank C, Magrane M, Apweiler R.基因本体注释(GOA)项目——GO在SWISS-PROT、TrEMBL和InterPro中的应用。compp Funct Genomics. 2003; 4:71-4。https://doi.org/10.1002/cfg.235.
- 81.
Ashburner M,Ball Ca,Blake Ja,Botstein D,Butler H,Cherry JM等。基因本体:生物学统一的工具。基因Ontol Consortium NAT Genet。2000; 25:25-9。https://doi.org/10.1038/75556.
- 82.
基因本体联盟。扩充基因本体知识库和资源。核酸研究2016;45:D331-8。https://doi.org/10.1093/nar/gkw1108.
- 83.
年轻MD,Wakefield MJ,Smyth GK,Oshlack A. RNA-SEQ的基因本体分析:选择偏差。基因组Biol。2010; 11:R14。https://doi.org/10.1186/gb-2010-11-2-R14.
- 84.
GOplot:一个R包,用于可视化地结合表达式数据和函数分析。生物信息学。2015;31:2912-4。https://doi.org/10.1093/bioinformatics/btv300..
- 85.
拉辛JS。RStudio:一个用于R和Sweave的平台独立IDE。应用经济学,2012;https://doi.org/10.1002/jae.1278.
- 86.
r核心团队。R:统计计算的语言和环境。统计计算基础。2020。
- 87。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ。基本的局部对齐搜索工具。中国生物医学工程杂志。1990;https://doi.org/10.1016/s0022 - 2836 (05) 80360 - 2.
- 88。
Götz S, García-Gómez JM, Terol J, Williams TD, Nagaraj SH, Nueda MJ,等。使用Blast2GO套件进行高通量功能注释和数据挖掘。核酸学报2008;36:36 - 35。https://doi.org/10.1093/nar/gkn176.
- 89。
Blazquez Ma,Green R,Nilsson O,Sussman Mr,Weigel D.Gibberellins通过激活叶状促进剂来促进拟南芥开花。植物细胞。1998年; 10:791-800。https://doi.org/10.1105/tpc.10.5.791.
致谢
我们感谢Andrea Weisert为优秀的技术帮助和饮食蜂蛋酒吧巧妙地提升植物。我们还感谢Volker Wissemann和Birgit Gemeinholzer在持续讨论该项目。BMBF资助项目的生物信息学支持“Bielefeld-Gießen-Bigi(Grant Number 031A533)”在德国网络中的生物信息学基础设施(DE。NBI)中的德国网络(DE。NBI)。这项工作由德国研究基金会(DFG,2547 / 12-1)获得的补助金。
第三方提交
稿件不在其他地方提交。
基金
这项工作由德国研究基金会(DFG,2547/12-1, - 2)获得的资助至A. B ..开放式访问资金,并通过Projekt交易组织。
作者信息
从属关系
贡献
DH获取、分析、解释数据并起草稿件,TG分析、解释数据a起草稿件,OR分析数据,AB构思设计作品,分析数据,修改稿件所有作者阅读并批准提交的版本。
通讯作者
伦理宣言
伦理批准并同意参与
不适用
同意出版
不适用。
相互竞争的利益
不适用。
附加信息
出版商的注意事项
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
补充信息
附加文件1:图S1。
生物多样性探测站样本位置的地图。
附加文件2:图S2。
注释概述:A:可以映射到T. Praatense.基因组,到一个已知的位点,并注释T. Praatense.基因组标识符。B:可以映射到未知T的转录本的分布.普延基因座。C:无法映射的转录物的分布T. Praatense.基因组。D:代表所有12个文库的整个转录组转录本的分布。
附加文件3:图S3。
选择的qRT-PCR分析T. Praatense.基因:A) tdn_146439/ENGase85A (coding A cytosolic beta-endo- n - acetylglucosaminidase (ENGase), B) tdn_69411/LTP(脂质转移蛋白,一种病理相关蛋白),C) tdn85889/PME44(果胶甲基酯酶44),D) K65_9861 P5CS (DELTA1-PYRROLINE-5-CARBOXYLATE SYNTHASE 1)T. Praatense.6个RNA提取物也用于构建转录组文库。转录组数据用TPM标记,所有样本均来自修剪(M)或未修剪(NM)植物。基因表达水平归一化到管家contig k65_5754(没有注释,但在整个转录组中表达相似)。如图所示,分别的contig相对于contig K65_5754的表达式的折叠变化的平均值,误差条表示标准差。在每个图表的右侧显示了每个转录组中各自基因的log2值,以便更好地比较表达模式。
附加文件4:图S4。
富集术语的浓缩分析。A:丰富的GO条款和非阳朔的温室样本。B:在割草和非割草场B样品中丰富的圆形术语。C:播种的富裕术语和非阳光领域的样品。有关相应GO编号的信息可以在表中找到第11页.
附加文件5:图S5。
表达了蒙盛的转录因子家庭成员,而不是割所T. Praatense.植物。y轴为各转录因子家族中上调转录因子的数量。转录组名称(GM, GNM, FaM, FaNM;FbM, FbNM)和转录因子家族在x轴上给出。对转录因子成员(GM vs GNM、FaM vs FaNM、FbM vs FbNM)的表达进行两两比较。在刈割或未刈割条件下,差异超过10%的contigs被标记为红色,5 - 9%的差异被标记为橙色。
附加文件6:图S6。
赤霉素处理植物的建筑特征及生长习性。A-E:在表型监测实验中测量、计数或计算植物特性。赤霉素处理过的植物,蓝色;控制植物,橙色。图表显示平均值和95%置信区间。时间是以星期为单位的。对照植株(左侧)与赤霉素处理植株(右侧)的生长习性,赤霉素处理约2周后再生长(F), 4周后再生长(G)。
附加文件7:表S1。
植物材料的采样位置概述。显示属于生物多样性探索或温室人群的字段的名称。以及位置,坐标和条件(割草/切割而非割草/未切割)。表S3。描述和分类前20度的来源和依据。表S4。每个测序库在修剪前后的reads数(转录组ID)。表S5。T。练习时态。表S6。单个转录om的总体对准率对参考转录组,80%的值良好。表S7。pratense植物特异性对不同数据库的注释。表S8。复制的质量。对于每个库复制,显示了1个以上的转录数为1。此外,显示了与相关副本之间共享的转录物数量,以及对副本唯一的成绩单数。通过计算与复制的成绩单总数相比,通过计算共享转录物的百分比来评估复制品的相似性。表S10。基于DE contigs的主要类。16个主要类别被开发来分组DE contigs。表S11。GO术语在个体转录组中特别富集。表S12。具有相应注释的共享contig。表向。有关讨论部分中描述的转录物的详细信息。表S14系列。引物序列。
附加文件8:表S2。
pratense转录组注释.
附加文件9:表S9。
用Deseq2分析所有转录组比较的转录组.
附加文件10:表S16。
TPM值为T.Pratense成绩单.
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
赫伯特,d。b。格罗斯,T。拉普,O。et al。转录组分析揭示了红三叶草刈割后再生过程中转录组的主要变化红车轴草).BMC植物BIOL.21日,95(2021)。https://doi.org/10.1186/s12870-021-02867-0.
收到:
接受:
发表:
关键字
- 三叶草
- 红三叶草
- RNAseq
- 再生反应
- 生物和非生物胁迫
- 现场条件
- 赤霉素类