抽象的
背景
Paphiopedilum是拖鞋最大的兰花。以前的研究表明,该属的系统发育关系并不熟悉,稀疏的分类群抽样记录了反转重复(IR)膨胀和小单拷贝(SSC)叶绿体基因组的收缩Paphiopedilum.
结果
在这里,我们对77个质体进行了测序、组装和注释Paphiopedilum种(大小范围152,130 - 164,092 bp)。除两个不稳定种的系统发育位置外,基于质体的系统发育解决了属间的关系。利用系统发育学和比较基因组学的方法来阐明植物的质体进化Paphiopedilum.质体系的Paphiopedilum除SSC区域外,其他区域的基因组结构和基因含量都比较保守。大单拷贝/反向重复区域(LSC/IR)的边界相对稳定,而反向重复区域和小单拷贝区域(IR/SSC)的边界在不同物种间存在差异。与IR/SSC边界移动相对应,该属叶绿体基因组经历IR扩展和SSC收缩。IR区域包含了一到六个SSC区域的基因。出乎意料的是,SSC区域的大小、基因顺序和基因含量都有很大的差异,尤其是亚群。Parvisepalum.此外,Paphiopedilum提供了正在恶化的证据ndh光自养植物的基因。估计的蛋白质编码基因的替代率显示了加速的进化速度clpP,psbH,psbZ.由于边界移位转移到IR区域的基因也具有更高的替代率。
结论
结果表明,在紫花苜蓿叶绿体基因组中,IR和SSC均有扩增和收缩Paphiopedilum具有致密采样,并且该属显示SSC区域的大小,基因阶和基因含量的变化。该属提供了一种探讨塑料进化动态的理想系统。
背景
高通量测序技术使获取叶绿体基因组(质体)序列更加实用[1].许多研究使用塑料数据来解决土地植物,叶绿体基因组进化和核苷酸取代的图案和率和核苷酸取代率之间的系统发育关系[2,3.,4,5].这些研究表明,叶绿体基因组在基因组的大小,基因组结构,并横跨被子植物基因置换率引人注目的变化。
虽然土地植物的平均叶绿体基因组是151 kB,但平均倒置重复(IR)区域为25 kB [6,7].已发表的研究表明,红外区域的质体大小和长度有很大的差异。4、8)。的质体大小天竺葵是242,575 BP,IR地区为87,724 bp [8].另一方面,在陆地植物的所有谱系中检测到IR区域损失[9]. 高通量测序提供了一个在更多群体中测试质体进化的好机会。
Paphiopedilum(Venus slipper)是拖鞋兰花中最大的属,主要分布在东南亚,其中一半的物种生长在岛屿上。的测序的质体Paphiopedilum显示IR区域的扩展,而SSC区域的大小和基因含量都大大减少[10,11,12,13].甚至是典型的SSC基因YCF1.,psaC,ndhD已经转移到IR区域Paphiopedilum armeniacum.[10].但是,以前的研究Paphiopedilum质体是基于稀疏分类单元的采样,并且在属水平上IR扩张/SSC收缩的模式是未知的。Paphiopedilum提供了研究对塑体基因组结构和序列演化的边界移位影响的动态的独特机会。
此外,还存在着许多未解决的系统发育问题Paphiopedilum.例如,以往的叶绿体标记无法解决深层的系统发育关系[14,15,16].解决关系Paphiopedilum将促进该属物种形成的研究。最近的系统发育分析表明该属和该组广泛的网状进化。Cochlopetalum在叶绿体基因树上不是单系的[14,15].此外,还有四种分类群的系统不确定性(P. Canhii.,p . fairrieanum,P. Hirsutissimum.,p . rungiyasanum)落在既定的部分/亚因子分组之外的异常形态[15,17].例如,P. Canhii.是来自越南的新描述种,建议属亚属[18,19]. 鉴于此Paphiopedilum是一种具有网状型进化和克隆繁殖的属,目前对该属的系统发育研究相当有限,这些事件对叶绿体基因组的演变的影响是未知的。Paphiopedilum是一种研究叶绿体基因组的演变并测试整个塑料序列是否可以解析属的系统的系统。
努力回答这些未解决的问题,我们使用了基因组撇渣方法来序列77雌噬菌素基因组Paphiopedilum.我们使用比较基因组学来研究Paphiopedilum叶绿体基因组包括四个Paphiopedilum以往研究中报道的质体序列[10,12,13,20]. 我们分析了来自81个国家的所有共享蛋白质编码基因的序列Paphiopedilum研究叶绿体基因组进化速率模式的样本。本研究的目的是1)重建该属的系统发育关系,特别是系统发育不稳定分类群的系统位置(P. Canhii.,p . fairrieanum,P. Hirsutissimum.,p . rungiyasanum),并测试叶绿体基因组是否可以解决最近分化的属的衍生类别;2)表征叶绿体基因组进化模式Paphiopedilum;3)计算编码基因的替代率,评估IR / SSC边界偏移的影响,并测试由于边界档导致的IR区域的基因是降低的替代率。
结果
质体系的Paphiopedilum
我们获得了66个完整的质体序列。其他11个质体序列有1个或2个缺口位于3个属间区域内AT含量较高的区域(的环境-trnG,trnE-trnT,trnP-psaJ).获得的质体基因组序列保存在GenBank中(登录号:MN587749 - MN587825)S1).测序的质体平均覆盖深度超过3000倍S1).我们包括了四个已发表的质体序列,即,p . armeniacum[10],p . dianthum[12],p . malipoense[20),而p . niveum[11在随后的分析中,共获得了81个该属的基因组Paphiopedilum.下载的质体序列Phragmipedium longifolium[10]被用作外组。
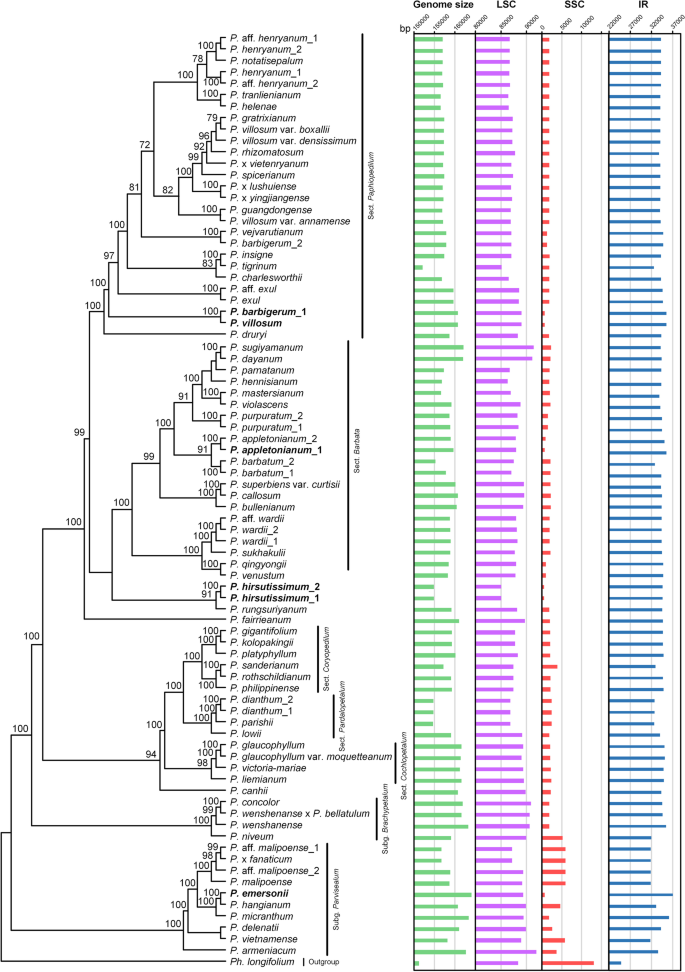
基因组的大小Paphiopedilum范围从152,130 bpP.螈164,092 bp埃默森P.(表S1),埃默森P.有最多的基因(134个基因)和最短的SSC区域之一(660 bp)。该属的质体基因组呈现典型的四部分结构,两个相同的IR副本被一个LSC区域和一个SSC区域分开(图)。S1).与其他高昂植物的塑料相比,Paphiopedilum对IRs进行了多次扩展。IR区域长度扩大至31,743 bp - 37,043 bp,其中8个样本IR区域长度大于35 kb(表1)S1),而SSC区长度收缩至524 bp - 5916 bp(图。1、表S1).此外,南中国海区域Paphiopedilum是基因转移、丢失和重排的热点(图。2,3.和4).SSC区域的大小变异主要是由于典型SSC基因向IR区域转移和缺失/伪基因化所致ndh基因。Subg。Parvisepalum与本属其他物种相比,具有相对较大的SSC区域(图。1和2、表S1).Pearson的相关性分析表明IR区域和SSC区域的长度强烈相关(r= - 0.8050,P < 0.001).
的基因顺序是保守的和的127到134的基因,包括76至81蛋白编码基因,38〜39的tRNA基因,八个rRNA,三至八个假,和20至25个基因在IR区域被复制(表组成S1和S2).除了IR区的重复外,TRNG-GCC重复了p . exul和P。等于off。Exul., 尽管trnq-uug.重复了P. Charlesworthii.,P.螈,巴比革兰变种.两份副本TRNG-GCC有一个核苷酸变异和两个trnq-uug.有八个核苷酸变化。基因密度范围为0.78至0.84,和P.螈具有最短的质体大小和最高的基因密度(表S1).质体基因组GC含量为34.7% ~ 36.3%,蛋白编码基因GC含量为29.7% ~ 46.1%。
此外,我们发现含有内含子的17个基因,其中包括6个tRNA基因(trna-ugc.,trnG-UCC,trnL-UAA,trnI-GAU,trnK-UUU,trnV-UAC)及11个蛋白质编码基因(atpF,clpP,ndhB,petB,petD,rpl2,rpl16.,rpoC1,RPS12.,RPS16,YCF3.).八个蛋白质编码基因包含一个内含子,其中三个(clpP,RPS12.,YCF3.)包含两个内含子(表1)S2).
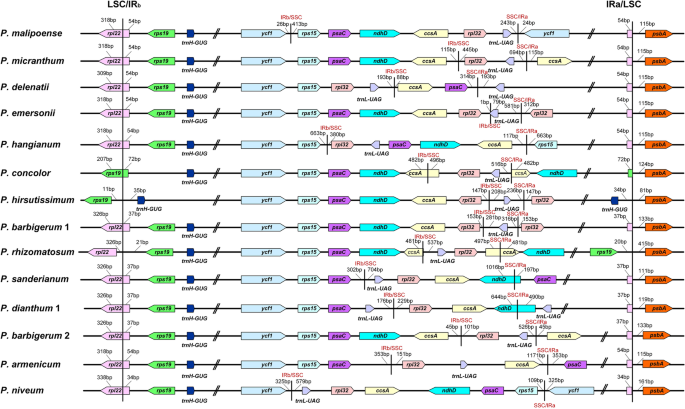
LSC /红外b边界相对稳定。虽然LSC / IRb交界处是开启的rpl22.在大多数物种(81 76个样本),则LSC / IRb交界处是开启的RPS19在p . concolor和p . wenshenanse×P颠茄属, 之间rpl22.和RPS19在P. Rhizomatosum.之间,RPS19和trnh-gug在P. Hirsutissimum.(图。4).与LSC/IR边界相比,IR/SSC边界Paphiopedilum物种之间变化(图。4).SSC / IR边界的大量变化主要是Subg。Parvisepalum.在大多数其他样品(81个样本中的56个)中,SSC / IR结的一端位于代际间隔区trnL-ccsA近的trnL-UAG,而SSC/IR的另一个连接点位于CCSA基因(图。2).
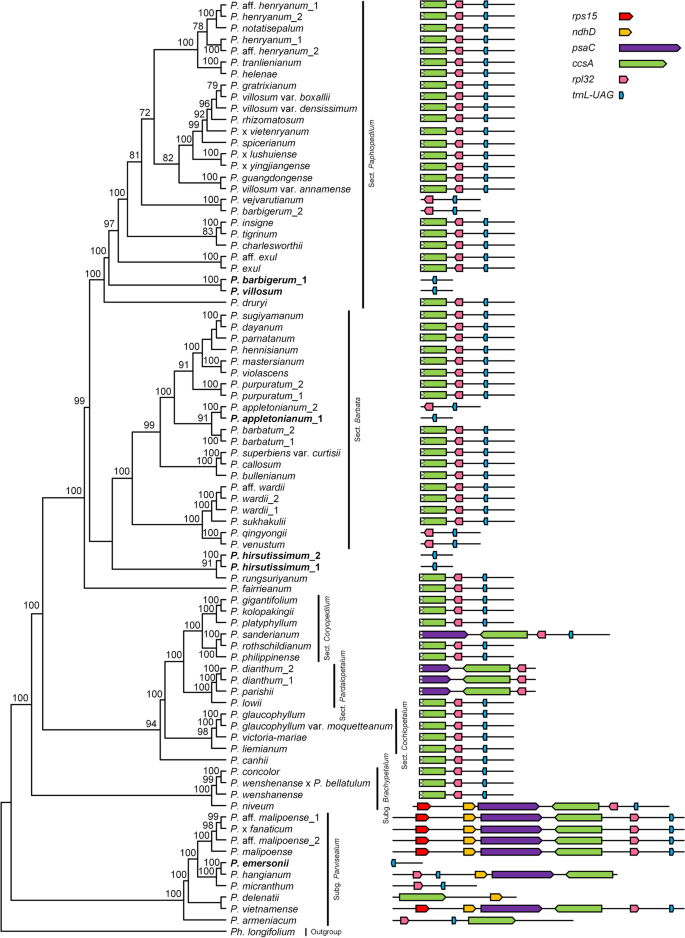
SSC区域的收缩导致典型的SSC基因转移到IR区域。一到六个来自SSC区域的基因转移到IR区域。例如YCF1.在所有测序样本中转移到IR区域,而ψndhD,psaC,RPS15在大多数物种中被纳入IR区域。的基因CCSA偶尔在IR区域扩展,以及trnL-UAG在中被转移到IR区域p . delenatii,p . dianthum,p . parishii(图。2).
基因组比较表明SSC区域Paphiopedilum在基因含量,基因阶和基因取向方面大大不同(图。2和4,S2).大多数物种的SSC区域包含trnL,rpl32.,部分CCSA,而五个物种的SSC区域正处于消失的边缘,p . appletonianum,P.barbigerum,埃默森P.,P. Hirsutissimum.,P. Villosum.只包含trnL-uag.在这个区域(图。2).此外,基因psaC和ψnadD在subg的6个样本中保存在SSC区域。Parvisepalum(图。2).此外,同一物种中可能存在两个方向不同的SSC副本[21].王和Lanfear [22]利用长读测序对陆生植物的结构异质性进行了检测,发现大部分陆生植物个体中都存在叶绿体结构异质性,因此没有考虑SSC区域的方向。根据基因含量和基因定位,将SSC区域分为12种类型(图2)。S2), VIII型为优势型(81个样本中有56个)(图。2).I型和II型在基因含量中是相同的,但在基因方向上有所不同rpl32.和trnL-UAG(图。S2).IX型和X型的基因含量也相同,但在IX型中,两个基因运行方向相反,而在X型中,两个基因运行方向相同(图2)。S2).类型XI和类型XII都有trnL-UAG,但XII型中有一个核苷酸已转移到IR区域。Subg。Parvisepalum有六种类型,而教派。Cochlopetalum只有一种类型(viii型)(图。2).当SSC类型绘制在系统发育树上时,结果表明,SSC类型不是血型特异性,即使是密切相关的物种也具有不同的SSC类型,例如Subg中的物种。Brachypetalum和subg。Parvisepalum(图。2).令人惊讶的是,SSC区域的基因含量具有嵌入式变异。例如,一个样本p . barbigerum包含trnL-UAG,而其他样本包含trnL-UAG和rpl32..两个样本p . appletonianum还具有不同的SSC类型(图。2).
基因的获得和损失Paphiopedilum还分析了样品。一些基因损失在整个属中共享(例如,一些ndh基因),而其他基因损失是谱系特异性的(见表)S3).大部分的ndh基因是伪基因化的(ΨndhD,ΨndhJ和ψndhK)或丢失(ndhA,ndhC,ndhE,ndhF,ndhG,ndhH,ndhI)Paphiopedilum种质体,除ndhB.本研究测序的81个样本中,有76个样本保留了完整的ndhB.此外,完整的开放式阅读框架ndhJ保存在23个样本中,包括4个门派样本。Cochlopetalum和19个样本。Paphiopedilum(表S3).基因ndhC和ndhK被保留为SUBG假。Parvisepalum和教派。Concoloria但在其他物种中消失了(TableS3).特别是在五个已测序的物种(p . barbatum,p . dayanum,p . platyphyllum,P. sugiyamanum.,P.螈),全部11个ndh基因丢失或伪基因化(图。2、表S3).
除了在ndh基因,我们发现过早终止诱导其他蛋白编码基因(cemA和YCF15.).大多数的注释副本(48份中的38份)cemA被保存为假基因,而YCF15.被保留作为假基因(表S3).的pseudogenizationcemA主要是由于聚结构的滑点和过早的止损密码子的出现,而伪血管生成化YCF15.是由于一个以上的提前终止密码子的出现。
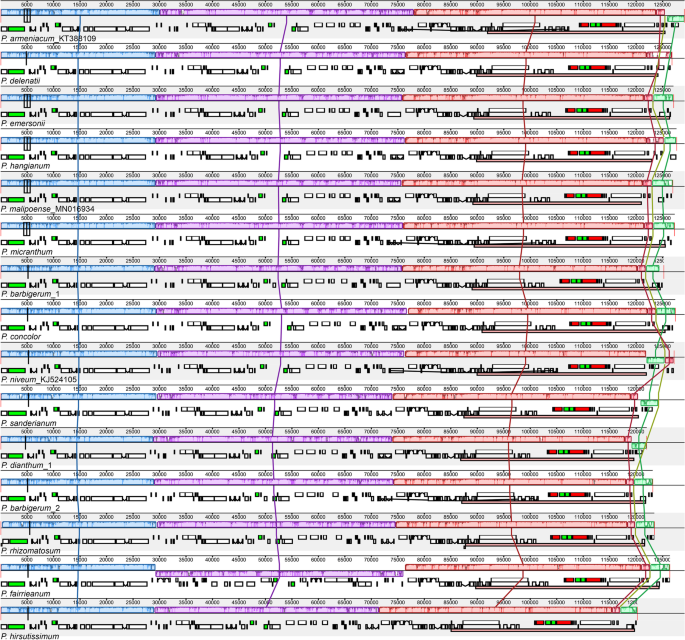
质体系的Paphiopedilum还显示多个结构重排。我们发现SSC区域的广泛结构变化,特别是在Subg中。Parvisepalum,包括SSC基因的反转和重组和IR / SSC边界的偏移(图。2,3.和4).特别是,我们发现了一个47 kB的反演跨越以季戊四醇四硝酸酯到clpP在p . fairrieanum,这是其他物种所没有的Paphiopedilum(无花果。3.,S1).
系统发育分析
对三个串联数据集进行系统发育分析。基于不同矩阵的ML和BI分析结果几乎相同,只是支持值不同,因此我们直接使用基于整个质体的结果。各部分之间的关系与先前的研究一致(图。1和2, 图。S3) [14,15].系统发育分析显示亚群。Parvisepalum在树的根部,然后SUBG。Brachypetalum,然后是subg中的五个部分。Paphiopedilum.将五个部分分为两种枝条,由派系形成一个思工。Cochlopetalum -(教派。Coryopedilum.,教派。Pardlopetalum.)和其他由派系形成的思考。Barbata和教派。Paphiopedilum,只有少数终末节在树中仍未解决,特别是种在组。Paphiopedilum(图。S3和S4).
此外,四种神秘的物种Paphiopedilum所有人都嵌套在subg中。Paphiopedilum.p . fairrieanum簇生的与节。Paphiopedilum教派。Barbata支持度高(BP = 100, PP = 1.00)的支P. Canhii.簇生的与节。Cochlopetalum -(教派。Coryopedilum.,教派。Pardlopetalum.)支(BP = 94, PP = 1.00);与此同时,P. rungsuriyanum.和P. Hirsutissimum.形成了一个支持值较高的支系(BP = 91, PP = 1.00),与其他支系的关系不明确(图2)。S3).在具有相同拓扑的树中删除四个分类群,但有一些分支的升高值(图。S4).
核苷酸替换率分析
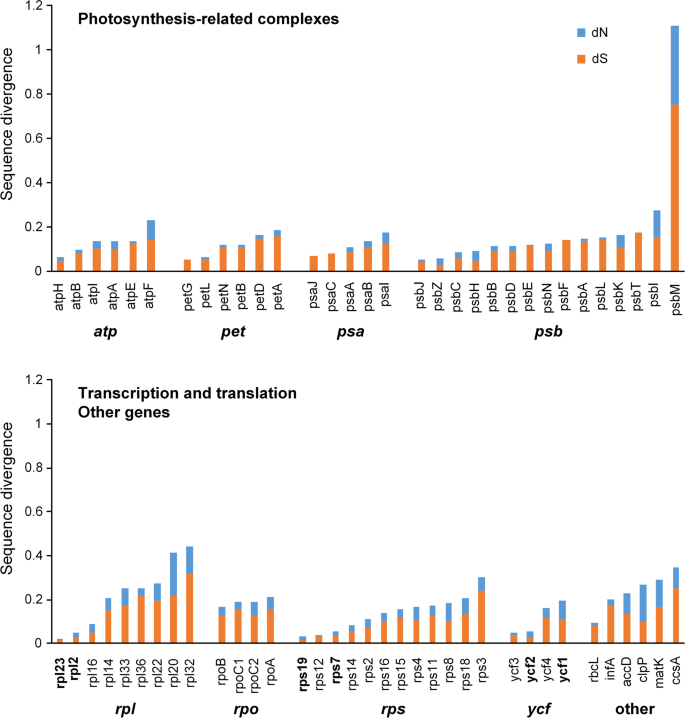
同义和非同义发散度均值较低(dN = 0.0471, dS = 0.1222), dS值几乎是dN的3倍(图3)。5、表S4).我们检测到正选择的信号clpP(DN / DS = 1.6561),psbH(dN/dS = 1.0318)psbZ(dN/dS) = 1.5768). 我们的分析表明,糖尿病肾病(dN)和糖尿病肾病综合征(dS)在糖尿病患者中均显著增加psbM(DN = 0.3585,DS = 0.7519)(图。5),这是由中帧移突变引起的p . niveum(kj524105)。SC区域中的基因比IR区域中的基因具有更高的替代率。出乎意料地,由于边界偏移导致的IR区域转移到IR区域的基因没有显示出较低的取代率(图。5、表S4).
讨论
发展史Paphiopedilum
我们基于整个塑料的文学发生决定了部分之间的关系,这与从部分细胞质DNA标记获得的最近似的文学发生是一致的(图。S3) [14,15].系统发育关系解决了组的单系性。Cochlopetalum具有高bootstrap支持(BP = 98, PP = 1.00),而在以往的研究中,该分支的解析较差[14,15].四种神秘的物种全部落在既定的部分之外。p . fairrieanum集群与教派。Paphiopedilum教派。Barbata(BP = 100, PP = 1.00)P. Canhii.集群与教派。Cochlopetalum -(教派。Coryopedilum.,教派。Pardlopetalum.)分枝具有中等支撑值(BP = 94, PP = 1.00)。S3).但是,其他两个神秘的分类征的系统发育位置(P. Hirsutissimum.和p . rungiyasanum)聚在一起,支持值高(BP = 99, PP = 1.00),与p . fairrieanum,教派。Paphiopedilum和教派。Barbata(图。S3).值得注意的是,去除这些物种后,一些支系的支持值略有增加。例如,节的支持值。Cochlopetalum增加到99(图。S4). 神秘分类群的不确定系统发育位置可能与叶绿体基因组中发现的异质性有关(未发表的数据)。节中的短分枝长度。Paphiopedilum建议他们最近的辐射,这意味着本节中的物种在他们的形态初步阶段。另一方面,长分支长度代表与密切相关的物种的分化。例如,长分支p . druryi可能是由于它在印度南部的孤立分布。
叶绿体基因组的进化Paphiopedilum
最大的 (埃默森P., 164,092 bp)及最小(P.螈,152,130bp)塑料大小不同〜12 kb,基因数范围为127至134基因(表S1).基因组大小和基因数量的变化主要是由于IR的膨胀和丧失ndh基因,基因组大小大于平均质体大小(151 kb) [6].与其它光自养兰花叶绿体基因组序列进行了比较石斛兰[23] 和槽舌[24],塑料Paphiopedilum在属水平上有更多的大小变异和结构变异(图。1、表S1).此外,该属的GC含量(34.7 ~ 36.3%)在属水平上也比兰科其他已测序属的GC含量变化更大,更低,如:兰花(36.8−37%)[25],石斛兰(37.31−37.68%)[23),而槽舌(35.3−35.5%)[24].GC含量较低可能与该属极长的at重复区域有关。我们发现在一些物种中AT重复长度超过100 bp,特别是在LSC区域有缺口的11个样本中(见表)S1).
LSC /红外b边界Paphiopedilum是相对稳定的;LSC /红外b居住在rpl22.而属的IR/SSC界限与其他兰科植物不同,且在属水平上存在差异(图2)。4).由于IR/SSC边界的不稳定性,叶绿体基因组Paphiopedilum经历IR膨胀和SSC收缩。一般陆地植物的IR为25 kb [6,7],虽然IR地区Paphiopedilum约为32 ~ 37 kb, IRs约占质体的40 ~ 45%。IR区域长度的增加是由于IR/SSC边界移动的结果。
在其他高昂的血统中也报告了大型IR扩展(几千碱基),包括金合欢和inga.[26],Erodium[27],西番莲[28,29),而天竺葵属植物[8,30].在天竺葵属植物IR向LSC和SSC区域扩展。然而,在Paphiopedilum主要包括前SSC基因。IR扩展到SSC区域也被报道在其他物种,例如尖形穆萨[31],Pedicularis ishidoyana[32),而香草[11,13,33].
塑料的大红外标准是有助于塑性稳定化,因为它们的缺失通常与基因令的严重变化相一致[34].IRs被认为是通过同源重组诱导修复机制来稳定质体[35].然而,大的IRs对该属的质体稳定性没有影响。随着IR/SSC边界的转移,SSC区域在Paphiopedilum已缩短和可变SSC长度(524 - 5913 BP)。SSC区域的收紧与损失有关ndh基因和前SSC基因转移到红外区域。例如,的524 bp的SSC区域P. Hirsutissimum.小于兰科的其他测序塑性小。其他未链接的光滑营养植物还记录了SSC地区的强烈缩小,例如番荔枝(2966个基点)36],细辛(0 - 14bp) [37],Lamprocapnos海棠(1645个基点)38],P. ishidoyana(27个碱基)32],天竺葵属植物(c. 6.7 kb) [8),而香草(c。2 KB)[11,13,33].有趣的是,其他经历SSC收缩的属在属的水平上具有相对稳定的SSC长度,例如,所有测序的质体天竺葵属植物共享一个6.7 kb的SSC区域[8,而SSC区域的长度Paphiopedilum它的变化更大。SSC收缩了细胞的质体Paphiopedilum与其他紧系SSC相比,具有独特的特征,包括基因含量变异、长度变异和基因重排(图1)。2,3.和4),根据拖鞋兰其他属的叶绿体基因组序列[10,11,39],建议发生SSC收缩Paphiopedilum从它的姊妹分支分裂。
除大小变化外,南中国海区域Paphiopedilum经历了广泛的基因重新排列(图。3.).组中大多数种的SSC区域。Concoloria,教派。Cochlopetalum,教派。Coryopedilum.,教派。Paphiopedilum和教派。Barbata保留trnL-UAG,rpl32.和截短CCSA片段(trnL-UAG(+)-rpl32(+)-ccsA-p(+),型viii,约1.8 kb)。在剩余的物种中,该区域的基因含量,基因阶数和长度是高度可变的(524-5913bp)(图。1和2,S2;表格S1).在其他高管培养物中,SSC区域中有两个基因簇,即, rpl32(+) -trnL-UAG(+) -CCSA( +) 和ndhD( −)-psaC( −)-RPS15(−)。在其他兰花中,如菊科[40],凤仙花[9,39],兰花[25],和蝴蝶氏植物阿芙罗狄蒂[41],在两个簇的基因是在相反的方向。然而,原rpl32.(+) -trnL-UAG(+) -CCSA(+)键仅在p . armeniacum[10].在该属的其他物种中,上述基因链被打破并转变为trnL-UAG(+) -rpl32.(+) -CCSA( +) 和trnL-UAG( −)-rpl32.( −)-CCSA(−)在大多数情况下,这意味着SSC区域存在反转和重组。基因排列了rpl32.和CCSA(通常在质体中分离)彼此相邻(如图。2和4).此外,我们在LSC地区找到了47千桶的反转p . fairieanum(图。3.,S1c) 。在LSC区域有一个61kb的倒位,以前在凤仙花formosanum[11].但这些倒置的机制尚不清楚。
完全的基因复制rpl32.,trnL-UAG,CCSA,ΨndhD,psaC,RPS15,YCF1.)由于IR地区的扩张而被记录。此外,还记录了IR区域之外的基因重复。我们发现了TRNG-GCC在两个物种的LSC中复制,而trnq-uug.在三个物种的LSC中重复,两次重复事件均发生在第。Paphiopedilum.第二份TRNG-GCC和trnq-uug.与其他兰花的序列相同,说明这两个拷贝可能是水平转移的,特别是trnq-uug..基因的两个拷贝有八个核苷酸变异。的重复trnq-uug.其他研究也有记录[42,43].
大部分的ndh基因被假基因化或从属中丢失,但功能ndhJ和ndhB在一些世系中被保存下来(表S3).考虑到ndhA,ndhE,ndhF,ndhG,ndhH,ndhI也迷失在P长叶[10]而在它们被保存c . formosanum[11] 和C.血吸虫[39,我们推断这些基因的丢失发生在共轭叶属的祖先中。伪拷贝ndhK和ndhC在大多数苏木样品中都保存了。Parvisepalum和subg。Brachypetalum(表S3)在Subg中丢失。Paphiopeilum,建议ndhK和ndhC在Subg的祖先迷失了。Paphiopeilum.考虑到保留ndhJ和ndhB属中的(表S3),我们推断ndhB和ndhJ可能是最近发生的,因为它们被反复地保留在末端类群中。
此外,5个物种中的4个拥有全部11个ndh基因伪种或丢失在岛上分布,另一个(P.螈)分布于内地。这五种植物都是光自养植物。此外,5种植物分别属于3个区(图。2)并嵌套在不同的植物中,表明有四个独立的ndh属级别的损失/假性化事件。上述证据提供了线索ndh在这个属中,复合体正在退化,并且ndh基因是不是谱系特异性,表明非常最近的一些pseudogenizationndh基因。独立退化ndh基因也被发现兰花[44].另外,整个质体都失去了ndh其他光合自养兰花的基因,例如Erycina pusilla[45],槽舌[24],p .阿佛洛狄忒[41),而香草普林洛杉矶[11]也曾在以前的研究中报道过.Lin等[46甚至提出损失ndh复合物可能增加了从飞营养营养到异养的生命史的转变。
Kim等人[8建议ndhF基因与IR / SSC结稳定性强烈相关,而NIU等人的结果13研究表明ndh与IR/SSC连接稳定性密切相关的基因。虽然IR/SSC的边界与其他兰花不同,但大部分都比较稳定Paphiopedilum物种,我们推断ndhF或ndh与属级的IR/SSC结稳定性无关。这个ndhF所有已测序物种的基因都丢失了Paphiopedilum,以及失去所有11种功能的5个物种ndh基因有稳定的IR/SSC连接(图。2). 我们推断,还有其他潜在的机制与该属的结构变异有关。
叶绿体基因的替代速率
属的替代率低(DN = 0.0471,DS = 0.1222)并在基因中变化,DS值几乎三倍的DN(图。5、表S4).我们检测到正选择的信号clpP(DN / DS = 1.6561),psbH(dN/dS = 1.0318)psbZ(dN / dS = 1.5768)。在一些被子植物谱系中,例如金合欢和inga.[26],西番莲[29),而硅宾[47],积极选择clpP,与蛋白质代谢有关。另外两个被积极选择的基因参与了光合作用:psbH是否在树荫下进行了积极选择选用[48],PSBZ显示肯定选择杜鹃pulchrum[49].
以往的研究表明,IR区域的基因比SC区域的基因具有更低的替代率[30,50,51].与兰花中的Sc区基因相比,IR区域中的基因也具有相对较低的替代速率[25].出乎意料,YCF1.迁移到IR区域,但仍有较高的替代率(dN = 0.0821, dS = 0.1140),且其替代率高于IR区域的其他基因。甚至psaC(dN = 0, dS = 0.0825)RPS15(dN = 0.0388, dS = 0.1183)转移到IR区域的基因在IR区域的分化程度高于其他基因。此外,CCSA(dN = 0.0932, dS = 0.2543)rpl32.(dN = 0.1222, dS = 0.3193)大多保留在SSC区域,且在SSC区域的分化程度高于其他基因(图1)。5).翁等[8发现了类似的现象天竺葵属植物在质体基因组中扩展的IR基因没有较低的替代率。有趣的是,光合作用相关基因的分化度相对于其他基因的分化度较低(图2)。5).
结论
分析叶绿体基因组是研究兰花分子演化的关键方法。然而,以前发表的兰花研究主要集中在异养种类和亚家族血症症。我们使用比较叶绿体基因组序列来揭示进化史Paphiopedilum.基因组的大小Paphiopedilum比其他被子植物略大,所有的物种Paphiopedilum经历IR膨胀和SSC收缩。IR区域的扩展范围从〜7 kB到〜12 kB,而IR扩展Paphiopedilum与原SSC基因转移到IR区域有关。细胞体的比较表明,红外光谱的膨胀与ndh损失促成属中的大小变化。因为在IR / SSC的界限的变化,的SSC区域的Paphiopedilum叶绿体基因组在长度和基因含量上表现出巨大的差异。Paphiopedilum为研究叶绿体基因组进化的动态和检测与IR/SSC边界变异相关的异质性速率提供了最好的机会。虽然基因组重排主要见于线粒体基因组(例如[52),这项研究提供了高度活跃的基因组重排在SSC区域的证据Paphiopedilum密集采样。的ndh基因Paphiopedilum经历了不同程度的退化,5个物种失去了/假基因化全部11个ndh基因。然而,这些基因的损失/假生化的基础的机制是未知的。这项研究揭示了在叶绿体基因组中发生的速度和进化变化的模式Paphiopedilum并提供了如何研究兰科的叶绿体基因组进化的良好举例。
方法
分类单元取样与建库
本研究测序的植物材料取自深圳市国家兰花保护研究中心(NOCC)S1),包括从GenBank下载的四个物种。共81个样本,代表63个物种,覆盖7个主要分支Paphiopedilum,还包括四种天然混合动力车和一个人造的杂种。基于以前的系统发育研究,P长叶被选为外群体。
采用CTAB法提取鲜叶总基因组DNA [53].本研究构建了350 bp插入的配对文库,并在Novogene(中国北京)Hiseq平台(150 bp)上进行测序。我们得到了每个样本20g的原始数据。
序列组装和注释
CLC Genomics Workbench v.10.1.1过滤高质量的对端reads (https://www.qiagenbioinformatics.com/),然后使用CLC Genomics Workbench v.10.1.1(不同的字大小和气泡大小)或novoplasy3.5 (K-mer = 39,大亚基从p . armeniacum作为种子序列)[54].组装的Contigs以遗传v.11.1.2(BioMatters,Inc。)合并以构建塑料草案。然后,我们映射到佐剂V.11.2中的塑料草案,检查含糊不清地区。塑料的注释在佐毒V.11.1.2(BioMatters,Inc。)中进行,再加上手动校正。
用细胞器生成塑料地图[55].IR和SC区域的边界由REPuter定义[56]的默认设置。我们计算了Geneious v.11.1.2的GC含量。然后,使用渐进式Mauve来可视化基因组结构的变化[57删除IRa。提取蛋白质编码区(CDS)、rRNA、tRNA、基因间间隔区(IGS)和内含子序列并进行比对,生成原始CDS、rRNA、tRNA、IGS和内含子序列。
系统发育分析
用于系统发育分析,68个蛋白质编码基因序列(ndhB包括),4个rRNA基因序列,30个tRNA基因序列,87个基因间间隔区(见表)S2,S5)用肌肉v3.8.1551对齐[58]使用默认设置并手动优化。用Gblocks v 0.91b去除IGS和内含子的排列不良区域[59]使用默认设置,但在多达一半的序列中允许有间隙。然后,将它们连接到不同的数据集中。我们获得了三个串联数据集:1)蛋白质编码序列(CDS)和rDNA;2) 基因间间隔区(IGS)、内含子和tRNA;和3)全质体(CD + IGS + 内含子 + 核糖核酸 + tRNA)。
最大似然(ML)的树木是从RAXml 8.2.4 [推断60.在GTRGAMMA模型中使用1000个引导复制。贝叶斯推理(BI)由MrBayes执行[61.].PartitionFinder v2.1.1 [62.用于确定在纠正的Akaike信息标准(AICC)和贪婪搜索下的基因/代际间隔物预定分区的最佳分区方案。分区方案用于以下分析。对于贝叶斯推理,一个寒冷和三个渐进式的Markov链条蒙特卡罗(MCMC)链被运行10,000,000次循环,并重复两次以避免杂散的结果。每1000代的一棵树被采样,每次运行都有一个烧伤的前2500个样本。
核苷酸替换率分析
我们在pamlv中使用了CODEML程序。4.9(模型) = 0) [63.]通过F3X4密码子模型计算67个蛋白质编码基因的平均非纯粹替代率(DN)和同义替代率(DS)。拆除区域被排除在速率估计(CleanData = 1)。从整个塑料推断的ML树用作输入树。
相关分析
IR长度和SCC长度的相关性在RStudio version 1.4.1106中计算[64.].使用cort .test函数进行评估p-值与皮尔逊相关检验。
数据和材料的可用性
本研究中产生的所有塑料沉积在NCBI数据库中(https://www.ncbi.nlm.nih.gov/) (GenBank登录号MN587749 - MN587825,见表S1),所有支持该结论的数据集均可在Dryad数字资源库中找到。在相关手稿被接受之前,这些数据将保持隐私。在本手稿中产生的所有其他数据可由通讯作者在合理的要求下提供。
缩写
- AIC:
-
akaike信息标准
- 地址:
-
腺苷胸腺嘧啶
- 双:
-
贝叶斯推理
- 英国石油公司:
-
自举概率
- 英国石油公司:
-
基对
- BS:
-
分支机构的支持
- 光盘:
-
蛋白质编码序列
- cp:
-
叶绿体
- CTAB:
-
Cetyltrimethylammonium溴化
- DN:
-
非同义
- dS:
-
同义突变
- GC:
-
鸟嘌呤 - 胞嘧啶
- GTR:
-
一般时间可逆
- IGS:
-
基因间序列
- 红外光谱:
-
反向重复
- IR.一个:
-
倒置重复A.
- IR.b:
-
倒置重复B.
- 美国国税局:
-
反向重复区
- kb:
-
千轴
- LSC:
-
大单副本
- ml:
-
最大似然
- 核糖体rna:
-
核糖体rna
- SC:
-
单拷贝
- SSC:
-
小单副本
- 图示:
-
转移RNA.
参考
- 1.
Wilkinson MJ,Szabo C,Ford CS,Yarom Y,Croxford Ae,Camp A等人。用下一代测序替换Sanger以改善植物参考DNA条形码的覆盖率和质量。SCI REP。2017; 7:46040。
- 2.
Jansen RK, Ruhlman TA种子植物质体基因组。在:博克R,努普V,编辑。叶绿体和线粒体的基因组学。纽约:施普林格;2012.103 - 26页。
- 3.
Tonti Filippini J、Nevill PG、Dixon K、Small I。我们能用1000个质体基因组做什么?植物J。2017;90(4):808–18.
- 4.
Barrett CF,Sinn Bt,Kennedy啊。异养兰氏兰属的前所未有的平行光合损失。mol Biol Evol。2019; 36(9):1884-901。
- 5.
在一个异养兰花复合体中,密集的次种取样揭示了快速和独立的质体降解轨迹。新植醇。2018;218(3):1192 - 204。
- 6.
Ruhlman TA, Jansen RK。开花植物的质体基因组。在:马利加P,主编。叶绿体生物技术:方法和协议。纽约:Humana公司出版社;2014年第3-38。
- 7。
Ruhlman TA, Jansen RK。畸变或类比?天竺葵科的非典型质体。在:Chaw S-M, Jansen RK,编辑。植物学研究进展,第85卷。剑桥:学术出版社;2018.p . 223 - 62。
- 8。
翁ML,Ruhlman Ta,Jansen RK。倒置重复的扩张不会降低替代率天竺葵属植物质体基因组。新植醇。2017;214(2):842 - 51。
- 9。
Mohanta TK, Khan A, Khan A, Abd_Allah EF, Al-Harrasi A.质体的基因丢失和进化。BioRxiv。2019.https://doi.org/10.1101/676304:676304.
- 10.
Kim HT, Kim JS, Moore MJ, Neubig KM, Williams NH, Whitten WM,等。7个新的完整的质体序列揭示了猖獗的独立缺失ndh横跨兰花的基因家族以及倒置重复/小单拷贝区域边界的相关不稳定性。PLoS ONE。2015; 10(11):E0142215。
- 11.
林春生,陈建军,黄玉涛,陈明涛,张文杰,等。的位置和转移ndh兰科植物叶绿体起源的基因。Sci众议员2015;5:9040。
- 12.
侯宁,王刚,朱艳,王丽,徐建军。稀有濒危草本植物叶绿体全基因组分析长瓣兜兰(芦笋:兰科西)。保守遗传资源。2018; 10(4):709-12。
- 13
牛志,薛强,朱松,孙军,刘伟,丁欣。4种兰花的完整质体序列:对兰科植物进化的认识和质体突变热点的利用。植物学报2017;8:715。
- 14
Chochai A,利奇IJ,Ingrouille MJ,菲MF。的分子系统学Paphiopedilum(Cypripedioideae;(兰科)基于核糖体ITS和质体序列。中国科学(d辑:地球科学)2012;
- 15
郭艳艳,罗永波,刘志军,王学强。网状进化和海平面波动共同推动了拖鞋兰的物种多样化(Paphiopedilum)在东南亚。Mol Ecol。2015; 24(11):2838-55。
- 16
蔡志聪,廖柏聪,高永志,陈振华,蒋永忠。系统发育和历史生物地理学PaphiopedilumPfitzer(兰科)基于核和塑性DNA。前植物SCI。2020; 11:126。
- 17。
Yap JW。马来拖鞋兰的分子和基因组进化(Paphiopedilum部分Barbata).伦敦:玛丽女王大学;2016.
- 18。
鲷GJ,Gruss O.Paphiopedilum亚属MegastaminodiumBraem&Gruss,一个新的子根属Paphiopedilum canhii.兰花消化。2011;75(3):164 - 5。
- 19。
Górniak M, Szlachetko DL, Kowalkowska AK, Bohdanowicz J, Canh CX。分类放置Paphiopedilum canhii(Cypripedioideae;基于细胞学、分子和微形态学证据。中国科学院地理科学与资源研究所。
- 20.
李丽琴,黄娟,陈丽娟,张启元,陈建斌。完整的叶绿体基因组麻栗坡兜兰(兰科)。线粒体DNA B. 2019;4(2): 2617-8。
- 21.
Palmer JD,Nugent JM,甲料洛杉矶。天竺葵叶绿体DNA的不寻常结构:三尺寸的倒置重复,广泛的基因重复,多重反转和两个重复家庭。Proc Natl Acad Sci USA。1987; 84(3):769-73。
- 22.
王W,Lanfear R.稳定和广泛的结构性异质在叶绿体基因组中,通过新的长读取量化方法揭示。BioRxiv。2019.https://doi.org/10.1101/692798:692798.
- 23.
牛Z,朱S,潘Ĵ,李力,孙健,丁X.对比分析石斛兰塑性突变热点的塑料和效用。SCI REP。2017; 7:2073。
- 24。
李志辉,马旭,王大勇,李云霞,王春伟,金晓辉。质体基因组的进化槽舌(兰科西)近期辐射。BMC EVOL BIOL。2019; 19(1):63。
- 25。
杨j-b,唐m,李h-t,张z-r,li d-z。完全叶绿体基因组的属兰花:阐明物种鉴定、系统发育和种群遗传分析。BMC Evol Biol. 2013;13(1):84。
- 26。
Dugas Dv,Hernandez D,Koenen EJ,Schwarz E,Straub S,Hughes Ce,等。模仿豆科植物塑料进化:IR扩展,串联重复扩展,加速进化率clpP.Sci众议员2015;5:16958。
- 27。
张军,张军,翁明良,等。可变的存在倒置重复和质体稳定性Erodium.Ann Bot。2016; 117(7):1209-20。
- 28。
Rabah SO, Shrestha B, Hajrah NH, Sabir MJ, Alharby HF, Sabir MJ,等。西番莲质体测序揭示了广泛的基因组重排。ĴSYST EVOL。2019; 57(1):1-14。
- 29。
王志强,王志强,王志强,等。质体基因组中基因组重排和核苷酸替换的高度加速西番莲亚属Decaloba.中国科学(d辑:地球科学)2019;
- 30.
朱安,郭伟,顾磊杰,范伟,Mower JP。质体反向重复的进化动力学:扩张、收缩和损失对替代率的影响。新植醇。2016;209(4):1747 - 56。
- 31.
香蕉叶绿体基因组的研究进展(小果野蕉对质体单子叶植物进化的洞察。PLoS ONE。2013; 8 (6): e67350。
- 32.
赵W-B,崔B-H, Kim J-H, Lee D-H, Lee J-H。完整的质体测序揭示了半寄生的SSC区域极度减少Pedicularis ishidoyana(列当科)。关键词:神经网络,神经网络,神经网络
- 33.
Vanillon的质体基因组香草pompona,兰科西。线粒体DNA B. 2017; 2(2):689-91。
- 34.
帕尔默·JD,汤普森WF。当一个大的反向重复序列丢失时,叶绿体DNA重排更为频繁。细胞。1982;29(2):537 - 50。
- 35.
王志强,王志强,王志强,等。陆地植物质体染色体的进化:基因含量、基因顺序、基因功能。植物学报。2011;76(3):273-97。
- 36.
Blazier JC,Ruhlman TA,翁M-L,拉赫曼SK,萨比尔JS,詹森RK。RNA的发散聚合酶被子植物质体基因组α亚基由基因组重排介导的。SCI代表2016; 6:24595。
- 37
Sinn BT, Sedmak DD, Kelly LM, Freudenstein JV。被子植物质体中小单拷贝区域的总复制:重排和反向重复不稳定性细辛.我是J机器人。2018; 105(1):71-84。
- 38
重组质体基因组的研究进展Lamprocapnos海棠:IR界限转移,反转和拆放变异。SCI批准。2018; 8(1):13568。
- 39
Kim JS, Kim HT, Kim J-H。单子叶植物最大的质体基因组:拖鞋兰中包含AT残基重复的新基因组类型凤仙花日本血吸虫.植物分子生物学代表2015; 33(5):1210至20年。
- 40。
Niu Z,Pan J,朱S,Li L,Xue Q,Liu W等人。完全塑性的比较分析Apostasia wallichii和Neuwiedia singapureana(Apostasioideae)揭示了光合兰花IR/SSC边界的不同进化动态。植物学报2017;8:1713。
- 41.
常C-C,林H-C,林I-P,Chow T-Y,Chen H-H,Chen W-H等。叶绿体基因组蝴蝶氏植物阿芙罗狄蒂兰科植物与禾本科植物进化速度的比较分析及其系统发育意义。中国生物医学工程学报。2006;23(2):279-91。
- 42.
过W,Grewe楼科博-Clark公司A,番W,段Z,亚当斯RP等人。内质体基因组并存的优势和不足化学计量的异构体Juniperus植物并在愚蠢的演变期间已经多次转移。基因组Biol Evol。2014; 6(3):580-90。
- 43.
易霞,高玲,王波,苏永杰,王涛Cephalotaxus奥利维(头孢噻肟):进化比较头孢菌裸子植物叶绿体dna和反向重复副本丢失的研究。生物医学进展。2013;5(4):688-98。
- 44.
Kim HT, Chase MW。质体基因的独立降解ndh基因家族在兰花属种兰花(兰科;附睾亚科)。公共科学图书馆一号。2017;12(11):e0187318。
- 45.
潘宜昌,廖德昌,吴福华,等。一株兰花模型植物的完整叶绿体基因组序列:Erycina pusilla适用于热带文心兰繁殖。PLoS ONE。7 (4): 2012; e34738。
- 46.
林春生,陈吉文,赵春春,肖华华,杨春娟,金晓辉,等。某些兰花叶绿体和核基因组中伴随NDH复杂相关基因的丢失。工厂j . 2017, 90(5): 994 - 1006。
- 47.
关键词:叶绿体,全基因阳性选择,同义替换率,重复,indel进化CLPP1基因。PLoS ONE。2008; 3(1):E0001386。
- 48。
高立志,刘玉玲,张德,李伟,高杰,刘玉玲,等选用叶绿体基因组促进了对不同生态环境的适应。Commun杂志。2019;2(1):278。
- 49。
沈静,李旭,朱旭,黄旭,金松杜鹃pulchrum杜鹃花科植物的比较遗传分析。森林。2020;11(2):158。
- 50.
Wolfe KH,Li WH,夏普首相。核苷酸替换率在植物线粒体、叶绿体和核DNA之间差异很大。美国科学院学报,1987年;84(24):9054–8.
- 51.
佩里·阿斯,沃尔夫·科赫。豆类叶绿体DNA中的核苷酸替换率取决于反向重复序列的存在。摩尔·伊沃。2002;55(5):501–8.
- 52.
李文杰,郭伟,李文杰,李文杰。一个植物属中重复介导的线粒体基因组重排的高和可变率。生物医学进展。2018;35(11):2773-85。
- 53.
道尔J,道尔J。一种用于少量新鲜叶片组织的快速DNA分离程序。植物化学公牛。1987;19(1):11–5.
- 54.
novo成形术:从全基因组数据重新组装细胞器基因组。核酸学报2017;45(4):e18。
- 55.
Greiner S, Lehwark P, Bock R. OrganellarGenomeDRAW (OGDRAW)版本1.3。1:细胞器基因组的图形可视化扩展工具包。核酸Res. 2019;47(W1):W59-64。
- 56.
引用本文:陈志强,陈志强,陈志强,陈志强:重复分析在基因组规模上的多重应用。核酸学报2001;29(22):4633-42。
- 57.
达林AE, Mau B, Perna NT. progressiveauve:与基因获得、丢失和重排的多基因组比对。PLoS ONE。5 (6): e0011147。2010;
- 58.
埃德加钢筋混凝土。肌肉:一种减少时间和空间复杂度的多序列比对方法。BMC Bioinform。2004;5(1):113。
- 59.
Talavera G,Castresana J.从蛋白质序列比对去除发散和模棱两可的嵌段后改善文学发生。系统中的生物学。2007; 56(4):564-77。
- 60。
基于最大可能性的系统发育分析与数千种分类群和混合模型。生物信息学。2006;22(21):2688 - 90。
- 61。
Ronquist F, Teslenko M, Mark P, Ayres DL, Darling A, Hohna S. MrBayes 3.2:跨大模型空间的有效贝叶斯系统发育推断和模型选择。系统医学杂志。2012;61(3):539 - 42。
- 62。
partionfinder 2:为分子和形态系统发育分析选择进化分区模型的新方法。生物医学进展。2017;34(3):772-3。
- 63。
杨志。PAML 4:最大似然法的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。
- 64。
RStudio团队。RStudio:马萨诸塞州R. Boston的集成开发环境;2015.可以从:http://www.rstudio.com/.
致谢
感谢饶文辉先生、黄洁先生和吴欣怡小姐的样本采集。
资金
基金资助:国家自然科学基金资助项目(No. 5130471);U1804117);国家重点研发计划项目(Nos. 2019YFD1000400, 2018YFD1000401和2019YFD1001000);河南省重点科研项目(17A180023);资助方没有参与研究的设计、数据分析和手稿的撰写。
作者信息
从属关系
贡献
YYG, ZJL是本研究的构思和设计。YYG、JXY、MZB、GQZ分析数据。YYG写了这篇论文。所有作者都已阅读并批准了最终稿。
相应的作者
道德宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
叶绿体基因组结构Paphiopedilum.一)Paphiopedilum charlesworthii, b)埃默森P.c)p . fairrieanum,及(d)P.越南语.图S2。发现了12种SSC类型Paphiopedilum.图S3。系统发育树(ml)Paphiopedilum基于整个质体。分支上面的数字是bootstrap值≥70和贝叶斯后验概率≥0.90。粗体的分支是四种不稳定种。图S4。系统发育树(ml)Paphiopedilum基于排除了四种不稳定物种的整个质体。分支上面的数字是bootstrap值≥70和贝叶斯后验概率≥0.90。
附加文件2:表S1。
本研究中包含的录取详情。表S2。在叶绿体基因组中鉴定的基因列表Paphiopedilum.表S3。基因损失和伪原Paphiopedilum.表S4。每个基因的非同义替代率(dN)、同义替代率(dS)和dN/dS。表S5。本研究中使用的代际间隔区域列表。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
郭,YY。,Yang, JX., Bai, MZ.等等。金星拖鞋叶绿体基因组的进化(Paphiopedilum):IR扩展,SSC收缩和高度重新排列的SSC区域。植物生物学21,248(2021)。https://doi.org/10.1186/s12870-021-03053-y
收到了:
接受:
发表:
关键词
- 兰科西
- Paphiopedilum
- Phylogenomics
- 塑料
- 边界班
- IR.
- SSC边界
- 基因丢失
- 伪原化学