抽象的
背景
理解小麦籽粒中自由天冬酰胺浓度的决定因素是必要的,以减少烘焙和烤的小麦产品中加工污染物丙烯酰胺的水平。虽然作物管理策略有助于减少天冬酰胺浓度,但育种者有限的选择选择该特征的遗传变异。天冬酰胺合成酶酶在植物中催化浅析中的临界步骤,并且在小麦中由具有不同表达谱的五种职业基因三合组编码。在这个家庭内,TaASN2基因在谷物发展期间高度表达,但是TaASN-B2型在某些品种中缺失。
结果
天冬酰胺合成酶基因家族的自然遗传多样性在不同的小麦品种中进行了评估,揭示了存在/不存在变异和其他多态性的实例,包括一些预测会影响编码蛋白功能的实例。存在与否TaASN-B2型在一系列英国和全球常见的小麦品种和相关物种中判定,表明包含这种基因的缺失已经存在于一些野生emmer小麦基因型中。表达分析证实了这一点TaASN2转录本只能在谷物中检测到,而Taasn3.1.在谷物发育的早期阶段高度表达基因。TaASN-A2型是最表达的TaASN2家庭在大多数测定的小麦品种。TaASN-B2型和TaASN-D2型以相似的,较低的品种具有相似的较低水平表达TaASN-B2型.表达式TaASN-A2型和TaASN-D2型没有增加弥补缺席TaASN-B2,总计TaASN2缺乏基因的品种表达量较低TaASN-B2型.因此,平均缺乏的野外产生的谷物中的自由天冬酰胺浓度较低TaASN-B2型虽然当硫缺乏导致的自由天冬酰胺累积到非常高浓度时,效果丢失。
结论
小麦基因型缺失的筛选TaASN-B2型基因可能是降低商品小麦籽粒中游离天门冬酰胺浓度的一种简单、快速的方法。
背景
天冬酰胺在其自由(可溶性非蛋白质)形式中是植物中重要的氮传输和储存分子(见[1]审查)。它也在非生物和生物胁迫中积累,并在氨解毒和活性氧/一氧化二氮的产生中具有潜在的作用(见[2]审查)。然而,游离的天冬酰胺也是丙烯酰胺(C3.H5.NO),是一种致癌污染物,在谷物、块茎、豆类和贮藏根的油炸、烘烤、烘烤和高温加工过程中形成(见[3.]). 丙烯酰胺在美国(USA)被列为极度危险物质,在欧盟(EU)被列为具有急性毒性的严重健康危害物质,在国际癌症研究机构(International Agency for Research on Cancer)被列为2A类致癌物(可能对人类致癌)[4.].
欧盟委员会领导了开发食品中丙烯酰胺水平的监管体系(见[3.]有关全面的审查)。目前欧盟在食品中对丙烯酰胺的调节(委员会法规(欧盟)2017/2158 [5.])指出,食物中的丙烯酰胺可能会增加所有年龄群体中消费者发展癌症的风险。它还为不同食品类型中的丙烯酰胺设定基准水平,并在将来设定最大水平的明确威胁(即,上述水平是非法销售产品的水平)。
在美国,联邦政府尚未引入相当的规定,尽管食品和药物管理局(FDA)发出了丙烯酰胺的行动计划[6.].但是,只要2005年,加利福尼亚州的司法部长向五家食品公司提出了一个诉讼,其中四个餐馆链,未能用警告标记他们的产品,以提醒消费者存在丙烯酰胺([3.]). 加州也有人因没有发出警告通知而对咖啡业提起私人诉讼。在其他国家已经采取了饮食丙烯酰胺的监管机构包括卫生加拿大、食品标准澳大利亚新西兰(FSANZ)和日本和香港当局(审查)。3.]). 同时,最近的一项研究发现了丙烯酰胺及其代谢物缩水甘油酰胺(glycidamide)的一个独特的突变“特征”,在多种人类肿瘤类型中都有发现[7.].这代表了最强烈的证据尚未在人类膳食丙烯酰胺摄入和癌症之间的联系。
降低作物原料中的游离天冬酰胺浓度将大大有助于食品工业遵守有关食品中丙烯酰胺的规定。在小麦中,在缺硫土壤中生长的植物或受病原体感染的植物中,籽粒中的游离天冬酰胺浓度通常较高。因此,作物管理策略,包括确保小麦在种植期间有足够的硫供应[8.],并免受病原体感染的保护[9.那10],是减少自由天冬酰胺浓度的最常见的策略。尽管如此,小麦育种者受到食品企业的压力,以开发品种,在谷物中减少自由芦笋浓度。虽然自由天冬酰胺浓度确实在基因型中变化,但具有相当高的遗传性[11那12那13],作物管理和其他环境(e)因子的巨大效果,既有se,也与遗传因子(g×e)组合,意味着低附加旁浓度的育种不会是一个简单的任务(在[3.]). 通过关联作图,已鉴定出天冬酰胺含量的几个数量性状位点(QTL),但研究间尚未发现共同的QTL[12那13].
通过从谷氨酰胺转移到天冬氨酸的氨基通过将氨基转移合成,以在通过天冬酰胺合成酶催化的反应中制备天冬酰胺和谷氨酸。谷物天冬酰胺合成酶基因系列包括每二倍体基因组的两种和五个基因[14]小麦科的成员每个基因组都有5个基因,分为4组:1、2、3(又分为3.1和3.2)和4(小麦L基因组AABBDD)和四倍体二粒小麦(T. Turgidum.;基因组AABB)基因型在B基因组中缺乏第2组基因(TaASN-B2型/TdASN-B2型) [14那15].来自普通小麦地兰春季的IWGSC Refseq V1.1基因组组装,缺乏该基因,但存在于品种的Cadenza [14]. 在四倍体小麦中,ASN-B2型在野生emmer小麦中缺席(T. Turgidum.ssp。dicoccoides.)基因型Zavitan,但存在于驯养的硬质小麦品种Svevo(T. Turgidum.l . ssp。硬质(Desf.)Husn.)[14].由于野生emmer小麦被认为是四倍体硬粒和六倍唾液常见小麦的B基因组供体[16],最可能的解释是ASN-B2型在一些品种中,产生六倍体小麦的杂交事件不止发生一次,涉及二粒小麦带和不带二粒小麦ASN-B2型[14].这将是一致的,所述证据来自种族数据的更广泛分析中发现的多种杂交[17].
存在/不存在的程度ASN-B2型在不同的基因型中特别有趣,因为TaASN2基因是谷类中天冬酰胺合成酶家族中表达最高的成员[18那19]并且它们在胚胎中的表达可能是确定整个谷物中的自由天冬酰胺浓度的关键因素[19].小麦植物携带CRISPR / CAS9在所有六个中诱导编辑TaASN2等位基因显示出其谷粒中游离天门冬酰胺浓度大大降低[20].这是自然的ASN-B2型删除可以代表小麦育种者的有价值的遗传变异,以利用谷物中的自由天冬酰胺含量。因此,在本研究中,特征在于小麦中天冬酰胺合成酶基因家族的自然遗传变异。存在/不存在ASN-B2型被筛选在英国和全球常见小麦品种的小组中,以及小麦祖母基因组和野生物种。删除TaASN-B2型是否与整体减少有关TaASN2转录物水平和谷物天冬酰胺浓度,并且可以是小麦育种计划的有用等位基因,以开发具有较低浓度的自由芦酰胺的品种。
结果
小麦芦笋合成酶基因家族的自然多样性
全长编码序列ASN公司以中国春小麦地方品种的基因为检索材料,对14个普通小麦品种和拼音小麦的基因组进行BLASTn搜索(T. Aestivum.ssp。spelta.)表征小麦天冬酰胺合成酶基因家族的自然等位基因变异。结果显示在表格中1,按基因名称排序[14那15]和中国Spring RefSeq v1.1基因组组装中相应注释的基因模型ID [21].对于每个正交基因,在翻译的蛋白质上进行分类免受耐受性(SIFT)分析的分类,以预测氨基酸序列的变化是否可能破坏蛋白质功能(在表中以黄色突出显示1)或被容忍(以绿色突出显示)。所有小麦品种的特定氨基酸变化的全部细节在附加文件中提供1,表S1。
几个小麦有缺失,多态性和存在/缺席变化ASN公司基因。例如,TaASN-B1型在SY-Mattis中被删除,而其他8个品种携带一个等位基因变体,基因型为16 外显子7的bp缺失,引入了一个移码,并将一个过早的终止密码子带入了框中。这种缺失的存在意味着该基因被预测编码一种375个氨基酸的蛋白质,其C端截短209个氨基酸,包括天冬酰胺合成酶结构域的一部分,表明该蛋白质可能是无功能的(补充文件1,图S1)。TaASN-B3.2在Norin 61,CDC Stanley和Lancer中被删除TAASN-A3.2.和TaASN-D3.2存在于所有分析的品种中,并显示出预测蛋白质功能的多态性(表1).相比之下,有12个小麦品种携带TAASN-A3.1.预测多态性的等位基因扰乱蛋白质功能(表1).一些品种携带预测的等位基因组合,以破坏多个天冬酰胺合成酶蛋白的功能。例如,CDC斯坦利携带预测的等位基因影响由酶编码的酶的功能TaASN-A1型那TaASN-B1型和TAASN-A3.1.,除了删除TaASN-B3.2,而SY Mattis携带TaASN-B1型和TaASN-B2型的破坏性等位基因TAASN-A3.1.和TaASN-D2型(表1).最常见的存在/不存在变异是TaASN-B2型,其中在其中八个基因型中删除,其中包括中国春天(表1).
描述的TaASN-B2型删除
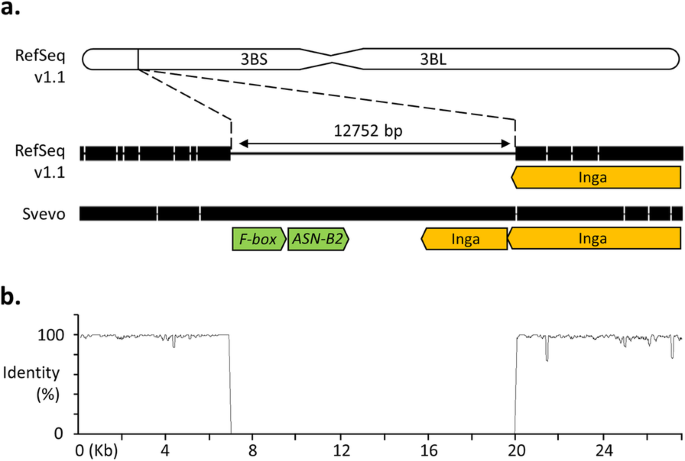
删除包含TaASN-B2型映射到中国春天Refseq V1.1基因组组件中的染色体臂3bs(图。1a).将已注释的Chinese Spring基因组与Svevo和Jagger基因组对应区域进行周边比对,Svevo和Jagger基因组均含有theASN-B2型基因,评估该基因座的其他特征(图。1A和B)。中国春天的缺失是关于静脉基因组和12,770bp相对于蛇形基因组的12,752bp。预测预测以编码F盒蛋白的推定的开放阅读框被检测到上游TaASN-B2型在删除区域(图。1一种)。在中国春天的删除下游和Svevo和Jagger的相应区域有一个大型,长的终端重复(LTR)Retrotransposon,inga.,属于Ty1-Copia家庭[22](图。1a),使用TREP数据库确认的身份[23].分析表中所示的基因型的其他基因组组装1透露,所有八种品种缺乏TaASN-B2型有相同的断点。
更广泛地筛选存在/不存在ASN-B2型
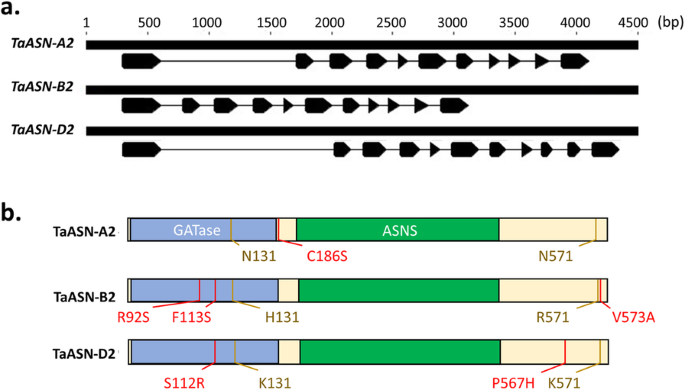
由于它们在测定小麦籽粒中游离天冬酰胺浓度方面的潜在作用,因此ASN2型在更广泛的小麦种质中探索了基因。三者的比较TaASN2Cadenza Genome的自主体显示它们共享常见的基因结构,每个基因结构含有11个外显子(图。2a) 是的。编码的蛋白质共享> 氨基酸水平上99%的同一性,同源异型基因之间只有8个多态性残基(图。2b) 是的。尽管这些多态性中有四个位于谷氨酰胺氨基转移酶(GATase)结构域(图。2b),预计没有根据SIFT分析影响蛋白质功能(表1).
的构造特征TaASN2来自凯兹达的基因和蛋白质。一种.外显子/内含子结构的图解表示TaASN-A2型那TaASN-B2型和TaASN-D2型同源异型[15].打开箭头表示外显子和线表示内含子。B..表示每个蛋白质的结构和相似性的图表TaASN2自制性,显示谷氨酰胺酰胺转移酶(Gatase)结构域(氨基酸约2-185)和天冬酰胺合成酶(Asns)结构域(氨基酸约210-450)。所有三种自主论不同的残留物在黄色中突出显示,而单个自制不同的残留物在红色中突出显示
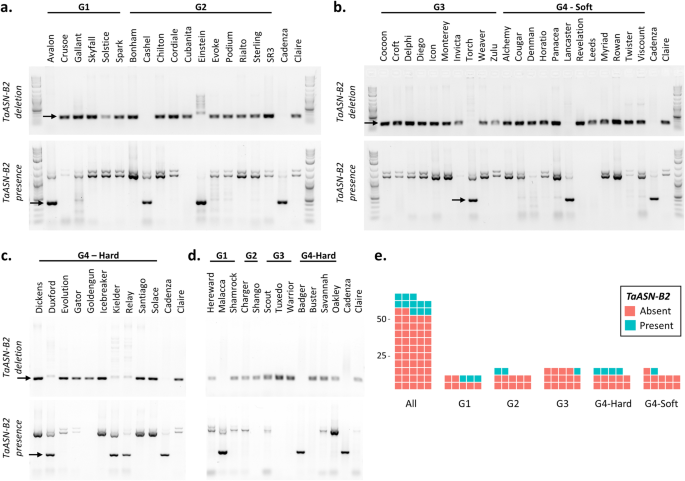
内含子1的长度在自制之间变化,是1104 bpTaASN-A2型, 1411 bp输入TaASN-D2型但只有175 bpTaASN-B2型(图。2a).设计一对冗余引物,从所有三个同源物的第一个内含子中扩增一个DNA片段,使反应产物根据大小易于区分,并检测是否存在TaASN-B2型.设计了第二对自制的引物,设计成含有缺失区域的上游和下游的含量TaASN-B2型只在携带这种缺失的基因型中扩增DNA片段。的存在TaASN-B2型因此,用第一引物对扩增了一个434 bp的产物,而用第二引物对扩增没有成功。分析结果如图所示。3.a-d,并在附加文件中总结1,表S2a。总的来说,TaASN-B2型被删除在63个冬季小麦品种中的52个(82.5%)(图。3.e)。缺失在饼干(G3)类(93.3%)中最常见(93.3%),在面包制作(G1)(70%)类中最不常见(图。3.e) 是的。另外一组24个全球小麦品种使用类似的PCR分析(附加文件)1,图S2a),结果见附加文件1,图S2b,并在附加文件中总结1,表S2b。这个TaASN-B2型在这些小麦中缺失不太常见于英国品种,仅在50%的基因型中存在(附加档案1、表开通。
存在/不存在TaASN-B2型在英国小麦品种。模拟:电泳凝胶PCR产物从分析中区分是否存在TaASN-B2型在两年内长大的英国小麦品种的集合中[11].一种。英国面粉厂1和2组硬面包小麦(2012-2013年种植)。湾英国面粉厂第3组和第4组软麦(2012-2013年种植)。c。英国面粉米勒集团4个硬母(在2012-2013年生长)。d。剩余的品种仅在2011 - 2012年增长。品种Cadenza和Claire被用作控制TaASN-B2型分别是在场和不在场。区别PCR产物用箭头表示。e。显示频率的图TaASN-B2型63个英国小麦品种的缺失,分为英国面粉加工组:G1(面包加工)、G2(面包加工潜力)、G3(软/饼干)、G4(饲料/其他)
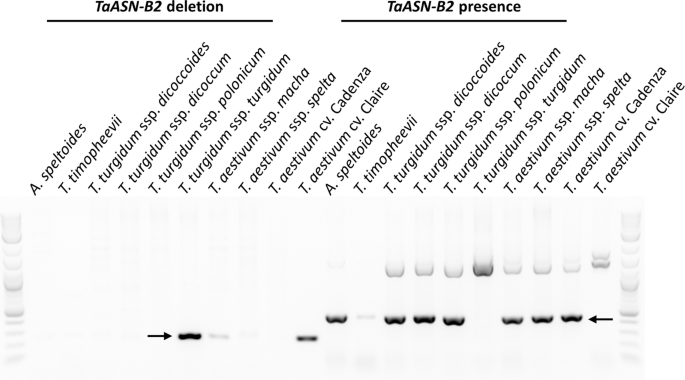
此外,还对其他小麦品种进行了筛选,以确定是否存在ASN-B2型基因(图。4.).一个ASN2型基因是在山羊草属speltoides(基因组BB);然而,虽然ASN-B2型该基因在一些四倍体小麦基因型(基因组AABB)中存在,在其他四倍体小麦基因型中缺失。4.).面食小麦(T. Turgidum.ssp。杜伦姆)在研究中检测的品种,Svevo和Kronos,显示有一个ASN-B2型基因,波兰小麦(T. Turgidum.ssp。polonicum)但在铆钉小麦中该基因缺失(T. Turgidum.ssp。Turgidum.). makha小麦的结果有些模糊(T马查)在存在的情况下,存在明显的积极结果ASN-B2型基因,但在测定中扩增的微弱条带进行缺失(图。4.). 重复实验时,该条带仍然存在(数据未显示),可能是由于样本中的遗传异质性。
小麦表达谱ASN公司发育过程中的基因
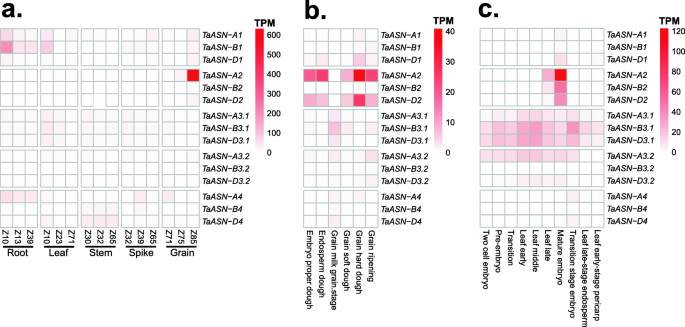
从公共RNA-SEQ数据集映射到IWGSC REFSEQ V1.1基因组组件的原始测序读取,以全面概述每个表达概况Taasn.基因。每个数据集的表达式值在附加文件中提供2作为每百万(TPM)值的平均转录物。在包含根,叶,茎,穗和谷物的数据集中,每个都在三个发展阶段进行采样[24],TaASN1转录水平在年轻根和叶子中是最高的,而这三个自主Taasn3.1.和taasn3.2.显示了更广泛的表达谱,在不同发育阶段的所有分析组织类型中检测到转录物(图。5.一种)。TaASN4同源语也被广泛表达,用TaASN-A4型转录物水平最高的根和尖峰组织,以及TaASN-B4型和TaASN-D4型在茎发动期间更高度表达(图。5.一种)。如前所述[18那19],TaASN2表现出谷物特异性表达谱,转录水平在Zadoks阶段85(Z85)最高,对应于软面团阶段[27](图。5.a) 是的。此外,TaASN-A2型占83.3%Taasn.Z85的晶粒组织的转录物,而TaASN-D2型仅贡献3.0%,与以前的结果一致[19].
进一步探索表达Taasn.以籽粒中的基因为例,分析了Azhurnaya品种籽粒发育6个阶段的表达数据集[25]. 如期而至[18那19],高记录水平TaASN-A2型和TaASN-D2型在五个发育阶段中发现,这些阶段加起来,占所有发育阶段的69%到86%Taasn.这些组织中的转录物(图。5.b) 是的。例外的是谷物-牛奶阶段Taasn3.1.转录水平高于TaASN2(图。5.b),如前所述[18].TaASN-B2型在该数据集中的可忽略水平下检测到转录物,表明其在Azhurnaya基因组中的缺失。
普通小麦品种AC-Barrie胚发育过程的表达数据分析[28]揭示了这一点TaASN2成熟胚胎阶段的转录物水平最高(图。5.C)。之中TaASN2homeologues,TaASN-A2型再次是最表达的基因,而TaASN-B2型和TaASN-D2型在类似水平表达(图。5.然而,c)。TaASN2在所有其他发育时间点,包括胚胎发育的早期阶段和胚乳和果皮组织中,转录本仅在可忽略的水平上检测到Taasn3.1.转录本更丰富(图。5.c) 是的。综上所述,这些数据证实了TaASN2在籽粒组织和成熟胚中,表现出更广泛的作用Taasn3.1.基因开发,包括胚胎发育的早期阶段。
变种间的变化Taasn.粮食开发期间的表达概况
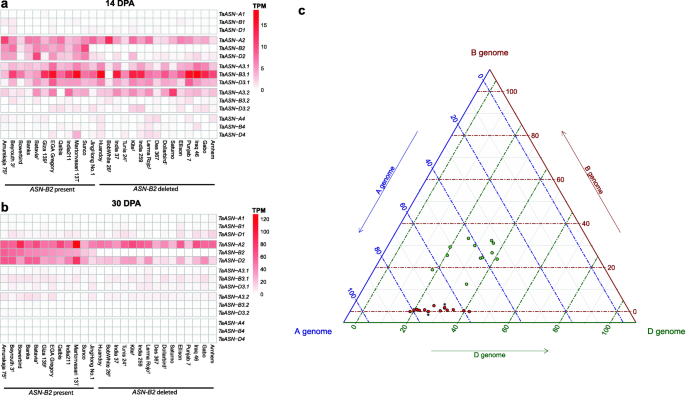
分析变异ASN公司从小麦花后14天(DPA)的籽粒样本和27个世界小麦品种的30个DPA中,绘制了小麦籽粒转录本水平和RNA-seq reads [28].在14 dpa,总计Taasn3.1.转录水平范围为2至32分钟,大于TaASN2在测定27个品种的22个中(图。6.a) ,与之前的结果一致[19]. 在30 DPA时,TaASN2自主体是所有品种中最高表达的斯坦氨酰胺合成酶基因(图。6.b) 是的。在后一个时间点,总计TaASN2转录水平显示基因型之间有很大的差异,从28到242个TPM(额外文件2).有几条线显得很低TaASN-B2型转录水平和该基因的缺失在其中5个株系中得到了PCR检测的证实(附加文件)1,图。S2B;附加文件1、表开通。还有易于检测的线条TaASN-B2型转录物(>5个TPM),其中4个株系证实了该基因的存在1,图。S2B;附加文件1、表开通。
表达式Taasn.来自RNA-SEQ映射数据的27个全球小麦品种的基因[28].一种。颗粒组织14dpa。湾晶粒组织30dpa。†表示存在或不存在的品种TaASN-B2型通过PCR确认c。三元图说明A,B和D基因组的相对贡献TaASN2家庭到总计TaASN230 DPA时各品种籽粒组织中基因的表达。品种TaASN-B2型目前用绿圆形表示,品种TaASN-B2型删除用红色圆圈表示
一个三元图,显示每个同源异型对整体的相对贡献TaASN2不同小麦品种在30dpa时籽粒中的转录水平(图。6.c) 他说TaASN-A2型转录水平通常大于TaASN-D2型在很低的品种TaASN-B2型转录水平(可能与这些品种中该基因的缺失有关)。相比之下,在品种上相对较高TaASN-B2型表达,TaASN-A2型和TaASN-D2型通常更均匀地表达,并在四个品种中,TaASN-D2型转录水平高于TaASN-A2型(图。6.c) 是的。然而,总体TPM值TaASN-A2型和TaASN-D2型在缺乏营养的品种中不高TaASN-B2型与那些相比TaASN-B2型存在(附加文件2),因此没有证据表明这些基因表达增加,以弥补缺乏TaASN-B2型抄本。
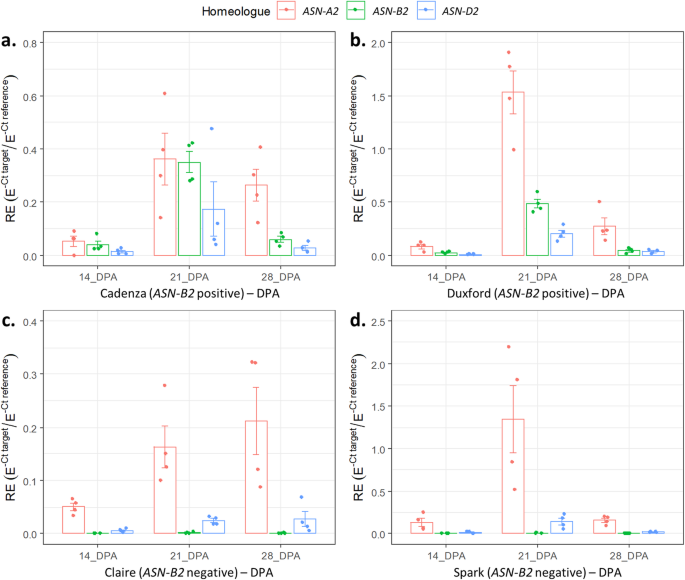
为了进一步研究表达式的动态性,提出了表达式TaASN2利用RT-qPCR对两个小麦品种的同源基因进行了分析TaASN-B2型(华彩乐章和杜克斯福德)和两个缺乏它的变体(火花和克莱尔)(图。7.).该实验的差异分析结果显示在附加文件中1,表S3a,显示显著影响(P < 0.001) of variety, timepoint, and homeologue, and the interactions between these factors, on relative expression levels. In Cadenza and Duxford, meanTaASN-A2型在所有时间点(14,21和28 DPA)的三个同源异型基因中,表达最高,而TaASN-D2型表达最低(图。7.A和B)。在两个品种中,意味着TaASN-B2型表达式大于TaASN-D2型在所有时间点和Cadenza在21dpa的表达,匹配水平TaASN-A2型表达式(图。7.一种)。在克莱尔和火花,意思TaASN-A2型表达式大于TaASN-D2型在所有样本中的表达,并显示对Cadenza和Duxford的时间点相似的表达动态(图。7.C和D)。值得注意的是,没有证据表明更高的表现TaASN-A2型或TaASN-D2型与Cadenza和Duxford相比,在克莱尔和火花中。
的贡献TaASN-B2型释放谷粒中天冬酰胺的浓度
对存在或不存在的品种进行筛选TaASN-B2型(图。3.,附加文件1,附加文件1,表S2)包括63个英国品种,在两个生长季节(2011-2012年和2012-2013年)在英国进行的田间试验中测定了谷物中的游离天冬酰胺浓度[11].这意味着评估可以由效果制成TaASN-B2型在谷物中自由芦笋浓度删除。在现场试验中的63个品种中,有11个拥有TaASN-B2型而52个没有。
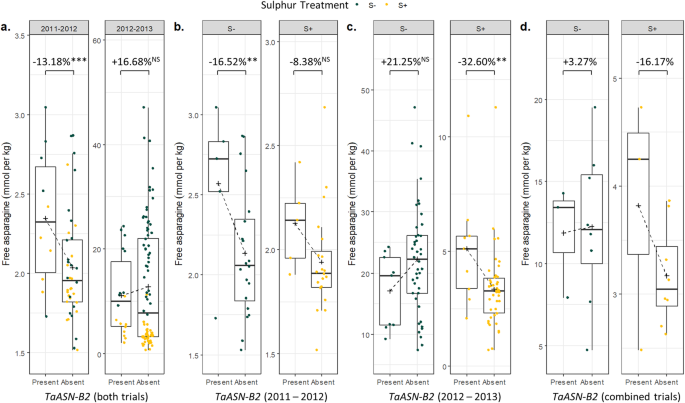
来自这些田间试验的谷物已经在图中产生,其中硫磺被提供或扣留[11].我们分析了效果TaASN-B2型与ANOVA的这些试验中的其他变量一起(附加文件1,表S3B),揭示了重要的(P < 0.001) effect of theTaASN-B2型在2011-2012现场试验中删除:品种没有TaASN-B2型相对于那些有13.18%的免费天冬酰胺TaASN-B2型(图。8.a) 是的。无明显影响(P > 0.05) of the deletion by itself in the 2012–2013 field trial, but there was a significant (P < 0.001)相互作用TaASN-B2型有/无和硫处理:无显著差异(P> 0.05)TaASN-B2型缺硫下,但品种无硫TaASN-B2型含有32.60%的自由芦笋(P < 0.01)TaASN-B2型(图。8.C)。
不同品种小麦籽粒中游离天冬酰胺的含量TaASN-B2型在2011-2012年和2012-2013年的两个实地试验中,基因要么存在要么不存在,显示了TaASN-B2型缺失在不同的硫处理(S-和S +)和基因存在/不存在之间的相互作用和治疗。(a)从2011-2012和2012-2013的反转换数据与S-+ +治疗中的浓度的试验绘制在一起。(b)从2011-2012赛季的后转换数据,浓度从S-an +治疗中分开绘制。(c)来自2012-2013季的后转换数据,S-+ S +治疗浓度分别绘制。(d)从两种试验中生长的11个品种的REML分析中的反变化预测手段。十字架表示每组的平均值。盒子展示了四分位数的范围和中位数。晶须分别显示在第75次和第25百分位数的四分位数范围的1.5倍以上的最小和最大值。主要效果的重要评分TaASN-B2型在(a)中取自单独的ANOVA分析在日志上进行E转换数据。在ANOVA分析后进行多次与Bonferroni校正进行比较TaASN-B2型2011-2012 (b)和2012-2013 (c)组比较P> 0.05, ** =P < 0.01, *** = P < 0.001
基于完整组合数据集的REML分析也识别出显着的效果TaASN-B2型以及相互作用TaASN-B2型和硫磺治疗(附加文件1,表S3B)。该分析还进一步证明了之间的相互作用TaASN-B2型硫处理逐年变化,如图所示。8.B和C,并且在年份和硫磺之间存在显着的相互作用。对两种试验共同的11个品种的预测与分析综合分析(总共分析的63种)表明了TaASN-B2型删除效应在不同生长季节的硫充足下最大伟大(图。8.d) 是的。尽管两个试验之间硫缺乏的影响差异很大,因此很难就硫缺乏的影响得出明确的结论TaASN-B2型基于REML分析的预测手段,在硫缺陷下。
讨论
自然删除ASN-B2型在小麦
在这项研究中,利用小麦基因组资源来表征包括完整的自然缺失TaASN-B2型基因和设计分子测定以追踪其在多种小麦种质中的频率。在分析的所有八个常见的小麦基因组组合中,这种删除事件的断点是相同的,强烈暗示这种等位基因的单一,共同来源。删除的区域紧邻TY1-Copia转座因子(无花果。1a)是小麦基因组中最丰富的LTR回收碳扫一类之一[29],是一种可能导致这种缺失的原因。这ASN-B2型基因在B基因组中完整AE。spottoides.(基因组BB)(图。4.),该基因组与驯化小麦品种的B基因组前体有关。然而,二粒小麦杂交事件的直接始祖(基因组AABB)尚未确定[16,所以坚定的结论的起源ASN-B2型不能绘制二倍体小麦祖细胞的删除。一些野生和驯养的四倍体小麦携带完整ASN-B2型基因,而在其他人是不存在的,这,正如其他地方所讨论的[14那17],建议在小麦进化史上发生独立的杂交事件。在更多样化的小麦种质收集中研究该基因座的单倍型变异,包括来自肥沃的新月内的不同群体的更野生和驯养的emmer小麦[16)的研究,可以帮助我们了解ASN-B2型删除。
在不同类别的英国冬小麦品种中,这种缺失现象出现的频率很高(图1)。3.e) 是的。该等位基因的不平衡分布可能是由于在英国小麦品种发育的早期选择中缺乏遗传多样性造成的,也可能是本研究分析的品种选择的产物,或者只是偶然发生的,表明该等位基因处于中性选择之下。然而,由于对植物适宜性或表现的积极影响,该等位基因也可能受到直接或间接选择的影响。如果是这样的话,它不太可能被选中,因为它与当前研究中描述的谷物中的游离天冬酰胺含量有关,因为基于天冬酰胺含量的选择是最近才做出的,如果有的话。基于小麦籽粒特异表达谱TaASN-B2型,其他可能解释缺失的积极选择的性状是收获前发芽抗性或其他一些研究显示与天冬酰胺浓度相关的品质性状[30].然而,其他研究没有发现天冬酰胺含量和许多烘焙品质性状之间的联系[12],所以这种关联需要持续的分析。进一步的可能性是,由于链接不平衡与该基因座的有益遗传变异,已经间接选择该缺失。在该地区其他农艺性状的标记特征联盟的证据将支持这一假设,尽管难以直接测试。更详细的功能表征ASN2型专注于谷物发育和品质性状的基因可能也揭示了这个基因的额外的,以前未被确认的作用;类似于酵母和哺乳动物细胞中天冬酰胺合成酶的兼职作用[31那32].
小麦天冬酰胺合成酶基因的表达谱
我们的表达数据确认了之前的研究结果[18那19]那种品种,TaASN-A2型是最表达的TaASN2谷粒发育中的同源物(图。5.那6.和7.).在12个品种中的五分之一TaASN-B2型已经被RNA-SEQ测定,TaASN-B2型贡献了更高比例的ASN2型比成绩单比TaASN-D2型,鉴于TaASN-D2型其他七个品种的表达量较大(图。6.c、 附加文件2).更高TaASN-B2型表达式结束TaASN-D2型在RT-QPCR的品种Cadenza和Duxford中也观察到,在整个取样时间过程中,从14〜28 dPa(图。7.).在每种缺乏TaASN-B2型那TaASN-A2型贡献的比例较大ASN2型比成绩单比TaASN-D2型(图。6.C)。相比之下,在品种中TaASN-B2型目前这三个同源异型基因的表达更为均匀,尽管TaASN-A2型仍然贡献了最大的比例ASN2型在大多数行抄本(图。6.然而,c)。B.一种sed on our combined RNA-seq and RT-qPCR data, the loss ofTaASN-B2型与整体减少相关ASN2型转录水平和没有代偿性上调的A或D同源异型,这是一致的影响,这种删除对粮食天冬酰胺浓度。这与其他生物合成途径形成对比,例如GA信号[33[其中反馈机制调节生物合成基因的转录水平,以补偿表达损失。
我们的表达分析还提供了额外的洞察潜在的作用Taasn3.1.在胚和籽粒发育过程中。在籽粒发育的早期,该基因的转录水平最为丰富,而在籽粒发育的早期,该基因的转录水平最为丰富TaASN2在晶粒发育后来占主导地位的转录物(图。5.和6.). 在大多数(但不是所有)品种中Taasn3.1.仍高于TaASN2甚至14 dpa,与前一项研究相比TaASN2被发现是胚胎中最高表达的天冬酰胺合成酶基因和在该发育阶段的常见小麦品种火花和加倍的单倍体线SR3中的胚乳和胚乳18那19].这很可能是由生长条件和发育速度的差异来解释的;事实上,即使在相同的生长条件下,Spark和SR3之间的发育速度和天冬酰胺合成酶基因表达也存在差异[19].因此,表征功能是有趣的Taasn3.1.在小麦中,有助于了解该基因在谷物中促成天冬酰胺生物合成的程度,以及其对粮食发育的重要性。该基因的等位基因变异也可以有助于减少谷物中的自由天冬酰胺含量,并且可以通过表征潜在的破坏性自然的影响来测试这个假设TAASN-A3.1.一些品种携带的等位基因(表1)或通过靶向突变。这一方法也可用于表征其他小麦天冬酰胺合成酶基因的功能,以帮助评估将该家族的遗传变异整合到小麦育种计划中的可能性。
减少谷物天冬酰胺含量的育种
这TaASN-B2型缺失在2011-2012场试验中减少了谷物免芦酰胺浓度的13.18%(图。8.a)在2012-2013场试验中,硫磺充足的32.60%减少了(图。8.C),表明该变异可以有助于减少谷物游离天门冬酰胺浓度在田间条件。2011-2012年,缺硫对游离天门冬酰胺浓度的影响较为温和[11]很可能是为什么的效果TaASN-B2型在两种硫处理中都有。相比之下,在2012-2013年的试验中,缺硫导致游离天冬酰胺浓度大幅增加,导致硫处理和TaASN-B2型(附加文件1,表S3b),并证明当游离天冬酰胺水平较高时,在严重缺硫的情况下,缺失不会产生影响(图。8.C)。这与先前的研究一致,显示出强环境(e)以及对自由天冬酰胺含量的遗传(g)影响,以及复杂的g×e相互作用[3.]. 这意味着TaASN-B2型通过采用适当的作物管理策略来减少压力,可以最大化删除。
这种回顾性分析TaASN-B2型无谷粒天冬酰胺水平的缺失是有限的,因为试验中的品种最初不是根据有无谷粒天冬酰胺来选择的TaASN-B2型,因此,有更多的品种没有TaASN-B2型比分析中的那些人要多。因此,描述TaASN-B2型在额外的和更多样化的环境中删除了复制的现场试验,包括更广泛的特征,以证实没有与此等位基因相关的有害性抗性效应。为了最大限度地减少其他遗传变异的影响,可以通过在常见的遗传背景下进行这些试验,其通过在近似同学系中进行,或通过直接诱导诱变诱变变异。
直接测量谷物中的游离天冬酰胺浓度是昂贵的,需要专业的分析设备,而且对种植者来说往往是不切实际的,但是,使用我们的简单PCR筛选可以使小麦育种家排除在硫充足的条件下生长时更可能在籽粒中具有高游离天冬酰胺浓度的基因型(图。8.b) 是的。虽然影响是轻微的,但这有助于降低与膳食丙烯酰胺相关的公共健康风险,并帮助食品制造商遵守其产品中丙烯酰胺存在的困难和不断变化的法规。由于其自然起源,这种缺失可以在所有调控环境中得到充分利用,就像其他品质性状在不同作物品种中得到利用一样。例如,由于镉转运蛋白上游的转座因子的自然插入,一些大麦基因型具有减少的镉积累,并且携带这种插入的等位基因可以不受限制地使用[34].
尽管这种特殊的变异对英国小麦育种家的影响可能有限,因为它在英国冬小麦品种中的频率很高(图。3.E,附加文件1,表S2A),可能有更多机会在世界其他地区应用此等位基因,因为只有50%的小麦品种来自各种全球品种的缺失(附加档案1、表开通。该小组包括来自澳大利亚、非洲和欧洲的小麦品种,因此在更广泛的小麦种质中探索这种缺失的频率是值得的。虽然目前的研究只包括两个硬粒小麦品种,但两者都携带ASN-B2型基因,可能表明历史杂交事件可能包括杜兰姆小麦的事件ASN-B2型频率高于常见小麦的基因。因此,杜兰姆小麦育种者可能通过鉴定和选择携带这种缺失的基因型来减少谷物中的自由天冬酰胺浓度。此外,因为杜兰姆小麦基因组是四倍体,所以那些缺乏的那些品种ASN-B2型自主体可能表现出比在六倍丙二醇中的颗粒常见小麦中的比例更大减少。杜兰姆小麦的主要用途是用于面食生产,虽然丙烯酰胺存在于面食中,但它处于相对较低的水平[35]. 然而,硬粒小麦谷物也被加入到谷物中,用于制造丙烯酰胺含量可能更高的产品,如比萨饼底料、皮塔面包和其他扁平面包。
虽然我们只检测到两种不同TaASN-D2型等位基因(一个只在品种SY MATTIS中发现),没有变化TaASN-A2型(表1),更加多样化的种质可能的更广泛的屏幕可能产生额外的天然变体,可以集成到育种程序中,以选择减少自由天冬酰胺浓度。以前的关联映射研究已鉴定QTL控制天冬酰胺含量[12那13]但是这些QTL没有映射到含有天冬酰胺合成酶基因的基因组的区域。然而,逆向遗传工具,例如EMS或CRISPR / CAS9诱导的诱变,提供了对小麦种质中不存在的工程师等位基因的潜力,包括由于多倍体的功能冗余而不太可能选择的隐性突变的组合基因组[36那37]. 最近的一项研究证明了这种方法的有效性,在这项研究中,通过编辑所有六种植物,发现游离天冬酰胺浓度降低了90%ASN2型使用CRISPR/Cas9的等位基因[20].此外,该基因的三种自主的存在允许选择可能允许育种者与其他谷物发育性状平衡减少自由天冬酰胺含量的等位基因敲除的组合。虽然这将是工程师的强大而快速的方法,并且表征潜在宝贵的遗传变异,但重要的是要注意当前在世界某些地区在植物育种中限制Crispr-Cas9在植物育种中的复杂和动态监管景观非常重要[38].
结论
对小麦天冬酰胺合成酶基因家族中自然等位基因变异的特征分析表明,缺失的基因不到13个 知识库包围TaASN-B2型这在英国冬小麦品种中出现的频率很高。在一些野生二粒小麦中也存在缺失现象,表明其起源于古代,并在驯化和现代育种过程中得以保留。在田间试验中,携带缺失的等位基因与游离天冬酰胺含量的降低有关,可以使用廉价的PCR方法来选择,以帮助育种家培育低天冬酰胺小麦品种。
方法
基因组分析
小麦的核苷酸序列数据ASN公司使用10+小麦基因组项目的喷炸工具获得来自不同小麦基因型的基因(https://weblast.ipk-gatersleben.de/wheat_ten_genomes/),基层基因组学项目(https://wheatis.tgac.ac.uk/grassroots-portal/blast) [39],以及Graingenes数据库(https://wheat.pw.usda.gov/cgi-bin/seqserve/blast_wheat.cgi) [40].一些ASN公司基因缺乏完整的序列信息,这些异常在附加文件中有描述1,表S4)。Geneous Prime 2020.1.2用于基因间的比对和序列同源性分析。意面小麦基因组的注释(T硬粒)品种svevo(https://www.interomics.eu/durum-wheat-genome) [41用于比较含的基因组区域TaASN-B2型与普通小麦的Refseq V1.1基因组中的相应区域(T. Aestivum.长白中国春(来自Ensembl植物)https://plants.ensembl.org/wheat.) [21].softberry-FGENESH工具[42用于鉴定包含12,770bp删除区域的推定基因TaASN-B2型来自德文队。该分析确定了五个推定的ORF。每个都用hmmscan分析[43]使用e值截止值0.05,确认存在TaASN-B2型和编码含有F盒PFAM结构域的蛋白质的第二基因(PF00646)。使用TREP中的BLAST工具(可转换元件平台)进行证实转座子注释(http://botserv2.uzh.ch/kelldata/trep-db/index.html.) [23]. 筛选分析[44通过将来自中国春季的每个ASN蛋白与来自其他小麦品种的正非基因编码的蛋白质进行比较来进行。为了TaASN-B2型, Jagger的蛋白质被用作参考。
植物材料与萌发
英国种子的种子是维持在Rothamsted的研究中或从这里研究的现场试验中产生的库存中获得[11].其他小麦品种是从USDA-ARS国家小谷物收集获得的(https://www.ars.usda.gov/)以及约翰·因内斯中心的种质资源部(www.seedstor.ac.uk.). 本研究中所有品种的名称在附加文件中列出1,表S2。种子在70%乙醇中孵育10分钟,进行种子表面灭菌 分钟,然后在20%(v/v)次氯酸钠溶液中60分钟 轻轻搅拌,以确保种子均匀灭菌。随后用无菌蒸馏水将种子洗涤四次,并在室温下使其在无菌培养基中在连续光照下萌发 mm湿滤纸上的皮氏培养皿,用Parafilm密封(Fisher Scientific Ltd.,拉夫伯勒,英国)。对于较老的种子,用箔纸包裹盘子,在4℃下孵化 °C 2-7天打破休眠,然后转入连续光照和室温下发芽。对于任何一种方法都不能发芽的种子,将胚胎解剖并放入90%的培养基中 含MS培养基的mm培养皿(4.4 g/L MS盐[45], 3%蔗糖(30 g/L), pH 5.8, 7 g/L琼脂)。然后这些培养皿用多聚膜密封,并在室温和连续光照下发芽。
DNA提取
根据制造商的说明书(南安普敦,英国南安普敦),使用Wizard®基因组DNA净化试剂盒从幼苗的叶片填充DNA。对于未使用上述方法进行发芽的种子,将胚胎从多种种子中沉积并一起研磨成用于通过CTAB方法进行DNA提取的细粉[46].使用NanoDrop™1000分光光度计(Thermo Fisher Scientific)检测DNA质量和丰度。
PCR测定以检测TaASN-B2型
同源异型引物ASN-B2-Deletion-F和ASN-B2-Deletion-R(附加文件1,表S5)设计用于对缺失位点的上游和下游进行退火,以便TaASN-B2型可以通过扩增232bp DNA片段来证明作为阳性结果。另一对引物扩增所有的第一内含子TaASN2自主,ASN-2-Universal-F和ASN-2-Universal-R(附加文件1,表S5),旨在测试和区分是否存在这三种情况TaASN2基于大小的同源序列。这些正向和反向引物扩增出大小为1363的DNA片段 英国石油公司,434 bp和1670 bp,对应于TaASN-A2型那TaASN-B2型和TaASN-D2型,分别。这两种引物组合用于验证存在或不存在ASN-B2。
这些引物用于25卷的PCR反应 μL使用1 × 梦幻塔克™ PCR主混合物(1.5 毫米氯化镁2)(Thermo Fisher Scientific,Epsom,英国),包括1μm的每个引物和50-150 ng基因组DNA。循环条件对于两个引物组是相同的:96°C的5分钟变性;32循环在96℃下的30°S变性,30℃在60.5℃下退火,1.5分钟在72℃下延伸;10分钟的最终延长72°C。通过电泳通过琼脂糖凝胶(1.0%w / v,溴化乙锭染色)的电泳分析反应,其中1kb加DNA梯(英国,英国),并在Geldoc成像系统(Biorad,USA)中使用紫外线可视化。
用一个单独的聚合酶链反应(PCR)来确认是否存在病毒ASN-B2型在一套24个全球小麦品种(附加档案1,图。S2)。该测定使用两组PCR中的引物,根据存在或不存在扩增不同的扩增子ASN-B2型. 所用引物为ASN-B2_qF1(P3)和ASN-B2_qR1(P4),扩增出125条DNA片段 带菌品种中的bpTaASN-B2型与ASN-B2泳CS泳F3(P1)和ASN-B2泳CS泳R1(P2)一起扩增189个DNA片段 缺糖品种的血压TaASN-B2型基因(附加文件)1,图S2)。PCR混合物共25份 微升,0.2 μM引物P1和P2以及0.24 μM引物P3和P4,1 × 标准Taq.缓冲液,250ng模板DNA和0.125μLTaq.聚合酶(新英格兰Biolabs,Ipswich,Ma,美国)。扩增使用以下条件进行:95℃,30秒;35循环:15℃,59℃,30秒,68°C为30秒;68°C 5分钟。通过用Sybr Safe(Apexbio,Houston,Tx,USA)染色的3%琼脂糖凝胶上的电泳分离扩增的DNA片段。每次反应预期189bp或125bp的单个扩增的DNA片段(附加文件1,图开通)。所有电泳凝胶的完整、未经裁剪的图像在附加文件中提供3..
RT定量PCR
两个普通小麦品种携带TaASN-B2型(Cadenza和Duxford)和两个缺乏这种基因的人(Claire和Spark)在一个温室里进行了随机区组设计。植物生长在单独的花盆中进行破坏性取样,每个时间点取四个重复。采用标准化的RNA提取方法,在花后14天、21天和28天三个时间点从胚组织中提取RNA[47].根据制造商的说明,使用ReliaPrep™RNA清理和浓缩系统(Promega)进一步清洗RNA。然后根据制造商的说明,使用RQ1 RNase-Free DNase (Promega)从这些样本中提取DNA,并使用NanoDrop™1000分光光度仪(Thermo Fisher Scientific)对RNA进行定量。
cDNA was synthesised by mixing 2 μg RNA in nuclease-free water with oligo-dT primer, dNTPs, and SuperScript™ III Reverse Transcriptase kit components (Invitrogen), according to the manufacturer’s instructions, and placing in a thermocycler using the following programme: 95 °C for 5 min; 60 °C for 60 min; 72 °C for 15 min.
RT-qPCR使用应用生物系统进行™ 7500实时PCR系统设置为ddCt(相对定量)模式。每个反应包含10个 μL SYBR绿色主混合物(应用生物系统),5 μL底漆母料(含0.04 在不含核酸酶的水中,每种引物和ROX参比染料的体积(mM)和5 μL cDNA(稀释至6 纳克/微升)。每个靶基因的表达相对于3个参考基因进行测定;GAPDH.那PROSM公司,SDH.. 所用引物的详细信息见附加文件1,表S5。
按照Rieu和Powers的描述计算相对表达值[48]. 使用Applied Biosystems 7500实时PCR软件2.0.5版计算Ct值,并使用LinRegPCR将导出的Rn数据转换为PCR效率数据[49].使用Genstat进行统计测试[50]为了解释实验的阻塞结构,图表绘制在r [51]使用包gppubr [52].
RNA序列数据分析
原始的RNA-SEQ从兰德斯中国春季的发展时间表中读取[24],来自多种Azhurnaya的谷物开发样本[25],来自各种AC Barrie的胚胎发育时间表[53]一组世界小麦品种在14dpa和30dpa的籽粒表达[28],从NCBI地理数据库下载(https://www.ncbi.nlm.nih.gov/geo/)按照前面描述的方法处理并映射到IWGSC RefSeq v1.1基因组[54].额外的contig对应于TaASN-B2型向参考基因组加入到蛋白质编码区上游和下游的编码序列和包括1kB序列以测定该基因的转录水平。使用自定义Python脚本将原始计数转换为TPM。使用GPLOTS包内的命令'HEATMAP'在R(v1.12.5019)中产生热量[55]. 三元图是使用ggtern包创建的[26在ggplot2中[56].TPM中的所有表达式数据在附加文件中显示2.
影响TaASN-B2型谷物中游离天冬酰胺的研究
2011-2012年和2012-2013年的现场试验数据[11]被用来调查TaASN-B2型用硫磺提供(S +)或扣留(S-)删除谷物天冬酰胺水平。如上所述,筛选来自该田间试验的品种,用于存在或不存在TaASN-B2型.数据日志E如前一项研究所述,转换为解释方差的异质性[11],在执行ANOVA和REML分析之前。分析和绘图在r [51]使用ggpubr包[52].在Genstat进行ANOVA和REML分析[50]来解释大田试验的裂区阻塞结构。
实地试验数据的重新分析包括识别存在/不存在的额外因素TaASN-B2型,随着品种嵌套的效果TaASN-B2型的因素。在个别年份进行的试验,是根据在[11]使用Anova,添加了TaASN-B2型的因素。通过使用REML算法,将两个试验合并的数据作为线性混合模型进行分析,考虑到两个试验中包含的不同设计结构和品种集,提供了存在/不存在的整体比较TaASN-B2型,以及两个试验中包含的品种之间的差异(允许对不同年份包含的品种进行比较)。
缩写
- 方差分析:
-
方差分析
- 爆炸:
-
基本的局部比对搜索工具
- CRISPR公司:
-
聚集的规则的短回文重复
- CTAB:
-
十六烷基三甲基溴化铵
- 分区:
-
黄花木后天数
- 特快专递:
-
甲烷磺酸乙酯
- 欧盟:
-
欧盟
- 食品和药物管理局:
-
食品药品监督管理局
- FSANZ:
-
澳大利亚食品标准机构和新西兰
- GA:
-
赤霉素
- 加酶:
-
谷氨酰胺amidotransferase
- IWGSC:
-
国际小麦基因组测序联盟
- kb:
-
千北面对
- 贷款利率:
-
长末端重复
- PCR:
-
聚合酶链反应
- QTL:
-
数量性状位点
- REML:
-
限制最大可能性
- TPM:
-
每百万成绩单
工具书类
- 1.
Lea PJ,Sodek L,Parry Ma,Shewry Pr,Halford Ng。芦笋在植物中。Ann Appl Biol。2007; 150(1):1-26。https://doi.org/10.1111/j.1744-7348.2006.00104.x.
- 2.
Oddy J、Raffan S、Wilkinson MD、Elmore JS、Halford NG。胁迫、养分和基因型:理解和管理小麦籽粒中天冬酰胺的积累。卡比农业生物园。2020;1(1):10.https://doi.org/10.1186/s43170-020-00010-x.
- 3.
Raffan S, Halford NG。食品中的丙烯酰胺:遗传和农艺解决方案的进展和前景。中国生物医学工程学报,2019;https://doi.org/10.1111/aab.12536.
- 4.
国际癌症研究机构。一些化工原料;国际癌症研究机构关于人类致癌风险评估的专著,第60卷。里昂:国际癌症研究机构;1994.
- 5.
欧盟委员会。委员会监管欧盟。2017/2158建立减缓措施和基准水平,减少食品中丙烯酰胺的存在。布鲁塞尔:欧盟;2017年。
- 6.
食品和药物管理局。工业指南,食品中的丙烯酰胺。College Park MD:食品和药物管理局;2016.
- 7.
Zhivagui M, Ng AWT, Ardin M, Churchwell MI, Pandey M, Renard C等。实验和泛癌症基因组分析显示,丙烯酰胺暴露对人类致癌有广泛贡献。基因组研究》2019;29(4):521 - 31所示。https://doi.org/10.1101/gr.242453.118.
- 8.
Raffan S, Oddy J, Halford NG。小麦中的硫反应及其对丙烯酰胺形成和食品安全的影响。中国生物医学工程学报,2010;https://doi.org/10.3390/ijms21113876.
- 9
Martinek P,Klem K,Vanova M,Bartackova V,Vecerkova L,Bucher P等人。氮营养,杀菌剂处理和小麦基因型对自由天冬酰胺和降低糖含量作为面包丙烯酰胺形成前体的影响。植物土壤环境。2009; 55(5):187-95。https://doi.org/10.17221/382-PSE.
- 10.
柯蒂斯TY,Powers SJ,Halford NG。杀菌剂处理对小麦游离氨基酸浓度和丙烯酰胺形成电位的影响。JAgric Food Chem。2016; 64(51):9689-96。https://doi.org/10.1021/acs.jafc.6b04520.
- 11.
Curtis TY,Powers SJ,Wang R,Halford NG。品种、栽培年限和硫供应对商品小麦籽粒游离天冬酰胺积累的影响。食品化学。2018;239:304–13.https://doi.org/10.1016/j.foodchem.2017.06.113.
- 12.
RAPP M,Schwadorf K,Sheer Wl,WürschumT,龙林CFH。评估小麦上天冬酰胺含量的变异和遗传建筑:植物育种有助于减少丙烯酰胺前体吗?Al Appl Genet。2018; 131(11):2427-37。https://doi.org/10.1007/s00122-018-3163-x.
- 13
emebiri lc。遗传变异和可用于繁殖小麦与低谷天冬酰胺的可能SNP标记,热加工产品中丙烯酰胺形成的主要前体。J SCI食物农业。2014; 94(7):1422-9。https://doi.org/10.1002/jsfa.6434.
- 14
Raffan S, Halford NG。谷门冬酰胺合成酶基因的系统发育分析。中国生物医学工程学报,2014;https://doi.org/10.1111/aab.12632.
- 15
徐浩,陈国强,杨国强,张国荣,黄杰,等。小麦天冬酰胺合成酶的基因组、生化和模拟分析。前植物科学。2018;8:2237.https://doi.org/10.3389/fpls.2017.02237.
- 16
Dubcovsky J,Dvorak J. Genome可塑性在驯化下多倍体麦成功的关键因素。科学。2007; 316(5833):1862-6。https://doi.org/10.1126/science.1143986.
- 17
国际小麦基因组测序联盟,中国科学院小麦研究所,北京,等。面包小麦祖先基因组的远古杂交。科学。2014;345:1250092-1——4。
- 18
高锐,黄杰,高锐。食品安全:小麦天门冬酰胺合成酶基因家族的结构与表达。中国粮食科学,2016;68:122-31。https://doi.org/10.1016/j.jcs.2016.01.010.
- 19
柯蒂斯蒂,raffans,wan y,国王r,gonzalez-uriarte a,halford ng。硫磺供应响应高低附加氨基小麦基因型籽粒基因表达模式。BMC基因组学。2019; 20(1):628。https://doi.org/10.1186/s12864-019-5991-8.
- 20。
raffan s,火花c,huttly a,海德l,martignago d,mead a等。小麦在谷物中大大减少了自由天冬酰胺的积累,由天冬酰胺合成酶基因的CRISPR / CAS9编辑产生TaASN2. 植物生物技术杂志。2021https://doi.org/10.1111/pbi.13573.
- 21。
国际小麦基因组测序联合会(IWGSC),Appels R,Eversole K,Feuillet C,Keller B,Rogers J等。利用全注释参考基因组改变小麦研究和育种的限制。科学。2018;361:eaar7191。
- 22。
柳条T,Keller B. Copia Retrotransposons在Triticeae,Rice和Copia Retroxposons的基因组比较分析拟南芥揭示了保守的古代进化谱系和独特的个体动力学Copia家庭。Genome Res。2007; 17(7):1072-81。https://doi.org/10.1101/gr.6214107.
- 23。
柳条T,Matthews de,Keller B. Trep:Triticeae重复元素的数据库。趋势植物SCI。2002; 7(12):561-2。https://doi.org/10.1016/S1360-1385(02)02372-5.
- 24。
陈建平,陈建平,等。面包小麦3B染色体的结构与功能分配。科学。2014;345:1249721-1——7。
- 25。
Ramírez-González RH, Borrill P, Lang D, Harrington SA, Brinton J, Venturini L, et al.;多倍体小麦的转录景观。科学。2018;361:eaar6089。
- 26。
汉密尔顿东北,费里M。ggtern:使用ggplot2的三元图。J统计软件。2018;8:3863.
- 27。
Zadoks JC,Chang TT,Konzak CF.谷物生长阶段的十进制代码。杂草研究,1974年;14(6):415–21.https://doi.org/10.1111/j.1365-3180.1974.tb01084.x.
- 28。
nirmal rc,furtado a,rigley c,henry rj。基因表达对小麦硬度的影响。Plos一个。2016; 11(10):E0164746。https://doi.org/10.1371/journal.pone.0164746.
- 29。
柳条(Wicker T, Gundlach H, Spannagl M, Uauy C, Borrill P, Ramírez-González RH,等。转座因子对面包小麦基因组结构和进化的影响。基因组医学杂志。2018;19:103。
- 30.
Simsek S,Ohm JB,Lu H,Rugg M,Berzonsky W,Alamri MS,等.小麦采前发芽对蛋白质理化变化的影响。食品农业科学杂志。2014;94(2):205–12.https://doi.org/10.1002/jsfa.6229.
- 31.
Noree C,Sirinonthanawech N,Wilhelm Je。Saccharomyces Cerevisiae Asn1和ASN2型是柿酰胺合成酶对帕拉洛,其伴随着响应营养应激而聚合的能力。SCI批准。2019; 9(1):278。https://doi.org/10.1038/s41598-018-36719-z.
- 32.
Noree C,Monfort E,Shotelersuk V。人天冬酰胺合成酶与有丝分裂纺锤体相关。生物门打开。2018;15:7(12).
- 33.
米德尔顿,Úbeda-Tomás S, Griffiths J, Holman T, Hedden P,等。数学模型阐明了转录反馈在赤霉素信号转导中的作用。国家自然科学基金资助项目:国家自然科学基金资助项目。https://doi.org/10.1073/pnas.1113666109.
- 34.
Lei GJ,Fujii Kashino M,Hisano H,Saisho D,Deng F,Yamaji N,et al.通过导入类苏库尔拉转座因子培育低镉大麦。纳特食物。2020;1(8):489–99.https://doi.org/10.1038/s43016-020-0130-x..
- 35.
欧洲食品安全管理局在食品链中的污染物(Contam Conse)。对食品中丙烯酰胺的科学意见。EFSA J. 2015; 13:4104。
- 36.
Krasileva KV、Vasquez Gross HA、Howell T、Bailey P、Paraiso F、Clissold L等。揭示多倍体小麦中隐藏的变异。美国国家科学院学报。2017;114(6):E913–21。https://doi.org/10.1073/pnas.1619268114.
- 37
张Y,Pribil M,Palmgren M,Gao C.一种加速粮食作物的改善方式。Nat Food。2020; 1(4):200-5。https://doi.org/10.1038/s43016-020-0051-8.
- 38
施密特SM,贝利斯勒M,弗洛默WB。农业基因组编辑的发展前景:许多国家已经免除或正在免除转基因作物的基因组编辑。EMO代表,2020年;19:e50680。
- 39
Bian X,Tyrrell S,Davey RP.草根生命科学数据基础设施。2017https://grassroots.tools..
- 40。
Blake VC、Woodhouse MR、Lazo GR、Odell SG、Wight CP、Tinker NA等。GrainGenes:遗传学家和育种家的集中小型谷物资源和数字平台。数据库。2019;baz065号。
- 41
Maccaferri M,Harris Ns,Twardziok So,Pasam Rk,Gundlach H,Spannagl M等人。杜兰姆小麦基因组突出了驯化签名和未来的改进目标。NAT Genet。2019; 51(5):885-95。https://doi.org/10.1038/s41588-019-0381-3.
- 42
索洛维耶夫、科萨雷夫P、塞列佐夫I、沃罗比耶夫D。真核基因、假基因和启动子的自动注释。基因组生物学。2006;7(S1):S10。https://doi.org/10.1186/gb-2006-7-s1-s10.
- 43
HMMER web服务器:2018更新。核酸图谱2018;46(W1): W200-4。https://doi.org/10.1093/nar/gky448.
- 44。
Sim-NL,Kumar P,Hu J,Henikoff S,Schneider G,Ng PC.SIFT-web服务器:预测氨基酸替代对蛋白质的影响。核酸研究,2012年;40(W1):W452–7。https://doi.org/10.1093/nar/gks539..
- 45。
Murashige T、Skoog F。烟草组织培养快速生长和生物测定的改良培养基。生理植物。1962;15(3):473–97.https://doi.org/10.1111/j.1399-3054.1962.tb08052.x..
- 46。
Sambrook J,Fritcech EF,Maniatis T.分子克隆:实验室手册:CSHL压力机;1989年。
- 47。
Chang S,Pureyear J,Cairney J.一种简单而有效的方法,用于从松树中分离RNA。植物分子Biol批准。1993; 11(2):113-6。https://doi.org/10.1007/BF02670468.
- 48。
Rieu I,Powers SJ。实时定量RT-PCR:设计、计算和统计。植物细胞。2009;21(4):1031–3.https://doi.org/10.1105/tpc.109.066001.
- 49。
拉姆加斯特C,Ruijter JM,Depez Rh,Moorman AF。无定量实时聚合酶链反应(PCR)数据的无假设分析。Neurosci Lett。2003; 339(1):62-6。https://doi.org/10.1016/s0304-3940(02)01423-4.
- 50
Windows 21st Edition的Genstat。VSN International,Hemel Hempstead,英国。2020。https://genstat.co.uk..
- 51
r核心团队。R:统计计算的语言和环境。维也纳,奥地利:统计计算基金会;2020。https://www.r-project.org/
- 52
Kassambara公司。ggpubr:基于“ggplot2”的出版物就绪绘图。R包版本0.4.0。2020https://CRAN.R-project.org/package=ggpubr
- 53
向德,奎利奇尼,刘志强,高平,潘勇,李Q,等。多倍体小麦及其二倍体祖先在胚胎发生和籽粒发育过程中的转录景观。植物细胞。2019;31(12):2888–911.https://doi.org/10.1105/TPC.19.00397.
- 54.
Pearce S,Vazquez Gross H,Herin SY,Hane D,Wang Y,Gu YQ,等.小麦XP:多倍体小麦RNA-seq表达数据库。BMC植物生物学。2015;15(1):299.https://doi.org/10.1186/s12870-015-0692-1..
- 55.
Warnes GR, Bolker B, Bonebakker L, Gentleman R, Huber W, Liaw A, Lumley T, Maechler M, Magnusson A, Moeller S, Schwartz M, Venables B. gplot:各种R编程工具绘制数据2013。R包版本2.12.1。2013.http://CRAN.R-project.org/package=gplots.
- 56.
Wickham H. GGPLOT2:用于数据分析的优雅图形。纽约:Springer-Verlag;2016.
致谢
不适用。
数据和材料的可用性
表达数据是从下载自https://www.ncbi.nlm.nih.gov/geo/并如结果部分中所述处理。用于生成表达图形的所有表达式值都在附加文件中呈现2.兰氏蛋白序列的氨基酸数据以前描述了中国春天的蛋白质序列[11].其他小麦品种的DNA和蛋白质序列来自“10+小麦基因组计划”(https://weblast.ipk-gatersleben.de/wheat_ten_genomes/),基层基因组学项目(https://wheatis.tgac.ac.uk/grassroots-portal/blast) [39],以及Graingenes数据库(https://wheat.pw.usda.gov/cgi-bin/seqserve/blast_wheat.cgi).
资金
Jo由BBSRC协作培训合作伙伴支持的合作伙伴:阅读和Mondelez英国R&D Ltd.SR的大学得到了生物技术和生物科学研究理事会(BBSRC)Swbio Icase DTP学生的支持,与合作伙伴:布里斯托大学,AHDB,KWS UK Ltd.,Saaten Union UK Ltd.,Ragt Seeds Ltd.,Syngenta UK Ltd.和Limagrain UK Ltd.NGH由BBSRC通过设计未来小麦计划(BB / P016855 / 1)支持Rothamsted Research。BBSRC是英国研究和创新的一部分。科罗拉多州立大学的研究部分由科罗拉多小麦研究基金会和科罗拉多小麦行政委员会资助。
作者信息
隶属关系
贡献
JO进行序列分析和注释,设计和运行分子分析,进行统计分析,并撰写手稿。RAR进行序列分析和注释,并设计和运行分子分析。MW设计并运行分子分析。KR进行了表达分析并撰写了手稿。SR进行序列分析和注释。AM1和AM2建议并指导实验设计和统计分析。JSE、IMdA和NCC为实验设计和数据分析提供建议;SP和NGH写了手稿。所有作者都阅读并批准了手稿。
作者的信息
不适用
相应的作者
道德宣言
伦理批准并同意参与
不适用。
同意出版
不适用。
相互竞争的利益
上述资助者在研究设计中没有任何作用;在数据的收集、分析或解释中;在写手稿时,或在决定出版时。
附加信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充资料
附加文件1
图S1。TaASN1等位基因多样性。Robigus、Julius、Norin 61、Mace和斯佩尔特小麦(野生型)编码ASN-B1全长蛋白的预测氨基酸序列,以及CDC Landmark、Claire、Jagger、Cadenza、Paragon、Arina、CDC Stanley和Lancer编码的截断蛋白(ASN-B1截断)。在后者中,预计第7外显子中16bp的缺失会在375氨基酸残基处引入一个提前终止密码子,用*表示。强调了保守的GATase和ASN合成酶结构域。图S2。用PCR方法来区分有无TaASN-B2型在24个全球小麦品种的集合中。一种。测定的示意图,以显示引物位置和预期的扩增子尺寸。具有引物P1和P2的189bp产品的扩增表明TaASN B2缺失,用引物P3和P4扩增125bp产物表明TaASN-B2型存在。每次反应中预期一个扩增的片段。湾来自测定的PCR产物的琼脂糖凝胶电泳。品种TaASN-B2型删除以红色突出显示,而存在的类型的品种以绿色突出显示。在凝胶的第一和最后孔中示出了100bp梯子,用于尺寸比较。品种中有五个携带TaASN-B2型删除和四个TaASN-B2型用于测定ASN在籽粒中的表达。每个品种的详细情况见表2。表S1。小麦ASN蛋白的自然变异。对于每一种蛋白质,绿色的阴影表示所有的氨基酸替换被预测是可以容忍的,编码的蛋白质被预测是功能性的。黄色/橙色的阴影表明,基于SIFT分析,至少有一种多态性被预测会破坏蛋白质功能。红色表示该品种的基因已被删除。每种蛋白质类型的全部细节在主表下方的键中提供,其中(T)表示该位置处的氨基酸取代预计可耐受,(APF)表示预测的变化将影响蛋白质功能。表S2。A.英国冬小麦名录(小麦)品种TaASN-B2型现在或缺席,由市场班级分开。湾普通小麦品种名单TaASN-B2型在24个全球小麦品种中存在或删除。提供ID、加入号和原产国。表S3。RT-qPCR和现场分析的意义值。a.采用Timepoint*Variety* homologue作为处理结构,Block/Subblock/Plot作为阻塞结构进行方差分析。b.田间试验数据方差分析和REML分析中各因素的显著性值。所有分析都是在对数转换数据上进行的。采用Block/MainPlot/SplitPlot作为随机模型,采用(TaASN-B2/Variety)*硫处理作为处理模型进行方差分析。REML分析采用Year/Block/MainPlot/SplitPlot为随机模型,Year*(TaASN-B2/Variety)*硫处理为处理模型。表S4。 Details of missing sequence data in ASN genes in some genome assemblies. Similarity among sequences was determined using all available sequence but some varieties had regions of ‘Ns’ within ASN genes, as indicated in the table below. Table S5. Primers used in this study.
附加文件2
平均每个TPM表达数据ASN公司RNA-seq数据集中的基因。
额外的文件3。
完整,在本研究中提出的凝胶电泳结果的不可折叠图像。
权利和权限
开放获取本文是根据知识共享署名4.0国际许可证授权的,该许可证允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供到知识共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可证中,除非在材料的信用额度中另有说明。如果材料未包含在文章的知识共享许可证中,并且您的预期用途不受法律法规的允许或超出允许的用途,您将需要直接获得版权持有人的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Oddy,J.,Alarcón-Reverte,R.,Wilkinson,M。等。从自然缺失中减少了小麦籽粒中的自由芦笋TaASN-B2型研究和开发天冬酰胺合成酶基因家族的多样性以提高小麦品质。BMC植物生物学21,302(2021)。https://doi.org/10.1186/s12870-021-03058-7.
收到了:
公认:
发表:
关键词
- 小麦
- 小麦
- 天冬酰胺代谢
- 丙烯酰胺
- 天冬酰胺合成酶
- 食品安全
- 小麦育种